Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
La microscopie vidéo sans lentille pour la dynamique et l’analyse Quantitative de la Culture de cellules adhérentes
Dans cet article
Résumé
La microscopie vidéo sans lentille permet de surveiller les cultures de cellules directement à l’intérieur de l’incubateur. Ici, les auteurs décrivent le protocole complet utilisé pour acquérir et analyser une acquisition 2,7 journée des cellules HeLa cultivées, conduisant à un ensemble de données 2.2 x 106 mesures de la morphologie des cellules individuelles et 10584 cycle cellulaire titres.
Résumé
Ici, nous démontrons que la lentille-gratuit vidéo microscopie permet de capturer simultanément la cinétique de milliers de cellules directement à l’intérieur de l’incubateur et qu’il est possible de surveiller et de quantifier les cellules isolées le long de plusieurs cycles cellulaires. Les auteurs décrivent le protocole complet permettant de surveiller et de quantifier une culture de cellules HeLa pour 2,7 jours. Tout d’abord, acquisition de la culture cellulaire est réalisée avec un microscope vidéo sans lentille, et puis les données sont analysées à la suite d’un processus en quatre étapes : reconstruction holographique de longueurs d’onde multiples, cellule-tracking, segmentation et la division cellulaire détection des cellules algorithmes. Ainsi, nous montrons qu’il est possible de rassembler un ensemble de données comportant plus de 10 000 titres du cycle cellulaire et de mesures morphologiques de plus de 2 x 106 cellules.
Introduction
Suivi tout au long de plusieurs cycles cellulaires des cellules de mammifères et de mesurer avec exactitude cell taille et cell masse sèche est une tâche difficile. Plusieurs techniques d’optiques sans étiquette sont capables d’accomplir cette tâche1,2: déphaseurs interférométrie3, microscopie holographique numérique (DHM)4,5,6, 7, quadriwave8,de l’interférométrie cisaillement latéral9 et volet quantitatif tomographie10,11. Ces méthodes ont abouti à nombreuses nouvelles perspectives sur la compréhension du cycle cellulaire des cellules de mammifères. Cependant, ils sont rarement associés à cellule automatique suivi des algorithmes et leur débit reste limitée lors de la mesure cellule trajectoires masse1 (N < 20 respectivement3,,4,5 , 6). c’est pourquoi une nouvelle méthode optique est nécessaire pour mesurer les trajectoires masse cellulaire avec statistiques grand (N > 1000).
Dans cet article, nous démontrent la capacité de la microscopie vidéo sans lentille image simultanément des milliers de cellules directement à l’intérieur de l’incubateur, et ensuite quantifier métriques de cellule unique le long des milliers de pistes cyclables de cellules individuelles. La microscopie exempte d’objectif est une phase quantitative imaging technique qui permet l’acquisition d’images de cellules sur un très grand champ de vision (typiquement plusieurs dizaines de mm2, ici 29,4 mm2)12,13 ,14,15. Plusieurs mesures au niveau de la cellule sont déterminées, par exemple, zone de cellules, cellules masse sèche, épaisseur de la cellule, la longueur axe majeur des cellules et rapport l / h12,15, de chaque image de cellule. Puis, en appliquant un algorithme de suivi des cellules, ces fonctionnalités peuvent être tracées pour chaque cellule en fonction de l’expérience de temps14,15. En outre, en détectant la présence de divisions cellulaires dans les traces de la cellule, il est possible d’extraire d’autres informations importantes telles que la cellule initiale poids sec (juste après la division cellulaire), la masse sèche de cellule final (juste avant la division cellulaire) et la cellule cycle de durée, c'est-à-dire, le temps entre deux divisions consécutives15. Toutes ces mesures peuvent être calculés avec de très bonnes statistiques (N > 1000) le grand champ de vision permettant généralement l’analyse de 200 à 10 000 cellules en une seule acquisition exempt de lentille.
Pour expliquer cette méthodologie basée sur la microscopie vidéo sans lentille, les auteurs décrivent le protocole pour surveiller et mesurer une culture de cellules HeLa pour 2,7 jours. Analyse des données est un processus en quatre étapes basé sur multi-longueur d’onde reconstruction holographique, suivi des cellules, segmentation de la cellule et des algorithmes de division cellulaire. Ici, on montre que la résolution spatiale et la fréquence d’images relativement rapide (une acquisition toutes les 10 minutes) obtenus avec cette configuration de microscopie vidéo sans lentille est compatible avec des algorithmes de suivi de cellule standard. L’analyse complète de ce jeu de données entraîne la mesure de 10 584 cellule titres plus complète des cycles cellulaires.
Pour résumer, la microscopie vidéo sans objectif est un outil puissant pour surveiller automatiquement des milliers de sans étiquette, non synchronisé et des cellules non modifiés par l’expérience ; chaque cellule étant suivie pendant plusieurs cycles cellulaires. Nos mesures fournissent ainsi la valeur moyenne de plusieurs paramètres des cellules, mais plus important encore, la variabilité inter cellulaire sur une large population de cellules.
Protocole
1. Culture cellulaire Acquisition de surveillance
- La croissance de cellules HeLa DMEM + glutamine (p. ex., GlutaMAX) additionné de 10 % (v/v) veau foetal inactivés par la chaleur de sérum et de 1 % la pénicilline et de streptomycine.
- Enduire les plaques de culture à fond de verre 6 puits avec la fibronectine (25 µg/mL) pendant 1 h. Les semences puis 2 x 104 cellules par puits.
- Lors de l’acquisition, changer le support tous les 3 jours.
- Pour l’acquisition de Time-lapse, utiliser le microscope objectif-free video (disponible dans le commerce).
NOTE : Ceci est basé sur la technique d’imagerie exempte d’objectif calcul, tel que décrit par Ozcan et al. 16 qui a été modifiée pour effectuer une surveillance continue dans un incubateur à une température de 37 ° C12,15. Il dispose d’un capteur d’image CMOS (semiconductor) oxyde de métal complémentaire avec un terrain de pixel de 1,67 µm et une zone d’imagerie de 6,4 x 4,6 mm2. Illumination de longueurs d’onde multiples est assurée par un multichip diode (LED) dispositif électroluminescent, qui offre un éclairage rouge, vert et bleu. Les longueurs d’onde sont centrés, 636, 521 et 452 nm respectivement, avec une largeur de bande spectrale de 25, 45 et 25 nm respectivement. Les rouge-vert-bleu (RVB) LED sont situés au-dessus d’un trou de 150 µm à une distance d’environ 5 cm des cellules. La puissance lumineuse mesurée à proximité de la LED est aussi basses que 10 µW à chaque longueur d’onde et le temps d’illumination est seulement une seconde par acquisition. Par conséquent, aucun problème de toxicité-photo n’est attendus lorsque des cultures de cellules sont observés au microscope vidéo sans lentille. - Mettre le récipient de culture cellulaire mis en contact avec le capteur CMOS (Figure 1). Utiliser un récipient avec une lamelle de verre au fond, ce qui est important pour la qualité de l’acquisition de lentille-free. Contenants en plastique peuvent également être utilisés, mais l’acquisition de lentille-libre sera dégradée en raison de la mauvaise qualité optique de ces conteneurs.
- Contrôler l’exempte d’objectif microscope video avec le logiciel d’acquisition (disponible dans le commerce), qui effectue une fois l’acquisition de Time-lapse et la reconstruction holographique. Les paramètres qui doivent être saisies par l’utilisateur dans l’interface du logiciel sont la cadence, la durée de l’expérience et le type de culture de cellules, cellules c.-à-d. adhérentes ou cellules flottantes.
- Définissez les paramètres d’entrée du logiciel acquisition. Définissez la cadence définie sur une acquisition toutes les 10 minutes et le type de culture cellulaire à « adherent ».
Remarque : Une cadence d’un acquisition toutes les 10 minutes est un bon compromis car il garantit une cellule fiable de suivi alors que la taille de l’ensemble de données qui en résulte à analyser est encore raisonnable. La fréquence d’images plus rapide réalisable avec cette configuration de microscopie exempte d’objectif est une acquisition toutes les 5 minutes. Ce qui augmente la framerate se traduit par un réchauffement excessif du capteur CMOS qui peut affecter la viabilité des cellules en culture. - Démarrer l’acquisition de Time-lapse, et l’algorithme de reconstruction holographique (disponible dans le commerce) traitera automatiquement l’acquisition exempt de lentille pour obtenir l’image de phase de la culture cellulaire. L’algorithme de reconstruction holographique est issu d’une phase de longueurs d’onde multiples récupération algorithme17 et fournit une image RVB de phase reconstruit dans la plage de [-π, + π] pour chaque cellule unique sur un grand champ de vision de 29,4 mm2. Le principe de l’holographie en ligne exempt de lentille est pour éclairer un échantillon mince (z = 0) avec une onde plane (d’amplitude 1 après normalisation) et de détecter sur un capteur CMOS de l’image d’intensité de diffraction ultérieure à une courte distance z = Z, généralement 1 mm après la échantillon. Dans notre configuration, l’éclairage est séquentiellement en 3 longueurs d’onde (λ1= 0,450 µm, λ2= 0.540 µm, λ3= 0,647 µm) pour mesurer les trois patrons de diffraction du même échantillon.
2. analyse de données de Culture cellulaire
- Pour cellule-tracking, utiliser l’algorithme de Trackmate, un plugin OpenSource de Fidji le suivi automatisé des particules unique18. At a tout d’abord, chargez l’acquisition complète de Time-lapse dans Fidji (commande de séquence de chargement image). En raison de la grande taille des ensembles de données, qui comporte généralement 400 images RVB de 29,7 Mo, il est plus rapide de les charger au format 8 bits. Ensuite le plugin Trackmate Fidji guide l’utilisateur à travers plusieurs étapes de l’algorithme de la cellule de suivi, à savoir une étape de détection de cellules, un stade de traqueur de cellules et plusieurs filtres appliqués à la détection de la cellule et les pistes calculées. À chaque étape, configurez les algorithmes et afficher les résultats immédiatement afin que, si nécessaire, on peut facilement naviguer en arrière pour réajuster les paramètres. Les différents menus de l’interface de Trackmate sont indiqués dans la Figure 2 avec les réglages utilisés pour l’expérience de cellules HeLa.
- Définir les paramètres d’entrée du logiciel acquisition comme suit (voir Figure 2). Définir le diamètre estimé blob à 15 pixels, le seuil de détecteur à 0.25, la distance maximale de liaison de 15 pixels, la distance max écart-fermeture 15 pixels et le nombre de places dans les titres à 3,5.
Remarque : Ces paramètres différeront évidemment d’une expérience à l’autre, en particulier le « blob estimée diamètre » qui correspond à la taille des cellules (ici donnée en pixels du µm 1.67). La même remarque s’applique à la « distance max relie » qui représentent la distance maximale parcourue par une cellule au sein de deux images consécutives (ici donnée en pixels du µm 1.67). Sa valeur optimale dépendra sur la motilité cellulaire, mais aussi sur la densité des cellules. - À la fin du processus de suivi des cellules, générer les résultats sous la forme de trois fichiers texte (« Analyse » bouton, voir Figure 2). Le fichier plus utile, nommé « Points dans les statistiques de titres », consistant en un tableau énumérant les cellules détectées tout avec leurs positions respectives (x, y) dans l’acquisition, leur numéro d’image et leur numéro de piste. Sur la base de ces données, effectuer la segmentation cellulaire et détecter des divisions cellulaires à l’aide des algorithmes dédiés (voir les fichiers de code supplémentaire). Les deux autres fichiers sont nommés « Liens statistiques de titres » et « Suivi statistique » et inclure tous les résultats portant sur les titres, par exemple les durées de piste, certaines lacunes détectées, suivre la trame initiale, etc..
- Utiliser l’algorithme de segmentation cellulaire (voir le dossier de code supplémentaires) pour extraire automatiquement plusieurs paramètres décrivant la morphologie des cellules de l’image de la phase de reconstruction. Ces mesures sont la surface de la cellule (S), la longueur d’axe majeur de cellule (L), la longueur d’axe mineur de cellule (l) et les proportions de la cellule (L/l). La phase Récupérée de microscopie exempt de lentille est proportionnelle à la densité et l’épaisseur de la couche de spécimen tel que discuté auparavant15.
- En plus des caractéristiques morphologiques de la cellule, extrait secs mesures de (CDM) de masse cellulaire quantitative la phase reconstituées13,19,20
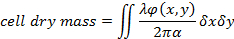 Équation (1)
Équation (1)
où φ(x,y) est la phase reconstituées MAJ19, λ la longueur d’onde et α = 1,8 x 10-4 m3/kg l’indice de réfraction spécifique qui concerne la variation de l’indice de réfraction pour sécher la masse1, 19. - Évaluer l’épaisseur de la cellule en utilisant une estimation de la différence entre l’indice de réfraction de cellule et les milieux de culture. La relation entre la cellule épaisseur h(x,y) et le déphasage mesurée est donnée par20:
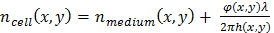 EQ. (2.1)
EQ. (2.1) EQ. (2.2)
EQ. (2.2)
avec Δn(x,y) = ncellule (x,y) - nmoyen (x,y), la différence d’indice de réfraction entre la cellule et les milieux de culture. Dans ce qui suit, supposons une valeur constante de Δn = 0,025, qui est estimée à partir de l’indice de réfraction mesuré dans les noyaux des cellules HeLa (n = 1.35511) et celle des milieux de culture de solution saline tamponnée au phosphate (PBS) (n = 1,33). Bien que la valeur d’épaisseur cellulaire est seulement indicative car elle repose sur une hypothèse, ses variations relatives sont significatives et peuvent être exploitées pour détecter les cellules en division. - Utiliser l’algorithme dédié (voir le dossier de code supplémentaires) pour détecter la présence de divisions cellulaires et puis extraire les pistes de cellule qui sont associés aux détection des divisions cellulaires, afin de détecter une division cellulaire, des cellules avec une épaisseur mesurée supérieure à 8 µm sont identifiés tout d’abord, puis vérifiez si une nouvelle cellule apparaît étroitement dans l’espace et temps. De cette façon, la détection des divisions cellulaires s’appuie sur deux critères robustes. Alternative et des méthodes plus élaborées pour la détection de la division cellulaire peuvent être appliqués au lensfree acquisition Time-lapse21,22,23.
Résultats
Pour le processus de reconstruction holographique, le terrain de la lumière est décrite par un champ scalaire A (où 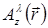 est la valeur complexe de A dans l’avion à distance z de la position de l’échantillon et latérale
est la valeur complexe de A dans l’avion à distance z de la position de l’échantillon et latérale 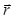 et à la longueur d’onde λ). Rayon est modelé par la théorie de Huygens-Fresnel qui fournit un noyau ...
et à la longueur d’onde λ). Rayon est modelé par la théorie de Huygens-Fresnel qui fournit un noyau ...
Discussion
Dans cet article, nous montrons que microscopie vidéo sans lentille peut être utilisée à l’intérieur d’un incubateur pour capturer la cinétique de milliers de cellules. Afin de décrire la méthodologie globale, nous a expliqué comment une acquisition Time-lapse 2,7 jours de culture, les cellules HeLa peut être analysée avec des algorithmes de suivi de cellule standard. Le résultat est un ensemble de données mettant en vedette 2.2 x 10 mesures de cellule de6 et 10 584 pistes du cycle cellulaire....
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs n’ont rien à reconnaître.
matériels
| Name | Company | Catalog Number | Comments |
| Cytonote lens-free video microscope | Iprasense | ||
| Horus acquisition software | Iprasense | ||
| 6-well glass bottom culture plates | MatTek corporation | Part No: P06G-0-14-F | |
| DMEM + GlutaMAX medium | Gibco | ||
| heat-inactivated fetal calf serum | Eurobio | ||
| penicillin and streptomycin | Gibco | ||
| Fibronectin | Sigma Aldrich | ||
| Matlab, image processing toolbox | Mathworks |
Références
- Zangle, T. A., Teitell, M. A. Live-cell mass profiling: an emerging approach in quantitative biophysics. Nat Methods. 11 (12), 1221-1228 (2014).
- Popescu, G., Park, K., Mir, M., Bashir, R. New technologies for measuring single cell mass. Lab Chip. 14 (4), 646-652 (2014).
- Reed, J., et al. Rapid, massively parallel single-cell drug response measurements via live cell interferometry. Biophys J. 101 (5), 1025-1031 (2011).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci. USA. 108 (32), 13124-13129 (2011).
- Girshovitz, P., Shaked, N. T. Generalized cell morphological parameters based on interferometric phase microscopy and their application to cell life cycle characterization. Biomed Opt Express. 3 (8), 1757-1773 (2012).
- Kemper, B., Bauwens, A., Vollmer, A., Ketelhut, S., Langehanenberg, P. Label-free quantitative cell division monitoring of endothelial cells by digital holographic microscopy. J Biomed Opt. 15 (3), (2010).
- Mir, M., Bergamaschi, A., Katzenellenbogen, B. S., Popescu, G. Highly sensitive quantitative imaging for monitoring single cancer cell growth kinetics and drug response. PLoS One. 9 (2), 1-8 (2014).
- Bon, P., Savatier, J., Merlin, M., Wattellier, B., Monneret, S. Optical detection and measurement of living cell morphometric features with single-shot quantitative phase microscopy. J Biomed Opt. 17 (7), (2012).
- Aknoun, S., et al. Living cell dry mass measurement usinq quantitative phase imaging with quadriwave lateral shearing interferometry: an accuracy and sensitivity discussion. J Biomed Opt. 20 (1), 1-4 (2015).
- Cotte, Y., et al. Marker-free phase nanoscopy. Nature Photonics. 7 (2), 113-117 (2013).
- Choi, W., et al. Tomographic phase microscopy. Nat Methods. 4 (9), 717-719 (2007).
- Kesavan, S. V., et al. High-throughput monitoring of major cell functions by means of lensfree video microscopy. Sci Rep. 4, 1-11 (2014).
- Zheng, G., Lee, S. A., Antebi, Y., Elowitz, M. B., Yang, C. The ePetri dish, an on-chip cell imaging platform based on subpixel perspective sweeping microscopy (SPSM). Proc Natl Acad Sci USA. 108 (41), 16889-16894 (2011).
- Pushkarsky, I., et al. Automated single-cell motility analysis on a chip using lensfree microscopy. Sci Rep. 4, 4717 (2014).
- Allier, C., et al. Imaging of dense cell cultures by multiwavelength lens-free video microscopy. Cytom Part A. 91 (5), 1-10 (2017).
- Su, T. -. W., Seo, S., Erlinger, A., Ozcan, A. High-throughput lensfree imaging and characterization of a heterogeneous cell solution on a chip. Biotechnol Bioeng. 102 (3), 856-868 (2009).
- Delacroix, R., et al. Cerebrospinal fluid lens-free microscopy: a new tool for the laboratory diagnosis of meningitis. Sci Rep. 7, 39893 (2017).
- Tinevez, J. -. Y., et al. TrackMate: an open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2016).
- Popescu, G. Optical imaging of cell mass and growth dynamics. Am J Physiol Physiol. 295 (2), 538-544 (2008).
- Liu, P. Y., et al. Cell refractive index for cell biology and disease diagnosis: past, present and future. Lab Chip. 16, 634-644 (2016).
- Rapoport, D. H., Becker, T., Mamlouk, A. M., Schicktanz, S., Kruse, C. A novel validation algorithm allows for automated cell tracking and the extraction of biologically meaningful parameters. PLoS One. 6 (11), e27315 (2011).
- Al-Kofahi, O., et al. Automated cell lineage construction: A rapid method to analyze clonal development established with murine neural progenitor cells. Cell Cycle. 5 (3), 327-335 (2006).
- Meijering, E., Dzyubachyk, O., Smal, I., van Cappellen, W. A. Tracking in cell and developmental biology. Semin Cell Dev Biol. 20 (8), 894-902 (2009).
- Posakony, J. W., England, J. M., Attardi, G. Mitochondrial growth and division during the cell cycle in HeLa cells. J Cell Biol. 74 (2), 468-491 (1977).
- Zocchi, E., et al. Expression of CD38 Increases Intracellular Calcium Concentration and Reduces Doubling Time in HeLa and 3T3 Cells. J Biol Chem. 273 (14), 8017-8024 (1979).
- Reitzer, L. J., Wice, B. M., Kennell, D. Evidence that glutamine, not sugar, is the major energy source for cultured HeLa cells. J Biol Chem. 254 (8), 2669-2676 (1979).
- Benedetti, A. D. E., Joshi-barve, S., Rinker-Schaeffer, C., Rhoads, R. E. Expression of Antisense RNA against Initiation Factor eIF-4E mRNA in HeLa Cells Results in Lengthened Cell Division Times, Diminished Translation Rates, and Reduced Levels of Both eIF-4E and the p220 Component of eIF-4F. Mol Cell Biol. 11 (11), 5435-5445 (1991).
- Kumei, Y., Nakajima, T., Sato, A., Kamata, N., Enomoto, S. Reduction of G1 phase duration and enhancement of c-myc gene expression in HeLa cells at hypergravity. J Cell Sci. 93 (2), 221-226 (1989).
- Ginzberg, M. B., Kafri, R., Kirschner, M. On being the right (cell) size. Science. 348 (6236), 1245075 (2015).
- Mathieu, E., et al. Time-lapse lens-free imaging of cell migration in diverse physical microenvironments. Lab Chip. 16 (17), 3304-3316 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon