Method Article
Секвенирование нового поколения и биоинформатики трубопровода для оценки генетических детерминант конституционных болезни
В этой статье
Резюме
Целевые секвенирование нового поколения является время - и экономически эффективного подхода, который становится все более популярным в болезни исследований и клинической диагностики. Протокол, описанные здесь представляет сложных рабочих процессов, необходимых для секвенирования и биоинформатики процесс, используемый для выявления генетических вариантов, влияющих на болезнь.
Аннотация
Секвенирование нового поколения (НГС) быстро революцию как проводится исследование генетических детерминант конституционных болезни. Техника очень эффективна с миллионами считывает последовательность, производится в короткие сроки и при относительно низких затратах. В частности целевые NGS способен фокус исследования геномной регионы особый интерес, на основе этой болезни исследования. Не только делает это далее снизить затраты и увеличить скорость этого процесса, но это снижает вычислительной бремя, которое часто сопровождает NGS. Хотя целевые NGS ограничено в некоторых регионах генома, предотвращения идентификации потенциальных Роман локусов интерес, он может быть отличную технику, когда сталкиваются с фенотипически и генетически гетерогенных болезнью, для которой существует ранее известных генетических ассоциаций. Ввиду сложного характера метода виртуализации важно строго придерживаться протоколы и методологий, с тем чтобы добиться последовательности читает высокого охвата и качества. Кроме того получив считывает последовательность, сложные биоинформатики рабочего процесса используется точно сопоставить считывает ссылку генома, позвонить варианты и чтобы убедиться, что варианты пройти показателей качества. Варианты также должны быть аннотацией и куратор на основании их клиническое значение, которое может быть стандартизирована, применяя Американский колледж медицинская генетика и геномика патогенности руководящие принципы. Методы, представленные в настоящем документе будет отображаться этапы создания и анализа данных NGS из целевой последовательности панели, с помощью панели ONDRISeq нейродегенеративные заболевания как модель, определить варианты, которые может иметь клиническое значение.
Введение
Как определение генетических детерминант различных условий берет на более высокий приоритет в научные исследования и в клинике, секвенирование нового поколения (НГС) оказалась высокой пропускной способностью и экономически эффективным инструментом для достижения этих целей1,2 ,3. За почти 40 лет, Сэнгер последовательности был золотым стандартом для выявления генетических вариантов4; Однако для заболеваний с генетической гетерогенностью или неизвестных генетической этиологии, многие гены возможного кандидата должны быть оценены, часто одновременно. В этом контексте, Сэнгер последовательности становится дорогим и трудоемким. Однако NGS включает массивной параллельной последовательности миллионы фрагментов ДНК, позволяя для времени и экономически эффективный способ одновременно обнаруживать широкий спектр генетической вариации в различных регионах генома.
Существует три типа NGS для секвенирования ДНК: 1) всего генома (РГ), последовательность 2) целом exome (WES) и 3) целевые последовательности5. WGS оценивает всю геномной содержание лица, в то время как Уэс включает последовательность только белок кодирование областей генома6. Целевые последовательности, напротив, сосредоточен на конкретных регионах генома, основанный на относительно небольшое число конкретных генов соединены общие патологические механизмы или известный клинический фенотип. С помощью этого подхода можно указаны экзонов или интронов или любой intergenic регионах ген или конкретной группы генов. Таким образом целевые последовательности может быть отличный подход, когда уже есть фонд кандидата генов, известно, что связано с болезнью интерес. Ориентации конкретных областей генома позволяет для ликвидации ненужным и неуместным генетические различия, которые могут облако или отвлекать от клинической интерпретации. В то время как РГ и Уэс производят большое количество качественных данных, объем данных может быть подавляющим. Не только это большое количество данных требует интенсивных вычислений биоинформатики анализа, но хранения данных часто может представлять проблемы7. Эта проблема хранения данных также добавляет дополнительные расходы РГ и Уэс, который часто не считается первоначально при расчете за счет виртуализации. Кроме того хотя он сокращается, стоимость РГ и Уэс остаются относительно высокими. Целевые последовательности может быть более экономически эффективным вариантом, особенно когда требуется секвенирования большого числа лиц.
Нейродегенеративных Онтарио болезни исследования инициатива (ONDRI) это мультиплатформенный, Провинциальный общесистемной, наблюдений когортное исследование, характеризующие пять нейродегенеративных заболеваний, в том числе: 1) болезни Альцгеймера и умеренными когнитивными нарушениями, 2). боковой амиотрофический склероз, 3) frontotemporal деменции, 4) болезнь Паркинсона и 5)8сосудистых когнитивных нарушений. Подгруппа геномики ONDRI стремится пролить свет как часть базовых характеристик этой когорты часто со скидкой, но чрезвычайно важные генетические ландшафт этих фенотипически и генетически гетерогенных заболеваний. Таким образом, нейродегенеративные заболевания являются подходящими кандидатами для NGS методологий и целевой виртуализации в частности.
Мы специально целевой группы NGS, ONDRISeq, чтобы виртуализировать 528 участников участвующих в ONDRI для регионов кодирвоания протеина 80 генов, которые были ранее связаны с пяти заболеваний интерес. С этой методологией мы имеем возможность использовать данные высокого качества NGS целенаправленным и эффективным образом. Дизайн и проверки панели ONDRISeq с несколько соответствий исследований была ранее описана, для которых ONDRISeq группа смогла выявить роман, редкие варианты возможных клиническое значение 72,2% 216 случаев, используемых для проверки группа 9. Хотя NGS технология продвинулась быстро и удивительно в последние годы многие исследователи сталкиваются с проблемой при обработке исходных данных в список вариантов использования, аннотированный10. Кроме того интерпретация вариантов может быть сложной, особенно когда сталкиваются с многие, которые являются редкими или Роман11.
Здесь мы опишем шаг за шагом, методология целевых NGS и связанные биоинформатики рабочего процесса, необходимых для ампликонов, вариант вызова и вариант, заметки, используя ONDRISeq исследование в качестве примера. После генерации данных NGS сырой последовательности файлов должны быть выровнены в геноме человека ссылку для того, чтобы точно вызвать варианты. Затем варианты должны отмечаться для выполнения последующих варианта курирование. Также мы будем объяснять нашей реализации американского колледжа стандартов и руководящих принципов медицинской генетики точно классифицировать вариант патогенности.
протокол
Для целей ONDRI этики протоколов и осознанного согласия были получены на основе исследовательской этики доски на Baycrest центр для престарелых уход (Торонто, Онтарио, Канада); Центр по вопросам наркомании и психического здоровья (Торонто, Онтарио, Канада); Элизабет Bruyère больница (Оттава, Онтарио, Канада); Больница Хэмилтон (Гамильтон, Онтарио, Канада); Центр наук здравоохранения Лондона (Лондон, Онтарио, Канада); МакМастер (Гамильтон, Онтарио, Канада); Оттава больницы (Оттава, Онтарио, Канада); Parkwood больница (Лондон, Онтарио, Канада); Больница Святого Михаила (Торонто, Онтарио, Канада); Саннибрук медицинских наук центр (Торонто, Онтарио, Канада); и Университет больница Западной сети Торонто (Торонто, Онтарио, Канада).
1. ДНК изоляции от образцов человеческой крови
-
Сбор образцов от участников последовательности в соответствии с соответствующим этики протоколов и осознанного согласия.
- Для получения ДНК высокого качества, нарисуйте образцов крови для целей добычи.
Примечание: ДНК можно также извлечь из слюны или буккально клеток, обеспечивая, что используется соответствующий комплект экстракции ДНК. - Если извлечение из крови, чтобы получить высокий урожай ДНК, собирать образца в трех 4 мл К2 ЭДТА трубы, обеспечивая образец общего объема ~ 12 мл.
- Центрифугуйте образцы крови для 20 минут, 750 x g фракции в верхней фазу плазмы, тонкий, средний этап лейкоцитов, а дно эритроцитов.
- Для получения ДНК высокого качества, нарисуйте образцов крови для целей добычи.
- Удалите плазмы из крови, дозирование с верхней части образца с одноразовой передачи пипетки. Обойтись в несколько 500 мкл аликвоты для хранения при температуре-80 ° C для будущих биохимические анализы или надлежащим образом отказаться от плазмы. Убедитесь, что для каждого образца используется новый, стерильной пипеткой.
- Извлечь ДНК из крови с кровью добыча комплект12 (Таблица материалов) согласно инструкциям производителя.
Примечание: Если получается образец тома, описанных выше, ~ 3 мл лейкоцитов будет получен использовать экстракции ДНК. - Измерения начальной концентрации ДНК в нг/мкл, используя полный спектр спектрофотометр13 (Таблица материалов), в соответствии с инструкциями производителя.
- Перейти непосредственно к шагу 2. Кроме того хранить ДНК при 4 ° C.
2. последовательность библиотеки подготовка
-
Выполните серийных разведений на образцы ДНК в течение трех дней, чтобы получить окончательный концентрации 5,0 ± 1,0 нг/мкл.
- Разбавьте 1 М трис-буфер рН 8,5 до 10 мкм деионизированной водой.
Примечание: Объем разреженных будет зависеть количество образцов ДНК, которые будет нужно быть разведен в последующих шагах. - Если выполнение ДНК разрежения непосредственно после шага 1.4, переходите к следующему шагу. Если не в тот же день, измерения концентрации ДНК, как это было сделано в шаге 1.4.
- На основе измеренных концентрация разбавлять 40 мкл ДНК ~ 10 нг/мкл с помощью 10 мкм трис буфер рН 8,5 и позволяют образца сидеть всю ночь при 4 ° C.
- Измерение концентрации ДНК с флуориметр14 , подходящие для количественного определения ДНК (Таблица материалов), согласно инструкциям производителя.
Примечание: Концентрация образца должна быть > 10 нг/мкл из-за более низкую чувствительность спектрофотометр, используемых ранее. - Основываясь на концентрации, Разбавьте 20 мкл ДНК до 10 нг/мкл, используя 10 мкм трис буфер рН 8,5 и позволяют образца сидеть всю ночь при 4 ° C.
- Измерение концентрации ДНК с флуориметр14, согласно инструкциям производителя.
- На основе концентрации разбавления 10 мкл ДНК 5 нг/мкл, используя 10 мкм трис-HCl рН 8,5 и позволяют образца сидеть всю ночь при 4 ° C.
- Разбавьте 1 М трис-буфер рН 8,5 до 10 мкм деионизированной водой.
-
Подготовьте библиотеку последовательности согласно инструкции производителя с целевой группы NGS соответствующих целевых обогащения комплект15 (таблица материалов). Убедитесь, что обогащение комплект подходит для используемой платформы NGS.
- Следуйте инструкции производителя16 , относительно plexity и объединение библиотек.
Примечание: Для ONDRISeq, библиотеки состоят из 12 образцов ДНК, объединили в наборах двух и запустить на NGS рабочего инструмента (Таблица материалов). Количество выборок, которые могут быть запущены в одном реакция будет зависеть от последовательности kit и используемой платформы. - Для достижения более качественных последовательности данных, выполните дополнительный шаг для проверки качества библиотеки ДНК, после tagmentation, описанные в инструкции производителя целевой обогащения комплект15.
- Анализ каждой библиотеки в трех экземплярах для обеспечения качества библиотека доходности.
- Если объединение библиотек, измерения концентрации ДНК с флуориметр14, в соответствии с инструкциями производителя. Используйте этот концентрации для определения объема каждой библиотеки ДНК в пул, чтобы получить эквимолярных соотношениях, рекомендованный целевой обогащения комплект используется.
- Следуйте инструкции производителя16 , относительно plexity и объединение библиотек.
3. следующего поколения последовательности
-
Последовательности библиотеки согласно рабочего инструмента NGS реагент комплект Производитель инструкции17,18 (таблица материалов).
- Подготовьте лист образца согласно инструкции производителя18 с помощью соответствующего NGS технологии программного обеспечения (Таблица материалов), которые будут импортированы в рабочего инструмента NGS рабочего процесса.
Примечание: Для целей ONDRISeq, выбран параметр приложения является «другой», с только FASTQ файлы просил (рис. 1). Последующие шаги будут обрабатывать эти файлы FASTQ, чтобы разрешить для полной настройки выравнивания и параметров качества. Однако если выбраны целевые последовательности, некоторые инструменты NGS возможность обрабатывать последовательности данных в VCF файлы сами. Инструкции производителя18 могут ознакомиться за полный выбор вариантов. - Если с помощью облачной вычислительной среды19 (Таблица материалов), войдите в систему при настройке последовательности запуска. Сделать это после нажатия кнопки «Последовательности» на домашней странице рабочего инструмента NGS.
- После библиотека денатурации18 согласно инструкциям производителя измерения концентрации ДНК библиотека с флуориметр14.
- Проверка качества библиотеки ДНК, с использованием системы соответствующих автоматизированных электрофореза и ДНК качества анализа комплект20 (Таблица материалов), в соответствии с инструкциями производителя.
- Чтобы преобразовать концентрации ДНК от нг/мкл Нм, используйте следующую формулу16
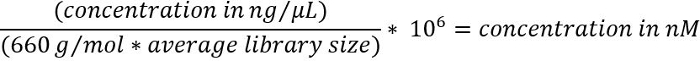
Примечание: Размер средняя библиотека будет характерных для цели обогащения комплект используется и могут быть получены из трассировки электрофореза, наблюдается в шаге 3.1.4. - Разбавить последовательности библиотеки до конечной концентрации 6 – 20 вечера, в соответствующих случаях и объем 600 мкл, согласно инструкции производителя21.
Примечание: Точная концентрация необходимых зависит от последовательности комплект используется. Проконсультируйтесь с производителем комплект обогащения для определения концентрации загрузку. - Разбавлять, Денатурировать и включают в себя положительный контроль последовательности библиотеки21, согласно инструкциям производителя.
- Вести журнал каждой последовательности запуска, которая включает в себя концентрации ДНК библиотека загружена (pM), процент положительных управления добавил, реагент картридж штрих, приложения, выбранного на шаге 3.1.1, количество индекс читает, Комплект обогащения используется, читать Длина и Имя образца листа.
Примечание: Время выполнения NGS рабочего документа будет зависеть инструмента, Комплект для обогащения и читать длины выбрана (4 – 56 h для секвенсора, используемых в этом эксперименте22).
- Подготовьте лист образца согласно инструкции производителя18 с помощью соответствующего NGS технологии программного обеспечения (Таблица материалов), которые будут импортированы в рабочего инструмента NGS рабочего процесса.
- По завершении последовательности выполнения доступ к «Запустить папке», которая включает в себя все выходы, перейдя к домашней странице NGS рабочего документа и нажав кнопку «Управление файлами». Переместите файлы на локальном диске для последующего доступа. Для отдельный параметр, на компьютере найдите файлы в облаке вычислительной среды19 , выбрав «Работает» на панели навигации. Выбор соответствующей последовательности запуска для перехода к странице запуска резюме. Выберите «Загрузить» для получения данных из облака. В диалоговом окне, которое появляется выберите файлы, FASTQ как тип файла для загрузки и нажмите кнопку «Скачать».
-
На странице Запуск резюме облачной вычислительной среды19,23перейдите к «Графики» для анализа качества последовательности, работать с различными фигурами, производимые вычислительной среды. Обратитесь к инструкции производителя23 для подробной информации о каждом рисунке производится.
- На странице запуска графики найдите рисунок с надписью «Данные по цикла». В разделе Диаграмма выберите «Интенсивность» и под канал выберите «Все каналы». Убедитесь, что этот сюжет интенсивности сигнала производится аналогично вырабатываемое виртуализации запускается, выступал в прошлом с же комплект обогащения и NGS рабочего инструмента.
Примечание: Это отражает процент интенсивности, проявленные каждой базы во всех 150 циклов. Цифра может варьироваться в зависимости от обогащения комплект используется, поэтому его необходимо сравнить с прошлых запусков последовательности той же панели. - В панели запуска навигации, чтобы найти индексации гистограммы контроля качества (КК), который находится на правой стороне страницы, перейдите на вкладку «Индексации QC». Убедитесь, что относительно равномерное распределение % читает определены (PF) наблюдается во всех образцах.
Примечание: Если любые образцы имеют гораздо меньше % читает определены (PF) чем остальная часть образцов, обратите внимание, что качество данных последовательности могут быть затронуты.
- На странице запуска графики найдите рисунок с надписью «Данные по цикла». В разделе Диаграмма выберите «Интенсивность» и под канал выберите «Все каналы». Убедитесь, что этот сюжет интенсивности сигнала производится аналогично вырабатываемое виртуализации запускается, выступал в прошлом с же комплект обогащения и NGS рабочего инструмента.
-
На странице Запуск резюме облачной вычислительной среды перейдите к метрики качества, нажав «Метрика» в рамках выполнения навигационной панели.
Примечание: Показатели обрезков будет зависеть от виртуализации платформы и обогащения комплект используется. Существует много метрик, которые могут быть использованы на основании инструкции производителя23, с помощью следующих шагов, выделив три, что особенно рекомендуется для контроля качества.- Под «Плотность (K/мм2)» обеспечение кластера плотность находится в пределах диапазона, рекомендованный комплект обогащения используется (в данном случае K 1200-1400/мм2).
- Под общей «% ≥Q30» убедитесь, что значение растворимость≥85%, отражающие качество чтения последовательности.
Примечание: Если меньше, чем этот порог 85%, обратите внимание, что качество последовательности может быть нарушена. - Под «Выравнивание (%)» убедитесь, что значение похож на % позитивных элемента управления, который был включен в последовательность запуска.
Примечание: Это действует как мера позитивного управления, таким образом, что только этот процент всего читает были найдены для выравнивания положительный контроль генома. Если был использован 1% положительный контроль будет ожидать, что выравнивание (%) будет ~ 1 – 5%.
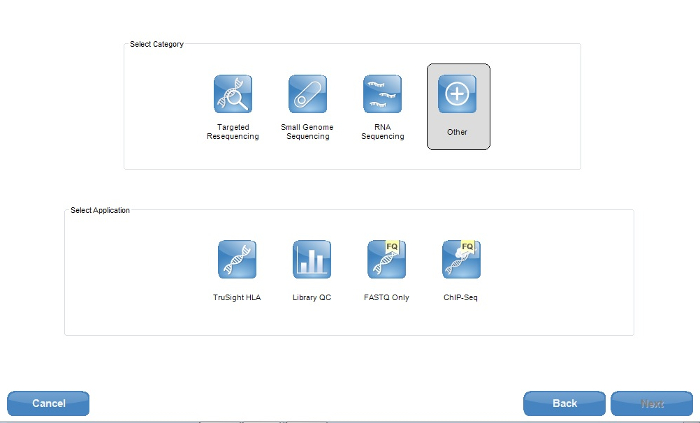
Рис: скриншот из NGS software технология (таблица материалов) образец приложения параметры листа создатель. Для целей ONDRISeq FASTQ только приложение используется. Однако если пользователь хотел бы другие файлы, созданные, например, VCF файлов, рекомендуется использовать приложение в пределах целевых resequencing категории. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
4. ампликонов и вариант вызова
- Для предварительной обработки данных, выберите соответствующее программное обеспечение для выравнивания raw файлов FASTQ в геноме человека ссылки и вызова варианты (Таблица материалов).
- Импорт FASTQ последовательности читает в программное обеспечение предварительной обработки данных.
Примечание: Для целей ONDRISeq, 48 FASTQ файлы, созданные из одной последовательности запуска 24 проб импортируются и обрабатываются с помощью программного обеспечения. Количество выборок, обрабатываются одновременно может варьироваться в зависимости от потребностей исследователь и размер панели NGS.- В рамках «Область переходов» щелкните правой кнопкой мыши и выберите «Создать папку». Имя папки таким образом, что нет ясности в отношении последовательности запуска, была выполнена.
- Из панели инструментов в верхней части выберите «Импорт». В раскрывающемся списке Список платформ виртуализации, показано выбрал платформу, с которой выполнялась последовательность.
Примечание: Для целей ONDRISeq, выбирается «Illumina». Однако если с помощью различных виртуализации платформы консультации инструкции изготовителя на оставшуюся FASTQ импорта шаги24. - В диалоговом окне перейдите и выберите FASTQ файлы из последовательности запуска, который обрабатывается. Убедитесь, что импортируемые файлы хранятся в и импортированных с локального диска, если используется компьютер с несколькими серверами.
- От «общие параметры» диалогового окна щелкните поле рядом с «Сопряженные гласит» если последовательность используется парных конец химия.
Примечание: В этом случае следует также два FASTQ образцы, ввозимые для каждого образца - один вперед и один обратный. - Сопряженные, читать информацию диалогового окна выберите «Сопряженные конец (вперед назад)» Если нападающий читать, что FASTQ файл появляется перед обратного чтения в списке файлов. Если файлы отображаются в обратном порядке, выберите «Мате пара (реверс вперед)». Установите парных чтения минимальное расстояние до 1 и максимальное расстояние до 1000, чтобы позволить для обнаружения мелких структурных перестановок внутри образца последовательности.
- От «Illumina параметры» диалогового окна, выберите «Удалить не читает», чтобы удалить чтений, которые не последовательности. NGS рабочего документа об исключении из мультиплексированных данных прежде чем экспортировать файлы FASTQ не флажок «MiSeq де мультиплексирования».
- Раскрывающемся списке «Показатель качества» выберите NGS трубопровода, который был использован для виртуализации. Выберите «Далее» в нижней части диалогового окна.
Примечание: Конвейер используется будет влиять на формат показателей качества файла FASTQ. Дополнительные сведения о какой трубопровода для выбора обратитесь к инструкции производителя24. - Новое диалоговое окно, выберите «сохранить» и «создать подпапки на единицу ванна положить каждый образец FASTQ файлы в их собственной отдельной папки. Выберите «Далее» в нижней части диалогового окна.
- Из нового диалогового окна выберите папку, созданную на шаге 4.2.1. Это где будет импортировать файлы FASTQ. Выберите «Готово» в нижней части диалогового окна и подождать до тех пор, пока FASTQ файлы импортируются. Чтобы увидеть состояние файла импорта, перейдите на вкладку «Процессы».
- Дизайн рабочего процесса внутри программное обеспечение для выполнения ампликонов и вариант вызова, в соответствии с инструкциями производителя.
Примечание: Этот рабочий процесс может меняться в зависимости от потребностей исследователь, но следующие шаги включают, что входит в целях ONDRISeq (рис. 2). Шаги в этом рабочем процессе может применяться к другим NGS ампликонов и вариант вызова программного обеспечения в соответствующих случаях. Все биоинформатики для целей ONDRI обработки выполняется со ссылкой на геном человека ссылка GRCH37/hg19, для последовательности обработки и анализа данных.- Карта читает секвенирования генома ссылку.
- При настройке, выберите ссылку генома при необходимости, обеспечение того, что это же ссылка генома, который используется для всех шагов биоинформатики.
- Маскировки режим раскрывающегося списка выберите «Без маскировки» таким образом, чтобы не регионов ссылка последовательности маскируются.
- Используйте по умолчанию, сопоставление параметров, назначенные программного обеспечения. Просмотрите инструкции производителя24 чтобы убедиться, что это приемлемо на основе целей исследования.
- Включите в рабочий процесс местные перестройки в геноме человека ссылки для разрешения любого чтения сопоставление ошибок, особенно вокруг варианты вставки удаление.
- Используйте местные перестройки параметры по умолчанию, присвоенный программного обеспечения. Просмотрите инструкции производителя24 чтобы убедиться, что это приемлемо на основе целей исследования.
- Удалите повторяющиеся сопоставленных читает, производимые методом ПЦР в протоколе NGS уменьшить эффект смещения амплификации PCR, который может производить ложных срабатываний25.
- Установите «максимум представительства меньшинств последовательности (%)», основанный на потребностях исследования.
Примечание: Параметр мягким, как используется для целей ONDRISeq, составляет 5%; Однако настройка по умолчанию программного обеспечения является более строгие 20%. Когда два читает очень похожи, этот параметр определяет, если последовательность с меньшим количеством чтения графов следует рассматривать ошибку последовательности от предвзятости амплификации PCR. Таким образом установив на 5%, меньшинство читать счетчик должен быть ≤ 5% большинства считывания количество до исправлениями должна быть идентична читать большинство.
- Установите «максимум представительства меньшинств последовательности (%)», основанный на потребностях исследования.
- Экспорт статистики в целевых регионах в виде покрытия резюме текстового файла из чтения треков, созданного на шаге 4.3.3. Игнорируйте матчи неспецифической и сломанной пар в настройках. Выберите папку на локальном диске для этих файлов.
- Экспорт файла карты (BAM) выравнивание двоичной последовательности для каждого образца из чтения треков, созданного на шаге 4.3.3. Это содержит последовательность выравнивания данных, при необходимости в будущем анализа. Выберите папку на локальном диске для этих файлов.
- Выберите метод вариант обнаружения для вызова варианты в пределах последовательности.
Примечание: Когда можно предположения о плоидности образцов, рекомендуется использовать фиксированный плоидности вариант обнаружения алгоритм, как используется для целей ONDRISeq. Если это предположение не может быть выполнено, обратитесь к инструкциям производителя24 определить лучший алгоритм для целей исследований.- При настройке, от фиксированной плоидности вариантные параметры параметры установите плоидности нужным для образца организма. Установите «требуемый вариант вероятности», или вероятность того, что вариант был правильно вызван в для того чтобы сохранить, 90,0%.
- Используйте следующие рекомендуемые параметры для общих фильтров: «Минимальная освещение» из 10 x, «Минимальный количество» 2, «Минимальной читать частота» 20%, «Игнорировать сломанной пар», игнорировать неспецифических матчей, основанный на «Читает», и «Минимум читать длина» 20.
Примечание: Эти параметры основаны на цели ONDRISeq. Обратитесь к инструкциям производителя24 чтобы убедиться, что они подходят для исследования. - Используйте следующие рекомендуемые параметры для фильтров шума: «Базовая качества фильтры» с «радиус соседства», сопоставление показателя качества 5, «минимальные Центральной качества» сопоставления Оценка 20 и показатель сопоставления «Минимум соседства качества» 15; «фильтр направление чтения» 5,0%; и «Относительная читать направление фильтра» значение 1,0%.
Примечание: Эти параметры основаны на цели ONDRISeq. Обратитесь к инструкциям производителя24 чтобы убедиться, что они подходят для исследования.
- Фильтровать варианты, которые были призваны на основании их совпадения с целевой группы в целевых регионах как указано в файле браузера расширяемый данных (кровать), позволяя только те варианты, которые имели место в пределах геномной областей, выбранных для целевой группы NGS сохранить.
Примечание: Файл кровать будет уникальным для целевых NGS панель, которая используется, основанный на регионах, Группа не в состоянии покрыть генома. - Экспорт из вариант трек, производится в шаге 4.3.7 вариант отчета в файл формата (VCF) вариант вызова. Выберите папку на локальном диске для этих файлов.
- Сохранить и установить процесс согласно инструкции производителя24, чтобы сделать его доступным в программное обеспечение «панели инструментов». Убедитесь, что рабочий процесс с именем таким образом, что это ясно в будущем, какие НГС группа подходит для.
- В диалоговом окне «Экспорт справочных данных» варианты во время установки установите все опции на «Пакет».
- В диалоговом окне «Установить местоположение» варианты во время установки нажмите кнопку «Установить рабочего процесса на локальном компьютере».
- Карта читает секвенирования генома ссылку.
- Запуск импорта FASTQ последовательности чтения файлов через процесс заказной биоинформатики в шаге 4.3, согласно инструкции производителя24.
- Определить рабочий процесс, разработанный в шаге 4.3 программного обеспечения «панели инструментов» и дважды щелкните его.
- В диалоговом окне, которое появляется найдите папки файлов FASTQ, которые были импортированы в шаге 4.2 в пределах «Область переходов». Выделите все папки, выбрав их в области «Навигация» и затем щелкните поле рядом с «Партии». Используйте кнопку со стрелкой вправо для перемещения файлов на «Выбранные элементы». Нажмите кнопку «Далее» в нижней части диалогового окна.
- В диалоговом окне Обзор «пакетного» для обеспечения были выбраны правильные файлы FASTQ и затем нажмите кнопку «Далее».
- Обзор следующие шаги рабочего процесса в диалоговом окне чтобы обеспечить правильные файлы и экспортировать места были выбраны при проектировании рабочего процесса в действии 4.3: «Карта читает в Справочник»; Удалить дубликаты сопоставленных читает»; «Создание статистики для целевых регионах»; «Экспорт БАМ»; «Экспорт табуляции текста;» «Фильтр на основе совпадения»; и «Экспортировать VCF»
- В заключительном шаге в диалоговом окне-«результат обработки» - выберите вариант «сохранить в Входной папке». Нажмите «Готово» в нижней части диалогового окна.
Примечание: Это означает, что файлы производится для каждого образца будет помещен в ту же папку, что хранит файл FASTQ в пределах данных предварительной обработки программного обеспечения.
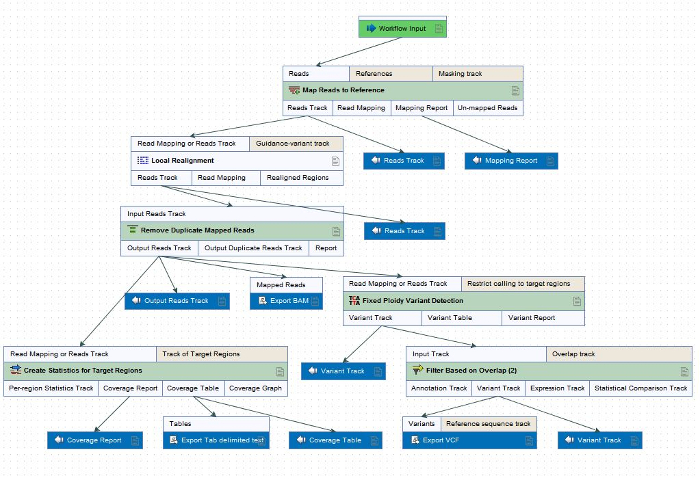
Рисунок 2: рабочий процесс для ампликонов и вариант вызова FASTQ файлы в пределах данных предварительной обработки программного обеспечения (таблица материалов) настроены для целей ONDRISeq. Действия в рабочем процессе могут быть применены к другим NGS ампликонов и вариант вызова программного обеспечения на основе потребностей исследователя. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
5. вариант аннотации
-
Скачать и настроить скрипт26 аннотировать вариации (ANNOVAR) для выполнения вариант аннотации каждого образца после VCF-файл.
- Скачать следующие базы данных от ANNOVAR быть включены в качестве аннотации: 1) RefSeq27 (ДОПОЛНЕНО августа 2015); 2) dbSNP13828 (Сентябрь 2014 обновление); 3 консорциум агрегации Exome29 (ExAC, версия 0.3 ноября 2015 года обновление); 4 Национальный сердца, легких и крови института Exome секвенирование проекта Европейской когорты30 (ESP, марта 2015 обновление); 5 1000 геномов проекта европейских когорты31 (1KGP, обновление августа 2015); 6) ClinVar32 (марта 2016 обновление); и 7) комбинированный аннотации зависимых истощения33 (CADD), сортировка нетерпимый терпимая(ый)34 (ПРОСЕЯТЬ) и PolyPhen-235.
Примечание: Геном координирует и всех баз данных, на который ссылается ANNOVAR передала генома человека построения GRCh37/hg19. Кроме того перечисленные версии базы данных, которые используются для целей ONDRISeq, при загрузке баз данных использовать самые современные доступные версии. - При желании настроить ANNOVAR для вывода полного списка аннотированных варианты, а также снижение сборник аннотированного варианта с использованием операции фильтра26.
Примечание: Сокращение списка могут быть настроены на основе потребностей исследователя. Для целей ONDRISeq, сокращение перечня аннотированного варианта не включают в себя варианты, которые происходят дальше, чем 15 баз из ближайшего экзона или любые варианты с частотой незначительные аллеля (ТБР) > 3% в любой из трех баз данных: 1) ExAC; 2) ESP; и 3) 1KGP. Этот шаг очень рекомендуется. - При желании настройте ANNOVAR выделить конкретные аллеля звонков, основанных на потребностях исследователь26.
Примечание: Для целей ONDRISeq, ANNOVAR оценивает последовательности вызовов для rs429358 аллели АРОЕ риска (C > T):p.C130R и rs7412 (C > T):p.R176C для вывода общего АРОЕ генотип, из которых существует шесть возможных комбинации, в том числе: 1) E2/E2; 2), E3/E2; 3) E4/E2; 4), E3/E3; 5) E4/E3; 6) E4/E4. Из этих шести возможных АРОЕ генотипов Е4/E4 является наиболее общепринятой генетическим фактором риска для развития болезни Альцгеймера поздним началом36.
- Скачать следующие базы данных от ANNOVAR быть включены в качестве аннотации: 1) RefSeq27 (ДОПОЛНЕНО августа 2015); 2) dbSNP13828 (Сентябрь 2014 обновление); 3 консорциум агрегации Exome29 (ExAC, версия 0.3 ноября 2015 года обновление); 4 Национальный сердца, легких и крови института Exome секвенирование проекта Европейской когорты30 (ESP, марта 2015 обновление); 5 1000 геномов проекта европейских когорты31 (1KGP, обновление августа 2015); 6) ClinVar32 (марта 2016 обновление); и 7) комбинированный аннотации зависимых истощения33 (CADD), сортировка нетерпимый терпимая(ый)34 (ПРОСЕЯТЬ) и PolyPhen-235.
-
Запрос базы данных заболеваний мутации (таблица материалы), чтобы определить, если варианты были ранее связаны с болезнью, разумных доказательств. Рассмотрим любые варианты, которые не были ранее зарегистрированы как Роман вариант.
- Оценить ANNOVAR аннотации от ClinVar, таким образом, что болезнь связанные варианты включают в себя любые классифицированы как вероятно патогенных и патогенных.
- Процесс сращивания варианты через предсказание в silico инструменты на основе сращивание анализ вариантов37 (SPANR) и человека сплайсинга Finder38 (ГФ, версия 3.0).
- Если обработка большое количество образцов, Сравните вариант звонков внутри каждого образца для определения, какие варианты являются общими для различных образцов. Сделать это вручную или с помощью специально сценария, позволяя для выявления возможных последовательности артефакты и загрязнение события.
Примечание: Для целей ONDRI, Пользовательский сценарий используется для аннотирования ANNOVAR выходных файлов, сравнивая их друг к другу. Сценарий включает аннотации, за вариант, с идентификатор субъекта любых других образцов, укрывательство же вариант, иначе называют вариант истории в когортное исследование. - Классификации на основе американского колледжа патогенности руководящие принципы медицинской генетики (ACMG)39, назначение каждого варианта классификации как один из следующих вариантов: 1) патогенных; 2) вероятно патогенных; 3) вариант неопределенной значимости; 4) скорее всего доброкачественные; или 5) доброкачественная.
Примечание: Для целей ONDRI, внутренний разработан сценарий Python используется для выполнения ACMG классификации на основе полуавтоматического. Хотя не используется для этого исследования, InterVar40 является аналогичным образом разработан инструмент, который может быть использован в Аналогичным образом. - Сэнгер последовательность любые варианты с охватом последовательности < 30 x и/или варианты, которые были определены в > 10% когортное исследование для проверки что они не являются секвенирования артефакты41.
Результаты
Методики, описанные здесь были применены к 528 участников образцы ДНК от лиц, которые были зачислены в ONDRI. Образцы были запущены на панели ONDRISeq в 22 работает 24 выборок на запуск. В целом, последовательности данных были полны решимости быть высокого качества с охватом среднего образца 78 ± 13 х и все отдельные прогоны выразил среднего образца покрытия > 30 x. Кроме того, в среднем, 94% всех целевых регионов были охвачены по крайней мере 20 x (Таблица 1).
Среднее 95,6% чтений были сопоставлены к последовательности ссылка и все ONDRISeq работает, имел > 90% читает сопоставлены (Таблица 1). Сопоставленные гласит, 92,0% имели Phred Оценка ≥Q30, только с одним запустить имея < 80% сопоставленных гласит, встреча этот показатель качества. Однако это по-прежнему отображается среднее освещение 79 x и 93% целевых регионов были охвачены по крайней мере 20 x.
| Параметр | Среднее (болезни) | Лучшее исполнение | Бедные производительность |
| Кластера плотность (x 103/мм2) | 1424 (±269) | 1347 | 1835 |
| Всего читает (106) | 43,1 (±6.0) | 48,7 | 47,4 |
| Сопоставлены читает (106) | 40.1 (±6.0) | 47,1 | 25,7 |
| Сопоставленные читает (%) | 95,6 (±1.3) | 96,8 | 92,6 |
| ≥Q30 Оценка качества PHRED (%) | 92,0 (±6.0) | 92 | 68,3 |
| Пример покрытия (x) | 78 (±13) | 99 | 51 |
Таблица 1: Виртуализация метрики качества для 22 работает на ONDRISeq.
Тематическое исследование: Выявление редких вариантов в PD пациента.
Чтобы продемонстрировать полезность наших целевых NGS рабочего процесса, мы представляем пример 68-летний, миастения, болезнь Паркинсона пациента. Образец ДНК был запущен на NGS рабочего инструмента (Таблица материалов) с помощью панели ONDRISeq наряду с 23 других образцов ONDRI. Запуск отображения кластера плотность 1555 x 103/мм2. Пациента в частности образец отображается среднее покрытие 76 x, 93,9% целевых регионов охватывает по меньшей мере 20 x.
После выполнения вариант вызова и аннотации с рабочим процессом пользовательского биоинформатики, пациент был найден до гавани 1351 вариантов в рамках экзонов и окружающие 250 bp 80 генов, включенных на панели ONDRISeq. Однако ANNOVAR трубопровод был в состоянии уменьшить количество вариантов, рассматривая вариант последовательности онтологию и ТБР, как описано выше. Это произвело список семи вариантов, которые прошли ручной курирование (рис. 3). Из этих семи вариантов два были определены как имеющие возможные клиническое значение. Этот процесс является конкретным потребностям ONDRI и было сделано путем выявления тех, которые встречаются относительно редко, населения в целом и nonsynonymous в онтологии, вызывая изменения в протеине. Ли вариант был ранее связан с болезнью, в silico предсказания deleteriousness белка и ACMG патогенности классификации вариантов также были использованы в этом процессе.
Первый из сокращение списка был гетерозиготных вариант, а именно LRRK2: c.T3939A, что приводит к нонсенс вариант p.C1313*. LRRK2 кодирует белок лейцин-богатые повторять 2 киназы, который обладает ГТФазы и киназы деятельность42. Кроме того быть среди ведущих причин семейная болезнь Паркинсона43известны мутации в пределах этого гена. Этот вариант представляет кодоном преждевременной остановки в пределах LRRK2, тем самым теряя аминокислотных остатков 1 314 – 2, 527. Это предотвращает перевод белка РАН сложных белков (Roc), Терминал C Roc (кор) и доменов киназы протеина, которые участвуют в функционировании как атипичная Rho ГТФазы, GTP белок и киназы протеина, соответственно и было предсказано чтобы нанести ущерб путем анализа в silico порожденных CADD (CADD Phred = 36). Этот вариант также редко с ТБР 0,004% и 0.01% в ExAC и ESP, соответственно и отсутствует от 1000G базы данных. Кроме того это единственный пациента из всех 528 виртуализации, кто носит этот вариант, который является роман, так как она не была ранее описана в заболевания мутация баз данных (Таблица материалов). Доверия вариант вызова было подтверждено его глубокое освещение 109 x. Наконец вариант оценивалась с AMCG стандартами и руководящими принципами для патогенности и был классифицирован как патогенных.
Пациент также осуществляется второй гетерозиготных вариант, NR4A2: c.C755A, что приводит к p.P252Q изменению Миссенс. Белков, кодируемых NR4A2, ядерных рецепторов Subfamily 4 группа членов 2, является фактор транскрипции, участвующих в поколение дофаминергические нейроны44 и мутации в пределах этого гена были ранее связаны с Паркинсона заболевание45. Замена неполярных пролина в полярных глютамин был предсказан повреждения в silico анализа прогнозирования порожденных CADD (CADD Phred = 21.1), но не на основе анализа, порожденных ПРОСЕЯТЬ или PolyPhen-2. Вариант является редким, с ТБР 0,004% в ExAC и отсутствие от ESP и 1000G. Вариант также была выявлена в ONDRI участник диагнозом сосудистых когнитивных нарушений, но не был описан ранее в базах данных заболеваний мутации. Этот вариант был охват только 18 x, однако, Сэнгер, последовательность будет выполняться для того, чтобы обеспечить ее действенность в пределах последовательности. Наконец вариант был определен неопределенное значение при оценке с ACMG стандартами и руководящими принципами для патогенности.
ONDRISeq панели и биоинформатики конвейер также может определить генотип АРОЕ каждого образца. Этот пациент был определен иметь АРОЕ генотип E3/E3.
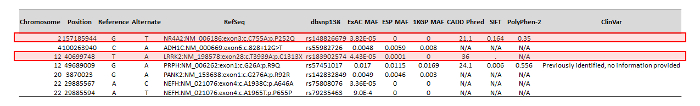
Рисунок 3: пример сокращения производства из ANNOVAR, отображение вручную куратор, аннотированного варианта. Снижение ANNOVAR выход из тематическое исследование по 68 лет, мужской, пациентов с болезнью Паркинсона. Аннотированного варианта куратор для выявления лиц, которые, скорее всего, иметь клиническую значимость, как обозначено красной коробки. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Обсуждение
На пути от извлечения образца ДНК для определения вариантов, которые могут представлять интерес при рассмотрении диагноз пациента, прогрессирования заболевания и возможных методов лечения важно признать разнообразный характер методологии, необходимых для последовательности и надлежащей обработки. Протокол, описанные здесь, является примером использования целевых NGS и последующих bioinformatic анализа необходимых для выявления редких вариантов потенциальных клиническое значение. В частности мы представляем подход подгруппы геномики ONDRI при использовании панели NGS специально ONDRISeq.
Признано, что эти методы были разработаны основанные на определенной платформе NGS и что есть другие платформы виртуализации и цели обогащения комплекты, которые могут быть использованы. Однако основаны на его ранних США продуктов питания и медикаментов (FDA) утверждения46был выбран NGS платформы и рабочего инструмента (Таблица материалов). Это разрешение отражает высокое качество виртуализации, которая может быть выполнена с протоколами NGS выбора и надежности, которые могут быть размещены на последовательности гласит.
Хотя получение точной последовательности чтения с глубиной охвата является очень важным, биоинформатики обработку, необходимую для окончательной редкий вариант анализа имеет жизненно важное значение и могут быть интенсивными. Из-за многочисленных источников ошибок, которые могут произойти в течение процесса виртуализации надежный биоинформатики трубопровода необходимо исправить для различных неточности, которые могут быть введены. Они могут возникать из перекосы в процессе сопоставления, усиление предвзятости, представленный амплификации PCR в подготовке библиотека и технология производства последовательности артефакты47. Независимо от того, программное обеспечение, используемое для выполнения чтения карт и вариант призвание есть общие способы уменьшить эти ошибки, включая местные перестройки, удаление дубликатов сопоставленных гласит и надлежащие параметры для контроля качества при вызове варианты. Кроме того параметры, выбранные при вариант вызова может различаться в зависимости от того, что является наиболее подходящим для изучения на руку11. Минимальное освещение и показатель качества вариант и окружающие нуклеотидов, которые были применены здесь были выбраны относительно создания баланса между соответствующим специфичность и чувствительность. Эти параметры были проверены для панели ONDRISeq, основанный на вариант вызова согласование с трех отдельных генетических методов, как описано ранее, включая: 1) на основе чипа генотипирования; 2) проба аллельные дискриминации; и 3) Сэнгер последовательности9.
После точной вариант вызова, чтобы определить те потенциальные клиническое значение, аннотации и курирование важны. Из-за своей платформы открытого доступа ANNOVAR является отличным инструментом для аннотации и предварительный вариант скрининг или ликвидации. Помимо того, легко доступны, ANNOVAR может быть применен к любой файл VCF, независимо от того, какая платформа виртуализации используется, и настраиваемые основывается на потребностях исследований26.
После аннотации должны толковаться варианты для определения, если они должны считаться клиническое значение. Не только делает этот процесс становится сложным, но это часто склонны к субъективности и человеческой ошибки. По этой причине ACMG установила руководящие принципы для оценки доказательств для патогенности любого варианта. Мы применяем не являются синонимами, редкий вариант основе ручной курирование подхода, который строится на основе этих руководящих принципов и обеспечиваются индивидуальной оценки каждого варианта, который способен пройти через конвейер с специально Python скрипт, что классифицирует варианты, основанные на руководящих принципах. Таким образом, каждый вариант присвоен рейтинг патогенных, вероятно патогенных, неопределенное значение, вероятно, доброкачественные, или доброкачественной, и мы можем добавить вариант курирование процесса стандартизации и прозрачности. Это очень важно признать, что специфика вариант курирование, за пределы конвейера биоинформатики, будет индивидуальный на основе потребностей научных исследований и поэтому выходит за рамки представленных методологий.
Хотя представленные здесь методы являются специфическими для ONDRI, описанные шаги могут быть переведены, при рассмотрении большого числа заболеваний конституционных интересов. Увеличением числа ассоциаций гена для многих фенотипов, целевых NGS обеспечивает гипотеза ориентированный подход, который можно использовать на предыдущих исследований, которая была проделана в области. Тем не менее существуют ограничения целевых NGS, и представил методологии. Сосредоточив внимание только на конкретных регионах генома, области обнаружения ограничены Роман аллели интерес. Таким образом, Роман генов или других геномной локусов, помимо тех, которые охватываются последовательности целей, которые могут быть раскрыты с WGS или Уэс подходы, не будут идентифицированы. Есть также регионы генома, который может быть трудно точно последовательность с NGS подходов, включая те с высокой степенью повторяющиеся последовательности48 или те, которые богаты контента GC49. К счастью существует при использовании целевых NGS, априори высокую степень знакомство с геномной регионами время виртуализации, и ли они могут представлять технические проблемы. Наконец обнаружение число вариантов Копировать от NGS данных в настоящее время не является стандартизированной50. Однако Биоинформатика решения этих проблем может быть на горизонте; новые вычислительные инструменты могут помочь проанализировать эти дополнительные формы изменчивости у ONDRI больных.
Несмотря на свои ограничения целенаправленных NGS-это возможность получения высокого качества данных, в рамках подхода, ориентированного на гипотезе, оставаясь при этом дешевле, чем ее коллеги РГ и Уэс. Не только подходит эта методология для эффективной и режиссер исследований, клинических осуществление целенаправленных NGS растет в геометрической прогрессии. Эта технология используется для ответа на многие различные вопросы, касающиеся молекулярные pathways различных заболеваний. Он также разрабатывается в точной диагностики при относительно низких затратах, когда против Уэс и РГ. Даже по сравнению с золотой стандарт Сэнгер виртуализации, целевых NGS можно переиграть в свое время - и эффективности затрат. По этим причинам важно для ученого или врач, который получает и использует данные NGS, например, поставляется в виде текста в лаборатории или клинических отчетов, чтобы понять комплекс «черный ящик», которая лежит в основе результаты. Методы, представленные в настоящем документе должно помочь пользователям понять этот процесс, лежащие в основе поколения и интерпретации данных NGS.
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Мы хотели бы поблагодарить всех участников ONDRI за их согласие и сотрудничество с нашего исследования. Спасибо ONDRI следователей (www. ONDRI.ca/people), включая наш ведущий следователь (MJS) и ONDRI управляющих комитетов: Исполнительный комитет, Руководящий комитет, публикации Комитета, рекрутинг Комитет, оценке платформ и группой управления проектом. Мы также благодарим Лондонский региональный центр геномики для их технического опыта. AAD поддерживается Alzheimer обществом Лондона и Мидлсекс мастеров выпускник исследовательских стипендий. SMKF поддерживается ALS Канада Тим э. Ноэль докторантура стипендий.
Материалы
| Name | Company | Catalog Number | Comments |
| 4 mL EDTA K2 tubes | Fisher Scientific | 02-689-4 | |
| 1 M Tris Buffer | Bio Basic Canada Inc. | SD8141 | |
| Gentra Puregene Blood Kit | Qiagen | 158389 | 1,000 mL Kit. This is the blood extraction kit, referred to in step 1.3. |
| NanoDrop-1000 Spectrophotometer | Thermo Fisher Scientific | ND-2000 | Replaced by the NanoDrop-2000 Spectrophotometer. This is the full-spectrum spectrophotometer, referred to in steps 1.4 and 2.1.2. |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | This is a fluorometer appropriate for the quantification of DNA, referred to in steps 2.1.4, 2.1.6, 2.2.3, and 3.1.3. |
| Nextera Rapid Custom Capture Enrichment Kit | Illumina, Inc. | FC-140-1009 | Specifically designed for the ONDRISeq panel, sequencing the exons of 80 genes, resulting in 971,388 base pairs of sequence in paired-end reads of 150 bases in length; 288 samples per kit. This is the target enrichment kit, referred to in steps 2.2, 2.2.2, 2.2.3, 3.1.5, 3.1.6, 3.4.1, and the Discussion. |
| 2100 BioAnalyzer | Agilent Technologies | G2939BA | This is a automated electrophoresis system, referred to in step 3.1.4. |
| High Sensitivity DNA Reagent Kit | Agilent Technologies | 5067-4626 | 110 Samples per kit; This is a DNA quality analysis kit, referred to in step 3.1.4. |
| MiSeq Reagent Kit v3 | Illumina, Inc. | MS-102-3003 | 600 Cycle Kit; This is the NGS desktop instrument reagent kit, referred to in step 3.1. |
| MiSeq Personal Genome Sequencer | Illumina, Inc. | SY-410-1003 | This is a NGS desktop instrument, referred to in steps 2.2.1, 3.1, 3.1.1, 3.1.2, 3.1.8, 3.2, 4.2.6, the Representative Results, and the Discussion. |
| Experiment Manager | Illumina, Inc. | This is NGS technology software, referred to in step 3.1.1 and Figure 1. https://support.illumina.com/sequencing/sequencing_software/experiment_manager/downloads.html | |
| BaseSpace | Illumina, Inc. | SW-410-1000 | This is a cloud-based computing environment, referred to in steps 3.1.2, 3.2, 3.3, 3.3.1, 3.3.2, 3.4, 3.4.1, 3.4.2 and 3.4.3. https://basespace.illumina.com/ |
| CLC Genomics Workbench 10.1.1 | Qiagen | 832000 | Open source options for data pre-processing are also available that can model the workflow used in this protocol. This is the software used for data pre-processing, referred to throughout step 4 and in Figure 2. |
| Annotate Variation | http://annovar.openbioinformatics.org/en/latest/user-guide/download/ | ||
| RefSeq | National Center for Biotechnology Information | https://www.ncbi.nlm.nih.gov/refseq/ | |
| dbSNP138 | National Center for Biotechnology Information | https://www.ncbi.nlm.nih.gov/projects/SNP/snp_summary.cgi?view+summary=view+summary&build_id=138 | |
| Exome Aggregation Consortium | Broad Institute | http://exac.broadinstitute.org/ | |
| National Heart, Lung, and Blood Institute Exome Sequencing Project European Cohort | University of Washington and the Broad Institute | http://evs.gs.washington.edu/EVS/ | |
| ClinVar | National Center for Biotechnology Information | https://www.ncbi.nlm.nih.gov/clinvar/ | |
| Combined Annotation Dependent Depletion | University of Washington and Hudson-Alpha Institute for Biotechnology | http://cadd.gs.washington.edu/ | |
| Sorting Intolerant from Tolerant | J. Craig Venter Instutite | http://sift.jcvi.org/ | |
| PolyPhen-2 | Brigham and Women's Hospital, Harvard Medical School | http://genetics.bwh.harvard.edu/pph2/ | |
| Human Gene Mutation Database | Qiagen | 834050 | This is a disease mutation database, referred to in step 5.2 and the Representative Results. https://portal.biobase-international.com/cgi-bin/portal/login.cgi?redirect_url=/hgmd/pro/start.php |
| Splicing-based Analysis of Variants | Frey lab, University of Toronto | http://tools.genes.toronto.edu/ | |
| Human Splicing Finder | Aix Marseille Université | http://www.umd.be/HSF3/HSF.shtml | |
| Other materials | |||
| Centrifuge | |||
| Disposable transfer pipets |
Ссылки
- Metzker, M. L. Sequencing technologies - the next generation. Nat Rev Genet. 11 (1), 31-46 (2010).
- Mardis, E. R. Next-generation DNA sequencing methods. Annu Rev Genomics Hum Genet. 9, 387-402 (2008).
- Shendure, J., Ji, H. Next-generation DNA sequencing. Nat Biotechnol. 26 (10), 1135-1145 (2008).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci U S A. 74 (12), 5463-5467 (1977).
- Farhan, S. M. K., Hegele, R. A. Exome Sequencing: New Insights into Lipoprotein Disorders. Current Cardiology Reports. 16 (7), (2014).
- Choi, M., et al. Genetic diagnosis by whole exome capture and massively parallel DNA sequencing. Proc Natl Acad Sci U S A. 106 (45), 19096-19101 (2009).
- Mardis, E. R. DNA sequencing technologies: 2006-2016. Nat Protoc. 12 (2), 213-218 (2017).
- Farhan, S. M., et al. The Ontario Neurodegenerative Disease Research Initiative (ONDRI). Can J Neurol Sci. 44 (2), 196-202 (2017).
- Farhan, S. M. K., et al. The ONDRISeq panel: custom-designed next-generation sequencing of genes related to neurodegeneration. NPJ Genom Med. (16032), 1-11 (2016).
- El-Metwally, S., Hamza, T., Zakaria, M., Helmy, M. Next-generation sequence assembly: four stages of data processing and computational challenges. PLoS Comput Biol. 9 (12), e1003345 (2013).
- Yohe, S., Thyagarajan, B. Review of Clinical Next-Generation Sequencing. Arch Pathol Lab Med. , (2017).
- Qiagen. . Gentra Puregene Handbook. , (2014).
- NanoDrop Technologies, Inc. . Spectrophotometer V3.5 User's Manual. , (2007).
- Invitrogen by Life Technologies. . Qubit 2.0 Fluorometer User Manual. Vol. Q32866. , (2010).
- Illumina, Inc. . Nextera Rapid Capture Enrichment Guide. , (2016).
- Illumina, Inc. . Nextera Rapid Capture Enrichment Reference Guide. , (2016).
- Rev. B. Illumina, Inc. . MiSeq Reagent Kit v3 Reagent Preparation Guide. , (2013).
- Illumina, Inc. . MiSeq System Guide. , (2015).
- . BaseSpace Sequence Hub Available from: https://basespace.illumina.com/dashboard (2017)
- Rev. B. Agilent Technologies. . Agilent High Sensitivity DNA Kit Guide. , (2013).
- Illumina, Inc. . MiSeq System Denature and Dilute Libraries Guide. , (2016).
- Illumina, Inc. . System Specification Sheet: MiSeq System. , (2016).
- . BaseSpace Sequence Hub Help Center Available from: https://help.basespace.illumina.com/ (2017)
- Qiagen. . Genomics Workbench 10.1.1 User Manual. , (2017).
- Ebbert, M. T., et al. Evaluating the necessity of PCR duplicate removal from next-generation sequencing data and a comparison of approaches. BMC Bioinformatics. 17, 239 (2016).
- Wang, K., Li, M., Hakonarson, H. ANNOVAR: functional annotation of genetic variants from high-throughput sequencing data. Nucleic Acids Res. 38 (16), e164 (2010).
- Leary, N. A., et al. Reference sequence (RefSeq) database at NCBI: current status, taxonomic expansion, and functional annotation. Nucleic Acids Res. 44 (D1), D733-D745 (2016).
- Kitts, A., Phan, L., Ward, M., Bradley Holmes, J. . The Database of Short Genetic Variation (dbSNP). , (2013).
- Lek, M., et al. Analysis of protein-coding genetic variation in 60,706 humans. Nature. 536 (7616), 285-291 (2016).
- Auton, A., et al. A global reference for human genetic variation. Nature. 526 (7571), 68-74 (2015).
- Landrum, M. J., et al. ClinVar: public archive of interpretations of clinically relevant variants. Nucleic Acids Res. 44 (D1), D862-D868 (2016).
- Kircher, M., et al. A general framework for estimating the relative pathogenicity of human genetic variants. Nat Genet. 46 (3), 310-315 (2014).
- Kumar, P., Henikoff, S., Ng, P. C. Predicting the effects of coding non-synonymous variants on protein function using the SIFT algorithm. Nat Protoc. 4 (7), 1073-1081 (2009).
- Adzhubei, I. A., et al. A method and server for predicting damaging missense mutations. Nat Methods. 7 (4), 248-249 (2010).
- Bertram, L., McQueen, M. B., Mullin, K., Blacker, D., Tanzi, R. E. Systematic meta-analyses of Alzheimer disease genetic association studies: the AlzGene database. Nat Genet. 39 (1), 17-23 (2007).
- Xiong, H. Y., et al. The human splicing code reveals new insights into the genetic determinants of disease. Science. 347 (6218), (2015).
- Desmet, F. O., et al. Human Splicing Finder: an online bioinformatics tool to predict splicing signals. Nucleic Acids Res. 37 (9), e67 (2009).
- Richards, S., et al. Standards and guidelines for the interpretation of sequence variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology. Genet Med. 17 (5), 405-424 (2015).
- Li, Q., Wang, K. InterVar: Clinical Interpretation of Genetic Variants by the 2015 ACMG-AMP Guidelines. Am J Hum Genet. 100 (2), 267-280 (2017).
- Yang, Z. L., Sun, G. L. High-frequency, low-coverage "false positives" mutations may be true in GS Junior sequencing studies. Scientific Reports. 7, (2017).
- Gandhi, P. N., Wang, X., Zhu, X., Chen, S. G., Wilson-Delfosse, A. L. The Roc domain of leucine-rich repeat kinase 2 is sufficient for interaction with microtubules. J Neurosci Res. 86 (8), 1711-1720 (2008).
- Goldwurm, S., et al. The G6055A (G2019S) mutation in LRRK2 is frequent in both early and late onset Parkinson's disease and originates from a common ancestor. J Med Genet. 42 (11), e65 (2005).
- Caiazzo, M., et al. Direct generation of functional dopaminergic neurons from mouse and human fibroblasts. Nature. 476 (7359), 224-227 (2011).
- Grimes, D. A., et al. Translated mutation in the Nurr1 gene as a cause for Parkinson's disease. Mov Disord. 21 (7), 906-909 (2006).
- Collins, F. S., Hamburg, M. A. First FDA authorization for next-generation sequencer. N Engl J Med. 369 (25), 2369-2371 (2013).
- Van der Auwera, G. A., et al. From FastQ data to high confidence variant calls: the Genome Analysis Toolkit best practices pipeline. Curr Protoc Bioinformatics. 43, 11-33 (2013).
- Treangen, T. J., Salzberg, S. L. Repetitive DNA and next-generation sequencing: computational challenges and solutions. Nat Rev Genet. 13 (1), 36-46 (2011).
- Shin, S., Park, J. Characterization of sequence-specific errors in various next-generation sequencing systems. Mol Biosyst. 12 (3), 914-922 (2016).
- Povysil, G., et al. panelcn.MOPS: Copy-number detection in targeted NGS panel data for clinical diagnostics. Hum Mutat. 38 (7), 889-897 (2017).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены