Method Article
다음-세대 시퀀싱과 생물 정보학 파이프라인 헌법 질병의 유전 결정 요인 평가 대상
요약
타겟된 다음-세대 시퀀싱 질병 연구 및 임상 진단에서 점점 인기 끌고있다는 시간 및 비용 효율적인 접근 이다. 여기에 설명 된 프로토콜 시퀀싱에 필요한 복잡 한 워크플로 및 질병을 유전 이체를 식별 하는 데 사용 하는 생물 정보학 과정을 제공 합니다.
초록
차세대 시퀀싱 (NGS)은 신속 하 게 헌법 질병의 유전 결정 요인에 대 한 연구를 수행 하는 방법을 혁명을. 기술은 짧은 기간에 그리고 상대적으로 낮은 비용으로 생산 되 고 시퀀싱 읽기의 수백만 매우 효율적입니다. 특히, 타겟된 NGS 연구의 질병에 따라 특정 관심의 게놈 영역을 집중 조사를 수 있다. 뿐만 아니라이 감소 비용을 추가 하 고 프로세스의 속도 증가 하지만 종종 NGS를 함께 계산 부담을 lessens. 타겟된 NGS는 게놈의 특정 지역에 제한, 관심의 잠재적인 소설 loci의 그것 수 있습니다 방지는 phenotypically 유전자 유형이 다른 질병에 직면 하는 우수한 기법 이전 알려진된 유전 연결입니다. 시퀀싱 기술의 복잡 한 특성, 그것은 밀접 하 게 시퀀싱 읽기 높은 범위와 품질을 달성 하기 위하여 프로토콜 및 방법론을 준수 하는 것이 중요. 또한, 연속 읽기 얻을 수 있습니다, 일단 읽기 참조 게놈, 변종, 호출 하 고 변형 품질 기준을 통과 되도록 정확 하 게 매핑됩니다 정교한 생물 정보학 워크플로 이용 된다. 변종 해야 합니다 또한 주석 하 고 큐레이터 미국 대학의 의료 유전학 및 유전체학 Pathogenicity 지침을 적용 하 여 표준화 될 수 있는 그들의 임상 중요성에 따라. 여기에 소개 하는 방법을 생성 하 고 모델, ONDRISeq 신경 질병 패널을 사용 하 여 변형 임상 의미의 수 있습니다 식별 하는 타겟된 시퀀싱 패널에서 NGS 데이터 분석 단계 표시 됩니다.
서문
연구 및 병원, 차세대 시퀀싱 (NGS) 증명 하는 이러한 목표1,2 달성 하기 위해 높은 처리량 및 비용 효율적인 도구가 될 정의 하는 다양 한 조건의 결정 하는 유전 요인으로에 더 높은 우선 순위에 걸립니다. ,3. 거의 40 년 동안, 생어 시퀀싱 유전 이체4;을 식별에 대 한 황금 표준에 있 었 그러나, 유전이 성분 또는 알 수 없는 유전 병 인 질병, 많은 가능한 후보 유전자 평가 되어야 합니다, 자주 동시에. 이러한 맥락에서 생어 시퀀싱 비싸고 시간이 많이 소요 된다. 그러나, NGS 포함 DNA 조각의 수백만의 대규모 병렬 시퀀싱 동시에 게놈의 다양 한 지역에 걸쳐 다양 한 유전 변이 검출 하기 위하여 비용 및 시간 효율적인 기법에 대 한 허용.
NGS 시퀀싱 dna의 세 가지 유형이 있다: 1) 전체-게놈 시퀀싱 (WGS), 2) 전체 exome 시퀀싱 (웨스), 및 3) 타겟된 시퀀싱5. WGS 웨스 포함만 단백질 코딩 영역6게놈 시퀀싱 하는 동안 개인의 전체 게놈 콘텐츠를 평가 합니다. 일반적인 병 적인 메커니즘에 의해 연결 된 또는 알려진 상대적으로 적은 특정 유전자에 따라 게놈의 특정 지역에 초점을 맞추고 반면, 타겟 시퀀싱, 임상 표현 형. exons 또는 introns, 또는 유전자 또는 유전자의 특정 그룹의 모든 intergenic 지역이이 접근을 사용 하 여 지정할 수 있습니다. 따라서, 타겟된 시퀀싱 때 이미 관심의 질병과 연관 되기 위하여 알려진 후보 유전자의 기초는 우수한 접근 될 수 있습니다. 게놈의 특정 영역을 대상으로 클라우드 또는 임상 해석에서 끌 수 있는 불필요 한과 무관 한 유전 변이의 제거에 대 한 수 있습니다. WGS와 웨스 높은-품질 데이터의 대량 생산, 데이터의 양을 압도 될 수 있습니다. 뿐만 아니라이 많은 양의 데이터 계산 집중적인 생물 정보학 분석을 필요로 하지만 데이터 저장 자주 문제7를 제시할 수 있습니다. 데이터 저장소의이 도전 또한 WGS와 웨스, 종종 처음으로 간주 되지 않습니다 시퀀싱 비용을 계산할 때 추가 비용을 추가 합니다. 또한, 그것 감소는 비록 WGS와 웨스의 비용이 상대적으로 높은 남아 있습니다. 개인의 많은 수의 연속은 필요한 때에 특히 타겟된 시퀀싱 비용 효율적인 옵션을 수 있습니다.
이니셔티브 (ONDRI)에 위치한는 온타리오 신경 퇴행 성 질환 연구는 멀티 플랫폼, 성 폭, 관측 코 호트 연구 특성화 등 5 신경 퇴행 성 질환: 1) 알 츠 하이 머 질환과 경미한 인지 장애, 2) 루 경화 증, 3) frontotemporal 치 매, 4) 파 킨 슨 병, 및 5) 혈관 인지 장애8. ONDRI 유전체학 네가 코이 호트의 기준선 특성화의 일환으로 이러한 phenotypically 유전자 유형이 다른 질병의 자주 할인, 아직 매우 중요 한 유전 풍경 명료를 목표로. 신경 퇴행 성 질환은 이렇게 특히 타겟 시퀀싱 NGS 방법론에 대 한 적절 한 후보자 이다.
우리는 사용자 정의-설계 대상된 NGS 패널, ONDRISeq, 시퀀싱 528 참가자 이전 관심의 5 개 질환에 연관 된 80 유전자의 단백질 코딩 영역에 대 한 ONDRI에 참여 하. 이 방법론, 우리 집중 하 고 효율적인 방식으로 높은-품질 NGS 데이터를 수 있습니다. 디자인 및 여러 색인 연구와 ONDRISeq 패널의 유효성 검사 이전 설명 하고있다, ONDRISeq 패널 했다 소설, 패널 유효성 검사에 사용 되는 216 사례의 72.2%에서 가능한 임상 의미의 희소 한 이체를 식별할 수 9. 비록 NGS 기술은 급속 하 게 전진 했다 및 현저 하 게 최근 몇 년 동안, 많은 연구원은 얼굴 도전 가능한, 주석이 달린 변종10의 목록으로 원시 데이터를 처리할 때. 또한, 변형 해석 수 복잡 한, 특히 많은 희귀 또는 소설11에 직면.
여기, 단계별 방식으로, 타겟된 NGS의 방법론 및 관련된 생물 정보학 워크플로 resequencing, 변종 전화와 variant는 ONDRISeq를 사용 하 여 주석을 예를 들어 공부에 필요한 설명 합니다. NGS 데이터 생성 후 원시 시퀀싱 파일 변형을 정확 하 게 호출 하려면 인간의 참조 게놈에 정렬 합니다. 변종 후속 변형 변호사 수행 하려면 하 고 다음 주석을 첨부 합니다. 우리는 또한 우리의 구현의 의료 유전학 표준 및 지침 정확 하 게 분류 변형 pathogenicity의 미국 대학 설명할 것입니다.
프로토콜
ONDRI의 목적을 위해 윤리 프로토콜 및 동의 가져온 노인 케어 (토론토, 온타리오, 캐나다)를 위한 Baycrest 센터에서 연구 윤리 보드에 따라 중독과 정신 건강 (토론토, 온타리오, 캐나다)를 위한 센터 엘리자베스 Bruyère 병원 (오타와, 온타리오, 캐나다); 해밀턴 종합 병원 (해밀턴, 온타리오, 캐나다); 런던 건강 과학 센터 (런던, 온타리오, 캐나다); 맥 매스 터 (해밀턴, 온타리오, 캐나다); 오타와 병원 (오타와, 온타리오, 캐나다); Parkwood 병원 (런던, 온타리오, 캐나다); 세인트 마이클 병원 (토론토, 온타리오, 캐나다); Sunnybrook 건강 과학 센터 (토론토, 온타리오, 캐나다); 그리고 대학 건강 네트워크 토론토 웨스턴 병원 (토론토, 온타리오, 캐나다).
1. DNA 격리 인간의 혈액 샘플에서
-
적절 한 윤리 프로토콜 및 동의 시퀀싱 참가자에서 샘플을 수집 합니다.
- 높은 품질의 DNA를, 추출의 목적에 대 한 혈액 샘플을 그립니다.
참고: DNA 또한 추출할 수 있습니다 타 액 이나 볼 셀에서 적절 한 DNA 추출 키트 사용. - DNA의 높은 수익률을 얻을 수 혈액에서 추출 3 4 mL K2 EDTA 튜브에 샘플 수집, 제공 하 고 총 볼륨의 샘플 ~ 12 ml.
- 백혈구의 중간 단계 및 적혈구의 아래 단계 플라즈마, 얇은의 위 단계에 750 x g 분수를 20 분에 대 한 혈액 샘플을 원심.
- 높은 품질의 DNA를, 추출의 목적에 대 한 혈액 샘플을 그립니다.
- 처분할 수 있는 이동 피 펫으로 샘플의 상단을 pipetting으로 혈액 샘플에서 플라스마를 제거 합니다. 적절 하 게 플라즈마를 삭제 하거나 미래 생 화 확 적인 분석-80 ° C에서 스토리지에 대 한 여러 500 µ L aliquots에 분배 합니다. 각 샘플에 대 한 새로운, 살 균 피 펫 사용 됩니다 확인 하십시오.
- 혈액 추출 키트12 (테이블의 재료) 제조업체의 지침에 따라 혈액 샘플에서 DNA를 추출 합니다.
참고: 위에서 설명한 볼륨의 샘플을 가져온 경우 ~ 3 mL 백혈구의 DNA 추출에 사용 하 여 얻을 수 있습니다. - 제조업체의 지침에 따라 ng / µ L 전체 스펙트럼 분 광 광도 계13 (자료 테이블)를 사용 하 여 초기 DNA 농도 측정 합니다.
- 2 단계 직접 진행. 또는, 4 ° c.에 DNA를 저장
2. 시퀀싱 라이브러리 준비
-
5.0 ± 1.0 ng / µ L의 최종 농도를 3 일에 걸쳐 DNA 샘플에 직렬 희석을 수행 합니다.
- 이온된 수 1 M Tris 버퍼 pH 8.5 ~ 10 µ M을 희석.
참고: 볼륨 희석 후속 단계에 희석 될 필요가 있을 것 이다 DNA 샘플 수에 따라 달라 집니다. - DNA 희석 단계 1.4 후 직접 수행 하는 경우 다음 단계를 진행 합니다. 그렇지 않으면 같은 날에 측정 DNA 농도 단계 1.4에서에서 완료 했다.
- ~ 10 ng / µ L 10 µ M Tris 버퍼 pH 8.5를 사용 하 여 DNA의 40 µ L를 희석 농도 측정에 기반, 및 4 ° c.에 밤새 앉아 샘플 허용
- 제조업체의 지침에 따라 fluorometer14 (자료 테이블), DNA의 정량화에 대 한 적절 한 DNA 농도 측정 합니다.
참고: 샘플의 농도 이어야 한다 > 이전에 사용 하는 분 광 광도 계의 낮은 감도 때문에 µ 10 ng/L. - 측정 농도, 및에 따라 10 ng / µ L 10 µ M Tris 버퍼 pH 8.5를 사용 하 여 DNA의 20 µ L를 희석 4 ° c.에 밤새 앉아 샘플 허용
- 제조업체의 지침에 따라 fluorometer14, DNA 농도 측정 합니다.
- 5 ng / µ L 10 µ M Tris HCl pH 8.5를 사용 하 여 DNA의 10 µ L를 희석 농도 측정에 기반, 및 4 ° c.에 밤새 앉아 샘플 허용
- 이온된 수 1 M Tris 버퍼 pH 8.5 ~ 10 µ M을 희석.
-
연속 타겟된 NGS 패널의 적절 한 대상 농축 키트15 (자료 테이블) 제조업체의 지침에 따라 라이브러리를 준비 합니다. 농축 키트 NGS 플랫폼 사용에 대 한 적절 한 있는지 확인 합니다.
- 따라 제조 업체의 지침16 는 plexity 및 라이브러리의 풀링.
참고: ONDRISeq에 대 한 라이브러리의 12 DNA 샘플, 2의 세트에 풀링된 구성 되며 NGS 데스크톱 악기 (자료 테이블)에서 실행. 단일 반응에서 실행할 수 있는 샘플 수는 시퀀싱 장비 및 사용 하는 플랫폼에 따라 달라 집니다. - 높은 품질 시퀀싱 데이터를 위해 tagmentation, 대상 농축 키트15의 제조 업체의 지침에 설명 된 다음 DNA 도서관 품질을 유효성을 선택적 단계를 수행 합니다.
- 각 라이브러리는 라이브러리의 품질을 보장 하기 위해 3 중에서 분석.
- 풀링 라이브러리, 제조업체의 지침에 따라 fluorometer14, DNA 농도 측정 합니다. 이 농도 사용 하 여 사용 되 고 대상 농축 키트에서 권장 하는 아데닌 비율을 얻기 위해 풀 각 DNA 라이브러리의 볼륨을 결정.
- 따라 제조 업체의 지침16 는 plexity 및 라이브러리의 풀링.
3. 차세대 시퀀싱
-
NGS 데스크톱 악기의 시 약 키트 제조 업체의 지침17,18 (자료 테이블)에 따라 라이브러리를 시퀀스.
- 제조 업체의 지침18 적절 한 NGS 기술 소프트웨어를 사용 (테이블의 자료), NGS 데스크톱 악기의 워크플로로 가져올 것이 따르면 샘플 시트를 준비 합니다.
참고: ONDRISeq의 목적에 대 한 응용 프로그램 옵션 선택은 '다른'만 FASTQ 파일 요청 (그림 1). 이후 단계는 정렬 및 품질 매개 변수 전체를 사용자 지정할 수 있도록 이러한 FASTQ 파일을 처리 합니다. 그러나, 타겟된 시퀀싱을 선택한 경우 일부 NGS 악기 VCF 파일 자체에 시퀀싱 데이터를 처리할 수 있습니다. 제조업체의 지침18 옵션의 전체 선택을 위해 상담 될 수 있습니다. - 클라우드 기반 컴퓨팅 환경19 (테이블의 재료)를 사용 하 여 로그인 실행 시퀀스를 설정할 때. NGS 데스크톱 악기 홈 페이지에 "시퀀싱"를 클릭 하면 이렇게.
- 라이브러리 변성18 제조업체의 지침에 따라, 다음 fluorometer14와 DNA 도서관 농도 측정 합니다.
- 제조업체의 지침에 따라 적절 한 자동된 전기 영동 시스템 및 DNA 품질 분석 키트20 (테이블의 재료)를 사용 하 여 DNA 도서관 품질을 확인 합니다.
- Nm ng / µ L에서 DNA 농도 변환 하려면 다음 수식16
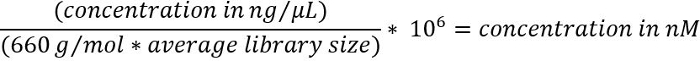
참고: 평균 라이브러리 크기 사용 되 고 대상 농축 키트에 적용 됩니다 그리고 전기 이동 법 추적 단계 3.1.4에서에서 관찰에서 얻을 수 있습니다. - 시퀀싱 라이브러리 6-20의 최종 농도를 희석, 오후, 그리고 제조 업체의 지침21에 따르면 600 μ의 볼륨.
참고: 필요한 정확한 농도 시퀀싱 키트 사용에 따라 달라 집니다. 적절 한 로드 농도 결정 하기 위해 농축 키트 제조 업체를 참조 하십시오. - 희석, 변성, 그리고 긍정적인 통제 시퀀싱 라이브러리21, 제조업체의 지침에 따라 포함.
- Length(s), 읽기의 모든 시퀀싱 실행, DNA 도서관 농도 로드 (오후), 긍정적인 컨트롤 추가의 비율, 시 약 카트리지 바코드, 단계 3.1.1, 인덱스 읽기, 사용, 농축 키트 수에서에서 선택한 응용 프로그램을 포함 하는 로그를 유지 하 고는 샘플 시트 이름입니다.
참고: NGS 데스크톱 악기의 실행된 시간 악기, 농축 키트에 의존 되며 길이 선택 (4-56 h22이 실험 사용 하는 시퀀서에 대 한)를 읽을.
- 제조 업체의 지침18 적절 한 NGS 기술 소프트웨어를 사용 (테이블의 자료), NGS 데스크톱 악기의 워크플로로 가져올 것이 따르면 샘플 시트를 준비 합니다.
- 시퀀싱 실행 완료 되 면 폴더에 액세스할 합니다 "실행", NGS 데스크톱 악기 홈 페이지를 탐색 하 고 "파일 관리"를 클릭 하 여 모든 출력을 포함 하. 이후 액세스를 위해 로컬 드라이브에 파일을 이동 합니다. 컴퓨터에서 별도 옵션에 대 한 탐색 패널에서 "실행"을 선택 하 여 내 클라우드 기반 컴퓨팅 환경19 파일을 찾을. 적절 한 시퀀싱 실행 요약 페이지로 이동 하려면 실행을 선택 합니다. 데이터를 얻기 위해 구름에서 "다운로드"를 선택 합니다. 나타나는 대화 상자에서 FASTQ 파일을 다운로드 하 고 "다운로드"를 클릭 파일 형식으로 선택 합니다.
-
클라우드 기반 컴퓨팅 환경19,23의 실행 요약 페이지에서 시퀀싱 컴퓨팅 환경에서 생산 하는 다양 한 수치와 실행의 품질 분석을 "차트"로 이동 합니다. 제조업체의 지침23 생산 각 그림에 대 한 자세한를 참조 하십시오.
- 차트 실행 페이지에서 "데이터에 의해 주기"를 표시 하는 그림을 찾아. 차트에서 "강도"를 선택 하 고 채널에서 "모든 채널"을 선택 합니다. 이 신호 강도 플롯 생산 시퀀싱 실행 같은 농축 키트와 NGS 데스크톱 악기 과거에서 제작한 비슷한 인지 확인 합니다.
참고:이 모든 150 사이클에 걸쳐 각 기초에 의해 표시 된 강도의 비율을 반영 한다. 그림 같은 패널의 과거 시퀀싱 실행에 비해 해야 합니다 이유는 사용 하 고, 농축 키트에 따라 달라질 수 있습니다. - 페이지의 오른쪽에 있는 인덱싱 품질 관리 (QC) 히스토그램을 찾이 실행된 탐색 패널에서 "인덱싱 QC" 탭을 선택 합니다. % 읽고 확인 (PF)의 비교적 균일 한 분포 모든 샘플에서 관찰 됩니다 확인 하십시오.
참고: 모든 샘플 있다면 훨씬 낮은 %를 읽고 식별 (PF) 샘플의 나머지 부분 보다, note 시퀀싱 데이터의 품질 영향을 받을 수 있습니다.
- 차트 실행 페이지에서 "데이터에 의해 주기"를 표시 하는 그림을 찾아. 차트에서 "강도"를 선택 하 고 채널에서 "모든 채널"을 선택 합니다. 이 신호 강도 플롯 생산 시퀀싱 실행 같은 농축 키트와 NGS 데스크톱 악기 과거에서 제작한 비슷한 인지 확인 합니다.
-
클라우드 기반 컴퓨팅 환경의 실행 요약 페이지에서 실행된 탐색 패널에서 "통계"를 클릭 하 여 품질 기준을 이동 합니다.
참고: 메트릭 컷-오프 사용 되 고 시퀀싱 플랫폼 및 농축 장비에 따라 달라 집니다. 많은 통계를 기반으로 제조 업체의 지침23, 다음 단계는 품질 관리에 대 한 좋습니다 3 개의 강조 활용할 수 있다.- "밀도 (K/m M2)"에서 클러스터 밀도 사용 되 고 농축 키트에서 권장 하는 범위 내에서 하는 것을 확인 (이 경우 1200-1400 K/m m2).
- 총 "% ≥Q30" 값이 ≥85%, 연속 읽기의 품질을 반영 인지 확인.
참고: 경우 85%의이 임계값 보다 낮은, note 시퀀싱의 품질을 손상 될 수 있습니다. - "정렬 (%)" 확인 값 시퀀싱 실행에 포함 된 긍정적인 컨트롤의 %와 비슷합니다.
참고:이 역할을 긍정적인 통제의 측정 총 읽습니다만이 백분율 긍정적인 제어 게놈에 정렬에 발견 되도록 합니다. 1% 긍정적인 제어를 사용 하는 경우 정렬 (%) ~ 1 ~ 5% 될 것 이라고 예상 될 것 이다.
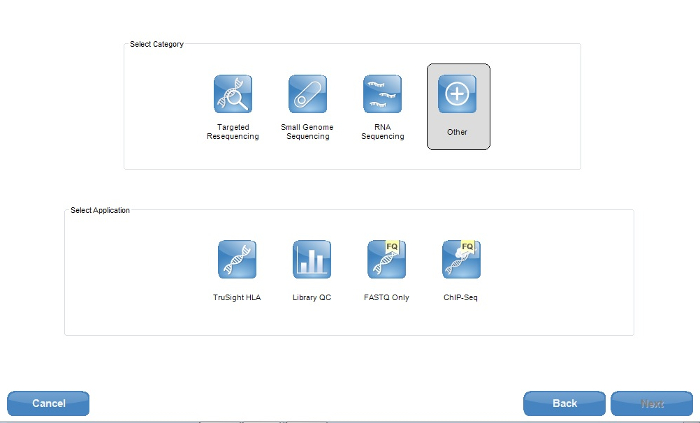
그림 1: NGS 기술 소프트웨어 (자료 테이블)의 스크린샷 샘플 시트 작성자 응용 프로그램 옵션. ONDRISeq에서는 FASTQ 유일한 응용 프로그램이 사용 됩니다. 그러나, 사용자 제작, VCF 파일 같은 다른 파일을 같은 것 이라고 하는 경우 대상된 resequencing 범주 내에서 응용 프로그램 사용 되는 것이 좋습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
4. resequencing 및 변형 호출
- 데이터 사전 처리에 대 한 인간의 참조 게놈을 원시 FASTQ 파일을 정렬 하 고 변종 (자료 테이블) 전화를 적절 한 소프트웨어를 선택 합니다.
- 가져오기 FASTQ 시퀀싱 사전 소프트웨어를 처리 하는 데이터를 읽습니다.
참고: ONDRISeq의 목적을 위해 24 샘플의 단일 시퀀싱 실행에서 생성 된 48 FASTQ 파일은 가져올 및 소프트웨어를 통해 처리. 한 번에 처리 하는 샘플 수는 연구자의 요구와 NGS 패널의 크기에 따라 달라질 수 있습니다.- "탐색 영역" 내에서 마우스 오른쪽 단추로 클릭 하 고 "새 폴더"를 선택 합니다. 이름 폴더는 시퀀싱으로 선명도 실행을 수행 했다.
- 상단에 도구 모음에서 "가져오기"를 선택 합니다. 드롭다운 목록에서 목록을 표시 하는 시퀀싱 플랫폼의 시퀀싱을 수행 하는 플랫폼을 선택 했다.
참고: ONDRISeq의 목적을 위해 "Illumina" 선택 됩니다. 그러나, 다른 시퀀싱 플랫폼 상담을 사용 하는 경우 FASTQ 가져오기의 나머지 부분에 대 한 제조업체의 지침24단계. - 대화 상자에서 탐색 하 고 선택은 FASTQ 파일은 시퀀싱에서 실행 하는 처리 되. 여러 서버 컴퓨터를 사용 하는 경우 가져올 파일에 저장 되 고 로컬 드라이브에서 가져온 확인 하십시오.
- "일반 옵션" 대화 상자에서 시퀀싱 짝된 끝 화학을 사용 하는 경우 "쌍 읽기" 옆에 상자를 클릭 합니다.
참고:이 경우에, 또한 이어야 한다-각 샘플에 대 한 가져온 두 FASTQ 샘플 한 앞으로 1 개의 반전. - 대화 상자의 정보를 읽고 쌍에서 FASTQ 파일은 파일 목록에서 역방향 읽기 전에 나타납니다 앞으로 읽으면 "쌍 간 (역회전)"를 선택 합니다. 파일은 반대 순서로 표시, "메이 페어 (역으로)"을 선택 합니다. 1 짝된 읽기 최소 거리 및 최대 거리 1000, 샘플 시퀀스 내에서 소규모 구조 재배열의 검출에 대 한 허용 하도록 설정 합니다.
- "제거 읽기 실패", 선택 "Illumina 옵션" 대화 상자에서 연속 실패 읽기 제거 하. NGS 데스크톱 악기 드 다중화 하는 경우 FASTQ 파일을 내보내기 전에 데이터는 "MiSeq 멀티플렉싱" 상자를 선택 하지 마십시오.
- "질 점수" 드롭다운 목록에서 시퀀싱에 대 한 활용은 NGS 파이프라인을 선택 합니다. 대화 상자 아래쪽에 "다음"을 선택 합니다.
참고: 사용 하는 파이프라인 FASTQ 파일 품질 점수 형식의 영향을 줍니다. 선택 하는 파이프라인에 대 한 자세한 내용은 제조업체의 지침24를 참조 하십시오. - 새로운 대화 상자, 선택 "저장" 및 "그들의 자신의 개인 폴더에 각 샘플의 FASTQ 파일을 넣어 목욕 단위당 하위 폴더를 만듭니다. 대화 상자 아래쪽에 "다음"을 선택 합니다.
- 새 대화 상자에서 단계 4.2.1에서에서 만든 폴더를 선택 합니다. 이것은 FASTQ 파일을 가져올 것 이다입니다. 대화 상자 아래쪽에 "완료"를 선택 하 고 FASTQ 파일을 가져올 때까지 기다립니다. 파일 가져오기의 상태를 보고 "프로세스" 탭을 클릭 합니다.
- Resequencing 및 변형 전화, 제조업체의 지침에 따라 수행을 하는 워크플로 디자인 합니다.
참고:이 워크플로 연구원의 요구에 따라 달라질 수 있습니다 있지만 다음 단계 포함 무엇 ONDRISeq (그림 2)의 목적을 위해 포함 됩니다. 이 워크플로 단계 다른 NGS resequencing 및 적절 한 변형 호출 소프트웨어에 적용할 수 있습니다. ONDRI의 목적을 위해 처리 하는 모든 생물 정보학 데이터 처리 및 분석의 일관성에 대 한 인간의 참조 게놈 GRCH37/hg19에 관하여 수행 됩니다.- 참조 게놈을 시퀀싱 읽기를 매핑하십시오.
- 를 구성할 때 모든 생물 정보학 단계에 사용 되는 동일한 참조 게놈 다는 것을 보장, 참조 게놈을 선택 합니다.
- 마스크 모드에서 드롭-다운 목록 "마스크" 아무 영역 참조 시퀀스의 마스크는 선택.
- 기본 매핑 소프트웨어에 의해 지정 된 옵션을 사용 합니다. 연구의 목적에 따라 제조 업체의 지침24 이 허용 되었는지 확인 하려면 검토 합니다.
- 모든 읽기 오류 매핑, 특히 삽입-삭제 변형 주변을 해결 하기 위해 인간의 참조 게놈을 워크플로 지방 재배치에 포함 합니다.
- 소프트웨어에 의해 지정 된 기본 로컬 재배치 옵션을 사용 합니다. 연구의 목적에 따라 제조 업체의 지침24 이 허용 되었는지 확인 하려면 검토 합니다.
- 가양성25생산할 수 있는 PCR 증폭 바이어스의 효과 줄이기 위해 NGS 프로토콜 내에서 PCR에 의해 생산의 매핑된 읽기 중복된 개를 제거 합니다.
- "최대의 대표 소수 시퀀스 (%)", 연구의 필요에 따라 설정 합니다.
참고: 관대 한 설정, ONDRISeq의 목적을 위해 사용 되는 5%; 그러나, 소프트웨어의 기본 설정은 더 엄격한 20%입니다. 두 개의 읽기 매우 비슷합니다,이 설정을 경우 적은 읽기 카운트 시퀀스 PCR 증폭 바이어스에서 시퀀싱 오류 간주 되어야 결정 합니다. 따라서, 설정 5%, 소수 읽을 수 대부분의 ≤ 5% 읽기 읽기 대부분 동일한 것으로 수정 될 수 있어야.
- "최대의 대표 소수 시퀀스 (%)", 연구의 필요에 따라 설정 합니다.
- 4.3.3 단계에서 생성 된 읽기 트랙에서 범위 요약 텍스트 파일의 형태로 대상 지역에 대 한 통계를 내보냅니다. 일반적인 선수 및 설정에서 깨진된 쌍을 무시 합니다. 이러한 파일에 대 한 로컬 드라이브에 대상을 선택 합니다.
- 4.3.3 단계에서 생성 된 읽기 트랙에서 각 샘플에 대 한 이진 순서 정렬 지도 (BAM) 파일을 내보냅니다. 이 분석을 나중에 필요한 경우 시퀀스 정렬 데이터를 포함 합니다. 이러한 파일에 대 한 로컬 드라이브에 대상을 선택 합니다.
- 호출 시퀀스 내에서 변형 변형 검출의 방법을 선택 하십시오.
참고: 가정 ploidy의 예제에 대 한 만들 수 있습니다 때 ONDRISeq의 목적을 위해 사용 되는 고정된 ploidy 변형 탐지 알고리즘 사용 될 것이 좋습니다. 이 가정은 만들 수 없습니다, 만약 연구의 목적에 대 한 최상의 알고리즘을 확인 하려면 제조업체의 지침24 를 참조 하십시오.- 구성, 고정된 ploidy에서 variant 매개 변수 옵션 설정 합니다 ploidy 샘플 유기 체에 적합. "필요한 변형 확률", 또는 variant 90.0%에서 유지 될 그것의 순서를 따라에서 제대로 호출 된 확률을 설정 합니다.
- 일반 필터에 대 한 설정을 권장 하는 다음을 사용: "최소"의 범위 10 x, "최소 개수" 2, 20%, "무시" 쌍을 깨진"," 최소 읽기 주파수""읽기"에 따라 일반적인 일치 무시 하 고"최소 길이 읽기"20.
참고: 이러한 매개 변수는 ONDRISeq의 목적에 근거한 다. 그들은 완료 되 고 연구에 적합 한 보장 하기 위해 제조 업체의 지침24 를 참조 하십시오. - 다음 권장 잡음 필터에 대 한 설정을 사용 하 여: "기본 품질 필터" "이웃 반경" 품질 평가 점수 5, "최소 중앙 품질의" 20의 점수 및 "최소 이웃 품질" 매핑 점수 15;의 매핑 매핑 5.0%;의 "읽기 방향 필터" 그리고 1.0% 의미의 "상대 읽기 방향 필터".
참고: 이러한 매개 변수는 ONDRISeq의 목적에 근거한 다. 그들은 완료 되 고 연구에 적합 한 보장 하기 위해 제조 업체의 지침24 를 참조 하십시오.
- 대상된 패널의 대상 영역 지정 된 브라우저 확장 데이터 (침대) 파일에만 변종 될 대상 NGS 패널 선정 게놈 영역에서 발생을 허용 그들의 중복에 따라 호출 된 변종 필터링 유지.
참고: 침대 파일 널리 사용 되 고는, 패널은 커버 할 수 있는 게놈의 지역에 따라 대상된 NGS 패널에 있을 것입니다. - 4.3.7 단계에서 생성 하는 변형 트랙에서 variant 호출 형식 (VCF) 파일에서 다른 보고서를 내보냅니다. 이러한 파일에 대 한 로컬 드라이브에 대상을 선택 합니다.
- 저장 하 고 소프트웨어의 "도구 상자"에서 사용할 수 있도록 제조 업체의 지침24에 따라 워크플로 설치 합니다. 그것은 미래에 분명 어떤 NGS 패널 적합 되도록 워크플로 이름이 있는지 확인 합니다.
- 설치 중 "내보내기 참조 데이터" 옵션 대화 상자에서 "번들"에 모든 옵션을 설정 합니다.
- 설치 중 "설치 위치" 옵션 대화 상자에서 "로컬 컴퓨터에 워크플로 설치"를 클릭 합니다.
- 참조 게놈을 시퀀싱 읽기를 매핑하십시오.
- 제조 업체의 지침24에 따라 사용자 지정된 생물 정보학 워크플로 단계 4.3에서에서 설계를 통해 가져온된 FASTQ 연속 읽기 파일을 실행 합니다.
- 소프트웨어의 "도구 상자"에서 4.3 단계에서 디자인 된 워크플로 식별 하 고 그것을 두 번 클릭 합니다.
- 대화 상자가 나타나면 내 영역 내에 "탐색" 4.2 단계에서 가져온 FASTQ 파일의 폴더를 찾습니다. 모든 폴더 "탐색 영역" 내에서 그들을 선택 하 여 강조 표시 한 다음 "일괄 처리" 옆에 있는 상자를 클릭 합니다. "요소 선택"으로 파일을 이동 하려면 오른쪽 화살표를 사용 합니다. 대화 상자 아래쪽에 "다음" 클릭 합니다.
- 대화 상자에서 "배치 개요" 검토 올바른 FASTQ 파일을 선택 하 고 다음 "다음"을 클릭 합니다.
- 검토 단계 4.3에서에서 워크플로 디자인할 때 워크플로의 올바른 파일을 확인 및 내보내기 위치 대화 상자에서 다음 단계 선정 됐다: "지도 읽고 참조로"; 중복 매핑된 읽기 제거 "; "대상 지역에 대 한 통계를 생성"; "BAM" 수출; "내보내기 탭 구분 텍스트"; "필터 기준 중복"; 및 "VCF 내보내기"
- 대화 상자에 마지막 단계에서 옵션을 선택-"결과 처리"-"저장 입력된 폴더에". 대화 상자 아래쪽에 "완료"를 클릭 합니다.
참고: 각 샘플에 대 한 파일 생성이 즉 사전 소프트웨어를 처리 하는 데이터 내에서 FASTQ 파일을 저장 하는 동일한 폴더에 배치 됩니다.
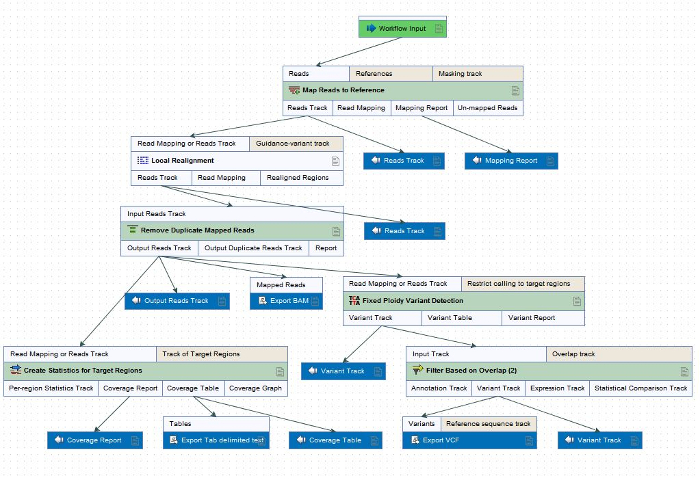
그림 2: resequencing와 FASTQ의 변형 호출에 대 한 워크플로 데이터 내에 소프트웨어 (자료 테이블) ONDRISeq의 목적에 맞게 전처리 파일. 워크플로의 단계는 다른 NGS resequencing에 적용할 수 있는 하 고 연구자의 요구에 따라 변형 호출 소프트웨어. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
5. 변형 주석
-
다운로드 하 고 수행 하는 각 샘플의 VCF 파일에 따라 변형 주석 주석 변형 (ANNOVAR)26 스크립트를 사용자 지정 합니다.
- 다음 데이터베이스에서 주석으로 포함 되도록 ANNOVAR 다운로드: 1) RefSeq27 (8 월 2015 업데이트); 2) dbSNP13828 (9 월 2014 업데이트); 3) Exome 집계 컨소시엄29 (ExAC, 버전 0.3 11 월 2015 업데이트); 4)는 국립 심장, 폐, 혈액 연구소 Exome 시퀀싱 프로젝트 유럽 코 호트30 (ESP, 3 월 2015 업데이트); 5) 1000 게놈 프로젝트 유럽 일대31 (1KGP, 8 월 2015 업데이트); 6) ClinVar32 (3 월 2016 업데이트); 그리고 7) 주석 결합 종속 고갈33 (CADD), 관용34 에서 유제품 정렬 (구별), PolyPhen-235.
참고: 게놈 조정 하 고 인간 게놈 빌드 GRCh37/hg19 ANNOVAR 참조 하는 모든 데이터베이스 참조. 또한, 나열 된 데이터베이스 버전은 사용 가능한 가장 최신 버전을 사용 하 여 데이터베이스를 다운로드 ONDRISeq의 목적을 위해 사용 되. - 원하는 경우, 사용자 지정 사용 하 여 주석 변종의 감소 편집 주석된 이체의 전체 목록을 출력 하는 ANNOVAR--필터 동작26.
참고: 감소 목록 수 사용자 지정할 수는 연구원의 요구에 따라. ONDRISeq의 목적에 대 한 주석된 이체의 감소 된 목록 포함 되지 않습니다 가까운 엑손에서 15 기지 보다 더 발생 하는 변형 또는 사소한 대립 유전자 주파수 (MAF) 어떤 이체 > 3 %3 데이터베이스에서: 1) ExAC; 2) ESP; 그리고 3) 1KGP. 이 단계는 매우 것이 좋습니다. - 원하는 경우 단일 연구원26의 요구에 따라 특정 대립 유전자 외침 ANNOVAR를 사용자 지정 합니다.
참고: ONDRISeq의 목적을 위해 ANNOVAR 평가 APOE 위험 대립 유전자 rs429358에 대 한 연속 호출 (C > T):p.C130R 및 rs7412 (C >는의 6 개의 가능한은 전반적인 APOE 유전자 형을 출력 하기 위해 T):p.R176C 조합, 포함: 1) e 2/e 2; 2) E3/E2; 3) E4/E2; 4) E3/E3; 5) E4/E3; 6) E 4/E4입니다. 이들 6 가능한 APOE genotypes e 4/E436늦은 개시 Alzheimer의 질병 개발 하기 위해 가장 일반적으로 허용된 유전 위험 요소 이다.
- 다음 데이터베이스에서 주석으로 포함 되도록 ANNOVAR 다운로드: 1) RefSeq27 (8 월 2015 업데이트); 2) dbSNP13828 (9 월 2014 업데이트); 3) Exome 집계 컨소시엄29 (ExAC, 버전 0.3 11 월 2015 업데이트); 4)는 국립 심장, 폐, 혈액 연구소 Exome 시퀀싱 프로젝트 유럽 코 호트30 (ESP, 3 월 2015 업데이트); 5) 1000 게놈 프로젝트 유럽 일대31 (1KGP, 8 월 2015 업데이트); 6) ClinVar32 (3 월 2016 업데이트); 그리고 7) 주석 결합 종속 고갈33 (CADD), 관용34 에서 유제품 정렬 (구별), PolyPhen-235.
-
질환 돌연변이 데이터베이스 (테이블의 재료) 변종 이전 질병, 합리적인 증거에 연관 된 경우 확인 하려면 쿼리 합니다. 새로운 변종으로 이전 보고 하지 않은 모든 이체를 고려 하십시오.
- ClinVar에서 ANNOVAR 주석을 평가, 어떤 분류로 가능성이 병원 성 또는 병원 성 질병 관련 된 변종 포함 되도록.
- 변종 철에 예측을 통해 접합 과정 도구 접합 기반 분석의 변종37 (SPANR)와 인간 접합 파인더38 (HSF, 버전 3.0).
- 샘플의 많은 수를 처리 하는 경우를 비교 하 여 어떤 변종 다양 한 샘플에 의해 공유 됩니다 확인 하려면 각 샘플 내에서 변형 전화. 이렇게 수동으로 또는 사용자 정의-설계 스크립트, 가능한 시퀀싱 유물과 오염 이벤트의 검출에 대 한.
참고: ONDRI의 목적을 위해 사용자 지정 스크립트를 서로 비교 하 여 ANNOVAR 출력 파일 주석을 사용 됩니다. 스크립트 주석, 변형, 같은 변종, 그렇지 않으면 variant의 역사 연구 일대에 불리를 은닉 하는 다른 샘플의 제목 id 당 통합. - 변종 미국 대학 의료 유전학 (ACMG) Pathogenicity 지침39, 할당 각 이체는 다음 중 하나로 분류에 따라 분류: 1) 병원 성; 2) 가능성이 병원 성; 3) 변종 불확 실한 의미; 4) 가능성이 양성; 또는 5) 양성.
참고: ONDRI의 목적을 위해 사내 설계 된 파이썬 스크립트를 반자동으로 ACMG 분류를 수행 하 사용 됩니다. 이 연구를 위해 사용 되지 않지만 InterVar40 유사한 방식으로 활용 될 수 있는 유사 하 게 설계 된 도구입니다. - 생어 시퀀싱 시퀀싱 보도 어떤 이체 < 30 x 및/또는 확인 된 유효성을 연구 코 호트의 > 10%에는 그들이 하지 유물41시퀀싱 변종.
결과
여기에 설명 된 방법론은 ONDRI에 등록 된 개인에서 528 참가자 DNA 샘플에 적용 했다. 샘플 실행 당 24 샘플 22 실행에서 ONDRISeq 패널에서 실행 했다. 전반적으로, 시퀀싱 데이터 78 ± 13 x의 의미 샘플 범위와 높은 품질의 수를 결정 하 고 모든 개별 실행 표현 의미 샘플 범위 > 30 x. 또한, 평균에, 모든 대상 영역의 94% 덮여 있었다 20 x (표 1).
읽기의 95.6% 참조 시퀀스 및 모든 ONDRISeq에 매핑된 했다 의미 했다 실행 > 90% 읽기의 매핑 (표 1). 매핑된 읽기 92.0% 했다 점수 ≥Q30, 단 하나 실행 데 Phred < 매핑된 읽기 품질 통계 회의의 80%. 그러나,이 실행 여전히 79 x의 의미 범위를 표시 하 고 대상 영역 했다의 93% 적용 적어도 20 배.
| 매개 변수 | 평균 (±sd) | 최고의 성능 | 가난한 성능 |
| (X 103/mm2) 클러스터 밀도 | 1424 (±269) | 1347 | 1835 |
| 총 읽기 (106) | 43.1 (±6.0) | 48.7 | 47.4 |
| 매핑된 읽기 (106) | 40.1 (±6.0) | 47.1 | 25.7 |
| 매핑된 읽기 (%) | 95.6 (±1.3) | 96.8 | 92.6 |
| Phred 품질 점수 ≥Q30 (%) | 92.0 (±6.0) | 92 | 68.3 |
| 샘플 범위 (x) | 78 (±13) | 99 | 51 |
표 1: 22에 대 한 품질 메트릭을 시퀀싱 ONDRISeq에서 실행 됩니다.
사례 연구: PD 환자에서 희소 한 이체의 Id입니다.
우리의 타겟된 NGS 워크플로 유틸리티를 보여, 우리는 68 세, 남성, 파 킨 슨 병 환자의 예 제시. DNA 샘플 NGS 데스크톱 악기 (자료 테이블) 다른 23 ONDRI 샘플 함께 ONDRISeq 패널을 사용 하 여에서 실행 되었다. 실행 표시 1,555 x 103/mm2의 클러스터 밀도. 환자의 특정 샘플 표시 76 x의 평균 범위, 대상의 93.9% 지역 적어도 20 배.
변형 전화 및 주석 사용자 지정 생물 정보학 워크플로 수행 후 환자는 exons 및 주변 250 하버 1351 변종 발견 ONDRISeq 패널에 포함 된 80 유전자의 혈압. 그러나, ANNOVAR 파이프라인 위에서 설명한 대로 변형 시퀀스 온톨로지와 농림 부, 고려 하 여 이체의 수를 줄일 수 있었습니다. 이 수동 큐레이터 (그림 3)를 받았다 7 변종의 목록 생산. 이 7 개의 이체에서 두 가능한 임상 의미를 가진 것으로 확인 되었다. 이 과정 ONDRI의 요구에 구체적 이며 일반 인구에서 비교적 드물다 고 그로 인하여 단백질에 변화를 일으키는 존재론에서 nonsynonymous 식별 하 여 이루어졌다. Variant 이전 질병와 관련 있었다, 여부는 단백질 deleteriousness의 실리콘에 예측 및 변형 ACMG pathogenicity 분류 또한이 과정에서 활용 했다.
감소 목록에서 확인 하는 첫번째 이었다 heterozygous 변종, 즉 LRRK2: c.T3939A, 넌센스 변형 p.C1313*의 결과로. LRRK2 단백질을 신 부유한 반복 키 2, GTPase와 키 니 아 제 활동42를 보유 하 고 인코딩합니다. 또한,이 유전자 내의 돌연변이43가족성 파 킨 슨 병 주요 원인 가운데 알려져 있습니다. 이 변종 LRRK2, 아미노 산 성 잔류물 1314-2, 527 잃는 그로 인하여 내 조 숙한 정지 codon를 소개 합니다. 이렇게 하면 복잡 한 단백질 (Roc)의 단백질의 Ras의 번역 Roc (오호), 및 각각, 비정형 Rho GTPase, GTP 바인딩 단백질 및 단백질 키 니 아 제, 기능에 참여 하 고 예상 했다 단백질 키 니 아 제 도메인의 C-터미널 CADD에 의해 생성 된 철에 분석에 의해 손상 될 수 (CADD Phred = 36). 이 변종 또한 드문 0.004%와 0.01 %ExAC ESP, 농림 부와 각각 이며 결 석 1000 G 데이터베이스에서. 또한, 이것은 운반은 소설 때문에 그것은 되어 이전에 설명 되지 않은 질환 돌연변이 데이터베이스 (테이블의 재료)이이 변형 시퀀스 모든 528에서 유일한 환자 이다. 변형 통화의 신뢰는 109 x의 깊은 범위에 의해 확인 되었다. 마지막으로, variant pathogenicity AMCG 표준 및 지침과 평가 고 병원 성 것으로 분류 했다.
환자는 또한 두 번째 heterozygous 변종, NR4A2실시: c.C755A, missense 변화 p.P252Q의 결과로. NR4A2, 핵 수용 체 Subfamily 4 그룹 A 회원 2, 의해 단백질은 녹음 방송 요인 dopaminergic 신경 세포44 의 생성에 관여 하 고이 유전자 내의 돌연변이 이전 파 킨 슨 병에 연관 된 질병45. 폴라 글루타민을 비-폴라 프롤린의 대체 실리콘에 예측 분석 CADD에 의해 생성에 의해 손상 될 전망 했다 (CADD Phred = 21.1), 아니라 선별 또는 PolyPhen-2에 의해 생성 된 분석. Variant은 ExAC 및 ESP를 1000 G 부재에 0.004%의 MAF와 드문 경우입니다. 변종 혈관 인지 장애 진단 ONDRI 참가자에 또한 발견 했다 하지만 질환 돌연변이 데이터베이스에서 이전에 설명 하지는. 그러나이 이체만 18 x의 범위를 했다,, 생어 시퀀싱 시퀀스 내의 그것의 타당성을 보장 하기 위해 수행 됩니다. 마지막으로, variant 불확 실한 의미 pathogenicity ACMG 표준 및 지침을 평가 하는 경우의 수를 결정 했다.
ONDRISeq 패널 및 생물 정보학 파이프라인은 각 샘플의 APOE 유전자 형을 확인할 수 있습니다. 이 환자는 E3/E3 APOE 유전자 형을 결정 했다.
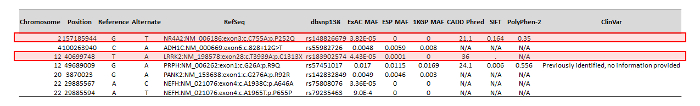
그림 3: 수동으로 큐레이터, 표시 하는 ANNOVAR에서 감소 된 출력의 예 주석 변종. 68 세, 남성, 파 킨 슨 병 환자의 사례 연구에서 감소 ANNOVAR 출력. 주석이 추가 된 이체는 그 임상 의미의 될 가능성이 가장 높은 빨간 상자에 의해 표시 된 대로 식별 하 큐레이터. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
토론
환자의 진단, 질병의 진행, 및 가능한 치료 옵션을 고려 관심이 있을 수 있습니다 변종 식별을 DNA 샘플 추출에서 경로에서 그것은 필요한 방법론의 여러 가지 특성을 인식 하는 것이 중요 시퀀싱 및 적절 한 데이터 처리. 여기에 설명 된 프로토콜 식별 잠재적인 임상 의미의 희소 한 이체 대상된 NGS 후속 bioinformatic 분석 필수의 활용의 예입니다. 특히, 우리는 접근 방식이 ONDRI 유전체학 비율 ONDRISeq 사용자 정의 설계 된 NGS 패널을 사용 하는 경우를 제시.
이러한 메서드는 특정 NGS 플랫폼 기반 개발 되었다 고 다른 시퀀싱 플랫폼 및 사용할 수 있는 대상 농축 키트는 인식 된다. 그러나, NGS 플랫폼 및 데스크톱 악기 (자료 테이블)의 초기 미국 식품 및 의약품 안전 청 (FDA) 승인46에 따라 선택 되었다. 이 권한 부여 NGS 프로토콜 선택 및 시퀀싱 읽기에 배치할 수 있는 안정성의 수행할 수 있는 고급 시퀀싱을 반영 한다.
범위의 깊이와 정확한 연속 읽기를 취득 하는 것이 매우 중요 하지만 최종 드문 변형 분석에 필요한 생물 정보학 처리 중요 하 고 계산 집중 수 있습니다. 시퀀싱 프로세스 내에서 발생할 수 있는 오류 많은 소스 때문에 강력한 생물 정보학 파이프라인 도입 될 수 있는 다양 한 부정확에 대 한 수정 해야 합니다. 그들은 매핑 프로세스, 라이브러리 준비 및 시퀀싱 유물47을 생산 하는 기술에서 PCR 증폭에 의해 도입 된 증폭 바이어스에에서 부정합에서 발생할 수 있습니다. 읽기 매핑 및 변형 호출을 수행 하는 데 사용 하는 소프트웨어에 상관 없이 지방 재배치, 중복 매핑된 읽기의 제거를 포함 하 고 이체를 호출할 때 품질 관리에 대 한 적절 한 매개 변수를 설정 이러한 오류를 줄일 수 있는 일반적인 방법 있다. 또한, 변형 전화 중 선택 매개 변수는 손11연구에 가장 적합에 따라 달라질 수 있습니다. 최소 범위와 품질 평가 점수는 변형 및 여기에 적용 된 주변 뉴클레오티드의 적절 한 특이성 및 감도 사이의 균형을 만들 선정 됐다. 3 개의 별도 유전 기법, 앞에서 설명한, 포함 한 변형 호출 색인에 따라 ONDRISeq 패널에 대 한 검증 된 이러한 매개 변수: 1) 칩 기반 유전형; 분석 결과 2) 유전자 차별; 그리고 3) 생어 시퀀싱9.
주석 및 큐레이터 정확한 변형 호출, 잠재적인 임상 의미의 파악에 따라 필수적입니다. 그것의 오픈 액세스 플랫폼으로 인해 ANNOVAR 주석 및 예비 변형 검사 또는 제거 하기 위한 훌륭한 도구입니다. 쉽게 접근할 수 있는 이상, ANNOVAR 시퀀싱 플랫폼 사용에 상관 없이 어떤 VCF 파일에 적용할 수 있습니다 그리고 사용자 정의 기반 연구26의 요구.
주석, 후 그들은 임상 의미의로 간주 해야 경우 확인 하 이체를 해석 해야 합니다. 뿐만 아니라이 과정, 복잡 한가 하지만 그것은 종종 인간의 오류 주관 하는 경향이. 이러한 이유로 ACMG pathogenicity의 모든 변종에 대 한 증거를 평가 하는 지침을 설정 했습니다. 우리는 비-동의어, 희귀 변종 기반 수동 변호사 접근, 건설은 이러한 지침에 따라 하 고 사용자 정의 설계 된 파이프라인을 통해 전달할 수 있는 각 변형을 개별적으로 평가 하 여 보호 적용 파이썬 스크립트를 지침에 따라 이체를 분류 합니다. 이 방법에서는, 각 변종 병원 성의 순위 할당 가능성이 병원 성, 불확 실한 의미, 가능성이 양성, 또는 양성, 변형 큐레이터 과정을 표준화 및 투명성을 추가할 수 있습니다. 그것은 생물 정보학 파이프라인 넘어 변형 변호사의 구체적인 연구의 요구에 따라 개별화 될 것을 인식 하는 것이 중요 하다 고 제시 하는 방법론의 범위를 넘어 따라서 했다.
여기에 제시 된 방법 ONDRI에 있지만, 관심의 헌법 질병의 많은 수를 고려할 때 설명 하는 단계 번역 될 수 있다. 유전자 협회의 수 많은 고기에 대 한 증가, 타겟된 NGS 필드에 완료 되었습니다 이전 연구에 활용할 수 있는 접근을 구동 하는 가설에 대 한 수 있습니다. 그러나, 대상된 NGS와 제시 하는 방법론에 한계가 있다. 게놈의 특정 지역에만 초점을 맞추고, 검색 분야 관심의 새로운 대립 유전자로 제한 됩니다. 따라서, 새로운 유전자 또는 그 이상의 다른 게놈 loci 밝혀질 수 있는 시퀀싱 대상에 의해 덮여 WGS와 웨스 접근, 식별 되지 것입니다. 또한 NGS 접근, 그 반복된 시퀀스48 의 높은 학위 또는 GC 콘텐츠49에서 부유한 그를 포함 하 여 정확 하 게 시퀀스에 어려울 수 있다 게놈 지역 있다. 다행히도, 타겟된 NGS를 활용 하면 시퀀싱 되 고 게놈 영역을 가진 친밀의 높은 수준의 선험적 이며 이러한 기술 도전 포즈 수 있습니다 여부. 마지막으로, 현재 NGS 데이터에서 복사 번호 이체의 탐지 표준화50아니다. 그러나, 이러한 우려에 대 한 생물 정보학 솔루션; 지평선에 있을 수 있습니다. 새로운 계산 도구 ONDRI 환자에서 이러한 추가 형태를 분석 하는 데 도움이 됩니다.
그 한계에도 불구 하 고 타겟된 NGS WGS와 웨스 대응 보다 저렴 하면서 가설 기반 접근 방식 내에서 높은-품질 데이터를 얻을 수 있다. 뿐만 아니라입니다이 방법론 적절 한 효율적이 고 감독 연구, 임상 구현의 대상된 NGS는 기 하 급수적으로 성장 하 고 있다. 이 기술은 다양 한 질병의 분자 경로 관한 많은 다른 질문에 대답을 사용 중입니다. 그것은 또한 상대적으로 낮은 비용 웨스와 WGS에 반대 하는 때에 정확한 진단 도구로 개발 되고있다. 골드 표준 생어 시퀀싱, 대상에 비해 경우에 NGS에 그것의 시간 및 비용-효율성 노르만족 수 있습니다. 이러한 이유로, 그것은 과학자 또는 받는 사람과 NGS 데이터, 예를 들어, 실험실 또는 임상 보고서에 텍스트로 전달를 사용 하 여 복잡 한 "블랙 박스" 결과 기본을 이해 하는 임상에 대 한 중요 한. 여기에 소개 하는 방법을 사용자가 생성 및 해석 NGS 데이터의 기본 프로세스 이해를 도와 야 한다.
공개
저자는 공개 없다.
감사의 말
우리는 그들의 동 및 우리의 연구 협력에 대 한 모든 ONDRI 참가자에 게 감사 하 고 싶습니다. 감사 합니다 ONDRI 수 사관 (www. ONDRI.ca/people), 우리 수 사관 (MJS), 및 경 세 위원회 ONDRI를 포함 하 여: 집행 위원회, 운영 위원회, 출판 위원회, 모집 위원회, 평가 플랫폼 및 프로젝트 관리 팀. 우리는 또한 런던 지역 게놈 센터 그들의 기술력에 대 한 감사합니다. AAD는 런던과 Middlesex 석사 대학원 연구 장학금의 알 츠 하이 사회에 의해 지원 됩니다. SMKF는 ALS 캐나다 팀 중 노엘 박사 친교에 의해 지원 됩니다.
자료
| Name | Company | Catalog Number | Comments |
| 4 mL EDTA K2 tubes | Fisher Scientific | 02-689-4 | |
| 1 M Tris Buffer | Bio Basic Canada Inc. | SD8141 | |
| Gentra Puregene Blood Kit | Qiagen | 158389 | 1,000 mL Kit. This is the blood extraction kit, referred to in step 1.3. |
| NanoDrop-1000 Spectrophotometer | Thermo Fisher Scientific | ND-2000 | Replaced by the NanoDrop-2000 Spectrophotometer. This is the full-spectrum spectrophotometer, referred to in steps 1.4 and 2.1.2. |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | This is a fluorometer appropriate for the quantification of DNA, referred to in steps 2.1.4, 2.1.6, 2.2.3, and 3.1.3. |
| Nextera Rapid Custom Capture Enrichment Kit | Illumina, Inc. | FC-140-1009 | Specifically designed for the ONDRISeq panel, sequencing the exons of 80 genes, resulting in 971,388 base pairs of sequence in paired-end reads of 150 bases in length; 288 samples per kit. This is the target enrichment kit, referred to in steps 2.2, 2.2.2, 2.2.3, 3.1.5, 3.1.6, 3.4.1, and the Discussion. |
| 2100 BioAnalyzer | Agilent Technologies | G2939BA | This is a automated electrophoresis system, referred to in step 3.1.4. |
| High Sensitivity DNA Reagent Kit | Agilent Technologies | 5067-4626 | 110 Samples per kit; This is a DNA quality analysis kit, referred to in step 3.1.4. |
| MiSeq Reagent Kit v3 | Illumina, Inc. | MS-102-3003 | 600 Cycle Kit; This is the NGS desktop instrument reagent kit, referred to in step 3.1. |
| MiSeq Personal Genome Sequencer | Illumina, Inc. | SY-410-1003 | This is a NGS desktop instrument, referred to in steps 2.2.1, 3.1, 3.1.1, 3.1.2, 3.1.8, 3.2, 4.2.6, the Representative Results, and the Discussion. |
| Experiment Manager | Illumina, Inc. | This is NGS technology software, referred to in step 3.1.1 and Figure 1. https://support.illumina.com/sequencing/sequencing_software/experiment_manager/downloads.html | |
| BaseSpace | Illumina, Inc. | SW-410-1000 | This is a cloud-based computing environment, referred to in steps 3.1.2, 3.2, 3.3, 3.3.1, 3.3.2, 3.4, 3.4.1, 3.4.2 and 3.4.3. https://basespace.illumina.com/ |
| CLC Genomics Workbench 10.1.1 | Qiagen | 832000 | Open source options for data pre-processing are also available that can model the workflow used in this protocol. This is the software used for data pre-processing, referred to throughout step 4 and in Figure 2. |
| Annotate Variation | http://annovar.openbioinformatics.org/en/latest/user-guide/download/ | ||
| RefSeq | National Center for Biotechnology Information | https://www.ncbi.nlm.nih.gov/refseq/ | |
| dbSNP138 | National Center for Biotechnology Information | https://www.ncbi.nlm.nih.gov/projects/SNP/snp_summary.cgi?view+summary=view+summary&build_id=138 | |
| Exome Aggregation Consortium | Broad Institute | http://exac.broadinstitute.org/ | |
| National Heart, Lung, and Blood Institute Exome Sequencing Project European Cohort | University of Washington and the Broad Institute | http://evs.gs.washington.edu/EVS/ | |
| ClinVar | National Center for Biotechnology Information | https://www.ncbi.nlm.nih.gov/clinvar/ | |
| Combined Annotation Dependent Depletion | University of Washington and Hudson-Alpha Institute for Biotechnology | http://cadd.gs.washington.edu/ | |
| Sorting Intolerant from Tolerant | J. Craig Venter Instutite | http://sift.jcvi.org/ | |
| PolyPhen-2 | Brigham and Women's Hospital, Harvard Medical School | http://genetics.bwh.harvard.edu/pph2/ | |
| Human Gene Mutation Database | Qiagen | 834050 | This is a disease mutation database, referred to in step 5.2 and the Representative Results. https://portal.biobase-international.com/cgi-bin/portal/login.cgi?redirect_url=/hgmd/pro/start.php |
| Splicing-based Analysis of Variants | Frey lab, University of Toronto | http://tools.genes.toronto.edu/ | |
| Human Splicing Finder | Aix Marseille Université | http://www.umd.be/HSF3/HSF.shtml | |
| Other materials | |||
| Centrifuge | |||
| Disposable transfer pipets |
참고문헌
- Metzker, M. L. Sequencing technologies - the next generation. Nat Rev Genet. 11 (1), 31-46 (2010).
- Mardis, E. R. Next-generation DNA sequencing methods. Annu Rev Genomics Hum Genet. 9, 387-402 (2008).
- Shendure, J., Ji, H. Next-generation DNA sequencing. Nat Biotechnol. 26 (10), 1135-1145 (2008).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci U S A. 74 (12), 5463-5467 (1977).
- Farhan, S. M. K., Hegele, R. A. Exome Sequencing: New Insights into Lipoprotein Disorders. Current Cardiology Reports. 16 (7), (2014).
- Choi, M., et al. Genetic diagnosis by whole exome capture and massively parallel DNA sequencing. Proc Natl Acad Sci U S A. 106 (45), 19096-19101 (2009).
- Mardis, E. R. DNA sequencing technologies: 2006-2016. Nat Protoc. 12 (2), 213-218 (2017).
- Farhan, S. M., et al. The Ontario Neurodegenerative Disease Research Initiative (ONDRI). Can J Neurol Sci. 44 (2), 196-202 (2017).
- Farhan, S. M. K., et al. The ONDRISeq panel: custom-designed next-generation sequencing of genes related to neurodegeneration. NPJ Genom Med. (16032), 1-11 (2016).
- El-Metwally, S., Hamza, T., Zakaria, M., Helmy, M. Next-generation sequence assembly: four stages of data processing and computational challenges. PLoS Comput Biol. 9 (12), e1003345 (2013).
- Yohe, S., Thyagarajan, B. Review of Clinical Next-Generation Sequencing. Arch Pathol Lab Med. , (2017).
- Qiagen. . Gentra Puregene Handbook. , (2014).
- NanoDrop Technologies, Inc. . Spectrophotometer V3.5 User's Manual. , (2007).
- Invitrogen by Life Technologies. . Qubit 2.0 Fluorometer User Manual. Vol. Q32866. , (2010).
- Illumina, Inc. . Nextera Rapid Capture Enrichment Guide. , (2016).
- Illumina, Inc. . Nextera Rapid Capture Enrichment Reference Guide. , (2016).
- Rev. B. Illumina, Inc. . MiSeq Reagent Kit v3 Reagent Preparation Guide. , (2013).
- Illumina, Inc. . MiSeq System Guide. , (2015).
- . BaseSpace Sequence Hub Available from: https://basespace.illumina.com/dashboard (2017)
- Rev. B. Agilent Technologies. . Agilent High Sensitivity DNA Kit Guide. , (2013).
- Illumina, Inc. . MiSeq System Denature and Dilute Libraries Guide. , (2016).
- Illumina, Inc. . System Specification Sheet: MiSeq System. , (2016).
- . BaseSpace Sequence Hub Help Center Available from: https://help.basespace.illumina.com/ (2017)
- Qiagen. . Genomics Workbench 10.1.1 User Manual. , (2017).
- Ebbert, M. T., et al. Evaluating the necessity of PCR duplicate removal from next-generation sequencing data and a comparison of approaches. BMC Bioinformatics. 17, 239 (2016).
- Wang, K., Li, M., Hakonarson, H. ANNOVAR: functional annotation of genetic variants from high-throughput sequencing data. Nucleic Acids Res. 38 (16), e164 (2010).
- Leary, N. A., et al. Reference sequence (RefSeq) database at NCBI: current status, taxonomic expansion, and functional annotation. Nucleic Acids Res. 44 (D1), D733-D745 (2016).
- Kitts, A., Phan, L., Ward, M., Bradley Holmes, J. . The Database of Short Genetic Variation (dbSNP). , (2013).
- Lek, M., et al. Analysis of protein-coding genetic variation in 60,706 humans. Nature. 536 (7616), 285-291 (2016).
- Auton, A., et al. A global reference for human genetic variation. Nature. 526 (7571), 68-74 (2015).
- Landrum, M. J., et al. ClinVar: public archive of interpretations of clinically relevant variants. Nucleic Acids Res. 44 (D1), D862-D868 (2016).
- Kircher, M., et al. A general framework for estimating the relative pathogenicity of human genetic variants. Nat Genet. 46 (3), 310-315 (2014).
- Kumar, P., Henikoff, S., Ng, P. C. Predicting the effects of coding non-synonymous variants on protein function using the SIFT algorithm. Nat Protoc. 4 (7), 1073-1081 (2009).
- Adzhubei, I. A., et al. A method and server for predicting damaging missense mutations. Nat Methods. 7 (4), 248-249 (2010).
- Bertram, L., McQueen, M. B., Mullin, K., Blacker, D., Tanzi, R. E. Systematic meta-analyses of Alzheimer disease genetic association studies: the AlzGene database. Nat Genet. 39 (1), 17-23 (2007).
- Xiong, H. Y., et al. The human splicing code reveals new insights into the genetic determinants of disease. Science. 347 (6218), (2015).
- Desmet, F. O., et al. Human Splicing Finder: an online bioinformatics tool to predict splicing signals. Nucleic Acids Res. 37 (9), e67 (2009).
- Richards, S., et al. Standards and guidelines for the interpretation of sequence variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology. Genet Med. 17 (5), 405-424 (2015).
- Li, Q., Wang, K. InterVar: Clinical Interpretation of Genetic Variants by the 2015 ACMG-AMP Guidelines. Am J Hum Genet. 100 (2), 267-280 (2017).
- Yang, Z. L., Sun, G. L. High-frequency, low-coverage "false positives" mutations may be true in GS Junior sequencing studies. Scientific Reports. 7, (2017).
- Gandhi, P. N., Wang, X., Zhu, X., Chen, S. G., Wilson-Delfosse, A. L. The Roc domain of leucine-rich repeat kinase 2 is sufficient for interaction with microtubules. J Neurosci Res. 86 (8), 1711-1720 (2008).
- Goldwurm, S., et al. The G6055A (G2019S) mutation in LRRK2 is frequent in both early and late onset Parkinson's disease and originates from a common ancestor. J Med Genet. 42 (11), e65 (2005).
- Caiazzo, M., et al. Direct generation of functional dopaminergic neurons from mouse and human fibroblasts. Nature. 476 (7359), 224-227 (2011).
- Grimes, D. A., et al. Translated mutation in the Nurr1 gene as a cause for Parkinson's disease. Mov Disord. 21 (7), 906-909 (2006).
- Collins, F. S., Hamburg, M. A. First FDA authorization for next-generation sequencer. N Engl J Med. 369 (25), 2369-2371 (2013).
- Van der Auwera, G. A., et al. From FastQ data to high confidence variant calls: the Genome Analysis Toolkit best practices pipeline. Curr Protoc Bioinformatics. 43, 11-33 (2013).
- Treangen, T. J., Salzberg, S. L. Repetitive DNA and next-generation sequencing: computational challenges and solutions. Nat Rev Genet. 13 (1), 36-46 (2011).
- Shin, S., Park, J. Characterization of sequence-specific errors in various next-generation sequencing systems. Mol Biosyst. 12 (3), 914-922 (2016).
- Povysil, G., et al. panelcn.MOPS: Copy-number detection in targeted NGS panel data for clinical diagnostics. Hum Mutat. 38 (7), 889-897 (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유