Method Article
Sequenziamento di nuova generazione e bioinformatica Pipeline per valutare fattori determinanti genetici della malattia costituzionale mirati
In questo articolo
Riepilogo
Mirati sequenziamento di nuova generazione è un approccio di tempo e costo-efficiente che sta diventando sempre più popolare in diagnostica clinica e ricerca di malattia. Il protocollo descritto qui presenta il complesso flusso di lavoro richiesto per la sequenza e il processo di bioinformatica utilizzato per identificare varianti genetiche che contribuiscono alla malattia.
Abstract
Sequenziamento di nuova generazione (NGS) sta rivoluzionando rapidamente come viene eseguita la ricerca nei determinanti genetici della malattia costituzionale. La tecnica è altamente efficiente con milioni di letture di sequenziamento prodotte in un breve lasso di tempo e a costi relativamente bassi. In particolare, NGS mirata è in grado di indagini di messa a fuoco di regioni genomiche di particolare interesse basato sulla malattia di studio. Non solo fa questo ulteriormente a ridurre i costi e aumentare la velocità del processo, ma diminuisce l'onere computazionale che spesso accompagna NGS. Anche se mirata NGS è limitato ad alcune regioni del genoma, impedendo l'identificazione di potenziali nuovi loci di interesse, può essere una tecnica eccellente di fronte a una malattia fenotipicamente e geneticamente eterogenea, per cui ci sono precedentemente conosciuto associazioni genetiche. A causa della natura complessa della tecnica del sequenziamento, è importante aderire strettamente al protocolli e metodologie al fine di ottenere letture di sequenziamento di elevata copertura e qualità. Ulteriormente, una volta ottenute sequenziamento letture, un flusso di lavoro sofisticato bioinformatica è utilizzato per accuratamente mappa letture di un genoma di riferimento, di chiamare varianti e affinché che le varianti passano parametri di qualità. Varianti anche devono essere annotate e curate basata sulla loro importanza clinica, che può essere standardizzato applicando l'American College of Medical Genetics e linee guida di patogenicità di genomica. I metodi presentati qui visualizzerà i passaggi coinvolti nella generazione e analisi di dati NGS da un pannello di sequenziamento mirato, tramite il pannello di malattia neurodegenerative ONDRISeq come un modello, per identificare le varianti che possono essere di importanza clinica.
Introduzione
Come definire i determinanti genetici di varie condizioni assume una priorità nella ricerca e nella clinica, sequenziamento di nuova generazione (NGS) sta dimostrando di essere uno strumento di alto-rendimento e conveniente per raggiungere questi obiettivi1,2 ,3. Per quasi 40 anni, Sanger sequenziamento era stato il gold standard per identificare varianti genetiche4; Tuttavia, per le malattie con eterogeneità genetica o eziologia genetica, molti geni possibili del candidato devono essere valutati, spesso contemporaneamente. In questo contesto, Sanger sequenziamento diventa costoso e richiede tempo. Tuttavia, NGS comporta sequenziamento massivo parallelo di milioni di frammenti di DNA, che consente per una tecnica efficiente di costo e di tempo rilevare contemporaneamente una vasta gamma di variazione genetica in varie regioni del genoma.
Ci sono tre tipi di NGS per il sequenziamento del DNA: 1) intero genoma di sequenziamento (WGS), 2) intero-dell'esoma sequenziamento (WES) e sequenziamento 3) mirati5. WGS valuta l'intero contenuto genomico di un individuo, mentre WES coinvolge solo le regioni di proteina-codificazione del genoma6di sequenziamento. La sequenziazione mirata, al contrario, si concentra su regioni specifiche del genoma basato su relativamente pochi geni specifici collegati da meccanismi patologici comuni o noti fenotipo clinico. Gli esoni o introni oppure qualsiasi regioni intergeniche di un gene o un gruppo specifico di geni possono essere specificati utilizzando questo approccio. Di conseguenza, sequenziamento mirato può essere un ottimo approccio quando c'è già una base di geni noti per essere associati con la malattia di interesse. Targeting per regioni specifiche del genoma consente per l'eliminazione del superfluo e irrilevante variazione genetica che può cloud o distrarre da interpretazione clinica. Mentre WGS e WES entrambi producono una grande quantità di dati di alta qualità, la quantità di dati può essere schiacciante. Non solo questa grande quantità di dati richiede l'analisi bioinformatica computazionalmente intensivi, ma archiviazione dati frequentemente possa presentare problemi7. Questa sfida di memorizzazione dei dati aggiunge anche costi aggiuntivi sia WGS e WES, che spesso non è considerato inizialmente quando si calcola la spesa di sequenziamento. Ulteriormente, anche se esso è in diminuzione, il costo di WGS e WES rimangono relativamente elevati. Sequenziamento mirato può essere un'opzione più conveniente, specialmente quando è richiesto il sequenziamento di un gran numero di individui.
The Ontario Neurodegenerative malattia Research Initiative (ONDRI) è uno studio di coorte multi-piattaforma, a livello provinciale, d'osservazione che caratterizza cinque malattie neurodegenerative, tra cui: 1) la malattia di Alzheimer e mild cognitive impairment, 2). sclerosi laterale amiotrofica, 3) la demenza frontotemporale, 4) morbo di Parkinson e 5) danno conoscitivo vascolare8. Il sottogruppo di genomica ONDRI mira a chiarire come parte della caratterizzazione della linea di base di questa coorte spesso scontato, eppure estremamente importante panorama genetico di queste malattie fenotipicamente e geneticamente eterogenee. Le malattie neurodegenerative sono così candidati appropriati per le metodologie di NGS e per la sequenziazione mirata in particolare.
Abbiamo personalizzato-progettato un pannello NGS mirato, ONDRISeq, sequenziare 528 partecipanti coinvolti in ONDRI per le regioni di codificazione della proteina di 80 geni che sono stati precedentemente associati con le cinque malattie di interesse. Con questa metodologia, siamo in grado di sfruttare i dati NGS di alta qualità in modo mirato ed efficiente. La progettazione e la convalida del pannello ONDRISeq con gli studi multipli di concordanza precedentemente è stato descritto, per il quale il pannello di ONDRISeq era in grado di identificare il romanzo, rare varianti di significato clinico possibile nel 72,2% dei 216 casi utilizzati per la convalida di pannello 9. Sebbene NGS technology ha avanzato rapidamente e notevolmente negli ultimi anni, molti ricercatori affrontano una sfida durante l'elaborazione dei dati grezzi in un elenco di varianti utilizzabili, con annotazioni10. Ulteriormente, interpretazione delle varianti può essere complessa, soprattutto quando di fronte a molti che sono rari o romanzo11.
Qui, descriviamo in maniera dettagliata, la metodologia di NGS mirati e il flusso di lavoro associato bioinformatica necessaria per nuova sequenza, variante chiamata e la variante annotazione utilizzando il ONDRISeq studia come un esempio. Dopo la generazione di dati NGS, file raw sequenziamento devono essere allineati al genoma umano riferimento per poter chiamare accuratamente varianti. Varianti quindi devono essere annotate al fine di eseguire la successiva variante curation. Spiegheremo anche nostra implementazione dell'American College of Medical Genetics standard e linee guida per classificare accuratamente variante patogenicità.
Protocollo
Ai fini di ONDRI, protocolli di etica e di consenso informato sono stati ottenuti basato sulle tavole di etica di ricerca presso Baycrest Centre for Geriatric Care (Toronto, Ontario, Canada); Centre for Addiction and Mental Health (Toronto, Ontario, Canada); Ospedale del Elizabeth Bruyère (Ottawa, Ontario, Canada); Ospedale generale di Hamilton (Hamilton, Ontario, Canada); Centro di Scienze di salute di Londra (London, Ontario, Canada); McMaster (Hamilton, Ontario, Canada); L'ospedale di Ottawa (Ottawa, Ontario, Canada); Parkwood Hospital (London, Ontario, Canada); Ospedale di s. Michele (Toronto, Ontario, Canada); Sunnybrook Health Sciences Centre (Toronto, Ontario, Canada); e University Health Network-Toronto Western Hospital (Toronto, Ontario, Canada).
1. isolamento DNA da campioni di sangue umano
-
Raccogliere campioni da partecipanti di sequenziamento secondo protocolli appropriati etica e consenso informato.
- Per ottenere il DNA di alta qualità, prelevare campioni di sangue ai fini dell'estrazione.
Nota: DNA può anche essere Estratto da saliva o cellule orali, assicurando che viene utilizzato un apposito kit di estrazione del DNA. - Se l'estrazione dal sangue, per ottenere un alto rendimento del DNA, raccogliere il campione in tre provette EDTA K2 da 4 mL, fornendo un campione di volume totale ~ 12 ml.
- Centrifugare i campioni di sangue per 20 min a 750 x g alla frazione in una fase superiore del plasma, sottile, fase intermedia dei leucociti e una fase di fondo degli eritrociti.
- Per ottenere il DNA di alta qualità, prelevare campioni di sangue ai fini dell'estrazione.
- Rimuovere il plasma dal campione di sangue pipettando fuori dalla parte superiore del campione con una pipetta monouso. Opportunamente scartare il plasma o dispensare in diverse aliquote di 500 µ l per conservazione a-80 ° C per future analisi biochimiche. Assicurarsi che una nuova pipetta sterile è utilizzata per ogni campione.
- Estrarre il DNA dal campione di sangue con sangue estrazione kit12 (Tabella materiali) secondo le istruzioni del produttore.
Nota: Se si ottiene un campione del volume sopra descritto, ~ 3 mL di leucociti si otterranno per utilizzare nell'estrazione del DNA. - Misurare la concentrazione di DNA iniziale in ng / µ l con un spettrofotometro di pieno-spettro13 (Tabella materiali), secondo le istruzioni del produttore.
- Procedere direttamente al passaggio 2. In alternativa, conservare il DNA a 4 ° C.
2. sequenziamento libreria preparazione
-
Effettuare diluizioni seriali sui campioni di DNA nel corso di tre giorni per ottenere una concentrazione finale di 5,0 ± 1.0 ng / µ l.
- Diluire 1 M Tris buffer pH 8,5 a 10 µM con acqua deionizzata.
Nota: Il volume diluito dipenderà il numero di campioni di DNA che dovranno essere diluiti nei passaggi successivi. - Se si esegue la diluizione del DNA direttamente dopo passo 1.4, procedere al passaggio seguente. Se non lo stesso giorno, misurare la concentrazione di DNA come è stato fatto nel passaggio 1.4.
- Sulla base della concentrazione misurata, diluire 40 µ l di DNA a ~ 10 ng / µ l, utilizzando 10 µM tampone Tris pH 8.5 e consentire al campione per sedersi durante la notte a 4 ° C.
- Misurare la concentrazione di DNA con un fluorimetro14 appropriato per la quantificazione del DNA (Tabella materiali), secondo le istruzioni del produttore.
Nota: La concentrazione del campione deve essere > 10 ng / µ l a causa della bassa sensibilità dello spettrofotometro utilizzato in precedenza. - Sulla base della concentrazione misurata, diluire 20 µ l di DNA in 10 ng / µ l, utilizzando 10 µM tampone Tris pH 8.5 e consentire al campione per sedersi durante la notte a 4 ° C.
- Misurare la concentrazione di DNA con il fluorimetro14, secondo le istruzioni del produttore.
- Sulla base della concentrazione misurata, diluire 10 µ l di DNA a 5 ng / µ l, utilizzando pH di Tris-HCl 10 µM 8.5 e consentire al campione per sedersi durante la notte a 4 ° C.
- Diluire 1 M Tris buffer pH 8,5 a 10 µM con acqua deionizzata.
-
Preparare il sequenziamento libreria secondo le istruzioni del produttore del pannello NGS mirata destinazione appropriata arricchimento kit15 (tabella materiali). Assicurarsi che il kit di arricchimento sia appropriato per la piattaforma NGS utilizzata.
- Seguire istruzioni16 per quanto riguarda la complessità e la messa in comune delle biblioteche del produttore.
Nota: Per ONDRISeq, librerie sono composte da 12 campioni di DNA, riuniti in gruppi di due ed eseguire lo strumento desktop di NGS (Tabella materiali). Il numero di campioni che possono essere eseguiti in una singola reazione dipenderà il kit di sequenziamento e la piattaforma utilizzata. - Per ottenere dati di sequenziamento di qualità superiore, eseguire il passaggio facoltativo per convalidare i qualità di libreria di DNA seguendo tagmentation, descritto nelle istruzioni del fabbricante della destinazione arricchimento kit15.
- Analizzare ogni libreria in triplice copia per garantire la qualità del rendimento biblioteca.
- Se il pool di librerie, misurare la concentrazione di DNA con il fluorimetro14, secondo le istruzioni del produttore. Utilizzare questa concentrazione per determinare il volume di ciascuna libreria di DNA alla piscina per ottenere i rapporti equimolari consigliati dal kit di arricchimento di destinazione viene utilizzato.
- Seguire istruzioni16 per quanto riguarda la complessità e la messa in comune delle biblioteche del produttore.
3. next-generation Sequencing
-
Sequenza la libreria secondo istruzioni17,18 (tabella materiali del produttore NGS desktop dello strumento dei reagenti kit).
- Preparare un foglio del campione secondo istruzioni18 utilizzando il software di tecnologia NGS appropriato (Tabella materiali), che sarà importato nel flusso di lavoro dello strumento desktop NGS del produttore.
Nota: Ai fini della ONDRISeq, l'opzione di applicazione scelto è 'altro', con solo i file FASTQ richiesto (Figura 1). I passaggi successivi elaborerà questi file FASTQ, per consentire completa personalizzazione dei parametri di qualità e di allineamento. Tuttavia, se viene scelto il sequenziamento mirato, alcuni strumenti NGS sono in grado di elaborare i dati di sequenziamento in file VCF stessi. Istruzioni18 del produttore può essere consultato per una selezione completa di opzioni. - Se si utilizza un basato su cloud computing ambiente19 (Tabella materiali), il login quando si imposta la sequenza di esecuzione. Eseguire questa operazione dopo aver cliccato su "Sequenziamento" NGS strumento desktop home page.
- In seguito biblioteca denaturazione18 secondo le istruzioni del produttore, misurare la concentrazione di libreria di DNA con il fluorimetro14.
- Convalidare la qualità di libreria di DNA usando un sistema per elettroforesi automatica appropriato e DNA qualità analisi kit20 (Tabella materiali), secondo le istruzioni del produttore.
- Per convertire la concentrazione di DNA da ng / µ l in nM, utilizzare la seguente formula16
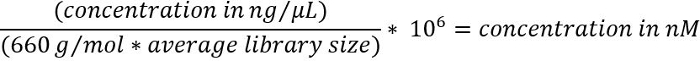
Nota: Dimensione media biblioteca sarà specifica per kit di arricchimento di destinazione utilizzato e può essere ottenuta dalla traccia elettroforesi osservata al punto 3.1.4. - Diluire la libreria di sequenziamento ad una concentrazione finale di 6-20 pM, come appropriato e il volume di 600 μL, secondo istruzioni21 di produttore.
Nota: La concentrazione esatta necessaria dipende dal sequenziamento kit utilizzato. Rivolgersi al produttore del kit di arricchimento per determinare la concentrazione di caricamento corretto. - Diluire, denaturare e includere un controllo positivo sequenziamento biblioteca21, secondo le istruzioni del produttore.
- Tenere un registro di ogni sequenza di esecuzione, che include la concentrazione di libreria DNA caricato (pM), la percentuale di controllo positivo aggiunto, codice a barre cartuccia di reagente, applicazione scelto al punto 3.1.1, numero di letture di indice, kit di arricchimento utilizzato, leggere lunghezze e la nome del foglio di esempio.
Nota: Il tempo di esecuzione dello strumento desktop NGS dipenderà lo strumento, kit di arricchimento e leggere lunghezze scelte (4 – 56 h per il sequencer usato in questo esperimento22).
- Preparare un foglio del campione secondo istruzioni18 utilizzando il software di tecnologia NGS appropriato (Tabella materiali), che sarà importato nel flusso di lavoro dello strumento desktop NGS del produttore.
- Al termine dell'esecuzione del sequenziamento, accedere alla "cartella di eseguire", che comprende tutte le uscite, navigando per la home page NGS strumento desktop e facendo clic su "Gestione file". Spostare i file in un'unità locale per un accesso successivo. Per un'opzione separata, in un computer, è possibile trovare i file all'interno basato su cloud computing ambiente19 selezionando "Runs" sul pannello di navigazione. Selezionare il sequenziamento appropriato eseguito per spostarsi nella pagina di riepilogo di eseguire. Seleziona "Scarica" per ottenere i dati dal cloud. Nella finestra di dialogo che appare, selezionare i file FASTQ come tipo di file da scaricare e fare clic su "Download".
-
Dalla pagina Run Riassunto del basata su cloud computing ambiente19,23, passare alla "Grafici" per analizzare la qualità della sequenziazione eseguita con le varie figure, prodotte dall'ambiente informatico. Consultare istruzioni23 per maggiori dettagli per quanto riguarda ogni figura prodotta del produttore.
- Dalla pagina eseguire grafici, trovare la figura con l'etichetta "Dati di ciclo". In grafico, selezionare "Intensità" e in canale selezionare "Tutti i canali". Assicurarsi che questa trama di intensità di segnale prodotta è simile a quella prodotta mediante sequenziamento esecuzioni eseguite in passato con lo stesso kit di arricchimento e strumento desktop NGS.
Nota: Ciò riflette la percentuale di intensità mostrata da ogni base attraverso tutti i 150 cicli. La figura può variare notevolmente a seconda del kit di arricchimento utilizzato, ragion per cui deve essere rispetto al passato corre di sequenziamento del pannello stesso. - Selezionare la scheda "Indicizzazione QC" all'interno del pannello di navigazione esecuzione per trovare l'indicizzazione istogramma di controllo qualità (QC), che si trova sul lato destro della pagina. Assicurare che una distribuzione relativamente uniforme di % legge identificato (PF) è osservata attraverso tutti i campioni.
Nota: Se i campioni hanno una % molto inferiore legge identificato (PF) rispetto al resto dei campioni, nota che la qualità dei dati di sequenziamento può risentirne.
- Dalla pagina eseguire grafici, trovare la figura con l'etichetta "Dati di ciclo". In grafico, selezionare "Intensità" e in canale selezionare "Tutti i canali". Assicurarsi che questa trama di intensità di segnale prodotta è simile a quella prodotta mediante sequenziamento esecuzioni eseguite in passato con lo stesso kit di arricchimento e strumento desktop NGS.
-
Dalla pagina Run Riassunto dell'ambiente basato su cloud computing, individuare le metriche di qualità facendo "Metriche" all'interno del pannello di navigazione gestito.
Nota: Il kit di piattaforma e l'arricchimento di sequenziamento utilizzato dipenderà metriche cut-off. Ci sono molti parametri che possono essere utilizzate basato su istruzioni23, del produttore con i seguenti passaggi evidenziando tre che sono altamente raccomandati per il controllo qualità.- Sotto "Densità (K/MM2)" garantire la densità di cluster è compreso nell'intervallo consigliato dal kit di arricchimento utilizzato (in questo caso 1.200 – 1.400 K/mm2).
- Sotto il totale "% ≥Q30" assicurarsi che il valore sia ≥ 85%, che riflette la qualità del sequenziamento si legge.
Nota: Se inferiore a questa soglia dell'85%, nota che la qualità della sequenziazione potrebbe essere compromessa. - Sotto "Allineato (%)" garantire che il valore è simile al % del controllo positivo è stato incluso nel sequenziamento eseguito.
Nota: Questo agisce come una misura di controllo positivo, tale che solo questa percentuale di totale letture sono stati trovati per allineare il genoma di controllo positivo. Se il controllo positivo 1% è stato utilizzato ci si aspetterebbe che l'allineato (%) sarebbe ~ 1 – 5%.
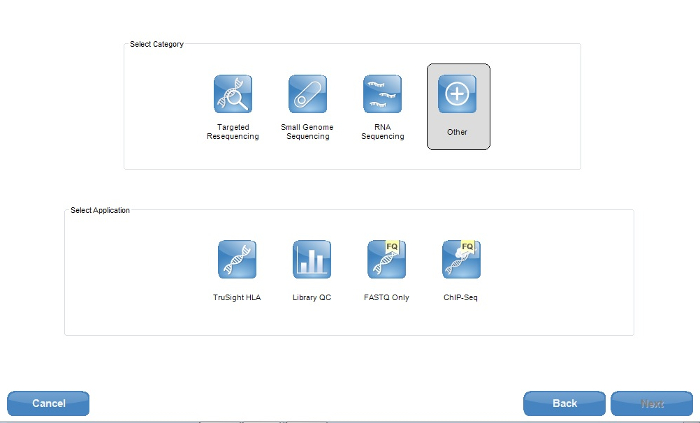
Figura 1: opzioni di applicazione del creatore di fogli di esempio Screenshot del software di tecnologia NGS (tabella materiali). Ai fini della ONDRISeq, l'unica applicazione di FASTQ viene utilizzato. Tuttavia, se l'utente desidera che altri file generati, ad esempio file VCF, è consigliabile che si utilizza un'applicazione all'interno della categoria risequenziamento mirata. Clicca qui per visualizzare una versione più grande di questa figura.
4. risequenziamento e variante chiamata
- Per la pre-elaborazione dei dati, selezionare software appropriato per allineare i file FASTQ raw per il genoma di riferimento umano e chiamare varianti (Tabella materiali).
- Consente di importare FASTQ sequenziamento letture il software di pre-elaborazione dei dati.
Nota: Ai fini della ONDRISeq, i 48 file FASTQ prodotti da una corsa singola sequenza di 24 campioni sono importati ed elaborati attraverso il software. Il numero di campioni trattati in una sola volta può variare secondo le esigenze del ricercatore e le dimensioni del pannello di NGS.- All'interno dell'Area di navigazione"", fare clic destro e selezionare "Nuova cartella". Nome della cartella in tale che non c'è chiarezza per quanto riguarda la sequenza di esecuzione che è stata eseguita.
- Nella barra degli strumenti nella parte superiore, selezionare "Importa". Dal menu a discesa elenco di piattaforme di sequenziamento mostrato ha scelto la piattaforma con cui è stato effettuato il sequenziamento.
Nota: Ai fini del ONDRISeq, "Illumina" è selezionato. Tuttavia, se si utilizza un diverso ordinamento piattaforma consultare le istruzioni del produttore per il resto della parte importatrice di FASTQ passaggi24. - Nella finestra di dialogo, spostarsi e selezionare la FASTQ file dal sequenziamento Esegui che è in fase di elaborazione. Garantire che i file importati vengono archiviati in e importati dall'unità locale, se si utilizza un computer con più server.
- Tra le "Opzioni generali" della finestra di dialogo, fare clic sulla casella accanto a "Paired letture" se l'ordinamento utilizzato accoppiati fine prodotti chimici per.
Nota: In questo caso, dovrebbe anche esserci due campioni FASTQ importati per ciascun campione - uno in avanti e una retromarcia. - Paired leggere le informazioni nella finestra di dialogo, selezionare "Paired-fine (avanti-indietro)" se l'avanti leggere file FASTQ viene visualizzata prima la lettura inversa nell'elenco dei file. Se i file vengono visualizzati nell'ordine opposto, selezionare "Mate-coppia (reverse-avanti)". Impostare la distanza minima lettura accoppiato a 1 e la distanza massima di 1000, per consentire la rilevazione delle riorganizzazioni strutturali di piccola scala all'interno delle sequenze di campione.
- Dalle opzioni"Illumina" nella finestra di dialogo, selezionare "Rimuovi fallito letture", per rimuovere le letture che non riuscito di sequenziamento. Se lo strumento desktop NGS de-multiplex i dati prima di esportare i file FASTQ non selezionare la casella di "MiSeq de-multiplexing".
- Nell'elenco a discesa "Punteggio di qualità", selezionare la Pipeline di NGS che è stato utilizzato per la sequenza. Selezionare "Avanti" nella parte inferiore della finestra di dialogo.
Nota: La pipeline utilizzata influirà il formato dei punteggi di qualità di file FASTQ. Per ulteriori informazioni su quali pipeline per selezionare, consultare istruzioni24 del produttore. - Dalla nuova finestra di dialogo, selezionare "Salva" e "creare sottocartelle per unità bagno mettere FASTQ file di ogni campione nella propria cartella individuale. Selezionare "Avanti" nella parte inferiore della finestra di dialogo.
- Nella finestra di dialogo nuovo, scegliere la cartella che è stata creata al punto 4.2.1. Questo è dove verranno importati i file FASTQ. Selezionare "Fine" nella parte inferiore della finestra di dialogo e attendere che i file FASTQ sono importati. Fare clic sulla scheda "Processi" per visualizzare lo stato dell'importazione file.
- Progettare un flusso di lavoro all'interno del software per eseguire la nuova sequenza e variante chiamata, secondo le istruzioni del produttore.
Nota: Questo flusso di lavoro può variare in base alle esigenze del ricercatore, ma i passaggi seguenti comprendono ciò che è incluso ai fini del ONDRISeq (Figura 2). La procedura descritta in questo flusso di lavoro può essere applicata ad altri NGS risequenziazione e variante chiamata software come appropriato. Tutti i bioinformatica trattamento per le finalità di ONDRI viene eseguita in riferimento al genoma umano riferimento GRCH37/hg19, per coerenza di elaborazione dei dati e analisi.- Mappare le letture di sequenziamento del genoma di riferimento.
- Durante la configurazione, scegliere il genoma di riferimento come appropriato, assicurandosi che sia lo stesso genoma di riferimento che viene utilizzato per tutti i passaggi di bioinformatica.
- Dalla modalità mascheramento elenco a discesa non selezionare "Nessun mascheramento" in modo che nessuna regione della sequenza di riferimento vengono mascherata.
- Utilizzare l'impostazione predefinita opzioni assegnate dal software di mappatura. Revisione istruzioni24 per verificare che questo sia accettabile del produttore basato sugli scopi della ricerca.
- Includere nel riallineamento al genoma umano riferimento per risolvere qualsiasi lettura errori di mappatura, in particolare che circonda varianti di inserzione-omissione locale del flusso di lavoro.
- Utilizzare le opzioni di riallineamento locale predefinito assegnate dal software. Revisione istruzioni24 per verificare che questo sia accettabile del produttore basato sugli scopi della ricerca.
- Rimuovere il duplicato letture mappate prodotte dalla PCR nell'ambito del protocollo NGS per ridurre l'effetto della compensazione dell'amplificazione di PCR, che può produrre falsi positivi25.
- Impostare la "massima rappresentazione della sequenza di minoranza (%)", in base alle esigenze della ricerca.
Nota: Un'impostazione indulgente, come usato ai fini della ONDRISeq, è 5%; Tuttavia, l'impostazione predefinita del software è più rigorose 20%. Quando due letture sono molto simili, questa impostazione determina se la sequenza con meno lettura conta deve essere considerata un errore di sequenza dal bias di amplificazione di PCR. Pertanto, l'impostazione 5%, la minoranza leggere conteggio deve essere ≤ 5% della maggioranza leggere conteggio essere corretto sia identica alla maggior parte leggere.
- Impostare la "massima rappresentazione della sequenza di minoranza (%)", in base alle esigenze della ricerca.
- Esportare le statistiche per le regioni di destinazione sotto forma di un file di testo del riepilogo di code coverage dal letti brani generati al punto 4.3.3. Ignorare non specifico partite e rotte coppie nelle impostazioni. Scegliere una destinazione sul disco locale per questi file.
- Esportare un file di mappa (BAM) di allineamento di sequenza binaria per ciascun campione dal letti brani generati al punto 4.3.3. Questo file contiene dati di allineamento di sequenza, se necessario in futuro analisi. Scegliere una destinazione sul disco locale per questi file.
- Scegliere un metodo di rilevazione variante di chiamare varianti all'interno della sequenza.
Nota: Quando l'ipotesi possono essere fatto circa la ploidia dei campioni, è consigliabile che venga utilizzato un algoritmo di rilevamento variante fissa ploidia, come viene utilizzato ai fini della ONDRISeq. Se questo presupposto non può essere effettuato, consultare istruzioni24 per determinare il miglior algoritmo per le finalità della ricerca del produttore.- Quando si configura, dalla ploidia fissa parametri variant opzioni impostate la ploidia come appropriato per l'organismo del campione. Impostare la "probabilità variante richiesta", ovvero la probabilità che una variante è stata chiamata correttamente in modo che esso debba essere conservato, a 90,0%.
- Utilizzare le seguenti impostazioni per i filtri generali consigliate: "Copertura minima" di 10 x, "Conte minimo" di 2, "Minimo leggere frequenza" del 20%, "Ignora rotto coppie", ignorare aspecifici corrispondenze basate su "Letture", e "Minimo leggere lunghezza" di 20.
Nota: Questi parametri sono basati su fini del ONDRISeq. Consultare istruzioni24 affinché che siano appropriate per la ricerca che viene fatta del produttore. - Utilizzare le seguenti impostazioni consigliate per i filtri di rumore: "Filtri di qualità di Base" con un "raggio di vicinato" mapping di Punteggio di qualità di 5, "minimo centrale qualità" mappatura Punteggio di 20, "minimo quartiere" mappatura Punteggio di qualità e di 15; un "filtro direzione di lettura" del 5,0%; e "Parente leggere filtro direzione" di significato di 1,0%.
Nota: Questi parametri sono basati su fini del ONDRISeq. Consultare istruzioni24 affinché che siano appropriate per la ricerca che viene fatta del produttore.
- Filtrare le varianti che sono state chiamate basate sulla loro sovrapposizione con destinazione regioni mirata del pannello come specificato dal file di dati Extensible Browser (BED), consentendo solo le varianti che si verificano all'interno delle regioni genomiche selezionate per il pannello NGS mirato per essere mantenute.
Nota: Il file letto sarà unico al pannello di NGS mirato che viene utilizzato, basate sulle regioni del genoma che il pannello è in grado di coprire. - Esportare un report variante in un file di formato (VCF) chiamata variante dalla pista variante prodotta nel passaggio 4.3.7. Scegliere una destinazione sul disco locale per questi file.
- Salvare e installare il flusso di lavoro ai sensi istruzioni24, del produttore per renderlo disponibile in "Toolbox" del software. Assicurare che il flusso di lavoro è denominato tale che è chiaro in futuro quale pannello NGS è adatto per.
- Nella finestra di dialogo con le opzioni "Esportazione dati di riferimento" durante l'installazione, è necessario impostare tutte le opzioni per "Bundle".
- Nella finestra di dialogo con le opzioni "Percorso di installazione" durante l'installazione, fare clic su "Installa il flusso di lavoro nel computer locale".
- Mappare le letture di sequenziamento del genoma di riferimento.
- Eseguire file letti FASTQ sequenziamento importati attraverso il flusso di lavoro personalizzato bioinformatica progettato al punto 4.3, secondo istruzioni24 del produttore.
- Identificare il flusso di lavoro progettato in fase 4.3 del software "casella degli strumenti" e fare doppio clic su esso.
- All'interno della finestra di dialogo che viene visualizzata, individuare le cartelle di file FASTQ che sono stati importati nel passaggio 4.2 all'interno della "zona di navigazione". Evidenziare tutte le cartelle selezionandoli all'interno della "zona di navigazione" e quindi fare clic sulla casella accanto a "Batch". Utilizzare la freccia rivolta a destra per spostare i file in "Elementi selezionati". Fare clic su "Avanti" nella parte inferiore della finestra di dialogo.
- All'interno della finestra di dialogo, rivedere la panoramica"Batch" per garantire i corretti file FASTQ sono stati selezionati e quindi fare clic su "Avanti".
- Recensione i seguenti passaggi del flusso di lavoro all'interno della finestra di dialogo per garantire i file corretti ed esportare percorsi sono stati selezionati durante la progettazione del flusso di lavoro in fase 4.3: "Mappa legge di riferimento"; Rimuovi Duplicate letture mappate"; "Creare statistiche per le regioni di destinazione"; "Esportare BAM"; "Testo delimitato da tabulazione di esportazione"; "Filtro basato sulla sovrapposizione"; e "Esporta VCF"
- All'interno il passaggio finale nella finestra di dialogo-"risultato di gestione" - selezionare l'opzione "Salva nella cartella di input". Fare clic su "Finish" nella parte inferiore della finestra di dialogo.
Nota: Questo significa che i file prodotti per ogni campione verrà inserito nella stessa cartella contenente il file FASTQ all'interno dei dati software di pre-elaborazione.
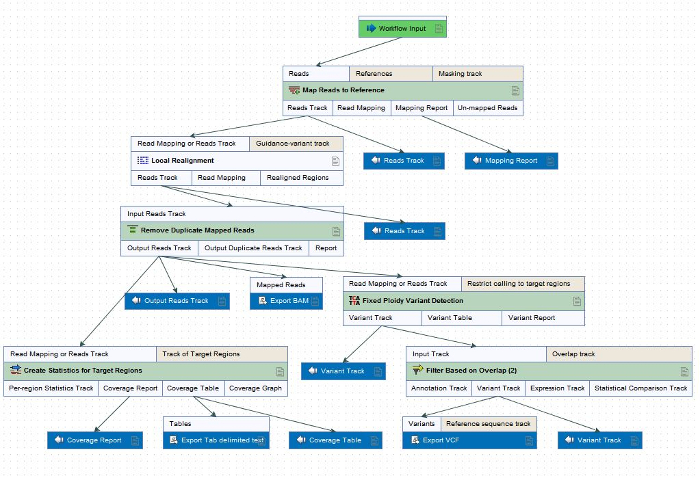
Figura 2: flusso di lavoro per la nuova sequenza e la variante chiamata di FASTQ files all'interno dei dati di pre-elaborazione software (tabella materiali) personalizzato ai fini della ONDRISeq. I passaggi del flusso di lavoro possono essere applicati a altri risequenziazione di NGS e variante chiamata software basato sulle esigenze del ricercatore. Clicca qui per visualizzare una versione più grande di questa figura.
5. variante annotazione
-
Scaricare e personalizzare lo script di26 annotare variazione (ANNOVAR) per eseguire la variante annotazione al momento il file VCF di ciascun campione.
- Scaricare i seguenti database da ANNOVAR essere inclusi come annotazioni: 1) RefSeq27 (aggiornamento agosto 2015); 2) dbSNP13828 (aggiornamento settembre 2014); 3) il Consorzio di aggregazione dell'esoma29 (ExAC, versione 0.3 novembre 2015 aggiornamento); 4) la nazionale cuore, polmone e sangue Istituto dell'esoma Sequencing progetto europeo coorte30 (ESP, aggiornamento marzo 2015); 5) il 1000 genomi progetto europeo coorte31 (1KGP, aggiornamento agosto 2015); 6) ClinVar32 (marzo 2016 aggiornamento); e 7) combinato annotazione lo svuotamento dipendente33 (CADD), ordinamento intollerante da tollerante34 (SIFT) e PolyPhen-235.
Nota: Genoma coordina e tutti i database a cui fa riferimento ANNOVAR cui Build genoma umano GRCh37/hg19. Inoltre, le versioni di database elencate sono quelli utilizzati ai fini della ONDRISeq, quando scaricare i database utilizzano le versioni più aggiornate disponibili. - Se lo si desidera, personalizzare ANNOVAR per uscita la lista completa delle varianti con annotazioni, come pure una ridotta compilation di varianti con annotazioni utilizzando l'opzione-- operazione di filtro26.
Nota: L'elenco ridotto può essere personalizzato in base alle esigenze del ricercatore. Ai fini della ONDRISeq, l'elenco ridotto di varianti con annotazioni non include varianti che si verificano oltre 15 basi dall'esone più vicina o eventuali varianti con una frequenza allelica minore (MAF) > 3% in uno qualsiasi dei tre database: 1) ExAC; 2) ESP; e 3) 1KGP. Questo passaggio è altamente raccomandato. - Se lo si desidera, è possibile personalizzare ANNOVAR per singolo allele specifiche chiamate sulla base delle esigenze del ricercatore26.
Nota: Ai fini della ONDRISeq, ANNOVAR valuta le chiamate del sequenziamento per la rs429358 di alleli APOE rischio (C > T):p.C130R e rs7412 (C > T):p.R176C per il genotipo APOE complessivo, di cui esistono sei possibili di uscita combinazioni, tra cui: 1) E2/E2; 2) E3/E2; 3) E4/E2; 4) E3/E3; 5) E4/E3; 6) E4/E4. Di questi sei possibili genotipi di APOE , E4/E4 è il fattore di rischio genetico più comunemente accettato per lo sviluppo di malattia di Alzheimer36.
- Scaricare i seguenti database da ANNOVAR essere inclusi come annotazioni: 1) RefSeq27 (aggiornamento agosto 2015); 2) dbSNP13828 (aggiornamento settembre 2014); 3) il Consorzio di aggregazione dell'esoma29 (ExAC, versione 0.3 novembre 2015 aggiornamento); 4) la nazionale cuore, polmone e sangue Istituto dell'esoma Sequencing progetto europeo coorte30 (ESP, aggiornamento marzo 2015); 5) il 1000 genomi progetto europeo coorte31 (1KGP, aggiornamento agosto 2015); 6) ClinVar32 (marzo 2016 aggiornamento); e 7) combinato annotazione lo svuotamento dipendente33 (CADD), ordinamento intollerante da tollerante34 (SIFT) e PolyPhen-235.
-
Interrogare i database di mutazione di malattia (tabella materiali) per determinare se le varianti sono stati precedentemente associati con la malattia, con ragionevoli evidenze. Prendere in considerazione tutte le varianti che precedentemente non sono state segnalate come una variante novella.
- Valutare le annotazioni di ANNOVAR da ClinVar, tale che le varianti di malattia-collegati includono qualsiasi classificati come probabile patogeni o patogeni.
- Processo di splicing varianti attraverso la previsione in silico strumenti basati su impionbatura analisi delle varianti37 (SPANR) e umano Splicing Finder38 (HSF, versione 3.0).
- Se l'elaborazione di un gran numero di campioni, confrontare le chiamate variante all'interno di ogni campione per determinare che le varianti sono condivisi da vari campioni. Eseguire questa operazione manualmente o con uno script personalizzati, consentendo per la rilevazione di sequenziamento possibili artefatti ed eventi di contaminazione.
Nota: Ai fini di ONDRI, uno script personalizzato viene utilizzato per annotare i file di output ANNOVAR confrontandole ad uno altro. Lo script incorpora un'annotazione, per variante, con l'ID di oggetto di qualsiasi altri campioni che harboring la stessa variante, altrimenti definita storia della variante nel gruppo di studio. - Classificare le varianti basate sull'American College of Medical Genetics (ACMG) patogenicità orientamenti39, assegnando ogni variante una classificazione come uno dei seguenti: 1) patogeni; 2) probabilmente patogeni; 3) variante di significato incerto; 4) probabilmente benigno; o 5) benigna.
Nota: Ai fini di ONDRI, uno script di Python progettato internamente viene utilizzato per eseguire classificazione ACMG su una base semi-automatica. Anche se non utilizzato per questo studio, InterVar40 è uno strumento similmente progettato che può essere utilizzato in modo analogo. - Sanger sequenza eventuali varianti con una copertura di sequenziamento di < 30 x e/o varianti che sono state identificate in > 10% della coorte Studio per convalidare che essi non sono sequenza artefatti41.
Risultati
Le metodologie descritte nel presente documento sono state applicate a 528 partecipanti campioni di DNA da individui che sono stati arruolati in ONDRI. Campioni sono stati analizzati in 22 piste di 24 campioni per Esegui nel pannello di ONDRISeq. Nel complesso, dati di sequenziamento sono stati determinati per essere di alta qualità con una copertura media del campione di 78 ± 13 x e tutte le esecuzioni individuali espresse una copertura medio campione > 30x. Ulteriormente, in media, il 94% di tutte le regioni dell'obiettivo sono stati coperti almeno 20 x (tabella 1).
Una media 95,6% di letture sono stati mappati per la sequenza di riferimento e tutti i ONDRISeq corre aveva > 90% di letture mappato (tabella 1). Della legge mappate, 92,0% aveva un Phred Punteggio ≥Q30, con una sola esecuzione avendo < 80% di letture mappate incontro questa metrica di qualità. Tuttavia, questa corsa ancora visualizzato una copertura media di 79 x e 93% del target regioni erano coperti almeno 20 volte.
| Parametro | Media (± DS) | Migliori prestazioni | Più povere prestazioni |
| Densità di cluster (x 103/mm2) | 1424 (±269) | 1347 | 1835 |
| Totale letture (106) | 43,1 (±6.0) | 48,7 | 47,4 |
| Mappato letture (106) | 40.1 (±6.0) | 47,1 | 25,7 |
| Mappata letture (%) | 95,6 (±1.3) | 96,8 | 92,6 |
| Qualità di Phred Punteggio ≥Q30 (%) | 92,0 (±6.0) | 92 | 68,3 |
| Copertura del campione (x) | 78 (± 13) | 99 | 51 |
Tabella 1: Sequenziamento metriche di qualità per 22 gira su ONDRISeq.
Caso di studio: Identificazione di varianti rare in un paziente del Palladio.
Per dimostrare l'utilità del nostro flusso di lavoro mirato di NGS, presentiamo l'esempio di un paziente di 68 anni, Maschio, morbo di Parkinson. Il campione di DNA è stato eseguito lo strumento desktop di NGS (Tabella materiali) utilizzando il pannello di ONDRISeq al fianco di 23 altri campioni ONDRI. L'Esegui visualizzata una densità di cluster di 1.555 x 103/millimetri2. Campione del paziente particolare visualizzato una copertura media di 76 x, con 93,9% del target regioni coperti almeno 20 volte.
Dopo aver eseguito la variante chiamata e annotazione con il flusso di lavoro personalizzato bioinformatica, il paziente è stato trovato per harbor 1351 varianti all'interno degli esoni e circostante 250 bp di 80 geni incluso sul pannello di ONDRISeq. Tuttavia, la pipeline ANNOVAR era in grado di ridurre il numero di varianti considerando sequenza variante ontologia e MAF, come descritto in precedenza. Ciò ha prodotto un elenco di sette varianti che hanno subito la curatela manuale (Figura 3). Da queste sette varianti, due sono stati identificati come avendo importanza clinica possibile. Questo processo è specifico per le esigenze di ONDRI e fu fatto identificando quelli che sono relativamente rare nella popolazione generale e sono nonsynonymous in ontologia provocando un cambiamento nella proteina. Se la variante era stato precedentemente associata con la malattia, le previsioni in silico di deleteriousness alla proteina e la classificazione di patogenicità ACMG delle varianti inoltre sono stati utilizzati in questo processo.
Il primo identificato dall'elenco ridotto era una variante eterozigote, vale a dire LRRK2: c.T3939A, conseguente la p.C1313* variante di sciocchezze. LRRK2 codifica per la proteina Leucine-Rich ripetere Kinase 2, che possiede attività GTPasica e la chinasi42. Inoltre, mutazioni in questo gene sono noti per essere tra le principali cause di malattia del Parkinson familiare43. Questa variante introduce un codone di stop prematuro all'interno LRRK2, perdendo così i residui dell'amminoacido 1.314 – 2, 527. Questo impedisce la traduzione della proteina RAS di proteine complesse (Roc), C-terminale del Roc (COR) e proteina chinasi domini, che sono coinvolti nel funzionamento come un atipico Rho GTPasi e GTP proteina chinasi di proteina, rispettivamente ed era stato previsto essere dannose dall'analisi in silico generati da CADD (CADD Phred = 36). Questa variante è anche rara con una MAF di 0,004% e 0,01% in ExAC ed ESP, rispettivamente ed è assente dal database di 1000G. Inoltre, questo è l'unico paziente fuori tutti i 528 sequenziati che porta questa variante, che è romanzo, dato che non è stato descritto precedentemente nei database di mutazione di malattia (Tabella materiali). La fiducia della variante chiamata è stata confermata dalla sua copertura profonda di x 109. Infine, la variante è stata valutata con l'AMCG standard e linee guida per la patogenicità ed è stata classificata come essendo patogeni.
Il paziente inoltre ha trasportato una seconda variante eterozigote, NR4A2: c.C755A, il p.P252Q cambiamento di senso sbagliato con conseguente. La proteina codificata da NR4A2, nucleare recettore sottofamiglia 4 gruppo A membro 2, è un fattore di trascrizione coinvolto nella generazione di dopaminergico neuroni44 e mutazioni in questo gene sono stati precedentemente associate con malattia di Parkinson malattia45. La sostituzione della prolina non polare per la glutamina polar era stato previsto per essere dannoso per l'analisi in silico Pronostico generato da CADD (CADD Phred = 21,1), ma non dall'analisi generati da SIFT o PolyPhen-2. La variante è rara, con una MAF di 0,004% in ExAC ed assenza da ESP e 1000G. La variante è stata anche identificata in un partecipante ONDRI diagnosticato con danno conoscitivo vascolare, ma precedentemente non è stata descritta nei database di mutazione di malattia. Questa variante ha avuto copertura di solo 18 x, tuttavia, Sanger sequenziamento verrà eseguita al fine di garantire la sua validità all'interno della sequenza. Infine, la variante è stata determinata per essere di importanza incerta quando valutati con l'ACMG standard e linee guida per la patogenicità.
La pipeline di bioinformatica e pannello di ONDRISeq è anche in grado di determinare il genotipo APOE di ogni campione. Questo paziente era determinato ad avere il genotipo APOE E3/E3.
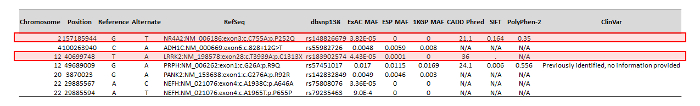
Figura 3: esempio di un output ridotto di ANNOVAR visualizzazione manualmente a cura, annotata varianti. L'output ANNOVAR ridotta dallo studio di caso di un paziente di 68 anni, Maschio, con il morbo di Parkinson. Varianti con annotazioni sono curate per identificare quelli che sono più probabilità di essere di importanza clinica, come indicato dalle caselle rosse. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Nel percorso dall'estrazione del campione di DNA per identificare varianti che possono essere di interesse quando si considera la diagnosi di un paziente, la progressione della malattia e possibili opzioni di trattamento, è importante riconoscere la natura multiforme della metodologia necessaria per sequenziamento e l'elaborazione dei dati appropriati. Il protocollo descritto nel presente documento è un esempio dell'utilizzo di NGS mirate e analisi bioinformatica successive essenziale per identificare varianti rare di potenziale rilevanza clinica. In particolare, vi presentiamo l'approccio adottato dal sottogruppo di genomica ONDRI quando si utilizza il pannello NGS ONDRISeq personalizzati.
È riconosciuto che questi metodi sono stati sviluppati basata su una specifica piattaforma NGS e che ci sono altre piattaforme di sequenziamento e kit di arricchimento di destinazione che può essere utilizzato. Tuttavia, lo strumento desktop e piattaforma NGS (Tabella materiali) è stato scelto in base il suoi primi US Food and Drug Administration (FDA) l'approvazione46. La presente autorizzazione riflette il sequenziamento di alta qualità che possa essere eseguito con i protocolli NGS di scelta e l'affidabilità che può essere posizionato sulle letture di sequenziamento.
Anche se ottenere letture accurate sequenziamento con la profondità di copertura è molto importante, l'elaborazione di bioinformatica richiesta per analisi di variante rara finale è vitale e può essere computazionalmente intensivo. A causa di numerose fonti di errori che possono verificarsi all'interno del processo di sequenziazione, una pipeline di bioinformatica robusto necessario correggere per le varie imprecisioni che possono essere introdotte. Essi possono derivare da disallineamenti nel processo di mapping, bias di amplificazione introdotto dall'amplificazione di PCR nella preparazione libreria e la tecnologia di produzione di manufatti di sequenziamento47. Non importa il software utilizzato per eseguire il mapping di lettura e la variante chiamata, ci sono modi comuni per ridurre questi errori tra cui locale riallineamento, rimozione di duplicati letture mappate e impostazione parametri appropriati per il controllo qualità quando si chiama varianti. Inoltre, i parametri scelti durante chiamata variante possono variare basato su ciò che è più appropriato per lo studio a mano11. La copertura minima e il Punteggio di qualità di una variante e i nucleotidi circostanti che sono stato applicati nel presente documento sono stati scelti da creare un equilibrio tra sensibilità e specificità appropriate. Questi parametri sono stati convalidati per il pannello di ONDRISeq basato su variante chiamata concordanza con tre distinte tecniche genetiche, come descritto in precedenza, tra cui: 1) basata su chip genotipizzazione; 2) test di discriminazione allelica; e 3) di sequenziamento Sanger9.
In seguito chiamata variante accurata, al fine di determinare quelli di potenziale rilevanza clinica, annotazione e curation sono essenziali. Grazie alla sua piattaforma di accesso aperto, ANNOVAR è uno strumento eccellente per sia annotazione e screening preliminare di variante o eliminazione. Oltre ad essere facilmente accessibile, ANNOVAR può essere applicato a qualsiasi file VCF, non importa quale piattaforma di sequenziamento viene utilizzato, ed è personalizzabile in base alle esigenze della ricerca26.
Dopo annotazione, varianti devono essere interpretati per determinare se sono da ritenersi per essere di importanza clinica. Non solo questo processo diventa complesso, ma spesso è incline a soggettività ed errori umani. Per questo motivo, l'ACMG ha fissato linee guida per valutare le prove di patogenicità di qualsiasi variante. Applichiamo un approccio curatela manuale base di variante non-sinonimo, raro, che è costruito sulla base di questi orientamenti e salvaguardato valutando singolarmente ogni variante che è in grado di passare attraverso la pipeline con un design personalizzato che script Python classifica le varianti sulla base degli orientamenti. In questo modo, ogni variante viene assegnata una classifica dei patogeni, probabile significato patogeno, incerta, probabilmente benigno o benigna, e siamo in grado di aggiungere standardizzazione e trasparenza per il processo di variante curation. Esso è importante riconoscere che le specifiche di variante curation, di là della pipeline di bioinformatica, saranno essere individualizzate in base alle esigenze della ricerca ed era quindi oltre l'ambito delle metodologie presentate.
Anche se i metodi presentati qui sono specifici di ONDRI, la procedura descritta può essere tradotto quando si considera un gran numero di malattie costituzionali di interesse. Aumentare il numero delle associazioni di gene per molti fenotipi, NGS mirata consente un'ipotesi di approccio che può capitalizzare la ricerca precedente che è stato fatto nel campo. Eppure, ci sono limitazioni a NGS mirati e la metodologia presentata. Concentrandosi solo su regioni specifiche del genoma, le aree della scoperta sono limitate a alleli novelli di interesse. Di conseguenza, nuovi geni o altri loci genomici di là di quelli coperti dagli obiettivi di sequenziamento, che potrebbero essere rivelati con WGS o WES approcci, non sarà identificato. Ci sono anche regioni all'interno del genoma che possono essere difficili da accuratamente sequenza con approcci NGS, compresi quelli con un elevato grado di sequenze ripetute48 o quelli che sono ricchi di contenuto di GC49. Fortunatamente, quando si utilizzano NGS mirati, c'è a priori un alto grado di familiarità con le regioni genomiche essere sequenziato, e se questi pongono sfide tecniche. Infine, il rilevamento di varianti di numero di copia dai dati NGS attualmente non è standardizzato50. Tuttavia, la bioinformatica soluzioni a queste preoccupazioni possono essere all'orizzonte; nuovi strumenti computazionali possono contribuire ad per analizzare queste ulteriori forme di variazione in ONDRI pazienti.
Nonostante i suoi limiti, NGS mirata è in grado di ottenere dati di alta qualità, all'interno di un approccio basato su ipotesi, pur rimanendo meno costosi rispetto ai suoi omologhi WGS e WES. Non solo è questa metodologia appropriata per ricerca efficiente e diretta, l'implementazione clinica di NGS mirati è in crescita esponenziale. Questa tecnologia sta usanda per rispondere a molte domande per quanto riguarda le vie molecolari di varie malattie. Inoltre è stato sviluppato in un preciso strumento diagnostico a costo relativamente basso quando si oppose a WES e WGS. Anche quando rispetto alla parità aurea Sanger sequenziamento, mirati NGS può prevalere nella sua tempo - e costo-efficienza. Per queste ragioni, è importante per uno scienziato o un clinico che riceve e utilizza dati NGS, per esempio, consegnati come testo in un laboratorio o un rapporto clinico, per capire il complesso "scatola nera" che è alla base dei risultati. I metodi presentati qui dovrebbero aiutare gli utenti a comprendere il processo alla base della generazione e interpretazione dei dati NGS.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Vorremmo ringraziare tutti i partecipanti ONDRI per il loro consenso e la cooperazione con il nostro studio. Grazie agli investigatori ONDRI (www. ONDRI.ca/people), tra cui il nostro investigatore (MJS) e il ONDRI direttivo comitati: il Comitato esecutivo, Comitato direttivo, Comitato di pubblicazione, Comitato di reclutamento, piattaforme di valutazione e management team di progetto. Ringraziamo anche il centro di genomica regionale di Londra per la loro competenza tecnica. AAD è supportato da Alzheimer Society di Londra e Middlesex Masters Graduate Research Scholarship. SMKF è supportato da ALS Canada Tim E. Noël Postdoctoral Fellowship.
Materiali
| Name | Company | Catalog Number | Comments |
| 4 mL EDTA K2 tubes | Fisher Scientific | 02-689-4 | |
| 1 M Tris Buffer | Bio Basic Canada Inc. | SD8141 | |
| Gentra Puregene Blood Kit | Qiagen | 158389 | 1,000 mL Kit. This is the blood extraction kit, referred to in step 1.3. |
| NanoDrop-1000 Spectrophotometer | Thermo Fisher Scientific | ND-2000 | Replaced by the NanoDrop-2000 Spectrophotometer. This is the full-spectrum spectrophotometer, referred to in steps 1.4 and 2.1.2. |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | This is a fluorometer appropriate for the quantification of DNA, referred to in steps 2.1.4, 2.1.6, 2.2.3, and 3.1.3. |
| Nextera Rapid Custom Capture Enrichment Kit | Illumina, Inc. | FC-140-1009 | Specifically designed for the ONDRISeq panel, sequencing the exons of 80 genes, resulting in 971,388 base pairs of sequence in paired-end reads of 150 bases in length; 288 samples per kit. This is the target enrichment kit, referred to in steps 2.2, 2.2.2, 2.2.3, 3.1.5, 3.1.6, 3.4.1, and the Discussion. |
| 2100 BioAnalyzer | Agilent Technologies | G2939BA | This is a automated electrophoresis system, referred to in step 3.1.4. |
| High Sensitivity DNA Reagent Kit | Agilent Technologies | 5067-4626 | 110 Samples per kit; This is a DNA quality analysis kit, referred to in step 3.1.4. |
| MiSeq Reagent Kit v3 | Illumina, Inc. | MS-102-3003 | 600 Cycle Kit; This is the NGS desktop instrument reagent kit, referred to in step 3.1. |
| MiSeq Personal Genome Sequencer | Illumina, Inc. | SY-410-1003 | This is a NGS desktop instrument, referred to in steps 2.2.1, 3.1, 3.1.1, 3.1.2, 3.1.8, 3.2, 4.2.6, the Representative Results, and the Discussion. |
| Experiment Manager | Illumina, Inc. | This is NGS technology software, referred to in step 3.1.1 and Figure 1. https://support.illumina.com/sequencing/sequencing_software/experiment_manager/downloads.html | |
| BaseSpace | Illumina, Inc. | SW-410-1000 | This is a cloud-based computing environment, referred to in steps 3.1.2, 3.2, 3.3, 3.3.1, 3.3.2, 3.4, 3.4.1, 3.4.2 and 3.4.3. https://basespace.illumina.com/ |
| CLC Genomics Workbench 10.1.1 | Qiagen | 832000 | Open source options for data pre-processing are also available that can model the workflow used in this protocol. This is the software used for data pre-processing, referred to throughout step 4 and in Figure 2. |
| Annotate Variation | http://annovar.openbioinformatics.org/en/latest/user-guide/download/ | ||
| RefSeq | National Center for Biotechnology Information | https://www.ncbi.nlm.nih.gov/refseq/ | |
| dbSNP138 | National Center for Biotechnology Information | https://www.ncbi.nlm.nih.gov/projects/SNP/snp_summary.cgi?view+summary=view+summary&build_id=138 | |
| Exome Aggregation Consortium | Broad Institute | http://exac.broadinstitute.org/ | |
| National Heart, Lung, and Blood Institute Exome Sequencing Project European Cohort | University of Washington and the Broad Institute | http://evs.gs.washington.edu/EVS/ | |
| ClinVar | National Center for Biotechnology Information | https://www.ncbi.nlm.nih.gov/clinvar/ | |
| Combined Annotation Dependent Depletion | University of Washington and Hudson-Alpha Institute for Biotechnology | http://cadd.gs.washington.edu/ | |
| Sorting Intolerant from Tolerant | J. Craig Venter Instutite | http://sift.jcvi.org/ | |
| PolyPhen-2 | Brigham and Women's Hospital, Harvard Medical School | http://genetics.bwh.harvard.edu/pph2/ | |
| Human Gene Mutation Database | Qiagen | 834050 | This is a disease mutation database, referred to in step 5.2 and the Representative Results. https://portal.biobase-international.com/cgi-bin/portal/login.cgi?redirect_url=/hgmd/pro/start.php |
| Splicing-based Analysis of Variants | Frey lab, University of Toronto | http://tools.genes.toronto.edu/ | |
| Human Splicing Finder | Aix Marseille Université | http://www.umd.be/HSF3/HSF.shtml | |
| Other materials | |||
| Centrifuge | |||
| Disposable transfer pipets |
Riferimenti
- Metzker, M. L. Sequencing technologies - the next generation. Nat Rev Genet. 11 (1), 31-46 (2010).
- Mardis, E. R. Next-generation DNA sequencing methods. Annu Rev Genomics Hum Genet. 9, 387-402 (2008).
- Shendure, J., Ji, H. Next-generation DNA sequencing. Nat Biotechnol. 26 (10), 1135-1145 (2008).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci U S A. 74 (12), 5463-5467 (1977).
- Farhan, S. M. K., Hegele, R. A. Exome Sequencing: New Insights into Lipoprotein Disorders. Current Cardiology Reports. 16 (7), (2014).
- Choi, M., et al. Genetic diagnosis by whole exome capture and massively parallel DNA sequencing. Proc Natl Acad Sci U S A. 106 (45), 19096-19101 (2009).
- Mardis, E. R. DNA sequencing technologies: 2006-2016. Nat Protoc. 12 (2), 213-218 (2017).
- Farhan, S. M., et al. The Ontario Neurodegenerative Disease Research Initiative (ONDRI). Can J Neurol Sci. 44 (2), 196-202 (2017).
- Farhan, S. M. K., et al. The ONDRISeq panel: custom-designed next-generation sequencing of genes related to neurodegeneration. NPJ Genom Med. (16032), 1-11 (2016).
- El-Metwally, S., Hamza, T., Zakaria, M., Helmy, M. Next-generation sequence assembly: four stages of data processing and computational challenges. PLoS Comput Biol. 9 (12), e1003345 (2013).
- Yohe, S., Thyagarajan, B. Review of Clinical Next-Generation Sequencing. Arch Pathol Lab Med. , (2017).
- Qiagen. . Gentra Puregene Handbook. , (2014).
- NanoDrop Technologies, Inc. . Spectrophotometer V3.5 User's Manual. , (2007).
- Invitrogen by Life Technologies. . Qubit 2.0 Fluorometer User Manual. Vol. Q32866. , (2010).
- Illumina, Inc. . Nextera Rapid Capture Enrichment Guide. , (2016).
- Illumina, Inc. . Nextera Rapid Capture Enrichment Reference Guide. , (2016).
- Rev. B. Illumina, Inc. . MiSeq Reagent Kit v3 Reagent Preparation Guide. , (2013).
- Illumina, Inc. . MiSeq System Guide. , (2015).
- . BaseSpace Sequence Hub Available from: https://basespace.illumina.com/dashboard (2017)
- Rev. B. Agilent Technologies. . Agilent High Sensitivity DNA Kit Guide. , (2013).
- Illumina, Inc. . MiSeq System Denature and Dilute Libraries Guide. , (2016).
- Illumina, Inc. . System Specification Sheet: MiSeq System. , (2016).
- . BaseSpace Sequence Hub Help Center Available from: https://help.basespace.illumina.com/ (2017)
- Qiagen. . Genomics Workbench 10.1.1 User Manual. , (2017).
- Ebbert, M. T., et al. Evaluating the necessity of PCR duplicate removal from next-generation sequencing data and a comparison of approaches. BMC Bioinformatics. 17, 239 (2016).
- Wang, K., Li, M., Hakonarson, H. ANNOVAR: functional annotation of genetic variants from high-throughput sequencing data. Nucleic Acids Res. 38 (16), e164 (2010).
- Leary, N. A., et al. Reference sequence (RefSeq) database at NCBI: current status, taxonomic expansion, and functional annotation. Nucleic Acids Res. 44 (D1), D733-D745 (2016).
- Kitts, A., Phan, L., Ward, M., Bradley Holmes, J. . The Database of Short Genetic Variation (dbSNP). , (2013).
- Lek, M., et al. Analysis of protein-coding genetic variation in 60,706 humans. Nature. 536 (7616), 285-291 (2016).
- Auton, A., et al. A global reference for human genetic variation. Nature. 526 (7571), 68-74 (2015).
- Landrum, M. J., et al. ClinVar: public archive of interpretations of clinically relevant variants. Nucleic Acids Res. 44 (D1), D862-D868 (2016).
- Kircher, M., et al. A general framework for estimating the relative pathogenicity of human genetic variants. Nat Genet. 46 (3), 310-315 (2014).
- Kumar, P., Henikoff, S., Ng, P. C. Predicting the effects of coding non-synonymous variants on protein function using the SIFT algorithm. Nat Protoc. 4 (7), 1073-1081 (2009).
- Adzhubei, I. A., et al. A method and server for predicting damaging missense mutations. Nat Methods. 7 (4), 248-249 (2010).
- Bertram, L., McQueen, M. B., Mullin, K., Blacker, D., Tanzi, R. E. Systematic meta-analyses of Alzheimer disease genetic association studies: the AlzGene database. Nat Genet. 39 (1), 17-23 (2007).
- Xiong, H. Y., et al. The human splicing code reveals new insights into the genetic determinants of disease. Science. 347 (6218), (2015).
- Desmet, F. O., et al. Human Splicing Finder: an online bioinformatics tool to predict splicing signals. Nucleic Acids Res. 37 (9), e67 (2009).
- Richards, S., et al. Standards and guidelines for the interpretation of sequence variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology. Genet Med. 17 (5), 405-424 (2015).
- Li, Q., Wang, K. InterVar: Clinical Interpretation of Genetic Variants by the 2015 ACMG-AMP Guidelines. Am J Hum Genet. 100 (2), 267-280 (2017).
- Yang, Z. L., Sun, G. L. High-frequency, low-coverage "false positives" mutations may be true in GS Junior sequencing studies. Scientific Reports. 7, (2017).
- Gandhi, P. N., Wang, X., Zhu, X., Chen, S. G., Wilson-Delfosse, A. L. The Roc domain of leucine-rich repeat kinase 2 is sufficient for interaction with microtubules. J Neurosci Res. 86 (8), 1711-1720 (2008).
- Goldwurm, S., et al. The G6055A (G2019S) mutation in LRRK2 is frequent in both early and late onset Parkinson's disease and originates from a common ancestor. J Med Genet. 42 (11), e65 (2005).
- Caiazzo, M., et al. Direct generation of functional dopaminergic neurons from mouse and human fibroblasts. Nature. 476 (7359), 224-227 (2011).
- Grimes, D. A., et al. Translated mutation in the Nurr1 gene as a cause for Parkinson's disease. Mov Disord. 21 (7), 906-909 (2006).
- Collins, F. S., Hamburg, M. A. First FDA authorization for next-generation sequencer. N Engl J Med. 369 (25), 2369-2371 (2013).
- Van der Auwera, G. A., et al. From FastQ data to high confidence variant calls: the Genome Analysis Toolkit best practices pipeline. Curr Protoc Bioinformatics. 43, 11-33 (2013).
- Treangen, T. J., Salzberg, S. L. Repetitive DNA and next-generation sequencing: computational challenges and solutions. Nat Rev Genet. 13 (1), 36-46 (2011).
- Shin, S., Park, J. Characterization of sequence-specific errors in various next-generation sequencing systems. Mol Biosyst. 12 (3), 914-922 (2016).
- Povysil, G., et al. panelcn.MOPS: Copy-number detection in targeted NGS panel data for clinical diagnostics. Hum Mutat. 38 (7), 889-897 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon