Method Article
Пробоподготовка к биоинформаматическому анализу метилирования ДНК: стратегия ассоциации для исследований ожирения и связанных с ним признаков
* Эти авторы внесли равный вклад
В этой статье
Резюме
В настоящем исследовании описывается рабочий процесс управления данными метилирования ДНК, полученными с помощью технологий микрочипов. Протокол демонстрирует этапы от подготовки образцов до анализа данных. Все процедуры подробно описаны, а на видео показаны значимые шаги.
Аннотация
Ожирение напрямую связано с образом жизни и связано с изменениями метилирования ДНК, которые могут вызвать изменения в адипогенезе и процессах хранения липидов, способствующих развитию заболевания. Мы демонстрируем полный протокол от отбора до анализа эпигенетических данных пациентов с ожирением и без него. Все шаги из протокола были протестированы и подтверждены в пилотном исследовании. В исследовании приняли участие 32 женщины, в котором 15 человек были классифицированы с ожирением по индексу массы тела (ИМТ) (45,1 ± 5,4 кг/м2); и 17 человек были классифицированы без ожирения в соответствии с ИМТ (22,6 ± 1,8 кг/м2). В группе с ожирением 564 участка CpG, связанных с жировой массой, были идентифицированы методом линейного регрессионного анализа. Сайты CpG находились в промоутерских регионах. Дифференциальный анализ обнаружил 470 CpGs гипометилированных и 94 гиперметилированных участка у лиц с ожирением. Наиболее гипометилированные обогащенные пути были в RUNX, сигнализации WNT и реакции на гипоксию. Гиперметилированные пути были связаны с секрецией инсулина, передачей сигналов глюкагона и Ca2+. Мы пришли к выводу, что протокол эффективно идентифицировал паттерны метилирования ДНК и метилирование ДНК, связанное с признаками. Эти паттерны могут быть связаны с измененной экспрессией генов, влияя на адипогенез и хранение липидов. Наши результаты подтвердили, что обесогенный образ жизни может способствовать эпигенетическим изменениям в ДНК человека.
Введение
Крупномасштабные омические технологии все чаще используются в исследованиях хронических заболеваний. Интересной особенностью этих методов является доступность большого количества генерируемых данных для научного сообщества. Поэтому возникла потребность в стандартизации протоколов, с тем чтобы можно было проводить техническое сопоставление между исследованиями. Настоящее исследование предлагает стандартизацию протокола для получения и анализа данных метилирования ДНК с использованием пилотного исследования в качестве применимого примера.
В современном образе жизни человека преобладает отрицательный расход энергии, приводящий к чрезмерному накоплению жировой ткани и, как следствие, развитию ожирения¹. Многие факторы увеличили показатели ожирения, такие как сидячий образ жизни, высококалорийные диеты и стрессовые процедуры. По оценкам Всемирной организации здравоохранения (ВОЗ), в 2016 году 1,9 миллиарда взрослых страдали ожирением, что означает, что более 20% населения мира имеет более 30 кг / м2 ИМТ2. Последнее обновление 2018 года показало, что распространенность ожирения в Соединенных Штатах Америки (США) была выше 42%3.
Эпигенетика — это структурная адаптация хромосомных областей для регистрации, передачи сигнала или увековечения измененных состояний активности4. Метилирование ДНК является обратимым химическим изменением в сайтах цитозин-гуанозиновых динуклеотидов (сайтах CpG), образуя 5-метилцитозин-pG (5mCpG). Он может модулировать экспрессию генов, регулируя доступ механизма транскрипции к ДНК5,6,7,8. В этом контексте важно понять, какие сайты CpG связаны с чертами, связанными с ожирением9. Многие факторы могут поддерживать или предотвращать сайт-специфическое метилирование ДНК. Необходимые ферменты для этого процесса, такие как ДНК-метилтрансферазы10 (DNTM) и десять-одиннадцать транслокаций (TETs), могут способствовать метилированию или деметилированию ДНК при воздействии окружающей среды11.
Учитывая растущий интерес к исследованиям метилирования ДНК за последние годы, выбор наиболее подходящей стратегии анализа для точного ответа на каждый вопрос был существенной проблемой исследователей12,13,14. Массив метилирования ДНК 450K является наиболее популярным методом, используемым в более чем 360 публикациях14 для определения профиля метилирования ДНК. Он может определять метилирование до 485 000 CpG, расположенных в 99% известных генов15. Тем не менее, этот массив был снят с производства и заменен НА EPIC, охватывающий 850 000 сайтов CpG. Настоящий протокол может применяться как для 450K, так и для EPIC16,17,18.
Протокол представлен шаг за шагом на рисунке 1 и включает в себя следующие этапы: отбор популяции, отбор проб, подготовка эксперимента, конвейер метилирования ДНК и анализ биоинформатики. Пилотное исследование, проведенное в нашей лаборатории, демонстрируется здесь, чтобы проиллюстрировать шаги предлагаемого протокола.
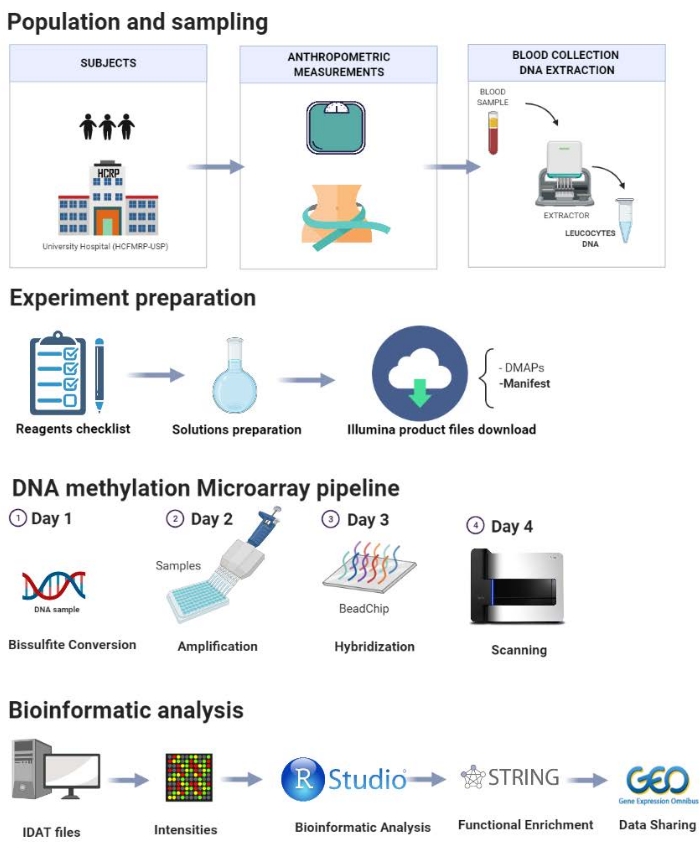
Рисунок 1: Схема представленного протокола. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
протокол
Комитет по этике Университетской больницы Медицинской школы Рибейран-Прету Университета Сан-Паулу (HCRP-USP) одобрил исследование (CAAE: 14275319.7.0000.5440). Участники подписали форму согласия, и все процедуры были проведены в соответствии с Хельсинкской декларацией.
1. Население и выборка
- Наберите участников с ИМТ >30 кг/м2 для исследования. Для настоящего исследования были набраны 32 женщины из популяции примесей19 в возрасте от 18 до 60 лет.
ПРИМЕЧАНИЕ: В HCRP-USP более высокие показатели ожирения наблюдались в основном у женщин20. Участники с ожирением, с ИМТ ≥30 кг/м2 (n = 15), были набраны из амбулаторной клиники метаболических заболеваний в HCRP-USP. Участники без ожирения, с ИМТ от 18,5 кг/м2 до 24,9 кг/м2 (n = 17), были набраны из других клиник и не имели тяжелых заболеваний; клинические данные приведены в таблице 1. - Исключить беременных женщин, кормящих матерей, курильщиков и употреблять алкоголь.
2. Антропометрия и состав тела
- Измерьте вес участников с помощью цифровой антропометрической шкалы весом 200 кг. Обязательно снимите обувь и лишнюю одежду.
- Оцените высоту с помощью стадиометра с градацией 0,5 см. Участникам было поручено стоять босиком, ногами вместе и руками по бокам21.
- Соберите информацию о жировой массе (FM) с помощью электрического биоимпеданса. После 12 ч голодания оцените с помощью пустого мочевого пузыря. Поместите участников в положение лежа на спине, с расставленными ногами и параллельными руками, не касаясь тела. Попросите участников снять все металлические аксессуары22.
- Получите окружность талии (WC) с помощью нерасширяемой измерительной ленты с градуировкой 0,1 мм. Лента была размещена горизонтально вокруг средней талии, чуть выше тазобедренных костей. Измерение произошло сразу после выдоха. По данным Центров по контролю и профилактике заболеваний (CDC), порог WC для женщин составляет 88 см23.
Таблица 1: Характеристики населения. Все переменные были параметрическими (p > 0,05, Шапиро Уилк), а различия оценивались с помощью независимого t-теста, в котором p < 0,05 считалось значимым. *p < 0,0001. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
3. Сбор биологического материала
- Собирают периферическую кровь после 12 ч голодания, как описано ранее24.
- Соберите 4 мл образцов цельной крови в пробирках с антикоагулянтом (например, ЭДТА) и храните их на льду для транспортировки.
4. Извлечение ДНК
- Центрифугируйте каждую пробирку, содержащую кровь по 2000 х г в течение 10 мин. Соберите около 300 мкл пышной шерсти (белая интерфаза).
- Извлеките ДНК из периферической крови с помощью коммерчески доступного инструмента (см. Таблицу материалов), как описано ранее25. Элюировать ДНК в 480 мкл сверхчистого H2O.
- Оцените концентрацию и качество ДНК с помощью флуорометра. Целостность оценивали с помощью анализа 1% агарозного геля25. Хранить в морозильной камере при -80 °C.
5. Подготовка перед анализом метилирования
- Убедитесь, что образцы имеют минимальную концентрацию ДНК (500 нг является идеальным начальным входом для начала), но разбавление может варьироваться между образцами на этом этапе.
- Некоторые реагенты не входят в комплект микрочипов26, поэтому приобретайте их отдельно27.
- Проверьте, содержит ли в комплекте все реагенты, используемые во время процедуры; они незаменимы. Проверьте срок годности всех реагентов. Убедитесь, что другие реагенты и растворы, не предусмотренные в наборе, также доступны (95% формамид /1 мМ ЭДТА, 0,1 N NaOH, абсолютный этанол, 2-пропанол).
ВНИМАНИЕ: Формамид является летучим реагентом; качество результатов может быть улучшено, если продукт свежий. Целостность и качество формамида и абсолютного этанола считаются критическими, поскольку они могут повлиять на процесс окрашивания, предложенный производителем. Выбираемые спирты должны быть сверхчистыми продуктами, специфичными для молекулярно-биологического анализа. - После получения комплекта микрочипов загрузите карты декодирования микросхем (DMAP). Эти файлы доступны на веб-сайте производителя через 24-48 часов после отправки комплекта и исключаются на срок годности28. Чтобы загрузить указанные файлы, выполните следующие действия.
- Убедитесь, что компьютер с доступом в Интернет доступен. Выполните загрузку с помощью программного клиента декодирования файлов.
- Перед установкой программы убедитесь, что компьютер имеет исходящий и входящий доступ, предоставленный учреждением; Клиент декодирования файлов не имеет ограничений безопасности.
- Войдите в программное обеспечение Decode File Client с помощью опции Access by BeadChip. Этот вход выполняется с использованием идентификатора пользователя MyIllumina и пароля. Регистрация на этой странице обязательна28.
- После входа в Клиент декодирования файлов введите информацию, указанную ниже, в соответствующие поля на домашней странице программного обеспечения28: Список штрих-кодов; Список идентификаторов коробок; Номер заказа на поставку (PO); и номер заказа на продажу (не забывайте включать буквы, которые следуют за цифрами).
- Выберите DMAP для загрузки в соответствии с номером штрих-кода в поле покупки и укажите место назначения в диалоговом окне, чтобы сохранить DMAP.
- Нажмите кнопку, чтобы начать загрузку.
ПРИМЕЧАНИЕ: В конце загрузки убедитесь, что загруженные папки не пусты, и храните файлы в безопасности, так как они будут необходимы для последнего шага для преобразования экспериментов в файлы данных интенсивности (файлы IDAT). После истечения срока действия Illumina может удалить данные DMAP из своей онлайн-базы данных.
6. Трубопровод метилирования ДНК
ПРИМЕЧАНИЕ: Эксперимент по метилированию ДНК разделен на 4 дня (рисунок 1). Следуйте рекомендациям и спецификациям производителя для получения точных результатов.
- День 1: Обработка бисульфита
- Начните с денатурации 500 нг геномной ДНК, затем добавьте конверсионные реагенты. Для выполнения этого шага следуйте рекомендациям наборов бисульфита и микрочипов29.
- Обработайте денатурированную ДНК 130 мкл бисульфитного конверсионного реагента в стандартной концентрации, предоставленной производителем.
- Инкубируйте трубку в термоциклере в течение 16 циклов при 95 °C в течение 30 с и 50 °C в течение 1 ч. Опустите термоциклер до 4 °C в течение 10 мин.
- Выполните этап промывки, добавив 100 мкл промывочного буфера, а затем центрифугирование на полной скорости в течение 30 с. Концентрация не предусмотрена в руководстве пользователя, и производитель стандартизирует объемы.
ПРИМЕЧАНИЕ: Этот реагент поставляется с комплектом и не нуждается в разбавлении или приготовлении.
- День 2: Амплификация - Анализ метилирования, ручной протокол
- Разогрейте печь гибридизации до 37 °C и дайте температуре уравновеситься.
- Нанесите этикетку со штрих-кодом на микропластинку объемом 0,8 мл.
- Разморозьте реагенты амплификации (Multi-Sample Amplification Mix 1, Random Primer Mix и Multi-Sample Amp Master Mix) при комнатной температуре (эти реагенты поставляются с комплектом и не нуждаются в разбавлении или приготовлении). Затем выполните мягкую инверсию по крайней мере в 10 раз, пока все содержимое трубок не будет перемешано.
- Распределите 20 мкл смеси для усиления нескольких образцов 1 (MA1) в колодцы пластины.
- Перенос 4 мкл образца ДНК с бисульфитной преобразованной пластины в соответствующие скважины на пластине 0,8 мл. На этом этапе запишите идентификатор оригинального образца ДНК на лабораторную форму слежения, соответствующую пластинчатым колодцам.
- Дозируйте 4 мкл 0,1 Н NaOH в каждую лунку пластины, содержащей смесь для амплификации нескольких образцов 1 (MA1) и образец ДНК.
- Запечатайте пластину пластиковым уплотнением.
- Вихрьте пластину, чтобы смешать реагенты с образцами при 1 600 об/мин в течение 1 мин. Инкубировать в течение 10 мин при комнатной температуре и аккуратно снять уплотнение.
- Пипетка 68 мкл случайной грунтовочной смеси (RPM) в каждую лунку пластины. Пипетка 75 мкл Multi-Sample Amp Master Mix (MSM) в каждую лунку пластины (без удаления предыдущего раствора). Используйте новое уплотнение для покрытия пластины.
ПРИМЕЧАНИЕ: Позаботьтесь о том, чтобы соединить колодцы с плитой и правильно сориентировать ее. - Вихрь герметичной пластины со скоростью 1 600 об/мин в течение 1 мин. Инкубировать в гибридизационной печи в течение 20-24 ч при 37 °C.
- День 3: Гибридизация - Анализ метилирования, ручной протокол
- Разогрейте блок до 37 °C.
- Разморозить содержимое трубки при комнатной температуре. Осторожно и осторожно инвертируйте трубки с раствором фрагментации (FMS) по крайней мере в 10 раз, чтобы тщательно перемешать содержимое.
- Осторожно снимите пластину с печи гибридизации. Импульсно-центрифуга пластины при 280 х г.
- Теперь снимите уплотнение с пластины. В каждую лунку пластины, содержащей образец, добавляют 50 мкл фрагментационного раствора (FMS) и снова запечатывают пластину. Вихрь пластины со скоростью 1 600 об/мин в течение 1 мин.
- Центрифугирование пластины при 280 x g в течение нескольких секунд (выполните импульсное центрифугирование). Затем поместите герметичную пластину на нагревательный блок при 37 °C в течение 1 ч.
- Если было решено приостановить эксперимент и заморозить пластину, разморозить ее при комнатной температуре и центрифугировать при 280 х г. Если он не заморожен, пропустите этот шаг и перейдите к шагу 6.3.7.
- Оставьте нагревательный блок для предварительного нагрева до 37 °C.
- Оставьте wash Buffer для размораживания при комнатной температуре. При размораживании инвертируйте его по крайней мере в 10 раз, чтобы перемешать содержимое.
- Снимите уплотнение с пластины.
- Добавьте 100 мкл осадков (PM1) к каждой лунке пластины, содержащей образцы.
- Снова запечатайте пластину пломбом.
- Вихрь пластины со скоростью 1 600 об/мин в течение 1 мин.
- Инкубировать при 37 °C в течение 5 мин.
- Затем поместите в импульсную центрифугу при 280 х г в течение 1 мин.
ПРИМЕЧАНИЕ: Установите центрифугу при 4 °C для следующего шага. - Осторожно снимите уплотнение с пластины и выбросьте его.
- Добавьте 300 мкл 2-пропанола (100%) в каждую скважину, содержащую образец.
- Запечатайте пластину с особой осторожностью, используя новое уплотнение. Следите за тем, чтобы не встряхнуть тарелку.
- Инвертируйте пластину не менее чем в 10 раз, перемешивая все содержимое.
- Инкубировать при 4 °C в течение 30 мин.
- Центрифуга при 3 000 х г при 4 °C в течение 20 мин.
ПРИМЕЧАНИЕ: В конце шага 6.3.20 немедленно снимите пластину центрифуги. - Снимите уплотнение с пластины и выбросьте его.
- Быстро переверните пластину и слейте жидкость на прокладку, чтобы выбросить супернатант. Затем ударьте по тарелке в сухом месте на бумажном полотенце, чтобы слить жидкость.
- Плотно постукивайте несколько раз в течение 1 мин или до тех пор, пока во всех колодцах не останется жидкости.
- Оставьте пластину перевернутой и непокрытой на 1 ч при комнатной температуре.
ПРИМЕЧАНИЕ: Ожидается увидеть синие гранулы на дне скважин. - Разогрейте печь гибридизации до 48 °C.
- Включите тепловой блок для предварительного нагрева. Подождите 20 минут для этого процесса.
- Оттаивайте раствор для ресусппензии, гибридизации и стирки при комнатной температуре. Инвертировать не менее 10x. Проверьте, чтобы в растворе не выпадала в осадок соль.
- Добавьте 46 мкл раствора для повторной суспензии, гибридизации и промывки (RA1) в каждую лунку пластины.
ПРИМЕЧАНИЕ: Все оставшиеся реагенты должны быть зарезервированы для этапов окрашивания и гибридизации. - Нанесите уплотнение на пластину. Используйте алюминиевое уплотнение для покрытия пластины. Держитесь плотно, чтобы выполнить герметизацию равномерно.
- Проверьте, все ли скважины надежно закрыты, чтобы избежать испарения.
- Аккуратно поместите недавно запечатанную пластину в печь гибридизации и инкубируйте при 48 °C в течение 1 ч.
- Вихрь пластины при 1 800 об/мин в течение 1 мин.
- Импульсная центрифуга при 280 х г.
- Перенесите пластину в тепловой блок при 95 °C в течение 1 ч.
- Подготовьте камеры гибридизации (Hyb), поместив их вместе. Распределите 400 мкл увлажняющего буфера (PB2) в резервуары Hyb-камер. Немедленно накройте крышку.
- Загрузите каждый образец с пластины в отверстие для входа в образец микрочипа.
- Загрузите вставки Hyb-камеры, содержащие микрочипы, в Hyb-камеру. Закройте камеру Hyb поперечно, чтобы избежать возможного смещения Hyb-камеры.
ПРИМЕЧАНИЕ: Гиб-камеры необходимо инкубировать при 48 °C в течение не менее 16 ч, но не более 24 ч.
- День 4: Окрашивание - Анализ метилирования, ручной протокол
- Повторно суспендируют раствор для окрашивания 4 330 мл 100% EtOH, получая конечный объем 350 мл. Энергично встряхните флакон с раствором для окрашивания 4, чтобы обеспечить полное повторное суспендирование. Хранить при комнатной температуре.
ПРИМЕЧАНИЕ: Окрашивающий раствор 4 можно хранить до 2 недель при температуре от 2 °C до 8 °C. - Извлеките камеры из печи гибридизации и дайте им остыть на скамейке в течение 30 минут перед открытием.
- Пока камера Hyb охлаждается, заполните два моечных контейнера с помощью Wash Buffer (200 мл на мойочный контейнер). Не забудьте пометить каждый контейнер как «Буфер промывки» (PB1 и PB2).
- Наполните аксессуары для выравнивания микрочипов 150 мл wash Buffer.
- Отделите пластиковые распорки и при необходимости очистите стекло.
- Чтобы промыть слайды микрочипа, прикрепите проволочную петлю к сетке и погрузите капающее оборудование в контейнеры с 200 мл Wash Buffer в общей сложности в 10 раз.
- Извлеките все вставки из камеры гибридизации и извлеките каждый массив из вкладышей. Снимите уплотнение.
ПРИМЕЧАНИЕ: Во время удаления не прикасайтесь к открытым матрицам. - При извлечении слайдов микрочипа из камеры гибридизации будьте осторожны, чтобы ничего не пролить.
- Мойте микрочипы в растворе буфера для стирки (PB1) медленно и легко, двигаясь вверх и вниз в 10 раз. Перейдите к свежему раствору PB1 и выполните промывку, медленно перемещая микрочипы в 10 раз вверх и вниз в растворе.
- Извлеките микрочип из контейнера и подтвердите отсутствие остатков.
- Используйте прозрачную распорку в верхней части каждого микрочипа и направьте распорки в соответствующие положения.
ПРИМЕЧАНИЕ: Будьте осторожны, используя только прозрачные распорки, а не белые. - Расположите планку выравнивания на аксессуаре выравнивания.
- Поместите стеклянную заднюю панель поверх прозрачной распорки; будьте осторожны, чтобы покрыть весь микрочип. Затем прикрепите металлические зажимы к проточным камерам. При необходимости используйте ножницы для обрезки концов проточной камеры распорок.
- Немедленно промыть резервуары камеры гибридизации с помощью ddH2O и почистить небольшой чистящей щеткой. Не допускайте, чтобы какой-либо увлажняющий буфер оставался в резервуаре.
- Перейдите к следующему шагу, отвечающему за микрочип расширения и окрашивания.
ПРИМЕЧАНИЕ: Оставьте все проточные камеры горизонтально установленными на лабораторном стенде. Повторно обрабатывайте их только тогда, когда весь этап окрашивания будет готов. Нагрейте раствор для повторной суспензии, гибридизации и промывки до температуры от 20 °C до 25 °C. Осторожно перемешайте до тех пор, пока в растворе не появится кристалл. - Поместите реагенты в стойку последовательно. Если они заморожены, оставьте их при комнатной температуре, а затем переверните их по крайней мере в 10 раз, чтобы смешать растворы.
- Наполните циркулятор воды до соответствующего уровня.
- Включите насос и установите температуру на 44 °C.
- Удалите все пузырьки, прикрепленные к стойке камеры.
- Выполните испытания на стойке камеры с помощью датчика температуры. Все тестируемые места должны находиться в диапазоне от 44 °C ± 0,5 °C.
ПРИМЕЧАНИЕ: Следующие шаги должны быть выполнены без перерыва. - Когда стойка достигнет температуры 44 °C, быстро поместите каждый узел в проточную камеру.
ПРИМЕЧАНИЕ: Для следующих шагов пипетка всех реагентов в заднюю стеклянную пластину. - Пипетка 150 мкл раствора для ресуспенсии, гибридизации и промывки (RA1). Затем инкубировать в течение 30 мин. Повторите этот шаг в общей сложности 5 раз.
- Пипетка 450 мкл окрашивающего раствора 1 (XC1) и инкубация в течение 10 мин.
- Пипетка 450 мкл окрашивающего раствора 2 (XC2) и инкубация в течение 10 мин.
- Пипетка 200 мкл двухцветного удлинительного мастер-микса (TEM) и инкубация в течение 15 мин.
- Пипетка 450 мкл 95% формамида/1 мМ ЭДТА и инкубация в течение 1 мин. Повторить дважды.
- Инкубировать в течение 5 мин.
- Снизить температуру камерной стойки до 32 °C (указано на Superior Two-Color Master Mix).
- Пипетка 450 мкл раствора для окрашивания 3 (XC3) и инкубация в течение 1 мин. Повторить 1x.
- Подождите, пока стойка в камере достигнет правильной температуры.
ПРИМЕЧАНИЕ: Чтобы прочитать изображение микрочипа сразу после процесса окрашивания, включите сканер на этом этапе. - Теперь дозируйте 250 мкл Superior Two-Color Master Mix (STM) с последующей 10-минутной инкубацией.
- Затем дозируют 450 мкл раствора для пятен 3 (XC3) с последующей инкубацией 1 мин. Повторите 1x.
- Подождите 5 минут, затем добавьте 250 мкл Anti-Stain 2-Color Master Mix (ATM) и инкубируйте в течение 10 минут.
- Теперь дозируйте 250 мкл Superior Two-Color Master Mix (STM) с последующей 10-минутной инкубацией.
- Затем дозируют 450 мкл раствора для пятен 3 (XC3) с последующей инкубацией 1 мин. Повторите 1x.
- Подождите 5 мин.
- Добавьте 250 мкл Anti-Stain 2-Color Master Mix (ATM) и инкубируйте в течение 10 минут.
- Добавьте 450 мкл раствора 3 (XC3) и инкубируйте в течение 1 мин, затем повторите снова.
- Подождите 5 мин.
ПРИМЕЧАНИЕ: Выполните следующую схему повторения: Этап 6.4.34, Этап 6.4.35. и этап 6.4.36; Этап 6.4.37, 6.4.38 и этап 6.4.39; Этап 6.4.34, этап 6.4.35 и этап 6.4.36. - Теперь сразу же снимите проточные камеры со стойки и аккуратно разместите их горизонтально на лабораторном стенде. Убедитесь, что температура окружающей среды.
- 6.4.41.Поместите 310 мл моющего раствора (ПБ1) на каждый микрочип в контейнер для мойки.
- Снимите металлические зажимы и стекло, и, наконец, микрочип.
- Поместите микрочипы на стойку для окрашивания и поместите их в контейнер для мойки, содержащий 10 растворов PB1. Убедитесь, что штрих-коды обращены в сторону от того, кто их обрабатывает, и что все микрочипы погружены в воду.
- Двигайтесь медленно и легко с движениями вверх и вниз 10x. Микрочип должен быть полностью извлечен из раствора, прежде чем снова погрузиться в воду. Затем дайте ему оставаться под водой в течение 5 минут.
- Энергично встряхните реагент Stain Solution 4 (XC4), чтобы обеспечить полное повторное суспендирование. Поместите 310 мл XC4 в контейнер для мойки.
ПРИМЕЧАНИЕ: Не оставляйте раствор в контейнере для мытья более 10 минут. - Слегка переместите подставку для окрашивания в другой контейнер с помощью XC4.
- Двигайтесь медленно и легко с движениями вверх и вниз 10x. Убедитесь, что микрочип полностью извлечен из раствора перед повторным погружением.
- Затем дайте микрочипу оставаться погруженным в течение 5 минут.
- Выдвиньте пятно из раствора и поместите его в держатель трубки со штрих-кодами вверх.
- Аккуратно извлеките микрочип с помощью фиксирующих щипцов и положите их на полку для высыхания.
- Сушите их с помощью вакуумного осушителя в течение 50-55 мин при 675 мм рт.ст. (0,9 бар).
- Очистите заднюю часть каждого микрочипа с помощью EtOH.
ВНИМАНИЕ: Избегайте прикосновения к полосам и не позволяйте EtOH капать на полосы каким-либо образом. - Перейдите к изображению микрочипа.
- Аккуратно храните микрочип в ящике для хранения при комнатной температуре.
ПРИМЕЧАНИЕ: Изображения микрочипов должны быть сделаны в течение 72 часов.
- Повторно суспендируют раствор для окрашивания 4 330 мл 100% EtOH, получая конечный объем 350 мл. Энергично встряхните флакон с раствором для окрашивания 4, чтобы обеспечить полное повторное суспендирование. Хранить при комнатной температуре.
7. Биоинформатический анализ
ПРИМЕЧАНИЕ: Необходимо изменить некоторые атрибуты типа микрочипа (например, arraytype = "450k" следует заменить на arraytype = "EPIC"). Автор конвейера ChAMP подробно описывает все поля, которые необходимо модифицировать для анализа массивов на странице Bioconductor пакета30.
- Образец листа
- Перенесите файлы IDAT на компьютер, чтобы продолжить анализ биоинформатики31. Создайте образец листа для выполнения анализа данных31.
ПРИМЕЧАНИЕ: Три столбца необходимы для этого файла электронной таблицы. Во-первых, это Sample_name. Этот столбец должен содержать имена или коды, используемые для идентификации образцов. Второе – это Sentrix_position. В этом столбце помещается соответствующий R01C01 образцов, причем R01 отвечает за строку чипа, а столбец называется C01. Третьим важным столбцом является Sentrix_ID, и он отвечает за получение номера, который идентифицирует микрочип. Убедитесь, что вы поставили правильные цифры. Можно добавить другую информацию на лист образца в виде значений переменных. В настоящий эксперимент были включены данные анализа металлов.
- Перенесите файлы IDAT на компьютер, чтобы продолжить анализ биоинформатики31. Создайте образец листа для выполнения анализа данных31.
- Трубопровод CHAMP
- Установите RStudio (V.4.0.2) на компьютер для выполнения анализа30.
ПРИМЕЧАНИЕ: Убедитесь, что компьютер имеет не менее 8 ГБ оперативной памяти. Анализ данных будет бороться или аварийно завершаться с меньшим объемом оперативной памяти. - После установки откройте RStudio и установите пакет ChAMP Bioconductor30 с помощью соответствующей командной строки (см. Дополнительный материал 1).
ПРИМЕЧАНИЕ: Если в конце установки возникает какая-либо ошибка, установите необходимые библиотеки ChAMP вручную, одну за другой26. - Теперь импортируйте необработанные данные и выполните анализ. Дополнительный материал 1.
champ.load() импортирует необработанные данные и выполняет процесс фильтрации, исключая зонды с низким уровнем обнаружения pval, многохитовые сайты, не-CG зонды, cpG-сайты рядом с SNP и CpG, расположенные в половых хромосомах.
champ.impute() выполняет условное исчисление KNN по умолчанию.
чавкать. QC извлекает графики качества данных (например, график плотности).
чавкать. SVD оценивает пакетные эффекты на основе предоставленных метаданных в образце листа.
champ.refbase() выполняет оценку и коррекцию типа ячейки.
чавкать. DMP( ) извлекает дифференциально метилированные положения и ассоциации признаков, такие как жировая масса, для проверки метилирования ДНК между группами и изменения параметров, как показано в Дополнительном материале 1.
- Установите RStudio (V.4.0.2) на компьютер для выполнения анализа30.
-
Обогащение (строковая база данных)
ПРИМЕЧАНИЕ: Функциональное обогащение используется для вывода биологических функций набора генов.- Чтобы обогатить данные в строке, выберите список целевых объектов для обогащения и использования в качестве входных данных из: https://string-db.org/cgi/input?sessionId=bywt5Gl8Bawy&input_page_active_form
=multiple_identifiers - Для организма выберите homo sapiens и нажмите кнопку Поиск.
- Для навигации по обогащению нажмите на кнопку Анализ.
- Чтобы обогатить данные в строке, выберите список целевых объектов для обогащения и использования в качестве входных данных из: https://string-db.org/cgi/input?sessionId=bywt5Gl8Bawy&input_page_active_form
-
Подача заявки на ГСО
- Депонируйте данные в общедоступный репозиторий, такой как GEO NCBI. Для этого зарегистрируйте учетную запись отправителя.
ПРИМЕЧАНИЕ: Завершите представление ГЭП (https://submit.ncbi.nlm.nih.gov/geo/submission/), загрузив лист метаданных с описательной информацией и протоколами для всего эксперимента и отдельных образцов. АрхивНЫЕ материалы GEO могут быть созданы в любом программном обеспечении для работы с электронными таблицами. Во-вторых, добавьте в каталог необработанный файл данных, созданный на основе сценария подсчета счетчика ячеек для всех библиотек. IDAT и связанные с ним файлы или матричный лист содержат ненормализованные данные. Третья папка — это обработанные файлы данных. Матричная таблица представляет собой электронную таблицу, содержащую окончательные, нормализованные значения, сопоставимые по строкам и образцам и предпочтительно обработанные, как описано в любой сопроводительной рукописи. - Поместите в каталог обработанные файлы данных, сгенерированные из скрипта подсчета счетчика ячеек для всех библиотек. Используйте учетные данные FTP-сервера отправителя GEO для передачи каталога, содержащего все три компонента.
- Депонируйте данные в общедоступный репозиторий, такой как GEO NCBI. Для этого зарегистрируйте учетную запись отправителя.
Результаты
После использования конвейера ChAMP в анализе было рассмотрено 409 887 зондов после всех фильтров (неквалифицированные CpG, не-CG зонды, CpG вблизи SNP, многохитовые участки и CpG, связанные с XY); схема представлена на рисунке 2.
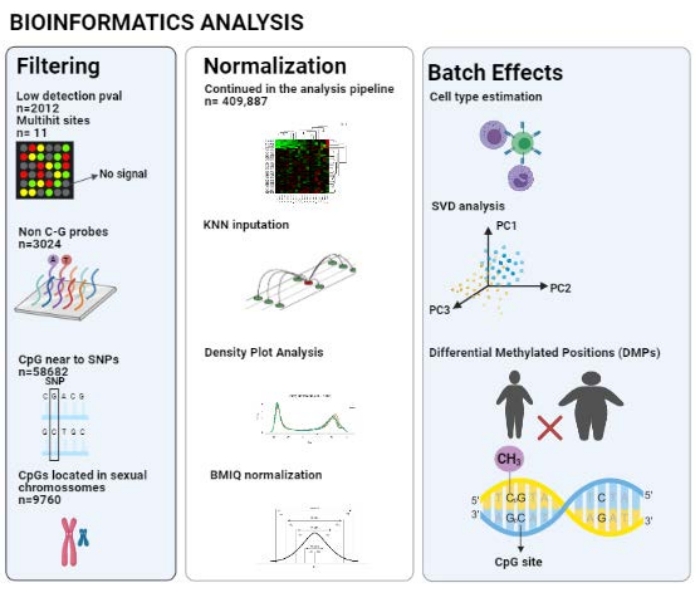
Рисунок 2: Конвейер анализа биоинформатики. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Кроме того, график плотности показал, что все образцы имели одинаковую плотность бета-распределения, связанную с этапами контроля качества. Этот анализ оценивает распределение бета-значений и указывает, есть ли какие-либо образцы, которые необходимо исключить. В этой партии на основе этого анализа не было исключено ни одного образца.
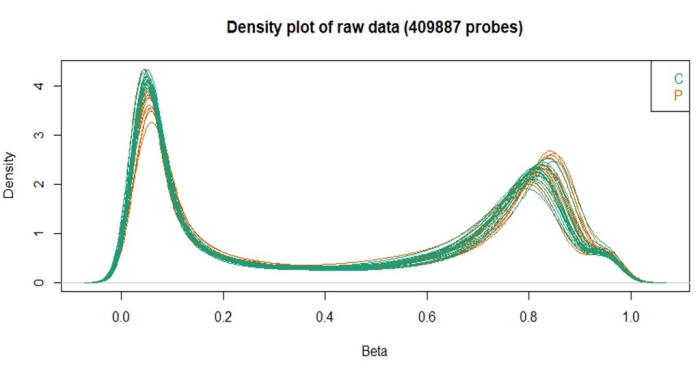
Рисунок 3: График плотности бета-значений, полученный с помощью пакета ChAMP. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Анализ разложения сингулярного значения (SVD) был использован для проверки основных компонентов, которые значительно повлияли на изменчивость данных метилирования ДНК. Данные показали, что ИМТ, WC (p < 1e10-5) и FM (p < 0,05) оказали значительное влияние на изменчивость данных (рисунок 4).
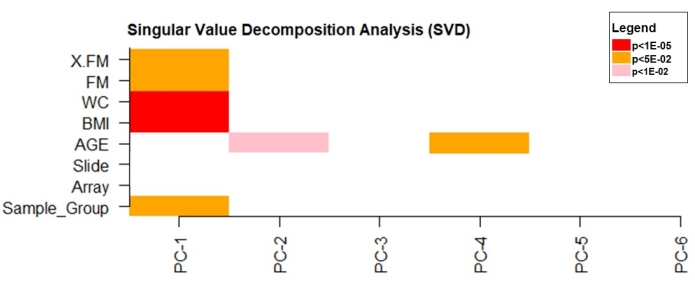
Рисунок 4: Анализ декомпозиции сингулярных значений. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Оценка типа клеток показала, что как естественные клетки-киллеры (NK), так и В-клетки были выше у женщин с ожирением (рисунок 5).
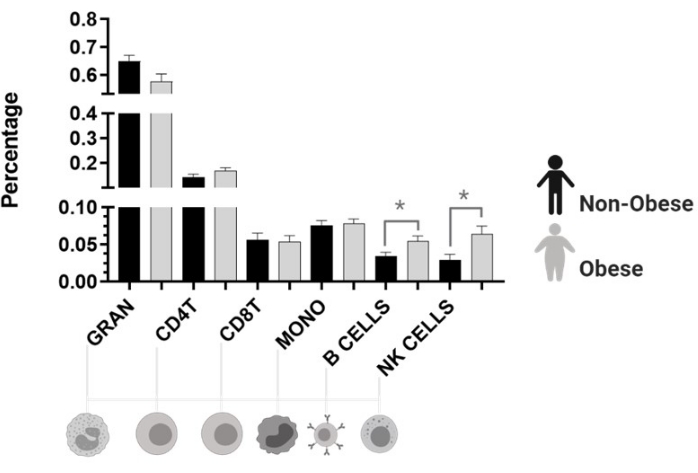
Рисунок 5: Фракции клеток, оцененные методом Хаусмана. Гран: гранулоцитарный. CD4T: хелпер Т-клетка, лимфоцит. CD8T: цитотоксические Т-клетки, лимфоциты. Моно: моноцит. В-клетка: В-лимфоцит. НК: естественные клетки-киллеры, лимфоциты. *p < 0,05. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Уровни метилирования ДНК между женщинами с ожирением и женщинами, не страдающими ожирением, различались до и после коррекции клеточного типа. До коррекции данных метилирования ДНК для типов клеток наблюдалось 43 463 дифференциально метилированных положения (DMP), а после этого оставалось 3 329 CpG. 445 участков CpG находились в межгенных регионах (IGR), а 2 884 были в генетических регионах, причем большинство из них находились в промоторной области (n = 1 438). Распределение по всем регионам было TSS1500 (n = 612), TSS200 (n = 826), 5'UTR (n = 390), первый экзон (n = 273), тело (n = 724) и 3'UTR (n = 59). Учитывая значения Δβ <-0,05 и >0,05, 162 CpG были гипометилированными и 576 были гиперметилированными у людей с ожирением по сравнению с лицами, не страдающими ожирением (рисунок 6). Данные доступны в базе данных GEO под регистрационным кодом GSE166611.
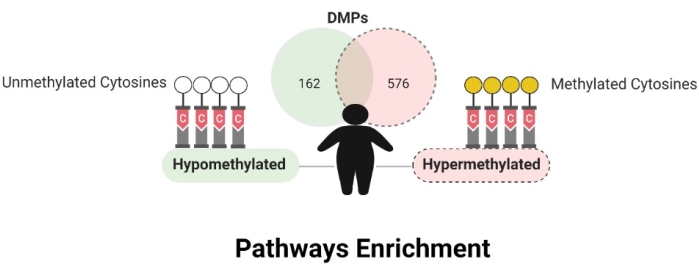
Рисунок 6: Дифференциально метилированные позиции. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Можно оценить различное метилирование между группами и найти участки CpG, связанные с конкретными признаками в исследованиях метилирования. Для исследований жировой массы было обнаружено 13 222 сайта CpG. 6 159 CpG в промоторной области были связаны с жировой массой, с 470 гиперметилированными и 94 гипометилированными (рисунок 7), и соответствующие гены были обогащены (таблица 2).
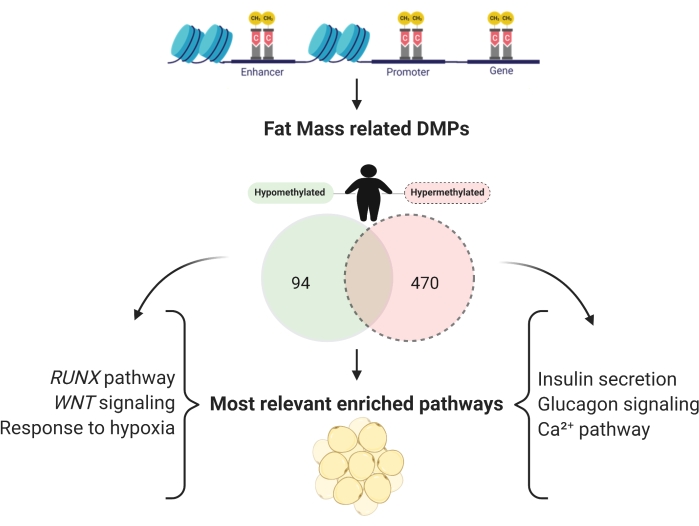
Рисунок 7: Промоторные области гипометилированных и гиперметилированных генов, независимо связанные с жировой массой. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Таблица 2: Функциональное обогащение генов из дифференциально метилированных участков CpG. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
Дополнительный материал 1: Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
Массивы метилирования ДНК являются наиболее часто используемыми методами доступа к метилированию ДНК из-за их соотношения затрат и выгод14. В настоящем исследовании описан подробный протокол с использованием коммерчески доступной платформы микрочипов для оценки метилирования ДНК в пилотном исследовании, проведенном в бразильской когорте. Полученные результаты пилотного исследования подтвердили эффективность протокола. На рисунке 3 показана сопоставимость образца и полная конверсия бисульфита32.
В качестве этапа контроля качества алгоритм ChAMP рекомендовал исключить сайты CpGs в процессе фильтрации. Цель исключения зондов заключается в улучшении анализа данных и устранении предвзятости. Низкокачественные CpG (p-значения ниже 0,05) были удалены для устранения экспериментального шума в наборе данных. Цели оставались пройденными при анализе плотностного графика. Zhou33 описал важность фильтрации CpG вблизи SNP, чтобы избежать несоответствий, неправильной интерпретации метилирования полиморфных цитозинов и изменения цвета конструкции зонда типа I34. Кроме того, поскольку на хромосомы XY дифференцированно влияет импринтинг, Heiss и Just35 подтвердили важность фильтрации этих зондов, потому что у самок проблемы с гибридизацией могут быть смешанными факторами35.
Дата истечения срока годности DMAP, дата открытия формамида, аналитическое качество абсолютного этанола и общее количество лейкоцитов считаются критическими шагами в протоколе.
Кроме того, согласно нашим наблюдениям, оценка типа клеток имеет важное значение при проведении биоинформатического анализа. Метод Хаусмана выполняет оценку типа клеток, как описано в исследовании Тяня30. Этот метод основан на 473 специфических сайтах CpG, которые могут предсказать процентное соотношение наиболее важных типов клеток, таких как гранулоциты, моноциты, В-клетки и Т-клетки36. Мы использовали рекомендуемую функцию "myRefbase" из пакета ChAMP. После оценки алгоритм ChAMP корректирует бета-значения и устраняет это смещение из набора данных. Этот шаг имеет решающее значение в исследованиях, посвященных ожирению, потому что эта популяция имеет значительную разницу в белых кровяных клетках из-за их хронического воспалительного состояния.
Мы изменили только оригинальную карту колпачков для общего уплотнения ПЦР в отношении модификации метода и устранения неполадок. После каждого процесса центрифугирования уплотнение меняли на новое. Мы не смогли использовать стандартное термоуплотнение и адаптировали его с помощью алюминиевой фольги вокруг пластины.
Хотя коммерческие анализы считались золотым стандартом для эпигенетических исследований, одним из ограничений протокола может быть специфичность реагентов и оборудования от уникального бренда37,38,39,40. Другим ограничением является отсутствие показателей, позволяющих определить правильный ход эксперимента41.
Стандартизация настоящего протокола представляет собой отличное руководство для эпигенетических исследований, уменьшая человеческие ошибки во время процесса и обеспечивая успешный анализ данных и сопоставимость между различными исследованиями.
Согласно нашим результатам, эксперименты по метилированию ДНК подходят для исследований, сравнивающих людей с ожирением и без него43. Кроме того, предлагаемый биоинформатический анализ обеспечивает высококачественные данные и может быть рассмотрен в крупномасштабных исследованиях.
Используя анализ SVD, мы определили, что признаки, связанные с ожирением (ИМТ, WC и FM), влияют на вариабельность данных метилирования ДНК. В качестве значимого результата оценка клеточного типа показывает, что как естественные клетки-киллеры (NK), так и В-клетки были выше у женщин с ожирением, чем у женщин без ожирения (рисунок 5). Более высокое количество этих клеток может быть объяснено низкосортным воспалительным состоянием этих людей44. Мы наблюдали, что пациенты с ожирением имеют гипо- и гиперметилированные CpG в промоторных областях генов, связанных с жировой массой. Большинство участков были гипометилированными, что может быть связано с естественным увеличением уровня активных форм кислорода (АФК) у этих людей. Это состояние окислительного стресса может способствовать возмущению гуанина в месте динуклеотида, образуя 8-гидрокси-2'-дезоксигуанозин (8-OHdG), что приводит к образованию 5mCp-8-OHdG динуклеотидного сайта и вызывает рекрутирование ферментов TET. Все эти события могут быть ответственны за стимулирование гипометилирования и гиперметилирования ДНК различными механизмами действия45.
Кроме того, скорость адипогенеза, по-видимому, увеличивается у людей с ожирением, причем примерно 10% новых клеток к старым клеткам46,47. Эпигенетические вклады, подчеркивая обесогенную среду, могут изменять скорость пролиферации и дифференцировки клеток, способствуя развитию жировой массы48. Эпигенетические изменения могут также влиять на адипогенные программы, облегчая или ограничивая их развитие. Первичные факторы транскрипции (PPARγ или C/EBPα) или сборка мультипротеиновых комплексов, расположенных в последующих промоторных областях, управляемых включением или исключением эпигенетических модифицирующих ферментов, регулируют экспрессию генов посредством гипер- или гипометилирования45. Путь PPARγ был ранее описан для изменения пути WNT, который имел гены, обогащенные в этом исследовании. Хотя до сих пор неизвестно, как передача сигналов WNT происходит во время адипогенеза, недавние исследования показали, что она может играть важную роль в метаболизме адипоцитов, особенно в обесогенных условиях49.
Раскрытие информации
Авторы этой статьи не имеют конфликта интересов и не имеют финансовой информации для декларирования.
Благодарности
Мы хотели бы поблагодарить Юаня Тяня, доктора философии (tian.yuan@ucl.ac.uk) за то, что он готов ответить на все сомнения относительно пакета ChAMP. Мы также благодарим Гильерме Теллеса, магистра наук, за его вклад как в технические, так и в научные вопросы, затронутые в настоящем документе; он высказал важные соображения относительно эпигенетики и методов захвата и форматирования видео (guilherme.telles@usp.br). Финансирование расходных материалов: Исследовательский фонд Сан-Паулу (FAPESP) (#2018/24069-3) и Национальный совет по научно-техническому развитию (CNPq: #408292/2018-0). Личное финансирование: (FAPESP: #2014/16740-6) и Программа академического превосходства от Координации для развития персонала высшего образования (CAPES: 88882.180020/2018-01). Данные будут общедоступными и свободно доступными без ограничений. Адрес корреспонденции в NYN (e-mail: nataliayumi@usp.br) или CBN (e-mail: carla@fmrp.usp.br).
Материалы
| Name | Company | Catalog Number | Comments |
| Absolute ethanol | J.T. Baker | B5924-03 | |
| Agarose gel | Kasvi | K9-9100 | |
| Electric bioimpedance | Quantum BIA 450 Q - RJL System | ||
| Ethylenediaminetetraacetic acid (EDTA) | Corning | 46-000-CI | |
| EZ DNA Methylation-Gold kit | ZymoResearch, Irvine, CA, USA | D5001 | |
| Formamide | Sigma | F9037 | |
| FMS—Fragmentation solution | Illumina | 11203428 | Supplied Reagents |
| HumanMethylation450 BeadChip | Illumina | ||
| Maxwell Instrument | Promega, Brazil | AS4500 | |
| MA1—Multi-Sample Amplification 1 Mix | Illumina | 11202880 | Supplied Reagents |
| MicroAmp Optical Adhesive Film | Thermo Fisher Scientific | 201703982 | |
| MSM—Multi-Sample Amplification Master Mix | Illumina | 11203410 | Supplied Reagents |
| NaOH | F. MAIA | 114700 | |
| PB1—Reagent used to prepare BeadChips for hybridization | Illumina | 11291245 | Supplied Reagents |
| PB2—Humidifying buffer used during hybridization | Illumina | 11191130 | Supplied Reagents |
| 2-propanol | Emsure | 10,96,34,01,000 | |
| RA1—Resuspension, hybridization, and wash solution | Illumina | 11292441 | Supplied Reagents |
| RPM—Random Primer Mix | Illumina | 15010230 | Supplied Reagents |
| STM—Superior Two-Color Master Mix | Illumina | 11288046 | Supplied Reagents |
| TEM—Two-Color Extension Master Mix | Illumina | 11208309 | Supplied Reagents |
| Ultrapure EDTA | Invitrogen | 155576-028 | |
| 96-Well Reaction Plate with Barcode (0.1mL) | ByoSystems | 4346906 | |
| 96-Well Reaction Plate with Barcode (0.8mL) | Thermo Fisher Scientific | AB-0859 | |
| XC1—XStain BeadChip solution 1 | Illumina | 11208288 | Supplied Reagents |
| XC2—XStain BeadChip solution 2 | Illumina | 11208296 | Supplied Reagents |
| XC3—XStain BeadChip solution 3 | Illumina | 11208392 | Supplied Reagents |
| XC4—XStain BeadChip solution 4 | Illumina | 11208430 | Supplied Reagents |
Ссылки
- Manna, P., Jain, S. Obesity, oxidative stress, adipose tissue dysfunction, and the associated health risks: causes and therapeutic strategies. Metabolic Syndrome and Related Disorders. 13 (10), 423-444 (2015).
- . Obesity Available from: https://www.who.int/health-topics/obesity#tab=tab_1 (2017)
- Adult Obesity Facts. Center for Disease Control and Prevention Available from: https://www.cdc.gov/obesity/data/adult.html (2021)
- Bird, A. Perceptions of epigenetics. Nature. 396, (2007).
- Kouzarides, T. Chromatin modifications, and their function. Cell. 128 (4), 693-705 (2007).
- Berger, F. The strictest usage of the term epigenetic. Seminars in Cell & Developmental Biology. 19 (6), 525-526 (2008).
- Henikoff, S., Greally, J. M. Epigenetics, cellular memory, and gene regulation. Current Biology. 26 (14), 644-648 (2016).
- Laker, R. C., et al. Transcriptomic and epigenetic responses to short-term nutrient-exercise stress in humans. Scientific Reports. 7 (1), 1-12 (2017).
- Wahl, S., et al. Epigenome-wide association study of body mass index, and the adverse outcomes of adiposity. Nature. 541, 81-86 (2017).
- Jin, B., Robertson, K. D. DNA methyltransferases, DNA damage repair, and cancer. Epigenetic Alterations in Oncogenesis. 754, 3-29 (2013).
- Jang, H. S., Shin, W. J., Lee, J. E., Do, J. T. CpG and non-CpG methylation in epigenetic gene regulation and brain function. Genes. 8 (6), 148 (2017).
- Shen, L., Waterland, R. A. Methods of DNA methylation analysis. Current Opinion in Clinical Nutrition & Metabolic Care. 10 (5), 576-581 (2007).
- Olkhov-Mitsel, E., Bapat, B. Strategies for discovery and validation of methylated and hydroxymethylated DNA biomarkers. Cancer Medicine. 1, 237-260 (2012).
- Kurdyukov, S., Bullock, M. DNA methylation analysis: choosing the right method. Biology. 5 (1), 3 (2016).
- Yong, W. -. S., Hsu, F. -. M., Chen, P. -. Y. Profiling genome-wide DNA methylation. Epigenetics & Chromatin. 9 (1), 1-16 (2016).
- Dugué, P. A., et al. Alcohol consumption is associated with widespread changes in blood DNA methylation: Analysis of cross-sectional and longitudinal data. Addiction Biology. 1, 12855 (2021).
- Colicino, E., et al. Blood DNA methylation sites predict death risk in a longitudinal study of 12, 300 individuals. Aging. 14, 14092-14124 (2020).
- Karlsson, L., Barbaro, M., Ewing, E., Gomez-Cabrero, D., Lajic, S. Genome-wide investigation of DNA methylation in congenital adrenal hyperplasia. The Journal of Steroid Biochemistry and Molecular Biology. 201, 105699 (2020).
- Ribeiro, R. R., Guerra-Junior, G., de Azevedo Barros-Filho, A. Bone mass in schoolchildren in Brazil: the effect of racial miscegenation, pubertal stage, and socioeconomic differences. Journal of Bone and Mineral Metabolism. 27 (4), 494-501 (2009).
- Filozof, C., et al. Obesity prevalence and trends in Latin-American countries. Obesity Reviews. 2 (2), 99-106 (2001).
- Chadid, S., Kreger, B. E., Singer, M. R., Bradlee, M. L., Moore, L. L. Anthropometric measures of body fat and obesity-related cancer risk: sex-specific differences in Framingham Offspring Study adults. International Journal of Obesity. 44 (3), 601-608 (2020).
- Nicoletti, C. F., et al. DNA methylation pattern changes following a short-term hypocaloric diet in women with obesity. European Journal of Clinical Nutrition. 74 (9), 1345-1353 (2020).
- Assessing Your Weight. Centers for Disease Control and Prevention Available from: https://www.cdc.gov/healthyweight/assessing/index.html (2020)
- Giavarina, D., Lippi, G. Blood venous sample collection: Recommendations overview and a checklist to improve quality. Clinical Biochemistry. 50 (10-11), 568-573 (2017).
- Duijs, F. E., Sijen, T. A rapid and efficient method for DNA extraction from bone powder. Forensic Science International: Reports. 2, 100099 (2020).
- . Infinium HD Methylation Assay, Manual Protocol Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/infinium_assays/infinium_hd_methylation/infinium-hd-methylation-guide-15019519-01.pdf (2015)
- Serrano, J., Snuderl, M. Whole genome DNA methylation analysis of human glioblastoma using Illumina BeadArrays. Glioblastoma. , 31-51 (2018).
- Leti, F., Llaci, L., Malenica, I., DiStefano, J. K. Methods for CpG methylation array profiling via bisulfite conversion. Disease Gene Identification. 1706, 233-254 (2018).
- Noble, A. J., et al. A validation of Illumina EPIC array system with bisulfite-based amplicon sequencing. PeerJ. 9, 10762 (2021).
- Tian, Y., et al. ChAMP: updated methylation analysis pipeline for Illumina BeadChips. Bioinformatics. 33, 3982-3984 (2017).
- Turinsky, A. L., et al. EpigenCentral: Portal for DNA methylation data analysis and classification in rare diseases. Human Mutation. 41 (10), 1722-1733 (2020).
- . The Chip Analysis Methylation Pipeline Available from: https://www.bioconductor.org/packages/release/bioc/vignettes/ChAMP/inst/doc/ChAMP.html (2020)
- Zhou, W., Laird, P. W., Shen, H. Comprehensive characterization, annotation and innovative use of Infinium DNA methylation BeadChip probes. Nucleic Acids Research. 45 (4), 22 (2017).
- Wanding, Z., Triche, T. J., Laird, P. W., Hui, S. SeSAMe: reducing artifactual detection of DNA methylation by Infinium BeadChips in genomic deletions. Nucleic Acids Research. 46 (20), 123 (2018).
- Heiss, J. A., Just, A. C. Improved filtering of DNA methylation microarray data by detection p values and its impact on downstream analyses. Clinical Epigenetics. 11, 15 (2019).
- Houseman, E. A., et al. DNA methylation arrays as surrogate measures of cell mixture distribution. BMC Bioinformatics. 13, 86 (2012).
- Valavanis, I., Sifakis, E. G., Georgiadis, P., Kyrtopoulos, S., Chatziioannou, A. A. A composite framework for the statistical analysis of epidemiological DNA methylation data with the Infinium human Methylation 450K BeadChip. IEEE Journal of Biomedical and Health Informatics. 18 (3), 817-823 (2014).
- Sun, N., Zhang, J., Zhang, C., Shi, Y., Zhao, B., Jiao, A., Chen, B. Using Illumina Infinium HumanMethylation 450K BeadChip to explore genomewide DNA methylation profiles in a human hepatocellular carcinoma cell line. Molecular Medicine Reports. 18 (5), 4446-4456 (2018).
- Moran, S., Arribas, C., Esteller, M. Validation of a DNA methylation microarray for 850,000 CpG sites of the human genome enriched in enhancer sequences. Epigenomics. 8 (3), 389-399 (2016).
- Bibikova, M., et al. High density DNA methylation array with single CpG site resolution. Genomics. 98 (4), 288-295 (2011).
- Lehne, B., et al. A coherent approach for analysis of the Illumina HumanMethylation450 BeadChip improves data quality and performance in epigenome-wide association studies. Genome Biology. 16 (1), 1-12 (2015).
- Wang, J., Zhang, H., Rezwan, F. I., Relton, C., Arshad, S. H., Holloway, J. W. Pre-adolescence DNA methylation is associated with BMI status change from pre- to post-adolescence. Clinical Epigenetics. 25, 64 (2021).
- Maugeri, A. The effects of dietary interventions on DNA methylation: implications for obesity management. International Journal of Molecular Sciences. 21, 8670 (2020).
- DeFuria, J., et al. B cells promote inflammation in obesity and type 2 diabetes through regulation of T-cell function and an inflammatory cytokine profile. Proceedings of the National Academy of Sciences. 110 (13), 5133-5138 (2013).
- Kietzmann, T., Petry, A., Shvetsova, A., Gerhold, J. M., Görlach, A. The epigenetic landscape related to reactive oxygen species formation in the cardiovascular system. British Journal of Pharmacology. 174, 1533-1554 (2017).
- Chase, K., Sharma, R. P. Epigenetic developmental programs and adipogenesis: implications for psychotropic induced obesity. Epigenetics. 8 (11), 1133-1140 (2013).
- Spalding, K. L., et al. Dynamics of fat cell turnover in humans. Nature. 453, 783-787 (2008).
- Ross, S. E., et al. Inhibition of adipogenesis by Wnt signaling. Science. 289, 950-953 (2000).
- Bagchi, D. P., et al. Wnt/β-catenin signaling regulates adipose tissue lipogenesis and adipocyte-specific loss is rigorously defended by neighboring stromal-vascular cells. Molecular Metabolism. 42, 101078 (2020).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены