Method Article
DNA 메틸화의 생물 정보학 분석에 대한 샘플 준비 : 비만 및 관련 특성 연구를위한 협회 전략
* 이 저자들은 동등하게 기여했습니다
요약
본 연구는 마이크로어레이 기술로 얻은 DNA 메틸화 데이터를 관리하기 위한 워크플로우를 기술한다. 이 프로토콜은 샘플 준비에서 데이터 분석에 이르는 단계를 보여 줍니다. 모든 절차가 자세히 설명되어 있으며 비디오에서는 중요한 단계를 보여줍니다.
초록
비만은 생활 양식과 직접 관련이 있으며 질병의 발달에 기여하는 지방 생성 및 지질 저장 과정의 변화를 일으킬 수있는 DNA 메틸화 변화와 관련이 있습니다. 우리는 선택에서 비만 유무에 관계없이 환자의 후성 유전 학적 데이터 분석에 이르기까지 완벽한 프로토콜을 보여줍니다. 프로토콜의 모든 단계는 파일럿 연구에서 테스트되고 검증되었습니다. 32 명의 여성이 연구에 참여했으며, 15 명의 개인이 체질량 지수 (BMI) (45.1 ± 5.4 kg / m2)에 따라 비만으로 분류되었습니다. 17 개인은 BMI (22.6 ± 1.8 kg / m2)에 따라 비만없이 분류되었습니다. 비만이 있는 그룹에서, 지방량과 관련된 564개의 CpG 부위가 선형 회귀 분석에 의해 확인되었다. CpG 부위는 프로모터 영역에 있었다. 차등 분석은 비만 환자에서 470 CpGs 하이포 메틸화 및 94 하이퍼 메틸화 부위를 발견했습니다. 가장 저메틸화 된 농축 경로는 RUNX, WNT 신호 전달 및 저산소증에 대한 반응이었습니다. 하이퍼메틸화 경로는 인슐린 분비, 글루카곤 신호전달, 및 Ca2+와 관련되었다. 우리는 프로토콜이 DNA 메틸화 패턴과 형질 관련 DNA 메틸화를 효과적으로 확인했다고 결론지었다. 이러한 패턴은 변경된 유전자 발현과 관련이있을 수 있으며, 지방 생성 및 지질 저장에 영향을 줄 수 있습니다. 우리의 결과는 비만 성 생활 방식이 인간 DNA의 후성 유전 적 변화를 촉진 할 수 있음을 확인했습니다.
서문
대규모 오믹스 기술은 만성 질환 연구에 점점 더 많이 사용되고 있습니다. 이러한 방법의 흥미로운 특징은 많은 양의 생성 된 데이터를 과학계에 제공한다는 것입니다. 따라서 연구 간의 기술적 비교를 허용하기 위해 프로토콜을 표준화하려는 요구가 제기되었습니다. 본 연구는 파일럿 연구를 적용 사례로 사용하여 DNA 메틸화 데이터를 획득하고 분석하기위한 프로토콜의 표준화를 제안합니다.
부정적인 에너지 소비는 현대 인간의 생활 방식에서 우세하며, 지방 조직의 과도한 축적과 결과적으로 비만의 발달로 이어집니다. 많은 요인들이 좌식증, 고칼로리 다이어트 및 스트레스가 많은 일상과 같은 비만율을 증가 시켰습니다. 세계 보건기구 (WHO)는 2016 년에 1.9 억 명의 성인이 비만이라고 추정했는데, 이는 세계 인구의 20 % 이상이 30kg / m2 이상의 BMI2를 가지고 있음을 의미합니다. 2018의 가장 최근의 업데이트에 따르면 미국 (미국)에서 비만의 유병률이 42 % 3보다 높았다.
후성유전학은 변화된 활동 상태를 등록, 신호 또는 영속화하기 위해 염색체 영역의 구조적 적응이다4. DNA 메틸화는 시토신-구아노신 디뉴클레오타이드 부위(CpG 부위)에서 가역적인 화학적 변화로, 5-메틸시토신-pG(5mCpG)를 형성한다. DNA5,6,7,8에 대한 전사 기계의 접근을 조절함으로써 유전자 발현을 조절할 수 있다. 이러한 맥락에서, 어떤 CpG 부위가 비만 관련 특성과 연관되어 있는지 이해하는 것이 필수적이다9. 많은 인자가 부위-특이적 DNA 메틸화를 지지하거나 예방할 수 있다. DNA 메틸트랜스퍼라제10(DNTMs) 및 10-10-17 전좌(TETs)와 같은 이 과정에 필요한 효소는 환경 노출하에서 DNA 메틸화 또는 탈메틸화를 촉진할 수 있다11.
지난 몇 년 동안 DNA 메틸화 연구에 대한 관심이 높아짐에 따라 각 질문에 정확하게 대답 할 수있는 가장 적절한 분석 전략을 선택하는 것이 연구자의 필수 관심사였습니다.12,13,14. 450K DNA 메틸화 어레이는 DNA 메틸화 프로파일을 결정하기 위해 360개 이상의 간행물14에서 사용되는 가장 널리 사용되는 방법입니다. 알려진 유전자의 99%에 위치한 최대 485,000 CpGs의 메틸화를 결정할 수 있다15. 그러나 이 어레이는 중단되고 EPIC로 대체되어 850,000개의 CpG 사이트를 커버합니다. 본 프로토콜은 450K 및 EPIC16,17,18 모두에 적용될 수 있다.
이 프로토콜은 도 1 에서 단계별로 제시되며 집단 선택, 샘플링, 실험 준비, DNA 메틸화 파이프라인 및 생물정보학 분석과 같은 단계로 구성됩니다. 우리 실험실에서 수행 된 파일럿 연구는 제안 된 프로토콜의 단계를 설명하기 위해 여기에서 시연됩니다.
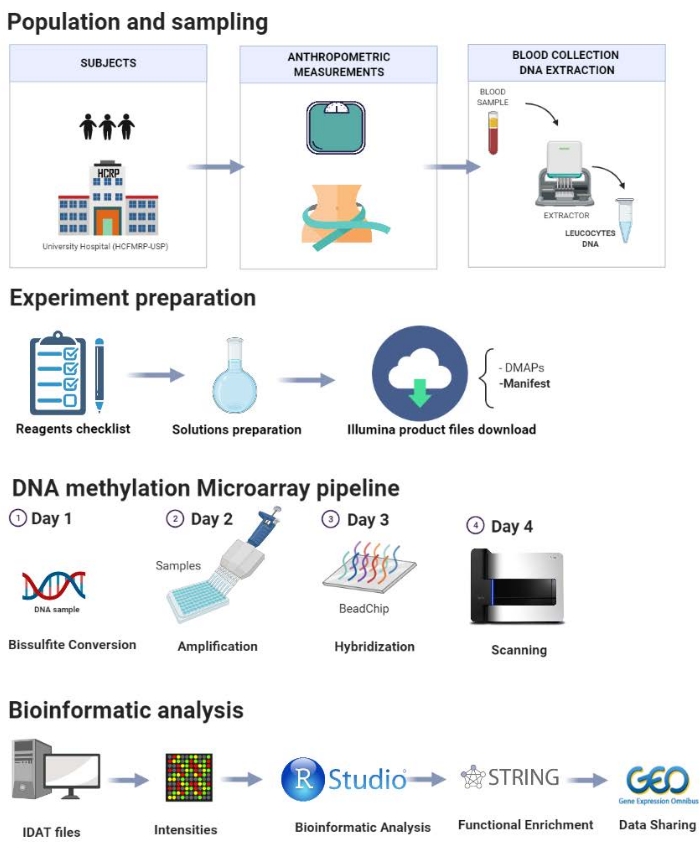
그림 1: 제시된 프로토콜의 회로도. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
상파울루 대학 (HCRP-USP)의 Ribeirão Preto Medical School University Hospital의 윤리위원회는 연구를 승인했습니다 (CAAE : 14275319.7.0000.5440). 참가자들은 동의서에 서명했으며 모든 절차는 헬싱키 선언에 따라 수행되었습니다.
1. 인구 및 샘플링
- 연구를 위해 BMI >30 kg / m2로 참가자를 모집하십시오. 본 연구를 위해, 18세에서 60세 사이의 혼혈 집단19에서 32명의 여성이 모집되었다.
참고 : HCRP-USP에서는 주로 여성20에서 높은 비만율이 관찰되었습니다. BMI ≥30 kg / m2 (n = 15)의 비만 참가자는 HCRP-USP의 대사 질환 외래 진료소에서 모집되었습니다. BMI가 18.5 kg / m2 와 24.9 kg / m2 (n = 17) 인 비만이없는 참가자는 다른 클리닉에서 모집되었으며 심각한 질병이 없었습니다. 임상 데이터는 표 1에 나타내었다. - 임신 중인 여성, 수유부, 흡연자 및 음주 여성은 제외합니다.
2. 인류학 및 신체 구성
- 200kg 디지털 인위적 척도를 사용하여 참가자의 체중을 측정하십시오. 신발과 과도한 의복을 제거하십시오.
- 0.5cm 눈금이있는 stadiometer를 사용하여 높이를 평가하십시오. 참가자들은 맨발로, 발을 함께 세우고, 팔을 옆으로 서도록 지시받았다.21
- 전기 생체 임피던스를 사용하여 지방 질량 (FM) 정보를 수집하십시오. 금식의 12 시간 후, 빈 요로 방광으로 평가하십시오. 참가자를 몸을 만지지 않고 다리를 벌리고 팔을 평행하게 한 상태에서 수핀 위치에 놓습니다. 참가자들에게 모든 금속 액세서리를 제거하도록 지시22.
- 0.1mm 졸업식의 확장할 수 없는 측정 테이프를 사용하여 허리 둘레(WC)를 구하십시오. 테이프는 엉덩이 뼈 바로 위의 중간 허리 주위에 수평으로 배치되었습니다. 측정은 숨을 쉴 직후에 발생했습니다. 질병 통제 예방 센터 (CDC)에 따르면 여성을위한 WC 컷오프는 88cm23입니다.
표 1: 인구 특성. 모든 변수는 파라메트릭(p > 0.05, Shapiro Wilk)이었고, 차이는 독립 t-검정을 사용하여 평가되었으며, 여기서 p < 0.05는 유의한 것으로 간주되었다. *p < 0.0001. 이 테이블을 다운로드하려면 여기를 클릭하십시오.
3. 생물학적 물질의 수집
- 앞서 설명한 대로 금식 12시간 후에 말초 혈액을 채취하십시오.24.
- 전혈 샘플 4 mL를 항응고제 (예 : EDTA)가있는 수집 튜브에 모으고 운송을 위해 얼음에 보관하십시오.
4. DNA 추출
- 혈액이 들어있는 각 튜브를 2,000 x g 에서 10분 동안 원심분리한다. 약 300 μL의 버피 코트 (흰색 인터페이즈)를 수집하십시오.
- 앞서 기술한 바와 같이 시판되는 기구(표 표 참조)를 사용하여 말초 혈액으로부터 DNA를 추출한다(표 참조). 480 μL의 초순수 H2O에서 DNA를 에루타냈다.
- 형광계를 사용하여 DNA 농도 및 품질을 평가합니다. 무결성을 1% 아가로스 겔 분석을 사용하여 평가하였다25. -80°C의 냉동실에 보관하십시오.
5. 메틸화 분석 전 준비
- 샘플이 최소 DNA 농도(500ng가 시작하기에 이상적인 초기 입력)를 갖는지 확인하지만, 희석은 이 단계에서 샘플마다 다를 수 있습니다.
- 일부 시약은 마이크로어레이 kit26에 포함되어 있지 않으므로 별도로 구입하십시오27.
- 키트에 절차 중에 사용되는 모든 시약이 포함되어 있는지 확인하십시오. 그들은 대체 할 수 없습니다. 모든 시약의 만료일을 확인하십시오. 키트에 제공되지 않은 다른 시약 및 용액도 사용할 수 있는지 확인하십시오(95% 포름아미드/1mM EDTA, 0.1N NaOH, 절대 에탄올, 2-프로판올).
주의: 포름아미드는 휘발성 시약입니다. 제품이 신선하면 결과의 품질을 향상시킬 수 있습니다. 포름아미드 및 절대 에탄올의 무결성 및 품질은 제조업체가 제안한 염색 공정에 영향을 줄 수 있기 때문에 중요한 것으로 간주됩니다. 선택한 알코올은 분자 생물학 분석에 특유한 초순수 제품이어야합니다. - 마이크로어레이 키트를 획득한 후 DMAP(칩 디코딩 맵)를 다운로드합니다. 이러한 파일은 키트가 배송 된 후 24-48 시간 후에 제조업체의 웹 사이트에서 사용할 수 있으며 만료일28에서 제외됩니다. 언급 된 파일을 다운로드하려면 아래 단계를 수행하십시오.
- 인터넷에 액세스할 수 있는 컴퓨터가 있는지 확인합니다. 소프트웨어 디코드 파일 클라이언트를 사용하여 다운로드를 수행하십시오.
- 프로그램을 설치하기 전에 컴퓨터에 기관에서 부여한 아웃바운드 및 인바운드 액세스가 있는지 확인하십시오. 디코드 파일 클라이언트에는 보안 제한이 없습니다.
- BeadChip에 의한 액세스 옵션을 사용하여 디코드 파일 클라이언트 소프트웨어에 로그인하십시오. 이 로그인은 MyIllumina 사용자 ID 및 암호를 사용하여 수행됩니다. 이 페이지에 등록해야 합니다28.
- 디코드 파일 클라이언트에 로그인 한 후 소프트웨어 홈 페이지 28의 해당 필드에 아래에 나열된 정보를 입력하십시오 : 바코드 목록; 박스 ID 목록; 구매 주문 번호(PO); 및 판매 주문 번호 (번호 뒤에 오는 문자는 포함하지 마십시오).
- 구매 상자의 바코드 번호에 따라 다운로드할 DMAP를 선택하고 대화 상자에서 DMAP를 저장할 대상을 지정합니다.
- 버튼을 클릭하여 다운로드를 시작하십시오.
참고: 다운로드가 끝나면 다운로드한 폴더가 비어 있지 않은지 확인하고 실험을 강도 데이터 파일(IDAT 파일)로 변환하는 마지막 단계에 필요하므로 파일을 안전하게 저장합니다. 만료 날짜 이후, 일루미나는 온라인 데이터베이스에서 DMAP 데이터를 제거할 수 있습니다.
6. DNA 메틸화 파이프라인
참고 : DNA 메틸화 실험은 4 일로 나뉩니다 (그림 1). 정확한 결과를 얻기 위해 제조업체의 권장 사항 및 사양을 따르십시오.
- 1일차: 바이설파이트 처리
- 먼저 500ng의 게놈 DNA를 변성시킨 다음 전환 시약을 첨가하십시오. 이 단계를 수행하려면 중아황산염 및 마이크로어레이 키트29의 권장 사항을 따르십시오.
- 변성된 DNA를 130 μL의 바이설파이트 전환 시약으로 제조자가 제공한 표준 농도로 처리한다.
- 튜브를 1시간 동안 95°C에서 30초 및 50°C에서 16 사이클 동안 열순환기에서 인큐베이션한다. 열사이클러를 10분 동안 4°C로 램프 다운한다.
- 세척 버퍼 100 μL를 첨가한 다음 30초 동안 전속력으로 원심분리하여 세척 단계를 수행하십시오. 농도는 사용자 가이드에 제공되지 않으며 제조업체는 볼륨을 표준화합니다.
참고: 이 시약은 키트와 함께 제공되며 희석하거나 준비할 필요가 없습니다.
- 2일차: 증폭 - 메틸화 분석, 수동 프로토콜
- 혼성화 오븐을 37°C로 예열하고 온도가 평형화되도록 한다.
- 바코드 라벨을 0.8 mL 마이크로플레이트에 바릅니다.
- 증폭 시약 (다중 샘플 증폭 믹스 1, 랜덤 프라이머 믹스 및 다중 샘플 앰프 마스터 믹스)을 실온에서 해동하십시오 (이러한 시약은 키트와 함께 제공되며 희석하거나 준비 할 필요가 없습니다). 그런 다음 튜브의 전체 내용물이 혼합 될 때까지 적어도 10x의 부드러운 반전을 수행하십시오.
- 20 μL의 다중 샘플 증폭 믹스 1(MA1)을 플레이트의 웰에 분주한다.
- 4 μL의 DNA 샘플을 바이설파이트 전환 플레이트로부터 0.8 mL 플레이트 상의 상응하는 웰로 옮긴다. 이 시점에서 원래의 DNA 샘플의 ID를 플레이트 웰에 해당하는 실험실 추적 양식에 기록하십시오.
- 4 μL의 0.1 N NaOH를 다중 샘플 증폭 믹스 1 (MA1) 및 DNA 샘플을 함유하는 플레이트의 각 웰에 분주한다.
- 플라스틱 씰로 플레이트를 밀봉하십시오.
- 플레이트를 볼텍스하여 시약을 샘플과 혼합하고 1분 동안 1,600 rpm에서 반응시킨다. 실온에서 10분 동안 인큐베이션하고 조심스럽게 밀봉을 제거한다.
- 68 μL의 랜덤 프라이머 믹스(RPM)를 플레이트의 각 웰에 피펫한다. 75 μL의 다중 샘플 앰프 마스터 믹스(MSM)를 플레이트의 각 웰에 피펫팅한다(이전 용액을 제거하지 않고). 새 씰을 사용하여 플레이트를 덮으십시오.
참고: 웰을 플레이트와 결합하고 올바르게 방향을 정하도록 주의하십시오. - 밀봉된 플레이트를 1분 동안 1,600 rpm에서 볼텍스한다. 혼성화 오븐에서 37°C에서 20-24 h 동안 인큐베이션한다.
- 3일차: 혼성화 - 메틸화 분석, 수동 프로토콜
- 블록을 37°C로 예열한다.
- 튜브의 내용물을 실온에서 해동하십시오. 단편화 용액(FMS) 튜브를 10x 이상 조심스럽게 부드럽게 뒤집어 내용물을 완전히 혼합합니다.
- 하이브리디제이션 오븐에서 플레이트를 조심스럽게 제거하십시오. 펄스-플레이트를 280 x g에서 원심분리한다.
- 이제 플레이트에서 씰을 제거하십시오. 샘플을 함유하는 플레이트의 각 웰에 50μL의 단편화 용액(FMS)을 첨가하고, 플레이트를 다시 밀봉한다. 플레이트를 1분 동안 1,600 rpm으로 볼텍스합니다.
- 플레이트를 280 x g 에서 몇 초 동안 원심분리한다(펄스 원심분리 수행). 이어서, 밀봉된 플레이트를 37°C에서 1시간 동안 가열 블록 상에 놓는다.
- 실험을 일시 중지하고 플레이트를 동결하기로 결정한 경우 실온에서 해동하고 280 x g에서 원심분리합니다. 고정되지 않은 경우 이 단계를 건너뛰고 6.3.7단계로 이동합니다.
- 가열 블록을 37°C로 예열하도록 방치한다.
- 세척 버퍼를 그대로 두어 실온에서 해동하십시오. 해동되면 내용물을 혼합하기 위해 적어도 10x를 뒤집습니다.
- 플레이트에서 씰을 분리합니다.
- 100 μL의 침전 용액 (PM1)을 샘플을 함유하는 플레이트의 각 웰에 첨가한다.
- 씰로 플레이트를 다시 밀봉하십시오.
- 플레이트를 1분 동안 1,600 rpm으로 볼텍스합니다.
- 37°C에서 5분 동안 인큐베이션한다.
- 이어서, 펄스 원심분리기에 1분 동안 280 x g 로 놓는다.
참고: 다음 단계를 위해 원심분리기를 4°C로 설정하십시오. - 조심스럽게 접시에서 씰을 제거하고 버리십시오.
- 300 μL의 2-프로판올 (100%)을 샘플을 함유하는 각 웰에 첨가한다.
- 새 씰을 사용하여 극도의주의를 기울여 플레이트를 밀봉하십시오. 접시를 흔들지 않도록주의하십시오.
- 플레이트를 적어도 10x 반전시켜 전체 내용물을 혼합하십시오.
- 4°C에서 30분 동안 인큐베이션한다.
- 4°C에서 3,000 x g 에서 20분 동안 원심분리한다.
참고: 단계 6.3.20이 끝나면 즉시 원심분리 플레이트를 제거하십시오. - 플레이트에서 씰을 제거하고 버립니다.
- 플레이트를 빠르게 뒤집고 패드에 액체를 배출하여 상층액을 버리십시오. 그런 다음 종이 타월의 건조한 부위에 접시를 치면 액체가 배출됩니다.
- 1 분 동안 또는 모든 웰에 액체가 없을 때까지 여러 번 단단히 두드리십시오.
- 플레이트를 뒤집어 놓고 실온에서 1 시간 동안 덮지 마십시오.
참고 : 우물 바닥에 파란색 알갱이가 보일 것으로 예상됩니다. - 혼성화 오븐을 48°C로 예열한다.
- 예열하려면 열 블록을 켭니다. 이 과정에 20 분이 소요됩니다.
- 실온에서 재현탁, 혼성화 및 세척 용액을 해동시킨다. 적어도 10x를 반전시킵니다. 용액 중에 침전된 염이 없는지 확인하십시오.
- 46 μL의 재현탁, 혼성화 및 세척 용액(RA1)을 플레이트의 각 웰에 첨가한다.
참고: 나머지 모든 시약은 염색 및 혼성화 단계를 위해 예약되어야 합니다. - 씰을 플레이트에 바릅니다. 알루미늄 씰을 사용하여 플레이트를 덮으십시오. 밀봉을 고르게 수행하기 위해 단단히 잡으십시오.
- 증발을 피하기 위해 모든 웰이 안전하게 닫혀 있는지 확인하십시오.
- 새로 밀봉된 플레이트를 혼성화 오븐에 조심스럽게 놓고 48°C에서 1시간 동안 인큐베이션한다.
- 플레이트를 1분 동안 1,800 rpm으로 볼텍스합니다.
- 280 x g에서 펄스 원심분리기.
- 플레이트를 95°C에서 1시간 동안 열 블록으로 옮긴다.
- 하이브리드화(Hyb) 챔버를 함께 배치하여 준비합니다. 400μL의 가습 버퍼(PB2)를 Hyb 챔버의 저장소에 분배합니다. 뚜껑을 즉시 켭니다.
- 플레이트로부터 각 샘플을 마이크로어레이의 샘플 엔트리 개구부로 로딩한다.
- 마이크로어레이가 포함된 Hyb 챔버 인서트를 Hyb 챔버에 로드합니다. Hyb 챔버의 가능한 이동을 피하기 위해 Hyb 챔버를 십자형으로 닫으십시오.
참고: Hyb 챔버는 48°C에서 최소 16시간 동안 배양되어야 하지만 24시간을 넘지 않아야 합니다.
- 4일차: 염색 - 메틸화 분석, 수동 프로토콜
- 염색 용액 4를 330 mL의 100% EtOH로 재현탁시켜, 350 mL의 최종 부피를 얻었다. 염색 용액 4 병을 강하게 흔들어 완전히 재현탁하십시오. 실온에서 유지하십시오.
참고: 염색액 4는 2°C와 8°C 사이의 온도에서 최대 2주 동안 보관할 수 있습니다. - 혼성화 오븐에서 챔버를 제거하고 열기 전에 30 분 동안 벤치에서 식히십시오.
- Hyb 챔버가 냉각되는 동안 두 개의 세척 용기에 워시 버퍼(세척 용기 당 200mL)를 채웁니다. 각 용기에 "세척 버퍼"(PB1 및 PB2)로 라벨을 붙이는 것을 잊지 마십시오.
- 마이크로어레이 정렬 액세서리를 150mL의 워시 버퍼로 채웁니다.
- 플라스틱 스페이서를 분리하고 필요한 경우 유리를 청소하십시오.
- 마이크로어레이 슬라이드를 세척하려면 와이어 루프를 그리드에 부착하고 물방울 장비를 200mL의 워시 버퍼로 용기에 담그면 총 10배가 됩니다.
- 하이브리드화 챔버에서 모든 인서트를 제거하고 인서트에서 각 어레이를 제거합니다. 씰을 분리합니다.
주: 제거하는 동안 노출된 매트릭스를 만지지 마십시오. - 하이브리드화 챔버에서 마이크로어레이 슬라이드를 분리하는 동안 아무 것도 흘리지 않도록 주의하십시오.
- 마이크로어레이를 세척 완충액(PB1) 용액으로 천천히 가볍게 세척하고, 10x 위아래로 움직인다. 신선한 PB1 용액으로 이동하고, 마이크로어레이를 용액 내에서 10x 상하로 천천히 이동시켜 세척을 수행한다.
- 용기에서 마이크로어레이를 분리하고 잔류물이 없는지 확인합니다.
- 각 마이크로어레이의 상단에 있는 투명 스페이서를 사용하고 스페이서를 해당 위치로 안내합니다.
참고: 흰색 스페이서가 아닌 투명 스페이서만 사용해야 합니다. - 정렬 액세서리에 정렬 막대를 놓습니다.
- 투명 스페이서의 상단에 유리 백 플레이트를 놓으십시오. 전체 마이크로어레이를 덮을 수 있도록 주의하십시오. 그런 다음 금속 클램프를 흐름 챔버에 부착하십시오. 필요한 경우 가위를 사용하여 유동 챔버 스페이서의 끝을 트리밍합니다.
- ddH2O를 사용하여 하이브리드화 챔버 저장소를 즉시 세척하고 작은 청소 브러시로 문지릅니다. 가습 버퍼가 저장고에 남아 있지 않도록하십시오.
- 확장 및 염색 마이크로어레이를 담당하는 다음 단계로 이동하십시오.
참고: 모든 유량실은 실험실 벤치에 수평으로 장착된 상태로 두십시오. 전체 염색 단계가 준비되었을 때만 다시 처리하십시오. 재현탁, 혼성화 및 세척 용액을 20°C 내지 25°C 사이의 온도로 가열한다. 용액에 결정이 보이지 않을 때까지 부드럽게 혼합하십시오. - 시약을 랙에 순서대로 놓습니다. 얼어 붙은 것이 있으면 실온에서 방치 한 다음 적어도 10x를 뒤집어 용액을 혼합하십시오.
- 물 순환기를 적절한 수준으로 채 웁니다.
- 펌프를 켜고 온도를 44 °C로 설정하십시오.
- 챔버 랙에 부착된 모든 기포를 제거합니다.
- 온도 프로브를 사용하여 챔버 랙에서 테스트를 수행합니다. 모든 테스트 장소는 44°C ~ 0.5°C 사이±야 합니다.
참고: 다음 단계는 중단 없이 수행해야 합니다. - 랙이 44°C의 온도에 도달하면 각 어셈블리를 신속하게 유량 챔버에 배치합니다.
참고: 다음 단계의 경우, 모든 시약을 후면 유리판에 피펫팅합니다. - 150 μL의 재현탁액, 혼성화 및 세척 용액 (RA1)의 피펫. 이어서, 30분 동안 인큐베이션한다. 이 단계를 총 5배 반복합니다.
- 450 μL의 염색 용액 1(XC1)을 피펫하고 10분 동안 인큐베이션한다.
- 450 μL의 염색 용액 2(XC2)를 피펫하고 10분 동안 인큐베이션한다.
- 200 μL의 투컬러 익스텐션 마스터 믹스(TEM)를 피펫하고 15분 동안 인큐베이션한다.
- 450 μL의 95% 포름아미드/1 mM EDTA의 피펫을 1분 동안 인큐베이션하고 두 번 반복한다.
- 5 분 동안 배양하십시오.
- 챔버 랙의 온도를 32°C로 낮춥니다(우수한 2색 마스터 믹스에 표시됨).
- 450 μL의 스테인 용액 3(XC3)을 피펫하고 1분 동안 인큐베이션하여 1x를 반복한다.
- 챔버의 랙이 올바른 온도에 도달할 때까지 기다립니다.
참고: 채색 과정 직후에 마이크로어레이 이미지를 읽으려면 이 단계에서 스캐너를 켜십시오. - 이제 250 μL의 우수한 2-컬러 마스터 믹스(STM)를 분배하고, 10분 인큐베이션하였다.
- 이어서, 450 μL의 스테인 용액 3(XC3)을 분주하고, 1분 인큐베이션하였다. 1x를 반복하십시오.
- 5분 동안 기다린 다음 250μL의 안티스테인 2-컬러 마스터 믹스(ATM)를 첨가하고 10분 동안 인큐베이션한다.
- 이제 250 μL의 우수한 2-컬러 마스터 믹스(STM)를 분배하고, 10분 인큐베이션하였다.
- 이어서, 450 μL의 스테인 용액 3(XC3)을 분주하고, 1분 인큐베이션하였다. 1x를 반복하십시오.
- 5 분 동안 기다리십시오.
- 250μL의 얼룩 방지 2-컬러 마스터 믹스(ATM)를 첨가하고 10분 동안 인큐베이션한다.
- 450 μL의 스테인 마이크로어레이 용액 3(XC3)을 첨가하고, 1분 동안 인큐베이션한 다음, 다시 반복한다.
- 5 분 동안 기다리십시오.
참고: 다음 반복 체계를 수행하십시오: 6.4.34단계, 6.4.35단계. 및 단계 6.4.36.; 단계 6.4.37., 6.4.38., 및 단계 6.4.39.; 6.4.34 단계, 6.4.35 단계 및 6.4.36 단계. - 이제 즉시 랙에서 유량실을 제거하고 조심스럽게 실험실 벤치에 수평으로 놓습니다. 온도가 주변 환경인지 확인하십시오.
- 6.4.41.각 마이크로어레이에 대한 세척액(PB1) 310 mL를 세척용기에 넣는다.
- 금속 클램프와 유리를 제거하고 마지막으로 마이크로 어레이를 제거하십시오.
- 마이크로어레이를 염색 랙 상에 놓고 10x PB1 용액이 들어있는 세척 용기에 넣는다. 바코드가 취급하는 사람과 마주보고 있고 모든 마이크로어레이가 물에 잠겨 있는지 확인하십시오.
- 위아래로 10x 움직임으로 천천히 가볍게 움직입니다. 마이크로어레이는 다시 잠기기 전에 용액에서 완전히 제거되어야 합니다. 그런 다음 5 분 동안 잠겨 있게하십시오.
- 스테인 용액 4 (XC4) 시약을 격렬하게 흔들어 총 재현탁을 보장합니다. XC4 310 mL를 세척 용기에 넣습니다.
참고 : 용액을 세척 용기에 10 분 이상 두지 마십시오. - 염색 스탠드를 XC4로 다른 용기로 가볍게 옮깁니다.
- 위아래로 10x 움직임으로 천천히 가볍게 움직입니다. 마이크로어레이가 다시 잠기기 전에 솔루션에서 완전히 제거되었는지 확인하십시오.
- 그런 다음 마이크로 어레이를 5 분 동안 침수 상태로 유지하십시오.
- 염색 스탠드를 용액 밖으로 꺼내 바코드가 위를 향하게하는 튜브 홀더에 놓습니다.
- 잠금 포셉을 사용하여 마이크로어레이를 조심스럽게 분리하고 선반에 올려 놓고 건조시킵니다.
- 진공 데시케이터를 사용하여 675 mm Hg (0.9 bar)에서 50-55 분 동안 건조시킵니다.
- EtOH를 사용하여 각 마이크로어레이의 뒷면을 청소합니다.
주의: 줄무늬를 만지지 말고 EtOH가 어떤 식으로든 줄무늬에 떨어지지 않도록 하십시오. - 마이크로어레이 이미지를 계속 진행합니다.
- 마이크로어레이를 조심스럽게 실온에서 보관 박스에 보관하십시오.
참고: 마이크로어레이 이미지는 72시간 이내에 촬영해야 합니다.
- 염색 용액 4를 330 mL의 100% EtOH로 재현탁시켜, 350 mL의 최종 부피를 얻었다. 염색 용액 4 병을 강하게 흔들어 완전히 재현탁하십시오. 실온에서 유지하십시오.
7. 생물정보학 분석
참고: 마이크로어레이 종류의 일부 속성을 변경할 필요가 있습니다(예: 배열 유형 = "450k"는 배열 유형 = "EPIC"로 대체되어야 함). ChAMP 파이프라인의 저자는 패키지30의 Bioconductor 페이지에서 배열을 분석하기 위해 수정해야 하는 모든 필드를 자세히 설명합니다.
- 샘플 시트
- IDAT 파일을 컴퓨터로 전송하여 생물 정보학 분석을 진행하십시오31. 데이터 분석을 수행할 샘플 시트를 생성한다31.
참고: 이 스프레드시트 파일에는 세 개의 열이 필수적입니다. 첫 번째는 Sample_name입니다. 이 열에는 샘플을 식별하는 데 사용되는 이름이나 코드가 포함되어야 합니다. 두 번째는 Sentrix_position입니다. 이 열은 샘플의 해당 R01C01을 배치하며, R01은 칩 행을 담당하고 컬럼은 C01이라고 합니다. 세 번째 필수 컬럼은 Sentrix_ID이며, 마이크로어레이를 식별하는 번호를 수신하는 역할을 한다. 올바른 숫자를 입력했는지 확인하십시오. 샘플 시트에 대한 다른 정보를 변수 값으로 추가할 수 있습니다. 본 실험에서, 금속 분석으로부터의 데이터가 포함되었다.
- IDAT 파일을 컴퓨터로 전송하여 생물 정보학 분석을 진행하십시오31. 데이터 분석을 수행할 샘플 시트를 생성한다31.
- ChAMP 파이프라인
- 컴퓨터에 RStudio(V.4.0.2)를 설치하여 분석을 수행합니다30.
주: 컴퓨터에 최소 8G의 RAM이 있는지 확인하십시오. 데이터 분석은 더 적은 양의 RAM으로 어려움을 겪거나 충돌합니다. - 설치 후 RStudio를 열고 각 명령줄을 사용하여 ChAMP Bioconductor package30 을 설치합니다( 보충 자료 1 참조).
참고: 설치가 끝날 때 오류가 발생하면 필요한 ChAMP 라이브러리를 수동으로 하나씩 설치하십시오26. - 이제 원시 데이터를 가져오고 분석을 수행합니다. 보충 자료 1을 참조하십시오.
champ.load() 는 원시 데이터를 가져오고 낮은 검출 pval, 다중 히트 부위, 비 CG 프로브, SNP 근처의 CpG 사이트 및 성 염색체에 위치한 CpGs가있는 프로브를 제외하고 필터링 프로세스를 수행합니다.
champ.impute() 는 기본적으로 KNN 전가를 수행합니다.
챔피언. QC 는 데이터 품질 그래프(예: 밀도 플롯 그래프)를 검색합니다.
챔피언. SVD 는 샘플 시트에 제공된 메타데이터를 기반으로 배치 효과를 평가합니다.
champ.refbase() 는 셀 유형 추정 및 보정을 수행합니다.
챔피언. DMP( ) 는 보충 자료 1에 표시된 것처럼 그룹 간의 DNA 메틸화를 확인하고 매개 변수를 변경하기 위해 지방 질량과 같은 차별적 인 메틸화 위치 및 형질 연관성을 검색합니다.
- 컴퓨터에 RStudio(V.4.0.2)를 설치하여 분석을 수행합니다30.
-
보강(문자열 DB)
참고 : 기능적 농축은 일련의 유전자의 생물학적 기능을 추론하는 데 사용됩니다.- 문자열의 데이터를 보강하려면 보강할 대상 목록을 선택하고 입력으로 사용합니다. https://string-db.org/cgi/input?sessionId=bywt5Gl8Bawy&input_page_active_form
=multiple_identifiers - 유기체의 경우 호모 사피엔스를 선택하고 검색 버튼을 누릅니다.
- 보강을 탐색하려면 분석 단추를 클릭합니다.
- 문자열의 데이터를 보강하려면 보강할 대상 목록을 선택하고 입력으로 사용합니다. https://string-db.org/cgi/input?sessionId=bywt5Gl8Bawy&input_page_active_form
-
GEO 제출
- NCBI의 GEO와 같은 공용 저장소에 데이터를 보관합니다. 이를 위해 제출자 계정에 등록하십시오.
참고: 전체 실험 및 개별 샘플에 대한 설명 정보 및 프로토콜이 포함된 메타데이터 시트를 다운로드하여 GEO 제출(https://submit.ncbi.nlm.nih.gov/geo/submission/)을 완료합니다. GEO 아카이브 제출물은 모든 스프레드시트 소프트웨어에서 만들 수 있습니다. 둘째, 모든 라이브러리에 대한 셀 레인저 개수 스크립트에서 생성된 원시 데이터 파일을 디렉터리에 추가합니다. IDAT 및 관련 파일 또는 행렬 워크시트에 정규화되지 않은 데이터가 포함되어 있습니다. 세 번째 폴더는 처리된 데이터 파일입니다. 행렬 테이블은 행과 샘플에 걸쳐 비교할 수 있는 최종, 정규화된 값을 포함하는 스프레드시트이며, 바람직하게는 수반되는 원고에 설명된 대로 처리됩니다. - 모든 라이브러리에 대한 셀 레인저 카운트 스크립트에서 생성된 처리된 데이터 파일을 디렉토리에 배치합니다. GEO 제출자의 FTP 서버 자격 증명을 사용하여 세 가지 구성 요소를 모두 포함하는 디렉터리를 전송합니다.
- NCBI의 GEO와 같은 공용 저장소에 데이터를 보관합니다. 이를 위해 제출자 계정에 등록하십시오.
결과
ChAMP 파이프라인을 사용한 후, 409,887개의 프로브가 모든 필터 후에 분석에서 고려되었다(규정되지 않은 CpGs, 비-CG 프로브, SNP 근처의 CpG, 멀티히트 부위 및 XY와 관련된 CpGs); 구성표는 그림 2에 나와 있습니다.
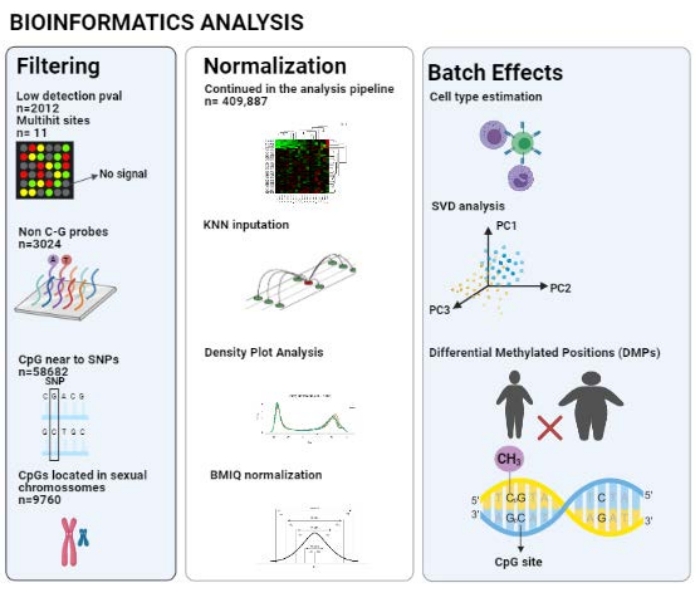
그림 2: 생물정보학 분석의 파이프라인. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
또한, 밀도 플롯은 모든 샘플이 품질 관리 단계와 관련된 베타 분포의 유사한 밀도를 가지고 있음을 밝혀 냈습니다. 이 분석은 베타 값의 분포를 평가하고 제외해야 할 샘플이 있는지 여부를 지적합니다. 이 배치에서, 어떠한 샘플도 이 분석에 기초하여 배제되지 않았다.
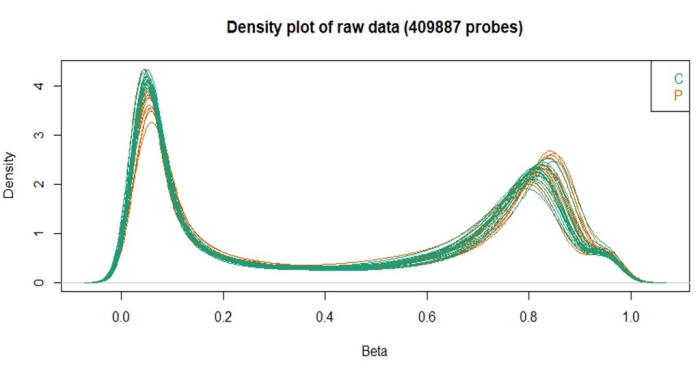
그림 3: ChAMP 패키지로 얻은 베타 값 밀도 그림. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
단수 값 분해 (SVD) 분석은 DNA 메틸화 데이터 가변성에 유의하게 영향을 미치는 주성분을 검증하기 위해 사용되었다. 데이터는 BMI, WC (p < 1e10-5) 및 FM (p < 0.05)이 데이터 변동성에 상당한 영향을 미친다는 것을 밝혀 냈습니다 (그림 4).
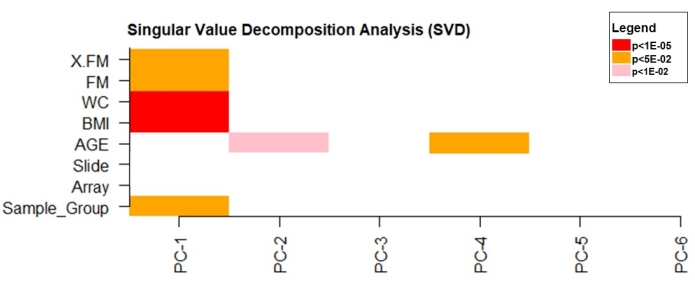
그림 4: 단수 값 분해 분석. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
세포 유형 추정은 자연 살해 세포 (NK)와 B 세포 모두 비만 여성에서 더 높았다는 것을 밝혀 냈습니다 (그림 5).
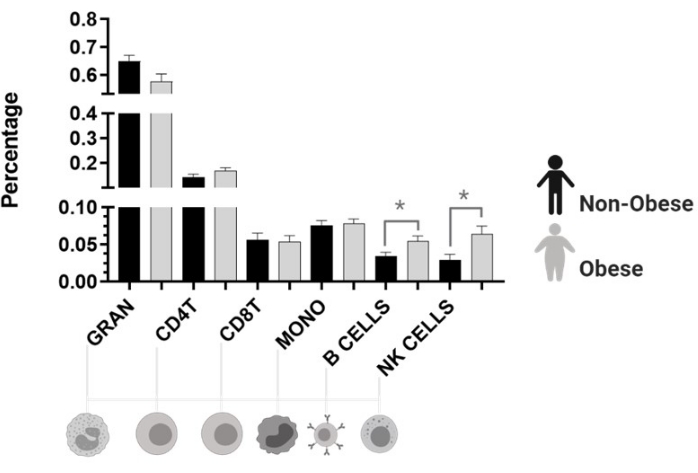
그림 5: Houseman의 방법에 의해 추정된 세포 분획. 그랜: 과립구. CD4T: 헬퍼 T 세포, 림프구. CD8T: 세포독성 T 세포, 림프구. 단핵구: 단핵구. B 세포: B 림프구. NK : 자연 살해 세포, 림프구. *p< 0.05입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
비만 여성과 비비만 여성 사이의 DNA 메틸화 수준은 세포형 교정 전후에 달랐다. 세포 유형에 대한 DNA 메틸화 데이터 보정 전에, 43,463개의 차등적 메틸화 위치(DMPs)가 관찰되었고, 3,329 CpGs는 그 후에도 유의하게 유지되었다. 445개의 CpG 부위가 인터제닉 영역(IGR)에 있었고, 2,884개가 프로모터 영역(n=1,438)에 가장 많이 있는 원성 영역에 있었다. 모든 영역을 따른 분포는 TSS1500 (n = 612), TSS200 (n = 826), 5'UTR (n = 390), 첫 번째 엑손 (n = 273), 몸체 (n = 724) 및 3'UTR (n = 59)이었다. Δβ 값 <-0.05 및 >0.05를 고려할 때, 162 CpGs는 하이포메틸화되었고 576은 비만인이 아닌 개인에 비해 비만에서 하이퍼메틸화되었다(도 6). 데이터는 GEO 데이터베이스에서 레지스터 코드 GSE166611로 사용할 수 있습니다.
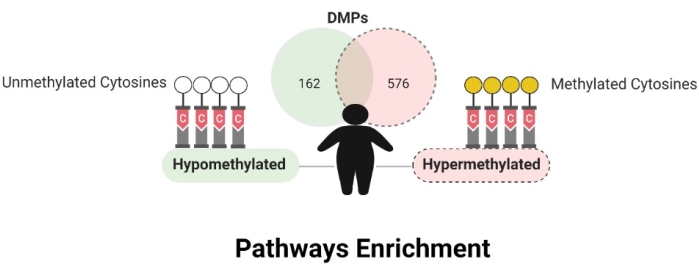
그림 6: 차등적으로 메틸화된 위치. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
그룹 간의 상이한 메틸화를 평가하고 메틸화 연구에서 특정 형질과 관련된 CpG 부위를 찾을 수 있다. 지방 질량 연구를 위해, 13,222 CpG 사이트가 발견되었다. 프로모터 영역에서 6,159 CpGs는 470개의 하이퍼메틸화 및 94개의 하이포메틸화(도 7)로 지방 질량과 관련되었고(도 7), 각각의 유전자는 농축되었다(표 2).
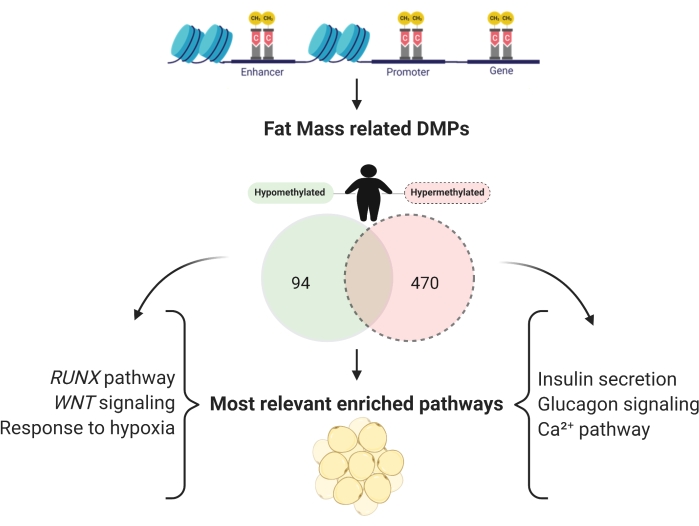
그림 7: 지방 질량과 독립적으로 관련된 하이포메틸화 및 하이퍼메틸화 유전자의 프로모터 영역. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
표 2: 차등적으로 메틸화된 CpG 부위로부터의 유전자의 기능적 농축. 이 테이블을 다운로드하려면 여기를 클릭하십시오.
보충 자료 1 :이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
DNA 메틸화 어레이는 비용 편익 비율14로 인해 DNA 메틸화에 접근하는 데 가장 많이 사용되는 방법입니다. 본 연구는 브라질 코호트에서 수행된 파일럿 연구에서 DNA 메틸화를 평가하기 위해 상업적으로 이용가능한 마이크로어레이 플랫폼을 사용하는 상세한 프로토콜을 기술하였다. 파일럿 연구에서 얻은 결과는 프로토콜의 효과를 확인했습니다. 도 3은 샘플 비교성 및 완전한 중아황산염 전환(bisulfite conversion32)을 도시한다.
품질 관리 단계로서 ChAMP 알고리즘은 필터링 프로세스 중에 CpGs 사이트를 제외하는 것이 좋습니다. 프로브를 제외하는 목적은 데이터 분석을 개선하고 편향을 제거하는 것입니다. 낮은 품질의 CpGs(0.05보다 낮은 p-값)를 제거하여 데이터 세트에서 실험 잡음을 제거했습니다. 목표는 밀도 플롯 분석에서 통과 된 채로 남아 있습니다. Zhou33은 불일치를 피하기 위해 SNP 근처에서 CpG를 필터링하는 것의 중요성, 다형성 시토신의 메틸화의 오해, 유형 I 프로브 설계의 스위치 색상 유발34의 중요성을 설명했습니다. 또한 XY 염색체가 각인에 의해 차별적으로 영향을 받기 때문에 Heiss와 Just35 는 여성에서 교잡 문제가 혼란스러운 요인이 될 수 있기 때문에 이러한 프로브를 필터링하는 것의 중요성을 강화했습니다35.
DMAPs 만료일, 포름아미드 개방일, 절대 에탄올의 분석 품질 및 총 백혈구 수는 프로토콜에서 중요한 단계로 간주됩니다.
또한, 우리의 관찰에 따르면, 세포 유형 추정은 생물 정보학 분석을 수행하는 데 필수적입니다. Houseman 방법은 Tian의 연구30에 설명 된대로 세포 유형 추정을 수행합니다. 이 방법은 과립구, 단핵구, B 세포 및 T 세포와 같은 가장 중요한 세포 유형의 백분율을 예측할 수 있는 473개의 특정 CpG 부위에 기초한다36. 우리는 ChAMP 패키지에서 권장되는 기능 "myRefbase"를 사용했습니다. 추정 후 ChAMP 알고리즘은 베타 값을 조정하고 데이터 세트에서 이러한 편향을 제거합니다. 이 단계는 비만에 초점을 맞춘 연구에서 매우 중요한데, 그 이유는 이 집단이 만성 염증 상태로 인해 백혈구에 상당한 차이가 있기 때문이다.
메서드 수정 및 문제 해결과 관련된 일반적인 PCR 씰에 대한 원래 캡 맵만 변경했습니다. 각각의 원심분리 과정 후에, 밀봉은 새로운 것에 대해 변경되었다. 우리는 표준 열 밀봉을 사용할 수 없었고 플레이트 주위에 알루미늄 호일을 사용하여 적응했습니다.
상업적 분석이 후성유전학 연구의 황금 표준으로 간주되었지만, 프로토콜의 한 가지 한계는 독특한 브랜드37,38,39,40의 시약 및 장비의 특이성일 수 있다. 또 다른 한계는 실험41의 정확한 진행을 식별할 수 있는 지표의 부족이다.
본 프로토콜의 표준화는 후성 유전학 연구를위한 훌륭한 지침이며, 프로세스 중 인적 오류를 줄이고 성공적인 데이터 분석 및 다른 연구 간의 비교 가능성을 허용합니다.
우리의 결과에 따르면, DNA 메틸화 실험은 비만이 있거나없는 개인을 비교하는 연구에 적합합니다43. 또한 제안 된 생물 정보학 분석은 고품질의 데이터를 제공했으며 대규모 연구에서 고려 될 수있었습니다.
SVD 분석을 사용하여, 우리는 비만 관련 형질 (BMI, WC 및 FM)이 DNA 메틸화 데이터의 변동성에 영향을 미친다는 것을 확인했다. 유의미한 결과로서, 세포형 추정은 자연살해세포(NK)와 B세포가 모두 비만이 없는 여성보다 비만 여성에서 더 높았음을 나타낸다(도 5). 이들 세포의 더 높은 카운트는 이들 개인들의 낮은 등급의 염증 상태에 의해 설명될 수 있다44. 우리는 비만 환자가 지방 질량과 관련된 유전자의 프로모터 영역에서 hypo- 및 hypermethylated CpGs를 가지고 있음을 관찰했다. 대부분의 부위는 하이포메틸화되었으며, 이는 이들 개체에서 반응성 산소 종 (ROS) 수준의 자연적 증가와 관련이있을 수 있습니다. 이러한 산화적 스트레스 조건은 디뉴클레오타이드 부위에서 구아닌 교란을 촉진하여 8-하이드록시-2'-데옥시구아노신(8-OHdG)을 형성하고, 이로 인해 5mCp-8-OHdG 디뉴클레오타이드 부위를 생성하고, TET 효소 모집을 야기할 수 있다. 이러한 모든 사건은 상이한 작용 메카니즘에 의해 DNA 하이포메틸화 및 하이퍼메틸화를 촉진시키는 역할을 할 수 있다45.
또한, 지방 생성의 비율은 비만 환자에서 증가하는 것으로 보이며, 오래된 세포에 대한 새로운 세포의 약 10 %46,47. 비만 환경을 강조하는 후성 유전 학적 기여는 세포의 증식 및 분화 속도를 변화시켜 지방 질량의 발달을 촉진 할 수 있습니다48. 후성 유전 학적 변화는 또한 아디포겐 프로그램에 영향을 미쳐 발달을 촉진하거나 제한 할 수 있습니다. 일차 전사 인자 (PPARγ 또는 C/EBPα) 또는 후성유전학적 변형 효소를 포함하거나 배제함으로써 조작되는 하류 프로모터 영역에 위치하는 다중 단백질 복합체의 조립은, 하이퍼- 또는 하이포메틸화를 통해 유전자 발현을 조절한다45. PPARγ 경로는 이전에 WNT 경로를 변경하기 위해 기술되었으며, 이는 본 연구에서 유전자가 풍부했다. 지방 생성 중에 WNT 신호 전달이 어떻게 발생하는지는 아직 알려지지 않았지만, 최근의 연구에 따르면 특히 비만 성 조건 하에서 지방 세포 대사에 필수적인 역할을 할 수 있다고보고되었습니다49.
공개
이 기사의 저자는 이해 상충이 없으며 선언 할 재정적 공개가 없습니다.
감사의 말
ChAMP 패키지에 대한 모든 의구심에 답할 수 있게 해주신 Yuan Tian, Ph.D. (tian.yuan@ucl.ac.uk)에게 감사드립니다. 우리는 또한이 논문의 기술 및 과학 문제에 대한 그의 공헌에 대해 Guilherme Telles, Msc.에게 감사드립니다. 그는 후성 유전학 및 비디오 캡처 및 서식 기술 (guilherme.telles@usp.br)에 관한 중요한 고려 사항을했습니다. 소모품 기금 : 상파울루 연구 재단 (FAPESP) (#2018/24069-3) 및 국립 과학 기술 개발 협의회 (CNPq : #408292 / 2018-0). 개인 기금 : (FAPESP : # 2014 / 16740-6) 및 고등 교육 직원 개발을위한 조정의 학업 우수 프로그램 (CAPES : 88882.180020 / 2018-01). 데이터는 공개적으로 공개되며 제한없이 자유롭게 사용할 수 있습니다. NYN (이메일 : nataliayumi@usp.br) 또는 CBN (이메일 : carla@fmrp.usp.br)에 대한 주소 서신.
자료
| Name | Company | Catalog Number | Comments |
| Absolute ethanol | J.T. Baker | B5924-03 | |
| Agarose gel | Kasvi | K9-9100 | |
| Electric bioimpedance | Quantum BIA 450 Q - RJL System | ||
| Ethylenediaminetetraacetic acid (EDTA) | Corning | 46-000-CI | |
| EZ DNA Methylation-Gold kit | ZymoResearch, Irvine, CA, USA | D5001 | |
| Formamide | Sigma | F9037 | |
| FMS—Fragmentation solution | Illumina | 11203428 | Supplied Reagents |
| HumanMethylation450 BeadChip | Illumina | ||
| Maxwell Instrument | Promega, Brazil | AS4500 | |
| MA1—Multi-Sample Amplification 1 Mix | Illumina | 11202880 | Supplied Reagents |
| MicroAmp Optical Adhesive Film | Thermo Fisher Scientific | 201703982 | |
| MSM—Multi-Sample Amplification Master Mix | Illumina | 11203410 | Supplied Reagents |
| NaOH | F. MAIA | 114700 | |
| PB1—Reagent used to prepare BeadChips for hybridization | Illumina | 11291245 | Supplied Reagents |
| PB2—Humidifying buffer used during hybridization | Illumina | 11191130 | Supplied Reagents |
| 2-propanol | Emsure | 10,96,34,01,000 | |
| RA1—Resuspension, hybridization, and wash solution | Illumina | 11292441 | Supplied Reagents |
| RPM—Random Primer Mix | Illumina | 15010230 | Supplied Reagents |
| STM—Superior Two-Color Master Mix | Illumina | 11288046 | Supplied Reagents |
| TEM—Two-Color Extension Master Mix | Illumina | 11208309 | Supplied Reagents |
| Ultrapure EDTA | Invitrogen | 155576-028 | |
| 96-Well Reaction Plate with Barcode (0.1mL) | ByoSystems | 4346906 | |
| 96-Well Reaction Plate with Barcode (0.8mL) | Thermo Fisher Scientific | AB-0859 | |
| XC1—XStain BeadChip solution 1 | Illumina | 11208288 | Supplied Reagents |
| XC2—XStain BeadChip solution 2 | Illumina | 11208296 | Supplied Reagents |
| XC3—XStain BeadChip solution 3 | Illumina | 11208392 | Supplied Reagents |
| XC4—XStain BeadChip solution 4 | Illumina | 11208430 | Supplied Reagents |
참고문헌
- Manna, P., Jain, S. Obesity, oxidative stress, adipose tissue dysfunction, and the associated health risks: causes and therapeutic strategies. Metabolic Syndrome and Related Disorders. 13 (10), 423-444 (2015).
- . Obesity Available from: https://www.who.int/health-topics/obesity#tab=tab_1 (2017)
- Adult Obesity Facts. Center for Disease Control and Prevention Available from: https://www.cdc.gov/obesity/data/adult.html (2021)
- Bird, A. Perceptions of epigenetics. Nature. 396, (2007).
- Kouzarides, T. Chromatin modifications, and their function. Cell. 128 (4), 693-705 (2007).
- Berger, F. The strictest usage of the term epigenetic. Seminars in Cell & Developmental Biology. 19 (6), 525-526 (2008).
- Henikoff, S., Greally, J. M. Epigenetics, cellular memory, and gene regulation. Current Biology. 26 (14), 644-648 (2016).
- Laker, R. C., et al. Transcriptomic and epigenetic responses to short-term nutrient-exercise stress in humans. Scientific Reports. 7 (1), 1-12 (2017).
- Wahl, S., et al. Epigenome-wide association study of body mass index, and the adverse outcomes of adiposity. Nature. 541, 81-86 (2017).
- Jin, B., Robertson, K. D. DNA methyltransferases, DNA damage repair, and cancer. Epigenetic Alterations in Oncogenesis. 754, 3-29 (2013).
- Jang, H. S., Shin, W. J., Lee, J. E., Do, J. T. CpG and non-CpG methylation in epigenetic gene regulation and brain function. Genes. 8 (6), 148 (2017).
- Shen, L., Waterland, R. A. Methods of DNA methylation analysis. Current Opinion in Clinical Nutrition & Metabolic Care. 10 (5), 576-581 (2007).
- Olkhov-Mitsel, E., Bapat, B. Strategies for discovery and validation of methylated and hydroxymethylated DNA biomarkers. Cancer Medicine. 1, 237-260 (2012).
- Kurdyukov, S., Bullock, M. DNA methylation analysis: choosing the right method. Biology. 5 (1), 3 (2016).
- Yong, W. -. S., Hsu, F. -. M., Chen, P. -. Y. Profiling genome-wide DNA methylation. Epigenetics & Chromatin. 9 (1), 1-16 (2016).
- Dugué, P. A., et al. Alcohol consumption is associated with widespread changes in blood DNA methylation: Analysis of cross-sectional and longitudinal data. Addiction Biology. 1, 12855 (2021).
- Colicino, E., et al. Blood DNA methylation sites predict death risk in a longitudinal study of 12, 300 individuals. Aging. 14, 14092-14124 (2020).
- Karlsson, L., Barbaro, M., Ewing, E., Gomez-Cabrero, D., Lajic, S. Genome-wide investigation of DNA methylation in congenital adrenal hyperplasia. The Journal of Steroid Biochemistry and Molecular Biology. 201, 105699 (2020).
- Ribeiro, R. R., Guerra-Junior, G., de Azevedo Barros-Filho, A. Bone mass in schoolchildren in Brazil: the effect of racial miscegenation, pubertal stage, and socioeconomic differences. Journal of Bone and Mineral Metabolism. 27 (4), 494-501 (2009).
- Filozof, C., et al. Obesity prevalence and trends in Latin-American countries. Obesity Reviews. 2 (2), 99-106 (2001).
- Chadid, S., Kreger, B. E., Singer, M. R., Bradlee, M. L., Moore, L. L. Anthropometric measures of body fat and obesity-related cancer risk: sex-specific differences in Framingham Offspring Study adults. International Journal of Obesity. 44 (3), 601-608 (2020).
- Nicoletti, C. F., et al. DNA methylation pattern changes following a short-term hypocaloric diet in women with obesity. European Journal of Clinical Nutrition. 74 (9), 1345-1353 (2020).
- Assessing Your Weight. Centers for Disease Control and Prevention Available from: https://www.cdc.gov/healthyweight/assessing/index.html (2020)
- Giavarina, D., Lippi, G. Blood venous sample collection: Recommendations overview and a checklist to improve quality. Clinical Biochemistry. 50 (10-11), 568-573 (2017).
- Duijs, F. E., Sijen, T. A rapid and efficient method for DNA extraction from bone powder. Forensic Science International: Reports. 2, 100099 (2020).
- . Infinium HD Methylation Assay, Manual Protocol Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/infinium_assays/infinium_hd_methylation/infinium-hd-methylation-guide-15019519-01.pdf (2015)
- Serrano, J., Snuderl, M. Whole genome DNA methylation analysis of human glioblastoma using Illumina BeadArrays. Glioblastoma. , 31-51 (2018).
- Leti, F., Llaci, L., Malenica, I., DiStefano, J. K. Methods for CpG methylation array profiling via bisulfite conversion. Disease Gene Identification. 1706, 233-254 (2018).
- Noble, A. J., et al. A validation of Illumina EPIC array system with bisulfite-based amplicon sequencing. PeerJ. 9, 10762 (2021).
- Tian, Y., et al. ChAMP: updated methylation analysis pipeline for Illumina BeadChips. Bioinformatics. 33, 3982-3984 (2017).
- Turinsky, A. L., et al. EpigenCentral: Portal for DNA methylation data analysis and classification in rare diseases. Human Mutation. 41 (10), 1722-1733 (2020).
- . The Chip Analysis Methylation Pipeline Available from: https://www.bioconductor.org/packages/release/bioc/vignettes/ChAMP/inst/doc/ChAMP.html (2020)
- Zhou, W., Laird, P. W., Shen, H. Comprehensive characterization, annotation and innovative use of Infinium DNA methylation BeadChip probes. Nucleic Acids Research. 45 (4), 22 (2017).
- Wanding, Z., Triche, T. J., Laird, P. W., Hui, S. SeSAMe: reducing artifactual detection of DNA methylation by Infinium BeadChips in genomic deletions. Nucleic Acids Research. 46 (20), 123 (2018).
- Heiss, J. A., Just, A. C. Improved filtering of DNA methylation microarray data by detection p values and its impact on downstream analyses. Clinical Epigenetics. 11, 15 (2019).
- Houseman, E. A., et al. DNA methylation arrays as surrogate measures of cell mixture distribution. BMC Bioinformatics. 13, 86 (2012).
- Valavanis, I., Sifakis, E. G., Georgiadis, P., Kyrtopoulos, S., Chatziioannou, A. A. A composite framework for the statistical analysis of epidemiological DNA methylation data with the Infinium human Methylation 450K BeadChip. IEEE Journal of Biomedical and Health Informatics. 18 (3), 817-823 (2014).
- Sun, N., Zhang, J., Zhang, C., Shi, Y., Zhao, B., Jiao, A., Chen, B. Using Illumina Infinium HumanMethylation 450K BeadChip to explore genomewide DNA methylation profiles in a human hepatocellular carcinoma cell line. Molecular Medicine Reports. 18 (5), 4446-4456 (2018).
- Moran, S., Arribas, C., Esteller, M. Validation of a DNA methylation microarray for 850,000 CpG sites of the human genome enriched in enhancer sequences. Epigenomics. 8 (3), 389-399 (2016).
- Bibikova, M., et al. High density DNA methylation array with single CpG site resolution. Genomics. 98 (4), 288-295 (2011).
- Lehne, B., et al. A coherent approach for analysis of the Illumina HumanMethylation450 BeadChip improves data quality and performance in epigenome-wide association studies. Genome Biology. 16 (1), 1-12 (2015).
- Wang, J., Zhang, H., Rezwan, F. I., Relton, C., Arshad, S. H., Holloway, J. W. Pre-adolescence DNA methylation is associated with BMI status change from pre- to post-adolescence. Clinical Epigenetics. 25, 64 (2021).
- Maugeri, A. The effects of dietary interventions on DNA methylation: implications for obesity management. International Journal of Molecular Sciences. 21, 8670 (2020).
- DeFuria, J., et al. B cells promote inflammation in obesity and type 2 diabetes through regulation of T-cell function and an inflammatory cytokine profile. Proceedings of the National Academy of Sciences. 110 (13), 5133-5138 (2013).
- Kietzmann, T., Petry, A., Shvetsova, A., Gerhold, J. M., Görlach, A. The epigenetic landscape related to reactive oxygen species formation in the cardiovascular system. British Journal of Pharmacology. 174, 1533-1554 (2017).
- Chase, K., Sharma, R. P. Epigenetic developmental programs and adipogenesis: implications for psychotropic induced obesity. Epigenetics. 8 (11), 1133-1140 (2013).
- Spalding, K. L., et al. Dynamics of fat cell turnover in humans. Nature. 453, 783-787 (2008).
- Ross, S. E., et al. Inhibition of adipogenesis by Wnt signaling. Science. 289, 950-953 (2000).
- Bagchi, D. P., et al. Wnt/β-catenin signaling regulates adipose tissue lipogenesis and adipocyte-specific loss is rigorously defended by neighboring stromal-vascular cells. Molecular Metabolism. 42, 101078 (2020).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유