È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
L'analisi fenotipica e l'isolamento di murini cellule staminali ematopoietiche e Lineage-committed progenitori
In questo articolo
Riepilogo
Un metodo per analizzare la distribuzione di progenitori midollari ematopoietiche citometria di flusso e per isolare in modo efficiente altamente purificate cellule staminali ematopoietiche (CSE) è descritto. La procedura di isolamento è essenzialmente basata su un arricchimento magnetico di c-kit + e le cellule di separazione delle cellule per purificare CSE per studi cellulari e molecolari.
Abstract
Il midollo osseo è il luogo principale in cui CSE e più mature cellule progenitrici del sangue del lignaggio risiedono e di differenziarsi in un organismo adulto. CSE costituiscono una popolazione minuti cellulare delle cellule pluripotenti in grado di generare tutte le linee cellulari del sangue per una vita-time 1. La dissezione molecolare di omeostasi CSE nel midollo osseo ha importanti implicazioni in ematopoiesi, oncologia e medicina rigenerativa. Abbiamo descritto il protocollo etichettatura con anticorpi fluorescenti e la procedura gating elettronico in citometria a flusso per segnare sottoinsiemi progenitrici ematopoietiche e la distribuzione CSE in singoli topi (Fig. 1). Inoltre, si descrive un metodo per arricchire ampiamente progenitori emopoietici, nonché a lungo termine (LT) ed a breve termine (ST) ricostituire CSE da sospensioni di cellule del midollo osseo da un pool di arricchimento magnetico di cellule che esprimono c-Kit. Preparato cellulare risultante può essere utilizzato per ordinare sottoinsiemi selezionati in vitro ein vivo studi funzionali (Fig. 2).
Entrambi gli osteoblasti trabecolari 2,3 e 4 endotelio sinusoidale costituiscono nicchie funzionali di supporto CSE nel midollo osseo. Diversi meccanismi nella nicchia osteoblastica, tra cui un sottoinsieme di N-caderina + 3 osteoblasti e l'interazione del Tie2 tirosina chinasi del recettore espresso in CSE con il suo angiopoietina-1 ligando 5 concorrono nel determinare quiescenza CSE. "Ibernazione" nel midollo osseo è fondamentale per proteggere HSCs dalla replica e la stanchezza finale sull'attività ciclistica eccessiva 6. Stimoli esogeni che agiscono sulle cellule del sistema immunitario innato, come Toll-like ligandi del recettore 7 e interferone alfa-6 può anche indurre la proliferazione e la differenziazione di cellule staminali progenitori impegnati nel lignaggio. Recentemente, una popolazione di cellule staminali di topo dormienti all'interno del lin - c-Kit + Sca-1 + CD150 + CD48 - CD34 - popolazione è stato descritto 8. Ordinamento delle cellule CD34 sulla base di un'espressione dalla ematopoietiche progenitori arricchita di sospensione cellulare come qui descritto permette l'isolamento di riposo sia auto-rinnovano LT-CSE e CSE ST-9. Una procedura simile basata su deplezione di cellule positive derivazione e smistamento di LT-HSC con CD48 e Flk2 anticorpi è stato precedentemente descritto 10. Nella presente relazione forniamo un protocollo per la caratterizzazione fenotipica ed ex vivo analisi del ciclo delle cellule di progenitori emopoietici, che possono essere utili per il monitoraggio ematopoiesi in diverse condizioni fisiologiche e patologiche. Inoltre, si descrive un procedimento per FACS smistamento CSE, che possono essere utilizzati per definire fattori e meccanismi che regolano l'automantenimento, espansione e la differenziazione in biologia cellulare e saggi di trasduzione del segnale così come per il trapianto.
Protocollo
1. Preparazione della sospensione di cellule dal midollo osseo
- Euthanize il mouse e collocare l'animale in una padella inox e spruzzo etanolo al 70% sulla sua all'addome e alla schiena. Raccogliere femore e tibia di zampe posteriori, e della colonna vertebrale.
- Accuratamente rimuovere tutti i residui dei tessuti molli della colonna vertebrale con un paio di forbici punta acuminata e dalle ossa delle gambe con una garza. Ossa conservare in una provetta Falcon da 50 ml con 30 ml di mezzo RPMI contenente il 10% inattivato al calore siero bovino fetale (FBS), supplementato con 50 pM β-mercaptoetanolo, 100 pM MEM Amminoacidi non essenziali, 1 mM MEM Piruvato di sodio, 2 mM L-glutammina, 100 pg / ml di streptomicina e 100 U / ml penicillina.
- Coprire il fondo di una malta precedentemente sterilizzato con filtri sterili.
- Trasferire le ossa nel mortaio e coprire con uno strato di filtri per assemblare una superficie di attrito.
- Preparare un tubo da 50 ml Falcon dotato di un filtro di Falcon.
- Rompete le ossacon l'aiuto di un pestello fino ad ottenere una sospensione omogenea cella.
- Trasferire la sospensione nel tubo.
- Aggiungi al 5 mL malta fresca di mezzo RPMI completo per lavare e raccogliere le cellule rimanenti.
- Ripetere gli ultimi due punti fino a quando le ossa omogeneizzati appaiono chiari.
- Trasferimento e filtrare in un nuovo tubo la sospensione cellulare per eliminare i detriti tessuto rimanente.
- Centrifugare la provetta a 1.500 rpm per 5 minuti e rimuovere con cautela il surnatante tramite aspirazione. Assicurarsi di non disturbare il pellet di cellule.
2. L'analisi fenotipica
- Risospendere e lavare cellule di singoli topi in 15 ml di PBS e il 2% FBS.
- Centrifugare la provetta a 1.500 rpm per 5 minuti e rimuovere con cautela il surnatante tramite aspirazione. Assicurarsi di non disturbare il pellet.
- Sospendere il pellet in 5 ml di PBS e il 2% FBS e cellule conteggio.
- Trasferire 1 x 10 6 cellule / pozzetto in 96 pozzetti a fondo tondopiastra.
- Centrifugare la piastra a 1.500 rpm per 5 min.
- Preparare una miscela di anticorpi monoclonali diretti contro i seguenti marcatori di superficie. Diluire come indicato in PBS e 2% FBS per colorare le cellule.
- CD4, CD8, CD3, CD45R, CD19, Gr1, Ter119, NK1.1 coniugati con PE-Cy5 1:200
- c-Kit coniugato APC 1:100
- Coniugato con biotina 01:50 SCA1
- Coniugato a FITC 1:50 CD34
- FcyR coniugato a PE 1:100
- IL-7R coniugato con PE-Cy7 1:100
- Eliminare il surnatante e aggiungere 50 microlitri di mix anticorpi monoclonali in ciascun pozzetto. Dissociarsi in singole cellule pipettando.
- Incubare le cellule per 20 minuti al buio a 4 ° C.
- Lavare le cellule due volte con 150 microlitri di PBS e il 2% FBS.
- Centrifugare la piastra a 1500 rpm per 5 minuti e scartare il supernatante capovolgendo il piatto.
- Diluire streptavidina in PBS e 2% FBS come indicato per colorare le cellule.
- Streptavidina coniugatogated di APC-Cy7 1:300
- Aggiungere 50 pl della soluzione ad ogni pozzetto. Dissociarsi in sospensione singola cella pipettando.
- Incubare le cellule per 10 minuti al buio a 4 ° C.
- Lavare le cellule due volte con 150 microlitri di PBS e il 2% FBS.
- Centrifugare la piastra a 1500 rpm per 5 min e scartare il surnatante.
- Risospendere le cellule in 100 pl di PBS e il 2% FBS.
- Acquisire campioni FACS.
3. Arricchimento magnetico di c-Kit + Cells
- Preparare una soluzione tampone contenente PBS, pH 7,2, 0,5% di albumina di siero bovino (BSA), e 2 mM EDTA.
- Risospendere il pellet di cellule di osso zucchine derivato da almeno 10 topi in 1 mL di soluzione tampone contenente c-kit coniugato APC mAb diluito 1:50.
- Incubare le cellule per 20 minuti al buio a 4 ° C.
- Lavare le cellule due volte con 40 mL di soluzione tampone.
- Centrifugare la provetta a 1.500 rpm per 5 minuti e rimuovere con cautela tha surnatante tramite aspirazione. Assicurarsi di non disturbare il pellet.
- Risospendere il pellet cellulare e aggiungere 50 ul di anti-APC microsfere per ciascun topo eutanasia direttamente al pellet.
- Mescolare bene e incubare per 20 minuti al buio a 4 ° C.
- Lavare le cellule aggiungendo 40 ml di soluzione tampone e centrifugare a 1500 rpm per 5 min. Aspirare il surnatante completamente.
- Risospendere il pellet cellulare in multipli di 4 mL di soluzione tampone ogni tre topi eutanasia.
- Inserire MACS colonne LS nel campo magnetico di un separatore adatto MACS. Utilizzare 1 colonna ogni tre topi eutanasia.
- Preparare le colonne risciacquando con 4 mL di soluzione tampone.
- Applicare sospensione cellulare sulle colonne.
- Raccogliere cellule non marcati che attraversano e lavare la colonna con 4 ml di soluzione tampone. Eseguire fasi di lavaggio con l'aggiunta di 4 ml di soluzione tampone tre volte. Aggiungi nuovo buffer quando il serbatoio si svuota colonna.
- Rimuovere le colonne dal separatore e Place ciascuno su 15 mL tubi falcon.
- Pipettare 4 mL di soluzione tampone su ogni colonna. Immediatamente lavare le cellule marcate magneticamente premendo lo stantuffo nella colonna.
- Centrifugare le provette a 1500 rpm per 5 minuti e rimuovere con cautela il surnatante tramite aspirazione.
- Risospendere ogni pellet con 1 mL di soluzione tampone e pellets di gruppo in un unico tubo di 15 ml Falcon.
4. Ordinamento di cellule staminali ematopoietiche e Lineage progenitori impegnati
- Preparare una miscela di anticorpi monoclonali diretti contro i seguenti marcatori di superficie. Diluire come indicato in 500 pl PBS e il 2% FBS per colorare le cellule.
- CD4, CD8, CD3, CD45R, CD19, Gr1, Ter119, NK1.1 coniugati con PE-Cy5 1:200
- c-Kit coniugato APC 1:100
- Sca-1 coniugato a biotina 1:50
- Coniugato a FITC 1:50 CD34
- FcyR coniugato a PE 1:100
- Centrifugare il tubo dal precesezione s a 1.500 rpm per 5 minuti e rimuovere con cautela il surnatante tramite aspirazione.
- Aggiungere la miscela mAbs al pellet. Dissociarsi in sospensione singola cella pipettando.
- Incubare le cellule per 20 minuti al buio a 4 ° C.
- Lavare le cellule due volte con 14mL di PBS e 2% FBS.
- Centrifugare la provetta a 1.500 rpm per 5 min e scartare il surnatante.
- Diluire streptavidina come indicato in 500 pl PBS e il 2% FBS per colorare le cellule.
- Streptavidina coniugata con APC-Cy7 1:300
- Aggiungere la soluzione del tubo. Dissociarsi in sospensione singola cella pipettando.
- Incubare le cellule per 10 minuti al buio a 4 ° C.
- Lavare le cellule due volte con 14 mL di PBS e il 2% FBS.
- Centrifugare la provetta a 1.500 rpm per 5 min e scartare il surnatante.
- Risospendere le cellule in 500 pl di PBS e il 2% FBS.
- Trasferire e filtrare la sospensione cellulare in un nuovo tubo 5 ml per elicessazione definitiva aggregati cellulari che possono interferire con la procedura di ordinamento.
- Aggiungere 5 pl di 7-AAD (usarlo diluito 1:100) alla sospensione cellulare al fine di escludere 7-AAD + cellule morte dalle popolazioni ordinati.
- Ordinare le celle con un FACSAria BD secondo i fenotipi seguenti:
- Cellule LKS: LIN - c-Kit + Sca-1 +
- LT-CSE: LKS CD34 -
- ST-CSE: LKS CD34 +
- PGM: lin - c-Kit + Sca-1 low FcyR alta CD34 +
- Deputati: lin - c-Kit + Sca-1 low FcyR bassa CD34 -
- CMP: lin - c-Kit + Sca-1 low FcyR bassa CD34 +
5. Risultati rappresentativi
La Figura 1 mostra e con l'esempio della procedura di gating elettronica apunteggio comuni progenitori linfoidi (CLPs) come Lin - / c-Kit Lo / interleuchina (IL)-7R + cellule; Lin - / c-Kit + / Sca-1 + (LKS) e Lin - / c-Kit + / Sca Lo -1 (c-Kit +) delle cellule, dalla c-Kit + gate, comuni progenitori mieloidi (CMP) sono identificati come FcyR Lo cellule CD34 +, progenitori dei granulociti / monociti (BPF) come FcyR hi cellule CD34 + e megacariociti / progenitori eritrocitari (MEP) come FcyR lo CD34 - cellule; dal cancello LKS, LT-HSC e ST-HSC sono identificati come CD34 - CD34 + e cellule, rispettivamente. Il midollo osseo sospensione cellulare può essere arricchito per c-kit + cellule con perline magnetiche (Fig. 2); LT-HSC e ST-HSC può quindi essere ordinati secondo CD34 espressione nonché altri progenitori secondo il fenotipo sopra descritto.
CSE può essere ana lisati in microscopia confocale immagini in diretta come mostrato nella figura 4, dove CD34 - cellule sono state colorate con il nucleotide-binding composto chinacrina 11 nonché rosso nucleare (DRAQ5). In CSE, ma non GMP, chinacrina-positivi vescicole sono visibili nel citoplasma 12. Inoltre, CSE ordinati possono essere utilizzati in esperimenti funzionali come mostrato in figura 5, che mostra l'aumento citosolico Ca 2 + in LT-HSC mediante attivazione dei recettori purinergici P2 per esposizione a-adenosina trifosfato (ATP) e ionomicina 12.
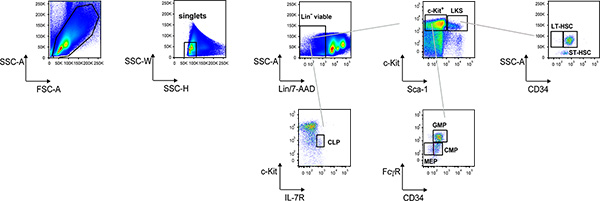
Figura 1. Rappresentante analisi dot plot di Lin-BM cellule. Procedura di gating elettronico a citofluorimetro per identificare e segnare progenitori ematopoietiche e CSE di sospensione di cellule del midollo osseo macchiato come descritto nel testo protocollo.
pload/3736/3736fig2.jpg "/>
Figura 2. Un sistema per l'arricchimento selettivo di c-Kit + cellule dal midollo osseo.
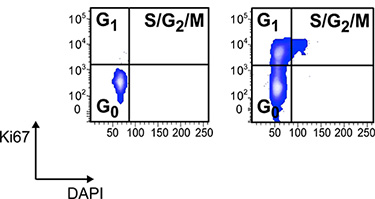
Figura 3 analisi del ciclo cellulare di elettronica LKS gated CD34 -. CSE nei controlli sani e nei topi con malattia infiammatoria intestinale colorati con anticorpo Ki-67 e DAPI. Le cellule sono state fissate e permeabilizzate con Lyse / Fix e Perm buffer seguita da colorazione con coniugato FITC Ki-67 e DAPI a 1 mg / ml. Cellule in bicicletta sono a malapena se non del tutto rilevabile nel LKS CD34 - vano HSCs di 12 controlli sani.

. Figura 4 live microscopia confocale imaging di FACS ordinati CD34 - CSE BPF e colorati con il nucleotide-binding quinacrine composto e nucleare rosso (DRAQ5). I quadranti di piccole dimensioni mostrano pos quinacrinevescicole itive rilevata nel citoplasma di cellule staminali, ma non GMP.

Figura 5. Citosolico Ca 2 + elevazioni in CSE ordinati caricate con Fura-2 e stimolate con ATP e ionomicina. Tracce da singole cellule vengono visualizzati. Tutte le cellule sono sensibili alle ATP extracellulare che mostravano un aumento di rilievo nel citosolico Ca 2 + dopo l'aggiunta di ATP.
Discussione
Il metodo qui descritto consente l'analisi rapida ed accurata di ematopoiesi in topi individuali (Figura 1). Questa analisi in diversi contesti sperimentali, inclusi i modelli murini di infiammazione, autoimmunità, immunodeficienza, malattie degenerative, malattie metaboliche e tumori, consente di indirizzare l'impatto delle condizioni patologiche su ematopoiesi. La Figura 3 mostra l'analisi di attività delle cellule ciclismo su LKS elettronicamente gated CD34...
Divulgazioni
Non ci sono conflitti di interesse dichiarati.
Riconoscimenti
Ringraziamo Nobuyuki Onai, Hitoshi Takizawa e Markus Manz per i preziosi consigli. Questo lavoro è stato finanziato dalla National Science Foundation svizzero, Lega svizzera contro il cancro e la Fondazione Ticinese per la Ricerca sul Cancro.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | |
| RPMI 1640 | Gibco | 42401 | |
| MEM NEAA 100X | Gibco | 11140 | |
| Sodio piruvato | Gibco | 11360 | |
| PenStrep | Gibco | 15070 | |
| PBS | Gibco | 20012 | |
| FBS | Gibco | 16000 | |
| Cella Filtro 40 micron | BD Falcon | 352340 | |
| 7-AAD Soluzione colorante | BD Pharmingen | 559925 | |
| Lyse / Fix tampone | BD Pharmingen | 558049 | |
| Perm tampone III | BD Pharmingen | 558050 | |
| Ki-67 | BD Pharmingen | 556026 | |
| DAPI | Invitrogen | D21490 | |
| CD4 (GK1.5) | eBioscience | 150041 | |
| CD8 (53-6,7) | eBioscience | 150081 | |
| CD3 (145-2C11) | eBioscience | 150031 | |
| CD45R (RA3-6B2) | eBioscience | 150452 | |
| CD19 (6D5) | eBioscience | 150193 | |
| Gr1 (RB6-8C5) | eBioscience | 155931 | |
| Tre119 (TER-119) | eBioscience | 155921 | |
| NK-1.1 (PK136) | eBioscience | 455941 | |
| c-kit (2B8) | eBioscience | 171171 | |
| Sca-1 (D7) | eBioscirienza | 135981 | |
| CD34 (RAM34) | eBioscience | 110341 | |
| FcyR (2.4G2) | eBioscience | 553145 | |
| Anti-APC microsfere | Miltenyi Biotec | 130-090-855 | |
| LS Colonne | Miltenyi Biotec | 130-042-401 |
Riferimenti
- Weissman, I. L. Stem cells: units of development, units of regeneration, and units in evolution. Cell. 100, 157-168 (2000).
- Calvi, L. M. Osteoblastic cells regulate the haematopoietic stem cell niche. Nature. 425, 841-846 (2003).
- Zhang, J. Identification of the haematopoietic stem cell niche and control of the niche size. Nature. 425, 836-841 (2003).
- Kiel, M. J., Yilmaz, O. H., Iwashita, T., Terhorst, C., Morrison, S. J. SLAM family receptors distinguish hematopoietic stem and progenitor cells and reveal endothelial niches for stem cells. Cell. 121, 1109-1121 (2005).
- Arai, F. Tie2/angiopoietin-1 signaling regulates hematopoietic stem cell quiescence in the bone marrow niche. Cell. 118, 149-161 (2004).
- Essers, M. A. IFNalpha activates dormant haematopoietic stem cells in vivo. Nature. 458, 904-908 (2009).
- Nagai, Y. Toll-like receptors on hematopoietic progenitor cells stimulate innate immune system replenishment. Immunity. 24, 801-812 (2006).
- Wilson, A. Hematopoietic stem cells reversibly switch from dormancy to self-renewal during homeostasis and repair. Cell. 135, 1118-1129 (2008).
- Osawa, M., Hanada, K., Hamada, H., Nakauchi, H. Long-term lymphohematopoietic reconstitution by a single CD34-low/negative hematopoietic stem cell. Science. 273, 242-245 (1996).
- Lo Celso, C., Scadden, D., D, . Isolation and Transplantation of Hematopoietic Stem Cells (HSCs. J. Vis. Exp. (2), e157 (2007).
- Romanello, M. Autocrine/paracrine stimulation of purinergic receptors in osteoblasts: contribution of vesicular ATP release. Biochem. Biophys. Res. Commun. 331, 1429-1438 (2005).
- Casati, A. Cell-autonomous regulation of hematopoietic stem cell cycling activity by ATP. Cell Death Differ. 18, 396-404 (2011).
- Bouma, G., Strober, W. The immunological and genetic basis of inflammatory bowel disease. Nat. Rev. Immunol. 3, 521-533 (2003).
- Takizawa, H., Regoes, R. R., Boddupalli, C. S., Bonhoeffer, S., Manz, M. G. Dynamic variation in cycling of hematopoietic stem cells in steady state and inflammation. J. Exp. Med. 208, 273-284 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon