È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Misurazione quantitativa della Invadopodia-mediata proteolisi della matrice extracellulare in singoli contesti e pluricellulari
In questo articolo
Riepilogo
Descriviamo il metodo di prototipo per la produzione di vetrini al microscopio a fluorescenza rivestite con gelatina per la visualizzazione invadopodia-mediata degradazione della matrice. Tecniche di calcolo utilizzando il software disponibile sono presentati per quantificare i livelli risultanti di proteolisi matrice di singole cellule all'interno di una popolazione mista e per gruppi multicellulari che comprende interi campi microscopici.
Abstract
Invasione cellulare nei tessuti locali è un processo importante nello sviluppo e omeostasi. Invasione Malregulated e il movimento delle cellule successiva è caratteristica di molteplici processi patologici, tra cui l'infiammazione, le malattie cardiovascolari e le metastasi delle cellule tumorali 1. Degradazione proteolitica focalizzata di matrice extracellulare (ECM), componenti della membrana basale epiteliale o endoteliale è un passo fondamentale nel dare inizio invasione cellulare. Nelle cellule tumorali, ampia analisi in vitro ha determinato che il degrado ECM si ottiene ventrali actina ricchi di strutture a membrana protrusione chiamati invadopodia 2,3. Forma Invadopodia in stretta apposizione della ECM, dove moderare ripartizione ECM attraverso l'azione di metalloproteinasi della matrice (MMP). La capacità delle cellule tumorali di formare invadopodia correla direttamente con la capacità di invadere stroma locale e componenti associati vascolari 3. _content "> Visualizzazione di invadopodia-mediata degradazione ECM delle cellule mediante microscopia a fluorescenza con colorante marcati con proteine della matrice rivestite su vetro coprioggetti è emerso come la tecnica più diffusa per valutare il grado di proteolisi matrice e cellulari potenziale invasivo 4,5. Qui descriviamo una versione del metodo standard per generare vetrini fluorescenza etichettati utilizzando un comune Oregon verde-488 coniugato gelatina. Questo metodo è facilmente scalata per produrre rapidamente un elevato numero di vetrini coprioggetto rivestiti. Mostriamo alcuni degli artefatti comuni microscopici che spesso si incontrano durante questa procedura e come questi possono essere evitati. Infine, si descrivono metodi standardizzati software prontamente disponibile per consentire la quantificazione della degradazione della matrice gelatinosa etichettati mediata da cellule singole e intere popolazioni cellulari. Le procedure descritte consentono di monitorare accurato e riproducibile invadopodiaattività, e può anche servire come una piattaforma per la valutazione dell'efficacia di espressione della proteina modulante o prove di anti-invasivi composti sulla degradazione della matrice extracellulare in impostazioni singole e multicellulari.
Protocollo
1. Produzione di Oregon verde coprioggetto rivestiti 488-gelatina
- Preparare un marcato 5% (w / w) stock gelatina / soluzione di saccarosio con l'aggiunta di 1,25 g di gelatina e 1,25 g di saccarosio in PBS fino ad un volume finale di 50 ml. Riscaldare la soluzione di gelatina magazzino a 37 ° C ed assicurare che sia completamente sciolto prima dell'uso. Conservare la miscela finale a 4 ° C.
- Pulire diametro di 13 mm # 1 vetrini coprioggetto ponendo un individuo in ciascun pozzetto di una piastra a 24 pozzetti di plastica coltura tissutale. Aggiungere 500 pl di 20% di acido nitrico in ciascun pozzetto e incubare per 30 min. Aspirare la soluzione di acido nitrico e lavare vetrini tre volte con acqua deionizzata.
- Coprioggetti cappotto con 500 pl di 50 ug / ml di poli-L-lisina (preparata da 0,1% di soluzione stock e diluito in acqua deionizzata) a ciascun pozzetto per 20 min a temperatura ambiente. Aspirare la soluzione e lavare tre volte con PBS. Poli-L-lisina rivestimento facilita anche il rivestimento e l'adesione del sovrastante laBeled gelatina.
- Aggiungere 500 pl di 0,5% glutaraldeide (preparato fresco prima dell'uso) a ciascun pozzetto ed incubare le piastre da 24 pozzetti in ghiaccio per 15 min. Aspirare e lavare tre volte con PBS freddo. Accertarsi di rimuovere tutte le tracce di PBS prima del rivestimento gelatina. Conservare le piastre in ghiaccio durante tutti i lavaggi fino gelatina viene aggiunto.
- Ricostituire il verde Oregon 488 coniugato gelatina come protocollo del produttore e per riscaldarla e il 5% senza etichetta gelatina / soluzione di saccarosio da (1.1) a 37 ° C. Diluire una parte Oregon verde 488 gelatina in otto parti di gelatina non marcato / saccarosio (ad esempio, 500 ml di Oregon verde 488 gelatina in 4 ml di miscela di gelatina 5%). Pipettare 100 ul del diluita 488-gelatina miscela (mantenuto a 37 ° C) su ciascun vetrino, usando abbastanza gelatina per rivestire il coprioggetto senza spargimento manuale (che può portare a rivestimento coprioggetto irregolare, come mostrato in Figura 3B). È importante mantenere la diluito 488-gelatina miscela a 37 °; C durante la procedura di rivestimento per evitare la solidificazione prematura. Da questo punto in avanti i coprioggetti devono essere tenuti all'oscuro il più possibile al fine di evitare photobleaching potenziale. Proteine ECM altri coniugati a fluorofori differenti può essere sostituito per Oregon verde 488 gelatina (vedi Discussione).
- Una volta che tutti i vetrini sono rivestite in una singola piastra, tenere la piastra a 24 pozzetti in un angolo e rimuovere l'eccesso gelatina da ogni pozzetto mediante aspirazione a vuoto. Incubare coprioggetto rivestiti al buio per 10 minuti a temperatura ambiente.
- Lavare i vetrini tre volte con PBS, poi aggiungere 500 microlitri di appena fatto 5 mg / ml di sodio boroidruro (NaBH4) per 15 min a temperatura ambiente per ridurre e inattivare glutaraldeide residua. Boroidruro di sodio è effervescente, e piccole bolle sarà evidente su e intorno ad ogni coprioggetto.
- Rimuovere la soluzione di NaBH 4 per aspirazione sotto vuoto con un movimento rapido spazzare intorno alla parte esterna di ogni pozzetto. Fare attenzione a non raccogliere eventuali coprioggetto galleggianti che si è staccato dal fondo della piastra di coltura dei tessuti durante il trattamento NaBH 4. Coprioggetti indipendente che galleggiano in alto può essere delicatamente spinto indietro fino in fondo bene, ma bisogna fare attenzione per evitare di danneggiare il rivestimento proteico. Lavare i pozzetti tre volte con PBS e incubare vetrini in etanolo al 70% per 30 min a temperatura ambiente.
- Usando una tecnica sterile, trasferire i coprioggetto contenenti lastre ad un IIA / B cappa tipo cultura della cella di flusso laminare e sciacquare vetrini tre volte con PBS sterile. A questo punto coprioggetto può essere memorizzato in PBS protetto dalla luce a 4 ° C per almeno due mesi.
- Trasferire coprioggetti essere utilizzata per i saggi di degradazione di un pozzo vuoto di una nuova piastra 24 così da rimozione accurata utilizzando un ago sterile e pinza. Equilibrare coprioggetti per 1-24 ore con mezzi completi appropriate per il tipo cellulare specifico da analizzare. La cura deve essere presa nonper invertire il coprioggetto o graffiare il rivestimento di gelatina (vedi Figura 3B).
2. Placcatura ed elaborazione di celle su Oregon verde coprioggetto rivestiti 488-gelatina al degrado del saggio ECM
- Seme 3-5x10 4 cellule su un vetrino coprioggetto entro ciascun pozzetto della piastra a 24 pozzetti.
- Condurre uno studio andamento nel tempo per determinare i tempi ottimali necessarie per l'attività di degradazione invadopodia per la linea cellulare particolare / tipo di interesse. Cellule più invasive richiedono un tempo tra 4-24 h per degradazione a diventare evidente, anche se questo intervallo può variare ampiamente e dovrebbero essere determinati empiricamente. Per sincronizzare l'attività invadopodia, le cellule possono essere trattate con inibitori di MMP (es., GM 6001) per un periodo di tempo desiderato, quindi lavare l'inibitore di consentire attività invadopodia procedere (per esempio, vedi 6).
- Risciacquare coprioggetti tre volte con PBS, poi fissare le cellule con 500 pl di 10% formalina tamponata fosfate per 15 min. Risciacquare tre volte con PBS e permeabilize per 4 min con 0,4% Triton X-100 in PBS. Risciacquare tre volte con PBS per rimuovere il Triton X-100.
- Cellule Label utilizzando qualsiasi protocollo standard per la colorazione di immunofluorescenza (vedi 7, per esempio) per co-cellule con etichettatura fluorescente falloidina coniugato per visualizzare filamenti di actina (F-actina) e per una proteina marker noto che localizza invadopodia (eg; cortactin 5, TKS5 8, o N-WASp 9). Ricordate di evitare l'uso di 488 marcati anticorpi secondari marcati con GFP o proteine se usando Oregon verde 488 o marcato con FITC gelatina per evitare interferenze di segnale.
- Montare coprioggetti colorati su vetrini per microscopio con attenzione invertendo il coprioggetto e appoggiandolo su una goccia di prolungare antifade Gold o reagente simile.
- Per valutare la degradazione della matrice, celle di immagine in canali appropriati utilizzando un microscopio confocale a fluorescenza o convenzionale. Degrado Gelatinaè visualizzato come aree scure sul vetrino a causa della rimozione proteolitica della gelatina fluorescente (Figura 4A). Etichettatura di cellule di actina e una proteina marker invadopodia consente di confermare invadopodia in siti di degradazione della matrice in immagini unite (Figura 4A).
- Attività di degradazione possono essere monitorate in tempo reale da imaging cellulare in tempo reale con fluorescente-tag proteine ricombinanti per monitorare la formazione e la degradazione della matrice invadopodia 5,10,11.
3. Quantificazione dei fluorescente degradazione Gelatina di misura degradazione normalizzato Matrix
Questa analisi fornisce la zona di degradazione della matrice normalizzata rispetto alla superficie delle cellule o il numero di cellule. È utile per analizzare interi campi di vista microscopico in cui sono presenti più celle che sono state trattate con siRNA collettivamente, fattori di crescita o agenti terapeutici. Per questa analisi dells, immagini raccolte in basso ingrandimento sono sufficienti per raccogliere in modo efficiente le informazioni sulle popolazioni di cellule.
- Aprire le immagini in ImageJ 12. ImageJ per microscopia può essere scaricato dal http://www.macbiophotonics.ca/imagej/ .
- Controllare le informazioni di scala scegliendo il comando di menu "Analizza / Set Scala." Queste informazioni importerà automaticamente con molti formati di file, ma possono essere inseriti manualmente, se necessario. Scala adeguata è necessario riportare le misure in micron piuttosto che pixel.
- Selezionare le misure appropriate per tenere traccia scegliendo l'opzione "Analizza / Set Misure". Verificare area e Limite di Soglia.
- Calcolare l'area di degradazione utilizzando l'immagine fluorescente gelatina (Figura 5A).
- Soglia l'immagine ("Immagine / Regola / Threshold") per impostare la pi superiore e inferiorevalori di intensità xel per selezionare le aree di degrado (evidenziata in rosso; Figura 5B). Nelle immagini successive, utilizzare il pulsante Imposta nella finestra di Threshold per impostare la soglia stessa per tutte le immagini come uno strumento oggettivo per selezionare l'area degrado.
- In alcuni casi, il coprioggetto può non essere perfettamente piana quando le immagini vengono acquisite. Ciò causa l'intensità della gelatina per cambiare in tutta l'immagine. Se questa variante crea problemi quando di soglia l'immagine, corretta per l'illuminazione uniforme in tutta la gelatina sottraendo lo sfondo ("Processo / Sottrai Sfondo") o per filtrazione con un filtro passa-banda ("Processo / FFT / filtro passa-banda") o una pseudo flatfield filtro ("Processo / Filtri / Pseudo Flatfield") fino a quando l'intensità sfondo è uniforme.
- Misurare l'area di matrice degrado ("Analizza / Analizza Particelle"). Nella finestra Analizza particelle, scegliere una dimensione delle particelle> 0 per rimuovere il rumore da tegli selezione. Mostra delinea per identificare le regioni di interesse (ROI). Verifica dei risultati di visualizzazione e di sintesi per mostrare le misurazioni. Se il disegno è specificamente indicato tutte le aree di degrado (Figura 5C), copiare la misurazione Area totale in un foglio elettronico. Se altri oggetti sono stati selezionati (come detriti), registrare solo le aree della ROI rilevante.
- Calcolare l'area della cella utilizzando il macchiato falloidina (F-actina) immagine (Figura 5D).
- Soglia l'immagine ("Immagine / Regola / Threshold") per impostare i superiori ed inferiori valori di intensità dei pixel in modo che i bordi delle celle sono selezionate (evidenziate in rosso; Figura 5E). Nelle immagini successive, utilizzare il pulsante Imposta nella finestra di Threshold per impostare la soglia stessa per tutte le immagini come uno strumento oggettivo per selezionare l'area della cella.
- 10 Misura l'area delle celle ("Analizza / Analizza Particelle"). Nel Analizza Partifinestra di Cles, scegliere una dimensione delle particelle> 0 per rimuovere il rumore dalla selezione. Mostra delinea per identificare le regioni per l'analisi (Figura 5F). Verifica dei risultati di visualizzazione e di sintesi per mostrare le misure della superficie. Non controllare includono fori se vi sono spazi tra le cellule in un cluster così i pixel non selezionati all'interno del cluster non verrà inclusa nel calcolo di celle. Scegliere OK.
- Copiare i risultati per superficie per il ROI rilevante in un foglio elettronico.
- Calcolare l'area di degrado gelatina per area totale di celle 13.
- Un approccio alternativo sarebbe di segnalare l'area di degradazione per numero di cellule da contare nuclei (Figura 5G). Questo è necessario se manipolazioni modificare l'area di cella tra diversi gruppi di trattamento rispetto. Conteggio automatico funziona meglio se i nuclei sono ben separati, uniforme di intensità e rotondo. Automaticamente contare nuclei ("Plugins / particelle Analisi / Contatore Nucleo"). Scegli la misura delle particelle più piccolo e più grande, un metodo di soglia e di un metodo di livellamento. Controllare Sottrarre sfondo, filtro Watershed, Aggiungi Particelle Manager ROI e Sommario Visualizza (Figura 5H).
- Se i nuclei si sovrappongono ampiamente o avere una forma irregolare o della trama, il conteggio automatico non può produrre un conteggio accurato (Figura 5H, frecce sulla destra). In questo caso, il conteggio manuale può essere facilitata utilizzando lo strumento contatore di cellule ("Plugins / particella Analisi / contatore di cellule"). Ciò manterrà conteggio come cellule sono contrassegnati durante un conteggio manuale (Figura 5I).
- Copiare il numero di cellule (nuclei) in un foglio elettronico. Calcolare l'area di degrado gelatina per numero totale di cellule.
4. Quantificazione dei fluorescente degradazione Gelatina di singole celle in una popolazione mista cellulare
Per valutare la degradazione della matrice risultante da cellule specifiche in una popolazione a parte altre celle nel campo (per esempio, rispetto a non transfettate cellule trasfettate), la procedura nella sezione 3 può essere modificato per misurare l'area di degrado in cellule singole. Un canale supplementare fluorescente è necessaria per marcare cellule trasfettate. In questo caso, le immagini maggiore ingrandimento e separate le cellule sono più facili da quantificare.
- Controllare le informazioni di scala scegliendo il comando di menu "Analizza / Set Scala." Selezionare le misure appropriate per tenere traccia scegliendo l'opzione "Analizza / Set Misure". Verificare area e Limite di Soglia.
- Per le singole celle che non si toccano, identificare ciascuna cella utilizzando la F-actina immagine (Figura 6A). Threshold l'immagine (vedere 3.9) (Figura 6B). È importante per catturare i bordi delle celle, ma ci possono essere foros all'interno che non sono inclusi nella soglia. Utilizzare i valori di intensità stessi attraverso le immagini per selezionare i bordi delle celle.
- Per misurare l'area delle celle, utilizzare "Analizza / analizzare particelle." Nella finestra Analizza particelle, scegli una taglia> 0 (per eliminare il disturbo), contorni Mostra, e controllare i risultati di visualizzazione, Aggiungi al Manager e sono dotate di fori (per la registrazione l'intera area all'interno del contorno). Scegliere OK e registrare la zona per ogni cella dalla finestra Risultati.
- Identificare quali cellule sono trasfettate (figura 6C).
- Identificare le aree di degrado utilizzando l'immagine fluorescente gelatina (Figura 6D). Se necessario, filtrare l'immagine gelatina per uniformare l'intensità di fondo (cfr. punto 3.6). Soglia per selezionare le aree di degrado, rendendo nota delle impostazioni di soglia (Figura 6E). Nelle immagini successive, utilizzare questi stessi valori di intensità superiore e inferiore (con il Setnella finestra Soglia) per una selezione oggettiva di aree di degrado.
- Misurare le aree di degradazione delle cellule. Sul thresholded immagine fluorescente gelatina, mostrano un profilo delle celle selezionando ROI nella finestra Gestione ROI e selezionare Misura (Figura 6F). Registrare i risultati e calcolare l'area normalizzato di degradazione / cella o area di cella.
5. Risultati rappresentativi
Lo schema generale della procedura è mostrato in Figura 1. La procedura implica la preparazione di vetrini e rivestimento con fluorescenza coniugato gelatina, placcatura di cellule sul coprioggetto rivestiti per permettere alle cellule di degradare la gelatina, il fissaggio e l'etichettatura delle cellule per analisi al microscopio a fluorescenza, la matrice di imaging fluorescente per valutare l'integrità della matrice, e oggettivamente quantificare il grado di degradazione della matrice di gelatina utilizzando il computer software.

Figura 1. Schema globale evidenziando i passaggi chiave coinvolti nella gelatina fluorescente rivestimento, placcatura cellulare, fissaggio e immunomarcatura, e la valutazione proteolisi matrice.
I passaggi chiave del procedimento coinvolti nella preparazione e coprioggetto rivestimento sono illustrate nella Figura 2.
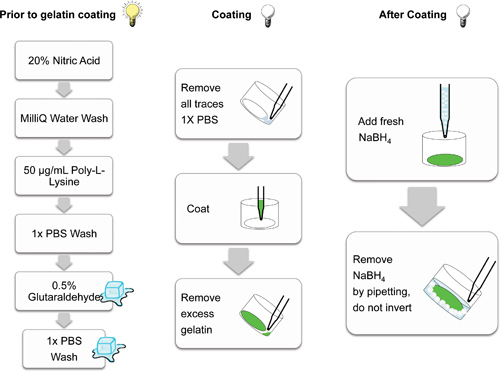
Figura 2. Schema dimostrazione dei singoli passaggi coinvolti nella preparazione vetrini per il rivestimento matrice di gelatina. Passi condotte nella luce (lampadina accesa), su ghiaccio (cubetti) e al buio (non illuminato lampadina) sono indicati cartone animato. Passi condotte nella guida buio prevenire photobleaching delle matrici fluorescenti.
Se correttamente eseguita, coprioggetto sono uniformemente rivestiti con Oregon verde 488-coniugato gelatin, visualizzando fluorescenza omogenea quando visualizzabili al microscopio (Figura 3A). Artefatti tipici che si possono verificare a causa di rivestimento improprio, la manipolazione, lo stoccaggio e l'utilizzo di vetrini rivestiti è riportata nella figura 3B.
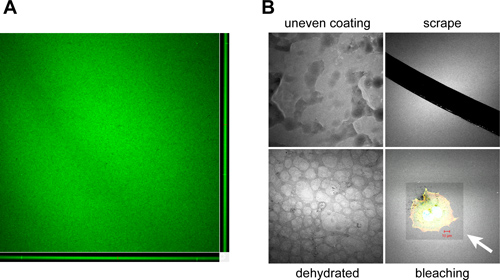
Esempi Figura 3. Di manufatti incontrati durante la preparazione di gelatina coprioggetto rivestito e la manipolazione. A. vista ortogonale di un confocale z-stack che mostra il tipico colore e la consistenza di un verde Oregon 488-coniugato coprioggetto gelatina rivestita prodotta utilizzando il protocollo prescritto. Coprioggetti dovrebbe avere un rivestimento omogeneo ~ 1-2 micron di spessore come mostrato nella XZ (basso) e YZ (destra) piani confocali B. artefatti che possono verificarsi durante il rivestimento e trattamento di gelatina rivestite coprioggetto includono:. Copertura improprio del coprioggetto durante il processo di rivestimento a causaalla scarsa miscelazione, solidificazione manuale diffusione o parziale della miscela di gelatina (non uniforme), rimozione della matrice rivestita segnando con aghi o pinze durante la movimentazione (scrape), essiccamento della superficie coprioggetto durante periodi di stoccaggio prolungati, con conseguente "cobblestone "aspetto (disidratato) e photobleaching della superficie fluorescente gelatina durante l'esposizione a causa di esposizione prolungata o ad alta intensità di luce (sbiancamento). Freccia bianca indica la zona che comprende una testa sbiancato placcato OSC19 e carcinoma a cellule squamose del collo. L'Oregon verde 488-coniugato gelatina è pseudocolored bianco per aumentare il contrasto dell'immagine. Bar, 10 micron.
Le matrici risultanti sottili prodotte durante questa procedura fornisce un mezzo sensibile per valutare la capacità delle cellule di degradare ECM. Figura 4 illustra un esempio di attività invadopodia da una cellula OSC19 piastrate su un verde-488 Oregon coprioggetto coniugato gelatinae fotografato dal convenzionale microscopia confocale nonché in volume di riempimento di rendering seguendo tre deconvoluzione dimensionale.

Figura 4. Esempi rappresentativi di attività invadopodia degradazione della matrice. A. Visualizzazione di invadopodia e proteolisi corrispondente matrice di gelatina. OSC19 cellule placcato su Oregon verde vetrini di gelatina 488-coniugati per 10 ore sono stati fissati ed etichettati con rodamina-coniugato falloidina (F-actina) e anti-cortactin anticorpi (visualizzato con un Alexa Fluor 647 anticorpo secondario e pseudocolored verde). Invadopodia sono evidenti come focali concentrazioni citoplasmatiche di F-actina e cortactin che si sovrappongono con le aree di gelatina di compensazione (buchi neri nella matrice) all'interno l'immagine unita. Regioni cofanetto contenente frecce indicano invadopodia individuale e aree di proteolisi matrice focale come mostrato nella ri allargataregioni inferiori. Bar, 10 micron. B.. Volume di riempimento visualizzazione di penetrazione invadopodia nella ECM. OSC19 cellule placcato e colorati come in (A) sono stati resi visivamente mediante l'ottenimento di 23 successive 0,32 micron ottici z-fette per un totale di 7,04 micron per rodamina-coniugato falloidina e Oregon verde 488-coniugato gelatina. Il file LSM nativo impostare per ogni canale è stato aperto nel AutoQuant X2.2 software e un 3D deconvoluzione cieca di ogni pila di immagini è stata effettuata utilizzando le impostazioni consigliate (10 iterazioni, rumore medio). Le immagini elaborate sono state salvate come TIFF pile che poi sono stati aperti in Elementi di sicurezza delle reti e visualizzate come una visione volume con alpha blending. Le LUT sono stati adeguati, e un sottovolume è stato creato per mostrare un bordo all'interno della cella in cui invadopodia sono presenti. Dorsale-edge vista dimostra invadopodia (rosso, frecce) inserito nel sottostante gelatina (verde). Ventrale-edge vista mostra invadopodia sporgente e le aree di degrado gelatina sotto il coprioggettocome regioni di regalo rosso nella matrice verde (punte di freccia). Il campo di immagine totale presentato è tagliata a 77 x 65 micron, la cella è ~ 60 x 40 micron.
La figura 5 mostra alcuni dei passi importanti per la quantificazione della degradazione della matrice normalizzato gelatina come descritto nel passaggio 3 del protocollo. Questa procedura è progettato per consentire la quantificazione obiettiva degradazione gelatina in un intero campo di vista, ed è adatto per la degradazione della matrice attribuita a molte cellule all'interno del campo.
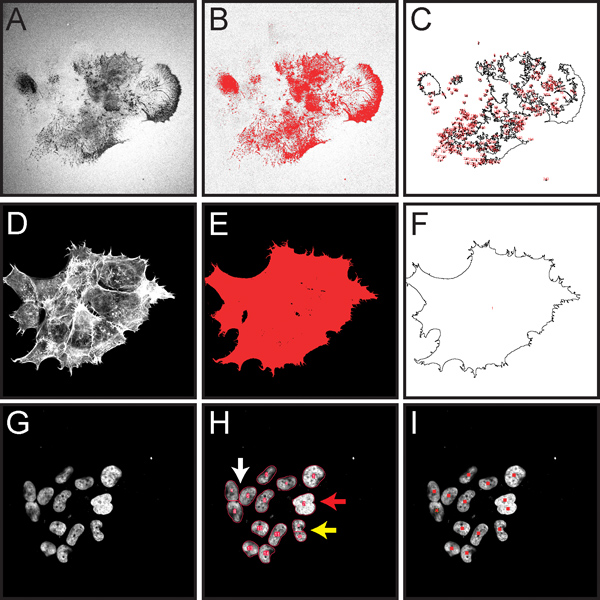
Figura 5. Schermo catturare immagini che dimostrano i passaggi chiave nel calcolo assistito quantificazione normalizzata degrado gelatina per le celle all'interno di una intera immagine al microscopio come descritto nella fase del protocollo 3. Tutte le immagini fluorescenti sono stati convertiti in scala di grigi per visualizzare meglio la soglia rossa e marcature ROI. A. Imetà di Oregon verde 488-coniugato gelatina, che indichi le zone scure ("buchi") in cui il degrado è verificato (punto 3.4). B. Thresholded immagine gelatina evidenziare le aree di degrado in rosso (fase 3.5). Disegno C. mostra ROI misurata per l'area di degradazione (passo 3,7). D. Rhodamine colorazione phalloidin di F-actina (passo 3,8). E. Thresholded immagine actina evidenziando superficie totale cella in rosso (passo 3.9). Disegno F. mostrando aree cellulari da misurare (step 3,10) . G. Immagine di DAPI macchiato di nuclei di cellule (passo 3.13). H. Rosso delinea evidenziano risultati di conteggio automatico nuclei (passo 3.13). Il filtro Watershed ha il potenziale per nuclei separati che si tocchino (freccia bianca). Se i nuclei si sovrappongono ampiamente, essi non possono essere separati in singoli oggetti (freccia rossa). Se un nucleo ha una forma irregolare, può essere separato in più oggetti (freccia gialla). I. </ Risultati> forti nuclei di marcatura nel corso di un conteggio manuale utilizzando lo strumento contatore di cellule (passo 3.14).
Figura 6. dimostra passi selezionare coinvolti nella degradazione quantificazione fluorescente gelatina da singole celle all'interno di una popolazione cellulare mista come descritto nel passaggio protocollo 4. Qui, la degradazione della matrice da cellule transfettate possono essere analizzati in una popolazione mista di cellule trasfettate e non transfettate.
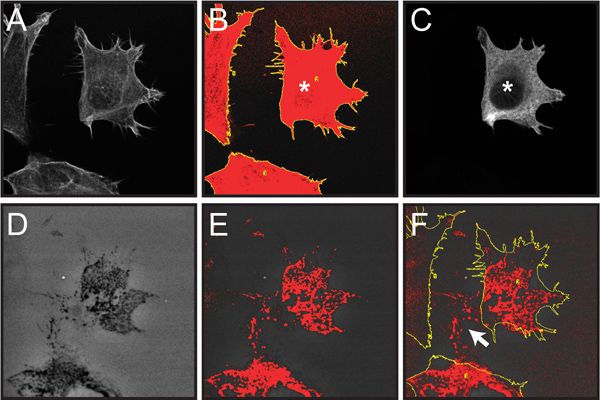
Figura 6. Schermata catturare immagini di passaggi necessari per quantificare il degrado gelatina da singole cellule transfettate all'interno di una popolazione di cellule. Quantificazione di una singola cellula trasfettata cortactin OSC19 overexpressing ricombinante fusa al tag epitopo FLAG è mostrato come esempio. Tutte le immagini fluorescenti sono stati convertiti in scala di grigi per visualizzare meglio la soglia rossa e marchi ROI gialli. A. immagine confocale di tre celle marcate con rodamina-falloidina (fase 4.2). B. Disegno della superficie cellulare totale sulla base di F-actina colorazione dopo l'applicazione della soglia e analizzare le funzioni di particelle (passo 4,2-3). C. immagine confocale di anti-FLAG immunomarcatura della popolazione di cellule dimostrando una singola cellula che esprime FLAG-tagged cortactin (contrassegnati con *) (fase 4.4). D. Immagine di Oregon verde 488-coniugato gelatina, che indichi le zone scure ("buchi") in cui il degrado ha si è verificato (fase 4.5) E. Thresholded immagine gelatina mettendo in evidenza le aree scure di degrado in rosso (fase 4.5). F. Thresholded sovrapposto gelatina immagine con contorni cellulari da pannello B (passo 4.6). Si noti che solo i pixel all'interno thresholded i contorni cellulari sono conteggiati nell'analisi. Aree di degrado al di fuori della posizione della cella corrente (freccia bianca) derivano da migrazione delle cellule attraverso la gelatina nel tempo e non sono incluDED nell'analisi.
Discussione
La possibilità di visualizzare le cellule che degradano la matrice extracellulare ha aiutato a scoprire i meccanismi molecolari impiegati nelle prime fasi di invasione cellulare. Lanciato da Wen-Tien Chen nei primi anni 1980 4,14,15, rivestimento proteine fluorescenti marcate extracellulari su vetrini per la successiva analisi al microscopio è emerso come la tecnica principale nella valutazione della funzione invadopodia in una vasta gamma di tipi di cellule. Il protocollo prescritto viene illustrato ...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Questo lavoro è stato sostenuto da fondi di dotazione della West Virginia University Mary Babb Randolph Cancer Center. Ringraziamo Susette Mueller (Georgetown University) e Laura Kelley per la consulenza precoce e assistenza. L'uso dello strumento di imaging Virginia Occidentale Microscopia University (supportato dal Babb Mary Randolph Cancer Center e, sovvenzioni NIH RR16440 P20, P30 e P30 RR032138 GM103488) è riconosciuto con gratitudine.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente / strumento | Azienda | Numero di catalogo | Commenti |
| gelatina | Sigma | G1890 | Suini pelle |
| saccarosio | Pescatore | BP220 | |
| Coprioggetto 12 millimetri | Pescatore | 50-121-5159 | |
| Piastre da 24 pozzetti | Pescatore | 08-772-1 | BD Falcon |
| acido nitrico | Ricca | R5326000 | Soluzione al 20% |
| poli-L-lisina | Scienze della microscopia elettronica | 19320-B | 0,1% soluzione |
| glutaraldeide | Sigma | G7526 | Soluzione 8% |
| Oregon verde 488-coniugato gelatina | Invitrogen | G-13186 | |
| sodio boroidruro | Sigma | 213462 | |
| Triton X-100 | Pescatore | BP151 | |
| rodamina-falloidina | Invitrogen | R415 | |
| anti-cortactin (clone 4F11) | Millipore | 05-180 | |
| Anticorpo anti-FLAG | Millipore | MAB3118 | |
| Alexa Fluor 647 di capra anti-IgG di topo | Invitrogen | A21235 | |
| Prolong antifade Oro | Invitrogen | P36930 | |
| LSM 510 microscopio confocale | Zeiss | ||
| ImageJ software | Di dominio pubblico | http://www.macbiophotonics.ca/imagej/ | |
| AutoQuant X2.2 software | Media Cibernetica | ||
| NIS Elements | Nikon |
Riferimenti
- Ridley, A. J. Life at the leading edge. Cell. 145, 1012-1022 (2011).
- Murphy, D. A., Courtneidge, S. A. The 'ins' and 'outs' of podosomes and invadopodia: characteristics, formation and function. Nat. Rev. Mol. Cell Biol. 12, 413-426 (2011).
- Linder, S., Wiesner, C., Himmel, M. Degrading Devices: Invadosomes in Proteolytic Cell Invasion. Annu. Rev. Cell. Dev. Biol. , (2010).
- Chen, W. T., Chen, J. M., Parsons, S. J., Parsons, J. T. Local degradation of fibronectin at sites of expression of the transforming gene product pp60src. Nature. 316, 156-158 (1985).
- Artym, V. V., Zhang, Y., Seillier-Moiseiwitsch, F., Yamada, K. M., Mueller, S. C. Dynamic interactions of cortactin and membrane type 1 matrix metalloproteinase at invadopodia: defining the stages of invadopodia formation and function. Cancer Res. 66, 3034-3043 (2006).
- Ayala, I., et al. Multiple regulatory inputs converge on cortactin to control invadopodia biogenesis and extracellular matrix degradation. J. Cell Sci. , (2008).
- Ammer, A. G., et al. Saracatinib impairs head and neck squamous cell carcinoma invasion by disrupting invadopodia function. J. Cancer Sci. Ther. 1, 52-61 (2009).
- Seals, D. F., et al. The adaptor protein Tks5/Fish is required for podosome formation and function, and for the protease-driven invasion of cancer cells. Cancer Cell. 7, 155-165 (2005).
- Yamaguchi, H., et al. Molecular mechanisms of invadopodium formation: the role of the N-WASP-Arp2/3 complex pathway and cofilin. J. Cell Biol. 168, 441-452 (2005).
- Kopp, P., et al. The kinesin KIF1C and microtubule plus ends regulate podosome dynamics in macrophages. Mol. Biol. Cell. 17, 2811-2823 (2006).
- Oser, M., et al. Cortactin regulates cofilin and N-WASp activities to control the stages of invadopodium assembly and maturation. J. Cell. Biol. 186, 571-587 (2009).
- Abramoff, M. D., Magalhaes, P. J., Ram, S. J. Image Processing with ImageJ. Biophotonics International. 11 (7), 36-42 (2004).
- Kelley, L. C., et al. Oncogenic Src requires a wild-type counterpart to regulate invadopodia maturation. J. Cell Sci. 123, 3923-3932 (2010).
- Chen, W. T., Singer, S. J. Fibronectin is not present in the focal adhesions formed between normal cultured fibroblasts and their substrata. Proc. Natl. Acad. Sci. U.S.A. 77, 7318-7322 (1980).
- Chen, W. T., Olden, K., Bernard, B. A., Chu, F. F. Expression of transformation-associated protease(s) that degrade fibronectin at cell contact sites. J. Cell Biol. 98, 1546-1555 (1984).
- Bharti, S., et al. Src-dependent phosphorylation of ASAP1 regulates podosomes. Mol. Cell Biol. 27, 8271-8283 (2007).
- Albrechtsen, R., Stautz, D., Sanjay, A., Kveiborg, M., Wewer, U. M. Extracellular engagement of ADAM12 induces clusters of invadopodia with localized ectodomain shedding activity. Exp. Cell Res. 317 (10), 195-209 (2011).
- Scott, R. W., et al. kinases are required for invasive path generation by tumor and tumor-associated stromal cells. J. Cell Biol. 191, 169-185 (2010).
- Mueller, S. C., Yeh, Y., Chen, W. T. Tyrosine phosphorylation of membrane proteins mediates cellular invasion by transformed cells. J. Cell. Biol. 119, 1309-1325 (1992).
- Bowden, E. T., Coopman, P. J., Mueller, S. C. Invadopodia: unique methods for measurement of extracellular matrix degradation in vitro. Methods Cell Biol. 63, 613-627 (2001).
- Baldassarre, M., et al. Dynamin participates in focal extracellular matrix degradation by invasive cells. Mol. Biol. Cell. 14, 1074-1084 (2003).
- Alexander, N. R., et al. Extracellular matrix rigidity promotes invadopodia activity. Curr. Biol. 18, 1295-1299 (2008).
- Artym, V. V., Yamada, K. M., Mueller, S. C. ECM degradation assays for analyzing local cell invasion. Methods Mol. Biol. 522, 211-219 (2009).
- Schoumacher, M., Goldman, R. D., Louvard, D., Vignjevic, D. M. Actin, microtubules, and vimentin intermediate filaments cooperate for elongation of invadopodia. J. Cell Biol. 189, 541-556 (2010).
- Clark, E. S., Whigham, A. S., Yarbrough, W. G., Weaver, A. M. Cortactin is an essential regulator of matrix metalloproteinase secretion and extracellular matrix degradation in invadopodia. Cancer Res. 67, 4227-4235 (2007).
- Yamaguchi, H., et al. Phosphoinositide 3-kinase signaling pathway mediated by p110alpha regulates invadopodia formation. J. Cell Biol. 193, 1275-1288 (2011).
- Li, A., et al. The actin-bundling protein fascin stabilizes actin in invadopodia and potentiates protrusive invasion. Curr. Biol. 20, 339-345 (2010).
- Yamaguchi, H., et al. Phosphatidylinositol 4,5-bisphosphate and PIP5-kinase Ialpha are required for invadopodia formation in human breast cancer cells. Cancer Sci. , (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon