È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Ad alta risoluzione strutturale Risonanza Magnetica del subcortex Umana
In questo articolo
Riepilogo
Here we present a protocol to determine the minimum number images that needed to be registered and averaged to resolve subcortical structures and test whether the individual layers of the LGN could be resolved in the absence of physiological noise.
Abstract
Il focus di questo studio è stato quello di testare i limiti di risoluzione della risonanza magnetica strutturale di un cervello post-mortem rispetto a vivere il cervello umano. La risoluzione della risonanza magnetica strutturale in vivo è in ultima analisi limitato dal rumore fisiologico, tra cui la pulsazione, la respirazione e il movimento della testa. Sebbene hardware di imaging continua a migliorare, è ancora difficile da risolvere strutture sulla scala millimetrica. Ad esempio, il primario visiva vie sensoriali sinapsi al corpo genicolato laterale (LGN), un relè di controllo visivo e nucleo nel talamo che normalmente è organizzato in sei strati monoculare interfogliati. Studi di neuroimaging non sono stati in grado di distinguere in modo affidabile questi strati a causa delle loro piccole dimensioni che sono di spessore inferiore a 1 mm.
Il limite di risoluzione di risonanza magnetica strutturale, in un cervello post-mortem è stata testata utilizzando più immagini mediate su una lunga durata (~ 24 ore). Lo scopo era quello di verificare se era possibile risolvere l'individuo layers del LGN in assenza di rumore fisiologico. Una densità protonica (PD) 1 sequenza di impulsi ponderato è stato utilizzato con diversi risoluzione e altri parametri per determinare il numero minimo di immagini necessarie per essere registrati e una media di distinguere in modo affidabile l'LGN e altre regioni sottocorticali. I risultati sono stati confrontati con le immagini acquisite nel vivere cervello umano. In vivo soggetti sono stati sottoposti a scansione per determinare gli effetti aggiuntivi di rumore fisiologica del numero minimo di scansioni PD necessari per differenziare strutture sottocorticali, utili in applicazioni cliniche.
Introduzione
Lo scopo di questa ricerca era di testare i limiti di risoluzione di MRI strutturale in assenza di rumore fisiologico. Proton densità (PD) immagini pesate sono state acquisite in un cervello post-mortem su una lunga durata (due ~ 24 sessioni hr) per determinare il numero minimo di immagini che dovevano essere registrati e una media di risolvere le strutture sottocorticali. Per confronto, PD immagini pesate sono stati acquisiti negli esseri umani che vivono su un numero di sessioni. In particolare, l'obiettivo è stato quello di verificare se sia possibile in un migliore dei casi per risolvere tutti i sei strati individuali del LGN umana, che sono circa 1 mm di spessore (Figura 1).
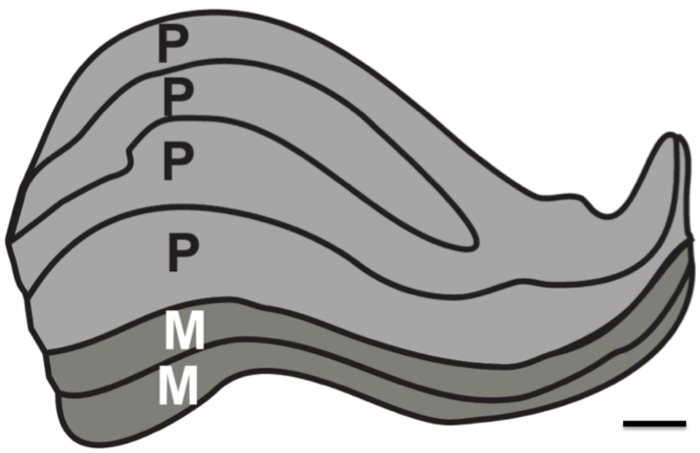
Figura 1. strati umano corpo genicolato laterale. Schematica della struttura laminare del LGN. Magnocellulari (M) strati sono costituiti da grandi neuronaledimensione della cella e densità cellulare più piccolo che sono responsabili per la risoluzione linee di movimento e del corso (layer 1-2, raffigurato in grigio scuro). Strati parvocellular (P) sono costituiti da dimensioni più piccole delle cellule neuronali e più grande densità cellulare che sono responsabili per la risoluzione fine-forma e colore (strati 4-6, raffigurato come grigio chiaro). Barra di scala 1 mm. Figura sulla base macchiato LGN umano 12.
La risoluzione spaziale di MRI è migliorata quando la dimensione della matrice è aumentata, e quando il campo-visivo (FOV) e spessore fetta sono diminuiti. Tuttavia, maggiore risoluzione diminuisce il rapporto segnale-rumore (SNR), che è proporzionale al volume voxel. SNR è anche proporzionale alla radice quadrata del numero di misurazioni. Negli esseri umani viventi, anche se più immagini possono essere acquisite nel corso di un numero di sessioni di imaging separati, la risoluzione massima è limitata dal rumore fisiologico, come la respirazione, pulsazioni circolatorio e movimento della testa.
Alto-Risoluzione (0,35 mm nel piano voxel) PD scansioni ponderati sono stati acquisiti. Scansioni PD migliorare grigio e bianco contrasto nel talamo 1, e producono immagini che minimizzano T 1 e T 2 effetti. La sua immagine dipende dalla densità di protoni in forma di acqua e macromolecole come proteine e grassi nel volume di imaging. L'aumento del numero di protoni in un risultato di tessuto in un segnale luminoso sopra l'immagine a causa della componente superiore longitudinale della magnetizzazione 2.
Scansioni PD ponderata sono stati raccolti in quanto forniscono un contrasto più elevato di strutture sottocorticali con il tessuto circostante. Altri contrasti, come T1 e immagini T2 provocano difficoltà nel delineare strutture sottocorticali come il LGN a causa di rapporti più piccoli contrasto-rumore, come determinato ƒ 1,3.
Allo stesso modo, studi precedenti hanno trovato che le immagini PD ponderata di formalina fisso cervelli post mortem resulted a differenze di contrasto più elevati tra materia grigia e bianca rispetto al T1 e immagini T2 che avevano simili grigio e bianco intensità dell'immagine materia 3,4. Le determinanti biofisiche sottostanti possono spiegare queste differenze. T1 (longitudinale) e T2 (trasversale) tempi di rilassamento dei protoni di idrogeno dipenderà da come l'acqua si muove all'interno del tessuto. Fissativi come il lavoro formalina da proteine cross-linking. Le differenze tra la mobilità dell'acqua sono ridotti tra i vari tipi di tessuto quando si usano fissativi. Contrasto dei tessuti T1 ridotto è stata osservata dopo la fissazione, mentre le differenze nella densità relativa di protoni all'interno tessuti cerebrali aumentati con la fissazione, fornendo una migliore differenziazione di contrasto 3, 4.
Precedenti studi hanno identificato il LGN in scansioni PD ponderato utilizzando un T 5,6,7 1.5, e alle 3 T scanner 8,9. È fondamentale per ottenere queste scansioni per poter delineare esattamente la portataLGN. Per mantenere la piena copertura dei nuclei sottocorticali, 18 fette PD ponderate sono state ottenute nel talamo. Ogni volume è stato ri-campionato a due volte la matrice 1024, (0,15 mm-aereo di dimensioni voxel), concatenati, movimento corretto e una media per produrre una alta risoluzione di immagini 3D delle strutture sottocorticali. Il numero ottimale di immagini PD richiesti per la seguente ricetta fetta era 5, riducendo il tempo di scansione a meno di 15 min in esseri umani viventi. Solo 1 immagine PD è stato richiesto di delimitare chiaramente le regioni subcorticali del cervello post-mortem, riducendo i tempi di scansione a meno di 3 minuti (Figura 2 e 3).
Un intero post mortem campione cervello fissati in formalina è stato scansionato da una donna che era morta di arresto cardiopolmonare all'età di 82 anni. Rassegna di documentazione medica ha rivelato che aveva: cronica malattia polmonare ostruttiva, chirurgia angina, triplo bypass 8 anni prima della morte, il cancro uterino trattati con isterectomia7 anni prima della morte, iperlipidemia, glaucoma, e la chirurgia della cataratta. Il campione è stato postmortem cervello immersione fissato in 10% formalina tamponata neutra per almeno 3 settimane a 4 ° C. Il cervello postmortem stato scansionato con lo stesso protocollo di imaging e con altri parametri nel corso di molte ore per il confronto di qualità dell'immagine . Verranno descritti i parametri ottimizzati per il protocollo.
Protocollo
1. partecipante e postmortem cervello Set-Up
NOTA: Tutte le immagini sono state acquisite utilizzando una T MRI scanner 3 con una bobina testa 32 canali e tutti scansione MRI è stata eseguita a temperatura ambiente, circa 20 ° C. Tutti i partecipanti erano di mano destra e hanno firmato un consenso informato. Ogni partecipante era in buona salute senza storia di disturbi neurologici. Il protocollo sperimentale è stato approvato e segue le linee guida di partecipanti York University umani Review Committee.
- Chiedi ogni partecipante di compilare e firmare un modulo di consenso del paziente che descrive le linee guida di sicurezza MRI e il protocollo di neuro-imaging.
- Per ogni partecipante, posizionare tappi per le orecchie in ciascun orecchio e garantire la loro testa con pastiglie per ridurre al minimo il movimento della testa.
- Per post mortem di imaging cerebrale, assicurarsi che il cervello è fissato prima di neuroimaging e è contenuto all'interno di un sacchetto o contenitore che si inserisce all'interno della testa-coil MRI. Posizionare il cervello post-mortem in testa-bobina con il suo asse z (superiore a inferior) allineato con il foro dello scanner. Il tronco encefalico (posteriore) deve essere rivolto verso i piedi del letto dello scanner.
- Mettere le mani cuscino vuoto intorno al cervello post mortem per ulteriore supporto.
2. Localizzazione e prescrizione subcortex
NOTA: Il talamo è una struttura a doppia lobate situato vicino al centro del cervello situata tra il mesencefalo e la corteccia cerebrale. Situato all'interno del talamo dorsale, LGN umano è una piccola struttura sottocorticale che si estende un massimo di ~ 10 mm.
- Per registrare un nuovo partecipante, aprire il software di imaging MRI e fare clic sulla scheda del paziente in alto a sinistra. Quindi fare clic su Registrati.
- Compilare le informazioni del paziente appropriato e quindi fare clic sulla scheda esame.
- Per ottenere una scansione localizzatore, fare clic sulla scheda esame Explorer per creare un nuovo protocollo. Osservare la finestra di set-up sullo schermo, fare clic sulla scheda di routine, e inserire i seguenti parametri: acquisizionetempo 28 sec, matrice di acquisizione di 160 × 160, 1 fetta, spessore di 1,6 mm le dimensioni voxel isotropico, FOV = 260 mm, fase FoV = 100%, risoluzione fetta = 69%, di fase e affettare fase parziale di Fourier = 6/8, TR = 3.15 ms, TE = 1.37 msec, Flip Angle = 8 °.
- Sovrapporre la casella di selezione della fetta utilizzata per acquisire le immagini PD sul localizzatore che coprono i nuclei subcorticali all'interno del talamo così come le strutture circostanti (Figura 4).
3. ad alta risoluzione dei parametri strutturali
- Creare un nuovo protocollo per ottenere scansioni PD ponderata ad alta risoluzione. Nella finestra di set-up sullo schermo, fare clic sulla scheda di routine, e immettere i seguenti parametri nel orientamento coronale: tempo di acquisizione 179 sec, matrice di acquisizione di 512 × 512, 0,3 × 0,3 × 1 mm 3 dimensioni voxel, TR = 3.25 sec , TE = 32 msec, flip angle = 120 °, acquisizione fetta intercalata, FoV leggere = 160 mm, FoV fase = 100%, imaging parallelo (grappa) conun fattore di accelerazione di 2.
- Utilizzare un Turbo Spin Echo sequenza, con una lunghezza Eco Treno di 5. Il primo eco a 32 msec è l'eco efficace per questa sequenza. Ridurre la larghezza di banda (BW) al minimo possibile, 40 Hz / pixel, per massimizzare il SNR. Per ridurre la durata della scansione, scegliere 18 fette, ogni 1 mm di spessore, con un FOV = 160 mm. Questa lastra fornisce una copertura sufficiente delle regioni subcorticali di interesse.
NOTA: Per l'identificazione affidabile di strutture sottocorticali, acquisire 5 piste con i parametri di cui sopra. La durata della scansione totale è soltanto ~ 15 min (Figura 5). Fat-saturazione non è stato impiegato.
- Utilizzare un Turbo Spin Echo sequenza, con una lunghezza Eco Treno di 5. Il primo eco a 32 msec è l'eco efficace per questa sequenza. Ridurre la larghezza di banda (BW) al minimo possibile, 40 Hz / pixel, per massimizzare il SNR. Per ridurre la durata della scansione, scegliere 18 fette, ogni 1 mm di spessore, con un FOV = 160 mm. Questa lastra fornisce una copertura sufficiente delle regioni subcorticali di interesse.
- In post-mortem di imaging cerebrale, identificazione certa delle strutture sottocorticali può essere osservato in una sola scansione con la durata totale di soli ~ 3 min seguendo lo stesso protocollo di scansione come in 3.1 (Figura 6).
Analisi 4. Immagine
NOTA: Per analizzare i dati MRI, utilizzare il FMRIB liberamente disponibili diSoftware Library (FSL) pacchetto disponibile per il download all'indirizzo (https://www.fmrib.ox.ac.uk/fsl/).
- Aprire una finestra di terminale, e convertire i file DICOM prime dallo scanner per ogni volume PD in un formato NIfTI con DICOM al convertitore NIfTI. Un certo numero dei quali sono liberamente disponibili per il download (ad es. Https://www.nitrc.org/projects/mricron). Nella riga di comando, digitare dcm2nii seguito dalla directory di ogni corsa PD immagine ponderato.
- In una finestra terminale ottenere i parametri della scansione PD originale. Digitare fslinfo nella riga di comando, seguito dalla scansione PD in formato NIfTI.
- Creare un alta risoluzione di immagine vuota volume bersaglio che ha due volte la risoluzione e la metà delle dimensioni voxel dato dai parametri dal fslinfo dalla scansione PD originale. L'ordine di input dei dati per questo comando sono i seguenti:
fslcreatehd
NOTA: Per esempio, se la scansione PD originale con i seguenti parametri come descritto al punto 3.1 sono raccolti (vale a dire, 512 × 512 a matrice, 18 fetta, 0,3 × 0,3 × 1 mm 3 dimensioni voxel, TR = 3,25 s), digitare il seguente nella finestra di comando:
fslcreatehd 1024 1024 36 1 0.15 0.15 3.25 0.5 0 0 0 4 blankhr.nii.gz - Definire la trasformazione utilizzando una matrice di identità. Digitare in qualsiasi editor di testo un file di testo salvato come 'identity.mat' che assomiglia a questo:
0 0 0
1 0 0
0 1 0
0 0 1 - Utilizzare il comando flirtare per applicare la trasformazione, si aumenta la frequenza ogni esecuzione ponderata PD originale di raddoppiare la risoluzione totale da una 512 ad una matrice 1024 e dimezzare le dimensioni voxel in ogni dimensione con conseguente risoluzione di 0,15 × 0,15× 0,5 millimetri 3. In una finestra di terminale per ogni volume PD, digitare il comando seguente flirtare cambiando i nomi originali e di uscita per corsa:
flirtare -interp sinc -in originalPD.nii.gz -ref blankhr.nii.gz -applyxfm -init identity.mat -out highresPD.nii.gz
NOTA: Dove originalPD.nii.gz è il volume della sorgente, blankhr.nii.gz è la risoluzione di uscita desiderata, e highresPD.nii.gz è il nome del volume di uscita. - Spostare tutte le immagini ad alta risoluzione in una nuova cartella, e creare una rotta in una finestra di terminale.
- Per ogni partecipante, concatenare tutte le immagini PD sovracampionati in un unico file 4D utilizzando fslmerge. In un tipo di finestra di terminale:
fslmerge -t concat_highresPD * .nii.gz
NOTA: Questo crea un file chiamato 4D concat_highresPD.nii.gz. - Movimento correggere il file concatenato utilizzando mcflirt 10. Questo strumento consente una registrazione robusta automatizzato per lineare (affine) immagini del cervello inter e intermodali. Seleziona unCorrezione 4 stadi, che utilizza l'interpolazione sinc (internamente) come un ulteriore passaggio di ottimizzazione per una maggiore precisione. In un tipo di finestra di terminale:
mcflirt -in concat_highresPD -out mcf_concat_highresPD.nii.gz -stages 4 -plots
NOTA: Questo crea un file chiamato 4D mcf_concat_highresPD.nii.gz. - Infine, creare il 3D significa immagine utilizzando fslmaths. In un tipo di finestra di terminale:
fslmaths mcf_concat_highresPD.nii.gz -Tmean mean_highresPD.nii.gz
NOTA: Questo crea un file 3D chiamato mean_highresPD.nii.gz che è di alta qualità - Visualizzare il risultato finale immagine 3D ad alta risoluzione con il comando fslview. Nella directory di cui l'immagine è, digitare quanto segue in una finestra terminale:
fslview mean_highresPD.nii.gz ". - Ispezionare profili di intensità di ROI in questione. Creare un ROI utilizzando fslview (questo può essere una linea verticale tracciata attraverso una regione del LGN per esempio). In fslview caricare l'immagine PD ad alta risoluzione. Fare clic sulla scheda Strumenti,quindi fare clic sulla scheda singola immagine per ingrandire l'immagine per disegnare ROI. Quindi, fare clic sulla scheda File seguita dalla scheda Crea maschera. Tracciare una linea nella ROI di interesse. Salvare il ROI facendo clic su File, quindi Salva con nome. Ripetere le maschere di linea per più aree all'interno del ROI per i confronti di intensità e di altri ROI in questione.
- Utilizzare il comando di 3dmaskdump AFNI analizzare l'intensità dell'immagine risultante. Nella directory di dove le immagini sono, usa il seguente comando in una finestra di terminale per estrarre le intensità di immagine e la posizione (dato come result_mask.txt) della maschera ROI:
3dmaskdump -o result_mask.txt -noijk -xyz -mask ROI_linemask.nii.gz PDaverage_image.nii.gz
Risultati
Una volta che il subcortex è prescritto all'interno del talamo, PD immagini pesate sono raccolte all'interno del riquadro di selezione della fetta (Figura 4). Il SNR migliorata aumentando il numero delle medie sia postmortem e scansioni in vivo. Per determinare la qualità dell'immagine, il SNR da diverse medie scansione è stato confrontato dividendo il segnale della regione del cervello medio per la deviazione standard in qualche zona fuori del cervello. Il SNR è stato calcolato...
Discussione
Questo studio descrive un protocollo ottimizzato in acquisizione e tecnica di analisi per ottenere alta risoluzione PD pesate di regioni sottocorticali. Un certo numero di parametri di scansione sono stati testati e modificato con i più significativi relativi alla dimensione della matrice, dimensioni voxel, e larghezza di banda per aumentare il SNR e diminuire il numero di acquisizioni, un passaggio fondamentale poter determinare strutture sottocorticali ad alta risoluzione. In concomitanza con l'individuazione dei...
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
The authors acknowledge the following funding sources, the Natural Sciences and Engineering Research Council of Canada (NSERC), the Dorothy Pitts Research Fund (NG), and the Nicky and Thor Eaton Research Fund. The authors acknowledge Kevin DeSimone, and Aman Goyal and for their knowledge in MRI acquisition and analysis expertise.
Materiali
| Name | Company | Catalog Number | Comments |
| Magnetom Trio 3T MRI | Siemens (Erlangen, Germany). | ||
| Vacuum cushion hand | Siemens | Mat No: 4765454 | Manufactured by: Johannes-Stark-Stk. 8 D-92224 Amberg |
Riferimenti
- Devlin, J. T., et al. Reliable identification of the auditory thalamus using multi-modal structural analyses. NeuroImage. 30 (4), 1112-1120 (2006).
- Fellner, F., et al. True proton density and T2-weighted turbo spin-echo sequences for routine MRI of the brain. Neuroradiology. 36 (8), 591-597 (1994).
- Schumann, C. M., Buonocore, M. H., Amaral, D. G. Magnetic resonance imaging of the post-mortem autistic brain. J Autism Dev Disord. 31 (6), 561-568 (2001).
- Tovi, M., Ericsson, A. Measurements of T1 and T2 over time in formalin-fixed human whole-brain specimens. Acta Radiol. 33 (5), 400-404 (1992).
- Fujita, N., et al. Lateral geniculate nucleus: anatomic and functional identification by use of MR imaging. Am J Neuroradiol. 22 (9), 1719-1726 (2001).
- Bridge, H., Thomas, O., Jbabdi, S., Cowey, A. Changes in connectivity after visual cortical brain damage underlie altered visual function. Brain. 131 (6), 1433-1444 (2008).
- Gupta, N., et al. Atrophy of the lateral geniculate nucleus in human glaucoma detected by magnetic resonance imaging. Br J Opthalmol. 93 (1), 56-60 (2009).
- Dai, H., et al. Assessment of lateral geniculate nucleus atrophy with 3T MR imaging and correlation with clinical stage of glaucoma. Am J Neuroradiol. 32 (7), 1347-1353 (2011).
- McKetton, L., Kelly, K. R., Schneider, K. A. Abnormal lateral geniculate nucleus and optic chiasm in human albinism. J Comp Neurol. 522 (11), 2680-2687 (2014).
- Jenkinson, M., Bannister, P., Brady, M., Smith, S. Improved optimization for the robust and accurate linear registration and motion correction of brain images. NeuroImage. 17 (2), 825-841 (2002).
- Dietrich, O., Raya, J. G., Reeder, S. B., Reiser, M. F., Schoenberg, S. O. Measurement of signal-to-noise ratios in MR images: influence of multichannel coils, parallel imaging, and reconstruction filters. J Magn Reson Imaging. 26 (2), 375-385 (2007).
- Andrews, T. J., Halpern, S. D., Purves, D. Correlated size variations in human visual cortex, lateral geniculate nucleus, and optic tract. J Neurosci. 17 (8), 2859-2868 (1997).
- Pfefferbaum, A., Sullivan, E. V., Adalsteinsson, E., Garrick, T., Harper, C. Postmortem MR imaging of formalin-fixed human brain. NeuroImage. 21 (4), 1585-1595 (2004).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon