Method Article
Filamentöz Ascomycete Huntiella omanensis'te CRISPR-Cas9 Aracılı Genom Düzenleme
Bu Makalede
Özet
CRISPR-Cas9 genom düzenleme sistemi, model ve model olmayan türlerde kullanılan kullanımı kolay bir genom editörüdür. Burada, model olmayan bir ipliksi askomisetit mantarının çiftleme genine erken dur kodonu tanıtmak için kullanılan bu sistemin protein bazlı bir versiyonunu sıyoruz.
Özet
CRISPR-Cas9 genom düzenleme sistemi, hem model hem de model olmayan türlerin genomlarına hassas değişiklikler getirmek için kullanılabilecek moleküler bir araçtır. Bu teknoloji, gen nakavtları ve knockins gelen hedeflenen bir yerde birkaç nükleotid giriş gibi daha spesifik değişiklikler için genom düzenleme yaklaşımları çeşitli için kullanılabilir. Genom düzenleme, genlerin kısmi fonksiyonel karakterizasyonu, transgenik organizmaların üretimi ve tanı araçlarının geliştirilmesi de dahil olmak üzere çok sayıda uygulama için kullanılabilir. Daha önce mevcut gen düzenleme stratejileri ile karşılaştırıldığında, CRISPR-Cas9 sistemi yeni türler kurmak kolay olduğu gösterilmiştir ve yüksek verimlilik ve özgüllük sahiptir. Bunun temel nedeni, düzenleme aracının genveya ilgi sırasını hedeflemek için bir RNA molekülü kullanması ve standart baz eşleştirme kurallarının kullanılabildiği göz önüne alındığında, hedef molekül tasarımını basit hale getirmesidir. Diğer genom düzenleme sistemlerine benzer şekilde CRISPR-Cas9 tabanlı yöntemler de etkin ve etkili dönüşüm protokollerinin yanı sıra hedefleyici RNA ve DNA moleküllerinin tasarımı için kaliteli dizi verilerine erişim gerektirir. Bu sistemin 2013 yılında piyasaya sürülmesinden bu yana, saccharomyces cerevisiae, Arabidopsis thaliana, Drosophila melanogaster ve Mus musculusgibi çeşitli model türlerin genetik olarak mühendislik için kullanılmıştır. Daha sonra, model olmayan türler üzerinde çalışan araştırmacılar sistemden yararlanarak mantarlarda ikincil metabolizma, bitkilerde nematod büyümesi ve hastalık direnci gibi çeşitli süreçlerde yer alan genlerin incelenmesiiçin kullandılar. Bu protokol aşağıda, Ceratocystidaceae ailesine ait filamentöz askolikente mantarı Huntiella ummanensisinincinsel döngüsünde yer alan bir genin kesilmesi için CRISPR-Cas9 genom düzenleme protokolünün kullanımını açıklamaktadır.
Giriş
Yüksek kaliteli artan kullanılabilirliği, tam olarak birleştirilmiş genomlar ve transkripsiyonlar büyük ölçüde organizmaların bir dizi biyolojik süreçlerin geniş bir yelpazede çalışma yeteneğini geliştirmiştir1. Bu hem model türler hem de model olmayan türler için de geçerlidir, bunların birçoğu biyolojik süreçlerin daha farklı bir anlayış sunabilir. Bu tür veriler gen keşfi, transkripsiyon ağlarının tanımlanması ve her biri kendi uygulamalarıyla birlikte gelen tüm genom ve transkripsiyon karşılaştırmaları için kullanılabilir. Ancak, genler önceden tahmin edilirken, açıklamalı ve putatif olarak daha önce hiç görülmemiş bir oranda farklı fonksiyonel yollara bağlanırken, bu genlerin fonksiyonel karakterizasyonu birçok tür için mevcut moleküler araç kitleri ile sınırlı olarak geride kalmıştır. Bu özellikle genomik verilerin üretilmesi nispeten kolay olduğu ancak daha fazla moleküler karakterizasyonunimkansızayakın olduğu 1,2model olmayan türler için de geçerlidir.
Mantar türlerinin biyolojisi için önemli olan spesifik genlerin fonksiyonlarının kısmi karakterizasyonu, mutant suşlarının fenotipik analizini takip eden nakavt veya nakavt deneylerinden elde edilebilir3. Bu iki sistem tamamen genetik mühendislik protokollerinin kullanılabilirliğine dayanır, en azından bir dönüşüm sistemi ve genetik düzenleme sistemi de dahil olmak üzere. Filamentöz mantarlar 4 çeşitli geliştirilmiştir farklı dönüşüm sistemleri bir dizivardır. Trichoderma harzianum5 ve Aspergillus niger6'dasırasıyla biyolistik ve elektroporasyona dayanan fiziksel sistemler geliştirilmiştir. Nörospora crassa7'dekalsiyum klorür veya lityum asetat gibi kimyasalları kullanan sistemler geliştirilmiştir. Son olarak, dönüşüm için Agrobacterium tumefaciens kullanımına dayanan biyolojik sistemler Ceratocystis albifundus8'debaşarıyla kullanılmıştır.
Farklı dönüşüm protokollerinin kullanılabilirliğinin aksine, genom düzenleme sistemleri daha az bulunur. Filamentöz mantarlarda yapılan geleneksel fonksiyonel karakterizasyon deneylerinin birçoğu, homoloji bölgeleri tarafından homoloji bölgeleri tarafından çevrili seçilebilir bir marker şeklinde bölünmüş marker nakavt yapısı kullanılmıştır3. Yöntem homoloji yönelimli dayanır (HR) DNA onarımı, hangi tesisleri nakavt yapı ve ilgi bölgesi arasında homolog rekombinasyon. Bu rekombinasyon olayı, ilgi geninin seçilebilir işaretleyici dizisiyle değiştirilmesiyle sonuçlanır. Ne yazık ki, bu Cercospora nicotianae10, Aspergillus fumigatus11 ve Grosmannia clavigera12dahil olmak üzere birçok türde başarılı iken , homolog rekombinasyon oranları farklı mantar türleri arasında son derece değişkendir, bu bazı türlerde verimsiz ve bazen kullanılamaz protokol yapma3.
Çinko-parmak nükleazları (ZFNs) ve transkripsiyon aktivatör benzeri efektör nükleazlar (TALENs) kullananlar da dahil olmak üzere diğer genom düzenleme sistemleri, özellikle özel ve hedefe yönelik değişiklikler yapma yetenekleri göz önüne alındığında, eski sistemlerde büyük bir gelişmeyi temsil eder13. Hem ZFN'ler hem de TALEN'ler bir nükleaz proteini ve belirli nükleotit dizilerini tanıyabilen bir proteinden oluşur13. Tanıma üzerine, nükleaz belirli mutasyonların girişini kolaylaştırabilir bir çift iplikçikli DNA kırılması neden olur. Genom değişikliklerini gerçekleştirmek için nükleotit dizisini tanıyan protein bölgesinin her deney için özel olarak tasarlanması gerekir. Çünkü protein-nükleik asit etkileşimleri düzenleme, tasarım ve her nakavt veya knockin deney için hedefleme molekülleri üreten rehberlik bu güven zor ve emek yoğun14,15. Bu zorlukların açıklayıcı, çok az ipliksi mantarlar bu sistemleri kullanarak genom düzenleme tabi tutulmuştür. Bir örnek pirinç patlama mantar, Magnaporthe oryzae16geliştirilen TALENs tabanlı sistemdir.
Genom düzenleme alanında belki de en büyük devrim CRISPR-Cas9 sisteminin keşfi ve sonraki gelişimioldu- bir RNA molekülü tarafından yönlendirilen bir ensonükleaz tarafından ilgi dizisinin hedeflenen bölünmesine olanak sağlayan bir genom editörü. Crispr-Cas9 sisteminin en büyük avantajı ilgi bölgesini hedeflemek için bir RNA molekülüne dayanması olduğu için protein-nükleik asit etkileşimlerine dayanan daha önce geliştirilmiş genom editörleri üzerinde bu gelişme büyük bir gelişmeydi. Bu, sistemin bir RNA-DNA etkileşimine dayandığı ve böylece standart baz eşleştirme kurallarından her deney15tasarlanırken yararlanılabildiği anlamına gelir.
Burada ayrıntılı olarak CRISPR-Cas9 sistemi üç ana bileşenden oluşur: tek bir kılavuz RNA (sgRNA), Cas9 enzimi ve donör DNA (dDNA)17. SgRNA protospacer denilen 20 nükleotit bölge yanı sıra iskele18denilen daha uzun bir bölgeden oluşur. Protospacer bölgesi, düzenleme sistemini hedef bölgeye yönlendirmek için kullanılır ve böylece her deneme için yeniden tasarlanır. İskele, ribonükleoproteini (RNP) oluşturmak için Cas9 enzimine fiziksel olarak bağlanan RNA bölgesidir ve böylece hedef alınacak bölge ne olursa olsun aynıdır. Cas9 enzimi fiziksel olarak hedef DNA'nın bölünmesini kolaylaştırır, bu bölgeyi tanımlamak için bir rehber olarak protospacer kullanarak19. Son bileşen olan dDNA isteğe bağlıdır ve kullanımı belirli deney20'yebağlıdır. DDNA, Cas9 enzimi tarafından kesilen bölgeye özel olarak yerleştirilmesi gereken diziyi barındırMaktadır ve bu nedenle genin genoma sokulduğu gen knockin deneyleri veya ilgi geninin yerine antibiyotik direnç geni veya diğer seçilebilir belirteçlerin sunulduğu gen nakavt deneyleri için idealdir. DDNA aynı zamanda genoma yeni diziler kgetirecek şekilde de tasarlanabilir. Örneğin, aşağıda ayrıntılı olarak, bir gen kesilme 21 gerektiğinde ilgi gen belirli bir bölgeye bir çerçeve durdurma kodonu tanıtmak mümkündür21. Diğer uygulamalar arasında genin belirli bölgelerinin mutasyonu, örneğin fonksiyonel bir etki alanı22veya etiketleme dizisi23'üngetirilmesi yer almaktadır.
CRISPR-Cas9 sistemini kullanmanın en önemli avantajı çok yönlülüğü24'tür. Bu adaptasyona bir örnek, Cas9 enziminin kullanılan dönüşüm sistemine bağlı olarak, üç formdan biri olan DNA, RNA veya proteinden birinde konak hücreye girebiliyor olmasıdır. DNA formunda tanıtıldığınızda, cas9 geni genellikle seçilebilir bir belirteç, sgRNA ifade etmek için bir kaset ve gerekirse, dDNA dizisi25kodlama bir kaset ile birlikte bir plazmid üzerinde yer almaktadır. Bu sistemin en önemli avantajı, sadece tek bir yapının hücreye dönüştürülmesi ve başarılı dönüşümün CRISRP-Cas9 aracılı genom düzenlemesi için gerekli tüm bileşenlerin bulunmasını sağlamasıdır. Ancak, bu yöntem ev sahibi türler için bir ifade sisteminin kullanılabilirliğine dayanır. Cas9'un DNA hasarına başarılı bir şekilde yol alabilmek için yüksek seviyelerde ifade edilmesi gerekir ve bu nedenle uygun ve potansiyel olarak özel bir organizatör gereklidir. Bu tür organizatörlerhenüz geliştirilmemiş olmayan model olmayan türler için, bu bir aşağılayıcı faktör olabilir ve böylece RNA veya protein formunda Cas9 tanıtmak için yeteneği daha cazip bir seçenek olabilir. RNA'nın hücreye girmesi, özellikle RNA'nın kararsız olması ve dönüşüm sürecinden sağ çıkamayabilmesi gibi kendi zorluklarını da beraberinde getirmektedir. Ayrıca, DNA veya RNA formunda tanıtıldıklarında, Cas9 gen dizisinin belirli ana bilgisayar sisteminde kullanılmak üzere kodon için optimize edilmesi gerekebilir17. Örneğin, Streptococcus pyogenes'in cas9 geni bir memeli konak hücresinde çalışmayabilir ve bir memeli hücresinde kullanılmak üzere kodon optimize edilmiş bir cas9 geni bir bitki hücresinde çalışmayabilir. Tüm bu zorluklar Cas9 protein formu kullanılarak aşılabilir, hangi, sgRNA ile birlikte, bir RNP içine monte edilebilir ve konak hücre 26 dönüştürülebilir26,27. Bu sistem herhangi bir endojen ifade sistemi veya kodon optimizasyonu güvenmez ve böylece model olmayan türlerin çoğunda çalışması gerekir. Protein bazlı sistemin dezavantajı, Agrobacteriumaracılı transfer gibi DNA tabanlı dönüşüm sistemleri ile uyumlu olmamasıdır. Bu nedenle, protein bazlı yöntemin işe yaraması için protoplastlara veya biyolistete dayananbir dönüşüm protokolünün mevcut olması gerekir. Bu RNP tabanlı sistem başarıyla filamentöz mantarlar, Fusarium oxysporum26 ve Mucor circinelloides27kullanılmıştır.
Huntiella umansis, Ceratocystidaceae ailesinin bir üyesi, kozmopolit bir mantar genellikle taze yaralı odunsu bitkiler28bulunur. Bu tür için yüksek kaliteli genom ve transkripsiyon verileri mevcut olmakla birlikte28,,29,30, hiçbir dönüşüm veya genom düzenleme protokolleri geliştirilmiştir. Bugüne kadar, H. ummanensis araştırma cinsel döngüsünün altında yatan genetik bileşenleri üzerinde duruldu29,31. Bu mantar tipik bir heterototik cinsel döngü sergiler, eşeyli üreme sadece MAT1-1 ve MAT1-2 ilişki tipleri31izole arasında meydana gelen . Buna karşılık, Yakından ilgili Huntiella moniliformis MAT1-2 izole bağımsız cinsel üreme yeteneğine sahiptir ve bir MAT1-1 ortağı yokluğunda bir cinsel döngü tamamlamak31. Cinsel yetenekleri bu fark, en azından kısmen, miyongen büyük bir fark nedeniyle, MAT1-2-7, H. ummanensis tam uzunlukta ve bozulmamış kopya barındırır nerede, gen ciddi H. moniliformis29,,31kesilir iken düşünülmektedir . Bu genin eşeyli üremedeki rolünü daha fazla karakterize etmek için H. ummanensisin MAT1-2-7 geni H. moniliformis21'degörülen kesilmeyi taklit etmek için kesildi.
Aşağıdaki protokolde H. ummanensisin dönüşümü ve CRISPR-Cas9 genom düzenleme sisteminin protein bazlı bir versiyonunu kullanarak MAT1-2-7 geninin kesilmesi ayrıntılı olarak açıklanmıştır. Bu protokol homolog rekombinasyon tabanlı gen replasmanı ve plazmid bazlı CRISPR-Cas9 genom düzenlemesi nin yaklaşımlarının başarısız lığı ile geliştirilmiştir.
Protokol
1. SGRNA'nın tasarımı ve sentezi
- SgRNA'nın bir parçasını oluşturacak potansiyel protospacer bölgelerini belirlemek için, hangi program kullanılıyorsa, arama işlevini kullanarak 5' NGG 3' üçüzleri için ilgi genini el ile arayın. Bu üçüzleri PAM dizileri olarak anons edin.
NOT: Potansiyel PAM ve protospacer dizilerini arayan ve açıklama ekleyen çeşitli yazılım programları mevcuttur.- Tanımlanan PAM dizilerinin her birinin 20 bp yukarı akışını seçin ve bu dizileri potansiyel protospacers olarak ek not ala.
- Tek bir protospacer'a karar vermek için, düşük kaliteli dizileri atmak için aşağıdaki filtreleme adımlarını gerçekleştirin.
- Potansiyel protospacer'lerin özgüllüğünü test etmek için PAM dizisini ve protospacer'ı tek bir dizide birleştirin ve tüm genoma karşı blastn sorgusu olarak kullanın. Genomdaki herhangi bir bölgeye benzerlik gösteren protouzaycıları hedef bölge dışında atın(Şekil 1A).
- RNA molekülünün Cas9 enzimine bağlanmak için doğru 3B yapıya katlanıp, cas9 enzimine özgü protospacer ve iskele dizisini içeren birleşik bir dizi oluşturun.
- Birleştirilmiş protospacer-iskele dizilerinin her birini bir RNA ikincil yapı tahmin aracına(Tablo Of Materials) yükleyin.
- En az serbest enerji ve merkezoit ikincil yapıları karşılaştırarak sonuçları analiz edin.
NOT: İdeal protospacer adayları aynı minimal serbest enerji ve merkezotoisek sekonder yapılara sahip olacaktır. Her iki ikincil yapı da beş halka yapısı tarafından kesilen üç kök halkadan oluşmalıdır. Yapılar ayrıca protospacer 'i temsil eden bölge hariç tüm yapı boyunca yüksek bağlama olasılıkları (kırmızı ile gösterilir) göstermelidir(Şekil 1B). Gerçek enerji değerleri ilgili değildir.
- Hedeflenen belirli bölgeye en yakın adayı seçerek yukarıdaki filtreleme adımlarını geçenadaylar arasından son bir aday seçin.
- SgRNA'yı tek bir RNA molekülü olarak sentezlemek için kullanılacak özel Cas9 enzimi ile uyumlu bir sgRNA sentez kiti(Tablos) kullanın (örn. Streptococcus pyogenes'tenCas9).
NOT: Aşağıdaki adımlar kullanılan SGRNA sentez kitine bağlı olabilir. Farklı bir kit kullanılması durumunda, üreticinin talimatlarına uyun. Alternatif olarak, sgRNA önceden sentezlenmiş sipariş edilebilir. RNA ile çalışırken, nükleaz içermeyen reaktifler ve tek kullanımlık maddeler kullanın.- Yukarıdaki adım 1.3'te seçilen 20 bp protospacer 5' sonunda G barındırmıyorsa, bu bölgeye Bir G ekleyin.
- Hedef sıranın 5' ucuna T7 yükseltici sırasını ekleyin. Bu sekans standarttır ve 5' TTCTAATACGACTCACTATAG 3'dür.
- Hedef dizinin 3' ucuna 14 nt çakışma sırası ekleyin. Bu sekans kitözgüdür ve burada kullanılan kit için 5' GTTTTAGAGCTAGA 3'tür.
- Elde edilen parça5' TTCTAATACGACTCACTATAG(N)20 GTTTTAGAGCTAGA, (N)20 seçilen protospacer önceden sentezlenmiş temsil eden sipariş (Malzeme Tablosu).
- Üreticiprotokolüne(Malzeme Tablosu)uygun olarak, oda sıcaklığında aşağıdaki reaktifleri birleştirir: 2 μL su, 10 μL reaksiyon tamponu, 5 μL sentezlenmiş protospacer dizisi, 1 μL 0,1 M DTT ve 2 μL transkripsiyon enzimi.
- Bu çözeltiyi 37 °C'de 30 dk kuluçkaya yatırın ve buza aktarın.
- 30 μL su ve 2 μL DNase I ekleyin, 37 °C'de 15 dakika daha karıştırın ve kuluçkaya yatırın.
- Elde edilen sgRNA'yı %2 agarose jel üzerinde görselleştirin.
2. sgRNA in vitro Dekolte Yeteneğini test
NOT: Bu adım isteğe bağlıdır, ancak önerilir.
- Seçilen sgRNA'nın hedef alacağı ve standart bir DNA polimeraz kullanarak bölgeyi güçlendireceği sitenin sırasını barındıran bir parçayı yükseltecek tasarım astarları.
NOT: Mümkünse, hedef sitede dekolte kolayca standart bir agarose jel üzerinde birbirinden ayırt edilebilir çok farklı boyutlarda iki parça üretecek şekilde astar tasarımı. - Yukarıda sentezlenen 30 nM sgRNA, 30 nM Cas9 proteini, 10x reaksiyon tamponu ve 10°L sudan oluşan bir çözeltiyi 10 dakika boyunca 25 °C'de kuluçkaya yatırarak sgRNA-Cas9 ribonükleoproteinini (RNP) birleştirin.
- Hedef bölgenin PCR ürününün RNP çözeltisine 3 nM'lik son konsantrasyona eklenmesiyle sgRNA'nın bölünme yeteneğini test edin.
- Çözeltiyi 37 °C'de 15 dk kuluçkaya yatırın.
- Dekolte reaksiyonu durdurmak için çözeltiye 3 μg proteinaz K ve 2 μg RNase ekleyin ve 10 dakika oda sıcaklığında kuluçkaya yatırın.
- Ortaya çıkan DNA parçalarını %2'lik agarose jelüzerinde görselleştirin. SgRNA, jel üzerinde beklenen boyutta iki bant gözlenirse in vivo deney için uygundur.
3. DDNA'nın tasarımı ve sentezi
- Hedeflenen genomik bölgeye tamamlayıcılık ve seçilebilir bir belirteç barındıran orta bölge olmak üzere üç bölgeden oluşan dDNA'yı tasarlayabilirsiniz(Malzeme Tablosu, Şekil 2).
NOT: Bu seçilebilir işaretçiye diğer özel diziler de eklenebilir. Bu özel deney için, seçilebilir işaretçiden hemen önce bir stop kodonu dizisi (5' TGA 3') eklenmiştir ve böylece genin içine çerçeve içi durdurma kodonu getirilebilir. DDNA önceden sentezlenebilir. Alternatif olarak, dDNA güçlendirilebilir ve adım adım, aşağıda ayrıntılı olarak PCR yaklaşımı örtüşme kullanılarak monte edilebilir.- 5' ve 3' yan bölgelerin yaklaşık 800 bp'sini yükseltmek için astartasarın. Seçilebilir işaretleyicinin sırasına tamamlayıcı olan 20 nt diziyi 5' bölgenin ters astarına ve 3' bölgenin ileri astarına ekleyin.
- Seçilebilir belirteci yükselten tasarım astarları, güçlendirilmiş ürünün direnç genini ve ilgi türlerinde çalıştığı bilinen bir organizatörü barındırmasını sağlar.
- Yüksek doğrulukta DNA polimeraz(Malzeme Tablosu)kullanarak, üç dDNA bölgesini güçlendirin. 5' ve 3' bölgeleri düzenlenen organizmanın gDNA'sından yükseltin. Seçilebilir işaretçiyi ilgili bir kaynaktan yükseltin.
- Tek bir reaksiyonda, güçlendirilmiş 5' bölgeyi seçilebilir işaretle birleştirin ve uzun menzilli, yüksek kaliteli DNA polimeraz kullanarak tüm bölgeyi güçlendirin.
- İkinci bir tek reaksiyonda, güçlendirilmiş 3' bölgeyi seçilebilir işaretle birleştirin ve uzun menzilli, yüksek kaliteli DNA polimeraz kullanarak tüm bölgeyi güçlendirin.
- Son olarak, önceki iki PCR ürününü tek bir reaksiyonda birleştirin ve tüm dDNA dizisini uzun menzilli, yüksek kaliteli DNA polimeraz ile güçlendirin.
- DNA parçasını %1 agarose jelüzerinde görselleştirin. İki veya daha fazla parçanın üretilmesi durumunda jel den doğru büyüklükteki parçayı jel arıtma kiti kullanarak arındırın.
4. Protoplastların çıkarılması
- Konidia üretmek için, 200 mL taze %2 malt ekstresi suyu (MEB) 1 cm x 1 cm misel kaplı agar bloğu ile 500 mL'lik bir şişeye aşılayın.
NOT: Tüm mantarlar konidia üretemez. Bu durumda misel de kullanılabilir. Bu genellikle protokolde daha fazla lysing enzimdaha yüksek konsantrasyonlarda gerektirecektir.- Sıvı kültürü 25 °C'de sallayarak 24 - 48 saat boyunca 120 rpm'de sallayarak inkübedin.
NOT: Bu kuluçka süresi ve sıcaklık H. ummansisiçin optimize edilmiştir. Bu diğer türler için optimize edilmesi gerekir. - Konidia hasat etmek için; sıvı kültürünü steril laboratuvar bezi (örneğin Miracloth) tabakası ile filtreleyin, conidial süspansiyonu 50 mL santrifüj tüplerine ve santrifüjü 3.220 x g'de 4 °C'de 10 dakika aktarın. Supernatant atın.
- Conidia çözeltisinin 5 mL su ve pipet 10°L'inde konidia'yı mikroskop kaydırağı üzerine yeniden askıya alın ve kapak kapağını kapatın. Sadece konidianın kurtarılmış olduğundan emin olmak için 40x büyütme altında bileşik mikroskop kullanarakgörselleştirin (Şekil 3A).
- 500 mL'lik bir şişede, 200 mL taze %1'lik MEB'i toplam resuspended konidia hacmiile aşıla.
- Sıvı kültürü 25 °C'de sallayarak 120 rpm'de 12 saate kadar sallayarak sıvı kültürü kuluçkaya yatırın.
NOT: Bu kuluçka süresi H. ummansisiçin optimize edilmiştir. Bu diğer türler için optimize edilmesi gerekir. - Mikropları hasat etmek için sıvı kültürünü 50 mL santrifüj tüpüne ve 3.220 x g'de 4 °C'de 10 dk.'ya aktarın. Supernatant atın.
- 1M sorbitol 10 mL kadar germlings resuspend.
- Pipet 10 μL mikroskop slayt üzerine germling çözeltisi ve kapak lı bir kapak. Sadece mikropların kurtarıldığından emin olmak için 40x büyütme altında bileşik bir mikroskop kullanarak görselleştirin(Şekil 3B).
NOT: Protokol burada duraklatılabilir. Mikropları -80 °C'de 1 M sorbitol'de saklayın.
- Sıvı kültürü 25 °C'de sallayarak 24 - 48 saat boyunca 120 rpm'de sallayarak inkübedin.
- Genç mikropların hücre duvarlarını lyse ve protoplasts serbest bırakmak için, steril bir 50 mL şişe çeşitli konsantrasyonlarda 9 mL lysing enzim in mikrop süspansiyon 1 mL ekleyin.
NOT: Farklı enzim konsantrasyonları ve kuluçka süreleri kullanılır ve Tablo 1'debulunabilir. Enzimler ve konsantrasyonları da mantar bağlı olarak değişebilir ve her tür için optimize edilmesi gerekir muhtemeldir.- Spor enzim çözeltisini 25 °C'de sallayarak bir kuluçka makinesinde 80 rpm'de 2-3 saat sallayarak kuluçkaya yatırın.
- Protoplast çözeltisini steril laboratuvar bezi tabakasından süzün ve 10 dakika boyunca 4 °C'de 1.810 x g'de santrifüj ile protoplastları toplayın. Supernatant atın.
NOT: Protoplastlar hücre duvarları olmayan hücrelerdir ve bu nedenle mekanik bozulmaya karşı çok hassastırlar. Özellikle pipetleme yaparken dikkatli bir şekilde hallettiğinizden emin olun. - 200 μL'lik STC tamponu(Malzeme Tablosu)protoplast peletini dikkatlice yeniden askıya alın.
- Protoplast çözeltisinin 10 μL'lik pipeti mikroskop slaytına ve kapak lı bir kapak la kaplayın. Sadece protoplastların kurtarıldığından emin olmak için 40x büyütme altında bileşik mikroskop kullanarak görselleştirin(Şekil 3C).
- Bir hemositometre kullanarak, yukarıdaki adımlarda oluşturulan protoplast sayısını sayın ve hesaplayın. Protoplast çözeltisini yaklaşık 5 x 106 protoplast içeren aliquots içine seyreltin.
NOT: Protokol burada duraklatılabilir. Protoplastları -80 °C'de STC tamponunda saklayın.
5. Protoplast ve PEG destekli dönüşüm ve transformant kurtarma
- Dönüşüme başlamak için, rnp çözeltisinin tek bir hacmi ve dDNA parçasının yaklaşık 6 μg'ı ile yaklaşık 5 x 106 protoplastları birleştirin.
NOT: Protoplastlar mekanik bozulmaya karşı çok hassastır. Özellikle pipetleme yaparken dikkatli bir şekilde hallettiğinizden emin olun.- Pipet kullanarak, protoplast çözeltisine yeni hazırlanmış %30 PTC çözeltisinin 1 mL'ini yavaşça damlatın ve çözeltiyi 20 dakika oda sıcaklığında kuluçkaya yatırın.
NOT: Bu adım hassas ve çok önemli bir adımdır. Taze hazırlanmış PTC çözeltisi kullandığınızdan emin olun ve hücre yüzeyinde hidrofobik bir tabaka oluşturarak, mümkün olduğunca yavaş ve eşit olarak hücreleri üzerinde çözüm bırakın. - Çözeltinin iyice karıştırıldığından emin olmak için protoplast çözeltisine 5 mL ozmotik kontrol ortamı (OCM) ve pipeti yavaşça ve yavaşça ekleyin.
- Protoplast çözeltisini 25 °C'de sallayarak bir inkübatörde 80 rpm'de bir gecede titretin.
- Pipet kullanarak, protoplast çözeltisine yeni hazırlanmış %30 PTC çözeltisinin 1 mL'ini yavaşça damlatın ve çözeltiyi 20 dakika oda sıcaklığında kuluçkaya yatırın.
- Dönüştürülmüş izolasyonları seçmek için, çözümü 5 boş 60 mm kültür plakasına bölün.
- Her kültür plakasına 30 μg/mL higromisin B ile takviye edilmiş 10 mL OCM agar ekleyin ve her plakayı yavaşça iyice karıştırmak için döndürün.
- 40 μg/mL higromisin B ile takviye osmotik kontrol orta agar 10 mL eklemeden önce agar ilk tabakası ayarlamak için izin verin.
- Agar'ın ikinci tabakasının kültürleri 25 °C'de ayarlamasına ve kuluçkaya yatırmasına izin verin, ta ki tek izole edilenler agarın her iki katmanından da büyüyene kadar.
- Başarılı bir şekilde dönüştürülmüş izolasyonları kurtarmak için, bireyin 40 μg/mL higromisin B ile desteklenen agar tabakası ile büyüme yeteneğine sahip izole taze malt özü agar (MEA) plakaları ile takviye 50 g/mL hygromycin B (MEA-50).
- Taze kültürleri 25 °C'de 5 gün kuluçkaya yatırın ve günlük büyüme kontrol edin. Bu medyada sürekli büyüme yeteneğine sahip kültürler başarıyla dönüştürülmüş ve daha fazla çalışma için kullanılabilir.
6. DDNA entegrasyonu ve stabilitesi onayı
- DDNA'nın hedef bölgedeki genoma entegre edildiğini doğrulamak için, öngörülen 5' ve 3' kesici kesici uç alanlarını çevreleyen astarlar tasarlar(Şekil 2).
- Bu iki astar seti ve yüksek kaliteli DNA polimeraz kullanarak iki PCR gerçekleştirin. Her iki PCR beklenen boyut ve dizi amplicons verim varsa, dDNA başarıyla hedef bölgeye entegre edilmiştir. Daha sonra, dDNA kararlı entegrasyonu için mutant leke değerlendirmek.
- DDNA'nın genoma entegre edildiğini ve vegetatif büyüme sırasında korunacağını doğrulamak için bir medya transferi testi yapın.
- Mea-50 ortamında aktif olarak büyüyen bir mutanttan misel kaplı bir agar bloğunu tamamlanmamış MEA ortamına aktarın. 25 °C'de 3 gün kuluçkaya yatırın.
- MEA-50 orta mea orta üzerinde büyüyen izole misel kaplı agar bir blok aktarın. 25 °C'de 3 gün kuluçkaya yatırın.
- Bu işlemi tekrarlayın, aktif olarak büyüyen misel transferi en az dört tur için tamamlanmamış orta için tamamlanır.
NOT: Eğer izole birçok transferden sonra MEA-50 ortamda sürekli büyüme yeteneğine sahipse, dDNA genoma entegre edilmiştir ve bitkisel büyüme ile sürdürülebilir. Mutant leke entegre dDNA sadece tek bir kopyasını varlığı için değerlendirilebilir.
- DDNA'nın genoma tek bir yerde entegre edildiğini doğrulamak için Güney leke analizi yapın.
- Üreticiprotokollerine uygun olarak HindIII ve EcoRI restri restriksiyon enzimleri kullanarak her mutant suşundan toplam 30 μg gDNA sindirin.
NOT: Restriksiyon enziminin seçimi araştırmacıya kalmış olsa da, restriksiyon enziminin tanıma bölgesinin dDNA dizisinde bulunmadığından emin olun. - Sindirilmiş gDNA'yı %0.75 agarose jel üzerinde ayırın ve STANDART prosedürleri kullanarak DNA'yı naylon membrana aktarın32.
- Membranı dDNA dizisini hedefleyen bir sonda kullanarak hibridizasyona tabi titretin.
- DDNA'nın kısa (300 bp) bölgesini yükseltmek için astarlar tasarla.
- Bu astarları kullanarak, bir PCR DIG etiketleme karışımı kullanarak prob sentez.
- Membran hibridizasyonu, tedavisi ve görselleştirme için standart prosedürleri kullanarak yeni sentezlenen prob kullanın32. Her şeritte sadece tek bir bant görülürse, dDNA genomda sadece tek bir yerde bulunur. Mutant suşu artık daha fazla fenotipik analiz ve fonksiyonel karakterizasyon deneyleri için kullanılabilir.
- Üreticiprotokollerine uygun olarak HindIII ve EcoRI restri restriksiyon enzimleri kullanarak her mutant suşundan toplam 30 μg gDNA sindirin.
7. Mutant suşlarının phenotipik analizi
- MAT geninin bozulmasının incelenmekte olan mantarın cinsel yetenekleri üzerinde bir etkisi olup olmadığını belirlemek için miyeting deneyleri gerçekleştirin.
NOT: Bu adım, incelenmekte olan belirli gen ve türlere bağlıdır. Bu durumda, hedef alan genin eşeyli üremede rol aldığı düşünülmekte dir ve böylece ilişki testleri yapılmıştır. Eğer genin, örneğin, eşeysiz üremeye dahil olduğu düşünülürse, o zaman conidial üretim gibi bir şey ölçülebilir.- Mutant suş heterotoritmik yeteneklerini test etmek için, bir mutant zorlanma yanı sıra zıt miyon türü bir suş ile birlikte taze MEA orta inoküle. H. ummansisdurumunda, plakaların kapaklarını kapalı tutun, ancak 7 gün boyunca oda sıcaklığında mühürlü ve kuluçka değil. Cinsel yapıların üretimi için görsel olarak değerlendirin.
- Mutant suşhomothallic yeteneklerini test etmek için, bir mutant zorlanma ile taze MEA orta aşılamak. H. ummansisdurumunda, plakaların kapaklarını kapalı tutun, ancak 7 gün boyunca oda sıcaklığında mühürlü ve kuluçka değil. Cinsel yapıların üretimi için görsel olarak değerlendirin.
- MAT geninin bozulmasının incelenmekte olan mantarın büyüme hızı üzerinde bir etkisi olup olmadığını belirlemek için büyüme hızı deneyleri gerçekleştirin.
- Agar içine büyük, steril pipet ucu arka tarafı takarak mutant ve wildtype suşlarının kültürlerin aktif büyüyen kenarından misel kaplı agar fişleri oluşturun.
- Bu agar fişleri ile taze MEA orta aşılamak. Her kültür türü için en az üç çoğaltma yapıldığından emin olun.
- 20 °C'de 3 günlük büyümeden sonra, büyümeyi iki dik çapta ölçün.
- Wildtype ve mutant suşları verileri karşılaştırın.
Sonuçlar
Yukarıda açıklanan protokol, model olmayan ascomycete, H. ummanensisbir çiftleme geni içine erken bir stop kodon giriş kolaylaştırdi. Bu süreç CRISPR-Cas9 genom düzenleme sisteminin bir versiyonunu kullanmaktadır ve bu protokoldeki en önemli adımlardan biri yüksek kaliteli bir sgRNA'nın tasarımı ve sentezidir. Şekil 1, bu molekülün nasıl tasarlandığını gösterir, a) özellikle ilgi genini hedefler ve genomdaki diğer bölgelere çok az benzerlik gösterir ve B) Cas9 proteini ile bağdaştırmak için doğru şekilde katlanır. SGRNA da etkili bir şekilde hedef bölge cleaving yeteneğine sahip olmalıdır. SGRNA'nın hedef lehimleme ve hedef bölgenin bölünmesine izin verme yeteneği, beklenen boyutta iki ürün elde ederek in vitroolarak gerçekleştirildi.
Başarılı bir dönüşüm gerçekleştikten sonra, dDNA'nın genoma sadece bir kez ve beklenen yerde entegre olduğundan emin olmak önemlidir. Şekil 2, doğru entegrasyon sitesi için potansiyel dönüştürücüleri taramak için kullanılabilecek ekleme sitelerini hedefleyen PCR astarlarının tasarımını göstermektedir. 5' ve 3' ekleme alanlarını çevreleyen astarlar tasarlayarak, amplifikasyon ancak dDNA'nın doğru bölgeye takılması halinde mümkündür. Şekil 4, prematüre stop kodonunun MAT1-2-7 genine doğru okuma çerçevesine girilmesini ve genin H. moniliformis'inkinebenzer şekilde kesileceğini göstermektedir. Ayrıca, Güney leke analizi dDNA yapısının genomdaki tek bir bölgeye entegre edildiğini gösterdi.
Protokolün başarısı mutant suşlarının henotibik analizi yle doğrulandı. MAT1-2-7 bozulma deneyi durumunda iki bağımsız mutant suş geliştirilmiştir. Her iki izole de, vegetatif radyal büyüme hızı önemli ölçüde azaltıldı, yeni çiftleme geninin pleiotropik etkisi düşündüren(Şekil 5). Ayrıca, mutant izole bir cinsel döngü tamamlayarak aciz, cinsel sporlar üretmek vermedi sadece olgunlaşmamış cinsel yapılar üreten(Şekil 5). Bu, kuluçkadan sonraki birkaç gün içinde tüm cinsel döngüyü tamamlayan yabani tip izolelerin aksineydi(Şekil 5).
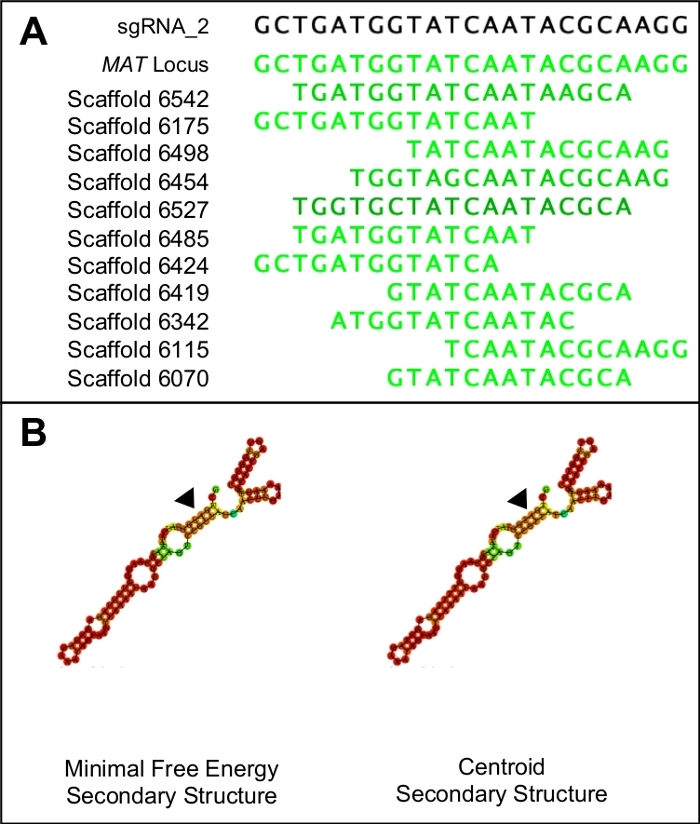
Şekil 1: Uygun bir sgRNA adayı nın seçilmesi.
(A) Uygun bir sgRNA sadece genomun hedef bölgesine benzerlik gösterecektir (bu durumda MAT lokus dizisi ile gösterilir). (B) Uygun bir sgRNA aynı minimal serbest enerji ve centroid ikincil yapılara sahip olacak, birincil adım döngüsünde üç kök döngü ve beş halka. Ayrıca, yapının büyük bir kısmı yüksek bağlama olasılıklarına sahip olacak (koyu turuncu ve kırmızı ile gösterilirken) daha düşük bağlama olasılıkları protospacer bölgesinde (siyah üçgenlerle gösterilir) görülmelidir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
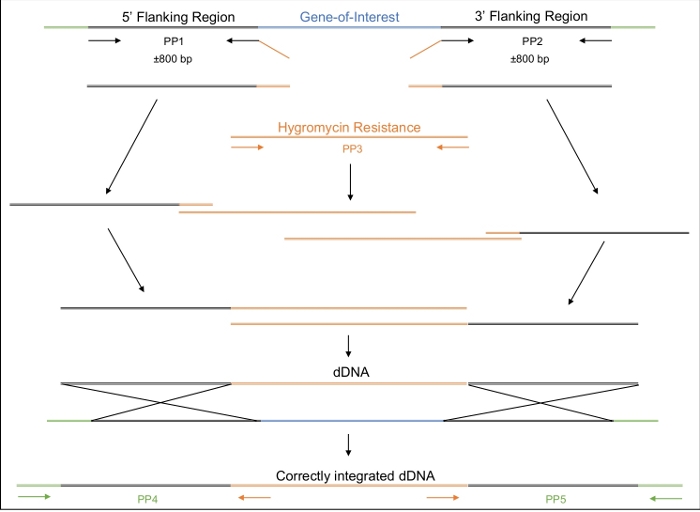
Şekil 2: DDNA'nın tasarımı, amplifikasyonu ve montajı.
Birinci ve ikinci astar çiftleri (PP1 ve PP2) ilgi geninin yaklaşık 800 bp upstream (5') ve 800 bp downstream (3') yükseltmek için kullanılır. PP1'in ters astarı ve PP2'nin ileri astarı higromisin direnç kasetine homoloji bölgeleri içerir. Üçüncü astar çifti tüm higromisin direnci kaseti güçlendirir. Adım adım olarak, çeşitli amfikonlar 5' bölgeden oluşan tüm dDNA, higromisin direnç kaseti ve 3' bölge bir araya getirilene kadar monte edilir. Hücreye dönüştürüldüğünde, dDNA Cas9 enziminin kesilmeye yönlendirildiği bölgede yeniden birleşerek ilgi genini higromisin direnci kasetiyle değiştirmeli. PP4 ve PP5, dDNA'nın uygun yerde genoma doğru şekilde sokulup yerleştirilmediğini belirlemek için kullanılabilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
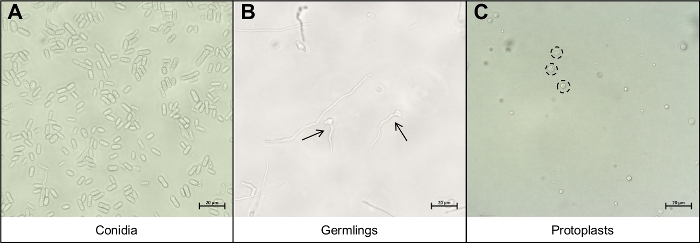
Şekil 3: Protoplast ekstraksiyon protokolü sırasında önemli olan farklı hücre tipleri.
(A) Konidia protokol için başlangıç malzemesi olarak kullanılır. Bu konidia çimlenme ve onlar kadar büyümek izin verilir (B) genç mikroplar. Genç mikropların ideal büyüme evresi iki siyah okla gösterilir. (B)üzerinde görülen diğer misel iplikçikleri bozulmaiçin çok olgundur ve kullanılmamalıdır. Protokolün son adımı siyah, noktalı daireler tarafından belirtilen (C) yuvarlak protoplasts, serbest bırakılmasıdır. Bu hücreler artık hücre duvarları var ve bu nedenle mekanik bozulmaya karşı çok duyarlıdırlar. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
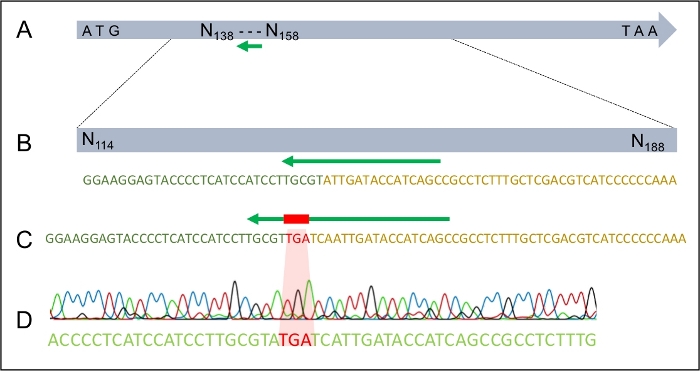
Şekil 4: TGA stop kodonunun H. ummansisin MAT1-2-7 genine başarılı bir şekilde entegrasyonu.
(A) Tam uzunlukta H. ummanensis MAT1-2-7 geni, yeşil ok ile gösterilen sgRNA hedef alanı ile. (B) H. ummanensis MAT1-2-7 geni içindeki sgRNA hedef bölgesinin büyütülmüş şeması. (C) DDNA'nın bir bölgesinin büyütülmüş şeması, h. ummanensisin MAT1-2-7 genine homolog kollarla çevrili dur kodonu gösterir. (D) Dur kodonunun MAT1-2-7 genine başarılı bir şekilde entegrasyonunu gösteren sanger dizisi kromatogramı. Wilson ve ark. 202021'dendeğiştirildi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 5: (A) yabani tipi izole ve (B) mutant izole arasındaki fenotipik farklılıklar.
Her paneldeki ilk üç resim, iki izole türünün cinsel yeteneklerindeki farklılıkları gösterir. Wildtype cinsel üreme sırasında olgun askomata formu izole ederken, askomatal boyun uçlarından sporların exudation ile komple, mutant herhangi bir cinsel spor lar üretmek olmayan sadece olgunlaşmamış cinsel yapılar oluşturur izole. Her paneldeki dördüncü görüntü, iki izole türünün büyüme hızı ve morfolojisi arasındaki farkı gösterir. Yabani tip izole çok daha hızlı ve daha fazla hava misel ile büyür iken, mutant yavaş gösterir ve agar içinde batırılır. Wilson ve ark. 202021'dendeğiştirildi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| Reaksiyon | Enzim Konsantrasyonu | Bozulma Süresi |
| A | 1.250 mg/mL | 180 dk |
| B | 1.875 mg/mL | 180 dk |
| C | 2.500 mg/mL | 150 dk |
| D | 3.750 mg/mL | 150 dk |
| E | 4.375 mg/mL | 120 dk |
| F | 5.000 mg/mL | 120 dk |
Tablo 1: Trichoderma harzianumdan lysing enzimleri ile germling/misel çözeltisinin bozulması. Farklı enzim konsantrasyonları farklı kuluçka dönemlerine karşılık gelir, daha düşük konsantrasyonlar daha uzun kuluçka gerektiren.
Tartışmalar
H. ummanensis in başarılı dönüşümü ve MAT1-2-7 geninin düzenlenmesi için protokol higromisin B21direnç için bir gen ile birlikte bir çerçeve erken stop codon tanıtılması ile gösterilmiştir . Bu, CRISPR-Cas9 genom düzenleme sisteminin protein bazlı bir versiyonu kullanılarak elde edilmiştir. Deney, sgRNA'nın in vitro transkripsiyonunu, dDNA'nın PCR tabanlı biraraya getirincesini ve bu iki nükleik asitin ticari olarak kullanılabilen Cas9 enzimi ile H. omanensisten çıkarılan protoplastlara dönüştürülmesini gerektirir.
Diğer birçok moleküler aracın kullanılabilirliğine dayanan diğer protokollerin aksine, yukarıda açıklanan protokol, moleküler alet kutusunun hala oldukça sınırlı olduğu türlerde başarıyla kullanılabilir21. Protokol sadece yerleşik bir dönüşüm sistemine ve tercihen tüm genom dizisi olan NGS verilerinin kullanılabilirliğine dayanır. Etkili bir dönüşüm sistemi, bunun mevcut olmadığı bir türde bazı optimizasyonlar gerekse de, çeşitli türler için birçok farklı protokol mevcuttur. Ayrıca, genom verileri türlerin en karanlık bile için giderek daha fazla kullanılabilir hale geliyor ve zaten mevcut değilse de novo oluşturmak daha kolay hale gelmektedir.
Protokolün uzunluğu göz önüne alındığında, değişikliklerin getirilebileceği ve sorun giderme nin gerekli olabileceği birçok adım vardır. Bu özellikle türlere özgü olarak kabul edilen adımlar için geçerlidir. Örneğin, bu protokolde deney için önemli hücre tipleri oluşturmak için belirli sıcaklıklarda ve belirli sürelerde yapılması gereken birçok kuluçka adımı vardır. Bu adımlar böylece türe özgü optimizasyon gerektirir. Mümkün olduğunda, bu protokolün farklı türlere aktarılmasına yardımcı olmak için belirli hücrelerin veya büyüme evrelerinin mikrografları sağlanmıştır (Şekil1). Protoplastları serbest bırakmak için mantar hücrelerinin hücre duvarlarını bozan enzimlerin türü ve konsantrasyonu da incelenmekte olan mantar türlerine özgü olacaktır. Bu protokolde, fusarium verticillioides33gibi türlerde protoplastların ekstraksiyonu için farklı enzim kombinasyonları kullanılırken, sadece bir lysing enzim kaynağı kullanılmaktadır. Bu adım tamamen hücre duvarının kimyasal haline bağlıdır ve bu nedenle türler bazında bir tür üzerinde optimize edilmesi gerekir.
Bu yöntem, bir ifade sistemine güven olmadığı için özellikle model olmayan türleri inceleyenler için önemlidir. CRISPR-Cas9 genom düzenleme sistemini kurmanın popüler bir yöntemi, Cas9 proteinini, sgRNA'yı ve tercih edilen hücrelere dönüşen bir veya iki plazmidden gelen dDNA'yı ifade etmektir. Bu durumda, Cas9 incelenen belirli organizmada ifade yüksek düzeyde yeteneğine sahip bir organizatör tarafından ifade edilmesi gerekir. Genel organizatörler ipliksi mantarlarda kullanılmak üzere geliştirilmiştir ve tüm türlerde uyumlu olmamakla birlikte, düşük seviyeli ifadeye izin verirler ve örneğin antibiyotik direnci genlerini ifade etmek için başarılı bir şekilde kullanılabilirler. Ancak bu organizatörler genellikle yüksek düzeyde ifadeye izin vermezler ve bu nedenle Cas9 proteinini ifade etmek için kullanılamazlar. CRISPR-Cas9 genom düzenleme sisteminin protein bazlı bir versiyonunun kullanılması bu sınırlamanın üstesinden gelir ve sgRNA ve dDNA'nın önceden üretilmiş bir Cas9 enzimi ile hücreye dönüştürülmesini sağlar.
H. ummanensis'te kullanılmak üzere bu protein bazlı sistemin geliştirilmesi, hem klasik split marker yaklaşımını hem de plazmid bazlı CRISPR-Cas9 sistemini kullanarak genom düzenlemede birçok başarısız girişimden sonra geldi. Verimlilik türlerden türlere farklılık gösterirken, bölünmüş marker yaklaşımı Alternaria alternata34,35ve C. nicotianae36gibi çeşitli türlerde %100 verimlilikle başarıyla kullanılmıştır. Buna karşılık, 80'den fazla bağımsız dönüşüm ve entegrasyon olayına rağmen, H. ummanensis'te bu sistemin etkinliği sıfırdı. Benzer şekilde, plazmid bazlı CRISPR-Cas9 sistemi, Trichoderma reesei (>93%)17 ve Penicillium chrysogenum (%100'e kadar)37'deyüksek verimlilikle başarıyla kullanılmıştır. Bu, yine, H. ummansisbu sistemin kullanışlılığı aksine. Cas9 proteininin yeterli ekspresyonu H. ummanoensis'te ulaşılamadı, ancak temizlik genlerinden tahmin edilen iki türe özgü organizatörler de dahil olmak üzere bir dizi potansiyel promotör denedi. Böylece bu sistem hiç kullanılamadı. CRISPR-Cas9 sisteminin protein bazlı versiyonunu kullanarak, ikisi entegre dDNA'yı doğru konumda barındıran birçok bağımsız transformant ortaya çıktı. Ayrıca, bu deney sadece bir kez denendi ve başarılı oldu- daha da bu sistemin kullanılabilir hangi kolaylığı gösteren.
Bu protokolün gelecekteki uygulamaları ceratocystidaceae diğer türlerde optimizasyon ve kullanımı içerir. Zaten bu türler için ngs veri zenginliği vardır30,38,39 ve ev sahibi özgüllük ile ilgili çalışmalar40, büyüme oranı ve virülans41 yapılmıştır. Bu çalışmalar, bu süreçlerde yer aldığı düşünülen genlerin fonksiyonel karakterizasyonu, dönüşüm ve genom düzenleme protokolünün kullanılabilirliği nedeniyle artık mümkün olacak araştırmalarla güçlendirilebilir.
Sonuç olarak, model olmayan türlerde önemli biyolojik süreçlerin altında yatan genlerin derinlemesi, kapsamlı biyolojik kaynakların ve moleküler araç kitlerinin varlığına dayanmayan kullanımı kolay genom düzenleme protokollerinin bulunması sayesinde daha erişilebilir hale gelmektedir. Model olmayan türlerin incelenmesi giderek kolaylaşıyor ve model türlerinde açıklığa kavuşturulacak standart biyolojik süreçlerden yeni yolların ve ilginç sapmaların keşfine olanak sağlayacaktır.
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Bu proje Pretoria Üniversitesi, Bilim ve Teknoloji Bölümü (DST)/Ulusal Araştırma Vakfı (NRF) Ağaç Sağlığı Biyoteknolojisi Mükemmeliyet Merkezi (CTHB) tarafından desteklenmiştir. Proje ayrıca Prof BD Wingfield'ın Fungal Genomic(Grant number: 98353) başkanı ve Dr. Wilson'ın NRF Doktora bursu (108548) tarafından desteklenmiştir. Hibe sahipleri, bu çalışmada ifade edilen görüş, bulgu ve sonuçların veya önerilerin araştırmacıların görüşlerini kabul ettiğini ve finansman kuruluşlarının bu konuda hiçbir sorumluluk kabul etmediğini kabul eder.
Malzemeler
| Name | Company | Catalog Number | Comments |
| EcoRI-HF | New England Biolabs, Ipswich, USA | R3101S | |
| EnGen Spy Cas9 NLS protein | New England Biolabs, Ipswich, USA | M0646T | Used to assemble the RNP |
| Eppendorf 5810 R centrifuge | Eppendorf, Hamberg, Germany | ||
| FastStart Taq DNA Polymerase | Sigma, St Louis, USA | 12032902001 | Standard DNA polyermase |
| GeneJET Gel Extraction Kit | ThermoFisher Scientific, Waltham, USA | K0691 | |
| HindIII-HF | New England Biolabs, Ipswich, USA | R3104S | |
| HiScribeTM T7 Quick High Yield RNA synthesis kit | New England Biolabs, Ipswich, USA | E2050S | |
| Hygromycin B from Streptomyces hygroscopicus | Sigma, St Louis, USA | 10843555001 | |
| Infors HT Ecotron Shaking Incubator | Infors AG, Bottmingen, Switzerland | ||
| LongAmp Taq DNA Polymerase | New England Biolabs, Ipswich, USA | M0323S | Long-range, high-fidelity DNA polymerase |
| Malt extract agar, 2% (MEA) | 20 g ME and 20 g agar in 1 l ddH20 | ||
| Malt extract | Sigma, St Louis, USA | 70167-500G | |
| Agar | Sigma, St Louis, USA | A5306 | |
| Malt Extract broth, 1% (MEB) | Sigma, St Louis, USA | 70167-500G | 2 g ME in 200 ml ddH20 |
| Malt Extract broth, 2% (MEB) | Sigma, St Louis, USA | 70167-500G | 4 g ME in 200 ml ddH20 |
| Miracloth | Merck Millipore, New Jersey, USA | 475855 | |
| Nylon membrane (positively charged) | Sigma, St Louis, USA | 11209299001 | |
| Osmotic control medium (OCM) | 0.3% yeast extract, 20% sucrose, 0.3% casein hydrolysate | ||
| Casein Hydrolysate | Sigma, St Louis, USA | 22090 | |
| Sucrose | Sigma, St Louis, USA | 84097 | |
| Yeast extract | Sigma, St Louis, USA | Y1625 | |
| Osmotic control medium (OCM) agar | Osmotic control medium (OCM) + 1% agar | ||
| Agar | Sigma, St Louis, USA | A5306 | |
| PCR DIG Labeling Mix | Sigma, St Louis, USA | 11585550910 | |
| Phusion High-Fidelity DNA Polymerase | ThermoFisher Scientific, Waltham, USA | F-530XL | High fidelity DNA polymerase |
| Plasmid pcb1004 | N/A | N/A | From: Carroll et al., 1994 |
| Presynthesized sgRNA | Inqaba Biotec, Pretoria, South Africa | Ordered as an synthesized dsDNA with specified sequence | |
| Proteinase K | Sigma, St Louis, USA | P2308 | |
| PTC Solution | 30% polyethylene glycol 8000 in STC buffer from above | ||
| Polyethylene glycol 8000 | Sigma, St Louis, USA | 1546605 | |
| RNase A | ThermoFisher Scientific, Waltham, USA | 12091021 | |
| RNAfold Webserver | Institute for Theoretical Chemistry, University of Vienna | N/A | http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi |
| RNAstructure | Mathews Lab | N/A | https://rna.urmc.rochester.edu/RNAstructureWeb/Servers/Predict1/Predict1.html |
| Sorbitol, 1 M | Sigma, St Louis, USA | 1617000 | 182.17g sorbitol in 1 l ddH20 |
| STC Buffer | 20% sucrose, 50 mM Tris-HCl pH 8.00 and 50 mM CaCl2 | ||
| Calcium chloride | Sigma, St Louis, USA | 429759 | |
| Tris-HCl pH 8.00 | Sigma, St Louis, USA | 10812846001 | |
| Sucrose | Sigma, St Louis, USA | 84097 | |
| Trichoderma harzianum lysing enzymes | Sigma, St Louis, USA | L1412 | |
| Zeiss Axioskop 2 Plus Ergonomic Trinocular Microscope | Zeiss, Oberkochen, Germany |
Referanslar
- Ekblom, R., Galindo, J. Applications of next generation sequencing in molecular ecology of non-model organisms. Heredity. 107, 1-15 (2011).
- Russell, J. J., et al. Non-model model organisms. BMC Biology. 15 (55), 1-31 (2017).
- Kück, U., Hoff, B. New tools for the genetic manipulation of filamentous fungi. Applied Microbiology and Biotechnology. 86, 51-62 (2010).
- Li, D., Tang, Y., Lin, J., Cai, W. Methods for genetic transformation of filamentous fungi. Microbial Cell Factories. 16 (168), 1-13 (2017).
- Lorito, M., Hayes, C. K., Di Pietro, A., Harman, G. E. Biolistic transformation of Trichoderma harzianum and Gliocladium virens using plasmid and genomic DNA. Current Biotechnology. 24, 349-356 (1993).
- Taylor, P., et al. Transformation of intact Aspergillus niger by electroporation. Bioscience, Biotechnology, and Biochemistry. 58 (12), 2224-2227 (2014).
- Dhawale, S. S., Paietta, J. V., Marzluf, G. A. A new, rapid and efficient transformation procedure for Neurospora. Current Genetics. 8, 77-79 (1984).
- Sayari, M., Van Der Nest, M. A., Steenkamp, E. T., Adegeye, O. O., Marincowitz, S. Agrobacterium-mediated transformation of Ceratocystis albifundus. Microbiological Research. 226, 55-64 (2019).
- Meyer, V. Genetic engineering of filamentous fungi- Progress, obstacles and future trends. Biotechnology Advances. 26, 177-185 (2008).
- You, B. J., Lee, M. H., Chung, K. R. Gene-specific disruption in the filamentous fungus Cercospora nicotianae using a split-marker approach. Archives of Microbiology. 191, 615-622 (2009).
- Gravelat, F. N., Askew, D. S., Sheppard, D. C. Targeted gene deletion in Aspergillus fumigatus using the hygromycin-resistance split-marker approach. Host-Fungus Interactions. 845, 119-130 (2012).
- Wang, Y., Diguistini, S., Bohlmann, J., Breuil, C. Agrobacterium-meditated gene disruption using split-marker in Grosmannia clavigera, a mountain pine beetle associated pathogen. Current Genetics. 56, 297-307 (2010).
- Wood, A. J., et al. Targeted genome editing across species using ZFNs and TALENs. Science. 333, 307 (2011).
- Mahfouz, M. M., Piatek, A., Neal, C. Genome engineering via TALENs and CRISPR/Cas9 systems: Challenges and perspectives. Plant Biotechnology. 12, 1006-1014 (2014).
- Wang, H., La Russa, M., Qi, L. S. CRISPR/Cas9 in genome editing and beyond. Annual Review of Biochemistry. 85 (1), 227-264 (2016).
- Arazoe, T., et al. Tailor-made TALEN system for highly efficient targeted gene replacement in the rice blast fungus. Biotechnology and Bioengineering. 112 (7), 1335-1342 (2015).
- Liu, R., Chen, L., Jiang, Y., Zhou, Z., Zou, G. Efficient genome editing in filamentous fungus Trichoderma reesei using the CRISPR/Cas9 system. Cell Discovery. 1, 1-11 (2015).
- Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J. A., Charpentier, E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337, 816-822 (2012).
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proceedings of the National Academy of Sciences. 109 (39), 2579-2586 (2012).
- Ran, F. A., Hsu, P. D., Wright, J., Agarwala, V., Scott, D. A., Zhang, F. Genome engineering using the CRISPR-Cas9 system. Cell. 8 (11), 2281-2308 (2013).
- Wilson, A. M., Wilken, P. M., Van Der Nest, M. A., Wing, M. J., Wing, B. D. The novel Huntiella omanensis mating gene, MAT1-2-7, is essential for ascomatal maturation. Fungal Genetics and Biology. 137, 103335 (2020).
- Miao, J., et al. Characterization of an N-terminal non-core domain of RAG1 gene disrupted Syrian Hamster model generated by CRISPR Cas9. Viruses. 10 (243), 10050243 (2018).
- Roberts, B., et al. Systematic gene tagging using CRISPR/Cas9 in human stem cells to illuminate cell organization. Molecular Biology of the Cell. 28 (21), 2854-2874 (2016).
- Schneider, S., Kirchner, M., Kirchner, M., Schneider, S. CRISPR-Cas: From the bacterial adaptive immune system to a versatile tool for genome engineering. Angewandte Chemie International Edition. 54 (46), 13508-13514 (2015).
- Nødvig, C. S., Nielsen, J. B., Kogle, M. E., Mortensen, U. H. A CRISPR-Cas9 system for genetic engineering of filamentous fungi. PLoS ONE. 10 (7), 1-18 (2015).
- Wang, Q., Cobine, P. A., Coleman, J. J. Efficient genome editing in Fusarium oxysporum based on CRISPR/Cas9 ribonucleoprotein complexes. Fungal Genetics and Biology. 117, 21-29 (2018).
- Nagy, G., et al. Development of a plasmid free CRISPR-Cas9 system for the genetic modification of Mucor circinelloides. Scientific Reports. 7 (16800), 1-10 (2017).
- Al-Subhi, A. M., Al-Adawi, A. O., Van Wyk, M., Deadman, M. L., Wingfield, M. J. Ceratocystis omanensis, a new species from diseased mango trees in Oman. Mycological Research. 110 (2), 237-245 (2006).
- Wilson, A. M., van der Nest, M. A., Wilken, P. M., Wingfield, M. J., Wingfield, B. D. Pheromone expression reveals putative mechanism of unisexuality in a saprobic ascomycete fungus. PLoS ONE. 13 (3), 0192517 (2018).
- van der Nest, M. A., et al. Draft genomes of Amanita jacksonii, Ceratocystis albifundus, Fusarium circinatum, Huntiella omanensis, Leptographium procerum, Rutstroemia sydowiana, and Sclerotinia echinophila. IMA Fungus. 5 (2), 472-485 (2014).
- Wilson, A. M., Godlonton, T., van der Nest, M. A., Wilken, P. M., Wingfield, M. J., Wingfield, B. D. Unisexual reproduction in Huntiella moniliformis. Fungal Genetics and Biology. 80, 1-9 (2015).
- Sambrook, J., Green, M. . Molecular cloning: A laboratory manual. , (2012).
- Ramamoorthy, V., Govindaraj, L., Dhanasekaran, M., Vetrivel, S., Kumar, K. K., Ebenezar, E. Combination of driselase and lysing enzyme in one molar potassium chloride is effective for the production of protoplasts from germinated conidia of Fusarium verticillioides. Journal of Microbiological Methods. , (2015).
- Lin, C., Yang, S. L., Wang, N., Chung, K. The FUS3 MAPK signaling pathway of the citrus pathogen Alternaria alternata functions independently or cooperatively with the fungal redox-responsive AP1 regulator for diverse developmental, physiological and pathogenic processes. Fungal Genetics and Biology. 47 (4), 381-391 (2010).
- Lin, C., Chung, K. Specialized and shared functions of the histidine kinase- and HOG1 MAP kinase-mediated signaling pathways in Alternaria alternata, a filamentous fungal pathogen of citrus. Fungal Genetics and Biology. 47 (10), 818-827 (2010).
- Choquer, M., et al. The CTB1 gene encoding a fungal polyketide synthase is required for cercosporin biosynthesis and fungal virulence of Cercospora nicotianae. Molecular Plant-Microbe Interactions. 18 (5), 468-476 (2005).
- Pohl, C., Kiel, J. A. K. W., Driessen, A. J. M., Bovenberg, R. A. L., Nygård, Y. CRISPR/Cas9 based genome editing of Penicillium chrysogenum. ACS Synthetic Biology. 5 (7), 754-764 (2016).
- van der Nest, M. A. M. A., et al. Draft genome sequences of Diplodia sapinea, Ceratocystis manginecans and Ceratocystis moniliformis. IMA Fungus. 5 (1), 135-140 (2014).
- Wingfield, B. D., et al. Draft genome sequences for Ceratocystis fagacearum, C. harringtonii, Grosmannia penicillata, and Huntiella bhutanensis. IMA Fungus. 7 (2), 317-323 (2016).
- Fourie, A., Van Der Nest, M. A., De Vos, L., Wingfield, M. J., Wingfield, B. D., Barnes, I. QTL mapping of mycelial growth and aggressiveness to distinct hosts in Ceratocystis pathogens. Fungal Genetics and Biology. 131, 103242 (2019).
- Lee, D. H., Roux, J., Wingfield, B. D., Wingfield, M. J. Variation in growth rates and aggressiveness of naturally occurring self-fertile and self-sterile isolates of the wilt pathogen Ceratocystis albifundus. Plant Pathology. 64 (5), 1103-1109 (2015).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır