Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
RNA Yapısı 'peroksidatif' ve 'Oksidatif' Hidroksil Radikal footprinting İzleme Denge Değişiklikler
Bu Makalede
Özet
Bu protokol, Mg (II) bağlı hidroksil radikal footprinting iki yöntem RNA üçüncül yapısının oluşumu ölçmek için nasıl kullanılacağını açıklar.
Özet
RNA molekülleri, biyolojide önemli bir rol oynamaktadır. , RNA genetik bilginin iletilmesi için ek olarak, regülatör, bağlayıcı veya katalizör olarak belirli bir biyolojik rolünü yerine getiren benzersiz üçüncül yapıları içine katlayabilirsiniz. Üçüncül temas oluşumu hakkında bilgi RNA molekülleri işlevini anlamak için önemlidir. Hidroksil radikalleri (• OH), yüksek reaktivite ve küçük boyutları nedeniyle nükleik asitlerin yapısını benzersiz probları 1 footprinting prob olarak kullanıldığında, hidroksil radikalleri DNA 1 ve RNA'nın 2 fosfodiester omurgası solvent erişilebilir yüzey haritası tek nükleotid çözünürlük gibi ince. Hidroksil radikal footprinting moleküller arası bir temas yüzeyi içerisinde nükleotidler, DNA-protein 1 ve RNA-protein komplekslerinin örneğin tanımlamak için kullanılır. Denge 3 ve 4 geçişler kinetik bir soluti bir fonksiyonu olarak hidroksil radikal footprinting yaparak tespit edilebilirdeğişken veya sırasıyla. Footprinting bir anahtar özelliği probu bu sınırlı maruz kalma (örneğin, 'tek-hit kinetiği), polimerin her bir nükleotid üniforması örnekleme sonuçları 5
Bu video makalede, RNA numune hazırlama ve Mg (II)-aracılı bir katlama izotermleri belirlenmesi göstermek için Tetrahymena ribozim P4-P6 etki alanı kullanın . H 2 O 2 (bu 'peroksidatif' protokolünün diyoruz) ve doğal olarak çözünmüş O 2 kullanan bir değerli, ancak yaygın olarak bilinen, alternatif (biz bu 'gerektiren bilinen bir hidroksil radikal footprinting protokolü kullanımını tanımlamak oksidatif 'protokolü). Veri azaltma, dönüşüm ve analiz işlemleri genel bir bakış sunulmuştur.
Protokol
1. Footprinting Reaktifler hazırlanması
- 100 mM sodyum cacodylate, 1 mM EDTA ve 1 M KCl içeren bir 10x reaksiyon tamponu hazırlayın. PH 7.4 'e ayarlayın. Filtre 0.2 mcM asetat filtre cihazı (Nalgene) kullanarak tampon. Not: 10x tampon doğrudan pipetlemeyin RNA yok.
- Tablo 1'de gösterildiği gibi her bir reaksiyon için titrasyon reaksiyon karışımı hazırlayın. Titrasyon karışımı hacmi (1x istenilen konsantrasyonda tampon ve Mg (II)), 1x tampon RNA 10μl eklemeden önce, 90 ul olmalıdır.
- 6.63M Üre, 20mm sodyum sitrat, 1mm EDTA, 0,25 mg / ml tRNA,% 0.025 ksilen cyanol,% 0.025 bromphenol mavi içeren bir RNAse T1 sindirim tampon hazırlayın. Bu tampon, 6 aya kadar 4 ° C saklanabilir.
- Fenton reaksiyonu 250 mM Fe (NH 4) 2 (SO 4) 2, 250 mM EDTA ve 500 mM sodyum L-askorbat taze olarak hazırlanan sulu çözeltiler gerektirir . Konsantrasyonları bölüm 1 not0,5 radikaller ve çeşitli kalsiyum iyon yoğunluğunun değil scavenge sodyum cacodylate gibi tampon denge deneyleri için en uygun olan.
- Peroksidatif (1.5.1) ve oksidatif (1.5.2) footprinting reaksiyon için Reaktifler.
- Peroksidatif Fenton reaksiyonu 100 mM Fe-EDTA çözeltisi ve 50 mM sodyum L-askorbat ve% 1.5 H 2 O 2 taze olarak hazırlanan sulu çözeltiler gerektirir . Fe-EDTA çözeltisi, demir üzerinde EDTA 1.1 kat fazla karıştırma Fe (NH 4) 2 (SO 4) 2 EDTA ile hazırlanmıştır .
- Sadece oksidatif parçalanma reaksiyonu (3.4.2) başlamadan önce, 2 ul ul Fe (NH 4) 2 (SO 4) 2 (250 mM), 2.2 EDTA ul (250 mM), 62.5 birleştirerek Fenton reaksiyon karışımı oluşturmak sodyum L-askorbat (500 mM), 22.5 ul 10x reaksiyon tamponu ve 0 footprinting reaksiyon karışımı (3.4.2) nihai konsantrasyonlarını ulaşmak için H 2 O 135,5 ul.10 mM 0.11 mM ve 6.61 mM, sırasıyla.
2. RNA footprinting Denemeye Hazırlık
- 50 nükleotidler, daha kısa (Integrated DNA Teknolojileri, www.idtdna.com gibi) satın alınması, RNA, DNA şablonları 6 in vitro transkripsiyon standart tarafından üretilen ya da olabilir . Footprinting deneyler sırasında RNA örnek bütünlüğünü korumak için 7 tavsiye edilen laboratuvar temizlik prosedürlerini izleyin .
- Defosforilasyon 8 in vitro transkripsiyonu RNA önce RNA'nın 5 'son 32 P etiketleme için gerekli. RNA 10 pmol kinasing tarafından uç-etiketlenmiş 9 çözüm fosfataz başarılı inaktivasyon sonra bulutlu olmalıdır.
- Denatüre jel elektroforezi kullanılarak radyoaktif etiketli RNA arındırın. RNA 0.3 M sodyum asetat (pH 5.5) ile iki sonraki jel ekstraksiyon tarafından kurtarılır. RNA ile 0,5 ml 1 ml etanol ve sonraki sulu çözeltisi karıştırılarak çöktürülmüşkuru buz üzerinde 1 dakika inkübasyon. 4 ° C, 0,5 saat süreyle 12.500 rpm örnek Spin. Süpernatantı atın ve RNA pelet% 70 etanol ile yıkayın. Ek santrifüj sonra süpernatantı ve pelet kuru vakum.
- 1x bir reaksiyon tüpünde RNA çözümü, etanol ve vakumlu kuru RNA ile çökelti Buffer.Pipet 30 ul 330 ul 32 P etiketli RNA örnekleri çözülür; referans merdiveni (adım 3.5) üretmek için bu tüp tutmak. 95 ısıtarak tamponlu RNA çözüm ° C 'de 2 dakika süreyle denatüre. Örnekleri 15 dakika oda sıcaklığında soğutun ve çözelti içinde tüplerin kapakları kondanseleri getirmek için hızlı bir spin vermek.
3. Footprinting Deney
- Elektroforez tarak, veri noktalarının analitik jel başına maksimum sayısını belirler. Biz 30 iyi bir tarak kullanın. Referans merdiven, 2 kuyu ve uncleaved kontrolü birini kaplar. 27 reaksiyon tüpleri hazırlayın. Son Mg (II) concentratititrasyon orta noktası etrafında büyüklük emirleri kapsayan bir logaritmik ölçekte ons eşit aralıklı olmalıdır.
- Ayrı ayrı 50 RNA ve Mg (II) çözeltileri ° C, 5 dakika boyunca inkübe edin. 100 ul son bir hacme ulaşmak için 1x tampon karşılık gelen miktarda Mg (II) 90 ul ile 10 ul RNA çözüm karıştırın. 50 - 30 dakika çözümler inkübe ° C
- Katlama oluşur sırasında çözümleri, 25 ° C'de 1 saat için muvazene edelim.
- Peroksidatif (3.4.1) veya oksidatif (3.4.2) hidroksil radikal footprinting reaksiyon.
- 2 ul Fe-EDTA (100 mM) damlacıkları koyarak peroksidatif reaksiyonu başlatın, 2 ul sodyum L-askorbat (50 mM) ve 2 ul H 2 O 2 (% 1.5) reaksiyon üst içinde birbirinden ayrılmış tüp RNA çözüm içeren ve dinç karıştırılarak footprinting reaksiyon başlatabilir. 300 ul soğuk mutlak etanol ekleyerek 15 saniye sonra tepki durdurun. Tüpler 3-5 kez çevirin. Çökelme, yıkama veadım 2.4 olarak pelet kurulayın.
- Oksidatif hidroksil radikal footprintingreaction taze hazırlanmış Fenton reaksiyon karışımı (1.6) 5 ul ekleyerek başlayın. 30 dakika boyunca inkübe 25 ° C Tüpler 3-5 kez çevirerek tepki ve karıştırın gidermek için soğuk mutlak etanol 300 ul ekleyin. Çökelme, yıkama ve kuru pelet gibi adım 2.4 'de belirtilen.
- Adım 1.3 'te hazırlanan çözüm kullanarak standart prosedürler 10 göre bir RNaz T1 sindirmek referans örnek oluşturun.
- 8 ul jel yükleme boyası II (Ambion) ve onaylamak RNA RNA pelet çözülür Geiger sayacı kullanarak yeniden süspanse.
- 11 Yük örnekleri iki referans ve kontrol dahil olmak üzere standart protokollere göre denatüre,% 8'lik poliakrilamid sıralama jel hazırlayın. 60 W Ayrı RNA parçaları - 2.5 saat için 75 W. Gecede bir depolama fosfor ekrana kurutulmuş jel Açığa. Tarama fosfor filmsiz otoradyografi, örneğin bir görüntüleme sistemi ile ekran. Fırtına 865 (GE Healthcare) veya Typhoon (GE Healthcare). Jel görüntü dosyası, bilgisayarınıza aktarın.
4. Veri Analizi
- Her bir nükleotid hidroksil radikal reaktivite analitik jel her bandın şiddeti nicel tarafından belirlenir. Karşıdan yükleyin, kurun ve açık SAFA, tek bant montaj ve ölçümü için açık kaynak kodlu yazılım kullanımı kolay bir 12, 13 kullanım kılavuzu yönergeleri izleyin. Kısaca, RNA dizisi olarak yükleyin. Txt dosyası. Jel dosyası olarak jel resmi izledi. Bant yoğunlukları ayarlayın, şerit tanımlamak, bir çapa şeritli seçin ve bir jel hizalama. Nükleotidleri Atama RNaz T1 sindirim merdiven referans gruplarından oluşur. Grubun integrallerin sayısal olarak ve potansiyel değişmez artıkları normalleştirmek ve atamak için normalleşme / colorplot özelliğini kullanabilirsiniz. Çıktı. Txt dosyası olarak kaydedin.
- SAFA temsil jel üzerinde şerit ve satır temsil eden sütunlar içeren bir elektronik tablo çıktılarıRNA parçası karşılık gelen bireysel bantları bant yoğunluğu tegrated. İlk olarak, çözücü erişilebilirlik gözle görülür bir değişim profili ile bitiş noktası Mg (II) konsantrasyonu (bizim örneğimizde 50 mM) Mg (II) örnek türetilmiş koruma profilinin karşılaştırarak ekran koruma siteleri de belirleyebilir. Değer ne kadar düşükse, daha nükleotid hidroksil radikalleri ve tersi saldırıya karşı korunmaktadır.
- Mg (II) konsantrasyonu karşı nükleotidler bireysel ya da gruplar normalize bant integraller katlanır izotermleri oluşturun. • - izotermleri fi = L + (L U) tarafından ayrı ayrı kesirli doygunluk ölçeklendirilir
 f grup (lar) entegre yoğunluğu gösterir analiz edilen L ve U geçiş için alt ve üst sınırları temsil eden ve
f grup (lar) entegre yoğunluğu gösterir analiz edilen L ve U geçiş için alt ve üst sınırları temsil eden ve 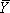 kesirli doygunluk 14 verilerin sığması non-lineer l kullanarakdoğu-Hill denklemi (1) kare analiz programı (ya Kökeni (OriginLab) veya GraphPad (GraphPad Software, Inc.) kullanın.)
kesirli doygunluk 14 verilerin sığması non-lineer l kullanarakdoğu-Hill denklemi (1) kare analiz programı (ya Kökeni (OriginLab) veya GraphPad (GraphPad Software, Inc.) kullanın.) 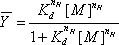
K d denge olduğu asitlik sabiti, [M] katlanır tepki aracılık değişken, bu örnekte Mg (II) konsantrasyonu gelir ve n H Hill katsayısı. Bu prosedür, kesirli doygunluk geçiş terazi, orta noktası belirler ve fenomenolojik bir geçiş sigmoidal olup olmadığını test sağlar. SAFA (Şekil 3B) tarafından analiz edilir ve yukarıda açıklandığı gibi uygun sıralama jeller (Şekil 3A) türetilen izotermleri Şekil 3C gösterilmiştir. Şekil 3'te gösterildiği gibi kesirli doygunluk izotermleri bir elektronik tablo ölçeklendirme tarafından oluşturulan denklem fraksiyonu kullanarak en uygun alt ve üst sınırları (U ve L) karşı SAFA tarafından oluşturulan değerleri = değer / (U-L) - L / (U - L).
5. Temsilcisi Sonuçlar:
Şekil 3, P4-P6 RNA • OH footprinting deneyler temsilcisi sonuçlar gösterir. Jel görüntüsünde (solda) T1 şeritli (Her G RNaz T1 parçalayarak) iyi tanımlanmış bantları nedeniyle olası tek nükleotid atama)) arka plan bölünme, sağ şeritte (b) az olduğunu gösterir, ve c) hidroksil radikali RNA indüklenmiş parçalanma iyi bir arka plan üzerindedir. RNA üçüncül yapısının oluşumu gösteren bantları bireysel ve gruplar entegre bant yoğunluğu azalan düşük ve yüksek Mg (II) geçiş bağlanmıştır. Tek veya bölünme değişiklikler eş zamanlı olarak 'koruma' olarak adlandırılır bitişik nükleotid grupları. Mg (II) yakından kristal yapısında gözlenen molekülünün çözücü ulaşılmaz bölgelere karşılık gelen karakteristik • OH korumaları P4-P6 RNA katlama aracılı 15
Katlanmış RNA molekülleri c bir çok farklı açık, yapısal ya da dinamik bir fenomen ile ilişkili olmamıştır üçüncül kişi koruma kapsamlarını (yani, nasıl arka bant yoğunlukları yakın düşüş) var. Böylece, bazı RNA molekülleri Şekil 3'te gösterildiği gibi, iyi tanımlanmış yapısal geçişler göstermek, ve bazı olmayacaktır. Band şiddetleri SAFA 12 analizi (Şekil 3B) tarafından sayısal ve normalize edilir. Çıkış • OH karşı koruma göreceli derecesi görselleştirmek bir 'termal' komplo. Renk geçişi, Mg (II) Ayrıca üzerine erişilebilirlik değişikliği açıklamaktadır. Sırasıyla kırmızı veya mavi, daha erişilebilir ve daha korumalı nükleotidler, Beyaz. Gölgeleme her bir derece, bir koruma eğrisi (Şekil 3C) olarak çizilen ve Hill denklemi (1) gibi bağlayıcı bir model ile analiz edilebilir bir sayısal değeri ile bağlanmıştır. Sabit disosiasyon Koruma 153-155 bağlı denge Koruma 163-164 karşılık gelen değeri yaklaşık iki kat fazladır.
ontent ">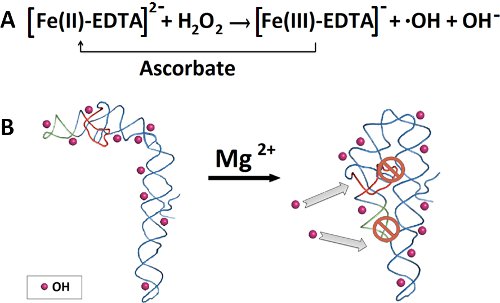
Şekil 1 Hidroksil radikal üretimi ve P4-P6 RNA katlanır. A) Fe (II), hidrojen peroksit, hidroksil radikalleri üretimi katalize eder. Askorbat demir demir Fe (III) geri azaltır. B) Mg (II) yokluğunda, hiçbir P4-P6 üçüncül yapısı, hidroksil radikalleri erişilebilir tüm omurga pozisyonları nüfuz ve tutunmaya izin oluşur. Mg (II) eklenmesi, sadece solvent erişilebilir omurga hidroksil radikalleri tarafından bölünmüş izin, P4-P6 katlayarak başlatır.
Şekil 2 hidroksil radikal footprinting deney için Anahat. A) Dephophorylation ve RNA 32 P 5'-etiketleme. B) 32 Arıtma denatüre poliakrilamid jel RNA P-etiketli. C) RNA bant eksizyonu sonraki RNA ekstraksiyonu ve etanol yağış. D) Prefolding veRNA katlanır. E) taze hazırlanmış Fenton reaksiyon karışımının eklenmesi hidroksil radikalleri oluşturur. F) poliakrilamid jel elektroforezi denatüre RNA parçası ayırma. G) SAFA yazılımı ile RNA parçaları kantitasyonu.
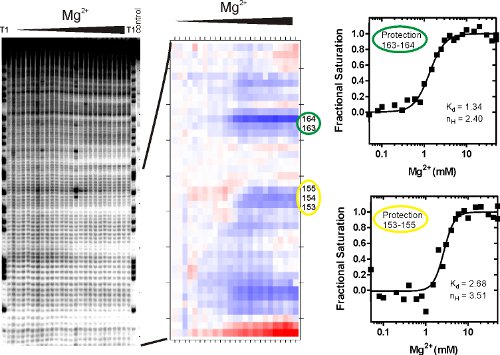
Şekil 3 peroksidatif Fenton footprinting reaksiyon sonra RNA parçaları Analizi. (A) RNA hidroksil radikalleri maruz kalmış ve parçalanma ürünleri denatüre poliakrilamid jel elektroforezi (PAGE) ile çözüldü. Resim, Mg (II) artan konsantrasyonu ile peroksidatif parçalanma ürünleri gösterir. Referans ve kontrol şeritleri etiketli. Bu görüntü dosyası, her bir band hacmi quantitates SAFA programı için girdi. (B) 'termal' komplo SAFA tarafından oluşturulur. (C) SAFA nükleotid sayısı çıkış entegre bant yoğunluğu ile ilgili bir tablo değerleri Hill equati olarak bağlayıcı bir model ile analiz(1). Koruma Bölgeleri, Mg (II) konsantrasyonunun bir fonksiyonu olarak bant ayrılmaz yoğunluğunda artışa göre seçilmiştir. K d RNA yarısı izlenmekte olan site katlanmış olduğu Mg (II) konsantrasyonu. Hill katsayısı, n H, bağlayıcı cooperativity hakkında bilgi verir ve bu özel site katlayarak yer alan magnezyum iyonlarının sayısı için daha düşük bir tahmin sağlar eğrisinin eğimi bir ölçüsüdür.
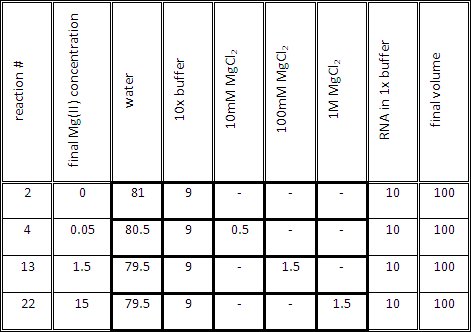
Tablo 1. Temsilcisi hacimleri farklı Mg (II) konsantrasyonu içeren RNA örnekleri üretmek için.
Tartışmalar
Hidroksil radikal footprinting nükleik asitlerin çözücü erişilebilir yüzey alanı değerlendirmek için değerli bir araçtır. Üçüncül yapısı 14 Kalitatif ve kantitatif oluşumu, iyon tipi ve konsantrasyonu, pH, sıcaklık, bağlayıcı proteinler veya katlama ko-faktörler gibi parametrelerin bir fonksiyonu olarak takip edilebilir . Yalındır ve ucuz bir protokol ve çıkan solvent erişilebilirlik ve tek nükleotid düzeyde katlanır bilgi zorlayıcı bir kombinasyon bu yöntem çok cazip hal...
Açıklamalar
Çıkar çatışması ilan etti.
Teşekkürler
Bu çalışma Sağlık RO1-GM085130 Ulusal Enstitüsü ve Ulusal Bilim Vakfı MCB0929394 bağışları ile destek verdi. Dr. Marion Schmidt Biz onun misafirperverliği ve onun laboratuar film bize izin verdiği için teşekkür ediyorum.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Name | Company | Cat# | |
| Sodium Cacodylate(Caution! Toxic) | Sigma-Aldrich | C4945-25g | |
| EDTA (0.5 M) | Ambion | AM9260G | |
| DEPC treated water | Ambion | AM9915G | |
| Sodium Acetate (3 M) | Ambion | AM9740 | |
| MgCl2 (1 M) | Ambion | AM9530G | |
| Urea | Ambion | AM9902 | |
| Sodium Citrate | Sigma-Aldrich | W302600 | |
| tRNA | Sigma-Aldrich | R-7876 | |
| Sodium-L-ascorbate | Sigma-Aldrich | A7631-25g | |
| Fe(NH4)2(SO4)2 . 6 H2O | Sigma-Aldrich | F1543-500g | |
| RNase T1 | Fermentas | EN0541 | |
| Hydrogen Peroxide (30%) | Sigma-Aldrich | 349887 |
Referanslar
- Tullius, T. D., Dombroski, B. A. Hydroxyl radical "footprinting": high-resolution information about DNA-protein contacts and application to lambda repressor and Cro protein. Proc. Natl. Acad. Sci. U.S.A. 83, 5469-5473 (1986).
- Celander, D. W., Cech, T. R. Iron(II)-ethylenediaminetetraacetic acid catalyzed cleavage of RNA and DNA oligonucleotides: similar reactivity toward single- and double-stranded forms. Biochemistry. 29, 1355-1361 (1990).
- Celander, D. W., Cech, T. R. Visualizing the higher order folding of a catalytic RNA molecule. Science. 251, 401-407 (1991).
- Sclavi, B., Sullivan, M., Chance, M. R., Brenowitz, M., Woodson, S. A. RNA folding at millisecond intervals by synchrotron hydroxyl radical footprinting. Science. 279, 1940-1943 (1998).
- Tullius, T. D., Dombroski, B. A., Churchill, M., Kam, L. Hydroxyl radical footprinting: a high-resolution method for mapping protein-DNA contacts. Methods. Enzym. 155, 537-558 (1987).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic. Acids. Res. 15, 8783-8798 (1987).
- Schlatterer, J. C., Brenowitz, M. Complementing global measures of RNA folding with local reports of backbone solvent accessibility by time resolved hydroxyl radical footprinting. Methods. 49, 142-147 (2009).
- Sambrook, J., Russell, D. W. . Molecular Cloning: A Laboratory Manual. , (2001).
- Zaug, A. J., Grosshans, C. A., Cech, T. R. Sequence-specific endoribonuclease activity of the Tetrahymena ribozyme: enhanced cleavage of certain oligonucleotide substrates that form mismatched ribozyme-substrate complexes. Biochemistry. 27, 8924-8931 (1988).
- Knapp, G. Enzymatic approaches to probing of RNA secondary and tertiary structure. Methods. Enzym. 180, 192-212 (1989).
- Slatko, B. E., Albright, L. M. Denaturing gel electrophoresis for sequencing. Curr. Protoc. Mol. Biol. Chapter 7, Unit 7.6-Unit 7.6 (2001).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. SAFA: semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. RNA. 11, 344-354 (2005).
- Simmons, K., Martin, J. S., Shcherbakova, I., Laederach, A. Rapid quantification and analysis of kinetic *OH radical footprinting data using SAFA. Methods. Enzym. 468, 47-66 (2009).
- Uchida, T., He, Q., Ralston, C. Y., Brenowitz, M., Chance, M. R. Linkage of monovalent and divalent ion binding in the folding of the P4-P6 domain of the Tetrahymena ribozyme. Biochemistry. 41, 5799-5806 (2002).
- Cate, J. H., Gooding, A. R., Podell, E., Zhou, K., Golden, B. L., Kundrot, C. E., Cech, T. R., Doudna, J. A. Crystal structure of a group I ribozyme domain: principles of RNA packing. Science. 273, 1678-1685 (1996).
- Petri, V., Brenowitz, M. Quantitative nucleic acids footprinting: thermodynamic and kinetic approaches. Curr. Opin. Biotechnol. 8, 36-44 (1997).
- Takamoto, K., Das, R., He, Q., Doniach, S., Brenowitz, M., Herschlag, D., Chance, M. R. Principles of RNA compaction: insights from the equilibrium folding pathway of the P4-P6 RNA domain in monovalent cations. J. Mol. Biol. 343, 1195-1206 (2004).
- Brenowitz, M., Senear, D. F., Shea, M. A., Ackers, G. K. Quantitative DNase footprint titration: a method for studying protein-DNA interactions. Methods. Enzym. 130, 132-181 (1986).
- Shcherbakova, I., Mitra, S. Hydroxyl-radical footprinting to probe equilibrium changes in RNA tertiary structure. Methods. Enzym. 468, 31-46 (2009).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for the study of protein association and assembly. Curr. Opinion. Chem. Biol. 10, 430-436 (2006).
- McGinnis, J. L., Duncan, C. D., Weeks, K. M. High-throughput SHAPE and hydroxyl radical analysis of RNA structure and ribonucleoprotein assembly. Methods. Enzym. 468, 67-89 (2009).
- Jonikas, M. A., Radmer, R. J., Laederach, A., Das, R., Pearlman, S., Herschlag, D., Altman, R. B. Coarse-grained modeling of large RNA molecules with knowledge-based potentials and structural filters. RNA. 15, 189-199 (2009).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır