Method Article
Gerçek zamanlı analiz bağlama, transkripsiyon, çeviri ve ciro hücresel etkinleştirme sırasında küresel olayları görüntülemek üzere transkripsiyon faktörü
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Bu iletişim kuralı ChIP-seq, 4sU seq, toplam RNA-seq ve hücre satırları ve Primer hücre için profil oluşturma ribozom Kombinatorik kullanımını açıklar. Transkripsiyon faktörü de novo transkripsiyon, RNA işlenmesi, ciro ve çeviri zamanla, bağlama ve olayların genel gidişatı harekete geçirmek ve/veya hızla değişen hücrelerde görüntüleme izleme değişiklikleri sağlar.
Özet
Harekete geçirmek, hücreleri hızlı bir şekilde onların fonksiyonel programları değiştirme ve, dolayısıyla, onların gen ifade profil. Büyük değişiklikler gen ekspresyonu gerçekleşir, örneğin, hücresel başkalaşım, morfogenez ve fonksiyonel stimülasyon (örneğin etkinleştirme bağışıklık hücreleri), sırasında veya yerel ortamdan uyuşturucu ve diğer etkenlere maruz kaldıktan sonra. Uyarıcı ve hücre türüne bağlı olarak, bu değişikliklerden hızla ve gen düzenlemesi mümkün herhangi bir düzeyde. Yanıt veren bir hücrenin tüm moleküler işlemler belirli bir uyarıcı/uyuşturucu görüntüleme Moleküler biyolojide en zor görevlerden biridir. Burada, tarif biz gen düzenlemesi çok katmanlı eşzamanlı analizini etkinleştiren bir iletişim kuralıdır. Biz, özellikle, transkripsiyon faktörü (Kromatin-immunoprecipitation-sıralama (ChIP-seq)) bağlama, de novo transkripsiyon (4-thiouridine-sıralama (4sU-seq)), mRNA işlemesi ve ciro gibi çeviri (ribozom karşılaştırın profil oluşturma). Bu yöntemleri birleştirerek, detaylı ve genom çapında elbette eylem görüntülemek mümkündür.
Yeni transkripsiyonu RNA sıralama hızla adapte veya bu 4sU maruz kalma (ne olursa olsun onlar yukarı - ya da downregulated olup) zamanında tüm genlerin transkripsiyon faaliyet gösteriyor bu yana sistemlerinin, değişen analiz ederken özellikle önerilir. Toplam RNA-seq ve ribozom profil oluşturma Kombinatorik kullanımı Ayrıca RNA ciro ve çeviri oranları hesaplama sağlar. Bioinformatic analizi yüksek üretilen iş sıralama sonuçlarının analizi ve verilerin yorumlanması için birçok araç sağlar. Oluşturulan veri Ayrıca co-transcriptional ve alternatif, sadece birkaç olası sonuçları söz Uçbirleştirme izleme sağlar.
Burada açıklanan kombine yaklaşım farklı model organizmalar veya Primer hücre de dahil olmak üzere hücre tipleri için uygulanabilir. Ayrıca, kalite kontrol, dahil olmak üzere kullanılan, her yöntem için detaylı iletişim kuralları sağlar ve olası sorunları ve tuzaklar tartışmak.
Giriş
Son yıllarda, RNA-sıralama (RNA-seq) bir hücre veya bir organizma1içindeki tüm ifade RNA'ların analiz etmek için Standart araç haline gelmiştir. Ancak, yanıt olarak belirli bir uyarıcı/ilaç adapte hücreleri tüm süreci anlamak için mRNA transkripsiyon işleme, ciro ve çeviri kadar tüm temel işlemler tam olarak belirlemek gereklidir. RNA transkripsiyonu kısa vadeli değişiklikler pek ölçülebilir toplam RNAseq tarafından faktörler örneğin RNA yarı-hayat ve transkripsiyon etkinlik, değişiklikleri toplam RNA'ın bağlıdır beri hücrelere uyarlaması yansıtan için zavallı bir şablon olan çevresel etkileri2,3. Gerçekten de, yeni sıralama teknikler geliştirilmiştir farklı adımları doğru şekilde birleştirildiğinde gen yönetmelik4 sürecinde bir analizini sağlayan. Bu iletişim kuralı, mRNA temel katmanları Yönetmeliği karşılaştırmalı bir şekilde izleme sağlar bazı oldukça kolayca uygulanabilir sıralama teknikleri birleştirmek açıklar. Transkripsiyon faaliyetini çözümleme için çeşitli yöntemler tarif edilmiştir, Cap-analizi gibi gen ifade (Kafes)5, yerli uzama transkript sıralama (NET-seq)6ve genom çapında nükleer çalıştırma (GRO-seq)7 , 8, hem de bromouridine-sıralama (Bru-seq) ve 4-thiouridine-sıralama (4sU-seq), metabolitleri kullanan bu yeni eklenen transkripsiyonu RNA9,10, sadece birkaç söz için. KAFES tam transkripsiyon başlangıç sitesini tanımlayan, NET-seq ve GRO-seq yön okuma hakkında daha doğru bilgi sunmak ve yalnızca yeni (ki burada açıklanan yöntemi) 4sU seq algılar ise RNA transkripsiyonu. Ancak, 4sU seq son derece hassas ve farklı zaman dilimlerini aktif hücreleri gibi (ki birkaç dakika içinde olur) mRNA işlemesi nicel değişiklikler değiştirme nicel transkripsiyon etkinliğini ölçmek için uygulanabilen9, 11,12. Ayrıca, 4sU seq genler9için RNA ciro oranları hesaplamak için RNA-seq ile birleştirmek idealdir. MRNA transkripsiyonu RNA polimeraz II (transkripsiyon faktörleri, Histon değişiklikler ve hatta genel harekete geçirmek/repressors transkripsiyon parçası olması gibi faktörler, çok sayıda tarafından sırayla etkilemiş RNAPII tarafından), yürütülen karmaşık. Nasıl pek çok gen/organizatörü/artırıcı bölge bir faktör tarafından bağlı olan test etmek için ChIP-seq, hangi şimdi birçok ticari olarak mevcut antikorlar13nedeniyle bu amaçla standart yöntemdir geliştirilmiştir. Her ne kadar ChIPseq nerede bind düzenleyen faktörler net bilgi verir, ancak, bu gerçekten transkripsiyon14değişikliklere yol Eğer yansıtmak zorunda değildir. Bu nedenle, ChIP-seq 4sU-seq ile gerçekleştirme biyolojik tür sorular için en ideal kombinasyonu. MRNA ve protein düzeyleri mutlaka15,16translasyonel veya post-translational potansiyel olarak önemli regülasyonu gösteren, ilişkilendirmek değil bu yana, düzenleme gen ekspresyonu, daha sonraki bir aşamada da oluşabilir düzeyi, bağlama. Yıl 2011, ribozom profil oluşturma ilk RNA-seq ile kombine edilmiş ve kütle spektrometresi17ile bazı duyarlılık sınırlar hala olduğundan hızla protein, oluşan değişiklikleri ölçmek için seçtiğiniz yöntemi şimdi. Nitekim, çeviri oranları bu tür yöntemleri elde nispeten iyi bir tahmin (hiç olmazsa için uzun vadeli değişiklikleri ölçülür) protein düzeyleri değişiklikleri teslim ve çeviri, örneğin süreci üzerinde daha detaylı bir görünümüne izin ver gösterilmiştir başlangıç tarafı ve alternatif çeviri belirlenmesi17çerçeveler. Dört yöntemi Kombinatorik kullanımı arasında çeşitli hücre tiplerinin, kararlı duruma kullanılabilir veya zaman-seri hızla değişen hücre11deneme. Bu kullanım transkripsiyon faktörü bağlama RNA transkripsiyonu, işleme ve çeviri etkileyen değişiklikler genom geniş bakış sağlar.
Protokol
Uygun olarak ve Helmholtz Zentrum München kurumsal, eyalet ve federal yönergeleri ile uyum içinde tüm yöntemlerdir.
1. hazırlık
- Deneysel kurulumunun bir zaman planı da dahil olmak üzere ayrıntılı bir plan yapmak, ne zaman hücre kültür ortamına 4sU ekleneceği ve her yöntem için hücre hasat zamanı. Biyolojik soru bağlı olarak zaman Puan örnek çizim, 4sU etiketleme zaman ve konsantrasyon (Tablo 1) için dikkatlice.
Not: 4sU hücre canlılığı üzerinde etkisini doğrulayın ve stres yanıt önceden ("Doğrula en uygun 4sU etiketleme koşullarda" temsilcisi sonuçları ve şekil 1bakınız). Bu her yöntemi en az bir örnek ile bir ön test gerçekleştirmek için tavsiye edilir. RNA/DNA miktarı ve kalitesini derin sıralama (iletişim kuralı bkz: adanmış parçaları) ve uygulama hızlı ama hücreleri nazik işleme sırasında deney için yeterli olup olmadığını doğrulayın. - Her yöntemi her zaman noktası için gerekli hücrelerin sayısını hesaplamak (kaba bir tahmini için birincil T hücreleri kullanırken bkz. Tablo 2 ). Ayrıca, toplam RNA 4sU RNA (örneğin, sadece zaman Puan çeviri oranları veya RNA ciro oranları hesaplanması için) sadece bundan daha az örnekleri sıralama göz önünde bulundurun. Onaylamak ve önemini (önerilen) sonuçları doğrulamak için en az bir biyolojik REPLICATE oluşturun.
Not: Önemlisi, tüm yöntemleri ve ardışık zaman puan için örnekleri hücreleri aynı başlangıç havuzundan olması gerekir. En az bir özel araştırmacı her yöntem için önerilir. - Önceden yer (örneğin, bölünmemeli 4sU, sikloheksimit, memeli lizis arabellek ve % 1 formaldehit) gerekli her şeyi hazırlayın. Bu 4sU etiketli RNA hücresel proteinler18polietilenin neden olabilir gibi 4sU eklenmesinden sonra parlak ışık, hücrelere kullanılmasını önlemek.
- Hemen önce tedavi (Şekil 2) bir şişeye ilgi tüm hücreleri havuz. (Örneğin, bir hemasitometre ile) hücreleri saymak ve her yöntem tedavi edilmezse denetim için gerekli miktarı kullanmak (Ayrıca 4sU-seq 4sU ile tedavi edilmezse kumandasında etiketlemek unutmayın). Kalan hücreleri tedavi ve hemen her zaman nokta ve yöntemi için gerekli sayısı bölünmüş. Hücreleri stres ısı veya CO2 düzeyleri değişiklikler nedeniyle en aza indirmek için mümkün olduğunca hızlı bir şekilde başa.
Not: Örneğin, örnekleri 1 h, 2 h, alınır ve 3 h tedavi sonra sonra dörtte birini hücreleri kullanılır için tedavi edilmezse denetim ve üç çeyrek tedavi kontrolü için kullanılır.
2. 4sU etiketleme
Not: Rädle, bu iletişim kuralı değiştirilir ve ark. 19 onların Protokolü metabolik 4sU ile etiketleme ile ilgili daha fazla bilgi için bakın. Tüm yöntemleri ve ardışık zaman puan için örnekleri hücreleri aynı başlangıç havuzundan kaynağı gerekir.
-
Başlangıç maddelerinin etiketlenmesi
- Tezcan bölünmemeli 4sU kullanmadan önce. 4sU her zaman yere doğrudan ilgi hücreleri içeren orta ekleyin (bakınız Tablo 2 önerilen T hücre numaraları için her zaman noktası en az 60 µg RNA) karışımı yavaşça ve geri kuluçka makinesi yerleştirin. 4sU kalan dispose (değil refreeze).
- Etiketleme sonunda, hücreleri (örneğin, hücre kazıyıcı) ve 330 x g, santrifüj (hangi yüksek g Kuvvetleri karşı koymak) polipropilen borular içinde 4 ° C'de 5 min için toplamak. Orta Aspire edin ve reaktif RNA izolasyonu için ekleyin (≥1 mL 3 x 106 hücreler, başına bkz: tavsiye için malzeme hangi kullanmak için) her tüp için. Tam olarak Pelet (3 x 106 hücre başına ≥1 mL) resuspend, (RT) oda sıcaklığında 5 min için kuluçkaya ve örnekleri-20 ° C'de dondurmak Örnekleri-20 ° C'de en az 1 ay depolanabilir.
Uyarı: RNA yalıtımı için kullanılan reaktifler cilt veya gözlerle temas alırken son derece tehlikeli. Bunlar özenle ele almak ve güvenlik talimatlarını. göz önünde bulundurun
-
RNA hazırlık kullanarak RNA izolasyon iletişim kuralı değiştirildi
- 0.2 mL kloroform 1 mL reaktif RNA izolasyonu için ücret ekleyin ve iyice karıştırın metabolik etiketleme iletişim kuralında (Adım 1-12, 2. belirtildiği gibi 15 s. devam et için sallayarak RNA hazırlık kullanarak değiştirildi trizol Protokolü) üzerinden Rädle ve ark. 19
- Ölçmek RNA konsantrasyonu (bkz. Tablo malzemeler), üreticinin yönergelerine göre. Bu RNA toplam RNA-seq için de kullanın (bkz. Adım 3. «Toplam RNA-seq) veya en az 1 ay boyunca-80 ° C'de depolayın.
-
Yeni transkripsiyonu RNA'ın thiol özel Biotinylation
- Toplam hücresel RNA'ın 30-80 µg ile başlayın. 60 µg RNA yeni transkripsiyonu RNA yeterli miktarda verim.
- Etiketleme tepki hazırlayın. Pipet (başına µg RNA) aşağıdaki sırayla: 1 µL 10 x Biotinylation arabellek, 7 µL RNA (1 µg nükleaz ücretsiz H seyreltilmiş RNA içeren2O) ve 2 µL biotin-HPDP (1 mg/mL). Biotin-HPDP son ve hemen pipetting tarafından karıştırın. Tüpler parlak ışığa maruz kalma önlemek için alüminyum folyo ile sarın. Biotin-HPDP bir alternatif için bkz. RT rotasyon ile 1,5 saat için kuluçkaya.
- 15.000 x g 2 dakika süreyle, santrifüj uygun 2 mL tüpler ( Tablo malzemelerigörmek) tüm biotinylated RNA önceden bükülmüş 2 mL tüp içine Pipette, kloroform eşit bir birim eklemek ve kuvvetle karıştırın. 2 için kuluçkaya-3 aşama ayrılmaya başlayana min ve kabarcıklar kaybolmaya başlar.
- 4 ° C'de 15 dak için 15.000 x g, santrifüj Dikkatle üst sulu faz yeni bir tüp içine aktarın.
- 2.3.3 adımları yineleyin. ve 2.3.4. bir kez. NaCl (5 M) hacmi ve isopropanol eşit bir hacmi yüzde 10'u sulu faz ekleyin. 4 ° C'de 20 dk için 20.000 x g, santrifüj Süpernatant atmak.
- Eşit bir cilt taze % 75 etanol hazırlanan ekleyin. 20.000 x g. atma süpernatant santrifüj kapasitesi, kısaca spin ve kalan etanol kaldırın. Pipet karıştırma tarafından 30-100 µL H2O (kullanım 1 µL H2O 1 µg giriş RNA--dan adım 2.3.1 başına) tam olarak resuspend.
- Elektroforetik analiz, RNA bütünlüğünü doğrulamak veya bir aliquot almak ve sonra doğrulayın.
-
Yeni ayrılması transkripsiyonu (etiketli) ve (etiketsiz) RNA önceden
- Paramagnetic boncuk ( Tablo malzemelerigörmek) 4 ° C deposundan kaldırmak ve onları oda sıcaklığına getirmek için en az 30 dakika stand izin verin. 65 ° c ısı 4sU çamaşır arabellek (örnek başına 3 mL)
- 100 mM dithiothreitol (DTT) çözüm hazırlamak. DTT Ultra-ince bir ölçekte tartmak ve gerekli miktarda nükleaz ücretsiz su ekleyin. Her zaman taze hazırlayın. Örnek başına 200 µL kullanın.
- Biotinylated RNA örnekleri (1 µg/µL) tabiatını ve hemen buza yerleştirmek 10 dk 65 ° c ısı. Biotinylated RNA 100 µL streptavidin boncuk ekleyin ve 15 dakika döndürme RT kuluçkaya.
- Uygun bir sütun yer (öneriler için bkz: Tablo reçetesi ) her biri için örnek manyetik stand içine ve her sütun 1 mL oda sıcaklığında 4sU çamaşır arabelleği ile önceden equilibrate.
Not: Bu yaklaşık 5-10 dakika sürer. Sütunlardan herhangi birine 5 dk sonra boşaltma başlatmak değil, eldivenli bir parmak ile sütun üzerine hafifçe bastırın. - Bir RNA/boncuk karışımı her sütun ortasına kadar geçerlidir. Etiketlenmemiş RNA kurtarılması gereken sürece akışı-aracılığıyla atın. Eğer öyleyse, akışı sayesinde ve en az ilk yıkama toplamak. RNA Rädle vd tarafından açıklandığı gibi kurtarması (Adım 1-7, 7. Kurtarma etiketsiz, ilişkisiz RNA)19.
- Üç kez (65 ° C'ye ısıtılmış adımından 2.4.2.) 0.9 mL 4sU çamaşır tampon ve 0.9 mL RT 4sU çamaşır tampon, sırasıyla yıkayın.
- Paramagnetic boncuk yeni transkripsiyonu RNA kurtarmak için kullanın. İyi dağınık RT paramagnetic boncuk bir tüp örnek ve her sütunun altında yer başına 400 µL pipet. 100 µL 100 mM DTT ile yeni transkripsiyonu RNA elute. İçin 3 dakika bekleyin ve 100 µL 100 mM DTT ile ikinci bir elüsyon gerçekleştirin. (İsteğe bağlı: elüsyon ve kurtarma işlemi yapma Rädle vdtarafından açıklandığı gibi) 19
- Mix yeni RNA/boncuk iyice 10 kat karıştırma pipet tarafından transkripsiyonu ve üreticinin kılavuz göre devam edin. 11 µL nükleaz ücretsiz RNA elute H2O.Quantify uygun bir yer kullanarak RNA ( Tablo malzemelerigörmek). RNA-80 ° C'de en az 1 ay depolanabilir.
Not: Yeni transkripsiyonu RNA cDNA kütüphaneler yeni nesil sıralama için hazırlamak için kullanılabilir (bir öneri kullanmak için hangi kiti için Malzemeler tablo bakınız) veya daha aşağı akım analizleri. 100 - 500 ng RNA (konuya bakın) en Kütüphane hazırlık kitleri için yeterli.
3. Toplam RNA-Seq
- Doğrudan sonra değiştirilmiş reaktif RNA izolasyon protokolü için toplam RNA-seq için kullanarak RNA hazırlık RNA etiketli 4sU RNA almak (bkz. Adım 2.2.2.)
- Kitaplık hazırlama, seyreltik bir RNA aliquot son konsantrasyonu 50-100 NG / µL. kullanmak gelince yeni transkripsiyonu RNA hazırlanması kitaplığı aynı seti. 100 - 500 ng RNA en Kütüphane hazırlık kitleri için yeterli.
4. ribozom profil oluşturma
Not: tüm yöntemleri ve ardışık zaman puan için hücreleri aynı başlangıç havuzundan örnekleri olması gerekir. Hangi kiti kullanmak için Tablo malzemeleriçin başvurmak için öneriler.
- Hazırlık ve yalıtım ribozomların parçaları (RPFs) korumalı:
- Her zaman noktası için hücreleri uygun miktarda kullanın (önerilen T hücre sayıları için Tablo 2 ' ye bakın). Sikloheksimit ile yapışık hücreleri üreticinin iletişim kuralında tanımlandığı gibi davran.
Dikkat: Sikloheksimit çok zehirli ve mutasyonlar neden olabilir. Ciltle temas ve inhalasyon kaçının. - Toplamak ve havuzu olmayan - ya da yarı - adherent hücrelerden her zaman bir polipropilen boru üzerine gelin ve hücre özgü orta (örneğin, T hücreleri için ilave RPMI) son konsantrasyon mL başına 1 x 106 hücre için ayarlayın. Sikloheksimit 0.1 mg/mL nihai bir konsantrasyon ile eklemek, polipropilen boru ters çevirme karıştırın ve 1 dk. için santrifüj hücreleri vasıl 330 x g 4 ° C'de 5 min için kuluçkaya Orta Aspire edin ve hücrelerin en az 10 mL sikloheksimit (son konsantrasyon 0.1 mg/mL) ile desteklenmiş PBS ile yıkayın.
- Vasıl 330 x g 4 ° C'de 5 dakika santrifüj hücreleri Orta Aspire edin ve 10 x 106 hücre başına 100 µL memeli hücre lizis arabellek ekleyin. Pipetting tarafından mix ve hücreleri tamamen koşullar için bir steril 22 25 lik bir iğne sınırdışı.
- Lysate hücre precooled 1,5 mL tüp aktarın. Periyodik silinme ile buzda 10 min için kuluçkaya. Santrifüj 20.000 x g lysate netleştirmek için 4 ° c de 10 dakika. Süpernatant precooled 1,5 mL tüp aktarın.
- 1:10 hazırlamak seyreltme, nükleaz ücretsiz su ve bir Spektrofotometre kullanarak kayıt A260okuma ile lysate. Nükleaz içermeyen bir boşluk ve bir 1:10 suyuyla memeli hücre lizis arabellek seyreltme standart olarak. Lysate göre aşağıdaki denklemi A260/mL konsantrasyonu hesaplayın:
(260 hücre lysate -260 memeli lizis arabellek) 10 seyreltme faktörü x260/mL = - 200 µL aliquots lysate, buz üzerinde oluşturmak ve nükleaz tedavi ile devam edin.
Not: İsteğe bağlı olarak, 100 µL aliquot için toplam RNA hazırlamak, 10 µL % 10 SDS ilave edilerek karıştırılır. 4 ° C'de depolayın ve 4.3.2 ile devam edin. Toplam RNA 4sU etiketli RNA üzerinden kullanarak önerilir (bkz. Adım 3. Toplam RNA-seq).
- Her zaman noktası için hücreleri uygun miktarda kullanın (önerilen T hücre sayıları için Tablo 2 ' ye bakın). Sikloheksimit ile yapışık hücreleri üreticinin iletişim kuralında tanımlandığı gibi davran.
- Ribozom footprinting
- Nükleaz tedavi hemen lysate donma olmadan gerçekleştirmek. 7.5 (önerilen kit ile birlikte) nükleaz birimlerinin her A260 lysate in için ekleyin. Örneğin: 80 A260/mL 0.2 mL 7,5 U/A260nükleaz x lysate x lysate = 120 U nükleaz.
Not: İsteğe bağlı olarak, üretici tarafından açıklandığı gibi sindirim için nükleaz titre. - Nazik karıştırma ile 45 dk RT, nükleaz tepki kuluçkaya. 200 µL aliquots lysate sıvı azot ile dondurma ve-80 ° C'de depolayın veya nükleaz tepki her 200 µL aliquot 15 µL RNase inhibitörü ekleyerek, durdurup 4.3.2 adımıyla devam edin.
- Nükleaz tedavi hemen lysate donma olmadan gerçekleştirmek. 7.5 (önerilen kit ile birlikte) nükleaz birimlerinin her A260 lysate in için ekleyin. Örneğin: 80 A260/mL 0.2 mL 7,5 U/A260nükleaz x lysate x lysate = 120 U nükleaz.
- RPFs arıtma
- Sindirmek nükleaz RPFs bir örnek çöz ve 15 µL RNase inhibitörü ekleyin. Buz üzerinde örnekleri tutmak.
- Üreticinin protokolüne göre RPFs arındırmak (sütun arıtma önerilir) ve bir Spektrofotometre RNA konsantrasyon ölçmek.
- rRNA tükenmesi
- Saf RPFs 5 µg rRNA tükenmesi için kullanın.
- Üreticinin iletişim kuralı (Adım 1-2, birincil rRNA tükenmesi) rRNA tükenmesi için izleyin. RRNA ölçü RNA konsantrasyonu bir Spektrofotometre RPFs tükenmiş.
- RPFs sayfa arıtma
- Kullanım 500 ng rRNA, sayfa arıtma RPFs tükenmiş.
- RNA kontrolü, örnekleri ve merdiven sayfa arıtma için hazır olun. Boya bir 0.5 mL microcentrifuge tüp yükleme RNA denetim ve 5 µL denaturing jel karışımı 5 µL. Yükleme, sırasıyla jel denaturing 10 µL ile Mix 10 µL her RPF in. Bir merdiven aliquot hazırlamak (4 µL 20/100 merdiven, 1 µL nükleaz ücretsiz su ve 5 µL denaturing jel yükleme boya). Her örnek ve crosscontamination önlemek için denetim arasında yük.
- Örnekleri ve merdiven 95 ° c 5 min için kuluçka tabiatını ve hemen buza koyun. 20 µL her örneğinin yükleme (isteğe bağlı olarak, 10 µL ve örnekleri 20 ° C'de kalan freeze yüklemek) 10 µL % 12 veya % 15 üre-polyacrylamide jel üzerine hazırlanan merdivenin ayrılmış. RNA denetiminin 10 µL yükleyin. Bromophenol mavi bant jel (180 V, ~ 70 dk) sonuna ulaşana kadar jel çalıştırmak (şekil 3).
- Leke 4 ° C'de üreticinin protokolüne göre jel RNA görselleştirmek için mavi ışık yayan bir karanlık-alan transilluminator kullanın. Jel dilimleri ~ 28 ve 30 karşılık gelen her örnek için tüketim nt uzunluğu. Referans olarak RNA kontrol edin ve bu tüketim.
Not: RPFs zor görünür. Dilimleri örnekleri görünür değilse bile - 28 ve 30 NT uzunluğunda iki oligos içeren - RNA denetimi tarafından belirtilen boyutta tüketim. - 0.5 mL microcentrifuge tüpler dibinde bir delik ile steril bir 20'lik iğne ponksiyon. Her jel dilim ayrı bir tüp ve şapkalı yer tüpler bir 1,5 mL tüp içine aktarın. Santrifüj 12.000 jel dilimleri tamamen 1,5 mL tüp içine parçalamak değil eğer g. tekrar Santrifüjü x 2 min için.
- RNA kesintiye jel dilimleri 400 µL nükleaz ücretsiz su, 40 µL amonyum asetat (5 M) ve 2 µL SDS (% 10) ile gelen her gecede 4 ° C'de elute
- Bulamaç 1,5 mL filtre tüpler (önerilen kit ile sağlanan) için 1 mL pipet ucu (wide-geçişli ipucu veya kendi kendine yapılan 1 mL ucu kesik ucuyla) aktarabilirsiniz. Ayırmak için 2.000 x g de 3 dk santrifüj RNA jel dilimleri eluted. Yavaşça sulu çözüm 1.5 mL tüp içine pipet. (Önerilen kit ile sağlanan) 2 µL glikojen ve 700 µL % 100 isopropanol ve mağaza eklemek için en az 1 h-20 ° C'de.
- 13.000 x g., 20 dk için 4 ° C'de atma süpernatant santrifüj kapasitesi. Pelet önceden soğutulmuş taze hazırlanmış %80 etanol ile 13.000 g. atma süpernatant x, 10 min için 4 ° C'de yıkama ve kuruması. 20 µL her örnek ve 8 µL nükleaz ücretsiz su RNA denetiminde resuspend. -20 ° C'de gerekirse depolar.
- Parçalanma, son onarım, 3' adaptör ligasyonu, ters transkripsiyon
- Üreticinin iletişim kuralı (parçalanma ve sonunda tamir, 3' bağdaştırıcısı ligasyonu ve ters transkripsiyon) tarafından açıklanan yordamı gerçekleştirin.
- CDNA sayfa arıtma
- Örnekler, RNA denetim ve merdiven sayfa arıtma için hazırlamak: Mix 10 µL her örnek ve RNA kontrol ile 10 µL jel boya, sırasıyla yükleme denaturing. Bir merdiven aliquot hazırlamak (4 20/100 µL merdiven, 1 µL nükleaz ücretsiz su, 5 jel boya yükleme denaturing µL). Her örnek ve crosscontamination önlemek için denetim arasında yük.
- Örnekleri ve merdiven 95 ° c 5 min için kuluçka tabiatını ve hemen buza koyun. 20 µL her örneğinin yükleme (isteğe bağlı olarak, 10 µL ve örnekleri 20 ° C'de kalan freeze yüklemek) bir % 10 polyacrylamide/7 - 8 M üre/TBE jel üzerine hazırlanan merdivenin 10 µL ayrılmış. RNA denetiminin 10 µL yükleyin. Mavi bromophenol tamamen jel (180 V, ~ 60 dk) dışında geçirir kadar jel çalıştırın.
- Leke 4 ° C'de üreticinin protokolüne göre jel RNA görselleştirmek ve jel dilimler ~ 70-80 nt için karşılık gelen her örnek için tüketim mavi ışık yayan bir karanlık-alan transilluminator kullanın.
- Basamakta 4.5.5 - 4.5.8 açıklandığı gibi devam etmek ve her örnek nükleaz ücretsiz 10 µL suda resuspend.
- cDNA daireselleşmesi
- Buz üstünde her örnek için aşağıdaki reaktifler birleştirerek bütün tepkiler için yeterli daireselleşmesi ana karışımı hazırlamak: 4.0 µL daireselleşmesi reaksiyon Mix, 2.0 µL ATP, 2.0 µL MnCl2ve 2.0 µL ligaz.
- Ana Mix 10 µL her örnek için ekleyin. Karışımı yavaşça ve kısaca santrifüj kapasitesi. Hemen yer buz üzerinde örnekleri örnekleri 2 h. için 60 ° C'de kuluçkaya.
- PCR güçlendirme
- PCR güçlendirme için üreticinin Protokolü (Adım 1-3, PCR güçlendirme) izleyin. Circularized cDNA 4 µL amplifikasyon birincil T hücreleri, 9 ÇSYİ devredir ile en iyi sonuçları elde etmek için kullanın.
- Kitaplıkları arındırmak ve üreticinin iletişim kuralı (Adım 4-8, PCR güçlendirme) göre boyut dağılımı kontrol edin. 140-160 bp güçlendirilmiş Kütüphanesi beklenen boyutudur (bkz. şekil 4).
- Sıralama kitaplıkları için üreticinin iletişim kuralı ve sıralama tesisi daha fazla yardım için başvurun.
5. chIP-Seq
Not: Bu protokol Blecher-Gönen değiştirilir ve ark. 14 onların Protokolü ChIP-devamı ile ilgili daha fazla bilgi için bakın Tüm yöntemleri ve ardışık zaman puan için örnekleri hücreleri aynı başlangıç havuzundan olması gerekir.
- Crosslinking ve hücreleri hasat
- (Önerilen T hücre sayıları için Tablo 2 ' ye bakın) hücreleri % 1 formaldehit bir ortamda cellspecific (örneğin, T hücreleri için ilave RPMI) oda sıcaklığında 10 dakika için son bir konsantrasyon ile her zaman için uygun sayıda Crosslink hafif sallanan ile. Çapraz reaksiyon 0,125 M son bir konsantrasyon için glisin eklenmesiyle durdurmak.
- 330 x g 4 ° C'de 5 dakika santrifüj hücreleri Süpernatant atın ve buz gibi PBS hücrelerde yıkayın. 5.1.2 iki kez adımları yineleyin ve hücre granül-80 ° C'de dondurmak Donmuş granül en az 6 ay süreyle saklanabilir.
- Hücre lizis ve Sonication
Not: tüm hücre lizis ve sonication adımları uyguladığınızda, buz üzerinde veya 4 ° C'de crosslink ters ve protein bozulması en aza indirmek için örnekleri tutulmalıdır.- Hücre topakları 1 mL buz gibi hücre lizis arabelleği (fosfataz inhibitörleri isteğe bağlı olarak eklemek) çekirdeği yalıtmak için taze ekledi proteaz inhibitörleri ile resuspend. Buz ve 4 ° C'de 5 min için 2600 x g, santrifüj 10 min için kuluçkaya
- Süpernatant Aspire edin ve 1 mL buz gibi çekirdek lizis arabelleği taze ekledi proteaz inhibitörleri ile çekirdeği Pelet resuspend (isteğe bağlı olarak: fosfataz inhibitörleri ekleyin). Buz üzerinde 10 min için kuluçkaya. 0,2 - 1.0 kb ortalama bir DNA boyutu kısmını oluşturmak için hücre solüsyon içeren temizleyicide (bkz şekil 5).
Not: Sonication koşullar gerek hücre tipine göre en iyi duruma getirilmiş ve daha fazla (örneğin, cep numarası, ses ve tampon) koşulları. Birincil T hücreleri için sonication için 20-25 devir (ayrıntılı açıklama için bkz: belgeleri) önerilir. - 20-50 µL yamultulmuş Kromatin ve 10 min 95 ° c ve hızlı geri crosslink gerçekleştirin ve Kromatin boyutu doğrulamak için sallayarak 1000 rpm için ısı aliquot al. 2-5 µL İndinavir K ekleyin ve 20 dk 56 ° C ve 1000 rpm sallayarak için kuluçkaya. Isı inactivation 10 min 95 ° C ve 1000 rpm sallayarak gerçekleştirir. Kromatin uygun bir kit ile arındırmak ( Tablo malzemelerigörmek). Kromatin boyutu % 1'özel jel üzerinde kontrol ve 100 bp artı Marker kullanın.
- DNA boyutu 0.2 - 20.000 x g ve 4 ° C'de 10 dakika için 1.0 kb kısmını bir ortalamaya sahip yamultulmuş Kromatin çözünmez Kromatin ve enkaz cips santrifüj kapasitesi. Süpernatant aktarmak için yeni bir tüp ve buz üzerinde tutun.
- 5-%10 sonicated Kromatin giriş olarak tutmak. -20 ° c (Adım 5.5.2 kullanılan) dondur.
- Çift antikor boncuk için
- Çift 10 µg antikor (örneğin, anti-RNA Pol II; anti Histon H3K36me3) 220 µL PBS içinde (% 0.5 BSA ve % 0,5 ile ara 20) 80 µL superparamagnetic boncuk G protein birleştiğinde için (bkz. Tablo reçetesi) döndürme ile oda sıcaklığında en az 1 h için.
- Tüpler bir mıknatıs yerleştirmek. Tüm boncuk için mıknatıs bağlıdır ve süpernatant kaldırmak kadar bekleyin. 6 µL ile daha fazla blok sonicated somon sperm DNA PBS ' (% 0.5 BSA ve % 0,5 ile ara 20) döndürme ile oda sıcaklığında 30 dakika.
- Tüpler bir mıknatıs yerleştirmek. Tüm boncuk mıknatıs için bağlı ve açık süpernatant kaldırmak kadar bekleyin. Boncuk çip IP arabelleği ile üç kez yıkayın.
- Kromatin Immunoprecipitation
- Kromatin çekirdeği lizis arabelleği taze ekledi proteaz inhibitörleri ile 1 mL toplam birime seyreltik (isteğe bağlı olarak, fosfataz inhibitörleri ekleyin). Taze ekledi proteaz inhibitörleri ile çip IP tampon Ekle (isteğe bağlı olarak, Ekle fosfataz inhibitörleri) 3 mL son hacmi için. Antikor için boncuk birleştiğinde 4 ° C veya buz üzerinde tutmak.
- Antikor için seyreltilmiş Kromatin Boncuk 5.3.3 adımından birleştiğinde ve gece nazik rotasyon ile 4 ° C'de kuluçkaya ekleyin.
- Yıkama ile (1 mL her, bkz: Malzemeler tablo) aşağıdaki tampon döndürme 5 min için oda sıcaklığında, tüpler mıknatıs üzerinde geri yerleştirin ve süpernatant kaldırın: yıkama arabellek ben, yıkama arabellek II, arabellek III yıkayın ve 2 x TE pH 8.0 anılan sıraya göre.
- Süpernatant atın ve ~ 5 dk'ya kuruması.
- Ters çapraz
- Örnekler mıknatıs kaldırın. 50 µL elüsyon arabellek ve protein-DNA komplekslerinde boncuk üzerinden elute için pipetting tarafından karıştırın.
- Bu adım üzerinden giriş sample(s) içerir. Elüsyon arabellek Sample(s) (arabellek oluşturma yonga örnekleri benzer tutmak için) 50 µL son hacmi için giriş eklemek ve proses yonga örnekleri ile birlikte.
- Mix 3 µL elüsyon arabellek ve 2 µL RNase (Dnaz ücretsiz). Her örnek için 5 µL Mix ekleyin ve 37 ° C'de 30 dk için kuluçkaya
- Mix 2.5 µL İndinavir K, 1 µL glikojen ve örnek başına 1,5 µL elüsyon arabellek. Her örnek (1 U İndinavir K ve örnek başına 20 µg glikojen) 5 µL Mix ekleyin ve 37 ° C'de 2 h için kuluçkaya
- 65 ° c gece (en az 4 h) örnekleri ters crosslinking gerçekleştirmek için sallayarak ile kuluçkaya.
- Tüpler en az 30 s ve transfer için mıknatıs yerleştirmek için yeni bir tüp süpernatant. Örnekleri-20 ° C'de 12 aya kadar donmuş olabilir.
- DNA arıtma
- İyi dağınık paramagnetic boncuk 140 µL örnek (2.3:1 oranı) 60 µL için ekleyin. Dikkatle iyice karıştırmak için 25 kez yukarı ve aşağı pipet. Sıvı her tüpün içinde homojen olduğundan emin olun. 2 dk. 4 dk için veya tüm boncuk olana mıknatıs üzerine boruları mıknatıs için bağlı ve süpernatant atmak bir yer için oda sıcaklığında kuluçkaya.
- Tüpler üzerinde mıknatıs bırakın ve taze hazırlanmış % 70 etanol 200 µL ekleyin. Tüpler 30 için kuluçkaya boncuk bozmadan s. Süpernatant atmak ve bir kez daha bu işlemi tekrarlayın. Etanol tamamen Aspire edin ve paramagnetic izin boncuk kuruması için 4 dak.
Not: Eksik etanol kaldırma ciddi DNA kurtarma azaltmak ve verim. Kuru kadar Pelet kuru. Aşırı pelet kurutma DNA kurtarma azaltmak ve verim. - Mıknatıs tüpler kaldırın ve 20 µL 10 mM Tris-HCl (pH 8.0) ekleyin. Yavaşça yukarı ve aşağı 25 kez iyice karıştırın tüm birimi pipette. Oda sıcaklığında 2 min için kuluçkaya. Tüpler geri 4 dk için mıknatıs yerleştirin ve süpernatant aktarmak için başka bir tüp.
- Uygun bir yer ile DNA miktarını ölçmek ( Tablo malzemelerigörmek).
- Çipi qPCR (100 µl H2O ve kullanım 2-5 µL qPCR için içinde seyreltik 1 µL) tarafından başarılı olduğunu doğrulayın. Belirli astar bir pozitif (protein ilgi bilinen bağlama sitesi) ve negatif kontrol (örneğin, sessiz ve/veya hedef değil protein ilgi bir gen) için kullanın.
Not: Kütüphane hazırlık 2 ile gerçekleştirilebilir çip DNA ng bağlı olarak Kiti ( Malzemeler tablo kullanmak için hangi kiti bir öneri için bakınız).
Sonuçlar
4 sU etiketleme: en iyi doğrulamak 4 sU etiketleme koşulları (apoptosis, nükleer stres, sitoplazmik stres), zaman ve konsantrasyon: 4sU yüksek düzeyde üretim ve işleme rRNA inhibe ve sitoplazmik yanı sıra nükleer stres30teşvik. Bu nedenle, faiz hücrelerinin 4sU kaynaklı stres gibi Apoptozis için test edilmelidir. Western blot analizi nükleer stres gösterir, p53 birikimi görüntülenmesi için önerilir sitoplazmik stres ve floresan aktif hücre (FACS) analiz apoptosis için sıralama görüntülemek fosfo-EIF2a düzeylerini artırma. Hücresel stres ikna etmek için yüksek düzeyde ve uzun pozlama 4sU veya thapsigargin veya arsenite gibi ilaçlar kullanılabilir. Apoptozis ya da hücre ölüm ikna etmek için hücreleri BH3I-1 ile tedavi edildi (500 ng/µL) veya inkübe 95 ° c (ısı şok) 5 min için. V/7-AAD boyama annexin apoptotik (Annexin V) ve (7-AAD) ölü hücreleri belirlemek için kullanıldı. Vitro etiketleme oluşturulan 500 µM 4sU ile 0,5 h için birincil Th1 hücre (son konsantrasyonu) veya 200 µM 4sU ile 1 saat de hücresel stres belirtileri indükler ne de apoptoz (şekil 1) ama yeterli 4sU birleşme için yol.
Zaman etiketleme RNA-da var kısaltılmış (≤5 min) göre artık bu etiketleme kez dizileri hangi intronic kısa ömürlü bir artış yol açar. Co-transcriptional dikişi oranları görselleştirmek için 4sU etiketleme kez 30 dk geçmemelidir. 4sUlabeling ile ilgili daha fazla bilgi için lütfen Rädle ve arkbakın. 19
Kalite kontrol: RNA bütünlük RNA işlenmesi sırasında büyük önem taşıyor. Electrophoretical analizi ile biotinylation sonra 4sU etiketli RNA RNA kalitesini kontrol etmek en uygun olan ( Tablo malzemelerigörmek). Özellikle için toplam RNA'ın sıralama kullanırken adım 2.2.2, izole RNA doğrulama göz önünde bulundurun. RNA bütünlük numarası (RIN) ≥8 (şekil 3) işleme daha fazla için RNA bütünlüğünü güvenceye almak için olmalıdır.
Electrophoretical analiz yeni transkripsiyonu RNA doğrulamak için de kullanılabilir. Yeni transkripsiyonu RNA toplam RNA çok daha az belirgin olan tipik rRNA bantları ile karşılaştırıldığında önemli ölçüde daha az olgun rRNA'lar içerdiğini unutmayın.
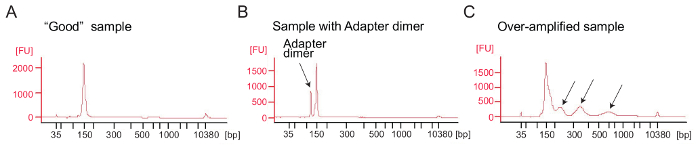
Ribozom profil oluşturma: PCR güçlendirme cDNA Kütüphanesi: cDNA amplifikasyon (Adım 4.9.1) iyi sıralama sonuçlar sağlamak için kritik bir adımdır. Güçlendirilmiş kitaplıkları tarafından electrophoretical analizi. Bir tepe etrafında iyi güçlendirilmiş kütüphanelerin gösterir 140-160 bp (şekil 4A). Aşırı miktarda bağdaştırıcısı dimer olmalıdır (şekil 4B) kaçınılması ve şu örnekleri daha fazla üreticinin iletişim kuralı (sayfa arıtma PCR ürünlerinin) göre sayfa arıtma yordamı kullanarak saf. Çok fazla şablon veya çok fazla PCR döngüleri beklenenden daha yüksek molekül ağırlıklı bantları, lekeli PCR ürünleri ve bağdaştırıcı dimer ürünleri (şekil 4 c) görünümünü tarafından karakterize aşırı amplifikasyon neden olabilir. Çoğu örnekleri için 1-5 µL olan circularized cDNA ve amplifikasyon 9 ÇSYİ devredir genellikle doğru PCR ürününün yeterli miktarda ortaya çıkarır.
Çip: Kromatin yamultma: Optimal kesme koşullar hücre türüne göre ayarlanması gerekir. Kesme koşulları (örneğin, döngü, yüksek veya düşük enerji sayısı) önceden belirleyin. Aynı sayıda hücre ve aynı birim için sınav amaç, daha düşük bir hücre yoğunluğu kesme verimliliği artırır bu yana kullanın. Bitti - veya altında-makaslama Kromatin önlemek için deneyin. Büyük Kromatin parçaları önemli ölçüde tıkanma tarafından ChIP sonuçları etkileyebilir ve aşırı yamultma epitopları için daha düşük bir bağlama verimliliği antikor tarafından önde gelen ilgi protein üzerinde yok edebilir. Yamultulmuş Kromatin ana kısmını yaklaşık 1.000 yaşındayken bu deneyde, en iyi sonuçları elde edildi kan basıncı ya da biraz daha düşük (şekil 5A).
Doğrulama ChIP qPCR tarafından: Çip başlamadan önce Eğer kullanılan antikor küçük parça için uygundur test etmek için tavsiye edilir (mümkünse, ChIP sınıf antikorlar kullanın) ChIP-qPCR tarafından. Kitaplık hazırlama başlamadan önce qPCR tarafından küçük parça için sıralama doğrulayın (bkz. Adım 5.6.5). Tasarım astar bir protein ilgi bilinen hedef siteye bağlayın. Tam hedef site içinde bir gen bilinmiyorsa, birkaç astar çiftleri gen ve ilişkili düzenleyici elemanlarının taramak için kullanılabilir. Ifng, RNAPII çip Th1 hücre için hangi transcriptionally upregulated üzerine stimülasyon ve aktin astar pozitif kontrol kullanılan. Bu genlerin Th1 hücre (şekil 5B) ifade değil beri Sox9 ve insülin bir negatif kontrol hizmet vermektedir. Normalde mRNA qPCR için kullanılan exon kapsayan astar kullanmayı unutmayın. Bir IgG denetimi kullanılan antikor özgüllük kanıtlamak için de kullanılabilir. Immunoprecipitated DNA ile uygun bir yer ölçülebilir ( Tablo malzemelerigörmek). Nonspecifically ilişkili DNA IgG denetim tarafından miktarda önemli ölçüde daha düşük faiz antikor tarafından bağlı DNA miktarının karşılaştırılması gereken.
Çoğaltır: biyolojik önemi kanıt: Kesinlikle aynı havuzun hücreleri tüm tedavi edilmezse ve tedavi örnekleri (Şekil 2) için aynı kimliğe sahip sağlamak için hücre başlayarak tüm yöntemleri için Kinetik deney gerçekleştirmek için önerilir. Yine de, bu küçük aliquots bir biyolojik Çoğalt (örneğin, qPCR, FACS analiz tarafından) örnekleri karşılaştırmak her yöntemi için ana zaman puan almak için tavsiye edilir. Bu her iki çoğaltır için tedavi tekrarlanabilir ve sıralama ile devam için kaba bir tahmin sağlar. Çoğaltır doğrulanmasını bioinformatical analiz yoluyla yapılmalıdır. Tekrarlanabilirlik sonuçlarının çoğaltır arasında FPKM değerleri arasındaki ilişki açısından değerlendirildi ve scatter araziler (şekil 6) kullanarak görüntülenir.
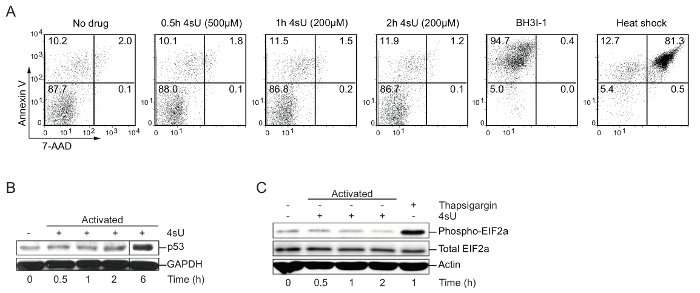
Resim 1: Doğrulama koşullardan hücre fizyolojisi perturbing olmadan optimum 4sU etiketleme (şekil--dan Davari ve ark. 11)
Hücre apoptosis FACS analizi tarafından tespiti(a): vitro üretilen hücreler tedavi (parantez içinde gösterilir) 4sU farklı konsantrasyonları ile 0,5 h, 1 h ve 2 h, Th0 anılan sıraya göre. BH3I-1 tedavi ısı şok (95 ° C'de 5 dk) 7-AAD tarafından belirlenen hücre ölümü ikna etmek için kullanıldı ise apoptosis Annexin V tarafından belirlenen ikna etmek için kullanıldı. (B) Western blot analizi 4sU p53 için tedavi ve T hücreleri aktive: örnekleri ile 200 µM 4sU harekete geçirmek, 500 µM 4sU ile etiketli 0,5 h saat noktası dışındaki belirtilen süre için etiketli. (C) Western blot analizi fosfo-EIF2a ve (B) olduğu gibi etiketleme aynı koşullarla aktif Th1 hücrelerdeki toplam EIF2a. Thapsigargin pozitif bir denetim olarak kullanıldı. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
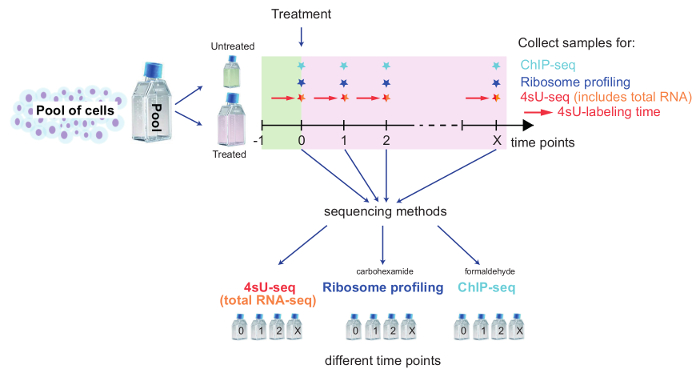
Resim 2: Genom genelindeki değişiklikleri izlemek için Kinetik bir kurulum şematik bakış
Bu düzen 4sU seq, toplam RNA-seq, ribozom profil oluşturma ve genom genelinde değişiklikler hücre tedavisi üzerine çalışmaya ChIP-seq birleştirmek için kurulum gösterilmektedir. Hücreleri havuz ve hücreleri tedavi edilmezse kontrolü için gerekli sayıda bir kenara koyun. Kalan hücreleri tedavi ve her yöntem ve saat noktası için bölünmüş. Tedavi edilmezse/tedavi hücreleri 4sU-seq açıklandığı gibi 4sU ile etiketleyin. Zaman puan ve her yöntem için örnekleri incelenmektedir belirli biyolojik soruda bağlıdır. Her zaman nokta ve yöntem örnekleri almak ve adanmış protokolünün bir parçası olarak izleyin. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
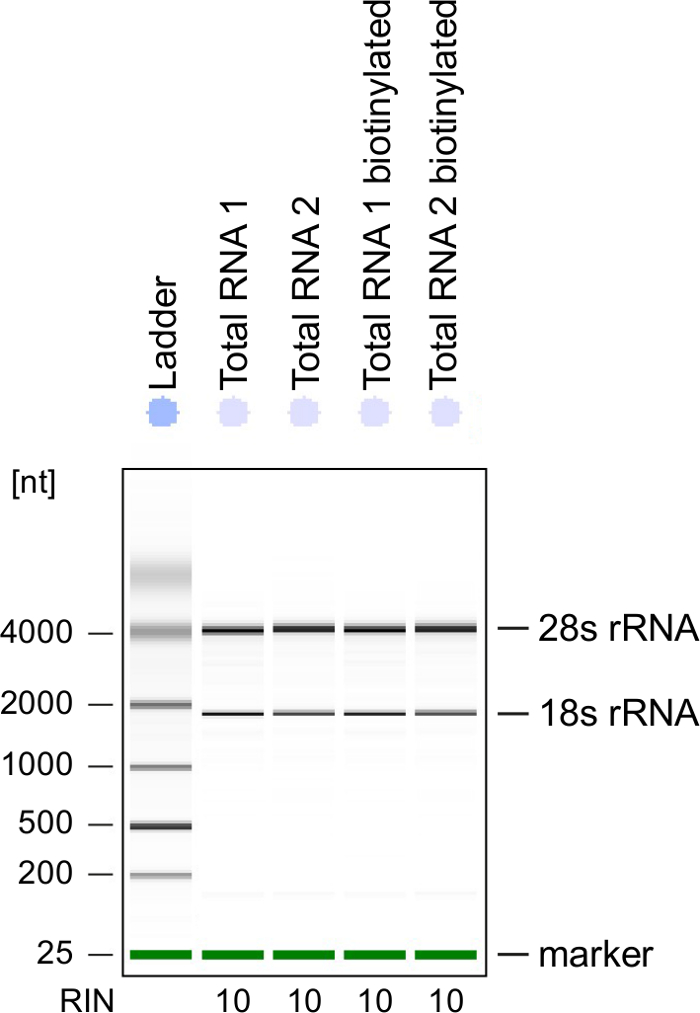
Şekil 3: kalite kontrol 4sU etiketli RNA'ın
Toplam RNA ve biotinylated RNA aktif Th1 hücrelerinden elde edilen bir Bioanalyzer üzerinde analiz edildi. 18s rRNA ve 28S rRNA gösterilir ve RNA bütünlük numarası (RIN) RNA bütünlüğünü belirlemek için aracı tarafından verilir. RIN ≥8 RNA bütünlüğünü güvenceye almak için olmalıdır. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Resim 4: Bioanalyzer profilleri ribozomların kitaplıkları profil oluşturma
(A) A iyi Kütüphane: örnek bir tepe beklenen boyutu aralığı (140-160 bp) gösterir ve başka hiçbir arıtma gereklidir. (B) Bu örnek gösterir aşırı bağdaştırıcısı dimer güçlendirilmiş ürün (120 bp) göreceli istenilen ürün (140-160 bp). Bu kütüphane daha fazla arıtma gerektirir. (C) aşırı güçlendirilmiş bir örnek: beklenenden daha yüksek molekül ağırlıklı tepeler ve lekeli PCR amplicons görünür (oklarla gösterilen). Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
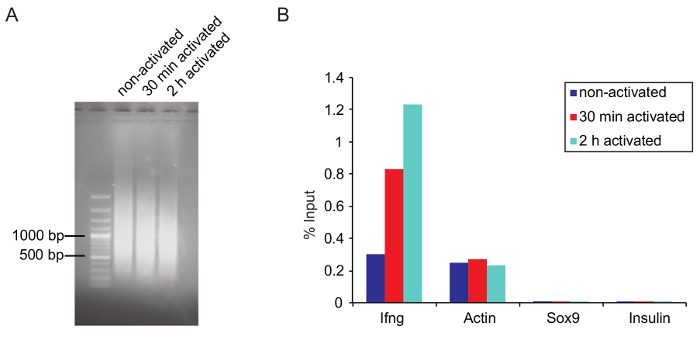
Şekil 5: Optimal Kromatin boyutu kesme ve çip doğrulama tarafından qPCR sonra
(A)özel jel resmi bir sonicator 25 döngüleri için sheared ve iletişim kuralında daha önce açıklandığı gibi saf üç örneklerinden yamultulmuş Kromatin en iyi parçası boyutunu gösterir. (B) toplam RNAPII küçük parça (anti-RNA Pol II, 8WG16, ab817) sonuçlarını Q-PCR giriş yüzdesi olarak temsil edilir. Sox9 ve insülin negatif denetimleri (hem genler aktive Th1 hücrelerde ifade edilir değil) iken Ifng ve aktin astar olumlu olarak kullanılmıştır. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
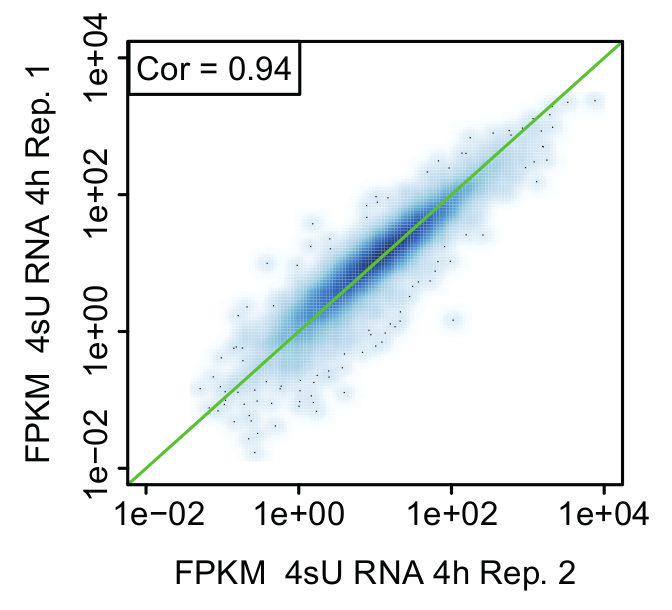
Şekil 6: Biyolojik karşılaştırılması (Davari ve ark. rakam çoğaltır. 11)
İfade değerleri (FPKM) arasında karşılaştırma temsilcisi dağılım çizim yeni kopya etmek (4sU) RNA 4 h sonra aktif Th1 hücre stimülasyon çoğaltır. Yeşil hat eşit FPKM değerleri gösterir ve sıralama korelasyon her mezarlığına belirtilir.
| Süresi (dk) etiketleme | Önerilen 4sU konsantrasyonu (µM) |
| 120 | 100 - 200 |
| 60 | 200 - 500 |
| 15 - 30 | 500 - 1000 |
| < 10 | 500 - 2000 |
Tablo 1: 4sU konsantrasyonları önerilir (Rädle, üzerinden ve ark. 19)
Önerilen 4sU konsantrasyonları çeşitli maddelerinin etiketlenmesi farklı zamanlarda belirtilir.
| Yöntemi | Cep numarası | RNA miktarı |
| 4sU etiketleme | ≥2 10 x7 | ≥60µg |
| Ribozom profil oluşturma | ≥2 x107 | |
| Çip seq | ≥2 10 x7 - 3 x 107 |
Tablo 2: Gerekli miktarda birincil T hücreleri
Her yöntem için gerekli birincil T hücrelerinin minimum tutar. Miktarları az diğer hücre tipleri kullanırken olabilir.
Tartışmalar
Gen düzenlemesi tüm süreç analiz tamamen hücresel adaptasyon belirli uyarıcı veya tedaviye yanıt olarak anlamak gereklidir. Toplam RNA-seq birleştirerek, 4sU seq, profil oluşturma ribozom ChIP-seq farklı zaman noktalarda bizi bir gen düzenlemesi zaman içinde ana süreçlerin kapsamlı bir analiz için. Biyolojik süreçlerin derin bir anlayış deneysel Kur hem de uygun zaman noktaları tanımlamak için gereklidir.
Gen düzenlemesi çalışma yöntemleri hızla geliştirmek beri bu iletişim kuralları hızlı değişikliklere adapte edilebilir. Yine de, onlar temel gen düzenleyici mekanizmaları hücre her türlü içinde çalışmak için en önemli bir yöntem sağlar. Burada, bazı tuzaklar ve bir bu yöntemleri kullanırken göz önünde bulundurun zorunda gerçekleri tartışmak.
Hücreleri: Hücrelerin son derece uygun olması gerekir ve birincil izole hücreler kullanıyorsanız, hücre popülasyonlarının saflığı (örneğin, FACS analiz için birincil T hücreleri) garanti gerekir. Hatta biraz stresli hücreleri bu çok duyarlı sıralama yöntemleri sonuçlarını etkilemek ve yeni kopya etmek veya çevrilmiş RNA miktarı düşük ve istenmeyen sonuçlar sıralama sonuçlarında stres yanıtının neden. Bu protokol için hücreleri cips belirtilen aralıklarla hız birincil T hücreleri için optimize edilmiştir. Böylece, hücre tipine göre hızı ayarlayın.
4 hücre fizyolojisi üzerinde sU etkileri: Özellikle hücre sayıları sınırlı olan hücresel fizyoloji üzerine 4sU ek için en az pertürbasyon doğrulama yukarıda belirtilen seçenekleri ek olarak, daha fazla ve/veya ek analiz, gerçekleştirilebilir. Hücre çoğalması üzerine etkileri sadece etiketli ve etiketsiz hücreleri sayarak katlama zaman hücre doğrulama test edilebilir. Genelde stres indüksiyon Ayrıca hücre morfolojisi ayirt nucleolin ve hücre çekirdeği boyama ile analiz ederek test olabilir. 4sU etkisini daha da doğrulamak için değiştirilmiş küresel gen ekspresyonu sayar etiketli toplam RNA okumak için etiketlenmemiş toplam RNA birleştiriliyor tarafından ölçülen.
Cep numaraları: Oluşturulan vitro T hücreler için en az miktarını tablo 2' de belirtilen hücreleri ile başlayan öneririz. Hücre tipine göre yöntem başına yeterli numara seçin. T hücreleri daha az sitoplazma ve diğer hücrelere göre RNA beri daha düşük miktarlarda büyük olasılıkla diğer hücreleri yeterli olacaktır. ChIP-seq için hücre sayıları son derece kullanılan antikor ve faiz hücrelerindeki protein ifade düzeyine göre değişir. Hücre sayıları transkripsiyon faktörleri kullanıldığında, özellikle düşük seviyelerde ifade edilir eğer artırılması gerekiyor ise Histon veya RNAPII çip için fewe hücreleri, kullanılabilir.
4 sU etiketleme ve RNA biotinylation: Yapışık hücreleri kullanırken 4sU etiketleme Rädle ve arktarafından açıklandığı gibi yönlendirilebilir. 19 hücreleri çok hızlı bir şekilde dahil 4sU beri bu süspansiyon, yapışık veya yarı yapışık hücreleri orta doğrudan eklenebilir.
Bu biotinylation RNA'ın 60-80 µg ile başlatmak için tavsiye edilir. Yine de, RNA düşük miktarda kullanılabilir, her ne kadar biz did değil sınav için daha az 30 µg. ekleyin (örneğin, GlycoBlue) bir coprecipitant ne zaman Pelet görmek zor ise RNA presipite. Duffy ve ark. da o methylthiosulfonate aktif biotin (MTS-biotin) daha verimli bir şekilde 4sU etiketli RNA ile HPDP-biotin31tepki verir göstermiştir. Bu nedenle, bu MTS-biotin, özellikle, daha az üridin artıkları (Duffy vd.; bkz: Arıtma 4sU etiketli RNA tarafından bahsedilen biotinylation iletişim kuralı aşağıdaki anlamlara sahip olma eğilimi, küçük RNA'ları Kurtarma için geçiş dikkate değer olabilir Deneysel yordamlar).
Yeni transkripsiyonu RNA kurtarma için paramagnetic boncuk veya RNA Temizleme boncuk seçtiğiniz kullanmak mümkündür. Her zaman bu kitleri ya da değil arındırmak için belirli RNA'ların dikkate almak. MiRNAs içinde ilgileniyorsanız, örneğin, belirli kitleri miRNA yakalama ve sıralama için kullanmayı düşünün.
Yeni transkripsiyonu RNA'ın miktar: Doğru yeni transkripsiyonu RNA ölçmek için uygun bir yer tarafından ölçüm yapılmalıdır ( Tablo malzemelerigörmek). 4sU maruz kalma 1 saat içinde yeni transkripsiyonu RNA toplam RNA'ın yaklaşık % 1-4 temsil eder. Yeni transkripsiyonu RNA 1 etiketli h aktive T hücreleri oluşur rRNA%11~ 90-94.
Ribozom profil oluşturma: Yöntem kurarken, özgün protokolünde önerilen 1.5 x nükleaz miktarda kullanarak doğru sindirim garanti belirledi. Ayrıca, herhangi bir yan etkisi nükleaz yüksek miktarda bildirilmiştir. Ribozomal proteinler tarafından bağlı RNA parçası oldukları sırada RPFs overdigest oldukça zor olduğundan, hala biraz en iyi nükleaz sindirim titre tutarı artırabilir.
Eğer daha az 500 ng RPF RNA'ın adım 4.4.2 kurtarıldı, rRNA tükenmesi ve havuzu saf RPFs RNA temiz & yoğunlaştırıcı-5 sütunları ile yineleyin. Alternatif olarak, iki aynı örnek üzerinde jel (Adım 4.5.3) birbirine yakın ve havuzu jel dilimleri yük sırasında RNA elüsyon jel (Adım 4.5.6) üzerinden.
Biz RPFs üzerinde bir jel mümkün olduğunca sıkı bir şekilde 28 ve 30 nt grup kesme öneririz. Bu istenmeyen parçaları rRNA'lar ve tRNA, hangi daha sonra kitaplığınıza parçası haline gelir ve sıralama okuma senin RPFs için azaltmak giderilmesinde yardımcı olur.
Ayrıca UV jel arıtma sırasında ışığı önlemek için önerilir. Bu RNA parçaları, aynı zamanda da sonunda ciddi Kütüphane hazırlanması ve sıralama sonuçları etkileyen pirimidin dimer nickler oluşturabilirsiniz.
Kütüphane hazırlama ve veri sıralama: Sıralama için uygun bir cDNA Kütüphanesi oluşturmak için ribozom Protokolü profil oluşturma sağlar. 4sU etiketleme tarafından oluşturulan örnek doğrudan herhangi bir uygun RNA sıralama kiti ile kütüphane hazırlık için kullanabilirsiniz. Özellikle kısa kullanarak kez, etiketleme henüz poliadenile, olmayabilir zaman yeni transkripsiyonu RNA beri hayır Poli-bir seçim yapılmalıdır. Bunun yerine, rRNA tükenmesi önlemek için önerilir gerçek örnek için sıralama derinliği azaltılması. T hücreleri kullanarak, biz 400 gerçekleştirilen ng (bağlı olarak kit, bkz: belgeleri), yeni kopya etmek ve toplam RNA'ın rRNA tükenmesi ile başladı ve çember PCR güçlendirme PCR önyargı en aza indirmek daha düşük. Kütüphane hazırlık başlangıç daha az malzeme ile gerçekleştirilebilir. PCR döngüleri Kütüphane karmaşıklık numaralarındaki hesap için optimize edilmelidir.
ChIP-seq için orada da birçok kitleri Kütüphane hazırlık için kullanılabilir. Bizim eller kitaplığında hazırlık de 2'den başlayarak çalıştı ng çip DNA'ın (malzeme kullanmak için hangi kiti bir öneri için bakınız). Sıralama sırasında Endeksi için renk dengesini kontrol ettiğinizden emin olun. Bir sıralama derinlik x 106 okuma 4sU seq, toplam RNA-seq ve ChIP-seq örnekleri için her ≥40 ve ≥80 x 106 okuma ribozom örnekleri profil oluşturma için tavsiye ediyoruz. Sıralama derinlik örnek ve aşağı akım Biyoinformatik analizi bağlıdır ve dikkatle düşünülmelidir. Cotranscriptional boşluklarına ayıran, 100 intronic okuma analiz etmek için bp eşleştirilmiş uç sıralama seçilmiş gerekir.
Önyargı sıralama: Sıralama transkripsiyon, çevirisi veya transkripsiyon faktörü bağlama küresel değişimler belirlerken altın standart haline gelmiştir. Son yıllarda içinde varolan yöntemleri için sınırları itilmiş veya RNA başlangıç giderek daha az miktarda sıralama için yeni teknikler geliştirilmiştir. Bu gürültü veya önyargı tanıttı cDNA amplifikasyon gerektirir. Son zamanlarda, benzersiz moleküler tanımlayıcıları (UMIs) deneysel olarak PCR tarafından tanıtılan çoğaltmaları tanımlamak için geliştirilmiştir. Son zamanlarda, bu UMIs sadece hafif sıralama güç ve yanlış bulma oranı fark gen ifade32için geliştirmek gösterilmiştir. Yine de, tüm sıralama Denetim Kitaplığı karmaşıklığı için kitaplıklarına için benzersiz moleküler tanımlayıcılarını (UMIs) kullanarak özellikle RNA düşük miktarda başlatırken ve birçok PCR döngüleri gerektiğinde düşünün.
Arabellek ve hisse senedi çözümleri: 4sU seq ve ribozom profil oluşturma için tüm arabellekleri nükleaz ücretsiz su kullanarak sıkı RNase free koşullar altında hazırlanmalıdır. Bu önceden yapılmış nükleaz ücretsiz NaCl, Tris-HCl, EDTA, sodyum sitrat ve su satın almak için tavsiye edilir. Nükleaz ücretsiz koşulları sağlamak için RNase decontaminating çözümünü Pipetler veya yüzeyleri temizlemek için kullanılabilir. ChIP-seq için tüm arabellekleri gerekir Dnaz boş en az ve oda sıcaklığında depolanabilir. Her zaman proteaz inhibitörleri ve isteğe bağlı olarak, fosfataz inhibitörleri kullanmadan önce ekleyin ve buz üzerinde tutun.
Biyoinformatik: Tüm sıralama veri (Yani, ChIP-seq, RNA-seq ve ribsosome profil oluşturma) analizini içerir Kalite kontrol (FastQC, kullanarakörneğin, http://www.bioinformatics.babraham.ac.uk/projects/fastqc/), bağdaştırıcı kesme (örneğin, ile cutadapt20) başvuru genom hücrelerin altında eğitim için eşleme tarafından takip. RNA-seq veri (Toplam ve 4sU seq) yanı sıra için veri profil oluşturma ribozom, spliced RNA-seq mapper, ContextMap 221gibi gereklidir. ChIP-seq unspliced veri hizalamaları için yeterli BWA-MEM22 kullanmaktır. Gen ifadesinin RPKM modeli (okuma kilobaz Exon milyon parçaları eşlenen başına başına)1, sayıları kullanarak bir program, örneğin featureCounts23gen başına okumak belirledikten sonra kullanılarak hesaplanabilir. ChIP-seq verilerden en yüksek arama için bir dizi program are elde edilebilir, örneğin, MAC'ler24 veya mücevher25. Daha da aşağı akım analizleri, R26özellikle Bioconductor proje27tarafından sağlanan araçlar kullanılarak, gerçekleştirilebilir.
Burada, normalleştirme 4sU ve toplam RNA düzeyleri ve ribozom profil oluşturma üzerinden translasyonel aktivite entegre büyük bir sorun olduğunu. Bu sorunu çözmek için bir klasik ev tutma genler düzeylere normalize etmek yaklaşımdır. Gürültü bireysel ev tutma genler için rastgele dalgalanmalar nedeniyle azaltmak için bu sadece bir kaç ev tutma genler medyan düzeyleri daha büyük bir küme için örneğin kullanılması önerilir > Eisenberg ve Levanon28 tarafından derlenmiş 3.000 ev tutma genler . RNA ciro oranları toplam RNA, normalleştirme medyan ciro dayanmaktadır oranları 4sU-e üzerinden hesaplanması için (5 h bir RNA half-life varsayarakörneğin,)29oranları. Bu ev tutma genler için genel değişiklik varsayar beri Ancak, çözümleme yaklaşımlar normalleştirme, örneğin, korelasyon tabanlı bir zaman-dizi gen grupları tanımlamak için farklı veri türleri kümeleme bağımsız kullanmanızı öneririz Transkripsiyon ve translasyon etkinleştirme sırasında farklı davranış ile. Bioinformatical tümleştirilmesi farklı veri türleri hakkında ayrıntılı bir açıklama için özgün yayın11' e bakın.
Ciro oranları ve veri tümleştirme analizi: Yarı-hayat bir çoğaltılmış gen denetim (MGC) tarafından belirlenen genel yöntemleri için karşılaştırma bir kısa bir süre önce kağıt33 yarı-hayat bu metabolik etiketleme yöntemleri (örneğin, diğer yöntemlerine göre elde edilen en iyi korelasyon gösterir misiniz Genel inhibisyonu transkripsiyon uyuşturucu tarafından). Ancak, half-life hesaplamalar arasındaki farklar ortaya çıkabilir ve açıklanan 15,34olmuştur belirtilmelidir. Sorunlar ve uzun süreli 4sU maruz kalma nedeniyle stres tepkisi tarafından tanıtıldı farklılıklar çoğu için hesap. Bu nedenle, 4sU etiketleme tarafından tanıtılan stres tepkisi dışlamak için vazgeçilmezdir. Daha fazla ciro oranları doğrulamak için MGCs kullanmanızı tavsiye ederiz.
Ayrıca, burada oluşturulan bir veri kümesi de bir daha bütünleştirici veri analizi (örneğin, uzun kodlamayan RNA'ların Yönetmeliği)35,36için kullanılabilir.
Açıklamalar
Yazarlar onlar rakip hiçbir mali çıkarları var bildirin.
Teşekkürler
Lars 4sU birincil T hücreleri için etiketleme kurmak için Dölken tavsiye için teşekkür ederiz; Elisabeth Graf ve Thomas Schwarzmayr Kütüphane nesiller ve sıralamanın önemli yardım için; Eick ve Andrew Flatley RNAPII ve T hücre antikorları sağlamak için dirk; N. Henriette Uhlenhaut ve Franziska Greulich ChIP-seq Kütüphane hazırlığında yardım için; Caroline C. Friedel hibe FR2938/7-1 ve CRC 1123 (Z2) Deutsche Forschungsgemeinschaft (DFG) üzerinden tarafından desteklenen; Elke Glasmacher hibe GL 870/1-1 Deutsche Forschungsgemeinschaft (DFG) ve Alman merkezi için diyabet araştırma (DZD), Helmholtz Zentrum München tarafından desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 4sU-labeling | |||
| 4-thiouridine (100 mg) | Carbosynth | 13957-31-8 | Prepare 50 mM stock in sterile H2O/PBS; store at –20°C in aliquots of 50-500 µl; do not refreeze. |
| 1.5 ml safe-lock tubes | Eppendorf | 30121589 | Optional |
| 1.5 ml screw-top polypropylene tubes | Sarstedt | 72692005 | Compatible with Dimethylformamide |
| 15 ml tubes | BD Falcon | 352096 | Compatible with Dimethylformamide |
| 2.0 ml screw-top polypropylene tubes | Sarstedt | 72694005 | Compatible with Dimethylformamide |
| 50 ml tubes | BD Falcon | 352070 | Compatible with Dimethylformamide |
| Agencourt RNAClean XP | Beckman Coulter | A63987 | We recommend to use these paramagnetic beads. Aliquot and store at 4°C |
| Chloroform | Sigma Aldirch | 372978 | WARNING – HAZARDOUS TO HEALTH |
| Dimethylformamide | Sigma Aldrich | D4551 | |
| Dithiothreitol (DTT) | Roth | 6908.1 | Prepare as 100 mM DTT in nuclease-free H2O; always prepare fresh |
| Ethanol | Merck | 1.00983.1000 | |
| EZ-Link Biotin-HPDP (50 mg) | Pierce | 21341 | Prepare 1 mg/ml stock solution by dissolving 50 mg Biotin-HPDP in 50 ml DMF. Gentle warming enhances solubilisation. Store at 4°C in aliquots of 1 ml. DMF dissolves some plastic materials. We recommend to use glass pipettes to transfer DMF from ist stock glass bottle to 50 ml Falcon tubes. |
| High Sensitivity DNA Kit | Agilent Technologies | 5067-4626 | |
| Isopropanol | Merck | 1.09634.1011 | |
| NaCl (5M) | Sigma Aldrich | 71386 | Stock solution |
| nuclease-free EDTA (500 mM ), pH 8.0 | Invitrogen | 15575-020 | Stock solution |
| Nuclease-free H2O | Sigma Aldrich | W4502 | Stock solution |
| nuclease-free Tris Cl (1M), pH 7.4 | Lonza | 51237 | Stock solution |
| Phase Lock Gel Heavy tubes (2.0 ml) | 5Prime | 2302830 | Use in step 1.3.4. |
| Polypropylene 15 ml centrifuge tubes | Greiner Bio-One | 188271 | Or equivalent; they have to tolerate up to 15,000 × g |
| QIAzol Lysis Reagent (200 ml) | Qiagen | 79306 | Use this or equivalent TRI reagent for RNA isolation, WARNING – CORROSIVE and HAZARDOUS TO HEALTH! Ensure immediate access to Phenol antidote (PEG-Methanol) |
| Qubit RNA HS assay kit | Life Technologies | Q32852 | Use this kit for quantifying RNA quantity in step 1.4.11 |
| RNeasy MinElute Kit | Qiagen | 74204 | Optional; includes Buffer RLT |
| Sodium citrat | Sigma Aldrich | C8532 | Prepare 1.6 M stock solution using nuclease-free water |
| Tween 20 | Sigma Aldrich | P1379 | |
| µMacs Streptavidin Kit | Miltenyi | 130-074-101 | Store at 4°C, includes µMacs columns used in step 1.4.6. (store at RT) |
| Cell viability and stress assay | |||
| PE Annexin V Apoptosis Detection Kit I | BD Biosciences | 559763 | Optional |
| Thapsigargin | Sigma-Aldrich | T9033 | Optional |
| p53 | abcam | ab26 | Optional |
| p-EIF2a (Ser51) | Cell Signaling | 9721 | Optional |
| BH3I-1 | Sigma-Aldrich | B 8809 | Optional |
| Buffers | |||
| 4sU Washing Buffer | store at RT | 100 mM Tris pH 7.4, 10 mM EDTA, 1 M NaCl, 0.1% Tween 20 in nuclease-free H2O | |
| Biotinylation Buffer (10x) | store at 4 °C | 100 mM Tris pH 7.4, 10 mM EDTA in nuclease-free water; make aliquots of 1 ml; store at 4°C | |
| RNA precipitation buffer | store at RT | 1.2 M NaCl, 0.8 M sodium citrate in nuclease free water. Prepare in advance under nuclease-free conditions. Store at room temperature in 50 ml falcon tubes. | |
| Equipment | |||
| 2100 Bioanalyzer instrument | Agilent | G2939BA | |
| RNA 6000 Nano Kit | Agilent | 5067-1511 | Use this kit to verify RNA integrity in step 1.3.10 |
| RNA 6000 Pico Kit | Agilent | 5067-1513 | Optional |
| UV/VIS spectrophotometer | Thermo Scientific | NanoDrop 1000 | Or equivalent. Use in step 1.2.2/3.1.8/3.3.3/3.4.3 |
| High-speed centrifuge | Thermo Scientific | Heraeus Multifuge X3R | Or equivalent equipment capable of reaching 13,000 × g |
| High-speed rotor | Thermo Scientific | Fiberlite F15-6 x 100y | |
| Adaptors for 15 ml tubes | |||
| Refrigerated table-top centrifuge | Eppendorf | 5430 R | Or equivalent. |
| Thermomixer | Eppendorf | Thermomixer C | Or equivalent. |
| Magnetic stand | Miltenyi Biotec | 130-042-109 | One stand holds 8 µMacs columns. |
| Ultra-fine scale | Mettler Toledo | ML204T | Or equivalent. |
| E-Gel iBase Power System | Invitrogen | G6400UK | For RNA gels; or equivalent. |
| E-Gel EX 1% agarose precast gels | Invitrogen | G4020-01 | For RNA gels; or equivalent. |
| DynaMag-2 Magnet-1 each | Life Technologies | 12321D | |
| RNaseZap | Sigma | R2020 | Optional |
| TruSeq stranded total RNA library prep kit | Illumina | RS-122-2201 | Or equivalent. For T cells we used 400 ng 4sU and Total RNA with 11 cycles for PCR amplification. rRNA depletion is included in this kit |
| Nanodrop | Thermo Scientific | use a Nanodrop or equivalent instrument to measure RNA concentration | |
| Ribosome Profiling | |||
| TruSeq Ribo Profile kit (Mammalian or Yeast) | Illumina | RPYSC12116 (Yeast) | |
| TruSeq Ribo Profile kit (Mammalian or Yeast) | Illumina | RPHMR12126 (Mammalian) | |
| Illustra MicroSpin S-400 HR Columns | GE Healthcare | 27-5140-01 | |
| RNA Clean & Concentrator-25 kit | Zymo Research | R1017 | |
| RNA Clean & Concentrator-5 kit | Zymo Research | R1015 | |
| Ribo-Zero Gold rRNA Removal Kit (Human/Mouse/Rat) | Illumina | MRZG126 or MRZG12324 | |
| (High Sensitivity DNA Kit) | Agilent Technologies | 5067-4626 | Already needed for 4sU-seq |
| All other consumables and equipment are listed in the User guide | !!! | Carefully read the user guide and order required consumables in advance (consider a long delivery time for some consumables e.g. gels) | |
| ChIP | |||
| 10 mM Tris-HCl (pH 8.0) | gereral lab supplier | ||
| 100 bp Plus Marker | Thermo Fisher | SM0323 | |
| 16 % Formaldehyde | Thermo Fisher | 28908 | Add to a final concentration of 1 % |
| 70% EtOH | gereral lab supplier | Always prepare fresh | |
| Agarose | gereral lab supplier | ||
| Agencourt RNAClean XP beads | Beckman Coulter | A63987 | We recommend to use these paramagnetic beads. Aliquot and store at 4°C |
| ChIP library preparation kit | KapaBiosystems | KK8504 | Or use the kit of your choice |
| DNA low bind microcentrifuge tubes | Eppendorf | Z666548-250EA | or equivalent |
| Dynabeads Protein G | Invitrogen | 10004D | Use these superparamagnetic beads coupled to protein G in step 4.3.1.; Bring to RT before use |
| Glycine | gereral lab supplier | Prepare a 2M stock solution | |
| Glycogen | Roche | 10-901-393-001 | |
| MinElute PCR Purification Kit | Qiagen | 28004 | Use this kit (or equivalent) to purify chromatin in step 4.2.4. |
| Phosphatase Inhibitor (PhosStop) | Roche | 4906837001 | Add freshly to the buffer and keep on ice |
| Power SYBRgreen Master mix | Thermo Fisher | 4367659 | |
| Protease Inhibitor (cOmplete, EDTA-free) | Roche | 11873580001 | Add freshly to the buffer and keep on ice |
| Proteinase K | Invitrogen | 25530049 | |
| Qubit dsDNA HS Assay kit | Invitrogen | Q32851 | Use this kit for quantifying DNA quantity in step 4.6.4. on a Qubit Fluorometer |
| Rnase, DNase free | Roche | 11-119915001 | |
| Salmon sperm (sonicated to around 100bp) | Sigma | D1626 | |
| TE pH 8.0 | gereral lab supplier | ||
| Antibodies (ChIP grade if possible) | |||
| anti-RNA Pol II [8WG16] | abcam | ab817 | |
| anti-Histon H3K36me3 | abcam | ab9050 | |
| or antibody of interest | |||
| Buffers | |||
| Binding/Blocking buffer | Store at RT | PBS with 0.5 % BSA and 0.5 % Tween 20 | |
| Cell-Lysis buffer | Store at RT | 5 mM Pipes [pH 8.0], 85 mM KCl, and 0.5 % NP40 | |
| ChIP IP buffer | Store at RT | 0.01 % SDS; 1. 1% Triton X-100;1.2 mM EDTA; 16.7 mM Tris-HCl, pH 8.1; 16.7 mM NaCl | |
| Elution buffer | Store at RT up to 6 months | 10 mM Tris-HCl (pH 8.0), 5 mM EDTA (pH 8.0), 300 mM NaCl and 0.5 % SDS | |
| Nuclei-Lysis buffer | Store at RT | 50 mM Tris [pH 8.0], 10 mM EDTA, and 1 % SDS | |
| Wash buffer I | Store at RT | 0.1 % SDS; 1 % Triton X-100; 2 mM EDTA; 20mM Tris-HCL pH 8.1; 150 mM NaCl | |
| Wash buffer II | Store at RT | 0.1 % SDS; 1 % Triton X-100; 2 mM EDTA; 20 mM Tris-HCL pH 8.1; 500 mM NaCl | |
| Wash buffer III | Store at RT | 0.25 M LiCl; 1% NP-40; 1 mM EDTA; 10 mM Tris-HCl, pH 8.1 | |
| Equipment | |||
| 2100 Bioanalyzer instrument | Agilent | G2939BA | use this instrument for electrophoretical analysis |
| Nanodrop | Thermo Scientific | ||
| Bioruptor TBX microtubes 1.5 ml | Diagenode | C30010010 | |
| or tubes special for your sonication device | |||
| Bioruptor sonication device or sonication device of your choice | Sonication of T cells with Bioruptor: 20 - 25 cycles (30 s on, 30 s off at high in two 1.5 ml bioruptor microtubes with 500 µl each tube) | ||
| Magnetic stand for tubes | |||
| Thermomixer | |||
| Agarose gel electrophoresis | |||
| Qubit Fluorometer | Thermo Scientific | Use this Fluorometer for quantifying low amounts of RNA/DNA |
Referanslar
- Mortazavi, A., Williams, B. A., McCue, K., Schaeffer, L., Wold, B. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nat Methods. 5 (7), 621-628 (2008).
- Chavez, S., Garcia-Martinez, J., Delgado-Ramos, L., Perez-Ortin, J. E. The importance of controlling mRNA turnover during cell proliferation. Curr Genet. 62 (4), 701-710 (2016).
- Rutkowski, A. J., et al. Widespread disruption of host transcription termination in HSV-1 infection. Nat Commun. 6, 7126 (2015).
- Ozsolak, F., Milos, P. M. RNA sequencing: advances, challenges and opportunities. Nat Rev Genet. 12 (2), 87-98 (2011).
- Carninci, P., et al. Genome-wide analysis of mammalian promoter architecture and evolution. Nat Genet. 38 (6), 626-635 (2006).
- Churchman, L. S., Weissman, J. S. Nascent transcript sequencing visualizes transcription at nucleotide resolution. Nature. 469 (7330), 368-373 (2011).
- Core, L. J., Waterfall, J. J., Lis, J. T. Nascent RNA sequencing reveals widespread pausing and divergent initiation at human promoters. Science. 322 (5909), 1845-1848 (2008).
- Hah, N., et al. A rapid, extensive, and transient transcriptional response to estrogen signaling in breast cancer cells. Cell. 145 (4), 622-634 (2011).
- Dolken, L., et al. High-resolution gene expression profiling for simultaneous kinetic parameter analysis of RNA synthesis and decay. RNA. 14 (9), 1959-1972 (2008).
- Paulsen, M. T., et al. Use of Bru-Seq and BruChase-Seq for genome-wide assessment of the synthesis and stability of RNA. Methods. 67 (1), 45-54 (2014).
- Davari, K., et al. Rapid Genome-wide Recruitment of RNA Polymerase II Drives Transcription, Splicing, and Translation Events during T Cell Responses. Cell Rep. 19 (3), 643-654 (2017).
- Windhager, L., et al. Ultrashort and progressive 4sU-tagging reveals key characteristics of RNA processing at nucleotide resolution. Genome Res. 22 (10), 2031-2042 (2012).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10 (10), 669-680 (2009).
- Blecher-Gonen, R., Barnett-Itzhaki, Z., Jaitin, D., Amann-Zalcenstein, D., Lara-Astiaso, D., Amit, I. High-throughput chromatin immunoprecipitation for genome-wide mapping of in vivo protein-DNA interactions and epigenomic states. Nat Protoc. 8 (3), 539-554 (2013).
- Schwanhausser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Larsson, O., Tian, B., Sonenberg, N. Toward a genome-wide landscape of translational control. Cold Spring Harb Perspect Biol. 5 (1), a012302 (2013).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nat Rev Mol Cell Biol. 16 (11), 651-664 (2015).
- Hafner, M., et al. Transcriptome-wide identification of RNA-binding protein and microRNA target sites by PAR-CLIP. Cell. 141 (1), 129-141 (2010).
- Radle, B., Rutkowski, A. J., Ruzsics, Z., Friedel, C. C., Koszinowski, U. H., Dolken, L. Metabolic labeling of newly transcribed RNA for high resolution gene expression profiling of RNA synthesis, processing and decay in cell culture. J Vis Exp. (78), (2013).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- Bonfert, T., Kirner, E., Csaba, G., Zimmer, R., Friedel, C. C. ContextMap 2: fast and accurate context-based RNA-seq mapping. BMC Bioinformatics. 16, 122 (2015).
- Li, H., Durbin, R. Fast and accurate long-read alignment with Burrows-Wheeler transform. Bioinformatics. 26 (5), 589-595 (2010).
- Liao, Y., Smyth, G. K., Shi, W. featureCounts: an efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2014).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biol. 9 (9), R137 (2008).
- Guo, Y., Mahony, S., Gifford, D. K. High resolution genome wide binding event finding and motif discovery reveals transcription factor spatial binding constraints. PLoS Comput Biol. 8 (8), e1002638 (2012).
- Team, R. D. C. . R: A Language and Environment for Statistical Computing. , (2016).
- Huber, W., et al. Orchestrating high-throughput genomic analysis with Bioconductor. Nature Methods. 12 (2), 115-121 (2015).
- Eisenberg, E., Levanon, E. Y. Human housekeeping genes, revisited. Trends Genet. 29 (10), 569-574 (2013).
- Friedel, C. C., Dolken, L. Metabolic tagging and purification of nascent RNA: implications for transcriptomics. Mol Biosyst. 5 (11), 1271-1278 (2009).
- Burger, K., et al. 4-thiouridine inhibits rRNA synthesis and causes a nucleolar stress response. RNA Biol. 10 (10), 1623-1630 (2013).
- Duffy, E. E., Rutenberg-Schoenberg, M., Stark, C. D., Kitchen, R. R., Gerstein, M. B., Simon, M. D. Tracking Distinct RNA Populations Using Efficient and Reversible Covalent Chemistry. Mol Cell. 59 (5), 858-866 (2015).
- Parekh, S., Ziegenhain, C., Vieth, B., Enard, W., Hellmann, I. The impact of amplification on differential expression analyses by RNA-seq. Sci Rep. 6, 25533 (2016).
- Baudrimont, A., et al. Multiplexed gene control reveals rapid mRNA turnover. Sci Adv. 3 (7), e1700006 (2017).
- Rabani, M., et al. Metabolic labeling of RNA uncovers principles of RNA production and degradation dynamics in mammalian cells. Nat Biotechnol. 29 (5), 436-442 (2011).
- Schlackow, M., Nojima, T., Gomes, T., Dhir, A., Carmo-Fonseca, M., Proudfoot, N. J. Distinctive Patterns of Transcription and RNA Processing for Human lincRNAs. Mol Cell. 65 (1), 25-38 (2017).
- Mukherjee, N., Calviello, L., Hirsekorn, A., de Pretis, S., Pelizzola, M., Ohler, U. Integrative classification of human coding and noncoding genes through RNA metabolism profiles. Nat Struct Mol Biol. 24 (1), 86-96 (2017).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır