Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
4 Boyutlu (x, y, z ve φ) Hiperspektral FRET Görüntüleme ve Analizi Kullanılarak Canlı Hücrelerde 3 Boyutlu cAMP Dağılımlarının Ölçümü
Bu Makalede
Özet
Fӧrster rezonans enerji transferi (FRET) tabanlı sensörlerin doğal düşük sinyal-gürültü oranı (SNR) nedeniyle, cAMP sinyallerinin ölçümü, özellikle üç uzamsal boyutta zor olmuştur. Burada, cAMP dağılımının üç mekansal boyutta ölçülemesine izin veren hiperspektral FRET görüntüleme ve analiz metodolojisini açıklıyoruz.
Özet
Siklik AMP, çok çeşitli hücresel ve fizyolojik aktivitelerde yer alan ikinci bir habercidir. Çeşitli çalışmalar, cAMP sinyallerinin bölümlere ayırdığını ve bölümlere ayırmanın cAMP sinyal yolu içindeki sinyal özgüllüğüne katkıda bulunduğunu göstermektedir. Fӧrster rezonans enerji transferi (FRET) bazlı biyosensörlerin geliştirilmesi, hücrelerdeki cAMP sinyallerini ölçme ve görselleştirme yeteneğini daha da ileriye taşıdı. Bununla birlikte, bu ölçümler genellikle verilerin yanlış yorumlanmasına neden olabilecek iki uzamsal boyutla sınırlıdır. Bugüne kadar, doğası gereği düşük sinyal-gürültü oranı (SNR) sergileyen FRET sensörlerinin kullanılmasındaki teknik sınırlamalar nedeniyle, üç uzamsal boyutta (x, y ve z) cAMP sinyallerinin çok sınırlı ölçümleri olmuştur. Buna ek olarak, geleneksel filtre tabanlı görüntüleme yaklaşımları, spektral çapraz konuşma, sınırlı sinyal gücü ve otofluoresans gibi çeşitli faktörler nedeniyle yerelleştirilmiş hücre altı bölgelerdeki cAMP sinyallerinin doğru ölçümü için genellikle etkisizdir. Bu sınırlamaların üstesinden gelmek ve FRET tabanlı biyosensörlerin birden fazla floresanla kullanılmasına izin vermek için, FRET verimliliklerini hesaplamak için spektral özgüllük ve FRET sinyallerini ek floresan etiketlerden gelen şaşırtıcı otoflüoresan ve/veya sinyallerden spektral olarak ayırma yeteneği sağlayan hiperspektral FRET görüntüleme ve analiz yaklaşımları geliştirdik. Burada, hiperspektral FRET görüntülemenin uygulanması için metodolojinin yanı sıra spektral karıştırma yapmak için ne az örneklenmiş ne de aşırı örneklenmiş uygun bir spektral kütüphane inşa etme ihtiyacını sunuyoruz. Pulmoner mikrovasküler endotel hücrelerinde (PMVEC) üç boyutlu cAMP dağılımlarının ölçümü için bu metodolojiyi sunarken, bu metodoloji cAMP'nin çeşitli hücre tiplerindeki mekansal dağılımlarını incelemek için kullanılabilir.
Giriş
Siklik adenozin monofosfat (cAMP), hücre bölünmesi, kalsiyum akışı, gen transkripsiyonu ve sinyal transdüksiyonu dahil olmak üzere önemli hücresel ve fizyolojik süreçlerde yer alan ikinci bir habercidir. Büyüyen bir kanıt gövdesi, hücrede sinyal özgüllüğünün elde edildiği cAMP bölmelerinin varlığını göstermektedir1, 2,3,4,5,6,7. Yakın zamana kadar, cAMP bölmeleme, farklı G bağlantılı reseptör agonistleri 8 ,9, 10,11tarafından indüklenen farklı fizyolojik veya hücresel etkilere dayanarakçıkarılmıştır. Daha yakın zamanda, FRET tabanlı floresan görüntüleme probları,12, 13,14hücresinde cAMP sinyallerinin doğrudan ölçümü ve gözlemlenmesi için yeni yaklaşımlar sağlamıştır.
Förster rezonans enerji transferi (FRET), donör ve alıcı moleküller arasında, moleküller yakın mesafedeyken radyatif olmayan bir şekilde enerji transferinin gerçekleştiği fiziksel bir olgudur15,16. FRET tabanlı floresan göstergelerin geliştirilmesiyle, bu fiziksel fenomen biyolojik uygulamalarda protein-protein etkileşimlerini incelemek için kullanılmıştır17, protein ko-lokalizasyonu18, Ca+2 sinyal19, gen ekspresyasyonu20, hücre bölünmesi21 ve döngüsel nükleotid sinyali. FRET tabanlı cAMP göstergeleri tipik olarak bir cAMP bağlama etki alanı, bir donör floroforu ve bir kabul edici florofor22'denoluşur. Örneğin, bu metodolojide kullanılan H188 cAMP sensör12,22, Turkuaz (donör) ve Venüs (kabul eden) floroforlar arasında sıkışmış, Epac'tan elde edilen bir cAMP bağlama alanından oluşur. Bazal koşullarda (ilişkisiz), Turkuaz ve Venüs, FRET'in floroforlar arasında meydana geldiği bir yönelimdedir. cAMP'nin bağlama alanına bağlanması üzerine, Turkuaz ve Venüs'ün birbirinden uzaklaşması ve FRET'te bir azalmaya neden olması için uygun bir değişiklik meydana gelir.
FRET tabanlı görüntüleme yaklaşımları, bir hücre içindeki cAMP sinyallerini araştırmak ve görselleştirmek için umut verici bir araç sunar. Bununla birlikte, mevcut FRET tabanlı mikroskobik görüntüleme teknikleri genellikle FRET'i hücre altı mekansal netlik ile ölçmek için yeterli sinyal gücü elde etmede kısmen başarılıdır. Bunun nedeni, birçok FRET muhabirinin sınırlı sinyal gücü, FRET verimliliğindeki değişiklikleri doğru bir şekilde ölçmek için gereken yüksek hassasiyet seviyesi ve hücresel otofluoresans23,24gibi şaşırtıcı faktörlerin varlığı da dahil olmak üzere çeşitli faktörlerden kaynaklanmaktadır. Sonuç genellikle zayıf SNR ile boğuşan ve FRET'teki hücre altı değişikliklerin görselleştirilmesini çok zor hale getiren bir FRET görüntüsüdür. Ek olarak, mekansal olarak lokalize cAMP sinyallerinin araştırılması neredeyse sadece iki mekansal boyutta gerçekleştirilmiştir ve eksenel cAMP dağılımı nadiren25olarak kabul edilmiştir. Bunun nedeni, düşük SNR'nin cAMP degradelerini üç uzamsal boyutta ölçme ve görselleştirme yeteneğini engellemesidir. Düşük SNR'li FRET sensörlerini kullanmanın sınırlamalarını aşmak için, FRET'i tek hücrelerde ölçmek için hiperspektral görüntüleme ve analiz yaklaşımları uyguladık25,26,27.
Hiperspektral görüntüleme yaklaşımları NASA tarafından uydu görüntülerinde bulunan karasal nesneleri ayırt etmek için geliştirilmiştir28,29. Bu teknikler o zamandan beri floresan mikroskopi alanı30spektral dedektörler sunan çeşitli ticari konfokal mikroskop sistemleri ile çevrilmiştir. Geleneksel (spektral olmayan) floresan görüntülemede, örnek bir bant geçiş filtresi veya lazer çizgisi kullanılarak heyecanlanır ve emisyon, genellikle floroforların tepe emisyon dalga boylarına uyacak şekilde seçilen ikinci bir bant geçiş filtresi kullanılarak toplanır. Bunun aksine, hiperspektral görüntüleme yaklaşımları, floresanemisyonu 26,31,32veya heyecan33 ,34'üntam bir spektral profilini belirli dalga boyu aralıklarında örneklemeyi amaçlamaktadır. Önceki çalışmalarımızda, hiperspektral görüntüleme ve analiz yaklaşımlarının, geleneksel filtre tabanlı FRET görüntüleme tekniklerine kıyasla hücrelerdeki FRET sinyallerinin daha iyi nicelleştirilmesini sunabileceğini gösterdik26. Burada, cAMP dağılımlarını üç mekansal boyutta ölçmek ve görselleştirmek için 4 boyutlu (x, y, z ve φ) hiperspektral FRET görüntüleme ve analizi yapmak için bir metodoloji sunuyoruz. Bu yaklaşımlar, agonist kaynaklı cAMP uzamsal gradyanlarının tek hücrelerde görselleştirilmesine izin verdi25. İlginçtir ki, agoniste bağlı olarak, hücrelerde cAMP gradyanları belirgin olabilir. Burada sunulan metodoloji, FRET ölçümlerinin doğruluğunu artırmak için tekdüze olmayan arka plan ve hücresel otofluoresans spektral karıştırmayı kullanır. Bu metodoloji pulmoner mikrovasküler endotel hücrelerinde (PMVEC) cAMP FRET biyosensörü kullanılarak gösterilmiş olsa da, metodoloji alternatif FRET muhabirleri veya alternatif hücre hatları ile kullanılmak üzere kolayca değiştirilebilir.
Protokol
Bu protokol, Güney Alabama Üniversitesi Kurumsal Hayvan Bakım ve Kullanım Komitesi tarafından onaylanan prosedürleri izler.
1. Görüntüleme için hücre, numune ve reaktif hazırlığı
- Daha önce açıklandığı gibi sıçan pulmoner mikrovasküler endotel hücrelerini (PMVEC) izole edin35.
NOT: Hücreler, Güney Alabama Üniversitesi, Mobile, AL'deki Hücre Kültürü Çekirdeği tarafından 100 mm hücre kültürü yemeklerinde izole edildi ve kültürlendi. - Tohum izole PMVEC'ler 25 mm yuvarlak cam kapak örtüleri üzerinde ve hücreler en az% 80 izdiah elde edene kadar (en az 24 saat) inkübatörde 37 ° C'de büyümelerini sağlar.
NOT: Hücreler ve hücre tipi çalışmadan çalışmaya değişebilir ve bu nedenle hücrelerin tohum ve büyümek için hücreye özgü hücre kültürü prosedürleri takip edilmelidir. Bu çalışmalarda kullanılan hücre tohumlama ve kültleştirme protokolü "Ek File_Cell Kültür ve Transfeksiyon" adlı dosyada ek bilgi olarak mevcuttur. - FRET biyosensörlü PMVEC'leri transfect ve 37 °C'de 48 saat kuluçkaya yatırın.
NOT: PMVEC'leri transfect protokolü ayrıca "Ek File_Cell Kültür ve Transfeksiyon" adlı ek bilgi dosyasında açıklanmıştır. - Görüntüleme gününde, Tyrode'un tamponu bir su banyosunda 37 °C'ye ısıtın.
NOT: Tyrode'un tamponu 145 mM NaCl, 4 mM KCl, 10 mM HEPES, 10 mM Glikoz, 1 mM MgCl 2 ve1 mM CaCl2'den oluşur - Transfected hücreler içeren bir kapak kapağını bir hücre odasına monte edin ve sızıntıyı önlemek için üst kısım bir montaj contası ile sabitleyin.
- Fazla medyayı veya yapışık hücreleri temizlemek için hassas bir görev silme kullanarak kapak kılıfının altını silin.
- Hücre odasına 800 μL çalışma tamponu ve 4 μL 5 mM nükleer etiket ekleyin ve 5 - 10 saniye boyunca hafifçe sallayın.
NOT: Hücre odasına monte edilen kapaklara tampon veya reaktif çözeltileri eklerken, yapışık hücreleri yerinden çıkarmak için çözeltiyi yavaşça ve hücre odasının kenarına eklediğinizden emin olun. 800 μL tampona 4 μL 5 mM nükleer etiket eklemek, 25 μM son nükleer etiket konsantrasyonu sağlar. HEK293 hücreleri gibi gevşek yapışan hücreler için, önce bir şişede nükleer etiket ve tamponu karıştırın ve ardından hücre odasına monte edilen kapaklara ekleyin. Bu, hücrelerin kapak kapağından kaldırılmasını önleyecektir. - Işıktan korumak için hücre haznesini alüminyum folyo ile örtün ve oda sıcaklığında 10 dakika kuluçkaya yatın.
- Reaktif Hazırlama: 199 μL tampona 1 μL 50 mM forskolin ekleyin. Bu, 800 μL tampon ile hazırlanan hücrelere eklendiğinde 50 μM'lik son bir forskolin konsantrasyonu üretecektir. 199 μL tamponda 1 μL DMSO da araç kontrolü olarak kullanılmak üzere hazırlanmalıdır.
NOT: Bu çalışmalarda forskolin, cAMP üretimini teşvik etmek için adenylyl siklaz aktivatörü olarak kullanılmaktadır. İstenirse, bu metodoloji, adenylyl siklaz, fosfoditeraz vb.
2. Görüntü alımı
- Spektral dedektörle donatılmış bir konfokal mikroskop kullanın.
NOT: Burada özetlenen tüm görüntü alma adımları, piyasada bulunan nikon A1R mikroskop sistemi kullanılarak geliştirilmiştir. Alternatif bir spektral mikroskop kullanıyorsanız bu adımların ayarlanması gerekebilir. Kararlı çalışma koşullarına ulaşmak için denemenin başlamasından en az 30 dakika önce tüm ekipmanların açık olduğundan emin olun. - 60x su daldırma hedefini seçin ve hedefe bir damla su ekleyin.
NOT: Yüksek çözünürlüklü canlı hücre görüntüleme için yüksek sayısal diyafram hedefi kullanılması önerilir. Bu çalışmalarda kullanılan amaç hakkında bilgi almak için lütfen Malzeme Listesi'ne bakın. - Yüklenen hücre odasını (adım 1.7'den itibaren) mikroskop aşamasına yerleştirin.
- Mikroskobun sağ tarafındaki filtre düğmesini ayarlayarak DFT (DAPI/FITC/TRITC) filtre kümesini seçin.
- cAMP FRET sensörüyü ifade eden hücreleri içeren bir görüş alanı seçmek için göz merceği kullanarak mikroskobu floresan geniş alan modunda çalıştırın.
NOT: Seçilen hücredeki donör veya kabul eden emisyon tepe dalga boyundaki FRET sinyalinin ortalama yoğunluğunun en az 100 yoğunluk birimi (A.U.) veya ifade hücresi olmayan bir bölgenin temel sinyalinin en az 4 katı olduğundan emin olun. Bu, NIS Elements yazılımında bulunan spektrum profili görüntüleyici kullanılarak onaylanabilir. İyi sinyalli bir hücre ararken, aşırı parlak hücrelerin atılması önerilir (tehlikeye atılabilirler). - NIS yazılımını açın, konfokal moda geçin, lazer kilitleme düğmesinin kilidini açın ve Canlı'ya tıklayın.
- Ekrandaki önizlemeye bakarak hücrelere odaklanmak için odak düğmesini kullanın.
- Yazılımdaki aygıt, edinme ve z yığını ayarlarını aşağıda açıklandığı gibi yapılandırın.
- Alım ayarları:
NOT: Kamera ve cihaz edinme ayarları daha önce edinilmiş bir görüntü kullanılarak uygulanabilir. Resmi açın, sağ tıklatın ve Kamera Ayarlarını Yeniden Kullan 'ıseçin.- A1 ayarları menüsünü açın, 405 nm ve 561 nm lazer hatlarına karşılık gelen kutuları işaretleyin, spektral dedektör için SD'yi seçin, çözünürlük için 10 ve kanallar için 31'i seçin.
NOT: A1 ayarları menüsü, A1 Plus Ayarları penceresinin sol üst köşesinde küçük bir dişli simgesi olarak gösterilir. Donör ekscitasyonu için 405 nm lazer, nükleer etiket ekscitasyonu için 561 nm lazer kullanılmaktadır. - Başlangıç ve bitiş dalga boy değerlerini seçerek dalga boyu aralığını (410 – 730 nm) ayarlayın.
- A1 ayarları menüsündeki binning/skip simgesine tıklayın ve 15 numaralı kutuyu seçin, ardından A1 ayarları menüsünde Tamam'a tıklayın.
NOT: Bu, 561 nm heyecan lazerine karşılık gelen dalga boyu kanalını kaldırmaktır (bu genellikle15. dalga boyu kanalıdır). Spektral bir yapı oluşturabilecek yapay olarak düşük bir sinyali önlemek için bu dalga boyu bandını kullanmamak önemlidir. Lazer hasarından korumak için dedektör elemanını kaplayan mekanik parmak nedeniyle sinyal bu bantta daha düşüktür. - Lazer yoğunluklarını sırasıyla 405 nm ve 561 nm lazerler için %8 ve %2, 149'da Si Hv (dedektör kazancı) ve 2,4 havadar disk ünitesi (AU) iğne deliği yarıçapı olarak ayarlayın.
NOT: Lazer yoğunluklarının cihazın yaşına ve lazerlerin durumuna bağlı olarak ayarlanması gerekebilir. Farklı örnekler veya deney grupları arasında lazer yoğunluklarını ayarlıyorsanız, aynı lazer yoğunluk oranını korumak önemlidir (örneğin, 8:2). Ek olarak, hızlı fotobleaching oluşturmak için çok parlak olmayan bir lazer yoğunluğu seçmek önemlidir. Dedektör kazancı, dedektör gürültüsünü en aza indirirken sinyal yoğunluğunu en üst düzeye çıkaracak şekilde ayarlanmalıdır. Bu çalışmalar için 149 kazanım kullanılmıştır. Yeterli sinyal-gürültü oranına (SNR) sahip görüntülerin alınması ile optik kesitin (konfokalite) korunması arasında denge olarak 2,4 AU'lık bir iğne deliği boyutu seçildi. İğne deliği boyutundaki artış SNR'yı arttırır, ancak konfokaliteyi azaltır. - Tarama hızını saniyede 0,25 spektral kareye ayarlayın, tarama yönü için tek yönlüye karşılık gelen simgeyi seçin, sayım için 4 girin ve tarama boyutu için 1024 x 1024 ayarlayın.
NOT: FRET sinyalleri zayıftır ve genellikle yavaş bir tarama hızı gerekir. 0,25 tarama hızı kullanılarak, spektral z yığınının alınması ~3 dakika içinde tamamlanır. Tarama hızı, kullanılan floroforlara bağlı olarak arttırılabilir veya azaltılabilir. Örneğin, eGFP gibi daha parlak floroforlar için daha hızlı tarama hızı (2 kare/saniye) kullanılabilir. Sayıma girilen sayı, görüntü alma sırasında gürültü azaltmada yardımcı olan 4 çerçeve ortalama değerine karşılık gelir. Çok kararlı numuneler için ve zamanın bir kısıtlama olmadığı durumlarda, geliştirilmiş SNR ile görüntü elde etmek için daha yüksek ortalama değerleri (16'ya kadar) kullanılabilir.
- A1 ayarları menüsünü açın, 405 nm ve 561 nm lazer hatlarına karşılık gelen kutuları işaretleyin, spektral dedektör için SD'yi seçin, çözünürlük için 10 ve kanallar için 31'i seçin.
- z-stack alma parametrelerini tanımla:
NOT: 2.10 adımlarında girilen değerlerin floresan etiket bağlama veya konsantrasyonundaki değişikliklere, etiket türüne, kullanılan etiket sayısına, hücre çizgisine ve hücre etiketleme yoğunluğunu ve/veya hücresel otoflüoresansı etkileyebilecek örnek hazırlamadaki diğer değişikliklere uyum sağlayacak şekilde ayarlanması gerekebilir. Edinme parametrelerini ayarlarken, fotobleaching'i en aza indirirken yeterli bir SNR elde etmeye özen edilmelidir. Ayrıca spektral FRET testini yapılandırırken parametrelerin tüm tedavi gruplarında iyi çalıştığından emin olunmalıdır. SNR'nin yeterli olduğundan ve fotobleachingin en aza indiriltiğinden emin olmak için önerilen parametre ayarlarıyla her tedavi grubunun bir denemesini çalıştırmanız önerilir.- Edinme denetimini görüntüle → ND edinme → tıklatarak ND edinme penceresiniaçın.
- ND dosyasını açılır pencereye kaydetmek için yolu/hedefi ve dosya adını girin.
- z serisinekarşılık gelen kutuyu işaretleyin.
- A1 Plus Ayarları penceresinde canlı olarak tıklayın. Bu, canlı bir görüntüleme penceresi açacaktır.
- Hücrenin üst kısmını seçmek için mikroskoptaki odak düğmesini ayarlayın ve geçerli konumu üst olarak ayarlamak için ND alma penceresinde Üst'ü tıklatın.
NOT: Tüm hücrenin z serisinde örneklenebilmesini sağlamak için hücrenin üst kısmının biraz üstüne odaklanılması önerilir. - Hücrenin altını seçmek için mikroskoptaki odak düğmesini ayarlayın ve geçerli konumu alt olarak ayarlamak için ND alım penceresinde Alt'ı tıklatın.
NOT: Tüm hücrenin örneklenebildiğinden emin olmak için hücrenin alt kısmının biraz altına odaklanın. - Adım boyutu için 1 μm girin, z tarama yönü için üst alt seçin ve z yığını elde etmek için ND Edinme penceresinde çalıştır'ı tıklayın.
NOT: Adım boyutu, üst ve alt konumlara (yani kat edilen mesafeye) bağlı olarak elde edilecek z dilimlerinin sayısını belirler. Görüntüleme hızı, z ekseni örneklemesi ve fotobleaching arasında bir uzlaşma olarak 1 μm adım boyutu seçildi. 2,4 AU'nun konfokal iğne deliği çapı ve 60x su daldırma hedefinin kullanılması, 1,73 μm optik kesit kalınlığı ile sonuçlandı. Bu nedenle, 1 μm adım boyutu Nyquist örnekleme kriterlerinin biraz altındadır, ancak bu, bir z-yığını elde etmek için gereken süreyi azaltmak için yapılmış bir uzlaşmadır. Hızı kritik olmayan çok kararlı numuneler için, z ekseni çözünürlüğünü artırmak için daha küçük bir z ekseni adımı ve muhtemelen daha küçük bir konfokal iğne deliği çapı kullanılabilir. Alt üst benzer sonuçlar vermeli ve z taraması sırasında oluşabilecek fotobleaching etkilerini değerlendirmek için kullanılabilir.
- Varsa Mükemmel Odak Sistemini (PFS) kurun:
NOT: PFS, sistemin görüntü alımı sırasında odak derinliğindeki dalgalanmaları telafi etmesine olanak tanır. PFS'yi kurmak için aşağıdaki adımlar kullanılabilir ve bu adımlar Nikon A1R sürümüne ve kullanılan NIS Öğeleri sürümüne bağlı olarak biraz değişebilir.- ND alma penceresindeki aralık simgesi tarafından tanımlanan simetrik modu vurgulayın.
- Mikroskobun ön yüzündeki PFS düğmesini açın (örnek aşamanın altındaki bölümde bulunan dikroik ayna düğmesinin 'in' olduğundan emin olun).
- PFS ofset denetleyicisinin ön yüzündeki düğmeyi kullanarak üst (saat yönünün tersine döndürün) ve altını (saat yönünde döndürün) yeniden tanımlayın.
- ND edinme penceresinde 'göreli'yi tıklatarak göreli bir z konumu/z derinliği tanımlayın.
- Mikroskobun ön yüzündeki belleğe tıklayın, böylece yazılım göreli z derinliğini ezberler.
- z-yığını alımı tamamlandıktan sonra, bir pipet kullanarak istediğiniz reaktifi (forskolin veya araç kontrolü) hafifçe ekleyin ve 10 dakika bekleyin.
NOT: Hücreleri rahatsız etmemek veya hücre odasının konumunu mikroskop XY aşaması içinde hareket ettirmemek için reaktifi çok nazikçe ekleyin; sonraki canlı görüntüde veya görüntüde, reaktif ekleme sırasında görüş alanının değişmediğini doğrulamak yararlıdır. 10 dakikalık bekleme süresi forskolin tedavisinin etkili olmasıdır. Alternatif bir tedavi kullanılırsa, bekleme süresinin ayarlanması gerekebilir. - 10 dakika sonra dosya adını değiştirin ve ND alma penceresinde çalıştır'ı tıklatın.
- İstatistiksel analiz için yeterli sonuçları elde etmek için en az 5 kapak için yukarıda belirtildiği gibi 2.11 – 2.13 adımlarını tekrarlayın (her tedavi grubu için n=5 – forskolin ve araç kontrolü).
- Spektral kitaplığı oluşturmak ve 2.9 ve 2.10 adımlarında belirtildiği gibi benzer alım ayarlarını kullanarak spektral görüntüler elde etmek için örnekler ve örnek boşluklar hazırlayın.
3. Görüntü analizi
NOT: Bu görüntüler, çalışmada bulunan tüm bireysel endmemberlerin saf spektrumunu içeren bir spektral kütüphane oluşturmak için kullanılacaktır. Spektral kütüphanedeki endmemberler, farklı floroforlar kullanılıyorsa çalışmadan çalışmaya değişebilir. Spektral kütüphaneyi oluşturmak için ayrıntılı bir prosedür "Ek File_Spectral Kütüphanesi" adlı ek bir bilgi dosyasında verilmiştir. Burada, .tiff dosyalarına veri dışa aktarmayı, doğrusal spektral karıştırmayı, FRET verimlilik ölçümlerini, üç boyutlu rekonstrüksiyonu ve cAMP düzeylerini tahmin ediyoruz. Görüntü analizi, ImageJ, Python, MATLAB veya CellProfiler gibi farklı görüntü analizi ve programlama platformları kullanılarak gerçekleştirilebilir. Bu çalışmalarda MATLAB betikleri kullanılmıştır.
- Görüntü verilerini dışarı aktar:
- 2.13 ve 2.14 adımlarında elde edilen spektral z yığını görüntülerine karşılık gelen aynı dosya adına sahip yeni klasörler oluşturun.
NOT: Verileri vermek için özetlenen aşağıdaki adımlar NIS Elements AR sürüm 4.30.01 için özeldir. Bu adımlar yazılımın sürümüne bağlı olarak biraz değişebilir. - DosyaPenceresi açacak olan Dosya 'yi tıklatın. 2.12. adımda alınan spektral görüntü dosyasına göz atın ve seçin ve Aç'ı tıklatın.
- Dosya yüklendikten sonra, Dosya→ ND belgesini → al/ver'i tıklatın.
- Açılır pencerede: 3.1.1 adımında oluşturulan klasöre göz atın ve seçin, Dosya türü için Etiketli Görüntü Biçimi'ni (TIF) seçin, ardından her kanal için Mono görüntü'yü seçin ve Bit derinliğini koru 'ya gidin.
NOT: Dosya öneki önceden oluşturulur; kolaylık sağlamak için bu değeri değiştirin. Dizin sırası seçilen Kanallara bağlı olarak değişir ve önce z dilimi konumuna ve ikinci dalga boyu bant numarasına göre dizin oluşturma için "z, c" görüntülemelidir. LUT'ları Uygula veya Yer Paylaşımları Ekle veya Nokta Adlarını Kullan'a karşılık gelen kutuların seçili olmadığından emin olun. - Tiff dosyalarını hedef klasöre tek tek tiff dosyaları olarak vermek için Dışarı Aktar'ı tıklatın.
- 2.13. adımda alınan spektral görüntü dosyalarını dışa aktarmak için 3.1.2 – 3.1.5 adımlarını yineleyin.
- 2.13 ve 2.14 adımlarında elde edilen spektral z yığını görüntülerine karşılık gelen aynı dosya adına sahip yeni klasörler oluşturun.
- Doğrusal spektral karıştırma:
- Programlama yazılımını açın.
NOT: Ham spektral görüntülerin karıştırılmasını sağlamak için özel olarak geliştirilen programlama komut dosyası, Güney Alabama Üniversitesi BioImaging ve BioSystems web sitesinde, Kaynaklar sekmesi altında (https://www.southalabama.edu/centers/bioimaging/resources.html) sağlanmaktadır. - "Doğrusal Karıştırma.m" etiketli dosyayı açın ve düzenleyici araç çubuğundaki çalıştır düğmesini tıklatın.
- NIS Elements yazılımı tarafından oluşturulan dışa aktarılan *.tif dosya sırasını içeren klasöre göz atın ve seçin.
- Wavelength ve Z-Sliceadlı yeni bir pencere açacak olan devam etmek için Tamam 'ı tıklatın.
- 3.2.4 adımında seçilen klasördeki ilk dosyanın dosya adını (z dilimi ve kanal numarası olmadan) kopyalayın ve "Görüntü Adını Girin" etiketli iletişim kutusunun ilk adımına yapıştırın.
- "Dalga boyu bantlarının sayısını girin" etiketli ikinci adımdaki kanal sayısını, üçüncü adımdaki "Z dilimlerinin sayısını girin" etiketli z dilimlerinin sayısını girin ve Tamam 'ıtıklatın.
NOT: Dalga boyu aralığını veya dalga boyu adım boyutunu ayarlama gibi alma ayarlarında değişiklik yapılırsa dalga boy bantlarının sayısı değişebilir. Z dilimlerinin sayısı da hücrenin yüksekliğine bağlı olarak değişebilir. - "Dalga boyu bilgi dosyasını seçin" etiketli açılır pencerede "Wavelength.mat" adlı dalga boyu dosyasına göz atın ve seçin ve aç'ı tıklatın.
- "Spektral kitaplık dosyasını seçin" etiketli yeni açılır pencerede "Library.mat" dosyasına göz atın ve seçin, aç'ı tıklatın ve dilimlerin karıştırılmama işinin bitmesini bekleyin.
NOT: Library.mat dosya hücre otofluorescence ve arka plan spektral imzaları ile birlikte her endmember fluorophore için saf spektra içeren bir dosyadır. Bu durumda, endmember floroforlar Turkuaz, Venüs ve DRAQ5 içerir. Arka plan spektral imzaları hücresel veya matris otofluoresans, kapaklı floresan ve coverlip kırınımını içerir. Wavelength.mat dosyası spektral görüntüyü elde etmek için kullanılan dalga boyu kanal bilgilerini içeren bir dosyadır. Örnek bir kitaplık dosyası ve dalga boyu dosyası Bioimaging ve Biosystems web sitesinde mevcuttur (3.2.1 altındaki nota bakın). Spektral kitaplık ve dalga boyu dosyalarının nasıl oluşturulacakları hakkında daha fazla bilgi için "Ek File_Spectral Kitaplığı" adlı ek bilgi dosyasına bakın. Her z dilimine karşılık gelen karıştırılmamış görüntüler, 3.2.3 adımında seçilen klasördeki karıştırma işlemi sırasında oluşturulan "Karıştırılmamış" adlı klasöre kaydedilir.
- Programlama yazılımını açın.
- FRET Verimlilik Hesaplaması:
- "multiFRRCF.m" adlı programlama komut dosyasını açın ve çalıştır'ı tıklatın.
NOT: Bu programlama dosyası Güney Alabama Üniversitesi Biyogörüntleme ve Biyosistem web sitesinden edinilebilir (adım 3.2.1 altındaki nota bakın). - "Kaç klasör yeniden kaydırılacağı" adlı açılır iletişim kutusuna çözümlemek için deneysel deneme sayısını girin ve Tamam 'ıtıklatın.
NOT: Her denemedeki görüntü verileri ayrı bir karıştırılmamış görüntü klasörüne kaydedilmelidir. Bu adım, analiz kodunun zaman kazandıran bir adım olarak birçok klasör üzerinde döngüye girmesine izin verir. - Karıştırılmamış klasörlere gözatıp seçin ve Tamam 'ıtıklatın.
NOT: "Klasöre gözat" açılır penceresinin açılma sayısı, önceki adımda "Yeniden kaydırılacağı klasör sayısı" iletişim kutusuna girilen sayıya bağlıdır. Klasörlere birbiri ardına göz atın ve seçin. - Yeni açılır pencerede, ilgili kutulara aşağıdaki bilgileri girin: ölçekleme faktörü 12,4, Eşik 5,6, X, Y ve Z Frekansı sırasıyla 5, 5 ve 1'dir ve yumuşatma algoritması Gauss'tır.
NOT: Ölçekleme faktörü piksel/μm cinsinden bir değerdir ve Z yönü örneklemeyi XY yönününkine ölçeklendirmek için kullanılacaktır. Ölçekleme faktörü, genellikle çoğu konfokal mikroskop sistemi için görüntüde meta veri olarak sağlanan görüntü piksel boyutundan elde edilir. Örneğin, görüntü 0,08 μm/piksel aralığıyla elde edilirse, ölçekleme faktörü 12,5 piksel/μm olmalıdır. Eşik değeri, görüntüleri eşik ve hücrenin ikili maskesini oluşturmak için kullanılır. Görüntü bağışçısı+alıcı yoğunluğuna göre optimum değerlerin bir listesini oluşturduk. Görüntünün parlak donör+alıcı yoğunluğu ve düşük arka planı varsa, yalnızca orta donör+alıcı yoğunluğuna ve/veya daha yüksek arka plana sahip görüntüler için 5,6 ile 6,5 arasında bir değer ve arka plandan daha düşük donör+alıcı yoğunluğuna sahip görüntüler için 7,5 ve üzeri bir değere sahipse, eşik değer olarak 4,5 kullanın. Frekans değeri, sonraki adımlarda dilimlemenin gerçekleştirildiği piksel sayısı aralığına karşılık gelir. Örneğin, hücrenin Z derinliği 1 μm adım boyutuna sahip 17 μm ise ve XY yönünde 12,5 piksel/μm ölçekleme faktörü kullanılıyorsa, 3 boyutlu görüntü veri kümesinin derinliği 212 piksel (Z yönü) olarak yeniden örneklenecektir. Girilen Z frekans değerine (örneğin, 1 piksel) bağlı olarak, 3 boyutlu görüntü veri kümesi, görüntü veri kümesinin en üstünden başlayarak yeniden dilimlenecek ve daha sonra aşağı doğru 1 piksellik artışlarla hareket ettirilir. Bu, 212 yeniden dizili görüntüyle sonuçlanır. Z Frekansı için daha büyük bir frekans değer aralığı girilirse, daha az yeniden dilimlenmiş görüntü oluşturulur. Yeniden kaydedilen görüntüler sonraki adımlarda kaydedilir. - Çalıştır'ı tıklatın ve tüm FRET ölçümleri ve yeniden dilimleme yapılana kadar bekleyin.
NOT: Üst dizinde, yeniden sınıflanmış gri tonlamalı FRET verimlilik görüntülerinin ve renkli (uygulanan bir renk haritası) FRET verimlilik görüntülerinin kaydedildiği ayrı bir klasör oluşturulur. Örneğin, X yönünde (YZ düzlemi) yeniden dizilen tüm gri tonlamalı ve renk haritası FRET görüntüleri "Resliced_XFRET" adlı bir klasöre kaydedilir. - Forskolin tedavileri ve araç kontrolleri öncesi ve sonrası tüm deneyler için benzer ayarlarla analizi tekrarlayın.
NOT: Bölüm 3.3'te belirtilen adımlar, 3 boyutlu FRET görüntü verileri oluşturmak için özel FRET analiz programlama komut dosyası için girilmesi gereken değerleri açıklar. Ancak, bu komut dosyası çalışırken aşağıdakiler de dahil olmak üzere çeşitli işlemler yürütür: görüntü verilerini yükleme, görüntü yığınları oluşturma, düzeltme, FRET verimlilik hesaplamaları oluşturma ve uygulama, hücre kenarlığı maskesi oluşturma ve uygulama, 3 boyutlu görüntü yeniden oluşturma, 3 boyutlu görüntüleri belirli aralıklarla (frekanslar) yeniden sıralama, FRET değişikliklerini görselleştirmek için bir renk haritası uygulama ve yeniden lisanslanan görüntü verilerini aynı dizine kaydetme. Ek ayrıntılar program komut dosyasına açıklama olarak eklenmiştir.
- "multiFRRCF.m" adlı programlama komut dosyasını açın ve çalıştır'ı tıklatın.
4. FRET verimliliğini cAMP seviyelerine eşleme
- 'Mapping_FRET Efficiency_to_cAMP_concentration.m' adlı programlama dosyasını açın ve ana pencerede çalıştır'ı tıklatın.
NOT: Dosya BioImaging ve BioSystems web sitesinde mevcuttur (3.2.1 altındaki nota bakın). Bu dosya gri tonlamalı FRET verimlilik görüntüleri okur ve karakteristik bir eğriye göre cAMP düzeylerine dönüştürür. Bu karakteristik eğri, Hill denklemi (aşağıda gösterilen üçüncü denklem) tarafından açıklanan literatür15,36'da belgelenen bir cAMP-to-FRET ilişkisi kullanır. Bununla birlikte, sağlam hücrelerdeki probun K d'sini tahmin etmek zordur ve hesaplamalarımızda 1 μM olduğunu varsayıyoruz. Bu nedenle, sonuçlar Kd. (yani, [cAMP] = x* Kd). FRET verimliliğini ölçmek ve FRET'i cAMP düzeyleriyle eşlemek için kullanılan denklemler aşağıda gösterilmiştir: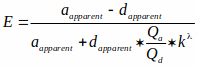
Burada E, FRET verimliliğidir vegörünür ve belirgin olan,sırasıyla alıcı ve donör görüntülerinin karıştırılmemiş piksel yoğunluklarıdır.
Qa ve Qd, kabul edenin ve bağışçının kuantum verimidir. Kφ denklemi FRET verimlilik denklemi içine dahil edildiğinde Qa ve Qd'nin iptal edildiğini unutmayın, kφ bir düzeltme faktörüdür: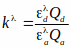
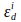 ve
ve 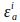 donör ve donör ekscitasyon dalga boyunda, i (405nm) donör ve kabul edenin yok olma katsayılarıdır.
donör ve donör ekscitasyon dalga boyunda, i (405nm) donör ve kabul edenin yok olma katsayılarıdır.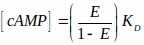
E, FRET verimliliği ve KD = Ayrışma sabiti = 1 μM'dir. - Gezinin ve ilk gri ölçekli FRET görüntüsünü seçin (adım 3.3.5'te kaydedildi) ve Tamam'ı tıklatın.
- cAMP sinyallerinin dağılımını üç boyutta incelemek için FRET/cAMP görüntülerini açın.
Sonuçlar
Bu protokol, canlı hücrelerde üç mekansal boyutta cAMP gradyanlarını ölçmek için hiperspektral FRET görüntüleme ve analiz yaklaşımlarının kullanımını açıklamaktadır. Verileri analiz ederken ve ölçerken dikkatli olunmesi gereken bu sonuçların oluşturulmasında birkaç önemli adım vardır. Bu önemli adımlar arasında uygun bir spektral kütüphanenin inşası, arka plan spektral karıştırması, hücre kenarlıklarını tanımlamak için eşik oluşturma ve FRET verimlilik hesaplamaları yer...
Tartışmalar
FRET biyosensörlerinin gelişimi, döngüsel nükleotid sinyallerinin tek hücrelerde ölçülmesini ve görselleştirilmesini sağladı ve hücre altı sinyal olaylarını görselleştirmek için büyük bir vaat var13,22,37,38. Bununla birlikte, FRET biyosensörlerinin kullanımı, birçok floresan protein bazlı FRET muhabirinin düşük sinyal-gürültü özellikleri ve FRET muhabirlerinin...
Açıklamalar
Dr. Leavesley ve Rich, spektral görüntüleme teknolojilerini ticarileştirmek için kurulan spectracyte, LLC bir start-up şirketine olan finansal ilgilerini açıkladılar. Ancak, bu protokolde açıklanan tüm prosedürler SpectraCyte, LLC ile ilişkili olmayan ticari olarak mevcut ürünler kullanılarak gerçek yapılmıştır.
Teşekkürler
Yazarlar, dr. Kees Jalink'i (Hollanda Kanser Enstitüsü ve van Leeuwenhoek İleri Mikroskopi Merkezi, Amsterdam, Hollanda) bize H188 cAMP FRET biyosensörü ve Kenny Trinh'i (Güney Alabama Mühendislik Fakültesi) özel olarak geliştirilen programlama komut dosyalarımızı çalıştırmak için geçen süreyi azaltmada teknik yardım sağladığı için kabul etmek istiyor.
Yazarlar fon kaynaklarını kabul etmek istiyor: Amerikan Kalp Derneği (16PRE27130004), Ulusal Bilim Vakfı; (1725937) NIH, S100D020149, S10RR027535, R01HL058506, P01HL066299).
Malzemeler
| Name | Company | Catalog Number | Comments |
| Attofluor Cell Chamber | Invitrogen | A7816 | Attofluor contains steel cell chambers and a rubber O-ring. Cell chamber holds the coverslip and O-ring provides a lock in mechanism to hold the buffer in cell chamber with out leakage |
| Dimethyl Sulfoxide (DMSO) | Fisher Scientific | BP231-100 | Solvent used to prepare stock solution forskolin. |
| DRAQ5 Fluoroscent Probe Solution | Thermo Scientific | 62252 | Nuclear label |
| Dulbecco Modified Eagle Medium (DMEM) | Gibco | 11965-092 | Contains nutrients and growth factors for the cells to grow and divide in the culture dishes. |
| Fetal Bovine Serum (FBS) | Sigma | F6178 | Growth factor suppliment that is added to culture medium, DMEM |
| Forskolin | Sigma | F3917 | Adenyly cyclase activator. |
| H188 Cyclic AMP FRET biosensor | Netherland Cancer Institute, Dr. K. Jalink | Gift | Plasmid encoding Turquoise (donor fluorophore), Venus (acceptor fluorophore), and binding domain obtained from Epac. |
| Image J | image.net | Free download | Another image processing platform used to extact spectral information and image processing. |
| Integrating Sphere | Ocean Optics | FOIS-1 | Used to measure illumination intensity of the laser line at different laser intensities (?). |
| Laminin Mouse Protein, Natural | Invitrogen | 23017-015 | Coverslips are coated with laminin and this helps in cell attachment, growth and motility of the cell. |
| Lipofectamine 3000 Transfection Kit | Invitrogen | L3000-015 | Transfection reagent used to transfect cells with H188 FRET biosensor |
| MATLAB | Mathworks | R2019a | Image processing operations (linear unmixing and FRET efficiency calculations) are performed by writing custom programs in MATLAB programming environment |
| Nikon A1R confocal microscope | Nikon Instruments | Nikon A1R | Spectral image acquisition is performed using confocal microscope. |
| Nikon Elements Software | Nikon Instruments | Software dongle | used to export and handle nd2 image files (multidimensional image files) that are aquired using Nikon A1R |
| NIST-Traceable Calibration Lamp | Ocean Optics | LS-1-CAL-INT | A lamp with a known spectrum for use as a standard |
| PBS pH 7.4 (1X) | Gibco | 10010-023 | coomonly used buffer suring cell culture |
| Pulmonary Microvascular Endothelial Cells (PMVECs) | In house - Cell culture core, Univeristy of South Alabama | Isolated from Rat pulmonary microvasculature | PMVECs form inner lining of a blood vessel. |
| Penicillin-Streptomycin (10,000 U/ml) | Gibco | 15140-122 | antibiotics are added to culture medum to prevent contamination of the cells. |
| Pre-Cleaned Gold Seal Micro Slides | Clay Adams | 3010 | Microscope slides used for cell fixation |
| ProLong Diamond Antifade Mounting Media | Invitrogen | P36961 | If samples are fixed using antifade mountant, then the later protects fluoroscent dyes and chromophores from fading. |
| Spectrometer | Ocean Optics | QE65000 | Used to measure spectral response of the light source (?) |
| Trypsin-EDTA (0.25%) | Gibco | 25200-056 | Digests the protein-protein bond between the cell and cell matrix and helps to disscociate and lift the cells during cell plating. |
| Tyrodes Buffer | Made in-house | Made in-house | Tyrodes buffer is used to make working solutions and to maintain cells in aqueous solution during image acquisition. |
| 6 Well Cell Culture Plate | Corning | 3506 | Laminin coated coverslips are placed in 6-well culture dish (one coverlisps/well). Cells along with medium is added into each well. |
| 25 mm Round Microscope Cover Slips | Fisher Scientific | 12545102 | Cells were grown on round glass coverslips |
| 60X Ojective | Nikon Instruments | Plan Apo VC 60X/1.2 WI ∞/0.15-0.18 WD 0.27 | water immersion and commonly used objective for cells |
Referanslar
- Corbin, J. D., Sugden, P. H., Lincoln, T. M., Keely, S. L. Compartmentalization of adenosine 3':5'-monophosphate and adenosine 3':5'-monophosphate-dependent protein kinase in heart tissue. The Journal of Biological Chemistry. 252, 3854-3861 (1977).
- Terrin, A., et al. PGE1 stimulation of HEK293 cells generates multiple contiguous domains with different [cAMP]: role of compartmentalized phosphodiesterases. The Journal of Cell Biology. 175, 441-451 (2006).
- Bacskai, B. J., et al. Spatially resolved dynamics of cAMP and protein kinase A subunits in Aplysia sensory neurons. Science. 260, 222-226 (1993).
- Iancu, R. V., Ramamurthy, G., Harvey, R. D. Spatial and temporal aspects of cAMP signalling in cardiac myocytes. Clinical and Experimental Pharmacology & Physiology. 35, 1343-1348 (2008).
- Brunton, L. L., Hayes, J. S., Mayer, S. E. Functional compartmentation of cyclic AMP and protein kinase in heart. Advances in Cyclic Nucleotide Research. 14, 391-397 (1981).
- Hohl, C. M., Li, Q. Compartmentation of cAMP in adult canine ventricular myocytes. Relation to single-cell free Ca2+ transients. Circulation Research. 69, 1369-1379 (1991).
- Rich, T. C., et al. A uniform extracellular stimulus triggers distinct cAMP signals in different compartments of a simple cell. Proceedings of the National Academy of Sciences of the United States of America. 98, 13049-13054 (2001).
- Sayner, S. L., Alexeyev, M., Dessauer, C. W., Stevens, T. Soluble adenylyl cyclase reveals the significance of cAMP compartmentation on pulmonary microvascular endothelial cell barrier. Circulation Research. 98, 675-681 (2006).
- Rich, T. C., Tse, T. E., Rohan, J. G., Schaack, J., Karpen, J. W. In vivo assessment of local phosphodiesterase activity using tailored cyclic nucleotide-gated channels as cAMP sensors. The Journal of General Physiology. 118, 63-78 (2001).
- Blackman, B. E., et al. PDE4D and PDE4B function in distinct subcellular compartments in mouse embryonic fibroblasts. Journal of Biological Chemistry. 286, 12590-12601 (2011).
- Sayner, S. L., et al. Paradoxical cAMP-induced lung endothelial hyperpermeability revealed by Pseudomonas aeruginosa ExoY. Circulation Research. 95, 196-203 (2004).
- Klarenbeek, J., Jalink, K. Detecting cAMP with an EPAC-based FRET sensor in single living cells. Methods in Molecular Biology. 1071, 49-58 (2014).
- Surdo, N. C., et al. FRET biosensor uncovers cAMP nano-domains at β-adrenergic targets that dictate precise tuning of cardiac contractility. Nature Communications. 8, 15031 (2017).
- Ponsioen, B., et al. Detecting cAMP-induced Epac activation by fluorescence resonance energy transfer: Epac as a novel cAMP indicator. EMBO Reports. 5, 1176-1180 (2004).
- Vogel, S. S., Thaler, C., Koushik, S. V. Fanciful FRET. Science's STKE. 2006, (2006).
- Clegg, R. M. The History of FRET: From conception through the labors of birth. Reviews in Fluorescence. 3, (2006).
- Giepmans, B. N. G., Adams, S. R., Ellisman, M. H., Tsien, R. Y. The fluorescent toolbox for assessing protein location and function. Science. 312, 217-224 (2006).
- Manzella-Lapeira, J., Brzostowski, J. A. Imaging protein-protein interactions by Förster resonance energy transfer (FRET) microscopy in live cells. Current Protocols in Protein Science. 93, 58 (2018).
- Cooper, D. M. F., Mons, N., Karpen, J. W. Adenylyl cyclases and the interaction between calcium and cAMP signalling. Nature. 374, 421-424 (1995).
- Sassone-Corsi, P. Coupling gene expression to cAMP signalling: role of CREB and CREM. The International Journal of Biochemistry & Cell Biology. 30, 27-38 (1998).
- Rebhun, L. I. Cyclic nucleotides, calcium, and cell division. International Review of Cytology. 49, 1-54 (1977).
- Klarenbeek, J., Goedhart, J., Van Batenburg, A., Groenewald, D., Jalink, K. Fourth-generation Epac-based FRET sensors for cAMP feature exceptional brightness, photostability and dynamic range: characterization of dedicated sensors for FLIM, for ratiometry and with high affinity. PLOS ONE. 10, 0122513 (2015).
- Leavesley, S. J., Rich, T. C. FRET: signals hidden within the noise. Cytometry Part A. 85, 918-920 (2014).
- Rich, T. C., Webb, K. J., Leavesley, S. J. Can we decipher the information content contained within cyclic nucleotide signals. The Journal of General Physiology. 143, 17-27 (2014).
- Annamdevula, N. S., et al. Spectral imaging of FRET-based sensors reveals sustained cAMP gradients in three spatial dimensions. Cytometry Part A. 93 (10), 1029-1038 (2018).
- Leavesley, S. J., Britain, A. L., Cichon, L. K., Nikolaev, V. O., Rich, T. C. Assessing FRET using spectral techniques. Cytometry Part A. 83, 898-912 (2013).
- Leavesley, S. J., Rich, T. C. Overcoming limitations of FRET measurements. Cytometry Part A. 89, 325-327 (2016).
- Fink, D. J. Monitoring Earcths Resources from Space. Technology Review. 75, 32-41 (1973).
- Goetz, A. F. H., Vane, G., Solomon, J. E., Rock, B. N. Imaging Spectrometry for Earth Remote Sensing. Science. 228, 1147-1153 (1985).
- Bücherl, C. A., Bader, A., Westphal, A. H., Laptenok, S. P., Borst, J. W. FRET-FLIM applications in plant systems. Protoplasma. 251, 383-394 (2014).
- Chen, Y., Mauldin, J. P., Day, R. N., Periasamy, A. Characterization of spectral FRET imaging microscopy for monitoring nuclear protein interactions. Journal of Microscopy. 228, 139-152 (2007).
- Zimmermann, T., Rietdorf, J., Girod, A., Georget, V., Pepperkok, R. Spectral imaging and linear un-mixing enables improved FRET efficiency with a novel GFP2-YFP FRET pair. FEBS Letters. 531, 245-249 (2002).
- Griswold, J. R., Annamdevula, N., Deal, J., Rich, T., Leavesley, S. Estimating FRET Efficiency using Excitation-Scanning Hyperspectral Imaging. Biophysical Journal. 112, 586 (2017).
- Favreau, P. F., et al. Excitation-scanning hyperspectral imaging microscope. Journal of Biomedical Optics. 19, 046010 (2014).
- King, J., et al. Structural and functional characteristics of lung macro- and microvascular endothelial cell phenotypes. Microvascular Research. 67, 139-151 (2004).
- Thaler, C., Koushik, S. V., Blank, P. S., Vogel, S. S. Quantitative multiphoton spectral imaging and its use for measuring resonance energy transfer. Biophysical Journal. 89, 2736-2749 (2005).
- Agarwal, S. R., et al. Compartmentalized cAMP signaling associated with lipid raft and non-raft membrane domains in adult ventricular myocytes. Frontiers in Pharmacology. 9, 332 (2018).
- Johnstone, T. B., Agarwal, S. R., Harvey, R. D., Ostrom, R. S. cAMP signaling compartmentation: Adenylyl cyclases as anchors of dynamic signaling complexes. Mol Pharmacol. , (2017).
- Zhang, J., Li, H., Chai, L., Zhang, L., Qu, J., Chen, T. Quantitative FRET measurement using emission-spectral unmixing with independent excitation crosstalk correction. Journal of Microscopy. 257, 104-116 (2015).
- Zhang, J., Lin, F., Chai, L., Wei, L., Chen, T. IIem-spFRET: improved Iem-spFRET method for robust FRET measurement. Journal of Biomedical Optics. 21, 105003 (2016).
- Levy, S., et al. SpRET: highly sensitive and reliable spectral measurement of absolute FRET efficiency. Microscopy and Microanalysis. 17, 176-190 (2011).
- West, S. J., et al. Hyperspectral Measurements Allow Separation of FRET Signals from Non-Uniform Background Fluorescence. Biophysical Journal. 112, 453 (2017).
- Annamdevula, N. S., et al. An approach for characterizing and comparing hyperspectral microscopy systems. Sensors. 13, 9267-9293 (2013).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır