É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Medição de distribuições 3-dimensionales cAMP em células vivas usando imagens e análises hiperespectrais de FRET 4-Dimensional (x, y, z e λ) Hiperespectrais
Neste Artigo
Resumo
Devido à relação sinal-ruído inerente (SNR) de sensores baseados em transferência de energia de ressonância (FRET) Fӧrster, a medição dos sinais cAMP tem sido desafiadora, especialmente em três dimensões espaciais. Aqui, descrevemos uma metodologia hiperespectral de imagem e análise de FRET que permite a medição da distribuição cAMP em três dimensões espaciais.
Resumo
O AMP cíclico é um segundo mensageiro que está envolvido em uma ampla gama de atividades celulares e fisiológicas. Vários estudos sugerem que os sinais de cAMP são compartimentados, e que a compartimentação contribui para a sinalização da especificidade dentro da via de sinalização cAMP. O desenvolvimento de biosensores baseados em transferência de energia de ressonância Fӧrster (FRET) promoveram a capacidade de medir e visualizar sinais cAMP nas células. No entanto, essas medidas são muitas vezes limitadas a duas dimensões espaciais, o que pode resultar em má interpretação dos dados. Até o momento, houve apenas medições muito limitadas de sinais cAMP em três dimensões espaciais (x, y e z), devido às limitações técnicas no uso de sensores FRET que inerentemente exibem baixa relação sinal/ruído (SNR). Além disso, as abordagens tradicionais de imagem baseadas em filtros são muitas vezes ineficazes para a medição precisa de sinais cAMP em regiões subcelulares localizadas devido a uma série de fatores, incluindo crosstalk espectral, força limitada do sinal e autofluorescência. Para superar essas limitações e permitir que os biosensores baseados em FRET sejam usados com fluoroforos múltiplos, desenvolvemos abordagens hiperespectrais de imagem e análise de FRET que fornecem especificidade espectral para calcular a eficiência do FRET e a capacidade de separar espectralmente os sinais FRET de confundir autofluorescência e/ou sinais de rótulos fluorescentes adicionais. Aqui, apresentamos a metodologia de implementação de imagens FRET hiperespectrais, bem como a necessidade de construir uma biblioteca espectral apropriada que não seja subamostra nem supersamplada para realizar descomprturações espectrais. Enquanto apresentamos essa metodologia para medição de distribuições tridimensionais de cAMP em células endoteliais microvasculares pulmonares (PMVECs), essa metodologia poderia ser usada para estudar distribuições espaciais de cAMP em uma gama de tipos celulares.
Introdução
Monofosfato de adenosina cíclica (cAMP) é um segundo mensageiro envolvido em processos celulares e fisiológicos chave, incluindo divisão celular, influxo de cálcio, transcrição genética e transdução de sinais. Um conjunto crescente de evidências sugere a existência de compartimentos cAMP na célula através dos quais a especificidade de sinalização é alcançada1,2,3,4,5,6,7. Até recentemente, a compartimentação cAMP era inferida com base em efeitos fisiológicos ou celulares distintos induzidos por diferentes agonistas receptores acopladosG 8,9,10,11. Mais recentemente, as sondas de imagem de fluorescência baseadas em FRET forneceram novas abordagens para a medição direta e observação de sinais cAMP em uma célula12,13,14.
A transferência de energia de ressonância förster (FRET) é um fenômeno físico no qual ocorre a transferência de energia entre moléculas doadoras e aceitadoras de forma não radiativa quando as moléculas estão próximas15,16. Com o desenvolvimento de indicadores fluorescentes baseados em FRET, esse fenômeno físico tem sido utilizado em aplicações biológicas para estudar interações proteína-proteína17, co-localização proteica18, Ca+2 sinalizando19, expressão genética20,divisão celular21 e sinalização nucleotídea cíclica. Os indicadores cAMP baseados em FRET normalmente consistem em um domínio de vinculação cAMP, um fluoróforo doador e um fluoróforoaceitor 22. Por exemplo, o sensor H188 cAMP12,22 utilizado nesta metodologia consiste em um domínio de ligação cAMP obtido da Epac, sanduíche entre fluoroforos turquesa (doador) e Vênus (aceitador). Em condições basais (sem entrada), Turquesa e Vênus estão em uma orientação de tal forma que o FRET ocorre entre os fluoroforos. Ao vincular o cAMP ao domínio de ligação, ocorre uma alteração conformacional de tal forma que Turquesa e Vênus se afastam, resultando em uma diminuição do FRET.
As abordagens de imagem baseadas em FRET oferecem uma ferramenta promissora para investigar e visualizar sinais cAMP dentro de uma célula. No entanto, as técnicas atuais de imagem microscópica baseadas em FRET são muitas vezes apenas parcialmente bem sucedidas em alcançar força de sinal suficiente para medir o FRET com clareza espacial subcelular. Isso se deve a vários fatores, incluindo a intensidade limitada do sinal de muitos repórteres da FRET, o alto nível de precisão necessário para quantificar com precisão as mudanças na eficiência do FRET e a presença de fatores de confusão, como a autofluorescência celular23,24. O resultado é muitas vezes uma imagem FRET que é atormentada por SNR fraco, tornando muito difícil a visualização de mudanças subcelulares no FRET. Além disso, a investigação de sinais cAMP espacialmente localizados tem sido realizada quase exclusivamente em apenas duas dimensões espaciais e a distribuição axial cAMP raramente foi considerada25. Isso é provável porque o SNR baixo impediu a capacidade de medir e visualizar gradientes cAMP em três dimensões espaciais. Para superar as limitações do uso de sensores FRET com baixo SNR, implementamos abordagens hiperespectrais de imagem e análise para medir o FRET em células únicas25,26,27.
Abordagens de imagem hiperespectral foram desenvolvidas pela NASA para diferenciar objetos terrestres presentes em imagens de satélite28,29. Essas técnicas foram traduzidas para o campo de microscopia de fluorescência30,com vários sistemas comerciais de microscópio confocal oferecendo detectores espectrais. Em imagens tradicionais (não espectrais) de fluorescência, a amostra é animada usando um filtro de passe de banda ou uma linha laser, e a emissão é coletada usando um segundo filtro de passe de banda, muitas vezes selecionado para corresponder ao comprimento de onda de emissão máxima do fluoróforo(s). Em contrapartida, as abordagens de imagem hiperespectral buscam amostrar um perfil espectral completo da emissão de fluorescência26,31,32 ou excitação33,34 em intervalos específicos de comprimento de onda. Em nossos estudos anteriores, mostramos que as abordagens de imagem e análise hiperespectral podem oferecer uma quantificação melhorada dos sinais DE TRAC nas células quando comparadas às técnicas tradicionais de imagem FRET baseadas emfiltros 26. Aqui, apresentamos uma metodologia para a realização de imagens e análises hiperespectrais de FRET hiperdimensionais (x, y, z e λ) para medir e visualizar distribuições cAMP em três dimensões espaciais. Essas abordagens permitiram a visualização de gradientes espaciais cAMP induzidos por agonista em célulasúnicas 25. Curiosamente, dependendo do agonista, gradientes cAMP podem ser aparentes nas células. A metodologia aqui apresentada utiliza o descompreamento espectral de fundo não uniforme e autofluorescência celular para melhorar a precisão das medições de FRET. Embora essa metodologia seja demonstrada em células endoteliais microvasculares pulmonares (PMVECs) utilizando um biosensor CAMP FRET, a metodologia poderia ser facilmente modificada para uso com repórteres fret alternativos ou linhas celulares alternativas.
Protocolo
Este protocolo segue os procedimentos aprovados pelo Comitê Institucional de Cuidados e Uso de Animais da Universidade do Sul do Alabama.
1. Preparação de células, amostras e reagentes para imagem
- Isolar as células endoteliais microvasculares pulmonares de ratos (PMVECs) como descrito anteriormente35.
NOTA: As células foram isoladas e cultivadas pelo Núcleo de Cultura Celular da Universidade do Sul do Alabama, Mobile, AL em pratos de cultura celular de 100 mm. - As sementes isolaram os PMVECs em tampas de vidro redondo de 25 mm e as deixam crescer na incubadora a 37 °C até que as células atinjam pelo menos 80% de confluência (pelo menos 24 horas).
NOTA: Células e tipo celular podem variar de estudo para estudo e, portanto, procedimentos de cultura celular específicos para células devem ser seguidos para sementes e células de cultivo. O protocolo de semeadura e cultivo de células utilizado nesses estudos está disponível como informação suplementar no arquivo denominado "Cultura e Transfecção File_Cell Suplementar". - Transfect PMVECs com um biosensor FRET e incubar por 48 horas a 37 °C.
NOTA: O protocolo para transfeccionar PMVECs também é descrito no arquivo de informações suplementares chamado "Cultura e Transfecção File_Cell Suplementar". - No dia da imagem, aqueça o tampão de Tyrode a 37 °C em um banho de água.
NOTA: O buffer de Tyrode é composto por 145 mM NaCl, 4 mM KCl, 10 mM HEPES, 10 mM De Glicose, 1 mM MgCl2 e 1 mM CaCl2 - Monte um deslizamento contendo células transfeinadas em uma câmara celular e proteja a parte superior com uma junta de montagem para evitar vazamentos.
- Limpe a parte inferior da tampa usando uma limpeza de tarefa delicada para limpar qualquer excesso de mídia ou células aderentes.
- Adicione 800 μL de tampão de trabalho e 4 μL de etiqueta nuclear de 5 mM à câmara celular e abale suavemente por 5 a 10 segundos.
NOTA: Ao adicionar soluções de tampão ou reagente a deslizamentos montados na câmara celular, certifique-se de adicionar a solução suavemente e ao lado da câmara celular para não desalojar células aderentes. Adicionar 4 μL de 5 mM de etiqueta nuclear a 800 μL de buffer faz 25 μM de concentração final de rótulo nuclear. Para células frouxamente aderentes, como células HEK293, misture o rótulo nuclear e o buffer em um frasco primeiro e, em seguida, adicione a tampas montadas na câmara celular. Isso evitará tirar as células da tampa. - Cubra a câmara celular com papel alumínio para proteger da luz e incubar por 10 minutos à temperatura ambiente.
- Preparação do reagente: Adicione 1 μL de 50 mM forskolin a 199 μL de tampão. Isso produzirá uma concentração final de forskolin de 50 μM quando adicionado às células que foram preparadas com 800 μL de tampão. 1 μL de DMSO em 199 μL de buffer também deve ser preparado para ser usado como controle de veículo.
NOTA: Nestes estudos, a forskolina é usada como ativadora de cyclase adenylyl para estimular a produção de cAMP. Se desejar, essa metodologia pode ser facilmente modificada para permitir o tratamento com reagentes alternativos para estimular ou inibir cíclase adenilyl, fosfodiesterases, etc.
2. Aquisição de imagens
- Use um microscópio confocal equipado com um detector espectral.
NOTA: Todas as etapas de aquisição de imagem descritas aqui foram desenvolvidas usando um sistema de microscópio Nikon A1R disponível comercialmente. Essas etapas podem precisar ser ajustadas se usar um microscópio espectral alternativo. Certifique-se de que todos os equipamentos estão ligados pelo menos 30 minutos antes do início do experimento, de modo a atingir condições de funcionamento estáveis. - Selecione o objetivo de imersão em água de 60x e adicione uma gota de água ao objetivo.
NOTA: Para imagens de células vivas de alta resolução, recomenda-se usar um objetivo de abertura numérica elevada. Consulte a Lista de Materiais para obter informações sobre o objetivo utilizado nestes estudos. - Coloque a câmara celular carregada (a partir da etapa 1.7) no estágio do microscópio.
- Selecione o conjunto de filtro DFT (DAPI/FITC/TRITC) afinando o botão do filtro no lado direito do microscópio.
- Opere o microscópio no modo widefield de fluorescência usando as oculares para selecionar um campo de visão contendo células expressando o sensor CAMP FRET.
NOTA: Certifique-se de que a intensidade média do sinal FRET no comprimento de onda de pico de emissão do doador ou aceitador na célula selecionada é de pelo menos 100 unidades de intensidade (A.U.) ou pelo menos 4X o sinal de linha de base de uma região sem células expressas. Isso pode ser confirmado usando o visualizador de perfil de espectro disponível no software NIS Elements. Ao procurar uma célula com bom sinal, é aconselhável descartar células excessivamente brilhantes (elas podem estar comprometidas). - Abra o software NIS, mude para o modo confocal, desbloqueie o botão de intertravamento a laser e clique em Live.
- Use o botão de foco para se concentrar nas células olhando para a visualização na tela.
- Configure as configurações do dispositivo, aquisição e pilha de z no software, conforme descrito abaixo.
- Configurações de aquisição:
NOTA: As configurações de aquisição de câmeras e dispositivos podem ser aplicadas usando uma imagem adquirida anteriormente. Abra a imagem, clique com o botão direito do mouse e selecione ReuseAr configurações da câmera.- Abra o menu de configurações do A1, verifique as caixas correspondentes a linhas laser de 405 nm e 561 nm, selecione SD para detector espectral, selecione 10 para resolução e 31 para canais.
NOTA: O menu de configurações do A1 é mostrado como um pequeno ícone de engrenagem no canto superior esquerdo da janela Configurações A1 Plus. Laser de 405 nm é usado para excitação de doadores e laser de 561 nm é usado para excitação de rótulo nuclear. - Defina a faixa de comprimento de onda (410 – 730 nm) selecionando valores de comprimento de onda de início e fim.
- Clique no ícone binning/skip no menu configurações A1 e selecione a caixa numerada 15 e clique em OK no menu de configurações A1.
NOTA: Isto é para remover o canal de comprimento de onda que corresponde ao laser de excitação de 561 nm (este é tipicamente o canal de comprimento deonda de 15º). É importante não usar essa faixa de comprimento de onda para evitar um sinal artificialmente baixo, que pode criar um artefato espectral. O sinal é menor nesta banda por causa do dedo mecânico que cobre o elemento detector para protegê-lo de danos a laser. - Defina as intensidades de laser para 8% e 2% para os lasers de 405 nm e 561 nm, respectivamente, Si Hv (ganho de detector) em 149, e um raio de 2,4 unidades de disco arejado (UA).
NOTA: As intensidades do laser podem ter que ser ajustadas dependendo da idade do instrumento e da condição dos lasers. Se ajustar as intensidades do laser entre diferentes amostras ou grupos experimentais, é importante manter a mesma proporção de intensidades de laser (por exemplo, 8:2). Além disso, é importante selecionar uma intensidade laser que não seja tão brilhante a ponto de criar fotobleaching rápido. O ganho do detector deve ser ajustado para maximizar a intensidade do sinal, minimizando o ruído do detector. Para esses estudos, foi utilizado um ganho de 149. Um tamanho de pinhole de 2,4 UA foi selecionado como um equilíbrio entre a aquisição de imagens com relação sinal-ruído suficiente (SNR) e a manutenção da secção óptica (confocalidade). Um aumento no tamanho do pinhole aumenta o SNR, mas diminui a confocalidade. - Defina a velocidade de varredura para 0,25 quadros espectrais por segundo, selecione o ícone correspondente à direção de varredura unidirecional, digite 4 para a contagem e defina 1024 x 1024 para o tamanho da varredura.
NOTA: Os sinais de FRET são fracos e muitas vezes é necessária uma velocidade de varredura lenta. Usando a velocidade de varredura de 0,25, a aquisição de uma pilha z espectral é concluída em ~3 minutos. A velocidade de varredura pode ser aumentada ou diminuída dependendo dos fluoroforos utilizados. Por exemplo, para fluoroforos mais brilhantes como eGFP, pode-se usar uma velocidade de varredura mais rápida (2 quadros/segundo). O número inserido sob contagem corresponde a um valor médio de quadro de 4, o que ajuda na redução de ruído durante a aquisição de imagem. Para amostras muito estáveis e onde o tempo não é uma restrição, valores de média mais elevados (até 16) podem ser usados para obter imagens com SNR melhorado.
- Abra o menu de configurações do A1, verifique as caixas correspondentes a linhas laser de 405 nm e 561 nm, selecione SD para detector espectral, selecione 10 para resolução e 31 para canais.
- Defina parâmetros de aquisição de pilhas z:
NOTA: Os valores inseridos nas etapas 2.10 podem precisar ser ajustados para acomodar alterações na ligação ou concentração de rótulos fluorescentes, tipo de rótulo, número de rótulos utilizados, linha celular e outras alterações na preparação da amostra que podem afetar a densidade de rotulagem celular e/ou autofluorescência celular. Ao ajustar os parâmetros de aquisição, deve-se tomar cuidado para obter um SNR suficiente, minimizando o fotobleaching. Além disso, ao configurar um ensaio espectral de FRET, deve-se tomar cuidado para garantir que os parâmetros funcionem bem em todos os grupos de tratamento. É aconselhável executar um teste de cada grupo de tratamento com as configurações de parâmetros propostas para garantir que o SNR seja suficiente e que a fotobleaching seja minimizada.- Abra a janela de aquisição da ND clicando em ver → controle de aquisição → aquisição da ND.
- Digite o caminho/destino e um nome de arquivo para salvar o arquivo ND na janela pop-up.
- Verifique a caixa correspondente à série Z.
- Clique ao vivo na janela Configurações A1 Plus. Isso abrirá uma janela de visualização ao vivo.
- Ajuste o botão de foco no microscópio para selecionar a parte superior da célula e clique em Topo na janela de aquisição ND para definir a posição atual como a parte superior.
NOTA: Sugere-se que se concentre ligeiramente acima da parte superior da célula para garantir que toda a célula seja amostrada na série z. - Ajuste o botão de foco no microscópio para selecionar a parte inferior da célula e clique em Inferior na janela de aquisição ND para definir a posição atual como a parte inferior.
NOTA: Concentre-se ligeiramente abaixo da parte inferior da célula para garantir que toda a célula seja amostrada. - Digite 1 μm para o tamanho da etapa, selecione a parte superior inferior para a direção z-scan e clique em executar na janela ND Acquisition para adquirir uma pilha z.
NOTA: O tamanho da etapa determina o número de fatias z que serão adquiridas dependendo dos locais superiores e inferiores (ou seja, a distância percorrida). Um tamanho de passo de 1 μm foi selecionado como um compromisso entre velocidade de imagem, amostragem de eixo z e fotobleaching. O uso do diâmetro do orifício confocal de 2,4 UA e o objetivo de imersão de água de 60x resultou em espessura de seção óptica de 1,73 μm. Assim, um tamanho de passo de 1 μm está ligeiramente abaixo dos critérios de amostragem de Nyquist, mas este é um compromisso que foi feito para reduzir o tempo necessário para adquirir uma pilha z. Para amostras muito estáveis, para as quais a velocidade não é crítica, uma etapa menor do eixo Z e possivelmente um diâmetro menor do pinóculo conferiocal podem ser usados para aumentar a resolução do eixo Z. A parte superior deve produzir resultados semelhantes e pode ser usada para avaliar quaisquer efeitos de fotobleaching que possam ocorrer durante a varredura z.
- Configure o Sistema de Foco Perfeito (PFS) se disponível:
NOTA: O PFS permite que o sistema compense flutuações na profundidade focal durante a aquisição de imagens. As etapas a seguir podem ser usadas para configurar pfs, e essas etapas podem variar ligeiramente dependendo da versão do Nikon A1R e da versão dos Elementos NIS usados.- Destaque o modo simétrico definido pelo ícone de alcance na janela de aquisição ND.
- Ligue o botão PFS na face frontal do microscópio (certifique-se de que o botão do espelhodicróico localizado na seção abaixo do estágio da amostra esteja 'in').
- Redefina a parte superior (gire no sentido anti-horário) e inferior (gire no sentido horário) usando o botão na face frontal do controlador de deslocamento PFS.
- Defina uma relativa z-position/z-profundidade clicando em 'relativo' na janela de aquisição ND.
- Clique na memória na face frontal do microscópio para que o software memorize a profundidade z relativa.
- Depois que a aquisição da pilha z estiver completa, adicione suavemente o reagente desejado (forskolin ou controle do veículo) usando uma pipeta e espere por 10 minutos.
NOTA: Adicione o reagente muito suavemente para não perturbar as células ou mover a posição da câmara celular dentro do estágio XY do microscópio; é útil verificar em uma visão ou imagem ao vivo subsequente que o campo de visão não mudou durante a adição de reagente. O tempo de espera de 10 minutos é para que o tratamento de forskolin faça efeito. Se um tratamento alternativo for usado, o tempo de espera pode precisar ser ajustado. - Após 10 minutos, altere o nome do arquivo e clique em executar na janela de aquisição ND.
- Repetição dos passos 2.11 – 2.13, conforme descrito acima, para pelo menos 5 deslizamentos de cobertura, de modo a obter resultados suficientes para análise estatística (n=5 para cada grupo de tratamento – forskolin e controle do veículo).
- Prepare amostras e amostras em branco para construir a biblioteca espectral e adquirir imagens espectrais usando configurações de aquisição semelhantes, conforme descrito nas etapas 2.9 e 2.10.
3. Análise de imagem
NOTA: Essas imagens serão usadas para construir uma biblioteca espectral contendo o espectro puro de todos os membros finais individuais presentes no estudo. Os membros finais na biblioteca espectral podem variar de estudo para estudo se diferentes fluoroforos forem usados. Um procedimento detalhado para construir a biblioteca espectral é fornecido em um arquivo de informações suplementares chamado "Biblioteca de File_Spectral Suplementar". Aqui, descrevemos dados de exportação para .tiff arquivos, descomprência espectral linear, medidas de eficiência de FRET, reconstrução tridimensional e estimativa dos níveis de cAMP. A análise de imagens pode ser realizada usando diferentes plataformas de análise de imagem e programação, como ImageJ, Python, MATLAB ou CellProfiler. Nesses estudos, foram utilizados scripts MATLAB.
- Dados de imagem de exportação:
- Crie novas pastas com o mesmo nome de arquivo correspondente às imagens espectrais da pilha z adquiridas nas etapas 2.13 e 2.14.
NOTA: As seguintes etapas descritas para exportar dados são específicas para nis elements ar versão 4.30.01. Essas etapas podem variar ligeiramente dependendo da versão do software. - Clique em Arquivo, que abrirá uma janela de arquivo. Navegue e selecione o arquivo de imagem espectral adquirido na etapa 2.12 e clique em Abrir.
- Uma vez que o arquivo seja carregado, clique em Arquivar→ documento ND de importação/exportação→ exportação.
- Na janela pop-up: navegue e selecione a pasta criada na etapa 3.1.1, selecione TIF (Tagged Image Format, formato de imagem marcada) para tipo arquivo e selecione imagem Mono para cada canal e Mantenha a profundidade de bits.
NOTA: O prefixo do arquivo será pré-gerado; alterar esse valor por conveniência. A ordem do Índice mudará dependendo dos canais selecionados e deverá exibir "z, c" para indexação de acordo com o local de z-slice primeiro e o número da banda de comprimento de onda em segundo lugar. Certifique-se de que as caixas correspondentes para aplicar LUTs ou Inserir sobreposições ou nomes de pontos de uso não sejam eleitas. - Clique em Exportar os arquivos de tiff para uma pasta de destino como arquivos de tiff individuais.
- Repetir passos 3.1.2 – 3.1.5 para exportar arquivos de imagem espectrais adquiridos na etapa 2.13.
- Crie novas pastas com o mesmo nome de arquivo correspondente às imagens espectrais da pilha z adquiridas nas etapas 2.13 e 2.14.
- Descomprturação espectral linear:
- Abra o software de programação.
NOTA: O script de programação desenvolvido sob medida para desmixar imagens espectrais brutas é fornecido no site da Universidade do Sul do Alabama BioImaging e BioSystems, sob a guia Recursos (https://www.southalabama.edu/centers/bioimaging/resources.html). - Abra o arquivo rotulado de "Linear Unmixing.m" e clique no botão executar na barra de ferramentas do editor.
- Navegue e selecione a pasta que contém a sequência de arquivos *.tif exportada gerada pelo software NIS Elements.
- Clique em OK para continuar, que abrirá uma nova janela chamada Wavelength e Z-Slice.
- Copie o nome do arquivo do primeiro arquivo (sem z-slice e número do canal) na pasta selecionada na etapa 3.2.4 e cole-a na primeira etapa da caixa de diálogo rotulada "Enter the Image Name".
- Digite o número de canais na segunda etapa intitulada "Digite o número de bandas de comprimento de onda", número de fatias z na terceira etapa rotulada "Digite o número de fatias Z" e clique em OK.
NOTA: O número de faixas de comprimento de onda pode mudar se forem feitas alterações nas configurações de aquisição, como ajustar a faixa de comprimento de onda ou o tamanho do passo do comprimento de onda. O número de fatias Z também pode mudar dependendo da altura da célula. - Navegue e selecione o arquivo de comprimento de onda chamado "Wavelength.mat" na janela pop-up rotulada "Selecione o arquivo de informações de comprimento de onda" e clique em abrir.
- Navegue e selecione o arquivo "Library.mat" na nova janela pop-up rotulada "Selecione o arquivo da biblioteca espectral", clique em abrir e aguarde até que o descompreamento das fatias seja concluído.
NOTA: O arquivo Library.mat é um arquivo que contém espectros puros para cada fluoróforo de membro final, juntamente com autofluorescência celular e assinaturas espectrais de fundo. Neste caso, os fluoroforos de fim incluem Turquesa, Vênus e DRAQ5. As assinaturas espectrais de fundo incluem autofluorescência celular ou matricial, fluorescência de deslizamento e difração de deslizamento. O arquivo Wavelength.mat é um arquivo que contém informações do canal de comprimento de onda usado para adquirir a imagem espectral. Um arquivo de biblioteca de exemplo e arquivo de comprimento de onda estão disponíveis no site bioimagem e biossistemas (veja a nota em 3.2.1). Para obter mais informações sobre como gerar biblioteca espectral e arquivos de comprimento de onda, consulte o arquivo de informações suplementares chamado "Biblioteca de File_Spectral Suplementar". Imagens não misturadas correspondentes a cada fatia z serão salvas na pasta chamada "Unmixed" criada durante o processo de descomprturação dentro da pasta que foi selecionada na etapa 3.2.3.
- Abra o software de programação.
- Cálculo de eficiência do FRET:
- Abra o script de programação chamado "multiFRRCF.m" e clique em executar.
NOTA: Este arquivo de programação está disponível no site da Universidade do Sul do Alabama Bioimagem e Biossistemas (ver nota abaixo do passo 3.2.1). - Digite o número de ensaios experimentais para analisar na caixa de diálogo pop-up chamada "quantas pastas para reslice" e clique em OK.
NOTA: Os dados de imagem de cada experimento devem ser salvos em uma pasta de imagem não misturada separada. Esta etapa simplesmente permite que o código de análise loop sobre muitas pastas como uma etapa de economia de tempo. - Navegue e selecione as pastas não misturadas e clique em OK.
NOTA: O número de vezes que a janela "Procurar pasta" é aberto depende do número inserido na caixa de diálogo "Quantas pastas para reslice" na etapa anterior. Navegue e selecione as pastas uma após a outra. - Na nova janela pop-up, insira as seguintes informações nas respectivas caixas: o fator de escala é 12.4, limiar é 5.6, X, Y, e Z Frequency são 5, 5 e 1, respectivamente, e o algoritmo de alisamento é gaussiano.
NOTA: O fator de escala é um valor em pixels/μm e será usado para dimensionar a amostragem de direção Z à da direção XY. O fator de escala é obtido a partir do tamanho do pixel da imagem, que geralmente é fornecido como metadados na imagem para a maioria dos sistemas de microscópio confocal. Por exemplo, se a imagem for adquirida com espaçamento de 0,08 μm/pixel, o fator de escala deve ser de 12,5 pixels/μm. O valor do limiar será usado para limiar das imagens e gerar uma máscara binária da célula. Criamos uma lista de valores ótimos com base na intensidade do doador de imagem+aceitador. Use 4,5 como valor de limiar se a imagem tiver um doador brilhante+intensidade aceitadora e baixo fundo, um valor entre 5,6 a 6,5 para imagens com apenas intensidade moderada de doador+aceitador e/ou fundo superior, e um valor de 7,5 e acima para imagens com intensidade doador+aceitante inferior ao fundo. O valor da frequência corresponde ao intervalo, em número de pixels, no qual o fatiamento é realizado nas etapas subsequentes. Por exemplo, se a profundidade Z da célula for de 17 μm com um tamanho de passo de 1 μm e um fator de escala de 12,5 pixels/μm for usado na direção XY, então a profundidade do conjunto de dados de imagem tridimensional será resamada em 212 pixels (sentido Z). Com base no valor de frequência Z inserido (por exemplo, 1 pixel), o conjunto de dados de imagem tridimensional será re fatiado a partir da parte superior do conjunto de dados de imagem e, em seguida, movendo-se em incrementos de 1 pixel para baixo. Isso resulta em 212 imagens relíquias. Se um intervalo de valor de frequência maior fosse inserido para a Frequência Z, então menos imagens relíquias seriam geradas. As imagens relíquias são salvas em etapas subsequentes. - Clique em executar e esperar até que todas as medidas e relicações do FRET sejam realizadas.
NOTA: Uma pasta separada é criada dentro do diretório pai para o qual imagens de eficiência FRET em escala de cinza relíquias e imagens coloridas (um colormap aplicado) são salvas imagens de eficiência do FRET. Por exemplo, todas as imagens FRET em escala de cinza e colormap reliced na direção X (plano YZ) são salvas em uma pasta chamada "Resliced_XFRET". - Repita a análise com configurações semelhantes para todos os experimentos – tratamentos antes e depois de forskolin e controles do veículo.
NOTA: As etapas mencionadas na seção 3.3 descrevem os valores a serem inserindo para o script de programação de análise fret personalizado para gerar dados de imagem FRET tridimensionais. No entanto, este script executa várias operações durante a execução, incluindo: carregando dados de imagem, criando pilhas de imagens, suavização, cálculos de eficiência de FRET, criação e aplicação de uma máscara de borda celular, reconstrução de imagem tridimensional, relicência de imagens tridimensionais em intervalos especificados (frequências), aplicando um colormap para visualizar alterações de FRET e salvar os dados de imagem relíquias para o mesmo diretório. Detalhes adicionais foram incluídos como comentários no roteiro do programa.
- Abra o script de programação chamado "multiFRRCF.m" e clique em executar.
4. Mapeando a eficiência do FRET para os níveis de cAMP
- Abra o arquivo de programação chamado 'Mapping_FRET Efficiency_to_cAMP_concentration.m' e clique em executar na janela principal.
NOTA: O arquivo está disponível no site bioimaging e biosystems (ver nota abaixo de 3.2.1). Este arquivo lê imagens de eficiência FRET em escala de cinza e as converte em níveis cAMP com base em uma curva característica. Esta curva característica usa uma relação cAMP-para-FRET documentada na literatura15,36 que é descrita pela equação de Hill (a terceira equação mostrada abaixo). No entanto, Kd da sonda em células intactas é difícil de estimar e assumimos que era 1 μM em nossos cálculos. Assim, os resultados são mostrados em função de Kd. (i.e., [cAMP] = x* Kd). Equações usadas para medir a eficiência do FRET e mapear os níveis de FRET para cAMP são mostradas abaixo: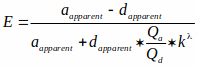
Onde E é a eficiência FRET, e umaparente e daparente são intensidades de pixels não misturadas de imagens aceitadoras e doadoras, respectivamente.
Qa e Qd são rendimentos quânticos de aceitador e doador. Note que Qa e Qd cancelam quando a equação para kλ é incorporada na equação de eficiência fret, kλ é um fator de correção: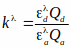
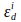 e
e 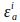 são coeficientes de extinção de doador e aceitador no comprimento de onda de excitação do doador, i (405nm).
são coeficientes de extinção de doador e aceitador no comprimento de onda de excitação do doador, i (405nm).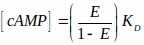
E é eficiência FRET e KD = Dissociação constante = 1 μM. - Navegue e selecione a primeira imagem FRET em escala cinza (salva na etapa 3.3.5) e clique em OK.
- Abra as imagens FRET/cAMP para inspecionar a distribuição de sinais cAMP em três dimensões.
Resultados
Este protocolo descreve o uso de abordagens hiperespectrais de imagem e análise de FRET para medir gradientes cAMP em três dimensões espaciais em células vivas. Existem várias etapas fundamentais envolvidas na geração desses resultados, para as quais é necessária uma atenção cuidadosa ao analisar e quantificar os dados. Essas etapas-chave incluem a construção de uma biblioteca espectral apropriada, descompramento espectral de fundo, limiar para identificar bordas celulares e cálculos de eficiência do FRET....
Discussão
O desenvolvimento de biosensores FRET permitiu a medição e visualização de sinais nucleotídeos cíclicos em células únicas, e há grande promessa de visualização de eventos de sinalização subcelular13,22,37,38. No entanto, o uso de biosensores FRET apresenta várias limitações, incluindo as características de baixo sinal-ruído de muitos repórteres fret à base de proteína fluor...
Divulgações
Os Drs. Leavesley e Rich revelam interesse financeiro em uma empresa iniciante, SpectraCyte, LLC, que foi formada para comercializar tecnologias de imagem espectral. No entanto, todos os procedimentos descritos neste protocolo foram realizados utilizando produtos comercialmente disponíveis não associados à SpectraCyte, LLC.
Agradecimentos
Os autores gostariam de reconhecer o Dr. Kees Jalink (Instituto de Câncer dos Países Baixos e o Centro de Microscopia Avançada van Leeuwenhoek, Amsterdã, Holanda) por nos fornecer o biosensor H188 cAMP FRET e Kenny Trinh (College of Engineering, University of South Alabama) para ajuda técnica na redução do tempo de tempo que leva para executar nossos scripts de programação desenvolvidos sob medida.
Os autores gostariam de reconhecer as fontes de financiamento: American Heart Association (16PRE27130004), National Science Foundation; (1725937) NIH, S100D020149, S10RR027535, R01HL058506, P01HL066299).
Materiais
| Name | Company | Catalog Number | Comments |
| Attofluor Cell Chamber | Invitrogen | A7816 | Attofluor contains steel cell chambers and a rubber O-ring. Cell chamber holds the coverslip and O-ring provides a lock in mechanism to hold the buffer in cell chamber with out leakage |
| Dimethyl Sulfoxide (DMSO) | Fisher Scientific | BP231-100 | Solvent used to prepare stock solution forskolin. |
| DRAQ5 Fluoroscent Probe Solution | Thermo Scientific | 62252 | Nuclear label |
| Dulbecco Modified Eagle Medium (DMEM) | Gibco | 11965-092 | Contains nutrients and growth factors for the cells to grow and divide in the culture dishes. |
| Fetal Bovine Serum (FBS) | Sigma | F6178 | Growth factor suppliment that is added to culture medium, DMEM |
| Forskolin | Sigma | F3917 | Adenyly cyclase activator. |
| H188 Cyclic AMP FRET biosensor | Netherland Cancer Institute, Dr. K. Jalink | Gift | Plasmid encoding Turquoise (donor fluorophore), Venus (acceptor fluorophore), and binding domain obtained from Epac. |
| Image J | image.net | Free download | Another image processing platform used to extact spectral information and image processing. |
| Integrating Sphere | Ocean Optics | FOIS-1 | Used to measure illumination intensity of the laser line at different laser intensities (?). |
| Laminin Mouse Protein, Natural | Invitrogen | 23017-015 | Coverslips are coated with laminin and this helps in cell attachment, growth and motility of the cell. |
| Lipofectamine 3000 Transfection Kit | Invitrogen | L3000-015 | Transfection reagent used to transfect cells with H188 FRET biosensor |
| MATLAB | Mathworks | R2019a | Image processing operations (linear unmixing and FRET efficiency calculations) are performed by writing custom programs in MATLAB programming environment |
| Nikon A1R confocal microscope | Nikon Instruments | Nikon A1R | Spectral image acquisition is performed using confocal microscope. |
| Nikon Elements Software | Nikon Instruments | Software dongle | used to export and handle nd2 image files (multidimensional image files) that are aquired using Nikon A1R |
| NIST-Traceable Calibration Lamp | Ocean Optics | LS-1-CAL-INT | A lamp with a known spectrum for use as a standard |
| PBS pH 7.4 (1X) | Gibco | 10010-023 | coomonly used buffer suring cell culture |
| Pulmonary Microvascular Endothelial Cells (PMVECs) | In house - Cell culture core, Univeristy of South Alabama | Isolated from Rat pulmonary microvasculature | PMVECs form inner lining of a blood vessel. |
| Penicillin-Streptomycin (10,000 U/ml) | Gibco | 15140-122 | antibiotics are added to culture medum to prevent contamination of the cells. |
| Pre-Cleaned Gold Seal Micro Slides | Clay Adams | 3010 | Microscope slides used for cell fixation |
| ProLong Diamond Antifade Mounting Media | Invitrogen | P36961 | If samples are fixed using antifade mountant, then the later protects fluoroscent dyes and chromophores from fading. |
| Spectrometer | Ocean Optics | QE65000 | Used to measure spectral response of the light source (?) |
| Trypsin-EDTA (0.25%) | Gibco | 25200-056 | Digests the protein-protein bond between the cell and cell matrix and helps to disscociate and lift the cells during cell plating. |
| Tyrodes Buffer | Made in-house | Made in-house | Tyrodes buffer is used to make working solutions and to maintain cells in aqueous solution during image acquisition. |
| 6 Well Cell Culture Plate | Corning | 3506 | Laminin coated coverslips are placed in 6-well culture dish (one coverlisps/well). Cells along with medium is added into each well. |
| 25 mm Round Microscope Cover Slips | Fisher Scientific | 12545102 | Cells were grown on round glass coverslips |
| 60X Ojective | Nikon Instruments | Plan Apo VC 60X/1.2 WI ∞/0.15-0.18 WD 0.27 | water immersion and commonly used objective for cells |
Referências
- Corbin, J. D., Sugden, P. H., Lincoln, T. M., Keely, S. L. Compartmentalization of adenosine 3':5'-monophosphate and adenosine 3':5'-monophosphate-dependent protein kinase in heart tissue. The Journal of Biological Chemistry. 252, 3854-3861 (1977).
- Terrin, A., et al. PGE1 stimulation of HEK293 cells generates multiple contiguous domains with different [cAMP]: role of compartmentalized phosphodiesterases. The Journal of Cell Biology. 175, 441-451 (2006).
- Bacskai, B. J., et al. Spatially resolved dynamics of cAMP and protein kinase A subunits in Aplysia sensory neurons. Science. 260, 222-226 (1993).
- Iancu, R. V., Ramamurthy, G., Harvey, R. D. Spatial and temporal aspects of cAMP signalling in cardiac myocytes. Clinical and Experimental Pharmacology & Physiology. 35, 1343-1348 (2008).
- Brunton, L. L., Hayes, J. S., Mayer, S. E. Functional compartmentation of cyclic AMP and protein kinase in heart. Advances in Cyclic Nucleotide Research. 14, 391-397 (1981).
- Hohl, C. M., Li, Q. Compartmentation of cAMP in adult canine ventricular myocytes. Relation to single-cell free Ca2+ transients. Circulation Research. 69, 1369-1379 (1991).
- Rich, T. C., et al. A uniform extracellular stimulus triggers distinct cAMP signals in different compartments of a simple cell. Proceedings of the National Academy of Sciences of the United States of America. 98, 13049-13054 (2001).
- Sayner, S. L., Alexeyev, M., Dessauer, C. W., Stevens, T. Soluble adenylyl cyclase reveals the significance of cAMP compartmentation on pulmonary microvascular endothelial cell barrier. Circulation Research. 98, 675-681 (2006).
- Rich, T. C., Tse, T. E., Rohan, J. G., Schaack, J., Karpen, J. W. In vivo assessment of local phosphodiesterase activity using tailored cyclic nucleotide-gated channels as cAMP sensors. The Journal of General Physiology. 118, 63-78 (2001).
- Blackman, B. E., et al. PDE4D and PDE4B function in distinct subcellular compartments in mouse embryonic fibroblasts. Journal of Biological Chemistry. 286, 12590-12601 (2011).
- Sayner, S. L., et al. Paradoxical cAMP-induced lung endothelial hyperpermeability revealed by Pseudomonas aeruginosa ExoY. Circulation Research. 95, 196-203 (2004).
- Klarenbeek, J., Jalink, K. Detecting cAMP with an EPAC-based FRET sensor in single living cells. Methods in Molecular Biology. 1071, 49-58 (2014).
- Surdo, N. C., et al. FRET biosensor uncovers cAMP nano-domains at β-adrenergic targets that dictate precise tuning of cardiac contractility. Nature Communications. 8, 15031 (2017).
- Ponsioen, B., et al. Detecting cAMP-induced Epac activation by fluorescence resonance energy transfer: Epac as a novel cAMP indicator. EMBO Reports. 5, 1176-1180 (2004).
- Vogel, S. S., Thaler, C., Koushik, S. V. Fanciful FRET. Science's STKE. 2006, (2006).
- Clegg, R. M. The History of FRET: From conception through the labors of birth. Reviews in Fluorescence. 3, (2006).
- Giepmans, B. N. G., Adams, S. R., Ellisman, M. H., Tsien, R. Y. The fluorescent toolbox for assessing protein location and function. Science. 312, 217-224 (2006).
- Manzella-Lapeira, J., Brzostowski, J. A. Imaging protein-protein interactions by Förster resonance energy transfer (FRET) microscopy in live cells. Current Protocols in Protein Science. 93, 58 (2018).
- Cooper, D. M. F., Mons, N., Karpen, J. W. Adenylyl cyclases and the interaction between calcium and cAMP signalling. Nature. 374, 421-424 (1995).
- Sassone-Corsi, P. Coupling gene expression to cAMP signalling: role of CREB and CREM. The International Journal of Biochemistry & Cell Biology. 30, 27-38 (1998).
- Rebhun, L. I. Cyclic nucleotides, calcium, and cell division. International Review of Cytology. 49, 1-54 (1977).
- Klarenbeek, J., Goedhart, J., Van Batenburg, A., Groenewald, D., Jalink, K. Fourth-generation Epac-based FRET sensors for cAMP feature exceptional brightness, photostability and dynamic range: characterization of dedicated sensors for FLIM, for ratiometry and with high affinity. PLOS ONE. 10, 0122513 (2015).
- Leavesley, S. J., Rich, T. C. FRET: signals hidden within the noise. Cytometry Part A. 85, 918-920 (2014).
- Rich, T. C., Webb, K. J., Leavesley, S. J. Can we decipher the information content contained within cyclic nucleotide signals. The Journal of General Physiology. 143, 17-27 (2014).
- Annamdevula, N. S., et al. Spectral imaging of FRET-based sensors reveals sustained cAMP gradients in three spatial dimensions. Cytometry Part A. 93 (10), 1029-1038 (2018).
- Leavesley, S. J., Britain, A. L., Cichon, L. K., Nikolaev, V. O., Rich, T. C. Assessing FRET using spectral techniques. Cytometry Part A. 83, 898-912 (2013).
- Leavesley, S. J., Rich, T. C. Overcoming limitations of FRET measurements. Cytometry Part A. 89, 325-327 (2016).
- Fink, D. J. Monitoring Earcths Resources from Space. Technology Review. 75, 32-41 (1973).
- Goetz, A. F. H., Vane, G., Solomon, J. E., Rock, B. N. Imaging Spectrometry for Earth Remote Sensing. Science. 228, 1147-1153 (1985).
- Bücherl, C. A., Bader, A., Westphal, A. H., Laptenok, S. P., Borst, J. W. FRET-FLIM applications in plant systems. Protoplasma. 251, 383-394 (2014).
- Chen, Y., Mauldin, J. P., Day, R. N., Periasamy, A. Characterization of spectral FRET imaging microscopy for monitoring nuclear protein interactions. Journal of Microscopy. 228, 139-152 (2007).
- Zimmermann, T., Rietdorf, J., Girod, A., Georget, V., Pepperkok, R. Spectral imaging and linear un-mixing enables improved FRET efficiency with a novel GFP2-YFP FRET pair. FEBS Letters. 531, 245-249 (2002).
- Griswold, J. R., Annamdevula, N., Deal, J., Rich, T., Leavesley, S. Estimating FRET Efficiency using Excitation-Scanning Hyperspectral Imaging. Biophysical Journal. 112, 586 (2017).
- Favreau, P. F., et al. Excitation-scanning hyperspectral imaging microscope. Journal of Biomedical Optics. 19, 046010 (2014).
- King, J., et al. Structural and functional characteristics of lung macro- and microvascular endothelial cell phenotypes. Microvascular Research. 67, 139-151 (2004).
- Thaler, C., Koushik, S. V., Blank, P. S., Vogel, S. S. Quantitative multiphoton spectral imaging and its use for measuring resonance energy transfer. Biophysical Journal. 89, 2736-2749 (2005).
- Agarwal, S. R., et al. Compartmentalized cAMP signaling associated with lipid raft and non-raft membrane domains in adult ventricular myocytes. Frontiers in Pharmacology. 9, 332 (2018).
- Johnstone, T. B., Agarwal, S. R., Harvey, R. D., Ostrom, R. S. cAMP signaling compartmentation: Adenylyl cyclases as anchors of dynamic signaling complexes. Mol Pharmacol. , (2017).
- Zhang, J., Li, H., Chai, L., Zhang, L., Qu, J., Chen, T. Quantitative FRET measurement using emission-spectral unmixing with independent excitation crosstalk correction. Journal of Microscopy. 257, 104-116 (2015).
- Zhang, J., Lin, F., Chai, L., Wei, L., Chen, T. IIem-spFRET: improved Iem-spFRET method for robust FRET measurement. Journal of Biomedical Optics. 21, 105003 (2016).
- Levy, S., et al. SpRET: highly sensitive and reliable spectral measurement of absolute FRET efficiency. Microscopy and Microanalysis. 17, 176-190 (2011).
- West, S. J., et al. Hyperspectral Measurements Allow Separation of FRET Signals from Non-Uniform Background Fluorescence. Biophysical Journal. 112, 453 (2017).
- Annamdevula, N. S., et al. An approach for characterizing and comparing hyperspectral microscopy systems. Sensors. 13, 9267-9293 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados