Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Mesure des distributions cAMP à 3 dimensions dans des cellules vivantes à l’aide de l’imagerie et de l’analyse FRET hyperspectrales en 4 dimensions (x, y, z et λ)
Dans cet article
Résumé
En raison du faible rapport signal/bruit (SNR) inhérent aux capteurs basés sur le transfert d’énergie par résonance Fӧrster (FRET), la mesure des signaux cAMP a été difficile, en particulier dans trois dimensions spatiales. Nous décrivons ici une méthodologie d’imagerie et d’analyse FRET hyperspectrale qui permet de mesurer la distribution de l’AMPc en trois dimensions spatiales.
Résumé
L’AMP cyclique est un deuxième messager impliqué dans un large éventail d’activités cellulaires et physiologiques. Plusieurs études suggèrent que les signaux cAMP sont compartimentés et que le compartimentation contribue à la spécificité de la signalisation dans la voie de signalisation cAMP. Le développement de biocapteurs basés sur le transfert d’énergie par résonance de Fӧrster (FRET) a renforcé la capacité de mesurer et de visualiser les signaux cAMP dans les cellules. Cependant, ces mesures sont souvent limitées à deux dimensions spatiales, ce qui peut entraîner une mauvaise interprétation des données. À ce jour, il n’y a eu que des mesures très limitées des signaux cAMP dans trois dimensions spatiales (x, y et z), en raison des limitations techniques liées à l’utilisation des capteurs FRET qui présentent intrinsèquement un faible rapport signal/bruit (SNR). En outre, les approches traditionnelles d’imagerie par filtre sont souvent inefficaces pour mesurer avec précision les signaux cAMP dans les régions subcellulaires localisées en raison d’une gamme de facteurs, notamment la diaphonie spectrale, la force limitée du signal et l’autofluorescence. Pour surmonter ces limites et permettre aux biocapteurs à base de FRET d’être utilisés avec plusieurs fluorophores, nous avons développé des approches d’imagerie et d’analyse FRET hyperspectrales qui fournissent une spécificité spectrale pour le calcul des efficacités FRET et la capacité de séparer spectralement les signaux FRET de l’autofluorescence confondante et / ou des signaux d’étiquettes fluorescentes supplémentaires. Nous présentons ici la méthodologie de mise en œuvre de l’imagerie FRET hyperspectrale ainsi que la nécessité de construire une bibliothèque spectrale appropriée qui n’est ni sous-échantillonnée ni suréchantillonnée pour effectuer un démélange spectral. Bien que nous présentions cette méthodologie pour la mesure des distributions tridimensionnelles de l’AMPc dans les cellules endothéliales microvasculaires pulmonaires (PMVEC), cette méthodologie pourrait être utilisée pour étudier les distributions spatiales de l’AMPc dans une gamme de types de cellules.
Introduction
L’adénosine monophosphate cyclique (AMPc) est un deuxième messager impliqué dans les processus cellulaires et physiologiques clés, notamment la division cellulaire, l’afflux de calcium, la transcription des gènes et la transduction du signal. Un nombre croissant de preuves suggère l’existence de compartiments cAMP dans la cellule à travers lesquels la spécificité de signalisation est atteinte1,2,3,4,5,6,7. Jusqu’à récemment, la compartimentation de l’AMPc était déduite sur la base d’effets physiologiques ou cellulaires distincts induits par différents agonistes des récepteurs couplés G8,9,10,11. Plus récemment, les sondes d’imagerie de fluorescence basées sur FRET ont fourni de nouvelles approches pour la mesure et l’observation directes des signaux cAMP dans une cellule12,13,14.
Le transfert d’énergie par résonance de Förster (FRET) est un phénomène physique dans lequel le transfert d’énergie se produit entre les molécules donneuses et acceptrices de manière non radiative lorsque les molécules sont à proximité15,16. Avec le développement d’indicateurs fluorescents basés sur FRET, ce phénomène physique a été utilisé dans des applications biologiques pour étudier les interactions protéine-protéine17,la colocalisationprotéique 18,la signalisation Ca+2 19,l’expressiongénique 20,la division cellulaire21 et la signalisation nucléotidique cyclique. Les indicateurs cAMP basés sur FRET se composent généralement d’un domaine de liaison cAMP, d’un fluorophore donneur et d’un fluorophore accepteur22. Par exemple, le capteur H188 cAMP12,22 utilisé dans cette méthodologie consiste en un domaine de liaison cAMP obtenu à partir d’Epac, pris en sandwich entre les fluorophores Turquoise (donneur) et Vénus (accepteur). Dans des conditions basales (non liées), la Turquoise et Vénus sont dans une orientation telle que fret se produit entre les fluorophores. Lors de la liaison de l’AMPc au domaine de liaison, un changement conformationnel se produit de sorte que turquoise et Vénus se séparent, ce qui entraîne une diminution de FRET.
Les approches d’imagerie basées sur FRET offrent un outil prometteur pour étudier et visualiser les signaux cAMP au sein d’une cellule. Cependant, les techniques actuelles d’imagerie microscopique basées sur FRET ne réussissent souvent que partiellement à obtenir une force de signal suffisante pour mesurer FRET avec une clarté spatiale subcellulaire. Cela est dû à plusieurs facteurs, notamment la force limitée du signal de nombreux rapporteurs FRET, le haut niveau de précision requis pour quantifier avec précision les changements dans l’efficacité FRET et la présence de facteurs de confusion, tels que l’autofluorescence cellulaire23,24. Le résultat est souvent une image FRET qui est en proie à un faible SNR, ce qui rend la visualisation des changements subcellulaires dans FRET très difficile. En outre, l’étude des signaux cAMP localisés dans l’espace a été presque exclusivement effectuée dans seulement deux dimensions spatiales et la distribution axiale cAMP a rarement été considérée comme25. Cela est probablement dû au fait qu’un faible SNR a entravé la capacité de mesurer et de visualiser les gradients de l’AMPc dans trois dimensions spatiales. Pour surmonter les limites de l’utilisation de capteurs FRET à faible SNR, nous avons mis en œuvre des approches d’imagerie et d’analyse hyperspectrales pour mesurer FRET dans des cellules individuelles25,26,27.
Des approches d’imagerie hyperspectrale ont été développées par la NASA pour différencier les objets terrestres présents dans les images satellites28,29. Ces techniques ont depuis été traduites dans le domaine de la microscopie à fluorescence30,avec plusieurs systèmes de microscope confocal commerciaux offrant des détecteurs spectraux. Dans l’imagerie de fluorescence traditionnelle (non spectrale), l’échantillon est excité à l’aide d’un filtre passe-bande ou d’une ligne laser, et l’émission est collectée à l’aide d’un deuxième filtre passe-bande, souvent sélectionné pour correspondre à la longueur d’onde d’émission maximale du ou des fluorophores. En revanche, les approches d’imagerie hyperspectrale cherchent à échantillonner un profil spectral complet de l’émission de fluorescence26,31,32ou de l’excitation33 ,34à des intervalles de longueur d’onde spécifiques. Dans nos études précédentes, nous avons montré que les approches d’imagerie et d’analyse hyperspectrales peuvent offrir une meilleure quantification des signaux FRET dans les cellules par rapport aux techniques traditionnelles d’imagerie FRET basées sur desfiltres 26. Nous présentons ici une méthodologie pour effectuer une imagerie et une analyse FRET hyperspectrales en 4 dimensions (x, y, z et λ) afin de mesurer et de visualiser les distributions cAMP en trois dimensions spatiales. Ces approches ont permis de visualiser les gradients spatiaux de l’AMPc induits par les agonistes dans des cellules individuelles25. Fait intéressant, selon l’agoniste, des gradients d’AMPc peuvent être apparents dans les cellules. La méthodologie présentée ici utilise le démélange spectral du fond non uniforme et de l’autofluorescence cellulaire pour améliorer la précision des mesures FRET. Bien que cette méthodologie soit démontrée dans les cellules endothéliales microvasculaires pulmonaires (PMVEC) à l’aide d’un biocapteur FRET cAMP, la méthodologie pourrait facilement être modifiée pour être utilisée avec d’autres rapporteurs FRET ou des lignées cellulaires alternatives.
Protocole
Ce protocole suit les procédures approuvées par le Comité institutionnel de soins et d’utilisation des animaux de l’Université de South Alabama.
1. Préparation des cellules, des échantillons et des réactifs pour l’imagerie
- Isoler les cellules endothéliales microvasculaires pulmonaires (PMVEC) du rat comme décrit précédemment35.
REMARQUE: Les cellules ont été isolées et cultivées par le noyau de culture cellulaire de l’Université de South Alabama, Mobile, AL sur des paraboles de culture cellulaire de 100 mm. - Ensemencez les PMVÉC isolés sur des couvercles de verre rond de 25 mm et laissez-les se développer dans l’incubateur à 37 °C jusqu’à ce que les cellules atteignent au moins 80 % de confluence (au moins 24 heures).
REMARQUE: Les cellules et le type de cellule peuvent varier d’une étude à l’autre et, par conséquent, les procédures de culture cellulaire spécifiques aux cellules doivent être suivies pour ensemencer et cultiver des cellules. Le protocole d’ensemencement et de culture cellulaire utilisé dans ces études est disponible en tant qu’information supplémentaire dans le fichier intitulé « Culture et transfection supplémentaires File_Cell ». - Transfecter les PMVEC avec un biocapteur FRET et incuber pendant 48 heures à 37 °C.
REMARQUE : Le protocole de transfection des PMVÉC est également décrit dans le fichier d’information supplémentaire intitulé « Supplément File_Cell culture et transfection ». - Le jour de l’imagerie, réchauffez le tampon de Tyrode à 37 °C au bain-marie.
REMARQUE: Le tampon de Tyrode se compose de 145 mM naCl, 4 mM KCl, 10 mM HEPES, 10 mM glucose, 1 mM MgCl2 et 1 mM CaCl2 - Montez un couvercle contenant des cellules transfectées dans une chambre à cellules et fixez le dessus avec un joint de montage pour éviter les fuites.
- Essuyez le bas du couvercle à l’aide d’un essuyage de tâche délicate pour nettoyer tout excès de support ou de cellules adhérentes.
- Ajouter 800 μL de tampon de travail et 4 μL d’étiquette nucléaire de 5 mM à la chambre cellulaire et basculer doucement pendant 5 à 10 secondes.
REMARQUE: Lorsque vous ajoutez des solutions tampons ou réactives à des couvercles montés dans la chambre cellulaire, assurez-vous d’ajouter la solution doucement et sur le côté de la chambre cellulaire afin de ne pas déloger les cellules adhérentes. L’ajout de 4 μL d’étiquette nucléaire de 5 mM à 800 μL de tampon donne une concentration finale de 25 μM d’étiquette nucléaire. Pour les cellules peu adhérentes telles que les cellules HEK293, mélangez d’abord l’étiquette nucléaire et le tampon dans un flacon, puis ajoutez-les aux couvercles montés dans la chambre cellulaire. Cela empêchera de soulever les cellules du couvercle. - Couvrir la chambre cellulaire avec du papier d’aluminium pour protéger de la lumière et incuber pendant 10 minutes à température ambiante.
- Préparation du réactif : Ajouter 1 μL de forskoline de 50 mM à 199 μL de tampon. Cela produira une concentration finale de forskoline de 50 μM lorsqu’elle sera ajoutée aux cellules préparées avec 800 μL de tampon. 1 μL de DMSO dans 199 μL de tampon doit également être préparé pour être utilisé comme contrôle du véhicule.
REMARQUE: Dans ces études, la forskoline est utilisée comme activateur de l’adénylyl cyclase pour stimuler la production d’AMPc. Si vous le souhaitez, cette méthodologie peut facilement être modifiée pour permettre un traitement avec des réactifs alternatifs pour stimuler ou inhiber l’adénylyl cyclase, les phosphodiestérases, etc.
2. Acquisition d’images
- Utilisez un microscope confocal équipé d’un détecteur spectral.
REMARQUE: Toutes les étapes d’acquisition d’images décrites ici ont été développées à l’aide d’un système de microscope Nikon A1R disponible dans le commerce. Ces étapes peuvent devoir être ajustées si vous utilisez un autre microscope spectral. Assurez-vous que tout l’équipement est allumé au moins 30 minutes avant le début de l’expérience afin d’atteindre des conditions de fonctionnement stables. - Sélectionnez l’objectif d’immersion dans l’eau 60x et ajoutez une goutte d’eau à l’objectif.
REMARQUE: Pour l’imagerie à haute résolution de cellules vivantes, il est recommandé d’utiliser un objectif à ouverture numérique élevée. Veuillez consulter la Liste des matériaux pour obtenir des renseignements sur l’objectif utilisé dans ces études. - Placez la chambre à cellules chargée (à partir de l’étape 1.7) sur l’étage du microscope.
- Sélectionnez le jeu de filtres DFT (DAPI/FITC/TRITC) en réglant le bouton de filtre sur le côté droit du microscope.
- Utilisez le microscope en mode grand champ de fluorescence à l’aide des oculaires pour sélectionner un champ de vision contenant des cellules exprimant le capteur FRET cAMP.
REMARQUE: Assurez-vous que l’intensité moyenne du signal FRET à la longueur d’onde maximale d’émission du donneur ou de l’accepteur dans la cellule sélectionnée est d’au moins 100 unités d’intensité (UA) ou d’au moins 4 fois le signal de base d’une région sans cellules d’expression. Cela peut être confirmé à l’aide de la visionneuse de profil de spectre disponible dans le logiciel NIS Elements. Lorsque vous recherchez une cellule avec un bon signal, il est conseillé de se débarrasser des cellules excessivement brillantes (elles peuvent être compromises). - Ouvrez le logiciel NIS, passez en mode confocal, déverrouillez le bouton de verrouillage laser et cliquez sur Live.
- Utilisez le bouton de mise au point pour faire la mise au point sur les cellules en regardant l’aperçu à l’écran.
- Configurez les paramètres de périphérique, d’acquisition et de pile z dans le logiciel, comme décrit ci-dessous.
- Paramètres d’acquisition :
REMARQUE: Les paramètres d’acquisition de l’appareil photo et de l’appareil peuvent être appliqués à l’aide d’une image précédemment acquise. Ouvrez l’image, faites un clic droit et sélectionnez Réutiliser les paramètres de l’appareil photo.- Ouvrez le menu des paramètres A1, cochez les cases correspondant aux lignes laser 405 nm et 561 nm, sélectionnez SD pour le détecteur spectral, sélectionnez 10 pour la résolution et 31 pour les canaux.
REMARQUE: Le menu des paramètres A1 s’affiche sous la forme d’une petite icône d’engrenage dans le coin supérieur gauche de la fenêtre Paramètres A1 Plus. Le laser 405 nm est utilisé pour l’excitation du donneur et le laser 561 nm est utilisé pour l’excitation de l’étiquette nucléaire. - Définissez la plage de longueurs d’onde (410 – 730 nm) en sélectionnant les valeurs de longueur d’onde de début et de fin.
- Cliquez sur l’icône binning/skip dans le menu des paramètres A1 et cochez la case numérotée 15, puis cliquez sur OK dans le menu des paramètres A1.
REMARQUE: Il s’agit de supprimer le canal de longueur d’onde qui correspond au laser d’excitation de 561 nm (il s’agit généralement du canal de longueurd’onde 15). Il est important de ne pas utiliser cette bande de longueur d’onde pour éviter un signal artificiellement bas, qui peut créer un artefact spectral. Le signal est plus faible dans cette bande en raison du doigt mécanique qui recouvre l’élément détecteur pour le protéger des dommages laser. - Réglez les intensités laser à 8% et 2% pour les lasers 405 nm et 561 nm, respectivement, Si Hv (gain du détecteur) à 149 et un rayon de sténopé de 2,4 unités de disque aéré (UA).
REMARQUE: Les intensités laser peuvent devoir être ajustées en fonction de l’âge de l’instrument et de l’état des lasers. Si vous ajustez les intensités laser entre différents échantillons ou groupes expérimentaux, il est important de maintenir le même rapport d’intensités laser (p. ex., 8:2). De plus, il est important de sélectionner une intensité laser qui n’est pas autant brillante qu’elle crée un photobleachage rapide. Le gain du détecteur doit être ajusté pour maximiser l’intensité du signal tout en minimisant le bruit du détecteur. Pour ces études, un gain de 149 a été utilisé. Une taille de sténopé de 2,4 UA a été choisie comme équilibre entre l’acquisition d’images avec un rapport signal/bruit (SNR) suffisant et le maintien de la section optique (confocalité). Une augmentation de la taille du sténopé augmente le SNR mais diminue la confocalité. - Réglez la vitesse de balayage sur 0,25 image spectrale par seconde, sélectionnez l’icône correspondant à unidirectionnel pour la direction de numérisation, entrez 4 pour le nombre et définissez 1024 x 1024 pour la taille de numérisation.
REMARQUE: Les signaux FRET sont faibles et une vitesse de balayage lente est souvent requise. En utilisant une vitesse de balayage de 0,25, l’acquisition d’une pile z spectrale est terminée en ~ 3 minutes. La vitesse de balayage peut être augmentée ou diminuée en fonction des fluorophores utilisés. Par exemple, pour les fluorophores plus brillants comme l’eGFP, une vitesse de balayage plus rapide (2 images / seconde) peut être utilisée. Le nombre saisi sous le nombre correspond à une valeur moyenne d’image de 4, ce qui contribue à la réduction du bruit lors de l’acquisition d’images. Pour les échantillons très stables et où le temps n’est pas une contrainte, des valeurs de moyenne plus élevées (jusqu’à 16) peuvent être utilisées pour obtenir des images avec un SNR amélioré.
- Ouvrez le menu des paramètres A1, cochez les cases correspondant aux lignes laser 405 nm et 561 nm, sélectionnez SD pour le détecteur spectral, sélectionnez 10 pour la résolution et 31 pour les canaux.
- Définissez les paramètres d’acquisition z-stack :
REMARQUE : Les valeurs saisies à l’étape 2.10 devront peut-être être ajustées pour tenir compte des changements dans la liaison ou la concentration des étiquettes fluorescentes, le type d’étiquette, le nombre d’étiquettes utilisées, la lignée cellulaire et d’autres changements dans la préparation des échantillons qui peuvent avoir une incidence sur la densité de marquage cellulaire et/ou l’autofluorescence cellulaire. Lors de l’ajustement des paramètres d’acquisition, il faut veiller à obtenir un SNR suffisant tout en minimisant le photobleachage. De plus, lors de la configuration d’un test FRET spectral, il faut veiller à ce que les paramètres fonctionnent bien dans tous les groupes de traitement. Il est conseillé d’effectuer un essai de chaque groupe de traitement avec les paramètres proposés pour s’assurer que le SNR est suffisant et que le photobleachage est minimisé.- Ouvrez la fenêtre d’acquisition ND en cliquant sur Afficher → contrôle d’acquisition → acquisition ND.
- Entrez le chemin/la destination et un nom de fichier pour enregistrer le fichier ND dans la fenêtre contextuelle.
- Cochez la case correspondant à la série z.
- Cliquez sur en direct dans la fenêtre Paramètres A1 Plus. Cela ouvrira une fenêtre de visualisation en direct.
- Ajustez le bouton de mise au point du microscope pour sélectionner le haut de la cellule et cliquez sur Haut dans la fenêtre d’acquisition ND pour définir la position actuelle comme étant en haut.
REMARQUE: Il est suggéré de se concentrer légèrement au-dessus du haut de la cellule pour s’assurer que toute la cellule est échantillonnée dans la série z. - Réglez le bouton de mise au point du microscope pour sélectionner le bas de la cellule et cliquez sur Bas dans la fenêtre d’acquisition ND pour définir la position actuelle en bas.
REMARQUE: Focalisation légèrement en dessous du bas de la cellule pour s’assurer que toute la cellule est échantillonnée. - Entrez 1 μm pour la taille de l’étape, sélectionnez haut-bas pour la direction z-scan et cliquez sur exécuter dans la fenêtre Acquisition ND pour acquérir une pile z.
REMARQUE : La taille des étapes détermine le nombre de tranches z qui seront acquises en fonction des emplacements supérieur et inférieur (c.-à-d. la distance parcourue). Une taille de pas de 1 μm a été choisie comme compromis entre la vitesse d’imagerie, l’échantillonnage sur l’axe Z et le photobleachage. L’utilisation du diamètre confocal du sténopé de 2,4 UA et de l’objectif d’immersion dans l’eau 60x a permis d’avoir une épaisseur de section optique de 1,73 μm. Par conséquent, une taille d’étape de 1 μm est légèrement inférieure aux critères d’échantillonnage de Nyquist, mais il s’agit d’un compromis qui a été fait pour réduire le temps nécessaire à l’acquisition d’une pile z. Pour les échantillons très stables, pour lesquels la vitesse n’est pas critique, un pas d’axe z plus petit et éventuellement un diamètre de sténopé confocal plus petit peuvent être utilisés pour augmenter la résolution de l’axe Z. Bottom-top devrait donner des résultats similaires et peut être utilisé pour évaluer les effets du photobleaching qui peuvent se produire pendant le scan z.
- Configurez le perfect focus system (PFS) si disponible :
REMARQUE: PFS permet au système de compenser les fluctuations de la profondeur focale lors de l’acquisition de l’image. Les étapes suivantes peuvent être utilisées pour configurer PFS, et ces étapes peuvent varier légèrement en fonction de la version du Nikon A1R et de la version de NIS Elements utilisée.- Mettez en surbrillance le mode symétrique défini par l’icône de plage dans la fenêtre d’acquisition ND.
- Allumez le bouton PFS sur la face avant du microscope (assurez-vous que le bouton du miroir dichroïque situé sur la section située sous l’étage d’échantillonnage est « in »).
- Redéfinissez le haut (rotation dans le sens inverse des aiguilles d’une montre) et le bas (rotation dans le sens des aiguilles d’une montre) à l’aide du bouton sur la face avant du contrôleur de décalage PFS.
- Définissez une position z relative/profondeur z en cliquant sur « relative » dans la fenêtre d’acquisition ND.
- Cliquez sur mémoire sur la face avant du microscope afin que le logiciel mémorise la profondeur z relative.
- Une fois l’acquisition de la pile z terminée, ajoutez doucement le réactif souhaité (forskoline ou contrôle du véhicule) à l’aide d’une pipette et attendez 10 minutes.
REMARQUE: Ajouter le réactif très doucement afin de ne pas déranger les cellules ou déplacer la position de la chambre cellulaire dans l’étage XY du microscope; il est utile de vérifier dans une vue ou une image en direct ultérieure que le champ de vision ne s’est pas déplacé lors de l’ajout de réactif. Le temps d’attente de 10 minutes est pour que le traitement à la forskoline prenne effet. Si un autre traitement est utilisé, le temps d’attente peut devoir être ajusté. - Après 10 minutes, modifiez le nom du fichier et cliquez sur Exécuter dans la fenêtre d’acquisition ND.
- Répétez les étapes 2.11 à 2.13 comme indiqué ci-dessus pour au moins 5 couvercles afin d’obtenir des résultats suffisants pour l’analyse statistique (n = 5 pour chaque groupe de traitement – forskoline et contrôle du véhicule).
- Préparer des échantillons et des blancs d’échantillons pour construire la bibliothèque spectrale et acquérir des images spectrales en utilisant des paramètres d’acquisition similaires à ceux décrits aux étapes 2.9 et 2.10.
3. Analyse d’images
REMARQUE: Ces images seront utilisées pour construire une bibliothèque spectrale contenant les spectres purs de tous les membres finaux individuels présents dans l’étude. Les membres finaux de la bibliothèque spectrale peuvent varier d’une étude à l’autre si différents fluorophores sont utilisés. Une procédure détaillée pour construire la bibliothèque spectrale est fournie dans un fichier d’information supplémentaire nommé « Bibliothèque File_Spectral supplémentaire ». Ici, nous décrivons l’exportation de données vers des fichiers .tiff, le démélange spectral linéaire, les mesures d’efficacité FRET, la reconstruction tridimensionnelle et l’estimation des niveaux cAMP. L’analyse d’images peut être effectuée à l’aide de différentes plates-formes d’analyse et de programmation d’images telles que ImageJ, Python, MATLAB ou CellProfiler. Dans ces études, des scripts MATLAB ont été utilisés.
- Exporter des données d’image :
- Créez de nouveaux dossiers avec le même nom de fichier correspondant aux images spectrales z-stack acquises aux étapes 2.13 et 2.14.
REMARQUE : Les étapes suivantes décrites pour exporter des données sont spécifiques à NIS Elements AR version 4.30.01. Ces étapes peuvent varier légèrement en fonction de la version du logiciel. - Cliquez sur Fichier, ce qui ouvrira une fenêtre de fichier. Parcourez et sélectionnez le fichier d’image spectrale acquis à l’étape 2.12 et cliquez sur Ouvrir.
- Une fois le fichier chargé, cliquez sur Fichier→ Importer/Exporter→ Exporter le document ND.
- Dans la fenêtre contextuelle : parcourez et sélectionnez le dossier créé à l’étape 3.1.1, sélectionnez Format d’image balisé (TIF) pour Type de fichier, puis sélectionnez Image mono pour chaque canal et Conserver la profondeur de bits.
REMARQUE : Le préfixe de fichier sera pré-généré ; modifier cette valeur pour plus de commodité. L’ordre d’index changera en fonction des canaux sélectionnés et doit afficher « z, c » pour l’indexation en fonction de l’emplacement de la tranche z en premier et du numéro de bande de longueur d’onde en second. Assurez-vous que les cases correspondant à Appliquer des LUT ou Insérer des superpositions ou Utiliser des noms de points ne sont pas sélectionnées. - Cliquez sur Exporter pour exporter les fichiers tiff vers un dossier de destination en tant que fichiers tiff individuels.
- Répétez les étapes 3.1.2 à 3.1.5 pour exporter les fichiers d’images spectrales acquis à l’étape 2.13.
- Créez de nouveaux dossiers avec le même nom de fichier correspondant aux images spectrales z-stack acquises aux étapes 2.13 et 2.14.
- Démélange spectral linéaire :
- Ouvrez le logiciel de programmation.
REMARQUE: Un script de programmation développé sur mesure pour démixer des images spectrales brutes est fourni sur le site Web de l’Université de South Alabama BioImaging and BioSystems, sous l’onglet Ressources (https://www.southalabama.edu/centers/bioimaging/resources.html). - Ouvrez le fichier intitulé « Linear Unmixing.m » et cliquez sur le bouton Exécuter dans la barre d’outils de l’éditeur.
- Parcourez et sélectionnez le dossier contenant la séquence de fichiers *.tif exporté générée par le logiciel NIS Elements.
- Cliquez sur OK pour continuer, ce qui ouvrira une nouvelle fenêtre appelée Longueur d’onde et Z-Slice.
- Copiez le nom de fichier du premier fichier (sans tranche z ni numéro de canal) dans le dossier sélectionné à l’étape 3.2.4 et collez-le dans la première étape de la boîte de dialogue intitulée « Entrez le nom de l’image ».
- Entrez le nombre de canaux dans la deuxième étape intitulée « Entrez le nombre de bandes de longueur d’onde », le nombre de tranches z dans la troisième étape intitulée « Entrez le nombre de tranches Z » et cliquez sur OK.
REMARQUE: Le nombre de bandes de longueurs d’onde peut changer si des modifications sont apportées aux paramètres d’acquisition, telles que le réglage de la plage de longueurs d’onde ou de la taille du pas de longueur d’onde. Le nombre de tranches Z peut également changer en fonction de la hauteur de la cellule. - Parcourez et sélectionnez le fichier de longueur d’onde appelé « Wavelength.mat » dans la fenêtre contextuelle intitulée « Sélectionnez le fichier d’informations sur la longueur d’onde » et cliquez sur Ouvrir.
- Parcourez et sélectionnez le fichier « Library.mat » dans la nouvelle fenêtre contextuelle intitulée « Sélectionnez le fichier de bibliothèque spectrale », cliquez sur Ouvrir et attendez que le démixage des tranches soit terminé.
REMARQUE: Le fichier Library.mat est un fichier contenant des spectres purs pour chaque fluorophore d’extrémité ainsi que l’autofluorescence cellulaire et les signatures spectrales de fond. Dans ce cas, les fluorophores des membres finaux comprennent la turquoise, Vénus et DRAQ5. Les signatures spectrales d’arrière-plan comprennent l’autofluorescence cellulaire ou matricielle, la fluorescence du glissement de couverture et la diffraction du glissement de couverture. Le fichier Wavelength.mat est un fichier contenant des informations sur le canal de longueur d’onde utilisé pour acquérir l’image spectrale. Un exemple de fichier de bibliothèque et de fichier de longueur d’onde est disponible sur le site Web de Bioimaging and Biosystems (voir la note sous 3.2.1). Pour plus d’informations sur la façon de générer une bibliothèque spectrale et des fichiers de longueur d’onde, reportez-vous au fichier d’informations supplémentaire nommé « Bibliothèque File_Spectral supplémentaire ». Les images non mélangées correspondant à chaque tranche z seront enregistrées dans le dossier appelé « Unmixed » créé pendant le processus de démixage dans le dossier sélectionné à l’étape 3.2.3.
- Ouvrez le logiciel de programmation.
- Calcul de l’efficacité FRET :
- Ouvrez le script de programmation appelé « multiFRRCF.m » et cliquez sur exécuter.
REMARQUE: Ce fichier de programmation est disponible sur le site Web de l’Université de South Alabama Bioimaging and Biosystems (voir la note à l’étape 3.2.1). - Entrez le nombre d’essais expérimentaux à analyser dans la boîte de dialogue contextuelle appelée « combien de dossiers à reselicer » et cliquez sur OK.
REMARQUE: Les données d’image de chaque expérience doivent être enregistrées dans un dossier d’image non mélangé séparé. Cette étape permet simplement au code d’analyse de passer en boucle sur de nombreux dossiers afin de gagner du temps. - Parcourez et sélectionnez le(s) dossier(s) non mélangé(s) et cliquez sur OK.
REMARQUE: Le nombre de fois que la fenêtre contextuelle « Rechercher un dossier » s’ouvre dépend du nombre entré dans la boîte de dialogue « Combien de dossiers à reslicer » à l’étape précédente. Parcourez et sélectionnez les dossiers l’un après l’autre. - Dans la nouvelle fenêtre contextuelle, entrez les informations suivantes dans les zones respectives: le facteur de mise à l’échelle est de 12,4, le seuil est de 5,6, X, Y et la fréquence Z sont respectivement de 5, 5 et 1, et l’algorithme de lissage est gaussien.
REMARQUE: Le facteur de mise à l’échelle est une valeur en pixels / μm et sera utilisé pour mettre à l’échelle l’échantillonnage de la direction Z à celle de la direction XY. Le facteur de mise à l’échelle est obtenu à partir de la taille en pixels de l’image, qui est généralement fournie sous forme de métadonnées dans l’image pour la plupart des systèmes de microscope confocal. Par exemple, si l’image est acquise avec un espacement de 0,08 μm/pixel, le facteur de mise à l’échelle doit être de 12,5 pixels/μm. La valeur de seuil sera utilisée pour seuilr les images et générer un masque binaire de la cellule. Nous avons créé une liste de valeurs optimales en fonction de l’intensité du donneur d’image + accepteur. Utilisez 4,5 comme valeur seuil si l’image a une intensité donneur+accepteur lumineuse et un arrière-plan faible, une valeur comprise entre 5,6 et 6,5 pour les images n’ayant qu’une intensité donneur+accepteur modérée et/ou un arrière-plan supérieur, et une valeur de 7,5 et plus pour les images ayant une intensité donneur+accepteur inférieure à l’arrière-plan. La valeur de fréquence correspond à l’intervalle, en nombre de pixels, auquel le découpage est effectué dans les étapes suivantes. Par exemple, si la profondeur Z de la cellule est de 17 μm avec une taille de pas de 1 μm et qu’un facteur d’échelle de 12,5 pixels/μm est utilisé dans la direction XY, la profondeur du jeu de données d’image en 3 dimensions sera rééchantillonnée à 212 pixels (direction Z). En fonction de la valeur de fréquence Z saisie (par exemple, 1 pixel), l’ensemble de données d’image en 3 dimensions sera retranché en commençant par le haut de l’ensemble de données d’image, puis en se déplaçant par incréments de 1 pixel vers le bas. Il en résulte 212 images resélquées. Si un intervalle de valeur de fréquence plus grand était entré pour la fréquence Z, moins d’images resélquées seraient générées. Les images resélquées sont enregistrées dans les étapes suivantes. - Cliquez sur Exécuter et attendez que toutes les mesures FRET et le découpage soient effectués.
REMARQUE : Un dossier distinct est créé dans le répertoire parent dans lequel sont enregistrées les images d’efficacité FRET en niveaux de gris rééliquées et les images d’efficacité FRET colorées (une carte de couleurs appliquée). Par exemple, toutes les images FRET en niveaux de gris et colormap réélquées dans la direction X (plan YZ) sont enregistrées dans un dossier appelé « Resliced_XFRET ». - Répétez l’analyse avec des paramètres similaires pour toutes les expériences – avant et après les traitements à la forskoline et les commandes du véhicule.
REMARQUE : Les étapes mentionnées à la section 3.3 décrivent les valeurs à entrer pour le script de programmation d’analyse FRET personnalisé afin de générer des données d’image FRET en 3 dimensions. Toutefois, ce script exécute plusieurs opérations en cours d’exécution, notamment : le chargement des données d’image, la création de piles d’images, le lissage, les calculs d’efficacité FRET, la création et l’application d’un masque de bordure de cellule, la reconstruction d’image en 3 dimensions, le redécoupage d’images en 3 dimensions à des intervalles spécifiés (fréquences), l’application d’une carte couleur pour visualiser les modifications FRET et l’enregistrement des données d’image resélquées dans le même répertoire. Des détails supplémentaires ont été inclus sous forme de commentaires dans le script du programme.
- Ouvrez le script de programmation appelé « multiFRRCF.m » et cliquez sur exécuter.
4. Mappage de l’efficacité FRET aux niveaux cAMP
- Ouvrez le fichier de programmation nommé 'Mapping_FRET Efficiency_to_cAMP_concentration.m' et cliquez sur exécuter dans la fenêtre principale.
REMARQUE : Le fichier est disponible sur le site Web de BioImaging and BioSystems (voir la note sous 3.2.1). Ce fichier lit les images d’efficacité FRET en niveaux de gris et les convertit en niveaux cAMP en fonction d’une courbe caractéristique. Cette courbe caractéristique utilise une relation cAMP-FRET documentée dans la littérature15,36 qui est décrite par l’équation de Hill (la troisième équation ci-dessous). Cependant, Kd de la sonde dans des cellules intactes est difficile à estimer et nous avons supposé qu’il était de 1 μM dans nos calculs. Par conséquent, les résultats sont présentés en fonction de Kd. (c.-à-d. [AMPc] = x* Kd). Les équations utilisées pour mesurer l’efficacité fret et mapper fret aux niveaux cAMP sont présentées ci-dessous :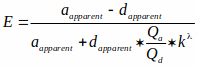
Où E est l’efficacité FRET, et unapparent et dapparent sont des intensités de pixels non mélangées d’images accepteurs et donneuses, respectivement.
Qa et Qd sont des rendements quantiques de l’accepteur et du donneur. Notez que Qa et Qds’annulent lorsque l’équation de kλ est incorporée dans l’équation d’efficacité FRET, kλ est un facteur de correction :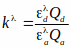
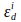 et
et 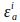 sont des coefficients d’extinction du donneur et de l’accepteur à la longueur d’onde d’excitation du donneur, i (405 nm).
sont des coefficients d’extinction du donneur et de l’accepteur à la longueur d’onde d’excitation du donneur, i (405 nm).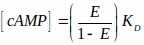
E est l’efficacité FRET et KD = constante de dissociation = 1 μM. - Naviguez et sélectionnez la première image FRET en niveaux de gris (enregistrée à l’étape 3.3.5) et cliquez sur OK.
- Ouvrez les images FRET/cAMP pour inspecter la distribution des signaux cAMP en trois dimensions.
Résultats
Ce protocole décrit l’utilisation d’approches hyperspectrales d’imagerie et d’analyse FRET pour mesurer les gradients cAMP dans trois dimensions spatiales dans des cellules vivantes. La génération de ces résultats implique plusieurs étapes clés, pour lesquelles une attention particulière est requise lors de l’analyse et de la quantification des données. Ces étapes clés comprennent la construction d’une bibliothèque spectrale appropriée, le démélange spectral de fond, le seuillage pour identifier...
Discussion
Le développement des biocapteurs FRET a permis la mesure et la visualisation des signaux nucléotidiques cycliques dans des cellules individuelles, et il existe de grandes promesses pour visualiser les événements de signalisation subcellulaire13,22,37,38. Cependant, l’utilisation de biocapteurs FRET présente plusieurs limites, notamment les faibles caractéristiques signal-bruit de nombre...
Déclarations de divulgation
Les Drs Leavesley et Rich divulguent leur intérêt financier dans une entreprise en démarrage, SpectraCyte, LLC, qui a été créée pour commercialiser des technologies d’imagerie spectrale. Cependant, toutes les procédures décrites dans ce protocole ont été effectuées à l’aide de produits disponibles dans le commerce non associés à SpectraCyte, LLC.
Remerciements
Les auteurs tiennent à remercier le Dr Kees Jalink (Institut néerlandais du cancer et centre van Leeuwenhoek pour la microscopie avancée, Amsterdam, Pays-Bas) pour nous avoir fourni le biocapteur H188 cAMP FRET et Kenny Trinh (College of Engineering, Université de South Alabama) pour leur aide technique afin de réduire le temps nécessaire à l’exécution de nos scripts de programmation développés sur mesure.
Les auteurs tiennent à remercier les sources de financement suivantes : American Heart Association (16PRE27130004), National Science Foundation; (1725937) NIH, S100D020149, S10RR027535, R01HL058506, P01HL066299).
matériels
| Name | Company | Catalog Number | Comments |
| Attofluor Cell Chamber | Invitrogen | A7816 | Attofluor contains steel cell chambers and a rubber O-ring. Cell chamber holds the coverslip and O-ring provides a lock in mechanism to hold the buffer in cell chamber with out leakage |
| Dimethyl Sulfoxide (DMSO) | Fisher Scientific | BP231-100 | Solvent used to prepare stock solution forskolin. |
| DRAQ5 Fluoroscent Probe Solution | Thermo Scientific | 62252 | Nuclear label |
| Dulbecco Modified Eagle Medium (DMEM) | Gibco | 11965-092 | Contains nutrients and growth factors for the cells to grow and divide in the culture dishes. |
| Fetal Bovine Serum (FBS) | Sigma | F6178 | Growth factor suppliment that is added to culture medium, DMEM |
| Forskolin | Sigma | F3917 | Adenyly cyclase activator. |
| H188 Cyclic AMP FRET biosensor | Netherland Cancer Institute, Dr. K. Jalink | Gift | Plasmid encoding Turquoise (donor fluorophore), Venus (acceptor fluorophore), and binding domain obtained from Epac. |
| Image J | image.net | Free download | Another image processing platform used to extact spectral information and image processing. |
| Integrating Sphere | Ocean Optics | FOIS-1 | Used to measure illumination intensity of the laser line at different laser intensities (?). |
| Laminin Mouse Protein, Natural | Invitrogen | 23017-015 | Coverslips are coated with laminin and this helps in cell attachment, growth and motility of the cell. |
| Lipofectamine 3000 Transfection Kit | Invitrogen | L3000-015 | Transfection reagent used to transfect cells with H188 FRET biosensor |
| MATLAB | Mathworks | R2019a | Image processing operations (linear unmixing and FRET efficiency calculations) are performed by writing custom programs in MATLAB programming environment |
| Nikon A1R confocal microscope | Nikon Instruments | Nikon A1R | Spectral image acquisition is performed using confocal microscope. |
| Nikon Elements Software | Nikon Instruments | Software dongle | used to export and handle nd2 image files (multidimensional image files) that are aquired using Nikon A1R |
| NIST-Traceable Calibration Lamp | Ocean Optics | LS-1-CAL-INT | A lamp with a known spectrum for use as a standard |
| PBS pH 7.4 (1X) | Gibco | 10010-023 | coomonly used buffer suring cell culture |
| Pulmonary Microvascular Endothelial Cells (PMVECs) | In house - Cell culture core, Univeristy of South Alabama | Isolated from Rat pulmonary microvasculature | PMVECs form inner lining of a blood vessel. |
| Penicillin-Streptomycin (10,000 U/ml) | Gibco | 15140-122 | antibiotics are added to culture medum to prevent contamination of the cells. |
| Pre-Cleaned Gold Seal Micro Slides | Clay Adams | 3010 | Microscope slides used for cell fixation |
| ProLong Diamond Antifade Mounting Media | Invitrogen | P36961 | If samples are fixed using antifade mountant, then the later protects fluoroscent dyes and chromophores from fading. |
| Spectrometer | Ocean Optics | QE65000 | Used to measure spectral response of the light source (?) |
| Trypsin-EDTA (0.25%) | Gibco | 25200-056 | Digests the protein-protein bond between the cell and cell matrix and helps to disscociate and lift the cells during cell plating. |
| Tyrodes Buffer | Made in-house | Made in-house | Tyrodes buffer is used to make working solutions and to maintain cells in aqueous solution during image acquisition. |
| 6 Well Cell Culture Plate | Corning | 3506 | Laminin coated coverslips are placed in 6-well culture dish (one coverlisps/well). Cells along with medium is added into each well. |
| 25 mm Round Microscope Cover Slips | Fisher Scientific | 12545102 | Cells were grown on round glass coverslips |
| 60X Ojective | Nikon Instruments | Plan Apo VC 60X/1.2 WI ∞/0.15-0.18 WD 0.27 | water immersion and commonly used objective for cells |
Références
- Corbin, J. D., Sugden, P. H., Lincoln, T. M., Keely, S. L. Compartmentalization of adenosine 3':5'-monophosphate and adenosine 3':5'-monophosphate-dependent protein kinase in heart tissue. The Journal of Biological Chemistry. 252, 3854-3861 (1977).
- Terrin, A., et al. PGE1 stimulation of HEK293 cells generates multiple contiguous domains with different [cAMP]: role of compartmentalized phosphodiesterases. The Journal of Cell Biology. 175, 441-451 (2006).
- Bacskai, B. J., et al. Spatially resolved dynamics of cAMP and protein kinase A subunits in Aplysia sensory neurons. Science. 260, 222-226 (1993).
- Iancu, R. V., Ramamurthy, G., Harvey, R. D. Spatial and temporal aspects of cAMP signalling in cardiac myocytes. Clinical and Experimental Pharmacology & Physiology. 35, 1343-1348 (2008).
- Brunton, L. L., Hayes, J. S., Mayer, S. E. Functional compartmentation of cyclic AMP and protein kinase in heart. Advances in Cyclic Nucleotide Research. 14, 391-397 (1981).
- Hohl, C. M., Li, Q. Compartmentation of cAMP in adult canine ventricular myocytes. Relation to single-cell free Ca2+ transients. Circulation Research. 69, 1369-1379 (1991).
- Rich, T. C., et al. A uniform extracellular stimulus triggers distinct cAMP signals in different compartments of a simple cell. Proceedings of the National Academy of Sciences of the United States of America. 98, 13049-13054 (2001).
- Sayner, S. L., Alexeyev, M., Dessauer, C. W., Stevens, T. Soluble adenylyl cyclase reveals the significance of cAMP compartmentation on pulmonary microvascular endothelial cell barrier. Circulation Research. 98, 675-681 (2006).
- Rich, T. C., Tse, T. E., Rohan, J. G., Schaack, J., Karpen, J. W. In vivo assessment of local phosphodiesterase activity using tailored cyclic nucleotide-gated channels as cAMP sensors. The Journal of General Physiology. 118, 63-78 (2001).
- Blackman, B. E., et al. PDE4D and PDE4B function in distinct subcellular compartments in mouse embryonic fibroblasts. Journal of Biological Chemistry. 286, 12590-12601 (2011).
- Sayner, S. L., et al. Paradoxical cAMP-induced lung endothelial hyperpermeability revealed by Pseudomonas aeruginosa ExoY. Circulation Research. 95, 196-203 (2004).
- Klarenbeek, J., Jalink, K. Detecting cAMP with an EPAC-based FRET sensor in single living cells. Methods in Molecular Biology. 1071, 49-58 (2014).
- Surdo, N. C., et al. FRET biosensor uncovers cAMP nano-domains at β-adrenergic targets that dictate precise tuning of cardiac contractility. Nature Communications. 8, 15031 (2017).
- Ponsioen, B., et al. Detecting cAMP-induced Epac activation by fluorescence resonance energy transfer: Epac as a novel cAMP indicator. EMBO Reports. 5, 1176-1180 (2004).
- Vogel, S. S., Thaler, C., Koushik, S. V. Fanciful FRET. Science's STKE. 2006, (2006).
- Clegg, R. M. The History of FRET: From conception through the labors of birth. Reviews in Fluorescence. 3, (2006).
- Giepmans, B. N. G., Adams, S. R., Ellisman, M. H., Tsien, R. Y. The fluorescent toolbox for assessing protein location and function. Science. 312, 217-224 (2006).
- Manzella-Lapeira, J., Brzostowski, J. A. Imaging protein-protein interactions by Förster resonance energy transfer (FRET) microscopy in live cells. Current Protocols in Protein Science. 93, 58 (2018).
- Cooper, D. M. F., Mons, N., Karpen, J. W. Adenylyl cyclases and the interaction between calcium and cAMP signalling. Nature. 374, 421-424 (1995).
- Sassone-Corsi, P. Coupling gene expression to cAMP signalling: role of CREB and CREM. The International Journal of Biochemistry & Cell Biology. 30, 27-38 (1998).
- Rebhun, L. I. Cyclic nucleotides, calcium, and cell division. International Review of Cytology. 49, 1-54 (1977).
- Klarenbeek, J., Goedhart, J., Van Batenburg, A., Groenewald, D., Jalink, K. Fourth-generation Epac-based FRET sensors for cAMP feature exceptional brightness, photostability and dynamic range: characterization of dedicated sensors for FLIM, for ratiometry and with high affinity. PLOS ONE. 10, 0122513 (2015).
- Leavesley, S. J., Rich, T. C. FRET: signals hidden within the noise. Cytometry Part A. 85, 918-920 (2014).
- Rich, T. C., Webb, K. J., Leavesley, S. J. Can we decipher the information content contained within cyclic nucleotide signals. The Journal of General Physiology. 143, 17-27 (2014).
- Annamdevula, N. S., et al. Spectral imaging of FRET-based sensors reveals sustained cAMP gradients in three spatial dimensions. Cytometry Part A. 93 (10), 1029-1038 (2018).
- Leavesley, S. J., Britain, A. L., Cichon, L. K., Nikolaev, V. O., Rich, T. C. Assessing FRET using spectral techniques. Cytometry Part A. 83, 898-912 (2013).
- Leavesley, S. J., Rich, T. C. Overcoming limitations of FRET measurements. Cytometry Part A. 89, 325-327 (2016).
- Fink, D. J. Monitoring Earcths Resources from Space. Technology Review. 75, 32-41 (1973).
- Goetz, A. F. H., Vane, G., Solomon, J. E., Rock, B. N. Imaging Spectrometry for Earth Remote Sensing. Science. 228, 1147-1153 (1985).
- Bücherl, C. A., Bader, A., Westphal, A. H., Laptenok, S. P., Borst, J. W. FRET-FLIM applications in plant systems. Protoplasma. 251, 383-394 (2014).
- Chen, Y., Mauldin, J. P., Day, R. N., Periasamy, A. Characterization of spectral FRET imaging microscopy for monitoring nuclear protein interactions. Journal of Microscopy. 228, 139-152 (2007).
- Zimmermann, T., Rietdorf, J., Girod, A., Georget, V., Pepperkok, R. Spectral imaging and linear un-mixing enables improved FRET efficiency with a novel GFP2-YFP FRET pair. FEBS Letters. 531, 245-249 (2002).
- Griswold, J. R., Annamdevula, N., Deal, J., Rich, T., Leavesley, S. Estimating FRET Efficiency using Excitation-Scanning Hyperspectral Imaging. Biophysical Journal. 112, 586 (2017).
- Favreau, P. F., et al. Excitation-scanning hyperspectral imaging microscope. Journal of Biomedical Optics. 19, 046010 (2014).
- King, J., et al. Structural and functional characteristics of lung macro- and microvascular endothelial cell phenotypes. Microvascular Research. 67, 139-151 (2004).
- Thaler, C., Koushik, S. V., Blank, P. S., Vogel, S. S. Quantitative multiphoton spectral imaging and its use for measuring resonance energy transfer. Biophysical Journal. 89, 2736-2749 (2005).
- Agarwal, S. R., et al. Compartmentalized cAMP signaling associated with lipid raft and non-raft membrane domains in adult ventricular myocytes. Frontiers in Pharmacology. 9, 332 (2018).
- Johnstone, T. B., Agarwal, S. R., Harvey, R. D., Ostrom, R. S. cAMP signaling compartmentation: Adenylyl cyclases as anchors of dynamic signaling complexes. Mol Pharmacol. , (2017).
- Zhang, J., Li, H., Chai, L., Zhang, L., Qu, J., Chen, T. Quantitative FRET measurement using emission-spectral unmixing with independent excitation crosstalk correction. Journal of Microscopy. 257, 104-116 (2015).
- Zhang, J., Lin, F., Chai, L., Wei, L., Chen, T. IIem-spFRET: improved Iem-spFRET method for robust FRET measurement. Journal of Biomedical Optics. 21, 105003 (2016).
- Levy, S., et al. SpRET: highly sensitive and reliable spectral measurement of absolute FRET efficiency. Microscopy and Microanalysis. 17, 176-190 (2011).
- West, S. J., et al. Hyperspectral Measurements Allow Separation of FRET Signals from Non-Uniform Background Fluorescence. Biophysical Journal. 112, 453 (2017).
- Annamdevula, N. S., et al. An approach for characterizing and comparing hyperspectral microscopy systems. Sensors. 13, 9267-9293 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon