Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Lipid Membran Dinamiği ve Membran-Protein Etkileşimleri için Benzersiz Bir Prob Olarak Nötron Spin Eko Spektroskopisi
Bu Makalede
Erratum Notice
Özet
Bu makalede lipid membranlarının nötron spin eko (NSE) çalışmalarında örnek hazırlama, veri azaltma ve veri analizi protokolleri açıklanmaktadır. Lipitlerin judicious döteryum etiketlemesi, üzerinde hayati biyolojik süreçlerin meydana geldiği mezoskopik uzunluk ve zaman ölçeklerinde farklı membran dinamiklerine erişim sağlar.
Özet
Lipid bilayerler hücre zarlarının ana matrisini oluşturur ve diğer hayati hücresel süreçlerin yanı sıra besin değişimi, protein-membran etkileşimleri ve viral tomurcuklanma için birincil platformdur. Verimli biyolojik aktivite için, hücre zarları hücrenin ve bölmelerinin bütünlüğünü koruyacak kadar sert, ancak proteinler ve fonksiyonel alanlar gibi membran bileşenlerinin yayılmasına ve etkileşime girmesine izin edecek kadar akışkan olmalıdır. Elastik ve akışkan membran özelliklerinin bu hassas dengesi ve biyolojik işlev üzerindeki etkileri, mezoskopik uzunluk ve zaman ölçekleri üzerinde kolektif membran dinamiklerinin daha iyi anlaşılmasıni gerektirir, örneğin, membran deformasyonları ve protein bağlama olayları. Bu dinamik aralığı etkili bir şekilde araştırabilecek teknikler arasında nötron spin ekosu (NSE) spektroskopisi vardır. Döteryum etiketleme ile birlikte NSE, bükme ve kalınlık dalgalanmalarının yanı sıra seçilen membran özelliklerinin mezoskopik dinamiklerine doğrudan erişmek için kullanılabilir. Bu makale, NSE tekniğinin kısa bir açıklamasını sağlar ve veri toplama ve azaltma talimatlarının yanı sıra örnek hazırlama ve döterasyon şemalarının ayrıntıları da dahil olmak üzere lipozomal membranlar üzerinde NSE deneyleri gerçekleştirme prosedürlerini özetlemektedir. Makale ayrıca bükme sertlik modülü, alan sıkıştırılabilirlik modülü ve düzlem içi viskozite gibi anahtar membran parametrelerini çıkarmak için kullanılan veri analizi yöntemlerini de tanıtır. NSE çalışmalarının biyolojik önemini göstermek için, NSE tarafından araştırılan membran fenomenlerinin seçilmiş örnekleri, yani katkı maddelerinin membran bükme sertliği üzerindeki etkisi, etki alanı oluşumunun membran dalgalanmaları üzerindeki etkisi ve membran-protein etkileşimlerinin dinamik imzası tartışılmaktadır.
Giriş
Hücre zarlarının ve işlevlerinin anlaşılması son birkaç on yılda önemli ölçüde gelişti. Hücre zarlarının hücre sınırlarını tanımlayan pasif lipid bilayerleri olarak eski görüşü ve membran proteinleri1, lipid bilayerlerinin hücresel sinyalizasyon, moleküler değişim ve protein fonksiyonu da dahil olmak üzere hayati biyolojik süreçleri düzenlemede önemli bir rol oynadığı dinamik bir modele dönüştü - birkaç 2,3,4,5,6. Hücre zarlarının son derece dinamik olduğunu, sürekli yeniden şekillendirme ve moleküler yeniden dağıtımdan geçtiğinin farkına varılması, membranların denge yapılarının ötesinde bilimsel keşifler çağrısında bulunmaktadır7,8,9. Buna göre, biyolojik ve biyoinspired lipid membranlarındaki çeşitli dinamik modları incelemek için birden fazla yaklaşım geliştirilmiştir. Bugüne kadar, bu çalışmaların çoğunluğu öncelikle difüzif moleküler hareketler10 , 11,12,13 ve makroskopik şekil dalgalanmaları14,15,16, ara membran dinamiklerini anlamada önemli bir boşluk bırakarak, yani birkaç 10-100'lük lipid moleküllerinden oluşan lipid montajlarının kolektif dalgalanmalarına odaklanmıştır. Bu dinamikler, birkaç on ila birkaç 100 Å'nın uzunluk ölçekleri ve zaman içinde alt ns'den birkaç yüz ns'ye kadar olan ölçekler üzerinde meydana gelir (bkz. Şekil 1),burada mezoskopik ölçekler olarak adlandırılır. Gerçekten de bu ölçeklerde anahtar biyolojik aktivite membran seviyesinde gerçekleşir17. Bu viral tomurcuklanmaiçerir 18, kanal gating19, ve membran-protein etkileşimleri20. Ayrıca, membran proteinleri 21,22'ninenerji manzarasının, proteinlerdeki konformasyonel değişikliklerin - düzenleyici rolleri için gerekli - kolektif membran dalgalanmalarının 23'lüğünde ns zaman ölçeklerinde gerçekleştiğini gösterdiğini belirtmek önemlidir, ayrıca mezoskopik dinamiklerin hücre zarlarının biyolojik işlevindeki önemini ve biyoinspired analoglarını20. Bu makale lipid zarlarındaki iki birincil mezoskopik dinamik moda, yani bükülme dalgalanmalarına ve kalınlık dalgalanmalarına odaklanmaktadır.
Bu dalgalanma modlarını doğrudan yoklamadaki temel zorluk, standart spektroskopi yöntemlerini kullanarak mekansal ve zamansal ölçeklerine aynı anda erişmenin zorluğudur. Diğer zorluk, doğrudan temas tekniklerinin16'yıölçmek için amaçladıkları dalgalanmaları etkileyebileceğidir. Bu, biyolojik membranların bileşimsel ve yapısal karmaşıklığı ile daha da şiddetlenir24,25, lipid etki alanı oluşumu26 , 27,28,29,30ve membran asimetrisi31 ,32,33dahil olmak üzere homojen olmayan membran özellikleri ile sonuçlanır- farklı membran özelliklerinin dinamiklerini anlamak için seçici problar talep eder. Neyse ki, bu zorluklar, doğal olarak gerekli uzunluk ve zaman ölçeklerine erişen nötron spin ekosu (NSE) gibi invaziv olmayan nötron spektroskopisi yöntemleriyle aşılabilir ve fizikokimyasal ortamlarını değiştirmeden seçici membran özelliklerinin çalışmalarını daha da mümkün kılar34. Gerçekten de, son birkaç yılda NSE spektroskopisi kolektif membran dinamiklerinin benzersiz ve güçlü bir probuna dönüştü35. Lipid membranları üzerinde yapılan NSE çalışmalarından elde edilensonuçlar,lipid membranlarının mekanik36,37 ve viskoelastik38,39 özellikleri hakkında yeni içgörüler üretmiş ve biyolojik işlevdeki potansiyel rollerine yeni bir ışık tutmuş40,41.
NSE spektroskopi tekniği, ilk olarak Mezei42tarafından önerilen bir interferometrik enstrüman tasarımına dayanmaktadır Nötronlar enstrümandan geçerken nötron dönüşünün presesyonunu kontrol etmek için bir dizi spin-flipper ve manyetik bobin kullanarak. Tasarım, manyetik alan elemanlarının örnek konumuna göre manyetik olarak yansıtılmasına dayanır (Şekil 1A). Bu, nötron ve numune arasındaki enerji değişiminin yokluğunda, nötronun cihazın ilk ve ikinci yarısında ters yönlerde aynı sayıda spin precession gerçekleştirdiğini ima eder (iki pressyon bobini arasındaki π-palete dikkat edin). Sonuç olarak, nötronun son spin durumu ilk duruma göre değişmeden kalır - spin-echo olarak adlandırılan bir fenomen (Şekil 1A'dakişeffaf nötron'a bakın). Bununla birlikte, nötron numuneyle enerjik bir şekilde etkileşime girdiğinde, enerji değişimi cihazın ikinci yarısındaki spin presesyonlarının sayısını değiştirerek farklı bir son spin durumuna yol sağlar (bkz. Şekil 1A). Bu deneysel olarak kutuplaşmada bir kayıp olarak tespit edilir, bu makalede daha sonra gösterileceği gibi. NSE tekniği hakkında daha fazla bilgi için, okuyucu özel teknik makalelere atıfta bulunulmaktadır42,43,44,45.
Burada, NSE ile erişilebilen uzunluk ve zaman ölçeklerinin kabaca bir tahminini sağlamak için basitleştirilmiş bir açıklama sunulmaktadır. Uzunluk ölçekleri ulaşılabilir dalgacı transferleri aralığına göre belirlenir, Q = 4π sin φ/φ, burada 2φ saçılma açısıdır ve φ nötron dalga boyudur. Q'nun dalga boyu aralığı ve spektrometrenin ikinci kolunun dönüş derecesine göre ayarlandıklarından biri görülebilir (bkz. Şekil 1A). NSE spektrometrelerinde tipik bir Qaralığı ~0.02-2 ş-146,47ve 0.01-4 ş-1'e kadardır ve son yükseltmelerle 48,49, uzaysal ölçeklere karşılık gelen ~1-600 Å'dır. Öte yandan, erişilebilir zaman ölçeği, nötron tarafından manyetik pressyon bobinleri içinde elde edilen toplam presezyon açısından (veya fazdan) hesaplanır ve50: 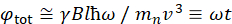 . Bu ifadede, t olarak tanımlanan Fourier
. Bu ifadede, t olarak tanımlanan Fourier 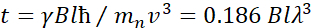
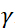 zamanıdır nötron nötron jinekagnetik oranı
zamanıdır nötron nötron jinekagnetik oranı  nerededir, bobin uzunluğudur ve
nerededir, bobin uzunluğudur ve 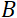 bobinin manyetik alanının gücüdür. Fourier zamanının kesinlikle enstrüman geometrisine, manyetik alan gücüne ve nötron dalga boyuna bağlı bir miktar olduğunu belirtmek gerekir. Örneğin, dalga boyu nötronları =
bobinin manyetik alanının gücüdür. Fourier zamanının kesinlikle enstrüman geometrisine, manyetik alan gücüne ve nötron dalga boyuna bağlı bir miktar olduğunu belirtmek gerekir. Örneğin, dalga boyu nötronları = 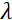 8 şve
8 şve  = 1,2 m ve
= 1,2 m ve 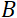 = 0,4 T'lik enstrüman ayarları kullanılarak, Fourier süresi t ~ 50 ns olarak hesaplanır. Deneysel olarak, Fourier zamanı, presesyon bobinlerindeki akımı değiştirerek (yani manyetik alan gücü) veya farklı nötron dalga boyları kullanılarak ayarlanır ve bu da ~ 1 ps ila 100 ns tipik NSE zaman ölçekleriyle sonuçlanır. Bununla birlikte, NSE spektrometrelerindeki son yükseltmeler daha uzun Fourier zamanlarına erişim sağladı, Heinz Maier-Leibnitz Zentrum51'deki J-NSE-Phoenix spektrometresinde ~400 ns'ye ve Oak Ridge National Lab 48'deki SNS-NSE spektrometresinde~400ns'ye kadar ve Institut Laue-Langevin (ILL) 49'daki IN15 NSE spektrometresinde~1.000ns'ye kadar.
= 0,4 T'lik enstrüman ayarları kullanılarak, Fourier süresi t ~ 50 ns olarak hesaplanır. Deneysel olarak, Fourier zamanı, presesyon bobinlerindeki akımı değiştirerek (yani manyetik alan gücü) veya farklı nötron dalga boyları kullanılarak ayarlanır ve bu da ~ 1 ps ila 100 ns tipik NSE zaman ölçekleriyle sonuçlanır. Bununla birlikte, NSE spektrometrelerindeki son yükseltmeler daha uzun Fourier zamanlarına erişim sağladı, Heinz Maier-Leibnitz Zentrum51'deki J-NSE-Phoenix spektrometresinde ~400 ns'ye ve Oak Ridge National Lab 48'deki SNS-NSE spektrometresinde~400ns'ye kadar ve Institut Laue-Langevin (ILL) 49'daki IN15 NSE spektrometresinde~1.000ns'ye kadar.
Membran dinamiklerinin uzunluk ve zaman ölçeğine doğrudan erişimin yanı sıra, NSE nötron izotop duyarlılığının doğal yeteneklerine sahiptir52. Özellikle, nötronların biyolojik sistemlerdeki en bol element olan hidrojenin izotoplarıyla farklı etkileşime girebilmeleri, protium döteryum ile değiştirildiğinde farklı bir nötron saçılma uzunluğu yoğunluğu,34 veya NSLD (kırılma50optik indeksine eşdeğer) ile sonuçlanır. Bu, genellikle belirli membran özelliklerini vurgulamak veya başkalarını gizlemek için kullanılan kontrast varyasyonu olarak bilinen bir yaklaşım sağlar — ikinci senaryoya kontrast eşleştirmesi denir. Kontrast değişiminin/eşleşmesinin sık uygulanması, suyun (NSLD = -0,56 × 10-6 ş-2)ağır su veya D 2 O (NSLD = 6) iledeğiştirilmesidir.4 × 10-6 ş-2) nötron sinyalini protiated lipid membranlarından yükseltmek için (NSLD ~ 0 × 10-6 ş-2). Bu yaklaşım membran yapısının çalışmalarında oldukça etkilidir, çünkü D2O'nun membranın kafa grubu bölgesine nüfuz etmesi, membran kalınlıklarının doğru bir şekilde belirlenmesine izin verir (bkz. Şekil 2A, sol panel) ve daha sofistike modeller uygulandığında farklı lipit alt gruplarının konumu53,54. Bu makalede, biyomimetik membranlarda kolektif dinamiklerin ve seçilmiş membran özelliklerinin çalışmaları için kontrast varyasyonunun kullanımına ilişkin bazı örnekler vurgulanmıştır.
Burada, NSE'nin dinamik ve fonksiyonel membran özelliklerine benzersiz içgörüler sağlamadaki etkinliği, NSE'nin model ve biyolojik olarak ilgili lipid membran sistemleri üzerinde yaptığı çalışmaların somut örnekleriyle, serbest duran membranlarda mezoscale dinamiklerine vurgu yaparak, lipozomal süspansiyonlar şeklinde gösterilmiştir. Düzlem içi membran dinamiklerinin NSE ölçümleri için okuyucu, otlatma-insidans nötron spin-echo spektroskopisi (GINSES)55,56 ve hizalanmış çok mller membran yığınlarının57 , 58,59,60ile ilgili diğer çalışmaları hakkında özel yayınlara atıfta bulunulmaktadır.
Basitlik için, bu makale iyi çalışılmış bir alan oluşturmada gösterilen üç farklı membran deuterasyon şemasını vurgulamaktadır, veya faz ayırma, lipid bilayer sistemi 1,2-dimyristoyl-sn-glisero-3-fosfokolin (DMPC) ve 1,2-distearoyl-sn-glisero-3-fosfokolin (DSPC) karışımları61,62. İki lipit, hidrokarbon zincir uzunluklarında (DMPC'de 14 karbon/kuyruk ve DSPC'de 18 karbon/kuyruk) ve jel-sıvı geçiş sıcaklıklarında (Tm, DMPC = 23 °C vs Tm, DSPC = 55 °C) bir uyumsuzluk ile karakterize edilir. Bu, DMPC:DSPC membranlarında karışımın üst ve alt geçiş sıcaklıkları arasındaki sıcaklıklarda yanal faz ayrımı ile sonuçlanır63. Burada göz önünde bulundurulan döterasyon şemaları, lipozomal membranlarda NSE ölçümlerinde erişilebilen farklı dinamik modları, yani bükme dalgalanmalarını, kalınlık dalgalanmalarını ve yanal etki alanlarının seçici bükme/kalınlık dalgalanmalarını göstermek için seçilir. Tüm lipid bileşimleri, DMPC ve DSPC'nin ticari olarak mevcut protiated ve perdeuterated varyantları kullanılarak 70:30 köstebek fraksiyonu ile hazırlanan DMPC:DSPC bilayerleri için rapor edilir. Tüm numune hazırlama adımları, D2O'da, 50 mg / mL lipit konsantrasyonuna sahip 4 mL lipozomal süspansiyona dayanmaktadır, toplam lipid kütlesi için Mtot = örnek başına 200 mg.
Protokol
1. Deney için gerekli olan döterasyon şeması
- Bükme dalgalanması ölçümleri için, D 2 O(%99,9)veya D 2 O tamponunda (örneğin,H2O yerine D2O ile hazırlanmış fosfat tamponu) tamamen protiated lipozomlar yapın. 133,4mg ile tamamen protiated DMPC (C 36 H72NO8P) ve DSPC (C44H88NO8P)
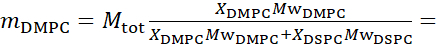 kullanın, burada XDMPC ve XDSPC, DMPC ve DSPC'nin köstebek fraksiyonlarıdır, burada sırasıyla 0.7 ve 0.3 olarak ayarlanır ve MwDMPC ve MwDSPC sırasıyla 677.9 g/mol ve 790.1 g/mol tarafından verilen azı dişleridir. Benzer şekilde, mDSPC = 66.6 mg. Bu döterasyon şeması, membran (NSLD ~ 0 × 10-6 ş-2)ile döterlenmiş tampon (NSLD ~ 6.4 × 10-6 ş-2)arasındaki saçılma kontrastını arttırır ve membran dalgalanmalarından gelen sinyali artırır (bkz. Şekil 2A sol panel).
kullanın, burada XDMPC ve XDSPC, DMPC ve DSPC'nin köstebek fraksiyonlarıdır, burada sırasıyla 0.7 ve 0.3 olarak ayarlanır ve MwDMPC ve MwDSPC sırasıyla 677.9 g/mol ve 790.1 g/mol tarafından verilen azı dişleridir. Benzer şekilde, mDSPC = 66.6 mg. Bu döterasyon şeması, membran (NSLD ~ 0 × 10-6 ş-2)ile döterlenmiş tampon (NSLD ~ 6.4 × 10-6 ş-2)arasındaki saçılma kontrastını arttırır ve membran dalgalanmalarından gelen sinyali artırır (bkz. Şekil 2A sol panel). - Belirli yanal membran özelliklerinin bükülme dinamiklerini ölçmek için, örneğin faz ayırma DMPC:DSPC membranlarındaki matris dinamikleri, protiated DMPC (C36H72NO8P) kullanın ve döterlenmiş, DSPC-d83 (C44H5NO8PD83, Mw 873.7 g/mol), böylece mDMPC = 128.8 mg ve mDSPC-d83 = 71.2 mg. Bu döterasyon şeması, istenmeyen DSPC bakımından zengin etki alanlarından saçılmayı en aza indirir ve DMPC bakımından zengin matristen bükme dalgalanmalarının seçici ölçümlerini sağlar (bkz. Şekil 2B orta).
NOT: Belirli bir kontrast eşleştirme şeması için gereken en uygun lipid döterasyonunu bulmak için, NIST Nötron Araştırmaları Merkezi64tarafından geliştirilen gibi mevcut web tabanlı saçılma uzunluğu yoğunluğu (SLD) hesap makinelerini kullanabilirsiniz. Bu web tabanlı arayüzler, çeşitli derecelerde döterasyona sahip lipitlerin SLD'lerinin ve lipit karışımlarının kolay hesaplanması için kullanıcı dostu araçlarla donatılmıştır. - Ortalama membran kalınlığı dalgalanmalarının NSE ölçümleri için (yanal kontrast olmadan), kurucu lipitlerin kuyruk deutere varyantlarını kullanın, yani, DMPC-d54 (C36H18NO8PD54, 732.3 g/mol) ve DSPC-d70 (C44H18NO8PD70), 860.1 g/mol)35,38, mDMPC-d54 = 133.0 mg ve mDSPC-d70 = 67.0 mg. Bu kontrast şeması (Şekil 2A, sağ panel) lipid kafa gruplarından saçılma sinyalini yükseltiyor (NSLD ~ 4.5 × 10-6 ş-2) kuyruk grubunu (NSLD ~ 6.4 × 10-6 Å -2)metre kalınlığındaki dalgalanmaların algılanmasını sağlayan deuterated tamponla karşılaştırarak.
- DMPC bakımından zengin matris gibi belirli membran bölmelerinin kalınlık dalgalanması çalışmaları için, DSPC bakımından zengin etki alanlarının kısırlaştırılmış arabellekle kontrastla eşleştiği ve birincil saçılma sinyalinin kuyruktan sönmüş DMPC bakımından zengin matrisin kafa grubu bölgesinden olması için, protiated DMPC lipidlerini kuyruk disuterated analoglarıyla, yani DMPC-d54 ile ikame ederek 1.2 adımda açıklanan stratejiyi kullanın.
2. Ekstrüzyon için lipid süspansiyonunun hazırlanması
- Örnek bileşimine bağlı olarak, numunedeki her bileşenin kütlesini hesaplayın. Genel bir kural olarak, birden fazla moleküler bileşene sahip örnekler için, bir bileşenin kütlesi, köstebek fraksiyonu, Xiile ağırlıklandırılmış ve M
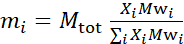 tot'un toplam kütle olduğu yerde, burada 200 mg olarak ayarlanmış, azı dişi kütlesi, Mw i , mole fraksiyonu ile ağırlıklandırılmıştır. Farklı döterasyon şemalarına sahip DMPC-DSPC lipid bilayerleri için yukarıdaki örneğe bakın.
tot'un toplam kütle olduğu yerde, burada 200 mg olarak ayarlanmış, azı dişi kütlesi, Mw i , mole fraksiyonu ile ağırlıklandırılmıştır. Farklı döterasyon şemalarına sahip DMPC-DSPC lipid bilayerleri için yukarıdaki örneğe bakın. - Dijital yarı mikro denge kullanarak, hesaplanan lipit kütlelerini (ve diğer örnek bileşenleri, örneğin proteinler, nanopartiküller vb.) tartın ve bunları bir şişeye veya yuvarlak tabanlı şişeye ekleyin - şişeyi veya şişeyi önceden tarttığını unutmayın. Tartılan bileşenleri bir davlumbazın içine manuel olarak karıştırarak çözmek için 1 mL çözücü ekleyin. Saf lipit örnekleri için kloroform veya etanol kullanın. Ek, lipid olmayan bileşenlere (örneğin, nanopartiküller) sahip numuneler için, tüm bileşenleri dağıtan ortak bir çözücü seçin.
- Küçük lipit miktarları (<10 mg) için bir stok çözeltisi hazırlayın ve gerekli hacmi karışıma pipetleyin.
NOT: Aşağıda açıklanan çözücü kurutma adımını önemli ölçüde yavaşlatacağından aşırı miktarda çözücü eklemeyin.
- Küçük lipit miktarları (<10 mg) için bir stok çözeltisi hazırlayın ve gerekli hacmi karışıma pipetleyin.
- Lipid çözeltisini, bir davlumbazın içinde, şişeyi yavaşça bir açıyla döndürürken şişeye bir inert gaz (örneğin azot, argon) hafifçe akışlayarak kurutun. Şişe duvarlarında ince bir kurutulmuş lipit filmi oluşturmak için şişeleri eğik konumda tutun, bu da kurumaya bile izin verir. Çözücü buharlaşmasını yavaşlatacak buharlaşma aracılı soğutmayı atlatmak için şişeyi aralıklı olarak 35 °C'de bir su banyosuna yerleştirin.
- Artık çözücüyü tamamen çıkarmak için şişeleri ~35 °C'de bir vakum fırınına gece boyunca yerleştirin. Doymamış lipitler için, oksidasyonu en aza indirmek için vakumları inert bir gazla temizleyin.
- Tam çözücü çıkarılmasını sağlamak için, lipid kuruduktan sonra şişeyi tartın ve ölçülen miktarda malzemenin ötesinde fazla kütle olmadığını onaylayın. Bunu, şişenin kütlesini kuruduktan sonra ölçülen kütleden çıkararak yapın. Fazla kütle varsa, numuneyi vakum altında 6 saat daha kurutun. Bu işlemi gerektiği gibi yineleyin.
- Lipid filmini 4 mL D2O veya D2O-tampon ile nemlendirerek 50 mg/mL'lik bir lipit konsantrasyonu elde edin. DMPC-DSPC karışımları gibi yüksek geçiş sıcaklıklarına sahip lipitler için, eşit karıştırma sağlamak için tamponu geçiş sıcaklığının (60 °C) üzerine ısıtın.
NOT: NSE deneyleri nispeten büyük numune hacimleri (~4 mL) gerektirdiğinden, numune başına ekstrüzyon sayısını en aza indirmek için gerekli arabelleğin yarısını, yani 2 mL'yi kullanarak numuneyi nemlendirmeyi düşünün (bkz. bölüm 3). Bu durumda, arabellek sonrası ekstrüzyonunun kalan yarısını ekleyin. Ekstrüzyonda kullanılan şırınnaların kapasitesinin 1 mL ile sınırlı olduğuna dikkat edin. Bu nedenle, 4 mL tampon ile nemlendirme dört ekstrüzyon seti gerektirir. - Lipid filmi tamamen çözünene ve şişenin duvarlarında artık görünmeyene kadar hidratlı lipit çözeltisini Vortex karıştırın. Bu aşamada, hidratlı lipitler multilameller vesikles ve mikron boyutlu çokmeller yığınları oluşturur ve süspansiyon süt beyazı görünür.
- Lipit yığınlarının kırılmasını kolaylaştırmak ve çokmelleriteyi azaltmak için, hidratlı lipit çözeltisinin şişesini tamamen donana kadar bir laboratuvar sınıfı dondurucuya (tercihen -80 °C dondurucu) yerleştirerek ve ardından lipid çözeltisi tamamen çözülene kadar şişeyi 35 °C'lik bir su banyosuna aktararak beş dondurma/çözülme döngüsü gerçekleştirin. Çözülen çözeltiyi homojen olana kadar girdap. Dört kez daha tekrarla.
NOT: Alternatif olarak, aseton ve kuru buz birleştirilerek hızlı donmaya karşı kuru bir buz banyosu hazırlanabilir.
3. Hidratlı lipit çözeltisinin ekstrüzyonu
- Ekstrüder kurulumunu iki membran desteği arasında bir polikarbonat membran kullanarak ve ek destek sağlamak için her iki tarafa iki kağıt filtre ekleyerek monte edin. Hedef lipozomal boyuta uyan gözenek boyutuna sahip bir polikarbonat membran kullanın (NSE deneyleri için ortak gözenek boyutları 50 nm ve 100 nm'dir - tipik olarak, 100 nm çapında lipozomlar daha az kısıtlı membran dalgalanmalarına izin verir, ancak eğrilik çalışmaları için daha küçük 50 nm lipozom kullanılabilir). Montajı tamamlamadan ve dış ekstrüder gövdesini sıkmadan önce polikarbonat membranın tamamen gerildiğine emin olun.
- Hava geçirmez cam şırınnalar kullanarak membran tertibatından birkaç kez~0,3mL D 2 O veya D2O-tampon geçirerek polikarbonat membranı nemlendirin. Numune hazırlamada kullanılan tamponun aynısını kullan. En az 10 dakika bekletin, ardından numuneyi tanıtmadan önce tamponu tamamen emin.
- 1 mL gaz geçirmez bir şırınnayı hazırlanan lipit çözeltisi ile doldurun ve ekstrüder cihazının bir ucuna yerleştirin. Ardından, karşı uca boş bir şırınna yerleştirin. Şırındlar ekstrüder tertibatına bağlandıktan sonra ekstrüder bloğuna yerleştirin.
- Yüksek geçiş sıcaklıklarına sahip doymuş lipitlerde olduğu gibi ekstrüzyon için yüksek sıcaklıklar gerekiyorsa (örneğin, DSPC, Tm = 55 °C), ekstrüder ısıtma bloğunu lipid geçiş sıcaklığının (örneğin, 60 °C) üzerine, ısıtma bloğunu sıcak bir tabağa yerleştirerek veya Şekil 3A'dagösterildiği gibi bir sirkülasyon banyosu kullanarak önceden ısıtın.
NOT: Bu adım, lipitlerin homojen bir şekilde karıştırılmasını sağlamak ve ekstrüzyon sırasında polikarbonat zarı yırtabilecek aşırı basınç uygulamaktan kaçınmak için çok önemlidir. Düşük geçiş sıcaklıklarına (<25 °C) sahip lipit numuneleri için ekstrüzyonu oda sıcaklığında gerçekleştirin. - Lipid çözeltisini ekstrüde etmek için, ekstrüder setini Şekil 3A'dagösterildiği gibi alüminyum/çelik çerçeveli programlanabilir bir şırınna pompasına takın. Sıcaklık kontrollü ekstrüzyonlar için, sıvı kanallı özel yapım bir ekstrüder tabanı ekleyin ve sirkülasyonlu bir su banyosuna takın.
- Şırınna pompasını, üretici el kitabını takiben 15-20 ekstrüzyon döngüsü gerçekleştirecek şekilde programla. Ekstrüde edildiğinde, lipid çözeltisinin rengi süt beyazından şeffaf opal mavisine (Şekil 3B, C), beklendiği gibi görünür ışığın dalga boyundan daha küçük olan son bir lipozomal boyutu gösterir. Şekil 3A'dagösterilen şırınna pompası türü için aşağıdaki adımları izleyin.
- Pompa ayarlarını yaparak başlayın. Hız düğmesini basılı tutun ve ekstrüzyon oranını (50,99 mL/s) girin, ardından Çap düğmesine basın ve şırınna çapını (4,606 mm) girin. Bu basamak değerini değiştirmek için ekrandaki her basamanın altındaki yukarı okları kullanın.
- Ekstrüder setini örnek şırınna ile sağa yerleştirin (bkz. Şekil 3A). Geri çekme ışığı yanana kadar Geri Çek düğmesine basın. Başlat'a basın ve numunenin sol (boş) şırınna dağıtmasını bekleyin.
- Örnek (sağda) şırınna tamamen boş olmadan hemen önce Durdur düğmesine basın. Dağıtılan hacmi kaydedin ve ekstrüzyon döngüsünü programlamak için kullanın. Ekranda 1. aşama (PH:01) görünene kadar Hız düğmesini basılı tutun. Daha önce kaydedilen dağıtılmış birimi girmek için Ses Seviyesi düğmesine basın. Bu aşamada, Geri Çekme ışığının kapalı olduğundan emin olun - bu numuneyi doğru yönde dağıtır.
- Hız düğmesine tekrar basın ve aşama 2'ye (PH:02) erişmek için en sağdaki yukarı oku kullanın. Daha önce kaydedilen dağıtılmış birimin aynı değerini girmek için Ses Seviyesi'ne basın. Bu aşamada, Geri Çek ışığı yanana kadar Geri Çek düğmesine basın - bu, numuneyi sola dağıtır.
- Bu döngüyü yinelemek için, Hız düğmesine tekrar basın ve aşama 3'e (PH:03) erişmek için en sağdaki yukarı oku kullanın. LP:SE ekranda görünene kadar Ses Düzeyi düğmesine basın ve 20 olarak ayarlayın. Bu, pompanın gerçekleştireceği döngü veya tekrar sayısıdır. Son olarak, Hız düğmesine basın, aşama 4'e (PH:04) erişin ve Durdur işlevine ulaşmak için Ses Seviyesi düğmesine basın. Pompa artık otomatik ekstrüzyon için ayarlanmıştır.
- Ekstrüzyon döngüsünü başlatmak için Başlat'a basın.
- Ekstrüde lipit süspansiyonu içeren şırınnayı temiz bir şişeye boşaltın ve depolama veya ölçümlere hazırlanın. Yüksek erime sıcaklığına sahip lipit numuneleri için numuneyi ölçülene kadar sıvı fazı geçişinin üzerinde saklayın. Aksi takdirde, numuneleri oda sıcaklığında tutun.
- Ekstrüde edilmiş numuneleri dondurmayın, çünkü donma veziklin patlamasına neden olur (süspansiyon tekrar süt beyazı olur).
4. Numuneler için NSE ölçümleri ve toplanan verilerin azaltılması
- NSE deneyinden önce, yeterli numune kalitesini sağlamak için mevcut yöntemleri kullanarak 3.7 adımından ekstrüde lipozomal örneği karakterize edin. NSE deneyleri için lipozomal süspansiyonların kalitesini değerlendirmek için kullanılabilecek potansiyel kömürleşme yöntemlerinin bir listesi, örneğin boyut dağılımı, multilamellarite, lateral membran yapısı, tartışma bölümünde yer almaktadır.
- Deneme için gereken Q aralığını ve ilgili cihaz ayarlarını belirleyin. Lipid bilayerlerin sertlik ölçümlerini bükmek için ~ (0.04 - 0.2) ş-1Q aralığı kullanın. Membran kalınlığı dalgalanmalarının çalışmaları için, membran kalınlığına karşılık gelen ~(0.04 - 0.2) ş-1 Q aralığı kullanın35,66,67.
NOT: Deney başlamadan önce cihaz bilimcisi ile deneysel kurulumu tartışın. Daha önce de belirtildiği gibi, numunenin SANS karakterizasyonu gereklidir, özellikle de seçici olarak sönmüş membranlarda olduğu gibi saçılma sinyalinin önceki bilgileri mevcut değilse. Alternatif olarak, NSE cihazında sınırlı bir Q aralığı üzerinden statik (kırınım olarak da bilinir) ölçümler çalıştırın ve bu tür ölçümlerin SANS'ye kıyasla çok daha uzun sürdüğü uyarısı. - Şırınd veya transfer pipet kullanarak, ekstrüde lipozomal süspansiyonları NSE kiriş hatlarında bulunan belirlenmiş numune hücrelerine yükleyin. Standart NSE örnek hücrelerinin 1, 2, 3 ve 4 mm kalınlıklarda geldiğini unutmayın. Tutarsız arka plan sinyalini makul bir yoğunlukta tutarken saçılma sinyalini optimize etmek için hücre kalınlığını seçin.
NOT: Genel bir kural olarak, deuterated tampondaki protiated lipozomlar için 1 veya 2 mm patlength ile örnek hücreler kullanın - daha kalın hücreler düzeltilmesi zor birden fazla saçılma etkisine neden olabilir. Daha yüksek döterasyon seviyelerine sahip lipozomlar için (örneğin, kuyruk kontrastı ile eşleşen lipozomlar veya tek protiated broşürlü asimetrik lipozomlar), numune daha büyük miktarlarda mevcutsa sayım istatistiklerini geliştirmek için daha kalın bir örnek hücre (örneğin, 3 veya 4 mm patlength) kullanmayı düşünün - bazen bu maliyet yasaklayıcı olabilir. - Arabellek için aynı örnek hücreyi hazırlayın. Lipozomal süspansiyondaki tamponun aynısını kullanın. Tampon üzerindeki ölçümler, yoğunluk normalleştirme ve arka plan (BKG) düzeltmeleri için gereklidir.
- Örnek hücrelerini NSE spektrometresinin örnek tutucusuna yerleştirin, ölçüm çalıştırmalarını programlayın ve yankı verilerini toplayın. İlk kez NSE kullanıcısı varsa ölçümlerin programlanması hakkında cihaz bilimciye danışın.
- Veri azaltma için gereken iki ek ölçüm kümesi gerçekleştirin: Çözünürlük (R) ve iletim (T) ölçümleri.
- Aynı ayarlar altında çalıştırılacak elastik saçılma referansı (örneğin karbon) üzerinde Çözünürlük(R)ölçümü gerçekleştirin; yani örnek ve tampon ölçümleri ile aynı dalgacı ve Fourier kez.
- İletilen nötron ışınının yoğunluğunu hesaplamak için numune ve tampon üzerinde iletim(T) ölçümleri gerçekleştirin (aşağıdaki adım 4.9'a bakın). İletim, nötrona bölünen numuneden veya tampondan gelen nötron sayımlarının oranı açık bir ışın için (yani boş bir numune konumuyla) sayıldığında hesaplanır.
- Toplanan verileri azaltmak için ölçümlerin gerçekleştirildiği NSE spektrometresi için özel veri azaltma yazılımını kullanın.
NOT: Farklı spektrometreler farklı yazılım veya kullanıcı arabirimleri kullanabilir. Aşağıda, Veri Analizi ve Görselleştirme Ortamı (DAVE) kullanılarak NSE veri azaltma örneği verilmiştir.68 NIST Nötron Araştırmaları Merkezi'ndeki NSE spektrometresi için özel olarak yazılmış yazılım.- DAVE yazılımını açın ve veri azaltma menüsünden NSE Verilerini Azalt'ı seçin. Birkaç açılır pencere görünecektir.
- Dosya menüsünden .echo Dosyalarını Aç'ı kullanarak veri dosyalarını farklı Q değerleri üzerinden yükleyin. Bu dosyalar spin yankı sinyalleri ile ham veri dosyalarına karşılık gelir ve dosya adında .echo uzantısına sahiptir. Dosya yükleme işlemi tamamlandıktan sonra, dosyalar kullanılabilir veri kümeleri altında gösterilir.
- Seçili dosyaya sağ tıklayın ve karşılık geldiği ölçüme göre etiketle; örneğin, Örnek, Hücre (boş hücre veya arabellek için) veya Çözünürlük.
- Veri Kümesi sekmesini kullanarak sinyal-gürültü oranını iyileştirmek için dedektör pixle'larını 2 x 2'de gruplandırın. Aynı binning'i tüm dosyalara uygulayın; örneğin Çözünürlük, Hücre ve Örnek.
- Tüm piksel grupları üzerindeki verileri inceleyin ve klavyedeki m tuşuna basarak sinyalleri zayıf olanları maskele (bkz. Şekil 4B). Aynı maskeyi fourier veya sonraki Fourier kez uygulamak için açılır pencereye erişmek için Enter tuşuna basın. Bu, veri azaltma sırasında herhangi bir noktada tek tek piksellere de uygulanabilir. Maskelenmiş pikseller yeşile döner.
- Toplanan verilerin her dedektör pikseli üzerinde bir yankı sinyali, yani faz akımı açısından kosinüs fonksiyonu şeklinde olduğundan emin olun (bkz. Şekil 4A).
NOT: Faz akımı nötron dönüşünün pressyon açısıyla orantılıdır; bu nedenle, Şekil 4A'dagösterildiği gibi faz akımını bir faz açısı olarak temsil etmek yaygındır. Darbeli kaynaklar üzerindeki ölçümler için, bir nötron darbesi içindeki olay nötron dalga boyunun bir işlevi olarak yankı sinyallerini elde etmek için verilere ek uçuş hesaplamaları süresi uygulanır. - Çözüm dosyasını takarak başlayın. Karşıya yüklenen dosya listesinden bir çözüm dosyası seçin ve dosyayı sağ tıklatın. İşlemleri Sığdır: Açılır menüden Yankıları Sığdır (Çözünürlük) seçeneğini belirleyin.
- Yankı sinyallerinin sığmasının, 4.8 adımında gereken Aparametresi de dahil olmak üzere bir dizi bağlantı parametresi sağladığından emin olun. Sığdırmalar aşağıdaki ifade kullanılarak otomatik olarak gerçekleştirilir.
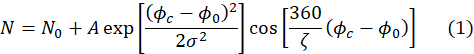
Burada, ζ yankı sinyalinin süresidir (yani Şekil 4A'dakikosinüs fonksiyonu), σ, olay nötron ışınının ortalama dalga boyu ve dalga boyu yayılımı ile belirlenen Gauss zarfının genişliğidir, Φc faz akımıdır ve Φ0, nötronların yaşadığı alan yoluna bağlı olan yankı noktasıdır50. Örnekle ilgili fiziksel bilgiler denklemdeki kosinüs işlevinin genliğiolan A'da kodlanır (1).
NOT: Gauss zarfının genişliği, enstrüman bilimcisi tarafından önceden belirlenmiş değerlere dayanmaktadır ve değiştirilmemelidir. Diğer paramterler, her piksel üzerindeki belirli yankı sinyaline takılan değişkenlerdir. - Elde edilen montaj parametrelerini, sığdırma kalitesini ve sığdırmanın ortalama kare sapmasını göstermek için her piksele tıklayarak sığdırma sonuçlarını inceleyin. Tüm dedektör üzerindeki her bağlantı parametresiyle ilişkili hatayı incelemek için Görüntü Seçenekleri'ni seçin ve ardından ilgi çekici bağlantı parametresini seçin. Bu, her piksel üzerinde bağlantı paramter değerine sahip bir harita oluşturur. Dedektör görüntüsüne sağ tıklayın. Seçili sığdırma parametresinin hata çubuğu eşlemesini gösteren bir açılır pencere görüntülenir.
- Belirli bir pikselin üzerine sığdırma tatmin edici değilse (örneğin, büyük hata çubuklarına sahip parameeterleri sığdırın), sinyali söz konusu pikselin üzerine yeniden takın. Bu pikseliseçin, Sığdır sekmesine basın ve sonra Piksele Sığdır 'abasın. Daha tatmin edici bir uyum elde etmek için Sığdırma sekmesine faz (Φ0) ve nokta (ζ) için yeni başlangıç parametreleri girin.
NOT: Takılan fazı Fourier Time'ın bir işlevi olarak çizmek yararlıdır. Bunu yapmak için, ana çizim penceresine gidin ve Aşamayı Sığdır v. Fourier Time'ı seçin. Bu arsa pürüzsüz ve sürekli olmalıdır. Bu çizimdeki süreksizlikleri inceleyin ve karşılık gelen pikselleri yeniden boyuta yeniden boyutlandırmayın.
- Karşıya yüklenen ve etiketlenen dosya listesinden ilgili dosyayı seçerek Örnek veya Hücre dosyasını azaltın.
- Tüm pikselleri inceleyin ve 4.7.5 adımında açıklandığı gibi kötü istatistiklere sahip olanları maskeleyin.
- Dosyayı sağ tıklatın ve İşlemleri Sığdır' ı seçin: Aşamaları İçe Aktar (Örnek, Hücre). Bu, aşamaları ve uygulanan maskeyi Çözüm dosyasından alır.
- Çözüm dosyası için daha önce açıklanan yordamı kullanarak yankı sinyallerini sığdırın (adım 4.7.8-4.7.10). Örnek ve Hücre dosyalarını sığdırmak için, çözünürlük sığar'dan alınan dönemin ve yankı faz noktasının değerlerini değiştirmeyin. Bu parametreler araçsal ayarlara bağlıdır ve örneklere göre değişmemelidir.
- Veri azaltma işlemine geçmeden önce, tüm veri dosyaları için ışın merkezini girin. Veri dosyasını seçin, Genel sekmesine gidin ve X ve Y ışın merkezi değerlerini girin. Bu değerler deneme sırasında kaydedilir.
- Örnek, Hücre ve Çözüm dosyalarına sığdıktan sonra, daha sonra veri analizi ve yorumlamada kullanılacak normalleştirilmiş ara saçılma işlevini hesaplayın. Bunu yapmak için, takılan dosyalar listesinden küçültülecek Örnek dosyaya sağ tıklayın ve açılır menüden I(Q) Hesapla'yı seçin. Çözünürlük ve Hücre (arabellek) dosyaları için giriş seçeneklerini ve Q yaylarının sayısını içeren bir pencere görüntülenir (bkz. adım 4.9). Gerekli tüm bilgiye girdikten sonra Tamam düğmesine basın. Sonuçlar yeni bir pencerede görünecektir.
NOT: Veri azaltma, normalleştirilmiş ara saçılma işlevi69'uelde etmek için aşağıdaki denkleme göre gerçekleştirilir.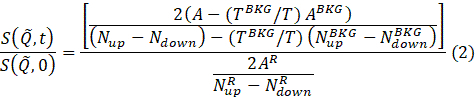
burada t Fourier zamanı, Nyukarı ve Naşağı, spin-flip olmayan ve spin-flip yapılandırmalarındaki nötron sayılarıdır (sırasıyla π/2-flipper kapalı ve π-flipper kapalı ve açık olarak ölçülür) ve üst simgeler, BKG ve R, sırasıyla 4.4 ve 4.6 adımlarında tanımlandığı gibi arka plan ve çözünürlük ölçümlerine karşılık gelir. Işın polarizasyonunun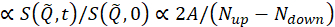 nötron ve numune arasındaki enerji değişimi nedeniyle spin durumunda değiştiğini unutmayın polarizasyonda bir düşüş olarak algılanır (birlikten).
nötron ve numune arasındaki enerji değişimi nedeniyle spin durumunda değiştiğini unutmayın polarizasyonda bir düşüş olarak algılanır (birlikten).
- Son olarak, normalleştirilmiş ara saçılma işlevinin Q bağımlılığını elde etmek için dedektör piksellerini Şekil 4B'de gösterildiği gibi Q-yayları halinde gruplandırın, S(Q,t) / S(Q,0). Bu teknik olarak veri binning olarak adlandırılır ve örneğin, örnekten sayım istatistikleri ve verilerin gruplanmış pikseller üzerinde beklenen standart sapması dikkate alınarak akıllıca yapılmalıdır.
- Güçlü saçılma örnekleri için, ortaya çıkan ara saçılma işlevi S ( Q ,t) / S(Q,0) üzerinde makul hata çubuklarını korurken dedektörü daha fazlaQarkına bölün. Bu, daha fazla Q veri noktası verir ve aşağıda açıklanan veri analizi prosedürü için önemlidir. Zayıf saçılan numuneler için, aşırı binning'in zayıf çürüme sinyallerine, yani S ( Q ,t) / S(Q,0) üzerindeki büyük hata çubuklarına neden olduğunu ve bunun büyük belirsizliklere neden olabileceğini unutmayın.
5. Veri analizi ve yorumlanması
- Yukarıdaki veri azaltmadan elde edilen normalleştirilmiş ara saçılma fonksiyonları olan S(Q,t) / S(Q,0), 2/370'likbir germe üslü ile gerilmiş bir üstel işleve uyarlayın.
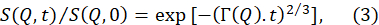
NOT: Bu uyumlara bir örnek Şekil 5B'de verilmiştir. S(Q,t) / S(Q,0) denklemine sığar (3) Q'ya bağımlı gevşeme oranlarını ΓQ) verir. - Q'nunbir işlevi olarak Γ ( Q ) çizin ve ilgili membran parametrelerini çıkarmak için uygun bir modele uygun.
Sonuçlar
Bükme dalgalanmalarına erişen NSE çalışmaları genellikle ~ (0,04 - 0,2) ş-1Q aralığı üzerinden gerçekleştirilir. Bu Q aralığı, membran kalınlığı ile bükme dinamiklerinin hakim olduğu lipozomal yarıçap arasındaki orta uzunluk ölçeklerine karşılık gelir. Genişletilmiş bir Q aralığı üzerinden ölçüm, lipozomal difüzyon ve intramembran dinamikleri de dahil olmak üzere ek dinamik modlara erişim sağlayabilir. NSE tarafından erişilen membran dinamiklerindeki çapraz geçi?...
Tartışmalar
NSE, lipid zarlarının mezoskopik dinamiklerinin çeşitli koşullar altında ölçülmesinde güçlü ve benzersiz bir tekniktir. NSE'nin etkin kullanımı numune kalitesine, nötron kontrastı ve belirli bir örnek için araştırılabilen erişilebilir dinamiklerin aralığına bağlıdır. Bu nedenle, başarılı NSE denemeleri gerçekleştirmek ve yüksek kaliteli veri toplamak için birkaç kritik adım gereklidir. Bir NSE deneyi sırasında nötron ışın süresinin etkili kullanımını sağlamanın önemli bir...
Açıklamalar
Yazarlar çıkar çatışması olmadığını ve açıklayacak hiçbir şeyi olmadığını beyan eder.
Teşekkürler
R. Ashkar, M.Nagao'ya teşekkür ediyor, L.-R. Stingaciu ve P. Zolnierczuk birçok yararlı tartışma ve kendi kiriş hatlarında NSE deneyleri ile sık sık yardım için. Yazarlar NIST ve ORNL'de nötron spin eko spektrometrelerinin kullanıldığını kabul eder. NIST'deki NSE spektrometresi, Ulusal Standartlar ve Teknoloji Enstitüsü ile Ulusal Bilim Vakfı arasında anlaşma no. DMR-1508249. ORNL'nin Spallation Nötron Kaynağı'ndaki NSE spektrometresi, ABD Enerji Bakanlığı Temel Enerji Bilimleri Ofisi Bilimsel Kullanıcı Tesisleri Bölümü tarafından desteklenmektedir. Oak Ridge Ulusal Laboratuvarı, ABD DOE Sözleşme No. DE-AC05-00OR22725.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Chloroform (biotech grade) | Sigma Aldrich | 496189 | Biotech. grade, ≥99.8%, contains 0.5-1.0% ethanol as stabilizer |
| Circulating water bath | Julabo | SE-12 | Heating Circulator with smart pump, programmable temperature settings, and external sensor connection for measurement and control |
| Deuterium Oxide | Cambridge Isotopes Laboratories | DLM-4 | Deuterated water; Heavy water (D2O) (D, 99.9%) |
| Digital Semi-Microbalance | Mettler Toledo | MS105 | Semi-micro balance with 120 g capacity, 0.01 mg readability, high resolution weighing cell, ergonomic doors, and pipette-check application |
| Ethanol (molecular biology grade) | Sigma Aldrich | E7023 | 200 proof ethanol for molecular biology applications |
| Glass Pipets | VWR | 36360-536 | Disposable Soda Lime glass Pasteur pipets |
| Glass Vials | Thermo Scientific | B7990-1 | Borosilicate glass vials with PTFE/Silione septum caps |
| Lab grade freezer | Fisher Scientific | IU2886D | Ultra-low temprature freezer (-86 to -50 C) for long-term storage of lipids and proteins |
| Lipids (protaited or perdeuterated) | Avanti Polar Lipids | varies by lipid | Lipids can be purchased from Avanti in powder form or in a chloroform solution with the required amounts and deuteration schemes. |
| Millipore water purifier | Millipore Sigma | ZRQSVP3US | Direct-Q® 3 UV Water Purification System which deliver both pure and ultrapure water with a built-in UV lamp to reduce the levels of organics for biological applications |
| Mini Extruder Set | Avanti Polar Lipids | 610020 | Mini-extruder set includes mini-extruder, heating block, 2 GasTight Syringes, and 2 O-rings, Polycarbonate Membranes, and Filter Supports |
| Quick Connect Fittings | Grainger | 2YDA1 and 2YDA7 | Push-button tube fittings for QuickConnect water circulation applications, e.g. high temperature vesicle extrusion |
| Syringe Pump | SyringePump.com | New Era-1000 | Fully programmable syringe pump for infusion and withdrawal; programs up to 41 pumping phases with adjustable pumping rates, dispensed volumes, and extrusion cycles |
| Ultrasonic bath | Fisher Scientific | CPX2800 | Temperature controlled ultra sonic bath with programmable functionality for degassing and ultrasonic applications |
| Vacuum Oven | Thermo Scientific | 3608 | 0.7 cu ft vaccum oven with built-in-high-limit thermostat guards against overheating |
| Vortex Mixer | Fisher Scientific | 02-215-414 | Variable speed, analog control that allows low rpm start-up for gentle shaking or high-speed mixing for vigorous vortexing of samples |
Referanslar
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175 (4023), 720-731 (1972).
- Andersen, O. S., Koeppe, R. E. Bilayer thickness and membrane protein function: an energetic perspective. Annual Review of Biophysics and Biomolecular Structure. 36, 107-130 (2007).
- Lundbæk, J. A., Collingwood, S. A., Ingólfsson, H. I., Kapoor, R., Andersen, O. S. Lipid bilayer regulation of membrane protein function: gramicidin channels as molecular force probes. Journal of The Royal Society Interface. 7 (44), 373-395 (2010).
- Bradley, R. P., Radhakrishnan, R. Curvature-undulation coupling as a basis for curvature sensing and generation in bilayer membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 117-124 (2016).
- Perozo, E., Cortes, D. M., Sompornpisut, P., Kloda, A., Martinac, B. Open channel structure of MscL and the gating mechanism of mechanosensitive channels. Nature. 418 (6901), 942-948 (2002).
- Jensen, M. &. #. 2. 1. 6. ;., Mouritsen, O. G. Lipids do influence protein function-the hydrophobic matching hypothesis revisited. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1666 (1-2), 205-226 (2004).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118 (6), 1099-1102 (2005).
- Katchalsky, A., Spangler, R. Dynamics of membrane processes. Quarterly Reviews of Biophysics. 1 (2), 127-175 (1968).
- Rheinstädter, M. C. Collective molecular dynamics in proteins and membranes (Review). Biointerphases. 3 (2), 83-90 (2008).
- Fujiwara, T., Ritchie, K., Murakoshi, H., Jacobson, K., Kusumi, A. Phospholipids undergo hop diffusion in compartmentalized cell membrane. The Journal of Cell Biology. 157 (6), 1071-1082 (2002).
- Hac, A. E., Seeger, H. M., Fidorra, M., Heimburg, T. Diffusion in two-component lipid membranes--a fluorescence correlation spectroscopy and monte carlo simulation study. Biophysical Journal. 88 (1), 317-333 (2005).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proceedings of the National Academy of Sciences of the United States of America. 107 (16), 7208-7213 (2010).
- Hormel, T. T., Kurihara, S. Q., Brennan, M. K., Wozniak, M. C., Parthasarathy, R. Measuring lipid membrane viscosity using rotational and translational probe diffusion. Physical Review Letters. 112 (18), 188101 (2014).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Advances in Colloid and Interface Science. 208, 225-234 (2014).
- Bassereau, P., Sorre, B., Lévy, A. Bending lipid membranes: Experiments after W. Helfrich's model. Advances in Colloid and Interface Science. 208, 47-57 (2014).
- Monzel, C., Sengupta, K. Measuring shape fluctuations in biological membranes. Journal of Physics D: Applied Physics. 49 (24), 243002 (2016).
- Deserno, M. Mesoscopic membrane physics: concepts, simulations, and selected applications. Macromolecular Rapid Communications. 30 (9-10), 752-771 (2009).
- Reynwar, B. J., et al. Aggregation and vesiculation of membrane proteins by curvature-mediated interactions. Nature. 447 (7143), 461-464 (2007).
- Haswell, E. S., Phillips, R., Rees, D. C. Mechanosensitive channels: what can they do and how do they do it. Structure. 19 (10), 1356-1369 (2011).
- Phillips, R., Ursell, T., Wiggins, P., Sens, P. Emerging roles for lipids in shaping membrane-protein function. Nature. 459 (7245), 379-385 (2009).
- Dill, K. A., Chan, H. S. From Levinthal to pathways to funnels. Nature Structural Biology. 4 (1), 10-19 (1997).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Lyman, E., Hsieh, C. -. L., Eggeling, C. From dynamics to membrane organization: experimental breakthroughs occasion a "modeling manifesto". Biophysical Journal. 115 (4), 595-604 (2018).
- Arriaga, L. R., et al. Dissipative curvature fluctuations in bilayer vesicles: Coexistence of pure-bending and hybrid curvature-compression modes. The European Physical Journal. E, Soft Matter. 31 (1), 105-113 (2010).
- Honerkamp-Smith, A. R., Veatch, S. L., Keller, S. L. An introduction to critical points for biophysicists; observations of compositional heterogeneity in lipid membranes. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1788 (1), 53-63 (2009).
- Veatch, S. L., Keller, S. L. Organization in lipid membranes containing cholesterol. Physical Review Letters. 89 (26), 268101 (2002).
- Heberle, F. A., et al. Bilayer thickness mismatch controls domain size in model membranes. Journal of the American Chemical Society. 135 (18), 6853-6859 (2013).
- Nickels, J. D., et al. The in vivo structure of biological membranes and evidence for lipid domains. PLOS Biology. 15 (5), 2002214 (2017).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387 (6633), 569-572 (1997).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews. Molecular Cell Biology. 9 (2), 112-124 (2008).
- Liu, S. -. L., et al. Orthogonal lipid sensors identify transbilayer asymmetry of plasma membrane cholesterol. Nature Chemical Biology. 13, 268 (2016).
- Rothman, J., Lenard, J. Membrane asymmetry. Science. 195 (4280), 743-753 (1977).
- Ashkar, R., et al. Neutron scattering in the biological sciences: progress and prospects. Acta Crystallographica Section D. 74 (12), 1129-1168 (2018).
- Woodka, A. C., Butler, P. D., Porcar, L., Farago, B., Nagao, M. Lipid bilayers and membrane dynamics: insight into thickness fluctuations. Physical Review Letters. 109 (5), 058102 (2012).
- Chakraborty, S., et al. How cholesterol stiffens unsaturated lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21896-21905 (2020).
- Arriaga, L. R., et al. Stiffening effect of cholesterol on disordered lipid phases: a combined neutron spin echo + dynamic light scattering analysis of the bending elasticity of large unilamellar vesicles. Biophysical Journal. 96 (9), 3629-3637 (2009).
- Nagao, M., Kelley, E. G., Ashkar, R., Bradbury, R., Butler, P. D. Probing elastic and viscous properties of phospholipid bilayers using neutron spin echo spectroscopy. The Journal of Physical Chemistry Letters. 8 (19), 4679-4684 (2017).
- Kelley, E. G., Butler, P. D., Ashkar, R., Bradbury, R., Nagao, M. Scaling relationships for the elastic moduli and viscosity of mixed lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (38), 23365-23373 (2020).
- Rickeard, B. W., et al. Transverse lipid organization dictates bending fluctuations in model plasma membranes. Nanoscale. 12 (3), 1438-1447 (2020).
- Nickels, J. D., et al. Mechanical properties of nanoscopic lipid domains. Journal of the American Chemical Society. 137 (50), 15772-15780 (2015).
- Mezei, F. Neutron spin echo: A new concept in polarized thermal neutron techniques. Zeitschrift für Physik A Hadrons and Nuclei. 255 (2), 146-160 (1972).
- Hayter, J. B., Penfold, J. Neutron spin-echo integral transform spectroscopy. Zeitschrift für Physik B Condensed Matter. 35 (2), 199-205 (1979).
- Monkenbusch, M., Richter, D., Imae, T., Kanaya, T., Furusaka, M., Torikai, N. . Neutrons in Soft Matter. , 147-182 (2011).
- Pynn, R., Mezei, F., Pappas, C., Gutberlet, T. . Neutron Spin Echo. , 159-177 (2003).
- Holderer, O., et al. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 19 (3), 034022 (2008).
- Schleger, P., et al. The long-wavelength neutron spin-echo spectrometer IN15 at the Institut Laue-Langevin. Physica B: Condensed Matter. 241-243, 164-165 (1997).
- Holderer, O., Zolnierczuk, P., Pasini, S., Stingaciu, L., Monkenbusch, M. A better view through new glasses: Developments at the Jülich neutron spin echo spectrometers. Physica B: Condensed Matter. 562, 9-12 (2019).
- Farago, B., et al. The IN15 upgrade. Neutron News. 26 (3), 15-17 (2015).
- Ashkar, R. Selective dynamics in polymeric materials: Insights from quasi-elastic neutron scattering spectroscopy. Journal of Applied Physics. 127 (15), 151101 (2020).
- Pasini, S., Holderer, O., Kozielewski, T., Richter, D., Phoenix Monkenbusch, M. J-NSE- Phoenix, a neutron spin-echo spectrometer with optimized superconducting precession coils at the MLZ in Garching. Review of Scientific Instruments. 90 (4), 043107 (2019).
- Svergun, D. I., Koch, M. H. J., Timmins, P. A., May, R. P. . Small Angle X-Ray and Neutron Scattering from Solutions of Biological Macromolecules. , (2013).
- Eicher, B., et al. Joint small-angle X-ray and neutron scattering data analysis of asymmetric lipid vesicles. Journal of Applied Crystallography. 50 (2), 419-429 (2017).
- Heberle, F. A., et al. Model-based approaches for the determination of lipid bilayer structure from small-angle neutron and X-ray scattering data. European Biophysics Journal. 41 (10), 875-890 (2012).
- Jaksch, S., Koutsioubas, A., Mattauch, S., Holderer, O., Frielinghaus, H. Long-range excitations in phospholipid membranes. Chemistry and Physics of Lipids. 225, 104788 (2019).
- Jaksch, S., et al. Influence of ibuprofen on phospholipid membranes. Physical Review E. 91 (2), 022716 (2015).
- Armstrong, C. L., et al. Effect of cholesterol on the lateral nanoscale dynamics of fluid membranes. European Biophysics Journal. 41 (10), 901-913 (2012).
- Rheinstädter, M. C., Häußler, W., Salditt, T. Dispersion relation of lipid membrane shape fluctuations by neutron spin-echo spectrometry. Physical Review Letters. 97 (4), 048103 (2006).
- Armstrong, C. L., Häußler, W., Seydel, T., Katsaras, J., Rheinstädter, M. C. Nanosecond lipid dynamics in membranes containing cholesterol. Soft Matter. 10 (15), 2600-2611 (2014).
- Nickels, J. D., et al. Lipid rafts: buffers of cell membrane physical properties. The Journal of Physical Chemistry B. 123 (9), 2050-2056 (2019).
- Michonova-Alexova, E. I., Sugár, I. P. Component and state separation in DMPC/DSPC lipid bilayers: a Monte Carlo simulation study. Biophysical Journal. 83 (4), 1820-1833 (2002).
- Sugár, I. P., Thompson, T. E., Biltonen, R. L. Monte Carlo simulation of two-component bilayers: DMPC/DSPC mixtures. Biophysical Journal. 76 (4), 2099-2110 (1999).
- Mabrey, S., Sturtevant, J. M. Investigation of phase transitions of lipids and lipid mixtures by sensitivity differential scanning calorimetry. Proceedings of the National Academy of Sciences. 73 (11), 3862-3866 (1976).
- . Neutron activation and scattering calculator Available from: https://www.ncnr.nist.gov/resources/activation/ (2021)
- Scott, H. L., et al. On the mechanism of bilayer separation by extrusion, or why your LUVs are not really unilamellar. Biophysical Journal. 117 (8), 1381-1386 (2019).
- Ashkar, R., et al. Tuning membrane thickness fluctuations in model lipid bilayers. Biophysical Journal. 109 (1), 106-112 (2015).
- Carrillo, J. -. M. Y., Katsaras, J., Sumpter, B. G., Ashkar, R. A computational approach for modeling neutron scattering data from lipid bilayers. Journal of Chemical Theory and Computation. 13 (2), 916-925 (2017).
- Azuah, R. T. DAVE: a comprehensive software suite for the reduction, visualization, and analysis of low energy neutron spectroscopic data. Journal of Research of the National Institute of Standards and Technology. 114 (6), 341-358 (2009).
- Van Hove, L. Correlations in space and time and born approximation scattering in systems of interacting particles. Physical Review. 95 (1), 249-262 (1954).
- Zilman, A. G., Granek, R. Undulations and dynamic structure factor of membranes. Physical Review Letters. 77 (23), 4788-4791 (1996).
- Kelley, E. G., Butler, P. D., Nagao, M. . Collective dynamics in model biological membranes measured by neutron spin echo spectroscopy. , 131-176 (2019).
- Zheng, Y., Michihiro, N., Dobrin, P. B. Bending elasticity of saturated and monounsaturated phospholipid membranes studied by the neutron spin echo technique. Journal of Physics: Condensed Matter. 21 (15), 155104 (2009).
- Sharma, V. K., Qian, S. Effect of an antimicrobial peptide on lateral segregation of lipids: a structure and dynamics study by neutron scattering. Langmuir. 35 (11), 4152-4160 (2019).
- Boggara, M. B., Faraone, A., Krishnamoorti, R. Effect of pH and Ibuprofen on the Phospholipid Bilayer Bending Modulus. The Journal of Physical Chemistry B. 114 (24), 8061-8066 (2010).
- Lee, J. -. H., et al. Thermal fluctuation and elasticity of lipid vesicles interacting with pore-forming peptides. Physical Review Letters. 105 (3), 038101 (2010).
- Chakraborty, S., Abbasi, A., Bothun, G. D., Nagao, M., Kitchens, C. L. Phospholipid bilayer softening due to hydrophobic gold nanoparticle inclusions. Langmuir. 34 (44), 13416-13425 (2018).
- Hoffmann, I., et al. Softening of phospholipid membranes by the adhesion of silica nanoparticles - as seen by neutron spin-echo (NSE). Nanoscale. 6 (12), 6945-6952 (2014).
- Watson, M. C., Brown, F. L. H. Interpreting membrane scattering experiments at the mesoscale: the contribution of dissipation within the bilayer. Biophysical Journal. 98 (6), 9-11 (2010).
- Seifert, U., Langer, S. A. Viscous modes of fluid bilayer membranes. Europhysics Letters (EPL). 23 (1), 71-76 (1993).
- Bingham, R. J., Smye, S. W., Olmsted, P. D. Dynamics of an asymmetric bilayer lipid membrane in a viscous solvent. EPL (Europhysics Letters). 111 (1), 18004 (2015).
- Rawicz, W., Olbrich, K. C., McIntosh, T., Needham, D., Evans, E. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophysical Journal. 79 (1), 328-339 (2000).
- Doktorova, M., LeVine, M. V., Khelashvili, G., Weinstein, H. A new computational method for membrane compressibility: bilayer mechanical thickness revisited. Biophysical Journal. 116 (3), 487-502 (2019).
- Evans, E., Needham, D. Physical properties of surfactant bilayer membranes: thermal transitions, elasticity, rigidity, cohesion and colloidal interactions. The Journal of Physical Chemistry. 91 (16), 4219-4228 (1987).
- Lesieur, S., Grabielle-Madelmont, C., Paternostre, M. T., Ollivon, M. Size analysis and stability study of lipid vesicles by high-performance gel exclusion chromatography, turbidity, and dynamic light scattering. Analytical Biochemistry. 192 (2), 334-343 (1991).
- Heberle, F. A., et al. Direct label-free imaging of nanodomains in biomimetic and biological membranes by cryogenic electron microscopy. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19943-19952 (2020).
- Cornell, C. E., Mileant, A., Thakkar, N., Lee, K. K., Keller, S. L. Direct imaging of liquid domains in membranes by cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19713-19719 (2020).
- Yao, X., Fan, X., Yan, N. Cryo-EM analysis of a membrane protein embedded in the liposome. Proceedings of the National Academy of Sciences of the United States of America. 117 (31), 18497-18503 (2020).
- Kučerka, N., Nieh, M. -. P., Katsaras, J. Fluid phase lipid areas and bilayer thicknesses of commonly used phosphatidylcholines as a function of temperature. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1808 (11), 2761-2771 (2011).
- Nielsen, J. E., Bjørnestad, V. A., Lund, R. Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin. Soft Matter. 14 (43), 8750-8763 (2018).
- Kučerka, N., et al. Lipid bilayer structure determined by the simultaneous analysis of neutron and X-ray scattering data. Biophysical Journal. 95 (5), 2356-2367 (2008).
- Kelley, E. G., Butler, P. D., Nagao, M. Scaling of lipid membrane rigidity with domain area fraction. Soft Matter. 15 (13), 2762-2767 (2019).
- Brüning, B. -. A., et al. Bilayer undulation dynamics in unilamellar phospholipid vesicles: Effect of temperature, cholesterol and trehalose. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1838 (10), 2412-2419 (2014).
- Kučerka, N., et al. Areas of monounsaturated diacylphosphatidylcholines. Biophysical Journal. 97 (7), 1926-1932 (2009).
- Sharma, V. K., Mamontov, E., Anunciado, D. B., O'Neill, H., Urban, V. S. Effect of antimicrobial peptide on the dynamics of phosphocholine membrane: role of cholesterol and physical state of bilayer. Soft Matter. 11 (34), 6755-6767 (2015).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in lipid membranes containing transmembrane peptides. Soft Matter. , (2021).
- Yu, J., et al. Structure and dynamics of lipid membranes interacting with antivirulence end-phosphorylated polyethylene glycol block copolymers. Soft Matter. 16 (4), 983-989 (2020).
- Stingaciu, L. -. R., et al. Revealing the dynamics of thylakoid membranes in living cyanobacterial cells. Scientific Reports. 6 (1), 19627 (2016).
- Stingaciu, L. -. R., O'Neill, H. M., Liberton, M., Pakrasi, H. B., Urban, V. S. Influence of chemically disrupted photosynthesis on cyanobacterial thylakoid dynamics in synechocystis sp. PCC 6803. Scientific Reports. 9 (1), 5711 (2019).
- Miller, I. R. Energetics of fluctuation in lipid bilayer thickness. Biophysical Journal. 45 (3), 643-644 (1984).
- Nagao, M. Observation of local thickness fluctuations in surfactant membranes using neutron spin echo. Physical Review E. 80 (3), 031606 (2009).
Erratum
Formal Correction: Erratum: Neutron Spin Echo Spectroscopy as a Unique Probe for Lipid Membrane Dynamics and Membrane-Protein Interactions
Posted by JoVE Editors on 8/06/2021. Citeable Link.
An erratum was issued for: Neutron Spin Echo Spectroscopy as a Unique Probe for Lipid Membrane Dynamics and Membrane-Protein Interactions. The Introduction, Protocol, and Representative Results sections have been updated.
In the Introduction, the fith pargraph was updated from:
Besides direct access to the length and time scale of membrane dynamics, NSE has the inherent capabilities of neutron isotope sensitivity52. Specifically, the ability of neutrons to interact differently with the isotopes of hydrogen, the most abundant element in biological systems, results in a different neutron scattering length density,34 or NSLD (the equivalent of the optical index of refraction50), when protium is substituted by deuterium. This enables an approach known as contrast variation, which is commonly used to highlight specific membrane features or conceal others — the latter scenario is referred to as contrast matching. A frequent application of contrast variation/matching is the substitution of water (NSLD = -0.56 × 10-6 Å-2) by heavy water or D2O (NSLD = 6.4 × 10-6 Å-2) to amplify the neutron signal from protiated lipid membranes (NSLD ~ 2 × 10-6 Å-2). This approach is highly effective in studies of membrane structure because the penetration of D2O into the headgroup region of the membrane allows accurate determination of the membrane thicknesses (see Figure 2A, left panel) and of the location of different lipid subgroups when more sophisticated models are applied53,54. This paper highlights some examples on the use of contrast variation for studies of collective dynamics in biomimetic membranes and select membrane features.
to:
Besides direct access to the length and time scale of membrane dynamics, NSE has the inherent capabilities of neutron isotope sensitivity52. Specifically, the ability of neutrons to interact differently with the isotopes of hydrogen, the most abundant element in biological systems, results in a different neutron scattering length density,34 or NSLD (the equivalent of the optical index of refraction50), when protium is substituted by deuterium. This enables an approach known as contrast variation, which is commonly used to highlight specific membrane features or conceal others — the latter scenario is referred to as contrast matching. A frequent application of contrast variation/matching is the substitution of water (NSLD = -0.56 × 10-6 Å-2) by heavy water or D2O (NSLD = 6.4 × 10-6 Å-2) to amplify the neutron signal from protiated lipid membranes (NSLD ~ 0 × 10-6 Å-2). This approach is highly effective in studies of membrane structure because the penetration of D2O into the headgroup region of the membrane allows accurate determination of the membrane thicknesses (see Figure 2A, left panel) and of the location of different lipid subgroups when more sophisticated models are applied53,54. This paper highlights some examples on the use of contrast variation for studies of collective dynamics in biomimetic membranes and select membrane features.
In the Protocol, step 1.1 was updated from:
For bending fluctuation measurements, make fully protiated liposomes in D2O (D 99.9%) or D2O-buffer (e.g., phosphate buffer prepared with D2O instead of H2O). Use fully protiated DMPC (C36H72NO8P) and DSPC (C44H88NO8P) with 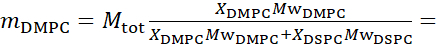 133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 2 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 2 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
to:
For bending fluctuation measurements, make fully protiated liposomes in D2O (D 99.9%) or D2O-buffer (e.g., phosphate buffer prepared with D2O instead of H2O). Use fully protiated DMPC (C36H72NO8P) and DSPC (C44H88NO8P) with 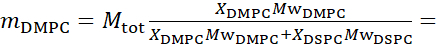 133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 0 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 0 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
In the Representative Results, the fist pagargaph was updted from:
NSE studies accessing bending fluctuations are typically performed over a Q-range of ~ (0.04 - 0.2) Å-1. This Q-range corresponds to intermediate length scales between the membrane thickness and the liposomal radius, where bending dynamics dominate. Measurement over an extended Q-range can give access to additional dynamic modes, including liposomal diffusion and intramembrane dynamics. For more details on the cross-over in membrane dynamics accessed by NSE, check these relevant publications25,71. It is important to emphasize that NSE signals are proportional to: 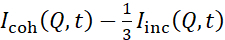 , where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~2 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
, where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~2 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
to:
NSE studies accessing bending fluctuations are typically performed over a Q-range of ~ (0.04 - 0.2) Å-1. This Q-range corresponds to intermediate length scales between the membrane thickness and the liposomal radius, where bending dynamics dominate. Measurement over an extended Q-range can give access to additional dynamic modes, including liposomal diffusion and intramembrane dynamics. For more details on the cross-over in membrane dynamics accessed by NSE, check these relevant publications25,71. It is important to emphasize that NSE signals are proportional to: 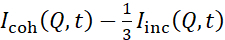 , where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~0 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
, where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~0 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
In the Representative Reults, Figure 2 was updated from:
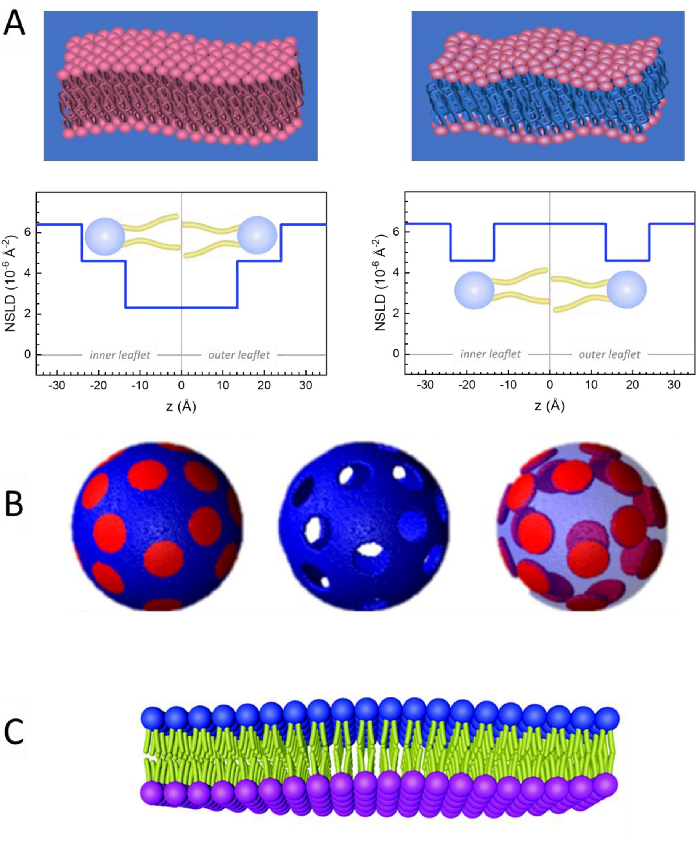
Figure 2: Examples of possible deuteration schemes in NSE experiments on lipid membranes. (A) Left: Fully contrasted membranes, e.g., protiated membranes in deuterated buffer, showing the NSLD profile along the normal to the membrane surface. The difference in the NSLD between the headgroup (~2 × 10-2 Å-2) and tail region (~4.5 × 10-6 Å-2) of the membrane is due to the headgroup hydration with deuterated buffer. Right: Tail-contrast matched membranes such that the hydrocarbon tail region of the membrane has the same NSLD as the buffer, as shown in the corresponding NSLD profile along the membrane normal. (B) Domain-forming membranes with two neutron contrast schemes where the domains (center) or the matrix (left) are contrast-matched to the buffer, enabling selective studies of matrix or domain dynamics, respectively. This figure has been modified from Nickels et al., JACS 201541. (C) Asymmetric membranes prepared by cyclodextrin exchange between protiated and deuterated lipid vesicles, resulting in the deuteration of one membrane leaflet while keeping the other leaflet protiated. This allows studies of the bending dynamics of the protiated leaflet and provides insights into the mechanical coupling between opposing leaflets in asymmetric membranes. This figure has been modified from Rickeard et al., Nanoscale 202040. Please click here to view a larger version of this figure.
to:
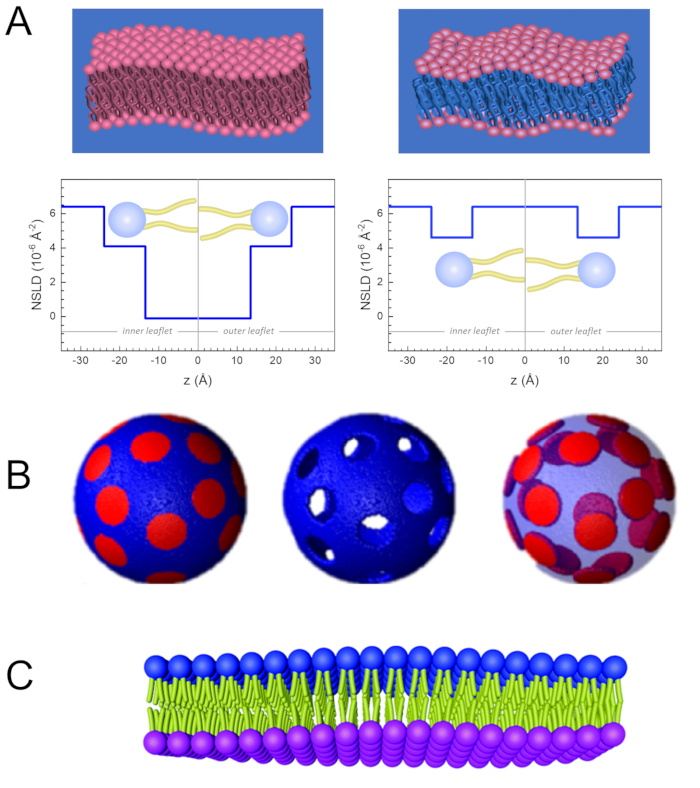
Figure 2: Examples of possible deuteration schemes in NSE experiments on lipid membranes. (A) Left: Fully contrasted membranes, e.g., protiated membranes in deuterated buffer, showing the NSLD profile along the normal to the membrane surface. The difference in the NSLD between the tail region (~0 × 10-2 Å-2) and headgroup region (~4.5 × 10-6 Å-2) of the membrane is due to the headgroup hydration with deuterated buffer. Right: Tail-contrast matched membranes such that the hydrocarbon tail region of the membrane has the same NSLD as the buffer, as shown in the corresponding NSLD profile along the membrane normal. (B) Domain-forming membranes with two neutron contrast schemes where the domains (center) or the matrix (left) are contrast-matched to the buffer, enabling selective studies of matrix or domain dynamics, respectively. This figure has been modified from Nickels et al., JACS 201541. (C) Asymmetric membranes prepared by cyclodextrin exchange between protiated and deuterated lipid vesicles, resulting in the deuteration of one membrane leaflet while keeping the other leaflet protiated. This allows studies of the bending dynamics of the protiated leaflet and provides insights into the mechanical coupling between opposing leaflets in asymmetric membranes. This figure has been modified from Rickeard et al., Nanoscale 202040. Please click here to view a larger version of this figure.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır