Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Нейтронная спиновая эхо-спектроскопия как уникальный зонд для динамики липидных мембран и мембранно-белковых взаимодействий
В этой статье
Erratum Notice
Резюме
В данной статье описываются протоколы подготовки образцов, восстановления данных и анализа данных в исследованиях нейтронного спинового эха (NSE) липидных мембран. Разумное дейтериевое маркировка липидов обеспечивает доступ к различной динамике мембран на мезоскопических масштабах длины и времени, в которых происходят жизненно важные биологические процессы.
Аннотация
Липидные бислои образуют основную матрицу клеточных мембран и являются основной платформой для обмена питательных веществ, белково-мембранных взаимодействий и вирусного бутонизации, среди других жизненно важных клеточных процессов. Для эффективной биологической активности клеточные мембраны должны быть достаточно жесткими, чтобы поддерживать целостность клетки и ее компартментов, но достаточно текучими, чтобы позволить мембранным компонентам, таким как белки и функциональные домены, диффундировать и взаимодействовать. Этот тонкий баланс свойств упругих и жидких мембран и их влияние на биологическую функцию требуют лучшего понимания динамики коллективной мембраны по мезоскопической длине и временным масштабам ключевых биологических процессов, например, деформаций мембраны и событий связывания белка. Среди методов, которые могут эффективно исследовать этот динамический диапазон, является спектроскопия нейтронного спинового эха (NSE). В сочетании с дейтериевой маркировкой NSE может использоваться для непосредственного доступа к колебаниям изгиба и толщины, а также к мезоскопической динамике отдельных особенностей мембраны. В данной статье приводится краткое описание методики NSE и описываются процедуры проведения экспериментов NSE на липосомальных мембранах, включая детали схем пробоподготовки и дейтерации, а также инструкции по сбору и сокращению данных. В статье также представлены методы анализа данных, используемые для извлечения ключевых параметров мембраны, таких как модуль жесткости изгиба, модуль сжимаемости площади и вязкость в плоскости. Чтобы проиллюстрировать биологическую значимость исследований NSE, обсуждаются отдельные примеры мембранных явлений, исследуемых NSE, а именно: влияние добавок на жесткость изгиба мембраны, влияние образования доменов на флуктуации мембраны и динамическую сигнатуру мембранно-белковых взаимодействий.
Введение
Понимание клеточных мембран и их функций замечательно изменилось за последние несколько десятилетий. Прежний взгляд на клеточные мембраны как на пассивные липидные бислои, которые определяют границы клеток и белки домашней мембраны1, постепенно трансформировался в динамическую модель, в которой липидные бислои играют важную роль в регулировании жизненно важных биологических процессов, включая клеточную сигнализацию, молекулярный обмен и функцию белка — и это2,3,4,5,6. Это осознание того, что клеточные мембраны очень динамичны, постоянно подвергаются ремоделированию и молекулярному перераспределению, побудило к научным исследованиям за пределами равновесных структур мембран7,8,9. Соответственно, было разработано несколько подходов к изучению различных динамических режимов в биологических и биоинспирируемых липидных мембранах. На сегодняшний день большинство этих исследований в основном сосредоточено на диффузных молекулярных движениях10,11,12,13 и макроскопических флуктуациях формы14,15,16,оставляя значительный пробел в понимании динамики промежуточных мембран, т. е. коллективных флуктуаций липидных сборок, состоящих из нескольких 10-100 молекул липидов. Эта динамика происходит на масштабах длины от нескольких десятков до нескольких 100 Å и во временных масштабах от суб-ns до нескольких сотен ns (см. Рисунок 1),называемых здесь мезоскопическими масштабами. Именно в этих масштабах происходит ключевая биологическая активность на мембранном уровне17. Это включает вирусное почкование18,канал19и мембранно-белковые взаимодействия20. Важно также отметить, что энергетический ландшафт мембранных белков21, 22показывает, что конформационные изменения в белках, необходимые для их регуляторной роли, происходят на временных масштабахns 23 коллективных флуктуаций мембран, что дополнительно подчеркивает важность мезоскопической динамики в биологической функции клеточных мембран и их биоинспирируемых аналогов20. В данной работе основное внимание уделяется двум основным мезоскопическим динамическим режимам в липидных мембранах, а именно флуктуациям изгиба и колебаниям толщины.
Основной проблемой при непосредственном провожде этих флуктуацийных режимов является сложность одновременного доступа к их пространственным и временным масштабам с использованием стандартных методов спектроскопии. Другая проблема заключается в том, что методы прямого контакта могут влиять на те же колебания, которые они предназначены для измерения16. Это еще более усугубляется композиционной и структурной сложностью биологических мембран24,25,что приводит к неоднородным особенностям мембраны, включая образование липидных доменов26,27, 28,29, 30и асимметрию мембран31, 32, 33,требуя селективных зондов для понимания динамики различных особенностей мембраны. К счастью, эти проблемы могут быть преодолены с помощью неинвазивных методов нейтронной спектроскопии, таких как нейтронное спиновое эхо (NSE), которые по своей сути получают доступ к требуемой длине и временным масштабам, а также позволяют дополнительно проводить исследования селективных особенностей мембран без изменения их физико-химической среды34. Действительно, за последние несколько лет спектроскопия NSE превратилась в уникальный и мощный зонд коллективной мембранной динамики35. Результаты исследований NSE на липидных мембранах дали новое представление о механическихсвойствах 36,37 и вязкоупругих38,39 липидных мембран и пролили новый свет на их потенциальную роль в биологической функции40,41.
Метод спектроскопии NSE основан на конструкции интерферометрического прибора, впервые предложенной Mezei42,с использованием серии спин-ласт и магнитных катушек для управления прецессией спина нейтрона, когда нейтроны пересекают инструмент. Конструкция опирается на магнитное зеркальное отражение элементов магнитного поля относительно положения образца(рисунок 1А). Это означает, что при отсутствии обмена энергией между нейтроном и образцом нейтрон выполняет одинаковое количество спиновых прецессий в противоположных направлениях в первой и второй половине инструмента (обратите внимание на π-флиппер между двумя катушками прецессии). В результате конечное спиновое состояние нейтрона остается неизменным относительно начального состояния - явление, называемое спин-эхо (см. прозрачный нейтрон на рисунке 1А). Однако, когда нейтрон энергетически взаимодействует с образцом, энергообмен изменяет число спиновых прецессий во второй половине инструмента, что приводит к другому конечному спиновому состоянию (см. Рисунок 1А). Это экспериментально обнаруживается как потеря поляризации, как будет показано далее в этой статье. Для получения более подробной информации о технике NSE читатель обращается к специальным техническим документам42,43,44,45.
Здесь представлено упрощенное описание, чтобы дать приблизительную оценку длины и временных масштабов, доступных с NSE. Масштабы длин определяются диапазоном достижимых волновекторных передач, Q = 4π sin θ/λ, где 2θ — угол рассеяния, а λ — длина волны нейтрона. Видно, что Q задается диапазоном длин волн и степенью вращения второго плеча спектрометра (см. рисунок 1А). Типичный Q-диапазонна спектрометрах NSE составляет ~0,02-2 Å-146,47и до 0,01-4 Å-1 с недавними обновлениями48,49,что соответствует пространственным масштабам ~1-600 Å. С другой стороны, доступная шкала времени вычисляется из общего угла прецессии (или фазы), приобретенного нейтроном в магнитных катушках прецессии, и обнаруживается, что она составляет50: 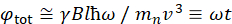 . В этом выражении t — время Фурье, определяемое как
. В этом выражении t — время Фурье, определяемое как 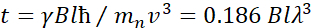 , где —
, где — 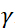 нейтронное гиромагнитное
нейтронное гиромагнитное  отношение, — длина катушки и
отношение, — длина катушки и 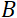 — сила магнитного поля катушки. Стоит отметить, что время Фурье — это величина, которая строго зависит от геометрии прибора, силы магнитного поля и длины волны нейтрона. Например, используя нейтроны длины волны
— сила магнитного поля катушки. Стоит отметить, что время Фурье — это величина, которая строго зависит от геометрии прибора, силы магнитного поля и длины волны нейтрона. Например, используя нейтроны длины волны 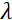 = 8 Å и настройки прибора
= 8 Å и настройки прибора  = 1,2 м и
= 1,2 м и 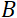 = 0,4 Тл, время Фурье вычисляется как t ~ 50 нс. Экспериментально время Фурье настраивается путем изменения тока в катушках прецессии (т. Е. Напряженность магнитного поля) или с использованием различных длин волн нейтронов, что приводит к типичным шкалам времени NSE от ~ 1 пс до 100 нс. Тем не менее, недавние обновления спектрометров NSE позволили получить доступ к более длинным временам Фурье, до ~ 400 нс на спектрометре J-NSE-Phoenix в Центре Хайнца Майера-Лейбница51 и спектрометре SNS-NSE в Национальной лаборатории Оук-Ридж48и до ~ 1000 нс на спектрометре IN15 NSE в Институте Лауэ-Ланжевена (ILL)49.
= 0,4 Тл, время Фурье вычисляется как t ~ 50 нс. Экспериментально время Фурье настраивается путем изменения тока в катушках прецессии (т. Е. Напряженность магнитного поля) или с использованием различных длин волн нейтронов, что приводит к типичным шкалам времени NSE от ~ 1 пс до 100 нс. Тем не менее, недавние обновления спектрометров NSE позволили получить доступ к более длинным временам Фурье, до ~ 400 нс на спектрометре J-NSE-Phoenix в Центре Хайнца Майера-Лейбница51 и спектрометре SNS-NSE в Национальной лаборатории Оук-Ридж48и до ~ 1000 нс на спектрометре IN15 NSE в Институте Лауэ-Ланжевена (ILL)49.
Помимо прямого доступа к длине и временной шкале мембранной динамики, NSE обладает присущими 52 возможностями чувствительности к изотопам нейтронов52. В частности, способность нейтронов по-разному взаимодействовать с изотопами водорода, наиболее распространенного элемента в биологических системах, приводит к различной плотности рассеяния нейтронов,34 или NSLD (эквивалент оптического показателя преломления50),когда протий замещается дейтерием. Это позволяет использовать подход, известный как вариация контраста, который обычно используется для выделения конкретных особенностей мембраны или сокрытия других — последний сценарий называется контрастным соответствием. Частым применением изменения/согласования контраста является замена воды (NSLD = -0,56 × 10-6 Å-2)тяжелой водой или D2O (NSLD = 6,4 × 10-6 Å-2)для усиления нейтронного сигнала от протиированных липидных мембран (NSLD ~ 0 × 10-6 Å-2). Этот подход очень эффективен в исследованиях структуры мембраны, поскольку проникновениеD2O в область головной группы мембраны позволяет точно определить толщину мембраны (см. Рисунок 2A,левая панель) и расположение различных липидных подгрупп при применении более сложных моделей53,54. В данной работе приводятся некоторые примеры использования вариации контраста для изучения коллективной динамики в биомиметических мембранах и отдельных особенностей мембран.
Здесь эффективность NSE в предоставлении уникального понимания динамических и функциональных свойств мембран иллюстрируется наглядными примерами исследований NSE на модельных и биологически значимых липидных мембранных системах с акцентом на мезомасштабную динамику в отдельно стоящих мембранах в виде липосомальных суспензий. Для NSE измерений динамики плоских мембран читатель обращается к специализированным публикациям по нейтронно-эхо-спектроскопии (GINSES)55, 56 и другим исследованиям выровненных многоламелярных мембранных стеков57,58,59,60.
Для простоты в данной работе выделены три различные схемы мембранной дейтрации, проиллюстрированные на хорошо изученной доменно-формирующей, или фазоразделяющей, липидной бислойной системе из смесей 1,2-димиристоил-sn-глицеро-3-фосфохолин (DMPC) и 1,2-дистеароил-sn-глицеро-3-фосфохолин (DSPC)61,62. Эти два липида характеризуются несоответствием длины углеводородной цепи (14 атомов углерода на хвост в DMPC против 18 атомов углерода на хвост в DSPC) и температуры перехода гель-жидкость (Tm, DMPC = 23 °C против Tm, DSPC = 55 °C). Это приводит к боковому фазовой разделению в мембранах DMPC:DSPC при температурах между верхними и нижними температурами перехода смеси63. Рассмотренные здесь схемы дейтерации выбраны для демонстрации различных динамических режимов, доступных в измерениях NSE на липосомальных мембранах, а именно флуктуаций изгиба, колебаний толщины и селективных флуктуаций изгиба/толщины боковых доменов. Все липидные композиции представлены для двухслоев DMPC:DSPC, приготовленных при моль фракции 70:30, с использованием коммерчески доступных протиированных и пердевтерированных вариантов DMPC и DSPC. Все этапы подготовки образца основаны на 4 мл липосомальной суспензии вD2Oс концентрацией липидов 50 мг/мл для общей липидной массы Mtot = 200 мг на образец.
протокол
1. Схема деитерации, необходимая для эксперимента
- Для измерения флуктуаций изгиба делают полностью протиатированные липосомы вD2O(D 99,9%) илиD2O-буфере (например, фосфатный буфер, приготовленный сD2O вместоH2O). Используйте полностью протиированный DMPC (C36H72NO8P) и DSPC (C44H88NO8P) с
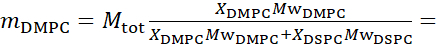 133,4 мг, где XDMPC и XDSPC являются молярными фракциями DMPC и DSPC, здесь установлены на 0,7 и 0,3 соответственно, а MwDMPC и MwDSPC - молярные веса, заданные 677,9 г / моль и 790,1 г / моль соответственно. Аналогично, mDSPC = 66,6 мг. Эта схема дейтерации увеличивает контраст рассеяния между мембраной (NSLD ~ 0 × 10-6 Å-2)и дейтерированным буфером (NSLD ~ 6,4 × 10-6 Å-2)и усиливает сигнал от мембранных волн (см. Рисунок 2A левая панель).
133,4 мг, где XDMPC и XDSPC являются молярными фракциями DMPC и DSPC, здесь установлены на 0,7 и 0,3 соответственно, а MwDMPC и MwDSPC - молярные веса, заданные 677,9 г / моль и 790,1 г / моль соответственно. Аналогично, mDSPC = 66,6 мг. Эта схема дейтерации увеличивает контраст рассеяния между мембраной (NSLD ~ 0 × 10-6 Å-2)и дейтерированным буфером (NSLD ~ 6,4 × 10-6 Å-2)и усиливает сигнал от мембранных волн (см. Рисунок 2A левая панель). - Для измерения динамики изгиба отдельных боковых характеристик мембраны, например, динамики матрицы в фазоразделяющих мембранах DMPC:DSPC, используют протиатированный DMPC (C36H72NO8P) и дейтерированный, DSPC-d83 (C44H5NO8PD83,Mw 873,7 г/моль), так что mDMPC = 128,8 мг и mDSPC-d83 = 71,2 мг. Эта схема дейтерации сводит к минимуму рассеяние от нежелательных доменов, богатых DSPC, что позволяет выборочно измерять флуктуации изгиба из матрицы, богатой DMPC (см. рисунок 2B в середине).
ПРИМЕЧАНИЕ: Чтобы найти оптимальную дейтрацию липидов, необходимую для конкретной схемы сопоставления контрастов, используйте доступные веб-калькуляторы плотности длины рассеяния (SLD), такие как тот, который разработан Центром нейтронных исследований NIST64. Эти веб-интерфейсы оснащены удобными инструментами для легкого расчета SLD липидов с различной степенью дейтерации, а также липидных смесей. - Для NSE измерений средней толщины мембраны (без бокового контраста) используют хвосто-дейтерированные варианты составляющих липидов, т.е. DMPC-d54 (C36H18NO8PD54,732,3 г/моль) и DSPC-d70 (C44H18NO8PD70,860,1 г/моль)35,38,такие, что mDMPC-d54 = 133,0 мг и мDSPC-d70 = 67,0 мг. Эта контрастная схема(рисунок 2A,правая панель) усиливает сигнал рассеяния от липидных головных групп (NSLD ~ 4,5 ×10 -6 Å-2)путем контрастного сопоставления хвостовой группы (NSLD ~ 6,4 × 10-6 Å-2)к дейтерированному буферу, позволяя обнаруживать колебания толщины мембраны.
- Для исследования флуктуаций толщины отдельных мембранных компартментов, например, матрицы, богатой DMPC, используйте ту же стратегию, описанную на этапе 1.2, путем замены протиированных липидов DMPC их хвостовыми дейтерированными аналогами, т.е. DMPC-d54, таким образом, что домены, богатые DSPC, контрастно соответствуют дейтерированному буферу, а первичный сигнал рассеяния происходит из области головной группы матрицы, богатой DMPC хвоста.
2. Подготовка липидной суспензии для экструзии
- Рассчитайте массу каждого компонента в образце в зависимости от состава образца. Как правило, для образцов с несколькими молекулярными компонентами масса компонента задается его молярной массой Mwi,взвешенной по его молярной фракции Xi,и нормируется по всем компонентам таким образом, что:
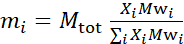 где Mtot - общая масса, установленная здесь на 200 мг. См. пример выше для липидных бислоев DMPC-DSPC с различными схемами дейтерации.
где Mtot - общая масса, установленная здесь на 200 мг. См. пример выше для липидных бислоев DMPC-DSPC с различными схемами дейтерации. - Используя цифровой полумикровес, взвесьте рассчитанные массы липидов (и других компонентов образца, например, белков, наночастиц и т. Д.) И добавьте их во флакон или колбу с круглым дном - не забудьте предварительно взвесить флакон или колбу. Добавьте 1 мл растворителя для растворения взвешенных компонентов, вручную перемешав внутри вытяжки. Для чистых липидных образцов используйте хлороформ или этанол. Для образцов с дополнительными, нелипидными компонентами (например, наночастицами) выбирайте общий растворитель, который диспергирует все компоненты.
- Для небольших количеств липидов (<10 мг) приготовьте бульон и пипетку необходимого объема в смесь.
ПРИМЕЧАНИЕ: Не добавляйте чрезмерное количество растворителя, так как это значительно замедлит этап сушки растворителя, описанный ниже.
- Для небольших количеств липидов (<10 мг) приготовьте бульон и пипетку необходимого объема в смесь.
- Высушите раствор липидов внутри капюшона, осторожно вливая в флакон инертный газ (например, азот, аргон), медленно вращая флакон под углом. Держите флаконы в наклонном положении, чтобы создать тонкую пленку высушенного липида на стенках флакона, которая позволит равномерно высохнуть. Периодически помещайте флакон на водяную баню при 35 °C, чтобы обойти охлаждение, опосредованное испарением, которое замедлит испарение растворителя.
- Поместите флаконы на ночь в вакуумную печь при температуре ~35 °C, чтобы полностью удалить остаточный растворитель. Для ненасыщенных липидов продувки вакуума инертным газом, чтобы свести к минимуму окисление.
- Чтобы обеспечить полное удаление растворителя, взвесьте флакон после высыхания липидов и убедитесь, что нет избыточной массы сверх измеренного количества материалов. Делают это путем вычитания массы флакона из измеренной массы после высыхания. При наличии избыточной массы высушите образец под вакуумом еще 6 ч. При необходимости повторите этот процесс.
- Увлажняют липидную пленку 4 млD2O илиD2O-буфера для получения концентрации липидов 50 мг/мл. Для липидов с высокими температурами перехода, таких как смеси DMPC-DSPC, нагревайте буфер до температуры выше температуры перехода (60 °C), чтобы обеспечить равномерное перемешивание.
ПРИМЕЧАНИЕ: Поскольку эксперименты NSE требуют относительно больших объемов образцов (~4 мл), рассмотрите возможность гидратации образца с использованием половины требуемого буфера, т.е. 2 мл, чтобы свести к минимуму количество экструзий на образец (см. раздел 3). В этом случае добавьте оставшуюся половину буфера после экструзии. Обратите внимание, что емкость шприцев, используемых при экструзии, ограничена 1 мл. Таким образом, гидратация 4 мл буфера потребует четырех наборов экструзии. - Вихрево смешайте гидратированный липидный раствор до тех пор, пока липидная пленка полностью не растворится и больше не будет видна на стенках флакона. На этом этапе гидратированные липиды образуют многоламелярные везикулы и многоцветные стеки микрон, а суспензия выглядит молочно-белой.
- Чтобы облегчить разрушение липидных стеков и уменьшить мультиламеллартность, выполните пять циклов замораживания/оттаивания, поместив флакон с гидратированным липидным раствором в морозильную камеру лабораторного класса (предпочтительно морозильную камеру с -80 °C) до полного замораживания, а затем переместив флакон на водяную баню с 35 °C до полного размораживания липидного раствора. Вихрь размороженный раствор до однородной однородной жижи. Повторите еще четыре раза.
ПРИМЕЧАНИЕ: В качестве альтернативы, ванну с сухим льдом можно подготовить для быстрого замораживания, сочетая ацетон и сухой лед.
3. Экструзия гидратированного липидного раствора
- Соберите установку экструдера, используя поликарбонатную мембрану между двумя мембранными опорами и добавив два бумажных фильтра с каждой стороны для обеспечения дополнительной поддержки. Используйте поликарбонатную мембрану с размером пор, который соответствует целевому размеру липосомы (общие размеры пор для экспериментов NSE составляют 50 нм и 100 нм - как правило, липосомы диаметром 100 нм допускают менее ограниченные флуктуации мембраны, но меньшие 50 нм липосомы могут быть использованы для исследований кривизны). Убедитесь, что поликарбонатная мембрана полностью растянута перед завершением сборки и затягивания внешнего корпуса экструдера.
- Гидратировать поликарбонатную мембрану, пропуская ~0,3 мл D2O или D2O-буфера несколько раз через мембранный узел с помощью герметичных стеклянных шприцев. Используйте тот же буфер, который используется при подготовке образцов. Оставьте его не менее чем на 10 минут, затем полностью высохите буфер перед введением образца.
- Залить подготовленным липидным раствором газонепроницаемый шприц 1 мл и вставить в один конец экструдера. Затем вставьте пустой шприц в противоположный конец. Как только шприцы будут подключены к экструдеру в сборе, поместите его в блок экструдера.
- Если для экструзии необходимы повышенные температуры, как в случае насыщенных липидов с высокими температурами перехода (например, DSPC, Tm = 55 °C), предварительно нагрейте нагревательный блок экструдера выше температуры липидного перехода (например, 60 °C), поместив нагревательный блок на конфорку или используя циркуляционную ванну, как показано на рисунке 3A.
ПРИМЕЧАНИЕ: Этот этап имеет решающее значение для обеспечения однородного смешивания липидов и предотвращения экстремального давления во время экструзии, которое может разорвать поликарбонатную мембрану. Для липидных образцов с низкими температурами перехода (<25 °C) выполняют экструзию при комнатной температуре. - Чтобы экструзить липидный раствор, прикрепите экструдер к программируемой шприцевой помпе с алюминиевой/стальной рамой, как показано на рисунке 3А. Для экструзий с контролируемой температурой добавьте специально изготовленное основание экструдера с жидкостным каналом и прикрепите к циркуляции водяной бани.
- Запрограммировать шприцевой насос на выполнение 15-20 циклов экструзии в соответствии с инструкцией производителя. При экструдировании цвет раствора липидов изменяется от молочно-белого до прозрачного опалового синего(рисунок 3B,C),что указывает на конечный размер липосомы, который меньше длины волны видимого света, как и ожидалось. Для типа шприцевого насоса, показанного на рисунке 3A,выполните следующие действия.
- Начните с настройки насоса. Удерживая нажатой кнопку Rate, введите скорость экструзии (50,99 мл/ч), затем нажмите кнопку Diameter (Diameter) и введите диаметр шприца (4,606 мм). Используйте стрелки вверх под каждой цифрой на экране, чтобы изменить значение этой цифры.
- Поместите комплект экструдера с образцом шприца справа (см. рисунок 3A). Нажимайте кнопку «Снять», пока не загорится индикатор вывода. Нажмите кнопку Пуск и подождите, пока образец попадет в левый (пустой) шприц.
- Нажмите кнопку Стоп непосредственно перед тем, как образец (справа) шприц полностью пуст. Запишите дозированный объем и используйте его для программирования цикла экструзии. Удерживайте кнопку «Скорость», пока на экране не появится фаза 1 (PH:01). Нажмите кнопку громкости, чтобы ввести дозированный объем, записанный ранее. На этом этапе убедитесь, что индикатор Withdraw выключен — это распределяет образец в правильном направлении.
- Нажмите кнопку Rate еще раз и используйте правую стрелку вверх для доступа к фазе 2 (PH:02). Нажмите Volume ( Громкость), чтобы ввести то же значение дозированного объема, которое было записано ранее. На этом этапе нажимайте кнопку «Вывести», пока не загорится индикатор «Вывести» — это выведет образец влево.
- Чтобы повторить этот цикл, снова нажмите кнопку Rate и используйте стрелку вверх вправо для доступа к фазе 3 (PH: 03). Нажимайте кнопку громкости, пока на экране не появится LP:SE, и установите для нее значение 20. Это количество циклов или повторов, которые будет выполнять насос. Наконец, нажмите кнопку Rate, перейдите к фазе 4 (PH: 04) и нажмите кнопку громкости, чтобы нажать функцию Stop. Насос теперь настроен на автоматическую экструзию.
- Нажмите кнопку Пуск, чтобы начать цикл экструзии.
- Опорожнить шприц, содержащий экструдированную липидную суспензию, в чистом флаконе и подготовить к хранению или измерениям. Для липидных образцов с высокой температурой плавления храните образец выше фазового перехода жидкости до тех пор, пока не будет измерено. В противном случае храните образцы при комнатной температуре.
- Не замораживайте экструдированные образцы, так как замораживание приведет к разрыву везикул (суспензия снова станет молочно-белой).
4. Измерения NSE для образца (образцов) и сокращение собранных данных
- Перед экспериментом NSE охарактеризуйте экструдированный липосомальный образец с этапа 3.7, используя доступные методы для обеспечения адекватного качества образца. Список потенциальных методов харкатеризации, которые могут быть использованы для оценки качества липосомальных суспензий для экспериментов NSE, например, распределение по размерам, многоламелларность, структура боковой мембраны, включен в дискуссионный раздел.
- Определите Q-диапазон и соответствующие настройки прибора, необходимые для эксперимента. Для измерения жесткости изгиба липидных бислоев используйте Q-диапазон ~(0,04 - 0,2) Å-1. Для исследования колебаний толщины мембраны используют Q-диапазон ~(0,04 - 0,2) Å-1, соответствующий толщине мембраны35,66,67.
ПРИМЕЧАНИЕ: Обсудите экспериментальную установку с ученым-прибористом до начала эксперимента. Как упоминалось ранее, характеристика образца SANS необходима, особенно если предварительная информация о сигнале рассеяния недоступна, как в селективно дейтерированных мембранах. В качестве альтернативы можно провести статические (также известные как дифракционные) измерения в ограниченном Q-диапазоне на приборе NSE с оговоркой, что такие измерения занимают гораздо больше времени по сравнению с SANS. - Используя шприц или передаточную пипетку, загрузите экструдированную липосомальную суспензию (суспензии) в назначенные ячейки образца, доступные на линиях пучка NSE. Обратите внимание, что стандартные образцы NSE бывают толщиной 1, 2, 3 и 4 мм. Выберите толщину ячейки таким образом, чтобы оптимизировать сигнал рассеяния, сохраняя при этом некогерентный фоновый сигнал с разумной интенсивностью.
ПРИМЕЧАНИЕ: Как правило, используйте образцы клеток с протяженностью 1 или 2 мм для протиированных липосом в дейтерированном буфере - более толстые клетки могут привести к множественным эффектам рассеяния, которые трудно исправить. Для липосом с более высоким уровнем дейтрации (например, липосомы, соответствующие контрасту хвоста или асимметричные липосомы с одиночными протитированными листочками) рассмотрите возможность использования более толстой клетки образца (например, 3 или 4 мм), чтобы улучшить статистику подсчета, если образец доступен в больших количествах - иногда это может быть непомерно дорого. - Подготовьте идентичную ячейку образца для буфера. Используйте тот же буфер, что и в липосомальной суспензии. Измерения на буфере необходимы для нормализации интенсивности и коррекции фона (BKG).
- Поместите ячейку (ячейки) образца в держатель образца спектрометра NSE, запрограммируем измерительные прогоны и соберите эхо-данные. Проконсультируйтесь с ученым-приборостроителем о программировании измерений, если вы впервые пользователь NSE.
- Выполните два дополнительных набора измерений, необходимых для уменьшения данных: измерения разрешения(R)и передачи(T).
- Выполнить измерение разрешения(R)на упругом эталоне рассеяния (например, углероде) — для запуска в тех же настройках; т.е. тот же волновой вектор и раз Фурье, что и измерения образца и буфера.
- Выполните измерения пропускания(T)на образце и буфере для расчета интенсивности передаваемого нейтронного пучка (см. шаг 4.9 ниже). Передача рассчитывается как отношение количества нейтронов от образца или буфера, деленное на количество нейтронов для открытого пучка (т.е. с пустым положением образца).
- Используйте специальное программное обеспечение для уменьшения данных для спектрометра NSE, на котором выполняются измерения, чтобы уменьшить собранные данные.
ПРИМЕЧАНИЕ: Различные спектрометры могут использовать различное программное обеспечение или пользовательские интерфейсы. Ниже приведен пример сокращения данных NSE с помощью среды анализа и визуализации данных (DAVE)68 программное обеспечение, специально написанное для спектрометра NSE в Центре нейтронных исследований NIST.- Откройте программное обеспечение DAVE и выберите «Уменьшить данные NSE» в меню сокращения данных. Появится несколько всплывающих окон.
- Загрузите файлы данных с различными значениями Q с помощью команды Открыть ECHO-файлы в меню файл. Эти файлы соответствуют файлам необработанных данных с сигналами спин-эха и имеют расширение .echo в имени файла. После завершения загрузки файла файлы будут отображаться под доступными наборами данных.
- Щелкните правой кнопкой мыши на выбранном файле и пометьте его в соответствии с измерением, которое он соответствует; т.е. Образец, Ячейка (для пустой ячейки или буфера) или Разрешение.
- Сгруппируйте пиксели детектора в 2 x 2, чтобы улучшить отношение сигнал/шум с помощью вкладки Набор данных. Применение одного и того же биннинга ко всем файлам; т.е. Разрешение, Ячейка и Образец.
- Проверьте данные по всем группам пикселей и замаскируйте данные с плохими сигналами (см. рисунок 4B),нажав клавишу m на клавиатуре. Нажмите клавишу ВВОД, чтобы открыть всплывающее окно, чтобы применить одну и ту же маску ко всем разам Фурье или последующим разам Фурье. Это также может быть применено к отдельным пикселям в любой момент во время сокращения данных. Замаскированные пиксели станут зелеными.
- Убедитесь, что собранные данные в виде эхо-сигнала, т.е. косинусной функции с точки зрения фазового тока, над каждым пикселем детектора (см. Рисунок 4А).
ПРИМЕЧАНИЕ: Фазовый ток пропорционален углу прецессии спина нейтрона; следовательно, обычно представляется фазовый ток в виде фазового угла, как показано на рисунке 4A. Для измерений на импульсных источниках к данным применяются дополнительные расчеты времени полета для получения эхо-сигналов в зависимости от длины волны падающего нейтрона в нейтронном импульсе. - Начните с установки файла разрешения. Выберите файл разрешения из списка загруженных файлов и щелкните его правой кнопкой мыши. Выберите «Операции подгонки: эхо-отсыпать (разрешение)» во всплывающем меню.
- Убедитесь, что припадки эхо-сигналов дают ряд параметров подгонки, включая параметр A,требуемый на шаге 4.8. Подгомка выполняется автоматически с помощью следующего выражения.
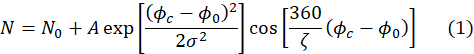
Здесь ζ — период эхо-сигнала (т. е. косинусная функция на рисунке 4A),σ — ширина гауссовской оболочки, определяемая средней длиной волны и разбросом длин волны падающего пучка нейтронов, Φc — фазовый ток, а Φ0 — точка эха, которая зависит от траектории поля, испытываемой нейтронами50. Физическая информация о образце кодируется в амплитуде, А,косинусной функции в уравнении (1).
ПРИМЕЧАНИЕ: Ширина гауссовской оболочки основана на значениях, заранее заданных ученым-приборостроителем, и не должна изменяться. Другие параметры представляют собой переменные, которые подогнаны к конкретному эхо-сигналу над каждым пикселем. - Проверьте результаты подгонки, щелкнув по каждому пикселю, чтобы показать результирующие параметры подгонки, качество посадки и среднее квадратное отклонение подгонки. Чтобы проверить ошибку, связанную с каждым параметром подгонки, по всему детектору, выберите Параметры изображения, а затем выберите интересующую параметр подгонки. Это сгенерирует карту со значением подходящего параметра над каждым пикселем. Щелкните правой кнопкой мыши на изображении детектора. Появится всплывающее окно с картой строки ошибок выбранного параметра подгонки.
- Если подгонка над определенным пикселем неудовлетворительна (например, подгонка паравечеров с большими полосами ошибок), перенастраить сигнал над этим конкретным пикселем. Выделите этот пиксель,перейдите на вкладку Подгонка, а затем нажмите кнопку По пикселю по выбору. Введите новые начальные параметры для фазы(Φ0)и периода (ζ) на вкладке Fitting для получения более удовлетворительного соответствия.
ПРИМЕЧАНИЕ: Полезно построить установленную фазу как функцию времени Фурье. Для этого перейдите в главное окно сюжета и выберите Fit Phase v. Fourier Time. Этот сюжет должен быть плавным и непрерывным. Проверьте разрывы на этом графике и переоборудуйте пиксели, которым они соответствуют.
- Уменьшите размер файла образца или ячейки, выбрав соответствующий файл из списка загруженных и помеченных файлов.
- Проверьте все пиксели и замаскируйте те, которые с плохой статистикой, как описано в шаге 4.7.5.
- Щелкните правой кнопкой мыши файл и выберите Операции подгонки: этапы импорта (образец, ячейка). При этом фазы и примененная маска импортируются из файла Resolution.
- Подгонка эхо-сигналов с помощью той же процедуры, описанной ранее для файла Resolution (шаги 4.7.8-4.7.10). При установке файлов Sample и Cell не изменяйте значения точки и фазы эха, импортированных из resolution fits. Эти параметры зависят от инструментальных настроек и не должны изменяться в зависимости от сэмплов.
- Прежде чем приступить к уменьшению данных, введите центр луча для всех файлов данных. Выберите файл данных, перейдите на вкладку Общие и введите центральные значения луча X и Y. Эти значения записываются во время эксперимента.
- После завершения подгонки файлов Sample, Cell и Resolution рассчитайте нормализованную промежуточную функцию рассеяния, которая будет использоваться позже при анализе и интерпретации данных. Для этого щелкните правой кнопкой мыши файл Sample, который нужно уменьшить из списка подходящих файлов, и выберите Calculate I(Q) во всплывающем меню. Появится окно с вариантами ввода для файлов Resolution и Cell (т.е. буфера), а также количеством Q-дуг (см. шаг 4.9). После ввода всей необходимой информации нажмите кнопку OK. Результаты появятся в новом окне.
ПРИМЕЧАНИЕ: Редукция данных выполняется по следующему уравнению для получения нормализованной промежуточной функции рассеяния69.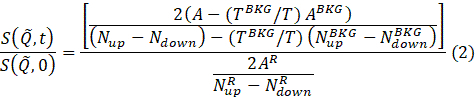
где t — время Фурье, Nвверх и Nвниз — количество нейтронов в конфигурациях без спин-флипа и спин-флипа (измеренное с выключенными π/2-ластами и π-флиппером, соответственно), а надстрочные индексы, BKG и R,соответствуют измерениям фона и разрешения, соответственно, как определено в шагах 4.4 и 4.6. Отметим, что поляризация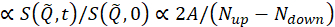 пучка, при этом изменение спинового состояния из-за обмена энергией между нейтроном и образцом обнаруживается как падение поляризации (от единицы).
пучка, при этом изменение спинового состояния из-за обмена энергией между нейтроном и образцом обнаруживается как падение поляризации (от единицы).
- Наконец, сгруппируйте пиксели детектора в Q-дуги,как показано на рисунке 4B, чтобы получить Q-зависимостьнормализованной промежуточной функции рассеяния, S(Q,t) / S(Q, 0 ). Технически это называется биннингом данных и должно быть сделано разумно, т.е. с учетом статистики подсчета из выборки и ожидаемого стандартного отклонения данных над сгруппированными пикселями.
- Для образцов с сильным рассеянием разделите детектор на большее количество Q-дуг, сохраняя при этом разумные полосы ошибок на результиимной промежуточной функции рассеяния, S(Q,t) / S(Q, 0 ). Это дает больше точек данных Q и важно для процедуры анализа данных, описанной ниже. Имейте в виду, что для слабо рассеянных образцов чрезмерное бинирование приводит к плохим сигналам распада, то есть большим полосам ошибок на S(Q,t) / S(Q, 0), что может привести к большим неопределенностям.
5. Анализ и интерпретация данных
- Подойдут нормализованные промежуточные функции рассеяния S(Q,t) / S(Q,0), полученные из приведенного выше сведения данных до растянутой экспоненциальной функции с растягивающей экспонентой 2/370.
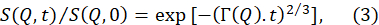
ПРИМЕЧАНИЕ: Пример этих подходов приведен на рисунке 5B. Припадки S(Q,t) / S(Q,0) к уравнению (3) дают Q-зависимые скорости релаксации Γ (Q). - График Γ(Q)как функция Q и подходит к подходящей модели для извлечения соответствующих параметров мембраны.
Результаты
Исследования NSE, обращающиеся к флуктуациям изгиба, обычно выполняются в Q-диапазоне ~ (0,04 - 0,2) Å-1. Этот Q-диапазон соответствует шкалам промежуточной длины между толщиной мембраны и липосомальным радиусом, где доминирует динамика изгиба. Измерение в расширенном Q-диапазоне может да...
Обсуждение
NSE является мощным и уникальным методом измерения мезоскопической динамики липидных мембран в различных условиях. Эффективное использование NSE зависит от качества образца, контраста нейтронов и диапазона доступной динамики, которая может быть исследована для данного образца. Таким о?...
Раскрытие информации
Авторы заявляют об отсутствии конфликта интересов и нечего раскрывать.
Благодарности
Р. Ашкар благодарит М. Нагао, Л.-Р. Стингачу и. Золнерчук за многие полезные дискуссии и за их частую помощь в экспериментах NSE на их соответствующих линиях луча. Авторы признают использование нейтронных спиновых эхо-спектрометров в NIST и ORNL. Спектрометр NSE в NIST поддерживается Центром рассеяния нейтронов высокого разрешения, партнерством между Национальным институтом стандартов и технологий и Национальным научным фондом по соглашению No. ДМР-1508249. Спектрометр NSE в источнике нейтронов ORNL поддерживается Отделом научных пользовательских установок Управления фундаментальных энергетических наук Министерства энергетики США. Национальная лаборатория Оук-Ридж управляется UT-Battelle, LLC в соответствии с контрактом NO 2 США. DE-AC05-00OR22725.
Материалы
| Name | Company | Catalog Number | Comments |
| Chloroform (biotech grade) | Sigma Aldrich | 496189 | Biotech. grade, ≥99.8%, contains 0.5-1.0% ethanol as stabilizer |
| Circulating water bath | Julabo | SE-12 | Heating Circulator with smart pump, programmable temperature settings, and external sensor connection for measurement and control |
| Deuterium Oxide | Cambridge Isotopes Laboratories | DLM-4 | Deuterated water; Heavy water (D2O) (D, 99.9%) |
| Digital Semi-Microbalance | Mettler Toledo | MS105 | Semi-micro balance with 120 g capacity, 0.01 mg readability, high resolution weighing cell, ergonomic doors, and pipette-check application |
| Ethanol (molecular biology grade) | Sigma Aldrich | E7023 | 200 proof ethanol for molecular biology applications |
| Glass Pipets | VWR | 36360-536 | Disposable Soda Lime glass Pasteur pipets |
| Glass Vials | Thermo Scientific | B7990-1 | Borosilicate glass vials with PTFE/Silione septum caps |
| Lab grade freezer | Fisher Scientific | IU2886D | Ultra-low temprature freezer (-86 to -50 C) for long-term storage of lipids and proteins |
| Lipids (protaited or perdeuterated) | Avanti Polar Lipids | varies by lipid | Lipids can be purchased from Avanti in powder form or in a chloroform solution with the required amounts and deuteration schemes. |
| Millipore water purifier | Millipore Sigma | ZRQSVP3US | Direct-Q® 3 UV Water Purification System which deliver both pure and ultrapure water with a built-in UV lamp to reduce the levels of organics for biological applications |
| Mini Extruder Set | Avanti Polar Lipids | 610020 | Mini-extruder set includes mini-extruder, heating block, 2 GasTight Syringes, and 2 O-rings, Polycarbonate Membranes, and Filter Supports |
| Quick Connect Fittings | Grainger | 2YDA1 and 2YDA7 | Push-button tube fittings for QuickConnect water circulation applications, e.g. high temperature vesicle extrusion |
| Syringe Pump | SyringePump.com | New Era-1000 | Fully programmable syringe pump for infusion and withdrawal; programs up to 41 pumping phases with adjustable pumping rates, dispensed volumes, and extrusion cycles |
| Ultrasonic bath | Fisher Scientific | CPX2800 | Temperature controlled ultra sonic bath with programmable functionality for degassing and ultrasonic applications |
| Vacuum Oven | Thermo Scientific | 3608 | 0.7 cu ft vaccum oven with built-in-high-limit thermostat guards against overheating |
| Vortex Mixer | Fisher Scientific | 02-215-414 | Variable speed, analog control that allows low rpm start-up for gentle shaking or high-speed mixing for vigorous vortexing of samples |
Ссылки
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175 (4023), 720-731 (1972).
- Andersen, O. S., Koeppe, R. E. Bilayer thickness and membrane protein function: an energetic perspective. Annual Review of Biophysics and Biomolecular Structure. 36, 107-130 (2007).
- Lundbæk, J. A., Collingwood, S. A., Ingólfsson, H. I., Kapoor, R., Andersen, O. S. Lipid bilayer regulation of membrane protein function: gramicidin channels as molecular force probes. Journal of The Royal Society Interface. 7 (44), 373-395 (2010).
- Bradley, R. P., Radhakrishnan, R. Curvature-undulation coupling as a basis for curvature sensing and generation in bilayer membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 117-124 (2016).
- Perozo, E., Cortes, D. M., Sompornpisut, P., Kloda, A., Martinac, B. Open channel structure of MscL and the gating mechanism of mechanosensitive channels. Nature. 418 (6901), 942-948 (2002).
- Jensen, M. &. #. 2. 1. 6. ;., Mouritsen, O. G. Lipids do influence protein function-the hydrophobic matching hypothesis revisited. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1666 (1-2), 205-226 (2004).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118 (6), 1099-1102 (2005).
- Katchalsky, A., Spangler, R. Dynamics of membrane processes. Quarterly Reviews of Biophysics. 1 (2), 127-175 (1968).
- Rheinstädter, M. C. Collective molecular dynamics in proteins and membranes (Review). Biointerphases. 3 (2), 83-90 (2008).
- Fujiwara, T., Ritchie, K., Murakoshi, H., Jacobson, K., Kusumi, A. Phospholipids undergo hop diffusion in compartmentalized cell membrane. The Journal of Cell Biology. 157 (6), 1071-1082 (2002).
- Hac, A. E., Seeger, H. M., Fidorra, M., Heimburg, T. Diffusion in two-component lipid membranes--a fluorescence correlation spectroscopy and monte carlo simulation study. Biophysical Journal. 88 (1), 317-333 (2005).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proceedings of the National Academy of Sciences of the United States of America. 107 (16), 7208-7213 (2010).
- Hormel, T. T., Kurihara, S. Q., Brennan, M. K., Wozniak, M. C., Parthasarathy, R. Measuring lipid membrane viscosity using rotational and translational probe diffusion. Physical Review Letters. 112 (18), 188101 (2014).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Advances in Colloid and Interface Science. 208, 225-234 (2014).
- Bassereau, P., Sorre, B., Lévy, A. Bending lipid membranes: Experiments after W. Helfrich's model. Advances in Colloid and Interface Science. 208, 47-57 (2014).
- Monzel, C., Sengupta, K. Measuring shape fluctuations in biological membranes. Journal of Physics D: Applied Physics. 49 (24), 243002 (2016).
- Deserno, M. Mesoscopic membrane physics: concepts, simulations, and selected applications. Macromolecular Rapid Communications. 30 (9-10), 752-771 (2009).
- Reynwar, B. J., et al. Aggregation and vesiculation of membrane proteins by curvature-mediated interactions. Nature. 447 (7143), 461-464 (2007).
- Haswell, E. S., Phillips, R., Rees, D. C. Mechanosensitive channels: what can they do and how do they do it. Structure. 19 (10), 1356-1369 (2011).
- Phillips, R., Ursell, T., Wiggins, P., Sens, P. Emerging roles for lipids in shaping membrane-protein function. Nature. 459 (7245), 379-385 (2009).
- Dill, K. A., Chan, H. S. From Levinthal to pathways to funnels. Nature Structural Biology. 4 (1), 10-19 (1997).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Lyman, E., Hsieh, C. -. L., Eggeling, C. From dynamics to membrane organization: experimental breakthroughs occasion a "modeling manifesto". Biophysical Journal. 115 (4), 595-604 (2018).
- Arriaga, L. R., et al. Dissipative curvature fluctuations in bilayer vesicles: Coexistence of pure-bending and hybrid curvature-compression modes. The European Physical Journal. E, Soft Matter. 31 (1), 105-113 (2010).
- Honerkamp-Smith, A. R., Veatch, S. L., Keller, S. L. An introduction to critical points for biophysicists; observations of compositional heterogeneity in lipid membranes. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1788 (1), 53-63 (2009).
- Veatch, S. L., Keller, S. L. Organization in lipid membranes containing cholesterol. Physical Review Letters. 89 (26), 268101 (2002).
- Heberle, F. A., et al. Bilayer thickness mismatch controls domain size in model membranes. Journal of the American Chemical Society. 135 (18), 6853-6859 (2013).
- Nickels, J. D., et al. The in vivo structure of biological membranes and evidence for lipid domains. PLOS Biology. 15 (5), 2002214 (2017).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387 (6633), 569-572 (1997).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews. Molecular Cell Biology. 9 (2), 112-124 (2008).
- Liu, S. -. L., et al. Orthogonal lipid sensors identify transbilayer asymmetry of plasma membrane cholesterol. Nature Chemical Biology. 13, 268 (2016).
- Rothman, J., Lenard, J. Membrane asymmetry. Science. 195 (4280), 743-753 (1977).
- Ashkar, R., et al. Neutron scattering in the biological sciences: progress and prospects. Acta Crystallographica Section D. 74 (12), 1129-1168 (2018).
- Woodka, A. C., Butler, P. D., Porcar, L., Farago, B., Nagao, M. Lipid bilayers and membrane dynamics: insight into thickness fluctuations. Physical Review Letters. 109 (5), 058102 (2012).
- Chakraborty, S., et al. How cholesterol stiffens unsaturated lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21896-21905 (2020).
- Arriaga, L. R., et al. Stiffening effect of cholesterol on disordered lipid phases: a combined neutron spin echo + dynamic light scattering analysis of the bending elasticity of large unilamellar vesicles. Biophysical Journal. 96 (9), 3629-3637 (2009).
- Nagao, M., Kelley, E. G., Ashkar, R., Bradbury, R., Butler, P. D. Probing elastic and viscous properties of phospholipid bilayers using neutron spin echo spectroscopy. The Journal of Physical Chemistry Letters. 8 (19), 4679-4684 (2017).
- Kelley, E. G., Butler, P. D., Ashkar, R., Bradbury, R., Nagao, M. Scaling relationships for the elastic moduli and viscosity of mixed lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (38), 23365-23373 (2020).
- Rickeard, B. W., et al. Transverse lipid organization dictates bending fluctuations in model plasma membranes. Nanoscale. 12 (3), 1438-1447 (2020).
- Nickels, J. D., et al. Mechanical properties of nanoscopic lipid domains. Journal of the American Chemical Society. 137 (50), 15772-15780 (2015).
- Mezei, F. Neutron spin echo: A new concept in polarized thermal neutron techniques. Zeitschrift für Physik A Hadrons and Nuclei. 255 (2), 146-160 (1972).
- Hayter, J. B., Penfold, J. Neutron spin-echo integral transform spectroscopy. Zeitschrift für Physik B Condensed Matter. 35 (2), 199-205 (1979).
- Monkenbusch, M., Richter, D., Imae, T., Kanaya, T., Furusaka, M., Torikai, N. . Neutrons in Soft Matter. , 147-182 (2011).
- Pynn, R., Mezei, F., Pappas, C., Gutberlet, T. . Neutron Spin Echo. , 159-177 (2003).
- Holderer, O., et al. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 19 (3), 034022 (2008).
- Schleger, P., et al. The long-wavelength neutron spin-echo spectrometer IN15 at the Institut Laue-Langevin. Physica B: Condensed Matter. 241-243, 164-165 (1997).
- Holderer, O., Zolnierczuk, P., Pasini, S., Stingaciu, L., Monkenbusch, M. A better view through new glasses: Developments at the Jülich neutron spin echo spectrometers. Physica B: Condensed Matter. 562, 9-12 (2019).
- Farago, B., et al. The IN15 upgrade. Neutron News. 26 (3), 15-17 (2015).
- Ashkar, R. Selective dynamics in polymeric materials: Insights from quasi-elastic neutron scattering spectroscopy. Journal of Applied Physics. 127 (15), 151101 (2020).
- Pasini, S., Holderer, O., Kozielewski, T., Richter, D., Phoenix Monkenbusch, M. J-NSE- Phoenix, a neutron spin-echo spectrometer with optimized superconducting precession coils at the MLZ in Garching. Review of Scientific Instruments. 90 (4), 043107 (2019).
- Svergun, D. I., Koch, M. H. J., Timmins, P. A., May, R. P. . Small Angle X-Ray and Neutron Scattering from Solutions of Biological Macromolecules. , (2013).
- Eicher, B., et al. Joint small-angle X-ray and neutron scattering data analysis of asymmetric lipid vesicles. Journal of Applied Crystallography. 50 (2), 419-429 (2017).
- Heberle, F. A., et al. Model-based approaches for the determination of lipid bilayer structure from small-angle neutron and X-ray scattering data. European Biophysics Journal. 41 (10), 875-890 (2012).
- Jaksch, S., Koutsioubas, A., Mattauch, S., Holderer, O., Frielinghaus, H. Long-range excitations in phospholipid membranes. Chemistry and Physics of Lipids. 225, 104788 (2019).
- Jaksch, S., et al. Influence of ibuprofen on phospholipid membranes. Physical Review E. 91 (2), 022716 (2015).
- Armstrong, C. L., et al. Effect of cholesterol on the lateral nanoscale dynamics of fluid membranes. European Biophysics Journal. 41 (10), 901-913 (2012).
- Rheinstädter, M. C., Häußler, W., Salditt, T. Dispersion relation of lipid membrane shape fluctuations by neutron spin-echo spectrometry. Physical Review Letters. 97 (4), 048103 (2006).
- Armstrong, C. L., Häußler, W., Seydel, T., Katsaras, J., Rheinstädter, M. C. Nanosecond lipid dynamics in membranes containing cholesterol. Soft Matter. 10 (15), 2600-2611 (2014).
- Nickels, J. D., et al. Lipid rafts: buffers of cell membrane physical properties. The Journal of Physical Chemistry B. 123 (9), 2050-2056 (2019).
- Michonova-Alexova, E. I., Sugár, I. P. Component and state separation in DMPC/DSPC lipid bilayers: a Monte Carlo simulation study. Biophysical Journal. 83 (4), 1820-1833 (2002).
- Sugár, I. P., Thompson, T. E., Biltonen, R. L. Monte Carlo simulation of two-component bilayers: DMPC/DSPC mixtures. Biophysical Journal. 76 (4), 2099-2110 (1999).
- Mabrey, S., Sturtevant, J. M. Investigation of phase transitions of lipids and lipid mixtures by sensitivity differential scanning calorimetry. Proceedings of the National Academy of Sciences. 73 (11), 3862-3866 (1976).
- . Neutron activation and scattering calculator Available from: https://www.ncnr.nist.gov/resources/activation/ (2021)
- Scott, H. L., et al. On the mechanism of bilayer separation by extrusion, or why your LUVs are not really unilamellar. Biophysical Journal. 117 (8), 1381-1386 (2019).
- Ashkar, R., et al. Tuning membrane thickness fluctuations in model lipid bilayers. Biophysical Journal. 109 (1), 106-112 (2015).
- Carrillo, J. -. M. Y., Katsaras, J., Sumpter, B. G., Ashkar, R. A computational approach for modeling neutron scattering data from lipid bilayers. Journal of Chemical Theory and Computation. 13 (2), 916-925 (2017).
- Azuah, R. T. DAVE: a comprehensive software suite for the reduction, visualization, and analysis of low energy neutron spectroscopic data. Journal of Research of the National Institute of Standards and Technology. 114 (6), 341-358 (2009).
- Van Hove, L. Correlations in space and time and born approximation scattering in systems of interacting particles. Physical Review. 95 (1), 249-262 (1954).
- Zilman, A. G., Granek, R. Undulations and dynamic structure factor of membranes. Physical Review Letters. 77 (23), 4788-4791 (1996).
- Kelley, E. G., Butler, P. D., Nagao, M. . Collective dynamics in model biological membranes measured by neutron spin echo spectroscopy. , 131-176 (2019).
- Zheng, Y., Michihiro, N., Dobrin, P. B. Bending elasticity of saturated and monounsaturated phospholipid membranes studied by the neutron spin echo technique. Journal of Physics: Condensed Matter. 21 (15), 155104 (2009).
- Sharma, V. K., Qian, S. Effect of an antimicrobial peptide on lateral segregation of lipids: a structure and dynamics study by neutron scattering. Langmuir. 35 (11), 4152-4160 (2019).
- Boggara, M. B., Faraone, A., Krishnamoorti, R. Effect of pH and Ibuprofen on the Phospholipid Bilayer Bending Modulus. The Journal of Physical Chemistry B. 114 (24), 8061-8066 (2010).
- Lee, J. -. H., et al. Thermal fluctuation and elasticity of lipid vesicles interacting with pore-forming peptides. Physical Review Letters. 105 (3), 038101 (2010).
- Chakraborty, S., Abbasi, A., Bothun, G. D., Nagao, M., Kitchens, C. L. Phospholipid bilayer softening due to hydrophobic gold nanoparticle inclusions. Langmuir. 34 (44), 13416-13425 (2018).
- Hoffmann, I., et al. Softening of phospholipid membranes by the adhesion of silica nanoparticles - as seen by neutron spin-echo (NSE). Nanoscale. 6 (12), 6945-6952 (2014).
- Watson, M. C., Brown, F. L. H. Interpreting membrane scattering experiments at the mesoscale: the contribution of dissipation within the bilayer. Biophysical Journal. 98 (6), 9-11 (2010).
- Seifert, U., Langer, S. A. Viscous modes of fluid bilayer membranes. Europhysics Letters (EPL). 23 (1), 71-76 (1993).
- Bingham, R. J., Smye, S. W., Olmsted, P. D. Dynamics of an asymmetric bilayer lipid membrane in a viscous solvent. EPL (Europhysics Letters). 111 (1), 18004 (2015).
- Rawicz, W., Olbrich, K. C., McIntosh, T., Needham, D., Evans, E. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophysical Journal. 79 (1), 328-339 (2000).
- Doktorova, M., LeVine, M. V., Khelashvili, G., Weinstein, H. A new computational method for membrane compressibility: bilayer mechanical thickness revisited. Biophysical Journal. 116 (3), 487-502 (2019).
- Evans, E., Needham, D. Physical properties of surfactant bilayer membranes: thermal transitions, elasticity, rigidity, cohesion and colloidal interactions. The Journal of Physical Chemistry. 91 (16), 4219-4228 (1987).
- Lesieur, S., Grabielle-Madelmont, C., Paternostre, M. T., Ollivon, M. Size analysis and stability study of lipid vesicles by high-performance gel exclusion chromatography, turbidity, and dynamic light scattering. Analytical Biochemistry. 192 (2), 334-343 (1991).
- Heberle, F. A., et al. Direct label-free imaging of nanodomains in biomimetic and biological membranes by cryogenic electron microscopy. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19943-19952 (2020).
- Cornell, C. E., Mileant, A., Thakkar, N., Lee, K. K., Keller, S. L. Direct imaging of liquid domains in membranes by cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19713-19719 (2020).
- Yao, X., Fan, X., Yan, N. Cryo-EM analysis of a membrane protein embedded in the liposome. Proceedings of the National Academy of Sciences of the United States of America. 117 (31), 18497-18503 (2020).
- Kučerka, N., Nieh, M. -. P., Katsaras, J. Fluid phase lipid areas and bilayer thicknesses of commonly used phosphatidylcholines as a function of temperature. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1808 (11), 2761-2771 (2011).
- Nielsen, J. E., Bjørnestad, V. A., Lund, R. Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin. Soft Matter. 14 (43), 8750-8763 (2018).
- Kučerka, N., et al. Lipid bilayer structure determined by the simultaneous analysis of neutron and X-ray scattering data. Biophysical Journal. 95 (5), 2356-2367 (2008).
- Kelley, E. G., Butler, P. D., Nagao, M. Scaling of lipid membrane rigidity with domain area fraction. Soft Matter. 15 (13), 2762-2767 (2019).
- Brüning, B. -. A., et al. Bilayer undulation dynamics in unilamellar phospholipid vesicles: Effect of temperature, cholesterol and trehalose. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1838 (10), 2412-2419 (2014).
- Kučerka, N., et al. Areas of monounsaturated diacylphosphatidylcholines. Biophysical Journal. 97 (7), 1926-1932 (2009).
- Sharma, V. K., Mamontov, E., Anunciado, D. B., O'Neill, H., Urban, V. S. Effect of antimicrobial peptide on the dynamics of phosphocholine membrane: role of cholesterol and physical state of bilayer. Soft Matter. 11 (34), 6755-6767 (2015).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in lipid membranes containing transmembrane peptides. Soft Matter. , (2021).
- Yu, J., et al. Structure and dynamics of lipid membranes interacting with antivirulence end-phosphorylated polyethylene glycol block copolymers. Soft Matter. 16 (4), 983-989 (2020).
- Stingaciu, L. -. R., et al. Revealing the dynamics of thylakoid membranes in living cyanobacterial cells. Scientific Reports. 6 (1), 19627 (2016).
- Stingaciu, L. -. R., O'Neill, H. M., Liberton, M., Pakrasi, H. B., Urban, V. S. Influence of chemically disrupted photosynthesis on cyanobacterial thylakoid dynamics in synechocystis sp. PCC 6803. Scientific Reports. 9 (1), 5711 (2019).
- Miller, I. R. Energetics of fluctuation in lipid bilayer thickness. Biophysical Journal. 45 (3), 643-644 (1984).
- Nagao, M. Observation of local thickness fluctuations in surfactant membranes using neutron spin echo. Physical Review E. 80 (3), 031606 (2009).
Erratum
Formal Correction: Erratum: Neutron Spin Echo Spectroscopy as a Unique Probe for Lipid Membrane Dynamics and Membrane-Protein Interactions
Posted by JoVE Editors on 8/06/2021. Citeable Link.
An erratum was issued for: Neutron Spin Echo Spectroscopy as a Unique Probe for Lipid Membrane Dynamics and Membrane-Protein Interactions. The Introduction, Protocol, and Representative Results sections have been updated.
In the Introduction, the fith pargraph was updated from:
Besides direct access to the length and time scale of membrane dynamics, NSE has the inherent capabilities of neutron isotope sensitivity52. Specifically, the ability of neutrons to interact differently with the isotopes of hydrogen, the most abundant element in biological systems, results in a different neutron scattering length density,34 or NSLD (the equivalent of the optical index of refraction50), when protium is substituted by deuterium. This enables an approach known as contrast variation, which is commonly used to highlight specific membrane features or conceal others — the latter scenario is referred to as contrast matching. A frequent application of contrast variation/matching is the substitution of water (NSLD = -0.56 × 10-6 Å-2) by heavy water or D2O (NSLD = 6.4 × 10-6 Å-2) to amplify the neutron signal from protiated lipid membranes (NSLD ~ 2 × 10-6 Å-2). This approach is highly effective in studies of membrane structure because the penetration of D2O into the headgroup region of the membrane allows accurate determination of the membrane thicknesses (see Figure 2A, left panel) and of the location of different lipid subgroups when more sophisticated models are applied53,54. This paper highlights some examples on the use of contrast variation for studies of collective dynamics in biomimetic membranes and select membrane features.
to:
Besides direct access to the length and time scale of membrane dynamics, NSE has the inherent capabilities of neutron isotope sensitivity52. Specifically, the ability of neutrons to interact differently with the isotopes of hydrogen, the most abundant element in biological systems, results in a different neutron scattering length density,34 or NSLD (the equivalent of the optical index of refraction50), when protium is substituted by deuterium. This enables an approach known as contrast variation, which is commonly used to highlight specific membrane features or conceal others — the latter scenario is referred to as contrast matching. A frequent application of contrast variation/matching is the substitution of water (NSLD = -0.56 × 10-6 Å-2) by heavy water or D2O (NSLD = 6.4 × 10-6 Å-2) to amplify the neutron signal from protiated lipid membranes (NSLD ~ 0 × 10-6 Å-2). This approach is highly effective in studies of membrane structure because the penetration of D2O into the headgroup region of the membrane allows accurate determination of the membrane thicknesses (see Figure 2A, left panel) and of the location of different lipid subgroups when more sophisticated models are applied53,54. This paper highlights some examples on the use of contrast variation for studies of collective dynamics in biomimetic membranes and select membrane features.
In the Protocol, step 1.1 was updated from:
For bending fluctuation measurements, make fully protiated liposomes in D2O (D 99.9%) or D2O-buffer (e.g., phosphate buffer prepared with D2O instead of H2O). Use fully protiated DMPC (C36H72NO8P) and DSPC (C44H88NO8P) with 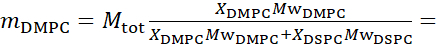 133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 2 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 2 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
to:
For bending fluctuation measurements, make fully protiated liposomes in D2O (D 99.9%) or D2O-buffer (e.g., phosphate buffer prepared with D2O instead of H2O). Use fully protiated DMPC (C36H72NO8P) and DSPC (C44H88NO8P) with 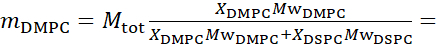 133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 0 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 0 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
In the Representative Results, the fist pagargaph was updted from:
NSE studies accessing bending fluctuations are typically performed over a Q-range of ~ (0.04 - 0.2) Å-1. This Q-range corresponds to intermediate length scales between the membrane thickness and the liposomal radius, where bending dynamics dominate. Measurement over an extended Q-range can give access to additional dynamic modes, including liposomal diffusion and intramembrane dynamics. For more details on the cross-over in membrane dynamics accessed by NSE, check these relevant publications25,71. It is important to emphasize that NSE signals are proportional to: 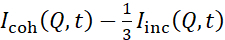 , where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~2 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
, where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~2 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
to:
NSE studies accessing bending fluctuations are typically performed over a Q-range of ~ (0.04 - 0.2) Å-1. This Q-range corresponds to intermediate length scales between the membrane thickness and the liposomal radius, where bending dynamics dominate. Measurement over an extended Q-range can give access to additional dynamic modes, including liposomal diffusion and intramembrane dynamics. For more details on the cross-over in membrane dynamics accessed by NSE, check these relevant publications25,71. It is important to emphasize that NSE signals are proportional to: 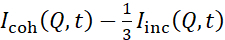 , where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~0 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
, where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~0 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
In the Representative Reults, Figure 2 was updated from:
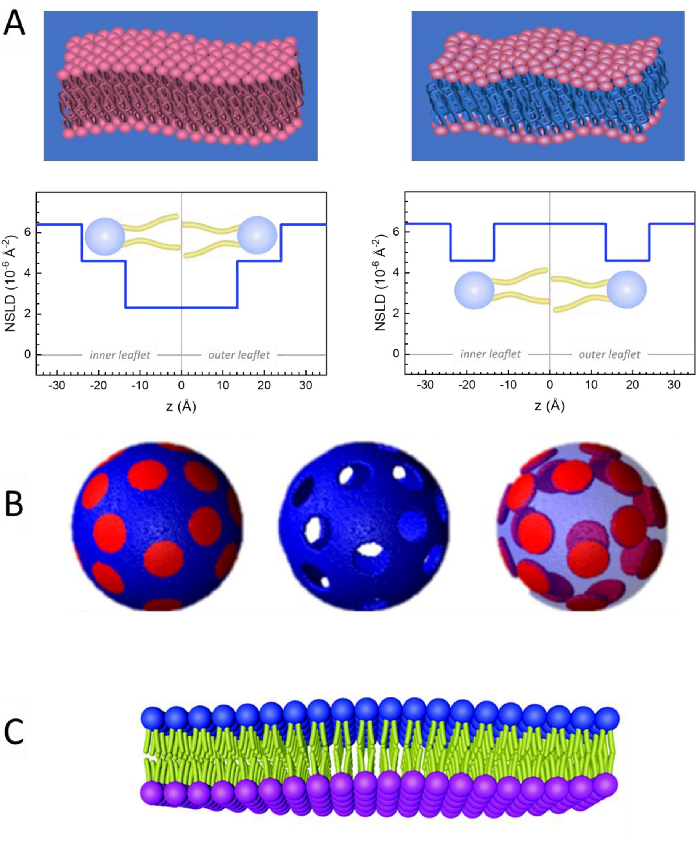
Figure 2: Examples of possible deuteration schemes in NSE experiments on lipid membranes. (A) Left: Fully contrasted membranes, e.g., protiated membranes in deuterated buffer, showing the NSLD profile along the normal to the membrane surface. The difference in the NSLD between the headgroup (~2 × 10-2 Å-2) and tail region (~4.5 × 10-6 Å-2) of the membrane is due to the headgroup hydration with deuterated buffer. Right: Tail-contrast matched membranes such that the hydrocarbon tail region of the membrane has the same NSLD as the buffer, as shown in the corresponding NSLD profile along the membrane normal. (B) Domain-forming membranes with two neutron contrast schemes where the domains (center) or the matrix (left) are contrast-matched to the buffer, enabling selective studies of matrix or domain dynamics, respectively. This figure has been modified from Nickels et al., JACS 201541. (C) Asymmetric membranes prepared by cyclodextrin exchange between protiated and deuterated lipid vesicles, resulting in the deuteration of one membrane leaflet while keeping the other leaflet protiated. This allows studies of the bending dynamics of the protiated leaflet and provides insights into the mechanical coupling between opposing leaflets in asymmetric membranes. This figure has been modified from Rickeard et al., Nanoscale 202040. Please click here to view a larger version of this figure.
to:
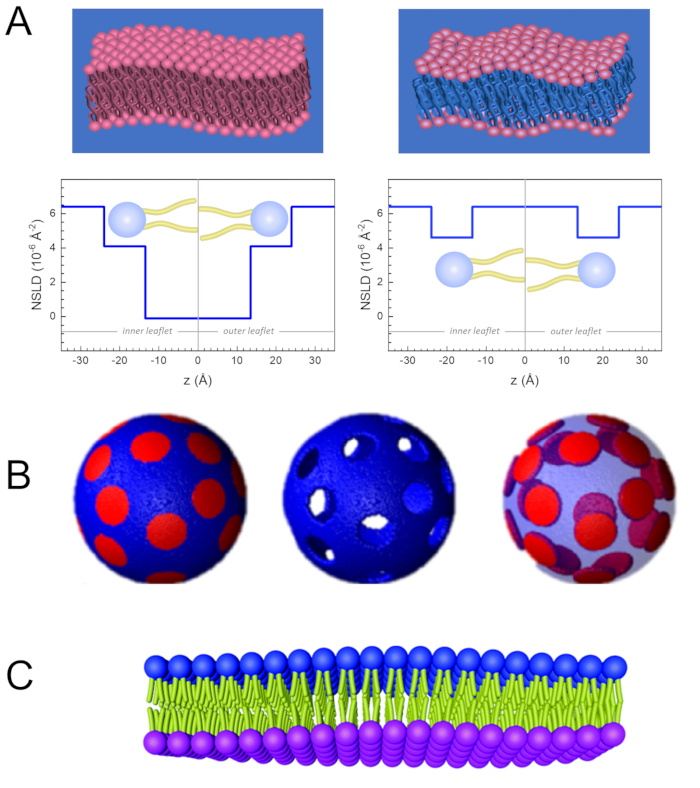
Figure 2: Examples of possible deuteration schemes in NSE experiments on lipid membranes. (A) Left: Fully contrasted membranes, e.g., protiated membranes in deuterated buffer, showing the NSLD profile along the normal to the membrane surface. The difference in the NSLD between the tail region (~0 × 10-2 Å-2) and headgroup region (~4.5 × 10-6 Å-2) of the membrane is due to the headgroup hydration with deuterated buffer. Right: Tail-contrast matched membranes such that the hydrocarbon tail region of the membrane has the same NSLD as the buffer, as shown in the corresponding NSLD profile along the membrane normal. (B) Domain-forming membranes with two neutron contrast schemes where the domains (center) or the matrix (left) are contrast-matched to the buffer, enabling selective studies of matrix or domain dynamics, respectively. This figure has been modified from Nickels et al., JACS 201541. (C) Asymmetric membranes prepared by cyclodextrin exchange between protiated and deuterated lipid vesicles, resulting in the deuteration of one membrane leaflet while keeping the other leaflet protiated. This allows studies of the bending dynamics of the protiated leaflet and provides insights into the mechanical coupling between opposing leaflets in asymmetric membranes. This figure has been modified from Rickeard et al., Nanoscale 202040. Please click here to view a larger version of this figure.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены