Method Article
تسلسل عميق متحيز من الحمض النووي الريبي الفيروسات من العينات السريرية
In This Article
Summary
This protocol describes a rapid and broadly applicable method for unbiased RNA-sequencing of viral samples from human clinical isolates.
Abstract
نحن هنا الخطوط العريضة لجيل القادم من بروتوكول تسلسل الحمض النووي الريبي التي تمكن دي نوفو المجالس ومكالمات البديل داخل مجموعة من العوامل الوراثية الفيروسية التي تم جمعها من مصادر السريرية والبيولوجية. هذه الطريقة غير منحازة والعالمي. ويستخدم البادئات العشوائية لتخليق [كدنا ولا يتطلب أي معرفة مسبقة من المحتوى تسلسل الفيروسي. قبل بناء المكتبة، يتم استخدام انتقائي الهضم أساس H-ريبونوكلياز في استنزاف غير المرغوب فيها RNA - بما في ذلك بولي (RA) الناقل والحمض النووي الريبي الريباسي - من عينة الحمض النووي الريبي الفيروسية. نضوب انتقائية يحسن كلا من جودة البيانات وعدد فريدة من نوعها يقرأ في المكتبات RNA تسلسل الفيروسية. وعلاوة على ذلك، يتم استخدام الخطوة "tagmentation" القائم على ترانسبوزاز في البروتوكول كما أنه يقلل من العام وقت البناء المكتبة. وقد مكن بروتوكول تسلسل عميق السريع لأكثر من 600 لاسا وفيروس إيبولا العينات بما في ذلك مجموعات من كل الدم والأنسجة المعزولة وغير قابلة للتطبيق على نطاق واسع لدراسات الجينوم الميكروبية الأخرى.
Introduction
الجيل القادم التسلسل الفيروسات من مصادر السريرية يمكن أن تبلغ نقل وبائيات الأمراض، فضلا عن المساعدة في دعم رواية التشخيص، وتطوير اللقاحات العلاجية. وقد سمح التوليف [كدنا باستخدام بادئات عشوائية للكشف والتجمع من الجينوم من المتباينة، وشارك في إصابة أو حتى الفيروسات الجديدة 1،2. كما هو الحال مع أساليب مشاركات أخرى، والملوثات غير المرغوب فيها وتشغل العديد من التسلسل يقرأ ويؤثر سلبا على نتائج التسلسل. المضيف وبولي (RA) RNA الناقل والملوثات الموجودة في العديد من مجموعات عينة الفيروسية الحالية.
يصف بروتوكول وسيلة فعالة وفعالة من حيث التكلفة لجينوم الفيروس تسلسل الحمض النووي الريبي عميقة على أساس غير متحيز الحمض النووي الريبي مجموع وما يليها. طريقة يستخدم لريبونوكلياز H نضوب انتقائية الخطوة 3 لإزالة المضيف الريباسي غير المرغوب فيها وRNA الناقل. نضوب انتقائية يثري المحتوى الفيروسي (الشكل 1) ويحسن الجودة الشاملة من تسلسل البيانات(الشكل 2) من العينات السريرية. وعلاوة على ذلك، يتم تطبيق tagmentation إلى البروتوكول كما أنه يقلل بشكل ملحوظ وقت البناء المكتبة. وقد استخدمت هذه الأساليب لتوليد بسرعة مجموعات كبيرة من البيانات من الايبولا وحمى لاسا جينوم فيروس 2،4،5، ويمكن استخدامها لدراسة مجموعة واسعة من فيروسات الحمض النووي الريبي. وأخيرا، فإن النهج لا يقتصر على عينات الإنسان؛ وقد تجلى فائدة نضوب انتقائية في عينات الأنسجة التي تم جمعها من القوارض المصابة لاسا ونماذج المرض الرئيسيات غير البشرية 5،6.

يعكس الشكل 1. إجمالي RNA المحتوى إثراء لاسا الفيروسات المحتوى عن طريق الانتقائية استنفاد ابتداء المضمون العام (RNA المدخلات) وإثراء فريد فيروس حمى لاسا (LASV) يقرأ (محتوى مكتبة) عند نضوب الريباسي من تسعة العزلات السريرية المختلفة. تم تعديل هذا الرقم من 6 . الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.
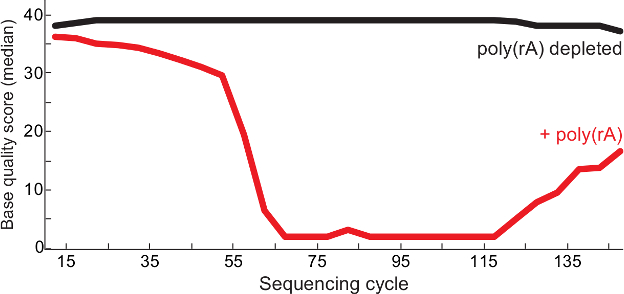
الشكل 2. العالي الجودة تسلسل بعد الناقل RNA نضوب. الصفات قاعدة الوسيطة في دورة تسلسل بولي (RA) -contaminated المكتبات اسا فيروس (الأحمر) والسيطرة (أي الناقل لوحظ في مكتبة، أسود) من تقرير QC 13. من القراءة (1) وقراءة 2 من نهاية يقترن يقرأ يتم دمج في ملف BAM مكتبة وتعرض عشرات الجودة في كل قاعدة. تم تعديل هذا الرقم من 6. الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.
والفيروسي RNA وما يليها تفاصيل بروتوكول إنشاء المكتبات مباشرة من استخراجRNA جمعها من العينات السريرية والبيولوجية. لضمان السلامة الشخصية، ينبغي المعطل جميع عينات مصل الدم والبلازما والأنسجة الفيروسية في مخازن مناسبة قبل استخراج الحمض النووي الريبي. في بعض التعطيل واستخراج مجموعات، يتم تضمين الناقل بولي (RA) RNA. سيتم إزالة هذا خلال ريبونوكلياز H خطوة نضوب انتقائية الأولية. واستنادا إلى الشفاء الكامل، وتركيز المتوقع من الحمض النووي الريبي الناقل هو 100 نانوغرام / ميكرولتر. في البروتوكول، ويستخدم 110 نانوغرام / ميكرولتر بنسبة ضئيلة DT RNA (تركيز الناقل 1.1x) للنضوب. إذا بولي (RA) الناقل غير موجود في العينة، لا ينبغي أن تضاف بعد ذلك بنسبة ضئيلة (DT) قبل نضوب.
تم تصميم بروتوكول التالية لمدة 24 ردود الفعل في شكل لوحة PCR (ما يصل الى 250 حجم ميكرولتر). وذكرت إصدار سابق من هذا البروتوكول في Matranga، وآخرون. 6.
Protocol
بيان الأخلاق: تم تجنيدهم لاسا مرضى حمى لهذه الدراسة باستخدام بروتوكولات التي وافقت عليها لجان بالكائن البشري في جامعة تولين، جامعة هارفارد، معهد برود، Irrua أخصائي تعليم مستشفى (ISTH)، المستشفى الحكومي كينيما (KGH)، وزارة ولاية أويو الصحة، إيبادان ونيجيريا وسيراليون وزارة الصحة سيراليون. تم علاج جميع المرضى الذين يعانون من مستوى مماثل من الرعاية وعرضت المخدرات ريبافيرين، أم لا قرروا المشاركة في الدراسة. حمى لاسا (LF) من المرضى، والمعاملة مع ريبافيرين اتبعت المبادئ التوجيهية الموصى بها حاليا وعرضت عموما بمجرد الاشتباه LF بقوة.
ونظرا لتفشي شديد للمرض الايبولا فيروس (EVD)، لا يمكن أن يكون وافق المرضى من خلال بروتوكولات موحدة لدينا. تم تقييم بدلا من ذلك استخدام عينات الزائدة سريرية من المرضى EVD والموافقة عليها من قبل مجالس المراجعة المؤسسية في سيراليون وفي جامعة هارفارد. وOFFIوقد منحت م لجنة سيراليون الأخلاق واستعراض العلمي، وزارة سيراليون الصحة والصرف الصحي، واللجنة هارفارد حول استخدام المواد البشرية تنازل عن الموافقة على تسلسل وجعل تسلسل الفيروسية المتاحة علنا تم الحصول عليها من عينات المريض والاتصال التي تم جمعها خلال تفشي الايبولا في سيراليون. تمنح هذه الهيئات أيضا استخدام البيانات السريرية والوبائية للعينات التي تم تحديدها دي التي تم جمعها من جميع المرضى EVD يشتبه تلقي الرعاية خلال الاستجابة لمقتضيات الفاشية. وافق سيراليون وزارة سييرا الصحة والصرف الصحي أيضا شحنات غير المعدية، والعينات غير البيولوجية من سيراليون إلى معهد برود وجامعة هارفارد للدراسات الجينية لعينات تفشي المرض.
1. الدناز المعالجة من الحمض النووي الريبي عينة (ما يصل إلى 55 ميكرولتر المستخلصة إجمالي RNA، ~ 4 ساعة)
- إعداد رد فعل الدناز في لوحة 96-جيدا PCR على الجليد في خزانة السلامة البيولوجية كما هو موضح في الجدول رقم 1 ، خطوة 1.1 (الحجم الكلي، 70 ميكرولتر / جيد). ملاحظة: مزيج الرئيسي يمكن أن يكون مستعدا.
- دوامة بلطف ودقة، ثم الطرد المركزي في 280 x ج في RT لمدة 1 دقيقة.
- احتضان عند 37 درجة مئوية لمدة 30 دقيقة.
- تنظيف باستخدام الحمض النووي الريبي المرحلة الصلبة عكسية الشلل (سبري) الخرز.
- الخرز RNA الحارة لRT لمدة 30 دقيقة.
- يهز بلطف الخرز RNA زجاجة ل resuspend أي الجسيمات المغناطيسية التي قد استقر. إضافة إلى حجم 1.8X (126 ميكرولتر) من الخرز RNA إلى الحمض النووي الريبي تعامل الدناز (70 ميكرولتر)، مزيج من قبل ماصة 10 مرات، واحتضان لمدة 5 دقائق على RT (الحجم الكلي بشكل جيد، 196 ميكرولتر).
- ضع الخليط على محطة المغناطيسية. انتظر الحل لمسح (5-10 دقيقة).
- إزالة الحل مسح بينما في المحطة بواسطة ماصة وتجاهل. بينما على المحطة، وغسل حبات من خلال تغطية بيليه مع الايثانول 70٪ واحتضان لمدة 1 دقيقة. إزالة الايثانول مع ماصة وتجاهل. كرر ما مجموعه اثنين من يغسل.
ملاحظة: عن طريق إعداد بدقة 70٪ الطازج وآخرونhanol أمر بالغ الأهمية، كما أن نسبة أعلى سيؤدي في غسل غير الكفء للجزيئات أصغر الحجم، في حين <70٪ من الإيثانول يمكن أن تسبب فقدان العينة 7. - حافظ على لوحة في محطة وترك مفتوحة للهواء الجاف. ملاحظة: تأكد من السماح حبات حتى يجف تماما حتى تبدأ حبات للقضاء.
- إضافة 55 ميكرولتر من nuclease خالية من المياه إلى لوحة لأزل الحمض النووي الريبي. إزالة لوحة من محطة لخلط حبات والمياه من قبل pipetting بدقة. ملاحظة: يمكنك أيضا استخدام كميات أقل من المياه (≤ 10 ميكرولتر) من أجل تركيز الحمض النووي الريبي مجموع.
- وضع لوحة على ظهر المحطة. الانتظار حتى يزيل حل لنقل بواسطة ماصة للالجديد أنبوب المسمار الحد الأقصى للتخزين طويل الأجل (-80 درجة مئوية). ضع 5 ميكرولتر الحمض النووي الريبي في الجديدة 96-جيدا لوحة PCR للنضوب (الخطوة 2.4).
- اختياري: حفظ وتمييع 1 ميكرولتر في 19 ميكرولتر المياه (01:20) لQRT-PCR من الريباسي (على سبيل المثال، 18S، 28S الريباسي) (الجدول 2) وعلامات الفيروسية 5
2. استنزاف الانتقائي للخاص بالريبوسوم والناقل RNA من عينة الحمض النووي الريبي الفيروسي (~ 4 ساعة)
- جعل التهجين 5X و 10x مخازن رد فعل ريبونوكلياز H، وnuclease خالية من المياه مع الناقل الأكريلاميد خطية كما هو موضح في الجدول رقم 1.
- إعداد رد فعل التهجين من خلال الجمع بين الحمض النووي الريبي مع oligos نضوب الريباسي (الجدول 3) وبنسبة ضئيلة (DT) على الجليد في لوحة 96-جيدا PCR كما هو موضح في الجدول رقم 1.
ملاحظة: مزيج الرئيسي يمكن أن يكون مستعدا. 50 femtograms (FG) من الحمض النووي الريبي الاصطناعية فريد (ERCCs 8) ويمكن أن يضاف لتتبع كل من عملية التسلسل الفيروسية والمحتملين المؤشر قراءة انتقال التلوث.- دوامة بلطف ودقة، ثم الطرد المركزي في 280 x ج في RT لمدة 1 دقيقة.
احتضان عند 95 درجة مئوية لمدة 2 دقيقة، وبطء التعلية إلى 45 درجة مئوية في -0.1 درجة مئوية في الثانية. وقفة thermocycler في 45 درجة مئوية.
- دوامة بلطف ودقة، ثم الطرد المركزي في 280 x ج في RT لمدة 1 دقيقة.
- إعداد مزيج رد فعل ريبونوكلياز H على الجليد وصفهد في الجدول رقم 1، ثم سخن على 45 درجة مئوية لمدة 2 دقيقة. ملاحظة: مزيج الرئيسي يمكن أن يكون مستعدا.
- يضاف مزيج ريبونوكلياز H قبل تسخينها إلى رد فعل التهجين في لوحة مع الحفاظ على لوحة في thermocycler في 45 درجة مئوية.
- تخلط جيدا طيف من قبل pipetting 6-8 مرات. احتضان عند 45 درجة مئوية لمدة 30 دقيقة أخرى. مكان على الجليد.
- إعداد مزيج رد فعل الدناز على الجليد كما هو موضح في الجدول رقم 1 ملاحظة: مزيج الرئيسي يمكن أن تكون مستعدة.
- إضافة إلى رد فعل ريبونوكلياز H في لوحة، ودوامة بلطف ودقة، ثم الطرد المركزي في 280 x ج في RT لمدة 1 دقيقة. احتضان عند 37 درجة مئوية لمدة 30 دقيقة.
- وقف رد فعل الدناز بإضافة 5 ميكرولتر 0.5 M EDTA. دوامة بلطف ودقة، ثم الطرد المركزي في 280 x ج في RT لمدة 1 دقيقة.
- تنظيف باستخدام الخرز RNA (راجع الخطوة 1.3) باستخدام حجم 1.8X (144 ميكرولتر) الخرز. أزل في 11 ميكرولتر من nuclease خالية من المياه. ملاحظة: للحصول على التخزين البارد آمن، المنضب مخزن RNوالعينات في -80 ° C O / N.
3. كدنا] التجميعي (~ 6 ساعات)
- مزيج الريباسي / الحمض النووي الريبي المنضب الناقل مع الاشعال عشوائية على الجليد في لوحة 96-جيدا PCR كما هو موضح في الجدول رقم 1، دوامة بلطف ودقة، ثم الطرد المركزي في 280 x ج في RT لمدة 1 دقيقة.
- حرارة الخليط إلى 70 درجة مئوية لمدة 10 دقيقة في thermocycler. مباشرة بعد تمسخ الحرارة، ووضع الحمض النووي الريبي على الجليد لمدة 1-5 دقيقة. لا تسمح الحمض النووي الريبي على الوقوف (حتى على الجليد) لمدة تزيد عن 5 دقائق قبل أول رد فعل حبلا.
- إعداد حبلا أولا مزيج رد فعل التوليف على الجليد كما هو موضح في الجدول رقم 1.
ملاحظة: A-مزيج الرئيسي قد يكون مستعدا.- إضافة إلى RNA / مزيج التمهيدي عشوائي في لوحة، ودوامة بلطف ودقة، ثم الطرد المركزي في 280 x ج في RT لمدة 1 دقيقة. احتضان في 22-25 درجة مئوية لمدة 10 دقيقة.
- احتضان عند 55 درجة مئوية في حاضنة الهواء لمدة 60 دقيقة. وضع لوحة على الجليد لإنهاء رد فعل. ليسمن المستحسن استخدام حاضنة الهواء لخلق ظاهرة الاحتباس التدريجي لأول رد فعل حبلا خلالها يصلب الاشعال وحبلا الأول يبدأ استطال: ه.
- إعداد حبلا الثانية مزيج رد فعل التوليف على الجليد كما هو موضح في الجدول رقم 1.
ملاحظة: A-مزيج الرئيسي قد يكون مستعدا.- إضافة إلى أول حبلا رد فعل التوليف في لوحة، ودوامة بلطف ودقة، ثم الطرد المركزي في 280 x ج في RT لمدة 1 دقيقة. احتضان لمدة 2 ساعة على 16 درجة مئوية (الحفاظ على الغطاء عند 25 درجة مئوية). لا تسمح درجة الحرارة إلى الارتفاع فوق 16 درجة مئوية.
- وضع لوحة على الجليد، ثم تعطيل رد فعل عن طريق إضافة 5 ميكرولتر من 0.5 M EDTA، مزيج بلطف ودقة، ثم الطرد المركزي في 280 x ج في RT لمدة 1 دقيقة.
- تنظيف مع حبات الحمض النووي (راجع الخطوة 1.3 لبروتوكول) باستخدام حجم 1.8X (153 ميكرولتر) من الخرز. أزل في 9 ميكرولتر من شطف العازلة (EB). حفظ 1 ميكرولتر لتقدير. استخدام 1 نانوغرام من [كدنا لsubsequeخطوات الإقليم الشمالي. إذا تركيز [كدنا منخفض جدا لاكتشاف واستخدام 4 ميكرولتر من [كدنا لtagmentation (راجع الخطوة 4.1).
- للتخزين البارد آمن وتخزين مزدوجة الذين تقطعت بهم السبل [كدنا في 4 درجات CO / N أو -20 درجة مئوية لمدة التخزين على المدى الطويل.
4. إعداد مكتبة - الحمض النووي مكتبة البناء (~ 4 ساعة)
- نقل 4 ميكرولتر من [كدنا لوحة 96-جيدا وحفظ ما تبقى من [كدنا لمحاولة ثانية إذا لزم الأمر.
- إعداد رد فعل tagmentation على الجليد كما هو موضح في الجدول رقم 1.
ملاحظة: A-مزيج الرئيسي قد يكون مستعدا. للحد من الخلفية والتكلفة الإجمالية، يتم تقليل الحجم الكلي للتفاعل tagmentation 20-10 ميكرولتر. كما كدنا] هو العامل المحدد، يتم تقليل كمية من أجهزة الصراف الآلي (أي transposome) المستخدمة في رد فعل أيضا لتقليل عدد مواقع التكامل.- إضافة مزيج tagmentation إلى كدنا] في لوحة، ودوامة بلطف ودقة وأجهزة الطرد المركزي في 280 x ج (في RT) لمدة 1 دقيقة.احتضان عند 55 درجة مئوية لمدة 5 دقائق، عقد في 10 درجة مئوية.
- مرة واحدة على 10 درجة مئوية، وعلى الفور إضافة 2.5 ميكرولتر من تحييد Tagment العازلة (NT) لإنهاء رد فعل. مزيج من قبل pipetting صعودا وهبوطا، ثم الطرد المركزي في 280 x ج (في RT) لمدة 1 دقيقة.
- في احتضان RT لمدة 5 دقائق.
- إعداد PCR التضخيم رد فعل على الجليد كما هو موضح في الجدول رقم 1.
- دوامة بلطف ودقة، ثم الطرد المركزي في 280 x ج في RT لمدة 1 دقيقة.
- أداء PCR في thermocycler باستخدام الشروط المبينة في الجدول 1.
ملاحظة: تم اقتراح 12 دورات PCR ل1 نانوغرام من [كدنا tagmented. ومع ذلك، العينات السريرية الفيروسية غالبا ما يكون مبالغ غير قابلة للكشف من كدنا]. لكميات قليلة من كدنا] (<1 نانوغرام)، استخدام ما يصل إلى 18 دورات PCR لإنشاء مكتبة كافية لالتسلسل.
- إعداد مكتبة - تنظيف وتجميع للتسلسل
- جلب عينة تصل إلى 50 ميكرولتر مع EB.
- تنظيف معحبات الحمض النووي (راجع الخطوة 1.3 لبروتوكول) باستخدام حجم 0.6x (30 ميكرولتر) الخرز. أزل في 15 ميكرولتر EB.
- تحديد تركيز مكتبة (الشكل 3) عن طريق إجراء تحليل المنطقة (150 إلى 1000 سنة مضت) باستخدام برنامج bioanalyzer 9، باستثناء dimers التمهيدي (~ 120 بي بي) من تحليل المنطقة. ملاحظة: بدلا من ذلك، QPCR يمكن استخدامها لتحديد مكتبات 10.
- مكتبات التجمع في أقل تركيز المولي من 1 نانومتر أو أكبر. إذا المكتبة أقل من 1 نانومتر، إضافة كمية صغيرة من مكتبة لتجمع (~ حجم 1X من المكتبات الأخرى) لالتقاط المعلومات تسلسل من هذه المكتبات.
- تجمع تنظيف مع حبات الحمض النووي 0.7X على النحو المبين أعلاه (راجع الخطوة 2). أزل في 15 ميكرولتر EB. ملاحظة: حجم حبات تعتمد على الحجم النهائي من حوض السباحة.
- تحليل تجمع 9. تحديد تركيز المولي عن طريق إجراء تحليل المنطقة (150 إلى 1000 سنة مضت) 9. ملاحظة: بدلا من ذلك، QPCR يمكن استخدامها لتحديد تجمع المكتبة 10 </ سوب>.
- تحميل المنظم في تركيز مكتبة 22:00 لتوليد 101 نقطة أساس، يقرأ إقران مع نهاية يقرأ الباركود ثنائي 11.
صورة جل من 4 ممثل فيروس إيبولا (EBOV) مكتبات الرقم 3. المكتبات شيدت من الإيبولا الفيروسات العينات السريرية. وتظهر مناطق مكتبة والتمهيدي dimers. الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.
النتائج
يتيح بروتوكول صفها توليد تسلسل جودة عالية يقرأ من المدخلات المنخفضة عينات الحمض النووي الريبي الفيروسية في حين إثراء للمحتوى الفيروسية فريدة من نوعها. كما هو مبين في الشكل رقم 1، أثرى بروتوكول محتوى فريد فيروس حمى لاسا ما لا يقل عن خمسة أضعاف في جميع العينات (مقارنة مع الضوابط غير المنضب) مع مليون نسخة على الأقل من 18S الريباسي (~ 100 خريج مجموع RNA). وبالمثل، يرتبط نجاح التسلسل أيضا مع كمية الفيروس في عينة معينة. باستخدام QRT-PCR كبديل لكمية الفيروسية، والعينات التي تحتوي على ~ 1000 نسخة أو أكثر الجينوم الفيروسي في معظم الأحيان خلق تجمعات كاملة (لا تظهر البيانات). وعلاوة على ذلك، ونضوب بولي (RA) الناقل يقلل تسلسل المبلمر المتجانس من ألف وتي في المكتبات، مما أدى إلى استعدادات أكثر نظافة وضمان تسلسل جودة أفضل يقرأ (الشكل 2). المكتبات النهائية من انخفاض مساهمة العينات السريرية الفيروسية غالبا ما يكون طول القطعة واسعة من 150 إلى 1000 سنة مضت(الشكل 3).
بعد التسلسل، للحد من العينة عدم التعرف والحديث المتبادل بين المكتبات ضمن تجمع 12، مؤشر فقط يقرأ بنتيجة جودة قاعدة 25 (Q25) وضمان يتم الاحتفاظ الصفر عدم التطابق أثناء عملية demultiplexing. يتم تجميع الجينوم الفيروسي باستخدام خط أنابيب المعلوماتية الحيوية معين للبحث عن الفيروسات المختلفة 2،4-6. تتوفر هذه الأدوات في https://github.com/broadinstitute/viral-ngs أو من خلال منصات السحابة التجارية 4.
| الخطوة 1.1: رد فعل الدناز | |
| كاشف | حجم في رد الفعل (ميكرولتر) |
| عازلة 10X الدناز | 7 |
| nuclease خالية من المياه | 6 |
| RNA الفيروسي المستخرج | 55 |
| الدناز (يو 2 / & #181؛ ل) | 2 |
| الحجم الكلي | 70 |
| الخطوة 2.1: عازلة 5X التهجين | |
| كاشف | حجم ل1 مل (ميكرولتر) |
| 5 M كلوريد الصوديوم | 200 |
| 1 M تريس، حمض الهيدروكلوريك (7.4 درجة الحموضة) | 500 |
| nuclease خالية من المياه | 300 |
| الحجم الكلي | 1000 |
| الخطوة 2.1: 10X رد فعل العازلة ريبونوكلياز H | |
| كاشف | حجم ل1 مل (ميكرولتر) |
| 5 M كلوريد الصوديوم | 200 |
| 1 M تريس، حمض الهيدروكلوريك (7.5 درجة الحموضة) | 500 |
| 1 م 2 MgCl | 200 |
| nuclease خالية من المياه | 500 |
| الحجم الكلي | 1000 |
| الخطوة 2.1: المياه ثمادة الأكريلاميد خطي إيث | |
| كاشف | حجم ل1 مل العازلة (ميكرولتر) |
| nuclease خالية من المياه | 992 |
| مادة الأكريلاميد الخطي (5 ملغ / مل) | 8 |
| الحجم الكلي | 1000 |
| الخطوة 2.2: رد فعل التهجين لاستنزاف انتقائية | |
| كاشف | حجم في رد الفعل (ميكرولتر) |
| 5X التهجين العازلة | 2 |
| مزيج بنسبة ضئيلة الريباسي استنفاد (100 ميكرومتر) | 1.22 |
| بنسبة ضئيلة (د) T (550 نانوغرام / ميكرولتر) | 1 |
| المعالجة الدناز الحمض النووي الريبي مجموع | ما يصل الى 5 |
| ارتفاع في الحمض النووي الريبي (وهذا هو اختياري) | 0.5 |
| الماء (مع الأكريلاميد خطية) | حتى تصل الى ما مجموعه 10 |
| إجمالي voluأنا | 10 |
| الخطوة 2.3: ريبونوكلياز H رد فعل لاستنزاف انتقائية | |
| كاشف | حجم في رد الفعل (ميكرولتر) |
| رد فعل العازلة 10X ريبونوكلياز H | 2 |
| الماء (مع الأكريلاميد خطية) | 5 |
| بالحرارة ريبونوكلياز H (5 U / ميكرولتر) | 3 |
| الحجم الكلي | 10 |
| الخطوة 2.4: الدناز آخر رد فعل استنزاف انتقائية | |
| كاشف | حجم في رد الفعل (ميكرولتر) |
| 10X الدناز العازلة | 7.5 |
| الماء (مع الأكريلاميد خطية) | 44.5 |
| المانع ريبونوكلياز (20 U / ميكرولتر) | 1 |
| ريبونوكلياز خالية الدناز الأول (2.72 U / ميكرولتر) | 2 |
| اجمالى حجم (مع رد فعل ريبونوكلياز H) | 75 |
| الخطوة 3.1: توليف [كدنا، التمهيدي عشوائي التهجين | |
| كاشف | حجم في رد الفعل (ميكرولتر) |
| الريباسي / المنضب الناقل RNA | 10 |
| 3 ميكروغرام التمهيدي عشوائي | 1 |
| الحجم الكلي | 11 |
| الخطوة 3.2: حبلا الأول رد فعل التوليف [كدنا | |
| كاشف | الحجم (ميكرولتر) |
| 5X الأولى الجديلة العازلة رد الفعل | 4 |
| 0.1 M DTT | 2 |
| مزيج 10 ملي dNTP | 1 |
| المانع ريبونوكلياز (20 U / ميكرولتر) | 1 |
| الناسخ العكسي (إضافة آخر) | 1 |
| الحجم الكلي (مع RNA أعلاه) | 20 |
| الخطوة 3.3: الشق الثاني رد فعل التوليف [كدنا | |
| كاشف | الحجم (ميكرولتر) |
| المياه خالية من ريبونوكلياز | 43 |
| 10X العازلة رد الفعل الثاني ستراند | 8 |
| مزيج 10 ملي dNTP | 3 |
| كولاي الحمض النووي يغاز (10 U / ميكرولتر) | 1 |
| كولاي البلمرة DNA الأول (10 U / ميكرولتر) | 4 |
| كولاي ريبونوكلياز H (يو 2 / ميكرولتر) | 1 |
| اجمالى حجم (مع 1 الحادي رد فعل حبلا) | 80 |
| الخطوة 4.2: رد فعل Tagmentation | |
| كاشف | الحجم (ميكرولتر) |
| Amplicon Tagment ميكس (ATM) | 1 |
| Tagment الحمض النووي العازلة (TD) د> | 5 |
| اجمالى حجم (مع كدنا]) | 10 |
| الخطوة 4.3: رد فعل مكتبة PCR | |
| كاشف | الحجم (ميكرولتر) |
| PCR ميكس ماجستير (الآلية) | 7.5 |
| مؤشر 1 التمهيدي (i7 و) | 2.5 |
| مؤشر 2 التمهيدي (i5 و) | 2.5 |
| اجمالى حجم (مع tagmented كدنا]) | 25 |
| خطوة 4.3.2: شروط الاسترداد مكتبة | |
| 72 ° C، 3 دقيقة | |
| 95 ° C، 30 ثانية | |
| ما يصل الى 18 دورات من 10 ثانية في 95 درجة مئوية، و 30 ثانية في 55 درجة مئوية، و 30 ثانية في 72 درجة مئوية | |
| 72 ° C، 5 دقيقة | |
| 10 ° C، إلى الأبد | |
FO: المحافظة على together.within الصفحات = "1"> الجدول 1: رد الفعل انشاء ومخازن خطوة بخطوة الجداول مع محتويات جميع المخازن ويمزج رد فعل.
الجدول 2: QRT-PCR الاشعال تسلسل الاشعال المستخدمة لقياس المضيف (18S الريباسي) والفيروسية المحتوى (ايبولا واسا). "KGH 'هو مستشفى كينيما الحكومة في سيراليون، حيث تم اختبار الاشعال الايبولا 2. "Kulesh 'هو المحقق الذي صمم التمهيدي تعيين 14.
الجدول 3: الحمض النووي الريبي الريباسي (الرنا الريباسي) استنفاد Oligos 195 50 النوكليوتيدات تسلسل طويلة مكملة لالريباسي البشري لاستنزاف انتقائية الخطوة 6. يرجى النقر هنا لتحميل هذا الملف.
| اسم بنسبة ضئيلة | تسلسل (5 'إلى 3') |
| ايبولا KGH مهاجم | GTCGTTCCAACAATCGAGCG |
| ايبولا KGH RV | CGTCCCGTAGCTTTRGCCAT |
| ايبولا KULESH مهاجم | TCTGACATGGATTACCACAAGATC |
| ايبولا KULESH RV | GGATGACTCTTTGCCGAACAATC |
| لاسا SL مهاجم | GTA AGC التقييم القطري المشترك GCD GYA AAB CC |
| لاسا SL RV | مجموعة الاهلي التقييم القطري المشترك CAG AAA RCT GGS AGC و |
| 18S الريباسي مهاجم | TCCTTTAACGAGGATCCATTGG |
| 18S الريباسي RV | CGAGCTTTTTAACTGCAGCAACT |
Discussion
النهج المبين تمكن قوية وعالمية، والتسلسل السريع، وكان يستخدم لتسلسل فيروس إيبولا خلال 2،4 2014 انتشار المرض. وذلك بربط نضوب انتقائية والتوليف [كدنا مع بناء مكتبة tagmentation، تم تخفيض الوقت عملية الشامل التي كتبها ~ 2 أيام من وسائل محول ربط السابقة. وفي الآونة الأخيرة، كان يعمل هذا البروتوكول من قبل المتعاونين الدوليين وغيرهم مع نجاحا كبيرا 15،16 وسيتم نشر إلى مختبرات في غرب أفريقيا لدعم البحوث والدراسات القائمة على علم الجينوم المحلية وتشخيص 17.
بروتوكول الموصوفة هنا يستخدم البادئات العشوائية لإعداد كدنا] للمكتبات الحمض النووي الريبي وما يليها الفيروسية. على عكس النهج RNA وما يليها الفيروسية السابقة، أنه لا يتطلب أي معرفة مسبقة من تسلسل البيانات أو تفصيلا وتستغرق وقتا طويلا التصميم التمهيدي للفيروس أو كليد محددة. ويمكن تطبيق هذا الأسلوب على أي عينة الحمض النووي الريبي الفيروسية. على سبيل المثال، كان يستخدم لتوليد محتوى الفيروسي من كل من الايبولاوعينات لاسا 6. ويمكن أيضا أن تستخدم بروتوكول لtranscriptomic المضيف، مشاريع التسلسل metagenomic واكتشاف الممرض 1.
ويستهدف خطوة حاسمة للبروتوكول ريبونوكلياز H الهضم، والإنتاجية العالية، وطريقة منخفضة التكلفة لإزالة الناقل غير المرغوب فيها والجيش الملكي النيبالي المضيف من العينات الفيروسية. الخطوة نضوب انتقائية للبروتوكول يستخدم العديد من المكونات، ويتطلب مهارة ودقة. ينبغي أن تؤخذ الوقت الاضافي والرعاية أثناء الإعداد الأولي.
حيث أن معظم مصل الدم والبلازما العينات السريرية غالبا ما يكون قليلا جدا مادة الحمض النووي والتلوث وفقدان العينة شائعة. لتجنب هذه المشاكل، يجب توخي الحذر خاصة عند استخدام هذا البروتوكول. أولا، RNA هو عرضة للتدهور. وبالتالي كل المناطق يجب أن تكون نظيفة وخالية من nucleases. ثانيا، لتحديد عينات مناسبة للاستخدام في هذا البروتوكول، المقايسات QRT-PCR RNA لكلا المضيف وفيروس ينبغي أن تستخدم لتقدير 5،6 . عند المقارنة بين المدخلات يرقى مع نتائج التسلسل من البروتوكول، والنجاح التسلسل (أي جيل من بيانات كافية لتجميع الفيروسي الكامل) المترابطة مع العينات التي تحتوي على ما لا يقل عن 100 خريج الحمض النووي الريبي مجموع و1،000 نسخة من الفيروس. وينبغي تجنب الثالث، والتعرض للمصادر البيئية من الأحماض النووية. يتم البروتوكول المذكورة هنا في مجلس الوزراء للسلامة الأحيائية لاحتياطات السلامة والحد من الملوثات البيئية. وعلاوة على ذلك، لدينا مجموعة والآخرون يلاحظون أن الإنزيمات التجارية قد تكون مصدرا آخر للتلوث الأحماض النووية البكتيرية في عينات المدخلات المنخفضة 6،18. استخدام مساحة عمل نظيفة (على سبيل المثال، PCR غطاء محرك السيارة، ومجلس الوزراء للسلامة الأحيائية) والضوابط السلبية (على سبيل المثال، الماء أو عازلة) سوف تساعد على التخفيف من وتتبع التلوث، على التوالي. للعينات مع <100 خريج من الحمض النووي الريبي مجموع، بولي فقط (RA) RNA الناقل، وليس الريباسي، ينبغي المنضب لضمان تحقيق نتائج التسلسل عالية الجودة مع الحد من فقدان المواد. للغايةعينات المدخلات المنخفضة، قد تكون أساليب كدنا] التضخيم أكثر ملاءمة 19، على الرغم من بولي (RA) الناقل يجب إزالة قبل تركيب كدنا].
استنزاف المضيف الريباسي يثري المحتوى الفيروسي في المكتبات التسلسل وينطبق على مجموعات عينة مختلفة بما في ذلك المصل أو البلازما، وأنواع متعددة من الأنسجة من القوارض والرئيسيات غير البشرية 5،6. في الكائنات غير البشرية، يقرأ محاذاة إلى 28S الريباسي بقيت بعد نضوب، مما يشير إلى 28S الريباسي أقل محفوظ بين البشر والأنواع الأخرى 6،20. عند استخدام هذا الأسلوب مع العزلات غير البشرية، قد يكون من الضروري لتكملة مع oligos الحمض النووي مكملة لتسلسل الريباسي المتباينة لل3،21 المضيف المحدد.
منذ البروتوكول هو غير متحيز، الفيروسية يقرأ قد لا تمثل سوى جزء صغير من المحتوى الكلي للمكتبة. على الرغم من أن الريباسي هو النوع الأكثر وفرة من الحمض النووي الريبي المضيفة وفقط نسبة صغيرة من الريباسي يقرأ (0؛ 1٪) وجدت بعد نضوب انتقائية، وجميع RNA المضيفة الأخرى (على سبيل المثال، مرنا) تبقى بعد نضوب وقد تكون مسؤولة عن يقرأ العديد من تسلسل من العينة. لذلك "الإفراط" (أي oversequencing) مكتبات فردية مطلوب من أجل الحصول على ما يكفي من التغطية للمكالمات التجمع والبديل الفيروسية. لدراستنا، ونحن نحاول تسلسل ~ 20 مليون يقرأ في عينة لدينا ما يكفي من العمق لتحليل الفيروسي المتغيرات الجيني وما يرتبط بها فضلا عن المحتوى metagenomic 2،5. لدراسات اكتشاف metagenomic والممرض، من المهم أن نلاحظ أن تتم إزالة تلويث الحمض النووي المضيف الدناز الهضم. لذلك قد تفقد الفيروسات ومسببات الأمراض الأخرى التي تحتوي على جينوم الحمض النووي خلال هذه العملية، ولكن قد لا يزال تكون متسلسلة وسيطة الحمض النووي الريبي.
Disclosures
The authors have no competing financial interests.
Acknowledgements
This work has been funded in part with Federal funds from the National Institutes of Health, Office of Director, Innovator (No.: DP2OD06514) (PCS) and from the National Institute of Allergy and Infectious Diseases, National Institutes of Health, Department of Health and Human Services, under Contracts (No:HHSN272200900018C, HHSN272200900049C and U19AI110818).
Materials
| Name | Company | Catalog Number | Comments |
| 96-well PCR Plates | VWR | 47743-953 | |
| Strips of Eight Caps | VWR | 47745-512 | |
| Nuclease-free water | Ambion | AM9937 | 50 ml bottle |
| TURBO DNase | Ambion | AM2238 | post RNA extraction step, 2 U/µl, buffer included |
| PCR cycler | any PCR cyclers | ||
| Agencourt RNAClean XP SPRI beads | Beckman Coulter Genomics | A63987 | beads for RNA cleanup |
| Real Time qPCR system | any system | ||
| DynaMag-96 Side Skirted Magnet | Invitrogen | 12027 | |
| 70% Ethanol | prepare fresh | ||
| qRT-PCR primers | IDT DNA | see Table 2 | |
| 5 M NaCl | Ambion | AM9760G | |
| 1 M Tris-HCl pH 7.4 | Sigma | T2663-1L | |
| 1 M Tris-HCl pH 7.5 | Invitrogen | 15567-027 | |
| 1 M MgCl2 | Ambion | AM9530G | |
| Linear acrylamide | Ambion | AM9520 | |
| DNA oligos covering entire rRNA region | IDT DNA | see Table 3, order lab-ready at 100 µM | |
| Oligo (dT) | IDT DNA | 40 nt long, desalted | |
| Hybridase Thermostable RNase H | Epicentre | H39100 | |
| RNase-free DNase Kit | Qiagen | 79254 | post selective depletion step |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Random Primers | Invitrogen | 48190-011 | mostly hexamers |
| 10 mM dNTP mix | New England Biolabs | N0447L | |
| SuperScript III Reverse Transcriptase | Invitrogen | 18080-093 | with first-strand buffer, DTT |
| Air Incubator | any air incubator cyclers | ||
| NEBNext Second Strand Synthesis (dNTP-free) Reaction Buffer | New England Biolabs | B6117S | 10x |
| E. coli DNA Ligase | New England Biolabs | M0205L | 10 U/μl |
| E. coli DNA Polymerase I | New England Biolabs | M0209L | 10 U/μl |
| E. coli RNase H | New England Biolabs | M0297L | 2 U/μl |
| 0.5 M EDTA | Ambion | AM9261 | |
| Agencourt AMPure XP SPRI beads | Beckman Coulter Genomics | A63881 | beads for DNA cleanup |
| Elution Buffer | Qiagen | 10 mM Tris HCl, pH 8.5 | |
| Quant-iT dsDNA HS Assay Kit | Invitrogen | Q32854 | |
| Qubit fluorometer | Invitrogen | Q32857 | |
| Nextera XT DNA Sample Prep Kit | Illumina | FC-131-1096 | |
| Nextera XT DNA Index Kit | Illumina | FC-131-1001 | |
| Tapestation 2200 | Agilent | G2965AA | |
| High Sensitivity D1000 reagents | Agilent | 5067-5585 | |
| High Sensitivity D1000 ScreenTape | Agilent | 5067-5584 | |
| BioAnalyzer 2100 | Agilent | G2939AA | |
| High Sensitivity DNA reagents | Agilent | 5067-4626 | |
| Library Quantification Complete kit (Universal) | Kapa Biosystems | KK4824 | alternative to tapestation, bioanalyzer for library quantification |
References
- Stremlau, M. H., et al. Discovery of novel rhabdoviruses in the blood of healthy individuals from West Africa. PLoS Negl Trop Dis. 9, e0003631(2015).
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345, 1369-1372 (2014).
- Morlan, J. D., Qu, K., Sinicropi, D. V. Selective depletion of rRNA enables whole transcriptome profiling of archival fixed tissue. PLoS One. 7, e42882(2012).
- Park, D. J., et al. Ebola Virus Epidemiology, Transmission, and Evolution during Seven Months in Sierra Leone. Cell. 161, 1516-1526 (2015).
- Andersen, K. G., et al. Clinical Sequencing Uncovers Origins and Evolution of Lassa Virus. Cell. 162, 738-750 (2015).
- Matranga, C. B., et al. Enhanced methods for unbiased deep sequencing of Lassa and Ebola RNA viruses from clinical and biological samples. Genome Biol. 15, 519(2014).
- Tang, F., et al. RNA-Seq analysis to capture the transcriptome landscape of a single cell. Nat Protoc. 5, 516-535 (2010).
- Jiang, L., et al. Synthetic spike-in standards for RNA-seq experiments. Genome Res. 21, 1543-1551 (2011).
- Agilennt Technologies. , Available from: http://www.agilent.com/cs/library/usermanuals/Public/G2946-90004_Vespucci_UG_eBook_(NoSecPack).pdf. (2015).
- Kapa Biosystems. , Available from: https://www.kapabiosystems.com/product-applications/products/next-generation-sequencing-2/library-quantification/ (2015).
- Illumina Technologies. , Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/system_documentation/miseq/preparing-libraries-for-sequencing-on-miseq-15039740-d.pdf. (2015).
- Kircher, M., Sawyer, S., Meyer, M. Double indexing overcomes inaccuracies in multiplex sequencing on the Illumina platform. Nucleic Acids Res. 40, 3(2012).
- Andrews, S. Babraham Bioinformatics. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Trombley, A. R., et al. Comprehensive panel of real-time TaqMan polymerase chain reaction assays for detection and absolute quantification of filoviruses, arenaviruses, and New World hantaviruses. Am J Trop Med Hyg. 82, 954-960 (2010).
- Hu, Y., et al. Serial high-resolution analysis of blood virome and host cytokines expression profile of a patient with fatal H7N9 infection by massively parallel RNA sequencing. Clin Microbiol Infect. 21, e1-4 713(2015).
- Simon-Loriere, E., et al. Distinct lineages of Ebola virus in Guinea during the 2014 West African epidemic. Nature. 524, 102-104 (2015).
- Folarin, O. A., Happi, A. N., Happi, C. T. Empowering African genomics for infectious disease control. Genome Biol. 15, 515(2014).
- Blainey, P. C., Quake, S. R. Digital MDA for enumeration of total nucleic acid contamination. Nucleic Acids Res. 39, 19(2011).
- Malboeuf, C. M., et al. Complete viral RNA genome sequencing of ultra-low copy samples by sequence-independent amplification. Nucleic Acids Res. 41, 13(2013).
- Gonzalez, I. L., Sylvester, J. E., Smith, T. F., Stambolian, D., Schmickel, R. D. Ribosomal RNA gene sequences and hominoid phylogeny. Mol Biol Evol. 7, 203-219 (1990).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nat Methods. 10, 623-629 (2013).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved