Method Article
Unvoreingenommene Tiefe Sequenzierung von RNA-Viren aus klinischen Proben
In diesem Artikel
Zusammenfassung
This protocol describes a rapid and broadly applicable method for unbiased RNA-sequencing of viral samples from human clinical isolates.
Zusammenfassung
Hier beschreiben wir eine Next-Generation - RNA - Sequenzierung Protokoll , das de novo Assemblierung und intra-Host - Variante Anrufe von viralen Genomen von klinischen und biologischen Quellen gesammelt werden können. Die Methode ist unvoreingenommen und universell; es verwendet zufällige Primer für die cDNA-Synthese und erfordert keine vorherige Kenntnis der viralen Sequenz Gehalt. Vor dem Aufbau der Bibliothek, selektive RNase H-basierte Verdauung wird verwendet, um unerwünschte RNA zu führen - einschließlich Poly (rA) Träger und ribosomale RNA - von der viralen RNA-Probe. Selective Depletion verbessert sowohl die Datenqualität und die Anzahl der eindeutigen liest in der viralen RNA-Sequenzierung Bibliotheken. Darüber hinaus ist ein Transposase-basierten 'tagmentation' Schritt wird in dem Protokoll verwendet wird, wie es allgemeine Bibliothek Bauzeit reduziert. Das Protokoll hat eine rasante tiefe Sequenzierung von mehr als 600 Lassa und Ebola-Virusproben-Sammlungen einschließlich aktiviert sowohl von Blut und Gewebe isoliert-und ist im Großen und Ganzen auf andere mikrobielle Genomstudien.
Einleitung
Next Generation Sequenzierung von Viren aus klinischen Quellen kann die Übertragung und die Epidemiologie von Infektionen informieren, sowie dazu beitragen, Unterstützung neuer diagnostischer, Impfstoff und therapeutische Entwicklung. cDNA - Synthese unter Verwendung von Zufallsprimer hat den Nachweis und die Montage von Genomen von divergent, Coinfizierung oder auch neuartige Viren 1,2 erlaubt. Wie bei anderen unvoreingenommene Methoden, besetzen unerwünschte Verunreinigungen viele Sequenzierung liest und negativ Sequenzierungsergebnisse auswirken. Host-und Poly (rA) Carrier-RNA sind Verunreinigungen, die in vielen bestehenden viralen Musterkollektionen.
Das Protokoll beschreibt eine effiziente und kostengünstige Möglichkeit der tiefen Sequenzierung Genomen RNA Virus basiert auf unvoreingenommene total RNA-seq. Das Verfahren nutzt eine RNase H selektive Depletion Schritt 3 unerwünschte Wirt ribosomalen und Carrier - RNA zu entfernen. Selektive Depletion reichert für virale Gehalt (Abbildung 1) und verbessert die Gesamtqualität der Sequenzierungsdaten(Abbildung 2) aus klinischen Proben. Außerdem wird tagmentation dem Protokoll angewendet, wie es deutlich Bibliothek Bauzeit reduziert. Diese Methoden wurden verwendet , um schnell große Datenmengen von Ebola und Lassa - Virus - Genome 2,4,5 erzeugen und kann verwendet werden , um ein breites Spektrum von RNA - Viren zu untersuchen. Schließlich wird der Ansatz nicht auf menschlichen Proben beschränkt; Die Nützlichkeit der selektive Depletion wurde auf Gewebeproben von Lassa-infizierte Nager und nicht-menschlichen Primaten - Krankheitsmodellen 5,6 gesammelt gezeigt.

Abbildung 1. Die Gesamt - RNA Inhalt Reflektiert Anreicherung von Lassa - Virus von Inhalt mit Selective Depletion. Beginnend Gesamtinhalt (RNA - Eingang) und Anreicherung von einzigartigen Lassa - Virus (LASV) liest (Library Inhalt) auf rRNA Depletion von neun verschiedenen klinischen Isolaten. Diese Zahl wurde von 6 geändert . Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
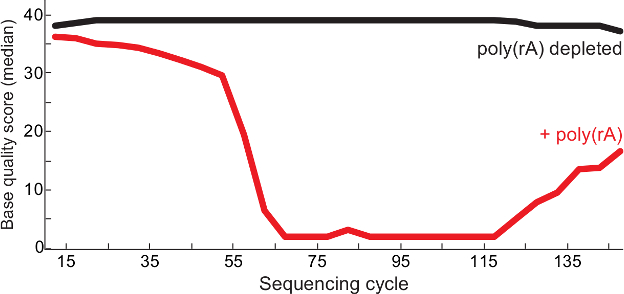
Abbildung 2. Höhere Qualität Sequenzierung Nach Carrier - RNA Depletion. Median Basisqualitäten pro Sequenzierzyklus von Poly (rA) -contaminated Bibliotheken Lassa - Virus (rot) und Kontrolle (kein Träger in der Bibliothek beobachtet, schwarz) von QC - Bericht 13. Beide lesen 1 und 2 gelesen gepaarter Ende liest in der Bibliothek BAM-Datei zusammengefügt und die Qualitätswerte werden an jeder Basis gezeigt. Diese Zahl hat sich von 6 geändert. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
Die virale RNA-seq Protokoll Details Bau von Bibliotheken direkt aus extrahiertenRNA gesammelt aus klinischen und biologischen Proben. Um sicherzustellen, dass die persönliche Sicherheit, alle viralen Serum, Plasma und Gewebeproben sollten in geeigneten Puffern vor der RNA-Extraktion inaktiviert werden. In einigen Inaktivierung und Extraktion-Kits, Träger Poly (rA) RNA ist im Preis inbegriffen; Dies wird während der anfänglichen RNase H selektive Depletion Schritt entfernt werden. Basierend auf vollständige Genesung, ist die erwartete Konzentration von Carrier-RNA 100 ng / ul. In dem Protokoll, 110 ng / & mgr; l Oligo-dT-RNA (1.1x Ladungsträgerkonzentration), zur Abreicherung eingesetzt. Wenn Poly (rA) Träger nicht in der Probe vorhanden ist, dann Oligo (dT) nicht hinzugefügt, um Erschöpfung vor werden.
Das folgende Protokoll wird für 24 Reaktionen in PCR-Plattenformat ausgelegt (bis zu 250 & mgr; l Volumen). Eine frühere Version dieses Protokolls wurde in Matranga, et al. 6.
Protokoll
Ethik-Anweisung: Lassa-Fieber-Patienten wurden für diese Studie rekrutiert unter Verwendung von Protokollen von menschlichen Probanden Ausschüsse an der Tulane Universität genehmigt, Harvard University, Broad Institute, Irrua Fach Teaching Hospital (ISTH), Kenema Regierungskrankenhaus (KGH), Oyo Staatsministerium für Gesundheit, Ibadan , Nigeria und Sierra Leone Ministerium für Gesundheit. Alle Patienten wurden mit einem ähnlichen Standard der Behandlung behandelt und das Medikament Ribavirin angeboten, auch nicht entschieden, sie in der Studie teilzunehmen. Für Lassa-Fieber (LF) der Patienten, die Behandlung mit Ribavirin folgte den derzeit empfohlenen Richtlinien und wurde in der Regel so schnell wie LF angeboten wurde stark vermutet.
Aufgrund der schweren Ausbruch für Ebola-Virus-Krankheit (EVD), Patienten, die nicht durch unsere Standardprotokolle zugestimmt werden konnte. Verwendung von klinischen überschüssigen Proben von EVD Patienten Stattdessen wurde von Institutional Review Boards in Sierra Leone und an der Harvard University bewertet und genehmigt. Die Office des Sierra Leone Ethik und Scientific Review Committee, die Sierra Leone Ministerium für Gesundheit und Hygiene, und der Harvard Ausschuss für die Verwendung von menschlichen Probanden haben einen Verzicht auf Zustimmung erteilt zu sequenzieren und öffentlich verfügbaren viralen Sequenzen von Patienten und Kontaktproben erhalten machen während des Ebola-Ausbruch in Sierra Leone gesammelt. Diese Stellen auch die Verwendung von klinischen und epidemiologischen Daten für de-identifizierte Proben aus allen Verdacht EVD Patienten während des Ausbruchs Antwort zu erhalten Pflege gesammelt gewährt. Die Sierra Leone Ministerium für Gesundheit und Hygiene auch die Verbringung von nicht-infektiösen, nicht-biologische Proben von Sierra Leone auf dem Broad Institute und der Harvard University für genomischen Untersuchungen bei Ausbruch Proben genehmigt.
1. DNase-Behandlung der Proben-RNA (bis zu 55 & mgr; l Extrahierte Gesamt-RNA, ~ 4 h)
- Stellen Sie die DNase - Reaktion in einer 96-Well PCR - Platte auf Eis in einem Bio - Sicherheitsschrank nach oben , wie in Tabelle 1 beschrieben , , Schritt 1.1 (Gesamtvolumen 70 ul / Vertiefung). Hinweis: Ein Master-Mix hergestellt werden können.
- Vortex sanft und gründlich, dann bei 280 × g bei RT Zentrifuge für 1 min.
- für 30 min bei 37 ° C inkubieren.
- Cleanup unter Verwendung von RNA Solid Phase Reversible Immobilisation (SPRI) Perlen.
- Warm RNA Perlen für 30 min auf RT.
- Sanft RNA Perlen Flasche schütteln alle magnetischen Partikel zu suspendieren, die bereits gelöst haben. In 1.8x Volumen (126 ul) von RNA-Perlen zu DNase behandelten RNA (70 & mgr; l), mischen Sie mit einer Pipette 10-mal und Inkubation für 5 min bei RT (Gesamtvolumen in gut 196 ul).
- Setzen Sie Mischung auf dem Magnetstation. Warten, bis die Lösung klar (5 - 10 min).
- Entfernen gelöscht Lösung, während auf der Station mit einer Pipette und entsorgen. Während auf der Station, waschen Perlen durch Pellet mit 70% Ethanol bedeckt und für 1 min inkubiert. Entfernen Ethanol mit Pipette und entsorgen. Wiederholen für insgesamt zwei Waschungen.
Hinweis: Die Verwendung genau 70% frisch zubereitet etHANOL ist kritisch, da ein höherer Prozentsatz in ineffizienten Waschen von kleineren Molekülen führen, während <70% Ethanolverlust der Probe 7 führen könnte. - Halten Sie Platte auf der Station und offen lassen, an der Luft trocknen. Hinweis: Stellen Sie sicher, dass die Kügelchen vollständig trocknen lassen, bis Perlen zu knacken beginnen.
- In 55 ul Nuklease-freies Wasser auf die Platte RNA zu eluieren. Entfernen Platte von der Station die Perlen zu mischen und Wasser durch sorgfältiges Pipettieren. Hinweis: Alternativ können Sie auch weniger Wasser (≤ 10 & mgr; l), um die Gesamt-RNA zu konzentrieren.
- Legen Sie die Platte wieder auf die Station. Warten Sie, bis die Lösung mit einer Pipette auf neue Schraubverschluss Rohr für die Langzeitlagerung (-80 ° C) zu übertragen löscht. Platz 5 ul RNA in neue 96-Well-PCR-Platte zur Abreicherung (Schritt. 2.4).
- Optional:. Speichern und verdünnen 1 ul in 19 & mgr; l Wasser (1:20) für qRT-PCR von rRNA (zB 18S, 28S rRNA) (Tabelle 2) und Virusmarker 5
2. Selektive Depletion von ribosomalen und Carrier-RNA aus Viral RNA-Probe (ca. 4 h)
- Machen Sie 5x Hybridisierung und 10x RNase H Reaktionspuffer und nukleasefreiem Wasser mit linearen Acrylamid Träger , wie in Tabelle 1 beschrieben.
- Set up Hybridisierungsreaktion von RNA mit rRNA Verarmungs Oligos (Tabelle 3) und Oligo (dT) auf Eis in einem 96-Well - PCR - Platte kombiniert , wie in Tabelle 1 beschrieben.
Hinweis: Ein Master-Mix hergestellt werden können. 50 Femtogramm (fg) einer einzigartigen synthetischen RNA (ERCCs 8) für die Verfolgung sowohl der viralen Sequenzierung und potenziellen Index gelesen Kreuzkontamination hinzugefügt werden.- Vortex sanft und gründlich, dann bei 280 × g bei RT Zentrifuge für 1 min.
bei 95 ° C inkubieren für 2 min, langsam erhöhende bis 45 ° C bei -0,1 ° C pro sec. Halten Sie den Thermocycler bei 45 ° C.
- Vortex sanft und gründlich, dann bei 280 × g bei RT Zentrifuge für 1 min.
- Richten Sie RNase H-Reaktionsmischung auf Eis, wie beschreibenVorwärmen d in Tabelle 1, dann bei 45 ° C für 2 min. Hinweis: Ein Master-Mix hergestellt werden können.
- Fügen Sie die vorgewärmte RNase H-Mix in die Hybridisierungsreaktion in der Platte, während die Platte bei 45 ° C in den Thermocycler zu halten.
- Gut mischen durch vorsichtiges Pipettieren 6 - 8 mal. für weitere 30 min bei 45 ° C inkubieren. Legen Sie auf dem Eis.
- Stellen Sie die DNase - Reaktionsmischung auf Eis, wie in Tabelle 1 beschrieben . Hinweis: Ein Master - Mix hergestellt werden können.
- In der RNase H-Reaktion in der Platte, Wirbel sanft und gründlich, dann bei 280 × g bei RT Zentrifuge für 1 min. für 30 min bei 37 ° C inkubieren.
- Stopp DNase Reaktion durch Zugabe von 5 ul 0,5 M EDTA. Vortex sanft und gründlich, dann bei 280 × g bei RT Zentrifuge für 1 min.
- Cleanup unter Verwendung von RNA-Kügelchen (siehe Schritt 1.3) mit einem 1,8x Volumen (144 ul) Perlen. Eluieren in 11 ul Nuklease-freies Wasser. Hinweis: Für einen sicheren Kaltlagerung, Lager abgereichertem RNA-Proben bei -80 ° C O / N.
3. cDNA Synthesis (~ 6 h)
- Mix rRNA / Träger-verarmten RNA mit zufälligen Primern auf Eis in einer 96-Well PCR - Platte , wie in Tabelle 1 beschrieben, Wirbel sanft und gründlich, dann bei 280 × g bei RT Zentrifuge für 1 min.
- Das Gemisch wird auf 70 ° C für 10 min in einem Thermocycler. Unmittelbar nach Hitzedenaturierung, legen Sie die RNA auf Eis für 1 - 5 min. Nicht in die RNA (auch auf Eis) stehen für länger als 5 Minuten vor dem ersten Strang Reaktion.
- Erstes Strangsynthese - Reaktionsmischung auf Eis, wie in Tabelle 1 beschrieben.
Hinweis: Ein Master-Mix hergestellt werden können.- In den RNA / Random-Primer-Mix in der Platte, Wirbel sanft und gründlich, dann bei 280 × g bei RT Zentrifuge für 1 min. Inkubieren bei 22 - 25 ° C für 10 min.
- bei 55 ° C inkubieren für 60 min in einem Luft Inkubator. Legen Sie die Platte auf Eis, um die Reaktion zu beenden. Nichte: Die Verwendung eines Luft-Inkubator wird empfohlen die Glühung Primern während dessen allmähliche Erwärmung der Erststrang-Reaktion zu erzeugen, und der erste Strang zu dehnen beginnt.
- Set Zweitstrangsynthese - Reaktionsmischung auf Eis, wie in Tabelle 1 beschrieben.
Hinweis: Ein Master-Mix hergestellt werden können.- In den ersten Strangsynthesereaktion in der Platte, Wirbel sanft und gründlich, dann bei 280 × g bei RT Zentrifuge für 1 min. Inkubieren für 2 Stunden bei 16 ° C (halten Deckel bei 25 ° C). Lassen Sie nicht die Temperatur über 16 ° C steigen.
- Legen Sie die Platte auf Eis, dann inaktivieren Reaktion durch Zugabe von 5 ul 0,5 M EDTA, vorsichtig mischen und gründlich, dann Zentrifuge bei 280 × g bei RT für 1 min.
- Cleanup mit DNA-Kügelchen (Schritt 1.3 für Protokoll sehen) unter Verwendung von 1,8x Volumen (153 ul) von Perlen. Eluieren in 9 & mgr; l Elutionspuffer (EB). Speichern 1 ul für die Quantifizierung. Verwenden Sie 1 ng cDNA für subsequent Schritte. Wenn cDNA-Konzentration zu niedrig ist, zu erfassen, verwenden 4 ul cDNA für tagmentation (siehe Schritt 4.1).
- Für einen sicheren Kaltlagerung, zu speichern doppelsträngige cDNA bei 4 ° CO / N oder -20 ° C zur Langzeitlagerung.
4. Bibliothek Vorbereitung - DNA Library Construction (~ 4 h)
- Transfer 4 ul cDNA in eine 96-Well-Platte und die verbleibende cDNA für einen zweiten Versuch zu speichern, wenn nötig.
- Stellen Sie die tagmentation Reaktion auf Eis, wie in Tabelle 1 beschrieben.
Hinweis: Ein Master-Mix hergestellt werden können. Um Hintergrund und die Gesamtkosten, das Gesamtvolumen der tagmentation Reaktion reduzieren wird von 20 auf 10 & mgr; l reduziert. Wie cDNA der begrenzende Faktor ist, wird die Menge von ATM verwendet (dh transposome) in der Reaktion ebenfalls verringert die Zahl der Integrationsstellen zu verringern.- Hinzufügen tagmentation Mischung cDNA in der Platte, vortex sanft und gründlich und Zentrifuge bei 280 xg (bei RT) für 1 min.Inkubieren bei 55 ° C für 5 min Halten bei 10 ° C.
- Nachdem bei 10 ° C, fügen Sie sofort 2,5 ul Neutralize Tagment Buffer (NT), um die Reaktion zu beenden. Mischen durch Pipettieren von oben und unten, dann Zentrifuge bei 280 xg (bei RT) für 1 min.
- 5 min bei RT inkubiert.
- Set up PCR Amplifikationsreaktion auf Eis , wie in Tabelle 1 beschrieben.
- Vortex sanft und gründlich, dann bei 280 × g bei RT Zentrifuge für 1 min.
- Führen PCR auf Thermocycler unter Verwendung der Bedingungen , die in Tabelle 1.
Hinweis: 12 PCR-Zyklen werden für 1 ng cDNA tagmented vorgeschlagen; jedoch virale klinischen Proben haben oft nicht nachweisbar Mengen an cDNA. Für geringe Mengen an cDNA (<1 ng), verwenden Sie bis 18 PCR-Zyklen bis genug Bibliothek für die Sequenzierung erstellen.
- Bibliothek Vorbereitung - Bereinigung und Bündelung für die Sequenzierung
- Bringen Probe bis zu 50 & mgr; l mit EB.
- Cleanup mitDNA-Kügelchen (Schritt 1.3 für Protokoll sehen) unter Verwendung von 0,6x Volumen (30 ul) Perlen. Eluieren in 15 ul EB.
- Die Konzentration der Bibliothek (Abbildung 3) durch die Durchführung Bereichsanalyse (150 bis 1.000 bp) mit Bioanalyzer Software 9, ohne Primer - Dimere (~ 120 bp) aus der Region Analyse. Hinweis: Alternativ können qPCR verwendet werden , um Bibliotheken zu quantifizieren 10.
- Pool-Bibliotheken auf der niedrigsten molaren Konzentration von 1 nM oder mehr beträgt. Wenn Bibliothek unter 1 nM ist, fügen Sie ein kleines Volumen von Bibliothek Pool (~ 1x Volumen von anderen Bibliotheken) Sequenzinformationen aus diesen Bibliotheken zu erfassen.
- Cleanup-Pool mit 0,7x DNA Perlen wie oben beschrieben (siehe Schritt 2). Eluieren in 15 ul EB. Hinweis: Volumen der Perlen auf dem endgültigen Volumen des Beckens ab.
- Analysieren Pool 9. Bestimmen Sie molare Konzentration nach Regionen Analyse der Durchführung (150 bis 1.000 bp) 9. Hinweis: Alternativ können qPCR verwendet werden Bibliothek Pool zu quantifizieren 10 </ Sup>.
- Last - Sequenzer auf 10.00 Uhr Bibliothek Konzentration 101 bp zu erzeugen, gepaart-End liest mit Dual - Barcode 11 liest.
Abbildung 3. Bibliotheken Hergestellt aus Ebola Virus klinischen Proben. Gel - Bild von 4 repräsentativen Ebola - Virus (EBOV) Bibliotheken. Regionen der Bibliothek und der Primer - Dimere werden angezeigt. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
Ergebnisse
Das beschriebene Protokoll ermöglicht die Erzeugung von qualitativ hochwertigen Sequenzierung liest aus Low-Input-Virus-RNA-Proben, während für einzigartige virale Inhalte zu bereichern. Wie in Abbildung 1 gezeigt, angereichert das Protokoll einzigartige Inhalte Lassa - Virus mindestens fünffach in allen Proben ( im Vergleich zu nicht-verarmte Kontrollen) mit mindestens einer Million Kopien von 18S rRNA (~ 100 pg Gesamt - RNA). Ebenso Sequenzierungs Erfolg auch mit der Menge des Virus in einer gegebenen Probe korreliert. Verwendung qRT-PCR als Surrogat für die Virusmenge, die Proben enthielten ~ 1.000 oder mehr viralen Genomkopien meist vollständige Baugruppen erstellt (Daten nicht gezeigt). Darüber hinaus Abreicherung von Poly (rA) Träger reduziert Homopolymer - Sequenzen A und T in Bibliotheken, in sauberer Vorbereitungen resultierende und eine bessere Qualität Sequenzierung sichergestellt liest (Abbildung 2). Endgültige Bibliotheken von extensiven viralen klinischen Proben haben oft ein breites Fragment Länge von 150 bis 1000 bp(Abbildung 3).
Nach der Sequenzierung Probe misidentification und Übersprechens zwischen Bibliotheken innerhalb eines Pools 12, nur Index liest mit einer Basisqualitätsfaktor von 25 (q25) und sicherzustellen , Null - Fehlpaarungen gehalten werden während des Demultiplexprozess zu reduzieren. Virale Genome werden mit einer Bioinformatik Pipeline spezifisch für divergent Viren 2,4-6 montiert. Diese Werkzeuge sind in https://github.com/broadinstitute/viral-ngs oder durch kommerzielle Cloud - Plattformen 4.
| Schritt 1.1: DNase Reaktion | |
| Reagens | Volumen pro Reaktion (ul) |
| 10x DNase Puffer | 7 |
| Nuklease-freies Wasser | 6 |
| Heraus viralen RNA | 55 |
| DNase (2 U / & #181; l) | 2 |
| Volle Lautstärke | 70 |
| Schritt 2.1: 5x Hybridisierungspuffer | |
| Reagens | Volume 1 ml (ul) |
| 5 M NaCl | 200 |
| 1 M Tris-HCl (pH 7,4) | 500 |
| Nuklease-freies Wasser | 300 |
| Volle Lautstärke | 1000 |
| Schritt 2.1: 10x RNase H - Reaktionspuffer | |
| Reagens | Volume 1 ml (ul) |
| 5 M NaCl | 200 |
| 1 M Tris-HCl (pH 7,5) | 500 |
| 1 M MgCl 2 | 200 |
| Nuklease-freies Wasser | 500 |
| Volle Lautstärke | 1000 |
| Schritt 2.1: Wasser wi-ten linearen Acrylamid | |
| Reagens | Volume 1 ml Puffer (ul) |
| Nuklease-freies Wasser | 992 |
| Lineares Acrylamid (5 mg / ml) | 8 |
| Volle Lautstärke | 1000 |
| Schritt 2.2: Hybridisierungsreaktion für die selektive Depletion | |
| Reagens | Volumen pro Reaktion (ul) |
| 5x Hybridisierungspuffer | 2 |
| rRNA-Depletion oligo-Mix (100 uM) | 1,22 |
| Oligo (d) T (550 ng / ul) | 1 |
| DNase-behandelte Gesamt-RNA | bis zu 5 |
| Spike-in-RNA (Dies ist optional) | 0,5 |
| Wasser (mit linearen acrylamid) | bringen bis zu 10 Gesamt |
| insgesamt Volumich | 10 |
| Schritt 2.3: RNase H - Reaktion für die selektive Depletion | |
| Reagens | Volumen pro Reaktion (ul) |
| 10x RNase H Reaction Buffer | 2 |
| Wasser (mit linearen acrylamid) | 5 |
| Thermostabile RNase H (5 U / ul) | 3 |
| Volle Lautstärke | 10 |
| Schritt 2.4: DNase Reaktion Post selektive Depletion | |
| Reagens | Volumen pro Reaktion (ul) |
| 10x DNase Buffer | 7.5 |
| Wasser (mit linearen acrylamid) | 44.5 |
| RNase-Inhibitor (20 U / ul) | 1 |
| RNase-freie DNase I (2,72 U / ul) | 2 |
| Das Gesamtvolumen (mit RNase H-Reaktion) | 75 |
| Schritt 3.1: cDNA - Synthese, Random - Primer - Hybridisierung | |
| Reagens | Volumen pro Reaktion (ul) |
| rRNA / Träger-verarmten RNA | 10 |
| 3 ug Zufallsprimer | 1 |
| Volle Lautstärke | 11 |
| Schritt 3.2: Der erste Strang der cDNA - Synthesereaktion | |
| Reagens | Volumen (ul) |
| 5x First Strand Reaction Buffer | 4 |
| 0,1 M DTT | 2 |
| 10 mM dNTP mix | 1 |
| RNase-Inhibitor (20 U / ul) | 1 |
| Reverse-Transkriptase (hinzufügen letzte) | 1 |
| Das Gesamtvolumen (mit RNA oben) | 20 |
| Schritt 3.3: Zweiter Strang cDNA - Synthesereaktion | |
| Reagens | Volumen (ul) |
| RNase-freies Wasser | 43 |
| 10x Second-Strand Reaction Buffer | 8 |
| 10 mM dNTP mix | 3 |
| E. coli DNA - Ligase (10 U / ul) | 1 |
| E. coli DNA Polymerase I (10 U / ul) | 4 |
| E. coli RNase H (2 U / ul) | 1 |
| Das Gesamtvolumen (mit 1. Strang - Reaktion) | 80 |
| Schritt 4.2: Tagmentation Reaktion | |
| Reagens | Volumen (ul) |
| Amplicon Tagment Mix (ATM) | 1 |
| Tagment DNA-Puffer (TD) | 5 |
| Das Gesamtvolumen (mit cDNA) | 10 |
| Schritt 4.3: Bibliothek PCR - Reaktion | |
| Reagens | Volumen (ul) |
| PCR Master Mix (NPM) | 7.5 |
| Index 1 Primer (i7) | 2.5 |
| Index 2 Primer (i5) | 2.5 |
| Das Gesamtvolumen (mit tagmented cDNA) | 25 |
| Schritt 4.3.2: Bibliothek PCR - Bedingungen | |
| 72 ° C, 3 min | |
| 95 ° C, 30 sec | |
| bis zu 18 Zyklen-10 sec bei 95 ° C, 30 sec bei 55 ° C, 30 sec bei 72 ° C | |
| 72 ° C, 5 min | |
| 10 ° C, für immer | |
Tabelle 1:. Reaction Set-up und Puffer Schritt- für -Schritt - Tabellen mit Inhalten aller Puffer und Reaktionsmischungen.
Tabelle 2: qRT-PCR - Primer - Sequenzen Primer für die Messung Host (18S rRNA) und virale (Ebola und Lassa) Inhalt.. "KGH" ist Kenema Regierungskrankenhaus in Sierra Leone, wo die Ebola - Primer 2 getestet wurden. 'Kulesh' ist der Ermittler, der Primer 14 gesetzt entworfen.
Tabelle 3: ribosomale RNA (rRNA) Depletion Oligos 195 50 Nukleotide lange Sequenzen , die komplementär zu menschlicher rRNA für die selektive Depletion Schritt 6.. Bitte hier klicken , um diese Datei herunterzuladen.
| Oligo - Name | Sequenz (5 'bis 3') |
| Ebola KGH FW | GTCGTTCCAACAATCGAGCG |
| Ebola KGH RV | CGTCCCGTAGCTTTRGCCAT |
| Ebola kulesh FW | TCTGACATGGATTACCACAAGATC |
| Ebola kulesh RV | GGATGACTCTTTGCCGAACAATC |
| Lassa SL FW | GTA AGC CCA GCD GYA AAB CC |
| Lassa SL RV | AAG CCA CAG AAA RCT GGS AGC A |
| 18S rRNA FW | TCCTTTAACGAGGATCCATTGG |
| 18S rRNA RV | CGAGCTTTTTAACTGCAGCAACT |
Diskussion
Das skizzierte Ansatz ermöglicht robuste, universelle, schnelle Sequenzierung und wurde zur Sequenzierung von Ebola - Virus während der 2014 Ausbruch 2,4 verwendet. Durch die Kopplung selektive Depletion und cDNA-Synthese mit tagmentation Bibliothekskonstruktion wurde die Gesamtprozesszeit um ~ 2 Tage reduziert von früheren Methoden Adaptorligation. In jüngerer Zeit wurde dieses Protokoll , das von internationalen Mitarbeiter und andere mit großem Erfolg 15,16 und wird eingesetzt , um Labors in Westafrika zu unterstützen lokale Genomik-basierte Studien und Diagnostik 17 eingesetzt.
Das hier beschriebene Protokoll verwendet Zufallsprimer cDNA-RNA-seq-Bibliotheken für die virale vorzubereiten. Im Gegensatz zu früheren viralen RNA-seq Ansätze, es erfordert keine a priori Kenntnis von Sequenzdaten oder aufwendige und zeitraubende Primerdesign für einen bestimmten Virus oder Klade. Das Verfahren kann auf jede viral RNA Probe angewendet werden. Beispielsweise wurde es verwendet, viralen Gehalt von sowohl Ebola zu erzeugenund Lassa Proben 6. Das Protokoll kann auch Projekte für Host transkriptomischen, Metagenom und Erreger Entdeckung Sequenzierung 1 verwendet werden.
Ein kritischer Schritt des Protokolls wird RNase H Verdau gezielte, ein Hochdurchsatz, kostengünstiges Verfahren zum Entfernen von unerwünschten Träger- und Host-RNA aus viralen Proben. Die selektive Depletion Schritt des Protokolls verwendet viele Komponenten und erfordert Geschicklichkeit und Genauigkeit. Extra-Zeit und Sorgfalt sollte bei der Ersteinrichtung genommen werden.
Da die meisten klinischen Serum- und Plasmaproben oft sehr wenig Nukleinsäurematerial aufweisen, sind Verunreinigungen und Probenverlust gemeinsam. Um diese Probleme zu vermeiden, sollten Sie besonders vorsichtig sein, wenn dieses Protokoll verwenden. Zunächst wird RNA sehr anfällig für Degradation; Daher sollten alle Bereiche sauber und frei von Nukleasen sein. Zweitens, um Proben zu identifizieren geeignet für den Einsatz in diesem Protokoll qRT-PCR - Assays für sowohl Wirts - RNA und Virus sollte zur Quantifizierung 5,6 verwendet werden . Wenn der Eingang Vergleich beträgt mit Sequenzierung ergibt sich aus dem Protokoll, Sequenzierung Erfolg (dh die Erzeugung von genügend Daten für die vollständige virale Montage) korreliert mit Proben , die mindestens 100 pg Gesamt - RNA und 1.000 Kopien des Virus enthalten. Drittens Einwirkung von Umweltquellen von Nukleinsäuren sollten vermieden werden. Das Protokoll hier skizzierte in einem Biosicherheitsschrank für Sicherheitsvorkehrungen durchgeführt und zur Begrenzung der Umweltschadstoffe. Darüber hinaus haben unsere Gruppe und anderen bemerkt , dass kommerzielle Enzyme bakterielle Nukleinsäuren in niedrigen Eingangsproben von verunreinigenden 6,18 eine andere Quelle sein kann. Die Verwendung eines sauberen Arbeitsplatz (zB PCR - Haube, biologische Sicherheit Kabinett) und Negativkontrollen (zB Wasser oder Puffer) wird dazu beitragen , lindern und Verschmutzung zu verfolgen sind. Bei Proben mit <100 pg Gesamt-RNA, nur Poly (rA) Carrier-RNA, nicht rRNA, sollte eine hohe Qualität Sequenzierergebnisse erschöpft sein, um sicherzustellen, während Materialverlust zu begrenzen. Für sehrniedrige Eingangsproben cDNA-Amplifikationsverfahren können 19 besser geeignet sein, obwohl Poly (rA) Träger sollte vor der cDNA - Synthese entfernt werden.
Die Abreicherung von Wirts rRNA reichert für virale Gehalt in Sequenzierungsbibliotheken und ist anwendbar auf verschiedene Probensammlungen einschließlich Serum oder Plasma, und mehrere Arten von Gewebe von Nagern und nicht-menschlichen Primaten 5,6. In nicht-menschlichen Organismen, liest Ausrichten zu 28S rRNA nach Erschöpfung blieb, was darauf hindeutet 28S rRNA ist weniger konserviert zwischen Menschen und anderen Spezies 6,20. Bei Verwendung dieser Methode mit nicht-humanen Isolate, kann es notwendig sein , mit DNA - Oligos zu ergänzen , die komplementär zu den divergierenden rRNA - Sequenzen der spezifischen Wirts 3,21.
Da das Protokoll unvoreingenommen ist, liest virale nur einen kleinen Bruchteil der gesamten Bibliothek Inhalt darstellen. Obwohl rRNA ist die häufigste Art des Wirts RNA und nur ein kleiner Prozentsatz der rRNA liest (0; 1%) werden nach der selektiven Erschöpfung, alle anderen Wirts - RNA (zB mRNA) bleiben nach Erschöpfung und Konto kann für viele Sequenzierung liest aus der Probe gefunden. Daher "Überabtasten" (dh oversequencing) wird einzelne Bibliotheken benötigt , um für Anrufe virale Montage und Variante genügend Deckung zu haben. Für unsere Studien versuchen wir zu Folge ~ 20 Millionen liest pro Probe 2,5 ausreichende Tiefe für die Analyse von viralen genomischen und die damit verbundenen Varianten sowie metagenomic Inhalt zu haben. Für metagenomic und Studien Entdeckung pathogen ist es wichtig, dass kontaminierende Wirts-DNA zu beachten, durch DNase-Verdau entfernt. Daher Viren und andere Pathogene, die DNA-Genome enthalten kann während des Prozesses jedoch RNA Zwischenprodukte können noch sequenzierenden verloren.
Offenlegungen
The authors have no competing financial interests.
Danksagungen
This work has been funded in part with Federal funds from the National Institutes of Health, Office of Director, Innovator (No.: DP2OD06514) (PCS) and from the National Institute of Allergy and Infectious Diseases, National Institutes of Health, Department of Health and Human Services, under Contracts (No:HHSN272200900018C, HHSN272200900049C and U19AI110818).
Materialien
| Name | Company | Catalog Number | Comments |
| 96-well PCR Plates | VWR | 47743-953 | |
| Strips of Eight Caps | VWR | 47745-512 | |
| Nuclease-free water | Ambion | AM9937 | 50 ml bottle |
| TURBO DNase | Ambion | AM2238 | post RNA extraction step, 2 U/µl, buffer included |
| PCR cycler | any PCR cyclers | ||
| Agencourt RNAClean XP SPRI beads | Beckman Coulter Genomics | A63987 | beads for RNA cleanup |
| Real Time qPCR system | any system | ||
| DynaMag-96 Side Skirted Magnet | Invitrogen | 12027 | |
| 70% Ethanol | prepare fresh | ||
| qRT-PCR primers | IDT DNA | see Table 2 | |
| 5 M NaCl | Ambion | AM9760G | |
| 1 M Tris-HCl pH 7.4 | Sigma | T2663-1L | |
| 1 M Tris-HCl pH 7.5 | Invitrogen | 15567-027 | |
| 1 M MgCl2 | Ambion | AM9530G | |
| Linear acrylamide | Ambion | AM9520 | |
| DNA oligos covering entire rRNA region | IDT DNA | see Table 3, order lab-ready at 100 µM | |
| Oligo (dT) | IDT DNA | 40 nt long, desalted | |
| Hybridase Thermostable RNase H | Epicentre | H39100 | |
| RNase-free DNase Kit | Qiagen | 79254 | post selective depletion step |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Random Primers | Invitrogen | 48190-011 | mostly hexamers |
| 10 mM dNTP mix | New England Biolabs | N0447L | |
| SuperScript III Reverse Transcriptase | Invitrogen | 18080-093 | with first-strand buffer, DTT |
| Air Incubator | any air incubator cyclers | ||
| NEBNext Second Strand Synthesis (dNTP-free) Reaction Buffer | New England Biolabs | B6117S | 10x |
| E. coli DNA Ligase | New England Biolabs | M0205L | 10 U/μl |
| E. coli DNA Polymerase I | New England Biolabs | M0209L | 10 U/μl |
| E. coli RNase H | New England Biolabs | M0297L | 2 U/μl |
| 0.5 M EDTA | Ambion | AM9261 | |
| Agencourt AMPure XP SPRI beads | Beckman Coulter Genomics | A63881 | beads for DNA cleanup |
| Elution Buffer | Qiagen | 10 mM Tris HCl, pH 8.5 | |
| Quant-iT dsDNA HS Assay Kit | Invitrogen | Q32854 | |
| Qubit fluorometer | Invitrogen | Q32857 | |
| Nextera XT DNA Sample Prep Kit | Illumina | FC-131-1096 | |
| Nextera XT DNA Index Kit | Illumina | FC-131-1001 | |
| Tapestation 2200 | Agilent | G2965AA | |
| High Sensitivity D1000 reagents | Agilent | 5067-5585 | |
| High Sensitivity D1000 ScreenTape | Agilent | 5067-5584 | |
| BioAnalyzer 2100 | Agilent | G2939AA | |
| High Sensitivity DNA reagents | Agilent | 5067-4626 | |
| Library Quantification Complete kit (Universal) | Kapa Biosystems | KK4824 | alternative to tapestation, bioanalyzer for library quantification |
Referenzen
- Stremlau, M. H., et al. Discovery of novel rhabdoviruses in the blood of healthy individuals from West Africa. PLoS Negl Trop Dis. 9, e0003631(2015).
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345, 1369-1372 (2014).
- Morlan, J. D., Qu, K., Sinicropi, D. V. Selective depletion of rRNA enables whole transcriptome profiling of archival fixed tissue. PLoS One. 7, e42882(2012).
- Park, D. J., et al. Ebola Virus Epidemiology, Transmission, and Evolution during Seven Months in Sierra Leone. Cell. 161, 1516-1526 (2015).
- Andersen, K. G., et al. Clinical Sequencing Uncovers Origins and Evolution of Lassa Virus. Cell. 162, 738-750 (2015).
- Matranga, C. B., et al. Enhanced methods for unbiased deep sequencing of Lassa and Ebola RNA viruses from clinical and biological samples. Genome Biol. 15, 519(2014).
- Tang, F., et al. RNA-Seq analysis to capture the transcriptome landscape of a single cell. Nat Protoc. 5, 516-535 (2010).
- Jiang, L., et al. Synthetic spike-in standards for RNA-seq experiments. Genome Res. 21, 1543-1551 (2011).
- Agilennt Technologies. , Available from: http://www.agilent.com/cs/library/usermanuals/Public/G2946-90004_Vespucci_UG_eBook_(NoSecPack).pdf. (2015).
- Kapa Biosystems. , Available from: https://www.kapabiosystems.com/product-applications/products/next-generation-sequencing-2/library-quantification/ (2015).
- Illumina Technologies. , Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/system_documentation/miseq/preparing-libraries-for-sequencing-on-miseq-15039740-d.pdf. (2015).
- Kircher, M., Sawyer, S., Meyer, M. Double indexing overcomes inaccuracies in multiplex sequencing on the Illumina platform. Nucleic Acids Res. 40, 3(2012).
- Andrews, S. Babraham Bioinformatics. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Trombley, A. R., et al. Comprehensive panel of real-time TaqMan polymerase chain reaction assays for detection and absolute quantification of filoviruses, arenaviruses, and New World hantaviruses. Am J Trop Med Hyg. 82, 954-960 (2010).
- Hu, Y., et al. Serial high-resolution analysis of blood virome and host cytokines expression profile of a patient with fatal H7N9 infection by massively parallel RNA sequencing. Clin Microbiol Infect. 21, e1-4 713(2015).
- Simon-Loriere, E., et al. Distinct lineages of Ebola virus in Guinea during the 2014 West African epidemic. Nature. 524, 102-104 (2015).
- Folarin, O. A., Happi, A. N., Happi, C. T. Empowering African genomics for infectious disease control. Genome Biol. 15, 515(2014).
- Blainey, P. C., Quake, S. R. Digital MDA for enumeration of total nucleic acid contamination. Nucleic Acids Res. 39, 19(2011).
- Malboeuf, C. M., et al. Complete viral RNA genome sequencing of ultra-low copy samples by sequence-independent amplification. Nucleic Acids Res. 41, 13(2013).
- Gonzalez, I. L., Sylvester, J. E., Smith, T. F., Stambolian, D., Schmickel, R. D. Ribosomal RNA gene sequences and hominoid phylogeny. Mol Biol Evol. 7, 203-219 (1990).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nat Methods. 10, 623-629 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten