Method Article
Imparziale sequenziamento profondo di RNA virus da campioni clinici
In questo articolo
Riepilogo
This protocol describes a rapid and broadly applicable method for unbiased RNA-sequencing of viral samples from human clinical isolates.
Abstract
Qui descriviamo un protocollo sequenza di RNA di nuova generazione che permette de novo assemblee e chiamate variante intra-host dei genomi virali raccolti da fonti clinici e biologici. Il metodo è imparziale e universale; utilizza primer casuali per la sintesi del DNA e non richiede alcuna conoscenza preliminare del contenuto di sequenza virale. Prima costruzione della libreria, selettivo digestione RNasi H-based è utilizzata per esaurire indesiderato RNA - vettore compreso poli (Ra) e RNA ribosomiale - dal campione di RNA virale. deplezione selettiva migliora sia la qualità dei dati e il numero di univoco legge in biblioteche RNA virali sequenziamento. Inoltre, passo un trasposasi-based 'tagmentation' è usato nel protocollo in quanto riduce il tempo complessivo di costruzione biblioteca. Il protocollo ha permesso un rapido sequenziamento profondo di oltre 600 campioni, tra cui Lassa e Ebola virus collezioni sia da sangue e tessuti isolati, ed è ampiamente applicabile ad altri studi di genomica microbica.
Introduzione
La nuova generazione di sequenziamento del virus da fonti cliniche può informare la trasmissione e l'epidemiologia delle infezioni, così come l'aiuto supporto romanzo diagnostica, vaccini e lo sviluppo terapeutico. sintesi del DNA utilizzando primer casuali ha permesso l'individuazione e l'assemblaggio dei genomi da divergenti, co-infettante o anche di nuovi virus 1,2. Come con altri metodi imparziali, contaminanti indesiderati occupano molti sequenziamento legge e un impatto negativo i risultati di sequenziamento. Host e poli (Ra) RNA carrier sono contaminanti presenti in molte collezioni di campioni virali esistenti.
Il protocollo descrive un modo efficiente ed economico di profondi genomi virali sequenza di RNA sulla base imparziale totale RNA-Seq. Il metodo utilizza una deplezione selettiva passo RNase H 3 per rimuovere indesiderati ribosomiale host e l'RNA carrier. Deplezione selettiva arricchisce di contenuti virali (Figura 1) e migliora la qualità complessiva dei dati di sequenziamento(Figura 2) da campioni clinici. Inoltre, tagmentation viene applicato al protocollo poiché riduce notevolmente i tempi di costruzione libreria. Questi metodi sono stati utilizzati per generare rapidamente grandi quantità di dati di Ebola e Lassa genomi virali 2,4,5 e possono essere utilizzati per studiare una vasta gamma di virus a RNA. Infine, il metodo non è limitato a campioni umani; l'utilità di esaurimento selettivo è stata dimostrata su campioni di tessuto raccolti da roditori Lassa-infette e modelli di malattia di primati non umani 5,6.

Figura 1. L'RNA totale contenuto riflette Arricchimento di Lassa Virus Content Utilizzando selettiva esaurimento. Partendo contenuto complessivo (ingresso RNA) e l'arricchimento di unico virus Lassa (LASV) legge (contenuto Library) sulla riduzione di rRNA da nove diversi isolati clinici. Questa cifra è stata modificata da 6 . Fai clic qui per vedere una versione più grande di questa figura.
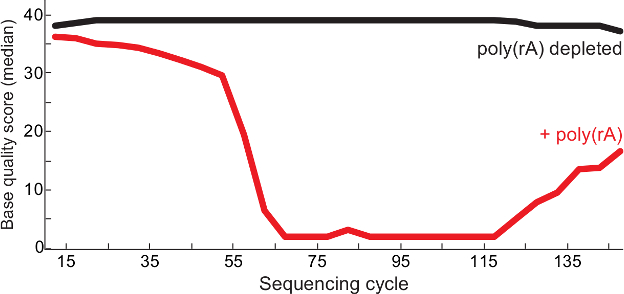
Figura 2. più di alta qualità Sequencing Dopo Carrier RNA esaurimento. Qualità di base mediani per ciclo di sequenziamento di poli (rA) -contaminated librerie virus Lassa (rosso) e di controllo (nessun vettore osservata in biblioteca, nero) dal rapporto QC 13. Sia in lettura 1 e 2 lettura di fine accoppiato letture vengono uniti nel file BAM biblioteca e il punteggio di qualità sono mostrati in ogni base. Questa cifra è stata modificata dal 6. Clicca qui per vedere una versione più grande di questa figura.
I dettagli del protocollo di costruzione virale RNA-Seq di librerie direttamente da estrattiRNA raccolti da campioni clinici e biologici. Per garantire la sicurezza personale, tutti i campioni di siero, plasma e del tessuto virali, devono essere inattivati nei buffer appropriato prima estrazione di RNA. In alcuni kit di inattivazione e di estrazione, vettore poli (rA) RNA è incluso; questo verrà rimosso durante la fase di svuotamento selettivo iniziale RNase H. Sulla base di recupero completo, la concentrazione atteso di RNA carrier è di 100 ng / ml. Nel protocollo, 110 ng / ml oligo dT RNA (concentrazione di portatori 1.1x) viene utilizzato per esaurimento. Se poly (rA) vettore non è presente nel campione, quindi oligo (dT) non dovrebbe essere aggiunto prima della deplezione.
Il seguente protocollo è stato progettato per 24 reazioni in formato piastra PCR (fino a 250 volumi mL). Una precedente versione di questo protocollo è stato segnalato in Matranga, et al. 6.
Protocollo
dichiarazione etica: pazienti febbre di Lassa sono stati reclutati per questo studio utilizzando protocolli approvati da comitati soggetti umani presso la Tulane University, Harvard University, Broad Institute, Irrua Specialist Teaching Hospital (ISTH), Ospedale governo Kenema (KGH), Oyo Ministero della Salute, Ibadan , Nigeria e Sierra Leone Ministero della Salute. Tutti i pazienti sono stati trattati con un livello simile di cura e sono stati offerti il farmaco ribavirina, anche se non hanno deciso di partecipare allo studio. Per la febbre Lassa (LF) dei pazienti, il trattamento con Ribavirina ha seguito le linee guida attualmente raccomandati ed è stato generalmente offerto non appena LF è stata fortemente sospettato.
A causa della grave epidemia di Ebola virus della malattia (EVD), i pazienti non potevano essere consentito attraverso i nostri protocolli standard. Invece l'uso di campioni in eccesso clinici di pazienti EVD è stato valutato e approvato da Institutional Review Boards in Sierra Leone e alla Harvard University. il Office della Etico e Review Comitato Scientifico la Sierra Leone, la Sierra Leone, Ministero della Salute e Igiene, e il Comitato di Harvard sulla uso di soggetti umani hanno concesso una rinuncia del consenso per sequenza e rendere sequenze virali pubblicamente disponibili ottenuti da campioni di pazienti e di contatto raccolti durante l'epidemia di Ebola in Sierra Leone. Questi corpi inoltre concesso l'uso dei dati clinici ed epidemiologici per campioni de-identificato raccolti da tutti i pazienti che ricevono cure EVD sospetti durante la risposta epidemia. Il Leone Ministero della Salute e Igiene Sierra ha inoltre approvato le spedizioni di non infettivi, i campioni non biologici da Sierra Leone a Broad Institute e Harvard University per gli studi genomici di campioni focolaio.
1. DNasi-trattamento di RNA del campione (fino a 55 ml estratti RNA totale, ~ 4 ore)
- Impostare la reazione DNasi in una piastra a 96 pozzetti PCR su ghiaccio in un armadio biosicurezza come descritto nella Tabella 1 , Step 1,1 (volume totale, 70 microlitri / pozzetto). Nota: Un mix master può essere preparato.
- Vortex delicatamente e accuratamente, quindi si centrifuga a 280 xg a temperatura ambiente per 1 min.
- Incubare a 37 ° C per 30 min.
- Cleanup utilizza in fase solida reversibile immobilizzazione (SPRI) perline RNA.
- perline RNA riscaldare a TA per 30 min.
- Agitare delicatamente bottiglia perline RNA per risospendere le particelle magnetiche che possono essere regolati. Aggiungere il volume 1,8x (126 ml) di perline RNA RNA DNasi-trattati (70 ml), mescolare con una pipetta 10 volte e incubare per 5 minuti a temperatura ambiente (volume totale di ben, 196 ml).
- Posizionare miscela sulla stazione magnetica. Attendere la soluzione per deselezionare (5 - 10 min).
- Rimuovere la soluzione eliminato mentre sulla stazione da pipetta e degli scarti. Mentre sulla stazione, lavare perline coprendo pellet con il 70% di etanolo e incubare per 1 min. Rimuovere etanolo con pipetta e degli scarti. Ripetere per un totale di due lavaggi.
Nota: L'utilizzo precisamente il 70% appena preparato etHANOL è critica, come una percentuale maggiore si tradurrà in lavaggio inefficiente delle molecole più piccole dimensioni, mentre <70% di etanolo potrebbe causare la perdita di campione 7. - Mantenere targa sulla stazione e lasciare aperta asciugare all'aria. Nota: Assicuratevi di lasciare le perline asciugare completamente fino perline cominciano a incrinarsi.
- Aggiungere 55 ml di acqua priva di nucleasi alla piastra per eluire RNA. Rimuovere la piastra dalla stazione di mescolare le perline e acqua pipettando accuratamente. Nota: In alternativa, utilizzare meno acqua (≤ 10 pl) per concentrare l'RNA totale.
- Mettere piastra posteriore della stazione. Attendere fino a quando la soluzione cancella per trasferire da pipetta nuovo tubo tappo a vite per la conservazione a lungo termine (-80 ° C). Mettere 5 ml di RNA in new 96-pozzetti PCR per esaurimento (passo. 2.4).
- Optional:. Salva e diluire 1 ml a 19 ml di acqua (1:20) per qRT-PCR di rRNA (ad esempio, 18S, 28S rRNA) (Tabella 2) e marcatori virali 5
2. L'esaurimento selettiva di ribosomiale e Carrier RNA da virale campione di RNA (~ 4 ore)
- Fare ibridazione 5x e 10x buffer di reazione RNasi H, e l'acqua priva di nucleasi con supporto acrilamide lineare come descritto nella tabella 1.
- Impostare reazione di ibridazione combinando RNA con oligonucleotidi deplezione rRNA (Tabella 3) e oligo (dT) sul ghiaccio in una piastra a 96 pozzetti PCR come descritto nella Tabella 1.
Nota: Un mix master può essere preparato. 50 femtogrammi (FG) di un RNA sintetico unico (ERCCs 8) possono essere aggiunti per il monitoraggio sia per il processo di sequenza virale e potenziale indice di lettura contaminazione incrociata.- Vortex delicatamente e accuratamente, quindi si centrifuga a 280 xg a temperatura ambiente per 1 min.
Incubare a 95 ° C per 2 min, lento rampa di 45 ° C a -0,1 ° C per sec. Pausa termociclatore a 45 ° C.
- Vortex delicatamente e accuratamente, quindi si centrifuga a 280 xg a temperatura ambiente per 1 min.
- Impostare mix di reazione RNase H sul ghiaccio come descrittod nella Tabella 1, quindi preriscaldare a 45 ° C per 2 min. Nota: Un mix master può essere preparato.
- Aggiungere la miscela preriscaldata RNase H della reazione di ibridazione in lamiera mantenendo la piastra nel termociclatore a 45 ° C.
- Mescolare bene delicatamente pipettando 6 - 8 volte. Incubare a 45 ° C per altri 30 min. Mettere in ghiaccio.
- Impostare la miscela di reazione DNasi sul ghiaccio, come descritto nella tabella 1. Nota: Un mix master può essere preparato.
- Aggiungere alla reazione RNasi H in lamiera, vortice delicatamente e accuratamente, quindi si centrifuga a 280 xg a temperatura ambiente per 1 min. Incubare a 37 ° C per 30 min.
- Smettere di reazione DNase con l'aggiunta di 5 ml di 0,5 M EDTA. Vortex delicatamente e accuratamente, quindi si centrifuga a 280 xg a temperatura ambiente per 1 min.
- Cleanup utilizzando perline di RNA (vedi punto 1.3) con un volume di 1,8 volte (144 ml) perline. Eluire in 11 ml di acqua priva di nucleasi. Nota: Per lo stoccaggio a freddo di sicurezza, negozio impoverito RNA campioni a -80 ° C O / N.
3. cDNA Synthesis (~ 6 ore)
- Mix rRNA / RNA carrier-impoverito con primer casuali sul ghiaccio in una piastra a 96 pozzetti PCR, come descritto nella tabella 1, vortex delicatamente e accuratamente, quindi si centrifuga a 280 xg a temperatura ambiente per 1 min.
- Riscaldare il miscuglio a 70 ° C per 10 minuti in un termociclatore. Subito dopo la denaturazione di calore, posizionare l'RNA in ghiaccio per 1-5 min. Non lasciare che l'RNA di stare (anche su ghiaccio) per più di 5 minuti prima del primo filamento reazione.
- Impostare mix reazione di sintesi del primo filamento sul ghiaccio come descritto nella Tabella 1.
Nota: Un maestro-mix può essere preparato.- Aggiungi RNA / random miscela di primer in lamiera, vortice delicatamente e accuratamente, quindi si centrifuga a 280 xg a temperatura ambiente per 1 min. Incubare a 22 - 25 ° C per 10 min.
- Incubare a 55 ° C in un incubatore d'aria per 60 min. Posizionare la piastra sul ghiaccio per terminare la reazione. None: L'uso di un incubatore aria è consigliabile creare progressivo riscaldamento del primo filamento di reazione durante il quale il primer ricottura e la prima parte inizia ad allungarsi.
- Impostare mix reazione di sintesi del secondo filone sul ghiaccio, come descritto nella tabella 1.
Nota: Un maestro-mix può essere preparato.- Aggiungi il primo filamento reazione di sintesi nel piatto, vortice delicatamente e accuratamente, quindi si centrifuga a 280 xg a temperatura ambiente per 1 min. Incubare per 2 ore a 16 ° C (tenere il coperchio a 25 ° C). Non permettere che la temperatura a salire sopra 16 ° C.
- Posizionare la piastra sul ghiaccio, poi inattivare reazione con l'aggiunta di 5 ml di 0,5 M EDTA, mescolare delicatamente e accuratamente, quindi si centrifuga a 280 xg a temperatura ambiente per 1 min.
- Pulizia con perline di DNA (vedi punto 1.3 per il protocollo) che utilizzano il volume 1,8 volte (153 ml) di perline. Eluire in 9 ml di tampone di eluizione (EB). Salva 1 ml per la quantificazione. Utilizzare 1 ng di cDNA per subsequepassi NT. Se la concentrazione cDNA è troppo bassa per rilevare, utilizzare 4 ml di cDNA per tagmentation (vedi punto 4.1).
- Per lo stoccaggio a freddo di sicurezza, negozio di doppio filamento cDNA a 4 ° CO / N o -20 ° C per una conservazione a lungo termine.
4. Biblioteca Preparazione - DNA Biblioteca Costruzioni (~ 4 ore)
- Trasferimento 4 ml di cDNA ad una piastra a 96 pozzetti e salvare il restante cDNA per un secondo tentativo se necessario.
- Impostare la reazione tagmentation su ghiaccio come descritto nella Tabella 1.
Nota: Un maestro-mix può essere preparato. Per ridurre il fondo e il costo complessivo, il volume totale della reazione tagmentation è ridotto da 20 a 10 microlitri. Come cDNA è il fattore limitante, la quantità di ATM (cioè, transposome) utilizzato nella reazione è anche ridotta per diminuire il numero di siti di integrazione.- Aggiungere mix tagmentation al cDNA nel piatto, vortice delicatamente e accuratamente e centrifugare a 280 g (a temperatura ambiente) per 1 min.Incubare a 55 ° C per 5 minuti, tenere a 10 ° C.
- Una volta a 10 ° C, aggiungere immediatamente 2,5 ml di Neutralizzare Tagment Buffer (NT) per terminare la reazione. Mescolare pipettando su e giù, quindi si centrifuga a 280 xg (a RT) per 1 min.
- Incubare a temperatura ambiente per 5 min.
- Impostare PCR reazione di amplificazione in ghiaccio come descritto nella Tabella 1.
- Vortex delicatamente e accuratamente, quindi si centrifuga a 280 xg a temperatura ambiente per 1 min.
- Eseguire la PCR sul termociclatore utilizzando le condizioni descritte in Tabella 1.
Nota: 12 cicli di PCR sono suggeriti per 1 ng di cDNA tagmented; tuttavia, campioni clinici virali hanno spesso quantità rilevabili di cDNA. Per basse quantità di cDNA (<1 ng), utilizzare fino a 18 cicli di PCR per creare abbastanza libreria per il sequenziamento.
- Preparazione Biblioteca - pulizia e mettere in comune per il sequenziamento
- Portare campione fino a 50 ml con EB.
- Pulizia conperline di DNA (vedi punto 1.3 per il protocollo) che utilizzano il volume 0.6x (30 ml) perline. Eluire in 15 microlitri EB.
- Determinare la concentrazione di biblioteca (figura 3) per lo svolgimento di analisi di regione (da 150 a 1000 bp) utilizzando il software bioanalizzatore 9, esclusi i dimeri di primer (~ 120 bp) da analisi regione. Nota: In alternativa, qPCR può essere utilizzato per quantificare le librerie 10.
- librerie piscina presso la più bassa concentrazione molare di 1 nm o superiore. Se biblioteca è inferiore a 1 nM, aggiungere una piccola quantità di biblioteca alla piscina (~ il volume 1x di altre biblioteche) per acquisire informazioni sulla sequenza da queste librerie.
- piscina Cleanup con 0,7x perline DNA come descritto in precedenza (vedi punto 2). Eluire in 15 microlitri EB. Nota: Volume di perline dipenderà dal volume finale della piscina.
- Analizzare piscina 9. Determinare concentrazione molare conducendo analisi regione (da 150 a 1000 bp) 9. Nota: In alternativa, qPCR può essere utilizzato per quantificare piscina libreria 10 </ Sup>.
- Carico sequencer a una concentrazione biblioteca 22:00 di generare 101 bp, abbinato-end legge con doppio codice a barre legge 11.
Figura 3. Biblioteche Costruito da virus Ebola campioni clinici. Immagine Gel di 4 rappresentante virus Ebola (EBOV) librerie. Regioni della biblioteca e di primer dimeri sono mostrati. Cliccate qui per vedere una versione più grande di questa figura.
Risultati
Il protocollo descritto consente la generazione di sequenziamento di alta qualità legge da campioni di RNA virale a basso input, arricchendo di contenuti virali unici. Come mostrato in figura 1, il protocollo arricchita unico contenuto virus Lassa almeno cinque volte in tutti i campioni (rispetto ai controlli non impoverito) con almeno un milione di copie del 18S rRNA (~ 100 pg di RNA totale). Allo stesso modo, il successo sequenziamento anche correlata con la quantità di virus in un dato campione. Utilizzando qRT-PCR come surrogato per quantità virale, i campioni che contenevano ~ 1.000 o più copie di genoma virale più spesso create assemblee pieno (dati non riportati). Inoltre, deplezione di poli (rA) portante, sequenze omopolimero di A e T in biblioteche, conseguente preparazioni puliti e garantire una migliore qualità sequenziamento letture (Figura 2). biblioteche definitivo campioni clinici virali a basso input spesso hanno un ampio frammento lunghezza da 150 a 1000 bp(Figura 3).
Dopo il sequenziamento, per ridurre errori di identificazione del campione e diafonia tra le biblioteche all'interno di una piscina di 12, solo indice di legge con un punteggio di qualità di base di 25 (Q25) e garantire a zero mancate corrispondenze sono mantenuti durante il processo di demultiplazione. Genomi virali sono assemblati utilizzando una pipeline bioinformatica specifico per virus divergenti 2,4-6. Questi strumenti sono disponibili presso https://github.com/broadinstitute/viral-ngs o attraverso piattaforme cloud commerciali 4.
| Fase 1.1: reazione DNasi | |
| Reagente | Volume per reazione (ml) |
| Buffer 10x DNase | 7 |
| acqua priva di nucleasi | 6 |
| RNA virale estratto | 55 |
| DNase (2 U / & #181; l) | 2 |
| Volume totale | 70 |
| Passo 2.1: tampone 5x ibridazione | |
| Reagente | Volume per 1 ml (ml) |
| 5 M NaCl | 200 |
| 1 M Tris-HCl (pH 7,4) | 500 |
| acqua priva di nucleasi | 300 |
| Volume totale | 1.000 |
| Fase 2.1: 10x tampone di reazione RNase H | |
| Reagente | Volume per 1 ml (ml) |
| 5 M NaCl | 200 |
| 1 M Tris-HCl (pH 7.5) | 500 |
| 1 M MgCl 2 | 200 |
| acqua priva di nucleasi | 500 |
| Volume totale | 1.000 |
| Fase 2.1: Acqua wacrilamide lineare esimo | |
| Reagente | Volume per 1 ml di tampone (ml) |
| acqua priva di nucleasi | 992 |
| acrilammide lineare (5 mg / ml) | 8 |
| Volume totale | 1.000 |
| Fase 2.2: reazione di ibridazione per la deplezione selettiva | |
| Reagente | Volume per reazione (ml) |
| 5x Tampone di ibridazione | 2 |
| oligo mix rRNA-esaurimento (100 micron) | 1.22 |
| Oligo (d) T (550 ng / ml) | 1 |
| DNasi trattati RNA totale | fino a 5 |
| Spike-in RNA (questo è facoltativo) | 0.5 |
| Acqua (con acrilammide lineare) | portare fino a 10 in totale |
| volu totaleme | 10 |
| Fase 2.3: RNase H reazione per deplezione selettiva | |
| Reagente | Volume per reazione (ml) |
| 10x RNase H tampone di reazione | 2 |
| Acqua (con acrilammide lineare) | 5 |
| Termostabile RNase H (5 U / ml) | 3 |
| Volume totale | 10 |
| Fase 2.4: messaggio reazione DNasi deplezione selettiva | |
| Reagente | Volume per reazione (ml) |
| 10x DNase Buffer | 7.5 |
| Acqua (con acrilammide lineare) | 44.5 |
| inibitore RNasi (20 U / ml) | 1 |
| DNasi RNasi-free I (2.72 U / ml) | 2 |
| Volume totale (con reazione RNasi H) | 75 |
| Fase 3.1: sintesi del DNA, Primer casuale ibridazione | |
| Reagente | Volume per reazione (ml) |
| rRNA / RNA carrier-impoverito | 10 |
| 3 mg di primer casuale | 1 |
| Volume totale | 11 |
| Fase 3.2: Primo aspetto reazione di sintesi del DNA | |
| Reagente | Volume (ml) |
| 5x First-Strand tampone di reazione | 4 |
| 0.1 M DTT | 2 |
| mix 10 mM dNTP | 1 |
| inibitore RNasi (20 U / ml) | 1 |
| trascrittasi inversa (aggiungere ultima) | 1 |
| Volume totale (con l'RNA sopra) | 20 |
| Fase 3.3: Secondo aspetto reazione di sintesi del DNA | |
| Reagente | Volume (ml) |
| acqua priva di RNasi | 43 |
| 10x tampone di reazione seconda Strand | 8 |
| mix 10 mM dNTP | 3 |
| E. coli DNA Ligase (10 U / ml) | 1 |
| E. coli DNA polimerasi I (10 U / ml) | 4 |
| E. coli RNase H (2 U / ml) | 1 |
| Volume totale (con 1 ° reazione filo) | 80 |
| Fase 4.2: reazione Tagmentation | |
| Reagente | Volume (ml) |
| Amplicon Tagment Mix (ATM) | 1 |
| Tagment DNA Buffer (TD) | 5 |
| Volume totale (con cDNA) | 10 |
| Passo 4.3: reazione Biblioteca PCR | |
| Reagente | Volume (ml) |
| PCR Master Mix (NPM) | 7.5 |
| Indice 1 Primer (i7) | 2.5 |
| Indice 2 Primer (i5) | 2.5 |
| Volume totale (con tagmented cDNA) | 25 |
| Punto 4.3.2: condizioni di PCR Biblioteca | |
| 72 ° C, 3 min | |
| 95 ° C, 30 sec | |
| fino a 18 cicli a 10 sec a 95 ° C, 30 sec a 55 ° C, 30 sec a 72 ° C | |
| 72 ° C, 5 min | |
| 10 ° C, per sempre | |
Tabella 1:. Reazione di set-up e buffer Step-by-Step tabelle con contenuto di tutti i buffer e miscele di reazione.
Tabella 2: qRT-PCR Primer sequenze primer utilizzati per la misurazione host (18S rRNA) e virali contenuti (Ebola e Lassa).. 'KGH' è Hospital Kenema governo in Sierra Leone, dove i primer di Ebola sono stati testati 2. 'Kulesh' è il ricercatore che ha progettato il primer set 14.
Tabella 3: RNA ribosomiale (rRNA) Depletion Oligos 195 lunghe sequenze di 50 nucleotidi complementari per rRNA umana per passo deplezione selettiva 6.. Cliccate qui per scaricare questo file.
| oligo Nome | Sequenza (5 'a 3') |
| Ebola KGH FW | GTCGTTCCAACAATCGAGCG |
| Ebola KGH RV | CGTCCCGTAGCTTTRGCCAT |
| Ebola Kulesh FW | TCTGACATGGATTACCACAAGATC |
| Ebola Kulesh RV | GGATGACTCTTTGCCGAACAATC |
| Lassa SL FW | GTA AGC CCA GCD GYA AAB CC |
| Lassa SL RV | AAG CCA CAG AAA RCT GGS AGC A |
| 18S rRNA FW | TCCTTTAACGAGGATCCATTGG |
| 18S rRNA RV | CGAGCTTTTTAACTGCAGCAACT |
Discussione
L'approccio delineato consente robusto, universale, rapida sequenza ed è stato utilizzato per il sequenziamento virus Ebola nel corso del 2014 2,4 scoppio. Accoppiando deplezione selettiva e sintesi del DNA con la costruzione biblioteca tagmentation, il tempo complessivo processo è stato ridotto di ~ 2 giorni da precedenti metodi adattatore legatura. Più di recente, questo protocollo è stato impiegato da collaboratori internazionali e gli altri con grande successo 15,16 e verrà distribuito ai laboratori in Africa occidentale per sostenere gli studi di ricerca genomica basata su locali e diagnostica 17.
Il protocollo qui descritto utilizza primer casuali per preparare cDNA per le librerie di RNA-Seq virali. A differenza dei precedenti approcci RNA-seq virali, essa non richiede una conoscenza a priori dei dati di sequenza o di progettazione Primer elaborato e richiede tempo per un virus o clade specifico. Il metodo può essere applicato a qualsiasi campione di RNA virale. Ad esempio, è stato utilizzato per generare contenuti virali sia Ebolae campioni Lassa 6. Il protocollo può essere utilizzato anche per la trascrittomica padrone di casa, progetti di sequenziamento metagenomiche e di scoperta patogeno 1.
Un passaggio fondamentale del protocollo è rivolto RNase H digestione, un alto rendimento, metodo a basso costo per la rimozione di vettore indesiderate e dell'RNA host da campioni virali. Il passo deplezione selettiva del protocollo utilizza molti componenti e richiede abilità e precisione. I tempi supplementari e la cura dovrebbe essere presa durante la configurazione iniziale.
Come la maggior parte dei campioni di siero e plasma clinici hanno spesso molto poco materiale acido nucleico, la contaminazione e la perdita del campione sono comuni. Per evitare questi problemi, particolare attenzione dovrebbe essere presa quando si utilizza questo protocollo. Innanzitutto, RNA è altamente suscettibile alla degradazione; quindi tutte le aree devono essere puliti e privi di nucleasi. In secondo luogo, per identificare i campioni adatti per l'utilizzo in questo protocollo, analisi qRT-PCR sia per l'RNA ospite e virus devono essere utilizzati per la quantificazione 5,6 . Quando si confrontano ingresso pari con i risultati di sequenziamento del protocollo, il successo sequenziamento (vale a dire, la generazione di dati sufficienti per l'assemblaggio virale pieno) correlati con i campioni che contenevano almeno 100 pg RNA totale e 1.000 copie del virus. Terzo, l'esposizione a fonti ambientali di acidi nucleici deve essere evitato. Il protocollo descritto qui viene eseguita in una cappa di biosicurezza per precauzioni di sicurezza e per limitare contaminanti ambientali. Inoltre, il nostro gruppo e gli altri hanno notato che gli enzimi commerciali possono essere un'altra fonte di contaminazione acidi nucleici batterici in campioni a basso input 6,18. L'utilizzo di uno spazio di lavoro pulito (ad esempio, la PCR cappuccio, armadio biosicurezza) e controlli negativi (ad esempio, acqua o buffer) aiuterà ad alleviare e tenere traccia di contaminazione, rispettivamente. Per i campioni con <100 pg di RNA totale, solo poli (rA) RNA carrier, non rRNA, dovrebbe essere esaurita per garantire risultati di sequenziamento di alta qualità, limitando la perdita di materiale. per moltocampioni di ingresso bassa, metodi cDNA-amplificazione può essere più adatto 19, anche se poly (rA) vettore dovrebbe essere rimosso prima della sintesi del DNA.
L'esaurimento di accoglienza rRNA arricchisce di contenuti virali nelle biblioteche di sequenziamento ed è applicabile alle collezioni di campioni diversi, tra cui siero o plasma, e diversi tipi di tessuti da roditori e primati non umani 5,6. Negli organismi non umani, legge l'allineamento di 28S rRNA è rimasto dopo l'esaurimento, suggerendo 28S rRNA è meno conservato tra gli esseri umani e le altre specie 6,20. Quando si utilizza questo metodo con isolati non-umani, può essere necessario integrare con oligonucleotidi di DNA complementare alle divergenti sequenze rRNA della specifica 3,21 host.
Dal momento che il protocollo è imparziale, virale legge può rappresentare solo una piccola frazione del contenuto totale biblioteca. Anche se rRNA è la specie più abbondante di RNA ospitante e solo una piccola percentuale di rRNA legge (0, 1%) si trovano dopo l'esaurimento selettiva, tutti gli altri RNA host (ad esempio, mRNA) rimarrà dopo l'esaurimento e può spiegare molti sequenziamento legge dal campione. Pertanto "sovracampionamento" (vale a dire, oversequencing) singole biblioteche è necessario al fine di avere una copertura sufficiente per le chiamate di assemblaggio e variante virale. Per i nostri studi, si cerca di sequenza ~ 20 milioni letture per campione di avere abbastanza profondità per l'analisi dei virus e varianti genomiche associate così come il contenuto metagenomica 2,5. Per gli studi di scoperta metagenomiche e patogeni, è importante notare che DNA contaminante host viene rimosso dalla DNasi digestione. Pertanto virus e altri agenti patogeni che contengono genomi di DNA possono essere persi durante il processo, tuttavia intermedi di RNA possono ancora essere sequenziato.
Divulgazioni
The authors have no competing financial interests.
Riconoscimenti
This work has been funded in part with Federal funds from the National Institutes of Health, Office of Director, Innovator (No.: DP2OD06514) (PCS) and from the National Institute of Allergy and Infectious Diseases, National Institutes of Health, Department of Health and Human Services, under Contracts (No:HHSN272200900018C, HHSN272200900049C and U19AI110818).
Materiali
| Name | Company | Catalog Number | Comments |
| 96-well PCR Plates | VWR | 47743-953 | |
| Strips of Eight Caps | VWR | 47745-512 | |
| Nuclease-free water | Ambion | AM9937 | 50 ml bottle |
| TURBO DNase | Ambion | AM2238 | post RNA extraction step, 2 U/µl, buffer included |
| PCR cycler | any PCR cyclers | ||
| Agencourt RNAClean XP SPRI beads | Beckman Coulter Genomics | A63987 | beads for RNA cleanup |
| Real Time qPCR system | any system | ||
| DynaMag-96 Side Skirted Magnet | Invitrogen | 12027 | |
| 70% Ethanol | prepare fresh | ||
| qRT-PCR primers | IDT DNA | see Table 2 | |
| 5 M NaCl | Ambion | AM9760G | |
| 1 M Tris-HCl pH 7.4 | Sigma | T2663-1L | |
| 1 M Tris-HCl pH 7.5 | Invitrogen | 15567-027 | |
| 1 M MgCl2 | Ambion | AM9530G | |
| Linear acrylamide | Ambion | AM9520 | |
| DNA oligos covering entire rRNA region | IDT DNA | see Table 3, order lab-ready at 100 µM | |
| Oligo (dT) | IDT DNA | 40 nt long, desalted | |
| Hybridase Thermostable RNase H | Epicentre | H39100 | |
| RNase-free DNase Kit | Qiagen | 79254 | post selective depletion step |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Random Primers | Invitrogen | 48190-011 | mostly hexamers |
| 10 mM dNTP mix | New England Biolabs | N0447L | |
| SuperScript III Reverse Transcriptase | Invitrogen | 18080-093 | with first-strand buffer, DTT |
| Air Incubator | any air incubator cyclers | ||
| NEBNext Second Strand Synthesis (dNTP-free) Reaction Buffer | New England Biolabs | B6117S | 10x |
| E. coli DNA Ligase | New England Biolabs | M0205L | 10 U/μl |
| E. coli DNA Polymerase I | New England Biolabs | M0209L | 10 U/μl |
| E. coli RNase H | New England Biolabs | M0297L | 2 U/μl |
| 0.5 M EDTA | Ambion | AM9261 | |
| Agencourt AMPure XP SPRI beads | Beckman Coulter Genomics | A63881 | beads for DNA cleanup |
| Elution Buffer | Qiagen | 10 mM Tris HCl, pH 8.5 | |
| Quant-iT dsDNA HS Assay Kit | Invitrogen | Q32854 | |
| Qubit fluorometer | Invitrogen | Q32857 | |
| Nextera XT DNA Sample Prep Kit | Illumina | FC-131-1096 | |
| Nextera XT DNA Index Kit | Illumina | FC-131-1001 | |
| Tapestation 2200 | Agilent | G2965AA | |
| High Sensitivity D1000 reagents | Agilent | 5067-5585 | |
| High Sensitivity D1000 ScreenTape | Agilent | 5067-5584 | |
| BioAnalyzer 2100 | Agilent | G2939AA | |
| High Sensitivity DNA reagents | Agilent | 5067-4626 | |
| Library Quantification Complete kit (Universal) | Kapa Biosystems | KK4824 | alternative to tapestation, bioanalyzer for library quantification |
Riferimenti
- Stremlau, M. H., et al. Discovery of novel rhabdoviruses in the blood of healthy individuals from West Africa. PLoS Negl Trop Dis. 9, e0003631(2015).
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345, 1369-1372 (2014).
- Morlan, J. D., Qu, K., Sinicropi, D. V. Selective depletion of rRNA enables whole transcriptome profiling of archival fixed tissue. PLoS One. 7, e42882(2012).
- Park, D. J., et al. Ebola Virus Epidemiology, Transmission, and Evolution during Seven Months in Sierra Leone. Cell. 161, 1516-1526 (2015).
- Andersen, K. G., et al. Clinical Sequencing Uncovers Origins and Evolution of Lassa Virus. Cell. 162, 738-750 (2015).
- Matranga, C. B., et al. Enhanced methods for unbiased deep sequencing of Lassa and Ebola RNA viruses from clinical and biological samples. Genome Biol. 15, 519(2014).
- Tang, F., et al. RNA-Seq analysis to capture the transcriptome landscape of a single cell. Nat Protoc. 5, 516-535 (2010).
- Jiang, L., et al. Synthetic spike-in standards for RNA-seq experiments. Genome Res. 21, 1543-1551 (2011).
- Agilennt Technologies. , Available from: http://www.agilent.com/cs/library/usermanuals/Public/G2946-90004_Vespucci_UG_eBook_(NoSecPack).pdf. (2015).
- Kapa Biosystems. , Available from: https://www.kapabiosystems.com/product-applications/products/next-generation-sequencing-2/library-quantification/ (2015).
- Illumina Technologies. , Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/system_documentation/miseq/preparing-libraries-for-sequencing-on-miseq-15039740-d.pdf. (2015).
- Kircher, M., Sawyer, S., Meyer, M. Double indexing overcomes inaccuracies in multiplex sequencing on the Illumina platform. Nucleic Acids Res. 40, 3(2012).
- Andrews, S. Babraham Bioinformatics. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Trombley, A. R., et al. Comprehensive panel of real-time TaqMan polymerase chain reaction assays for detection and absolute quantification of filoviruses, arenaviruses, and New World hantaviruses. Am J Trop Med Hyg. 82, 954-960 (2010).
- Hu, Y., et al. Serial high-resolution analysis of blood virome and host cytokines expression profile of a patient with fatal H7N9 infection by massively parallel RNA sequencing. Clin Microbiol Infect. 21, e1-4 713(2015).
- Simon-Loriere, E., et al. Distinct lineages of Ebola virus in Guinea during the 2014 West African epidemic. Nature. 524, 102-104 (2015).
- Folarin, O. A., Happi, A. N., Happi, C. T. Empowering African genomics for infectious disease control. Genome Biol. 15, 515(2014).
- Blainey, P. C., Quake, S. R. Digital MDA for enumeration of total nucleic acid contamination. Nucleic Acids Res. 39, 19(2011).
- Malboeuf, C. M., et al. Complete viral RNA genome sequencing of ultra-low copy samples by sequence-independent amplification. Nucleic Acids Res. 41, 13(2013).
- Gonzalez, I. L., Sylvester, J. E., Smith, T. F., Stambolian, D., Schmickel, R. D. Ribosomal RNA gene sequences and hominoid phylogeny. Mol Biol Evol. 7, 203-219 (1990).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nat Methods. 10, 623-629 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon