Method Article
臨床サンプルからのRNAウイルスの公平なディープシーケンシング
要約
This protocol describes a rapid and broadly applicable method for unbiased RNA-sequencing of viral samples from human clinical isolates.
要約
ここでは、 デノボアセンブリおよび臨床的および生物学的供給源から収集されたウイルスゲノムのホスト内のバリアントの呼び出しを可能にする次世代のRNAの塩基配列決定プロトコルの概要を説明します。この方法は、公平かつ普遍的です。それは、cDNA合成のためのランダムプライマーを使用し、ウイルス配列のコンテンツの予備知識を必要としません。含むポリ(RA)キャリアとリボソームRNA - - ウイルスRNAサンプルからライブラリー構築する前に、選択のRNase Hベースの消化は、不要なRNAを枯渇させるために使用されます。選択的枯渇は、データ品質とウイルスRNAシーケンシングライブラリを読み込んでユニークな数の両方を向上させます。それは全体のライブラリー構築時間を低減するようにまた、トランスポザーゼ系」tagmentation」ステップは、プロトコルで使用されています。プロトコルは、血液および組織の両方からの600以上のラッサ熱とエボラウイルスサンプル-を含むコレクションの急速な深いシーケンシングは、隔離、および他の微生物ゲノム研究に広く適用可能である可能となりました。
概要
臨床情報源からのウイルスの次世代シーケンシングは、送信および感染症の疫学、ならびに診断ヘルプサポート小説、ワクチンや治療の開発に知らせることができます。ランダムプライマーを用いてcDNA合成は、検出およびアセンブリ発散からゲノムの共感染あるいは新規ウイルス1,2を可能にしました。他の公正な方法と同様に、不要な汚染物質は、多くのシーケンシングを読み取って、負の配列決定の結果に影響を与える占めます。ホストおよびポリ(RA)キャリアRNAは、多くの既存のウイルスサンプルのコレクションに存在する汚染物質です。
プロトコルは公平全RNA-seqのに基づいて、深いシーケンシングRNAウイルスのゲノムの効率的かつ費用対効果の高い方法を説明します。この方法は、不要なホストリボソームとキャリアRNAを除去するためのRNase H選択的枯渇のステップ3を利用しています。選択的枯渇は、ウイルスコンテンツ( 図1)のために豊かにし、配列決定データの全体的な品質を向上させます臨床サンプルから( 図2)。また、tagmentationは、著しくライブラリー構築時間を低減するようなプロトコルに適用されます。これらの方法は、迅速にエボラおよびラッサウイルスゲノム2,4,5の大規模なデータセットを生成するために使用されており、RNAウイルスの広い範囲を研究するために使用することができます。最後に、このアプローチは、ヒトのサンプルに限定されるものではありません。選択的な枯渇の有用性は、ラッサ感染げっ歯類および非ヒト霊長類疾患モデル5,6から採取した組織サンプルで実証されました。

図1.全RNAのコンテンツは、コンテンツ全体(RNA入力)を開始しています。選択的枯渇を使用したラッサウイルス、コンテンツの充実を反映し、ユニークなラッサウイルス(LASV)の濃縮は、9つの異なる臨床分離株からのrRNA枯渇時(ライブラリコンテンツ)を読み込みます。この図は、6から変更されています。 この図の拡大版をご覧になるにはこちらをクリックしてください。
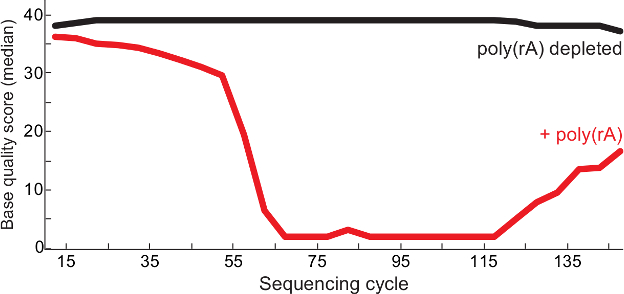
図2.品質向上シングキャリアRNA枯渇。ポリシークエンシングサイクル(RA)あたりの中央値ベースの資質は、ラッサウイルスライブラリ(赤)とQCレポート13からの制御(黒ライブラリに観察されなかったキャリアを、)-contaminated 後 。両方が1を読み、ペアエンドの2を読んで、ライブラリのBAMファイルにマージされ、品質スコアは、各ベースで示されている読み込みます。この図は、6から変更されている。 この図の拡大版をご覧になるにはこちらをクリックしてください。
抽出から直接ライブラリのウイルスRNA-seqのプロトコルの詳細建設RNAは、臨床的および生物学的サンプルから収集します。個人の安全を確保するために、全てのウイルスの血清、血漿および組織サンプルは、RNA抽出前に適切な緩衝液で不活性化されるべきです。いくつかの不活性化および抽出キットにおいて、キャリアポリ(RA)RNAが含まれています。これは初期のRNase H選択的枯渇ステップの間に削除されます。完全な回復に基づいて、キャリアRNAの予想濃度は100 ngの/μlです。プロトコルでは、110 ngの/μlのオリゴdT RNA(1.1倍のキャリア濃度)を枯渇するために使用されます。ポリ(RA)担体が試料中に存在しない場合は、オリゴ(dT)が枯渇する前に添加されるべきではありません。
以下のプロトコルは、(250μlの容量まで)PCRプレート形式で24の反応のために設計されています。このプロトコルの以前のバージョンはMatranga らに報告された。6。
プロトコル
倫理文:ラッサ熱患者はチューレーン大学、ハーバード大学、ブロード研究所、Irrua専門教育病院(ISTH)、ケネマ政府病院(KGH)、健康、イバダンのオヨ州省でのヒト被験者委員会によって承認されたプロトコルを使用して、この研究のために募集されました、ナイジェリアと健康のシエラレオネ省。すべての患者は、彼らが研究に参加することを決めたかどうか、治療の同様の規格で処理し、薬物リバビリン提供されました。ラッサ熱(LF)の患者について、リバビリンによる治療は、現在、推奨ガイドラインに従い、一般的に、すぐにLFが強く疑われたとして提供されていました。
エボラウイルス病(EVD)に深刻な流行に、患者は、当社の標準的なプロトコルを使用して同意することができませんでした。 EVD患者からの臨床過剰サンプルの代わりに使用することは評価され、シエラレオネで、ハーバード大学の制度審査委員会によって承認されました。オフィヒト対象の使用に関するシエラレオネ倫理と科学審査委員会、保健衛生のシエラレオネ省、およびハーバード大学委員会のCEは、シーケンスに同意の放棄を付与しているし、患者と接触したサンプルから得られた公的に利用可能なウイルス配列を作りますシエラレオネエボラ流行中に収集されました。これらの機関はまた、流行の応答の間にケアを受けてすべての疑いのあるEVDの患者から採取匿名化されたサンプルについての臨床および疫学データの使用を認めました。シエラレオネ保健省と衛生にも流行サンプルのゲノム研究のためのシエラレオネからのブロード研究所、ハーバード大学への非感染性、非生物学的サンプルの出荷を承認しました。
サンプルRNAの1 DNアーゼ処理(最大55μlを抽出した全RNAを、〜4時間)
- 表1に記載されているように安全キャビネットに氷上で96ウェルPCRプレート中のDNase反応を設定します、1.1(総容量、70μL/ウェル)をステップ。注:マスターミックスを調製することができます。
- やさしく、徹底的にボルテックスは、その後、1分間室温で280×gで遠心します。
- 37℃で30分間インキュベートします。
- RNA固相可逆的固定化(SPRI)ビーズを使用してクリーンアップ。
- 暖かいRNAビーズを30分間室温に。
- 静かに落ち着いた可能性のある磁性粒子を再懸濁し、RNAビーズボトルを振ります。 DNase処理RNA(70マイクロリットル)へのRNAビーズの1.8倍の容量(126μl)を追加し、ピペットで10回を混合し、(まあ、196μlの総容量)を室温で5分間インキュベートします。
- 磁気ステーションに混合物を置きます。 ( - 10分5)をクリアするためのソリューションを待ちます。
- ピペットおよび廃棄によってステーション上ながらクリアソリューションを削除します。ステーションの間に、70%エタノールでペレットを被覆することにより、ビーズを洗浄し、1分間インキュベートします。ピペットでエタノールを除去し、廃棄します。 2回の洗浄の合計のために繰り返します。
注:正確には70%新たに調製されたらを使用します<70%エタノールは、サンプル7の損失を引き起こす可能性があり、一方、より高い割合は、より小さなサイズの分子の非効率的な洗浄になりますようhanolは、非常に重要です。 - ステーションにプレートを維持し、空気乾燥にオープンにしてください。注:ビーズが割れ始めるまでビーズが完全に乾燥することを可能にしてください。
- RNAを溶出するためにプレートにヌクレアーゼフリー水55μlを添加します。徹底的にピペッティングしてビーズと水を混合するステーションからプレートを取り外します。注:別の方法として、全RNAを濃縮するために、少ない水(≤10μl)を使用します。
- 背面ステーションにプレートを置きます。解決策は、長期保存のための新しいスクリューキャップチューブにピペットで転送するためにクリアするまで待ちます(-80°C)。枯渇のための新しい96ウェルPCRプレートに5μlのRNAを置きます(ステップ2.4)。
- オプション:保存し、rRNAの定量RT-PCRのために19μlの水(1時20分)に1μlのを希釈( 例えば 、18S、28S rRNAの)( 表2)およびウイルスマーカー5
ウイルスRNAサンプルからリボソームとキャリアRNAの2選択的枯渇(〜4時間)
- 表1に記載されているように直鎖状アクリルアミドキャリアと5倍ハイブリダイゼーションおよび10倍のRNase Hの反応緩衝液、およびヌクレアーゼフリー水を作ります。
- 表1に記載したように、96ウェルPCRプレート中、氷上でのrRNA枯渇オリゴ( 表3)を用いてRNAを合成し、オリゴ(dT)でハイブリダイゼーション反応をセットアップします。
注:マスターミックスを調製することができます。ユニークな合成RNA(ERCCs 8)の50フェムトグラム(FG)は、ウイルスの配列決定プロセスや潜在的な指標読み交差汚染の両方を追跡するために追加することができます。- やさしく、徹底的にボルテックスは、その後、1分間室温で280×gで遠心します。
毎秒-0.1℃で45℃までゆっくりランプ、2分間95℃でインキュベートします。 45℃でサーモサイクラーを一時停止します。
- やさしく、徹底的にボルテックスは、その後、1分間室温で280×gで遠心します。
- 説明するように氷上でのRNase H反応ミックスを設定します表1のdは、次に2分間45℃で予熱します。注:マスターミックスを調製することができます。
- 45℃でサーモサイクラー中でプレートを維持しながらプレートにハイブリダイゼーション反応に予備加熱されたRNアーゼHミックスを加えます。
- 8回 - 穏やかにピペッティング6でよく混ぜます。さらに30分間45℃でインキュベートします。氷の上に置きます。
- 表1に記載したように氷上でのDNase反応ミックスを設定注意:マスターミックスを調製することができます。
- 、プレート内のRNase H反応に追加やさしく、徹底的にボルテックスし、1分間室温で280×gで遠心します。 37℃で30分間インキュベートします。
- 5μlの0.5 M EDTAを添加することにより、DNアーゼ反応を停止します。やさしく、徹底的にボルテックスは、その後、1分間室温で280×gで遠心します。
- 1.8倍の容量(144μl)をビーズを用いてRNAビーズを使用してクリーンアップ(ステップ1.3を参照してください)。ヌクレアーゼフリー水11μlの溶出。注:安全な低温貯蔵のために、ストアはRNを枯渇しました-80°C O / Nサンプル。
3. cDNA合成(〜6時間)
- ミックスのrRNA / 96ウェルPCRプレート中の氷上でランダムプライマーとキャリア枯渇RNA、表1に記載されているように、渦やさしく、徹底的に、その後1分間室温で280×gで遠心します。
- サーモサイクラー中で10分間70℃で混合物を加熱します。 5分 - すぐに熱変性した後、1氷上でRNAを置きます。 RNAは、従来の第一鎖の反応により長い5分間(でも、氷上で)放置することはできません。
- 表1に記載したように氷上で第一鎖合成反応ミックスを設定します。
注:マスターミックスを調製することができます。- その後、1分間室温で280×gで遠心し、RNA /プレート内のランダムプライマーミックス、やさしく、徹底的にボルテックスに追加します。 10分間25°C - 22でインキュベートします。
- 60分間空気インキュベーター中、55℃でインキュベートします。反応を停止し、氷上でプレートを置きます。しませんE:空気インキュベーターの使用は、プライマーのアニーリングおよび第一鎖が細長くし始める時の第一鎖反応の漸進的な温暖化を作成することをお勧めします。
- 表1に記載したように氷上で第二鎖合成反応ミックスを設定します。
注:マスターミックスを調製することができます。- 、プレート内の第一鎖合成反応に追加やさしく、徹底的にボルテックスし、1分間室温で280×gで遠心します。 16°C(25°Cで蓋を保つ)で2時間インキュベートします。温度が16°Cを超えないようにしてください。
- 次いで1分間室温で280×gで遠心分離し、静かによく混ぜ、0.5 M EDTAを5μlを加えて反応を不活性化、氷上にプレートを置きます。
- ビーズの1.8倍の容量(153μl)を用いてDNAビーズ(プロトコルのためのステップ1.3を参照)でクリーンアップ。溶出緩衝液(EB)の9μlの溶出。定量化のための1μLを保存します。 subsequeのcDNA 1ngのを使用しますNTの手順を実行します。 cDNA濃度は、検出tagmentationのcDNAを4μlのを使用することが低すぎる場合(ステップ4.1参照)。
- 長期保存のための安全な冷蔵、C°4°CO / Nまたは-20ストア二本鎖cDNAのため。
4.ライブラリの準備 - DNAライブラリーの構築(〜4時間)
- cDNAの転送4μlを96ウェルプレートに、必要な場合は、2番目の試みのために残りのcDNAを保存します。
- 表1に記載されているように氷上でtagmentation反応を設定します。
注:マスターミックスを調製することができます。背景と全体的なコストを削減するために、tagmentation反応の総容量は、20〜10マイクロリットルから減少されます。 cDNAを制限する要因であるように、反応に用いられるATM( すなわち、transposome)の量はまた、組み込み部位の数を減少させるために減少されます。- 1分間(室温で)280×gで静かに、徹底的に、プレートにcDNAに渦をtagmentationミックスを追加し、遠心分離機。5分間55℃でインキュベートし、10℃で保持します。
- いったん10℃で、すぐに反応を終了するために中和するTagmentバッファー(NT)の2.5μlを添加します。その後、1分間(室温で)280×gで遠心し、上下にピペッティングにより混和します。
- 室温で5分間インキュベートします。
- 表1に記載されているように氷上でPCR増幅反応を設定します。
- やさしく、徹底的にボルテックスは、その後、1分間室温で280×gで遠心します。
- 表1に記載の条件を用いて、サーマルサイクラーにPCRを行います。
注:PCRの12サイクルはtagmented cDNAの1 ngのために提案されています。しかし、ウイルスの臨床サンプルは、多くの場合、cDNAの検出不可能な量を持っています。 cDNAを(<1 ngの)低量のために、配列決定のために十分なライブラリーを作成するために、PCRの18サイクルまで使用しています。
- ライブラリの準備 - クリーンアップおよび配列決定のためのプール
- EB 50μlにサンプルを起動します。
- とクリーンアップ0.6x量(30μl)をビーズを用いたDNAビーズ(プロトコルのためのステップ1.3を参照してください)。 15μlのEBで溶出します。
- 領域分析からプライマーダイマー(〜120 bp)を除く、バイオアナライザソフトウェア9を使用して、領域分析(150〜1,000 bp)を行うことにより、ライブラリーの濃度( 図3)を決定します 。注:また、定量PCRは、ライブラリ10を定量するために使用することができます。
- 1 nMもしくはそれ以上の最低のモル濃度でプールライブラリ。ライブラリが1nm未満の場合、これらのライブラリーからの配列情報を取得するために、プール(他のライブラリの〜1倍容量)に、ライブラリの少量を加えます。
- 上記のように0.7倍のDNAビーズとクリーンアッププール(ステップ2を参照)。 15μlのEBで溶出します。注:ビーズのボリュームプールの最終容積に依存するであろう。
- プール9を分析します。領域分析(150〜1,000 bp)の9を行うことにより、モル濃度を決定します。注:また、定量PCRは、ライブラリープール10を定量するために使用することができます</ SUP>。
- デュアルバーコード11を読み取ると10pMの101塩基対を生成するためのライブラリ濃度のロードシーケンサは、ペアエンドを読み込みます。
4代表的なエボラウイルス(EBOV)ライブラリのエボラウイルス臨床サンプル。ゲル画像から構築図3.ライブラリ 。ライブラリとプライマーダイマーの領域が示されている。 この図の拡大版をご覧になるにはこちらをクリックしてください。
結果
記載されているプロトコルは、固有のウイルスのコンテンツを充実しつつ、高品質なシーケンシングの発生は、低入力ウイルスRNAサンプルから読み取ることができます。 図1に示すように 、プロトコルは、一意ラッサウイルス含有量が、少なくとも18S rRNAの少なくとも一つ万枚(約100 pgのトータルRNA)を用いて(非枯渇した対照と比較して)すべての試料の5倍濃縮されました。同様に、シークエンシングの成功はまた、与えられた試料中のウイルス量と相関していました。ウイルス量の代用〜ほとんどの場合、完全なアセンブリを作成した1000以上のウイルスゲノムコピーを含有サンプルとして定量RT-PCRを使用して(データは示さず)。また、ポリデプレッション(RA)キャリアクリーナー製剤が得られる、ライブラリー中のAとTのホモポリマー配列を低減し、より高品質の配列決定を確保する( 図2)を読み出します。低入力から最終的なライブラリーは、ウイルスの臨床サンプルは、多くの場合、150〜1,000塩基対から広い断片長を有します( 図3)。
配列決定の後、プール12内のライブラリ間のサンプルの誤認やクロストークを低減するために、唯一のインデックスが25(Q25)のベース品質スコアで読み取り、ゼロ不一致が分離プロセス中に保たれていることを確認します。ウイルスゲノムは、発散ウイルス2,4-6のための具体的なバイオインフォマティクスパイプラインを使用して組み立てられています。これらのツールは、https://github.com/broadinstitute/viral-ngsで、または商用クラウドプラットフォーム4を介して使用できます。
| ステップ1.1:DNアーゼ反応 | |
| 試薬 | 反応あたり容量(μL) |
| 10倍のDNaseバッファー | 7 |
| ヌクレアーゼフリー水 | 6 |
| 抽出されたウイルスRNA | 55 |
| DNアーゼ(2 U /&#181;リットル) | 2 |
| 全容積 | 70 |
| ステップ2.1:5×ハイブリダイゼーションバッファー | |
| 試薬 | 1ミリリットルのための容量(μL) |
| 5 M NaClを | 200 |
| 1 Mトリス-HCl(pH7.4) | 500 |
| ヌクレアーゼフリー水 | 300 |
| 全容積 | 千 |
| ステップ2.1:10倍のRNase H反応バッファー | |
| 試薬 | 1ミリリットルのための容量(μL) |
| 5 M NaClを | 200 |
| 1 Mトリス-HCl(pH7.5) | 500 |
| 1 MのMgCl 2 | 200 |
| ヌクレアーゼフリー水 | 500 |
| 全容積 | 千 |
| ステップ2.1:水のwi番目の直鎖状アクリルアミド | |
| 試薬 | 1ミリリットルバッファの容量(μL) |
| ヌクレアーゼフリー水 | 992 |
| 直鎖状アクリルアミド(5 mg / mlで) | 8 |
| 全容積 | 千 |
| ステップ2.2:選択的枯渇するためのハイブリダイゼーション反応 | |
| 試薬 | 反応あたり容量(μL) |
| 5×ハイブリダイゼーションバッファー | 2 |
| rRNAを枯渇オリゴミックス(100μM) | 1.22 |
| オリゴ(d)のT(550 NG /μL) | 1 |
| DNアーゼ処理した総RNA | 5まで |
| スパイクにRNA(これはオプションです) | 0.5 |
| (直鎖状アクリルアミドとの)水 | 10総に持ち出します |
| 総volu私に | 10 |
| ステップ2.3:選択的枯渇のためのRNase H反応 | |
| 試薬 | 反応あたり容量(μL) |
| 10倍のRNase H反応緩衝液 | 2 |
| (直鎖状アクリルアミドとの)水 | 5 |
| 耐熱性リボヌクレアーゼH(5 U /μl)を | 3 |
| 全容積 | 10 |
| ステップ2.4:DNアーゼ反応後の選択的枯渇 | |
| 試薬 | 反応あたり容量(μL) |
| 10倍のDNaseバッファー | 7.5 |
| (直鎖状アクリルアミドとの)水 | 44.5 |
| RNaseインヒビター(20 U /μl)を | 1 |
| RNaseフリーのDNase I(2.72 U /μl)を | 2 |
| (RNアーゼHの反応を含む)の合計量 | 75 |
| ステップ3.1:cDNA合成、ランダムプライマーハイブリダイゼーション | |
| 試薬 | 反応あたり容量(μL) |
| rRNA /キャリアが枯渇したRNA | 10 |
| 3μgのランダムプライマー | 1 |
| 全容積 | 11 |
| 第一鎖cDNA合成反応:3.2手順 | |
| 試薬 | 容量(μL) |
| 5Xファーストストランド反応緩衝液 | 4 |
| 0.1 M DTT | 2 |
| 10mMのdNTPミックス | 1 |
| RNaseインヒビター(20 U /μl)を | 1 |
| 逆転写酵素(最後の追加) | 1 |
| (上記RNAとの)総容量 | 20 |
| ステップ3.3:第二鎖cDNA合成反応 | |
| 試薬 | 容量(μL) |
| RNaseフリー水 | 43 |
| 10倍の第二鎖反応緩衝液 | 8 |
| 10mMのdNTPミックス | 3 |
| 大腸菌 DNAリガーゼ(10 U /μl)を | 1 |
| 大腸菌 DNAポリメラーゼI(10 U /μl)を | 4 |
| 大腸菌のRNase H(2 U /μl)を | 1 |
| (第1鎖反応を含む)全体積 | 80 |
| ステップ4.2:Tagmentation反応 | |
| 試薬 | 容量(μL) |
| アンプリコンTagmentミックス(ATM) | 1 |
| Tagment DNAバッファー(TD) | 5 |
| (cDNAでの)総体積 | 10 |
| ステップ4.3:ライブラリーのPCR反応 | |
| 試薬 | 容量(μL) |
| PCRマスターミックス(NPM) | 7.5 |
| インデックス1プライマー(I7) | 2.5 |
| インデックス2プライマー(I5) | 2.5 |
| (tagmented cDNAでの)総体積 | 25 |
| ステップ4.3.2:ライブラリPCR条件 | |
| 72℃、3分 | |
| 95℃、30秒 | |
| 95℃で18サイクル、10秒、55℃で30秒、30秒まで72℃で | |
| 72℃、5分 | |
| 永遠に10°C、 | |
表1:反応セットアップおよびバッファステップバイステップの全てのバッファとの反応混合物の内容を持つテーブル。
表2:定量RT-PCRプライマー配列ホスト(18SリボソームRNA)およびウイルス(エボラおよびラッサ)含量を測定するために使用したプライマー。 「KGHは「エボラプライマーは2を試験したシエラレオネ、中ケネマ政府病院です。 「Kuleshは'プライマーセット14設計研究者です。
表3:リボソームRNA(rRNAの)枯渇オリゴ選択的枯渇のステップ6のためのヒトのrRNAに相補的な195の50ヌクレオチド長の配列が、 このファイルをダウンロードするにはこちらをクリックしてください。
| オリゴ名 | 配列(5 'から3') |
| エボラKGH FW | GTCGTTCCAACAATCGAGCG |
| エボラKGH RV | CGTCCCGTAGCTTTRGCCAT |
| エボラKULESH FW | TCTGACATGGATTACCACAAGATC |
| エボラKULESH RV | GGATGACTCTTTGCCGAACAATC |
| ラッサSL FW | GTA AGC CCA GCD GYAのAABのCC |
| ラッサSL RV | AAG CCA CAG AAA RCT GGS AGC A |
| 18S rRNAのFW | TCCTTTAACGAGGATCCATTGG |
| 18S rRNAのRV | CGAGCTTTTTAACTGCAGCAACT |
ディスカッション
概説的なアプローチは、堅牢、普遍的、迅速なシーケンシングを可能にし、2014年流行の2,4の間にエボラウイルスの配列を決定するために使用されました。 tagmentationライブラリー構成の選択的枯渇及びcDNA合成を結合することにより、全体の処理時間は、前のアダプターライゲーション方法から1~2日後に減少しました。最近では、このプロトコルは大成功15,16との国際共同研究者らによって採用されたと地元ゲノミクスベースの調査研究および診断17をサポートするために西アフリカのラボに展開されます。
ここで説明するプロトコルは、ウイルスRNA-seqのライブラリのcDNAを準備するために、ランダムプライマーを使用しています。前回のウイルスRNA-seqのアプローチとは異なり、それは特定のウイルスまたはクレードのための配列データや精巧で時間のかかるプライマー設計の事前知識を必要としません。この方法は、任意のウイルスRNA試料に適用することができます。例えば、エボラウイルスの両方からコンテンツを生成するために使用されましたそして、ラッササンプル6。プロトコルは、ホスト・トランスクリプトーム、メタゲノムおよび病原体検出の配列決定プロジェクト1のために使用することができます。
プロトコルの重要なステップは、RNアーゼH消化、高スループット、ウイルスのサンプルから不要なキャリアと宿主RNAを除去するための低コストの方法を対象としています。プロトコルの選択的枯渇のステップは、多くのコンポーネントを使用し、スキルと精度を必要とします。余分な時間とケアは、初期設定時に注意すべきです。
ほとんどの臨床血清および血漿サンプルは、多くの場合、非常に少ない核酸物質を有するので、汚染および試料の損失が一般的です。このプロトコルを使用する場合、これらの問題を回避するために、特別な注意が払われるべきです。まず、RNAが分解に対して非常に敏感です。したがって、すべてのエリアは清潔でヌクレアーゼの自由であるべきです。第二に、このプロトコルで使用するのに適したサンプルを識別するために、宿主RNAとウイルスの両方のためのqRT-PCRアッセイは、定量5,6のために使用されるべきです。入力を比較すると、少なくとも100 pgのトータルRNAとウイルスの千のコピーを含まれるサンプルと相関プロトコル、シークエンシングの成功(フルウイルス構築のための十分なデータがすなわち、発生)からの配列決定の結果に相当します。第三に、核酸の環境源への曝露を避けるべきです。ここで概説プロトコルは、安全上の注意事項は、環境汚染物質を制限するための安全キャビネットで行われます。また、当社グループなどが市販酵素は、低入力サンプル6,18中の細菌の核酸を汚染する別の源になる可能性があることに気づきました。クリーンな作業領域( 例えば 、PCRフード、安全キャビネット)および陰性対照( 例えば 、水または緩衝液)の使用は、それぞれ、軽減と汚染を追跡します。 <全RNAの100 pgの、唯一のポリ(RA)キャリアRNA、ないのrRNAとサンプルについては、材料の損失を制限しながら、高品質の配列決定結果を確実にするために枯渇されるべきです。以下のために非常にポリ(RA)キャリアはcDNA合成の前に除去しなければならないが、低入力サンプルは、cDNAの増幅方法は、19より適切であり得ます。
宿主rRNAの枯渇は、配列ライブラリー中のウイルス含有量を富化し、血清又は血漿、及びげっ歯類および非ヒト霊長類5,6からの組織の複数のタイプを含む異なるサンプルのコレクションに適用可能です。ヒト以外の生物では、28S rRNAのに整列させることは28S rRNAのは、人間と他の種6,20間の少ない保存されて示唆し、枯渇した後に残った読み取り。非ヒト分離株で、この方法を使用する場合、特定のホスト3,21の発散rRNA配列に相補的なDNAオリゴと補完する必要があるかもしれません。
プロトコルは公平であるので、ウイルスは、全ライブラリの内容のごく一部を表すことができる読み取ります。 rRNAが宿主RNAの最も豊富な種であるとrRNAのわずかな割合は、(読み込みが0;)1%が選択的枯渇後に発見され、他のすべての宿主RNA( 例えば 、mRNA)が枯渇した後に残る、多くのシーケンシングは、サンプルからの読み取りを説明することができます。したがって「オーバーサンプリング」( すなわち 、oversequencing)個々のライブラリは、ウイルスアセンブリおよびバリアントの呼び出しのための十分なカバレッジを持っているために必要とされます。私たちの研究のために、私たちは順番にしよう〜2000万は、ウイルスゲノムおよび関連する変異体の解析のために十分な深さだけでなく、メタゲノムコンテンツ2,5を持っているために、サンプルごとに読み込みます。メタゲノムおよび病原体検出の研究のために、汚染宿主DNAは、DNアーゼ消化によって除去されることに留意することが重要です。したがって、ウイルスおよびDNAゲノムを含む他の病原体は、しかし、RNA中間体がまだ配列決定され得る、プロセスの間に失われることがあります。
開示事項
The authors have no competing financial interests.
謝辞
This work has been funded in part with Federal funds from the National Institutes of Health, Office of Director, Innovator (No.: DP2OD06514) (PCS) and from the National Institute of Allergy and Infectious Diseases, National Institutes of Health, Department of Health and Human Services, under Contracts (No:HHSN272200900018C, HHSN272200900049C and U19AI110818).
資料
| Name | Company | Catalog Number | Comments |
| 96-well PCR Plates | VWR | 47743-953 | |
| Strips of Eight Caps | VWR | 47745-512 | |
| Nuclease-free water | Ambion | AM9937 | 50 ml bottle |
| TURBO DNase | Ambion | AM2238 | post RNA extraction step, 2 U/µl, buffer included |
| PCR cycler | any PCR cyclers | ||
| Agencourt RNAClean XP SPRI beads | Beckman Coulter Genomics | A63987 | beads for RNA cleanup |
| Real Time qPCR system | any system | ||
| DynaMag-96 Side Skirted Magnet | Invitrogen | 12027 | |
| 70% Ethanol | prepare fresh | ||
| qRT-PCR primers | IDT DNA | see Table 2 | |
| 5 M NaCl | Ambion | AM9760G | |
| 1 M Tris-HCl pH 7.4 | Sigma | T2663-1L | |
| 1 M Tris-HCl pH 7.5 | Invitrogen | 15567-027 | |
| 1 M MgCl2 | Ambion | AM9530G | |
| Linear acrylamide | Ambion | AM9520 | |
| DNA oligos covering entire rRNA region | IDT DNA | see Table 3, order lab-ready at 100 µM | |
| Oligo (dT) | IDT DNA | 40 nt long, desalted | |
| Hybridase Thermostable RNase H | Epicentre | H39100 | |
| RNase-free DNase Kit | Qiagen | 79254 | post selective depletion step |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Random Primers | Invitrogen | 48190-011 | mostly hexamers |
| 10 mM dNTP mix | New England Biolabs | N0447L | |
| SuperScript III Reverse Transcriptase | Invitrogen | 18080-093 | with first-strand buffer, DTT |
| Air Incubator | any air incubator cyclers | ||
| NEBNext Second Strand Synthesis (dNTP-free) Reaction Buffer | New England Biolabs | B6117S | 10x |
| E. coli DNA Ligase | New England Biolabs | M0205L | 10 U/μl |
| E. coli DNA Polymerase I | New England Biolabs | M0209L | 10 U/μl |
| E. coli RNase H | New England Biolabs | M0297L | 2 U/μl |
| 0.5 M EDTA | Ambion | AM9261 | |
| Agencourt AMPure XP SPRI beads | Beckman Coulter Genomics | A63881 | beads for DNA cleanup |
| Elution Buffer | Qiagen | 10 mM Tris HCl, pH 8.5 | |
| Quant-iT dsDNA HS Assay Kit | Invitrogen | Q32854 | |
| Qubit fluorometer | Invitrogen | Q32857 | |
| Nextera XT DNA Sample Prep Kit | Illumina | FC-131-1096 | |
| Nextera XT DNA Index Kit | Illumina | FC-131-1001 | |
| Tapestation 2200 | Agilent | G2965AA | |
| High Sensitivity D1000 reagents | Agilent | 5067-5585 | |
| High Sensitivity D1000 ScreenTape | Agilent | 5067-5584 | |
| BioAnalyzer 2100 | Agilent | G2939AA | |
| High Sensitivity DNA reagents | Agilent | 5067-4626 | |
| Library Quantification Complete kit (Universal) | Kapa Biosystems | KK4824 | alternative to tapestation, bioanalyzer for library quantification |
参考文献
- Stremlau, M. H., et al. Discovery of novel rhabdoviruses in the blood of healthy individuals from West Africa. PLoS Negl Trop Dis. 9, e0003631(2015).
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345, 1369-1372 (2014).
- Morlan, J. D., Qu, K., Sinicropi, D. V. Selective depletion of rRNA enables whole transcriptome profiling of archival fixed tissue. PLoS One. 7, e42882(2012).
- Park, D. J., et al. Ebola Virus Epidemiology, Transmission, and Evolution during Seven Months in Sierra Leone. Cell. 161, 1516-1526 (2015).
- Andersen, K. G., et al. Clinical Sequencing Uncovers Origins and Evolution of Lassa Virus. Cell. 162, 738-750 (2015).
- Matranga, C. B., et al. Enhanced methods for unbiased deep sequencing of Lassa and Ebola RNA viruses from clinical and biological samples. Genome Biol. 15, 519(2014).
- Tang, F., et al. RNA-Seq analysis to capture the transcriptome landscape of a single cell. Nat Protoc. 5, 516-535 (2010).
- Jiang, L., et al. Synthetic spike-in standards for RNA-seq experiments. Genome Res. 21, 1543-1551 (2011).
- Agilennt Technologies. , Available from: http://www.agilent.com/cs/library/usermanuals/Public/G2946-90004_Vespucci_UG_eBook_(NoSecPack).pdf. (2015).
- Kapa Biosystems. , Available from: https://www.kapabiosystems.com/product-applications/products/next-generation-sequencing-2/library-quantification/ (2015).
- Illumina Technologies. , Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/system_documentation/miseq/preparing-libraries-for-sequencing-on-miseq-15039740-d.pdf. (2015).
- Kircher, M., Sawyer, S., Meyer, M. Double indexing overcomes inaccuracies in multiplex sequencing on the Illumina platform. Nucleic Acids Res. 40, 3(2012).
- Andrews, S. Babraham Bioinformatics. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Trombley, A. R., et al. Comprehensive panel of real-time TaqMan polymerase chain reaction assays for detection and absolute quantification of filoviruses, arenaviruses, and New World hantaviruses. Am J Trop Med Hyg. 82, 954-960 (2010).
- Hu, Y., et al. Serial high-resolution analysis of blood virome and host cytokines expression profile of a patient with fatal H7N9 infection by massively parallel RNA sequencing. Clin Microbiol Infect. 21, e1-4 713(2015).
- Simon-Loriere, E., et al. Distinct lineages of Ebola virus in Guinea during the 2014 West African epidemic. Nature. 524, 102-104 (2015).
- Folarin, O. A., Happi, A. N., Happi, C. T. Empowering African genomics for infectious disease control. Genome Biol. 15, 515(2014).
- Blainey, P. C., Quake, S. R. Digital MDA for enumeration of total nucleic acid contamination. Nucleic Acids Res. 39, 19(2011).
- Malboeuf, C. M., et al. Complete viral RNA genome sequencing of ultra-low copy samples by sequence-independent amplification. Nucleic Acids Res. 41, 13(2013).
- Gonzalez, I. L., Sylvester, J. E., Smith, T. F., Stambolian, D., Schmickel, R. D. Ribosomal RNA gene sequences and hominoid phylogeny. Mol Biol Evol. 7, 203-219 (1990).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nat Methods. 10, 623-629 (2013).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved