Method Article
临床样品RNA病毒的无偏深度测序
摘要
This protocol describes a rapid and broadly applicable method for unbiased RNA-sequencing of viral samples from human clinical isolates.
摘要
在这里,我们勾勒出下一代RNA测序协议,使从头组件和从临床和生物学来源收集的病毒基因组的帧内主机变种呼叫。该方法是无偏的和普遍的;它采用cDNA合成随机引物,并且需要病毒序列内容的先验知识。图书馆建设之前,选择基于H-RNA酶消化来消耗多余的RNA - 包括聚(RA)载体和核糖体RNA - 从病毒RNA样品。选择性耗尽同时提高数据质量和独特的数目在病毒RNA测序文库的读取。此外,基于转座-'tagmentation'步骤在协议中使用,因为它减少总库施工时间。该协议使快速深度测序超过600拉沙和埃博拉病毒样本,包括来自血液和组织分离,并广泛适用于其它微生物基因组学研究的集合。
引言
从临床来源的病毒的新一代测序可以通知传播和感染的流行病学,以及帮助支持新的诊断,疫苗和治疗的发展。使用随机引物合成cDNA已经允许从发散的,共同的侵染的基因组或甚至新的病毒1,2的检测和组装。与其他方式偏见,有害的污染物,会占用大量的测序读取和测序结果产生负面影响。主机和聚(RA)的载体RNA存在于许多现有的病毒样品收集污染物。
该协议描述了基于偏总RNA-SEQ深度测序RNA病毒基因组的高效率和成本效益的方法。该方法利用一种核糖核酸酶H选择性枯竭步骤3,删除不需要的主机核糖体和载体RNA。选择性耗竭丰富了病毒内容( 图1),提高了测序数据的整体质量( 图2)从临床样品。此外,tagmentation被应用到,因为它显著降低了图书馆的建设时间的协议。这些方法已被用于快速产生埃博拉病毒和拉沙病毒基因组2,4,5的大型数据集,并且可以用于研究广泛的RNA病毒。最后,该方法不限于人类样品;选择性消耗的效用已证明来自拉沙感染啮齿动物和非人类灵长类动物疾病模型5,6-收集组织样品。

图1.总RNA内容反映拉沙病毒含量采用选择性枯竭的富集。从九个不同的临床分离后,开始枯竭rRNA的整体内容(RNA输入)和独特的拉沙病毒(LASV)富集读取(库内容)。这个数字已经从6修改 点击此处查看该图的放大版本。
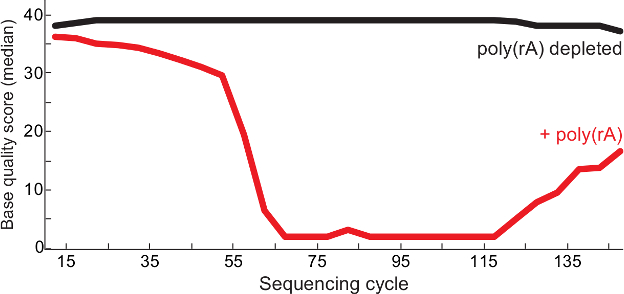
图2.更高质量测序后载体的RNA耗尽。每聚测序循环(RA)中值碱品质-contaminated拉沙病毒库(红色),并从质量控制报告13对照(无载体库观察,黑色)。双方念1,阅读配对末端的2读取合并库中的BAM文件,并在每个基地的质量分数表示。这个数字已经从6修改。 请点击此处查看本图的放大版本。
病毒RNA-SEQ协议的细节建设库直接从提取从临床和生物样品收集的RNA。为了确保人身安全,所有病毒血清,血浆和组织样品应前RNA提取适当的缓冲灭活。在有些失活和提取试剂盒,载体聚(RA)的RNA包括在内;这将在最初的RNA酶H选择性消耗步骤中除去。基于完全恢复,载体RNA的预期浓度为100纳克/微升。在协议中,110纳克/微升寡聚dT的RNA(1.1倍载流子浓度)用于耗竭。如果聚(RA)的载体是不存在于样品中,那么寡(dT)不应之前耗尽加入。
以下协议被设计用于PCR板格式24反应(高达250微升体积)。该协议的早期版本据报道,Matranga 等。6。
研究方案
伦理学声明:拉沙热患者被招募为这项研究使用在杜兰大学,哈佛大学,Broad研究所,Irrua专科教学医院(ISTH),凯内马政府医院(KGH),卫生,伊巴丹的奥约州教育部批准由人类受试者委员会协议,尼日利亚和健康的塞拉利昂部。所有患者的护理的类似标准处理,然后提供的药物利巴韦林,无论它们是否决定参加了这项研究。拉沙热(LF)患者,利巴韦林的治疗遵循了目前建议的准则和普遍尽快LF被强烈怀疑提供。
由于对埃博拉病毒病(EVD)的严重非典疫情,患者无法通过我们的标准协议同意。而是使用从EVD患者的临床标本过多的评价,并在塞拉利昂和哈佛大学通过机构审查委员会批准。该OFFI塞拉利昂道德教育和科学审查委员会,健康和卫生的塞拉利昂部,以及哈佛委员会的人类受试者的使用CE授予同意豁免进行排序,并从病人和接触的样品获得公开可用的病毒序列塞拉利昂爆发埃博拉期间收集。这些机构还授予使用从应对疫情期间接受照顾所有可疑EVD收集患者去确定样本的临床和流行病学资料。健康和卫生的塞拉利昂卫生部还核准了塞拉利昂非感染性,非生物样本的出货量Broad研究所和哈佛大学的爆发样本的基因组学研究。
样品RNA(最多55微升提取总RNA,〜4小时)的DNA酶1.治疗
- 如表1中所述,设置在上在生物安全柜冰的96孔PCR平板的DNA酶反应步骤1.1(总体积70微升/孔)。注:主拌匀即可准备。
- 轻轻涡旋后透,然后在280 xg离心离心在RT 1分钟。
- 孵育在37℃下30分钟。
- 清理利用RNA固相可逆固定(SPRI)珠。
- 温暖的RNA珠至室温30分钟。
- 轻轻摇动RNA珠瓶悬浮可能已经解决任何磁性颗粒。添加的RNA珠DNA酶处理的RNA(70微升)1.8倍量(126微升),用移液管混合10次,(在井196微升总体积)在室温孵育5分钟。
- 将混合磁站。等待溶液来清除(5 - 10分钟)。
- 同时取出吸管,丢弃站上清理解决方案。而站上,通过覆盖沉淀用70%乙醇洗珠并孵育1分钟。用吸管和丢弃除去乙醇。重复总共两次洗涤的。
注意:使用精确的70%新鲜配制等hanol是关键的,因为较高的百分比会导致较小尺寸的分子的低效洗涤,而<70%的乙醇会导致样品7的损耗。 - 保持站上盘和平仓离场风干。注意:一定要允许直到珠开始破解珠完全干燥。
- 添加55微升不含核酸酶的水至板,以洗脱RNA。从站删除板被彻底吹打到珠和水混合。注意:可替换地,以集中的总RNA使用较少的水(≤10微升)。
- 将板回站。等到溶液清除由吸管转移到新的螺旋盖管长期贮存(-80℃)。放置5微升的RNA在新的96孔PCR板耗尽(步骤2.4)。
- 可选:保存并在rRNA基因的定量RT-PCR检测19微升水(1:20)稀释1微升( 例如 ,18S,28S rRNA的)( 表2)和病毒标记物5
从病毒RNA核糖体样品和载体RNA 2.选择性耗竭(〜4小时)
- 使5倍的杂交和10倍核糖核酸酶H反应缓冲剂,和无核酸酶水与线性丙烯酰胺载体,如表1所述。
- 通过如表1中所述的RNA与rRNA的耗尽寡聚( 表3)和寡聚(dT)在冰上在96孔PCR板组合成立杂交反应。
注:主拌匀即可准备。一个独特的合成RNA(ERCCs 8)的50毫微微克(FG)可以添加跟踪这两个病毒测序过程和潜力指数读交叉污染。- 轻轻涡旋后透,然后在280 xg离心离心在RT 1分钟。
在95℃下2分钟,慢速倾斜孵育至45℃,以每秒-0.1℃。暂停在45℃的热循环。
- 轻轻涡旋后透,然后在280 xg离心离心在RT 1分钟。
- 设置核糖核酸酶H反应混合物在冰的描述d在表1中 ,然后预热在45℃下2分钟。注:主拌匀即可准备。
- 预热的RNA酶H混合物添加到杂交反应中板,同时保持在温度循环器的板在45℃。
- 8倍 - 轻柔吹打6拌匀。孵育在45℃另外30分钟。广场上冰。
- 如表1中的描述设置在冰上DNA酶反应混合物注:A主混合物可以制备。
- 添加到板中的RNA酶H反应,涡旋轻轻和彻底,然后在280 xg离心离心在RT 1分钟。孵育在37℃下30分钟。
- 通过加入5微升的0.5M EDTA停止DNA酶反应。轻轻涡旋后透,然后在280 xg离心离心在RT 1分钟。
- 清理利用RNA珠(见步骤1.3),使用1.8倍体积(144微升)珠。洗脱在11微升不含核酸酶的水。注意:为了安全冷库,存储耗尽RN一个样品在-80℃O / N。
3. cDNA合成(〜6小时)
- 混合的rRNA /在96孔PCR平板在冰上随机引物载体贫RNA为表1中所述,旋涡轻轻和彻底,然后在280 xg离心离心在RT 1分钟。
- 加热该混合物至70℃下,在热循环10分钟。热变性后,立即放在冰上1 RNA的 - 5分钟。不允许的RNA静置(甚至在冰上)超过5分钟的第一链反应之前。
- 如表1中所述,设置在冰上第一链合成反应混合物中。
注:主结构可能准备。- 加入RNA /随机引物混合物中板,旋涡轻轻和彻底,然后在280 xg离心离心在RT 1分钟。孵育在22 - 25℃下进行10分钟。
- 在空气培养箱中60分钟孵育在55℃。放置在冰上的板以终止反应。不E:使用空气培养箱时,建议创建其间的引物退火和第一链开始伸长第一链反应的逐步升温。
- 如表1中所述,设置在冰上第二链合成反应混合物中。
注:主结构可能准备。- 添加到板中的第一链合成反应,涡旋轻轻和彻底,然后在280 xg离心离心在RT 1分钟。在16℃(保持盖在25℃)孵育2小时。不要让温度超过16℃。
- 放置在冰上的板,然后通过加入5微升的0.5M EDTA的灭活反应,轻轻调匀,然后在280 xg离心离心在RT 1分钟。
- 清理与DNA的珠子(见步骤1.3协议)使用的珠1.8倍体积(153微升)。洗脱在洗脱缓冲液9微升(EB)。保存1微升进行量化。使用subseque 1纳克的cDNANT步骤。如果cDNA的浓度太低而不能检测,使用4微升的cDNA为tagmentation(参见步骤4.1)。
- 为安全冷库,存储在双链cDNA 4°的CO / N或-20℃下长期贮存。
4.图书馆准备 - DNA文库构建(〜4小时)
- 如果需要传递4μl的cDNA的一个96孔板并保存其余的cDNA第二次尝试。
- 如表1中的描述设置在冰上tagmentation反应。
注:主结构可能准备。以减少背景和总成本,该tagmentation反应的总体积为20〜10微升减少。作为cDNA的是限制因素,自动柜员机( 即,转座子)的反应中所用的量也减少到减少整合位点的数目。- 在280 XG(室温)1分钟轻轻地,彻底地离心机添加tagmentation组合成cDNA中板,旋涡。孵育在55℃进行5分钟,保持在10℃。
- 一旦在10℃下,立即加入2.5微升抵消Tagment缓冲液(NT),以结束该反应。通过吹吸混合和向下,然后在280 xg离心(在RT)离心1分钟。
- 孵育在室温下5分钟。
- 如表1中所述,设置在冰上的PCR扩增反应。
- 轻轻涡旋后透,然后在280 xg离心离心在RT 1分钟。
- 使用表1中描述的条件的热循环进行PCR。
注:PCR的12个周期建议为1纳克tagmented的cDNA;然而,病毒的临床标本往往检测不到的数额cDNA的。对于少量的cDNA(<1纳克)中,使用最多的PCR 18个循环来进行测序创造足够的库。
- 图书馆准备 - 清理并汇集了测序
- 把样品多达50微升与EB。
- 清理与DNA珠(见步骤1.3协议)用0.6倍体积(30微升)珠。洗脱在15微升的EB。
- 通过进行使用生物分析仪软件9,但不包括区域分析引物二聚体(〜120 bp)的区域分析(150至1000碱基对)确定库( 图3)的浓度。注意:可替换地,定量PCR可用于定量库10。
- 池库在1nm或更大的最低摩尔浓度。如果库低于1nm时,添加库的一个小体积池(其他库的〜1倍体积),以捕获从这些库序列信息。
- 与上面所列0.7倍DNA珠清理池(见步骤2)。洗脱在15微升的EB。注意:珠体积将取决于池的最终体积。
- 分析池9。通过进行区域分析(150至1000碱基对)9确定摩尔浓度。注意:可替换地,定量PCR可用于定量文库池10 </ SUP>。
- 晚上10点库集中载荷序产生101 bp的配对末端读取与双条形码读取11。
图3.从图书馆埃博拉病毒的临床标本构成。4代表埃博拉病毒(EBOV)库凝胶图像。图书馆和引物二聚体的区域所示。 请点击此处查看该图的放大版本。
结果
所描述的协议使高品质的测序的产生,同时丰富独特的病毒含量低输入病毒RNA样品读取。正如图1所示,该协议富集独特拉沙病毒含量至少与18S rRNA基因(约100皮克总RNA)中的至少一百万份的五倍所有样品(相对于非耗尽控制)。同样地,测序的成功还与给定样品中病毒的量相关。使用定量RT-PCR,作为病毒的数量的替代,包含〜最多创建的完整组件1000以上病毒基因组拷贝的样品(未示出数据)。此外,聚(RA)载体的耗竭减少了A和T的均聚物序列文库,导致清洁制剂,确保更好的质量测序读取( 图2)。从低输入病毒的临床标本库,最终往往具有广阔的片段长度为150至1000基点( 图3)。
测序后,减少池12内图书馆之间的样品误认和串扰,只指数与基质量分数25(Q25)的读取,并确保零错配在解复用过程中保存。病毒基因组均使用特定的发散病毒2,4-6生物信息学管道装配。这些工具可在https://github.com/broadinstitute/viral-ngs或通过商业云平台4。
| 步骤1.1:DNA酶反应 | |
| 试剂 | 每反应体积(微升) |
| 10X DNA酶缓冲 | 7 |
| 不含核酸酶的水 | 6 |
| 提取病毒RNA | 55 |
| DNA酶(2U /&#181; L) | 2 |
| 总成交量 | 70 |
| 步骤2.1:5X杂交缓冲液 | |
| 试剂 | 为1mL容量(微升) |
| 5 M氯化钠 | 200 |
| 的1M的Tris-HCl(pH 7.4)中 | 500 |
| 不含核酸酶的水 | 300 |
| 总成交量 | 1000 |
| 步骤2.1:10X核糖核酸酶H反应缓冲液 | |
| 试剂 | 为1mL容量(微升) |
| 5 M氯化钠 | 200 |
| 的1M的Tris-HCl(pH7.5)中 | 500 |
| 1M的氯化镁 | 200 |
| 不含核酸酶的水 | 500 |
| 总成交量 | 1000 |
| 步骤2.1:水(W)第i个线性丙烯酰胺 | |
| 试剂 | 1毫升缓冲液体积(微升) |
| 不含核酸酶的水 | 992 |
| 线性丙烯酰胺(5毫克/毫升) | 8 |
| 总成交量 | 1000 |
| 步骤2.2:选择性枯竭杂交反应 | |
| 试剂 | 每反应体积(微升) |
| 5X杂交缓冲液 | 2 |
| rRNA基因耗尽寡聚混合物(100μM) | 1.22 |
| 寡核苷酸(D)T(550纳克/微升) | 1 |
| DNA酶处理的总RNA | 高达5 |
| 穗的RNA(这是可选) | 0.5 |
| 水(与线性丙烯酰胺) | 带来高达10总 |
| 总VOLU我 | 10 |
| 步骤2.3:选择性枯竭核糖核酸酶H反应 | |
| 试剂 | 每反应体积(微升) |
| 10倍的核糖核酸酶H反应缓冲液 | 2 |
| 水(与线性丙烯酰胺) | 五 |
| 热稳定酶H(5U /微升) | 3 |
| 总成交量 | 10 |
| 步骤2.4:DNA酶反应后选择性枯竭 | |
| 试剂 | 每反应体积(微升) |
| 10X DNA酶缓冲 | 7.5 |
| 水(与线性丙烯酰胺) | 44.5 |
| RNA酶抑制剂(20U /微升) | 1 |
| 无RNA酶的DNA酶I(2.72单位/微升) | 2 |
| 总成交量(含核糖核酸酶H反应) | 75 |
| 步骤3.1:cDNA合成,随机引物杂交 | |
| 试剂 | 每反应体积(微升) |
| rRNA基因/载耗尽的RNA | 10 |
| 3微克随机引物 | 1 |
| 总成交量 | 11 |
| 步骤3.2:第一链cDNA合成反应 | |
| 试剂 | 体积(微升) |
| 5X第一链反应缓冲液 | 4 |
| 0.1 M DTT | 2 |
| 10毫米dNTP混合物 | 1 |
| RNA酶抑制剂(20U /微升) | 1 |
| 逆转录(添加最后一个) | 1 |
| 总成交量(含上述RNA) | 20 |
| 步骤3.3:第二链cDNA合成反应 | |
| 试剂 | 体积(微升) |
| 无RNA酶的水 | 43 |
| 10X第二链反应缓冲液 | 8 |
| 10毫米dNTP混合物 | 3 |
| 大肠杆菌 DNA连接酶(10U /微升) | 1 |
| 大肠杆菌 DNA聚合酶I(10U /微升) | 4 |
| 大肠杆菌核糖核酸酶H(2 U /μL) | 1 |
| 总成交量(含第一链反应) | 80 |
| 步骤4.2:Tagmentation反应 | |
| 试剂 | 体积(微升) |
| 扩增子Tagment混合(ATM)的 | 1 |
| Tagment的DNA缓冲液(TD)的 | 五 |
| 总成交量(用cDNA) | 10 |
| 步骤4.3:图书馆PCR反应 | |
| 试剂 | 体积(微升) |
| PCR主要混合液(NPM) | 7.5 |
| 指数1底漆(I7) | 2.5 |
| 指数2底漆(I5) | 2.5 |
| 总成交量(含tagmented的cDNA) | 25 |
| 步骤4.3.2:图书馆PCR条件 | |
| 72°C,3分钟 | |
| 95℃,30秒 | |
| 多达18个周期10 95℃秒,55℃30秒,30秒,72℃ | |
| 72℃,5分钟 | |
| 10°C,永远 | |
表1:反应设置和缓冲区一步一步的所有缓冲区和反应混合物表的内容。
表2:定量RT-PCR的引物序列用于测量主机(18S rRNA基因)和病毒(埃博拉和拉沙)内容引物。 "KGH'是凯内马政府医院在塞拉利昂,那里的埃博拉引物进行了测试2。 "Kulesh'是谁设计引物组14研究者。
表3:核糖体RNA(rRNA的)枯竭寡核苷酸 195至选择性枯竭步骤6人rRNA互补的50个核苷酸长序列。 请点击此处下载此文件。
| 寡名称 | 序列(5'至3') |
| 埃博拉KGH FW | GTCGTTCCAACAATCGAGCG |
| 埃博拉KGH RV | CGTCCCGTAGCTTTRGCCAT |
| 埃博拉KULESH FW | TCTGACATGGATTACCACAAGATC |
| 埃博拉KULESH RV | GGATGACTCTTTGCCGAACAATC |
| 拉沙SL FW | GTA AGC CCA GCD GYA AAB CC |
| 拉沙SL RV | AAG CCA CAG AAA RCT GGS AGC的 |
| 18S rRNA基因FW | TCCTTTAACGAGGATCCATTGG |
| 18S rRNA基因RV | CGAGCTTTTTAACTGCAGCAACT |
讨论
概述的方法能够健壮,通用,快速排序,并用于2014年爆发期间2,4测序埃博拉病毒。通过偶合选择性消耗和cDNA合成用tagmentation文库构建,整个过程的时间是通过从前面的适配器连接方法〜2天缩短。最近,该协议受雇于国际合作者和其他人取得了巨大成功和15,16将被部署到西非的实验室,以支持当地基于基因组学研究的研究和诊断17。
这里所描述的协议使用随机引物来制备cDNA为病毒RNA-SEQ库。不同于以往的病毒RNA-SEQ方法,它不需要序列数据或复杂和费时的引物设计为特定的病毒或分化体的先验知识。该方法可以适用于任何病毒RNA样品。例如,它被用来产生从两个埃博拉病毒内容和拉沙样品6。该协议也可以被用于主机转录,宏基因组和病原体发现测序项目1。
该协议的一个关键步骤是针对核糖核酸酶H消化,高通量,从病毒样本去除多余的载体和宿主RNA低成本的方法。该协议的选择性耗尽步骤使用了许多部件,并需要技巧性和准确性。额外的时间和精力应该在初始设置期间服用。
因为大多数临床血清和血浆样品通常具有非常少的核酸材料,污染和样品损失是常见的。为了避免这些问题,应特别注意使用此协议时作出。首先,RNA是非常容易退化;因此,所有的区域应清洁,无核酸酶。第二,以确定适用于该协议使用的样品,应该用于定量5,6-两种宿主RNA和病毒定量RT-PCR测定。当从协议测序结果测序成功比较输入量( 即,代全病毒装配足够的数据)与含有至少100pg的总RNA和病毒的1000份样本相关。应避免第三,暴露于核酸环境来源。这里列出的协议是在安全防范生物安全柜和限制环境污染物进行。此外,我们的组和其他已经注意到商业酶可以是低输入样本6,18在污染细菌的核酸的另一个来源。用干净的工作空间( 如 PCR罩,生物安全柜)和阴性对照( 例如 ,水或缓冲液)将有助于减轻分别和跟踪污染。用于与样品<100pg的总RNA,只聚(RA)载体RNA,不rRNA基因,应该被耗尽,以确保高质量的测序结果,同时限制材料的损失。对于非常低输入样本,cDNA的扩增方法可以是更合适的19,虽然聚(RA)载体应之前的cDNA合成中除去。
主机的rRNA的耗尽丰富在测序文库的病毒含量,并适用于不同的样本的集合,包括血清或血浆,和多种类型的来自啮齿类和非人灵长类5,6-组织。在非人类生物,读取对准28S rRNA的仍然耗尽后,提示28S rRNA的是人类和其它物种6,20之间不太保守。当使用这种方法与非人类分离,它可能有必要对特定的主机3,21的发散rRNA序列互补的DNA寡核苷酸来补充。
由于该协议是公正的,病毒会读取仅代表总库内容的一小部分。虽然rRNA基因是宿主RNA品种最丰富,仅rRNA基因的一小部分读取(0; 1%)被发现选择性耗尽之后,所有其它宿主RNA( 例如 ,mRNA)的仍将耗尽后及可能占许多测序从样品中读取。因此,"过采样"( 即 oversequencing)单个图书馆需要,以有足够的覆盖面为病毒组装和变异电话。对于我们的研究中,我们试图序列每个样品〜20000000读取具有用于病毒基因组和相关的变异体的分析深度不够以及宏基因组内容2,5。对于宏基因组和病原体发现研究中,要注意的是污染宿主DNA通过DNA酶消化除去是很重要的。因此,包含的DNA基因组的病毒和其他病原体的过程中会丢失,但是RNA中间体可以仍然进行测序。
披露声明
The authors have no competing financial interests.
致谢
This work has been funded in part with Federal funds from the National Institutes of Health, Office of Director, Innovator (No.: DP2OD06514) (PCS) and from the National Institute of Allergy and Infectious Diseases, National Institutes of Health, Department of Health and Human Services, under Contracts (No:HHSN272200900018C, HHSN272200900049C and U19AI110818).
材料
| Name | Company | Catalog Number | Comments |
| 96-well PCR Plates | VWR | 47743-953 | |
| Strips of Eight Caps | VWR | 47745-512 | |
| Nuclease-free water | Ambion | AM9937 | 50 ml bottle |
| TURBO DNase | Ambion | AM2238 | post RNA extraction step, 2 U/µl, buffer included |
| PCR cycler | any PCR cyclers | ||
| Agencourt RNAClean XP SPRI beads | Beckman Coulter Genomics | A63987 | beads for RNA cleanup |
| Real Time qPCR system | any system | ||
| DynaMag-96 Side Skirted Magnet | Invitrogen | 12027 | |
| 70% Ethanol | prepare fresh | ||
| qRT-PCR primers | IDT DNA | see Table 2 | |
| 5 M NaCl | Ambion | AM9760G | |
| 1 M Tris-HCl pH 7.4 | Sigma | T2663-1L | |
| 1 M Tris-HCl pH 7.5 | Invitrogen | 15567-027 | |
| 1 M MgCl2 | Ambion | AM9530G | |
| Linear acrylamide | Ambion | AM9520 | |
| DNA oligos covering entire rRNA region | IDT DNA | see Table 3, order lab-ready at 100 µM | |
| Oligo (dT) | IDT DNA | 40 nt long, desalted | |
| Hybridase Thermostable RNase H | Epicentre | H39100 | |
| RNase-free DNase Kit | Qiagen | 79254 | post selective depletion step |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Random Primers | Invitrogen | 48190-011 | mostly hexamers |
| 10 mM dNTP mix | New England Biolabs | N0447L | |
| SuperScript III Reverse Transcriptase | Invitrogen | 18080-093 | with first-strand buffer, DTT |
| Air Incubator | any air incubator cyclers | ||
| NEBNext Second Strand Synthesis (dNTP-free) Reaction Buffer | New England Biolabs | B6117S | 10x |
| E. coli DNA Ligase | New England Biolabs | M0205L | 10 U/μl |
| E. coli DNA Polymerase I | New England Biolabs | M0209L | 10 U/μl |
| E. coli RNase H | New England Biolabs | M0297L | 2 U/μl |
| 0.5 M EDTA | Ambion | AM9261 | |
| Agencourt AMPure XP SPRI beads | Beckman Coulter Genomics | A63881 | beads for DNA cleanup |
| Elution Buffer | Qiagen | 10 mM Tris HCl, pH 8.5 | |
| Quant-iT dsDNA HS Assay Kit | Invitrogen | Q32854 | |
| Qubit fluorometer | Invitrogen | Q32857 | |
| Nextera XT DNA Sample Prep Kit | Illumina | FC-131-1096 | |
| Nextera XT DNA Index Kit | Illumina | FC-131-1001 | |
| Tapestation 2200 | Agilent | G2965AA | |
| High Sensitivity D1000 reagents | Agilent | 5067-5585 | |
| High Sensitivity D1000 ScreenTape | Agilent | 5067-5584 | |
| BioAnalyzer 2100 | Agilent | G2939AA | |
| High Sensitivity DNA reagents | Agilent | 5067-4626 | |
| Library Quantification Complete kit (Universal) | Kapa Biosystems | KK4824 | alternative to tapestation, bioanalyzer for library quantification |
参考文献
- Stremlau, M. H., et al. Discovery of novel rhabdoviruses in the blood of healthy individuals from West Africa. PLoS Negl Trop Dis. 9, e0003631(2015).
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345, 1369-1372 (2014).
- Morlan, J. D., Qu, K., Sinicropi, D. V. Selective depletion of rRNA enables whole transcriptome profiling of archival fixed tissue. PLoS One. 7, e42882(2012).
- Park, D. J., et al. Ebola Virus Epidemiology, Transmission, and Evolution during Seven Months in Sierra Leone. Cell. 161, 1516-1526 (2015).
- Andersen, K. G., et al. Clinical Sequencing Uncovers Origins and Evolution of Lassa Virus. Cell. 162, 738-750 (2015).
- Matranga, C. B., et al. Enhanced methods for unbiased deep sequencing of Lassa and Ebola RNA viruses from clinical and biological samples. Genome Biol. 15, 519(2014).
- Tang, F., et al. RNA-Seq analysis to capture the transcriptome landscape of a single cell. Nat Protoc. 5, 516-535 (2010).
- Jiang, L., et al. Synthetic spike-in standards for RNA-seq experiments. Genome Res. 21, 1543-1551 (2011).
- Agilennt Technologies. , Available from: http://www.agilent.com/cs/library/usermanuals/Public/G2946-90004_Vespucci_UG_eBook_(NoSecPack).pdf. (2015).
- Kapa Biosystems. , Available from: https://www.kapabiosystems.com/product-applications/products/next-generation-sequencing-2/library-quantification/ (2015).
- Illumina Technologies. , Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/system_documentation/miseq/preparing-libraries-for-sequencing-on-miseq-15039740-d.pdf. (2015).
- Kircher, M., Sawyer, S., Meyer, M. Double indexing overcomes inaccuracies in multiplex sequencing on the Illumina platform. Nucleic Acids Res. 40, 3(2012).
- Andrews, S. Babraham Bioinformatics. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Trombley, A. R., et al. Comprehensive panel of real-time TaqMan polymerase chain reaction assays for detection and absolute quantification of filoviruses, arenaviruses, and New World hantaviruses. Am J Trop Med Hyg. 82, 954-960 (2010).
- Hu, Y., et al. Serial high-resolution analysis of blood virome and host cytokines expression profile of a patient with fatal H7N9 infection by massively parallel RNA sequencing. Clin Microbiol Infect. 21, e1-4 713(2015).
- Simon-Loriere, E., et al. Distinct lineages of Ebola virus in Guinea during the 2014 West African epidemic. Nature. 524, 102-104 (2015).
- Folarin, O. A., Happi, A. N., Happi, C. T. Empowering African genomics for infectious disease control. Genome Biol. 15, 515(2014).
- Blainey, P. C., Quake, S. R. Digital MDA for enumeration of total nucleic acid contamination. Nucleic Acids Res. 39, 19(2011).
- Malboeuf, C. M., et al. Complete viral RNA genome sequencing of ultra-low copy samples by sequence-independent amplification. Nucleic Acids Res. 41, 13(2013).
- Gonzalez, I. L., Sylvester, J. E., Smith, T. F., Stambolian, D., Schmickel, R. D. Ribosomal RNA gene sequences and hominoid phylogeny. Mol Biol Evol. 7, 203-219 (1990).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nat Methods. 10, 623-629 (2013).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。