Method Article
هندسة طفرات اكتساب الوظيفة غير المتجانسة المنشأ في الخلايا الجذعية البشرية المكونة للدم والخلايا السلفية
In This Article
Summary
تعد الاستراتيجيات الجديدة لنمذجة الطفرات الجسدية بأمانة في الخلايا الجذعية المكونة للدم والخلايا السلفية (HSPCs) ضرورية لدراسة بيولوجيا الخلايا الجذعية المكونة للدم والأورام الخبيثة الدموية بشكل أفضل. هنا ، يتم وصف بروتوكول لنمذجة طفرات كسب الوظيفة غير المتجانسة في HSPCs من خلال الجمع بين استخدام CRISPR / Cas9 ونقل المانحين rAAV المزدوج.
Abstract
طوال حياتهم ، تكتسب الخلايا الجذعية والسلفية المكونة للدم (HSPCs) طفرات جسدية. بعض هذه الطفرات تغير الخصائص الوظيفية ل HSPC مثل الانتشار والتمايز ، وبالتالي تعزيز تطور الأورام الخبيثة الدموية. مطلوب معالجة جينية فعالة ودقيقة ل HSPCs لنمذجة وتوصيف وفهم العواقب الوظيفية للطفرات الجسدية المتكررة بشكل أفضل. يمكن أن يكون للطفرات تأثير ضار على الجين وتؤدي إلى فقدان الوظيفة (LOF) أو ، في تناقض صارخ ، قد تعزز الوظيفة أو حتى تؤدي إلى خصائص جديدة لجين معين ، تسمى كسب الوظيفة (GOF). على عكس طفرات LOF ، تحدث طفرات GOF بشكل حصري تقريبا بطريقة متغايرة الزيجوت. لا تسمح بروتوكولات تحرير الجينوم الحالية بالاستهداف الانتقائي للأليلات الفردية ، مما يعوق القدرة على نمذجة طفرات GOF غير المتجانسة. هنا ، نقدم بروتوكولا مفصلا حول كيفية هندسة طفرات النقاط الساخنة GOF غير المتجانسة في HSPCs البشرية من خلال الجمع بين الإصلاح الموجه بوساطة CRISPR / Cas9 وتقنية AAV6 المؤتلفة لنقل قالب مانح الحمض النووي بكفاءة. الأهم من ذلك ، أن هذه الاستراتيجية تستخدم نظام مراسل فلوري مزدوج للسماح بتتبع وتنقية HSPCs التي تم تحريرها بنجاح بشكل غير متجانس. يمكن استخدام هذه الاستراتيجية للتحقيق بدقة في كيفية تأثير طفرات GOF على وظيفة HSPC وتطورها نحو الأورام الخبيثة الدموية.
Introduction
مع تطور تقنية التكرارات القصيرة المتباعدة بانتظام (CRISPR) / Cas9 ، تمت إضافة أداة جديدة وقوية للغاية إلى مجموعة أدوات العلماء. تسمح هذه التقنية بالهندسة الدقيقة للجينوم وقد أثبتت أنها مفيدة للغاية ليس فقط لأغراض البحث (تمت مراجعتها في Hsu et al.1) ولكن في الآونة الأخيرة تمت ترجمتها بنجاح إلى الإعداد السريري2،3،4. تعتمد استراتيجيات تحرير كريسبر / كاس 9 على نشاط بروتين كاس 9 والحمض النووي الريبي أحادي الدليل (sgRNA)5،6،7. في الخلية المضيفة ، يتم توجيه بروتين Cas9 إلى موقع معين في الحمض النووي مكمل لتسلسل sgRNA وسيقدم كسر الحمض النووي المزدوج (DSB). بمجرد إنشاء DSB ، هناك آليتان رئيسيتان ومتنافستان للإصلاح يمكن أن تحدثا: الانضمام النهائي غير المتماثل (NHEJ) والإصلاح الموجه بالتماثل (HDR). NHEJ هي آلية إصلاح معرضة للخطأ ، وتستخدم في الغالب مما يؤدي إلى عمليات الإدراج والحذف (indels) ، في حين أن HDR ، باستخدام الكروماتيد الشقيق كقالب إصلاح ، دقيق للغاية ولكنه يقتصر على المرحلة S أو G2 من دورة الخلية8. في هندسة الجينوم ، يمكن استخدام HDR للتعديل المستهدف للحمض النووي من خلال توفير قالب مانح محاط بأذرع تماثلية مماثلة لطرفي الحمض النووي ل DSB الناجم عن Cas9 (الشكل 1). يمكن أن يؤثر نوع قالب المانحين المستخدم ل HDR بشكل كبير على كفاءة التحرير. بالنسبة للهندسة الوراثية في HSPCs البشرية ، تم وصف النمط المصلي 6 (AAV6) المرتبط بالغدي مؤخرا بأنه وسيلة ممتازة لتسليم قوالب الحمض النووي أحادية الشريط 9,10.
يمكن استخدام هندسة الجينوم CRISPR / Cas9 علاجيا لتصحيح الطفرات الضارة11 ولكن يمكن استخدامها أيضا لإدخال طفرات مسببة للأمراض في الحمض النووي لنمذجة تطور السرطان12. يتطور سرطان الدم ، مثل اللوكيميا ، عن طريق الاكتساب المتسلسل للطفرات الجسدية في HSPCsالصحية 13,14. تؤدي الأحداث الجينية المبكرة إلى ميزة تكاثرية نسيلية ، مما يؤدي إلى تكون الدم النسيلي ذي الإمكانات غير المحددة (CHIP)15,16. سيؤدي المزيد من اكتساب الطفرات في النهاية إلى تحول اللوكيميا وتطور المرض. يمكن العثور على طفرات جسدية في الجينات التي تتحكم في التجديد الذاتي والبقاء والانتشار والتمايز17.
يسمح إدخال الطفرات الفردية عبر هندسة الجينوم في HSPCs الصحية بنمذجة عملية سرطان الدم التدريجي هذه بدقة. العدد المحدود من الطفرات المتكررة الموجودة في الأورام النخاعية مثل ابيضاض الدم النخاعي الحاد (AML)18,19 يجعل هذا المرض قابلا بشكل خاص لإعادة تلخيصه باستخدام أدوات هندسة الجينوم.
يمكن أن تظهر الطفرات الجسدية على أليل واحد فقط (طفرات أحادية الأليليك / غير متجانسة الزيجوت) أو على كلا الأليلين (طفرات ثنائية الأليليك / متماثلة الزيجوت) ويمكن أن يكون لها تأثيرات عميقة على وظيفة الجين ، مما قد يتسبب في فقدان الوظيفة (LOF) أو اكتساب الوظيفة (GOF). تؤدي طفرات LOF إلى انخفاض (إذا تأثر أليل واحد) أو كامل (إذا تأثر كلا الأليلين LOF) من الجين ، بينما تؤدي طفرات GOF إلى زيادة التنشيط أو وظيفة جديدة للجين. عادة ما تكون طفرات GOF متغايرة الزيجوت20.
الأهم من ذلك ، أن الزيجوت (غير المتجانس مقابل متماثل الزيجوت) له آثار كبيرة على محاولة نمذجة الطفرة بأمانة. لذلك ، فإن المعالجة المستهدفة لأليل واحد فقط من الجين ضرورية لهندسة طفرات GOF الساخنة غير المتجانسة الزيجوت. يؤدي NHEJ المعرض للخطأ إلى أطوال مختلفة21 يمكن أن تؤدي إلى عواقب بيولوجية متفاوتة وغير متوقعة. ومع ذلك ، نظرا لأن NHEJ هو برنامج الإصلاح السائد الذي تستخدمه الخلايا بعد إدخال DSB ، فإن معظم منصات CRISPR / Cas9 المستخدمة حاليا لمعالجة HSPCs لا تسمح بالتنبؤ بدقة بالنتيجة الجينية22,23. في المقابل ، فإن إدخال كسر مزدوج الخيوط بوساطة كريسبر / كاس9 (DSBs) ، جنبا إلى جنب مع استخدام قوالب مانحي الحمض النووي القائمة على ناقلات الفيروسات الغدية المؤتلفة (rAAV) لهندسة الجينوم عبر HDR ، يسمح بإدخال الطفرات الخاصة بالأليلات في HSPCsالبشرية 11,24. يمكن إجراء التكامل المتزامن لتسلسل متحولة ونوع بري (WT) إلى جانب مراسلات فلورية مميزة على الأليلات الفردية لاختيار النمط الجيني غير المتجانس (الشكل 2). يمكن الاستفادة من هذه الاستراتيجية كأداة قوية لتوصيف آثار طفرات GOF الساخنة المتكررة ، اللوكيميا ، غير المتجانسة الزيجوت على وظيفة HSPC ، وبدء المرض ، وتقدمه.
في هذه المقالة ، يتم توفير بروتوكول مفصل للهندسة الفعالة لطفرات GOF غير المتجانسة المتحورة بشكل متكرر في HSPCs البشرية الأولية. تجمع هذه الاستراتيجية بين استخدام CRISPR / Cas9 وتحويل AAV6 مزدوج لتوفير قوالب WT وقوالب مانحة للحمض النووي الطافر للجيل المحتمل من طفرة GOF غير المتجانسة. على سبيل المثال ، سيتم عرض هندسة الطفرات المتكررة من النوع 1 (حذف 52 نقطة أساس) في جين الكالريتيكولين (CALR)25. تم العثور على طفرة GOF غير المتجانسة في exon 9 من CALR بشكل متكرر في اضطرابات التكاثر النقوي مثل كثرة الصفيحات الأساسية (ET) والتليف النقوي الأولي (PMF)26. CALR هو بروتين مقيم في الشبكة الإندوبلازمية له في المقام الأول وظيفة مراقبة الجودة في عملية طي البروتينات المركبة حديثا. يمكن تقسيم هيكلها إلى ثلاثة مجالات رئيسية: مجال طرفي أميني (N) ومجال P غني بالبرولين ، والذي يشارك في وظيفة مرافقة البروتين ، ومجال C ، الذي يشارك في تخزين الكالسيوم وتنظيمه27,28. تتسبب طفرات CALR في إزاحة إطار +1 ، مما يؤدي إلى نسخ نهاية C-terminal ممتدة جديدة وفقدان إشارة الاحتفاظ بالشبكة الإندوبلازمية (ER) (KDEL). وقد تبين أن CALR الطافر يربط مستقبلات الثرومبوبويتين (TPO) ، مما يؤدي إلى إشارات مستقلة عن TPO مع زيادة الانتشار29.
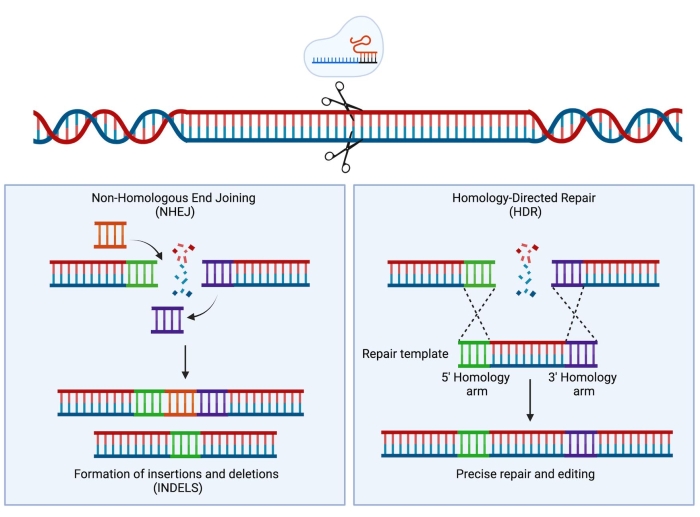
الشكل 1: إصلاح NHEJ و HDR. تمثيل تخطيطي مبسط لآليات إصلاح NHEJ و HDR بعد إدخال كسر مزدوج تقطعت بهم السبل في الحمض النووي. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
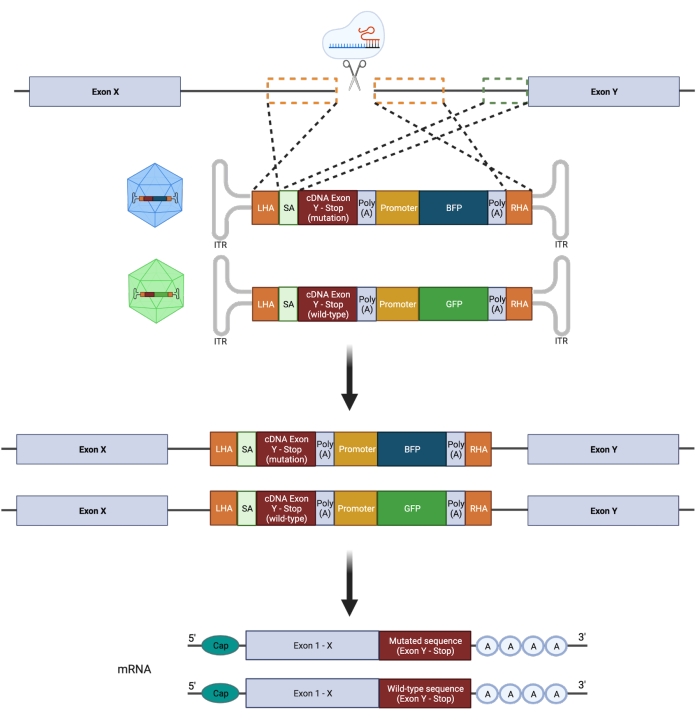
الشكل 2: نظرة عامة تخطيطية لاستراتيجية تحرير HDR biallelic. تمثيل تخطيطي يوضح دمج قوالب المانحين في الأليلات المستهدفة متبوعة بترجمتها إلى mRNAs العاملة. تشير المربعات المنقطة البرتقالية إلى المناطق المقابلة لذراع التماثل الأيسر (LHA) وذراع التماثل الأيمن (RHA). الحجم المثالي ل HAs هو 400 نقطة أساس لكل منهما. يمثل المربع المنقط الأخضر المنطقة المقابلة لتسلسل SA. حجم SA هو 150 نقطة أساس. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
Protocol
يتطلب هذا البروتوكول استخدام CD34 + HSPCs المشتقة من الجهات المانحة ويتطلب موافقة أخلاقية من مجالس المراجعة المؤسسية المحلية (IRB) والموافقة المستنيرة الموقعة. تم عزل CD34 + HSPCs المستخدمة في هذا البروتوكول من دم الحبل السري (UCB) للولادات المدوية (>34 أسبوعا من الحمل). تم الحصول على موافقة مستنيرة من الأمهات قبل الولادة ، وتم الحصول على الموافقة الأخلاقية لجمع UCB (موافقة IRB: 31-322 ex 18/19) من جامعة غراتس الطبية. يمكن العثور على قائمة كاملة بالمواد المستخدمة في هذا البروتوكول في جدول المواد.
1. تصميم sgRNA وتقييم كفاءات القطع
- ابحث عن موقع الطفرة المطلوبة والنص الصحيح على أداة قاعدة بيانات عبر الإنترنت (على سبيل المثال ، COSMIC ، https://cancer.sanger.ac.uk/cosmic).
- حدد sgRNA المجاور للطفرة (الاستهداف الخارجي) أو في intron السابق (الاستهداف الداخلي) باستخدام أداة تصميم sgRNA. في هذا البروتوكول ، يتم استخدام الأداة عبر الإنترنت من Benchling. انظر جدول المواد للحصول على قائمة بالأدوات الممكنة عبر الإنترنت لتصميم sgRNA.
- حدد الخيار + (إنشاء) > تسلسل الحمض النووي > استيراد تسلسلات الحمض النووي > استيراد من قواعد البيانات. أدخل الجين المطلوب ، على سبيل المثال ، CALR وحدد الإنسان كنوع > البحث > حدد النسخة الصحيحة (على سبيل المثال ، CALR-201 -ENST00000316448) > استيراد. حدد المنطقة التي تريد تقديم فاصل DS فيها (على سبيل المثال ، intron 7).
- حدد خيار CRISPR على الجانب الأيمن من الشاشة وحدد تصميم الأدلة وتحليلها. حدد دليلا واحدا ، واحتفظ بطول الدليل إلى 20 نقطة أساس ، وتسلسل PAM للتحرير باستخدام SpCas9 (NGG).
- اختر دليلا بدرجة عالية على الهدف (فرصة عالية للتحرير في الموضع المطلوب) ودرجة عالية خارج الهدف (فرصة منخفضة للتحرير في المواقع غير المرغوب فيها). اختر ثلاثة أدلة على الأقل لاختبارها من أجل العثور على أفضل أداء sgRNA. اطلب sgRNA ك sgRNA اصطناعي معدل كيميائيا من بائع تجاري.
ملاحظة: من المستحسن في هذه المرحلة طلب كمية صغيرة من sgRNAs الاصطناعية للشاشة الأولية. بمجرد تحديد sgRNA جيد الأداء ، تابع بترتيب أكبر من sgRNA المحدد. - ذوبان الجليد 2 × 10 5-5 × 105 CD34 + HSPCs. نقل الخلايا إلى 10 مل من RPMI1640 قبل التسخين مع استكمال المضادات الحيوية 1 ٪ (على سبيل المثال ، البنسلين / الستربتومايسين).
ملاحظة: يجب استخدام CD34+ HSPCs بنقاوة >90٪ لتحقيق أفضل النتائج وأكثرها قابلية للتكرار. - جهاز طرد مركزي عند 350 × جم في درجة حرارة الغرفة (RT) لمدة 10 دقائق. عد الخلايا باستخدام مقياس الهيموسيتول وقم بتعليق الخلايا في وسط SFEM II المكمل ب 0.2٪ بنسلين / ستربتومايسين (P / S) ، 100 نانوغرام / مل ثرومبوبويتين (TPO) ، 100 نانوغرام / مل عامل الخلايا الجذعية (SCF) ، 100 نانوغرام / مل من التيروزين كيناز الشبيه ب FMS 3 ليجند (FLT3L) ، 100 نانوغرام / مل إنترلوكين -6 (IL-6) ، 35 نانومتر UM171 ، و 0.75 ميكرومتر StemRegenin1 (SR1) بتركيز 2.5 × 105 خلايا / مل. احتضان عند 37 درجة مئوية / 5٪ CO2 لمدة 48 ساعة - 72 ساعة.
ملاحظة: سيشار إلى الوسيط الكامل من الآن فصاعدا باسم وسيط الاحتفاظ HSPC. - اجمع الخلايا في أنبوب سعة 15 مل وعد الخلايا. تحقق من صلاحية الخلية عن طريق استبعاد تريبان الأزرق.
- قبل البدء ، قم بتشغيل نظام النقل وحدد خيار cuvettes. حدد البرنامج المناسب وحل nucleofection ل HSPCs (انظر جدول المواد). بين 2 × 10 5 و5 × 106 خلايا يمكن أن تكون نواة في كوفيت واحد 100 ميكرولتر. ضع جانبا عددا صغيرا من الخلايا (على سبيل المثال ، 1 × 10 5-2 × 105) للاحتفاظ بها في الثقافة لمدة 48 ساعة لاستخدامها كعنصر تحكم في WT.
- تحضير مجمع RNP. في أنبوب سعة 1.5 مل ، أضف 15 ميكروغرام من Cas9 و 8 ميكروغرام من sgRNA (النسبة المولية 1: 2.5) واحتضانها عند 25 درجة مئوية لمدة 10 دقائق في كتلة تسخين.
- أثناء احتضان مجمع RNP ، قم بالطرد المركزي للخلايا عند 350 × جم في RT لمدة 5 دقائق وتخلص من المادة الطافية باستخدام ماصة. تعليق الخلايا في 100 ميكرولتر من محلول nucleofection.
- امزج الخلايا مع مجمع RNP وانقلها إلى كوفيت. اضغط برفق على الكوفيت لإزالة أي فقاعات هواء محتملة قد تكون تشكلت أثناء النقل.
- أدخل الكوفيت في حامل نظام النقل وقم بالكهرباء الخلايا باستخدام برنامج DZ-100.
- مباشرة بعد التثقيب الكهربائي ، أضف 400 ميكرولتر من وسط الاحتفاظ HSPC الدافئ مسبقا بدون P / S.
- انقل الخلايا ذات ماصة نقل دقيقة إلى صفيحة استزراع تحتوي على وسط احتفاظ HSPC مسبق التسخين بدون P / S. اعتمادا على رقم الخلية ، استخدم لوحة استزراع مناسبة (لوحة 24 أو 12 أو 6 آبار) من أجل الوصول إلى كثافة تتراوح بين 0.25 × 10 6 و 1 × 106 خلايا / مل.
- انقل اللوحة إلى الحاضنة عند 37 درجة مئوية / 5٪ CO2. احتضان الخلايا nucleofected لمدة 6-8 ساعات.
- بعد 6-8 ساعات ، قم بإزالة الوسط القديم واستبدله بوسط احتفاظ HSPC طازج مسبق التسخين مكمل ب P / S. الخلايا بتركيز يتراوح بين 2.5 × 10 5 و 5 × 105 وانقلها إلى لوحة زراعة الخلايا (لوحة 24 أو 12 أو 6 آبار). احتضان الخلايا لمدة 48 ساعة عند 37 درجة مئوية / 5٪ CO2.
- حصاد 2 × 10 5 خلايا وأجهزة الطرد المركزي في 350 × غرام في RT لمدة5 دقائق. قبل البدء ، اضبط كتل التسخين على 65 درجة مئوية و 98 درجة مئوية.
- تخلص من المادة الطافية وقم بتعليق الخلايا في 1 مل من 1x DPBS في أنبوب سعة 1.5 مل. جهاز طرد مركزي عند 350 × جم في RT لمدة 5 دقائق.
- تخلص من المادة الطافية وقم بتعليق الخلايا في 50 ميكرولتر من محلول استخراج الحمض النووي. دوامة لمدة 15 ثانية.
- احتضان لمدة 6 دقائق عند 65 درجة مئوية. دوامة لمدة 15 ثانية واحتضان لمدة 2 دقيقة عند 98 درجة مئوية.
- تضخيم منطقة DSB بواسطة PCR باستخدام البادئات التي تولد amplicon من حوالي 400-600 bp مع DSB في المركز. استخدم 0.5-1 ميكرولتر من الحمض النووي المستخرج لتفاعل PCR.
ملاحظة: نظرا لتكوين محلول استخراج الحمض النووي ، لا يمكن قياس تركيزات الحمض النووي بدقة عن طريق القياس الطيفي. - قم بتشغيل منتج PCR باستخدام سلم الحمض النووي على هلام أغاروز 1.5٪ عند 100 فولت لمدة 45-60 دقيقة. ضع الجل على ضوء أزرق أو مصباح للأشعة فوق البنفسجية.
- استخرج شريط الحمض النووي بالحجم الصحيح من الجل باستخدام مجموعة متاحة تجاريا وفقا لتعليمات الشركة المصنعة (انظر جدول المواد).
- تسلسل العينات عن طريق تسلسل سانجر باستخدام التمهيدي الأمامي أو التمهيدي العكسي من PCR. بالنسبة لمنتجات تفاعل البوليميراز المتسلسل التي يتراوح طولها بين 400 و600 نقطة أساس ، يلزم 75 نانوغرام من الحمض النووي للتسلسل بتركيز 5 نانوغرام / مل في حجم إجمالي قدره 15 ميكرولتر.
- تحليل كفاءة تحرير sgRNAs عن طريق تحميل ملفات التسلسل إلى برنامج مصمم لحساب كفاءة التحرير من خلال تحديد الإدراج والحذف الناتج عن sgRNA. حدد sgRNA الأفضل أداء للمتابعة.
ملاحظة: يتم سرد الأدوات المخصصة عبر الإنترنت في جدول المواد. يتطلب هذا التحليل ملفات .ab1 الخاصة ب HSPCs المنقولة و WT وتسلسل sgRNA وتسلسل PAM.
2. بناء ناقلات الإصلاح الموجه بالتماثل (HDR)
- تصميم قالب HDR
ملاحظة: يجب تصميم قالبين HDR: قالب لتسلسل WT وقالب للتسلسل المتحور.- استيراد التسلسل الجينومي وتسلسل الترميز (CDS) للجين المطلوب إلى برنامج مناسب للاستنساخ الجزيئي (يمكن العثور على أدوات مخصصة مدرجة في جدول المواد). من ملف التسلسل الجينومي ، صمم ذراع التماثل الأيسر (HA) عن طريق تحديد 400 نقطة أساس بشكل مثالي على الطرف 5 'من DSB. الصق هذا التسلسل في ملف جديد.
- إذا تم استهداف intron باستخدام sgRNA ، فيجب تضمين تسلسل متقبل لصق (SA). حدد آخر 150 نقطة أساس من intron المستهدف والصقه بعد HA الأيسر. إذا تم استهداف exon مباشرة ، فإن هذا التسلسل ليس ضروريا (الشكل 3).
- من ملف CDS ، حدد cDNA محل الاهتمام. في حالة استهداف تسلسل خارجي ، تأكد من أن cDNA يبدأ على الفور في اتجاه مجرى موقع DSB ويتضمن جميع الإكسونات التالية للجين محل الاهتمام بالإضافة إلى كودون التوقف. في حالة استهداف intron ، تأكد من أن cDNA يبدأ بالكودون الأول من exon التالي. كودون تحسين cDNA وإدخاله بعد تسلسل SA. استخدم أدوات مخصصة عبر الإنترنت لهذا الغرض (جدول المواد).
- أدخل إشارة 3 'polyadenylation (PolyA) بعد cDNA محل الاهتمام (على سبيل المثال ، SV40 أو bGH) (الجدول 1). بعد PolyA ، أدخل تسلسل المروج (على سبيل المثال ، فيروس تشكيل تركيز الطحال [SFFV] أو polyubiquitin C [UBC] ، الجدول 1) للبروتين الفلوري.
- أدخل تسلسل البروتين الفلوري (أي إما GFP أو BFP أو mCherry). أدخل إشارة PolyA ثانية ولكن مختلفة.
ملاحظة: سيؤدي إدخال تسلسل PolyA مختلف إلى تجنب مشاكل مثل إعادة التركيب البكتيري للبلازميد أو مشاكل محاذاة التسلسل بسبب تسلسلين متطابقين في القالب. - من ملف التسلسل الجينومي ، صمم HA الصحيح عن طريق تحديد 400 نقطة أساس بشكل مثالي على الطرف 3 'من DSB. أدخل هذا التسلسل بعد PolyA الثاني.
- قم بإنشاء نسخة من القالب بأكمله وقم بتعديل cDNA محل الاهتمام بحيث يحتوي على تسلسل الطفرة المطلوبة.
- استبدل بروتين الفلورسنت ببروتين فلوري مختلف. على سبيل المثال ، إذا تم استخدام GFP لقالب WT ، فقم باستبداله مع BFP أو mCherry للقالب المتحول. بناء واستنساخ قوالب HDR في بلازميد pAAV-MCS (أو غيرها من العمود الفقري المناسب).
ملاحظة: يمكن إنتاج الأجزاء المطلوبة للتجميع إما عن طريق تفاعل البوليميراز المتسلسل أو طلبها تجاريا ويجب أن تحتوي على تسلسلات متداخلة مع الأجزاء المجاورة لها. إذا كانت الطفرة محل الاهتمام عبارة عن طفرة نقطية أو إدخال صغير أو حذف صغير ، فيمكن إنتاج cDNA المتحور بواسطة تفاعل البوليميراز المتسلسل عن طريق إجراء طفرات موجهة للموقع على قالب HDR الذي يحتوي على WT cDNA. - قم بتحويل الإشريكية القولونية المختصة بالمنتج المجمع باستخدام طريقة الصدمة الحرارية. قبل البدء ، ضع البكتيريا لتذوب على الثلج لمدة 10 دقائق.
- أضف 2 ميكرولتر من المنتجات المجمعة إلى 50 ميكرولتر من البكتيريا. امزج المحتويات عن طريق النقر بلطف.
- ضع العينات على الثلج لمدة 30 دقيقة. نقل العينات إلى مجموعة حرارية إلى 42 درجة مئوية لمدة 30 ثانية.
- نقل العينات على الجليد لمدة 5 دقائق. أضف 450 ميكرولتر من وسط SOC بدرجة حرارة الغرفة واحتضان العينات عند 37 درجة مئوية لمدة 1 ساعة.
- انشر العينات على ألواح أجار LB تحتوي على الأمبيسلين. لكل عينة ، انشر 100 ميكرولتر من ثلاثة تخفيفات مختلفة من المحلول البكتيري (غير مخفف ، 1: 5 ، و 1:10) من أجل الحصول على ألواح أجار LB مع مستعمرات مفردة يمكن قطفها. احتضان الأطباق طوال الليل عند 37 درجة مئوية.
- في اليوم التالي ، اختر ثلاث مستعمرات لكل عينة. انقل المستعمرات إلى أنابيب سعة 15 مل مع أغطية تحتوي على 4 مل من وسط LB المكمل بالأمبيسلين واحتضانها طوال الليل في شاكر عند 37 درجة مئوية.
- حصة 500 ميكرولتر من المحلول البكتيري من كل مستعمرة وتخزينها في الثلاجة لاستخدامها لاحقا. قم بإجراء تحضير مصغر لاستخراج الحمض النووي البلازميد وفقا لتعليمات الشركة المصنعة.
- أرسل العينات لتسلسل سانجر لتأكيد التجميع الصحيح للبلازميدات. استخدم ما يكفي من البادئات الموزعة في جميع أنحاء البلازميد لضمان تأكيد التسلسل دون انقطاع ، وتغطية كل منطقة بشكل مثالي مرتين بقراءات أمامية وعكسية.
- أضف 200 ميكرولتر من المحلول البكتيري الذي يحتوي على البلازميد المجمع بشكل صحيح إلى 200 مل من وسائط LB المكملة بالأمبيسلين واحتضانها في شاكر طوال الليل عند 37 درجة مئوية.
- قم بإجراء إعداد ميدي أو ماكسي لاستخراج الحمض النووي للبلازميد وفقا لتعليمات الشركة المصنعة. قم بتخزين الحمض النووي البلازميد عند -20 درجة مئوية.
- إعداد AAV6 المؤتلف
ملاحظة: يجب إجراء تحضيرين منفصلين ل AAV6: أحدهما ل rAAV6 مع قالب WT HDR والآخر ل rAAV6 مع قالب HDR المتحور. يصف هذا القسم الخطوات اللازمة لإعداد فيروس واحد فقط.- قم بإذابة خلايا HEK293T ، انقل الخلايا إلى 10 مل من DMEM الدافئة مسبقا مع 10٪ FBS و 1٪ P / S و 25 mM HEPES وأجهزة الطرد المركزي عند 350 × جم عند RT لمدة 5 دقائق.
- الخلايا بتركيز 1 × 105 خلايا / مل وانقلها إلى دورق مناسب (على سبيل المثال ، دورق 175 سم2 ). انقل القارورة إلى حاضنة عند 37 درجة مئوية / 5٪ CO2.
ملاحظة: يجب إذابة خلايا HEK293T مسبقا للسماح للخلايا بالتعافي التام والحصول على عدد كاف من الخلايا (11 × 107 خلايا ضرورية لإنتاج فيروس واحد). يوصى باستخدام الخلايا بعد ثلاثة ممرات على الأقل والحفاظ على الخلايا أقل من 20 مقطعا. يجب تقسيم خلايا HEK293T ثلاث مرات في الأسبوع. مع الحفاظ على الخلايا ، يجب ألا تتجاوز 70٪ -80٪ التقاء. يجب أيضا استكمال DMEM المستخدم في إعداد AAV بالفعل ب L-glutamine وبيروفات الصوديوم. - اجمع الخلايا في أنبوب سعة 50 مل وأجهزة طرد مركزي بسرعة 350 × جم عند RT لمدة 5 دقائق.
- قم بتعليق الخلايا في 20 مل من DMEM المكملة ب 10٪ FBS و 1٪ P / S و 25 mM HEPES وعد باستخدام مقياس الدم. استخدم تريبان بلو لاستبعاد الخلايا الميتة.
- بذرة 3 × 106 خلايا في 20 مل من DMEM مكملة ب 10٪ FBS و 1٪ P / S و 25 mM HEPES في دورق 175 سم2 . لإنتاج فيروس واحد ، قم بإعداد ما لا يقل عن أربع قوارير 175 سم2 . احتضان الخلايا عند 37 درجة مئوية / 5٪ CO2 لمدة 3 أيام.
ملاحظة: من الأفضل تنفيذ هذه الخطوة يوم الجمعة لأنها تسمح للخلايا بالتمدد خلال عطلة نهاية الأسبوع. - حصاد وعد خلايا HEK293T.
- لتحضير أحد الفيروسات ، قم بإعداد عشرة أطباق مقاس 150 مم يحتوي كل منها على 11 × 106 HEK293T في 20 مل من DMEM مكملة ب 10٪ FBS و 1٪ P / S و 25 mM HEPES.
- ضع الأطباق في الحاضنة عند 37 درجة مئوية / 5٪ CO2 لمدة 24 ساعة. تخلص بعناية من الوسط القديم واستبدله ب 20 مل من DMEM الخالي من المضادات الحيوية المكمل ب 10٪ FBS و 25 mM HEPES و 1 mM من زبدات الصوديوم. قبل المتابعة ، تحقق من أن التقاء الخلايا لا يزيد عن 80٪.
- قم بإعداد أنبوبين سعة 15 مل يحتويان على خلطات النقل. إلى الأنبوب 1 ، أضف 5 مل من وسط المصل المختزل ، و 60 ميكروغرام من بلازميد HDR المتحور rAAV6 ، و 220 ميكروغرام من pDGM6 (البلازميد المساعد). إلى الأنبوب 2 ، أضف 5 مل من وسط المصل المختزل و 1120 ميكرولتر من محلول بولي إيثيلين أمين 1 مجم / مل (PEI ، كاشف النقل).
- أضف محتويات الأنبوب 2 إلى الأنبوب 1 والدوامة لمدة 30 ثانية. احتضن لمدة 15 دقيقة في RT لضمان التغليف السليم للحمض النووي في مذيل جزيرة الأمير إدوارد.
ملاحظة: لا تحتفظ بالمحلول لأكثر من 20 دقيقة. - أضف بعناية قطرة قطرة 1.1 مل من المحلول إلى كل طبق ووزعه عن طريق الدوران برفق. ضع الأطباق في الحاضنة عند 37 درجة مئوية / 5٪ CO2 لمدة 48 ساعة.
- بعد 48 ساعة ، أضف 250 ميكرولتر من 0.5 M EDTA إلى كل طبق وضع الأطباق في الحاضنة لمدة 10 دقائق. احصد الخلايا عن طريق غسلها من الطبق ونقلها إلى أنبوب طرد مركزي سعة 500 مل أو أنابيب متعددة سعة 50 مل.
- جهاز طرد مركزي عند 2000 × جم لمدة 10 دقائق عند 4 درجات مئوية. تخلص من المادة الطافية. لضمان الإزالة الكاملة للطافي ، يتم تشغيل جهاز الطرد المركزي مرة أخرى عند 2000 × جم لمدة 1 دقيقة عند 4 درجات مئوية.
- تخلص من أي مادة طافية متبقية. أي طافت متبقي يمكن أن يضعف تنقية الفيروس.
- قم بفك الحبيبات عن طريق الدوامة واستخراج الفيروس باستخدام مجموعة تنقية AAV (انظر جدول المواد) وفقا لتعليمات الشركة المصنعة. بدلا من ذلك ، قم بإجراء الاستخراج بواسطة الطرد المركزي الفائق المتدرج لليوديكسانول30,31.
- القسمة على الفيروس المنقى وتخزينها في -80 درجة مئوية. قم بمعايرة AAV وظيفيا أو حدد عيار AAV بواسطة PCR القطيرات الرقمية (ddPCR)32 من أجل تحديد ظروف النقل المثلى.
- كرر هذه العملية لإعداد بلازميد rAAV6 WT HDR.
- معايرة AAV بواسطة ddPCR
- قبل البدء ، اضبط كتل التسخين على 65 درجة مئوية و 98 درجة مئوية.
- استخراج الحمض النووي الفيروسي عن طريق إضافة 15 ميكرولتر من محلول استخراج الحمض النووي إلى 5 ميكرولتر من الفيروس. دوامة لمدة 15 ثانية.
- احتضان لمدة 6 دقائق عند 65 درجة مئوية. دوامة لمدة 15 ثانية. احتضان لمدة 2 دقيقة عند 98 درجة مئوية.
- استخدم الحمض النووي المستخرج (وهو تخفيف 1: 4 للحمض النووي الفيروسي) لتحضير التخفيفات التسلسلية (1: 400 ، 1: 40000 ، 1: 160،000 ، 1: 640،000) مع خالية من النيوكلياز (nf) H2O ل ddPCR.
ملاحظة: يمكن تخزين التخفيفات عند -20 درجة مئوية إذا لم يتم إجراء ddPCR على الفور. - حدد ثلاثة من التخفيفات ل ddPCR. يعتمد اختيار التخفيف المراد استخدامه على تركيز الفيروس. على نحو مفضل ، استخدم ثلاثة تخفيفات مختلفة (على سبيل المثال ، 1: 40,000 و 1: 160,000 و 1: 640,000) للعثور على أفضل تخفيف ضمن حد الكشف عن الجهاز.
- الحفاظ على الكواشف على الجليد أثناء العمل. قم بإعداد مزيج رئيسي (سيتم قياس جميع العينات في نسخ مكررة). لكل تفاعل ، قم بإعداد ما يلي: 12.5 ميكرولتر من ddPCR Supermix للمجسات ، بدون dUTP ، 1.25 ميكرولتر من فحص PrimeTime Std qPCR ، AAV-ITR (انظر جدول المواد) ، و 6.25 ميكرولتر nf-H2O.
- امزج 5 ميكرولتر من الحمض النووي مع 20 ميكرولتر من المزيج الرئيسي. قم بإجراء هذا للتخفيفات 1: 40,000 و 1: 160,000 و 1: 640,000.
- ضع خرطوشة في حامل الخرطوشة. أضف 70 ميكرولتر من زيت توليد القطيرات إلى المجسات في الصف المقوم بالزيت. احرص على عدم توليد أي فقاعات.
- أضف 20 ميكرولتر من حجم العينة في الصف المقوم كعينة. احرص على عدم توليد أي فقاعات.
- أغلق الخرطوشة بالحشية وضعها في مولد القطرات. قم بإنشاء القطرات عن طريق الضغط على زر البدء في الجهاز ، وإزالة الحشية ، ونقل بعناية ، باستخدام ماصة متعددة القنوات ، 40 ميكرولتر من القطرات المتولدة إلى لوحة ذات 96 بئرا. اعمل ببطء لتجنب تدمير القطرات وفقاعات الهواء.
- أغلق اللوحة المكونة من 96 بئرا بورق الثقب (الشريط الأحمر متجها لأعلى) باستخدام مانع تسرب لوحة PCR. ضع اللوحة في جهاز حراري للحرارة.
- اضبط مستوى الصوت على 40 ميكرولتر ، ودرجة حرارة الغطاء على 105 درجة مئوية ، ومعدلات المنحدر على 2 درجة مئوية / ثانية. قم بتشغيل برنامج PCR كما هو موضح في الجدول 2.
- قم بتشغيل قارئ القطيرات قبل 30 دقيقة من الاستخدام. افتح البرنامج على سطح المكتب.
- أدخل تخطيط اللوحة وحدد ABS في علامة تبويب التجربة و ddPCR Supermix في علامة التبويب Supermix. ثم اضغط أولا على PRIME ، متبوعا بالنقر فوق FLUSH SYSTEM على البرنامج لبدء النظام.
- أدخل اللوحة في القارئ. ابدأ التشغيل، وعند الانتهاء، قم بتصدير البيانات كملف CSV لتحليلها لاحقا كجدول بيانات.
ملاحظة: الأرقام التي تم إنشاؤها بواسطة البرنامج هي نسخ الجينوم (GC) لكل ميكرولتر. يجب ضربها في عوامل التخفيف: GC / μL × 5 (تخفيف الحمض النووي في المزيج الرئيسي) × التخفيف الأولي (أي 40000 أو 160000).
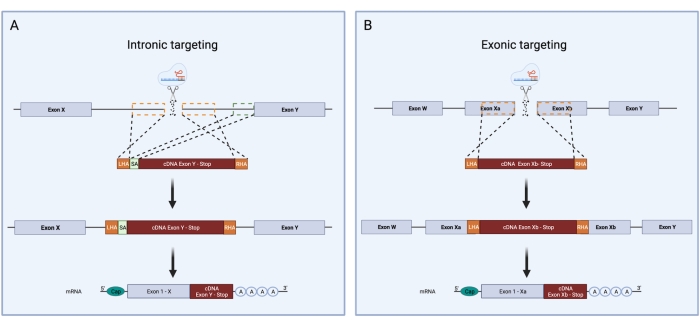
الشكل 3: نظرة عامة تخطيطية على الاستهداف الداخلي والخارجي أثناء تنفيذ تقنية CRISPR/Cas9 HDR. مقارنة تخطيطية بين استراتيجيات الاستهداف intronic و exonic ل CRISPR / Cas9 HDR الضربة القاضية. (أ) أثناء الاستهداف الداخلي، يحدث كسر مزدوج الشريط في إنترون الحمض النووي (DNA). يتكون قالب HDR من LHA وتسلسل cDNA و RHA. يتطلب الاستهداف الداخلي ، بالإضافة إلى ذلك ، وجود متقبل شريحة يحتوي على موقع لصق 3 '، ونقطة الفرع ، وقناة polypyrimidin. هذا يسمح بالربط الصحيح. يمثل المربع المنقط الأخضر المنطقة المقابلة لتسلسل SA. حجم SA هو 150 bp. (B) يعتمد الاستهداف Exonic على إنتاج كسر مزدوج تقطعت به السبل مباشرة في exon. يتكون قالب HDR من LHA وتسلسل cDNA و RHA. تشير المربعات المنقطة البرتقالية إلى المناطق المقابلة ل LHA و RHA. الحجم المثالي ل HAs هو 400 نقطة أساس لكل منهما. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
| المروجين | ||||
| اسم | طول | وصف | ||
| سف | 492 نقطة أساس | الطحال التركيز تشكيل فيروس المروج. مروج قوي للثدييات. معبرا عنها بشكل أساسي | ||
| يو بي سي | 400 نقطة أساس | المروج المستمدة من جين يوبيكويتين C البشري. أعرب بشكل أساسي. | ||
| سي إم في | 508 نقطة أساس | المروج المستمدة من الفيروس المضخم للخلايا. قد تحتوي على منطقة محسن. أعرب بشكل أساسي. مروج قوي للثدييات. يمكن إسكاتها. | ||
| EF-1a | 1182 نقطة أساس | عامل استطالة الترجمة حقيقية النواة البشرية 1 مروج ألفا. أعرب بشكل أساسي. مروج قوي للثدييات. | ||
| إي إف إس | 200-300 نقطة أساس | EF-1 ألفا إنترون شكل قصير أقل | ||
| CAG | 584 نقطة أساس | مروج هجين للثدييات يحتوي على محسن CMV المبكر (C) ، ومروج بيتا أكتين الدجاج (A) ، ومتقبل لصق لجين بيتا جلوبين الأرنب (G). أعرب بشكل أساسي. | ||
| إشارات بولي أدينيل (PolyA) | ||||
| اختصار | طول | وصف | ||
| SV40 بوليا | 82-122 نقطة أساس | فيروس سيميان 40 إشارة بولي أدينيل | ||
| بي جي إتش بوليا | 224 نقطة أساس | إشارة هرمون النمو البقري polyadenylation | ||
| rbGlob بوليا | 56 نقطة أساس | أرنب بيتا غلوبين إشارة polyadenylation | ||
الجدول 1: المروجون وإشارات polyadenylation.
| درج | درجة الحرارة | الوقت | دورات |
| تنشيط الإنزيم | 95 درجة مئوية | 10 دقائق | 1x |
| تمسخ | 94 درجة مئوية | 30 ثانية | 42 ضعفا |
| الصلب | 60 درجة مئوية | 1 دقيقة | |
| امتداد | 72 درجة مئوية | 30 ثانية | |
| تعطيل الإنزيم | 98 درجة مئوية | 10 دقائق | 1x |
| مسك | 4 درجة مئوية | ∞ |
الجدول 2: برنامج PCR بالقطرات الرقمية.
3. تحرير HSPCs
- نقل ونقل HSPCs
- قم بإذابة CD34 + HSPCs ونقل الخلايا إلى 10 مل من RPMI الدافئ مسبقا المكمل بجهاز طرد مركزي P / S. بنسبة 1٪ عند 350 × جم عند RT لمدة 10 دقائق.
- قم بتعليق الخلايا في وسط الاحتفاظ HSPC إلى تركيز 2.5 × 10 5 خلايا / مل واحتضانها عند 37 درجة مئوية /5 ٪ CO2 لمدة 48 ساعة - 72 ساعة.
- حصاد الخلايا في أنبوب 15 مل. عد الخلايا وتحقق من صلاحية الخلية عن طريق استبعاد تريبان الأزرق. بين 2 × 10 5 و5 × 106 خلايا يمكن أن تكون نواة في كفيت واحد. قم بإعداد العدد المناسب من cuvettes اعتمادا على رقم الخلية المحسوب.
- تحضير مجمع RNP. في أنبوب سعة 1.5 مل ، أضف 15 ميكروغرام من Cas9 و 8 ميكروغرام من sgRNA (النسبة المولية 1: 2.5) واحتضانها عند 25 درجة مئوية لمدة 10 دقائق في كتلة تسخين.
- أثناء احتضان مجمع RNP ، قم بالطرد المركزي للخلايا عند 350 × جم لمدة 5 دقائق وتخلص من المادة الطافية.
- تعليق الخلايا في 100 ميكرولتر من محلول nucleofection. امزج الخلايا مع مجمع RNP وانقلها إلى كوفيت.
- اضغط برفق على الكوفيت لإزالة أي فقاعات هواء متبقية قد تكون تشكلت أثناء النقل.
- أدخل الكوفيت في حامل نظام النقل وقم بالكهرباء الخلايا كما هو موضح سابقا في قسم تصميم sgRNA.
- مباشرة بعد التثقيب الكهربائي ، أضف 400 ميكرولتر من وسط الاحتفاظ ب HSPC الدافئ مسبقا بدون P / S وانقل الخلايا باستخدام ماصة نقل دقيقة إلى لوحة زراعة الأنسجة التي تحتوي على 500 ميكرولتر من وسط الاحتفاظ HSPC الدافئ مسبقا بدون P / S. اعتمادا على رقم الخلية ، استخدم لوحة استزراع مناسبة (لوحة 24 أو 12 أو 6 آبار) من أجل الوصول إلى كثافة تتراوح بين 0.25 × 106 و 1 × 106 خلايا / مل. انقل اللوحة إلى الحاضنة عند 37 درجة مئوية / 5٪ CO2.
- قم بإذابة القوارير التي تحتوي على rAAVs المجمدة على الجليد. قم بتحويل الخلايا عن طريق سحب الكمية المثلى من كل rAAV6 إلى تعليق الخلية. قم بإجراء عملية النقل في غضون 20-30 دقيقة بعد التثقيب الكهربائي للحصول على كفاءات نقل عالية.
ملاحظة: يجب تحديد التركيز الأمثل لكل فيروس (فيروس واحد لقالب WT HDR وفيروس منفصل آخر لقالب HDR المتحور) تجريبيا. عادة ، 5000-10000 GC / خلية (على سبيل المثال ، 5 × 109 GC ل 1 × 106 خلايا) يؤدي إلى نقل عالي. - امزج تعليق الخلية برفق مع الماصة. احتضان الخلايا المحولة لمدة 6-8 ساعات عند 37 درجة مئوية / 5٪ CO2. بعد 6-8 ساعات ، اجمع الخلايا في أنبوب وجهاز طرد مركزي بسرعة 350 × جم في RT لمدة 5 دقائق.
- تخلص من الوسط القديم واستبدله بوسط احتفاظ HSPC جديد مسبق التسخين مكمل ب P / S.
- قم بتعليق الخلايا بتركيز يتراوح بين 2.5 × 105-5 × 105 خلايا / مل وانقلها إلى صفيحة زراعة الخلايا المعالجة بالأنسجة (24 أو 12 أو 6 آبار لوحة). احتضان الخلايا لمدة 48 ساعة عند 37 درجة مئوية / 5٪ CO2 قبل الشروع في الفرز.
- فرز التدفق ل HSPCs المهندسة التي تحمل طفرة GOF غير المتجانسة
- احصد الخلايا في أنبوب سعة 15 مل وأجهزة طرد مركزي عند 350 × جم عند RT لمدة 5 دقائق. قم بإزالة المادة الطافية ، وقم بتعليق الخلايا في 1 مل من DPBS التي تحتوي على 0.1٪ BSA ، وأجهزة الطرد المركزي مرة أخرى عند 350 × جم عند RT لمدة 5 دقائق.
- تخلص من المادة الطافية وقم بتعليق الخلايا في حجم مناسب من DPBS + 0.1٪ BSA اعتمادا على رقم الخلية.
ملاحظة: يوصى بتعليق الخلايا بحجم لا يقل عن 200 ميكرولتر ولا يتجاوز 1 × 107 خلايا / مل لتقليل خطر انسداد الفارز. - نقل الخلايا إلى أنبوب FACS معقم مزود بغطاء. أضف 7-AAD أو صبغة صلاحية أخرى (اعتمادا على توافقها مع بروتينات الفلورسنت) إلى معلق الخلية لاستبعاد الخلايا الحية / الميتة.
- فرز الخلايا الحية التي تكون إيجابية مزدوجة لبروتينات مراسل الفلورسنت في أنبوب تجميع يحتوي على 200 ميكرولتر من وسط الاحتفاظ HSPC.
ملاحظة: يجب إضافة عناصر التحكم المناسبة التي تتكون من خلايا تم تحويلها فقط باستخدام AAVs لضمان البوابات المناسبة أثناء إجراء الفرز. - قم بالطرد المركزي للخلايا المصنفة عند 350 × جم عند RT لمدة 5 دقائق وتعليق الخلايا في وسط الاحتفاظ HSPC بتركيز 2.5 × 105 خلايا / مل لمزيد من التوسع في الثقافة أو استخدم الخلايا مباشرة للمقايسات الوظيفية.
4. تأكيد نجاح تحرير الجينات
- استخراج الحمض النووي الجينومي
- قبل البدء ، اضبط كتل التسخين على 65 درجة مئوية و 98 درجة مئوية. حصاد 2 × 10 5 خلايا وجهاز طرد مركزي محرر بالجينوم ومنقى بالفرز بمعدل 350 × جم لمدة5 دقائق.
- استخراج gDNA كما هو موضح سابقا. إما استخدام محلول الحمض النووي مباشرة لتفاعل البوليميراز المتسلسل أو تخزينه عند -20 درجة مئوية.
- داخل وخارج PCR
- تصميم اثنين من الشعير لتضخيم موقع الإدراج 5 '(الشكل 4A): التمهيدي إلى الأمام 1 يستهدف الموضع الجينومي خارج ذراع التماثل الأيسر. التمهيدي عكس 2 يستهدف التسلسل المتكامل.
- بالنسبة لتفاعل البوليميراز المتسلسل الداخل والخارج ، قم بإعداد مزيج PCR التالي لكل تفاعل:
10 ميكرولتر من PCR Master Mix (2x ؛ انظر جدول المواد)
1 ميكرولتر من التمهيدي إلى الأمام 1 (مخزون 10 ميكرومتر)
1 ميكرولتر من عكس التمهيدي 2 (مخزون 10 ميكرومتر)
0.5-1.0 ميكرولتر من الحمض النووي المستخرج
H2O خال من النيوكلياز إلى حجم نهائي يبلغ 20 ميكرولتر
ملاحظة: بالنسبة لأكثر من تفاعل واحد ، ينصح بإعداد مزيج رئيسي يحتوي على تفاعل إضافي واحد لحساب أخطاء سحب العينات. من المهم تضمين عنصر تحكم غير قالب وقالب DNA من عينة معالجة وهمية. - قم بتشغيل تفاعلات PCR باستخدام برنامج الدراجات الحرارية المناسب (انظر تعليمات الشركة المصنعة).
ملاحظة: ينصح أولا باختبار زوج التمهيدي للحصول على درجة حرارة التلدين المثلى. يمكن القيام بذلك عن طريق حساب Tm ثم عن طريق تشغيل PCR باستخدام دراجة حرارية يمكنها أداء درجة حرارة متدرجة. - قم بتشغيل منتجات تفاعل البوليميراز المتسلسل باستخدام سلم الحمض النووي على هلام الأغاروز 1.5٪ عند 100 فولت لمدة 45-60 دقيقة (الشكل 4 ب). ضع الجل على ضوء أزرق أو مصباح للأشعة فوق البنفسجية. استئصال العصابات من هلام.
- استخرج الحمض النووي من العصابات باستخدام مجموعة استخراج هلام الحمض النووي. أرسل عينات تفاعل البوليميراز المتسلسل المستخرجة مع البادئات المناسبة لتسلسل سانجر لتأكيد التكامل الصحيح والسلس ل cDNA المطلوب في موضع الجين الداخلي.
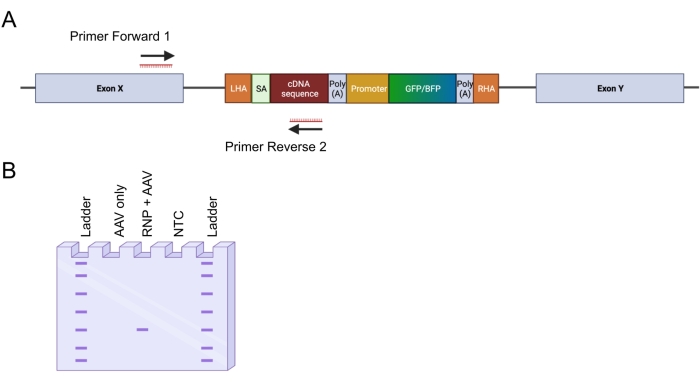
الشكل 4: التحقق من التكامل الجينومي عن طريق تفاعل البوليميراز المتسلسل الداخل والخارج. (أ) تمثيل تخطيطي لاستراتيجية تفاعل البوليميراز المتسلسل الداخلة والخارجة. في الاستراتيجية المصورة ، تم تصميم اثنين من الاشعال. يستهدف التمهيدي الأمامي 1 الموضع الجينومي خارج LHA ، ويستهدف التمهيدي العكسي 2 التسلسل المحسن للكودون. (ب) تمثيل تخطيطي للرحلان الكهربائي بهلام الأغاروز. فقط الخلايا المحررة بنجاح (RNP + AAV) ستولد منتج PCR أثناء تفاعل البوليميراز المتسلسل الداخل والخارج ، في حين أن العينات غير المحررة (AAV فقط) لن تولد منتج PCR. اختصار: NTC = عنصر تحكم غير قالب. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
النتائج
من خلال تطبيق البروتوكول الموصوف أعلاه ، تم إدخال طفرات CALR من النوع 1 غير المتجانسة بشكل متكرر في HSPCs المشتقة من دم الحبل السري. تتكون هذه الطفرة من حذف 52 نقطة أساس في exon 9 (آخر إكسون من CALR) ، مما ينتج عنه إزاحة إطار +1 ، مما يؤدي إلى ترجمة مجال C-terminal جديد موجب الشحنة26,33. لإدخال طفرة CALR في موضع الجين الداخلي ، تم اعتماد استراتيجية استهداف إنترونيكية في المنبع من exon 9 ، لأن هذا من شأنه أن يتحايل على أي تغييرات غير مرغوب فيها في تسلسل الترميز في الحالات التي لم يتم فيها إصلاح DSB الناجم عن Cas9 عبر آلية HDR. في هذه الحالة المحددة ، تم تصميم sgRNA ل intron 7 بسبب توفر تسلسلات عالية على الهدف ومنخفضة خارج الهدف بالاقتران مع أذرع التماثل المواتية (عدم وجود تكرار التسلسل ؛ الشكل 5 أ).
ثم تم تصميم نموذجين للمانحين وتعبئتهما في ناقلات AAV6. من أجل تمكين الربط الصحيح من الإكسونات الداخلية إلى cDNA المتكامل ، تحتوي قوالب المانحين على (i) تسلسل SA بما في ذلك موقع لصق 3 '، ونقطة الفرع ، وقناة polypyrimidine ، (ii) تسلسل cDNA المحسن للكودونات من exons 8-9 ، إما يحتوي على WT (CALR WT) أو التسلسل المتحور (CALRDEL ) بما في ذلك كودون التوقف ، (iii) فيروس سيميان 40 (SV40) إشارة polyA ، (iv) تشفير تسلسلي لبروتين فلوري تحت سيطرة المروج الداخلي المنفصل ، مروج فيروس تشكيل تركيز الطحال (SFFV) ، متبوعا ب (v) إشارة polyA لهرمون النمو البقري (bGH). تم تصميم قالب المانحين الذي يحتوي على CALR WT cDNA ليحتوي على شريط GFP ، بينما تم تصميم قالب المانحين الذي يحتوي على تسلسل CALRDEL cDNA ليحتوي على شريط BFP. كان البناء بأكمله محاطا ب HA يسار ويمين (الشكل 5 أ).
بعد يومين من النقل مع مجمع RNP والنقل مع فيروسات rAAV6 ، تم تحليل الخلايا عن طريق قياس التدفق الخلوي. يمكن اكتشاف أربع مجموعات سكانية رئيسية: (i) الخلايا التي لا تعبر عن GFP ولا BFP ، والتي تمثل الخلايا التي لا تحتوي على تحرير الجينوم القائم على HDR ، (ii) الخلايا الموجبة فقط ل GFP ، والتي تمثل تلك التي دمجت بنية WT فقط ، (iii) الخلايا الإيجابية فقط ل BFP ، والتي تمثل تلك التي دمجت البنية المتحورة فقط ، و (iv) الخلايا الإيجابية المزدوجة GFP و BFP ، تمثل الخلايا التي دمجت كلا من WT والتسلسلات المتحولة (الشكل 5 ب). من أجل الحصول على مجموعات نقية من HSPCs تحمل طفرة CALR من النوع 1 غير المتجانسة ، تم فرز الخلايا الإيجابية المزدوجة عن طريق قياس التدفق الخلوي. تم استخدام HSPCs التي تم فيها طرقها تسلسلين WT كخلايا تحكم (GFP + mCherry + ؛ الشكل 5 ب). البديل الصالح للاستخدام كخلايا تحكم هو HSPCs مع تكامل ثنائي الأليل للبروتينات الفلورية في موضع ملاذ آمن (أي AAVS1 ؛ غير معروض). أشار عد الخلايا عن طريق استبعاد التريبان الأزرق الذي تم إجراؤه على HSPCs التي تم فرزها إلى أن أكثر من 90٪ من الخلايا كانت قابلة للحياة.
تم تأكيد التكامل السلس على الهدف للبنيات من خلال تطبيق استراتيجية تفاعل البوليميراز المتسلسل الداخل والخارج (الشكل 6 أ). في هذه الحالة المحددة ، أجرينا مقطعي تفاعل البوليميراز المتسلسل المنفصلين للداخل والخارج ، أحدهما لتسلسل CALR WT المقطوع (الممر 1 من الرحلان الكهربائي الهلامي في الشكل 6A) والآخر لتسلسل CALRDEL الذي تم طرقه (الممر 2 من الرحلان الكهربائي الهلامي في الشكل 6A). أكد تسلسل سانجر الذي تم إجراؤه على الحمض النووي المستخرج من نطاقات الهلام الإدخال الصحيح لتسلسل WT والتسلسل المتحور في CALRDEL / WT HSPCs (الشكل 6 ب).
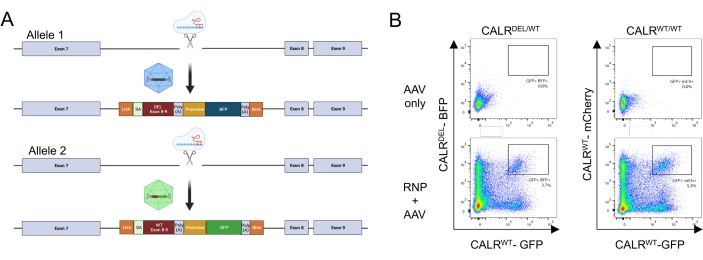
الشكل 5: توليد HSPCs التي تحمل طفرة CALR غير المتجانسة. (أ) مخطط تمثيلي يصور استراتيجية التحرير لإدخال طفرة CALR غير المتجانسة الزيجوت. يستهدف مجمع RNP الإنترون بين exon 7 و exon 8 من جين CALR . اثنان من AAVs ، أحدهما يحتوي على exons المتحورة 8-9 و BFP والآخر يحتوي على WT exons 8-9 و GFP ، سيكون بمثابة قوالب إصلاح المانحين وسيعزز تكامل التسلسل المتحور في أليل واحد ودمج تسلسل WT في الأليل المتبقي. (ب) مخططات قياس التدفق الخلوي التمثيلية التي تصور التعبير عن GFP و BFP أو GFP و mCherry 48 ساعة بعد نقل ونقل HSPCs. الرجاء النقر هنا لعرض نسخة أكبر من هذا الشكل.
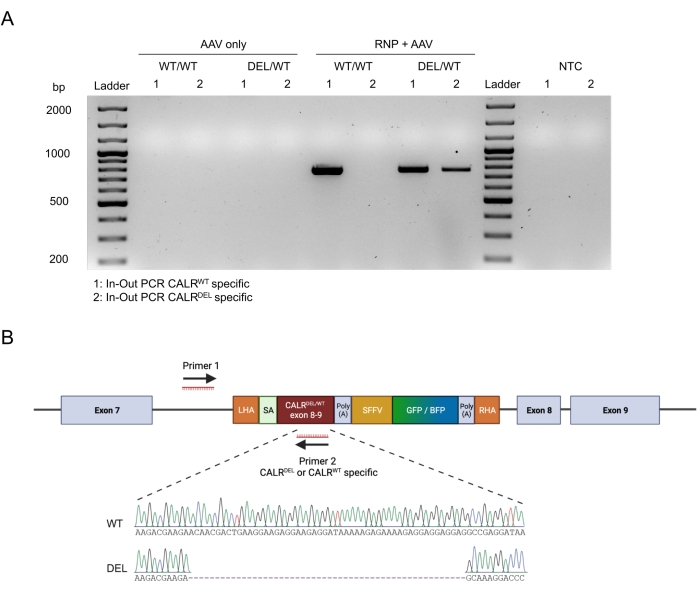
الشكل 6: التحقق من صحة طفرة CALR غير المتجانسة الناجحة في HSPCs. (أ) الرحلان الكهربائي الهلامي من منتجات تفاعل البوليميراز المتسلسل الداخل والخارج الذي يتم إجراؤه على الحمض النووي الجينومي المستخرج من عناصر تحكم AAV و CALR WT / WT و CALRDEL / WT. تم استخدام سلم الحمض النووي 100 bp. اختصار: NTC = عنصر تحكم غير قالب. (ب) نتائج تسلسل سانجر التي تم الحصول عليها من تفاعل البوليميراز المتسلسل الداخل والخارج الذي تم إجراؤه على CALRDEL / WT مما يؤكد التكامل الناجح ل WT والتسلسلات المتحورة. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
Discussion
يمثل التلاعب الجيني الفعال والدقيق ل HSPCs الأولية البشرية فرصة عظيمة لاستكشاف وفهم العمليات التي تؤثر على تكون الدم الطبيعي ، والأهم من ذلك ، التحول اللوكيمي للخلايا المكونة للدم.
في هذا البروتوكول ، تم وصف استراتيجية فعالة لهندسة HSPCs البشرية للتعبير عن طفرات GOF غير المتجانسة المتكررة استفاد هذا الإجراء من تقنية CRISPR / Cas9 ونواقل rAAV6 كمتبرعين لقوالب الحمض النووي لإدخال تسلسل WT والحمض النووي الطافر بدقة في مواقع الجينات الداخلية. يسمح اقتران cDNAs المهندسة (WT والطافرة) ببروتينات مراسل الفلورسنت المنفصلة بإثراء وتتبع الخلايا ذات الحالة غير المتجانسة النهائية.
تقدم هذه الاستراتيجية العديد من المزايا مقارنة بالطرق القائمة على الفيروسات العدسية (LV) المستخدمة بشكل متكرر. تتمثل إحدى المزايا الرئيسية في أن النظام المستند إلى CRISPR / Cas9 يسمح بالتحرير الدقيق في المواقع الداخلية ، مما يؤدي إلى الحفاظ على المروجين الداخليين والعناصر التنظيمية. هذا يؤدي إلى التجانس في التعبير عن الجين المحرر في الخلايا ، وهو هدف يصعب تحقيقه عند استخدام طريقة قائمة على LV. يؤدي نقل الجينات مع ناقلات الجهد المنخفض إلى تكامل شبه عشوائي للجين مع تفضيل المواقع النشطة نسخيا34. يمكن أن يترجم هذا إلى الإفراط في التعبير عن الجين المنقول وعدم التجانس بين الخلايا المحررة ، مما يؤدي في النهاية إلى صعوبات في التحقيق في دور الطفرات والتفاعلات الجينية وتحليلها. الميزة الثانية هي أن النظام الموصوف ، كونه نظام تحرير خاص بالموقع ، يزيل مخاطر الطفرات الإدراجية35.
تسمح استراتيجية المراسل الفلوري المزدوج بالإثراء الدقيق وتتبع الخلايا التي تم تحريرها بنجاح على كلا الأليلين ، حيث يدمج أحد الأليل WT cDNA والأليل الآخر يدمج تسلسل cDNA المتحور. تمثل الخلايا التي تعبر عن مراسل واحد فقط إما التكامل أحادي الأليل فقط أو التكامل ثنائي الأليل لقوالب HDR مع نفس المراسل الفلوري. لا يمكن تمييز كلا السيناريوهين بدقة إلا إذا تم إنتاج الحيوانات المستنسخة المشتقة من خلية واحدة وتحليلها بشكل فردي. ومع ذلك ، فإن HSPCs لديها قدرة تكاثرية محدودة فقط في المختبر ، وعندما يتم الاحتفاظ بها في الثقافة لفترات طويلة من الزمن ، تبدأ HSPCs في التمايز إلى ذرية أكثر نضجا وتفقد قدرتها على التجديد الذاتي والتطعيم. وهذا يجعل اختيار وتوسيع الحيوانات المستنسخة أحادية الخلية التي تؤوي الطفرة غير المتجانسة المرغوبة غير مجدية. يسمح تطبيق استراتيجية البروتين الفلوري المزدوج والإثراء عن طريق قياس التدفق الخلوي للخلايا التي تحمل طفرة متغايرة الزيجوت بتجاوز المشاكل التي تسببها الثقافة الممتدة في المختبر .
في هذا المثال المحدد ، تم إثبات بنجاح أنه يمكن هندسة HSPCs وفرزها بكفاءة من أجل الحصول على مجموعات نقية من HSPCs تحمل طفرة CALRDEL / WT غير المتجانسة الزيجوت.
ومع ذلك ، لا يقتصر هذا النظام على هندسة طفرات إزاحة الإطار غير المتجانسة ، ولكن يمكن أيضا اعتماده بسهولة لإنشاء أنواع أخرى من الطفرات ، بما في ذلك الطفرات الخاطئة وغير المنطقية. من خلال تطبيق مجموعات مختلفة من AAVs التي تحتوي على WT أو تسلسلات متحولة مع بروتينات مراسلة فلورية مختلفة ، يمكن أيضا استخدام هذا النظام لإدخال طفرات متماثلة الزيجوت (النقل المتزامن مع اثنين من rAAVs كلاهما يحمل cDNA الطافر ولكن مراسلين فلوريين مختلفين) أو حتى تصحيح الطفرات (النقل المتزامن مع اثنين من AAVs كلاهما يحمل WT cDNA ولكن مراسلين فلورسنت مختلفين). بالإضافة إلى ذلك ، من المهم الإشارة إلى أن هذه الاستراتيجية لا تقتصر على إدخال طفرات GOF المسرطنة. في الواقع ، يمكن استخدام البروتوكول الموصوف لاستراتيجيات بديلة متعددة بما في ذلك خروج الجينات ، واستبدال الجينات 36,37 ، والضربة القاضية المستهدفة لجينات التحوير (أي مستقبلات المستضد الخيمري)38 ، وحتى لتصحيح الطفرات المسببة للأمراض11,39.
كما ثبت أن استراتيجية الجمع بين CRISPR / Cas9 و AAV6 مع العديد من مراسلي الفلورسنت قابلة للتطبيق في العديد من أنواع الخلايا الأخرى بما في ذلك الخلايا التائية والخلايا المتغصنة البلازمية والخلايا الجذعية المستحثة متعددة القدرات والخلايا الجذعية العصبية والخلايا الجذعية لمجرى الهواء 24،38،40،41،42،43،44 . يمكن تنفيذ هذه الاستراتيجية لإنتاج الخلايا التائية لمستقبلات المستضد الخيمري (CAR) الفائقة. على سبيل المثال ، تم نشره مؤخرا أن الضربة القاضية بوساطة CRISPR / Cas9 لجين TGFBR2 في الخلايا التائية CAR T تزيد بشكل كبير من وظيفتها في البيئة الدقيقة الغنية بالورم TGF-βالقمعية 45. يمكن أن يوفر مثل هذا النهج بروتوكولا من خطوة واحدة لكل من هندسة الخلايا التائية للتعبير عن CAR وضرب جين TGFBR2 عن طريق إدخال CAR على وجه التحديد في كلا أليلات جين TGFBR2. علاوة على ذلك ، يمكن أن يكون هذا النهج مفيدا أيضا في توليد خلايا CAR T العالمية من خلال دمج CAR في جين ثابت ألفا لمستقبلات الخلايا التائية (TRAC)46,47.
لزيادة قابلية التكاثر وضمان التحرير الفعال للخلايا ، يجب الاهتمام ببعض الاعتبارات المهمة. تكمن النقاط الحاسمة الرئيسية لضمان التحرير الناجح للخلايا في (i) اختيار sgRNA ، (ii) تصميم قالب HDR ، و (iii) إنتاج rAAV6.
يعد اختيار sgRNA جيد الأداء أمرا بالغ الأهمية لأنه سيحدد الحد الأقصى لعدد الأليلات التي يمكن دمج قالب HDR فيها. نظرا للعديد من البرامج المتوفرة الآن ، تم تبسيط البحث عن sgRNAs المرشحة. من خلال تحديد منطقة الاهتمام ، يمكن للبرنامج اقتراح سلسلة من sgRNAs مع درجة على الهدف ودرجة خارج الهدف تشير إلى فرص التحرير في الموضع المطلوب والمواقع غير المرغوب فيها ، على التوالي. يتم حساب هذه الدرجات بناء على نماذج التسجيل المنشورة مسبقا48,49. على الرغم من أن هذه نقطة انطلاق جيدة لاختيار sgRNA جيد الأداء ، إلا أن أداء sgRNA يحتاج إلى تأكيد لأن أدائه المتوقع في السيليكو لا يتوافق دائما مع sgRNA الفعال في المختبر. لذلك ، يوصى بشدة بتصميم واختبار ثلاثة sgRNAs على الأقل لزيادة فرص العثور على أفضل sgRNA. بمجرد تحديد sgRNA حقيقي جيد الأداء ، يقترح المضي قدما في تصميم قالب HDR.
يجب أخذ الاحتياطات في الاعتبار عند تصميم قالب HDR. يجب أن يمتد كل من ذراعي التماثل الأيسر والأيمن (LHA و RHA ، على التوالي) على 400 نقطة أساس في اتجاه المنبع والمصب لموقع قطع sgRNA ، على التوالي ، حيث يمكن أن يؤدي HAs الأقصر إلى انخفاض ترددات HDR. يعتمد حجم cDNA الذي يمكن إدخاله عبر HDR على قدرات التعبئة والتغليف ل AAVs ، والتي تبلغ حوالي 4.7 كيلو بايت. نظرا للعناصر العديدة الإلزامية داخل قالب HDR (LHA ، RHA ، SA ، PolyA ، المروج ، وتسلسل المراسل الفلوري) ، فإن المساحة المتبقية ل cDNA المتحور أو WT محدودة. هذا لا يمثل مشكلة إذا كانت الطفرة المطلوبة تقع بالقرب من نهاية 3 'من الجين أو في الجينات ذات CDS القصير الشامل. ومع ذلك ، في الحالات التي تقع فيها الطفرة بالقرب من جانب بداية النسخ (TSS) للجينات ذات CDS الطويل (يتجاوز مساحة التعبئة المتبقية من AAV) ، قد لا يكون هذا النهج الموصوف ممكنا. للتحايل على هذه المشكلة ، تم مؤخرا تطوير استراتيجية تعتمد على تقسيم قالب HDR إلى اثنين من AAVs بواسطة Bak وزملاؤه. تعتمد هذه الاستراتيجية على تكاملين منفصلين بوساطة HDR للحصول على التكامل السلس النهائي للجينالكبير 50.
تعد جودة الفيروس وعياره من العوامل الإضافية التي يمكن أن تصنع أو تكسر هندسة الجينوم الناجحة للخلايا. للحصول على عائد مثالي ، من المهم عدم السماح ل HEK293T بالوصول إلى نقطة التقاء كاملة مع الحفاظ عليها في الثقافة. من الناحية المثالية ، يجب تقسيم خلايا HEK293T عند الوصول إلى التقاء 70٪ -80٪. بالإضافة إلى ذلك ، لا ينبغي استزراع HEK293T لفترات طويلة من الزمن لأن هذا يمكن أن يقلل من قدرتها على إنتاج الفيروس. تحتاج خلايا HEK293T الجديدة إلى إذابة بعد 20 مقطعا. يعد الحصول على عيار عالي من الفيروسات أمرا مهما لزيادة كفاءة التجارب وقابليتها للتكرار. سوف يترجم التتر الفيروسي المنخفض إلى كميات كبيرة من محلول rAAV المطلوب لنقل HSPCs. كقاعدة عامة ، يجب ألا يتجاوز محلول rAAV المضاف إلى الخلايا النووية 20٪ من الحجم الكلي لوسط الاحتفاظ HSPC. قد تؤدي الكميات الكبيرة من محلول AAV إلى زيادة موت الخلايا ، وانخفاض التكاثر ، وضعف كفاءة النقل. في حالة انخفاض عيار الفيروس ، يوصى بزيادة تركيز الفيروس.
باختصار ، يقدم هذا البروتوكول نهجا قابلا للتكرار لمعالجة HSPCs البشرية بدقة وكفاءة من خلال الاستخدام المتزامن لقوالب المانحين CRIPSR / Cas9 و rAAV6 مع مراسلين فلورسنت مزدوجين إضافيين. وقد أثبت هذا النهج أنه أداة عظيمة في دراسة بيولوجيا الخلايا الجذعية المكونة للدم الطبيعية والمساهمات التي تقدمها الطفرات في تكوين الكريات البيض.
Disclosures
ليس لدى المؤلفين ما يكشفون عنه.
Acknowledgements
يتم دعم هذا العمل من خلال منح من صندوق العلوم النمساوي (FWF ؛ رقم P32783 و I5021) إلى A.R. كما يتم توفير تمويل إضافي ل A.R. من قبل الجمعية النمساوية للطب الباطني (زمالة جوزيف سكودا) ، والجمعية النمساوية لأمراض الدم والأورام (OeGHO; منحة البحوث السريرية) ، و MEFOgraz. T.K. هو زميل خاص في جمعية اللوكيميا وسرطان الغدد الليمفاوية.
Materials
| Name | Company | Catalog Number | Comments |
| 175 cm2 Cell Culture Flask, Vent Cap, TC-treated | Corning | 431080 | |
| 150 mm x 25 mm dishes | Corning | 430599 | |
| 293T | DSMZ | ACC 635 | https://www.dsmz.de/collection/catalogue/details/culture/ACC-635 |
| 4D Nucleofector Core Unit | Lonza | - | For nucleofection of human HSPCs use the DZ-100 program. |
| 4D Nucleofector X Unit | Lonza | - | |
| 500 ml Centrifuge Tube | Corning | 431123 | |
| 7-AAD | BD Biosciences | 559925 | |
| AAVpro Purification Kit | Takara | 6666 | |
| Alt-R S.p. Cas9 Nuclease V3 | Integrated DNA Technologies (IDT) | 1081058 | |
| Avanti JXN-30 Ultracentrifuge | Beckman Coulter | - | |
| Benchling sgRNA design tool | Online tool for sgRNA design: http://www.benchling.com/crispr | ||
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A7906-100G | |
| C1000 Touch Thermal Cycler | Bio-Rad | - | |
| Chemically modified synthetic sgRNA | Synthego | Website: https://www.synthego.com/products/crispr-kits/synthetic-sgrna Sequence for the sgRNA targeting intron 7 of CALR: 5’-CGCCTGTAATCCTCGCCCAG-3’ An 80 nucleotide SpCas9 scaffold is added to the 20 nucleotide RNA sequence to complete the sgRNA. Chemical modifications of 2'-O-Methyl are added to the first and last 3 bases and 3' phosphorothioate bonds are added in the first 3 and last 2 bases. *Alternatively chemically modified synthetic sgRNAs can be acquired from IDT (https://eu.idtdna.com/site/order/oligoentry/index/crispr) and Trilink (https://www.trilinkbiotech.com/custom-oligos) | |
| CHOPCHOP sgRNA design tool | Online tool for sgRNA design: http://chopchop.cbu.uib.no | ||
| Costar 24-well Clear TC-treated Multiple Well Plates | Corning | 3526 | |
| CRISPick sgRNA design tool | Online tool for sgRNA design: https://portals.broadinstitute.org/gppx/crispick/public | ||
| CRISPOR sgRNA design tool | Online tool for sgRNA design: http://crispor.tefor.net | ||
| ddPCR 96-Well Plates | Bio-Rad | 12001925 | |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863024 | |
| DG8 Cartridges for QX200/QX100 Droplet Generator | Bio-Rad | 1864008 | |
| DG8 Gaskets for QX200/QX100 Droplet Generator | Bio-Rad | 1863009 | |
| DreamTaq Green PCR Master Mix (2X) | Thermo Scientific | K1081 | |
| Droplet Generation Oil for Probes | Bio-Rad | 1863005 | |
| Dulbecco’s Modified Eagle Medium (DMEM) with high glucose | Sigma-Aldrich | D6429-6X500ML | |
| Dulbecco’s Phosphate Buffered Saline (DPBS) | Sigma-Aldrich | D8537-500ML | |
| FACSAria Fusion | BD Biosciences | - | |
| Falcon 5 mL Round Bottom | Corning | 352054 | |
| Fetal Bovine Serum (FBS) Good Forte (heat inactivated), 500 ml | Pan Biotech | P40-47500 | |
| FlowJo 10.8.0 | BD Biosciences | - | |
| GenAgarose L.E. | Inno-train | GX04090 | |
| GeneRuler 100 bp Plus DNA Ladder | Thermo Scientific | SM0321 | |
| Gibson Assembly Master Mix | New England Biolabs Inc. (NEB) | E2611L | |
| HEK293T | |||
| HEPES solution | Sigma-Aldrich | H0887-100ML | |
| ICE | Synthego | https://ice.synthego.com | |
| IDT codon optimization tool | IDT | https://www.idtdna.com/pages/tools/codon-optimization-tool | |
| IDT sgRNA design tool | Online tool for sgRNA design: https://www.idtdna.com/site/order/designtool/index/CRISPR_CUSTOM | ||
| LB Broth (Lennox) EZMix powder microbial growth medium | Sigma-Aldrich | L7658-1KG | |
| LB Broth with agar (Lennox) EZMix powder microbial growth medium | Sigma-Aldrich | L7533-1KG | |
| Midori Green Advance | Nippon Genetics | MG04 | |
| Monarch Plasmid Miniprep Kit | NEB | T1010L | |
| Monarch DNA Gel Extraction Kit | NEB | T1020L | |
| NEB 5-alpha Competent E. coli (High Efficiency) | NEB | C2987U | |
| Nuclease-Free Water, 5X100 ml | Ambion | AM9939 | |
| NucleoBond Xtra Midi | Macherey-Nagel | 740410 | |
| Opti-MEM, Reduced Serum Medium, 500 ml | Gibco | 31985070 | |
| P3 Primary Cell 4D-Nucleofetor X Kit L | Lonza | V4XP-3024 | The Lonza Primary P3 solution is supplied as a 2.25 mL P3 Primary Cell Nucleofector Solution and 0.5 mL Supplement 1. To reconstitute, add the Supplement 1 to the P3 Primary Cell Nucleofector Solution and mix. |
| pAAV-MCS2 | Addgene | 46954 | |
| PCR Plate Heat Seal Foil, pierceable | Bio-Rad | 1814040 | |
| pDGM6 | Addgene | 110660 | |
| Penicillin-Streptomycin (P/S) | Gibco | 15140122 | |
| Polyethylenimine (PEI) | Polysciences | 23966 | Add 50 mL of PBS 4.5 pH (made with HCl) to 50 mg of PEI in a tube. Dissolve by placing the tube in a 70°C water bath and vortexing every 10 minutes until the solution is dissolved. After the solution has reached RT, filter sterilze through a 0.22 μm filter, make 1120 μL aliquots, and store at -80°C. |
| Polystyrene Test Tube, with Snap Cap | |||
| Primers | Eurofins | - | Primers were ordered from Eurofins (eurofinsgenomics.eu) as unmodified salt free custom oligos. The primers were designed by using PRIMER-Blast (https://www.ncbi.nlm.nih.gov/tools/primer-blast/) Primer 1 Fwd: AAGTGATCCGTTCGCCATGAC; Primer 2 Rev CALR WT specific: ACGTCCTCTTCCTCGTCCTC; Primer 2 Rev CALR DEL specific: CCAACCCTGGAGACACGCTTC |
| PrimeTime qPCR Primer Assay | IDT | - | PrimeTime qPCR Probe Assays (1 probe/2 primers) that can be ordered from IDT (https://eu.idtdna.com/site/order/qpcr/assayentry). Scale: Std - qPCR Assay 500 reactions; Primer 1 Forward (5'-3') : GGAACCCCTAGTGATGGAGTT; Primer 2 Reverse (5'-3'): CGGCCTCAGTGAGCGA; Probe (5'-3'): CACTCCCTCTCTGCGCGCTCG; 5' Dye/3' Quencher: FAM/ZEN/IBFQ; Primer to probe ratio: 3.6 |
| PX1 PCR Plate Sealer | Bio-Rad | 1814000 | |
| QuantaSoft Software | Bio-Rad | ||
| Quick Extract DNA Extraction Solution | Lucigen | QE0905T | |
| QX200 Droplet Generator | Bio-Rad | 1864002 | |
| QX200 Droplet Reader | Bio-Rad | ||
| Recombinant human Flt3-ligand | Peprotech | 300-19 | |
| Recombinant human IL-6 | Peprotech | 200-06 | |
| Recombinant Human SCF | Peprotech | 300-07 | |
| Recombinant Human TPO | Peprotech | 300-18 | |
| RPMI 1640 | Sigma-Aldrich | R8758-6X500ML | |
| SnapGene | Dotmatics | Molecular cloning software https://www.snapgene.com *Alternatively also Benchling (https://www.benchling.com) and Geneious (https://www.geneious.com) can be used. | |
| Soc outgrowth medium | NEB | B9020S | |
| Sodium-butyrate | Sigma-Aldrich | B5887-1G | |
| Stem Regenin 1 (SR1) | Biogems | 1224999 | |
| StemSpan SFEM II | STEMCELL Technologies | 9655 | |
| TAE Buffer (Tris-acetate-EDTA) 50X | Thermo Scientific | B49 | |
| TIDE | http://shinyapps.datacurators.nl/tide/ | ||
| Trypan blue 0.4% | Sigma-Aldrich | T8154-100ML | |
| TrypLE (with phenol red), 500 ml | Thermo Scientific | 16605-028 | |
| UltraPure 0.5: EDTA, pH 8.0, 100 ml | Thermo Scientific | 15575-038 | |
| UM171 | STEMCELL Technologies | 72914 | |
| Vector Builder codon optimization tool | Vector Builder | https://en.vectorbuilder.com/tool/codon-optimization.html |
References
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Gillmore, J. D., et al. CRISPR-Cas9 in vivo gene editing transthyretin amyloidosis. New England Journal of Medicine. 385 (6), 493-502 (2021).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and β-thalassemia. New England Journal of Medicine. 384 (3), 252-260 (2021).
- Stadtmauer, E. A., et al. CRISPR-engineered T cells in patients with refractory cancer. Science. 367 (6481), (2020).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Heyer, W. D., Ehmsen, K. T., Liu, J. Regulation of homologous recombination in eukaryotes. Annual Review of Genetics. 44, 113-139 (2010).
- Veldwijk, M. R., et al. Pseudotyped recombinant adeno-associated viral vectors mediate efficient gene transfer into primary human CD34+ peripheral blood progenitor cells. Cytotherapy. 12 (1), 107-112 (2010).
- Song, L., et al. High-efficiency transduction of primary human hematopoietic stem cells and erythroid lineage-restricted expression by optimized AAV6 serotype vectors in vitro and in a murine xenograft model in vivo. PLoS One. 8 (3), 58757 (2013).
- Dever, D. P., et al. CRISPR/Cas9 β-globin gene targeting in human haematopoietic stem cells. Nature. 539 (7629), 384-389 (2016).
- Drost, J., et al. Sequential cancer mutations in cultured human intestinal stem cells. Nature. 521 (7550), 43-47 (2015).
- Jan, M., et al. Clonal evolution of preleukemic hematopoietic stem cells precedes human acute myeloid leukemia. Science Translational Medicine. 4 (149), (2012).
- Corces-Zimmerman, M. R., Hong, W. J., Weissman, I. L., Medeiros, B. C., Majeti, R. Preleukemic mutations in human acute myeloid leukemia affect epigenetic regulators and persist in remission. Proceedings of the National Academy of Sciences of the United States of America. 111 (7), 2548-2553 (2014).
- Jaiswal, S., et al. Clonal hematopoiesis and risk of atherosclerotic cardiovascular disease. New England Journal of Medicine. 377 (2), 111-121 (2017).
- Genovese, G., et al. Clonal hematopoiesis and blood-cancer risk inferred from blood DNA sequence. New England Journal of Medicine. 371 (26), 2477-2487 (2014).
- Papaemmanuil, E., et al. Genomic classification and prognosis in acute myeloid leukemia. New England Journal of Medicine. 374 (23), 2209-2221 (2016).
- Cancer Genone Atlas Research Network. Genomic and epigenomic landscapes of adult de novo acute myeloid leukemia. New England Journal of Medicine. 368 (22), 2059-2074 (2013).
- Ball, M., List, A. F., Padron, E. When clinical heterogeneity exceeds genetic heterogeneity: thinking outside the genomic box in chronic myelomonocytic leukemia. Blood. 128 (20), 2381-2387 (2016).
- Varmus, H. E. The molecular genetics of cellular oncogenes. Annual Review of Genetics. 18, 553-612 (2003).
- Cox, D. B. T., Platt, R. J., Zhang, F. Therapeutic genome editing: Prospects and challenges. Nature Medicine. 21 (2), 121-131 (2015).
- Tothova, Z., et al. Multiplex CRISPR/Cas9-based genome editing in human hematopoietic stem cells models clonal hematopoiesis and myeloid neoplasia. Cell Stem Cell. 21 (4), 547-555 (2017).
- Mandal, P. K., et al. Efficient ablation of genes in human hematopoietic stem and effector cells using CRISPR/Cas9. Cell Stem Cell. 15 (5), 643-652 (2014).
- Bak, R. O., Dever, D. P., Porteus, M. H. CRISPR/Cas9 genome editing in human hematopoietic stem cells. Nature Protocols. 13 (2), 358-376 (2018).
- Foßelteder, J., et al. Human gene-engineered calreticulin mutant stem cells recapitulate MPN hallmarks and identify targetable vulnerabilities. Leukemia. , (2023).
- Nangalia, J., et al. Somatic CALR mutations in myeloproliferative neoplasms with nonmutated JAK2. New England Journal of Medicine. 369 (25), 2391-2405 (2013).
- Merlinsky, T. R., Levine, R. L., Pronier, E. Unfolding the role of calreticulin in myeloproliferative neoplasm pathogenesis. Clinical Cancer Research. 25 (10), 2956-2962 (2019).
- Belčič Mikič, T., Pajič, T., Zver, S., Sever, M. The contemporary approach to CALR-positive myeloproliferative neoplasms. International Journal of Molecular Sciences. 22 (7), 3371 (2021).
- How, J., Hobbs, G. S., Mullally, A. Mutant calreticulin in myeloproliferative neoplasms. Blood. 134 (25), 2242-2248 (2019).
- Grieger, J. C., Choi, V. W., Samulski, R. J. Production and characterization of adeno-associated viral vectors. Nature Protocols. 1 (3), 1412-1428 (2006).
- Zolotukhin, S., et al. Recombinant adeno-associated virus purification using novel methods improves infectious titer and yield. Gene Therapy. 6 (6), 973-985 (1999).
- Aurnhammer, C., et al. Universal real-time PCR for the detection and quantification of adeno-associated virus serotype 2-derived inverted terminal repeat sequences. Human Gene Therapy Methods. 23 (1), 18-28 (2011).
- Klampfl, T., et al. Somatic mutations of calreticulin in myeloproliferative neoplasms. New England Journal of Medicine. 369 (25), 2379-2390 (2013).
- Bulcha, J. T., Wang, Y., Ma, H., Tai, P. W. L., Gao, G. Viral vector platforms within the gene therapy landscape. Signal Transduction and Targeted Therapy. 6 (1), 1-24 (2021).
- Montini, E., et al. The genotoxic potential of retroviral vectors is strongly modulated by vector design and integration site selection in a mouse model of HSC gene therapy. The Journal of Clinical Investigation. 119 (4), 964-975 (2009).
- Vaidyanathan, S., et al. Targeted replacement of full-length CFTR in human airway stem cells by CRISPR-Cas9 for pan-mutation correction in the endogenous locus. Molecular Therapy. 30 (1), 223-237 (2022).
- Cromer, M. K., et al. Gene replacement of α-globin with β-globin restores hemoglobin balance in β-thalassemia-derived hematopoietic stem and progenitor cells. Nature Medicine. 27 (4), 677-687 (2021).
- Wiebking, V., et al. Genome editing of donor-derived T-cells to generate allogenic chimeric antigen receptor-modified T cells: Optimizing αβ T cell-depleted haploidentical hematopoietic stem cell transplantation. Haematologica. 106 (3), 847-858 (2021).
- Wilkinson, A. C., et al. Cas9-AAV6 gene correction of beta-globin in autologous HSCs improves sickle cell disease erythropoiesis in mice. Nature Communications. 12 (1), 1-9 (2021).
- Dever, D. P., et al. CRISPR/Cas9 genome engineering in engraftable human brain-derived neural stem cells. iScience. 15, 524-535 (2019).
- Laustsen, A., et al. Interferon priming is essential for human CD34+ cell-derived plasmacytoid dendritic cell maturation and function. Nature Communications. 9 (1), 1-14 (2018).
- Bak, R. O., et al. Multiplexed genetic engineering of human hematopoietic stem and progenitor cells using CRISPR/Cas9 and AAV6. eLife. 6, 27873 (2017).
- Nakauchi, Y., et al. The cell type-specific 5hmC landscape and dynamics of healthy human hematopoiesis and TET2-mutant preleukemia. Blood Cancer Discovery. 3 (4), 346-367 (2022).
- Vaidyanathan, S., et al. selection-free gene repair in airway stem cells from cystic fibrosis patients rescues CFTR function in differentiated epithelia. Cell Stem Cell. 26 (2), 161-171 (2020).
- Tang, N., et al. TGF-β inhibition via CRISPR promotes the long-term efficacy of CAR T cells against solid tumors. JCI Insight. 5 (4), 133977 (2020).
- Georgiadis, C., et al. Long terminal repeat CRISPR-CAR-coupled "universal" T cells mediate potent anti-leukemic effects. Molecular Therapy. 26 (5), 1215-1227 (2018).
- Ren, J., et al. Multiplex genome editing to generate universal CAR T cells resistant to PD1 inhibition. Clinical Cancer Research. 23 (9), 2255-2266 (2017).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Doench, J. G., et al. Optimized sgRNA design to maximize activity and minimize off-target effects of CRISPR-Cas9. Nature Biotechnology. 34 (2), 184-191 (2016).
- Bak, R. O., Porteus, M. H. CRISPR-mediated integration of large gene cassettes using AAV donor vectors. Cell Reports. 20 (3), 750-756 (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved