Method Article
ヒト造血幹細胞および前駆細胞における発癌性ヘテロ接合性機能獲得変異の改変
要約
造血幹細胞生物学および造血悪性腫瘍をよりよく研究するためには、造血幹細胞および前駆細胞(HSPC)の体細胞変異を忠実にモデル化するための新しい戦略が必要です。ここでは、CRISPR/Cas9とデュアルrAAVドナー形質導入の使用を組み合わせることにより、HSPCにおけるヘテロ接合機能獲得変異をモデル化するためのプロトコルについて説明します。
要約
その生涯を通じて、造血幹および前駆細胞(HSPC)は体細胞変異を獲得します。これらの変異のいくつかは、増殖や分化などのHSPC機能特性を変化させ、それによって血液悪性腫瘍の発症を促進します。HSPCの効率的かつ正確な遺伝子操作は、再発性の体細胞突然変異の機能的影響をモデル化し、特徴付け、よりよく理解するために必要です。突然変異は遺伝子に有害な影響を及ぼし、機能喪失(LOF)をもたらすか、まったく対照的に、機能を強化したり、機能獲得(GOF)と呼ばれる特定の遺伝子の新しい特性を引き起こしたりする可能性があります。LOF変異とは対照的に、GOF変異はほぼ独占的にヘテロ接合方式で発生します。現在のゲノム編集プロトコルでは、個々の対立遺伝子の選択的標的化はできず、ヘテロ接合型のGOF変異をモデル化する能力を妨げています。ここでは、効率的なDNAドナーテンプレート導入のために、CRISPR/Cas9を介した相同性指向修復と組換えAAV6技術を組み合わせることにより、ヒトHSPCにおけるヘテロ接合型GOFホットスポット変異を設計する方法に関する詳細なプロトコルを提供します。重要なことに、この戦略は、ヘテロ接合的に編集されたHSPCの追跡と精製を可能にするために、デュアル蛍光レポーターシステムを利用しています。この戦略は、GOF変異がHSPCの機能にどのように影響するか、および血液悪性腫瘍への進行を正確に調査するために使用できます。
概要
クラスター化された規則的に間隔を空けた短い回文反復(CRISPR)/ Cas9技術の開発により、新しい非常に強力な機器が科学者のツールキットに追加されました。この技術はゲノムの精密なエンジニアリングを可能にし、研究目的(Hsu et al.1でレビュー)だけでなく、最近では臨床現場への変換にも成功していることが証明されています2,3,4。CRISPR/Cas9編集戦略は、Cas9タンパク質とシングルガイドRNA(sgRNA)の活性に依存しています5,6,7。宿主細胞では、Cas9タンパク質はsgRNA配列に相補的なDNAの特定の部位に誘導され、DNA二本鎖切断(DSB)を導入します。DSBが生成されると、非相同末端結合(NHEJ)と相同性指向修復(HDR)の2つの主要な競合する修復メカニズムが発生する可能性があります。NHEJはエラーが発生しやすく、主に挿入と欠失(インデル)につながる修復メカニズムですが、姉妹染色分体を修復テンプレートとして使用するHDRは非常に正確ですが、細胞周期のSまたはG2期に限定されます8。ゲノム工学では、Cas9誘導性DSBの両DNA末端と同一の相同性アームに隣接するドナーテンプレートを提供することにより、HDRをDNAの標的修飾に利用できます(図1)。HDR に使用されるドナーテンプレートのタイプは、編集効率に大きな影響を与える可能性があります。ヒトHSPCにおける遺伝子工学に関して、アデノ随伴ウイルス血清型6(AAV6)は、一本鎖DNA鋳型の送達のための優れた媒体であることが最近記載されている9、10。
CRISPR/Cas9ゲノム工学は、有害な変異を修正するために治療的に使用することができます11が、癌の発生をモデル化するためにDNAに病原性突然変異を導入するために利用することもできます12。白血病などの血液がんは、健康なHSPCにおける体細胞変異の順次獲得を介して発症します13,14。初期の遺伝的事象はクローン増殖の利点をもたらし、不確定な可能性(CHIP)のクローン造血をもたらします15,16。突然変異のさらなる獲得は、最終的に白血病の形質転換と病気の発症につながります。体細胞変異は、自己複製、生存、増殖、および分化を制御する遺伝子に見られます17。
ゲノムエンジニアリングを介して個々の突然変異を健康なHSPCに導入することで、この段階的な白血病発生プロセスを正確にモデル化することができます。急性骨髄性白血病(AML)18,19などの骨髄性新生物に見られる再発性変異の数が限られているため、この疾患はゲノム工学ツールを使用して再現するのに特に適しています。
体細胞変異は、一方の対立遺伝子(単対立遺伝子/ヘテロ接合型変異)または両方の対立遺伝子(双対立遺伝子/ホモ接合型変異)にのみ出現し、遺伝子の機能に大きな影響を与える可能性があり、機能喪失(LOF)または機能獲得(GOF)を引き起こす可能性があります。LOF変異は、遺伝子のLOFの低下(一方の対立遺伝子が影響を受ける場合)または完全な(両方の対立遺伝子が影響を受ける場合)をもたらすが、GOF変異は、遺伝子の活性化または新規機能の増加をもたらす。GOF変異は、典型的にはヘテロ接合型20である。
重要なことに、接合性(ヘテロ接合対ホモ接合)は、突然変異を忠実にモデル化する試みに大きな影響を及ぼします。したがって、遺伝子の1つの対立遺伝子のみの標的操作は、ヘテロ接合ホットスポットGOF変異を設計するために必要です。エラーが発生しやすいNHEJは、さまざまな長さのインデル21につながり、さまざまな予測不可能な生物学的結果につながる可能性があります。しかし、NHEJはDSBの導入後に細胞によって採用された主要な修復プログラムであるため、HSPCを操作するために現在使用されているほとんどのCRISPR/Cas9プラットフォームでは、遺伝的転帰を正確に予測することができません22,23。対照的に、CRISPR/Cas9を介した二本鎖切断(DSB)の導入と、HDRを介したゲノムエンジニアリングのための組換えアデノ随伴ウイルス(rAAV)ベクターベースのDNAドナーテンプレートの使用を組み合わせることで、ヒトHSPCにおける変異の対立遺伝子特異的挿入が可能になります11,24。変異体と野生型(WT)配列の同時統合を、個々の対立遺伝子上の異なる蛍光レポーターと組み合わせて実行して、ヘテロ接合遺伝子型を選択できます(図2)。この戦略は、HSPCの機能、疾患の開始、および進行に対する再発性、白血病性、ヘテロ接合性のGOFホットスポット変異の影響を正確に特徴付けるための強力なツールとして活用できます。
この記事では、初代ヒトHSPCにおける反復変異ヘテロ接合型GOF変異の効率的なエンジニアリングのための詳細なプロトコルを提供します。この戦略は、CRISPR/Cas9とデュアルAAV6形質導入の使用を組み合わせて、ヘテロ接合型GOF変異の将来の生成のためのWTおよび変異DNAドナーテンプレートを提供します。一例として、カルレティキュリン(CALR)遺伝子における再発性1型変異(52bp欠失)の工学が図25に示される。CALRのエクソン9におけるヘテロ接合型GOF変異は、本態性血小板血症(ET)や原発性骨髄線維症(PMF)などの骨髄増殖性疾患に再発見られます26。CALRは小胞体常在タンパク質であり、主に新規合成タンパク質の折り畳み過程における品質管理機能を有する。その構造は、タンパク質のシャペロン機能に関与するアミノ(N)末端ドメインとプロリンリッチPドメイン、およびカルシウム貯蔵と調節に関与するCドメインの3つの主要なドメインに分けることができます27,28。CALR変異は+1フレームシフトを引き起こし、新規の伸長したC末端の転写と小胞体(ER)保持シグナル(KDEL)の喪失につながります。変異型CALRはトロンボポエチン(TPO)受容体に結合することが示されており、それによって増殖の増加を伴うTPO非依存性シグナル伝達をもたらす29。
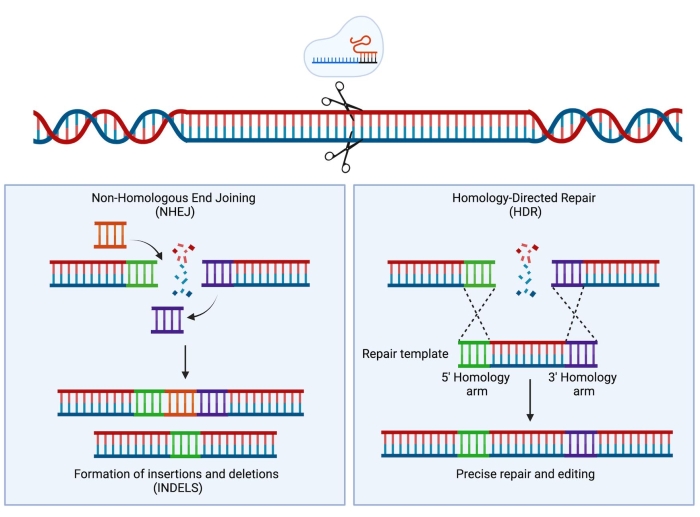
図1:NHEJとHDRの修復。 DNAに二本鎖切断を導入した後のNHEJおよびHDR修復メカニズムの簡略化された概略図。 この図の拡大版を表示するには、ここをクリックしてください。
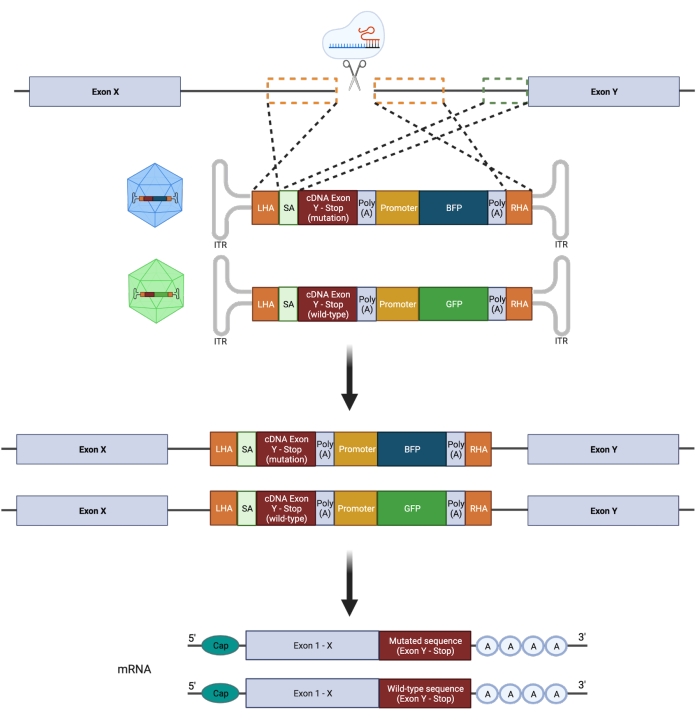
図2:双対立遺伝子HDR編集戦略の概略図。 ドナーテンプレートの標的対立遺伝子への統合とそれに続く機能的mRNAへの翻訳を示す概略図。オレンジ色の点線のボックスは、左相同性アーム(LHA)および右相同性アーム(RHA)に対応する領域を示す。HA の理想的なサイズは、それぞれ 400 bp です。緑色の点線のボックスは、SA配列に対応する領域を表す。SAのサイズは150 bpです。 この図の拡大版を表示するには、ここをクリックしてください。
プロトコル
このプロトコルは、健康なドナー由来のCD34+ HSPCの使用を必要とし、地元の治験審査委員会(IRB)からの倫理的承認と署名されたインフォームドコンセントを必要とします。このプロトコルで使用されたCD34+ HSPCは、正期分娩(妊娠34週>)の臍帯血(UCB)から分離されました。出産前に母親からインフォームドコンセントが得られ、グラーツ医科大学からUCBの収集に関する倫理的承認が得られました(IRB承認:31-322 ex 18/19)。このプロトコルで使用される材料の完全なリストは、 材料表に記載されています。
1. sgRNAの設計と切断効率の評価
- 目的の突然変異の場所と正しい転写産物をオンラインデータベースツール(COSMIC、https://cancer.sanger.ac.uk/cosmic など)で検索します。
- sgRNA設計ツールを使用して、変異に隣接するsgRNA(エキソンターゲティング)または前のイントロン(イントロンターゲティング)を選択します。このプロトコルでは、ベンチリングのオンラインツールが使用されます。sgRNA設計のための可能なオンラインツールのリストについては、 材料の表 を参照してください。
- [DNA 配列> DNA 配列のインポート] > [データベースからインポート] > [+ (作成)] オプションを選択します。目的の遺伝子(例:CALR)を入力し、種としてヒトを選択します > 検索> 正しい転写物(例:CALR-201 -ENST00000316448)を選択し、インポート>。DSブレークを導入する関心領域を選択します(例:イントロン7)。
- 画面の右側にある [CRISPR ] オプションを選択し、[ ガイドの設計と分析] を選択します。単一のガイドを選択し、 ガイドの長さを20 bpに保ち、SpCas9(NGG)で編集するためのPAMシーケンスを選択します。
- オンターゲットスコアが高い(目的の遺伝子座で編集する可能性が高い)とオフターゲットスコアが高い(不要な遺伝子座で編集する可能性が高い)ガイドを選択します。最高のパフォーマンスを発揮するsgRNAを見つけるために、テストするガイドを少なくとも3つ選択してください。sgRNAを化学修飾合成sgRNAとして市販のベンダーに注文してください。
注:この段階では、初期スクリーニング用に少量の合成sgRNAを注文することをお勧めします。良好な性能のsgRNAが同定されたら、選択したsgRNAのより大きなオーダーに進みます。 - 2 x 10 5-5 x 105 CD34+ HSPCを解凍します。 細胞を、1%抗生物質(ペニシリン/ストレプトマイシンなど)を添加した10 mLの予熱したRPMI1640に移します。
注:純度>90%のCD34+ HSPCを使用して、最良かつ最も再現性のある結果を得る必要があります。 - 室温(RT)で350 x g で10分間遠心分離します。血球計算盤で細胞をカウントし、0.2%ペニシリン/ストレプトマイシン(P / S)、100 ng / mLトロンボポエチン(TPO)、100 ng / mL幹細胞因子(SCF)、100 ng / mL FMS様チロシンキナーゼ3リガンド(FLT3L)、100 ng / mLインターロイキン-6(IL-6)、35 nM UM171、および0.75 μMステムレゲニン1(SR1)を添加したSFEM II培地に細胞を2.5 x 105 細胞/ mLの濃度に懸濁します。37°C/5%CO2で48時間〜72 時間インキュベートします。
注: 今後、完全な培地を HSPC 保持培地と呼びます。 - 細胞を15 mLチューブに集め、細胞を数えます。トリパンブルー排除により細胞生存率を確認する。
- 開始する前に、トランスフェクションシステムをオンにして、キュベットのオプションを選択します。HSPCに適したプログラムと核放出溶液を選択します(材料表を参照)。2 x 10 5 から5 x 106 の細胞を 1 つの 100 μL キュベットで核形成できます。少数の細胞(例えば、1 x 10 5-2 x 105)を48時間培養して、WTコントロールとして使用する。
- RNPコンプレックスを準備します。1.5 mLチューブに、15 μgのCas9と8 μgのsgRNA(モル比1:2.5)を加え、加熱ブロック内で25°Cで10分間インキュベートします。
- RNP複合体がインキュベートしている間に、細胞を350 x g のRTで5分間遠心分離し、ピペットを使用して上清を廃棄します。細胞を100 μLの核オフクション溶液に懸濁します。
- 細胞をRNP複合体と混合し、キュベットに移します。キュベットを軽くたたいて、転送中に形成された可能性のある最終的な気泡を取り除きます。
- キュベットをトランスフェクションシステムのホルダーに挿入し、DZ-100プログラムで細胞をエレクトロポレーションします。
- エレクトロポレーションの直後に、P/Sを含まない400 μLの予熱したHSPC保持培地を追加します。
- 細胞数に応じて、適切な培養プレート(24、12、または6ウェルプレート)を使用して、P/Sなしで事前に温めたHSPC保持培地を含む培養プレートに細胞を移します。
- プレートを37°C/5%CO2でインキュベーターに移します。核細胞を6〜8時間インキュベートします。
- 6〜8時間後、古い培地を取り出し、P / Sを添加した新鮮な予熱したHSPC保持培地と交換します。 細胞を2.5 x 10 5から5 x 105の濃度で懸濁し、細胞培養プレート(24、12、または6ウェルプレート)に移します。細胞を37°C/5%CO2で48時間インキュベートします。
- 2 x 10 5細胞を回収し、350 x gでRTで5分間遠心分離します。開始する前に、加熱ブロックを65°Cと98°Cに設定してください。
- 上清を廃棄し、1.5 mLチューブ中の1 mLの1x DPBSに細胞を懸濁します。350 x g でRTで5分間遠心分離します。
- 上清を捨て、細胞を50 μLのDNA抽出溶液に懸濁します。15秒間渦。
- 65°Cで6分間インキュベートします。 15秒間ボルテックスし、98°Cで2分間インキュベートします。
- DSBを中心にして約400〜600bpのアンプリコンを生成するプライマーを用いてPCRによりDSBの領域を増幅する。PCR反応には、0.5〜1 μLの抽出DNAを使用します。
注:DNA抽出溶液の組成により、分光光度法ではDNA濃度を正確に定量することはできません。 - DNAラダーを使用してPCR産物を1.5%アガロースゲル上で100 Vで45〜60分間実行します。ゲルを青色光またはUVトランスイルミネーターに置きます。
- 製造元の指示に従って市販のキットを使用して、ゲルから正しいサイズのDNAバンドを抽出します( 材料表を参照)。
- PCRからのフォワードプライマーまたはリバースプライマーを使用したサンガーシーケンシングによりサンプルをシーケンスします。長さが400〜600 bpのPCR産物の場合、総容量15 μLで5 ng/mLの濃度でシーケンシングするには、75 ngのDNAが必要です。
- sgRNAによって生成される挿入と欠失を特定することによって編集効率を計算するように設計されたプログラムにシーケンスファイルをアップロードすることにより、sgRNAの編集効率を分析します。最もパフォーマンスの高いsgRNAを選択して続行します。
注意: 専用のオンラインツールは 、材料の表に記載されています。この解析には、トランスフェクトされたHSPC、WT HSPCの.ab1ファイル、sgRNA配列、およびPAM配列が必要です。
2. 相同性指向修復(HDR)ベクトル構築
- HDRテンプレートのデザイン
注:WTシーケンスのテンプレートと変異シーケンスのテンプレートの2つのHDRテンプレートを設計する必要があります。- 目的の遺伝子のゲノム配列とコード配列(CDS)を分子クローニング用の適切なソフトウェアにインポートします(専用ツールは 材料表に記載されています)。ゲノム配列ファイルから、理想的にはDSBの5'末端で400 bpを選択して、左相同性アーム(HA)を設計します。このシーケンスを新しいファイルに貼り付けます。
- イントロンがsgRNAで標的化されている場合、スプライスアクセプター(SA)配列を含める必要があります。ターゲットイントロンの最後の150 bpを選択し、左側のHAの後に貼り付けます。エクソンが直接標的化されている場合、この配列は必要ありません(図3)。
- CDS ファイルから目的の cDNA を選択します。エキソン配列が標的となる場合は、cDNAがDSB部位のすぐ下流から始まり、目的の遺伝子の以下のすべてのエクソンと終止コドンが含まれていることを確認してください。イントロンを標的とする場合は、cDNAが次のエクソンの最初のコドンから始まることを確認してください。cDNAをコドン最適化し、SA配列の後に挿入します。この目的のために専用のオンラインツールを使用してください(資料表)。
- 目的のcDNA(SV40またはbGHなど)の後に3'ポリアデニル化(PolyA)シグナルを挿入します(表1)。PolyAに続いて、蛍光タンパク質のプロモーター配列(例えば、脾臓焦点形成ウイルス[SFFV]またはポリユビキチンC[UBC]、 表1)を挿入する。
- 蛍光タンパク質の配列(すなわち、GFP、BFP、またはmCherryのいずれか)を挿入します。2 つ目の異なる PolyA 信号を挿入します。
注:異なるPolyA配列を挿入することで、プラスミドの細菌組換えなどの問題や、テンプレート内の2つの同一の配列による配列アラインメントの問題を回避できます。 - ゲノム配列ファイルから、理想的にはDSBの3'末端で400 bpを選択して、適切なHAを設計します。このシーケンスを 2 番目の PolyA の後に挿入します。
- テンプレート全体のコピーを作成し、目的の突然変異の配列が含まれるように目的のcDNAを変更します。
- 蛍光タンパク質を別の蛍光タンパク質と交換する。たとえば、GFPがWTテンプレートに使用された場合、変異テンプレートのBFPまたはmCherryと交換します。HDRテンプレートを構築し、pAAV-MCSプラスミド(または他の適切なバックボーン)にクローニングします。
注:アセンブリに必要なフラグメントは、PCRまたは商業的に注文して製造でき、隣接するフラグメントと重複する配列を含む必要があります。目的の変異が点突然変異、小さな挿入、または小さな欠失である場合、WT cDNAを含むHDRテンプレート上で部位特異的突然変異誘発を行うことにより、変異したcDNAをPCRによって作製することができる。 - コンピテント 大腸菌 をヒートショック法を使用して組み立てられた製品で変換します。始める前に、バクテリアを氷の上に10分間置きます。
- 組み立てた製品2 μLを50 μLのバクテリアに加えます。軽くフリックして内容物を混ぜます。
- サンプルを氷上に30分間置きます。サンプルを42°Cに設定したサーモブロックに30秒間移します。
- サンプルを氷上で5分間移します。室温のSOC培地450 μLを加え、サンプルを37°Cで1時間インキュベートします。
- サンプルをアンピシリンを含むLB寒天プレートに広げます。各サンプルについて、細菌溶液の3つの異なる希釈液(未希釈、1:5、および1:10)を100 μL広げて、ピック可能な単一コロニーを有するLB寒天プレートを得た。プレートを37°Cで一晩インキュベートします。
- 翌日、サンプルごとに3つのコロニーを選びます。アンピシリンを添加した4 mLのLB培地を含むキャップ付きの15 mLチューブにコロニーを移し、37°Cのシェーカーで一晩インキュベートします。
- 各コロニーから500 μLの細菌溶液を分注し、後で使用するために冷蔵庫に保管します。ミニプレップを実行して、製造元の指示に従ってプラスミドDNAを抽出します。
- サンガーシーケンシング用のサンプルを送信して、プラスミドの正しいアセンブリを確認します。プラスミド全体に分散した十分なプライマーを使用して、配列の確認が中断されないようにし、理想的には各領域を順リードと逆リードで2回カバーします。
- 正しく組み立てられたプラスミドを含む細菌溶液200 μLを、アンピシリンを添加した200 mLのLB培地に加え、シェーカーで37°Cで一晩インキュベートします。
- ミディプレップまたはマキシプレップを実行して、製造元の指示に従ってプラスミドDNAを抽出します。プラスミドDNAを-20°Cで保存します。
- 組換えAAV6調製
注:2つの別々のAAV6準備を実行する必要があります:1つはWT HDRテンプレートを使用したrAAV6用、もう1つは変異したHDRテンプレートを使用したrAAV6用です。このセクションでは、1 つのウイルスのみを準備するために必要な手順について説明します。- HEK293T細胞を解凍し、10%FBS、1%P/S、および25 mM HEPESを添加した10 mLの予熱DMEMに細胞を移し、350 x g でRTで5分間遠心分離します。
- 細胞を1 x 105 細胞/mLの濃度で懸濁し、適切なフラスコ(例えば、175 cm2 フラスコ)に移します。フラスコを37°C/5%CO2のインキュベーターに移します。
注:HEK293T細胞は、細胞が完全に回復し、十分な数の細胞を得るために、事前に解凍する必要があります(1つのウイルスの産生には11 x 107 細胞が必要です)。最低3継代後にセルを使用し、細胞を20継代未満に保つことをお勧めします。HEK293T細胞は週に3回分割する必要があります。細胞を維持しながら、これらは70%〜80%のコンフルエントを超えてはなりません。AAV製剤に使用されるDMEMには、L-グルタミンとピルビン酸ナトリウムがすでに補給されているはずです。 - 細胞を50 mLチューブに回収し、350 x g でRTで5分間遠心分離します。
- 10%FBS、1%P / S、および25 mM HEPESを添加した20 mLのDMEMに細胞を懸濁し、血球計算盤でカウントします。死細胞排除にはトリパンブルーを使用してください。
- 175 cm2フラスコに10%FBS、1%P/S、および25 mM HEPESを添加した20 mLのDMEMに3 x 106細胞を播種します。1つのウイルスを生産するために、少なくとも4つの175 cm2フラスコを準備します。細胞を37°C/5%CO2で3日間インキュベートします。
注:この手順は、週末にセルを拡張できるため、金曜日に実行するのが最適です。 - HEK293T細胞を収穫して数えます。
- ウイルスの1つを調製するために、10%FBS、1%P / S、および25 mM HEPESを添加した20 mLのDMEM中に11 x 106 HEK293Tを含む150 mmディッシュをそれぞれ10個準備します。
- 皿を37°C / 5%CO2 のインキュベーターに24時間入れます。古い培地を慎重に廃棄し、10%FBS、25 mM HEPES、および1 mM酪酸ナトリウムを添加した20 mLの抗生物質を含まないDMEMと交換します。先に進む前に、細胞のコンフルエントが80%を超えていないことを確認してください。
- トランスフェクションミックスを入れる15 mLチューブを2本用意します。チューブ1に、5 mLの還元血清培地、60 μgのrAAV6変異HDRプラスミド、および220 μgのpDGM6(ヘルパープラスミド)を加えます。チューブ2に、5 mLの還元血清培地と1120 μLの1 mg/mLポリエチレンイミン溶液(PEI、トランスフェクション試薬)を加えます。
- チューブ2の内容物をチューブ1に追加し、30秒間渦巻きます。RTで15分間インキュベートして、DNAをPEIミセルに適切にカプセル化します。
注意: 溶液を20分以上保管しないでください。 - 各皿に1.1 mLの溶液を慎重に滴下し、穏やかに回転させて分配します。皿を37°C / 5%CO2 のインキュベーターに48時間入れます。
- 48時間後、各皿に250 μLの0.5 M EDTAを加え、皿をインキュベーターに10分間入れます。細胞を皿から洗い流して回収し、500 mL遠沈管または複数の50 mLチューブに移します。
- 2,000 x g で4°Cで10分間遠心分離します。 上澄み液を捨てる。上清を完全に除去するには、2,000 x g で4°Cで1分間遠心分離します。
- 残っている上清を捨てます。上清が残っていると、ウイルスの精製が損なわれる可能性があります。
- ボルテックスによってペレットを緩め、製造元の指示に従ってAAV精製キット( 材料の表を参照)を使用してウイルスを抽出します。あるいは、ヨージキサノール勾配超遠心30、31による抽出を行う。
- 精製したウイルスを分注し、-80°Cで保存します。 最適な形質導入条件を決定するために、AAVを機能的に滴定するか、デジタルドロップレットPCR(ddPCR)32によってAAV力価を定量します。
- rAAV6 WT HDRプラスミドの調製のためにこのプロセスを繰り返します。
- ddPCRによるAAV滴定
- 開始する前に、加熱ブロックを65°Cと98°Cに設定してください。
- 5 μLのウイルスに15 μLのDNA抽出溶液を加えてウイルスDNAを抽出します。15秒間渦。
- 65°Cで6分間インキュベートします。 15秒間渦。98°Cで2分間インキュベートします。
- 抽出したDNA(ウイルスDNAの1:4希釈)を使用して、ddPCR用のヌクレアーゼフリー(nf)H2Oによる段階希釈(1:400、1:40,000、1:160,000、1:640,000)を調製します。
注:ddPCRがすぐに実行されない場合は、希釈液を-20°Cで保存できます。 - ddPCR用の希釈液を3つ選択します。使用する希釈液の選択は、ウイルスの濃度によって異なります。好ましくは、3つの異なる希釈液(例えば、1:40,000、1:160,000、および1:640,000)を使用して、機械の検出限界内にある最良の希釈物を見つける。
- 作業中は試薬を氷上に保管してください。マスターミックスを準備します(すべてのサンプルが重複して測定されます)。各反応について、プローブ用 ddPCR スーパーミックス 12.5 μL、dUTP なし、プライムタイム Std qPCR アッセイ 1.25 μL、AAV-ITR ( 材料表参照)、および 6.25 μL nf-H2O を準備します。
- 5 μLのDNAを20 μLのマスターミックスと混合します。1:40,000、1:160,000、および1:640,000の希釈に対してこれを実行します。
- カートリッジをカートリッジホルダーに入れます。70 μLの液滴生成オイルをオイル建ての列のプローブに追加します。気泡が発生しないように注意してください。
- サンプル単位の行に20 μLのサンプル量を追加します。気泡が発生しないように注意してください。
- カートリッジをガスケットで密封し、液滴発生器に入れます。マシンのスタートボタンを押して液滴を生成し、ガスケットを取り外し、マルチチャンネルピペットで生成された液滴の40 μLを96ウェルプレートに慎重に移します。液滴や気泡の破壊を避けるためにゆっくりと作業してください。
- PCRプレートシーラーを使用して、96ウェルプレートをピアスフォイル(赤い縞模様を上向き)で密封します。プレートをサーモサイクラーに入れます。
- 容量を40 μL、蓋の温度を105°C、ランプレートを2 °C/sに設定します。 表 2 の説明に従って PCR プログラムを実行します。
- 使用の30分前に液滴リーダーの電源を入れてください。デスクトップでソフトウェアを開きます。
- プレートレイアウトを入力し、[実験]タブで [ABS ]を選択し、[スーパーミックス]タブで ddPCRスーパーミックス を選択します。次に、最初に PRIMEを押してから、ソフトウェアの [システムのフラッシュ ]をクリックしてシステムを開始します。
- プレートをリーダーに入れます。実行を開始し、終了したら、データを CSV ファイルとしてエクスポートし、後でスプレッドシートとして分析します。
注:ソフトウェアによって生成される数値は、μLあたりのゲノムコピー(GC)です。これらに希釈係数を掛ける必要があります:GC/μL x 5(マスターミックス中のDNAの希釈)x初期希釈(すなわち、40,000または160,000)。
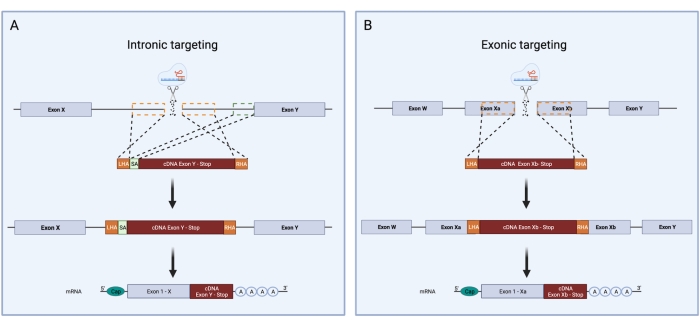
図3:CRISPR/Cas9 HDRノックイン中のイントロニックおよびエキソンターゲティングの概略図。 CRISPR/Cas9 HDRノックインのイントロニックターゲティング戦略とエキソンターゲティング戦略の概略比較。(A)イントロニックターゲティング中に、DNAのイントロンに二本鎖切断が導入される。HDRテンプレートは、LHA、cDNA配列、およびRHAで構成されています。イントロニックターゲティングには、さらに、3'スプライス部位、分岐点、およびポリピリミジントラクトを含むスライスアクセプターの存在が必要です。これにより、正しいスプライシングが可能になります。緑色の点線のボックスは、SA配列に対応する領域を表す。SAのサイズは150bpである。 (B)エキソンターゲティングは、エクソンに直接二本鎖切断を生成することに依存している。HDRテンプレートは、LHA、cDNA配列、およびRHAで構成されています。オレンジ色の点線のボックスは、LHAおよびRHAに対応する領域を示します。HA の理想的なサイズは、それぞれ 400 bp です。 この図の拡大版を表示するには、ここをクリックしてください。
| プロモーター | ||||
| 名前 | 長さ | 形容 | ||
| ティッカー | 492 bp | 脾臓焦点形成ウイルスプロモーター。強力な哺乳類プロモーター。構成的に表現 | ||
| ティッカー | 400 bp | ヒトユビキチンC遺伝子由来のプロモーター。構成的に表現された。 | ||
| ティッカー | 508 bp | サイトメガロウイルス由来のプロモーター。エンハンサー領域を含んでいてもよい。構成的に表現された。強力な哺乳類プロモーター。沈黙させることができます。 | ||
| EF-1a | 1182 bp | ヒト真核生物の翻訳伸長因子1アルファプロモーター。構成的に表現された。強力な哺乳類プロモーター。 | ||
| ティッカー | 200-300 BP | EF-1アルファイントロンレスショートフォーム | ||
| ティッカー | 584 bp | CMV早期エンハンサー(C)、ニワトリベータアクチンプロモーター(A)、およびウサギベータグロビン遺伝子のスプライシングアクセプター(G)を含むハイブリッド哺乳動物プロモーター。構成的に表現された。 | ||
| ポリアデニル化(PolyA)シグナル | ||||
| 略称 | 長さ | 形容 | ||
| SV40ポリア | 82-122 BP | サルウイルス40ポリアデニル化シグナル | ||
| bGH ポリア | 224 bp | ウシ成長ホルモンポリアデニル化シグナル | ||
| rbGlob PolyA | 56 bp | ウサギベータグロビンポリアデニル化シグナル | ||
表1:プロモーターおよびポリアデニル化シグナル。
| 歩 | 温度 | 時間 | サイクル |
| 酵素活性化 | 95°C | 10分 | 1倍速 |
| 変性 | 94°C | 30秒 | 42倍速 |
| アニーリング | 60°C | 1 分 | |
| 延長 | 72°C | 30秒 | |
| 酵素の失活 | 98°C | 10分 | 1倍速 |
| 持つ | 4°C | ∞ |
表2:デジタル液滴PCRプログラム。
3. HSPCの編集
- HSPCのトランスフェクションおよび形質導入
- CD34+ HSPCを解凍し、細胞を1%P/Sを添加した10 mLの予熱RPMIに移します。 遠心分離機、350 x g 、室温で10分間。
- 細胞をHSPC保持培地に2.5 x 10 5細胞/mLの濃度まで懸濁し、37°C/5 %CO2で48時間から72 時間インキュベートします。
- 細胞を15 mLチューブで回収します。細胞をカウントし、トリパンブルー排除により細胞生存率を確認する。2 x 10 5と5 x 106 細胞の間で、単一のキュベット内で核形成することができます。計算したセル数に応じて適切な数のキュベットを用意します。
- RNPコンプレックスを準備します。1.5 mLチューブに、15 μgのCas9と8 μgのsgRNA(モル比1:2.5)を加え、加熱ブロック内で25°Cで10分間インキュベートします。
- RNP複合体がインキュベートされている間に、細胞を350 x g で5分間遠心分離し、上清を廃棄します。
- 細胞を100 μLの核オフクション溶液に懸濁します。細胞をRNP複合体と混合し、キュベットに移します。
- キュベットを軽くたたいて、移動中に形成された可能性のある残留気泡を取り除きます。
- キュベットをトランスフェクションシステムのホルダーに挿入し、sgRNA設計のセクションで前述したように細胞をエレクトロポレーションします。
- エレクトロポレーションの直後に、P/Sを含まない400 μLの予温めたHSPC保持培地を加え、P/Sを含まない500 μLの予め加温したHSPC保持培地を含む組織培養プレートに微細トランスファーピペットで細胞を移します。 細胞数に応じて、適切な培養プレート(24、12、または6ウェルプレート)を使用して、0.25 x 10 6から1 x 106細胞/mLの間の密度に到達します。プレートを37°C/5%CO2でインキュベーターに移します。
- 凍結したrAAVを含むバイアルを氷上で解凍します。各rAAV6の最適量を細胞懸濁液にピペッティングすることによって細胞を形質導入する。エレクトロポレーション後20〜30分以内に形質導入を行い、高い形質導入効率を実現します。
注:各ウイルスの最適濃度(WT HDRテンプレート用の1つのウイルスと変異したHDRテンプレート用の別のウイルス)は実験的に決定する必要があります。通常、5,000〜10,000 GC /細胞(例えば、1 x 106細胞に対して5 x 109 GC)は高い形質導入をもたらす。 - 細胞懸濁液をピペットで穏やかに混合します。形質導入した細胞を37°C/5%CO2で6〜8時間インキュベートします。6〜8時間後、細胞をチューブに集め、RTで350 x g で5分間遠心分離します。
- 古い培地を廃棄し、P/Sを添加した新しい予熱したHSPC保持培地と交換します。
- 細胞を2.5 x 10 5-5 x10 5細胞/mLの濃度で懸濁し、組織処理した細胞培養プレート(24、12、または6ウェルプレート)に移します。ソーティングに進む前に、細胞を37°C/5%CO2で48時間インキュベートします。
- ヘテロ接合型GOF変異を有する改変HSPCのフローソーティング
- 細胞を15 mLチューブで回収し、350 x g でRTで5分間遠心分離します。上清を除去し、0.1% BSAを含む1 mLのDPBSに細胞を懸濁し、350 x g でRTで5分間遠心分離します。
- 上清を廃棄し、細胞数に応じてDPBS+0.1%BSAの適量で細胞を懸濁する。
注:ソーターを詰まらせるリスクを減らすために、細胞を最小容量200 μLで懸濁し、1 x 107 細胞/ mLを超えないようにすることをお勧めします。 - 細胞をキャップ付きの滅菌FACSチューブに移します。7-AADまたはその他の生存率色素(蛍光タンパク質との適合性に応じて)を細胞懸濁液に追加して、生細胞/死細胞を排除します。
- 蛍光レポータータンパク質に対して二重陽性の生細胞を、200 μLのHSPC保持培地を含む収集チューブに選別します。
注:選別手順中に適切なゲーティングを確保するために、AAVのみで形質導入された細胞で構成される適切なコントロールを追加する必要があります。 - 選別した細胞をRTで350 x g で5分間遠心分離し、細胞をHSPC保持培地に2.5 x 105 細胞/mLの濃度で懸濁して培養液でさらに増殖させるか、細胞を直接機能アッセイに使用します。
4. 遺伝子編集成功の確認
- ゲノムDNA抽出
- 開始する前に、加熱ブロックを65°Cと98°Cに設定してください。 2 x 10 5ゲノム編集およびソート精製された細胞を回収し、350 x gで5分間遠心分離します。
- 前述のようにgDNAを抽出します。DNA溶液をPCRに直接使用するか、-20°Cで保存してください。
- インアウトPCR
- 5'挿入部位を増幅するために2つのプライマーを設計します(図4A):プライマーフォワード1は、左相同性アームの外側のゲノム遺伝子座を標的にします。プライマーリバース2は、組込み配列を標的とする。
- インアウトPCRでは、反応ごとに以下のPCRミックスを準備します。
10 μL の PCR マスターミックス (2x; 材料表を参照)
1 μL プライマーフォワード 1 (10 μM ストック)
1 μL プライマーリバース 2 (10 μM ストック)
0.5-1.0 μLの抽出されたDNA
ヌクレアーゼフリーのH2Oを最終容量20 μLまで
注:複数の反応の場合は、ピペッティングエラーを考慮して、1つの追加の反応を含むマスターミックスを準備することをお勧めします。非テンプレートコントロールとモック処理サンプルからのテンプレートDNAを含めることが重要です。 - 適切なサーマルサイクリングプログラムを使用してPCR反応を実行します(製造元の指示を参照)。
注:最初にプライマーペアをテストして、最適なアニーリング温度を得ることをお勧めします。これは、Tmを計算し、次に温度勾配を実行できるサーモサイクラーでPCRを実行することによって行うことができます。 - DNAラダーを使用してPCR産物を1.5%アガロースゲル上で100 Vで45〜60分間実行します(図4B)。ゲルを青色光またはUVトランスイルミネーターに置きます。ゲルからバンドを切り取ります。
- DNAゲル抽出キットを使用してバンドからDNAを抽出します。抽出したPCRサンプルをサンガーシーケンシング用の適切なプライマーとともに送信し、内因性遺伝子座での目的のcDNAの正確かつシームレスな統合を確認します。
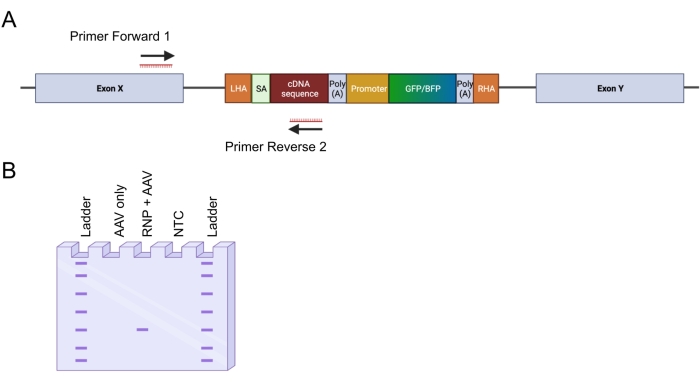
図4:インアウトPCRによるゲノム統合の検証 。 (A)インアウトPCR戦略の概略図。描かれた戦略では、2つのプライマーが設計されました。プライマーフォワード1はLHA外のゲノム遺伝子座を標的とし、プライマーリバース2はコドン最適化配列を標的とする。(B)アガロースゲル電気泳動の模式図。正常に編集された細胞(RNP + AAV)のみがインアウトPCR中にPCR産物を生成しますが、編集されていないサンプル(AAVのみ)はPCR産物を生成しません。省略形: NTC = 非テンプレート制御。 この図の拡大版を表示するには、ここをクリックしてください。
結果
上記のプロトコールを適用することにより、ヘテロ接合型1型CALR変異が臍帯血由来HSPCに再現性よく導入された。この変異は、エクソン9(CALRの最後のエクソン)の52 bpの欠失で構成され、+1フレームシフトをもたらし、新規の正に荷電したC末端ドメイン26,33の翻訳につながります。内因性遺伝子座にCALR変異を導入するために、Cas9誘発DSBがHDRメカニズムを介して修復されない場合にコード配列の望ましくない変化を回避するため、エクソン9の上流にあるイントロニックターゲティング戦略が採用されました。この特定のケースでは、イントロン7用のsgRNAは、良好な相同性アーム(配列反復の欠如;図5A)。
次に、2つのドナーテンプレートを設計し、AAV6ベクターでパッケージ化しました。内因性エクソンから組み込まれたcDNAへの正しいスプライシングを可能にするために、ドナーテンプレートには、(i)3'スプライス部位、分岐点、およびポリピリミジントラクトを含むSA配列、(ii)WT(CALRWT)または変異配列(CALRDEL)終止コドンを含む、(iii)サルウイルス40(SV40)polyAシグナル、(iv)別個の内部プロモーターの制御下にある蛍光タンパク質をコードする配列、脾臓焦点形成ウイルス(SFFV)プロモーター、続いて(v)ウシ成長ホルモン(bGH)polyAシグナルを含む。CALR WT cDNAを含むドナーテンプレートはGFPカセットを含むように設計されましたが、CALRDELのcDNA配列を含むドナーテンプレートはBFPカセットを含むように設計されました。構成全体は、左右のHAに隣接していました(図5A)。
RNP複合体によるトランスフェクションおよびrAAV6ウイルスによる形質導入の2日後に、細胞をフローサイトメトリーで分析しました。4つの主要な集団を検出することができた:(i)GFPもBFPも発現しない細胞、HDRベースのゲノム編集のない細胞、(ii)GFPのみ陽性の細胞、WT構築物のみを組み込んだ細胞、(iii)変異した構築物のみを組み込んだ細胞を表すBFPのみ陽性の細胞、および(iv)GFPとBFPのダブルポジティブ細胞、 WTおよび変異配列の両方を組み込んだ細胞を表す(図5B)。ヘテロ接合型 1型CALR 変異を有するHSPCの純粋な集団を得るために、ダブルポジティブ細胞をフローサイトメトリーで選別した。2つのWT配列をノックインしたHSPCをコントロール細胞として使用しました(GFP + mCherry+; 図5B)。コントロール細胞として使用する有効な代替手段は、セーフハーバー遺伝子座に蛍光タンパク質の二対立遺伝子組み込みを有するHSPCである(すなわち、AAVS1;図示せず)。選別されたHSPCに対して実施されたトリパンブルー排除による細胞計数は、細胞の90%以上が生存可能であることを示した。
コンストラクトのシームレスなオンターゲット統合は、インアウトPCR戦略を適用することによって確認されました(図6A)。この特定のケースでは、ノックインされたCALRWT配列(図6Aのゲル電気泳動のレーン1)とノックインされたCALRDEL配列(図6Aのゲル電気泳動のレーン2)の2つの別々のインアウトPCRを実行しました。ゲルバンドから抽出したDNAに対してサンガーシーケンシングを行ったところ、CALRDEL/WT HSPCにWTおよび変異配列が正しく挿入されていることが確認されました(図6B)。
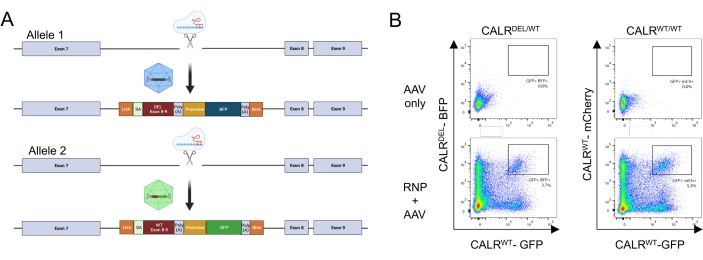
図5:ヘテロ接合型CALR変異を有するHSPCの生成 。 (a)ヘテロ接合型 CALR 変異の挿入のための編集戦略を示す代表的なスキーム。RNP複合体は、 CALR 遺伝子のエクソン7とエクソン8の間のイントロンを標的とする。変異したエクソン8〜9とBFPを含むAAVと、WTエクソン8〜9とGFPを含むAAVの2つのAAVは、ドナー修復テンプレートとして機能し、変異配列の統合を促進します 一方の対立遺伝子および残りの対立遺伝子におけるWT配列の統合。(B)HSPCのトランスフェクションおよび形質導入後のGFPおよびBFPまたはGFPおよびmCherryの発現を48時間後に描いた代表的なフローサイトメトリープロット。 この図の拡大版を表示するには、ここをクリックしてください。
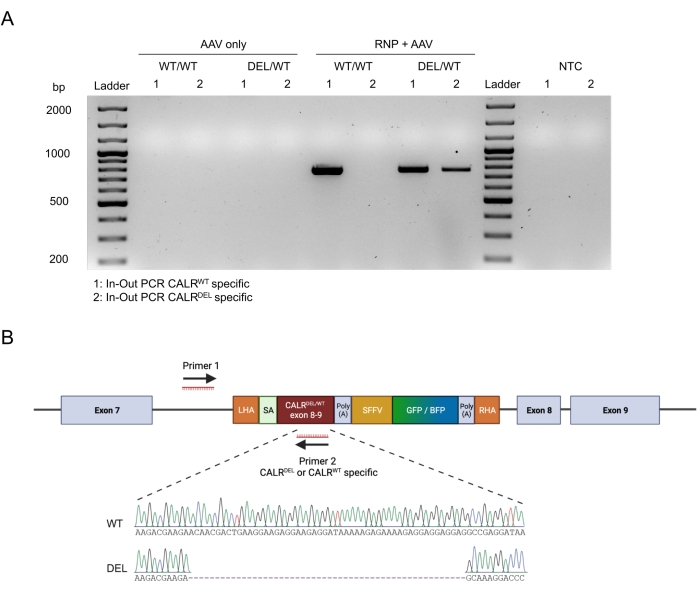
図6:HSPCにおけるヘテロ接合型CALR変異の成功の検証 。 (A)AAVコントロール、CALR WT /WT、およびCALRDEL / WTから抽出されたゲノムDNAに対して実行されたインアウトPCRの産物からのゲル電気泳動。100 bpのDNAラダーを使用した。省略形: NTC = 非テンプレート制御。(B)CALR DEL/WTで実行されたインアウトPCRから得られたサンガーシーケンシングの結果は、WT と変異配列の統合が成功したことを確認しています。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
ヒト初代HSPCの効率的かつ正確な遺伝子操作は、正常な造血、そして最も重要なことに、造血細胞の白血病形質転換に影響を与えるプロセスを探求し理解する絶好の機会を表しています。
このプロトコルでは、再発性ヘテロ接合型GOF変異を発現するようにヒトHSPCを設計するための効率的な戦略が説明されました。この手順では、CRISPR/Cas9テクノロジーとrAAV6ベクターをDNAテンプレートのドナーとして利用して、WTおよび変異DNA配列を内因性遺伝子座に正確に挿入しました。改変されたcDNA(WTおよび変異体)を分離された蛍光レポータータンパク質と結合させることで、決定的なヘテロ接合状態にある細胞の濃縮と追跡が可能になります。
この戦略は、頻繁に使用されるレンチウイルス(LV)ベースの方法と比較していくつかの利点を提示します。主な利点の1つは、CRISPR/Cas9ベースのシステムにより、内因性遺伝子座の正確な編集が可能になり、内因性プロモーターと調節要素が保存されることです。これは、細胞内で編集された遺伝子の発現の均一性をもたらし、LVベースの方法が使用される場合には達成不可能な目標である。LVベクターによる遺伝子導入は、転写活性部位34を優先して遺伝子の半ランダム組込みをもたらす。これは、移入された遺伝子の過剰発現と編集された細胞間の不均一性に変換され、最終的には突然変異と遺伝子相互作用の役割を調査および分析することが困難になります。第二の利点は、記載されたシステムが、部位特異的編集システムであることであり、挿入突然変異誘発のリスクを排除することである35。
二重蛍光レポーター戦略は、両方の対立遺伝子で正常に編集された細胞の正確な濃縮と追跡を可能にし、一方の対立遺伝子がWT cDNAを統合し、もう一方の対立遺伝子が変異したcDNA配列を統合します。単一のレポーターのみを発現する細胞は、同じ蛍光レポーターを有するHDRテンプレートの単対立遺伝子統合または双対立遺伝子統合のいずれかを表す。どちらのシナリオも、単一細胞由来のクローンを作製し、個別に分析した場合にのみ正確に区別できます。しかし、HSPCは in vitroでの増殖能力が限られており、長期間培養しておくと、より成熟した子孫に分化し始め、自己複製能力と生着能力を失います。これにより、所望のヘテロ接合変異を有する単一細胞クローンの選択および増殖が不可能になる。ヘテロ接合変異を有する細胞に対するフローサイトメトリーによる二重蛍光タンパク質戦略および濃縮の適用は、長時間の in vitro 培養によって誘発される問題を回避することを可能にする。
この特定の例では、ヘテロ接合性のCALR DEL/WT変異を有するHSPCの純粋な集団を得るために、HSPCを効率的に操作および分類できることが実証されました。
しかし、このシステムは、ヘテロ接合型フレームシフト変異のエンジニアリングに限定されず、ミスセンス変異やナンセンス変異を含む他の変異タイプを作成するために容易に採用することもできます。WTを含むAAVまたは異なる蛍光レポータータンパク質を有する変異配列の異なる組み合わせを適用することにより、このシステムは、ホモ接合型変異の導入(変異cDNAを有するが蛍光レポーターが異なる2つのrAAVによる同時形質導入)または変異の補正(WT cDNAを有するが蛍光レポーターが異なる2つのAAVによる同時形質導入)にも利用することができる。さらに、この戦略は発癌性GOF変異の導入に限定されないことに言及することが重要です。実際、記載されたプロトコルは、遺伝子ノックアウト、遺伝子置換36,37、導入遺伝子(すなわち、キメラ抗原受容体)の標的ノックイン38、さらには疾患の原因となる突然変異の補正11,39を含む複数の代替戦略に利用することができる。
CRISPR/Cas9およびAAV6を複数の蛍光レポーターと組み合わせる戦略は、T細胞、形質細胞様樹状細胞、人工多能性幹細胞、神経幹細胞、気道幹細胞など、他の多くの細胞タイプにも適用できることが示されています24,38,40,41,42,43,44.この戦略は、優れたキメラ抗原受容体(CAR)T細胞の産生のために実施することができる。例えば、CAR-T細胞におけるTGFBR2遺伝子のCRISPR/Cas9媒介ノックアウトが、抑制性TGF-βに富む腫瘍微小環境におけるそれらの機能を大幅に増加させることが最近発表された45。このようなアプローチは、CARを発現するようにT細胞を設計し、TGFBR2遺伝子の両方の対立遺伝子にCARを特異的に挿入する部位によってTGFBR2遺伝子をノックアウトするためのワンステッププロトコルを提供する可能性があります。さらに、このアプローチは、CARをT細胞受容体アルファ定数(TRAC)遺伝子46,47に組み込むことによってユニバーサルCAR T細胞を生成するのにも有用であり得る。
再現性を高め、細胞の効率的な編集を保証するには、いくつかの重要な考慮事項に注意する必要があります。細胞の編集を成功させるための主な重要なポイントは、(i)sgRNAの選択、(ii)HDRテンプレートの設計、および(iii)rAAV6の生産にあります。
良好な性能のsgRNAの選択は、HDRテンプレートを統合できる対立遺伝子の最大数を決定するため、非常に重要です。現在利用可能な多数のソフトウェアにより、候補sgRNAの検索が簡素化されました。関心領域を選択することにより、ソフトウェアは、所望の遺伝子座および望ましくない遺伝子座での編集の可能性をそれぞれ示すオンターゲットスコアおよびオフターゲットスコアを有する一連のsgRNAを提案することができる。これらのスコアは、以前に公開されたスコアリングモデル48、49に基づいて計算される。これは良好な性能のsgRNAを選択するための良い出発点ですが、 インシリコでの 予測性能はin vitroで効率的なsgRNAに必ずしも対応しないため、sgRNAの性能を確認する必要があります。したがって、最良のsgRNAを見つける可能性を高めるために、少なくとも3つのsgRNAを設計してテストすることを強くお勧めします。真の良好な性能を発揮するsgRNAが特定されたら、HDRテンプレートの設計を進めることをお勧めします。
HDRテンプレートを設計する際には、予防措置を考慮する必要があります。左右の相同性アーム(それぞれLHAとRHA)は、HAが短くなるとHDR周波数が低下する可能性があるため、それぞれsgRNAカット部位の上流と下流にそれぞれ400 bpにまたがる必要があります。HDR を介して 導入できるcDNAのサイズは、AAVのパッケージング機能(約4.7kb)に依存します。HDRテンプレート内で必須の多数の要素(LHA、RHA、SA、PolyA、プロモーター、および蛍光レポーター配列)により、変異またはWT cDNAの残りのスペースは限られています。これは、目的の突然変異が遺伝子の3'末端近くにある場合、または全体的に短いCDSを持つ遺伝子にある場合は問題になりません。しかしながら、突然変異が長いCDSを有する(AAVの残りのパッキングスペースを超える)遺伝子の転写開始側(TSS)の近くに位置する場合、この記載されたアプローチは実行不可能であり得る。この問題を回避するために、HDRテンプレートを2つのAAVに分割することに依存する戦略が最近Bakらによって開発されました。この戦略は、大きな遺伝子50の最終的なシームレスな統合を得るために、2つの別個のHDR媒介統合に依存する。
ウイルスの品質とその力価は、細胞のゲノム工学の成功を左右する追加の要因です。最適な収量を得るには、HEK293Tを培養液で維持しながら完全なコンフルエントに達しさせないことが重要です。理想的には、HEK293T細胞は、70%〜80%のコンフルエントに達したときに分割する必要があります。さらに、HEK293Tはウイルス産生能力を低下させる可能性があるため、長期間培養しないでください。新しいHEK293T細胞は、20継代後に解凍する必要があります。高いウイルス力価を得ることは、実験の効率と再現性を高めるために重要です。ウイルス力価が低いと、HSPCの形質導入に必要な大量のrAAV溶液に変換されます。原則として、核細胞に添加されるrAAV溶液は、HSPC保持培地の総体積の20%を超えてはなりません。AAV溶液の量が多いと、細胞死の増加、増殖の低下、形質導入効率の低下につながる可能性があります。したがって、ウイルス力価が低い場合は、ウイルスをさらに濃縮することをお勧めします。
要約すると、このプロトコルは、CRIPSR/Cas9およびrAAV6ドナーテンプレートと追加のデュアル蛍光レポーターを同時に使用することにより、ヒトHSPCを正確かつ効率的に操作するための再現可能なアプローチを提供します。このアプローチは、正常な造血幹細胞生物学と突然変異が白血病発生にもたらす寄与を研究する上で優れたツールであることが証明されています。
開示事項
著者は開示するものは何もありません。
謝辞
この研究は、オーストリア科学基金(FWF、番号P32783およびI5021)からA.R.への助成金によってサポートされています。臨床研究助成金)、およびMEFOgraz。T.K.は白血病・リンパ腫協会の特別研究員です。
資料
| Name | Company | Catalog Number | Comments |
| 175 cm2 Cell Culture Flask, Vent Cap, TC-treated | Corning | 431080 | |
| 150 mm x 25 mm dishes | Corning | 430599 | |
| 293T | DSMZ | ACC 635 | https://www.dsmz.de/collection/catalogue/details/culture/ACC-635 |
| 4D Nucleofector Core Unit | Lonza | - | For nucleofection of human HSPCs use the DZ-100 program. |
| 4D Nucleofector X Unit | Lonza | - | |
| 500 ml Centrifuge Tube | Corning | 431123 | |
| 7-AAD | BD Biosciences | 559925 | |
| AAVpro Purification Kit | Takara | 6666 | |
| Alt-R S.p. Cas9 Nuclease V3 | Integrated DNA Technologies (IDT) | 1081058 | |
| Avanti JXN-30 Ultracentrifuge | Beckman Coulter | - | |
| Benchling sgRNA design tool | Online tool for sgRNA design: http://www.benchling.com/crispr | ||
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A7906-100G | |
| C1000 Touch Thermal Cycler | Bio-Rad | - | |
| Chemically modified synthetic sgRNA | Synthego | Website: https://www.synthego.com/products/crispr-kits/synthetic-sgrna Sequence for the sgRNA targeting intron 7 of CALR: 5’-CGCCTGTAATCCTCGCCCAG-3’ An 80 nucleotide SpCas9 scaffold is added to the 20 nucleotide RNA sequence to complete the sgRNA. Chemical modifications of 2'-O-Methyl are added to the first and last 3 bases and 3' phosphorothioate bonds are added in the first 3 and last 2 bases. *Alternatively chemically modified synthetic sgRNAs can be acquired from IDT (https://eu.idtdna.com/site/order/oligoentry/index/crispr) and Trilink (https://www.trilinkbiotech.com/custom-oligos) | |
| CHOPCHOP sgRNA design tool | Online tool for sgRNA design: http://chopchop.cbu.uib.no | ||
| Costar 24-well Clear TC-treated Multiple Well Plates | Corning | 3526 | |
| CRISPick sgRNA design tool | Online tool for sgRNA design: https://portals.broadinstitute.org/gppx/crispick/public | ||
| CRISPOR sgRNA design tool | Online tool for sgRNA design: http://crispor.tefor.net | ||
| ddPCR 96-Well Plates | Bio-Rad | 12001925 | |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863024 | |
| DG8 Cartridges for QX200/QX100 Droplet Generator | Bio-Rad | 1864008 | |
| DG8 Gaskets for QX200/QX100 Droplet Generator | Bio-Rad | 1863009 | |
| DreamTaq Green PCR Master Mix (2X) | Thermo Scientific | K1081 | |
| Droplet Generation Oil for Probes | Bio-Rad | 1863005 | |
| Dulbecco’s Modified Eagle Medium (DMEM) with high glucose | Sigma-Aldrich | D6429-6X500ML | |
| Dulbecco’s Phosphate Buffered Saline (DPBS) | Sigma-Aldrich | D8537-500ML | |
| FACSAria Fusion | BD Biosciences | - | |
| Falcon 5 mL Round Bottom | Corning | 352054 | |
| Fetal Bovine Serum (FBS) Good Forte (heat inactivated), 500 ml | Pan Biotech | P40-47500 | |
| FlowJo 10.8.0 | BD Biosciences | - | |
| GenAgarose L.E. | Inno-train | GX04090 | |
| GeneRuler 100 bp Plus DNA Ladder | Thermo Scientific | SM0321 | |
| Gibson Assembly Master Mix | New England Biolabs Inc. (NEB) | E2611L | |
| HEK293T | |||
| HEPES solution | Sigma-Aldrich | H0887-100ML | |
| ICE | Synthego | https://ice.synthego.com | |
| IDT codon optimization tool | IDT | https://www.idtdna.com/pages/tools/codon-optimization-tool | |
| IDT sgRNA design tool | Online tool for sgRNA design: https://www.idtdna.com/site/order/designtool/index/CRISPR_CUSTOM | ||
| LB Broth (Lennox) EZMix powder microbial growth medium | Sigma-Aldrich | L7658-1KG | |
| LB Broth with agar (Lennox) EZMix powder microbial growth medium | Sigma-Aldrich | L7533-1KG | |
| Midori Green Advance | Nippon Genetics | MG04 | |
| Monarch Plasmid Miniprep Kit | NEB | T1010L | |
| Monarch DNA Gel Extraction Kit | NEB | T1020L | |
| NEB 5-alpha Competent E. coli (High Efficiency) | NEB | C2987U | |
| Nuclease-Free Water, 5X100 ml | Ambion | AM9939 | |
| NucleoBond Xtra Midi | Macherey-Nagel | 740410 | |
| Opti-MEM, Reduced Serum Medium, 500 ml | Gibco | 31985070 | |
| P3 Primary Cell 4D-Nucleofetor X Kit L | Lonza | V4XP-3024 | The Lonza Primary P3 solution is supplied as a 2.25 mL P3 Primary Cell Nucleofector Solution and 0.5 mL Supplement 1. To reconstitute, add the Supplement 1 to the P3 Primary Cell Nucleofector Solution and mix. |
| pAAV-MCS2 | Addgene | 46954 | |
| PCR Plate Heat Seal Foil, pierceable | Bio-Rad | 1814040 | |
| pDGM6 | Addgene | 110660 | |
| Penicillin-Streptomycin (P/S) | Gibco | 15140122 | |
| Polyethylenimine (PEI) | Polysciences | 23966 | Add 50 mL of PBS 4.5 pH (made with HCl) to 50 mg of PEI in a tube. Dissolve by placing the tube in a 70°C water bath and vortexing every 10 minutes until the solution is dissolved. After the solution has reached RT, filter sterilze through a 0.22 μm filter, make 1120 μL aliquots, and store at -80°C. |
| Polystyrene Test Tube, with Snap Cap | |||
| Primers | Eurofins | - | Primers were ordered from Eurofins (eurofinsgenomics.eu) as unmodified salt free custom oligos. The primers were designed by using PRIMER-Blast (https://www.ncbi.nlm.nih.gov/tools/primer-blast/) Primer 1 Fwd: AAGTGATCCGTTCGCCATGAC; Primer 2 Rev CALR WT specific: ACGTCCTCTTCCTCGTCCTC; Primer 2 Rev CALR DEL specific: CCAACCCTGGAGACACGCTTC |
| PrimeTime qPCR Primer Assay | IDT | - | PrimeTime qPCR Probe Assays (1 probe/2 primers) that can be ordered from IDT (https://eu.idtdna.com/site/order/qpcr/assayentry). Scale: Std - qPCR Assay 500 reactions; Primer 1 Forward (5'-3') : GGAACCCCTAGTGATGGAGTT; Primer 2 Reverse (5'-3'): CGGCCTCAGTGAGCGA; Probe (5'-3'): CACTCCCTCTCTGCGCGCTCG; 5' Dye/3' Quencher: FAM/ZEN/IBFQ; Primer to probe ratio: 3.6 |
| PX1 PCR Plate Sealer | Bio-Rad | 1814000 | |
| QuantaSoft Software | Bio-Rad | ||
| Quick Extract DNA Extraction Solution | Lucigen | QE0905T | |
| QX200 Droplet Generator | Bio-Rad | 1864002 | |
| QX200 Droplet Reader | Bio-Rad | ||
| Recombinant human Flt3-ligand | Peprotech | 300-19 | |
| Recombinant human IL-6 | Peprotech | 200-06 | |
| Recombinant Human SCF | Peprotech | 300-07 | |
| Recombinant Human TPO | Peprotech | 300-18 | |
| RPMI 1640 | Sigma-Aldrich | R8758-6X500ML | |
| SnapGene | Dotmatics | Molecular cloning software https://www.snapgene.com *Alternatively also Benchling (https://www.benchling.com) and Geneious (https://www.geneious.com) can be used. | |
| Soc outgrowth medium | NEB | B9020S | |
| Sodium-butyrate | Sigma-Aldrich | B5887-1G | |
| Stem Regenin 1 (SR1) | Biogems | 1224999 | |
| StemSpan SFEM II | STEMCELL Technologies | 9655 | |
| TAE Buffer (Tris-acetate-EDTA) 50X | Thermo Scientific | B49 | |
| TIDE | http://shinyapps.datacurators.nl/tide/ | ||
| Trypan blue 0.4% | Sigma-Aldrich | T8154-100ML | |
| TrypLE (with phenol red), 500 ml | Thermo Scientific | 16605-028 | |
| UltraPure 0.5: EDTA, pH 8.0, 100 ml | Thermo Scientific | 15575-038 | |
| UM171 | STEMCELL Technologies | 72914 | |
| Vector Builder codon optimization tool | Vector Builder | https://en.vectorbuilder.com/tool/codon-optimization.html |
参考文献
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Gillmore, J. D., et al. CRISPR-Cas9 in vivo gene editing transthyretin amyloidosis. New England Journal of Medicine. 385 (6), 493-502 (2021).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and β-thalassemia. New England Journal of Medicine. 384 (3), 252-260 (2021).
- Stadtmauer, E. A., et al. CRISPR-engineered T cells in patients with refractory cancer. Science. 367 (6481), (2020).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Heyer, W. D., Ehmsen, K. T., Liu, J. Regulation of homologous recombination in eukaryotes. Annual Review of Genetics. 44, 113-139 (2010).
- Veldwijk, M. R., et al. Pseudotyped recombinant adeno-associated viral vectors mediate efficient gene transfer into primary human CD34+ peripheral blood progenitor cells. Cytotherapy. 12 (1), 107-112 (2010).
- Song, L., et al. High-efficiency transduction of primary human hematopoietic stem cells and erythroid lineage-restricted expression by optimized AAV6 serotype vectors in vitro and in a murine xenograft model in vivo. PLoS One. 8 (3), 58757 (2013).
- Dever, D. P., et al. CRISPR/Cas9 β-globin gene targeting in human haematopoietic stem cells. Nature. 539 (7629), 384-389 (2016).
- Drost, J., et al. Sequential cancer mutations in cultured human intestinal stem cells. Nature. 521 (7550), 43-47 (2015).
- Jan, M., et al. Clonal evolution of preleukemic hematopoietic stem cells precedes human acute myeloid leukemia. Science Translational Medicine. 4 (149), (2012).
- Corces-Zimmerman, M. R., Hong, W. J., Weissman, I. L., Medeiros, B. C., Majeti, R. Preleukemic mutations in human acute myeloid leukemia affect epigenetic regulators and persist in remission. Proceedings of the National Academy of Sciences of the United States of America. 111 (7), 2548-2553 (2014).
- Jaiswal, S., et al. Clonal hematopoiesis and risk of atherosclerotic cardiovascular disease. New England Journal of Medicine. 377 (2), 111-121 (2017).
- Genovese, G., et al. Clonal hematopoiesis and blood-cancer risk inferred from blood DNA sequence. New England Journal of Medicine. 371 (26), 2477-2487 (2014).
- Papaemmanuil, E., et al. Genomic classification and prognosis in acute myeloid leukemia. New England Journal of Medicine. 374 (23), 2209-2221 (2016).
- Cancer Genone Atlas Research Network. Genomic and epigenomic landscapes of adult de novo acute myeloid leukemia. New England Journal of Medicine. 368 (22), 2059-2074 (2013).
- Ball, M., List, A. F., Padron, E. When clinical heterogeneity exceeds genetic heterogeneity: thinking outside the genomic box in chronic myelomonocytic leukemia. Blood. 128 (20), 2381-2387 (2016).
- Varmus, H. E. The molecular genetics of cellular oncogenes. Annual Review of Genetics. 18, 553-612 (2003).
- Cox, D. B. T., Platt, R. J., Zhang, F. Therapeutic genome editing: Prospects and challenges. Nature Medicine. 21 (2), 121-131 (2015).
- Tothova, Z., et al. Multiplex CRISPR/Cas9-based genome editing in human hematopoietic stem cells models clonal hematopoiesis and myeloid neoplasia. Cell Stem Cell. 21 (4), 547-555 (2017).
- Mandal, P. K., et al. Efficient ablation of genes in human hematopoietic stem and effector cells using CRISPR/Cas9. Cell Stem Cell. 15 (5), 643-652 (2014).
- Bak, R. O., Dever, D. P., Porteus, M. H. CRISPR/Cas9 genome editing in human hematopoietic stem cells. Nature Protocols. 13 (2), 358-376 (2018).
- Foßelteder, J., et al. Human gene-engineered calreticulin mutant stem cells recapitulate MPN hallmarks and identify targetable vulnerabilities. Leukemia. , (2023).
- Nangalia, J., et al. Somatic CALR mutations in myeloproliferative neoplasms with nonmutated JAK2. New England Journal of Medicine. 369 (25), 2391-2405 (2013).
- Merlinsky, T. R., Levine, R. L., Pronier, E. Unfolding the role of calreticulin in myeloproliferative neoplasm pathogenesis. Clinical Cancer Research. 25 (10), 2956-2962 (2019).
- Belčič Mikič, T., Pajič, T., Zver, S., Sever, M. The contemporary approach to CALR-positive myeloproliferative neoplasms. International Journal of Molecular Sciences. 22 (7), 3371 (2021).
- How, J., Hobbs, G. S., Mullally, A. Mutant calreticulin in myeloproliferative neoplasms. Blood. 134 (25), 2242-2248 (2019).
- Grieger, J. C., Choi, V. W., Samulski, R. J. Production and characterization of adeno-associated viral vectors. Nature Protocols. 1 (3), 1412-1428 (2006).
- Zolotukhin, S., et al. Recombinant adeno-associated virus purification using novel methods improves infectious titer and yield. Gene Therapy. 6 (6), 973-985 (1999).
- Aurnhammer, C., et al. Universal real-time PCR for the detection and quantification of adeno-associated virus serotype 2-derived inverted terminal repeat sequences. Human Gene Therapy Methods. 23 (1), 18-28 (2011).
- Klampfl, T., et al. Somatic mutations of calreticulin in myeloproliferative neoplasms. New England Journal of Medicine. 369 (25), 2379-2390 (2013).
- Bulcha, J. T., Wang, Y., Ma, H., Tai, P. W. L., Gao, G. Viral vector platforms within the gene therapy landscape. Signal Transduction and Targeted Therapy. 6 (1), 1-24 (2021).
- Montini, E., et al. The genotoxic potential of retroviral vectors is strongly modulated by vector design and integration site selection in a mouse model of HSC gene therapy. The Journal of Clinical Investigation. 119 (4), 964-975 (2009).
- Vaidyanathan, S., et al. Targeted replacement of full-length CFTR in human airway stem cells by CRISPR-Cas9 for pan-mutation correction in the endogenous locus. Molecular Therapy. 30 (1), 223-237 (2022).
- Cromer, M. K., et al. Gene replacement of α-globin with β-globin restores hemoglobin balance in β-thalassemia-derived hematopoietic stem and progenitor cells. Nature Medicine. 27 (4), 677-687 (2021).
- Wiebking, V., et al. Genome editing of donor-derived T-cells to generate allogenic chimeric antigen receptor-modified T cells: Optimizing αβ T cell-depleted haploidentical hematopoietic stem cell transplantation. Haematologica. 106 (3), 847-858 (2021).
- Wilkinson, A. C., et al. Cas9-AAV6 gene correction of beta-globin in autologous HSCs improves sickle cell disease erythropoiesis in mice. Nature Communications. 12 (1), 1-9 (2021).
- Dever, D. P., et al. CRISPR/Cas9 genome engineering in engraftable human brain-derived neural stem cells. iScience. 15, 524-535 (2019).
- Laustsen, A., et al. Interferon priming is essential for human CD34+ cell-derived plasmacytoid dendritic cell maturation and function. Nature Communications. 9 (1), 1-14 (2018).
- Bak, R. O., et al. Multiplexed genetic engineering of human hematopoietic stem and progenitor cells using CRISPR/Cas9 and AAV6. eLife. 6, 27873 (2017).
- Nakauchi, Y., et al. The cell type-specific 5hmC landscape and dynamics of healthy human hematopoiesis and TET2-mutant preleukemia. Blood Cancer Discovery. 3 (4), 346-367 (2022).
- Vaidyanathan, S., et al. selection-free gene repair in airway stem cells from cystic fibrosis patients rescues CFTR function in differentiated epithelia. Cell Stem Cell. 26 (2), 161-171 (2020).
- Tang, N., et al. TGF-β inhibition via CRISPR promotes the long-term efficacy of CAR T cells against solid tumors. JCI Insight. 5 (4), 133977 (2020).
- Georgiadis, C., et al. Long terminal repeat CRISPR-CAR-coupled "universal" T cells mediate potent anti-leukemic effects. Molecular Therapy. 26 (5), 1215-1227 (2018).
- Ren, J., et al. Multiplex genome editing to generate universal CAR T cells resistant to PD1 inhibition. Clinical Cancer Research. 23 (9), 2255-2266 (2017).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Doench, J. G., et al. Optimized sgRNA design to maximize activity and minimize off-target effects of CRISPR-Cas9. Nature Biotechnology. 34 (2), 184-191 (2016).
- Bak, R. O., Porteus, M. H. CRISPR-mediated integration of large gene cassettes using AAV donor vectors. Cell Reports. 20 (3), 750-756 (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved