Method Article
Infinium检测的大型SNP基因分型应用
摘要
的协议,描述了使用Illumina的Infinium测定进行大规模基因分型。这些检测能可靠百万基因型的SNP位点在数百个人的DNA样本中三天。一旦产生,这些基因型可以用来检查协会与各种不同的疾病或表型。
摘要
在人类基因组基因分型的变体已被证明是一种有效的方法来识别与表型的遗传关联。家庭或群体内变异的分布可以方便识别疾病的遗传因素。基因分型BeadChip芯片的Illumina的面板允许调查人员基因型数千或数百万的单核苷酸多态性(SNPs),或分析其他基因变异体,如拷贝数,在大量的DNA样本。这些SNP可扩散到整个基因组或对象中的特定区域,以便最大限度地电位的发现。该Infinium检测进行了优化,以快速产生高品质,准确的结果。通过适当的设置,一个技术人员可以从几百处理,以每周超过一千DNA样本,根据数组的类型。此法可指导用户完成每一步,从基因组DNA,并与阵列的扫描结束。使用恰当reageNTS,样品被放大,分散,沉淀,重悬浮,杂交到芯片上,由单一的基础上,延长染色和扫描在任一个ISCAN或扫描您好高分辨率光学成像系统。一整夜步骤是必需的,以扩增DNA。 DNA变性,并通过全基因组扩增等温扩增,因此,不存在PCR是必需的。样品在第二步骤中过夜杂交到阵列。在第三天,将样品准备进行扫描和分析。扩增的DNA可能会在储存大批量,使磁珠阵列要在一周的每一天处理,从而最大限度地提高吞吐量。
引言
输入的单核苷酸多态性(SNP)是识别与疾病相关的风险的变体的一个重要方法。从历史上看,基因分型实验的范围已经由现有技术的限制。凝胶电泳为基础的基因分型方法在采样和SNP-吞吐量有限的[1]。开发这些分析往往是劳动密集型的,依托周边的变体优化该地区的化妆和结构[1]。 TaqMan探针基因分型检测,由Life Technologies公司开发,可以快速并以最少的技术人员参与运行大量样本[2],但SNP复用限制继续基因型的总数限制在远低于百万每天[3,4 。 Sequenom的的iPlex平台还可以运行多个样本一次,但是,只有不到百个SNP可复用在一起,吞吐量相对较低的整体[5]。 Beckman Coulter公司的SNP流技术理论上可以每天生产超过三百万基因型,但这种技术限制项目范围上限为每次反应仅48个SNPs [4,6]。而GoldenGate的检测可以在数百个样品的单核苷酸多态性或数千每天处理近二百年的DNA样本,打字超过三千个SNP一次[4,7时,每基因型的价格是不是有先进的,超高通量技术竞争力。为了处理每天数百万的基因型,需要较大的全基因组关联研究的规模,阵列杂交检测已成为市场上最具成本效益的选择。
Affymetrix公司的线阵列杂交和Illumina的线的Infinium为基础的阵列允许潜在的数百个样品平行[4,8],以输入在成千上万的SNP或数百万。这些SNP位点可以分散在整个基因组中,在局部利益的地区,如前OMES,或定制到用户的偏好。这些阵列具有不仅能够准确基因型每采样1万个SNPs一次,也是衡量拷贝数变异,可能亮相染色体异常的好处。的Infinium对齐OMNI BeadChip芯片阵列目前每天有基因型高达每样近五百万的标记,包括五十万定制的位点,在多达近百个样品的能力。
由于很多疾病有遗传成分,这些大型的实验可以寻找与疾病相关的基因是至关重要的。高通量基因分型提供了高效基因型代样品设置足够大,以令人信服的检测遗传协会在较低的次要等位基因频率。全基因组基因分型项目,可以用来定位的区域具有统计学显著病例对照等位基因频率或拷贝数差异[9,10,11]。根据美国国家人类基因诺姆研究所,全基因组关联研究,导致1,490单独的出版物2008年11月25日和2013年1月25日之间,从8,283个SNP的发现与p值小于1×10 -5词干( 见包含http:// www.genome.gov/gwastudies/)。这些研究,其研究条件,从高至睾丸癌,从广义的方法由全基因组分析得到受益。在这样的情况下,利息整个区域可能已经逃脱了通知打字的范围太局限。因此,对于大规模的关联分析,全基因组基因分型技术是选择的技术。
不同版本的Infinium检测的存在,每一个用于特定类型的数组使用。该InfiniumUltra分析,在下面讨论的深度,适用于许多12 - 或24-样品阵列芯片。这些通常每基因型DNA样本超过十万个SNP,并重点对Targeted地区,如外显子组或自定义面板。可能需要的其它类型的芯片,如全基因组基因分型阵列的其它测定的版本。然而,由于所有Infinium测定都有一个共同的基础,主要区别仅由试剂名,试剂量,或者确切的染色试剂过程中,完善了一个实验版本的技术往往可以普遍应用。其他阵列,如甲基化阵列,可以使用一个几乎相同的协议,以及。必须小心,以仅使用所需使用的芯片型测定的版本。某些类型的,如那些测量基因表达水平,可能需要使用一个nonInfinium协议。
样品必须分批进行处理。例如,与InfiniumUltra测定中,预杂交试剂管中含有足够量来运行96个样品,并且在管不能再冷冻。因此,样品必须在同一时间运行在96批样品。样品将在冷杉放大T日。经过约1小时的钳工,样品必须在对流烘箱中于20-24小时进行加热。翌日,将近4小时将用于粉碎,沉淀,并再悬浮的样品,在该点的样品可以被冷冻以供将来使用,或杂交到芯片上。装载芯片需要近2小时,在此之后,样品将被通宵杂交16-24小时。到了第三天,染色和延伸步骤约需4小时。再一个小时将用于洗涤,涂布,并干燥该晶片。最后,该阵列被扫描时,这可能需要15-60分钟/芯片,这取决于所用的类型。
标准实验室安全和清洁注意事项适用。虽然扩增不基于PCR的,前和扩增后的程序不同的工作站上是必要的,以减少污染的可能性。在跟踪表必须先登录才能利用每试剂盒提供的试剂的标识号。 ReageNTS应立即使用前分装前解冻,倒了好几次。需要输入的DNA必须是高品质的基因组DNA(260/280的1.6-2.0吸光度比值,低于3.0二百三十○分之二百六十零吸光度比值),通过标准方法分离和量化与荧光。 DNA降解往往是低质量的测定结果的一个因素。通常情况下,200 ng的DNA是必需的,但这个数额可能会因某些芯片类型。 Tecan公司一个液体处理机器人可以自动完成许多步骤的协议,并最大限度地减少人为错误的因素。
研究方案
第一天
1。准备
- 免除200纳克的DNA陷入了深深的井,96个样品板。至少有96个样品(全板)必须镀以确保没有试剂将被浪费。标示板与套件中提供的条形码贴纸和离心它。
- 以归卷,离开样品在抽屉或通风柜一夜之间蒸发的液体。松散覆盖板盖或纸巾,以防止灰尘。
2。放大
警告:样品不应该进行扩增,除非4小时将可在翌日的碎片,沉淀和再悬浮的步骤。
- 除去该公司提供的箱管从-20℃冷冻箱(MA1,MA2及MSM中的一个管是足够的每一组96个样本),标记为"前"或包。设置在板凳上MA2和男男性接触者的管解冻。打开正确校准烘箱,并设置为37℃。
- 使用试剂盆和一个10微升,8道移液器,取4μL的DNA悬浮缓冲液中,每孔再水化样品。这是没有必要放弃每列之间的枪头如果小心不要接触液体。
- 用8通道移液器,取20微升的MA1到板的每个孔。对于每一个新的试剂,使用新鲜的试剂盆地。用可重复使用的密封,脉冲离心机,并在1600转的微孔板摇床1分钟旋涡覆盖板。
- 2.4)在室温下孵育至少30分钟。
- 分配4微升的0.1N的NaOH到板的各孔中。用可重复使用的密封,脉冲离心机,并在1600转1分钟旋涡覆盖板。
- 在室温下孵育10分钟。
- 免除34微升MA2入板的各孔中。
- 免除38微升的MSM到板的每个孔。覆盖板的可重复使用的密封,脉冲离心机,并为涡旋在1600转1分钟。
- 放入烤箱20-24小时。
两日
3。碎片
- 从"后1"删除FMS管-20°C箱(一管就足够了每组96个样本)。解冻板凳上或在室温水浴。插入MIDI板插入到桌面微量样品孵化体系后,打开加热块上,并设置为37°C。
- 一旦管解冻,从烤箱和脉冲离心机除去样品板。免除25微升的FMS到DNA板的每个孔中。更换盖,脉冲式离心机,和涡流在1600 rpm下离心1分钟。
- 在热块培养板1小时。
4。沉淀
- 从"后3"4°C包装盒中取出PM1管或包和温暖至室温(一管就足够了每组96个样本)。从热集团移除板k和脉冲离心机。
- 分配50μl的PM1的入板的各孔中。更换盖,脉冲式离心机,和涡流在1600 rpm下离心1分钟。
- 在热块培养板5分钟。
- 从加热块取出板,将其关闭,并丢弃板盖。免除155微升100%的异丙醇到该板的各孔中。一个新的盖子捂得严严实实,并手动翻转板多次混合。
- 放置板在4℃下至少30分钟。打开冷冻离心机,并设置为4℃至冷却。
- 平衡板和离心机在4℃下静置20分钟,在3000×g下。
- 检查板的底部不反转,并且确认该样本是沉淀在蓝色沉淀。如果没有小球可以再次看出,离心机。拿纸巾。
- 丢弃盖子并迅速通过翻转板和覆盖纸巾台式用力拍打它去除液体。一旦该板块是我nverted,注意不要回复它,而任何液体仍然存在。反复敲击板对台式直到所有液体被删除。
- 在一个试管架,反转设置板,晾干。在室温下孵育1小时。
5。悬浮
- 从"后2"删除RA1 -20°C箱和解冻在室温水浴。打开烤箱,并将其设置为48°C。
- 一旦解冻,取23微升RA1到样品板的每个孔中。整个瓶RA1的不倾入盆内;节省30毫升后。如果样品将在当天晚些时候进行杂交到阵列,将RA1到4℃凉爽。如果样品将在以后的日子中运行,标签的瓶子和重新冻结的RA1。
- 热封新盖到板上。脉冲离心机的板,并将其放置在烘箱中1小时。
- 从烘箱中并涡旋在1,800 rpm离心1分钟,取出板。
样品可以安全地为hELD在这个阶段长达一个星期。板可以储存和样品可能会有所调整,为珠芯片应用程序做准备,如果必要的。如果继续进行过程中,离开该板在室温下,把加热块上。否则,存储板在-20℃下
6。杂交
警告:样品不应该进行杂交,除非5.5小时可在翌日的染色和洗涤步骤。
- 打开加热块上,并将其设置为95°C。打开烤箱,并将其设置为48°C。
- 一旦温度稳定,孵育板20分钟加热块上。
- 而板变性,从4℃取出的珠片的框中,然后在板凳上进行设置。拿一瓶XC4的(从室温套件),并添加330毫升100%的乙醇。充分摇匀,并在室温下孵育过夜。制备酰胺/ EDTA混合物(95%甲酰胺,0.2%EDTA(0.5M),4.8%H 2 O的体积比),并冻结在不同的15毫升增量。过量的甲酰胺可堆存。
- 从加热块中取出的样品板。在室温下孵育30分钟。
- 虽然该板块冷却,准备HYB室。一室将需要处理每四珠芯片。
放置在HYB室基部的顶部的橡胶垫,对准基座的正面较厚的孔。蚀刻到基条码符号应该仍然可见( 见图2)。 - 使用1,000微升移液器,免除400微升PB2到每个雕刻成底座八加湿水库。 PB2可以在"后3"箱或包被发现。放置HYB室盖在基座上,并通过关闭两个扣紧在对角线相对的两侧的第一扣两端。
- 从他们各自的箱子取出珠芯片的个人银牌包,但是,为了尽量减少芯片的世博会一定要通风透光,还没有打开它们。仔细扫描芯片的条码,并记录在其中,他们将在一个跟踪表被加载,以及他们所来自框的ID的顺序。彻底了解每一个单独的DNA样本将在珠片分配是必要的。
- 从他们各自的箱子取出珠芯片的个人银牌包,但是,为了尽量减少对芯片的暴露在空气和光线,还没有打开它们。仔细扫描芯片的条码,并记录在其中,他们将在一个跟踪表被加载,以及他们所来自框的ID的顺序。彻底了解每一个单独的DNA样本将在珠片分配是必要的。
- 就在30分钟冷却完成后,打开银色珠片包。从透明的塑料套中取出芯片。不碰珠,放置在HYB室插入每一个珠片,定向芯片条码与蚀刻到刀片的顶部表面的条码符号。
- 剥离并丢弃样品板的盖子。取15微升样品从样品板并慢慢分配在入口到阵列( 见图3)。临时股东必须小心放置正确的样品的正确珠芯片在正确的位置,匹配先前记录的珠芯片订单。多通道移液管可用于分配液体的芯片,但事先考虑必须采取吸样本只数可以被安全地放置在胎圈芯片,作为在平板上的行数并不总是匹配的数目排在一个芯片上。
- 一旦每个样本被放置其对应的阵列上,目视检查芯片不涂在液体中的气泡或地区。如果存在这些问题,轻轻摇动插入。如果有必要,更多的液体,可以添加到数组中。小心添加正确的DNA样本。
- 打开HYB室的d将插入的加湿水库。芯片的条形码应放置在蚀刻到HYB室基部的条形码。更换HYB室盖,并通过关闭钩子上斜对面两侧先关闭所有四个钩子。
- 孵育HYB室中的烘箱中16-24小时。移动它们时要小心不要倾斜室。
- 如果一个以上的板要被处理,则返回步骤6.2。处理超过24珠片在一天内不建议为了处理双方大量消耗品或芯片时,尽量减少人为错误的可能性,并尽量减少处理芯片在今后的步骤所需的时间量,因为必须小心应当被用来限制其暴露于空气中尽可能。一个瓶子XC4的是足以支持多达24珠芯片。
第三天
7。染色准备
- 从烤箱中取出的HYB室和孵化在室温德温度25分钟。如果在处理两个以上的HYB室,交错的双拆除每10分钟。为了保持芯片的干燥,还没有打开HYB室。
- 而HYB腔冷却,从"后1"删除XC1,XC2,TEM,STM和ATM的管-20°C箱和板凳解冻的设置。拿一瓶RA1从"后2"盒在-20°C(或4℃,如果重复使用来自前一天的试剂)和解冻在室温水浴。解冻95%甲酰胺的管为好。对于已处理,两管各试剂,将10毫升RA1,15毫升甲酰胺,和150毫升XC3(在室温下,公司提供的包装盒上找到)8珠芯片将是必需的。
- 对于每一个处理芯片,一是搏命一塑料流通梅开二度,两个金属扣,以及一个塑料垫片是必需的。对于塑料垫片,只使用了很清楚的;分开并丢弃不透明隔板。此外,两个珠芯片洗碗和一个珠子芯片托盘将是需要的,以及一个液体装配站和一个塑料装配杆。
- 喷洒他们用乙醇和擦拭干燥清洁玻片。
- 打开该附加到流过腔室的机架的热水浴中,将其设定为44℃。轻轻摇动室机架驱逐任何气泡。
- 当HYB室冷却25分钟,填补洗菜与PB1(大约200毫升)。 PB1可以在"后4"室温箱被发现。
- 睁一只HYB室。通过抓住密封在一个角部,并轻轻剥离角( 见图4)从胎圈芯片取下盖子密封。一旦密封被删除,立即放置在一个芯片珠芯片托盘和淹没在PB1不碰珠。重复,直到托盘充满了珠片,同时注意不要让任何芯片干燥。
- 轻轻摇动托盘,以消除任何气泡,离开淹没1分钟的芯片。填写第二个洗菜与PB1。再次搅动托盘。
- 移动的珠片盘进入第二洗的菜。轻轻搅动,让浸泡1分钟,然后再搅拌。
- 在液体流过组装工段,放置4黑色塑料流动通过括号中的凹槽。填充站与PB1,直到液体几乎达到8装配在方括号(约150毫升)中的高度。
- 从纸盒中取出一珠芯片,并将其放置到装配站上一个塑料流通支撑。芯片条形码应该高于蚀刻到组装工位的条形码符号被对齐。重复其他三款芯片,可以在装配站内适合。
- 广场上的珠片顶部的透明塑料垫片。隔离器的外边缘应该装配站内环绕支架。重复淹没在PB1的其他芯片。
- 通过在凹槽拟合它放置在装配站的塑料装配杆。轻轻地放在载玻片上的塑料垫片的顶部由推压装配杆滑动的后端,慢慢地降低前面到液体中。在滑动的顶部的槽应该朝下,芯片的条形码的正上方,使珠芯片和滑动在条形码端之间的间隙。重复淹没在PB1的其他芯片。对于一个完整的流程,通过装配图, 见图5。
- 检查芯片与载玻片之间的气泡。如果出现气泡,轻轻抬起滑动的条形码结束,然后再试一次。持久的气泡可以从玻璃用实验室纸毛巾(的Kimwipe)擦拭。
- 捕捉周围的载玻片两个金属扣,一是对条形码结束和一个朝后。该扣的边缘应握在芯片下的塑性流动,通过支撑。重复淹没在PB1每一个芯片。
- 拆下完成流过组件和水平放置在B恩奇。小心不要小费垂直装配,使载玻片下的液体逃脱。如果有更多的筹码都在等待在洗脸盘,他们可以在相同的PB1进行组装。如果其他芯片都在等待在原来的HYB室,丢弃所有液体在装配站和洗碗,并返回到步骤7.6。
- 一旦所有的珠芯片在其流过组件,取一对手术剪,切隔离物的两端关闭时,在靠近玻璃滑动的可能。
8。染色和扩展
- 验证流通室架已经达到44℃,在多个位置有一个温度探头。如果温度超过半度是关闭的,调节水浴的温度。
- 通过滑动装配下来,钩住支架顶部放置一个珠片流通过装配在室内机架上。胎圈芯片的背面侧,应触摸室架。该g小姑娘幻灯片应朝外,用槽在顶部形成的试剂水库。重复每一个珠片总成。
- 用200μl移液管,由液体分配到玻璃储添加150微升RA1to流过组件。孵育30秒。重复5倍。
- 使用1,000微升移液管,由液体分配到玻璃储添加450微升XC1to流过组件。孵育10分钟。
- 添加450微升XC2以流过组件的液体分配到玻璃储存。孵育10分钟。
- 加入200μl的TEM向流过组件的液体分配到玻璃储存。孵育15分钟。
- 添加450微升95%甲酰胺/ EDTA混合到流过组件的液体分配到玻璃储存。孵育1分钟。重复1X。
- 孵育5分钟,流过组件。
- 设置热水浴的温度升在STM的管(或37°C如果显示没有)isted。确保每个STM管具有所列出的相同的温度。
- 添加450微升XC3到流过组件。孵育1分钟。重复1X。
- 当热水浴温度已达到所需温度时,通过液体分配到玻璃储加250μLSTM到流过组件。孵育10分钟。
- 添加450微升XC3到流过组件的液体分配到玻璃储存。孵育1分钟。重复1X。
- 孵育5分钟,流过组件。
- 加入250μlATM向流过组件的液体分配到玻璃储存。孵育10分钟。
- 添加450微升XC3到流过组件的液体分配到玻璃储存。孵育1分钟。重复1X。
- 孵育5分钟,流过组件。
- 加入250μlSTM的流通通过液体分配到玻璃储集。孵育10分钟。
- ADD4 50微升XC3到流过组件的液体分配到玻璃储存。孵育1分钟。重复1X。
- 孵育5分钟,流过组件。
- 重复步骤8.14至8.19 1X。
- 从腔机架上卸下流通组件和关闭水浴。
9。洗涤和封
- 填写染色珠芯片洗菜与PB1(约315毫升)。两个垂直洗碗和一个垂直珠芯片托盘,将需要与至少一个真空歧管。
- 通过插入卡扣和支架之间的薄金属条,然后旋转拆卸流通组件。抛开载玻片,取出珠芯片。立即将珠片在垂直珠芯片托盘和淹没在PB1。重复每一个流过装配,注意要面对EAC在同一方向H珠芯片和减少其暴露于空气中。
- 轻轻搅动珠芯片托盘,赶走气泡。孵育淹没珠子芯片PB1 5分钟。与XC4填充第二个垂直洗菜前一天准备。再次搅动珠芯片托盘。
- 将珠芯片托盘,洗碟充满了XC4。轻轻摇动托盘,赶走气泡。孵育5分钟,再次搅动。
- 在一个平滑的运动,拉从洗菜珠芯片托盘和试管架设置,让每一个珠片的脸珠向上。带自锁镊子,滑珠芯片出纸盘并将其放置在一个试管架。重复每一个珠片。
- 芯片的试管架小心转移到真空干燥器中。关闭和打开真空,确保适当的密封。孵育50-55分钟。如果有必要,孵化期间热身扫描仪。
10。扫描
- 屠RN关闭真空并缓慢的压力恢复到大气压。检查珠芯片都是干的。如果需要的话,擦拭边缘和用纸巾芯片的底部,以消除任何液体或碎屑。
- 启动扫描软件和移动珠芯片扫描托盘。通过激活解码文件的客户端软件并输入所需的珠芯片的条形码,以及它们对应的框的ID下载芯片的解码文件(DMAPS)。未扫描的珠片可以安全地储存在干燥,避光的地方长达72小时。
- 一旦芯片正确放置在托盘和解码文件是由软件识别,开始扫描。
结果
经过适当处理的珠片应显示明亮,鲜明的红色和绿色激光的光强度,同时扫描。在图6中,ISCAN扫描软件显示一个标准的基因组DNA样品成功地杂交到一个定制面板SNP基因分型阵列。在延长和染色步骤均连接的标记的核苷酸在两个激光器的光的荧光。作为这些核苷酸选择性地延伸的胎圈的寡核苷酸链杂交的片段化的DNA链,并作为该寡核苷酸链被设计成终止于所述变体的位点,由此产生的信号的颜色和强度可以用来确定出现在等位基因SNP位点。
用户生成的样品板,其中CHIPID和位置相匹配的样品ID和临床信息,以及特定于阵列的单核苷酸多态性明显,直接从公司获得的,都是必要的,以便导入SCA新新人类创新输出文件到GenomeStudio分析软件。例如样张可以在公司的网站上找到。一旦GenomeStudio项目建成后,基因型可以由软件自动生成的最终强度集群来获得。虽然多种显示选项存在,默认的视图,规范-R(归一化强度值)与规范 - 西塔(等位基因强度比),往往是最简单的情节来区分三个不同的集群。大多数SNP位点应该显示一个,两个或三个簇,取决于次要等位基因频率。
这三个集群代表样品展示AA,AB,BB或等位基因( 见图7)。这些集群可以通过,或者通过单击并拖动彩色圆圈上的强度地块手动EDI直接从该公司进口的标准簇位置的模板,通过选择"集群中的所有单核苷酸多态性"从软件的分析工具栏被分配的基因型 t调用。样品的强度曲线(红色,紫色或蓝色)的颜色指示调用(AA,AB,BB或分别);黑色表示没有电话。通过在软件的完整资料窗格中列出的SNP位点滚动,聚类每个SNP可以查看或召回。一旦簇被分配令人满意的电话,该软件的完整资料窗格中列出的基因型为每个单独的样品在每一个特定的SNP。上表中的列选择选项可以切换数据格式。
数据可以通过分析工具栏或直接从完整的数据表,深入分析出口。
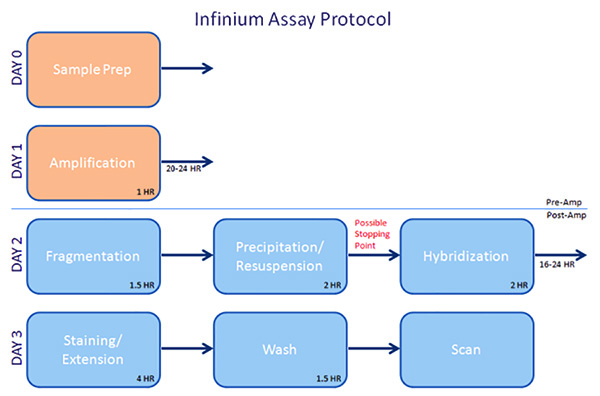
图1。概述- Infinium检测协议。683/50683fig1highres.jpg"目标="_blank">点击这里查看大图。

图2。完整的HYB商会基地和垫子,加上盖子。[12] 点击这里查看大图 。
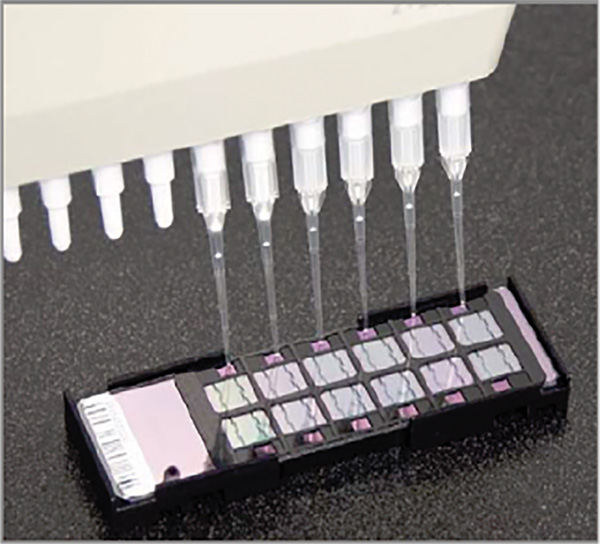
图3载入BeadChip芯片-分装在进气口的样本[12] 点击这里查看大图像。

图4卸下BeadChip芯片Coverseal -抓住封印在拐角处,轻轻地撕掉角[12] 点击这里查看大图 。

图5。完整BeadChip芯片流通过组件 - BeadChip芯片从与一个隔片载玻片分离和结合有扣。>点击这里查看大图。
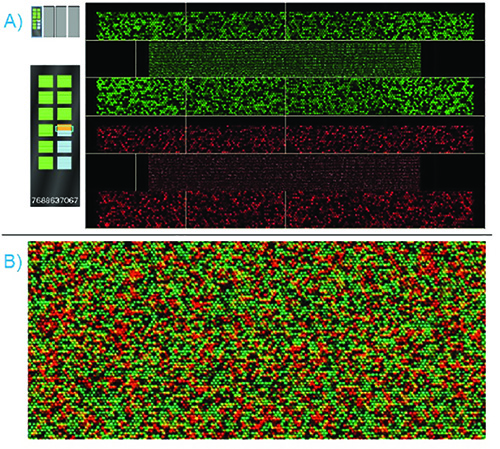
图6。成功BeadChip芯片扫描 - A)本BeadChip芯片进行扫描既具有红色和绿色激光,扫描软件显示两者同时进行。通过强度QC节将突出BeadChip芯片显示屏的左边绿色。失败的强度QC节将突出红色的BeadChip芯片显示屏上。 二)一旦扫描完成后,软件会覆盖在红色和绿色显示。将显示一个放大的图像。每一个人珠子的颜色和强度表示等位基因存在。 点击这里查看大图 。
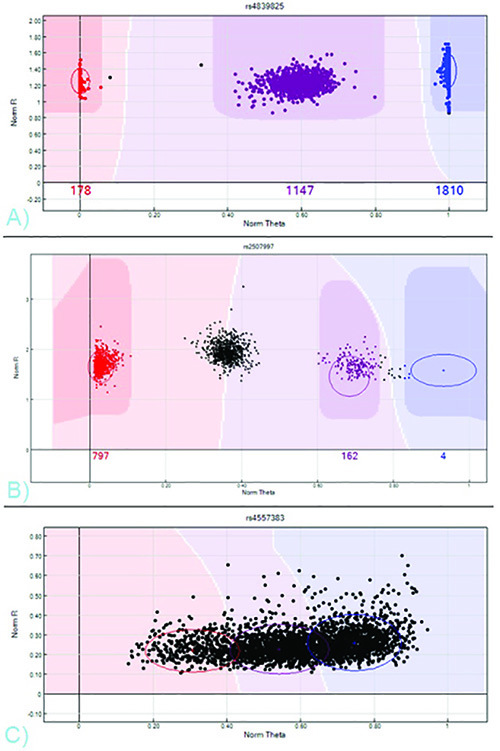
图7。 SNP NORM-R与NORM-THETA集群概况 - A)与代表AA,AB和BB基因型,红色,紫色,蓝色B)的SNP需要编辑三个不同的聚类有效的SNP。中间的集群,这应该是纯合子AB,保留未调用。该BB集群误称为AB型。C)性能较差的单核苷酸多态性。无基因型可以从这个强度图来获得,因为没有明显的集群存在。 点击这里查看大图 。
讨论
大规模基因分型应用已被用来更好地了解潜在许多人类疾病的遗传机制。任何显著变种通过全基因组关联分析发现可以标记一个候选区域进行进一步的研究。此外,基因型数据是对测序项目质量控制的好工具。
为了最大限度地提高样品通量,多个样品板可以被放大和存储在它们分散,再悬浮的状态。八板可以在一天被放大,结合在第一个24小时的协议的多个批次,并提供足够的材料为〜2-8天的芯片的处理。如果放大板被预先储存起来,并且如果新的样本被杂交到芯片扫描开始于上一次运行后,立即处理,而不需要暂停以等待附加的样品制备连续运行。因此,虽然样本将需要三天时间进行完整的测定中,数据可以被每天产生。 Assuming24芯片每天都在处理,5天工作制可让市民能在12样品珠芯片运行在1000的DNA样本。如果任何一个步骤或试剂已经失败,但是,多批次可能会有风险表现不佳的任何修正才能应用。错误可能逃脱的通知,直到阵列扫描或分析,因此,如果吞吐量最大化,数百名在协议中的不同阶段的样本可能已经收到了同样的故障处理时发现。由于失去了试剂和数据无法恢复,用户必须权衡这些风险对需要加速工作流程。
该GenomeStudio分析软件是第一次有机会真正了解基因分型过程的成功与否。如果规范-R与规范 - 西塔强度曲线是正常聚集,平均拆借利率的样本(共SNP位点的百分比成功类型的)应该接近99%,虽然这个数值变化SLIghtly根据数组类型。从与拆借利率低于85-90%为低任何样品的数据是不可靠,应该被丢弃。对于质量控制的目的,结果应该比任何可能的先前已知基因型时。如果没有这样的数据存在,故意重复样品在验证板或阵列安置一个有用的工具。这些重复的对应放在独立的芯片,板,批次或项目;代后其基因型检查。虽然具体的质量控制约束,根据研究者的偏好有所不同,常见的SNP约束是基于样本呼叫成功,Hardy-Weinberg平衡,或病例与对照组之间missingness,而普通样品的限制是基于话费,孟德尔不一致,或交叉引用的X染色体杂合性临床性别数据[13]。
如有任何问题,在控制面板,在分析套件中,可以提交给公司为了确定原因。这些控件通常可以缩小问题最有可能的步骤或试剂失效。如果通过的Infinium基因分型实验中发现任何感兴趣的SNP位点,它们的强度曲线应该是双重检查中GenomeStudio集群的错误进一步的研究进行之前。
一个失败的Infinium基因分型实验可能是由于人为处理错误或劣质输入的DNA。样品定量必须准确和精确。为了达到最佳效果,任何试剂加入到任何样品或芯片必须由协议设置音量进行分配。移液器必须正确校准。试剂不应期满后运行,不应该被再次冷冻,一旦解冻,保存为RA1试剂。为了尽量减少可能的染色和扩展错误,甲酰胺/ EDTA的混合物应新鲜每月编制。所有的-20°C试剂应存放在只有手动除霜冰柜。在ST中使用的所有实验设备癌宁,协议的延伸,洗部分应彻底用清水和中性清洁剂后,立即停止使用冲洗。在HYB室加湿水库应擦洗用试管刷和温和的清洁剂。载玻片应,用10%漂白剂进行洗涤的指示,由他们的用户手册,每周一次。
披露声明
这篇文章的作者有没有竞争的经济利益披露。
致谢
资助这项工作得到了美国国立卫生研究院P20 GM103456,美国国立卫生研究院RC2 AR058959,和NIH R56 AI063274提供
材料
| Name | Company | Catalog Number | Comments |
| Consumable or Equipment | Manufacturer | Part Number | Minimum Required for 96 Samples |
| 0.8 ml Deep Well Plate | Thermo Scientific | AB-0765 | 1 |
| Plate Mats | Thermo Scientific | AB-0674 | 2 |
| Reagent Basin | Fisher Scientific | 13-681-502 | 9 |
| Heat-seal Sheets | Thermo Scientific | AB-0559 | 1 |
| Flow-through Spacer | Fisher Scientific | NC9563984 | 6 |
| Pipette tips - 200 μl | Rainin | GP-L200F | 192 |
| Pipette tips - 10 μl | Rainin | GP-L10F | 16 |
| Pipette tips - 1,000 μl | Rainin | GP-L1000F | 16 |
| DNA Suspension Buffer | Teknova | T0220 | 0.5 ml |
| 0.1 N NaOH | Fisher Scientific | AC12419-0010 | 0.5 ml |
| Isopropanol (HPLC grade) | Fisher Scientific | A451 | 15 ml |
| Ethanol (200-proof) | Sigma-Aldrich | 459836 | 330 ml |
| Formamide (100%) | Thomas Scientific | C001K38 | 15 ml |
| EDTA (0.5 M) | Amresco | E177 | 0.2 ml |
| 10 μl 8-channel Pipette | Rainin | L8-10XLS | 1 |
| 200 μl 8-channel Pipette | Rainin | L8-200XLS | 2 |
| 1,000 μl Single-channel Pipette | Rainin | L-1000XLS | 1 |
| Microplate Shaker | VWR | 13500-890 | 1 |
| Refrigerated Microplate Centrifuge | VWR | BK369434 | 1 |
| Hybridization Oven | Illumina | SE-901-1001 | 1 |
| Hybex Microsample Incubator | SciGene | 1057-30-0 | 1 |
| Hybex MIDI Heat Block Insert | Illumina | BD-60-601 | 1 |
| Heat Sealer | Thermo Scientific | AB-0384 | 1 |
| Hyb Chamber w/ Insert and Mat | Illumina | BD-60-402 | 2 |
| Surgical Scissors | Fisher Scientific | 13-804-20 | 1 |
| Flow Through Assembly Parts | Illumina | WG-10-202 | 8 |
| Wash Rack and Dish | Illumina | BD-60-450 | 1 |
| Genepaint Chamber Rack | Tecan | 760-800 | 1 |
| Temperature Probe | Illumina | A1-99-109 | 1 |
| Staining Rack and Dish | Illumina | WG-10-207 | 1 |
| Self-Closing Tweezers | Ted Pella, Inc | 5374-NM | 1 |
| Vacuum Manifold | Ted Pella, Inc | 2240 | 1 |
| iScan or HiScan | Illumina | - | 1 |
参考文献
- Shi, M. M. Enabling large-scale pharmacogenetic studies by high-throughput mutation detection and genotyping technologies. Clin. Chem. 47 (2), 164-172 (2001).
- Livak, K. J. SNP genotyping by the 5'-nuclease reaction. Methods Mol. Biol. 212, 129-148 (2003).
- Seeb, J. E., Pascal, C. E., Ramakrishnan, R., Seeb, L. W. SNP genotyping by the 5'-nuclease reaction: advances in high throughput genotyping with non-model organisms. Methods in Mol. Biol. 578, 277-292 (2009).
- Bayés, M., Gut, I. G., ed, . Overview of Genotyping. Molecular Analysis and Genome Discovery. , 1-23 (2011).
- Lee, Y., Seifert, S. N., Fornadel, C. M., Norris, D. E., Lanzaro, G. C. Single-Nucleotide Polymorphisms for High-Throughput Genotyping of Anopheles arabiensis in East and Southern Africa. J. Med. Entomol. 49 (2), 307-315 (2012).
- Liu, Z. SNP Genotyping Platforms. Next generation sequencing and whole genome selection in aquaculture. , 123-132 (2011).
- Shen, R., Fan, J. B., et al. High-throughput SNP genotyping on universal bead arrays. Mutat. Res./Fundam and Mol. Mech. of Mutagenesis. 573 (1), 70-82 (2005).
- Ragoussis, J. Genotyping technologies for all. Drug Discov. Today: Technol. 3 (2), 115-122 (2006).
- Wu, C. C., Shete, S., Jo, E. J., et al. Whole-Genome Detection of Disease-Associated Deletions or Excess Homozygosity in a Case-Control Study of Rheumatoid Arthritis. Hum. Mol. Genet. , (2012).
- Green, E. K., Hamshere, M., Forty, L., et al. Replication of bipolar disorder susceptibility alleles and identification of two novel genome-wide significant associations in a new bipolar disorder case-control sample. Mol. Psychiatry. , (2012).
- Shete, S., Hosking, F. J., Robertson, L. B., et al. Genome-wide association study identifies five susceptibility loci for glioma. Nat. Genet. 41 (8), 899-904 (2009).
- Inc Illumina. . Infinium HD Ultra User Guide 11328087 RevB-1. , 15-101 (2009).
- Turner, S., Armstrong, L. o. r. e. n. L., Yuki Bradford, ., et al. Quality Control Procedures for Genome Wide Association Studies. Curr Protoc Hum Genet. , 1-19 (2011).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。