Method Article
Infinium Assay pour Grands génotypage SNP Applications
Dans cet article
Résumé
Un protocole est décrit qui utilise les tests de Infinium d'Illumina pour effectuer le génotypage à grande échelle. Ces dosages peuvent fiable génotype millions de SNP à travers des centaines d'échantillons d'ADN individuels en trois jours. Une fois généré, ces génotypes peuvent être utilisés pour vérifier les associations avec une variété de différentes maladies ou phénotypes.
Résumé
variantes de génotypage du génome humain s'est avérée être une méthode efficace pour identifier les associations génétiques des phénotypes. La distribution des variantes au sein des familles ou des populations peut faciliter l'identification des facteurs génétiques de la maladie. Le panneau d'Illumina de BeadChips de génotypage permet aux enquêteurs de génotype des milliers ou des millions de polymorphismes nucléotidiques simples (SNP) ou d'analyser d'autres variantes génomiques, tels que le nombre de copie, à travers un grand nombre d'échantillons d'ADN. Ces SNP peuvent être répartis sur tout le génome ou ciblées dans des régions spécifiques afin de maximiser le potentiel de découverte. L'essai Infinium a été optimisé pour obtenir rapidement de haute qualité, des résultats fiables. Avec une configuration correcte, un seul technicien peut traiter de quelques centaines à plus d'un millier d'échantillons d'ADN par semaine, selon le type de tableau. Ce test guide les utilisateurs à travers chaque étape, en commençant par l'ADN génomique et se terminant avec le balayage de la matrice. Utilisation de bienséance Reagents, les échantillons sont amplifiés, fragmenté, précipité, remis en suspension, hybridé à la puce, prolongée par une base unique, souillé, et scanné soit sur un système d'imagerie optique à haute résolution iScan ou Salut Scan. Une étape d'une nuit est nécessaire pour amplifier l'ADN. L'ADN est dénaturé et isotherme amplifié par amplification du génome entier et, par conséquent, pas de PCR est nécessaire. Les échantillons sont hybridées aux puces à ADN au cours d'une seconde étape d'une nuit. Dès le troisième jour, les échantillons sont prêts à être numérisée et analysée. L'ADN amplifié peut être stocké en grandes quantités, ce qui permet des arrangements de billes à traiter chaque jour de la semaine, en maximisant ainsi le débit.
Introduction
Taper polymorphismes nucléotidiques simples (SNP) est une méthode clé de l'identification des variantes de risque associés à la maladie. Historiquement, le champ d'application de génotypage a été limitée par la technologie disponible. méthodes de génotypage basés sur l'électrophorèse sur gel sont limitées dans l'échantillon-et SNP-débit [1]. Le développement de ces tests peut souvent être main-d'œuvre, en s'appuyant sur la composition et la structure de la région entourant la variante pour l'optimisation [1]. TaqMan de génotypage, développé par Life Technologies, peuvent exécuter un grand nombre d'échantillons rapidement et avec la participation de technicien minimal [2], mais les restrictions SNP-multiplexage continuent de limiter le nombre total de génotypes à bien moins d'un million par jour [3,4 ]. La plate-forme iPlex Sequenom peut également exécuter plusieurs échantillons à la fois, mais, étant donné que moins d'une centaine de SNPs peuvent être multiplexés, ce débit est comparable globalement faible [5]. La technologie de flux SNP de Beckman Coulterpourrait théoriquement produire plus de trois millions de génotypes par jour, mais cette gamme limites de la technologie du projet à un maximum de seulement quarante-huit SNPs par réaction [4,6]. Bien que l'essai GoldenGate peut traiter près de deux cents échantillons d'ADN chaque jour sur des centaines ou des milliers de SNP par exemple, le prix par génotype n'est pas compétitif avec des techniques ultra-haut débit de pointe lors de la saisie de plus de trois mille SNP à la fois [4,7 ]. Afin de traiter plusieurs millions de génotypes par jour, à l'échelle requise pour les grandes études d'association pangénomique, analyses tableau d'hybridation sont devenus l'option la plus rentable sur le marché.
La ligne de Affymetrix de tableaux d'hybridation et de la ligne d'Illumina de tableaux basés sur Infinium permettent potentiellement des centaines d'échantillons à taper sur des centaines de milliers ou des millions de SNP en parallèle [4,8]. Ces SNP peuvent être dispersés sur l'ensemble du génome, localisée dans les régions d'intérêt, tels que les exomes, ou sur mesure selon les préférences de l'utilisateur. Ces tableaux ont l'avantage d'être non seulement en mesure de déterminer le génotype précision d'un million de SNPs par échantillon à la fois, mais aussi de mesurer la variation du nombre de copies, des anomalies chromosomiques potentiellement dévoilement. Infinium alignés tableaux OMNI BeadChip ont actuellement la capacité de génotype à près de cinq millions de marqueurs par échantillon, y compris un demi-million de loci de coutume, sur un maximum de près d'une centaine d'échantillons chaque jour.
Comme la plupart des maladies ont une composante génétique, ces expériences à grande échelle peuvent être crucial dans la recherche des gènes associés à la maladie. Génotypage à haut débit permet de production efficace de génotype dans l'échantillon fixe suffisante pour détecter de façon convaincante association génétique à des fréquences des allèles mineurs inférieurs. Projets de génotypage du génome entier peuvent être utilisés pour localiser des régions avec statistiquement significative cas-contrôle la fréquence des allèles ou des différences du nombre de copies [9,10,11]. Selon le National Human GeInstitut de recherche nome, les études d'association du génome a conduit à 1490 publications distinctes entre le 25 Novembre 2008 et le 25 Janvier 2013, découlant de la découverte de 8283 SNP avec une p-valeur inférieure à 1 x 10 -5 (voir http:// www.genome.gov/gwastudies/). Ces études, qui fait des recherches sur des conditions allant de la hauteur de cancer du testicule, bénéficié de l'approche globale offerte par une analyse de l'ensemble du génome. Dans de tels cas, des régions entières d'intérêt pourraient avoir échappé à l'attention avait la portée de taper trop restrictive. Ainsi, pour les analyses d'association à grande échelle, une technique de génotypage du génome est la technique de choix.
Différentes versions de l'essai Infinium existent, chacun destiné à être utilisé avec des types spécifiques de tableaux. Le dosage InfiniumUltra, discuté en détail ci-dessous, est approprié pour beaucoup 12 - ou 24-échantillons des puces de tableau. Ce génotype souvent plus de cent mille SNP par échantillon d'ADN et de se concentrer sur trégions argeted, comme sur exome ou personnalisés panneaux. D'autres versions de dosage peuvent être nécessaires pour d'autres types de puces, telles que les matrices de génotypage du génome entier. Cependant, comme tous les tests Infinium partagent une base commune et diffèrent principalement que par les noms de réactifs, les volumes de réactifs ou de la procédure de réactif de coloration exacte, techniques perfectionnées sur une seule version d'essai peuvent souvent être universellement appliquées. D'autres tableaux, comme les tableaux de méthylation, peuvent utiliser un protocole pratiquement identique, aussi bien. Il faut prendre soin de n'utiliser que la version du dosage requis pour le type de puce en cours d'utilisation. Certains types, tels que ceux mesurant le niveau d'expression des gènes, peuvent nécessiter l'utilisation d'un protocole de nonInfinium.
Les échantillons doivent être traitées par lots. Par exemple, avec le dosage InfiniumUltra, tubes de réactif de pré-hybridation contiennent un volume suffisant pour exécuter 96 échantillons, et les tubes ne peuvent être recongelés. Par conséquent, les échantillons doivent être analysés en lots de 96 échantillons à la fois. Les échantillons seront amplifiés sur les sapinst jour. Après environ 1 heure de travail d'établi, les échantillons doivent être chauffés dans un four à convection pendant 20-24 h. Le lendemain, près de 4 heures sera consacré fragmentation, la précipitation, et remise en suspension des échantillons, à quel point les échantillons peuvent être soit congelés pour une utilisation ultérieure ou hybridées sur la puce. Chargement puces prend près de 2 heures, après quoi les échantillons seront hybrides pendant une nuit pour 16-24 heures. Le troisième jour, l'étape de coloration et d'extension prend ~ 4 heures. Une heure supplémentaire sera consacré à laver, de revêtement, et le séchage des copeaux. Enfin, les tableaux sont analysés, ce qui peut prendre de 15 à 60 min / puce, selon le type utilisé.
Les précautions de sécurité de laboratoire et de propreté standard s'appliquent. Bien que basée sur la PCR de l'amplification n'est pas, les postes de travail séparés pour pré-et postamplification procédures sont nécessaires afin de minimiser la probabilité de contamination. Le numéro d'identification de chaque réactif du kit fourni dans l'utilisation doit être connecté sur une feuille de suivi. Reagents doivent être décongelés avant utilisation et immédiatement inversés plusieurs fois avant la distribution. L'ADN doit être dactylographié doit être de l'ADN génomique de haute qualité (260/280 rapport d'absorbance de 1,6-2,0, 260/230 rapport d'absorbance inférieure à 3,0), isolé par des méthodes classiques et quantifiés avec un fluorimètre. La dégradation de l'ADN est souvent un facteur contribuant dans les résultats d'analyse de qualité médiocre. Typiquement, 200 ng d'ADN est nécessaire, si ce montant peut varier pour certains types de puces. Un robot de liquide de traitement Tecan peut automatiser de nombreuses étapes du protocole et de minimiser l'erreur humaine comme un facteur.
Protocole
Day One
Une. Préparation
- Distribuer 200 ng d'ADN dans une plaque de 96 échantillons de puits profonds. Au moins 96 échantillons (plaques complètes) doivent être étalées afin de s'assurer qu'aucun réactif sera gaspillé. Étiqueter la plaque avec une étiquette code à barres fournis par le kit et centrifuger.
- Afin de normaliser les volumes, laisser les échantillons dans un tiroir ou une hotte fermée la nuit pour évaporer le liquide. Recouvrir la plaque lâche avec un couvercle ou une serviette en papier pour protéger de la poussière.
2. Amplification
AVERTISSEMENT: les échantillons ne doivent pas subir une amplification à moins de 4 heures sera disponible le jour suivant pour les étapes de fragmentation, les précipitations, et remise en suspension.
- Retirez le boîtier fourni par l'entreprise ou le paquet de tubes étiquetés "Pre" du congélateur à -20 ° C (un tube de MA1, MA2, et MSM est suffisant pour chaque série de 96 échantillons). Réglez tubes de MA2 et MSM sur le banc à dégeler. Mettez correctement calibréfour et à 37 ° C.
- L'utilisation d'un bassin de réactif et un pl 10, 8 canaux pipette, distribuer 4 pi de tampon de resuspension ADN dans chaque puits pour réhydrater les échantillons. Il n'est pas nécessaire de jeter les embouts de pipette entre chaque colonne si l'on prend soin de ne pas toucher le liquide.
- Avec une pipette à 8 canaux, passer 20 pi de MA1 dans chaque puits de la plaque. Pour chaque nouveau réactif, utiliser un bassin de réactif frais. Couvrir la plaque avec un joint réutilisable, impulsion centrifugeuse, et le vortex pendant 1 min à 1600 rpm sur un agitateur de microplaques.
- 2,4) à incuber à température ambiante pendant au moins 30 min.
- Distribuer 4 pl de NaOH 0,1 N dans chaque puits de la plaque. Couvrir la plaque avec le joint réutilisable, impulsion centrifugeuse, et le vortex pendant 1 min à 1600 rpm.
- Incuber à température ambiante pendant 10 min.
- Distribuer 34 ul de MA2 dans chaque puits de la plaque.
- Distribuer 38 ul de MSM dans chaque puits de la plaque. Couvrir la plaque avec lejoint réutilisable, impulsion centrifugeuse, et le vortex pendant 1 min à 1600 rpm.
- Placez au four pendant 20-24 h.
Deuxième jour
3. Fragmentation
- Retirer les tubes de la FMS "Post-1" -20 ° C case (un tube est suffisant pour chaque ensemble de 96 échantillons). Décongeler sur le banc ou dans un bain d'eau à température ambiante. Après l'insertion de l'insert MIDI-plaque dans un système de table pour micro d'incubation, mettez le bloc de feu et à 37 ° C.
- Une fois les tubes sont décongelés, retirer la plaque du four et l'échantillon impulsion centrifugeuse. Distribuer 25 ul de FMS dans chaque puits de la plaque de l'ADN. Replacez le couvercle, impulsion centrifugeuse, et le vortex à 1600 rpm pendant 1 min.
- Incuber la plaque dans le bloc de la chaleur pendant 1 heure.
4. Précipitation
- Retirez le tube de PM1 de "Post-3" C boîte de 4 ° ou emballer et chaud à la température ambiante (un tube est suffisant pour chaque série de 96 échantillons). Retirer la plaque du bloc thermiquek et impulsion centrifugeuse.
- Distribuer 50 ul de PM1 dans chaque puits de la plaque. Replacez le couvercle, impulsion centrifugeuse, et le vortex à 1600 rpm pendant 1 min.
- Incuber la plaque à bloc chauffant pendant 5 minutes.
- Retirer la plaque du bloc thermique, l'éteindre, et jeter le couvercle de la plaque. Distribuer 155 pl d'isopropanol à 100% dans chaque puits de la plaque. Couvrir hermétiquement avec un nouveau couvercle et retourner manuellement la plaque plusieurs fois pour mélanger.
- Placer la plaque dans 4 ° C pendant un minimum de 30 minutes. Mettez centrifugeuse réfrigérée et réglé à 4 ° C pour refroidir.
- Équilibrer la plaque et centrifuger à 4 ° C pendant 20 min à 3000 x g.
- Inspecter le fond de la plaque sans l'inverser et confirment les échantillons sont précipités dans une pastille bleue. Si aucun des pastilles peuvent être revus, centrifugeuse. Obtenez des serviettes en papier.
- Jeter le couvercle et retirez rapidement le liquide en inversant la plaque et en le frappant avec force sur la paillasse recouverte de serviettes en papier. Une fois la plaque inverted, prendre soin de ne pas revenir tout en reste du liquide. Appuyez plusieurs fois sur la plaque contre la paillasse jusqu'à ce que tout le liquide est retiré.
- Placer la plaque sur un porte-tube à essai, inversé, à sécher. Incuber à température ambiante pendant 1 heure.
5. La remise en suspension
- Retirer RA1 de "Post-2" -20 ° C boîte et dégel dans la salle de bain d'eau à température. Allumez le four et réglez-le à 48 ° C
- Une fois décongelé, distribuer 23 ul de RA1 à chaque puits de la plaque de l'échantillon. Ne pas verser toute la bouteille de RA1 dans le bassin; économiser 30 ml pour plus tard. Si les échantillons seront hybridés sur les réseaux tard dans la journée, placez le RA1 dans un refroidisseur C 4 °. Si les échantillons seront exécutés à une date ultérieure, l'étiquette de la bouteille et recongeler RA1.
- De thermo-scellage d'un nouveau couvercle sur la plaque. Pulse-centrifuger la plaque et placez-le dans le four pendant 1 heure.
- Retirer la plaque du four et tourbillon à 1800 rpm pendant 1 min.
Les échantillons peuvent être en toute sécurité hchamp à ce stade jusqu'à une semaine. Les plaques peuvent être stockées, et les échantillons peuvent être réorganisés pour se préparer à l'application de la puce de perles, si nécessaire. Si le traitement de la procédure, laissez la plaque à température ambiante et mettre sur le bloc thermique. Sinon, stocker la plaque à -20 ° C.
6. Hybridation
AVERTISSEMENT: Les échantillons doivent pas subir hybridation moins 5,5 h sont disponibles le jour suivant pour la coloration et étapes de lavage.
- Allumez le bloc de feu et à 95 ° C. Allumez le four et réglez-le à 48 ° C
- Une fois la température stabilisée, incuber la plaque sur le bloc chauffant pendant 20 min.
- Tandis que la plaque est la dénaturation, enlever la boîte de copeaux de perles de 4 ° C et le mettre sur le banc. Prenez une bouteille de XC4 (de la trousse à température ambiante) et ajouter 330 ml d'éthanol à 100%. Secouez bien et incuber à température ambiante pendant une nuit. Préparer formamide / EDTA mélange (95% de formamide, 0,2%EDTA (0,5 M), 4,8% de H 2 O en volume) et de geler en incréments de 15 ml à chaque fois. L'excès de formamide peut être stocké.
- Retirer la plaque de l'échantillon à partir du bloc thermique. Incuber à température ambiante pendant 30 min.
- Tandis que la plaque se refroidit, préparer la chambre de hyb. Une chambre sera nécessaire pour tous les quatre puces perles transformés.
Placer un tapis de caoutchouc sur la partie supérieure du fond de chambre de hyb, en alignant le trou plus épais avec l'avant de la base. Le symbole de code à barres gravé dans la base doit encore être visible (voir la figure 2). - En utilisant une pipette de 1000 pi, distribuer 400 pi de PB2 dans chacun des huit réservoirs d'humidification sculptés dans la base. PB2 peut être trouvé dans la boîte ou paquet «post-3". Placer le couvercle de la chambre de hyb sur la base et serrer deux extrémités par deux agrafes de fermeture sur des côtés opposés en diagonale premier.
- Retirez les paquets d'argent individuels de copeaux de perles de leurs boîtes respectives, mais, afin de minimiser l'exposition des pucesAssurez-vous de l'air et de la lumière, ne pas ouvrir encore eux. Analyser soigneusement les codes à barres des puces et enregistrer l'ordre dans lequel ils seront chargés sur une feuille de suivi, ainsi que les identifiants de la boîte d'où ils venaient. Une compréhension approfondie de l'endroit où chaque échantillon d'ADN individuel sera distribué sur les puces de perles est nécessaire.
- Retirez les paquets d'argent individuels de copeaux de perles de leurs boîtes respectives, mais, afin de minimiser l'exposition des puces à l'air et la lumière, ne pas ouvrir encore eux. Analyser soigneusement les codes à barres des puces et enregistrer l'ordre dans lequel ils seront chargés sur une feuille de suivi, ainsi que les identifiants de la boîte d'où ils venaient. Une compréhension approfondie de l'endroit où chaque échantillon d'ADN individuel sera distribué sur les puces de perles est nécessaire.
- Juste avant les 30 min refroidir a terminé, ouvrez les paquets de copeaux de perles d'argent. Retirer les morceaux de leurs manches en plastique transparent. Sans toucher les billes, placez chaque puce de perles sur un insert de chambre de hyb, orientant la pucecode-barres avec le symbole de code à barres gravé dans la surface supérieure de l'insert.
- Décoller et jeter le couvercle de la plaque de l'échantillon. Prendre 15 pl d'échantillon à partir de la plaque d'échantillons et distribuer lentement sur l'entrée de la matrice (voir figure 3). Soin extraordinaire doit être prise pour placer l'échantillon correct sur la puce de cordon adéquat dans la bonne position, correspondant à la commande de puce de perles enregistrée plus tôt. Une pipette à canaux multiples peut être utilisé pour distribuer le liquide sur les puces, mais prévoyance doivent être prises pour aspirer seulement le nombre d'échantillons qui peut être placé en toute sécurité sur la puce de talon, comme le nombre de lignes sur une plaque ne correspond pas toujours au nombre de rangées sur une puce.
- Une fois chaque échantillon a été placé sur son tableau correspondant, inspecter visuellement la puce de bulles ou régions non revêtues en liquide. Si ces problèmes existent, secouez-la délicatement l'insert. Si nécessaire, davantage de liquide peut être ajouté au tableau. Prenez soin d'ajouter l'échantillon d'ADN correcte.
- Ouvrez la une de la chambre de hybd placer les inserts sur les réservoirs d'humidification. Le code à barres de la puce doit être placé au-dessus du code à barres gravé dans le fond de chambre de hyb. Replacez le couvercle de la chambre de hyb et fermez toutes les quatre crochets en fermant les agrafes sur les côtés opposés en diagonale premier.
- Incuber la chambre de hyb au four pendant 16 à 24 h. Prenez soin de ne pas incliner les chambres lors de leur déplacement.
- Si plus d'une plaque doit être traitée, retourner à l'étape 6.2. Traitement de plus de 24 puces de perles dans une journée n'est pas recommandé pour à la fois réduire la probabilité d'une erreur humaine lors de la manipulation d'une grande quantité de consommables ou de copeaux et de minimiser la quantité de temps nécessaire pour traiter les jetons dans les étapes à venir, que les soins doivent être prises pour limiter leur exposition à l'air autant que possible. Une bouteille de XC4 est suffisante pour jusqu'à 24 puces de perles.
Troisième jour
7. Préparation coloration
- Enlever les chambres de hyb du four et laisser incuber à manger température pendant 25 min. Si le traitement de plus de deux chambres de hyb, échelonner leur double élimination toutes les 10 min. Afin de garder les puces de séchage, ne pas encore ouvrir les chambres de hyb.
- Alors que les chambres de hyb sont de refroidissement, retirer les tubes de XC1, XC2, TEM, STM et ATM de la "Post-1" -20 ° C case et mettre sur le banc à dégeler. Prenez une bouteille de RA1 de la boîte "Post-2" à -20 ° C (ou 4 ° C si la réutilisation du réactif de la veille) et dégel dans un bain d'eau à température ambiante. Décongeler un tube de 95% de formamide ainsi. Pour 8 perles puces transformés, deux tubes de chaque réactif, 10 ml, 15 ml RA1 formamide, et 150 ml XC3 (qui se trouve dans une boîte fourni par l'entreprise à température ambiante) sera nécessaire.
- Pour chaque puce traitée, diapositive d'un verre, une entretoise d'écoulement au travers du plastique, deux agrafes métalliques, et une entretoise en plastique seront nécessaires. Pour l'entretoise en plastique, utilisez uniquement le clair un; séparer et jeter l'entretoise opaque. En outre, deux plats et de lavage de puce perlesun plateau de jeu de billes sera nécessaire, avec une station d'assemblage de liquide et une barre d'assemblage plastique.
- Nettoyez les lames de verre par pulvérisation avec de l'éthanol et de l'essuyer les sécher.
- Allumez le bain d'eau chaude qui est attaché à la porte de la chambre accréditive et le mettre à 44 ° C. Agitez doucement la porte de la chambre à déloger les bulles.
- Lorsque la chambre de hyb a refroidi pendant 25 minutes, remplir un plat de lavage avec PB1 (environ 200 ml). PB1 peut être trouvé dans la zone de température "Post-4" chambre.
- Ouvrez une chambre de hyb. Retirer le joint du couvercle d'une puce de perles en saisissant le joint dans un coin et doucement le pelage en diagonale (voir figure 4). Une fois que le joint est enlevé, de placer immédiatement la puce dans un plateau de jetons de talon et plonger dans PB1 sans toucher les billes. Répétez jusqu'à ce que le bac est rempli de copeaux de perles, en prenant soin de ne pas laisser puce sec.
- Agiter doucement le plateau pour enlever les bulles et laisser les puces immergées pendant 1 min.Remplissez un deuxième plat de lavage avec PB1. Agiter à nouveau le plateau.
- Déplacer le plateau de jetons de perles dans le deuxième plat de lavage. Agiter doucement et laisser tremper pendant 1 min, puis agiter à nouveau.
- Dans le liquide poste d'assemblage dynamique, placer quatre plastique accolades accréditives noir dans les rainures. Remplir le poste PB1 à ce que le liquide atteigne à peu près la hauteur des crochets d'assemblage de huit (environ 150 ml).
- Retirer une puce de perles du bac et le placer dans le poste d'assemblage sur un doublé accréditives plastique. Le code à barres de la puce doit être aligné au-dessus du symbole de code à barres gravé dans le poste d'assemblage. Répétez l'opération pour les trois autres puces qui peuvent tenir dans le poste d'assemblage.
- Placez une entretoise en plastique transparente sur le dessus d'une puce de perles. Les bords extérieurs de l'élément d'espacement doivent entourer les supports à l'intérieur de la station d'assemblage. Répétez l'opération pour les autres puces immergés dans le PB1.
- Placer la barre de montage en plastique dans le poste d'assemblage par emboîtement au-dessus des rainures. Placer délicatement une lame de verre sur le dessus de l'entretoise en plastique en appuyant sur l'extrémité arrière de la lame sur la barre de montage et abaisser lentement l'avant dans le liquide. La rainure sur la partie supérieure de la lame doit être tourné vers le bas, directement au-dessus du code à barres de la puce, en laissant un espace entre la puce de talon et le coulisseau à la fin du code à barres. Répétez l'opération pour les autres puces immergés dans le PB1. Pour une représentation complète de montage dynamique, voir Figure 5.
- Vérifiez bulles entre la puce et un toboggan. Si des bulles apparaissent, soulever délicatement la lame à la fin du code à barres et essayez à nouveau. Bulles persistantes peuvent être nettoyés de verre avec une serviette en laboratoire de papier (Kimwipe).
- Enclencher deux agrafes métalliques autour de la lame de verre, une vers l'extrémité de code à barres et l'autre vers l'arrière. Les bords des agrafes doit saisir le plastique de renfort accréditives en vertu de la puce. Répétez l'opération pour chaque puce immergé dans PB1.
- Retirer les ensembles accréditives complétées et placer horizontalement sur le bench. Prenez soin de ne pas basculer l'ensemble verticalement, permettant au liquide sous la lame de verre de s'échapper. Si plus de jetons sont en attente dans le bac de lavage, ils peuvent être réunis sous le même PB1. Si d'autres jetons sont en attente dans leurs chambres d'hyb originaux, jeter tout liquide dans le poste d'assemblage et laver la vaisselle, et retourner à l'étape 7.6.
- Une fois toutes les puces de perles sont dans leurs assemblées accréditives, prendre une paire de ciseaux chirurgicaux et coupez les deux extrémités de l'entretoise loin, plus près de la lame de verre que possible.
8. Coloration et extension
- Vérifiez que le porte-chambre écoulement continu a atteint 44 ° C, dans des positions multiples d'une sonde de température. Si la température est décalée de plus d'un demi-degré, ajuster la température du bain d'eau.
- Placez un ensemble de flux par puce de perles sur la grille de la chambre en faisant glisser l'ensemble vers le bas et accrocher l'accolade au sommet. La face arrière de la puce de talon doit toucher la grille de la chambre. Le gdiapositive jeune fille doit être tourné vers l'extérieur, avec la rainure vers le haut pour former un réservoir de réactif. Répétez l'opération pour chaque ensemble de puce de perles.
- En utilisant une pipette de 200 ul, 150 ul RA1to ajouter les ensembles à circulation continue par la distribution du liquide dans le réservoir de verre. Incuber pendant 30 sec. Répétez 5x.
- En utilisant une pipette de 1000 pi, ajouter 450 ul XC1to les assemblées accréditives par la distribution du liquide dans le réservoir de verre. Incuber pendant 10 min.
- Ajouter 450 ul XC2 aux assemblées accréditives par la distribution du liquide dans le réservoir de verre. Incuber pendant 10 min.
- Ajouter 200 pi de TEM aux assemblées accréditives par la distribution du liquide dans le réservoir de verre. Incuber pendant 15 min.
- Ajouter 450 ul 95% de formamide / EDTA mélange aux assemblées accréditives par la distribution du liquide dans le réservoir de verre. Incuber pendant 1 min. Répétez 1x.
- Incuber les ensembles accréditives pour 5 min.
- Régler le bain d'eau chaude à la température de lexisté sur les tubes de la STM (ou 37 ° C si aucun n'est représenté). Assurez-vous que chaque tube STM a la même température indiqué.
- Ajouter 450 ul XC3 aux assemblées accréditives. Incuber pendant 1 min. Répétez 1x.
- Lorsque la température du bain d'eau chaude a atteint la température désirée, ajouter 250 μLSTM aux ensembles à circulation continue par la distribution du liquide dans le réservoir de verre. Incuber pendant 10 min.
- Ajouter 450 ul XC3 aux assemblées accréditives par la distribution du liquide dans le réservoir de verre. Incuber pendant 1 min. Répétez 1x.
- Incuber les ensembles accréditives pour 5 min.
- Ajouter 250 ul ATM aux assemblées accréditives par la distribution du liquide dans le réservoir de verre. Incuber pendant 10 min.
- Ajouter 450 ul XC3 aux assemblées accréditives par la distribution du liquide dans le réservoir de verre. Incuber pendant 1 min. Répétez 1x.
- Incuber les ensembles accréditives pour 5 min.
- Ajouter 250 ul STM à l'écoulement continuassemblées par la distribution du liquide dans le réservoir en verre. Incuber pendant 10 min.
- Add4 50 pi XC3 aux assemblées accréditives par la distribution du liquide dans le réservoir de verre. Incuber pendant 1 min. Répétez 1x.
- Incuber les ensembles accréditives pour 5 min.
- Répétez les étapes 8.14 à 8.19 1x.
- Retirer les ensembles accréditives de l'armoire de la chambre et éteindre le bain d'eau.
9. Laver et étanchéité
- Remplir un plat de lavage de puce coloration de perle avec PB1 (environ 315 ml). Deux plats de lavage verticales et une verticale plateau de jetons de talon seront nécessaires, et aussi d'au moins un collecteur à vide.
- Démonter un ensemble de flux continu en insérant une barre métallique mince entre les fermoirs et l'accolade, puis faire pivoter. Mettez de côté la lame de verre et enlever la puce de perles. Placer immédiatement la puce de talon dans le bac à puce perle verticale et plonger dans PB1. Répétez l'opération pour chaque ensemble de flux continu, en prenant soin de faire face each puce de talon dans la même direction et en minimisant leur exposition à l'air.
- Agiter doucement le plateau de jetons de perle pour éliminer les bulles. Incuber les copeaux de billes immergées dans PB1 pendant 5 min. Remplissez le deuxième plat de lavage vertical avec le XC4 préparé la veille. Agiter à nouveau le plateau de jetons de talon.
- Déplacez le plateau de jetons de talon au plat de lavage remplie de XC4. Agiter doucement le plateau pour éliminer les bulles. Incuber pendant 5 min, puis bien mélanger.
- En un seul mouvement, tirez le plateau de jetons de perle de l'antenne de lavage et de mettre sur une grille de tube à essai, de sorte que les perles sur chaque face de coupe de talon haut. Avec des pincettes autobloquants, faites glisser une puce de perles du bac et placer sur une grille de tube à essai. Répétez l'opération pour chaque puce de perles.
- Soigneusement transférer le support de tube à essai de puces dans un dessiccateur à vide. Fermez et mettez le vide, assurant une bonne étanchéité. Incuber pendant 50-55 min. Si nécessaire, chauffer le scanner pendant l'incubation.
10. Numérisation
- Turn l'aspirateur et revenez lentement à la pression atmosphérique. Vérifiez que les puces de billes sont sèches. Si nécessaire, essuyez les bords et le fond de la puce avec une serviette en papier pour enlever tout liquide ou de débris.
- Activez le logiciel de numérisation et changer les jetons de perles de plateau de balayage. Télécharger les fichiers de décodage de la puce (de DMAPS) en activant le logiciel client de décodage de fichiers et la saisie des codes-barres aux pépites de perles désirées, avec leurs cartes d'identité de zone correspondant. Copeaux de perles non analysés peuvent en toute sécurité être stockés dans un endroit sec et sombre jusqu'à 72 h.
- Une fois que les puces sont bien en place dans le bac et les fichiers de décodage sont reconnus par le logiciel, lancer la numérisation.
Résultats
Une puce de perles bien-traité devrait afficher des intensités de lumière laser rouge et vert lumineux et distincts pendant la numérisation. Dans la figure 6, le logiciel de numérisation iScan affiche un échantillon d'ADN génomique norme hybride avec succès à une coutume panneau tableau SNP de génotypage. Les nucléotides marqués qui ont été attachés pendant la prolongation et de coloration étapes fluorescence sous les lumières des deux lasers. Comme ces nucléotides s'étendent de façon sélective la chaîne oligonucléotidique du bourrelet hybridée au brin de l'ADN fragmenté, et que les chaînes oligonucléotidiques sont conçues pour se terminer au niveau du site de la variante, la couleur et l'intensité du signal résultant peut être utilisé pour déterminer les allèles présents à l' place SNP.
Une feuille généré par les utilisateurs échantillon, ce qui correspond ID d'échantillon et les renseignements cliniques à CHIPID et la position, et un SNP manifeste spécifique au réseau, obtenu directement à partir de l'entreprise, sont à la fois nécessaire pour importer le scales fichiers de sortie de nner dans le logiciel d'analyse GenomeStudio. Feuilles exemple d'échantillons peuvent être trouvés sur le site Web de l'entreprise. Une fois le projet GenomeStudio est construit, les génotypes peuvent être obtenus à partir des pôles d'intensité résultant générés automatiquement par le logiciel. Bien que plusieurs options d'affichage existent, la vue par défaut, Norm-R (une valeur d'intensité normalisée) vs Norm-Theta (un ratio de l'intensité allélique), est souvent l'intrigue la plus facile de différencier trois groupes distincts. La plupart des SNP devraient montrer un, deux, ou trois groupes, en fonction de la fréquence de l'allèle mineur.
Les trois groupes représentent des échantillons démontrant AA, AB ou BB allèles (voir Figure 7). Ces groupes peuvent être affectés génotypes soit par l'importation d'un modèle de position de regroupement standard directement à partir de l'entreprise, en sélectionnant «Cluster Tous les SNP" de la barre d'outils d'analyse du logiciel, ou en cliquant et en faisant glisser les cercles colorés sur les parcelles d'intensité à l'EDI manuellement t appelle. La couleur de l'échantillon sur le terrain de l'intensité (rouge, violet ou bleu) indique que l'appel (AA, AB ou BB, respectivement); noir indique aucun appel. En faisant défiler les SNP répertoriés dans le volet de données complète du logiciel, le regroupement pour chaque SNP peut être consulté ou rappelé. Une fois que les groupes sont affectés appels satisfaisants, le volet de données complète du logiciel répertorie les génotypes pour chaque échantillon à chaque SNP particulier. L'option Colonne Sélecteur dessus de la table peut basculer formats de données.
Les données peuvent être exportées, soit par la barre d'outils d'analyse ou directement à partir de la table de données complet pour une analyse en profondeur.
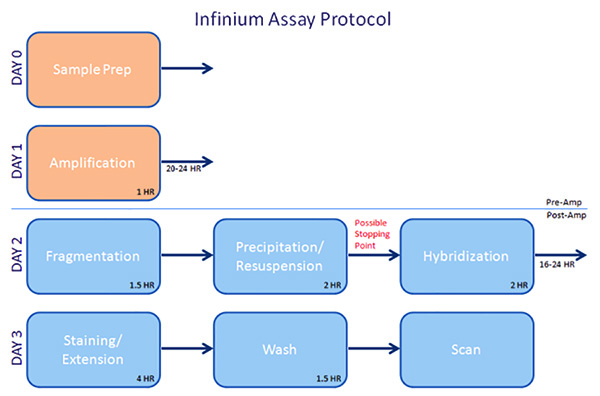
Figure 1. Aperçu - Infinium protocole de test.683/50683fig1highres.jpg "target =" _blank "> Cliquez ici pour agrandir l'image.

Figure 2. Complète Hyb Chambre base et tapis, plus couvercle. [12] Cliquez ici pour agrandir l'image .
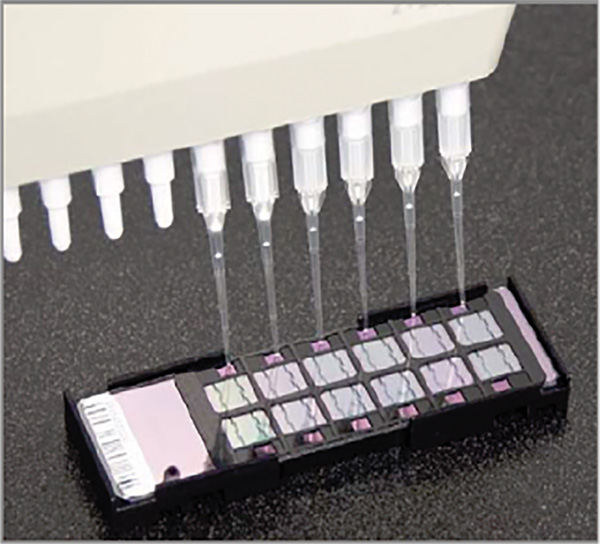
Figure 3 Chargement d'un BeadChip -.. Déposer l'échantillon sur les ports d'entrée [12] Cliquez ici pour agrandir l'image.

Figure 4 Retrait de la BeadChip Coverseal -.. Saisissez le joint dans le coin et retirez délicatement diagonale [12] Cliquez ici pour agrandir l'image .

Figure 5. Ensemble complet BeadChip accréditives - Le BeadChip est séparée d'une lame de verre avec une entretoise et lié avec des agrafes.> Cliquez ici pour agrandir l'image.
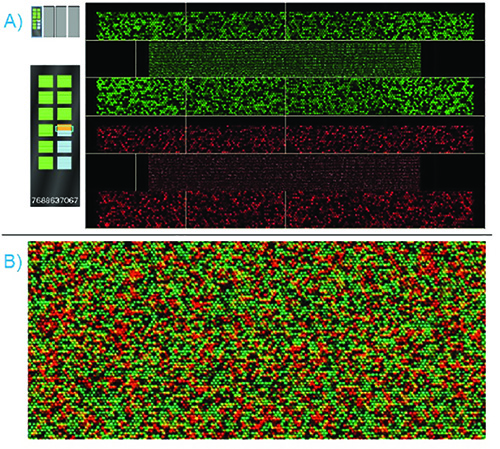
Figure 6. Le succès de numérisation BeadChip - A) Le BeadChip est scanné à la fois un laser rouge et vert, les logiciels de numérisation affiche à la fois simultanément. Sections passant intensité QC mettront en lumière verte sur l'écran de BeadChip à gauche. Sections défaut intensité QC mettra en lumière rouge sur l'écran de BeadChip. B) Une fois la numérisation terminée, le logiciel superposer les affichages rouges et verts. Une image zoomée s'affiche. La couleur et l'intensité de chaque bille individuelle indique l'allèle présent. Cliquez ici pour agrandir l'image .
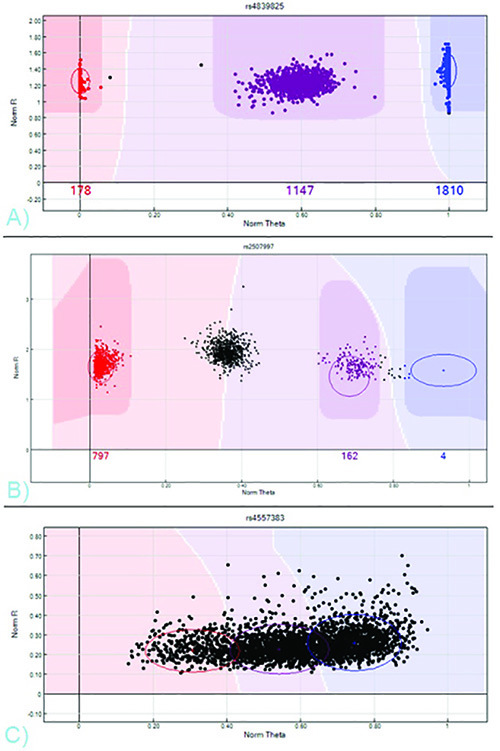
Figure 7. SNP NORM-R vs-Theta NORM Profils du cluster - A) Un SNP valide avec trois groupes distincts représentant AA, AB, BB et génotypes, de couleur rouge, violet et bleu B) Un SNP nécessitant édition.. Le groupe du milieu, qui doit être homozygote AB, est laissé non appelé. Le groupe BB est appelée à tort AB. C) Un SNP peu performant. Pas de génotypes peuvent être obtenus à partir de cette parcelle d'intensité, comme aucun grappes distinctes existent. Cliquez ici pour agrandir l'image .
Discussion
Applications de génotypage à grande échelle ont été utilisées pour mieux comprendre le mécanisme génétique sous-jacente de nombreuses maladies humaines. La découverte d'une variante importante grâce à une analyse de génome d'association peut signaler une région candidate pour une étude plus approfondie. En outre, les données de génotype est un bon outil pour le contrôle de la qualité des projets de séquençage.
Pour maximiser le débit d'échantillons, de plaques échantillons multiples peuvent être amplifiés et stockés dans leurs Etats, remis en suspension fragmentés. Huit plaques peuvent être amplifiées en une seule journée, en combinant les 24 premières heures du protocole pour plusieurs lots et de fournir suffisamment de matière pour ~ 2-8 jours de la puce de traitement. Si les plaques amplifiés sont stockées à l'avance, et si de nouveaux échantillons sont hybridées à des puces immédiatement après la numérisation commence sur la course précédente, le traitement peut fonctionner en continu sans avoir besoin de faire une pause pour la préparation de l'échantillon supplémentaire. Par conséquent, bien que les échantillons auront trois jours pour subir la complèteessai, les données peuvent être générés quotidiennement. copeaux de Assuming24 sont traitées chaque jour, une semaine de travail de 5 jours permet depuis plus de 1000 échantillons d'ADN pour être exécuté sur une puce de perles 12-échantillon. Si une étape ou réactif a échoué, cependant, plusieurs lots pourraient être à risque de mauvaise performance avant toute correction peut être appliquée. Des erreurs peuvent échapper à l'attention jusqu'à ce que les réseaux sont scannés ou analysés, par conséquent, si le débit est maximisé, des centaines d'échantillons à différents stades du protocole pourraient déjà avoir reçu le même traitement défectueux lors de la découverte. Comme les réactifs et les données perdues ne peuvent pas être récupérés, l'utilisateur doit peser ces risques contre la nécessité d'un flux de travail accéléré.
Le logiciel d'analyse GenomeStudio est la première occasion de mesurer réellement la réussite du processus de génotypage. Si la norme R-vs parcelles d'intensité Norm-Theta sont bien regroupés, le taux moyen de l'appel (pour cent du total SNP tapé avec succès) des échantillons devrait approcher 99%, bien que cette valeur varie sliselon ghtly le type de tableau. Les données de n'importe quel échantillon avec un taux inférieur à 85-90% d'appels n'est pas digne de confiance et doivent être jetés. À des fins de contrôle de la qualité, les résultats doivent être comparés à des génotypes déjà connues chaque fois que possible. Si aucune donnée n'existe, intentionnelle duplication de l'échantillon est un outil utile pour vérifier la plaque ou tableau placements. Ces paires en double doivent être placés sur des puces séparées, des assiettes, des lots, ou des projets; leurs génotypes vérifiés lors de la génération. Bien que les contraintes QC spécifiques varient en fonction de la préférence de l'enquêteur, les contraintes communes SNP sont basés sur la réussite exemple d'appel, Hardy-Weinberg, ou manquantes entre les cas et les témoins, tandis que les contraintes de l'échantillon communes sont basés sur les taux d'appel, les incohérences de Mendel, ou des références croisées de X-chromosome hétérozygotie des données cliniques entre les sexes [13].
En cas de problème, le tableau de bord des contrôles, a trouvé dans la suite de l'analyse, peut être soumis à laentreprise afin de déterminer la cause. Ces contrôles peuvent souvent limiter la question à l'étape la plus probable ou l'échec de réactif. Si des SNP d'intérêt se trouvent à travers une expérience de génotypage Infinium, leurs parcelles d'intensité doivent être vérifiées dans GenomeStudio pour les erreurs de regroupement avant est effectué de nouvelles recherches.
Une expérience de génotypage Infinium pas est probablement dû à une erreur de manipulation humaine ou l'ADN d'entrée de mauvaise qualité. quantification de l'échantillon doit être exacte et précise. Pour de meilleurs résultats, tous les réactifs ajoutés à un échantillon ou puce doivent être dispensés au volume fixé par le protocole. Les pipettes doivent être correctement calibrés. Réactifs ne doivent pas être exécutés après expiration et ne doivent pas être recongelés une fois décongelé, sauf pour le réactif RA1. Afin de minimiser les erreurs de coloration et de vulgarisation possibles, le mélange formamide / EDTA doit être préparé chaque mois. Tous les -20 C ° réactifs doivent être stockés dans des congélateurs à dégivrage non automatique seulement. Tout le matériel de laboratoire utilisé dans la mAining, de vulgarisation, et lavage des parties du protocole doivent être rincés abondamment à l'eau et un détergent doux dès désuétude. Les réservoirs d'humidification dans la chambre de hyb doivent être lavés avec une brosse en tube à essai et un détergent doux. Les lames de verre doivent être lavés avec l'eau de Javel à 10%, selon les instructions de leurs manuels de l'utilisateur, une fois par semaine.
Déclarations de divulgation
Les auteurs de cet article n'ont pas d'intérêts financiers concurrents de divulguer.
Remerciements
Le financement de ce travail a été fourni par le NIH P20 GM103456, NIH RC2 AR058959, et les NIH R56 AI063274
matériels
| Name | Company | Catalog Number | Comments |
| Consumable or Equipment | Manufacturer | Part Number | Minimum Required for 96 Samples |
| 0.8 ml Deep Well Plate | Thermo Scientific | AB-0765 | 1 |
| Plate Mats | Thermo Scientific | AB-0674 | 2 |
| Reagent Basin | Fisher Scientific | 13-681-502 | 9 |
| Heat-seal Sheets | Thermo Scientific | AB-0559 | 1 |
| Flow-through Spacer | Fisher Scientific | NC9563984 | 6 |
| Pipette tips - 200 μl | Rainin | GP-L200F | 192 |
| Pipette tips - 10 μl | Rainin | GP-L10F | 16 |
| Pipette tips - 1,000 μl | Rainin | GP-L1000F | 16 |
| DNA Suspension Buffer | Teknova | T0220 | 0.5 ml |
| 0.1 N NaOH | Fisher Scientific | AC12419-0010 | 0.5 ml |
| Isopropanol (HPLC grade) | Fisher Scientific | A451 | 15 ml |
| Ethanol (200-proof) | Sigma-Aldrich | 459836 | 330 ml |
| Formamide (100%) | Thomas Scientific | C001K38 | 15 ml |
| EDTA (0.5 M) | Amresco | E177 | 0.2 ml |
| 10 μl 8-channel Pipette | Rainin | L8-10XLS | 1 |
| 200 μl 8-channel Pipette | Rainin | L8-200XLS | 2 |
| 1,000 μl Single-channel Pipette | Rainin | L-1000XLS | 1 |
| Microplate Shaker | VWR | 13500-890 | 1 |
| Refrigerated Microplate Centrifuge | VWR | BK369434 | 1 |
| Hybridization Oven | Illumina | SE-901-1001 | 1 |
| Hybex Microsample Incubator | SciGene | 1057-30-0 | 1 |
| Hybex MIDI Heat Block Insert | Illumina | BD-60-601 | 1 |
| Heat Sealer | Thermo Scientific | AB-0384 | 1 |
| Hyb Chamber w/ Insert and Mat | Illumina | BD-60-402 | 2 |
| Surgical Scissors | Fisher Scientific | 13-804-20 | 1 |
| Flow Through Assembly Parts | Illumina | WG-10-202 | 8 |
| Wash Rack and Dish | Illumina | BD-60-450 | 1 |
| Genepaint Chamber Rack | Tecan | 760-800 | 1 |
| Temperature Probe | Illumina | A1-99-109 | 1 |
| Staining Rack and Dish | Illumina | WG-10-207 | 1 |
| Self-Closing Tweezers | Ted Pella, Inc | 5374-NM | 1 |
| Vacuum Manifold | Ted Pella, Inc | 2240 | 1 |
| iScan or HiScan | Illumina | - | 1 |
Références
- Shi, M. M. Enabling large-scale pharmacogenetic studies by high-throughput mutation detection and genotyping technologies. Clin. Chem. 47 (2), 164-172 (2001).
- Livak, K. J. SNP genotyping by the 5'-nuclease reaction. Methods Mol. Biol. 212, 129-148 (2003).
- Seeb, J. E., Pascal, C. E., Ramakrishnan, R., Seeb, L. W. SNP genotyping by the 5'-nuclease reaction: advances in high throughput genotyping with non-model organisms. Methods in Mol. Biol. 578, 277-292 (2009).
- Bayés, M., Gut, I. G. Overview of Genotyping. Molecular Analysis and Genome Discovery. ed, , 2nd, John Wiley & Sons, Ltd. Chichester, UK. 1-23 (2011).
- Lee, Y., Seifert, S. N., Fornadel, C. M., Norris, D. E., Lanzaro, G. C. Single-Nucleotide Polymorphisms for High-Throughput Genotyping of Anopheles arabiensis in East and Southern Africa. J. Med. Entomol. 49 (2), 307-315 (2012).
- Liu, Z. SNP Genotyping Platforms. Next generation sequencing and whole genome selection in aquaculture. , Wiley-Blackwell. 123-132 (2011).
- Shen, R., Fan, J. B., et al. High-throughput SNP genotyping on universal bead arrays. Mutat. Res./Fundam and Mol. Mech. of Mutagenesis. 573 (1), 70-82 (2005).
- Ragoussis, J. Genotyping technologies for all. Drug Discov. Today: Technol. 3 (2), 115-122 (2006).
- Wu, C. C., Shete, S., Jo, E. J., et al. Whole-Genome Detection of Disease-Associated Deletions or Excess Homozygosity in a Case-Control Study of Rheumatoid Arthritis. Hum. Mol. Genet. , (2012).
- Green, E. K., Hamshere, M., Forty, L., et al. Replication of bipolar disorder susceptibility alleles and identification of two novel genome-wide significant associations in a new bipolar disorder case-control sample. Mol. Psychiatry. , (2012).
- Shete, S., Hosking, F. J., Robertson, L. B., et al. Genome-wide association study identifies five susceptibility loci for glioma. Nat. Genet. 41 (8), 899-904 (2009).
- Inc Illumina. Infinium HD Ultra User Guide 11328087 RevB-1. , 15-101 (2009).
- Turner, S., Armstrong, L. orenL., Yuki Bradford,, et al. Quality Control Procedures for Genome Wide Association Studies. Curr Protoc Hum Genet. , 1-19 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon