Method Article
Infinium Ensaio para a grande escala Applications genotipagem
Neste Artigo
Resumo
Um protocolo é descrito que utiliza ensaios Infinium Illumina para realizar genotipagem em larga escala. Estes ensaios podem genótipo de forma confiável milhões de SNPs em centenas de amostras de DNA individuais em três dias. Uma vez gerados, os genótipos pode ser usado para verificar as associações com uma variedade de diferentes doenças ou fenótipos.
Resumo
Genotipagem variantes no genoma humano provou ser um método eficiente para identificar associações genéticas com fenótipos. A distribuição de variantes dentro das famílias ou populações pode facilitar a identificação dos fatores genéticos da doença. Painel de BeadChips genotipagem da Illumina permite aos investigadores ao genótipo milhares ou milhões de polimorfismos de nucleotídeo único (SNPs) ou analisar outras variantes genômicas, tais como número de cópias, através de um grande número de amostras de DNA. Estes SNPs podem ser espalhados por todo o genoma ou alvo em regiões específicas, a fim de maximizar o potencial de descoberta. O ensaio Infinium foi otimizado para produzir de alta qualidade, resultados precisos rapidamente. Com uma configuração adequada, um único técnico pode processar a partir de algumas centenas a mais de um milhar de amostras de ADN por semana, dependendo do tipo de matriz. Este ensaio orienta os usuários através de cada passo, começando com DNA genômico e terminando com a varredura da matriz. Usando decoro REAGEnts, as amostras são amplificados, fragmentado, precipitado, ressuspenso, hibridizado com o chip, prorrogado por uma única base, manchado, e digitalizadas em cada um de alta resolução sistema de imageamento óptico iScan ou Hi Scan. Um passo durante a noite é necessário para amplificar o ADN. O DNA é desnaturado e isotermicamente amplificado por amplificação de todo o genoma e, portanto, não é necessária a PCR. As amostras são hibridizadas para as matrizes durante um segundo passo durante a noite. No terceiro dia, as amostras estão prontas para serem digitalizados e analisados. O ADN amplificado pode ser armazenados em grandes quantidades, o que permite matrizes grânulo para ser processado a cada dia da semana, maximizando assim o rendimento.
Introdução
Digitando polimorfismos de nucleotídeo único (SNPs) é um método fundamental de identificar variantes de risco associados à doença. Historicamente, o âmbito de experiências de genotipagem tiver sido limitados pela tecnologia disponível. Métodos de genotipagem baseadas em electroforese em gel estão limitadas na amostra e SNP throughput [1]. O desenvolvimento destes ensaios muitas vezes pode ser trabalhoso, contando com a composição e estrutura da região em torno da variante de otimização [1]. Ensaios TaqMan genotipagem, desenvolvido pela Life Technologies, pode executar um grande número de amostras de forma rápida e com o mínimo envolvimento técnico [2], mas as restrições SNP-multiplexação continuam a limitar o número total de genótipos para bem menos de um milhão por dia [3,4 ]. Plataforma Iplex da Sequenom também pode executar muitas amostras de uma só vez, mas, como menos de cem SNPs podem ser multiplexados em conjunto, o throughput é comparativamente baixa global [5]. Tecnologia fluxo SNP da Beckman Coulterpoderia, teoricamente, produzir mais de três milhões de genótipos por dia, mas esta faixa de limites de tecnologia de projeto para um máximo de apenas quarenta e oito SNPs por reação [4,6]. Enquanto o ensaio GoldenGate pode processar quase duas centenas de amostras de DNA de cada dia em centenas ou milhares de SNPs por amostra, o preço por genótipo não é competitivo com, técnicas avançadas de ultra-alto rendimento ao digitar mais de três mil SNPs de uma só vez [4,7 ]. A fim de processar vários milhões de genótipos por dia, a escala necessária para grandes estudos de associação do genoma, os ensaios matriz de hibridização tornaram-se a opção mais rentável no mercado.
Linha de Affymetrix de matrizes de hibridização e linha de matrizes à base de Infinium da Illumina permitir potencialmente centenas de amostras a ser digitado em centenas de milhares ou milhões de SNPs em paralelo [4,8]. Estes SNPs podem ser espalhados por todo o genoma, localizada em regiões de interesse, como a exomes, ou personalizado com a preferência do usuário. Estas matrizes têm a vantagem de não apenas ser capaz de genótipo com precisão um milhão de SNPs por amostra de uma vez, mas também para medir a variação do número de cópias, anormalidades cromossômicas potencialmente revelando. Infinium alinhados matrizes OMNI BeadChip atualmente tem a capacidade de genótipo até quase cinco milhões de marcadores por amostra, incluindo meio milhão de loci costume, em até cerca de cem amostras de cada dia.
Como a maioria das doenças têm um componente genético, essas experiências em larga escala pode ser crucial na busca de genes associados com a doença. De alta capacidade para a geração de genotipagem permite genótipo eficiente no exemplo define grande o suficiente para detectar de forma convincente de associação genética em freqüências alélicas menores inferiores. Projectos de genotipagem todo o genoma pode ser usado para localizar regiões com freqüência do alelo de caso-controle estatisticamente significativa ou diferenças no número de cópias [9,10,11]. De acordo com o National Human GeInstituto de Pesquisa de nome, estudos de associação ampla de genoma levou a 1.490 publicações separadas entre 25 de novembro de 2008 e 25 de janeiro de 2013, decorrente da descoberta de SNPs 8283 com um valor de p menor que 1 x 10 -5 (ver http:// www.genome.gov/gwastudies/). Esses estudos, que pesquisou as condições que vão desde a altura ao câncer testicular, beneficiou da abordagem ampla proporcionada por uma análise de todo o genoma. Em casos como estes, regiões inteiras de interesse poderia ter escapado à atenção tinha o escopo de digitação sido demasiado restritiva. Assim, para análises de associação em grande escala, uma técnica de genotipagem de todo o genoma é a técnica de escolha.
Existem diferentes versões do ensaio Infinium, cada um projetado para uso com tipos específicos de arrays. O ensaio InfiniumUltra, discutidos em profundidade a seguir, é adequado para muitos 12 - ou lascas de matriz 24 de amostra. Estes, muitas vezes o genótipo mais de cem mil SNPs por amostra de DNA e se concentrar em targeted regiões, como em exome ou personalizados painéis. Outras versões do ensaio podem ser necessárias para outros tipos de chips, como as matrizes de genotipagem de todo o genoma. No entanto, como todos os ensaios Infinium compartilham uma base comum e diferem principalmente apenas pelos nomes de reagentes, os volumes de reagentes, ou o procedimento exato reagente coloração, técnicas aperfeiçoadas em uma versão ensaio muitas vezes pode ser universalmente aplicado. Outras matrizes, como matrizes de metilação, pode usar um protocolo quase idênticas, como bem. Cuidados devem ser tomados ao utilizar apenas a versão de ensaio necessários para o tipo de chip em uso. Alguns tipos, como as que medem nível de expressão de genes, pode exigir o uso de um protocolo de nonInfinium.
As amostras devem ser processadas em lotes. Por exemplo, com o ensaio de InfiniumUltra, tubos de ensaio de pré-hibridação conter um volume suficiente para executar 96 amostras, e os tubos não podem voltar a ser congelados. Portanto, as amostras devem ser executados em lotes de 96 amostras de cada vez. As amostras serão amplificados sobre os abetosdia t. Após aproximadamente 1 hora de benchwork, as amostras devem ser aquecidos num forno de convecção durante 20-24 hr. No dia seguinte, cerca de 4 horas, será gasto fragmentação, precipitação, e ressuspensão das amostras, no ponto em que as amostras podem ser congeladas para uso futuro ou hibridadas com o chip. Carregando fichas leva quase 2 horas, após o qual as amostras serão hibridizados durante a noite por 16-24 horas. No terceiro dia, o passo de coloração e extensão leva ~ 4 horas. Uma hora mais será gasto lavagem, revestimento, secagem e os chips. Finalmente, as matrizes são verificados, o que pode levar 15-60 min / chip, dependendo do tipo usado.
As precauções padrão de segurança de laboratório e limpeza aplicar. Embora a amplificação não é à base de RCP, as estações de trabalho separados para pré-e postamplification procedimentos são necessários a fim de minimizar a probabilidade de contaminação. O número de cada reagente fornecido pelo kit em uso de identificação deverá ser registrado em uma folha de rastreamento. Reagents devem ser descongeladas imediatamente antes da utilização e invertido várias vezes antes de dispensar. O DNA a ser digitado deve ser ADN genómico de alta qualidade (260/280 razão de absorvâncias de 1,6-2,0, 260/230 razão de absorvâncias de abaixo de 3,0), isolado através de métodos padrão e quantificados com um fluorómetro. Degradação de DNA é muitas vezes um fator que contribui para os resultados do ensaio de baixa qualidade. Normalmente, é necessária a 200 ng de ADN, embora esta quantidade pode variar de alguns tipos de chip. A-manipulação líquido Tecan robô pode automatizar muitas etapas do protocolo e minimizar o erro humano como um fator.
Protocolo
Day One
1. Preparação
- Dispensar a 200 ng de ADN em, uma placa de 96 amostras de poços profundos. Pelo menos 96 amostras (placas completos) deve ser banhado para garantir que nenhum reagente será desperdiçado. Rotular a placa com um adesivo de código de barras fornecido pelo kit e centrifugar isso.
- A fim de normalizar os volumes, deixar as amostras numa gaveta ou exaustor de fumos durante a noite para evaporar o líquido. Cobrir a placa solta com uma tampa ou papel toalha para impedir a entrada de poeira.
2. Amplificação
AVISO: As amostras não devem ser submetidos a amplificação, a menos de 4 horas estará disponível no dia seguinte para as etapas de fragmentação, precipitação e ressuspensão.
- Remova a caixa fornecido pelo empresa ou pacote de tubos rotulados "Pré" do freezer -20 ° C (um tubo de MA1, MA2, e MSM é suficiente para cada conjunto de 96 amostras). Definir tubos de MA2 e HSH no banco para descongelar. Ligue devidamente calibradoforno e ajustado para 37 ° C.
- Utilizando uma bacia de reagente e um pi 10, 8 canais de pipeta, dispensar 4 ul de DNA buffer ressuspensão em cada poço para re-hidratar as amostras. Não é necessário descartar as pontas de pipeta entre cada coluna se o cuidado de não tocar líquido.
- Com uma pipeta de 8 canais, 20 ul de dispensar MA1 em cada poço da placa. Para cada novo reagente, use uma bacia de reagente fresco. Cobrir a placa com um vedante reutilizável, pulso-centrífuga, e vortex durante 1 min a 1600 rpm num agitador de microplacas.
- 2.4) Incubar à temperatura ambiente durante pelo menos 30 min.
- Dispensar 4 ul de NaOH 0,1 N a cada poço da placa. Cobrir a placa com a vedação reutilizável, pulso-centrífuga, e vortex durante 1 min a 1600 rpm.
- Incubar à temperatura ambiente durante 10 min.
- Dispensar 34 ul de MA2 em cada poço da placa.
- Dispensar 38 ul de MSM em cada poço da placa. Cobrir a placa com oselo reutilizável, pulso-centrífuga, e vortex por 1 min a 1.600 rpm.
- Coloque no forno por 20-24 horas.
Dia dois
3. Fragmentação
- Remova os tubos FMS do "Post-1" -20 ° C caixa (um tubo é suficiente para cada conjunto de 96 amostras). Descongelar no banco ou num banho de água à temperatura ambiente. Depois de inserir a inserção MIDI-plate em um sistema de mesa microsample incubação, ligue o bloco de aquecimento e ajuste a 37 ° C.
- Uma vez que os tubos são descongelados, retire a placa da amostra do forno e pulso-centrífuga. Dispensar 25 mL de SFM em cada poço da placa de ADN. Substituir a tampa, pulso-centrífuga, e no vórtice 1600 rpm durante 1 min.
- Incubar placa em bloco de aquecimento por 1 hora.
4. Precipitação
- Retire o tubo PM1 de "Post-3" caixa de 4 ° C ou embalar e quente à temperatura ambiente (um tubo é suficiente para cada conjunto de 96 amostras). Retire a placa do bloco de calork e pulso-centrífuga.
- Dispensar 50 ul de PM1 em cada poço da placa. Substituir a tampa, pulso-centrífuga, e no vórtice 1600 rpm durante 1 min.
- Incubar placa em bloco de aquecimento por 5 min.
- Retire a placa do bloco de aquecimento, desligue-o, e descartar a tampa da placa. Dispensar 155 mL de isopropanol a 100% a cada poço da placa. Tampe com uma nova tampa e inverter manualmente a placa várias vezes para misturar.
- Colocar no prato 4 ° C durante um mínimo de 30 min. Ligue centrífuga refrigerada e ajustado para 4 ° C e arrefecer.
- Equilibre a placa e centrifugar a 4 ° C durante 20 min a 3.000 x g.
- Inspeccionar o fundo da placa, sem invertendo-a e confirmar as amostras são precipitados num sedimento azul. Se não há aglomerados pode ser visto, centrifugar novamente. Obter toalhas de papel.
- Descartar a tampa e remover rapidamente o líquido invertendo a placa e batendo-o com força na bancada coberta em toalhas de papel. Uma vez que a placa é inverted, tome cuidado para não revertê-lo enquanto qualquer líquido permanece. Repetidamente bata a placa contra a bancada, até que todo o líquido é removido.
- Coloque o prato em um rack tubo de ensaio, invertido, para secar. Incubar à temperatura ambiente durante 1 hora.
5. Ressuspensão
- Remover RA1 de "Post-2" -20 ° C e caixa de degelo na sala banho de água de temperatura. Ligue o forno e coloque-o a 48 ° C.
- Uma vez descongelado, dispensar 23 ul de RA1 a cada poço da placa de amostra. Não despeje toda a garrafa de RA1 na bacia; salvar 30 ml para mais tarde. Se as amostras serão hibridizados para as matrizes mais tarde naquele dia, coloque o RA1 em um C mais frio de 4 °. Se as amostras será realizada em uma data posterior, rotular o frasco e voltar a congelar o RA1.
- Heat-selar uma nova tampa para o prato. Pulso-centrífuga a placa e coloque-o no forno por 1 hora.
- Remova a placa do forno e vórtice a 1.800 rpm durante 1 min.
As amostras podem ser seguramente held neste estágio por até uma semana. As placas podem ser armazenadas, e as amostras podem ser reorganizados para se preparar para a aplicação de chip grânulo, se necessário. Se continuar com o processo, deixar a placa à temperatura ambiente e ligar o bloco de calor. Caso contrário, a chapa de armazenar a -20 ° C.
6. Hibridização
AVISO: As amostras não devem ser submetidos a hibridização, a menos de 5,5 horas estão disponíveis no dia seguinte para a coloração e lavar passos.
- Ligue o bloco de calor e configurá-lo para 95 ° C. Ligue o forno e coloque-o a 48 ° C.
- Uma vez que a temperatura tenha estabilizado, incubar a placa sobre o bloco de aquecimento durante 20 min.
- Enquanto a placa é desnaturação, remover a caixa de batatas fritas talão de 4 ° C e coloque-o no banco. Pegue uma garrafa de XC4 (a partir de kit em temperatura ambiente) e adicione 330 ml de etanol 100%. Agite bem e incubar à temperatura ambiente durante a noite. Preparar a mistura de formamida / EDTA (95% formamida, 0,2%EDTA (0,5 M), 4,8% de H 2 O, em volume) e congelar em incrementos de 15 ml separados. O excesso de formamida podem ser estocados.
- Remova a placa de amostra a partir do bloco de calor. Incubar à temperatura ambiente durante 30 min.
- Enquanto a placa está esfriando, prepare a câmara hyb. Uma câmara será necessária para cada quatro chips de talão processados.
Coloque um tapete de borracha no topo da base da câmara hyb, alinhando o furo mais espessa com a parte frontal da base. O símbolo de código de barras gravadas na base ainda deve ser visível (ver Figura 2). - Usando uma pipeta de 1.000 ul, 400 ul de dispensar PB2 em cada um dos oito reservatórios de humidificação entalhados na base. PB2 pode ser encontrada na caixa "Post-3" ou bando. Coloque a tampa da câmara hyb na base e apertar as duas extremidades ao fechar dois fechos em lados opostos diagonalmente primeiro.
- Retirar as embalagens individuais de prata de fichas de talão das suas respectivas caixas, mas, a fim de minimizar a exposição dos 'chipsCertifique-se de ar e luz, ainda não abri-los. Analisar cuidadosamente os códigos de barras dos chips e gravar a ordem em que eles serão carregados em uma folha de rastreamento, juntamente com as identificações de caixa de onde vieram. Uma compreensão completa de onde cada amostra de DNA individual será distribuído sobre os chips do grânulo é necessário.
- Retirar as embalagens individuais de prata de fichas de talão das suas respectivas caixas, mas, a fim de minimizar a exposição das fichas de ar e luz, mas não abri-los. Analisar cuidadosamente os códigos de barras dos chips e gravar a ordem em que eles serão carregados em uma folha de rastreamento, juntamente com as identificações de caixa de onde vieram. Uma compreensão completa de onde cada amostra de DNA individual será distribuído sobre os chips do grânulo é necessário.
- Pouco antes dos 30 min arrefecer foi concluída, abrir os pacotes de chips cordão de prata. Retire as fichas de suas mangas de plástico transparente. Sem tocar as contas, colocar todas as fichas pérola sobre uma pastilha de câmara hyb, orientando o chipCódigo de barras com o símbolo de código de barras gravado na superfície superior da inserção.
- Retire e descarte a tampa da placa de amostra. Tomar 15 ul de amostra a partir da placa de amostra e lenta dispensar na entrada da matriz (ver Figura 3). Cuidados devem ser tomados Extraordinária para colocar a amostra correta do chip talão correta na posição correta, que corresponde ao fim de chip talão gravado anteriormente. Uma pipeta de canais múltiplos pode ser utilizado para distribuir o líquido sobre as fichas, mas prudência deve ser feita para aspirar apenas o número de amostras que pode ser colocado de forma segura sobre o chip de grânulo, tal como o número de linhas em uma placa nem sempre coincide com o número de linhas em um chip.
- Uma vez que cada amostra foi colocada em sua matriz correspondente, inspecione visualmente o chip para bolhas ou regiões não revestidos no líquido. Se existem esses problemas, agite levemente o insert. Se necessário, mais líquido pode ser adicionado à matriz. Tome cuidado para adicionar a amostra de DNA correto.
- Abra a uma câmara hybd colocar as inserções ao longo dos reservatórios umidificação. O código de barras do chip deve ser colocado sobre o código de barras gravada na base da câmara de hyb. Recoloque a tampa da câmara de hyb e fechar todos os quatro fechos fechando os grampos em lados opostos na diagonal primeiro.
- Incubar a câmara hyb no forno por 16-24 horas. Tome cuidado para não inclinar as câmaras quando se deslocam-los.
- Se mais de uma placa está a ser processado, volte ao passo 6.2. Processando mais de 24 chips de talão em um dia não é recomendado, a fim de tanto minimizar a probabilidade de erro humano no manuseio de uma grande quantidade de materiais de consumo ou chips e para minimizar a quantidade de tempo necessário para processar as fichas em etapas futuras, como o cuidado deve ser tomadas para limitar a exposição ao ar, tanto quanto possível. Uma garrafa de XC4 é suficiente para até 24 chips de grânulo.
Terceiro dia
7. Coloração Preparação
- Retirar as câmaras hyb do forno e incubar a te quartomperatura para 25 min. Se o processamento de mais de duas câmaras hyb, escalonar sua dupla remoção a cada 10 min. A fim de manter as fichas de secagem, ainda não abrir as câmaras hyb.
- Enquanto as câmaras hyb estão esfriando, remover os tubos de XC1, XC2, TEM, STM, ATM e do "Post-1" -20 ° C e definir caixa no banco para descongelar. Pegue uma garrafa de RA1 do "Post-2" caixa a -20 ° C (ou 4 ° C, se a reutilização do reagente do dia anterior) e descongelar em banho-maria a temperatura ambiente. Descongele um tubo de 95% formamida também. Para 8 chips talão processados, dois tubos de cada reagente, 10 ml, 15 ml RA1 formamida, e 150 ml XC3 (encontrado em uma sala de temperatura, caixa fornecido pelo empresa) será necessária.
- Para cada chip de processamento, um slide de vidro, um plástico cinta flow-through, dois fechos de metal, e um espaçador de plástico será necessária. Para o espaçador de plástico, use somente a clara um; separar e descartar o espaçador opaco. Além disso, dois pratos e lavagem de chip talãouma bandeja de chip talão será necessário, juntamente com uma estação de montagem líquido e uma barra de montagem de plástico.
- Limpe as lâminas de vidro de pulverização com etanol e limpando-os secar.
- Ligue o banho de água quente, que é conectada ao rack câmara de fluxo e configurá-lo para 44 ° C. Agite suavemente o rack de câmara para desalojar as bolhas.
- Quando a câmara de hyb esfriou durante 25 min, encher um prato de lavagem com PB1 (aproximadamente 200 ml). PB1 pode ser encontrada na caixa de temperatura "Post-4" sala.
- Abra uma câmara hyb. Remova a vedação da tampa de um chip talão segurando o selo em um canto e delicadamente descascar na diagonal (ver Figura 4). Uma vez que o selo é removido, coloque imediatamente o chip em uma bandeja de chip talão e submergir no PB1, sem tocar as esferas. Repita até que o tabuleiro está cheio de fichas de contas, tendo o cuidado para não deixar qualquer chip seco.
- Agite suavemente a bandeja para remover quaisquer bolhas e deixar os chips submersos por 1 min.Encha um segundo prato de lavagem com PB1. Agitar a bandeja novamente.
- Mova o tabuleiro de chips de contas para o segundo prato de lavagem. Agite suavemente e deixe de molho por 1 min, em seguida, agitar novamente.
- Na estação de montagem líquido flow-through, coloque quatro cintas plásticas escoamento negros nas ranhuras. Encha com a estação de PB1, até o líquido quase atinge a altura de oito suportes de montagem (aproximadamente 150 ml).
- Remover um chip talão da bandeja e coloque-o na estação de montagem em uma cinta de escoamento plástico. O código de barras de chips devem estar alinhados acima do símbolo de código de barras gravado na estação de montagem. Repita o procedimento para os outros três chips que podem caber dentro da estação de montagem.
- Colocar um espaçador de plástico transparente no topo de um chip de grânulo. As bordas externas do espaçador deve rodear os suportes no posto de montagem. Repita o procedimento para os outros chips submersos no PB1.
- Coloque a barra de plástico de montagem na estação de montagem por encaixe ao longo dos sulcos. Suavemente colocar uma placa de vidro na parte superior do espaçador de plástico, empurrando a extremidade traseira da lâmina contra a barra de montagem e lentamente baixar a frente para dentro do líquido. A ranhura na parte superior do slide deve ficar voltada para baixo, diretamente acima do código de barras do chip, deixando uma lacuna entre o chip talão eo slide no final de código de barras. Repita o procedimento para os outros chips submersos no PB1. Para um diagrama completo de montagem de fluxo contínuo, ver Figura 5.
- Verifique se há bolhas entre o chip e slide. Se aparecerem bolhas, levante suavemente o slide no final de código de barras e tente novamente. Bolhas persistentes podem ser eliminados a partir do vidro com uma toalha de papel de laboratório (Kimwipe).
- Encaixe dois fechos de metal em torno da lâmina de vidro, uma no final de código de barras e um para trás. As bordas dos grampos devem prender a braçadeira de fluxo através de plástico sob o chip. Repita o procedimento para todos os chips submerso em PB1.
- Remova os conjuntos escoamento concluídos e colocar horizontalmente no bench. Tome cuidado para não derrubar a montagem vertical, permitindo que o líquido sob a lâmina de vidro para escapar. Se mais fichas estão esperando na bandeja de lavagem, que pode ser montado sob o mesmo PB1. Se outros chips estão esperando em suas câmaras hyb originais, descarte todo o líquido na estação de montagem e lavar pratos, e volte ao passo 7.6.
- Uma vez que todos os chips de contas estão em suas assembléias escoamento, levar um par de tesouras cirúrgicas e cortar ambas as extremidades do espaçador fora, tão perto da lâmina de vidro possível.
8. Coloração e Extensão
- Verificar que a prateleira da câmara de fluxo atingiu 44 ° C em várias posições, com uma sonda de temperatura. Se a temperatura estiver fora por mais do que meio grau, regular a temperatura da água do banho.
- Coloque um conjunto de fluxo de chip talão na prateleira câmara deslizando o conjunto para baixo e ligar a chave no topo. O lado de trás do chip talão deve tocar o bastidor câmara. O gslides moça deve estar voltado para fora, com a ranhura na parte superior para formar um reservatório de reagente. Repita o procedimento para cada montagem de chips talão.
- Usando uma pipeta de 200 mL, adicionar 150 ul RA1to os conjuntos de escoamento por distribuir o líquido para dentro do reservatório de vidro. Incubar por 30 seg. Repita 5x.
- Usando uma pipeta de 1000 mL, adicionar 450 ul XC1to os conjuntos de escoamento por distribuir o líquido para dentro do reservatório de vidro. Incubar durante 10 min.
- Adicionar 450 ul XC2 aos conjuntos de escoamento por distribuir o líquido para dentro do reservatório de vidro. Incubar durante 10 min.
- Adicionar 200 ul de ETM para os conjuntos de escoamento por distribuir o líquido para dentro do reservatório de vidro. Incubar durante 15 min.
- Adicionar 450 ul de 95% de mistura de formamida / EDTA para os conjuntos de escoamento por distribuir o líquido para dentro do reservatório de vidro. Incubar durante 1 min. Repetir 1x.
- Incubar as assembléias escoamento para 5 min.
- Defina o banho de água quente com a temperatura lIsted sobre os tubos de STM (ou 37 ° C, se não é mostrado). Certifique-se de cada tubo STM tem a mesma temperatura listados.
- Adicionar 450 mL XC3 às assembléias escoamento. Incubar durante 1 min. Repetir 1x.
- Quando a temperatura do banho de água quente tiver atingido a temperatura desejada, adicionar 250 μLSTM aos conjuntos de escoamento por distribuir o líquido para dentro do reservatório de vidro. Incubar durante 10 min.
- Adicionar 450 ul XC3 aos conjuntos de escoamento por distribuir o líquido para dentro do reservatório de vidro. Incubar durante 1 min. Repetir 1x.
- Incubar as assembléias escoamento para 5 min.
- Adicionar 250 ul de ATM para os conjuntos de escoamento por distribuir o líquido para dentro do reservatório de vidro. Incubar durante 10 min.
- Adicionar 450 ul XC3 aos conjuntos de escoamento por distribuir o líquido para dentro do reservatório de vidro. Incubar durante 1 min. Repetir 1x.
- Incubar as assembléias escoamento para 5 min.
- Adicionar 250 ul de STM para o fluxo atravésconjuntos por distribuir o líquido para dentro do reservatório de vidro. Incubar durante 10 min.
- ADD4 50 ul XC3 aos conjuntos de escoamento por distribuir o líquido para dentro do reservatório de vidro. Incubar durante 1 min. Repetir 1x.
- Incubar as assembléias escoamento para 5 min.
- Repita os passos 8,14-8,19 1x.
- Remova os conjuntos escoamento do rack câmara e desligue a água do banho.
9. Lavagem e Vedação
- Encha uma coloração pérola prato lavagem chip com PB1 (aproximadamente 315 ml). Serão necessários dois pratos de lavagem verticais e um tabuleiro de fichas cordão vertical, juntamente com pelo menos um colector de vácuo.
- Desmonte um conjunto de fluxo através da inserção de uma barra de metal fina entre os colchetes ea cinta e depois girando. Separe a lâmina de vidro e retire o chip talão. Imediatamente coloque o chip talão na bandeja a vertical chip de talão e submergir no PB1. Repita o procedimento para cada montagem flow-through, tomando cuidado para enfrentar each chip de talão na mesma direção e minimizando sua exposição ao ar.
- Agite suavemente a bandeja do chip talão para remover as bolhas. Incubar os chips talão submersas em PB1 por 5 min. Preencha o segundo vertical, lavagem de prato com o XC4 preparado no dia anterior. Agitar bandeja de chip de talão de novo.
- Mova a bandeja do chip talão para o prato cheio de lavagem XC4. Agite suavemente a bandeja para remover as bolhas. Incubar durante 5 min e agita-se de novo.
- Em um movimento suave, puxe a bandeja do chip talão do prato de lavagem e definir em um rack tubo de ensaio, de modo que as contas em cada rosto chip de talão para cima. Com uma pinça de travamento automático, deslize um chip talão para fora do tabuleiro e coloque em um rack tubo de ensaio. Repita o procedimento para cada chip de talão.
- Transferir cuidadosamente o rack tubo de ensaio de chips para um exsicador de vácuo. Feche e ligue a vácuo, garantindo uma vedação adequada. Incubar por 50-55 min. Se necessário, aquecer o scanner durante a incubação.
10. Exploração
- Turn o vácuo e, lentamente, regressar a pressão atmosférica. Verifique se os chips de contas estão secos. Se necessário, limpe as bordas e fundo do chip com uma toalha de papel para remover qualquer líquido ou detritos.
- Ative o software de digitalização e mover as fichas de contas para a bandeja de digitalização. Baixar arquivos de decodificação do chip (dmaps) ativando o software cliente de decodificação de arquivos e inserir os códigos de barras de chips talão desejados, juntamente com seus respectivos IDs de caixa. Fichas talão Não Verificadas pode seguramente ser armazenado em uma área escura seco por até 72 horas.
- Quando as fichas estão encaixadas corretamente na bandeja e decodificar os arquivos são reconhecidos pelo software, iniciar a digitalização.
Resultados
Um chip talão devidamente processado deve mostrar intensidades vermelhas e verdes brilhantes e distintas de luz a laser durante a digitalização. Na Figura 6, o software de digitalização iScan exibe uma amostra de ADN genómico padrão hibridizaram para um painel de costume matriz genotipagem. Os nucleotídeos marcados que foram anexados durante as etapas de extensão e de coloração fluorescente sob as luzes dos dois lasers. Como estes nucleótidos estender selectivamente cadeia de oligonucleótido do talão hibridado com a cadeia de DNA fragmentado, e como as cadeias de oligonucleótidos foram concebidos para terminar no local da variante, a cor e a intensidade do sinal resultante pode ser utilizado para determinar os alelos presentes no local SNP.
Uma folha gerado por usuários da amostra, que corresponde IDs de amostra e informações clínicas para chipID e posição, e um manifesto específico SNP-array, obtido diretamente da empresa, são necessárias a fim de importar o scaarquivos de saída nner para o software de análise de GenomeStudio. Folhas de amostra Exemplo pode ser encontrada no site da empresa. Uma vez que o projeto GenomeStudio é construído, os genótipos podem ser obtidos a partir dos grupos de intensidade resultantes gerados automaticamente pelo software. Apesar de existirem várias opções de exibição, o modo de exibição padrão, Norm-R (um valor de intensidade normalizada) vs Norm-Theta (um rácio de intensidade alélicas), é muitas vezes o enredo mais fácil de diferenciar três grupos distintos. A maioria dos SNPs deve mostrar uma, duas, ou três grupos, dependendo da freqüência do alelo menor.
Os três grupos representam amostras demonstrando AA, AB, ou alelos BB (ver Figura 7). Estes grupos podem ser atribuídos genótipos ou através da importação de um modelo de posição agrupamento padrão diretamente da empresa, selecionando "Cluster Todos os SNPs" na barra de ferramentas de análise do software, ou clicando e arrastando os círculos coloridos nas parcelas de intensidade para EDI manualmente t chama. A cor da amostra no gráfico de intensidade (vermelho, roxo ou azul) indica que a chamada (AA, AB ou BB, respectivamente); preto indica nenhuma chamada. Ao percorrer os SNPs listados no painel de dados completa do software, o agrupamento para cada SNP podem ser vistos ou recolhidos. Uma vez que os clusters são atribuídos chamadas satisfatórios, o painel de dados completa do software lista os genótipos para cada amostra individual em cada SNP particular. A opção Seletor de coluna acima da tabela pode alternar formatos de dados.
Os dados podem ser exportados através de barra de ferramentas de análise ou diretamente da tabela de dados completa para análise em profundidade.
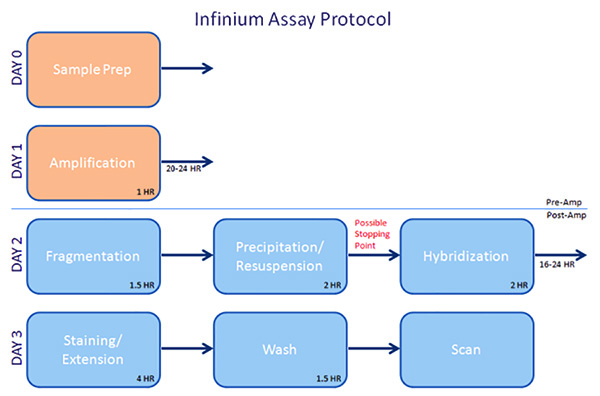
Figura 1. Visão geral - Infinium Ensaio protocolo.683/50683fig1highres.jpg "target =" _blank "> Clique aqui para ver imagem ampliada.

Figura 2. Completa Hyb Câmara de base e esteira, além de tampa. [12] Clique aqui para ver a imagem ampliada .
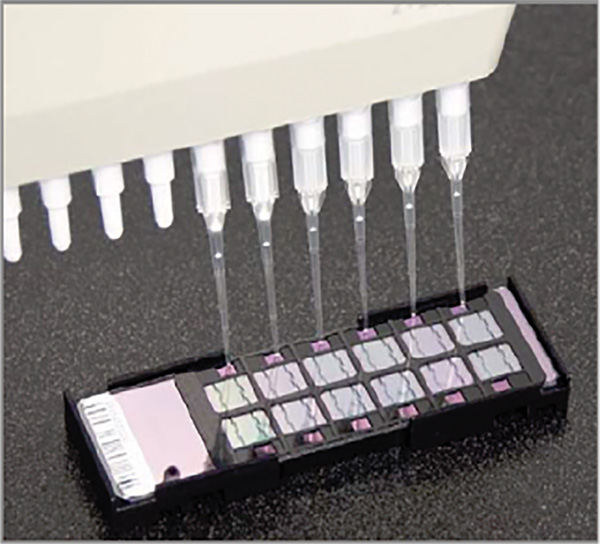
Figura 3 Carregando um BeadChip -.. Dispensar a amostra sobre as portas de entrada [12] Clique aqui para ver maior imagem.

Figura 4 Retirar o BeadChip Coverseal -.. Segure o selo no canto e delicadamente descascar diagonal [12] Clique aqui para ver a imagem ampliada .

Figura 5. Completa Assembléia BeadChip Flow-through - O BeadChip é separado de uma lâmina de vidro com um espaçador e preso com grampos.> Clique aqui para ver imagem ampliada.
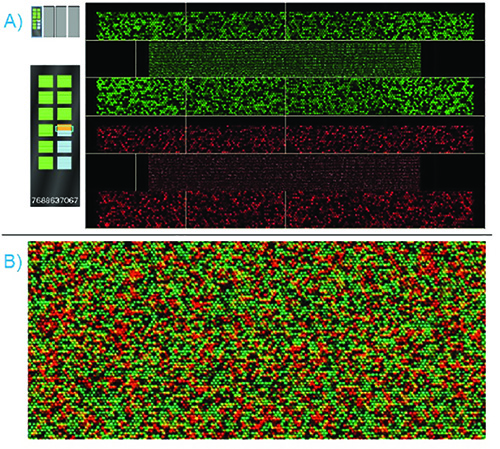
Figura 6. Successful BeadChip Scan - A) O BeadChip é digitalizada com ambos um laser vermelho e verde; o software de digitalização exibe ambos simultaneamente. Seções passam intensidade QC irá destacar verde no visor BeadChip para a esquerda. Seções falhando intensidade QC irá destacar vermelho no visor BeadChip. B) Quando a digitalização estiver concluída, o software irá sobrepor os displays vermelhos e verdes. Uma imagem zoom-in é mostrado. A cor ea intensidade de cada talão indivíduo indica o alelo presente. Clique aqui para ver a imagem ampliada .
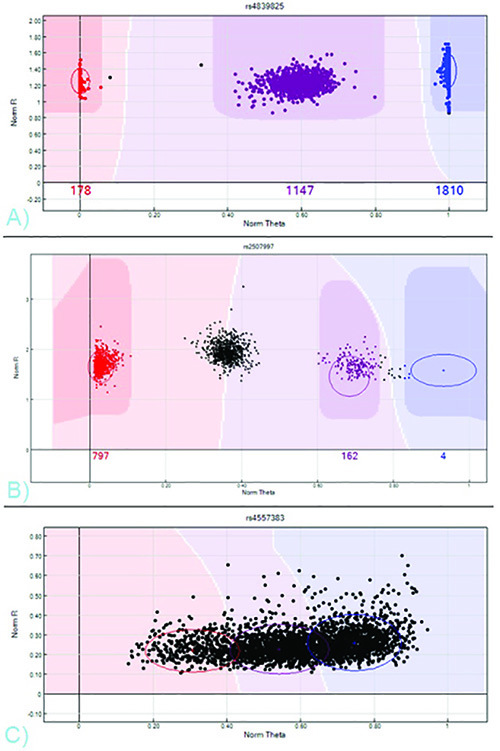
Figura 7. SNP NORM-R vs NORM-teta Perfis de Cluster - A) A SNP válido com três grupos distintos que representam AA, AB, e os genótipos BB, de cor vermelho, roxo e azul B) A edição exigindo SNP.. O cluster de meio, que deve ser homozigotos AB, é deixado sem chama. O cluster BB é erroneamente chamado de AB. C) A SNP pobre desempenho. Não genótipos podem ser obtidas a partir deste enredo intensidade, como não existem grupos distintos. Clique aqui para ver a imagem ampliada .
Discussão
Aplicações de genotipagem em larga escala têm sido usados para compreender melhor o mecanismo genético subjacente muitas doenças humanas. A descoberta de qualquer variante significativa através de uma análise de associação do genoma pode sinalizar uma região candidata para um estudo mais aprofundado. Além disso, os dados do genótipo é uma boa ferramenta para o controle de qualidade em projetos de seqüenciamento.
Para maximizar o rendimento da amostra, várias placas de amostra podem ser amplificados e armazenados em seus fragmentados, estados suspensão. Oito placas pode ser amplificado em um único dia, combinando a primeira 24 horas do protocolo para vários lotes e fornecer material suficiente para ~ 2-8 dias de processamento de chip. Se as placas são armazenadas previamente amplificados, e se novas amostras são cruzados para fichas imediatamente após a digitalização começa na corrida anterior, o processamento pode ser executado de forma contínua, sem a necessidade de fazer uma pausa para a preparação da amostra adicional. Portanto, apesar de amostras terá três dias para submeter-se ao completoensaio, os dados podem ser gerados por dia. Fichas Assuming24 são processados a cada dia, uma semana de trabalho de cinco dias permite a mais de um mil amostras de DNA para serem executados em um chip de talão de 12 amostras. Se um dos passos ou reagente falhou, no entanto, vários lotes pode estar em risco para o mau desempenho antes de qualquer correção pode ser aplicada. Os erros podem passar despercebidos até que as matrizes são digitalizados ou analisado, portanto, se o rendimento é maximizado, centenas de amostras em vários estágios do protocolo já poderia ter recebido o mesmo tratamento com defeito após o descobrimento. Como os reagentes e os dados perdidos não podem ser recuperados, o usuário deve pesar os riscos contra a necessidade de um fluxo de trabalho acelerado.
O software de análise de GenomeStudio é a primeira oportunidade de medir realmente o sucesso do processo de genotipagem. Se a Norma-R vs parcelas intensidade Norm-Teta estão devidamente agrupados, a taxa média de chamada (por cento do total de SNPs digitado com sucesso) das amostras deve se aproximar de 99%, embora este valor varia slightly dependendo do tipo de matriz. Os dados a partir de qualquer amostra com uma taxa de chamada menor que 85-90% não é confiável e deve ser descartada. Para fins de controle de qualidade, os resultados devem ser comparados a qualquer genótipos previamente conhecidos sempre que possível. Se esses dados não existe, intencional duplicação da amostra é uma ferramenta útil na verificação da placa ou matriz colocações. Estes pares duplicados devem ser colocados em chips separados, placas, lotes ou projectos; seus genótipos verificado após geração. Enquanto constrangimentos específicos QC variar de acordo com a preferência do investigador, as restrições comuns SNP são baseados em sucesso chamada de exemplo, o equilíbrio de Hardy-Weinberg, ou falta completa entre casos e controles, enquanto que as restrições de amostras comuns são baseados em taxas de chamada, inconsistências mendelianas, ou referências cruzadas do cromossomo X heterozygosity aos dados clínicos de gênero [13].
Se algum problema surge, o Dashboard Controles, encontrado na suíte de análise, podem ser submetidos àcompanhia, a fim de determinar a causa. Esses controles muitas vezes pode limitar o problema ao passo provável ou falha de reagentes. Se algum SNPs de interesse são encontrados através de um experimento genotipagem Infinium, suas parcelas de intensidade deve ser verificada em GenomeStudio por erros de agrupamento antes de mais pesquisas sejam efetuadas.
Um experimento de genotipagem Infinium não é provavelmente devido a um erro de processamento humano ou DNA entrada de má qualidade. Amostra quantificação deve ser exato e preciso. Para melhores resultados, os reagentes adicionados a qualquer amostra ou chip tem de ser vertido para o volume definido pelo protocolo. As pipetas devem ser devidamente calibrado. Os reagentes não devem ser executados após o vencimento e não devem ser novamente congelados, uma vez descongelado, para salvar o reagente RA1. A fim de minimizar possíveis erros de coloração e de extensão, a mistura de formamida / EDTA deve ser preparado todos os meses. Todos os -20 ° C reagentes devem ser armazenados em freezers apenas o manual-degelo. Todo material de laboratório usado na ruaaining, de extensão e de lavagem de partes do protocolo deve ser lavada cuidadosamente com água e detergente neutro imediatamente após desuso. Os reservatórios de umidificação na câmara hyb deve ser limpo com uma escova de tubo de ensaio e detergente neutro. As lâminas de vidro devem ser lavados com água sanitária a 10%, conforme as instruções dos seus manuais do usuário, uma vez por semana.
Divulgações
Os autores deste artigo não têm concorrentes interesses financeiros para divulgar.
Agradecimentos
O financiamento para este trabalho foi fornecido pelo NIH P20 GM103456, NIH RC2 AR058959, e NIH R56 AI063274
Materiais
| Name | Company | Catalog Number | Comments |
| Consumable or Equipment | Manufacturer | Part Number | Minimum Required for 96 Samples |
| 0.8 ml Deep Well Plate | Thermo Scientific | AB-0765 | 1 |
| Plate Mats | Thermo Scientific | AB-0674 | 2 |
| Reagent Basin | Fisher Scientific | 13-681-502 | 9 |
| Heat-seal Sheets | Thermo Scientific | AB-0559 | 1 |
| Flow-through Spacer | Fisher Scientific | NC9563984 | 6 |
| Pipette tips - 200 μl | Rainin | GP-L200F | 192 |
| Pipette tips - 10 μl | Rainin | GP-L10F | 16 |
| Pipette tips - 1,000 μl | Rainin | GP-L1000F | 16 |
| DNA Suspension Buffer | Teknova | T0220 | 0.5 ml |
| 0.1 N NaOH | Fisher Scientific | AC12419-0010 | 0.5 ml |
| Isopropanol (HPLC grade) | Fisher Scientific | A451 | 15 ml |
| Ethanol (200-proof) | Sigma-Aldrich | 459836 | 330 ml |
| Formamide (100%) | Thomas Scientific | C001K38 | 15 ml |
| EDTA (0.5 M) | Amresco | E177 | 0.2 ml |
| 10 μl 8-channel Pipette | Rainin | L8-10XLS | 1 |
| 200 μl 8-channel Pipette | Rainin | L8-200XLS | 2 |
| 1,000 μl Single-channel Pipette | Rainin | L-1000XLS | 1 |
| Microplate Shaker | VWR | 13500-890 | 1 |
| Refrigerated Microplate Centrifuge | VWR | BK369434 | 1 |
| Hybridization Oven | Illumina | SE-901-1001 | 1 |
| Hybex Microsample Incubator | SciGene | 1057-30-0 | 1 |
| Hybex MIDI Heat Block Insert | Illumina | BD-60-601 | 1 |
| Heat Sealer | Thermo Scientific | AB-0384 | 1 |
| Hyb Chamber w/ Insert and Mat | Illumina | BD-60-402 | 2 |
| Surgical Scissors | Fisher Scientific | 13-804-20 | 1 |
| Flow Through Assembly Parts | Illumina | WG-10-202 | 8 |
| Wash Rack and Dish | Illumina | BD-60-450 | 1 |
| Genepaint Chamber Rack | Tecan | 760-800 | 1 |
| Temperature Probe | Illumina | A1-99-109 | 1 |
| Staining Rack and Dish | Illumina | WG-10-207 | 1 |
| Self-Closing Tweezers | Ted Pella, Inc | 5374-NM | 1 |
| Vacuum Manifold | Ted Pella, Inc | 2240 | 1 |
| iScan or HiScan | Illumina | - | 1 |
Referências
- Shi, M. M. Enabling large-scale pharmacogenetic studies by high-throughput mutation detection and genotyping technologies. Clin. Chem. 47 (2), 164-172 (2001).
- Livak, K. J. SNP genotyping by the 5'-nuclease reaction. Methods Mol. Biol. 212, 129-148 (2003).
- Seeb, J. E., Pascal, C. E., Ramakrishnan, R., Seeb, L. W. SNP genotyping by the 5'-nuclease reaction: advances in high throughput genotyping with non-model organisms. Methods in Mol. Biol. 578, 277-292 (2009).
- Bayés, M., Gut, I. G. Overview of Genotyping. Molecular Analysis and Genome Discovery. ed, , 2nd, John Wiley & Sons, Ltd. Chichester, UK. 1-23 (2011).
- Lee, Y., Seifert, S. N., Fornadel, C. M., Norris, D. E., Lanzaro, G. C. Single-Nucleotide Polymorphisms for High-Throughput Genotyping of Anopheles arabiensis in East and Southern Africa. J. Med. Entomol. 49 (2), 307-315 (2012).
- Liu, Z. SNP Genotyping Platforms. Next generation sequencing and whole genome selection in aquaculture. , Wiley-Blackwell. 123-132 (2011).
- Shen, R., Fan, J. B., et al. High-throughput SNP genotyping on universal bead arrays. Mutat. Res./Fundam and Mol. Mech. of Mutagenesis. 573 (1), 70-82 (2005).
- Ragoussis, J. Genotyping technologies for all. Drug Discov. Today: Technol. 3 (2), 115-122 (2006).
- Wu, C. C., Shete, S., Jo, E. J., et al. Whole-Genome Detection of Disease-Associated Deletions or Excess Homozygosity in a Case-Control Study of Rheumatoid Arthritis. Hum. Mol. Genet. , (2012).
- Green, E. K., Hamshere, M., Forty, L., et al. Replication of bipolar disorder susceptibility alleles and identification of two novel genome-wide significant associations in a new bipolar disorder case-control sample. Mol. Psychiatry. , (2012).
- Shete, S., Hosking, F. J., Robertson, L. B., et al. Genome-wide association study identifies five susceptibility loci for glioma. Nat. Genet. 41 (8), 899-904 (2009).
- Inc Illumina. Infinium HD Ultra User Guide 11328087 RevB-1. , 15-101 (2009).
- Turner, S., Armstrong, L. orenL., Yuki Bradford,, et al. Quality Control Procedures for Genome Wide Association Studies. Curr Protoc Hum Genet. , 1-19 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados