需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
siRNA的筛选识别泛素和泛素样系统压力表生物学途径在培养的哺乳动物细胞
摘要
在这里,我们描述了一种方法来执行对象的siRNA“ubiquitome”屏幕来识别对缺氧的HIF1A介导的细胞应答的新型泛素和泛素样调节剂。这可以适用于任何生物学途径,其中一个强大的读出报告基因活性的是可用的。
摘要
与泛素和泛素样分子(UBLs)的蛋白质的翻译后修饰是一种新兴的一种调节多种生物学途径包括缺氧反应,proteostasis,DNA损伤反应和转录的动态细胞信号传导网络。为了更好地理解UBLs如何调控与人类疾病的途径,我们还编制了人类的siRNA“ubiquitome”库包括针对所有已知和预测的UBL系统路径组件1,186 siRNA双链池。该库可筛选针对一系列表达不同的生物途径的记者,以确定细胞系的UBL组件作为该途径中的问题的正或负调节。在这里,我们描述了利用这个库来识别ubiquitome,监管机构对缺氧的使用基于转录的荧光素酶报告的HIF1A介导的细胞应答的协议。初步试验发展阶段执行三个阶段进行屏幕之前建立的细胞系的合适的筛选参数:一级,二级和三级/反褶积审查。采用有针对性的在全基因组的siRNA库正变得越来越受欢迎,因为它仅提供了对通路成员与研究者最感兴趣申报的优势。尽管siRNA的筛选的固有的局限性,尤其是假阳性引起siRNA的脱靶效应,有问题的途径的真正新颖稳压器的识别胜过这些缺点,这可以通过执行一系列仔细进行的对照实验来克服。
引言
修改与泛素和泛素样分子(UBLs)蛋白质代表一个广阔的生化系统,调节多种生物学途径和应激反应。共价附着UBLs对它们的靶蛋白可以有不同的结果调节的稳定性,定位,功能,或在基板1的相互作用组。首先建立了泛素相关修饰UBL酶的步骤,现在作为修饰范式与大多数UBLs,包括相扑,NEDD8,ISG15和FAT10。对于发生变形例中,UBL二甘氨酸基序的羧酸酯基团通过一个E1活化酶首先被激活以形成被转移到一个E2结合酶的活性位点半胱氨酸的高能量硫醇。在E2然后用基板结合的E3连接酶相互作用介导的UBL转移到(通常情况下)的目标赖氨酸残基产生支链(异肽)联动2。连续多轮的修改可发生建立异肽链到衬底上,这对泛素可以发生通过其任何7赖氨酸,或通过其N-末端甲硫氨酸创建线性泛素链。这些修改形成离散拓扑结构具有不同的用途,如创造新的互动的图案和靶蛋白的降解之前,UBL去除专科蛋白酶。在泛素的情况下,有两种酶E1,E2 30-40结合酶,至少有600 E3连接酶和大约100去泛素化(配音)。而途径较少膨胀为其他10个左右UBLs,整体ubiquitome复杂能提供巨大的多样性在特定的UBL修饰的生物学结果。然而,虽然在UBL生物学的重大进展已经作出,大多数这些ubiquitome组件的精确细胞的作用尚不清楚。
使用短整型的erfering核糖核酸(siRNAs)的已成为在反向遗传学的有力工具,由于siRNA的特异性靶向细胞mRNA为破坏的能力,允许在不同的生物的上下文3被检个体的基因的作用。全基因组的屏幕已经被用于识别和验证的许多细胞过程的新的调节剂,并已创建接触到更广泛的科学界大量有用的数据。然而,在整个基因组中的屏幕已经被证明是非常有用的,有针对性的屏幕变得越来越受欢迎,因为它们更便宜,更快速,仅涉及对基因组的成员,其中,调查者是最感兴趣的更少的数据管理和报告。因此,为了更好地了解哪些细胞过程UBL的家庭成分都参与了,我们还编制了人类的siRNA库中针对所有已知和预测的ubiquitome的组成部分。这包括UBLs,E1活化酶,E2缀合酶,E3连接酶,含蛋白质和配音泛素结合结构域(UBD)。这个库可以用于对多种的不同的生物学问题报告细胞系的屏幕,从而使公正鉴定新的UBL组件监管这些途径。
以下协议说明如何执行严格的有针对性的siRNA ubiquitome屏幕识别HIF1A依赖性反应缺氧新型稳压器。在正常的氧张力,HIF1A受脯氨酰羟化,导致它的希佩尔林道(VHL)E3连接酶复合体4被认可和有针对性的退化。缺氧抑制脯氨酰羟化导致HIF1A的稳定和其随后的结合缺氧反应元件(的HRE)驱动的基因表达。在这里,我们描述了一个屏幕采用U20S骨肉瘤细胞的海兰三个串联拷贝的控制下稳定表达萤火虫荧光素酶poxia反应元件(U20S-HRE细胞)5。这个协议可以适于任何生物学途径是否健全读出的报告基因活性的是可以实现的,并且可以加上适当的阳性和阴性对照。
研究方案
1,实验开发阶段
注:启动siRNA筛选之前,化验发展阶段关键是要订出与报道细胞系筛选的重要参数。关键是要投入显著努力,在这个阶段,因为这将支持屏幕的未来的成功。
- 表征缺氧反应性U20S-HRE报告细胞系,生长2×75平方厘米的烧瓶U20S-HRE细胞中的Dulbeccos 80-90%汇合改良的Eagle培养基(DMEM),补充有10%FBS。
- 抽吸从细胞的一个烧瓶中的介质,用10ml PBS洗涤两次,并通过加入2ml的0.05%胰蛋白酶-EDTA溶液,并在37℃温育5分钟,分离细胞。将细胞再悬浮于8ml DMEM 10%FBS和移液管上下形成均匀的细胞悬浮液。
- 移取20微升悬浮液的细胞计数室,一式两份,并插入腔到自动化单元耦合NTER。单击单元格计数器软件“显示图片”,并确保细胞是关注的焦点。点击“伯爵”,并记下细胞密度。重复此操作的其他重复的,并计算平均细胞密度。稀释的细胞用DMEM 10%胎牛血清至60,000个细胞/ ml的浓度。
- 使用多道移液器,添加稀释细胞至三个相同的列( 例如 A5-H5,A6-H6和A7-H7)的5无菌白墙的96孔测定板,并转移到100微升(6000个细胞)加湿37℃培养箱中在5%CO 2过夜。注意:这些板块将被用来确定缺氧依赖性荧光素酶的输出为一系列缺氧暴露:0小时,2小时,6小时,10小时和24小时。
- 在翌日的结束,记录时间和在24小时低氧板添加到缺氧工作站设置为1%的氧。第二天早晨,算算时间10小时,6小时和2小时前的24小时盘是由于被删除神父OM缺氧工作站。在这些时间中,10小时,6小时和2小时板添加到缺氧工作站。
- 准备加入30ml 2×结合荧光素酶裂解/测定缓冲液(50mM的Tris磷酸盐pH值为7.8; 16毫摩尔MgCl 2; 2mM的DTT,2%的Triton-X-100,30%甘油,1毫摩尔ATP,1%BSA; 0.25毫荧光素和8微米的Na 4 P 2 O 7)。注:按所列顺序添加的组件,并允许BSA至少30分钟加入萤光素和Na 4 P 2 O 7的前溶解。
- 从缺氧工作站在适当的时候删除所有板块。添加100μl的2倍的荧光素酶裂解/测定缓冲液加入到分析板的相应孔中,并盖上透明薄膜。摇板振荡器上在板10分钟,以500rpm的转速,以彻底裂解细胞。
- 在板堆转移到一个自动96孔板光度计机架上。根据“协议”菜单,选择“绝对595”,并突出显示所有WELLS被下了“好选择”菜单读取屏幕上的板图。单击“运行”来衡量每个发光以及然后计算每块板的平均读数。计算缺氧依赖性折叠增加的报告基因活性的,通过将每缺氧板的平均读数由常氧(0小时缺氧)控制盘的平均读数。注意:它必须遵循一个强大的缺氧依赖性荧光素酶反应,以适合于筛选。阿五十倍增加,报告基因活性被认为是优秀的。
- 创建一个包含四个高控制(HIF1A干扰)重复(井A1-D1),四低控制(FIH1干扰)复制(井A12-D12)主控板,四缓冲区只控制重复(井E1-H1)和四非靶siRNA对照重复(E12-H12),其中所有的siRNA池是在200nM的浓度。注:高和低控制是基于已知的途径稳压器,增加和DECR成立缓解缺氧反应分别。另外,使用该库的一个子集研发分析可用于识别适当的控制。
- 使用多道移液器,转移10μL的每个控件的siRNA以无菌白色围墙的试验板。准备0.1微升转染试剂转染混合物在10微升降低血清培养基(1:100)每孔,并转移10微升转染混合物中各控制良好。吸管上下简单地混合,并留下平板休息20-60分钟,使转染复合物的形成。
- 制备75,000个细胞/ ml的细胞悬液与U20S-HRE细胞的第二个烧瓶。使用多道移液器,添加细胞悬浮液的80微升(6000个细胞)到每个转染混合物。板转移至饱和湿度37℃培养箱中培养,用5%的CO 2为24小时,然后转移到一个缺氧工作站,另有24小时,如步骤进行荧光素酶检测1.6-1.8。
- 计算使用公式Z = 1的高,低控制Z因子 - [3×(高控+低控制的标准偏差的标准偏差)/(平均高控制 - 低控制的平均值)。注意:0.5-1之间的一个值表明优良的测定,并应争取。
2,主屏幕
注意:一旦从分析开发阶段这些基本条件是在这里,主屏幕可以以一式三份使用以下协议来进行在96孔板格式。
- 增长7×75 厘米的烧瓶U20S-HRE细胞在DMEM中80%-90%汇合补充有10%体积/体积的胎牛血清(FCS)。
注:以下步骤2.2-2.13应进行第1天。 - 通过与步骤1.9和解冻了系列稀释的siRNA ubiquitome升说明制备含200微升每个控制的主控板启动的复制1IBRARY了至少30分钟。注意:每个孔含有4的siRNA组装在17×96孔板用列1和12的池留空为对照。
- 在层流罩,标签(或条形码)17无菌白色壁测定板用从数字1-17盖子,对应于所述的siRNA库系列的每个板。
- 离心主控板和所述siRNA ubiquitome库简要地(在2000×g离心1分钟),以确保所有的siRNA积累在井的底部。
- 使用自动化的液体分配器,以机器人“邮票”10微升来自主控制板上各控制到17测定板,使用相同的96 - 孔前端堆叠为每个板。
- 使用一个新的96孔头堆栈的每个17库板,转移10μl的每个ubiquitome的siRNA以使用自动化液体分配器,其相应的试验板。
- 准备40毫升转染试剂用于使用比ES 17测定板建立于1.10。注意:这包括附加20毫升以帐户为“死体积”,在小区分配器。 “死体积”指的是液体的体积在细胞分配器管网络内持续存在。
- 使用自动细胞分配器在17测定平板10微升转染试剂转移至每个孔中。简要地摇动测定板在振荡器上(在500rpm下1分钟),以允许siRNA和转染试剂的充分混合,然后离开仍然在室温下搅拌20-60分钟,使siRNA的:转染试剂复合物的形成。
- 用10ml PBS洗涤2 75 厘米烧瓶U20S-HRE细胞两次,并用2毫升胰蛋白酶-EDTA分离,在37℃5分钟。添加8毫升的DMEM 10%FBS到每个烧瓶和吸管上下几次来创建一个均匀的细胞悬液。从两烧瓶结合的细胞并转移到无菌的50毫升塑料管。
- 吹打计算细胞浓度从如在步骤1.3中所述的自动细胞计数器的两个读数20微升细胞一式两份至细胞计数室,并计算平均细胞浓度。
- 通过用DMEM 10%胎牛血清稀释至每毫升75,000细胞在无菌的塑料容器中的浓度制备155毫升细胞悬浮液。注意:这个量是足够的了17片,并包括额外的25毫升的细胞分配器中的死体积。
- 无菌磁力搅拌器加至细胞悬浮液中,并将其置于搅拌器设置在层流罩内,以限制细胞聚集。平衡自动饮水机细胞与细胞悬液,将其设置为免除80微升每孔细胞(6,000细胞)。
- 把每个17板,进而到自动细胞分配器和分配单元。记录中的5个组的时间和层叠板然后将堆叠在潮湿的37℃培养箱中在5%CO 2的24小时。注意:终浓度siRNA的池将是20纳米的100微升总体积。
注:以下步骤2.14-2.15,应进行第2天。 - 启动第2天的第二个重复,按照一天1所示的复制1(2.2-2.13)的程序。
- 转染后24小时,从在无菌环境中复制1传热板到缺氧工作站设置在1%氧气和离开放置24小时以诱导HRE记者。
注:以下步骤,2.16-2 .20,应进行第3天。 - 启动第三复制,继天1所列的复制1(2.2-2.13)的程序。
- 转染后24小时,从在无菌环境中复制2传热板到缺氧工作站设置在1%氧气和离开放置24小时以诱导HRE记者。
- 前两小时,复制1板是被从缺氧工作站除去,如在1.6中所述制得200毫升2倍的荧光素酶裂解/测定缓冲液中。注:本卷中cludes另外20毫升保证缓冲水库仍远覆盖。
- 删除重复1检测板从工作站缺氧后24小时低氧暴露和使用自动化液体分配器,加入100μl的2倍荧光素酶裂解/分析缓冲液到每个检测板,盖上透明薄膜。
- 摇动的小区摇床板10分钟,在500rpm下,以彻底裂解细胞。依次将每个板光度计酶标仪和记录发光如步骤1.8中所述。
注:以下步骤2.21-2.22,应进行第4天。 - 转染后24小时,从在无菌环境中复制3传热板到缺氧工作站设置在1%氧气和离开放置24小时以诱导HRE记者。
- 由用于重复1以下步骤,2.18-2 .20阅读复制2片。
注意:下面的步骤2.23,应进行第5天。 - 通过下面的内容复制3板步骤用于重复1 ,2.18-2 .20。
- 编译主屏幕的所有3个重复,并计算用1.12给出的公式每块板的Z参数。注意:如果任板具有小于0.5的Z参数,考虑重复该板以提高数据质量。
- 通过使用下面的公式计算方差(CV)的系数为高和低控制:控制控制/平均100×标准差。注:简历应该尽可能的低,这是合理的预期10-15%的变化。如果变化是显著较高,可考虑重复板。
- 。×100另外,计算出非目标(新台币[ - / - (意味着高控制低控制的平均值)(siRNA的得分意味着高控制)]:通过使用下面的公式计算出每盘各的siRNA%的激活)倍各的siRNA通过使用公式[NT的样本数据/平均值]。为百分之激活和NT倍,计算3 replic的平均阿泰和编译的值。
- 使用原始数据,计算出斯皮尔曼等级相关系数(SRCC),以确定如何紧密三个重复平板关联使用公式:
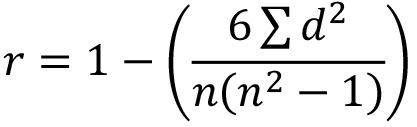
其中r = SRCC; D =每对队伍和对数据的n =数的两个数字之间的差异。注:重复板之间的良好关系将给接近1的值如果一个复制显示了对其他2片低SRCC,进一步调查,并考虑重复该板块。 - 决定哪些siRNA是足够的兴趣二次筛选(80)。采用用户自定义的切销( 如选择显示小于5%或超过95%%的激活,或小于0.5新台币倍或超过150 NT倍所有的siRNA)或应用生物信息学或基于知识的推理来搜索集群打属于同一UBL途径中。注意:通常,这些方法的组合决定了的siRNA都考虑二次筛选。
3,二次网
注意:一个确定次级屏幕进行基于一个最大的从主屏幕80的siRNAs的兴趣。这个数量可以方便地进行,以镀上一个单一的96孔板中每个重复使用全控制按主屏幕(参见2.2)。这是非常有用的,以确认在主屏幕中确定的监管机构可重复触发相同的表型,这将有助于完善上打应该通过到最终的专/反褶积屏幕进行分流的决定。
- 生长1×75 厘米烧瓶U20S-HRE细胞在DMEM中80-90%汇合时,用10%的FBS。
- 注意,塔板数和主画面的命中位置,并计划他们的次级屏幕樱桃采摘底版新的位置,离开柱1的D 12免费为对照。制备所述控制底版如1.9但与50微升的每个控制的总体积。
- 解冻第二系列稀释的ubiquitome库(预留樱桃采摘单个的siRNA池)最少30分钟。离心板短暂(2,000×g离心1分钟)然后加入50微升的二次筛选的siRNA,以他们的新板位置上的樱桃采摘主板。
- 使用概述了以下修改主屏相同的基本协议进行了二次筛选:(一)运行在同一天的所有三个重复,每重复一个实验板及(b)调整试剂的量相应。
4,大专/解卷积屏幕
注:大专或卷积筛选上最大的从二级屏20的siRNA进行。这个步骤是使用每个单独的siRNA从原来的大便检查击倒的效果升四。通常情况下,从每个池的至少两个单独的siRNA双链体应该非法相同的表型具有合理的置信度,所观察到的表型不是由于siRNA的脱靶效应。为了进一步增加在这个阶段信心,靶向基因在质询额外个体siRNA双链体可以被设计并测试它们引出的给定表型的能力。这些实验的结果可随后被用于计算在H得分,其中H = 0.6或以上( 即 ,其中至少有3星单独的siRNA引起的表型的)被认为是可以接受的6。
- 长1 75 厘米烧瓶U20S-HRE细胞在DMEM中80-90%汇合,用10%的FBS。
- 按规划每一击的四个独立的siRNA双链上的第三屏幕主盘位置,使第1列和12个免费的控件创建一个板图的第三屏幕。准备控制主控板为1.9,但总体积50微升每个控件的。
- 解冻解卷积/个人的siRNA板,并根据板图(4.2)加50微升的各个siRNA(即200纳米)的入第三主屏板的各孔中,以允许3×10微升重复和舒适的20微升过量。
- (一)运行在一个试验板在同一天的所有三个重复,每个重复及(b)调整试剂的量相应:使用具有以下修改概述了主要画面基本相同的协议进行三次筛选
注意:在理想情况下的阈值各个siRNA双链应设置在同一严紧切断用作池。然而,这可能是可接受的10-20%,以放松的阈值siRNA个体,特别是其中至少一个其它双工落在设定的阈值。这是值得铭记的siRNA的单独效应可能小于该中池,并且相反地,在某些情况下,siRNA个体可能显示在分离的细胞表型上的更强的效果比当它存在于池(即使当浓度为占)。
结果
在筛选之前,缺氧反应U20S-HRE细胞的建立。 U20S-HRE细胞表达报告基因构建由萤火虫荧光素酶融合的低氧反应元件的3个连续拷贝,其在暴露于缺氧( 图1A)约束的HIF1A/HIF1B异二聚体的下游。单元被放置在一个缺氧工作站的时间范围,以确定哪些低氧产生用于筛选的最有效的响应。 U20S细胞表达HIF2A水平低,因此,荧光素酶的读数在很大程度上依赖于缺氧介导的HIF1A稳定7。曝光U20S-H...
讨论
使用在哺乳动物细胞中的全基因组siRNA筛选已被证明是在确定不同的生物途径新颖监管极其宝贵的。这里,我们已经描述了使用靶向ubiquitome siRNA筛选,以识别对缺氧的HIF1A介导的细胞响应的调节剂。有针对性的屏幕也变得越来越有吸引力,因为他们一般都比较便宜,更快,更容易管理,只有对通路组件中,研究者有兴趣7,10,11的报告。
它把一个重大努力进入检验发?...
披露声明
作者什么都没有透露。
致谢
这项工作是由威康信托基金会,葛兰素史克(GSK)和苏格兰研究所细胞信号(即现在的MRC蛋白质磷酸化和泛素化单元的一部分)的支持。
材料
| Name | Company | Catalog Number | Comments |
| Automated Liquid Dispenser | Fluid-X | XPP-721 | http://www.fluidx.eu/BIOTRACK/xpp-721-liquid-handling-system.html |
| White Walled Assay Plate | Greiner Bio One | 655083 | http://www.greinerbioone.com/en/row/articles/catalogue/article/37_11/13221/ |
| Clear Plate Film | Perkin Elmer | 1450-461 | http://www.perkinelmer.co.uk/Catalog/Product/ID/1450-461 |
| siRNA library | Thermo Scientific | On-Target Plus | http://www.thermoscientificbio.com/rnai-and-custom-rna-synthesis/sirna/on-targetplus-sirna/search-gene/ |
| Transfection reagent | Invitrogen | Lipofectamine RNAimax | http://www.invitrogen.com/site/us/en/home/Products-and-Services/Applications/Protein-Expression-and-Analysis/Transfection-Selection/lipofectamine-rnaimx.html |
| Reduced Serum Medium | Invitrogen | Optimem | http://products.invitrogen.com/ivgn/product/31985062?ICID=search-product |
| DMEM | Invitrogen | 41965-039 | http://products.invitrogen.com/ivgn/product/41965039# |
| FBS | Invitrogen | 16000-044 | https://products.invitrogen.com/ivgn/product/16000044?ICID=search-product# |
| Tryspin-EDTA | Invitrogen | 25300-054 | https://products.invitrogen.com/ivgn/product/25300054?ICID=search-product# |
参考文献
- Hochstrasser, M. Origin and function of ubiquitin-like proteins. Nature. 458, 422-429 (2009).
- Schulman, B. A., Harper, J. W. Ubiquitin-like protein activation by E1 enzymes: the apex for downstream signalling pathways. Nat Rev Mol Cell Biol. 10, 319-331 (2009).
- Siomi, H., Siomi, M. C. On the road to reading the RNA-interference code. Nature. 457, 396-404 (2009).
- Kaelin, W. G., Ratcliffe, P. J. Oxygen sensing by metazoans: the central role of the HIF hydroxylase pathway. Mol Cell. 30, 393-402 (2008).
- Melvin, A., Mudie, S., Rocha, S. The chromatin remodeler ISWI regulates the cellular response to hypoxia: role of FIH. Mol Biol Cell. 22, 4171-4181 (2011).
- Bhinder, B., Djaballah, H. A simple method for analyzing actives in random RNAi screens: introducing the "H Score" for hit nomination & gene prioritization. Comb Chem High Throughput Screen. 15, 686-704 (2012).
- Bett, J. S., et al. The P-body component USP52/PAN2 is a novel regulator of HIF1A mRNA stability. Biochem J. 451, 185-194 (2013).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J Biomol Screen. 4, 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nat Methods. 6, 569-575 (2009).
- Stagg, H. R., et al. The TRC8 E3 ligase ubiquitinates MHC class I molecules before dislocation from the ER. J Cell Biol. 186, 685-692 (2009).
- Zhang, Y., et al. RNF146 is a poly(ADP-ribose)-directed E3 ligase that regulates axin degradation and Wnt signalling. Nat Cell Biol. 13, 623-629 (2011).
- Jackson, A. L., et al. Expression profiling reveals off-target gene regulation by RNAi. Nat Biotechnol. 21, 635-637 (2003).
- Birmingham, A., et al. 3' UTR seed matches, but not overall identity, are associated with RNAi off-targets. Nat Methods. 3, 199-204 (2006).
- . Whither RNAi. Nat Cell Biol. 5, 489-490 (2003).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。