JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
배양 된 포유 동물 세포의 생물 학적 경로의 유비퀴틴과 유비퀴틴 같은 시스템 레귤레이터를 확인하는 siRNA를 상영
요약
여기, 우리는 저산소증에 HIF1A - 매개 세포 반응의 새로운 유비퀴틴과 유비퀴틴 같은 규제를 식별 할 수있는 대상의 siRNA "ubiquitome"화면을 수행하는 방법을 설명합니다. 이 기자 활동 중 강력한 읽기가 사용할 수있는 모든 생물학적 경로에 적용 할 수 있습니다.
초록
유비퀴틴과 유비퀴틴 같은 분자 (UBLs)와 단백질의 번역 후 변형은 저산소증 응답, proteostasis, DNA 손상 반응 및 전사 등 다양한 생물 학적 경로를 조절하는 동적 인 세포 신호 전달 네트워크로 부상하고있다. 더 나은 UBLs이 인간의 질병에 관련된 경로를 조절하는 방법을 이해하기 위해, 우리는 인간의 siRNA를 컴파일 "ubiquitome"모든 알려진 UBL 시스템 경로의 구성 요소를 예측을 대상으로 1,186 siRNA의 이중 풀로 구성된 라이브러리입니다. 이 라이브러리는 UBL 성분은 당해 통로의 포지티브 또는 네거티브 레귤레이터 역할 결정하는 다양한 생물학적 경로의 기자 발현 세포주의 범위에 대해 스크리닝 할 수있다. 여기에서, 우리는 전사 기반의 루시 페라 제 기자를 사용하여 저산소증에 HIF1A - 매개 세포 반응의 ubiquitome - 레귤레이터를 식별하기 위해이 라이브러리를 사용하는 프로토콜에 대해 설명합니다. 초기 분석 개발 단계1 차, 2 차 및 3 차 / 컨볼 루션 스크리닝 : 세 단계로 화면을 수행하기 전에 세포주의 선별 적합한 매개 변수를 설정하기 위해 수행된다. 그것은 단지 조사가 가장 관심있는 통로의 구성원에 대한보고의 이점을 제공으로 전체 게놈의 siRNA 라이브러리를 통해 대상의 사용은 점점 더 인기를 끌고있다. siRNA의 심사의 본질적인 한계에도 불구하고, 특히 가양는 siRNA를 표적 이탈 효과로 인한 문제의 경로의 진정한 새로운 규제의 식별은주의 깊게 수행 제어 일련의 실험을 수행함으로써 극복 할 수있다 이러한 단점을보다 큽니다.
서문
유비퀴틴과 유비퀴틴 같은 분자 (UBLs)와 단백질의 변형은 다양한 생물 학적 경로와 스트레스 반응을 조절하는 광대 한 생화학 시스템을 나타냅니다. 타겟 단백질에 UBLs의 공유 결합은 안정성, 현지화, 기능 또는 기판 (1)의 interactome를 조절하는 다양한 결과를 가질 수 있습니다. UBL 수정의 기초가되는 효소 단계는 첫 번째 유비퀴틴 설립, 지금 SUMO, NEDD8, ISG15 및 FAT10를 포함한 대부분의 UBLs와 수정을위한 패러다임의 역할을 하였다. 수정이 발생하는 경우, UBL의 diglycine 모티프의 카르 복실 그룹은 제 E2 컨쥬 효소의 활성 사이트에 전송된다 시스테인 고 에너지 티올을 형성하기 위해 E1 활성화 효소에 의해 활성화된다. E2는 (보통) 측쇄 (isopeptide)을 생성 대상 라이신 잔기가 결합이 상 UBL의 이동을 중재하는 기판 - 결합 E3 리가 아제와 상호 작용. 수정의 연속 라운드 선형 유비퀴틴 체인을 만들 유비퀴틴을위한 일곱 라이신의를 통해 발생할 수있는 기판 상에 또는 N-말단 메티오닌을 통해 isopeptide 체인을 구축하기 위해 발생할 수 있습니다. 이러한 수정은 이전의 전문 단백질 분해 효소에 의해 UBL 제거에 새로운 상호 작용 모티브를 생성하고 분해를 위해 단백질을 대상으로 다양한 목적으로 별도의 토폴로지를 형성한다. 유비퀴틴의 경우 두 개의 E1 효소, 30 ~ 40 E2 활용시키는 효소, 적어도 600 E3 ligase라는 약 100 deubiquitylating 효소 (더빙)가있다. 경로가 다른 10 정도 UBLs 덜 팽창 동안, 전반적인 ubiquitome 복잡성은 특정 UBL 개질 생물학적 결과에 큰 다양성을 제공한다. UBL 생물학의 주요 발전하였으나 그러나, 이러한 ubiquitome 구성 요소의 대부분의 정확한 세포의 역할은 잘 알려져 있지 않은 상태이다.
짧은 INT의 사용erfering 리보 핵산 (siRNA를)은 개별 유전자의 역할은 다른 생물 학적 맥락 3에서 조사 할 수 있도록 구체적으로 인해 파괴 세포의 mRNA를 표적으로하는 siRNA를의 능력에 역 유전학의 강력한 도구로 떠오르고있다. 전체 게놈 화면은 많은 세포 과정의 새로운 규제를 확인하고 유효성을 검사하는 데 사용되었으며, 넓은 과학 사회에 접근 할 유용한 데이터의 재산을 만들었습니다. 전체 게놈 스크린은 매우 유용 입증하면서 그러나, 대상 화면들이 저렴 점점 인기를 끌고있다, 빨리 만 조사가 가장 관심있는 게놈의 구성원에 더 적은 데이터 관리 및보고를 포함한다. 따라서, 더 나은 세포 프로세스 UBL 가족 구성 요소에 관여하는 이해하기 위해, 우리는 모든 알려진 ubiquitome의 구성 요소를 예측을 대상으로하는 인간의 siRNA 라이브러리를 컴파일합니다. 이것은 UBLs, E1 활성화 효소, E2의 공액을 포함효소, E3의 리가 제, 유비퀴틴 결합 도메인 (UBD) 함유 단백질과 더빙. 이 라이브러리는 그러므로 이러한 경로를 지배하는 신규 UBL 성분의 공정한 식별을 허용 별개 생물학적 문제 리포터 세포주의 넓은 범위에 대하여 스크린 할 수있다.
다음 프로토콜은 엄격한 대상의 siRNA에게 저산소증에 HIF1A 의존 반응의 새로운 규제를 식별 할 수 ubiquitome 화면을 수행하는 방법에 대해 설명합니다. 정상 산소 분압에서 HIF1A는 폰 Hippel 린다 우 (VHL) E3 리가 단지 4 인식하고 분해 대상이됩니다 프 롤릴 수산화이 적용됩니다. 저산소증은 HIF1A의 안정화로 이어지는 프 롤릴 하이드 록을 억제하고 그 이후의 저산소증 반응 요소 (HRES)에 결합하는 유전자 발현을 구동 할 수 있습니다. 여기서, 우리는 안정적 하이록 세 탠덤 복사본 제어 하에서 반딧불 루시퍼 라제를 발현 U20S 골육종 세포를 사용하여 화면을 설명poxia 응답 요소 (HRE U20S-세포) 5. 리포터 활성의 강력한 판독이 달성 적절한 양성 및 음성 대조군으로 결합 할 수있는 경우이 프로토콜은 모든 생물학적 경로에 대해 적응 될 수있다.
프로토콜
1. 분석 개발 단계
참고 : 이전에 siRNA의 화면을 시작으로, 분석 개발 단계는 기자 셀 라인과 심사에 중요한 매개 변수를 설정하는 것이 중요합니다. 이것이 화면의 미래의 성공을 뒷받침하므로이 단계에서 상당한 노력을 투자하는 것이 필수적입니다.
- U20S-HRE 기자 세포주의 저산소증 응답의 특성을, 10 % FBS와 보충 이글 중간 (DMEM) 수정 Dulbeccos 80-90 % 년 합류에 2 × 75cm에게 U20S-HRE 세포의 2 플라스크를 성장.
- 기음 셀 중 하나 술병에서 미디어는 10 ㎖의 PBS로 두 번 씻어 2 ML 0.05 % 트립신 - EDTA (에틸렌 다이아 민 테트라 초산) 솔루션을 추가하고 5 분 동안 37 ° C에서 배양하여 세포를 분리. 8 ML의 DMEM 균일 한 세포 현탁액을 만들 수 상하 10 % FBS와 피펫을 Resuspend 세포.
- 피펫 (20) 중복의 셀 카운팅 챔버 정지 μL, 그리고 자동화 된 셀 커플에 실을 삽입NTER. 셀 카운터 소프트웨어의 "디스플레이 이미지"를 클릭하고 세포의 초점이되어 있는지 확인합니다. "횟수"를 클릭하고 셀 밀도를 기록해 둡니다. 다른 중복에 대해이 단계를 반복하고 평균 셀 밀도를 계산합니다. 60000 세포 / ml의 농도로 DMEM 10 % FBS를 가진 세포를 희석.
- 멀티 채널 피펫을 사용하여 같은 세 개의 열 (예 : A5-H5, A6-H6과 A7-H7) 5 멸균 흰색 벽으로 둘러싸인 96 - 웰 분석 플레이트 및 전송로에 희석 된 세포 100 μL (6000 셀)를 추가 밤새 5 % CO 2에서 가습 37 ° C의 배양기. 참고 : 1 시간, 2 시간, 6 시간, 10 시간 및 24 시간 :이 판은 저산소 노출의 범위에 대한 저산소증 의존 루시 페라 제의 출력을 결정하는 데 사용됩니다.
- 다음 하루의 끝에서, 시간을 기록하고 1 %의 산소에 저산소증 워크 스테이션 세트에 24 시간 저산소증 판을 추가합니다. 24 시간 플레이트 FR을 제거 할 예정이다하기 전에 다음과 같은 아침 시간 10 시간, 6 시간, 2 시간을 계산저산소증 워크 스테이션 옴. 이 시간에, 저산소증의 워크 스테이션에 10 시간, 6 시간, 2 시간 플레이트를 추가합니다.
- 16 mM의 MgCl2를, 2 mM의 DTT, 2 % 트리톤 - X-100, 30 % 글리세롤, 1 mM의 ATP, 1 % BSA, 0.25 mM의 2 배 결합 루시퍼 용해 / 분석 완충액 30 ㎖ (50 mM 트리스 인산염 pH가 7.8을 준비 루시페린 8 μM 나 4 P 2 O 7). 참고 : 나열된 순서대로 구성 요소를 추가하고 BSA 적어도 30 분은 루시페린과 나 4 P 2 O 7의 추가 전에 해산 할 수 있습니다.
- 적절한 시간에 저산소증 워크 스테이션에서 모든 플레이트를 제거합니다. 분석 플레이트의 적절한 우물에 배 루시퍼 용해 / 분석 완충액 100 μl를 추가하고 투명 필름 커버. 철저하게 세포를 용해하기 위해 500 rpm에서 10 분 동안 접시 통에 접시를 흔들어.
- 자동화 된 96 웰 플레이트 루미 랙에 플레이트 스택을 전송합니다. "프로토콜"메뉴에서 "복근 595"을 선택하고 모든 아를 강조LS는 "잘 선택"메뉴에서 화면 플레이트지도에서 읽을 수 있습니다. 그럼 각 판의 평균값을 계산 각각의 발광을 측정하기 위해 "실행"을 클릭합니다. 정상 산소 상태 (0 시간 저산소증) 제어 판의 평균값에 의해 각 저산소증 판의 평균 독서를 분할하여 기자 활동의 저산소증 의존 접이식 증가를 계산합니다. 참고 :이 검사에 적합하도록 강력한 저산소증 의존 루시 페라 제의 반응을 관찰하는 것이 필수적입니다. 기자 활동의 5-10 배의 증가가 우수한 것으로 간주됩니다.
- 4 단 제어 (HIF1A의 siRNA)은 복제 (웰스 A1-D1), (FIH1의 siRNA) (웰스 A12-D12) 복제 개의 낮은 컨트롤이 포함 된 마스터 제어 판을 만들어, 네 개의 버퍼는 반복 실험 (우물 E1-H1)를 제어 네 모든 siRNA를 풀이 200 ㎚의 농도에서 아르 비 표적 siRNA를 제어 복제물 (E12-H12). 참고 : 높고 낮은 컨트롤이 증가하고 DECR 알려진 경로의 규제에 따라 설립각각 저산소증 반응을 완화. 대안 적으로, 라이브러리의 서브 세트를 사용하여 분석 개발은 적절한 컨트롤을 식별하기 위해 사용될 수있다.
- 멀티 채널 피펫을 사용하여 멸균 흰색 벽으로 둘러싸인 분석 플레이트에 각 컨트롤의 siRNA의 10 μl를 전송합니다. 10 μL의 0.1 μL의 형질 전환 시약의 형질 믹스를 준비 감소 혈청 배지 (1:100)도 각 컨트롤에 잘 및 전송 형질 믹스 10 μL 당. 최대 피펫 믹스 다운 간략하게하고, 형질 복합체 형성을 할 수 있도록 20 ~ 60 분 동안 휴식을 판을 둡니다.
- U20S-HRE 셀들의 제 플라스크로 75000 세포 / ml의 세포 현탁액을 준비한다. 멀티 채널 피펫을 사용하여, 각각의 형질 전환 혼합물에 세포 현탁액 80 μL (6000 세포)를 추가한다. 24 시간 동안 5 % CO 2 가습 37 ° C 배양기에 플레이트를 전송 한 후 추가로 24 시간 동안 저산소 워크 스테이션에 전송하고 단계에 설명 된대로 루시 페라 제 분석을 수행 1.61.8.
- [3 × (높은 컨트롤 + 저 제어의 표준 편차) / (높은 제어의 의미 - - 낮은 컨트롤의 평균을)] = 1 식 Z를 사용하여 높고 낮은 컨트롤에 대한 Z-계수를 계산한다. 주 : 0.5 ~ 1 사이의 값은 훌륭한 분석을 나타내며위한 노력을해야한다.
2. 기본 화면
주 : 분석 개발 단계에서 이러한 기본 조건이 준비되면, 주 화면은 다음 프로토콜을 이용하여 96 웰 플레이트 포맷 중으로 수행 될 수있다.
- DMEM 80-90 % 년 합류로 7 × 75cm에게 U20S-HRE 세포의 2 플라스크를 성장 10 %의 V 보충 / V 소 태아 혈청 (FCS).
참고 : 다음은 2.2-2.13은 1 일에 실시해야합니다 단계를 반복합니다. - 단계 1.9 및 해동 중의 siRNA ubiquitome 리터의 희석 시리즈에 설명 된대로 각 컨트롤의 200 μl를 포함하는 마스터 제어 판을 준비하여 복제 1 시작30 분의 최소 ibrary. 참고 : 각이 잘 컨트롤 비어 열 1 12 17 X 96 - 웰 플레이트에 조립 4 siRNA가 풀을 포함하고 있습니다.
- 층류 후드에서, 라벨 (또는 바코드) 숫자 1-17에서 뚜껑 17 멸균 흰색 벽으로 둘러싸인 분석 플레이트를,의 siRNA 라이브러리 시리즈의 각 판에 해당.
- 마스터 컨트롤 플레이트를 원심 분리기와 siRNA의 모든 siRNA가이 우물의 바닥에 축적하기 위해 라이브러리를 짧게 (2,000 XG에 1 분) ubiquitome.
- 각 플레이트에 대해 동일한 96 웰 팁 스택을 사용하여 17 분석 플레이트에 자동화 된 액체 디스펜서 로봇을 "스탬프"마스터 제어 판에서 각 컨트롤의 10 μl를 사용합니다.
- 17 라이브러리 판의 각각의 신선한 96 웰 팁 스택, 전송 각 10 μl를 사용하여 자동화 된 액체 디스펜서를 사용하여 해당 분석 플레이트에의 siRNA를 ubiquitome.
- 비 ES를 사용하여 분석 플레이트 (17)에 대해 40 ㎖ 형질 감염 시약을 준비1.10에 수립 된. 참고 :이 셀 디스펜서에서 "죽은 양"을 설명하기 위해 추가 20 ㎖를 포함한다. "죽은 볼륨은"셀 디스펜서 튜브 네트워크 내에서 지속적으로 존재하는 액체의 양을 의미한다.
- 17 분석 플레이트의 각 웰에 형질 전환 시약의 10 μl를 전송하는 자동화 된 세포의 디스펜서를 사용합니다. 형질 전환 시약 복합체 형성 : 간단히 다음 siRNA를 할 수 있도록 20 ~ 60 분 동안 실온에서 여전히 남겨, siRNA의 형질 전환 및 시약을 완전히 혼합 할 수 있도록 진탕 (500 rpm에서 1 분)에 대한 분석 플레이트를 흔들.
- 10 ㎖ PBS로 두 번 두 75cm에게 U20S-HRE 세포의 2 플라스크를 세척하고 5 분 동안 37 ° C에서 2 ML 트립신-EDTA로 분리합니다. 균질 한 세포 현탁액을 만들 위아래로 각각의 플라스크에 피펫으로 8 ML의 DMEM을 여러 번 10 % FBS를 추가합니다. 두 플라스크에서 세포를 결합하고 멸균 50 ML 플라스틱 튜브에 전송할 수 있습니다.
- 피펫으로 세포 농도를 계산20 μL의 셀 카운팅 챔버 중복 된 셀 및 단계 130에 설명 된대로 자동 세포 계수기의 두 판독에서 보통 세포 농도를 산출한다.
- 멸균 플라스틱 용기에 밀리리터 당 75,000 세포의 농도로 DMEM 10 % FBS로 희석하여 세포 현탁액 155 ㎖를 준비. 참고 :이 볼륨은 17 플레이트에 충분하며, 세포 디스펜서 죽은 볼륨에 대한 추가 25 ML을 포함한다.
- 세포의 응집을 제한하는 층류 후드 내부 설정 교반기에 세포 현탁액과 장소에 멸균 자석 교반기를 추가합니다. 세포 현탁액과 자동화 된 세포 디스펜서 평형 잘 당 세포의 80 μL (6000 셀)를 분배하기 위해 설정합니다.
- 자동화 된 세포 디스펜서에, 차례 차례로, 17 판을 각각 배치하고 세포를 분배. 5 그룹의 시간과 스택 플레이트를 기록 한 후 24 시간 동안 5 % CO 2에서 습기가 37 ° C 배양기에서 스택을 배치합니다. 참고 : 최종 농도를siRNA를 풀은 100 ㎕의 총 부피의 20 nm의 것입니다.
참고 : 다음은 2.14-2.15는 2 일에 실시해야합니다 단계를 반복합니다. - 복제 1 (2.2-2.13)에 대해 1 일에 설명 된 절차에 따라, 2 일에 두 번째 복제를 시작합니다.
- 형질 전환 24 시간 후, 저산소증 워크 스테이션에 무균 환경에서 복제 1에서 전송 플레이트는 1 % 산소로 설정하고 HRE 기자를 유도하기 위해 24 시간 동안 둡니다.
참고 : 다음은 2.16-2.20는 3 일에 실시해야합니다 단계를 반복합니다. - 복제 1 (2.2-2.13)에 대해 1 일에 설명 된 절차에 따라, 세 번째 복제를 시작합니다.
- 형질 전환 24 시간 후, 저산소증 워크 스테이션에 무균 환경에서 복제 2에서 전송 플레이트는 1 % 산소로 설정하고 HRE 기자를 유도하기 위해 24 시간 동안 둡니다.
- 2 시간 전에, 1 판이 저산소증 워크 스테이션에서 제거하여야한다 복제 1.6에 설명 2X 루시페라아제 용해 / 분석 완충액 200 ㎖를 준비한다. 참고 :이 볼륨에버퍼 저장이 잘 덮여 유지하기 위해 추가로 20 ㎖를 cludes.
- , 24 시간 저산소증 노출과 자동화 된 액체 디스펜서를 사용한 후 저산소증 워크 스테이션에서 1 분석 플레이트를 복제 각 분석 플레이트에 2 배 루시퍼 용해 / 분석 완충액 100 μl를 추가하고 투명 필름 커버 제거합니다.
- 철저 용균 500 rpm으로 10 분 동안 세포 쉐이커에 플레이트를 흔들어. 단계 1.8에 설명 된대로 루미 플레이트 리더 기록 발광에 차례로 각각의 판을 전송합니다.
참고 : 다음은 2.21-2.22는 4 일에 실시해야합니다 단계를 반복합니다. - 형질 전환 24 시간 후, 저산소증 워크 스테이션에 무균 환경에서 복제 3 전송 플레이트는 1 % 산소로 설정하고 HRE 기자를 유도하기 위해 24 시간 동안 둡니다.
- 복제 1에 사용되는 다음 단계 2.18-2.20하여 복제 2 판을 읽어보십시오.
주 : 다음 단계 2.23는 5 일에 실시한다. - 다음으로 복제 3 판을 읽고복제 1에 사용 2.18-2.20 단계.
- 기본 화면의 모든 3 개의 반복을 컴파일하고 1.12의 식을 사용하여 각 판의 Z 계수를 계산합니다. 주 : 모든 플레이트는 0.5 미만의 Z 팩터를 가지고있는 경우, 데이터의 품질을 향상시키기 위해이 플레이트를 반복 참고.
- 다음과 같은 수식을 사용하여 높고 낮은 컨트롤에 대한 분산 (CV)의 계수를 계산 컨트롤의 컨트롤 / 평균 100 × 표준 편차. 주 : CV 가능한 한 낮아야, 그것은 15 % 변화를 예상하는 것이 합리적이다. 편차가 상당히 높은 경우에, 플레이트를 반복 참고.
- . × 100 또한, 비 표적을 계산 (NT [- - / (높은 제어의 의미 낮은 컨트롤의 평균) (siRNA의 점수 높은 제어의 의미)] : 다음과 같은 수식을 사용하여 판마다 각각의 siRNA %의 활성화를 계산 식 [NT의 샘플 데이터 / 평균]을 사용하여 각각의 siRNA) 배. %의 활성화 및 NT-방면 모두에 대해, 세 replic의 평균을 계산ATES와 값을 컴파일합니다.
- 원시 데이터를 사용하여, 세 개의 복제에 플레이트 식을 이용하여 상관 관계를 결정하는 방법에 밀접 스피어의 순위 상관 계수 (SRCC)를 계산한다 :
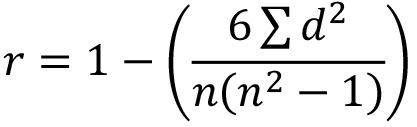
여기서, R = SRCC; D = 각 계급 쌍의 데이터 쌍의 N = 수의 두 숫자의 차이. 참고 :. 하나의 복제가 다른 2 판에 대해 낮은 SRCC가 표시되면 판은 복제 사이의 상관 관계는 1 가까운 값을 줄 것이다, 더 조사하고 그 판을 반복하는 것이 좋습니다. - (80까지) 2 차 심사에 충분한 관심이되는 siRNA를 결정합니다. (예를 들어, 5 % 미만의 이상 95 % %의 활성화, 이하 0.5 NT 배 또는 1.5 이상 NT 배를 보여주는 모든 siRNA를 선택) 사용자 정의 컷 오프를 사용하거나 클러스터를 검색하는 생물 정보학 또는 지식 기반 추론을 적용 같은 UBL 경로에서 떨어지는 안타. 참고 :일반적으로, 이러한 접근 방식의 조합의 siRNA가 차 심사에 촬영을 결정합니다.
3. 보조 화면
참고 : 확실한 보조 화면이 기본 화면에서 관심의 80 siRNA를 최대 기준으로 수행합니다. 이 숫자는 편리하게 기본 화면에 따라 전체 컨트롤과 하나의 96 웰 플레이트에 도금 각 복제 (2.2 참조)로 수행 할 수 있습니다. 그것은 기본 화면에서 식별 된 규제 재현성 같은 표현형을 유도하고,이 최종 차 / 디컨 볼 루션 화면을 통해 수행되어야한다 명중되는 환자 분류 결정을 정제에 도움이되는지 확인하는 것이 매우 유용하다.
- 10 % FBS와 DMEM 80-90 % 년 합류에 1 개의 x 75cm에게 U20S-HRE 세포의 2 플라스크를 성장.
- 판 번호와 기본 화면 히트 위치를 확인하고, 열 1을 떠나, 보조 스크린 체리 마스터 플레이트를 포착에 새 위치를 계획D 컨트롤에 대한 무료 12. 1.9에서와 같이하지만, 50 μL의 각 컨트롤의 전체 볼륨 제어 마스터 판을 준비합니다.
- 최소 30 분의 ubiquitome 라이브러리의 두 번째 희석 시리즈 (체리 따기 개인의 siRNA 풀에 따로) 해동. 판 짧게 (2,000 XG에 1 분)을 원심 분리 한 후 체리에 새로운 판 위치에 보조 화면 siRNA를 50 μl를 추가 마스터 플레이트를 들었다.
- 다음 수정을 기본 화면에 대한 설명 동일한 기본 프로토콜을 사용하여 보조 화면을 수행한다 : (a) 복제 당 하나의 분석 플레이트에서 같은 날에 세 개의 반복을 실행하고 (b)에 따라 시약의 볼륨을 조정합니다.
4. 차 / 디컨 볼 루션 화면
참고 : 차 또는 컨볼 루션 검사가 보조 화면에서 20 siRNA를 최대에서 수행됩니다. 이 단계는 일본어 똥에서 각각의 siRNA를 이용하여 넉다운의 효과를 조사하는 것이다네 L. 일반적으로, 각 풀에서 적어도 두 개의 개별 siRNA의 이중 가닥 관찰 된 표현형은 siRNA를 표적 이탈 효과로 인해이 아니라고 확신의 합리적인 학위를 가지고 동일한 표현형을 이끌어 낼 것이다. 또이 단계에서 신뢰성을 높이기 위해, 문제의 유전자를 표적으로 추가적인 개별의 siRNA 이중 가닥을 설계하고 관련 표현형을 유도하는 능력에 대해 시험 할 수있다. 이 실험의 결과는 H는 = 0.6 이상 (즉, 5 개인 siRNA를 중 적어도 3 표현형을 유도 곳이) 6 허용 간주되는 H 점수를 계산하는 데 사용할 수 있습니다.
- 10 % FBS와 DMEM 80-90 % 년 합류 한 75cm에게 U20S-HRE 세포의 2 플라스크를 성장.
- 컨트롤에 대한 무료 열 1과 12를 떠나, 차 화면 마스터 접시에 명중 된 각각의 네 개의 개별 siRNA의 이중 가닥의 위치를 계획하여 차 화면 접시지도를 만듭니다. 1.9으로 만 총 컨트롤 마스터 플레이트를 준비합니다50 μL의 각 컨트롤의 볼륨.
- 디컨 볼 루션 / 개인 siRNA의 판을 해동 세 × 10 μL의 복제하고 편안한 20 ㎕의 초과를 허용하는 판지도 (4.2)에 따라 차 화면 마스터 플레이트의 각 웰에 각각의 siRNA를 (200 NM)의 50 μl를 추가 .
- () 복제 당 하나의 분석 플레이트에서 같은 날에 세 개의 반복을 실행하고 (b)에 따라 시약의 볼륨을 조정 : 다음 수정을 기본 화면에 대한 설명 동일한 기본 프로토콜을 사용하여 차 화면을 수행
참고 : 이상적으로 개인의 siRNA 이중 가닥에 대한 임계 값이 같은 엄격한 설정해야 차단 풀로. 그러나, 적어도 하나의 다른 양면은 설정된 임계 값 내에 특히 여기서 개별적인 siRNA가 10-20 %의 임계 값을 완화하도록 허용 될 수있다. 그것은 siRNA를의 개별 효과는보다 적은 수 있다는 것을 염두에두고 가치 베어링입니다반대로 수영장, 일부의 경우 개별 된 siRNA는 (농도를 차지하더라도) 풀에 존재하는 경우에 비해 분리의 세포 표현형에 강한 효과를 표시 할 수 있습니다.
결과
이전 검사에, U20S-HRE 세포의 저산소증 응답이 설정됩니다. U20S-HRE 세포는 하류 저산소증에 노출 (그림 1A)에 HIF1A/HIF1B의 이종 구속 된 저산소증 응답 요소의 세 가지 직렬 사본의 융합 반딧불 루시 페라로 구성된 기자의 구조를 표현한다. 세포를 저산소 노출 스크리닝을위한 가장 효율적인 반응을 생성하는 확립 배의 범위 저산소증 워크 스테이션에 배치된다. U20S 세포는 따라서 루시 페...
토론
포유 동물 세포의 게놈 전체의 siRNA 스크린의 사용은 별개의 생물 학적 경로의 새로운 규제를 식별하는 매우 가치있는 것으로 입증되었습니다. 여기서, 우리는 저산소증 HIF1A - 매개 세포 반응의 레귤레이터를 식별하기 위해 타겟 ubiquitome의 siRNA를 화면의 사용을 설명했다. 그들은 일반적으로 저렴 빠르고, 쉽게 관리 할 수 만 수사관 7, 10, 11, 관심있는 통로의 구성 요소에 대한 보고서입니?...
공개
저자가 공개하는 게 없다.
감사의 말
이 작품은 웰컴 트러스트 (Wellcome Trust), 글락소 스미스 클라인 (GSK)과 세포 신호 전달을위한 스코틀랜드 연구소 (MRC 단백질 인산화 및 Ubiquitylation 단위의 지금 부분)에 의해 지원되었다.
자료
| Name | Company | Catalog Number | Comments |
| Automated Liquid Dispenser | Fluid-X | XPP-721 | http://www.fluidx.eu/BIOTRACK/xpp-721-liquid-handling-system.html |
| White Walled Assay Plate | Greiner Bio One | 655083 | http://www.greinerbioone.com/en/row/articles/catalogue/article/37_11/13221/ |
| Clear Plate Film | Perkin Elmer | 1450-461 | http://www.perkinelmer.co.uk/Catalog/Product/ID/1450-461 |
| siRNA library | Thermo Scientific | On-Target Plus | http://www.thermoscientificbio.com/rnai-and-custom-rna-synthesis/sirna/on-targetplus-sirna/search-gene/ |
| Transfection reagent | Invitrogen | Lipofectamine RNAimax | http://www.invitrogen.com/site/us/en/home/Products-and-Services/Applications/Protein-Expression-and-Analysis/Transfection-Selection/lipofectamine-rnaimx.html |
| Reduced Serum Medium | Invitrogen | Optimem | http://products.invitrogen.com/ivgn/product/31985062?ICID=search-product |
| DMEM | Invitrogen | 41965-039 | http://products.invitrogen.com/ivgn/product/41965039# |
| FBS | Invitrogen | 16000-044 | https://products.invitrogen.com/ivgn/product/16000044?ICID=search-product# |
| Tryspin-EDTA | Invitrogen | 25300-054 | https://products.invitrogen.com/ivgn/product/25300054?ICID=search-product# |
참고문헌
- Hochstrasser, M. Origin and function of ubiquitin-like proteins. Nature. 458, 422-429 (2009).
- Schulman, B. A., Harper, J. W. Ubiquitin-like protein activation by E1 enzymes: the apex for downstream signalling pathways. Nat Rev Mol Cell Biol. 10, 319-331 (2009).
- Siomi, H., Siomi, M. C. On the road to reading the RNA-interference code. Nature. 457, 396-404 (2009).
- Kaelin, W. G., Ratcliffe, P. J. Oxygen sensing by metazoans: the central role of the HIF hydroxylase pathway. Mol Cell. 30, 393-402 (2008).
- Melvin, A., Mudie, S., Rocha, S. The chromatin remodeler ISWI regulates the cellular response to hypoxia: role of FIH. Mol Biol Cell. 22, 4171-4181 (2011).
- Bhinder, B., Djaballah, H. A simple method for analyzing actives in random RNAi screens: introducing the "H Score" for hit nomination & gene prioritization. Comb Chem High Throughput Screen. 15, 686-704 (2012).
- Bett, J. S., et al. The P-body component USP52/PAN2 is a novel regulator of HIF1A mRNA stability. Biochem J. 451, 185-194 (2013).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J Biomol Screen. 4, 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nat Methods. 6, 569-575 (2009).
- Stagg, H. R., et al. The TRC8 E3 ligase ubiquitinates MHC class I molecules before dislocation from the ER. J Cell Biol. 186, 685-692 (2009).
- Zhang, Y., et al. RNF146 is a poly(ADP-ribose)-directed E3 ligase that regulates axin degradation and Wnt signalling. Nat Cell Biol. 13, 623-629 (2011).
- Jackson, A. L., et al. Expression profiling reveals off-target gene regulation by RNAi. Nat Biotechnol. 21, 635-637 (2003).
- Birmingham, A., et al. 3' UTR seed matches, but not overall identity, are associated with RNAi off-targets. Nat Methods. 3, 199-204 (2006).
- . Whither RNAi. Nat Cell Biol. 5, 489-490 (2003).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유