Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
siRNA Screening zu Ubiquitin und Ubiquitin-ähnlichen System Regler der biologischen Stoffwechselwege in kultivierten Säugerzellen identifizieren
In diesem Artikel
Zusammenfassung
Hier beschreiben wir eine Methode, um eine gezielte siRNA "ubiquitome"-Bildschirm, um neue Ubiquitin und Ubiquitin-ähnliche Regulatoren des HIF1A-vermittelte zelluläre Antwort auf Hypoxie zu identifizieren durchzuführen. Dies kann zu jedem biologischen Weg, wo ein robuster Auslesen der Reporteraktivität ist angepasst werden.
Zusammenfassung
Posttranslationale Modifikation von Proteinen mit Ubiquitin und Ubiquitin-artige Moleküle (UBLs) als dynamische Zellsignalisierungsnetz, die verschiedene biologische Pfade einschließlich der Hypoxie-Antwort, Proteostase, die die DNA-Reparatur und Transkription reguliert Schwellen. Um besser zu verstehen, wie UBLs regeln für die menschliche Krankheitsbahnen, haben wir eine menschliche siRNA zusammengestellt "ubiquitome" Bibliothek, bestehend aus 1.186 siRNA-Duplex-Pools gezielt alle bekannten und vorhergesagten Komponenten der UBL-System Wege. Diese Bibliothek kann gegen eine Reihe von Zelllinien, die Reporter von verschiedenen biologischen Wege, um zu bestimmen, die UBL Komponenten wirken als positive oder negative Regulatoren des Weges in Frage zu sehen sein. Hier beschreiben wir ein Protokoll unter Verwendung dieser Bibliothek in ubiquitome-Regulatoren des HIF1A-vermittelte zelluläre Antwort auf Hypoxie mit einem Transkriptions-basierte Luciferase-Reporter identifizieren. Eine erste Testentwicklungsstadiumdurchgeführt, um geeignete Screening-Parameter der Zelllinie, bevor Sie den Bildschirm in drei Stufen festgelegt: Primär-, Sekundär-und Tertiär / Entfaltung Screening. Der Einsatz von über gesamte Genom siRNA-Bibliotheken gezielt wird immer beliebter, denn es bietet den Vorteil der Berichterstattung nur auf die Mitglieder der Weg, mit dem die Ermittler am meisten interessiert sind. Trotz inhärenten Grenzen der siRNA-Screening, insbesondere Falsch-Positiven von siRNA Off-Target-Effekte verursacht, die Identifizierung neuer Regulatoren echt der Wege in Frage, überwiegen diese Mängel, die durch die Durchführung einer Reihe von sorgfältig durchgeführten Kontrollexperimente überwunden werden können.
Einleitung
Modifikation von Proteinen mit Ubiquitin und Ubiquitin-ähnlichen Molekülen (UBLs) stellt eine expansive biochemischen Systems, die verschiedene biologische Pfade und Stress-Reaktionen reguliert. Die kovalente Bindung von UBLs ihre Zielproteine können verschiedene Ergebnisse Regulierung der Stabilität, Lokalisierung, Funktion oder interactome des Substrats 1 aufweisen. Die enzymatischen Schritte UBL Änderung zugrunde liegenden wurden zunächst für Ubiquitin gegründet, und jetzt als Paradigma für die Modifikation mit den meisten UBLs, einschließlich SUMO, NEDD8, ISG15 und FAT10 zu dienen. Für die Modifikation auftritt, wird die Carboxylatgruppe des UBL Diglycin Motiv zunächst durch einen aktivierenden Enzym E1 aktiviert, um einen Hochenergie-thiol, die dem Cystein des aktiven Zentrums eines konjugierenden Enzym E2 übertragen wird, zu bilden. Die E2 interagiert dann mit einem substratgebundenen E3-Ligase, um die Übertragung des UBL auf (in der Regel) eine Ziellysinrest Schaffung eines verzweigtkettigen (Isopeptid) Zugstange 2 zu vermitteln. Aufeinanderfolgende Runden der Modifikation kann auftreten Isopeptid Ketten auf dem Substrat, die für Ubiquitin kann durch jedes der sieben Lysine auftreten oder durch ihre N-terminale Methionin zu bauen, um lineare Ubiquitin-Ketten zu erzeugen. Diese Änderungen bilden diskrete Topologien mit verschiedensten Zwecke wie die Erstellung von neuen Interaktionsmotiven und Targeting-Proteine für den Abbau vor UBL Entfernung durch Fach Proteasen. Im Fall von Ubiquitin zwei E1 Enzyme, 30-40 E2 konjugierenden Enzyme, mindestens 600 E3-Ligasen und etwa 100 deubiquitylating Enzyme (DUBs). Während die Wege sind für die anderen 10 oder so UBLs weniger expansiv, bietet die Gesamt ubiquitome Komplexität große Vielfalt in der biologischen Ergebnis einer bestimmten UBL Änderung. , Während die großen Fortschritte in der UBL Biologie gemacht worden sind, die genauen zellulären Rollen von der Mehrheit dieser ubiquitome Komponenten bleiben jedoch unbekannt.
Die Verwendung von short interfering Ribonukleinsäure (siRNA) als ein mächtiges Werkzeug in der reversen Genetik aufgrund der Fähigkeit von siRNAs, um gezielt zellulären mRNAs für die Zerstörung entstanden, so dass die Rolle einzelner Gene in verschiedenen biologischen Zusammenhängen 3 untersucht werden. Gesamtgenom-Bildschirme wurden zur Identifizierung und Validierung neuer Regulatoren vieler zellulärer Prozesse und haben eine Fülle von nützlichen Daten zugänglich zu weiteren wissenschaftlichen Community. Doch während des gesamten Genoms Bildschirme äußerst nützlich erwiesen, sind gezielte Bildschirme immer beliebter, da sie billiger sind, schneller, beinhalten weniger Datenmanagement und Bericht nur auf Mitglieder des Genoms, in dem die Ermittler am meisten interessiert. Deshalb, um besser zu verstehen, welche zellulären Prozesse UBL Familien Komponenten beteiligt sind, haben wir eine menschliche siRNA-Bibliothek gezielt alle bekannten und vorhergesagten Komponenten des ubiquitome zusammengestellt. Dies beinhaltet die UBLs, E1 aktivierende Enzyme, E2 konjugierendeEnzyme, E3-Ligasen, Ubiquitin-bindende Domäne (UBD) enthaltende Proteine und DUBs. Diese Bibliothek kann auf Bildschirm gegen ein breites Spektrum von Reporter-Zelllinien von verschiedenen biologischen Fragestellungen verwendet werden, so dass die unvoreingenommene Identifizierung neuer Komponenten UBL über diese Wege.
Das folgende Protokoll beschreibt, wie eine strenge gezielte siRNA ubiquitome Bildschirm, um neue Regulatoren der HIF1A abhängige Reaktion auf Hypoxie zu identifizieren durchzuführen. Unter normalen Sauerstoffspannung ist HIF1A unterliegen Prolylhydroxylierung, die es von der Von Hippel Lindau (VHL)-E3-Ligase-Komplex 4 erkannt und für den Abbau gezielt verursacht. Hypoxie hemmt Prolylhydroxylierung, die zur Stabilisierung der HIF1A und die anschließende Bindung an Hypoxie-Response-Elemente (HRE), um die Genexpression zu fahren. Hier mit U20S Osteosarkom-Zellen unter der Kontrolle von drei Tandemkopien der Hy stabil exprimieren Luciferase beschreiben wir eine BildschirmHypoxie-Response-Faktor (U20S-HRE-Zellen) 5. Dieses Protokoll kann für jeden biologischen Weg angepasst werden, wenn ein robustes Auslesen der Reporteraktivität erreichbar ist und mit geeigneten positiven und negativen Kontrollen gekoppelt werden kann.
Protokoll
1. Assay Development Stage
Hinweis: vor dem Beginn des siRNA-Bildschirm, ist ein Assay Entwicklungsphase entscheidend wichtige Parameter für das Screening mit dem Reporter-Zelllinie festzulegen. Es ist wichtig, erhebliche Anstrengungen in dieser Phase zu investieren, da dies den zukünftigen Erfolg des Bildschirms zu untermauern.
- Um die Reaktionsfähigkeit der Hypoxie-U20S-HRE-Reporterzelllinie zu charakterisieren, wachsen 2 x 75 cm 2-Kolben von U20S-HRE-Zellen zu 80-90% Konfluenz in Dulbecco modifiziertes Eagle-Medium (DMEM) mit 10% FBS.
- Saugen Sie die Medien aus einem Kolben von Zellen, zweimal waschen mit 10 ml PBS und lösen die Zellen durch Zugabe von 2 ml 0,05% Trypsin-EDTA-Lösung und Inkubation bei 37 ° C für 5 min. Die Zellen in 8 ml DMEM 10% FBS und Pipette auf und ab, um eine homogene Zellsuspension erstellen.
- Je 20 ul der Suspension auf eine Zellzählung Kammer, in zweifacher Ausfertigung, und die Kammer einfügen in eine automatisierte Zelle counter. Klicken Sie auf "Anzeigebild" auf der Zellzähler Software und sicherzustellen, dass die Zellen im Fokus. Klicken Sie auf "Count" und notieren Sie sich die Zelldichte. Wiederholen Sie diesen Vorgang für die andere doppelte und berechnen die durchschnittliche Zelldichte. Verdünne die Zellen mit DMEM 10% FBS in einer Konzentration von 60.000 Zellen / ml.
- Mit einer Mehrkanal-Pipette 100 ul (6.000 Zellen) der verdünnten Zellen auf die gleichen drei Spalten (z. B. A5-H5, H6 A6-und A7-H7) von 5 sterilen weißen Mauern 96-Well-Assay-Platten, und Transfer zum einer befeuchteten 37 ° C Inkubator bei 5% CO 2 über Nacht. Hinweis: diese Platten verwendet werden, um die Hypoxie-abhängigen Luciferase-Ausgang für eine Reihe von Hypoxie Engagements bestimmen: 0 h, 2 h, 6 h, 10 h und 24 h.
- Am Ende des folgenden Tages, notieren Sie die Zeit und fügen Sie die 24-Stunden-Hypoxie Platte auf die Hypoxie-Workstation auf 1% Sauerstoff. Am nächsten Morgen, die Berechnung der Zeit 10 h, 6 h und 2 h vor der 24-Stunden-Platte aufgrund fr entfernt werdenom die Hypoxie-Workstation. Zu diesen Zeiten, fügen Sie die 10 h, 6 h und 2 h Platten an der Hypoxie-Workstation.
- Vorbereitung 30 ml 2x kombiniert Luciferase Lyse / Assay-Puffer (50 mM Tris-Phosphat pH 7,8, 16 mM MgCl 2, 2 mM DTT, 2% Triton-X-100, 30% Glycerin, 1 mM ATP, 1% BSA, 0,25 mM Luciferin und 8 &mgr; M Na 4 P 2 O 7). Anmerkung: Die Komponenten in der aufgeführten Reihenfolge und damit die BSA mindestens 30 min vor der Zugabe von Luciferin und Na 4 P 2 O 7 zu lösen.
- Entfernen Sie alle Platten von der Hypoxie-Workstation zu gegebener Zeit. 100 l 2x Luciferase-Lyse / Assay-Puffer in die entsprechenden Vertiefungen der Testplatte und Abdeckung mit Klarsichtfolie. Schütteln Sie die Platten auf einem Plattenschüttler für 10 min bei 500 Umdrehungen pro Minute, um gründlich zu lysieren die Zellen.
- Übertragen Sie die Plattenstapel zu einem automatisierten 96-Well-Platte Luminometer Rack. Unter dem Menü "Protokolle", wählen Sie "abs 595", und markieren Sie alle wells in der On-Screen-Platte Karte unter dem Menü "Auswahl gut" gelesen werden. Klicken Sie auf "Run", um Lumineszenz von jedem messen und berechnen dann die durchschnittliche Lese jeder Platte. Berechnen Sie die Hypoxie-abhängigen facher Anstieg der Reporteraktivität durch Division der Durchschnittswert der einzelnen Hypoxie Platte durch die durchschnittliche Lese der Normoxie (0 h Hypoxie) Steuerplatte. Hinweis: Es ist wichtig, um eine robuste Hypoxie-abhängigen Luciferase-Reaktion zu beobachten für das Screening geeignet sind. Ein fünfzehnfachen Erhöhung der Reporteraktivität gilt als ausgezeichnet.
- Erstellen Sie ein Master-Steuerplatte, die vier Hochsteuer (HIF1A siRNA) Wiederholungen (wells A1-D1), vier Low-Regelung (FIH1 siRNA) repliziert (Brunnen A12-D12) enthält, nur vier Puffer Kontrolle Wiederholungen (Brunnen E1-H1) und vier Nicht-Ziel-siRNA Kontrollwiederholungen (E12-H12), wobei alle Pools siRNA in einer Konzentration von 200 nM. Hinweis: High-und Low-Steuerelemente werden basierend auf bekannten Weg Regulatoren, und erhöhen decr gegründeterleichtern die Hypoxie-Antwort auf. Alternativ kann der Assay-Entwicklung unter Verwendung einer Teilmenge der Bibliothek verwendet werden, um geeignete Steuerungen zu identifizieren.
- Mit einer Mehrkanal Pipette 10 ul jeder Kontroll-siRNA in einen sterilen weißen Mauern Testplatte. Bereiten Transfektion Mischung von 0,1 ul Transfektionsreagenz in 10 ul reduzierten Serummedium (1:100) pro Vertiefung und Transfer 10 ul Transfektionsmischung jeder Steuer gut. Pipette auf und ab kurz zu mischen, und lassen Sie die Platte für 20-60 min ruhen der Transfektion Komplexbildung zu ermöglichen.
- Bereiten Sie eine Zellsuspension von 75.000 Zellen / ml mit der zweiten Flasche U20S-HRE-Zellen. Mit einer Mehrkanal-Pipette 80 ul (6.000 Zellen) der Zellsuspension in jede Transfektion Mix. Übertragen Sie die Platte an einem befeuchteten 37 ° C Inkubator mit 5% CO 2 für 24 Stunden, dann zu einer Hypoxie-Workstation übertragen für weitere 24 Stunden und führen Luciferase-Assays wie in den Schritten 1,6-1.8.
- Berechnen Sie den Z-Faktor für den hohen und niedrigen Kontrollen mit Hilfe der Formel Z = 1 - [3 x (Standardabweichung der hohen Steuer + Standardabweichung der niedrigen Steuer) / (Mittelwert der hohen Steuer - Mittel der niedrigen Steuer)]. Hinweis: Ein Wert zwischen 0,5 bis 1 zeigt eine ausgezeichnete Test und sollte angestrebt werden.
2. Primäre Bildschirm
Hinweis: einmal diese Grundbedingungen aus der Testentwicklungsphase vorhanden sind, der primäre Bildschirm kann in dreifacher Ausfertigung in 96-Well-Plattenformat mit dem folgenden Protokoll durchgeführt werden.
- Wachsen 7 x 75 cm 2-Kolben von U20S-HRE-Zellen 80-90% Konfluenz in DMEM, ergänzt mit 10% v / v fötalem Rinderserum (FCS).
Hinweis: Die folgenden Schritte 2,2-2,13 sollte am Tag 1 durchgeführt werden. - Initiieren Sie ein Replikat hergestellt, indem ein Master-Steuerplatte, die 200 ul jeder Kontrolle, wie in Schritt 1.9 und Auftauen eine Verdünnungsreihe der siRNA ubiquitome l beschriebenibrary für mindestens 30 min. Hinweis: enthält jede auch einen Pool von vier siRNAs in 17 x 96-Well-Platten mit den Spalten 1 und 12 zusammengesetzt leer für Kontrollen.
- In einer Steril, Etikett (oder Bar-Code) 17 sterile weiße Mauern Testplatten mit Deckel aus Zahlen von 1 bis 17, entsprechend jeder Platte des siRNA-Bibliothek-Serie.
- Zentrifugieren Sie die Master-Steuerplatte und die siRNA-Bibliothek ubiquitome kurz (1 min bei 2.000 xg), um sicherzustellen, dass alle siRNAs sich am Boden des Brunnens.
- Verwenden Sie eine automatische Flüssigkeitsspender, um Roboter "Stempel" 10 ul jeder Kontrolle von der Hauptsteuerplatte auf den 17 Testplatten mit dem gleichen 96-Loch-Spitze-Stack für jede Platte.
- Verwendung einer frischen 96 Vertiefungen Spitze Stapel für jedes der Bibliotheksplatten 17, Transfer 10 ul jeder ubiquitome siRNA zu seinem entsprechenden Testplatte unter Verwendung eines automatisierten Flüssigkeitsabgabevorrichtung.
- Bereiten Sie 40 ml Transfektionsreagenz für die 17 Testplatten mit den Verhältnis es1.10 tablished. Hinweis: Diese enthält zusätzlich 20 ml für die "Totvolumen" in der Zellspender zu berücksichtigen. Der "tote Volumen" bezieht sich auf das Volumen der Flüssigkeit kontinuierlich im Rohrleitungsnetz in der Zelle vorhanden Spender.
- Eine automatisierte Zellspender zu 10 &mgr; l Transfektionsreagenz in jede Vertiefung der 17-Assayplatten übertragen. Kurz schütteln die Testplatten auf einem Schüttler (1 min bei 500 Umdrehungen pro Minute), um eine gute Durchmischung von siRNA und Transfektionsreagenz zu ermöglichen, dann lassen noch bei Raumtemperatur für 20-60 min auf siRNA ermöglichen: Transfektionsreagenz Komplexbildung.
- Zweimal waschen mit zwei 75 cm 2-Kolben von U20S-HRE-Zellen mit 10 ml PBS und nehmen mit 2 ml Trypsin-EDTA bei 37 ° C für 5 min. In 8 ml DMEM 10% FBS zu jedem Kolben und Pipette nach oben und unten mehrere Male, um eine homogene Zellsuspension erstellen. Kombinieren Sie Zellen aus beiden Flaschen und Transfer in ein steriles 50 ml-Kunststoffrohr.
- Berechnen Zellkonzentration durch Pipettieren20 ul Zellen in zweifacher Ausfertigung zu einer Zellzählung Kammer und die Berechnung der durchschnittliche Zellkonzentration von zwei Lesungen eines automatisierten Zellzähler wie in Schritt 1.3 beschrieben.
- Planen 155 ml Zellsuspension verdünnt mit DMEM 10% FBS in einer Konzentration von 75.000 Zellen pro ml in sterile Kunststoffbehälter. Hinweis: Dieses Volumen ist ausreichend für 17 Platten und enthält zusätzlich 25 ml für das Totvolumen in der Zellspender.
- In einer sterilen Magnetrührer zu der Zellsuspension und auf ein Rührwerk im Inneren des Steril gesetzt, um die Zellklumpenbildung zu begrenzen. Äquilibrieren die automatisierte Zellspender mit der Zellsuspension und setzen ihn auf 80 ul der Zellen pro Well (6000 Zellen) zu verzichten.
- Legen Sie jede der 17 Platten, die wiederum auf die automatisierte Zellspender und verzichten Zellen. Aufzeichnen der Zeit und Stapel Platten in Gruppen von 5 platzieren Stapel in einer feuchten 37 º C-Inkubator bei 5% CO 2 für 24 Stunden. Hinweis: die EndkonzentrationsiRNA Pool wird 20 nM in 100 ul Gesamtvolumen sein.
Hinweis: Die folgenden Schritte 2,14-2,15 sollte am Tag 2 durchgeführt werden. - Initiieren Sie die zweite Wiederholungs am Tag 2 nach dem Verfahren in Tag 1 für replizieren 1 (2,2 bis 2,13) skizziert.
- Nach 24 Stunden der Transfektion, Übertragungsplatten aus Replikat 1 in einer sterilen Umgebung zu einer Hypoxie-Workstation mit 1% Sauerstoff eingestellt und lassen für 24 Stunden, um die HRE-Reporter zu induzieren.
Hinweis: Die folgenden Schritte 2,16-2,20 sollte an Tag 3 durchgeführt werden. - Initiieren Sie die dritte Replikat nach dem Verfahren in Tag 1 für replizieren 1 (2,2 bis 2,13) skizziert.
- Nach 24 Stunden der Transfektion, Übertragungsplatten aus Replikat 2 in einer sterilen Umgebung zu einer Hypoxie-Workstation mit 1% Sauerstoff eingestellt und lassen für 24 Stunden, um die HRE-Reporter zu induzieren.
- Zwei Stunden vor replizieren 1 Platten sind der Hypoxie-Arbeitsstation entfernt werden, bereiten 2x 200 ml Luciferase-Lyse / Assay-Puffer wie in 1.6 beschrieben. Hinweis: dieses Volumen inumfasst weitere 20 ml, um sicherzustellen, der Pufferspeicher bleibt gut abgedeckt.
- Entfernen replizieren ein Assay-Platten aus der Hypoxie-Workstation nach 24 Stunden Hypoxie-Exposition und mit Hilfe eines automatisierten Flüssigkeitsspender, 100 l 2x Luciferase-Lyse / Assay-Puffer zu jedem Testplatte und Abdeckung mit Klarsichtfolie.
- Schütteln der Platten auf einem Zell Schüttler für 10 min bei 500 rpm, um gründlich die Zellen zu lysieren. Übertragen Sie jede Platte wiederum mit einem Luminometer Plattenlesegerät und Platten Lumineszenz, wie in Schritt 1.8 beschrieben.
Hinweis: Die folgenden Schritte 2,21-2,22 sollte am Tag 4 durchgeführt werden. - Nach 24 Stunden der Transfektion, Übertragungsplatten aus Wiederholungs 3 in einer sterilen Umgebung zu einer Hypoxie-Workstation mit 1% Sauerstoff eingestellt und lassen für 24 Stunden, um die HRE-Reporter zu induzieren.
- Lesen Sie das Replikat 2 Platten durch folgende Schritte 2,18-2,20 für replizieren 1 verwendet wird.
Hinweis: der folgende Schritt 2.23 sollte an Tag 5 durchgeführt werden. - Lesen Sie die Wiederholungs 3 Platten von folgenden2,18-2,20 Schritt für replizieren 1 verwendet wird.
- Kompilieren aller drei Replikate des primären Bildschirm, und berechnen Sie den Z-Faktor für jede Platte nach der Formel in 1.12 gegeben. Hinweis: wenn eine Platte hat eine Z-Faktor von weniger als 0,5, betrachten Wiederholung dieser Platte, um die Datenqualität zu verbessern.
- Berechnen Sie den Variationskoeffizient (CV) für die hohen und die niedrigen Steuerelementen mithilfe der folgenden Formel: 100 x Standardabweichung der Kontrolle / durchschnittlich Kontrolle. Hinweis: Lebenslauf sollte so niedrig wie möglich sein, ist es sinnvoll, von 10 bis 15% Abweichung zu erwarten. Wenn Variation ist deutlich höher, betrachten Wiederholung der Platte.
- Berechnen Prozent Aktivierung jeder siRNA pro Platte mit Hilfe der folgenden Formel: [(Mittelwert der hohen Steuer - siRNA score) / (Mittelwert der hohen Steuer - Mittel der niedrigen Steuer)] x 100 Darüber hinaus berechnen die Nicht-Ziel-(NT. )-fache der einzelnen siRNA unter Verwendung der Formel [Sample data / Mittelwert von NT]. Für beide Prozent-Aktivierung und NT-fach, berechnen Sie den Durchschnitt der drei replicAtes und kompilieren Sie die Werte.
- Mit den Rohdaten, die Berechnung der Spearman Rangkorrelationskoeffizient (SRCC), um zu bestimmen, wie eng die drei Wiederholungsplatten korrelieren mit der Formel:
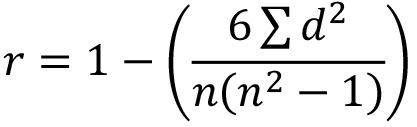
wobei r = SRCC; d = Differenz zwischen den beiden Zahlen in jedem Paar von Reihen und n = Anzahl der Paare von Daten. Hinweis:. Eine gute Korrelation zwischen Platte Wiederholungen werden Werte in der Nähe, um 1 Wenn ein Replikat zeigt niedrigen SRCC gegen die anderen 2 Platten, weiter zu untersuchen und prüfen, zu wiederholen, dass Platte. - Entscheiden Sie, welche siRNAs für Sekundärscreening (bis zu 80) ausreichender Interesse sind. Beschäftigen benutzerdefinierte Cut-Offs (zB Auswahl aller siRNAs, die weniger als 5% oder über 95% Prozent-Aktivierung, oder weniger als 0,5 NT-fach oder mehr als 1,5 NT-fach) oder wenden Bioinformatik oder wissensbasierte Argumentation für Cluster suchen trifft innerhalb der gleichen UBL Weg fallen. Hinweis:Normalerweise wird eine Kombination dieser Methoden bestimmt, welche siRNA in sekundären Screening genommen.
3. Sekundäre Bildschirm
Hinweis: eine bestätigende sekundären Bildschirm wird auf der Grundlage einer maximal 80 siRNAs von Interesse aus der primären Bildschirm durchgeführt. Diese Zahl kann bequem mit jeder Wiederholung mit vollen Kontrollen nach dem ersten Bildschirm auf einem einzigen 96-Loch-Platte plattiert (siehe 2.2) durchgeführt werden. Es ist sehr nützlich, um zu bestätigen, dass die in der primären Bildschirm identifiziert Regula reproduzierbar entlocken den gleichen Phänotyp, und dies wird in die Verfeinerung der Triage-Entscheidungen, auf die trifft sollte bis zum letzten tertiären / Entfaltung Bildschirm durchgeführt werden zu unterstützen.
- Wachsen 1 x 75 cm 2-Kolben-HRE U20S-Zellen 80-90% Konfluenz in DMEM mit 10% FBS.
- Notieren Sie sich die Kennzeichen und den Standort der primären Bildschirm Hits, und planen ihre neue Position auf dem zweiten Bildschirm Kirschen gepflückt Masterplatte, so dass die Spalten 1 eind 12 kostenlos für Kontrollen. Bereiten Sie die Masterplatte wie in 1.9, aber mit einem Gesamtvolumen von jedem Steuer von 50 pl.
- Tauen Sie eine zweite Verdünnungsreihe der ubiquitome Bibliothek (beiseite für cherry-picking einzelnen siRNA-Pools) für mindestens 30 min. Zentrifugieren Sie die Platten kurz (1 min bei 2.000 xg) fügen Sie dann 50 ul der sekundären Bildschirm siRNAs, um ihre neue Platte Orten auf der Kirschen gepflückt Masterplatte.
- Führen Sie den zweiten Bildschirm mit der gleichen Basisprotokoll für den primären Bildschirm mit folgenden Änderungen vor: (a) alle drei Wiederholungen laufen am selben Tag in einer Testplatte pro Wiederholung und (b) einstellen Volumina von Reagenzien entsprechend.
4. Tertiär / Entfaltung Bildschirm
Hinweis: tertiäre oder Entfaltung Screening ist auf maximal 20 siRNAs aus dem sekundären Bildschirm durchgeführt. Dieser Schritt besteht darin, die Wirkung der Zuschlags prüfen unter Verwendung jedes einzelnen siRNA vom ursprünglichen Pool von vier. Normalerweise sollte mindestens zwei Einzel siRNA-Doppelstränge aus jedem Pool denselben Phänotyp unerlaubt auf ein vernünftiges Maß an Vertrauen, dass der beobachtete Phänotyp ist nicht durch siRNA Off-Target-Effekte haben. Um das Vertrauen in diesem Stadium weiter zu erhöhen, können zusätzliche einzelne siRNA-Duplexe Targeting das betreffende Gen konstruiert und auf ihre Fähigkeit, die gegebenen Phänotyp hervorrufen getestet werden. Ergebnisse aus diesen Experimenten kann dann verwendet werden, um die Punktzahl H, wobei H = 0,6 oder mehr (das heißt, wenn mindestens 3 der 5 einzelnen siRNAs hervorrufen den Phänotyp) als akzeptabel 6 zu berechnen.
- Wachsen einem 75 cm 2-Kolben-HRE U20S-Zellen 80-90% Konfluenz in DMEM mit 10% FBS.
- Erstellen Sie eine Platte Karte für den tertiären Bildschirm durch die Planung der Lage der vier einzelnen siRNA-Doppelstränge von jedem Treffer auf dem Bildschirm tertiären Masterplatte, so dass die Spalten 1 und 12 frei für Kontrollen. Bereiten Sie die Masterplatte wie in 1.9, sondern mit einem GesamtLautstärke der einzelnen Steuer von 50 ul.
- Tauen die Entfaltung / siRNA einzelnen Platten und 50 ul der einzelnen siRNAs (200 nM) in jede Vertiefung des tertiären Bildschirm Masterplatte entsprechend der Platten Karte (4.2) für drei x 10 ul Wiederholungen und eine komfortable 20 ul Über ermöglichen .
- Führen Sie die tertiäre Bildschirm mit der gleichen Basisprotokoll für den primären Bildschirm beschrieben mit folgenden Änderungen: (a) alle drei Wiederholungen laufen am selben Tag in einer Testplatte pro Wiederholung und (b) einstellen Volumina von Reagenzien entsprechend
Hinweis: Idealerweise sollte die Schwelle für die einzelnen siRNA-Doppelstränge sollten die gleiche Stringenz eingestellt werden abgeschnitten als für den Pool. Jedoch kann es akzeptabel sein, um die Schwelle von 10-20% für einzelne siRNAs entspannen, insbesondere wenn zumindest einer anderen Duplex innerhalb der eingestellten Schwellenwert fällt. Es lohnt sich, unter Berücksichtigung, dass die individuelle Wirkung von siRNAs kann weniger sein als die derder Pool, und umgekehrt, in einigen Fällen einzelne siRNAs kann eine stärkere Wirkung auf die zelluläre Phänotyp in Isolation, als wenn es in den Pool (auch bei Konzentration wird berücksichtigt) existiert zu zeigen.
Ergebnisse
Vor dem Screening wird die Hypoxie-Reaktionsfähigkeit der HRE U20S-Zellen etabliert. U20S-HRE-Zellen exprimieren ein Reporterkonstrukt, bestehend aus Leuchtkäfer-Luciferase stromabwärts drei Tandemkopien der Hypoxie-Antwortelement, das von der HIF1A/HIF1B Heterodimer bei Einwirkung von Hypoxie (1A) gebunden ist, kondensiert ist. Die Zellen werden in einer Hypoxie-Workstation für eine Reihe von mal zu ermitteln, welche Hypoxie Exposition produziert die wirksamste Antwort für das Screening gestellt. ...
Diskussion
Die Verwendung von genomweiten siRNA-Bildschirme in Säugerzellen hat sich als äußerst wertvoll bei der Identifizierung neuer Regulatoren von verschiedenen biologischen Wege zu sein. Hier haben wir den Einsatz einer gezielten ubiquitome siRNA Bildschirm, um Regulatoren des HIF1A-vermittelte zelluläre Antwort auf Hypoxie zu identifizieren beschrieben. Gezielte Bildschirme werden immer attraktiver, da sie in der Regel billiger, schneller, einfacher zu verwalten und Bericht nur auf den Weg Komponenten, in denen die Ermi...
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Diese Arbeit wurde von The Wellcome Trust, Glaxosmithkline (GSK) und der schottischen Institut für Zelluläre Signaltransduktion (jetzt Teil des MRC Protein-Phosphorylierung und Ubiquitinierung Unit) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Automated Liquid Dispenser | Fluid-X | XPP-721 | http://www.fluidx.eu/BIOTRACK/xpp-721-liquid-handling-system.html |
| White Walled Assay Plate | Greiner Bio One | 655083 | http://www.greinerbioone.com/en/row/articles/catalogue/article/37_11/13221/ |
| Clear Plate Film | Perkin Elmer | 1450-461 | http://www.perkinelmer.co.uk/Catalog/Product/ID/1450-461 |
| siRNA library | Thermo Scientific | On-Target Plus | http://www.thermoscientificbio.com/rnai-and-custom-rna-synthesis/sirna/on-targetplus-sirna/search-gene/ |
| Transfection reagent | Invitrogen | Lipofectamine RNAimax | http://www.invitrogen.com/site/us/en/home/Products-and-Services/Applications/Protein-Expression-and-Analysis/Transfection-Selection/lipofectamine-rnaimx.html |
| Reduced Serum Medium | Invitrogen | Optimem | http://products.invitrogen.com/ivgn/product/31985062?ICID=search-product |
| DMEM | Invitrogen | 41965-039 | http://products.invitrogen.com/ivgn/product/41965039# |
| FBS | Invitrogen | 16000-044 | https://products.invitrogen.com/ivgn/product/16000044?ICID=search-product# |
| Tryspin-EDTA | Invitrogen | 25300-054 | https://products.invitrogen.com/ivgn/product/25300054?ICID=search-product# |
Referenzen
- Hochstrasser, M. Origin and function of ubiquitin-like proteins. Nature. 458, 422-429 (2009).
- Schulman, B. A., Harper, J. W. Ubiquitin-like protein activation by E1 enzymes: the apex for downstream signalling pathways. Nat Rev Mol Cell Biol. 10, 319-331 (2009).
- Siomi, H., Siomi, M. C. On the road to reading the RNA-interference code. Nature. 457, 396-404 (2009).
- Kaelin, W. G., Ratcliffe, P. J. Oxygen sensing by metazoans: the central role of the HIF hydroxylase pathway. Mol Cell. 30, 393-402 (2008).
- Melvin, A., Mudie, S., Rocha, S. The chromatin remodeler ISWI regulates the cellular response to hypoxia: role of FIH. Mol Biol Cell. 22, 4171-4181 (2011).
- Bhinder, B., Djaballah, H. A simple method for analyzing actives in random RNAi screens: introducing the "H Score" for hit nomination & gene prioritization. Comb Chem High Throughput Screen. 15, 686-704 (2012).
- Bett, J. S., et al. The P-body component USP52/PAN2 is a novel regulator of HIF1A mRNA stability. Biochem J. 451, 185-194 (2013).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J Biomol Screen. 4, 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nat Methods. 6, 569-575 (2009).
- Stagg, H. R., et al. The TRC8 E3 ligase ubiquitinates MHC class I molecules before dislocation from the ER. J Cell Biol. 186, 685-692 (2009).
- Zhang, Y., et al. RNF146 is a poly(ADP-ribose)-directed E3 ligase that regulates axin degradation and Wnt signalling. Nat Cell Biol. 13, 623-629 (2011).
- Jackson, A. L., et al. Expression profiling reveals off-target gene regulation by RNAi. Nat Biotechnol. 21, 635-637 (2003).
- Birmingham, A., et al. 3' UTR seed matches, but not overall identity, are associated with RNAi off-targets. Nat Methods. 3, 199-204 (2006).
- . Whither RNAi. Nat Cell Biol. 5, 489-490 (2003).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten