需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
PIP-on-a-chip:蛋白质 - 磷酸肌醇相互作用的无标记研究
摘要
我们在微流体平台的背景下提供支持的脂质双层,以使用基于pH调节的无标记方法研究蛋白质 - 磷酸肌醇相互作用。
摘要
许多细胞蛋白质与膜表面相互作用以影响基本的细胞过程。这些相互作用可以针对膜内的特定脂质组分,如在磷酸肌醇(PIP)的情况下),以确保特定的亚细胞定位和/或活化。已广泛研究PIP和细胞PIP结合结构域以更好地了解其在细胞生理学中的作用。我们对支持的脂质双层(SLB)进行了pH调节测定,作为研究蛋白质-PIP相互作用的工具。在这些研究中,pH敏感的邻 -磺酰罗丹明B缀合的磷脂酰乙醇胺用于检测蛋白质-PIP相互作用。当蛋白质与含PIP的膜表面结合时,调节界面电位( 即局部pH值的变化),使探针的质子化状态发生变化。通过使用磷脂酶C delta1 Pleckstr提出了pH调节测定的成功应用的案例研究在同源性(PLC-δ1PH)结构域和磷脂酰肌醇4,5-二磷酸(PI(4,5)P 2 )相互作用为例。这种相互作用的表观解离常数( K d,app )为0.39±0.05μM,类似于其他人获得的K d值。如前所述,PLC-δ1PH结构域是PI(4,5)P 2特异性,显示与磷脂酰肌醇4-磷酸的结合较弱,并且不与纯磷脂酰胆碱SLB结合。 PIP片上测定优于传统的PIP结合测定,包括但不限于低样品体积和无配体/受体标记要求,测试高和低亲和力膜相互作用的能力与小和大分子,提高信噪比。因此,PIP-on-a-chip方法的使用将有助于阐明各种膜相互作用的机制。此外,这种方法可能是你的确定调节蛋白质与膜相互作用的能力的治疗剂。
引言
无数的相互作用和生物化学过程发生在二维流体的膜表面上。真核细胞中的膜封闭的细胞器不仅在生物化学过程及其相关的蛋白质组学中而且在其脂质组成中是独特的。一类特殊的磷脂是磷酸肌醇(PIP)。即使它们包括只有1%的细胞脂质组的,它们在信号转导,自噬和膜运输至关重要的作用,其中包括1,2,3,4。通过细胞PIP激酶肌醇头部基团的磷酸化动态产生七个PIP头基是单- ,双- ,或三-磷酸化5。另外,PIP定义膜的亚细胞同一性,并且用作含有一种或多种磷酸苷酶的蛋白质/酶的专门的膜对接位点itide结合结构域,例如,pleckstrin同源(PH),PHOX同源性(PX),和底物蛋白N端同源性(ENTH)6,7。最好研究的PIP结合结构域之一是在高纳摩尔 - 低微摩尔范围亲和力8内与磷脂酰肌醇4,5-二磷酸(PI(4,5)P 2 )特异性相互作用的磷脂酶C(PLC)-δ1PH结构域,9,10,11。
已经开发了多种定性和定量的体外方法,用于研究这些相互作用的机制,热力学和特异性。在最常用的PIP结合测定中,表面等离子体共振(SPR),等温量热法(ITC),核磁共振(NMR)光谱,脂质体浮选/沉降测定和脂质印迹(脂肪印迹/ PIP条)12,13。即使这些被广泛使用,它们都具有许多缺点。例如,SPR,ITC,和NMR需要大量的样品,昂贵的仪器,和/或受过训练的人员12,13的。一些测定法形式,例如基于抗体的脂质的印迹利用的PIP的水可溶形式,并在非生理方式12,14,15,16呈现它们。此外,脂质印迹不能可靠定量,它们经常导致假阳性/阴性的观察12,17,18。为了克服这些挑战并改进当前的工具集,在上下文中基于支持的脂质双层(SLB)建立了一种新的无标记方法 icrofluidic平台,将其成功地应用于蛋白质-PIP相互作用的研究( 图1)19。
用于检测蛋白质-PIP相互作用的策略是基于pH调节感测。这涉及一种pH敏感的染料,其具有直接与磷脂酰乙醇胺脂质头部组20缀合的邻 -磺酰罗丹明B( o SRB)。 o SRB-POPE探针( 图2A )在低pH下是高度荧光的,在高pH下淬灭,在7.5mol%含PI(4,5)P 2的SLB中具有约6.7的pKa( 图5B )。 PLC-δ1PH结构域已被广泛使用,用于验证蛋白质-PIP结合方法由于其对PI(4,5)P 2( 图5A)高特异性21,22"> 23,24,25 .Hence,我们的理由是,PLC-δ1PH结构域可以用来测试其结合至PI(4,5)P 2通过PIP-上的单芯片测定法。该PH结构域构建体在此研究中使用具有净正电荷(等电点8.4),并且因此吸引OH -离子( 图5C)在结合PI(4,5)P 2含SLBs中,PH结构域所带来的OH -离子的膜表面,这反过来又调制所述界面电势并转移öSRB-POPE( 图5C)26,随着PH结构域浓度的函数,荧光被猝灭( 图6A)的质子化状态。最后,将归一化的数据是适合于结合等温线以确定PH结构域-P1(4,5)P 2相互作用的亲和力( 图6B , 6C )。/ P>
在这项研究中,提供了详细的方案,以在微流体平台内进行蛋白质结合含PIP的SLBs。该协议使读者将微流体装置和囊泡制备装配成SLB形成和蛋白质结合。此外,提供了用于提取PLC-δ1PH域-PI(4,5)P 2相互作用的亲和度信息的数据分析方向。
Access restricted. Please log in or start a trial to view this content.
研究方案
1.清洁玻璃盖板
- 稀释7倍的清洁溶液(参见材料表 )用100毫米深硼硅酸盐玻璃皿的去离子水将其稀释7倍,并将其在平板热板上加热至95℃,持续20分钟,或直到混浊溶液变澄清。
注意:解决方案将会很热,请注意避免身体受伤。 7x清洁溶液是[氧化 - (氧化(膦酰氧基)磷酰基]氧磷酰基]磷酸六钠,2-(2-丁氧基乙氧基)乙醇,1,4-双(2-乙基己氧基)-1,4-二氧代丁烷钠-2-磺酸盐和无害添加剂。 - 使用镊子,在铝染色架上沿交替方向排列盖玻片(40毫米长x 22毫米宽×0.16-0.19毫米高)。在每个盖玻片之间保持空位,以防止它们彼此接触。
- 将染色架完全淹没在清洁溶液中。保留盖玻片解决方案1小时。
注意:当水蒸发时,继续加入新鲜的去离子水以保持溶液浓度不变。 - 将染色架从清洁溶液中取出,并用大量的去离子水清洗盖玻片以除去洗涤剂。
- 用氮气干燥盖玻片(每个染色架约5分钟),然后将盖玻片放置在窑炉中550℃下6小时退火。
注意:这是平滑玻璃表面粗糙特征的关键步骤。 - 将盖玻片放在塑料容器中,使其远离灰尘。
2.制作微图案PDMS块
- 在大塑料称重舟皿中以10:1(w / w)的比例混合聚二甲基硅氧烷(PDMS)预聚物和固化剂,并在真空中真空脱气1小时,真空强度为500托或更低。
注意:PDMS是一种透明和惰性的硅氧烷聚合物。获得所需的PDMS块t(0.5厘米),将55克预聚物与5.5克固化剂混合。 - 将含有相同SU-8微图案的多个重复的硅母版置于大(10厘米基径)的塑料称重船中,并倒入脱气的PDMS中。然后,在60℃的干燥炉中固化过夜。
注意:微图案尺寸足够大,可以用肉眼看到。每个图案包含八个微通道(100μm宽×40μm高×1cm长),间隔40μm( 图1A , 1B )。在纳米加工设备27中制造由硬烘烤的SU-8光致抗蚀剂组成的硅晶片模具。对于主制备的其它方法包括氢氟酸(HF)蚀刻,高温水腐蚀,xurography和3D印刷28,29,30,31。 COMMERC也可以使用硅制备的主要来源。微流体装置的图案设计有起草软件(见材料表)。包含图案设计的原始文件作为补充文件"Pattern Design.dwg"提供,可以直接提供给制造商。 - 轻轻地用手剥离硅胶主板上的PDMS。使用外科手术刀和尺子标记矩形中每个微图案的边界。然后,将PDMS切成块。
- 如图3B所示 ,每个微通道的两端用活检穿孔(1.0mm孔直径)打入16孔(每块8个入口和8个出口)。
- 在每个PDMS块上涂抹胶带,以保护微图案免受损坏和灰尘。将它们存放在塑料容器中。
3.准备小单层囊泡(SUVs)
注意:阴性对照双层组成是99.5mol%1-棕榈酰-2-油酰-sn-甘油-3-磷酸胆碱(POPC)和0.5mol% 邻磺酰罗丹明B-1-棕榈酰-2-油酰-sn-甘油-3-磷酸乙醇胺( o SRB-教皇)。测试双层组成为92.0mol%POPC,0.5mol% o SRB-POPE和7.5mol%的L-α-磷脂酰肌醇-4-磷酸(PI4P)或L-α-磷脂酰肌醇-4,5-二磷酸酯(PI( 4,5)P 2 )。以下是制备92.0mol%POPC,0.5mol% o SRB-POPE和7.5mol%含PI(4,5)P 2的SUV的方法。 øSRB-POPE在本研究中使用的合成先前被描述20。
- 计算需要混合的POPC,PI(4,5)P 2和o SRB-POPE的体积,占以下总量:2.22mg POPC,0.26mg PI(4,5)P 2 ,和2.1×10 -5毫克邻 SRB-POPE的。
- 吸取计算的POPC体积,PI(4,5)P 2 ,<将SRB-POPE注入单个20mL玻璃闪烁瓶中。
注意:POPC和o SRB-POPE储备溶液含有氯仿溶液,而PI(4,5)P 2 (和其他磷酸肌醇)储备液中加入氯仿:甲醇:水(20:9:1)溶剂混合物。为了准确,使用前用氯仿将吸液管吸干。为安全起见,此步骤应在化学通风柜内进行。 - 将混合物在化学通风柜内的氮气流中干燥10分钟,或直到溶剂蒸发,并在小瓶的底部形成薄的脂质膜。
- 在10 mTorr的真空强度下真空干燥混合物≥3h,以除去任何残留的有机溶剂。
注意:3小时干燥时间足以满足本协议中描述的SUV制剂规模,这足以满足50次实验。如果需要更大规模的SUV制备,则可以进行隔夜干燥。 - 再加水用20mL HEPES,100mM NaCl,pH7.0的5mL运行缓冲液将其置于35kHz的操作频率的超声波浴中30分钟。
注意:混合步骤3.1中指定的脂质的量并在步骤3.5中加入5mL的运行缓冲液,得到0.5mg / mL的囊泡悬浮液。在这个阶段,囊泡悬浮液是单层和多层囊泡的混合物( 图2B )。该解决方案将显示为粉色/紫色,相对浑浊。 - 用液氮和水浴在40℃下冻融囊泡悬浮液以获得单层囊泡。重复冻融10次。
注:(可选)以确保不存在在悬浮液中的非单层囊泡的,离心步骤可以应用32,33。 - 使用脂质挤出机将小泡悬浮液通过0.10μm轨道蚀刻的聚碳酸酯膜挤出(参见表格)丰富了SUV。重复挤压10次。
注意:可以使用15-mL锥形管来收集挤出的囊泡。该溶液在此阶段应无浊度。可以通过动态光散射(DLS)确认SUV直径( 图2C )。 SUV最适合于形成SLB 34 。 - 用铝箔盖住锥形管,并保存在4°C直到准备使用。
4.组装微流控装置
- 通过使用水洗瓶将去离子水喷射穿过孔,测试PDMS块的入口和出口堵塞。然后用氮气干燥PDMS块。
注意:此步骤还将清除可能已被捕获在通道内和/或通道之间的任何灰尘颗粒。如有必要,用氮气除尘预清洁的盖玻片,以除去任何灰尘颗粒。 - 放置PDMS块并预先清洁氧气等离子体系(见表格)样品室内的盖玻片(见第1节),用氧等离子体暴露45秒,功率设定为75瓦,氧气流速为10厘米3 /分,真空强度为200毫托。
- 在氧等离子体处理后立即将PDMS块的图案化表面立即与盖玻片接触。轻轻按压以清除接触部位的任何气泡。
- 将设备放置在100℃的热平板上3分钟以增强粘合。
- 使用带有100%乙醇的湿式无绒毛擦拭( 例如 kimwipe)从设备的顶部(PDMS)和底部(玻璃)中除去任何灰尘颗粒。然后,将设备贴在玻璃显微镜载玻片的顶部。
注意:不要使用过量的乙醇,并避免乙醇进入入口和出口通道。建议在设备仍然热的情况下执行此步骤,以确保乙醇立即蒸发。玻璃微型振荡玻片可以储存在100%乙醇溶液中,以除去灰尘和其他污染物。
5.形成支持的脂质双层(SLB)
- 将100μL来自步骤3.8的PI(4,5)P 2的SUV转移到0.65mL的微量离心管中。通过加入6.4μL0.2N盐酸将溶液的pH调节至〜3.2。
注意:用配有微量pH探针的pH计确认溶液的pH值(参见表格)。 - 通过入口将10μLpH调整后的SUV溶液吸入每个通道,并通过移液管施加压力,直到溶液到达出口。将吸头从吸液管中取出并将其连接到设备上。
- 对每个通道重复上述步骤,然后在室温下孵育10分钟。
注意:在器件组装后,应立即执行将囊泡注入微通道。 - 入口和出口管的切割套件ea长60厘米(入口管)和8厘米(出口管)长。
注意:对角切割管道并创建锋利的边缘,可以更轻松地将管道插入入口和出口。出口管应具有弧形。入口管的长度可以根据显微镜设置而变化。管的内径为0.05厘米。 - 使用镊子将出口管道连接到设备,然后将设备粘贴到显微镜平台上。
- 将入口管的一端浸没在25 mL运行缓冲液中,包含在锥形管中,并将其胶带,以确保管道固定。
- 将锥形管放置在比设备更高的地面(约20厘米)上,以便通过重力流将溶液推入微通道;可以使用实验室插孔来实现这一点。
- 对于每个入口管,使用注射器从管道的自由端抽出1 mL运行缓冲液。从进样口取下移液器吸头并插入入口管的自由端进入设备。
注意:将一滴运行的缓冲液引入入口以减少在此步骤期间气泡被引入通道的可能性。入口管连接到设备后,使用无毛巾擦去多余的缓冲液。 - 重复上述步骤将所有入口管件连接到设备。
注意:通过通道的流动缓冲液有助于去除过量的囊泡,并将双层平衡到实验条件。 - 打开显微镜控制软件(见表格材料)。在左面板上,点击"显微镜"选项卡,选择"10X"目标。
- 点击工具栏上的"Live",然后点击"Alexa 568"图标图标。使用精细和校准调节旋钮,专注于微通道。
- 扫描设备以检查SLB和通道的质量( 图8 )。然后,点击工具栏上的"FL快门关闭"图像图标。
- 点击"采集"选项卡,在"基本调整"下选择"曝光时间"。将曝光时间设置为"200 ms"。
- 在左侧面板上,点击"多维采集",并在滤镜菜单下选择红色通道(标记为"Alexa 568")。然后点击"时间流逝"菜单,将时间间隔设置为5分钟,持续时间为30分钟,然后单击"开始"。
注意:根据持续时间(30分钟)和流速(〜1.0μL/ min),使用30μL运行缓冲液来平衡通道内的SLB, 即所有8个通道使用240μL运行缓冲液。 - 选择"测量"选项卡下的"圆"工具,并在任意通道中绘制一个圆。选择圆圈时,右键单击,然后选择"属性"。在"配置文件"选项卡下,选中"全部T"查看荧光强度作为时间的函数。
- 在进行下一步之前,确保该曲线达到平台,表示平衡。
- 将缓冲溶液降低到与设备相等的地面以停止流动。
- 保存延时文件。
6.测试与PI(4,5)含P 2的SLB的PLC-δ1PH域相互作用
- 使用运行缓冲液作为稀释剂准备PH域的稀释液。
注意:由于设备内有8个通道,因此至少要使用两个通道进行空白控制。选择每边的远端,并使用剩余的通道进行蛋白质稀释。测试以下PH域浓度:0.10,0.25,0.50,1.00和2.50μM。每次稀释约200μL足以在30分钟内达到平衡。 - 一次一个,分离每个出口管,并将200μL的每种蛋白质稀释液加入出口通道使用移液器。不要施加任何压力,让重力做好工作。从吸液管中取出尖端并将其连接到微流体装置。
- 对每个通道重复上述步骤,并确保在此过程中气泡不会引入通道。
- 将进样管降低到微流体装置下方的地面,以开始将蛋白质流过微通道。将管道的自由端粘贴到废物容器。流动蛋白质稀释30分钟。
注意:这将反转流程,并允许将蛋白质引入通道。所需的平衡和体积的蛋白质稀释的时间将取决于相互作用的亲和力。 - 在软件的左面板上,在"时间段"选项卡下,单击"开始"开始再次成像。
- 重复步骤5.15以确保达到平衡。实验完成后,节省时间文件。
7.评估膜流动性
注意:荧光恢复在漂白(FRAP)实验之后,应使用每批新的SUV和清洁的玻璃盖玻片进行,以确保SLB是流体。
- 按照以前的步骤清洁玻璃盖玻片(1.1-1.6),制造微图案化PDMS块(2.1-2.5),制备SUV(3.1-3.8),组装微流体装置(4.1-4.5)和形成SLB(5.1-5.17)描述。
- 将显微镜聚焦在双层上,将曝光时间设置为200 ms,并将合并为1。
- 使用532 nm,2 mW激光束(13μM半径)照相漂白圆点。光漂白后立即开始在前45秒内每3秒拍摄一系列图像,然后在剩余时间内持续30秒(总持续时间15分钟)。数据采集完成后,保存延时文件。
- 选择漂白的area使用圆形绘图工具提取荧光强度值作为时间的函数。此外,选择附近的两个区域,一个对应于未漂白区域,一个对应于没有任何SLB的区域。
- 使用以下公式计算荧光恢复率( y ):
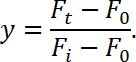
注意:这里F t表示漂白区域的强度作为时间的函数, F i表示漂白前的荧光强度(在这种情况下使用"1"作为归一化值); F 0是背景强度。 - 绘制荧光恢复(y轴)作为时间(x轴)的函数以产生FRAP曲线。然后,将数据拟合为单个指数函数,如下所示:
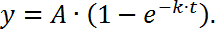
注意:这里A代表移动分数 k是移动部分的动力学常数,用于计算半衰期( t 1/2 ):
- 使用t 1/2计算扩散常数( D ):

注意:这里R表示激光束半径(13μm)。用于流体双层扩散常数应为≥1.0微米2 / s 以下 。
8.处理数据
注意:数据分析的例程将取决于显微镜,图像处理软件和正在使用的曲线拟合软件。
- 打开之前(从步骤5.17)和之后(从步骤6.6)滴定PH域的时间推移文件。转到每个时间流逝文件的最后一帧,并对所有微通道进行线扫描以获得荧光强度数据作为距离(以像素为单位)的函数( 图6A )。将数据传输到电子表格软件。
- 对于每个微通道(PH域加法之前和之后),采样通道内的一些数据和表示基线的通道的任一侧( 图7A )。
注意:空白通道荧光强度应保持不变。所有其他通道被标准化为空白通道,所以至关重要的是有两个空白通道,并确保其荧光强度保持一致。 - 应用下面的公式将数据规范化为空白通道;在图7B中提供了样本计算。

注意:这里ΔF 蛋白表示蛋白质通道的背景扣除荧光强度Δ; F blank表示空白通道的背景减去的荧光强度, 前和后参考蛋白质滴定步骤之前和之后, α表示校正因子,其是蛋白质通道和空白通道的荧光强度之比([ F 蛋白 / F 空白 ] pre )。由于荧光强度作为PH域浓度的函数而降低,归一化数据将为负值。 - 使用曲线拟合软件(参见材料表 ),绘制归一化荧光作为PH域浓度的函数,并适合Langmuir等温线( 图6C ):
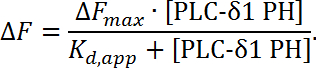
注意:这里ΔF表示荧光强度相对于荧光最大变化的变化rescence强度当蛋白的饱和浓度的情况下(ΔF 最大 ),而K个D,应用程序表示表观解离常数,它等于本体蛋白质浓度([PLC-δ1PH]),在该50%的覆盖率(膜结合复合物)。这是基于平衡的结合测量,因此归一化数据适合于简单的Langmuir等温线,以提取表观实验参数K d,app 。基于拟合软件, PLC-δ1PH-PI(4,5)P 2相互作用的K d,app为0.39±0.05μM( 图6B )。
Access restricted. Please log in or start a trial to view this content.
结果
我们使用pH调节测定法研究了PIP-on-a-chip微器件中的PLC-δ1PH域-PI(4,5)P 2相互作用( 图1 )。通过详细的方案,我们演示了如何准备和组装微流体装置组件,制备小单层囊泡(SUV)( 图2 ),在装置内形成SLB( 图3 ),并测试蛋白质与含有PIP的SLB的结合。 图4中描绘了典型的SLB...
Access restricted. Please log in or start a trial to view this content.
讨论
每个PIP变体虽然浓度低,但存在于特定细胞器的细胞溶质表面,其中它们有助于建立独特的物理组成和细胞器膜1的功能特异性。一个的PIP的最重要的用途是作为一个特定的对接平台需要特定亚细胞定位和/或活化6,7蛋白质的众人。由于其在细胞生理学和疾病中的作用,在生理学相关的背景下研究体外蛋白质-PIP相互作用的能力是?...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者没有什么可以披露的。
致谢
部分支持DS和CEC的授权AI053531(NIAID,NIH); SS和PSC由授权N00014-14-1-0792(ONR)支持。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| Coverslip | |||
| Glass Coverslips: Rectangles | Fisher Scientific | 12-544B | 22 x 40 x 0.16 - 0.19 mm, No. 1 1/2; Borosilicate Glass |
| 7X Cleaning Solution | MP Biomedicals | 976670 | Detergent |
| PYREX Crystallizing Dish | Corning | 3140-190 | Borosilicate glass dish with a flat bottom; Diameter x Height (190 x 100 mm); Distributor: VWR (89090-700) |
| Sentry Xpress 2.0 | Paragon Industries | SC-2 | Kiln |
| Name | Company | Catalog Number | Comments |
| PDMS | |||
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 4019862 | Polydimethylsiloxane (PDMS); Distributor: Ellsworth Adhesives |
| PYREX Desiccator | VWR | 89134-402 | Vacuum Rated |
| Biopsy punch | Harris | 15110-10 | Harris Uni-Core; 1.0 mm diameter; Miltex Biopsy Punch with Plunger (Cat. No. 15110-10) can be used as an alternative |
| Name | Company | Catalog Number | Comments |
| Device | |||
| Plasma Cleaning System | PlasmaEtch | PE25-JW | 2-stage Direct Drive Oil Vacuum Pump, O2 service (Krytox Charged) |
| Digital Hot Plate | Benchmark | H3760-H | Purchased through Denville Scientific (Cat. No. 1005640) |
| Frosted Micro Slides | VWR | 48312-003 | Frosted, Selected, and Precleaned; Made of Swiss Glass; Thickness: 1 mm; Dimensions: 75 x 25 mm; GR 144 |
| Name | Company | Catalog Number | Comments |
| Mold | |||
| AutoCAD | Autodesk | v.2016 | Drafting software for the photomask design |
| Photomask | CAD/Art Services | N/A | Design with black background and clear features was printed at 20k dpi resolution on a transparent mask (5 x 7 in) by CAD/Art Services |
| Silicone Wafers | University Wafer | 1575 | Prime Grade, Single Side Polished; 100 mm (4 inch) Diameter; 525 um Thickness |
| SU-8 50 | MicroChem Corp. | N/A | Negative Tone Photoresist; Penn State Nanofabrication Facility Property |
| SU-8 Developer | MicroChem Corp. | N/A | Penn State Nanofabrication Facility Property |
| Name | Company | Catalog Number | Comments |
| SUV | |||
| 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine | Avanti Polar Lipids | 850457C | POPC |
| L-α-phosphatidylinositol-4-phosphate | Avanti Polar Lipids | 840045X | PI4P |
| L-α-phosphatidylinositol-4,5-bisphosphate | Avanti Polar Lipids | 840046X | PI(4,5)P2 |
| 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphoethanolamine | Avanti Polar Lipids | 850757C | POPE; Required for the synthesis of oSRB-POPE |
| Lissamine Rhodamine B Sulfonyl Chloride (mixed isomers) | ThermoFisher Scientific | L-20 | Required for the synthesis of oSRB-POPE |
| pH Sensitive Fluorescent Lipid Probe (oSRB-POPE) | In-house | N/A | In-house Synthesis (Huang D. et al. 2013) |
| Glass Scintillation Vial | VWR | 66022-065 | 20 mL volume capacity |
| Aquasonic 250D | VWR | N/A | Ultrasonic Water Bath |
| Nuclepore Track-Etched Membranes | Whatman | 110605 | Polycarbonate Membrane; Diameter: 25 mm; Pore Size: 0.1 um; Distributor: Sigma-Aldrich |
| Chloroform | VWR | CX1054-6 | HPLC grade |
| LIPEX Extruder | Transferra Nanosciences | T.001 | LIPEX 10 mL Thermobarrel Extruder |
| Viscotek 802 DLS | Malvern Instruments | N/A | Dynamic Light Scattering; Penn State X-Ray Crystallography Facility Property |
| Name | Company | Catalog Number | Comments |
| Data Analysis | |||
| GraphPad Prism | GraphPad Software | v.6 | Curve-fitting software for data analysis |
| Name | Company | Catalog Number | Comments |
| Microscope | |||
| Axiovert 200M Epifluorescence Microscope | Carl Zeiss Microscopy | N/A | Microscope |
| AxioCam MRm Camera | Carl Zeiss Microscopy | N/A | Camera |
| X-Cite 120 | Excelitas Technologies | N/A | Light Source |
| Alexa 568 Filter Set | Carl Zeiss Microscopy | N/A | Ex/Em 576/603 nm |
| AxioVision LE64 v.4.9.1.0 Software | Carl Zeiss Microscopy | N/A | Image Processing Software |
| Name | Company | Catalog Number | Comments |
| Other | |||
| Tips | VWR | 10034-132 | 200 uL pipette tips; Thin and smooth tip for applying the protein solution into the microfluidic channel |
| Tips | VWR | 53509-070 | 10 uL pipette tips; Thin and smooth tip for applying the vesicle solution into the microfluidic channel |
| Orion Star A321 pH meter | Thermo Scientific | STARA3210 | pH meter |
| Orion micro pH probe | Thermo Scientific | 8220BNWP | micro pH probe |
| N-(2-Hydroxyethyl)-Piperazine-N'-(2-Ethanesulfonic Acid) | VWR | VWRB30487 | HEPES, Free Acid |
| Sodium Chloride | VWR | BDH8014-2.5KGR | NaCl |
| Tubing | Allied Wire & Cable | TFT-200-24 N | Internal Diameter: 0.020-0.026 inches (0.051-0.066 cm); Wall Thickness: 0.010 inches (0.025 cm); Flexible Polytetrafluoroethylene Thin-Wall Tubing; Natural Color |
| Nitrogen Gas - Industrial | Praxair | N/A | Local Provider |
| Oxygen Gas - Industrial | Praxair | N/A | Local Provider |
| Liquid Nitrogen | Praxair | N/A | Local Provider |
参考文献
- Di Paolo, G., De Camilli, P. Phosphoinositides in cell regulation and membrane dynamics. Nature. 443 (7112), 651-657 (2006).
- Shewan, A., Eastburn, D. J., Mostov, K. Phosphoinositides in cell architecture. Cold Spring Harb Perspect Biol. 3 (8), a004796(2011).
- Picas, L., Gaits-Iacovoni, F., Goud, B. The emerging role of phosphoinositide clustering in intracellular trafficking and signal transduction. F1000Res. 5, (2016).
- Lystad, A. H., Simonsen, A. Phosphoinositide-binding proteins in autophagy. FEBS Lett. 590 (15), 2454-2468 (2016).
- Balla, T. Phosphoinositides: Tiny lipids with giant impact on cell regulation. Physiol Rev. 93, 1019-1137 (2013).
- Lemmon, M. A. Membrane recognition by phospholipid-binding domains. Nat Rev Mol Cell Biol. 9 (2), 99-111 (2008).
- Kutateladze, T. G. Translation of the phosphoinositide code by PI effectors. Nat Chem Biol. 6 (7), 507-513 (2010).
- Harlan, J. E., Hajduk, P. J., Yoon, H. S., Fesik, S. W. Pleckstrin homology domains bind to phosphatidylinositol-4,5-bisphosphate. Nature. 371 (6493), 168-170 (1994).
- Garcia, P., et al. The pleckstrin homology domain of phospholipase C-delta 1 binds with high affinity to phosphatidylinositol 4,5-bisphosphate in bilayer membranes. Biochemistry. 34 (49), 16228-16234 (1995).
- Lemmon, M. A., Ferguson, K. M., O'Brien, R., Sigler, P. B., Schlessinger, J. Specific and high-affinity binding of inositol phosphates to an isolated pleckstrin homology domain. Proc Natl Acad Sci U S A. 92 (23), 10472-10476 (1995).
- Flesch, F. M., Yu, J. W., Lemmon, M. A., Burger, K. N. Membrane activity of the phospholipase C-delta1 pleckstrin homology (PH) domain. Biochem J. 389, 435-441 (2005).
- Narayan, K., Lemmon, M. A. Determining selectivity of phosphoinositide-binding domains. Methods. 39 (2), 122-133 (2006).
- Scott, J. L., Musselman, C. A., Adu-Gyamfi, E., Kutateladze, T. G., Stahelin, R. V. Emerging methodologies to investigate lipid-protein interactions. Integr Biol (Camb). 4 (3), 247-258 (2012).
- Dowler, S., Currie, R. A., Downes, C. P., Alessi, D. R. DAPP1: A dual adaptor for phosphotyrosine and 3-phosphoinositides. Biochem J. 342, 7-12 (1999).
- He, J., et al. Molecular basis of phosphatidylinositol 4-phosphate and ARF1 GTPase recognition by the FAPP1 pleckstrin homology (PH) domain. J Biol Chem. 286 (21), 18650-18657 (2011).
- Ceccarelli, D. F., et al. Non-canonical interaction of phosphoinositides with pleckstrin homology domains of Tiam1 and ArhGAP9. J Biol Chem. 282 (18), 13864-13874 (2007).
- Huang, S., Gao, L., Blanchoin, L., Staiger, C. J. Heterodimeric capping protein from Arabidopsis is regulated by phosphatidic acid. Mol Biol Cell. 17 (4), 1946-1958 (2006).
- Yu, J. W., et al. Genome-eide analysis of membrane targeting by S. cerevisiae pleckstrin homology domains. Mol Cell. 13 (5), 677-688 (2004).
- Jung, H., Robison, A. D., Cremer, P. S. Detecting protein-ligand binding on supported bilayers by local pH modulation. J Am Chem Soc. 131 (3), 1006-1014 (2009).
- Huang, D., Zhao, T., Xu, W., Yang, T., Cremer, P. S. Sensing small molecule interactions with lipid membranes by local pH modulation. Anal Chem. 85 (21), 10240-10248 (2013).
- Saxena, A., et al. Phosphoinositide binding by the pleckstrin homology domains of Ipl and Tih1. J Biol Chem. 277 (51), 49935-49944 (2002).
- Knödler, A., Mayinger, P. Analysis of phosphoinositide-binding proteins using liposomes as an affinity matrix. Biotechniques. 38 (6), 858-862 (2005).
- Baumann, M. K., Swann, M. J., Textor, M., Reimhult, E. Pleckstrin homology-phospholipase C-delta1 interaction with phosphatidylinositol 4,5-bisphosphate containing supported lipid bilayers monitored in situ with dual polarization interferometry. Anal Chem. 83 (16), 6267-6274 (2011).
- Saliba, A. E., et al. A quantitative liposome microarray to systematically characterize protein-lipid interactions. Nat Methods. 11 (1), 47-50 (2014).
- Arauz, E., Aggarwal, V., Jain, A., Ha, T., Chen, J. Single-molecule analysis of lipid-protein interactions in crude cell lysates. Anal Chem. 88 (8), 4269-4276 (2016).
- Best, Q. A., Xu, R., McCarroll, M. E., Wang, L., Dyer, D. J. Design and investigation of a series of rhodamine-based fluorescent probes for optical measurements of pH. Org Lett. 12 (14), 3219-3221 (2010).
- Lee, J., Choi, K. H., Yoo, K. Innovative SU-8 lithography techniques and their applications. Micromachines. 6 (1), 1-18 (2014).
- Poyton, M. F., Sendecki, A. M., Cong, X., Cremer, P. S. Cu(2+) binds to phosphatidylethanolamine and increases oxidation in lipid membranes. J Am Chem Soc. 138 (5), 1584-1590 (2016).
- Karasek, P., Grym, J., Roth, M., Planeta, J., Foret, F. Etching of glass microchips with supercritical water. Lab Chip. 15 (1), 311-318 (2015).
- Thomas, M. S., et al. Print-and-peel fabrication for microfluidics: what's in it for biomedical applications? Ann Biomed Eng. 38 (1), 21-32 (2010).
- Waheed, S., et al. 3D printed microfluidic devices: enablers and barriers. Lab Chip. 16 (11), 1993-2013 (2016).
- Axmann, M., Schutz, G. J., Huppa, J. B. Single molecule fluorescence microscopy on planar supported bilayers. J Vis Exp. (105), e53158(2015).
- Barenholz, Y., et al. A simple method for the preparation of homogeneous phospholipid vesicles. Biochemistry. 16 (12), 2806-2810 (1977).
- Castellana, E. T., Cremer, P. S. Solid supported lipid bilayers: From biophysical studies to sensor design. Surface Science Reports. 61 (10), 429-444 (2006).
- Hamai, C., Yang, T., Kataoka, S., Cremer, P. S., Musser, S. M. Effect of average phospholipid curvature on supported bilayer formation on glass by vesicle fusion. Biophys J. 90 (4), 1241-1248 (2006).
- Tero, R. Substrate effects on the formation process, structure and physicochemical properties of supported lipid bilayers. Materials. 5 (12), 2658-2680 (2012).
- Ferguson, K. M., Lemmon, M. A., Schlessinger, J., Sigler, P. B. Structure of the high affinity complex of inositol trisphosphate with a phospholipase C pleckstrin homology domain. Cell. 83 (6), 1037-1046 (1995).
- Simonsson, L., Hook, F. Formation and diffusivity characterization of supported lipid bilayers with complex lipid compositions. Langmuir. 28 (28), 10528-10533 (2012).
- Cong, X., Poyton, M. F., Baxter, A. J., Pullanchery, S., Cremer, P. S. Unquenchable surface potential dramatically enhances Cu(2+) binding to phosphatidylserine lipids. J Am Chem Soc. 137 (24), 7785-7792 (2015).
- Robison, A. D., et al. Polyarginine interacts more strongly and cooperatively than polylysine with phospholipid bilayers. J Phys Chem B. 120 (35), 9287-9296 (2016).
- Robison, A. D., Huang, D., Jung, H., Cremer, P. S. Fluorescence modulation sensing of positively and negatively charged proteins on lipid bilayers. Biointerphases. 8 (1), 1(2013).
- Tabaei, S. R., et al. Formation of cholesterol-rich supported membranes using solvent-assisted lipid self-assembly. Langmuir. 30 (44), 13345-13352 (2014).
- Johnson, S. J., et al. Structure of an adsorbed dimyristoylphosphatidylcholine bilayer measured with specular reflection of neutrons. Biophys J. 59 (2), 289-294 (1991).
- Koenig, B. W., et al. Neutron reflectivity and atomic force microscopy studies of a lipid bilayer in water adsorbed to the surface of a silicon single crystal. Langmuir. 12 (5), 1343-1350 (1996).
- Tanaka, M., Sackmann, E. Polymer-supported membranes as models of the cell surface. Nature. 437 (7059), 656-663 (2005).
- Renner, L., et al. Supported lipid bilayers on spacious and pH-responsive polymer cushions with varied hydrophilicity. J Phys Chem B. 112 (20), 6373-6378 (2008).
- Wagner, M. L., Tamm, L. K. Tethered polymer-supported planar lipid bilayers for reconstitution of integral membrane proteins: Silane-polyethyleneglycol-lipid as a cushion and covalent linker. Biophys J. 79 (3), 1400-1414 (2000).
- Pace, H., et al. Preserved transmembrane protein mobility in polymer-supported lipid bilayers derived from cell membranes. Anal Chem. 87 (18), 9194-9203 (2015).
- Braunger, J. A., Kramer, C., Morick, D., Steinem, C. Solid supported membranes doped with PIP2: Influence of ionic strength and pH on bilayer formation and membrane organization. Langmuir. 29 (46), 14204-14213 (2013).
- Paridon, P. A., de Kruijff, B., Ouwerkerk, R., Wirtz, K. W. Polyphosphoinositides undergo charge neutralization in the physiological pH range: A 31P-NMR study. Biochim Biophys Acta. 877 (1), 216-219 (1986).
- Liu, C., Huang, D., Yang, T., Cremer, P. S. Monitoring phosphatidic acid formation in intact phosphatidylcholine bilayers upon phospholipase D catalysis. Anal Chem. 86 (3), 1753-1759 (2014).
- Saad, J. S., et al. Structural basis for targeting HIV-1 Gag proteins to the plasma membrane for virus assembly. Proc Natl Acad Sci U S A. 103 (30), 11364-11369 (2006).
- Hsu, N. Y., et al. Viral reorganization of the secretory pathway generates distinct organelles for RNA replication. Cell. 141 (5), 799-811 (2010).
- Del Campo, C. M., et al. Structural basis for PI(4)P-specific membrane recruitment of the Legionella pneumophila effector DrrA/SidM. Structure. 22 (3), 397-408 (2014).
- Kolli, S., et al. Structure-function analysis of vaccinia virus H7 protein reveals a novel phosphoinositide binding fold essential for poxvirus replication. J Virol. 89 (4), 2209-2219 (2015).
- Cho, N. J., et al. Phosphatidylinositol 4,5-bisphosphate is an HCV NS5A ligand and mediates replication of the viral genome. Gastroenterology. 148 (3), 616-625 (2015).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。