Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
PIP-on-a-chip: une étude sans étiquette des interactions protéines-phosphoinositide
Dans cet article
Résumé
Nous présentons ici une bicouche lipidique supportée dans le contexte d'une plate-forme microfluidique pour étudier les interactions protéines-phosphoinositides en utilisant une méthode sans étiquette basée sur la modulation du pH.
Résumé
De nombreuses protéines cellulaires interagissent avec les surfaces membranaires pour affecter les processus cellulaires essentiels. Ces interactions peuvent être dirigées vers un composant lipidique spécifique dans une membrane, comme dans le cas des phosphoinositides (PIP), pour assurer une localisation et / ou une activation sous-cellulaires spécifiques. Les PIP et les domaines de liaison cellulaire PIP ont été largement étudiés pour mieux comprendre leur rôle dans la physiologie cellulaire. Nous avons appliqué un test de modulation du pH sur les bicouches lipidiques supportées (SLB) comme outil pour étudier les interactions protéines-PIP. Dans ces études, la phosphatidyléthanolamine conjuguée à l' ortho -Sulforhodamine B est utilisée pour détecter les interactions protéine-PIP. Lors de la liaison d'une protéine à une surface de membrane contenant du PIP, le potentiel interfacial est modulé ( c.-à-d. Le changement du pH local), en déplaçant l'état de protonation de la sonde. Une étude de cas de l'utilisation réussie du test de modulation de pH est présentée en utilisant la phospholipase C delta1 PleckstrDans le domaine de l'homologie (PLC-δ1 PH) et de l'interaction phosphatidylinositol 4,5-bisphosphate (PI (4,5) P 2 ) à titre d'exemple. La constante de dissociation apparente ( K d, app ) pour cette interaction était de 0,39 ± 0,05 μM, similaire à K d, valeurs d' application obtenues par d'autres. Comme précédemment observé, le domaine PLC-δ1 PH est PI (4,5) P 2 spécifique, montre une liaison plus faible au phosphatidylinositol 4-phosphate et ne se lie pas aux SLB de phosphatidylcholine pure. Le dosage PIP-on-a-chip est avantageux par rapport aux essais traditionnels de liaison au PIP, y compris, mais sans s'y limiter, le faible volume de l'échantillon et les exigences d'étiquetage du ligand / récepteur, la capacité de tester des interactions membranaires à haute et faible affinité avec les petits et les petits De grandes molécules et un rapport signal / bruit amélioré. En conséquence, l'utilisation de l'approche PIP-on-a-chip facilitera l'élucidation des mécanismes d'une large gamme d'interactions membranaires. En outre, cette méthode pourrait être possibleDans l'identification des agents thérapeutiques qui modulent la capacité des protéines à interagir avec les membranes.
Introduction
De nombreuses interactions et processus biochimiques se déroulent sur des surfaces à membrane bidimensionnellement fluides. Les organites membranaires dans les cellules eucaryotes sont exclusives non seulement dans les procédés biochimiques et leur protéome associé, mais également dans leur composition lipidique. Une classe exceptionnelle de phospholipides est la phosphoinositide (PIP). Bien qu'ils ne représentent que 1% du lipidome cellulaire, ils jouent un rôle crucial dans la transduction du signal, l'autophagie et le trafic de la membrane, entre autres 1 , 2 , 3 , 4 . La phosphorylation dynamique du groupe de tête d'inositol par les PIP kinases cellulaires donne naissance à sept groupes de tête PIP mono-, bis- ou tris-phosphorylés 5 . En outre, les PIP définissent l'identité subcellulaire des membranes et servent de sites d'ancrage membranaire spécialisés pour les protéines / enzymes contenant un ou plusieurs phosphoinosItide-binding domains, par exemple, Pleckstrin Homology (PH), Phox Homology (PX) et epsin N-terminal Homology (ENTH) 6 , 7 . L'un des domaines de liaison au PIP le mieux étudié est le domaine de phospholipase C (PLC) -δ1 PH qui interagit spécifiquement avec le phosphatidylinositol 4,5-bisphosphate (PI (4,5) P 2 ) dans une affinité de gamme micromolaire élevée à haut nanomolaire 8 , 9 , 10 , 11 .
Une variété de méthodes qualitatives et quantitatives in vitro ont été développées et utilisées pour étudier le mécanisme, la thermodynamique et la spécificité de ces interactions. Parmi les tests de liaison au PIP les plus couramment utilisés, on trouve la résonance plasmonique de surface (SPR), la calorimétrie isotherme (ITC), la spectroscopie de résonance magnétique nucléaire (RMN), le test de flottation / sédimentation de liposomes et les transferts de lipides (Fat-blots / PIP-strips)12 , 13 . Bien que ceux-ci soient largement utilisés, ils ont tous de nombreux inconvénients. Par exemple, le SPR, l'ITC et la RMN nécessitent de grandes quantités d'échantillons, d'instruments coûteux et / ou de personnel formé 12 , 13 . Certains formats de dosage tels que des lipides à base d'anticorps utilisent des formes hydrosolubles de PIP et les présentent de manière non physiologique 12 , 14 , 15 , 16 . En outre, les transferts de lipides ne peuvent être quantifiés de manière fiable et ils ont souvent abouti à des observations fausses positives / négatives 12 , 17 , 18 . Pour surmonter ces défis et améliorer l'ensemble d'outils actuel, une nouvelle méthode sans étiquette a été établie sur la base d'une bicouche lipidique supportée (SLB) dans le contexte d'am Icrofluidic platform, qui a été appliquée avec succès à l'étude des interactions protéine-PIP ( figure 1 ) 19 .
La stratégie utilisée pour détecter les interactions protéines-PIP est basée sur la détection de la modulation du pH. Cela implique un colorant sensible au pH qui a ortho -Sulforhodamine B ( o SRB) directement conjugué au groupe 20 de tête de lipide de phosphatidyléthanolamine. La sonde o SRB-POPE (figure 2A) est hautement fluorescent à faible pH et désactivé à un pH élevé avec un pKa d' environ 6,7 à moins de 7,5% en moles de PI (4,5) P2 contenant SLBS (figure 5B). Le domaine de PLC-δ1 PH a été largement utilisé pour valider les méthodologies de liaison aux protéines-PIP en raison de sa spécificité élevée vis-à-vis de PI (4,5) P 2 ( Figure 5A ) 21 , 22 ,"> 23 , 24 , 25. Par conséquent, nous avons estimé que le domaine PLC-δ1 PH peut être utilisé pour tester sa liaison à PI (4,5) P 2 à travers le test PIP-on-a-chip. La construction de domaine PH utilisé dans cette étude a une charge positive nette (pI 8,4) et attire ainsi OH -. ions (Figure 5C) Lors de la liaison de PI (4,5) P2 contenant SLBS, le domaine PH apporte les ions OH - au surface de la membrane, ce qui module le potentiel interfacial et déplace l'état de protonation de o SRB-POPE (Figure 5C) 26. en fonction de la concentration PH de domaine, la fluorescence est éteinte (Figure 6A). Enfin, les données normalisées est S'adapter à une isotherme de liaison pour déterminer l'affinité de l'interaction PH-PI (4,5) P 2 ( Figure 6B , 6C ).
Dans cette étude, un protocole détaillé est fourni pour effectuer la liaison des protéines aux SLB contenant du PIP dans une plate-forme microfluidique. Ce protocole permet au lecteur d'assembler le dispositif microfluidique et la préparation des vésicules à la formation de SLB et à la liaison des protéines. De plus, le mode d'analyse de données pour extraire des informations affinité pour le domaine de PH PI-PLC-δ1 (4,5) P 2 interaction sont fournis.
Protocole
1. Nettoyage des couvertures de verre
- Diluez la solution de nettoyage 7x (voir la table des matériaux ) 7 fois avec de l'eau désionisée dans un plat en verre de borosilicate de 100 mm de profondeur avec un fond plat et chauffez-le jusqu'à 95 ° C sur une plaque chauffante chaude pendant 20 minutes ou jusqu'à ce que la solution trouble devienne claire .
REMARQUE: la solution sera chaude, faites attention pour éviter les blessures corporelles. La solution de nettoyage 7x est un mélange exclusif d'hexasodium [oxido- [oxido (phosphonatooxy) phosphoryl] oxyphosphoryl] phosphate, 2- (2-butoxyéthoxy) éthanol, 1,4-bis (2-éthylhexoxy) -1,4-dioxobutane -2-sulfonate et ajouts non dangereux. - À l'aide de pincettes, organiser les lamelles (40 mm de longueur x 22 mm de largeur x 0,16-0,19 mm de hauteur) sur une crémaillère en aluminium dans des directions alternées. Gardez une tache vide entre chaque lamelle pour éviter qu'elles ne se touchent.
- Enlevez complètement la crémaillère avec les lamelles dans la solution de nettoyage. Gardez les lamelles dansLa solution pendant 1 h.
REMARQUE: À mesure que l'eau s'évapore, continuez à ajouter de l'eau désionisée fraîche pour maintenir inchangée la concentration de la solution. - Retirez les supports de coloration de la solution de nettoyage et lavez les lamelles avec de grandes quantités d'eau désionisée pour enlever le détergent.
- Séchez les lamelles avec de l'azote gazeux (environ 5 min par support de teinture), puis placez les lamelles dans un four pendant 6 h à 550 ° C pour le recuit.
REMARQUE: Il s'agit d'une étape cruciale qui lisse les fonctions grossières sur la surface du verre. - Placez les lamelles dans un récipient en plastique et retirez-les de la poussière.
2. Fabrication de blocs PDMS micropatternés
- Mélanger le prépolymère de polydiméthylsiloxane (PDMS) et l'agent de durcissement à 10: 1 (p / p) dans un grand bateau de pesage en plastique et le dégager sous vide pendant 1 h avec une résistance au vide à 500 Torr ou moins.
REMARQUE: PDMS est un polymère de silicone transparent et inerte. Pour obtenir le bloc PDMS souhaité tHickness (0,5 cm), mélanger 55 g du prépolymère avec 5,5 g de l'agent de durcissement. - Placez le maître de silicium qui contient plusieurs répliques du même micropatteur SU-8 dans un grand bateau de pesée en plastique de diamètre de base de 10 cm et versez le PDMS dégazé. Ensuite, curez-le dans un four sec à 60 ° C pendant la nuit.
REMARQUE: Les micropatrans sont assez grands pour être visibles à l'œil nu. Chaque motif contient 8 microcanaux (100 μm de largeur x 40 μm de hauteur x 1 cm de longueur) avec un intervalle de 40 μm entre ( Figure 1A , 1B ). On a fabriqué un moule en forme de plaquette de silicium constitué d'un photorésist SU-8 cuit au four dur dans une installation de nanofabrication 27 . D'autres approches pour la préparation de maître comprennent la gravure à l'acide fluorhydrique (HF), la gravure à l'eau à haute température, la xurographie et l'impression 3D 28 , 29 , 30 , 31 . CommercLes sources de préparation de silicium peuvent également être utilisées. Les modèles pour le dispositif microfluidique ont été conçus avec un logiciel de dessin (voir tableau de matériaux). Le fichier original contenant la conception du motif est fourni sous forme de fichier supplémentaire "Pattern Design.dwg", qui peut être fourni directement au fabricant. - Doucement, retirez le PDMS du maître de silicium avec les mains. Marquez les limites de chaque micropattern dans des rectangles en utilisant un scalpel chirurgical et une règle. Puis, couper le PDMS en blocs.
- Punch 16 trous (8 entrées et 8 sorties par bloc) aux deux extrémités de chaque microcane avec un poinçon de biopsie (diamètre de trou de 1,0 mm) comme indiqué sur la figure 3B .
- Appliquer du ruban adhésif sur chaque bloc PDMS pour protéger les microprocesseurs des dommages et de la poussière. Rangez-les dans un récipient en plastique.
3. Préparation des petites vésicules unilamellaires (SUV)
REMARQUE: composition bicouche de contrôle négatifEst 99,5% en moles de 1-palmitoyl-2-oléoyl- sn -glycéro-3-phosphocholine (POPC) et 0,5% en moles d' ortho- Sulforhodamine B-1-palmitoyl-2-oléoil- sn -glycéro-3-phosphoéthanolamine ( o SRB- LE PAPE). Composition bicouche de test est de 92,0 mol% POPC, 0,5 mol% o SRB-POPE, et 7,5% en moles soit de la L-α-phosphatidylinositol-4-phosphate (PI4P) ou L-α-phosphatidylinositol-4,5-bisphosphate (PI ( 4,5) P 2 ). Ci - dessous, la procédure de préparation de 92,0% en moles de POPC, 0,5 mol% o SRB-POPE, et 7,5% en mole PI (4,5) P2 VUS contenant. La synthèse de o SRB-POPE utilisée dans cette étude a été précédemment décrite 20 .
- Calculer le volume de POPC, PI (4,5) P 2 et o SRB-POPE qui doit être mélangé, ce qui représente les quantités totales suivantes: 2,22 mg de POPC, 0,26 mg de PI (4,5) P 2 , Et 2,1 x 10 -5 mg de o SRB-POPE.
- Pipettez le volume calculé de POPC, PI (4,5) P 2 , o SRB-POPE dans un seul flacon de scintillation en verre de 20 mL.
REMARQUE: les solutions stock POPC et o SRB-POPE sont livrées dans une solution chloroforme, tandis que le stock PI (4,5) P 2 (et autres phosphoinositides) se trouve dans un mélange de solvants chloroforme: méthanol: eau (20: 9: 1). Pour une précision, mouillez la pointe de la pipette avec du chloroforme avant de l'utiliser. Pour plus de sécurité, cette étape doit avoir lieu dans une hotte chimique. - Sécher le mélange dans un courant d'azote gazeux dans une hotte chimique pendant 10 min ou jusqu'à ce que le solvant s'évapore et un mince film lipidique se forme au fond du flacon.
- Desserrer le mélange sous vide pendant ≥ 3 h à une résistance au vide de 10 mTorr pour éliminer tout solvant organique résiduel.
REMARQUE: Le temps de dessiccation de 3 h est suffisant pour l'échelle de la préparation SUV décrite dans ce protocole, ce qui est suffisant pour 50 expériences. Si une plus grande échelle de préparation SUV est nécessaire, une dessiccation peut être effectuée. - Rehydrate le dr(20 mM de HEPES, 100 mM de NaCl, à pH 7,0) et le placer dans un bain à ultrasons à une fréquence de fonctionnement de 35 kHz pendant 30 min à la RT.
NOTE: Le mélange des quantités de lipides spécifiées à l'étape 3.1 et l'addition de 5 ml de tampon de roulement à l'étape 3.5 donne une suspension de vésicules à 0,5 mg / mL. À ce stade, la suspension de vésicules est un mélange de vésicules unilamellaires et multilamellaires ( figure 2B ). La solution apparaîtra en couleur rose / violet et relativement trouble. - Congeler-décongeler la suspension de vésicule avec de l'azote liquide et un bain d'eau à 40 ° C pour obtenir des vésicules unilamellaires. Répétez le gel-décongélation 10 fois.
REMARQUE: (Facultatif) Pour assurer l'absence de vésicules non-unilamellaires dans la suspension, l'étape de centrifugation peut être appliquée 32 , 33 . - Extruder la suspension de vésicules à travers une membrane de polycarbonate gravée à la piste de 0,10 μm à l'aide d'une extrudeuse lipidique (voirTable des matières) pour enrichir les VUS. Répétez l'extrusion 10 fois.
REMARQUE: Un tube conique de 15 ml peut être utilisé pour collecter les vésicules extrudées. La solution devrait être vide de turbidité à ce stade. Le diamètre du VUS peut être confirmé par diffusion dynamique de la lumière (DLS) ( Figure 2C ). Les VUS sont les mieux adaptés pour former des SLB 34 . - Couvrir le tube conique avec du papier d'aluminium et le conserver à 4 ° C jusqu'à ce qu'il soit prêt à l'emploi.
4. Assemblage du dispositif microfluidique
- Testez les entrées et les sorties du bloc PDMS pour le blocage en jetant de l'eau désionisée à travers les trous à l'aide d'une bouteille de lavage à l'eau. Ensuite, sécher le bloc PDMS avec de l'azote gazeux.
REMARQUE: Cette étape éliminera également toutes les particules de poussière qui auraient pu être piégées à l'intérieur et / ou entre les canaux. Si nécessaire, couvre-lits pré-nettoyés dépoussiérés avec de l'azote gazeux pour enlever les particules de poussière. - Placez le bloc PDMS et le pré-nettoyéLamelle coulissante (voir la section 1) à l'intérieur du système de plasma d'oxygène (voir tableau de matériaux) échantillon pour exposer avec du plasma d'oxygène pendant 45 s avec réglage de puissance à 75 Watts, vitesse d'écoulement d'oxygène à 10 cm 3 / min et résistance au vide à 200 mTorr .
- Placez la surface à motifs du bloc PDMS en contact avec la lamelle immédiatement après le traitement par plasma à l'oxygène. Appuyez doucement pour éliminer les bulles d'air sur les sites de contact.
- Placez l'appareil sur une plaque chauffante à 100 ° C pendant 3 min pour améliorer la liaison.
- Utilisez un chiffon humide sans peluches ( p. Ex . Kimwipe) à 100% d'éthanol pour éliminer les particules de poussière du dessus (PDMS) et du fond (verre) de l'appareil. Ensuite, tapez le périphérique sur une glissière en verre.
REMARQUE: N'utilisez pas une quantité excessive d'éthanol et évitez l'éthanol dans les canaux d'entrée et de sortie. Il est recommandé d'effectuer cette étape pendant que l'appareil est encore chaud pour s'assurer que l'éthanol s'évapore immédiatement. Micre de verreLes diapositives d'oscopie peuvent être stockées dans une solution à 100% d'éthanol pour éliminer la poussière et d'autres contaminants.
5. Création de bavures lipidiques supportées (SLB)
- Transférer 100 μL de VUS contenant PI (4,5) P 2 de l'étape 3.8 dans un tube de microcentrifugeuse de 0,65 ml. Ajuster le pH de la solution à ~ 3,2 en ajoutant 6,4 μl d'acide chlorhydrique 0,2 N.
REMARQUE: Confirmer le pH de la solution avec un pHmètre équipé d'une sonde pH pH (voir tableau de matériaux). - Pipeter 10 μL de la solution de SUV ajustée au pH dans chaque canal à travers l'entrée et appliquer une pression à travers la pipette jusqu'à ce que la solution atteigne la sortie. Détachez la pointe de la pipette et laissez-la attachée à l'appareil.
- Répétez l'étape ci-dessus pour chaque canal, puis incubez l'appareil pendant 10 min à la RT.
REMARQUE: l'injection de vésicules dans les microcanaux doit être effectuée immédiatement après l'assemblage du dispositif. - Couper les ensembles de tuyaux d'entrée et de sortieCh 60 cm (tube d'entrée) et 8 cm (tube de sortie) long, respectivement.
REMARQUE: en coupant le tube en diagonale et en créant un bord tranchant, il est plus facile d'insérer le tube dans les entrées et sorties. Le tube de sortie devrait avoir une forme d'arc. La longueur du tube d'entrée peut varier en fonction de la configuration du microscope. Le diamètre intérieur du tube est de 0,05 cm. - À l'aide de pinces, raccordez le tube de sortie au périphérique, puis tapez l'appareil sur un microscope.
- Immergez une extrémité de la tubulure d'entrée dans 25 ml de tampon de fonctionnement contenu dans un tube conique et tapez-le pour vous assurer que le tube est bien fixé.
- Placer le tube conique sur un sol plus élevé (~ 20 cm) que l'appareil afin de pousser la solution à travers les microcanaux par écoulement par gravité; Une prise de laboratoire peut être utilisée pour y parvenir.
- Pour chaque tube d'entrée, utiliser une seringue pour dessiner 1 mL de tampon courant à partir de l'extrémité libre de la tubulure. Retirez la pointe de la pipette de l'entrée et insérez laExtrémité libre du tube d'entrée dans l'appareil.
REMARQUE: Introduire une baisse de la mémoire tampon en cours d'exécution sur l'entrée pour réduire la probabilité qu'une bulle d'air soit introduite dans le canal pendant cette étape. Une fois que le tube d'entrée est attaché à l'appareil, utilisez un chiffon sans peluches pour éliminer l'excès de tampon. - Répétez l'étape ci-dessus pour connecter toutes les pièces d'entrée du tube à l'appareil.
REMARQUE: Le flux de tampon de fonctionnement à travers les canaux permet d'éliminer les excès de vésicules et d'équilibrer la bicouche dans des conditions expérimentales. - Ouvrez le logiciel de contrôle du microscope (voir la table des matières). Sur le panneau de gauche, cliquez sur l'onglet "Microscope" et choisissez l'objectif "10X".
- Cliquez sur "Live", puis sur les icônes d'image "Alexa 568" sur la barre d'outils. En utilisant les boutons de réglage fin et de course, mettez l'accent sur les microcanaux.
- Numérisez l'appareil pour vérifier la qualité des SLB et des canaux ( Figure 8 ). Alors,Cliquez sur l'icône d'image "Shutter Closed" sur la barre d'outils.
- Cliquez sur l'onglet "Acquisition", et sous "Réglages de base", sélectionnez "Temps d'exposition". Réglez le temps d'exposition sur "200 ms".
- Sur le panneau de gauche, cliquez sur «Acquisition multidimensionnelle» et, dans le menu des filtres, sélectionnez le canal rouge (marqué «Alexa 568»). Ensuite, cliquez sur le menu "time lapse", réglez l'intervalle de temps à 5 min, durée à 30 min, puis cliquez sur "Démarrer".
REMARQUE: en fonction de la durée (30 min) et du débit (~ 1,0 μL / min), 30 μL de tampon de fonctionnement est utilisé pour équilibrer le SLB dans un canal, c'est-à - dire que 240 μL de tampon de fonctionnement sont utilisés pour les 8 canaux. - Sélectionnez l'outil "Cercle" sous l'onglet "Mesurer" et tracez un cercle dans n'importe quel canal. Cliquez avec le bouton droit de la souris pendant que le cercle est sélectionné et choisissez "Propriétés". Sous l'onglet "Profil", cochez "tout T" pour afficher leIntensité de fluorescence en fonction du temps.
- Assurez-vous que cette courbe atteint un plateau, ce qui indique l'équilibre, avant de passer à l'étape suivante.
- Abaissez la solution tampon à un sol égal comme dispositif pour arrêter le flux.
- Enregistrez le fichier de temporisation.
6. Test PLC-δ1 interaction PH de domaine avec PI (4,5) P 2 -Contenant SLBS
- Préparer les dilutions du domaine PH à l'aide du tampon courant en tant que diluant.
REMARQUE: Étant donné qu'il existe huit canaux dans l'appareil, utilisez au moins deux d'entre eux pour les contrôles vierges. Choisissez les extrémités de chaque côté et utilisez les canaux restants pour les dilutions de protéines. Les concentrations de domaine PH suivantes ont été testées: 0,10, 0,25, 0,50, 1,00 et 2,50 μM. Environ 200 μL de chaque dilution était suffisant pour atteindre l'équilibre dans les 30 minutes. - Un à la fois, détachez chaque tube de sortie et appliquez 200 μL de chaque dilution de protéines dansLe canal de sortie en utilisant une pipette. N'appliquez aucune pression, laissez la gravité faire le travail. Détachez la pointe de la pipette et laissez-la attachée au dispositif microfluidique.
- Répétez l'étape ci-dessus pour chaque canal et assurez-vous que les bulles d'air ne sont pas introduites dans les canaux pendant ce processus.
- Abaissez le tube d'entrée à un sol sous le dispositif microfluidique pour commencer à écouler la protéine à travers les microcanaux. Tapez l'extrémité libre du tube dans un récipient à déchets. Réglez les dilutions de protéines pendant 30 min.
REMARQUE: cela inversera le flux et permettra l'introduction de la protéine dans les canaux. Le temps d'équilibrage et le volume des dilutions de protéines nécessaires dépendent de l'affinité de l'interaction. - Sur le panneau gauche du logiciel, sous l'onglet "Time Lapse", cliquez sur "Démarrer" pour recommencer l'imagerie.
- Répétez l'étape 5.15 pour vous assurer que l'équilibre est atteint. Lorsque l'expérience est terminée, enregistrez le laps de tempsfichier.
7. Évaluation de la fluidité des membranes
REMARQUE: les expériences de récupération de fluorescence Après Photobleaching (FRAP) doivent être effectuées avec chaque nouveau lot de SUV et lamelles de verre nettoyées pour s'assurer que les SLB sont fluides.
- Suivez la procédure de nettoyage des lamelles de verre (1.1-1.6), la fabrication de blocs PDMS micropatternés (2.1-2.5), la préparation de SUV (3.1-3.8), l'assemblage du périphérique microfluidique (4.1-4.5) et la formation de SLB (5.1-5.17) comme précédemment Décrit.
- Concentrez-vous sur le microscope sur la bicouche, réglez le temps d'exposition à 200 ms, et le passage à 1.
- Photo-blanchir un point circulaire à l'aide d'un rayon laser de 532 nm, 2 mW (rayon 13 μM). Immédiatement après le photoblanchage, commencez à capturer une série d'images toutes les 3 s pour les 45 premières s, suivies d'un intervalle de 30 s pour le reste du temps (durée totale de 15 minutes). Une fois l'acquisition de données terminée, enregistrez le fichier de temporisation.
- Sélectionnez le blanchi aRea en utilisant l'outil de dessin en cercle pour extraire les valeurs d'intensité de fluorescence en fonction du temps. En outre, sélectionnez deux autres zones à proximité, l'une correspondant à une région non blanchie et une correspondant à une région sans SLB.
- Calculer la récupération de fluorescence ( y ) en utilisant l'équation suivante:
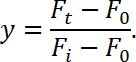
NOTE: Ici F t représente l'intensité de la région blanchie en fonction du temps, F i représente l'intensité de fluorescence avant le blanchiment (utiliser "1" comme valeur normalisée dans ce cas); F 0 est l'intensité du fond. - Reconstituez la récupération de fluorescence (axe des y) en fonction du temps (axe des abscisses) pour générer une courbe FRAP. Ensuite, ajustez les données à une seule fonction exponentielle comme suit,
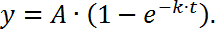
REMARQUE: Ici A représente la fraction mobile et k est la constante cinétique pour la fraction mobile, qui est utilisée pour calculer le demi-temps de récupération ( t 1/2 ):
- Utilisez le t 1/2 pour calculer la constante de diffusion ( D ):

REMARQUE: ici R représente le rayon du rayon laser (13 μm). La constante de diffusion pour une bicouche fluide doit être ≥1,0 μm 2 / s.
8. Traitement des données
REMARQUE: La routine de l'analyse des données dépend du microscope, du logiciel de traitement d'image et du logiciel d'ajustement des courbes utilisées.
- Ouvrez les fichiers de temporisation antérieurs (à partir de l'étape 5.17) et après (à partir de l'étape 6.6) en titrant le domaine PH. Passez à la dernière image de chaque fichier de temporisation et faites un balayage de ligne sur tous les microcanaux pour obtenir le fluorescEn fonction de la distance en pixels ( figure 6A ). Transférez les données vers une feuille de calcul.
- Pour chaque microcanal (avant et après l'ajout de domaine PH), échantillonner des données dans le canal et de chaque côté du canal représentant la ligne de base ( Figure 7A ).
REMARQUE: L'intensité de fluorescence du canal vierge devrait rester inchangée. Tous les autres canaux sont normalisés sur le canal vierge, il est donc essentiel d'avoir deux canaux vierges et de s'assurer que leurs intensités de fluorescence restent cohérentes les unes avec les autres. - Appliquer la formule ci-dessous pour normaliser les données sur le canal vierge; Un calcul d'échantillon est fourni à la figure 7B .

REMARQUE: Ici, la protéine ΔF représente l'intensité de fluorescence soustraite par le fond du canal protéique, Δ; F blanc représente l'intensité de fluorescence soustraite par le fond du canal vierge, avant et après se référer aux étapes de titrage avant et après, et α représente le facteur de correction qui est le rapport des intensités de fluorescence du canal protéique et du canal vierge ([ F protéine / F blanc ] pré ) à une étape de titrage pré-protéine. Comme l'intensité de la fluorescence diminue en fonction de la concentration du domaine PH, les données normalisées seront négatives. - À l'aide d'un logiciel de montage en courbe (voir Tableau des matériaux ), tracer la fluorescence normalisée en fonction de la concentration du domaine PH et s'adapter à une isotherme de Langmuir ( Figure 6C ):
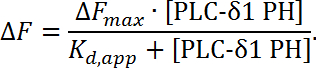
NOTE: Ici ΔF représente la variation de l'intensité de fluorescence par rapport au changement maximal de fluoIntensité de la fluorescence lorsqu'une concentration de saturation de la protéine est présente ( ΔF max ), alors que l'application K d, représente la constante de dissociation apparente, qui est égale à la concentration en protéine en vrac ( [PLC-δ1 PH] ) à laquelle 50% de couverture ( Complexe lié à la membrane) est atteint. Il s'agit d'une mesure de liaison basée sur l'équilibre, de sorte que les données normalisées sont adaptées à une simple isotherme de Langmuir pour extraire un paramètre expérimental apparent K d, application . Sur la base du logiciel de montage , l'application K d, application pour PLC-δ1 PH-PI (4,5) P 2 est de 0,39 ± 0,05 μM ( Figure 6B ).
Résultats
Nous avons utilisé le test de modulation du pH pour étudier l'interaction PLC-δ1 PH-PI (4,5) P 2 dans un micro-dispositif PIP-on-a-chip ( Figure 1 ). Grâce à un protocole détaillé, nous avons démontré comment préparer et assembler les composants des dispositifs microfluidiques, fabriquer de petites vésicules unilamellaires (VUS) ( Figure 2 ), former des SLB dans un dispositif ( Figu...
Discussion
Chaque variante PIP, bien que faiblement concentrée, est présente sur la surface cytosolique d'organites spécifiques où elle contribue à l'établissement d'une composition physique unique et de la spécificité fonctionnelle de la membrane organellaire 1 . L'une des utilisations les plus importantes des PIP est une plate-forme d'amarrage spécifique pour la multitude de protéines nécessitant une localisation et / ou une activation sous-cellulaire spécifiques
Déclarations de divulgation
Les auteurs n'ont rien à dévoiler.
Remerciements
DS et la CCE ont été soutenus, en partie, par la subvention AI053531 (NIAID, NIH); SS et PSC ont été soutenus par la subvention N00014-14-1-0792 (ONR).
matériels
| Name | Company | Catalog Number | Comments |
| Coverslip | |||
| Glass Coverslips: Rectangles | Fisher Scientific | 12-544B | 22 x 40 x 0.16 - 0.19 mm, No. 1 1/2; Borosilicate Glass |
| 7X Cleaning Solution | MP Biomedicals | 976670 | Detergent |
| PYREX Crystallizing Dish | Corning | 3140-190 | Borosilicate glass dish with a flat bottom; Diameter x Height (190 x 100 mm); Distributor: VWR (89090-700) |
| Sentry Xpress 2.0 | Paragon Industries | SC-2 | Kiln |
| Name | Company | Catalog Number | Comments |
| PDMS | |||
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 4019862 | Polydimethylsiloxane (PDMS); Distributor: Ellsworth Adhesives |
| PYREX Desiccator | VWR | 89134-402 | Vacuum Rated |
| Biopsy punch | Harris | 15110-10 | Harris Uni-Core; 1.0 mm diameter; Miltex Biopsy Punch with Plunger (Cat. No. 15110-10) can be used as an alternative |
| Name | Company | Catalog Number | Comments |
| Device | |||
| Plasma Cleaning System | PlasmaEtch | PE25-JW | 2-stage Direct Drive Oil Vacuum Pump, O2 service (Krytox Charged) |
| Digital Hot Plate | Benchmark | H3760-H | Purchased through Denville Scientific (Cat. No. 1005640) |
| Frosted Micro Slides | VWR | 48312-003 | Frosted, Selected, and Precleaned; Made of Swiss Glass; Thickness: 1 mm; Dimensions: 75 x 25 mm; GR 144 |
| Name | Company | Catalog Number | Comments |
| Mold | |||
| AutoCAD | Autodesk | v.2016 | Drafting software for the photomask design |
| Photomask | CAD/Art Services | N/A | Design with black background and clear features was printed at 20k dpi resolution on a transparent mask (5 x 7 in) by CAD/Art Services |
| Silicone Wafers | University Wafer | 1575 | Prime Grade, Single Side Polished; 100 mm (4 inch) Diameter; 525 um Thickness |
| SU-8 50 | MicroChem Corp. | N/A | Negative Tone Photoresist; Penn State Nanofabrication Facility Property |
| SU-8 Developer | MicroChem Corp. | N/A | Penn State Nanofabrication Facility Property |
| Name | Company | Catalog Number | Comments |
| SUV | |||
| 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine | Avanti Polar Lipids | 850457C | POPC |
| L-α-phosphatidylinositol-4-phosphate | Avanti Polar Lipids | 840045X | PI4P |
| L-α-phosphatidylinositol-4,5-bisphosphate | Avanti Polar Lipids | 840046X | PI(4,5)P2 |
| 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphoethanolamine | Avanti Polar Lipids | 850757C | POPE; Required for the synthesis of oSRB-POPE |
| Lissamine Rhodamine B Sulfonyl Chloride (mixed isomers) | ThermoFisher Scientific | L-20 | Required for the synthesis of oSRB-POPE |
| pH Sensitive Fluorescent Lipid Probe (oSRB-POPE) | In-house | N/A | In-house Synthesis (Huang D. et al. 2013) |
| Glass Scintillation Vial | VWR | 66022-065 | 20 mL volume capacity |
| Aquasonic 250D | VWR | N/A | Ultrasonic Water Bath |
| Nuclepore Track-Etched Membranes | Whatman | 110605 | Polycarbonate Membrane; Diameter: 25 mm; Pore Size: 0.1 um; Distributor: Sigma-Aldrich |
| Chloroform | VWR | CX1054-6 | HPLC grade |
| LIPEX Extruder | Transferra Nanosciences | T.001 | LIPEX 10 mL Thermobarrel Extruder |
| Viscotek 802 DLS | Malvern Instruments | N/A | Dynamic Light Scattering; Penn State X-Ray Crystallography Facility Property |
| Name | Company | Catalog Number | Comments |
| Data Analysis | |||
| GraphPad Prism | GraphPad Software | v.6 | Curve-fitting software for data analysis |
| Name | Company | Catalog Number | Comments |
| Microscope | |||
| Axiovert 200M Epifluorescence Microscope | Carl Zeiss Microscopy | N/A | Microscope |
| AxioCam MRm Camera | Carl Zeiss Microscopy | N/A | Camera |
| X-Cite 120 | Excelitas Technologies | N/A | Light Source |
| Alexa 568 Filter Set | Carl Zeiss Microscopy | N/A | Ex/Em 576/603 nm |
| AxioVision LE64 v.4.9.1.0 Software | Carl Zeiss Microscopy | N/A | Image Processing Software |
| Name | Company | Catalog Number | Comments |
| Other | |||
| Tips | VWR | 10034-132 | 200 uL pipette tips; Thin and smooth tip for applying the protein solution into the microfluidic channel |
| Tips | VWR | 53509-070 | 10 uL pipette tips; Thin and smooth tip for applying the vesicle solution into the microfluidic channel |
| Orion Star A321 pH meter | Thermo Scientific | STARA3210 | pH meter |
| Orion micro pH probe | Thermo Scientific | 8220BNWP | micro pH probe |
| N-(2-Hydroxyethyl)-Piperazine-N'-(2-Ethanesulfonic Acid) | VWR | VWRB30487 | HEPES, Free Acid |
| Sodium Chloride | VWR | BDH8014-2.5KGR | NaCl |
| Tubing | Allied Wire & Cable | TFT-200-24 N | Internal Diameter: 0.020-0.026 inches (0.051-0.066 cm); Wall Thickness: 0.010 inches (0.025 cm); Flexible Polytetrafluoroethylene Thin-Wall Tubing; Natural Color |
| Nitrogen Gas - Industrial | Praxair | N/A | Local Provider |
| Oxygen Gas - Industrial | Praxair | N/A | Local Provider |
| Liquid Nitrogen | Praxair | N/A | Local Provider |
Références
- Di Paolo, G., De Camilli, P. Phosphoinositides in cell regulation and membrane dynamics. Nature. 443 (7112), 651-657 (2006).
- Shewan, A., Eastburn, D. J., Mostov, K. Phosphoinositides in cell architecture. Cold Spring Harb Perspect Biol. 3 (8), a004796 (2011).
- Picas, L., Gaits-Iacovoni, F., Goud, B. The emerging role of phosphoinositide clustering in intracellular trafficking and signal transduction. F1000Res. 5, (2016).
- Lystad, A. H., Simonsen, A. Phosphoinositide-binding proteins in autophagy. FEBS Lett. 590 (15), 2454-2468 (2016).
- Balla, T. Phosphoinositides: Tiny lipids with giant impact on cell regulation. Physiol Rev. 93, 1019-1137 (2013).
- Lemmon, M. A. Membrane recognition by phospholipid-binding domains. Nat Rev Mol Cell Biol. 9 (2), 99-111 (2008).
- Kutateladze, T. G. Translation of the phosphoinositide code by PI effectors. Nat Chem Biol. 6 (7), 507-513 (2010).
- Harlan, J. E., Hajduk, P. J., Yoon, H. S., Fesik, S. W. Pleckstrin homology domains bind to phosphatidylinositol-4,5-bisphosphate. Nature. 371 (6493), 168-170 (1994).
- Garcia, P., et al. The pleckstrin homology domain of phospholipase C-delta 1 binds with high affinity to phosphatidylinositol 4,5-bisphosphate in bilayer membranes. Biochemistry. 34 (49), 16228-16234 (1995).
- Lemmon, M. A., Ferguson, K. M., O'Brien, R., Sigler, P. B., Schlessinger, J. Specific and high-affinity binding of inositol phosphates to an isolated pleckstrin homology domain. Proc Natl Acad Sci U S A. 92 (23), 10472-10476 (1995).
- Flesch, F. M., Yu, J. W., Lemmon, M. A., Burger, K. N. Membrane activity of the phospholipase C-delta1 pleckstrin homology (PH) domain. Biochem J. 389, 435-441 (2005).
- Narayan, K., Lemmon, M. A. Determining selectivity of phosphoinositide-binding domains. Methods. 39 (2), 122-133 (2006).
- Scott, J. L., Musselman, C. A., Adu-Gyamfi, E., Kutateladze, T. G., Stahelin, R. V. Emerging methodologies to investigate lipid-protein interactions. Integr Biol (Camb). 4 (3), 247-258 (2012).
- Dowler, S., Currie, R. A., Downes, C. P., Alessi, D. R. DAPP1: A dual adaptor for phosphotyrosine and 3-phosphoinositides. Biochem J. 342, 7-12 (1999).
- He, J., et al. Molecular basis of phosphatidylinositol 4-phosphate and ARF1 GTPase recognition by the FAPP1 pleckstrin homology (PH) domain. J Biol Chem. 286 (21), 18650-18657 (2011).
- Ceccarelli, D. F., et al. Non-canonical interaction of phosphoinositides with pleckstrin homology domains of Tiam1 and ArhGAP9. J Biol Chem. 282 (18), 13864-13874 (2007).
- Huang, S., Gao, L., Blanchoin, L., Staiger, C. J. Heterodimeric capping protein from Arabidopsis is regulated by phosphatidic acid. Mol Biol Cell. 17 (4), 1946-1958 (2006).
- Yu, J. W., et al. Genome-eide analysis of membrane targeting by S. cerevisiae pleckstrin homology domains. Mol Cell. 13 (5), 677-688 (2004).
- Jung, H., Robison, A. D., Cremer, P. S. Detecting protein-ligand binding on supported bilayers by local pH modulation. J Am Chem Soc. 131 (3), 1006-1014 (2009).
- Huang, D., Zhao, T., Xu, W., Yang, T., Cremer, P. S. Sensing small molecule interactions with lipid membranes by local pH modulation. Anal Chem. 85 (21), 10240-10248 (2013).
- Saxena, A., et al. Phosphoinositide binding by the pleckstrin homology domains of Ipl and Tih1. J Biol Chem. 277 (51), 49935-49944 (2002).
- Knödler, A., Mayinger, P. Analysis of phosphoinositide-binding proteins using liposomes as an affinity matrix. Biotechniques. 38 (6), 858-862 (2005).
- Baumann, M. K., Swann, M. J., Textor, M., Reimhult, E. Pleckstrin homology-phospholipase C-delta1 interaction with phosphatidylinositol 4,5-bisphosphate containing supported lipid bilayers monitored in situ with dual polarization interferometry. Anal Chem. 83 (16), 6267-6274 (2011).
- Saliba, A. E., et al. A quantitative liposome microarray to systematically characterize protein-lipid interactions. Nat Methods. 11 (1), 47-50 (2014).
- Arauz, E., Aggarwal, V., Jain, A., Ha, T., Chen, J. Single-molecule analysis of lipid-protein interactions in crude cell lysates. Anal Chem. 88 (8), 4269-4276 (2016).
- Best, Q. A., Xu, R., McCarroll, M. E., Wang, L., Dyer, D. J. Design and investigation of a series of rhodamine-based fluorescent probes for optical measurements of pH. Org Lett. 12 (14), 3219-3221 (2010).
- Lee, J., Choi, K. H., Yoo, K. Innovative SU-8 lithography techniques and their applications. Micromachines. 6 (1), 1-18 (2014).
- Poyton, M. F., Sendecki, A. M., Cong, X., Cremer, P. S. Cu(2+) binds to phosphatidylethanolamine and increases oxidation in lipid membranes. J Am Chem Soc. 138 (5), 1584-1590 (2016).
- Karasek, P., Grym, J., Roth, M., Planeta, J., Foret, F. Etching of glass microchips with supercritical water. Lab Chip. 15 (1), 311-318 (2015).
- Thomas, M. S., et al. Print-and-peel fabrication for microfluidics: what's in it for biomedical applications?. Ann Biomed Eng. 38 (1), 21-32 (2010).
- Waheed, S., et al. 3D printed microfluidic devices: enablers and barriers. Lab Chip. 16 (11), 1993-2013 (2016).
- Axmann, M., Schutz, G. J., Huppa, J. B. Single molecule fluorescence microscopy on planar supported bilayers. J Vis Exp. (105), e53158 (2015).
- Barenholz, Y., et al. A simple method for the preparation of homogeneous phospholipid vesicles. Biochemistry. 16 (12), 2806-2810 (1977).
- Castellana, E. T., Cremer, P. S. Solid supported lipid bilayers: From biophysical studies to sensor design. Surface Science Reports. 61 (10), 429-444 (2006).
- Hamai, C., Yang, T., Kataoka, S., Cremer, P. S., Musser, S. M. Effect of average phospholipid curvature on supported bilayer formation on glass by vesicle fusion. Biophys J. 90 (4), 1241-1248 (2006).
- Tero, R. Substrate effects on the formation process, structure and physicochemical properties of supported lipid bilayers. Materials. 5 (12), 2658-2680 (2012).
- Ferguson, K. M., Lemmon, M. A., Schlessinger, J., Sigler, P. B. Structure of the high affinity complex of inositol trisphosphate with a phospholipase C pleckstrin homology domain. Cell. 83 (6), 1037-1046 (1995).
- Simonsson, L., Hook, F. Formation and diffusivity characterization of supported lipid bilayers with complex lipid compositions. Langmuir. 28 (28), 10528-10533 (2012).
- Cong, X., Poyton, M. F., Baxter, A. J., Pullanchery, S., Cremer, P. S. Unquenchable surface potential dramatically enhances Cu(2+) binding to phosphatidylserine lipids. J Am Chem Soc. 137 (24), 7785-7792 (2015).
- Robison, A. D., et al. Polyarginine interacts more strongly and cooperatively than polylysine with phospholipid bilayers. J Phys Chem B. 120 (35), 9287-9296 (2016).
- Robison, A. D., Huang, D., Jung, H., Cremer, P. S. Fluorescence modulation sensing of positively and negatively charged proteins on lipid bilayers. Biointerphases. 8 (1), 1 (2013).
- Tabaei, S. R., et al. Formation of cholesterol-rich supported membranes using solvent-assisted lipid self-assembly. Langmuir. 30 (44), 13345-13352 (2014).
- Johnson, S. J., et al. Structure of an adsorbed dimyristoylphosphatidylcholine bilayer measured with specular reflection of neutrons. Biophys J. 59 (2), 289-294 (1991).
- Koenig, B. W., et al. Neutron reflectivity and atomic force microscopy studies of a lipid bilayer in water adsorbed to the surface of a silicon single crystal. Langmuir. 12 (5), 1343-1350 (1996).
- Tanaka, M., Sackmann, E. Polymer-supported membranes as models of the cell surface. Nature. 437 (7059), 656-663 (2005).
- Renner, L., et al. Supported lipid bilayers on spacious and pH-responsive polymer cushions with varied hydrophilicity. J Phys Chem B. 112 (20), 6373-6378 (2008).
- Wagner, M. L., Tamm, L. K. Tethered polymer-supported planar lipid bilayers for reconstitution of integral membrane proteins: Silane-polyethyleneglycol-lipid as a cushion and covalent linker. Biophys J. 79 (3), 1400-1414 (2000).
- Pace, H., et al. Preserved transmembrane protein mobility in polymer-supported lipid bilayers derived from cell membranes. Anal Chem. 87 (18), 9194-9203 (2015).
- Braunger, J. A., Kramer, C., Morick, D., Steinem, C. Solid supported membranes doped with PIP2: Influence of ionic strength and pH on bilayer formation and membrane organization. Langmuir. 29 (46), 14204-14213 (2013).
- Paridon, P. A., de Kruijff, B., Ouwerkerk, R., Wirtz, K. W. Polyphosphoinositides undergo charge neutralization in the physiological pH range: A 31P-NMR study. Biochim Biophys Acta. 877 (1), 216-219 (1986).
- Liu, C., Huang, D., Yang, T., Cremer, P. S. Monitoring phosphatidic acid formation in intact phosphatidylcholine bilayers upon phospholipase D catalysis. Anal Chem. 86 (3), 1753-1759 (2014).
- Saad, J. S., et al. Structural basis for targeting HIV-1 Gag proteins to the plasma membrane for virus assembly. Proc Natl Acad Sci U S A. 103 (30), 11364-11369 (2006).
- Hsu, N. Y., et al. Viral reorganization of the secretory pathway generates distinct organelles for RNA replication. Cell. 141 (5), 799-811 (2010).
- Del Campo, C. M., et al. Structural basis for PI(4)P-specific membrane recruitment of the Legionella pneumophila effector DrrA/SidM. Structure. 22 (3), 397-408 (2014).
- Kolli, S., et al. Structure-function analysis of vaccinia virus H7 protein reveals a novel phosphoinositide binding fold essential for poxvirus replication. J Virol. 89 (4), 2209-2219 (2015).
- Cho, N. J., et al. Phosphatidylinositol 4,5-bisphosphate is an HCV NS5A ligand and mediates replication of the viral genome. Gastroenterology. 148 (3), 616-625 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon