Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
PIP-on-a-chip: безвредное исследование взаимодействия протеин-фосфоинозитид
В этой статье
Резюме
Здесь мы представляем поддерживаемый липидный бислой в контексте микрофлюидной платформы для изучения взаимодействия белок-фосфоинозитид с использованием метода без меток, основанного на модуляции рН.
Аннотация
Многочисленные клеточные белки взаимодействуют с поверхностями мембран, чтобы влиять на существенные клеточные процессы. Эти взаимодействия могут быть направлены на конкретный липидный компонент внутри мембраны, как в случае фосфоинозитидов (ПГИ), для обеспечения специфической субклеточной локализации и / или активации. PIPs и клеточные PIP-связывающие домены были широко изучены, чтобы лучше понять их роль в клеточной физиологии. Мы применили анализ модуляции рН на поддерживаемых липидных бислоях (SLB) в качестве инструмента для изучения взаимодействия белка с PIP. В этих исследованиях для определения белково-PIP-взаимодействий используется рН-чувствительный орто- сульфоподадим B-конъюгированный фосфатидилэтаноламин. При связывании белка с поверхностью, содержащей PIP-мембрану, межфазный потенциал модулируется ( т. Е. Изменяется локальный рН), изменяя состояние протонирования зонда. Пример успешного использования анализа модуляции рН представлен с использованием фосфолипазы C delta1 PleckstrВ гомологии (PLC-δ1 PH) и фосфатидилинозитол 4,5-бисфосфат (PI (4,5) P 2 ). Очевидная константа диссоциации ( K d, app ) для этого взаимодействия составляла 0,39 ± 0,05 мкМ, аналогично K d, значениям приложения, полученным другими. Как было отмечено ранее, ПЛК-δ1 домен РНА PI (4,5) P 2 специфический, показывает более слабое не связывание по отношению к фосфатидилинозитолам 4-фосфата, а не связывание с чистым фосфатидилхолином SLBS. Анализ PIP-на-чипе является преимуществом по сравнению с традиционными анализами связывания PIP, включая, но не ограничиваясь ими, низкий объем образца и отсутствие требований к маркировке лигандов / рецепторов, способность тестировать взаимодействия с высокой и низкой аффинностью мембран как с малыми, так и с малыми Больших молекул и улучшенного соотношения сигнал / шум. Соответственно, использование подхода PIP-на-чипе облегчит выяснение механизмов широкого спектра мембранных взаимодействий. Кроме того, этот метод потенциально может бытьSed при идентификации терапевтических средств, которые модулируют способность белка взаимодействовать с мембранами.
Введение
Мириады взаимодействий и биохимические процессы протекают на поверхности двумерных жидкостных мембран. Мембранные органеллы в эукариотических клетках уникальны не только в биохимических процессах и их ассоциированном протеоме, но также и в их составе липидов. Одним исключительным классом фосфолипидов является фосфоинозитиды (ПГИ). Несмотря на то, что они составляют лишь 1% клеточного липидома, они играют решающую роль в передаче сигналов, аутофагии и распространении мембран, среди прочих 1 , 2 , 3 , 4 . Динамическое фосфорилирование головной группы инозитола клеточными киназами PIP приводит к появлению семи головных групп PIP, моно-, бис- или трисфосфорилированных 5 . Кроме того, PIP определяют субклеточную идентичность мембран и служат специализированными местами для стыковки мембран для белков / ферментов, содержащих один или несколько фосфоиновНапример, гомологию Плекстрина (PH), гомологию Фокса (PX) и эпсиновую N-терминальную гомологию (ENTH) 6 , 7 . Одним из наиболее изученных PIP-связывающих доменов является домен фосфолипазы C (PLC) -δ1 PH, который специфически взаимодействует с 4,5-бисфосфатом фосфатидилинозитола (PI (4,5) P 2 ) в пределах сродства к высокому наномолярно-низкому микромолярному диапазону 8 , 9 , 10 , 11 .
Разработаны различные качественные и количественные методы in vitro и используются для изучения механизма, термодинамики и специфичности этих взаимодействий. Среди наиболее часто используемых анализов на PIP-связывание - поверхностный плазмонный резонанс (SPR), изотермическая калориметрия (ITC), спектроскопия ядерного магнитного резонанса (ЯМР), липосомная флотация / седиментационный анализ и липид-блоты (Fat-blots / PIP-strip)12 , 13 . Несмотря на то, что они широко используются, все они имеют множество недостатков. Например, SPR, ITC и NMR требуют больших объемов выборки, дорогостоящих приборов и / или обученного персонала 12 , 13 . В некоторых форматах анализа, таких как липид-блоты на основе антител, используются водорастворимые формы ПГИ и представлены их нефизиологическим образом 12 , 14 , 15 , 16 . Кроме того, липидные пятна не могут быть надежно оценены количественно, и они часто приводили к ложным положительным / отрицательным наблюдениям 12 , 17 , 18 . Чтобы преодолеть эти проблемы и улучшить текущий набор инструментов, был создан новый метод без этикетки на основе поддерживаемого липидного бислоя (SLB) в контексте am icrofluidic платформы, который был успешно применен к изучению белок-ПУМ взаимодействий (Рисунок 1) 19.
Стратегия, используемая для обнаружения взаимодействий белок-PIP, основана на измерении модуляции pH. Это включает pH-чувствительный краситель, который имеет орто- сульфамодинамик B ( o SRB), непосредственно конъюгированный с группой 20 липидов липидов фосфатидилэтаноламина. Зонд o SRB-POPE ( рис. 2A ) сильно флуоресцентен при низком значении pH и гасит при высоком рН pKa около 6,7 в пределах 7,5 мол.% PI (4,5) P 2 -содержащих SLB ( рис. 5B ). PLC-δ1 домен PH широко используется для проверки методологий связывания белка с PIP из-за его высокой специфичности к PI (4,5) P 2 ( фиг.5A ) 21 , 22 ,"> 23 , 24 , 25. Мы полагали, что домен PLC-δ1 PH может быть использован для проверки его связывания с PI (4,5) P 2 с помощью анализа PIP-на-чипе. используемый в данном исследовании имеет суммарный положительный заряд (РI 8.4), и , следовательно , привлекает ОН -. ионы (рис 5с) После связывания с PI (4,5) P 2 отработанный SLBS, домен PH приносит ОН - ионов в Мембрана, которая, в свою очередь, модулирует межфазный потенциал и сдвигает состояние протонирования o SRB-POPE ( рис. 5C ) 26. В зависимости от концентрации домена PH флуоресценция гасится ( рис. 6A ). Наконец, нормированные данные Подходит для изотермы связывания для определения аффинности взаимодействия PH-домена-PI (4,5) P 2 ( рис. 6B , 6C ).
В этом исследовании представлен подробный протокол для связывания белка с PIP-содержащими SLB в микрофлюидной платформе. Этот протокол позволяет читателю собирать микрожидкостное устройство и препарат везикул для образования SLB и связывания с белком. Кроме того, предоставляются направления для анализа данных для извлечения информации о сродстве для взаимодействия PLC-δ1 PH-PI (4,5) P 2 .
Access restricted. Please log in or start a trial to view this content.
протокол
1. Очистка стеклянных крышек
- Разбавьте 7-кратный очищающий раствор (см. Таблицу материалов ) в 7 раз с деионизированной водой в чашке из боросиликатного стекла толщиной 100 мм с плоским дном и нагрейте его до 95 ° C на ровной плите в течение 20 минут или до тех пор, пока не станет ясно, ,
ПРИМЕЧАНИЕ. Решение будет горячим, соблюдайте осторожность, чтобы избежать телесных повреждений. 7x Чистящий раствор представляет собой запатентованную смесь гексанатрия [оксидо [оксидо (фосфонатоокси) фосфорил] оксифосфорил] фосфата, 2- (2-бутоксиэтокси) этанола, 1,4-бис (2-этилгексокси) -1,4-диоксобутана натрия -2-сульфонатные и неопасные добавки. - Используя пинцет, расположите покровные стекла (длина 40 мм х 22 мм шириной х 0,16-0,19 мм) на алюминиевой окрасочной стойке в чередующихся направлениях. Держите пустое пятно между каждым покровным слоем, чтобы они не касались друг друга.
- Полностью погрузите окрашивающую стойку с помощью покровных в очищающий раствор. Держите покровные вРаствор в течение 1 часа.
ПРИМЕЧАНИЕ. Когда вода испаряется, продолжайте добавлять свежую деионизированную воду, чтобы поддерживать концентрацию раствора без изменений. - Выньте окрашивающие стеллажи из очищающего раствора и промойте покровные стекла обильным количеством деионизированной воды, чтобы удалить моющее средство.
- Высушите покровные стекла газообразным азотом (приблизительно 5 минут на стойку для окрашивания), а затем поместите покровные стекла в печь на 6 часов при 550 ° C для отжига.
ПРИМЕЧАНИЕ. Это важный шаг, который сглаживает грубые свойства на поверхности стекла. - Поместите покровные стекла в пластиковый контейнер и держите их подальше от пыли.
2. Изготовление блоков микрокомпилированных PDMS
- Смешать полидиметилсилоксановый (PDMS) форполимер и отвердитель при соотношении 10: 1 (мас. / Мас.) На большой пластиковой взвешенной лодке и дегазировать в вакууме в течение 1 часа с силой вакуума при 500 Торр или менее.
ПРИМЕЧАНИЕ. PDMS представляет собой прозрачный и инертный силиконовый полимер. Чтобы получить желаемый блок PDMS t(0,5 см), смешайте 55 г форполимера с 5,5 г отвердителя. - Поместите кремниевый мастер, который содержит несколько повторов одного и того же микротрубочки SU-8 в большой пластиковой весовой лодке диаметром 10 см и вылейте дегазированную PDMS. Затем вылечите его в сухом духовке при 60 ° C в течение ночи.
ПРИМЕЧАНИЕ. Микрокассеты достаточно большие, чтобы их можно было видеть невооруженным глазом. Каждый образец содержит 8 микроканалов (ширина 100 мкм x 40 мкм, высота х 1 см) с интервалом 40 мкм между ними ( рис. 1А , 1В ). Кремниевая пластинчатая форма, состоящая из твердого печеного фоторезиста SU-8, была изготовлена в установке 27 нанообработки. Другие подходы к основному приготовлению включают плавление фтористоводородной кислоты (HF), высокотемпературное травление водой, кюрографию и трехмерную печать 28 , 29 , 30 , 31 . CommercТакже могут быть использованы источники для подготовки кремния. Образцы микрожидкостного устройства были разработаны с использованием программного обеспечения для составления чертежей (см. Таблицу материалов). Исходный файл, содержащий шаблон, предоставляется в виде дополнительного файла «Pattern Design.dwg», который может быть предоставлен непосредственно производителю. - Нежно отделите PDMS от кремниевого мастера руками. Отметьте границы каждого микрошара в прямоугольниках с помощью хирургического скальпеля и линейки. Затем отрежьте PDMS на блоки.
- Перфорируйте 16 отверстий (8 входов и 8 выходов на блок) на обоих концах каждого микроканала с помощью биопсийного пуансона (диаметр отверстия 1,0 мм), как показано на рисунке 3B .
- Нанесите ленту поверх каждого блока PDMS, чтобы защитить микронапряжения от повреждений и пыли. Храните их в пластиковом контейнере.
3. Подготовка малых однослойных везикул (внедорожников)
ПРИМЕЧАНИЕ. Двухслойная композиция с отрицательным контролемсоставляет 99,5% мол 1-пальмитоил-2-oleoyl- зп глицеро-3-фосфохолин (POPC) и 0,5 моль% орто- сульфородамина В-1-пальмитоил-2-oleoyl- зп глицеро-3-фосфоэтаноламин (о SRB- ПАПА). Испытуемая двухслойная композиция составляет 92,0 мол.% POPC, 0,5 мол.% O SRB-POPE и 7,5 мол.% L-α-фосфатидилинозитол-4-фосфата (PI4P) или L-α-фосфатидилинозитол-4,5-бисфосфата (PI ( 4,5) P 2 ). Ниже приведена методика получения 92,0 мол.% POPC, 0,5 мол.% O SRB-POPE и 7,5 мол.% PI (4,5) P 2 -содержащих внедорожников. Синтез o SRB-POPE, использованный в этом исследовании, был ранее описан 20 .
- Рассчитайте объем POPC, PI (4,5) P 2 и o SRB-POPE, которые необходимо смешивать, что составляет следующие общие количества: 2,22 мг POPC, 0,26 мг PI (4,5) P 2 , И 2,1 × 10 -5 мг o SRB-POPE.
- Пипеткой рассчитанный объем POPC, PI (4,5) P 2 , o SRB-POPE в один стеклянный сцинтилляционный флакон объемом 20 мл.
ПРИМЕЧАНИЕ: исходные растворы POPC и o SRB-POPE входят в раствор хлороформа, тогда как запас PI (4,5) P 2 (и других фосфоинозитидов) поступает в смесь растворителей хлороформ: метанол: вода (20: 9: 1). Для точности смажьте наконечник пипетки хлороформом перед его использованием. Для безопасности этот шаг должен проходить внутри химического вытяжного шкафа. - Высушите смесь в потоке газообразного азота внутри химического вытяжного шкафа в течение 10 минут или до тех пор, пока растворитель не испарится и на нижней части флакона образуется тонкая липидная пленка.
- Высушить смесь под вакуумом в течение 3 ч при вакуумировании 10 мТорр для удаления остаточного органического растворителя.
ПРИМЕЧАНИЕ. Время высыхания 3 ч достаточно для шкалы подготовки SUV, описанной в этом протоколе, что подходит для 50 экспериментов. Если требуется более крупная шкала подготовки внедорожника, может быть выполнено высушивание в течение ночи. - Восстановить(20 мМ HEPES, 100 мМ NaCl, при pH 7,0) и помещают его в ультразвуковую ванну при рабочей частоте 35 кГц в течение 30 мин при комнатной температуре.
ПРИМЕЧАНИЕ. Смешивание количеств липидов, указанных на этапе 3.1, и добавление 5 мл бегущего буфера на стадии 3.5 дает суспензию везикулы при 0,5 мг / мл. На этом этапе суспензия везикул представляет собой смесь однослойных и многослойных везикул ( фиг. 2В ). Решение будет выглядеть розово-фиолетовым цветом и относительно мутным. - Замораживание-оттаивание суспензии пузырьков жидким азотом и водяной баней при 40 ° C для получения однослойных везикул. Повторите замораживание-оттепель 10 раз.
ПРИМЕЧАНИЕ. (Необязательно). Для обеспечения отсутствия неламеллярных везикул в суспензии можно применять операцию центрифугирования 32 , 33 . - Экструдируйте суспензию везикул через 0,10 мкм трек-травленную поликарбонатную мембрану с использованием липидного экструдера (см.Таблица материалов) для обогащения внедорожников. Повторите экструзию 10 раз.
ПРИМЕЧАНИЕ. 15-мл коническую трубку можно использовать для сбора экструдированных везикул. На этом этапе решение должно быть не мутным. Диаметр SUV можно подтвердить с помощью динамического рассеяния света (DLS) ( рис. 2C ). Внедорожники лучше всего подходят для формирования SLB 34 . - Накройте коническую трубку алюминиевой фольгой и храните при температуре 4 ° C до готовности к использованию.
4. Сборка микрожидкостного устройства
- Протестируйте входы и выходы блока PDMS для блокировки, пропуская деионизированную воду через отверстия с помощью бутылки для промывки водой. Затем высушите блок PDMS газообразным азотом.
ПРИМЕЧАНИЕ. На этом этапе также будут удалены любые частицы пыли, которые могли быть захвачены внутри и / или между каналами. Если необходимо, очистите очищенные покровные стекла газообразным азотом для удаления любых частиц пыли. - Поместите блок PDMS и предварительно очищенную(См. Раздел 1) внутри кислородной плазменной системы (см. Таблицу материалов) камеру для экспонирования с кислородной плазмой в течение 45 с с установкой мощности при 75 Вт, скоростью потока кислорода при 10 см 3 / мин и силой вакуума при 200 мТорр ,
- Поместите узорную поверхность блока PDMS в контакт с покровным стеклом сразу же после обработки кислородной плазмой. Нажмите мягко, чтобы удалить пузырьки воздуха на местах контакта.
- Поместите устройство на ровную горячую плиту при 100 ° C в течение 3 минут, чтобы улучшить склеивание.
- Используйте влажную безворсовую салфетку ( например, кимвипе) со 100% этанолом для удаления пыли с верхней части (PDMS) и нижней части (стекла) устройства. Затем закрепите устройство на стекле на стекле.
ПРИМЕЧАНИЕ. Не используйте чрезмерное количество этанола и избегайте попадания этанола во входные и выходные каналы. Рекомендуется выполнить этот шаг, пока устройство остается горячим, чтобы гарантировать, что этанол немедленно испарится. Стеклянный микрофонOscopy slides можно хранить в 100% -ном растворе этанола для удаления пыли и других загрязнений.
5. Формирование поддерживаемых липидных бислоев (SLB)
- Передайте 100 мкл PI (4,5) P 2 -содержащих внедорожников с шага 3.8 в микроцентрифужную пробирку 0,65 мл. Отрегулируйте рН раствора до ~ 3,2 путем добавления 6,4 мкл 0,2 н. Соляной кислоты.
ПРИМЕЧАНИЕ. Подтвердите pH раствора с помощью рН-метра, снабженного зондом с микро-рН (см. Таблицу материалов). - Внесите 10 мкл раствора SUV с регулируемым pH в каждый канал через вход и нанесите давление через пипетку до тех пор, пока раствор не достигнет выхода. Отсоедините наконечник от пипетки и оставьте его прикрепленным к устройству.
- Повторите вышеуказанный шаг для каждого канала, а затем инкубируйте устройство в течение 10 минут при комнатной температуре.
ПРИМЕЧАНИЕ. Впрыскивание везикул в микроканалы должно выполняться сразу после сборки устройства. - Режущие наборы впускных и выпускных трубок eaХ 60 см (входная трубка) и 8 см (выпускная трубка), соответственно.
ПРИМЕЧАНИЕ. Резка трубки по диагонали и создание острой кромки облегчает установку трубки в отверстия и выпускные отверстия. Выпускная трубка должна иметь форму дуги. Длина впускной трубы может варьироваться в зависимости от установки микроскопа. Внутренний диаметр трубки составляет 0,05 см. - Используя пинцет, подключите выпускную трубу, установленную к устройству, и затем заклейте устройство на ступень микроскопа.
- Погрузите один конец впускной трубки, установленный в 25 мл бегущего буфера, содержащегося в конической трубке, и закрепите его лентой, чтобы убедиться, что трубка закреплена.
- Поместите коническую трубку на более высокую поверхность (~ 20 см), чем устройство, чтобы проталкивать раствор через микроканалы через гравитационный поток; Для этого можно использовать лабораторный домкрат.
- Для каждой входной трубки используйте шприц для натягивания 1 мл бегущего буфера из свободного конца трубки. Удалите наконечник пипетки с впускного отверстия и вставьтеСвободный конец впускной трубы в устройство.
ПРИМЕЧАНИЕ. Внесите капельку буфера на вход, чтобы уменьшить вероятность появления пузырька воздуха в канале на этом этапе. После того, как впускная трубка присоединяется к устройству, используйте безворсовую салфетку для удаления избыточного буфера. - Повторите описанный выше шаг, чтобы соединить все элементы впускного трубопровода с устройством.
ПРИМЕЧАНИЕ. Проточный буферный поток через каналы помогает удалять избыточные пузырьки и уравновешивать двухслойный слой до экспериментальных условий. - Откройте программное обеспечение для управления микроскопом (см. Таблицу материалов). На левой панели щелкните вкладку «Микроскоп» и выберите цель «10X».
- Нажмите «Live», а затем «Alexa 568» значки изображений на панели инструментов. Используя тонкие ручки регулировки курса, сосредоточьтесь на микроканалах.
- Просканируйте устройство, чтобы проверить качество SLB и каналов ( рисунок 8 ). Затем,Щелкните значок «FL Shutter Closed» на панели инструментов.
- Перейдите на вкладку «Приобретение», а в разделе «Основные настройки» выберите «Время экспозиции». Установите время экспозиции равным «200 мс».
- На левой панели нажмите «Многомерное приобретение», а в меню «Фильтры» выберите красный канал (отмечен как «Alexa 568»). Затем нажмите «Время», установите интервал времени до 5 минут, продолжительность до 30 минут и нажмите «Пуск».
ПРИМЕЧАНИЕ. В зависимости от продолжительности (30 мин) и скорости потока (~ 1,0 мкл / мин) для выравнивания SLB в канале используется 30 мкл рабочего буфера, т.е. для всех 8 каналов используется 240 мкл рабочего буфера. - Выберите инструмент «Круг» на вкладке «Измерение» и нарисуйте круг в любом канале. Щелкните правой кнопкой мыши, когда выбран круг, и выберите «Свойства». На вкладке «Профиль» проверьте «все T», чтобы просмотретьИнтенсивность флуоресценции как функция времени.
- Убедитесь, что эта кривая достигает плато, которое указывает на равновесие, прежде чем перейти к следующему шагу.
- Опустите буферный раствор на равную поверхность как устройство, чтобы остановить поток.
- Сохраните файл временной задержки.
6. Тестирование взаимодействия доменов PLC-δ1 PH с PI (4,5) P 2 -содержащими SLB
- Подготовьте разведения домена PH, используя бегущий буфер в качестве разбавителя.
ПРИМЕЧАНИЕ. Поскольку в устройстве имеется восемь каналов, используйте для их пустых элементов как минимум два из них. Выбирайте дальние концы с каждой стороны и используйте оставшиеся каналы для разведения белка. Были испытаны следующие концентрации домена PH: 0,10, 0,25, 0,50, 1,00 и 2,50 мкМ. Приблизительно 200 мкл каждого разведения было достаточным для достижения равновесия в течение 30 мин. - По одному, отсоедините каждую выпускную трубку и нанесите 200 мкл каждого разведения белка вВыходной канал с использованием пипетки. Не применяйте никакого давления, дайте гравитации выполнить работу. Отсоедините наконечник от пипетки и оставьте его прикрепленным к микрожидкостному устройству.
- Повторите вышеуказанный шаг для каждого канала и убедитесь, что воздушные пузырьки не введены в каналы во время этого процесса.
- Опустите входную трубу на землю под микрожидкостное устройство, чтобы начать протекать через микроканалы. Проденьте свободный конец трубки в контейнер для отходов. Поток белковых разведений в течение 30 мин.
ПРИМЕЧАНИЕ. Это приведет к обратному потоку и позволяет вводить белок в каналы. Необходимое время для уравновешивания и объем необходимых разведений белка зависит от аффинности взаимодействия. - На левой панели программного обеспечения на вкладке «Время ожидания» нажмите «Пуск», чтобы начать визуализацию снова.
- Повторите шаг 5.15, чтобы убедиться, что равновесие достигнуто. Когда эксперимент будет завершен, сэкономьте времяфайл.
7. Оценка мембранной текучести
ПРИМЕЧАНИЕ. Для каждой новой партии внедорожников и очищенных покровных покрытий для стекла должны выполняться эксперименты по флуоресценции после фотообесцвечивания (FRAP), чтобы гарантировать, что SLB являются текучими.
- Следуйте процедуре по очистке стеклянных покровных (1.1-1.6), изготовлению микропланктонных блоков PDMS (2.1-2.5), подготовке внедорожников (3.1-3.8), сборке микрожидкостного устройства (4.1-4.5) и формированию SLB (5.1-5.17), как ранее описано.
- Фокусируйте микроскоп на двухслойном слое, установите время экспозиции на 200 мс, а биннинг - на 1.
- Фото-отбеливать круговое пятно с использованием лазерного луча 532 нм, 2 мВт (радиус 13 мкМ). Сразу же после фотообесцвечивания начните захватывать серию изображений каждые 3 с в течение первых 45 с, за которым следует интервал 30 с в течение оставшегося времени (общая продолжительность 15 мин). После завершения сбора данных сохраните файл временной задержки.
- Выберите отбеленный aИспользуя инструмент рисования кругов для извлечения значений интенсивности флуоресценции в зависимости от времени. Кроме того, выберите еще две области рядом, одну из которых соответствует небеленой области, а другая соответствует области без каких-либо SLB.
- Рассчитайте восстановление флуоресценции ( y ), используя следующее уравнение:
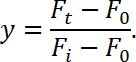
ПРИМЕЧАНИЕ. Здесь F t представляет интенсивность отбеленной области как функцию времени, F i представляет интенсивность флуоресценции перед отбеливанием (используйте в этом случае «1» в качестве нормированной величины); F 0 - интенсивность фона. - Восстановление флуоресценции участка (ось y) в зависимости от времени (по оси x) для генерации кривой FRAP. Затем подгоните данные к одной экспоненциальной функции следующим образом:
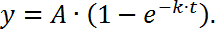
ПРИМЕЧАНИЕ. Здесь A представляет собой подвижную фракцию и k - кинетическая константа для подвижной фракции, которая используется для вычисления времени полувыведения до восстановления ( t 1/2 ):
- Используйте t 1/2 для расчета константы диффузии ( D ):

ПРИМЕЧАНИЕ. Здесь R представляет радиус лазерного луча (13 мкм). Константа диффузии для жидкого бислоя должна быть ≥ 1,0 мкм 2 / с.
8. Обработка данных
ПРИМЕЧАНИЕ. Процедура анализа данных будет зависеть от микроскопа, программного обеспечения для обработки изображений и используемого программного обеспечения для подбора кривой.
- Откройте файлы с временной задержкой (начиная с шага 5.17) и после (с шага 6.6), титрируя домен PH. Перейдите к последнему кадру каждого временного файла и выполните сканирование линии по всем микроканалам для получения флуоресценцииВ зависимости от расстояния в пикселях ( рис. 6A ). Перенесите данные в программное обеспечение для работы с электронными таблицами.
- Для каждого микроканала (до и после добавления домена PH) проведите несколько данных в канале и по обе стороны канала, представляющего базовую линию ( рисунок 7A ).
ПРИМЕЧАНИЕ. Интенсивность флуоресценции пустого канала должна оставаться неизменной. Все остальные каналы нормированы на пустой канал, поэтому важно иметь два пустых канала и следить за тем, чтобы их интенсивности флуоресценции оставались согласованными друг с другом. - Примените приведенную ниже формулу, чтобы нормализовать данные на пустой канал; Расчет выборок представлен на рисунке 7B .

ПРИМЕЧАНИЕ. Здесь белок ΔF представляет фокусную вычитаемую интенсивность флуоресценции белкового канала, Δ; F blank представляет фокусную вычитаемую интенсивность флуоресценции пустого канала, pre и post относятся к стадиям титрования до и после белков, а α представляет собой поправочный коэффициент, который представляет собой отношение интенсивностей флуоресценции белкового канала и пустого канала ([ F белок / F бланк ] до ) на стадии предварительного белкового титрования. Поскольку интенсивность флуоресценции уменьшается в зависимости от концентрации домена PH, нормированные данные будут отрицательными по значению. - Используя программное обеспечение для подгонки кривой (см. Таблицу материалов ), нарисуйте нормированную флуоресценцию в зависимости от концентрации домена PH и примените к изотерме Ленгмюра ( рис. 6C ):
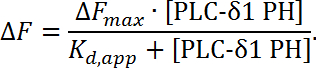
ПРИМЕЧАНИЕ. Здесь ΔF представляет собой изменение интенсивности флуоресценции относительно максимального изменения флюоресценцииКогда концентрация насыщения белка присутствует ( ΔF max ), тогда как K d, приложение представляет собой кажущуюся константу диссоциации, которая равна концентрации объемного белка ( [PLC-δ1 PH] ), при которой 50% охвата ( Мембрано-связанный комплекс). Это измерение привязки на основе равновесия, поэтому нормированные данные пригодны для простой изотермы Ленгмюра для извлечения видимого экспериментального параметра K d, приложение . На основе программного обеспечения для установки K d, приложение для взаимодействия PLC-δ1 PH-PI (4,5) P 2 составляет 0,39 ± 0,05 мкМ ( рис. 6B ).
Access restricted. Please log in or start a trial to view this content.
Результаты
Мы использовали пробу рН модуляции для изучения ПЛК-б1 PH домен-PI (4,5) P 2 взаимодействие в рамках Микроприбор PIP-на-чипе (рисунок 1). По подробному протоколу мы продемонстрировали, как подготовить и собрать компоненты микрожидкостного устройства, изг...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Каждый вариант PIP, хотя и при низких концентрациях, присутствует на цитозольной поверхности конкретных органелл, где они способствуют установлению уникального физического состава и функциональной специфичности органелларной мембраны 1 . Одним из наиболее важных примене?...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
DS и CEC были частично поддержаны грантом AI053531 (NIAID, NIH); SS и PSC поддерживались грантом N00014-14-1-0792 (ONR).
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| Coverslip | |||
| Glass Coverslips: Rectangles | Fisher Scientific | 12-544B | 22 x 40 x 0.16 - 0.19 mm, No. 1 1/2; Borosilicate Glass |
| 7X Cleaning Solution | MP Biomedicals | 976670 | Detergent |
| PYREX Crystallizing Dish | Corning | 3140-190 | Borosilicate glass dish with a flat bottom; Diameter x Height (190 x 100 mm); Distributor: VWR (89090-700) |
| Sentry Xpress 2.0 | Paragon Industries | SC-2 | Kiln |
| Name | Company | Catalog Number | Comments |
| PDMS | |||
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 4019862 | Polydimethylsiloxane (PDMS); Distributor: Ellsworth Adhesives |
| PYREX Desiccator | VWR | 89134-402 | Vacuum Rated |
| Biopsy punch | Harris | 15110-10 | Harris Uni-Core; 1.0 mm diameter; Miltex Biopsy Punch with Plunger (Cat. No. 15110-10) can be used as an alternative |
| Name | Company | Catalog Number | Comments |
| Device | |||
| Plasma Cleaning System | PlasmaEtch | PE25-JW | 2-stage Direct Drive Oil Vacuum Pump, O2 service (Krytox Charged) |
| Digital Hot Plate | Benchmark | H3760-H | Purchased through Denville Scientific (Cat. No. 1005640) |
| Frosted Micro Slides | VWR | 48312-003 | Frosted, Selected, and Precleaned; Made of Swiss Glass; Thickness: 1 mm; Dimensions: 75 x 25 mm; GR 144 |
| Name | Company | Catalog Number | Comments |
| Mold | |||
| AutoCAD | Autodesk | v.2016 | Drafting software for the photomask design |
| Photomask | CAD/Art Services | N/A | Design with black background and clear features was printed at 20k dpi resolution on a transparent mask (5 x 7 in) by CAD/Art Services |
| Silicone Wafers | University Wafer | 1575 | Prime Grade, Single Side Polished; 100 mm (4 inch) Diameter; 525 um Thickness |
| SU-8 50 | MicroChem Corp. | N/A | Negative Tone Photoresist; Penn State Nanofabrication Facility Property |
| SU-8 Developer | MicroChem Corp. | N/A | Penn State Nanofabrication Facility Property |
| Name | Company | Catalog Number | Comments |
| SUV | |||
| 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine | Avanti Polar Lipids | 850457C | POPC |
| L-α-phosphatidylinositol-4-phosphate | Avanti Polar Lipids | 840045X | PI4P |
| L-α-phosphatidylinositol-4,5-bisphosphate | Avanti Polar Lipids | 840046X | PI(4,5)P2 |
| 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphoethanolamine | Avanti Polar Lipids | 850757C | POPE; Required for the synthesis of oSRB-POPE |
| Lissamine Rhodamine B Sulfonyl Chloride (mixed isomers) | ThermoFisher Scientific | L-20 | Required for the synthesis of oSRB-POPE |
| pH Sensitive Fluorescent Lipid Probe (oSRB-POPE) | In-house | N/A | In-house Synthesis (Huang D. et al. 2013) |
| Glass Scintillation Vial | VWR | 66022-065 | 20 mL volume capacity |
| Aquasonic 250D | VWR | N/A | Ultrasonic Water Bath |
| Nuclepore Track-Etched Membranes | Whatman | 110605 | Polycarbonate Membrane; Diameter: 25 mm; Pore Size: 0.1 um; Distributor: Sigma-Aldrich |
| Chloroform | VWR | CX1054-6 | HPLC grade |
| LIPEX Extruder | Transferra Nanosciences | T.001 | LIPEX 10 mL Thermobarrel Extruder |
| Viscotek 802 DLS | Malvern Instruments | N/A | Dynamic Light Scattering; Penn State X-Ray Crystallography Facility Property |
| Name | Company | Catalog Number | Comments |
| Data Analysis | |||
| GraphPad Prism | GraphPad Software | v.6 | Curve-fitting software for data analysis |
| Name | Company | Catalog Number | Comments |
| Microscope | |||
| Axiovert 200M Epifluorescence Microscope | Carl Zeiss Microscopy | N/A | Microscope |
| AxioCam MRm Camera | Carl Zeiss Microscopy | N/A | Camera |
| X-Cite 120 | Excelitas Technologies | N/A | Light Source |
| Alexa 568 Filter Set | Carl Zeiss Microscopy | N/A | Ex/Em 576/603 nm |
| AxioVision LE64 v.4.9.1.0 Software | Carl Zeiss Microscopy | N/A | Image Processing Software |
| Name | Company | Catalog Number | Comments |
| Other | |||
| Tips | VWR | 10034-132 | 200 uL pipette tips; Thin and smooth tip for applying the protein solution into the microfluidic channel |
| Tips | VWR | 53509-070 | 10 uL pipette tips; Thin and smooth tip for applying the vesicle solution into the microfluidic channel |
| Orion Star A321 pH meter | Thermo Scientific | STARA3210 | pH meter |
| Orion micro pH probe | Thermo Scientific | 8220BNWP | micro pH probe |
| N-(2-Hydroxyethyl)-Piperazine-N'-(2-Ethanesulfonic Acid) | VWR | VWRB30487 | HEPES, Free Acid |
| Sodium Chloride | VWR | BDH8014-2.5KGR | NaCl |
| Tubing | Allied Wire & Cable | TFT-200-24 N | Internal Diameter: 0.020-0.026 inches (0.051-0.066 cm); Wall Thickness: 0.010 inches (0.025 cm); Flexible Polytetrafluoroethylene Thin-Wall Tubing; Natural Color |
| Nitrogen Gas - Industrial | Praxair | N/A | Local Provider |
| Oxygen Gas - Industrial | Praxair | N/A | Local Provider |
| Liquid Nitrogen | Praxair | N/A | Local Provider |
Ссылки
- Di Paolo, G., De Camilli, P. Phosphoinositides in cell regulation and membrane dynamics. Nature. 443 (7112), 651-657 (2006).
- Shewan, A., Eastburn, D. J., Mostov, K. Phosphoinositides in cell architecture. Cold Spring Harb Perspect Biol. 3 (8), a004796(2011).
- Picas, L., Gaits-Iacovoni, F., Goud, B. The emerging role of phosphoinositide clustering in intracellular trafficking and signal transduction. F1000Res. 5, (2016).
- Lystad, A. H., Simonsen, A. Phosphoinositide-binding proteins in autophagy. FEBS Lett. 590 (15), 2454-2468 (2016).
- Balla, T. Phosphoinositides: Tiny lipids with giant impact on cell regulation. Physiol Rev. 93, 1019-1137 (2013).
- Lemmon, M. A. Membrane recognition by phospholipid-binding domains. Nat Rev Mol Cell Biol. 9 (2), 99-111 (2008).
- Kutateladze, T. G. Translation of the phosphoinositide code by PI effectors. Nat Chem Biol. 6 (7), 507-513 (2010).
- Harlan, J. E., Hajduk, P. J., Yoon, H. S., Fesik, S. W. Pleckstrin homology domains bind to phosphatidylinositol-4,5-bisphosphate. Nature. 371 (6493), 168-170 (1994).
- Garcia, P., et al. The pleckstrin homology domain of phospholipase C-delta 1 binds with high affinity to phosphatidylinositol 4,5-bisphosphate in bilayer membranes. Biochemistry. 34 (49), 16228-16234 (1995).
- Lemmon, M. A., Ferguson, K. M., O'Brien, R., Sigler, P. B., Schlessinger, J. Specific and high-affinity binding of inositol phosphates to an isolated pleckstrin homology domain. Proc Natl Acad Sci U S A. 92 (23), 10472-10476 (1995).
- Flesch, F. M., Yu, J. W., Lemmon, M. A., Burger, K. N. Membrane activity of the phospholipase C-delta1 pleckstrin homology (PH) domain. Biochem J. 389, 435-441 (2005).
- Narayan, K., Lemmon, M. A. Determining selectivity of phosphoinositide-binding domains. Methods. 39 (2), 122-133 (2006).
- Scott, J. L., Musselman, C. A., Adu-Gyamfi, E., Kutateladze, T. G., Stahelin, R. V. Emerging methodologies to investigate lipid-protein interactions. Integr Biol (Camb). 4 (3), 247-258 (2012).
- Dowler, S., Currie, R. A., Downes, C. P., Alessi, D. R. DAPP1: A dual adaptor for phosphotyrosine and 3-phosphoinositides. Biochem J. 342, 7-12 (1999).
- He, J., et al. Molecular basis of phosphatidylinositol 4-phosphate and ARF1 GTPase recognition by the FAPP1 pleckstrin homology (PH) domain. J Biol Chem. 286 (21), 18650-18657 (2011).
- Ceccarelli, D. F., et al. Non-canonical interaction of phosphoinositides with pleckstrin homology domains of Tiam1 and ArhGAP9. J Biol Chem. 282 (18), 13864-13874 (2007).
- Huang, S., Gao, L., Blanchoin, L., Staiger, C. J. Heterodimeric capping protein from Arabidopsis is regulated by phosphatidic acid. Mol Biol Cell. 17 (4), 1946-1958 (2006).
- Yu, J. W., et al. Genome-eide analysis of membrane targeting by S. cerevisiae pleckstrin homology domains. Mol Cell. 13 (5), 677-688 (2004).
- Jung, H., Robison, A. D., Cremer, P. S. Detecting protein-ligand binding on supported bilayers by local pH modulation. J Am Chem Soc. 131 (3), 1006-1014 (2009).
- Huang, D., Zhao, T., Xu, W., Yang, T., Cremer, P. S. Sensing small molecule interactions with lipid membranes by local pH modulation. Anal Chem. 85 (21), 10240-10248 (2013).
- Saxena, A., et al. Phosphoinositide binding by the pleckstrin homology domains of Ipl and Tih1. J Biol Chem. 277 (51), 49935-49944 (2002).
- Knödler, A., Mayinger, P. Analysis of phosphoinositide-binding proteins using liposomes as an affinity matrix. Biotechniques. 38 (6), 858-862 (2005).
- Baumann, M. K., Swann, M. J., Textor, M., Reimhult, E. Pleckstrin homology-phospholipase C-delta1 interaction with phosphatidylinositol 4,5-bisphosphate containing supported lipid bilayers monitored in situ with dual polarization interferometry. Anal Chem. 83 (16), 6267-6274 (2011).
- Saliba, A. E., et al. A quantitative liposome microarray to systematically characterize protein-lipid interactions. Nat Methods. 11 (1), 47-50 (2014).
- Arauz, E., Aggarwal, V., Jain, A., Ha, T., Chen, J. Single-molecule analysis of lipid-protein interactions in crude cell lysates. Anal Chem. 88 (8), 4269-4276 (2016).
- Best, Q. A., Xu, R., McCarroll, M. E., Wang, L., Dyer, D. J. Design and investigation of a series of rhodamine-based fluorescent probes for optical measurements of pH. Org Lett. 12 (14), 3219-3221 (2010).
- Lee, J., Choi, K. H., Yoo, K. Innovative SU-8 lithography techniques and their applications. Micromachines. 6 (1), 1-18 (2014).
- Poyton, M. F., Sendecki, A. M., Cong, X., Cremer, P. S. Cu(2+) binds to phosphatidylethanolamine and increases oxidation in lipid membranes. J Am Chem Soc. 138 (5), 1584-1590 (2016).
- Karasek, P., Grym, J., Roth, M., Planeta, J., Foret, F. Etching of glass microchips with supercritical water. Lab Chip. 15 (1), 311-318 (2015).
- Thomas, M. S., et al. Print-and-peel fabrication for microfluidics: what's in it for biomedical applications? Ann Biomed Eng. 38 (1), 21-32 (2010).
- Waheed, S., et al. 3D printed microfluidic devices: enablers and barriers. Lab Chip. 16 (11), 1993-2013 (2016).
- Axmann, M., Schutz, G. J., Huppa, J. B. Single molecule fluorescence microscopy on planar supported bilayers. J Vis Exp. (105), e53158(2015).
- Barenholz, Y., et al. A simple method for the preparation of homogeneous phospholipid vesicles. Biochemistry. 16 (12), 2806-2810 (1977).
- Castellana, E. T., Cremer, P. S. Solid supported lipid bilayers: From biophysical studies to sensor design. Surface Science Reports. 61 (10), 429-444 (2006).
- Hamai, C., Yang, T., Kataoka, S., Cremer, P. S., Musser, S. M. Effect of average phospholipid curvature on supported bilayer formation on glass by vesicle fusion. Biophys J. 90 (4), 1241-1248 (2006).
- Tero, R. Substrate effects on the formation process, structure and physicochemical properties of supported lipid bilayers. Materials. 5 (12), 2658-2680 (2012).
- Ferguson, K. M., Lemmon, M. A., Schlessinger, J., Sigler, P. B. Structure of the high affinity complex of inositol trisphosphate with a phospholipase C pleckstrin homology domain. Cell. 83 (6), 1037-1046 (1995).
- Simonsson, L., Hook, F. Formation and diffusivity characterization of supported lipid bilayers with complex lipid compositions. Langmuir. 28 (28), 10528-10533 (2012).
- Cong, X., Poyton, M. F., Baxter, A. J., Pullanchery, S., Cremer, P. S. Unquenchable surface potential dramatically enhances Cu(2+) binding to phosphatidylserine lipids. J Am Chem Soc. 137 (24), 7785-7792 (2015).
- Robison, A. D., et al. Polyarginine interacts more strongly and cooperatively than polylysine with phospholipid bilayers. J Phys Chem B. 120 (35), 9287-9296 (2016).
- Robison, A. D., Huang, D., Jung, H., Cremer, P. S. Fluorescence modulation sensing of positively and negatively charged proteins on lipid bilayers. Biointerphases. 8 (1), 1(2013).
- Tabaei, S. R., et al. Formation of cholesterol-rich supported membranes using solvent-assisted lipid self-assembly. Langmuir. 30 (44), 13345-13352 (2014).
- Johnson, S. J., et al. Structure of an adsorbed dimyristoylphosphatidylcholine bilayer measured with specular reflection of neutrons. Biophys J. 59 (2), 289-294 (1991).
- Koenig, B. W., et al. Neutron reflectivity and atomic force microscopy studies of a lipid bilayer in water adsorbed to the surface of a silicon single crystal. Langmuir. 12 (5), 1343-1350 (1996).
- Tanaka, M., Sackmann, E. Polymer-supported membranes as models of the cell surface. Nature. 437 (7059), 656-663 (2005).
- Renner, L., et al. Supported lipid bilayers on spacious and pH-responsive polymer cushions with varied hydrophilicity. J Phys Chem B. 112 (20), 6373-6378 (2008).
- Wagner, M. L., Tamm, L. K. Tethered polymer-supported planar lipid bilayers for reconstitution of integral membrane proteins: Silane-polyethyleneglycol-lipid as a cushion and covalent linker. Biophys J. 79 (3), 1400-1414 (2000).
- Pace, H., et al. Preserved transmembrane protein mobility in polymer-supported lipid bilayers derived from cell membranes. Anal Chem. 87 (18), 9194-9203 (2015).
- Braunger, J. A., Kramer, C., Morick, D., Steinem, C. Solid supported membranes doped with PIP2: Influence of ionic strength and pH on bilayer formation and membrane organization. Langmuir. 29 (46), 14204-14213 (2013).
- Paridon, P. A., de Kruijff, B., Ouwerkerk, R., Wirtz, K. W. Polyphosphoinositides undergo charge neutralization in the physiological pH range: A 31P-NMR study. Biochim Biophys Acta. 877 (1), 216-219 (1986).
- Liu, C., Huang, D., Yang, T., Cremer, P. S. Monitoring phosphatidic acid formation in intact phosphatidylcholine bilayers upon phospholipase D catalysis. Anal Chem. 86 (3), 1753-1759 (2014).
- Saad, J. S., et al. Structural basis for targeting HIV-1 Gag proteins to the plasma membrane for virus assembly. Proc Natl Acad Sci U S A. 103 (30), 11364-11369 (2006).
- Hsu, N. Y., et al. Viral reorganization of the secretory pathway generates distinct organelles for RNA replication. Cell. 141 (5), 799-811 (2010).
- Del Campo, C. M., et al. Structural basis for PI(4)P-specific membrane recruitment of the Legionella pneumophila effector DrrA/SidM. Structure. 22 (3), 397-408 (2014).
- Kolli, S., et al. Structure-function analysis of vaccinia virus H7 protein reveals a novel phosphoinositide binding fold essential for poxvirus replication. J Virol. 89 (4), 2209-2219 (2015).
- Cho, N. J., et al. Phosphatidylinositol 4,5-bisphosphate is an HCV NS5A ligand and mediates replication of the viral genome. Gastroenterology. 148 (3), 616-625 (2015).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены