Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
PIP-on-a-chip: un estudio sin etiquetas de las interacciones proteína-fosfoinositida
En este artículo
Resumen
Aquí presentamos una bicapa lipídica soportada en el contexto de una plataforma microfluídica para estudiar las interacciones proteína-fosfoinositido utilizando un método libre de etiquetas basado en la modulación del pH.
Resumen
Numerosas proteínas celulares interactúan con las superficies de la membrana para afectar los procesos celulares esenciales. Estas interacciones pueden dirigirse hacia un componente lipídico específico dentro de una membrana, como en el caso de fosfoinositidos (PIP), para asegurar una localización y / o activación subcelular específica. Los PIP y los dominios celulares de unión a PIP se han estudiado ampliamente para comprender mejor su papel en la fisiología celular. Se aplicó un ensayo de modulación del pH en bicapas lipídicas (SLB) como una herramienta para estudiar las interacciones proteína-PIP. En estos estudios, se usó fosfatidiletanolamina conjugada con orto- sulforeamina B sensible al pH para detectar interacciones proteína-PIP. Tras la unión de una proteína a una superficie de membrana que contiene PIP, se modula el potencial interfacial ( es decir, cambio en el pH local), desplazando el estado de protonación de la sonda. Se presenta un estudio de caso del uso exitoso del ensayo de modulación del pH usando fosfolipasa C delta1 Pleckstren la homología (PH PLC-δ1) dominio y fosfatidilinositol 4,5-bifosfato (PI (4,5) P 2) la interacción como un ejemplo. La aparente constante de disociación ( K d, app ) para esta interacción fue 0,39 ± 0,05 μ M, similar a K d, app valores obtenidos por otros. Como se observó anteriormente, el dominio PH PLC-δ1 es PI (4,5) P 2 específico, muestra unión más débil hacia fosfatidilinositol 4-fosfato, y no se une a SLB de fosfatidilcolina pura. El ensayo de PIP sobre un chip es ventajoso con respecto a los ensayos de unión a PIP tradicionales, incluyendo, pero sin limitarse a, bajo volumen de muestra y sin requisitos de marcado de ligando / receptor, la capacidad de probar interacciones de membrana de alta y baja afinidad con Grandes moléculas, y una relación señal / ruido mejorada. En consecuencia, el uso del enfoque PIP-on-a-chip facilitará la elucidación de mecanismos de una amplia gama de interacciones de membrana. Además, este método podría ser uEn la identificación de terapéuticos que modulan la capacidad de la proteína para interactuar con las membranas.
Introducción
Las innumerables interacciones y procesos bioquímicos tienen lugar en superficies de membrana fluidas bidimensionales. Los organelos cerrados con membrana en células eucariotas son únicos no sólo en los procesos bioquímicos y su proteoma asociado, sino también en su composición lipídica. Una clase excepcional de fosfolípidos es phosphoinositides (PIPs). Aunque constituyen sólo el 1% del lipidoma celular, desempeñan un papel crucial en la transducción de señales, autofagia y tráfico de membranas, entre otros 1 , 2 , 3 , 4 . La fosforilación dinámica del grupo cabeza de inositol por quinasas PIP celular da lugar a siete grupos de cabeza PIP que son mono-, bis- o tris-fosforilados 5 . Adicionalmente, los PIPs definen la identidad subcelular de las membranas y sirven como sitios de acoplamiento de membrana especializados para proteínas / enzimas que contienen uno o más fosfoinosPor ejemplo, Homología de Pleckstrin (PH), Homología de Phox (PX), y Homología N-terminal de epsina (ENTH) 6 , 7 . Uno de los dominios mejor estudiados de unión PIP-es fosfolipasa C (PLC) dominio PH -δ1 que interactúa específicamente con fosfatidilinositol 4,5-bifosfato (PI (4,5) P 2) dentro de una alta afinidad rango micromolar nanomolar bajo 8 , 9 , 10 , 11 .
Se han desarrollado una variedad de métodos cualitativos y cuantitativos in vitro para estudiar el mecanismo, la termodinámica y la especificidad de estas interacciones. Entre los ensayos de unión a PIP más comúnmente utilizados están la resonancia de plasmón de superficie (SPR), la calorimetría isotérmica (ITC), la espectroscopia de resonancia magnética nuclear (NMR), el ensayo de flotación / sedimentación de liposomas y las transferencias lipídicas (tiras de grasa / PIP)12 , 13 . A pesar de que se utilizan ampliamente, todos ellos tienen muchas desventajas. Por ejemplo, SPR, ITC y RMN requieren grandes cantidades de muestra, instrumentación costosa y / o personal capacitado 12 , 13 . Algunos formatos de ensayo tales como las transferencias lipídicas basadas en anticuerpos utilizan formas de PIP solubles en agua y las presentan de una manera no fisiológica 12 , 14 , 15 , 16 . Además, las transferencias de lípidos no se pueden cuantificar de forma fiable y con frecuencia han dado lugar a falsas observaciones positivas / negativas 12 , 17 , 18 . Para superar estos desafíos y mejorar el conjunto actual de herramientas, se estableció un nuevo método libre de etiquetas basado en una bicapa lipídica soportada (SLB) en el contexto de am Icrofluidic plataforma, que se aplicó con éxito al estudio de la proteína-PIP interacciones ( Figura 1 ] [ 19] .
La estrategia empleada para detectar las interacciones proteína-PIP se basa en la detección de la modulación del pH. Esto implica un colorante sensible al pH que tiene orto -Sulforhodamine B (o SRB) directamente conjugado con grupo de cabeza fosfatidiletanolamina lípidos 20. La sonda o SRB-POPE (Figura 2A) es altamente fluorescente a pH bajo y se inactivó a pH alto con un pKa en torno a 6,7 dentro de 7,5% en moles de PI (4,5) P 2 SLB que contiene (figura 5B). Dominio PH PLC-δ1 se ha utilizado ampliamente para la validación de metodologías proteína-PIP-de unión debido a su alta especificidad hacia PI (4,5) P 2 (Figura 5A) 21, 22,"> 23, 24, 25 .Hence, razonó que el dominio PLC-δ1 PH se puede utilizar para poner a prueba su unión a PI (4,5) P 2 a través del ensayo de PIP-on-a-chip. El dominio PH constructo Utilizado en este estudio tiene una carga positiva neta (pI 8.4), y por lo tanto atrae a OH - iones ( Figura 5C ). Al unirse a PI (4,5) P 2 -contiene SLBs, el dominio PH lleva a OH - superficie de la membrana, que a su vez modula el potencial interfacial y desplaza el estado de protonación de O SRB-POPE (Figura 5C) 26. en función de la concentración de dominio PH, la fluorescencia se inactiva (figura 6A). por último, la transmisión de datos normalizada es ajustarse a una isoterma de unión para determinar la afinidad de la interacción PH dominio-PI (4,5) P 2 (Figura 6B, 6C). En este estudio, se proporciona un protocolo detallado para realizar la unión de proteínas a PIB que contienen SLBs dentro de una plataforma microfluídica. Este protocolo lleva al lector a ensamblar el dispositivo microfluídico y la preparación de la vesícula a la formación de SLB y la unión a proteínas. Además, las direcciones para el análisis de datos para extraer información afinidad por el PLC-δ1 PH dominio de PI (4,5) la interacción P 2 se proporcionan.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Limpieza de los cubreobjetos de vidrio
- Solución diluida de limpieza 7x (ver Tabla de Materiales) 7 veces con agua desionizada en un recipiente de vidrio de borosilicato de profundidad 100 mm con un fondo plano y calentar hasta 95 ° C sobre una placa caliente nivel durante 20 min o hasta que la solución turbia se vuelve clara .
NOTA: La solución estará caliente, tenga cuidado para evitar lesiones corporales. La Solución de Limpieza 7x es una mezcla patentada de hexafluorofosfato de hexasodio [oxido (fosfonatooxi) fosforil] oxifosforil], 2- (2-butoxietoxi) etanol, 1,4-bis (2-etilhexoxi) -1,4-dioxobutano -2-sulfonato y adiciones no peligrosas. - Utilizando pinzas, coloque los cubreobjetos (40 mm de longitud x 22 mm de ancho x 0,16-0,19 mm de altura) en una rejilla de aluminio en direcciones alternadas. Mantenga un lugar vacío entre cada cubreobjetos para evitar que se toquen.
- Sumerja completamente la rejilla de tinción con los cubreobjetos en la solución de limpieza. Mantenga los cubreobjetos enLa solución durante 1 h.
NOTA: A medida que el agua se evapora, agregue agua desionizada fresca para mantener la concentración de la solución sin cambios. - Saque las bandejas de coloración de la solución de limpieza y lave las cubreobjetos con cantidades abundantes de agua desionizada para retirar el detergente.
- Seque los cubreobjetos con gas nitrógeno (aproximadamente 5 minutos por rejilla de tinción) y luego coloque los cubreobjetos en un horno durante 6 h a 550 ° C para el recocido.
NOTA: Este es un paso crucial que suaviza las características ásperas de la superficie del vidrio. - Coloque los cubreobjetos en un recipiente de plástico y mantenerlos alejados del polvo.
2. Fabricación de bloques PDMS micropatternados
- Mezclar el prepolímero de polidimetilsiloxano (PDMS) y el agente de curado a una relación 10: 1 (p / p) en un barco pesado de plástico grande y desgasearlo en vacío durante 1 h con una resistencia al vacío de 500 Torr o menos.
NOTA: PDMS es un polímero de silicona transparente e inerte. Para obtener el bloque PDMS deseado t(0,5 cm), mezclar 55 g del prepolímero con 5,5 g del agente de curado. - Coloque el maestro de silicio que contiene múltiples réplicas de la misma micropattern SU-8 en un barco de pesada de plástico grande (10 cm de diámetro de base) y vierte el PDMS desgasificado. Luego, se cura en un horno seco a 60 ° C durante la noche.
NOTA: Los micropatterns son lo suficientemente grandes para ser vistos a simple vista. Cada patrón contiene 8 microcanales (100 μm de ancho x 40 μm de altura x 1 cm de longitud) con un espaciamiento de 40 μm entre ellos ( Figura 1A , 1B ). Se fabricó un molde de oblea de silicio compuesto por material fotorresistente SU-8 horneado duro en una instalación de nanofabricación 27 . Otros enfoques para la preparación principal incluyen ácido fluorhídrico (HF) grabado, agua de alta temperatura grabado, xurografía, y la impresión 3D 28 , 29 , 30 , 31 . CommercTambién se pueden usar fuentes de preparación maestro de silicio. Los patrones para el dispositivo microfluídico fue diseñado con un software de dibujo (ver tabla de materiales). El archivo original que contiene el diseño del patrón se proporciona como un archivo suplementario "Pattern Design.dwg", que se puede proporcionar directamente al fabricante. - Suavemente, despegue el PDMS del maestro de silicio con las manos. Marque los límites de cada micropatrón en rectángulos usando un bisturí quirúrgico y una regla. A continuación, corte el PDMS en bloques.
- Perforar 16 agujeros (8 entradas y 8 salidas por bloque) en ambos extremos de cada microcanal con un punzón de biopsia (diámetro de agujero de 1,0 mm) como se indica en la Figura 3B .
- Aplique cinta sobre cada bloque de PDMS para proteger los micropatrones de daños y polvo. Guárdelos en un recipiente de plástico.
3. Preparación de pequeñas vesículas unilamelares (SUV)
NOTA: Composición bicapa de control negativoes 99,5% en moles de 1-palmitoil-2-oleoil sn-glicero-3-fosfocolina (POPC) y 0,5% en moles orto- Sulforodamina B-1-palmitoil-2-oleoil sn-glicero-3-fosfoetanolamina (O SRB- PAPA). Composición bicapa de prueba es 92,0% en moles de POPC, 0,5 mol% o SRB-POPE, y 7,5% en moles de cualquiera de L-α-fosfatidilinositol-4-fosfato (PI4P) o L-α-fosfatidilinositol-4,5-bifosfato (PI ( 4,5) P $ ₂ $). A continuación se muestra el procedimiento para la preparación de 92.0 mol% POPC, 0,5 mol% o SRB-POPE, y 7,5% en moles de PI (4,5) P 2 SUVs que contiene. La síntesis de O SRB-POPE utilizado en este estudio fue descrito previamente 20.
- Calcular el volumen de POPC, PI (4,5) P 2, y o SRB-POPE que necesita mixto, que representa las siguientes cantidades totales: 2,22 mg de POPC, 0,26 mg de PI (4,5) P 2, y 2,1 x 10 -5 mg de o SRB-POPE.
- Pipetear el volumen calculado de POPC, PI (4,5) P 2 , o SRB-POPE en un único vial de centelleo de vidrio de 20 ml.
NOTA: Las soluciones madre POPC y o SRB-POPE vienen en una solución de cloroformo, mientras que las existencias PI (4,5) P 2 (y otras fosfoinositidas) vienen en una mezcla de disolventes de cloroformo: metanol: agua (20: 9: 1). Para mayor precisión, humedezca la punta de la pipeta con cloroformo antes de usarla. Por seguridad, este paso debe tener lugar dentro de una campana de humo químico. - Se seca la mezcla en una corriente de gas nitrógeno dentro de una campana extractora de humos durante 10 minutos o hasta que el disolvente se evapore y se forme una fina película lipídica en la parte inferior del vial.
- Desecar la mezcla bajo vacío durante ≥ 3 h a una resistencia al vacío de 10 mTorr para eliminar cualquier disolvente orgánico residual.
NOTA: El tiempo de desecación de 3 h es suficiente para la escala de preparación de SUV descrita en este protocolo, la cual es adecuada para 50 experimentos. Si se requiere una escala mayor de preparación de SUV, se puede realizar una desecación durante la noche. - Rehidratar el dr(HEPES 20 mM, NaCl 100 mM, pH 7,0) y colocarlo en un baño ultrasónico a una frecuencia operativa de 35 kHz durante 30 min a TA.
NOTA: La mezcla de las cantidades de lıpidos especificadas en la etapa 3.1 y la adición de 5 ml de tampón corriente en la etapa 3.5 produce una suspensión de vesıculas a 0,5 mg / ml. En esta etapa, la suspensión de vesículas es una mezcla de vesículas unilamelares y multilamelares ( Figura 2B ). La solución aparecerá de color rosa / morado en color y relativamente turbio. - Congelar-descongelar la suspensión de vesículas con nitrógeno líquido y baño de agua a 40 ° C para obtener vesículas unilamelares. Repita el congelamiento-descongelamiento 10 veces.
NOTA: (Opcional) Para asegurar la ausencia de vesículas no unilamelares en la suspensión, se puede aplicar la etapa de centrifugación 32 , 33 . - Extruir la suspensión de vesículas a través de una membrana de policarbonato grabada en pista de 0,10 μm utilizando un extrusor de lípidos (véaseTabla de materiales) para enriquecer para SUVs. Repetir la extrusión 10 veces.
NOTA: Se puede usar un tubo cónico de 15 mL para recoger las vesículas extruidas. La solución debe estar vacía de turbiedad en esta etapa. SUV diámetro se puede confirmar a través de dispersión de luz dinámica (DLS) ( Figura 2C ]. SUVs son los más adecuados para formar SLBs 34 . - Cubra el tubo cónico con papel de aluminio y guárdelo a 4 ° C hasta que esté listo para su uso.
4. Montaje del dispositivo microfluídico
- Pruebe las entradas y salidas del bloque PDMS para bloquear inyectando agua desionizada a través de los orificios usando una botella de lavado con agua. A continuación, seque el bloque PDMS con gas nitrógeno.
NOTA: Este paso también eliminará las partículas de polvo que podrían haber quedado atrapadas dentro y / o entre canales. Si es necesario, limpie previamente los cubreobjetos con gas nitrógeno para eliminar las partículas de polvo. - Coloque el bloque PDMS y el bloque pre-limpiadocubreobjetos (ver sección 1) en el interior del sistema de plasma de oxígeno (véase la tabla de materiales) cámara de la muestra para exponer con plasma de oxígeno durante 45 s con ajuste de potencia a 75 vatios, velocidad de flujo de oxígeno a 10 cm 3 / min, y la fuerza de vacío a 200 mTorr .
- Coloque la superficie modelada del bloque PDMS en contacto con la cubreobjetos inmediatamente después del tratamiento con plasma de oxígeno. Presione suavemente para eliminar cualquier burbuja de aire en los sitios de contacto.
- Coloque el dispositivo en una placa caliente nivelada a 100 ° C durante 3 min para mejorar la unión.
- Utilice un paño húmedo sin pelusa ( por ejemplo, kimwipe) con etanol al 100% para eliminar las partículas de polvo de la parte superior (PDMS) y la parte inferior (vidrio) del dispositivo. A continuación, cinta el dispositivo en la parte superior de una diapositiva de cristal de microscopio.
NOTA: No use una cantidad excesiva de etanol y evite que el etanol entre en los canales de entrada y salida. Se recomienda realizar este paso mientras el dispositivo está todavía caliente para asegurarse de que el etanol se evapora inmediatamente. Micr micrófonoLos portaobjetos de oscopia se pueden almacenar en una solución de etanol al 100% para eliminar el polvo y otros contaminantes.
5. Formación de bicapas lipídicas (SLB)
- Transferir 100 l de PI (4,5) P 2 SUVs que contiene desde el paso 3.8 en un tubo de microcentrífuga de 0,65 ml. Ajustar el pH de la solución a ~ 3.2 añadiendo 6,4 μl de ácido clorhídrico 0,2 N.
NOTA: Confirmar el pH de la solución con un medidor de pH equipado con una sonda de micro-pH (ver tabla de materiales). - Pipetee 10 μl de la solución de SUV con pH ajustado en cada canal a través de la entrada y aplique presión a través de la pipeta hasta que la solución alcance la salida. Quite la punta de la pipeta y dejarla conectada al dispositivo.
- Repita el paso anterior para cada canal e incube el dispositivo durante 10 min a TA.
NOTA: La inyección de vesículas en microcanales se debe realizar inmediatamente después del montaje del dispositivo. - Cortar conjuntos de tubos de entrada y salidaCh 60 cm (tubo de entrada) y 8 cm (tubo de salida) de largo, respectivamente.
NOTA: Cortar el tubo en diagonal y crear un borde afilado, facilita la inserción del tubo en las entradas y salidas. El tubo de salida debe tener forma de arco. La longitud del tubo de entrada puede variar en función de la configuración del microscopio. El diámetro interno del tubo es de 0,05 cm. - Usando pinzas, conecte el conjunto de tubos de salida al dispositivo, y luego cinta el dispositivo en una etapa de microscopio.
- Sumergir un extremo del conjunto de tubos de entrada en 25 mL de solución tampón de funcionamiento contenida en un tubo cónico y grabarlo para asegurarse de que el tubo está asegurado.
- Coloque el tubo cónico en un terreno más alto (~ 20 cm) que el dispositivo para empujar la solución a través de los microcanales a través del flujo por gravedad; Un gato del laboratorio se puede utilizar para lograr esto.
- Para cada tubo de entrada, utilice una jeringa para extraer 1 ml de tampón corriente del extremo libre de la tubería. Retire la punta de la pipeta de la entrada yLibre del tubo de entrada en el dispositivo.
NOTA: Introduzca una gota de buffer de funcionamiento en la entrada para reducir la probabilidad de que se introduzca una burbuja de aire en el canal durante este paso. Después de que el tubo de entrada esté unido al dispositivo, utilice un paño sin pelusa para eliminar el exceso de tampón. - Repita el paso anterior para conectar todas las piezas de tubo de entrada al dispositivo.
NOTA: El flujo de buffer de funcionamiento a través de los canales ayuda a eliminar el exceso de vesículas y equilibrar la bicapa a condiciones experimentales. - Abra el software de control del microscopio (ver tabla de materiales). En el panel izquierdo, haga clic en la pestaña "Microscopio" y elija el objetivo "10X".
- Haga clic en "Live" y luego "Alexa 568" iconos de imagen en la barra de herramientas. Utilizando las perillas de ajuste fino y de curso, enfoque los microcanales.
- Explore el dispositivo para comprobar la calidad de los SLB y los canales ( Figura 8 ). Entonces,Haga clic en el icono de imagen "FL Shutter Closed" en la barra de herramientas.
- Haga clic en la pestaña "Adquisición" y en "Ajustes básicos" seleccione "Tiempo de exposición". Ajuste el tiempo de exposición a "200 ms".
- En el panel izquierdo, haga clic en "Adquisición multidimensional" y en el menú de filtros seleccione el canal rojo (marcado como "Alexa 568"). A continuación, haga clic en el menú "lapso de tiempo", ajuste el intervalo de tiempo a 5 min, duración a 30 min y haga clic en "Inicio".
NOTA: Basándose en la duración (30 min) y en el caudal (~ 1,0 μL / min), se utilizan 30 μl de tampón corriente para equilibrar el SLB dentro de un canal, es decir , se utilizan 240 μl de tampón de funcionamiento para los 8 canales. - Seleccione la herramienta "Círculo" en la pestaña "Medir" y dibuje un círculo en cualquier canal. Haga clic con el botón derecho del ratón mientras se selecciona el círculo y seleccione "Propiedades". En la pestaña "Perfil", marque "todo T" para ver elIntensidad de fluorescencia en función del tiempo.
- Asegúrese de que esta curva alcanza una meseta, lo que indica equilibrio, antes de pasar al siguiente paso.
- Baje la solución tampón a una tierra igual como el dispositivo para detener el flujo.
- Guarde el archivo de lapso de tiempo.
6. Ensayo de la interacción del dominio PH del PLC-δ1 con los SLBs que contienen PI (4,5) P 2
- Preparar diluciones del dominio PH usando el tampón de funcionamiento como un diluyente.
NOTA: Ya que hay ocho canales en el dispositivo, utilice al menos dos de ellos para controles en blanco. Escoja los extremos de cada lado y use los canales restantes para diluciones de proteínas. Se ensayaron las siguientes concentraciones de dominio de PH: 0,10, 0,25, 0,50, 1,00 y 2,50 mu M. Aproximadamente 200 μl de cada dilución fue suficiente para alcanzar el equilibrio en 30 minutos. - Uno a la vez, separe cada tubo de salida y aplique 200 μl de cada dilución de proteína enEl canal de salida utilizando una pipeta. No aplique ninguna presión, deje que la gravedad haga el trabajo. Separar la punta de la pipeta y dejarla unida al dispositivo microfluídico.
- Repita el paso anterior para cada canal y asegúrese de que no se introduzcan burbujas de aire en los canales durante este proceso.
- Baje el tubo de entrada a un suelo por debajo del dispositivo microfluídico para comenzar a fluir la proteína a través de los microcanales. Tape el extremo libre de la tubería a un contenedor de residuos. Flujo de las diluciones de proteínas durante 30 min.
NOTA: Esto invertirá el flujo y permitirá que la proteína se introduzca en los canales. El tiempo de equilibrado y el volumen de diluciones de proteínas necesarias dependerá de la afinidad de la interacción. - En el panel izquierdo del software, bajo la pestaña "Lapso de tiempo", haga clic en "Iniciar" para comenzar la creación de imágenes de nuevo.
- Repita el paso 5.15 para asegurarse de que se alcanza el equilibrio. Cuando el experimento esté completo, guarde el lapso de tiempoarchivo.
7. Evaluación de la Fluidez de la Membrana
NOTA: Los experimentos de recuperación de fluorescencia después del fotoblanqueo (FRAP) deben realizarse con cada nuevo lote de SUVs y cubreobjetos de vidrio limpiado para asegurar que los SLBs son fluidos.
- Siga el procedimiento para limpiar los cubreobjetos de vidrio (1.1-1.6), fabricando bloques PDMS micropatternados (2.1-2.5), preparando SUV (3.1-3.8), ensamblando el dispositivo microfluídico (4.1-4.5) y formando SLBs (5.1-5.17) como anteriormente Descrito.
- Enfoque el microscopio en la bicapa, ajuste el tiempo de exposición a 200 ms y el binning a 1.
- Foto-blanquear un punto circular utilizando un haz láser de 532 nm, 2 mW (radio de 13 μM). Inmediatamente después del fotoblanqueo, comience a capturar una serie de imágenes cada 3 s durante los primeros 45 s, seguido de un intervalo de 30 s durante el resto del tiempo (15 min de duración total). Una vez completada la adquisición de datos, guarde el archivo de tiempo de espera.
- Seleccione el blanqueado aUtilizando la herramienta de dibujo circular para extraer los valores de intensidad de fluorescencia en función del tiempo. Además, seleccione dos áreas más cercanas, una correspondiente a una región no blanqueada y una correspondiente a una región sin ningún SLB.
- Calcular la recuperación de fluorescencia ( y ) utilizando la siguiente ecuación:
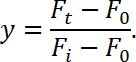
NOTA: Aquí F t representa la intensidad de la región blanqueada en función del tiempo, F i representa la intensidad de fluorescencia antes del blanqueo (utilice "1" como valor normalizado en este caso); F 0 es la intensidad de fondo. - Trazar la recuperación de fluorescencia (eje y) en función del tiempo (eje x) para generar una curva FRAP. Luego, ajuste los datos a una sola función exponencial como sigue,
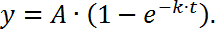
NOTA: Aquí A representa la fracción móvil y k es la constante cinética de la fracción móvil, que se utiliza para calcular el tiempo medio de recuperación ( t 1/2 ):
- Utilice el t 1/2 para calcular la constante de difusión ( D ):

NOTA: Aquí R representa el radio del rayo láser (13 μm). La constante de difusión para una bicapa fluida debe ser ≥1,0 μm 2 / s.
8. Procesamiento de datos
NOTA: La rutina del análisis de datos dependerá del microscopio, del software de procesamiento de imágenes y del software de ajuste de curva que se esté utilizando.
- Abra los archivos time lapse de antes (desde el paso 5.17) y después (desde el paso 6.6) titulando el dominio PH. Ir al último cuadro de cada archivo de lapso de tiempo y hacer una línea de exploración a través de todos los microcanales para obtener el fluorescEn función de la distancia en píxeles ( Figura 6A ). Transfiera los datos a un programa de hoja de cálculo.
- Para cada microcanal (antes y después de la adición del dominio PH), muestre algunos datos dentro del canal ya cada lado del canal que representa la línea de base ( Figura 7A ).
NOTA: La intensidad de fluorescencia del canal en blanco debería haber permanecido sin cambios. Todos los otros canales están normalizados al canal en blanco, por lo que es crucial tener dos canales en blanco y asegurarse de que sus intensidades de fluorescencia permanecen coherentes entre sí. - Aplique la siguiente fórmula para normalizar los datos al canal en blanco; Se proporciona un cálculo de muestra en la figura 7B .

NOTA: Aquí, la proteína ΔF representa la intensidad de fluorescencia restante de fondo del canal de proteína, Δ; F en blanco representa el fondo se resta la intensidad de fluorescencia del canal en blanco, pre y post se refieren a antes y después de las etapas de valoración de proteínas, y α representa el factor de corrección que es la relación de las intensidades de fluorescencia del canal de la proteína y el canal en blanco ([ F proteína / F en blanco ] pre ) en una etapa de titulación pre-proteína. Puesto que la intensidad de fluorescencia disminuye como una función de la concentración de dominio PH, los datos normalizados serán negativos en valor. - Utilizando un software de ajuste de curvas (véase Tabla de Materiales ), trace la fluorescencia normalizada en función de la concentración del dominio PH y ajuste a una isoterma de Langmuir ( Figura 6C ):
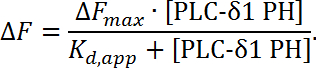
NOTA: Aquí ΔF representa el cambio en la intensidad de fluorescencia en relación con el cambio máximo en fluo(ΔF max ), mientras que el K d, app representa la constante de disociación aparente, que es igual a la concentración de proteína en masa ( [PLC-δ1 PH] ) a la que el 50% de cobertura ( Complejo ligado a la membrana). Esta es una medición de enlace basada en el equilibrio de modo que los datos normalizados se ajustan a una isoterma simple de Langmuir para extraer un parámetro experimental aparente K d, app . Basado en el software de ajuste de la K d, aplicación para PLC-δ1 PH-PI (4,5) P 2 interacción es de 0,39 ± 0,05? M (Figura 6B).
Access restricted. Please log in or start a trial to view this content.
Resultados
Se utilizó el ensayo de modulación del pH para estudiar la PLC-δ1 dominio PH-PI (4,5) P 2 interacción dentro de un PIP-on-a-chip microdevice ( Figura 1 ]. A través de un protocolo detallado, hemos demostrado cómo preparar y ensamblar los componentes del dispositivo microfluídico, hacer pequeñas vesículas unilamelares (SUVs) ( Figura 2 ), forma SLBs dentro de un dispositivo ( Figura 3
Access restricted. Please log in or start a trial to view this content.
Discusión
Cada variante PIP, aunque a bajas concentraciones, está presente en la superficie citosólica de organelos específicos donde contribuyen al establecimiento de una composición física única y especificidad funcional de la membrana organelar 1 . Uno de los usos más importantes de los PIPs es como una plataforma de acoplamiento específica para la multitud de proteínas que requieren localización subcelular específica y / o la activación [ 6 ,
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
DS y CEC fueron apoyados, en parte, por la concesión AI053531 (NIAID, NIH); SS y PSC fueron apoyados por la subvención N00014-14-1-0792 (ONR).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Coverslip | |||
| Glass Coverslips: Rectangles | Fisher Scientific | 12-544B | 22 x 40 x 0.16 - 0.19 mm, No. 1 1/2; Borosilicate Glass |
| 7X Cleaning Solution | MP Biomedicals | 976670 | Detergent |
| PYREX Crystallizing Dish | Corning | 3140-190 | Borosilicate glass dish with a flat bottom; Diameter x Height (190 x 100 mm); Distributor: VWR (89090-700) |
| Sentry Xpress 2.0 | Paragon Industries | SC-2 | Kiln |
| Name | Company | Catalog Number | Comments |
| PDMS | |||
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 4019862 | Polydimethylsiloxane (PDMS); Distributor: Ellsworth Adhesives |
| PYREX Desiccator | VWR | 89134-402 | Vacuum Rated |
| Biopsy punch | Harris | 15110-10 | Harris Uni-Core; 1.0 mm diameter; Miltex Biopsy Punch with Plunger (Cat. No. 15110-10) can be used as an alternative |
| Name | Company | Catalog Number | Comments |
| Device | |||
| Plasma Cleaning System | PlasmaEtch | PE25-JW | 2-stage Direct Drive Oil Vacuum Pump, O2 service (Krytox Charged) |
| Digital Hot Plate | Benchmark | H3760-H | Purchased through Denville Scientific (Cat. No. 1005640) |
| Frosted Micro Slides | VWR | 48312-003 | Frosted, Selected, and Precleaned; Made of Swiss Glass; Thickness: 1 mm; Dimensions: 75 x 25 mm; GR 144 |
| Name | Company | Catalog Number | Comments |
| Mold | |||
| AutoCAD | Autodesk | v.2016 | Drafting software for the photomask design |
| Photomask | CAD/Art Services | N/A | Design with black background and clear features was printed at 20k dpi resolution on a transparent mask (5 x 7 in) by CAD/Art Services |
| Silicone Wafers | University Wafer | 1575 | Prime Grade, Single Side Polished; 100 mm (4 inch) Diameter; 525 um Thickness |
| SU-8 50 | MicroChem Corp. | N/A | Negative Tone Photoresist; Penn State Nanofabrication Facility Property |
| SU-8 Developer | MicroChem Corp. | N/A | Penn State Nanofabrication Facility Property |
| Name | Company | Catalog Number | Comments |
| SUV | |||
| 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine | Avanti Polar Lipids | 850457C | POPC |
| L-α-phosphatidylinositol-4-phosphate | Avanti Polar Lipids | 840045X | PI4P |
| L-α-phosphatidylinositol-4,5-bisphosphate | Avanti Polar Lipids | 840046X | PI(4,5)P2 |
| 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphoethanolamine | Avanti Polar Lipids | 850757C | POPE; Required for the synthesis of oSRB-POPE |
| Lissamine Rhodamine B Sulfonyl Chloride (mixed isomers) | ThermoFisher Scientific | L-20 | Required for the synthesis of oSRB-POPE |
| pH Sensitive Fluorescent Lipid Probe (oSRB-POPE) | In-house | N/A | In-house Synthesis (Huang D. et al. 2013) |
| Glass Scintillation Vial | VWR | 66022-065 | 20 mL volume capacity |
| Aquasonic 250D | VWR | N/A | Ultrasonic Water Bath |
| Nuclepore Track-Etched Membranes | Whatman | 110605 | Polycarbonate Membrane; Diameter: 25 mm; Pore Size: 0.1 um; Distributor: Sigma-Aldrich |
| Chloroform | VWR | CX1054-6 | HPLC grade |
| LIPEX Extruder | Transferra Nanosciences | T.001 | LIPEX 10 mL Thermobarrel Extruder |
| Viscotek 802 DLS | Malvern Instruments | N/A | Dynamic Light Scattering; Penn State X-Ray Crystallography Facility Property |
| Name | Company | Catalog Number | Comments |
| Data Analysis | |||
| GraphPad Prism | GraphPad Software | v.6 | Curve-fitting software for data analysis |
| Name | Company | Catalog Number | Comments |
| Microscope | |||
| Axiovert 200M Epifluorescence Microscope | Carl Zeiss Microscopy | N/A | Microscope |
| AxioCam MRm Camera | Carl Zeiss Microscopy | N/A | Camera |
| X-Cite 120 | Excelitas Technologies | N/A | Light Source |
| Alexa 568 Filter Set | Carl Zeiss Microscopy | N/A | Ex/Em 576/603 nm |
| AxioVision LE64 v.4.9.1.0 Software | Carl Zeiss Microscopy | N/A | Image Processing Software |
| Name | Company | Catalog Number | Comments |
| Other | |||
| Tips | VWR | 10034-132 | 200 uL pipette tips; Thin and smooth tip for applying the protein solution into the microfluidic channel |
| Tips | VWR | 53509-070 | 10 uL pipette tips; Thin and smooth tip for applying the vesicle solution into the microfluidic channel |
| Orion Star A321 pH meter | Thermo Scientific | STARA3210 | pH meter |
| Orion micro pH probe | Thermo Scientific | 8220BNWP | micro pH probe |
| N-(2-Hydroxyethyl)-Piperazine-N'-(2-Ethanesulfonic Acid) | VWR | VWRB30487 | HEPES, Free Acid |
| Sodium Chloride | VWR | BDH8014-2.5KGR | NaCl |
| Tubing | Allied Wire & Cable | TFT-200-24 N | Internal Diameter: 0.020-0.026 inches (0.051-0.066 cm); Wall Thickness: 0.010 inches (0.025 cm); Flexible Polytetrafluoroethylene Thin-Wall Tubing; Natural Color |
| Nitrogen Gas - Industrial | Praxair | N/A | Local Provider |
| Oxygen Gas - Industrial | Praxair | N/A | Local Provider |
| Liquid Nitrogen | Praxair | N/A | Local Provider |
Referencias
- Di Paolo, G., De Camilli, P. Phosphoinositides in cell regulation and membrane dynamics. Nature. 443 (7112), 651-657 (2006).
- Shewan, A., Eastburn, D. J., Mostov, K. Phosphoinositides in cell architecture. Cold Spring Harb Perspect Biol. 3 (8), a004796(2011).
- Picas, L., Gaits-Iacovoni, F., Goud, B. The emerging role of phosphoinositide clustering in intracellular trafficking and signal transduction. F1000Res. 5, (2016).
- Lystad, A. H., Simonsen, A. Phosphoinositide-binding proteins in autophagy. FEBS Lett. 590 (15), 2454-2468 (2016).
- Balla, T. Phosphoinositides: Tiny lipids with giant impact on cell regulation. Physiol Rev. 93, 1019-1137 (2013).
- Lemmon, M. A. Membrane recognition by phospholipid-binding domains. Nat Rev Mol Cell Biol. 9 (2), 99-111 (2008).
- Kutateladze, T. G. Translation of the phosphoinositide code by PI effectors. Nat Chem Biol. 6 (7), 507-513 (2010).
- Harlan, J. E., Hajduk, P. J., Yoon, H. S., Fesik, S. W. Pleckstrin homology domains bind to phosphatidylinositol-4,5-bisphosphate. Nature. 371 (6493), 168-170 (1994).
- Garcia, P., et al. The pleckstrin homology domain of phospholipase C-delta 1 binds with high affinity to phosphatidylinositol 4,5-bisphosphate in bilayer membranes. Biochemistry. 34 (49), 16228-16234 (1995).
- Lemmon, M. A., Ferguson, K. M., O'Brien, R., Sigler, P. B., Schlessinger, J. Specific and high-affinity binding of inositol phosphates to an isolated pleckstrin homology domain. Proc Natl Acad Sci U S A. 92 (23), 10472-10476 (1995).
- Flesch, F. M., Yu, J. W., Lemmon, M. A., Burger, K. N. Membrane activity of the phospholipase C-delta1 pleckstrin homology (PH) domain. Biochem J. 389, 435-441 (2005).
- Narayan, K., Lemmon, M. A. Determining selectivity of phosphoinositide-binding domains. Methods. 39 (2), 122-133 (2006).
- Scott, J. L., Musselman, C. A., Adu-Gyamfi, E., Kutateladze, T. G., Stahelin, R. V. Emerging methodologies to investigate lipid-protein interactions. Integr Biol (Camb). 4 (3), 247-258 (2012).
- Dowler, S., Currie, R. A., Downes, C. P., Alessi, D. R. DAPP1: A dual adaptor for phosphotyrosine and 3-phosphoinositides. Biochem J. 342, 7-12 (1999).
- He, J., et al. Molecular basis of phosphatidylinositol 4-phosphate and ARF1 GTPase recognition by the FAPP1 pleckstrin homology (PH) domain. J Biol Chem. 286 (21), 18650-18657 (2011).
- Ceccarelli, D. F., et al. Non-canonical interaction of phosphoinositides with pleckstrin homology domains of Tiam1 and ArhGAP9. J Biol Chem. 282 (18), 13864-13874 (2007).
- Huang, S., Gao, L., Blanchoin, L., Staiger, C. J. Heterodimeric capping protein from Arabidopsis is regulated by phosphatidic acid. Mol Biol Cell. 17 (4), 1946-1958 (2006).
- Yu, J. W., et al. Genome-eide analysis of membrane targeting by S. cerevisiae pleckstrin homology domains. Mol Cell. 13 (5), 677-688 (2004).
- Jung, H., Robison, A. D., Cremer, P. S. Detecting protein-ligand binding on supported bilayers by local pH modulation. J Am Chem Soc. 131 (3), 1006-1014 (2009).
- Huang, D., Zhao, T., Xu, W., Yang, T., Cremer, P. S. Sensing small molecule interactions with lipid membranes by local pH modulation. Anal Chem. 85 (21), 10240-10248 (2013).
- Saxena, A., et al. Phosphoinositide binding by the pleckstrin homology domains of Ipl and Tih1. J Biol Chem. 277 (51), 49935-49944 (2002).
- Knödler, A., Mayinger, P. Analysis of phosphoinositide-binding proteins using liposomes as an affinity matrix. Biotechniques. 38 (6), 858-862 (2005).
- Baumann, M. K., Swann, M. J., Textor, M., Reimhult, E. Pleckstrin homology-phospholipase C-delta1 interaction with phosphatidylinositol 4,5-bisphosphate containing supported lipid bilayers monitored in situ with dual polarization interferometry. Anal Chem. 83 (16), 6267-6274 (2011).
- Saliba, A. E., et al. A quantitative liposome microarray to systematically characterize protein-lipid interactions. Nat Methods. 11 (1), 47-50 (2014).
- Arauz, E., Aggarwal, V., Jain, A., Ha, T., Chen, J. Single-molecule analysis of lipid-protein interactions in crude cell lysates. Anal Chem. 88 (8), 4269-4276 (2016).
- Best, Q. A., Xu, R., McCarroll, M. E., Wang, L., Dyer, D. J. Design and investigation of a series of rhodamine-based fluorescent probes for optical measurements of pH. Org Lett. 12 (14), 3219-3221 (2010).
- Lee, J., Choi, K. H., Yoo, K. Innovative SU-8 lithography techniques and their applications. Micromachines. 6 (1), 1-18 (2014).
- Poyton, M. F., Sendecki, A. M., Cong, X., Cremer, P. S. Cu(2+) binds to phosphatidylethanolamine and increases oxidation in lipid membranes. J Am Chem Soc. 138 (5), 1584-1590 (2016).
- Karasek, P., Grym, J., Roth, M., Planeta, J., Foret, F. Etching of glass microchips with supercritical water. Lab Chip. 15 (1), 311-318 (2015).
- Thomas, M. S., et al. Print-and-peel fabrication for microfluidics: what's in it for biomedical applications? Ann Biomed Eng. 38 (1), 21-32 (2010).
- Waheed, S., et al. 3D printed microfluidic devices: enablers and barriers. Lab Chip. 16 (11), 1993-2013 (2016).
- Axmann, M., Schutz, G. J., Huppa, J. B. Single molecule fluorescence microscopy on planar supported bilayers. J Vis Exp. (105), e53158(2015).
- Barenholz, Y., et al. A simple method for the preparation of homogeneous phospholipid vesicles. Biochemistry. 16 (12), 2806-2810 (1977).
- Castellana, E. T., Cremer, P. S. Solid supported lipid bilayers: From biophysical studies to sensor design. Surface Science Reports. 61 (10), 429-444 (2006).
- Hamai, C., Yang, T., Kataoka, S., Cremer, P. S., Musser, S. M. Effect of average phospholipid curvature on supported bilayer formation on glass by vesicle fusion. Biophys J. 90 (4), 1241-1248 (2006).
- Tero, R. Substrate effects on the formation process, structure and physicochemical properties of supported lipid bilayers. Materials. 5 (12), 2658-2680 (2012).
- Ferguson, K. M., Lemmon, M. A., Schlessinger, J., Sigler, P. B. Structure of the high affinity complex of inositol trisphosphate with a phospholipase C pleckstrin homology domain. Cell. 83 (6), 1037-1046 (1995).
- Simonsson, L., Hook, F. Formation and diffusivity characterization of supported lipid bilayers with complex lipid compositions. Langmuir. 28 (28), 10528-10533 (2012).
- Cong, X., Poyton, M. F., Baxter, A. J., Pullanchery, S., Cremer, P. S. Unquenchable surface potential dramatically enhances Cu(2+) binding to phosphatidylserine lipids. J Am Chem Soc. 137 (24), 7785-7792 (2015).
- Robison, A. D., et al. Polyarginine interacts more strongly and cooperatively than polylysine with phospholipid bilayers. J Phys Chem B. 120 (35), 9287-9296 (2016).
- Robison, A. D., Huang, D., Jung, H., Cremer, P. S. Fluorescence modulation sensing of positively and negatively charged proteins on lipid bilayers. Biointerphases. 8 (1), 1(2013).
- Tabaei, S. R., et al. Formation of cholesterol-rich supported membranes using solvent-assisted lipid self-assembly. Langmuir. 30 (44), 13345-13352 (2014).
- Johnson, S. J., et al. Structure of an adsorbed dimyristoylphosphatidylcholine bilayer measured with specular reflection of neutrons. Biophys J. 59 (2), 289-294 (1991).
- Koenig, B. W., et al. Neutron reflectivity and atomic force microscopy studies of a lipid bilayer in water adsorbed to the surface of a silicon single crystal. Langmuir. 12 (5), 1343-1350 (1996).
- Tanaka, M., Sackmann, E. Polymer-supported membranes as models of the cell surface. Nature. 437 (7059), 656-663 (2005).
- Renner, L., et al. Supported lipid bilayers on spacious and pH-responsive polymer cushions with varied hydrophilicity. J Phys Chem B. 112 (20), 6373-6378 (2008).
- Wagner, M. L., Tamm, L. K. Tethered polymer-supported planar lipid bilayers for reconstitution of integral membrane proteins: Silane-polyethyleneglycol-lipid as a cushion and covalent linker. Biophys J. 79 (3), 1400-1414 (2000).
- Pace, H., et al. Preserved transmembrane protein mobility in polymer-supported lipid bilayers derived from cell membranes. Anal Chem. 87 (18), 9194-9203 (2015).
- Braunger, J. A., Kramer, C., Morick, D., Steinem, C. Solid supported membranes doped with PIP2: Influence of ionic strength and pH on bilayer formation and membrane organization. Langmuir. 29 (46), 14204-14213 (2013).
- Paridon, P. A., de Kruijff, B., Ouwerkerk, R., Wirtz, K. W. Polyphosphoinositides undergo charge neutralization in the physiological pH range: A 31P-NMR study. Biochim Biophys Acta. 877 (1), 216-219 (1986).
- Liu, C., Huang, D., Yang, T., Cremer, P. S. Monitoring phosphatidic acid formation in intact phosphatidylcholine bilayers upon phospholipase D catalysis. Anal Chem. 86 (3), 1753-1759 (2014).
- Saad, J. S., et al. Structural basis for targeting HIV-1 Gag proteins to the plasma membrane for virus assembly. Proc Natl Acad Sci U S A. 103 (30), 11364-11369 (2006).
- Hsu, N. Y., et al. Viral reorganization of the secretory pathway generates distinct organelles for RNA replication. Cell. 141 (5), 799-811 (2010).
- Del Campo, C. M., et al. Structural basis for PI(4)P-specific membrane recruitment of the Legionella pneumophila effector DrrA/SidM. Structure. 22 (3), 397-408 (2014).
- Kolli, S., et al. Structure-function analysis of vaccinia virus H7 protein reveals a novel phosphoinositide binding fold essential for poxvirus replication. J Virol. 89 (4), 2209-2219 (2015).
- Cho, N. J., et al. Phosphatidylinositol 4,5-bisphosphate is an HCV NS5A ligand and mediates replication of the viral genome. Gastroenterology. 148 (3), 616-625 (2015).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados