Method Article
完整蛋白质复合物的化学交联和质谱分析研究多亚基蛋白质组合体的体系结构
摘要
蛋白质复合体的结构对其功能至关重要。结合各种质谱技术证明了研究其组装的强大功能。我们提供化学交联和本机质谱的协议, 并展示这些互补技术如何帮助阐明 multi-subunit 蛋白组件的结构。
摘要
蛋白质与他们的配体相互作用形成活跃的和动态的组合, 执行各种细胞功能。因此, 阐明这些相互作用是理解细胞过程的基础。然而, 许多蛋白质复合体是动态组件, 不能通过传统的结构技术来获取。质谱分析有助于这些组件的结构研究, 特别是各种质谱技术的结合, 为它们的结构安排提供了宝贵的见解。
在本文中, 我们描述了两种互补质谱技术的应用和组合, 即化学交联结合质谱和本机质谱。化学交联是用化学试剂在近距离接触氨基酸的共价键。在消化与蛋白酶, 交联的 di 肽通过质谱和蛋白质相互作用站点被发现。在质谱仪的气相中, 对完整的蛋白质组合进行了分析。它揭示了蛋白质 stoichiometries 以及蛋白质和配体相互作用。因此, 这两种技术都提供了有关蛋白质-配体组件结构的补充信息, 并且在先前的研究中证明了它们的结合力。
引言
蛋白质组装的结构研究对细胞过程的理解变得尤为重要。因此, 许多技术已经开发和改进的结构生物学1。然而, 这些技术有时是有限的, 因为其应用的大小, 灵活性, 或异质性的蛋白质复合物的调查。质谱法可以处理这些挑战, 因此, 成为一个强大的工具在结构生物学2,3,4,5。然而, 质谱法的最大优点是能够在复杂和异构混合物中明确识别蛋白质,6。为此, 蛋白质通常是用 endoproteinases 消化的, 由此产生的肽混合物通过液相色谱法分离, 直接洗到质谱仪中。肽质量随后被确定和前体离子被选择为进一步分裂的多肽。然后在已知的数据库中通过搜索肽和相应的片段质量来识别蛋白质。这个过程不仅允许识别多肽/蛋白质, 而且他们的修饰的修改, 导致前体肽和片段离子的大规模转移进行修改7。结构质谱中的许多技术都是基于这一原则4,8。例如, 标记技术如氢氘交换9,10, 化学标记策略11,12, 或羟基根脚打印13,14, 在一定条件下洞察蛋白质的表面可及性。
另一种技术是 (化学) 交联, 涉及氨基酸的共价键联系通过其功能组密切接近。为此, 化学试剂、UV 激活试剂或氨基酸被使用15,16。交联后, 通常用蛋白酶水解蛋白质, 用液相色谱-耦合质谱法分析交叉连接的 di 肽。通过数据库检索来识别交联产品, 但是, 需要使用专门的软件, 连接各种蛋白质和不同区域的肽序列17,18,19。使用化学 cross-linkers 的好处是, 它可以被用于几乎每一个感兴趣的蛋白质复合体, 不需要加入紫外线-激活氨基酸, 这只能实现时, 表达的蛋白质感兴趣的宿主细胞。因此, 交联是一个多才多艺的工具, 并成功地使用了许多结构研究, 甚至大型蛋白质组件20。
另一方面, 完整的蛋白质复合物的质谱学 (有时被称为 "本机质谱"), 涉及对完整的蛋白质和蛋白质复合物的分析而不水解为多肽。它揭示了蛋白质复合物的组成、异质性、化学计量、拓扑和亚基相互作用21,22。结合离子流动性, 本机质谱进一步允许确定其构象23,24。这使得它成为一个强有力的工具, 对蛋白质复合物的结构研究, 难以评估传统的结构技术。然而, 本机质谱需要分析缓冲, 以保持价蛋白相互作用在电喷雾电离。这通常是通过使用水, 挥发性的缓冲区, 如醋酸铵25。此外, 仪器的修改是必要的, 以防止分离过程中传输到质谱仪的气相中26。应用这种方法, 对许多 (大) 蛋白复合物进行了分析。令人印象深刻的例子是研究完整的核糖体27, ATP 酶28, 或病毒29。
当地质谱和交联的结合在以前的研究中证明特别成功。例如, stoichiometries 的伴侣配合物, 包括一个意想不到的 Hsp70 二聚体, 可以获得从质谱实验的完整的蛋白质复合物, 而化学交联揭示了蛋白质的安排程序集30,31。在不同的研究中, 研究了修饰修饰对完整的 ATP 合酶复合体的影响。本机质谱法提供了洞察到蛋白质复合体的稳定性存在或缺乏磷酸化或乙酰性32,33。一个比较交联策略34然后揭示了蛋白质复合体在不同条件下的构象变化。
在这里, 我们提供的蛋白质鉴定的协议质谱, (化学) 交联, 和本机质谱, 包括数据分析和解释 (图 1)。在两个良好蛋白配合物的帮助下, 从这些方法中获得的互补结果的结合显示为35。我们的协议可以适用于任何蛋白质组装, 可以在一定纯度和浓度纯化。这种方法在某些情况下受交联数据的数据分析限制,即, 所使用的数据库的大小决定了所需的搜索空间和时间。此外, 通常需要手动验证已识别的交, 并进一步减少输出。本机质谱主要受样品质量的限制,例如, 在纯化过程中使用的缓冲器和加, 以及通过水和挥发性缓冲液交换它们的可能性。然而, 为结构分析纯化的蛋白质复合物通常具有成功分析我们的协议所需的质量。
研究方案
1. 蛋白质复合物的纯化
- 根据优化的标准协议准备蛋白质复合物。
注意: 该协议是在这里演示的 RvB1/B2 复合体从毛 thermophilum和甲磷酸合成酶 (CPS) 从大肠杆菌。RvB1/B2 和 CPS 被纯化为描述的36,37。每个蛋白质复合体都需要一个单独的纯化协议。相应地调整协议。无胺缓冲剂, 如磷酸盐缓冲盐水 (PBS) 或 2-4-(2-羟乙基) 嗪-1-基] 磺酸 (HEPES) 应用于化学交联。如果可能, 在净化过程中更换缓冲液。
注意: 不要使用任何方法或试剂来扰乱蛋白质复合体的原生组装。
2. 质量谱蛋白鉴定
-
凝胶电泳
注: 有不同的凝胶系统可用, 每个实验室使用自己的设置。根据凝胶系统调整条件。在整个协议中佩戴手套和实验外套作为角是质谱分析中最常见的污染物。- 应用7.5 µL 4x 样品缓冲器和3µL 10x 还原剂 (最终浓度 50 mM 糖 ()) 到20µL 蛋白样品。使用10µM CPS 或2.5 µM RvB1/B2。旋转下来1分钟在 16200 x g 和热为10分钟在70° c。
注: 当使用不同的凝胶系统时, 调节所需的体积和加热温度。 - 准备一个 4-12% 梯度凝胶电泳。用清水稀释连续缓冲液20次, 填充电泳室。使用 2-(n-吗) 磺酸 (MES) 的蛋白质的更小的分子量 (2-200 kDa) 和 3-(n-吗) 磺酸 (拖把) 缓冲为更大的蛋白质 (14-200 kDa)。将0.5 毫升抗氧化剂添加到内腔。
注: 当使用不同的凝胶系统准备运行的缓冲和凝胶根据制造商的协议。 - 在第一个腔中加载一个合适的蛋白质标记物 (pre-stained, 不, 不同的分子大小), 并将蛋白质样本加载到剩余的腔中。
- 分离蛋白35分钟 (MES) 或50分钟 (拖把) 在 200 v
注: 调整不同凝胶系统的电泳时间和电压。 - 要染色的蛋白质带, 转移到一个凝胶染色盒和覆盖的凝胶与水基马斯亮染色溶液。在一个水平的凝胶振动筛上通宵和室温孵育。
- 用水代替染色液, Destain 凝胶。重复此步骤几次 (大约3-5 次), 直到凝胶背景显示清楚。
- 应用7.5 µL 4x 样品缓冲器和3µL 10x 还原剂 (最终浓度 50 mM 糖 ()) 到20µL 蛋白样品。使用10µM CPS 或2.5 µM RvB1/B2。旋转下来1分钟在 16200 x g 和热为10分钟在70° c。
-
凝胶消化
注意: 为了避免污染, 请在整个消化协议中使用 HPLC 级溶剂, 并采取以下步骤 (即, 质谱分析)。在使用和过滤水和碳酸氢铵之前, 准备好所有的解决方案。- 用手术刀切割由凝胶中的蓝色马斯亮着色的蛋白质带。小心地将蛋白质带切成大约1毫米 x 1 毫米的小块. 用不同的蛋白质带水冲洗手术刀。用水和乙腈 (ACN) 清洗凝胶带。
- 用带碘的烷基化半胱氨酸残留物减少二硫化硫键, 用胰蛋白酶消化蛋白质, 如前所述38;通常酵素: 蛋白质比率1:20 由1:100 使用。在消化协议中使用100毫米碳酸氢铵。
- 两步提取多肽。
- 首先用碳酸氢铵和 ACN 孵育凝胶片, 并收集含有多肽的上清液。第二, 用 5% (v/v) 甲酸和 ACN 孵育凝胶件。每一个步骤孵育15分钟。结合两个清。通过在真空离心机中蒸发溶剂来干燥提取的多肽。
注: 干肽可贮存在-20 ° c 以下几个月。
- 首先用碳酸氢铵和 ACN 孵育凝胶片, 并收集含有多肽的上清液。第二, 用 5% (v/v) 甲酸和 ACN 孵育凝胶件。每一个步骤孵育15分钟。结合两个清。通过在真空离心机中蒸发溶剂来干燥提取的多肽。
-
液相色谱-耦合质谱法
- 在 2% ACN/0.1% 甲酸中溶解干的多肽。将超声溶液中的多肽溶解2-3 分钟, 在离心机上旋转 16200 x g, 30 分钟, 将样品转移到样瓶中。
注意: 根据蛋白质量调整音量。对于 well-stained 马斯亮带, 我们使用20µL。 - 使用样向纳米 LC/ms 系统注入5µL 的样品。将肽混合物加载到反相 C18 柱 (C18, 150 µm 识别, 2 厘米, 5 µm 孔径) 到淡化, 并将肽类集中在网上。
- 使用 0.1% (v/v) 甲酸作为流动相 A 和 80% ACN/0.1% (v/v) 甲酸作为移动阶段 b. 在反相 C18 分析柱上分离肽 (C18, 75 µm 身份, 50 厘米, 3 µm 孔大小) 使用梯度 4-80% B (含0.1% 甲酸) 在 300 nL/最小65分钟
注: nanospray 离子源用于将洗肽转移到质谱仪中。 - 使用 (典型) MS 条件: 1.6 kV 的喷雾电压;毛细管温度为250° c;归一化碰撞能量为30。在数据相关模式下操作质谱仪。
- 获取质量分析器 (例如, orbitrap) (m/z 350−1,600) 的 MS 频谱, 分辨率为 7万, 而自动增益控制目标为 3 x 106。在 1 x 105的自动增益控制目标中, 选择20最强烈的 HCD 碎片离子。在三十年代动态排除先前选定的离子. 排除单个带电离子以及无法识别电荷状态的离子。
注: 使用锁质量选项39进行了质谱的内部校准。
- 在 2% ACN/0.1% 甲酸中溶解干的多肽。将超声溶液中的多肽溶解2-3 分钟, 在离心机上旋转 16200 x g, 30 分钟, 将样品转移到样瓶中。
-
数据库搜索
注意: 有不同的软件可用于数据库搜索。例如, MaxQuant40可自由使用。- 使用 pXtract 转换工具 (http://pfind.ict.ac.cn/downloads.html) 将原始文件转换为. mgf 文件。
- 使用典型搜索参数执行数据库搜索: 数据库、swissprot;多肽质量耐受, 10 ppm;碎片质量公差, 0.5 Da;酶, 胰蛋白酶;错过了站点 , 2 ;可变的修改, carbamidomethylation (半胱氨酸) 和氧化 (蛋氨酸)。
注意: 根据实验设置更改搜索参数。 - 检查数据库搜索结果。评估蛋白质评分, 确定多肽的数量, 多肽评分和质量准确度;序列覆盖率返回所确定的蛋白质覆盖率。
注意: 每个搜索引擎都有其各自的评分算法。用串联质谱的质量评价多肽评分系统。一个良好的频谱的主要信号应显示 (完整) 离子系列, 以验证所确定的肽。通常, 肽评分是概率的,即, 肽评分是一个衡量确定的肽序列与获得的频谱匹配的可能性。蛋白质评分通常是从多肽分数中获得的, 并且被用来将蛋白质排列在被鉴定的蛋白质列表中。
3. 完整的蛋白质复合物的质谱 (本机质谱)
- 电喷雾电离用金涂层发射剂的制备
- 使用微拉拔器从玻璃毛细管中制备 nanoflow 排放源, 如前所述25,41。使用0.78 毫米内径的硼硅酸盐毛细管。
注: 通过改变拔针的参数, 可以对试样的尖端形状和尺寸进行修改和调整。可提供不同内径和壁厚的毛细血管。 - 用导电材料 (如、金或钯) 将玻璃毛细管涂上;使用溅射涂布机产生金等离子体是常见的。按照制造商的指示, 以获得良好的质量涂层。
注: 在使用普通毛细管电压时, 涂层应足够足以获得稳定的喷雾 (见下文)。
- 使用微拉拔器从玻璃毛细管中制备 nanoflow 排放源, 如前所述25,41。使用0.78 毫米内径的硼硅酸盐毛细管。
- 本品质谱样品的制备
注: 盐、洗涤剂或大量的甘油与电喷雾电离不相容。因此, 净化缓冲器由一个挥发性的水缓冲液交换。200毫米醋酸铵是常用的。使用大小排除自旋列或超滤设备进行缓冲交换。在某些情况下, 复杂的稳定性或活动可能会受到缓冲交换的影响。仔细评估质谱, 检查复杂的活动。如有必要, 将因子或添加剂添加到分析缓冲区。- 使用大小排除自旋列快速缓冲交换。通过离心 1000 x g 和4° c 去除存储缓冲, 1 分钟, 放弃流经。洗涤三次, 加入500µL 200 毫米醋酸铵, 然后进行离心。将蛋白质样本的20µL 到柱上, 离心机在 1000 x g 和4° c 下为4分钟。
注: 蛋白质复合物的浓度应为1-10 µM。如果非挥发性元件仍干扰分析, 则可重复此过程。 - 在同一实验中, 浓缩蛋白质样品并交换缓冲液使用离心过滤器。使用孔径小于所分析的蛋白质大小50% 的过滤膜。
- 将蛋白质样品转移到过滤装置中, 加入200毫米醋酸铵。向下旋转 1.5万 x g, 并丢弃流经。加入200毫米醋酸铵, 重复离心。重复此步骤多次。按照制造商的说明离心速度。在4° c 时进行离心。
警告:膜蛋白在过滤器的膜上趋于沉淀。
- 将蛋白质样品转移到过滤装置中, 加入200毫米醋酸铵。向下旋转 1.5万 x g, 并丢弃流经。加入200毫米醋酸铵, 重复离心。重复此步骤多次。按照制造商的说明离心速度。在4° c 时进行离心。
- 使用大小排除自旋列快速缓冲交换。通过离心 1000 x g 和4° c 去除存储缓冲, 1 分钟, 放弃流经。洗涤三次, 加入500µL 200 毫米醋酸铵, 然后进行离心。将蛋白质样本的20µL 到柱上, 离心机在 1000 x g 和4° c 下为4分钟。
- 完整蛋白质复合物的质谱分析
注: 有不同类型的质谱仪从不同的制造商, 可以修改为本机质谱,如, 四极飞行时间 (Q-时间) 质谱计或 orbitrap 质谱。下面所述的协议是在 Q 飞行的仪器上进行的。- 在毛细血管支架上放置一个镀金毛细管, 用1-4 µL 的蛋白质样品填充毛细管。用镊子打开针尖。
- 将毛细管支架与纳米电喷雾源连接, 调整毛细管位置。将毛细管尖端放在 0.5-1.5 厘米的位置。使用80-150 升/小时 nanoflow 气体启动喷雾和调整气体流量, 以保持稳定的喷雾。
- 在 Q 飞行仪的调页中调整参数。典型的起动条件是: 毛细管电压, 1.50 伏;锥电压, 80 伏;射频透镜1能量, 80 V;碰撞能量, 20 V;孔径 3, 13.6 v. 修改这些参数以获得良好的质谱。通过单击 "获取" 按钮开始获取, 并将尽可能多的扫描组合起来以获得良好的质量频谱。
注: 我们建议至少结合100扫描。
- 完整蛋白复合物的串联质谱分析
- 获得如上所述的质量谱 (协议3.3 节)。选择蛋白质复合物的前驱离子。
- 在捕获文件中从 ms 模式更改为 ms/毫秒。将 ms/毫秒的选择设置为前体质量。
- 开始在低碰撞能量的收购。结合几个扫描 (大约20扫描), 以验证正确的前兆质量的选择。增加碰撞能量, 直到蛋白质复合离解。要获得一个良好的质量谱结合至少500扫描。
注: 剥离复合物有时强度低。结合尽可能多的扫描可能会增加分辨率和信噪比。
- 完整蛋白配合物的溶出解离
注意: 为了在蛋白质复合体中获得更多的蛋白质相互作用的洞察力, 可以进行溶液解离。- 如上文所述, 准备蛋白质样品 (3.2 节)。将溶剂添加到蛋白质样品中, 或将 pH 值转化为游离完整的 sub-complexes。典型的溶剂有甲醇、异丙醇和 ACN;通过添加醋酸或氨水溶液来改变乙酸铵的 pH 值。如上文所述获得质谱 (3.3 和3.4 节)。
- 改变溶剂量或 pH 值范围, 产生各种 sub-complexes。通常, 溶剂的用量是 5-50% (最终浓度), 一个典型的 pH 值范围是4-9。从低浓度的溶剂 (5%) 或 pH 值的细微变化开始, 获得质量谱 (见3.3 节)。
- 增加溶剂量或改变 pH 值逐步直到 sub-complexes 在溶液中生成。获取质谱分析 sub-complexes。
- 校准数据
注: 通过碘化铯 (CsI) 溶液对获得的质谱进行外部校准。- 在1毫升水中溶解100毫克 CsI。
- 获取 CsI 的大量频谱。随着上述蛋白质复合物的分析, 在相同的m/z范围内, 改变碰撞能量以获得 CsI 簇。
警告: CsI 迅速沉淀在发射器尖端和污染锥。获得足够的频谱所需的扫描次数。完成后从源中卸下发射器。 - 使用获得的质量频谱和 CsI 参考文件制作校准文件。
- 将校准应用于获得的质谱。
警告: 质量谱的校准可能是原始数据的永久性改变。如果需要 non-calibrated 谱, 请制作文件的备份副本。
- 数据处理和分析
注: 有许多自由使用的软件工具用于数据分析的本机质谱;例如, Massign42或 UniDec43。下面的协议描述了在仪器软件的帮助下的手工数据分析以及对复杂样本的 Massign 的使用。该软件非常适合于分析复杂的质谱。按照联机提供的说明使用程序 (http://massign.chem.ox.ac.uk/)。- 用于数据分析, 通过调整平滑参数来平滑光谱。通过调整参数来确定质心光谱。利用仪器软件工具计算蛋白质复合体峰值包络的两个相邻峰的复杂质量。
警告: 过于密集的平滑可能会导致数据丢失 (例如, 丢失绑定的配体)。 - 对于 Massign42的分析, 生成质量频谱的峰值列表。线性数据点的频谱和平滑。使用各种软件工具分配蛋白质复合物, 计算复杂的成分, 或模拟复杂的峰值信封。
- 用于数据分析, 通过调整平滑参数来平滑光谱。通过调整参数来确定质心光谱。利用仪器软件工具计算蛋白质复合体峰值包络的两个相邻峰的复杂质量。
4. 化学交联结合质谱法
注: 有许多交联战略可利用。在这里, 我们描述的使用双 (sulfosuccinimidyl) 辛二 (BS3), 胺反应交, 这是常用的研究蛋白质-蛋白质相互作用。
- 溶解1.43 毫克 BS3 在100µL 水中, 准备一个25毫米的股票溶液。
注: 其他试剂如 disuccinimidyl 辛二 (DSS) 不是水溶性的, 通常在二甲基亚砜中溶解。一些 cross-linkers 也可在重 isotopically 标记的形式。在 MS 谱中, 重稳定同位素的加入会产生交联 di 肽的峰值对, 这有助于数据评估。当使用差异标记 cross-linkers, 两个变种的股票解决方案准备和混合1:1。
注意: 为了避免由亚甲基亚砜分离的蛋白质复合物, 在交联反应之前, 高度浓缩的溶液会被水或缓冲液所稀释。 - 将 BS3 添加到蛋白质复合物中。使用不同数量的 BS3 范围从 0.5-5 毫米, 以确定最佳的交浓度。在 thermomixer 中孵育反应混合物在25° c 为 1 h。使用4-12% 梯度凝胶和执行 SDS 页评估交联结果 (参见图 2中的一个例子)。
注: 当非链控制中不可见的高分子量蛋白带时, 获得了最佳浓度的 BS3, 而单亚基仍然可见 (图 2)。 - 将交联反应与最佳 BS3 浓度重复。在 thermomixer 中孵育反应混合物在25° c 为 1 h。
注: 某些蛋白复合物在室温下不稳定。交联反应也可以在冰上进行;然而, 反应时间需要调整。 -
通过添加胺缓冲剂 (如、50-100 mM 三缓冲器、pH 值7.5、最终浓度) 或执行乙醇沉淀去除任何残留交联试剂来淬火交联反应。
- 在反应混合物中加入水或缓冲液以达到200µL 的最终体积. 添加600µL ice-cold 乙醇和20µL 3 M 醋酸钠, pH 5.3。在-20 ° c 的2小时或过夜进行彻底混合和孵育。
注: 另外, 蛋白质可在80° c 下沉淀30分钟或液态氮。 - 在 16200 x g 和4° c 下旋转30分钟, 小心地除去上清液。
- 用1毫升 ice-cold 80% (v/五) 乙醇清洗颗粒。在 16200 x g 和4° c 下旋转30分钟, 小心地除去上清液。在真空离心机中烘干颗粒。
- 在反应混合物中加入水或缓冲液以达到200µL 的最终体积. 添加600µL ice-cold 乙醇和20µL 3 M 醋酸钠, pH 5.3。在-20 ° c 的2小时或过夜进行彻底混合和孵育。
- 执行交联蛋白的 SDS 页。切割凝胶带和消化的蛋白质在凝胶如上所述 (2.1 节和 2.2)。
注: 在溶液中消化也可以进行, 但是, 它通常需要额外的分离步骤 (例如, 被消化的肽的大小排除色谱)。 -
如上所述, 执行液相色谱耦合质谱法 (2.3 节)。由于交联多肽通常是低丰度, 应用以下变化的质谱分析, 以增加分析深度的交联样品。
- 在液相色谱分离过程中使用较长的渐变 (例如, 90 分钟而不是65分钟, 请参阅上面的)。
- 从 HCD 碎片中排除双电荷肽。
注: 双荷肽通常为 intra-cross 链多肽 ("环肽" 或 "1 型" 交)。 - 在使用差异标记交联试剂时, 在分析过程中使用 "峰值拾取" 选项,即, 在质谱中定义质量差的峰值对的存在触发了 HCD 碎片。
注: "峰值拾取" 选项可能无法在每种质谱仪上使用。
- 使用叮软件18来识别交联的 di 肽。使用最小化的数据库进行标识。典型的搜索参数有: 仪器谱、HCD;酶, 胰蛋白酶;最大.错过了站点 , 3 ;可变的修改, 氧化 (蛋氨酸) 和 carbamidomethylation (半胱氨酸);交, BS3;最小肽长度, 4;最大.多肽长度, 100;最小肽质量, 400 Da;最大.肽质量, 1万 Da;罗斯福, 1%。
注意: 使用差异标记 cross-linkers 时, 需要配置链接器引起的大量增加。还有其他常用软件用于交标识,例如、xQuest17、MassMatrix19或 XlinkX15。 - 根据碎片谱的质量评估数据库搜索结果。交的可接受光谱应在合理的信噪比下显示两种肽 (至少4相邻离子) 的离子系列。
注意: 当使用差异标记的交联试剂时, MS 谱中的峰值对可以作为附加的质量控制。 - 如果需要, 使用软件工具 (例如, XVis, XiNET) 可视化蛋白质交互网络中的交联结果。使用条形图或圆形图形来可视化蛋白质相互作用。
注意: 两种软件工具都可以在 web 服务器上自由使用。按照有关网站 (https://xvis.genzentrum.lmu.de/和 http://crosslinkviewer.org/) 的详细说明进行操作。
结果
蛋白质的结构分析和它们形成的复合物是理解它们功能的基础。质谱法对结构研究有很大的贡献, 因为它可以适用于几乎所有的复杂的兴趣, 而不考虑大小或样品的异质性。我们使用两个良好蛋白复合物来举例说明该协议;首先, 从C. thermophilum dodecamer RvB1/B2, 其次是来自大肠杆菌的杂 octameric CPS 复合体.
首先, 我们确定了两个复合体的蛋白质成分。为此, 蛋白质被分离了 SDS 页 (图 2) 和凝胶带被切开了从胶凝体。在对蛋白质进行凝胶消化后, 用液相色谱-质谱联用和多肽分析了多肽混合物, 并对其进行了数据库搜索。按照这个工作流程, 我们确定了两个高置信度复合体的所有蛋白质亚基,即, 观察到高数量的多肽具有合理的多肽分数, 从而产生了所有蛋白质亚基的高序列覆盖率 (表1).
然后, 我们用本机质谱分析了完整的 RvB1/B2 复合物 (图 3A)。质谱显示两个物种, 一个在大约 8000 m/z和另一个物种在大约 1100万m/z。这些种类的计算质量对应于 hexameric (RvB1)3(RvB2)3环 (大约 310 kDa) 和 dodecameric 双 (RvB1)6(RvB2)6 (约 620 kDa)。双峰系列显示两个种群;这些起源于他的标签和标签 RvB2 亚基在复合物中的混合物。RvB1/B2 复合体的晶体结构以前获得了36 , 并显示了双的排列 (图 3B)。因此, 本机质谱证实了完整 dodecamer 的化学计量, 并进一步揭示了稳定的 sub-complex。此外, 还确定了共存种群。
为了识别 RvB1/B2 复合体中的蛋白质相互作用点, 我们用化学交联的纯化复合物与 BS3 交。我们首先滴定在交联反应中的 BS3 量, 以确定最佳浓度。BS3 是胺特异性和共价键连接赖氨酸侧链以及蛋白质的 N 总站。交联反应后的 SDS 页 (图 2A)。非连接的复合体显示了 RvB1 和 RvB2 亚基。在反应混合物中加入 BS3, 导致蛋白质带在较高分子量的共价键。SDS 凝胶显示, 随着非链蛋白亚基的减少, BS3 量增加, 交联物种数量增多。然后, 我们从凝胶中剪切蛋白质带, 并遵循上面提供的协议来识别蛋白质相互作用的部位。给出了一个交联 di 肽的例子 (图 3C)。光谱显示两种肽的 y 离子系列证实了这种蛋白质相互作用。总共, 我们获得了14蛋白质相互作用, 包括四交之间的亚基 RvB1 和 RvB2 和两个交之间的两个副本 RvB2 (表 2)。BS3 交联的结果在一个交互网络 (图 3D) 中可视化, 显示内相互作用以及不同亚基间的相互作用。内部分子交表明, RvB1 和 RvB2 亚基折叠的方式, N-和 c-终端领域是在接近接近。注意, 在这种情况下, 内的相互作用不能与同一亚基的分子相互作用区分开来。两个亚基之间的分子间交也被观察到。其中, RvB1 和 RvB2 之间的两个分子交可以在验证交联方法的结构中可视化。其它分子交位于不包括在晶体结构中的挠性回路中。我们还确定了 RvB2 中含有相同肽序列的两个交。这些交可以明确地归类为分子, 因为它们必须来源于同一蛋白质的两个拷贝 (图 3D)。我们的交联实验揭示了复合体内的蛋白质相互作用点, 但也在蛋白质亚基内部提供了对它们的结构安排的洞察力, 这也可以通过现有的晶体结构 (图 3B) 来确认。
我们研究的第二个蛋白质复合体是 CPS。本机质谱 (图 4A) 显示了6000和 1.2万m/z之间的三蛋白复合物。最大的复合体 640 kDa 对应于原封 octamer。更小的复合体代表二个 sub-complexes;小的和大的 CPS 亚基 (160 kDa) 的二聚体和一个杂聚丙烯, 包含每个亚基 (320 kDa) 的两个拷贝。这些 sub-complexes 提供第一洞察到蛋白质汇编;即, 大的和小的亚基是直接接触 (由异质-二聚体揭示) 和聚丙烯可能是一个产品的两个聚。为了获得完整的 CPS 复合体结构布置的更多信息, 我们进行了杂 octamer 和异聚丙烯的串联质谱 (ms)。在这两种情况下, 与前体分离的小亚基都表明小亚基位于组件的外围 (图 4B)。实际上, 在可用晶体结构 (图 4C)44中, 小亚基是外围的。
还进行了化学交联使用 BS3 交。利用增加的量, 增强了 CPS 子单元的共价键连接。在消化蛋白质和分析的肽如上所述, 许多蛋白质相互作用在大亚基和一个交在小亚基被获得了 (表 2)。此外, 类似于 RvB1/B2 复合体, 我们发现两个分子交之间的大型 CPS 亚基的两个副本。这些交安置二个大子单元面对彼此在他们的 C 终端边。在先前的一项研究中, 结合结构质谱和计算建模35, 我们确定了大亚单位中的三种额外的相互作用, 这很可能来源于由大亚基的两个拷贝的接口晶体结构和获得的模型 (图 4C和表 2)。这些交互允许由四大亚基组成的 CPS 核心复合体的排列。然而, 在小和大亚基之间没有 inter-subunit 交观察。通过检查可用的晶体结构 (图 4C), 很明显, 复合体的 tetrameric 核心之间的相互作用表面, 包括大亚基和外围小亚基非常小, 这可能解释缺乏 inter-subunit 的互动。这是证实的本地质谱, 这表明小亚基容易离解从完整的复合体很可能由于一个小的结合接口。然而, 在 CPS 复合体中的蛋白质相互作用结合化学交联和本机质谱可以推断它们的结构排列 (图 4D)。
综合起来, 本机质谱和化学交联结合质谱鉴定的交叉连接多肽, 允许重组的结构安排, 两个例子配合。当化学交联显露了蛋白质亚基的安排, 例如 RvB1 和 RvB2 之间的相互作用或在 CPS 的 tetrameric 核心之内, 本机质谱提供了蛋白质 stoichiometries 原封复合体和流行的亚。在 CPS 的情况下, 两个亚基之间的分子相互作用可以通过化学交联来观察, 本机质谱表明每个大亚基与一个小亚基交互 (图 4D)。串联质谱法提出了复合体中小亚基的外围位置和两个亚基之间的小界面。
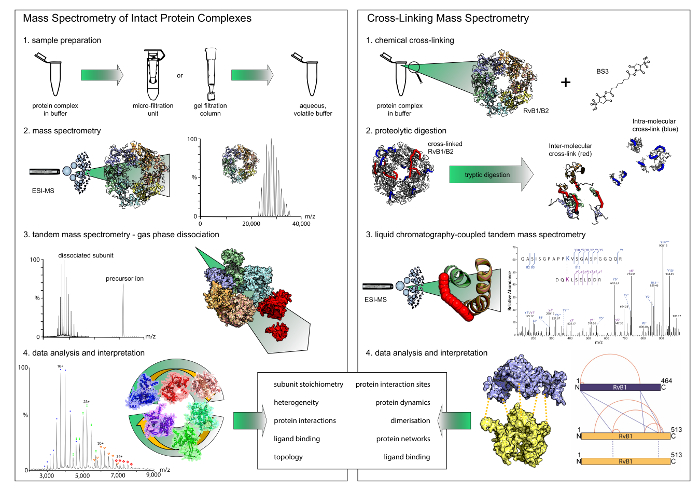
图 1: 本机质谱和交联的工作流程.这两种技术都提供了互补的结果。当本机质谱显示 stoichiometries 和相互作用模块, 交联提供洞察到蛋白质相互作用站点在复合体之内。请注意, 化学交联只揭示二元相互作用。(A) 本机质谱中的第一步是使用过滤器或凝胶过滤柱对挥发性和水缓冲液进行缓冲交换。完整的蛋白质复合物的质谱, 然后揭示其化学计量。在串联质谱实验中, 外周亚基是离解的。(B) 对于化学交联, 蛋白质复合体用交联试剂孵育。然后用液相色谱-耦合质谱法对交联蛋白进行消化。请单击此处查看此图的较大版本.
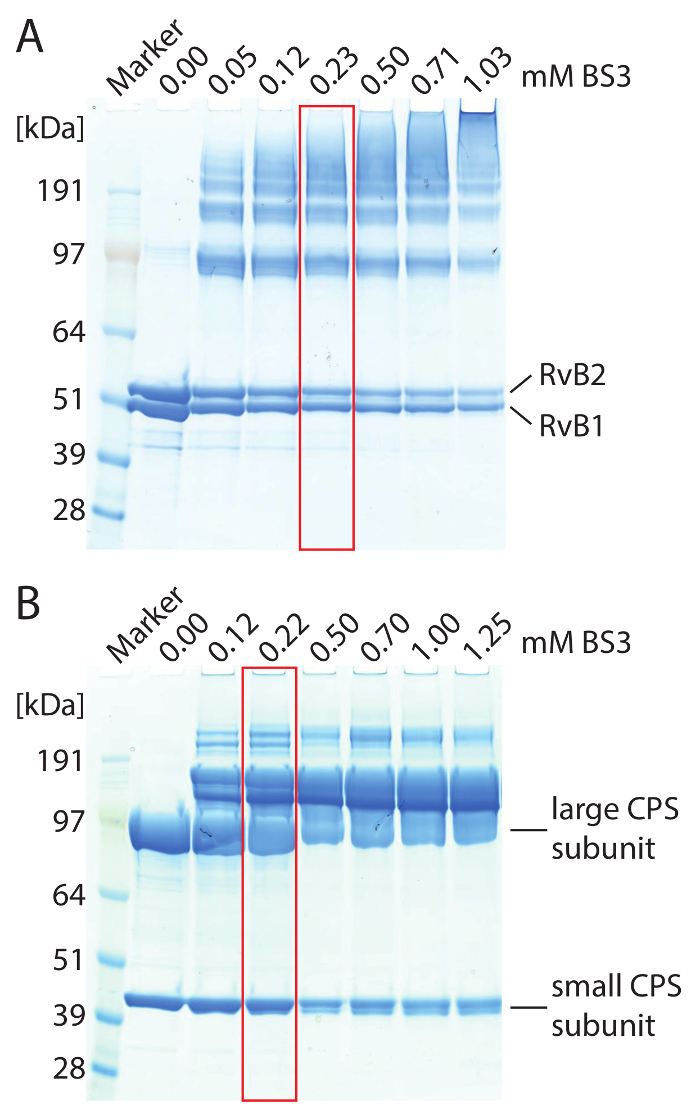
图 2: 交联 RvB1/B2 (a) 和 CPS (B) 复合物的 SDS 页.(A) 2.5 µM RvB1/B2 被装入每个胶凝体车道。BS3 的浓度是不同的。非交联 RvB1/B2 显示两个蛋白质亚基在大约 50 kDa。BS3 的加入导致蛋白质亚基的共价键连接, 导致蛋白质带在更高的分子量。交联种的数量随着 BS3 浓度的升高而增加。最佳的交联条件被突出 (红色)。(B) 10 µM CPS 是每条凝胶车道加载的。得到了大 (90 kDa) 和小 (40 kDa) CPS 亚基。BS3 的加入导致蛋白质亚基的共价键连接, 导致蛋白质带在更高的分子量。最佳的交联条件被突出 (红色)。请单击此处查看此图的较大版本.
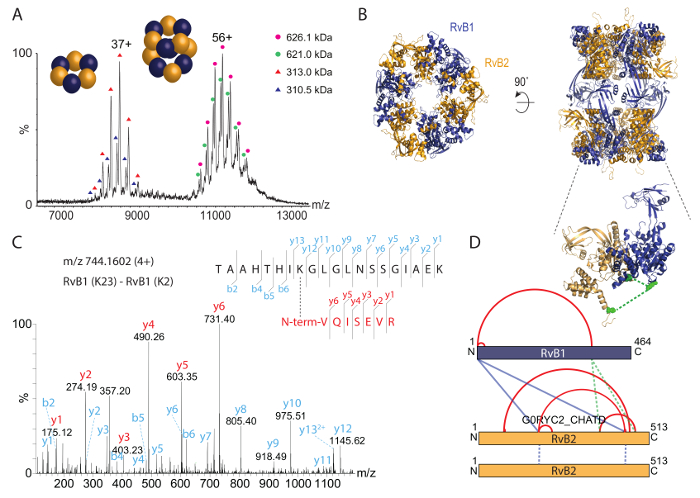
图 3: 本机质谱和 RvB1/B2 络合物的化学交联.(A) 本机质谱揭示了两种 RvB1/B2;完整的 dodecamer (即, (RvB1)6(RvB2)6) 在大约1.1万到 1.2万m/z和 hexameric 环 (RvB1)3(RvB2)3在大约 8000 m/z。这两种物种都显示了由他的标签和标签 RvB2 的两个种群。已从35中修改了频谱。(B) 显示 RvB1/B2 的晶体结构 (PDB ID 4WVY)。交替 RvB1 和 RvB2 亚基形成两个 hexameric 环。(C) 交联的 di 肽的破碎谱。RvB1 的 n-端部与 K23 RvB1. y-离子系列分别获得了双肽 (红和青色)。(D) 在 RvB1/B2 复合体中获得的内部和 inter-protein 的相互作用。交以红色显示, inter-cross 链接以蓝色显示。插入显示两个分子交之间的 RvB1 和 RvB2 亚基可以可视化的晶体结构 (绿色, 插入)。来自两个 RvB2 副本的交互显示为蓝色虚线。请单击此处查看此图的较大版本.
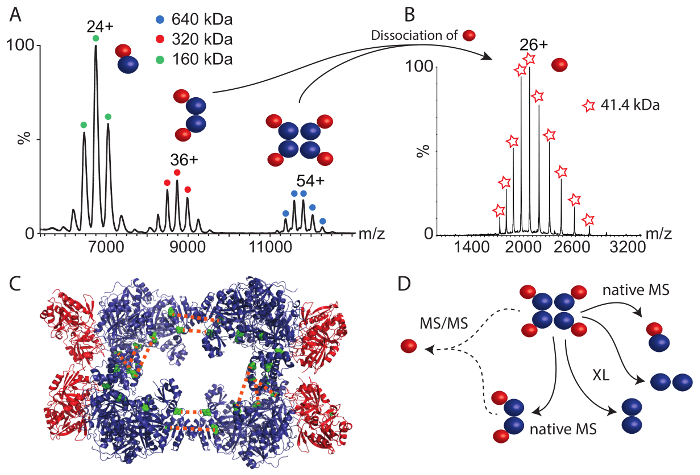
图 4: CPS 的本机质谱和化学交联.(A) CPS 的本地质量谱显示了三个复合体。异质二聚体 (160 kDa), 杂聚丙烯 (320 kDa) 和异 octamer (640 kDa)。已从35中修改了频谱。(B) tetrameric 和 octameric cps 复合物的串联质谱法揭示了小 cps 亚基的离解。(C) 显示 CPS 的晶体结构 (PDB ID 1BXR)。大亚基形成一个 tetrameric 的核心, 小亚基位于复合体的外围。在大亚单位的两个拷贝之间的分子间交显示 (绿色)。(D) 大型和小型 CPS 子单元的交互。当地质谱显示亚和建议的小亚基的外围位置。化学交指示 CPS tetrameric 核心的安排。请单击此处查看此图的较大版本.
表 1: 数据库搜索结果.用液相色谱-质谱联用和数据库检索对蛋白质进行了鉴定。给出了蛋白质的名称、加入数量、描述以及蛋白质质量。列出了蛋白质的评分、每种蛋白质的观察光谱数以及观察到的肽序列的数量。五多肽与最高的吉祥物肽分数列出每个蛋白质亚基。请单击此处下载此文件.
表 2: 在 RvB1/B2 和 CPS 中观察到的交叉链接给出了络合物的亚基和交联的残余物。交 (intra-or 分子) 的类型是从重叠的肽序列或先前的研究中发现的35。请单击此处下载此文件.
讨论
为 multi-subunit 蛋白复合物的质量谱结构分析提供了协议。这两种技术, 在议定书中描述, 主要是提供互补的结果, 并非常适合于了解蛋白质 (-配体) 的结构安排, 这是很难用传统的结构技术研究。本机质谱分析亚和稳定的相互作用模块, 提供了对蛋白质 stoichiometries 以及蛋白质相互作用的洞察。另一方面, 交叉链接产生直接接触点的信息。根据所使用的交, 可以或应该在分析中包含一定的灵活性。
所提供的协议一般易于执行, 且不耗时。整个协议可以在一周内执行, 并且可以应用到几乎所有的蛋白质复合物, 虽然, 一定数量的蛋白质复合体是需要成功的分析。样品制备简单, 不需要特别纯化的蛋白质复合物。然而, 一个常见的缺陷是样品的污染在样品制备过程中的质量谱蛋白的鉴定。在大多数情况下, 这些污染物包括来自灰尘、皮肤或毛发的角。因此, 在样品制备过程中应特别注意佩戴手套和实验室大衣、过滤水缓冲液和使用高纯度溶剂, 以谱蛋白的鉴定。其他污染的蛋白质, 如伴侣, 通常在蛋白质纯化过程中被引入,例如, 在使用亲和标记时。在这些情况下, 应该改进蛋白质纯化, 例如通过增加洗涤步骤。在任何情况下, 在数据库搜索过程中, 通过省略分类过滤器 (即, 搜索所有物种的蛋白质), 可以很容易地识别样本中的蛋白质污染物。如果只观察到少量的肽 (即, 就可以获得低蛋白质覆盖率), 即使有足够的样本, 在消化过程中也可能需要使用不同的蛋白酶。在一般的胰蛋白酶产生足够多肽;然而, 在某些情况下, 如膜蛋白或膜领域的蛋白质, 胰裂解点的数量减少和其他酶靶向疏水性氨基酸是一个更好的选择。
在仪器仪表方面, 本机质谱仪是一种特别改装的仪器, 它在转移到气相中保持价的相互作用。介绍了几种仪器类型, 包括 Q 飞行和 orbitrap 仪器。虽然修改后的 Q 飞行时间质谱仪是商业可用于本地质谱自几年以来, 后者是最近才引入, 并在大多数情况下需要专门修改45。然而, 高分辨率仪器的应用允许研究多重配体的结合及其量化46,47 , 并有望用于将来的应用程序。
用液相色谱-耦合质谱法鉴定交联的 di 肽, 可采用少量修改的标准程序。然而, 数据库搜索是限制因素, 因为专业软件很少能处理大型数据库, 并且需要减少含有蛋白质亚基的数据库。最近的研究使用质谱-裂解 cross-linkers 靶蛋白相互作用在整个细胞裂解48,49。在串联质谱实验中使用化学 cross-linkers 的方法主要是产生线性多肽 (由交), 这可以通过进一步的分裂和线性肽的数据库搜索来识别, 这就减少了搜索时间和计算搜索空间。然而, 为了进行这些实验, 需要离子阱质量分析仪或带有离子阱的混合质谱仪。一般而言, 由于假阳性是一个重要的问题, 交联多肽的质谱往往通过其片段谱的质量来进行人工验证, 从而极大地延长了数据分析时间。因此, 在没有进一步验证步骤的情况下开发健壮的评分系统是未来的潜在应用。改进数据分析和减少误报次数的一种方法是引入错误发现率计算, 并将其应用于交联数据集50。
总的来说, 这里描述的技术可以通过进一步的质谱技术 (例如, 共价键标记) 来补充, 以增加分析的输出。协议的其他修改和改进可以很容易地实现。作为这样比较交联34揭开构象变化在蛋白质汇编。在本族质谱学的进一步发展现在允许分析膜蛋白51,52及其与血脂的相互作用28,52,53,54.本机质谱的高分辨率质谱仪的新发展扩展了应用和配体结合,例如, 脂类与膜蛋白的结合, 现在可以包括在分析中45,46. 与计算建模方法相结合, 这些技术可以提供不同分辨率55的结构模型。如果没有晶体结构可用于完整的复杂或单一的亚基, 质谱可以提供第一洞察到蛋白质相互作用和拓扑结构的未知的复杂。根据所使用的技术和所获得的结果, 可以获得未知复杂度的低分辨率模型56,57,58。如果有晶体结构或同源模型, 从质谱学得到的结构信息可以产生甚至接近本机模型59。
与其他结构技术相比, 质谱法具有要求样品量低的优点, 可以处理异质样品, 适用于无限大小的蛋白质配合物。此外, 质谱法允许研究动态蛋白质系统。在溶液中存在的蛋白质或蛋白质复合体的不同种群通常一起进行分析, 因此, 与其他需要选择某些种群的结构技术不同, 所有构象都在分析, 并在一个实验中被征税。定量交联方法最近被引入34,60,61 , 并有望用于描述不同条件下的构象变化的未来应用。
披露声明
作者没有什么可透露的。
致谢
我们感谢我们的同事进行了有益的讨论。我们还感谢 Ilme Schlichting 和卡尔-彼得 Hopfner 提供蛋白质复合物。我们承认联邦教育和研究部 (BMBF、ZIK 方案、03Z22HN22)、欧洲区域发展基金 (EFRE、ZS/2016/04/78115) 和 MLU 哈雷-维滕贝格的资助和惠康信托基金 (109854/658Z/15/Z) 到美联社
材料
| Name | Company | Catalog Number | Comments |
| Chemicals, Reagents, Consumables | |||
| 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid (HEPES) | Sigma Aldrich | H4034 | buffer |
| Acetic acid | Sigma Aldrich | 695092 | pH |
| Acetonitrile Optima LC/MS | Fisher Chemicals | A955-1 | solvent |
| Amicon Ultra centrifugal devices (different MWCO) | Millipore | i.e. UFC500396 | buffer exchange, concentration |
| Ammonium acetate solution | Sigma Aldrich | A2706 | native MS |
| Ammonium bicarbonate | Sigma Aldrich | 9830 | in-gel digestion |
| Ammonium solution | Sigma Aldrich | 9859 | pH |
| Bis(Sulfosuccinimidyl)suberate-d0 (BS3-d0) | Thermo Scientific | 21590 | cross-linker |
| Bis(Sulfosuccinimidyl)suberate-d4 (BS3-d4) | Thermo Scientific | 21595 | cross-linker |
| Caesium iodide | Sigma Aldrich | 203033 | calibration |
| Calcium chloride | Sigma Aldrich | C5670 | in-gel digestion |
| Capillaries (1.0 OD × 0.78 ID × 100 L mm) | Harvard Apparatus | 30-0038 | native MS |
| Disuccinimidyl suberate (DSS) | Thermo Scientific | 21655 | cross-linker |
| DL-Dithiothreitol | Sigma Aldrich | D5545 | in-gel digestion |
| DMSO (dimethyl sulfoxide) | Sigma Aldrich | D8418 | solvent |
| Ethanol | Fisher Chemicals | BP2818 | solvent |
| Formic acid Optima LC/MS | Fisher Chemicals | A117-50 | solvent supplement |
| Instant Blue Coomassie staining solution | expedeon | ISB1L | staining solution for gel electrophoresis |
| Invitrogen NuPAGE 4-12% Bis-Tris gels (1 mm, 10 well) | life technologies | NP0323BOX | gel electrophoresis |
| Iodoacetamide | Sigma Aldrich | I1149 | In-gel digestion |
| Isopropyl alcohol, HPLC grade | Fisher Chemicals | P750717 | solvent |
| Methanol | Fisher Chemicals | A456-212 | solvent |
| Micro BioSpin 6 columns | BioRad | 732-6222 | buffer exchange |
| NuPAGE Antioxidant | invitrogen | NP0005 | running buffer, gel electrophoresis |
| NuPAGE LDS Sample Buffer (4 ×) | invitrogen | NP0007 | sample loading buffer (non-reducing) for gel electrophoresis |
| NuPAGE MES SDS Running buffer | life technologies | NP0001 | gel electrophoresis |
| NuPAGE MOPS SDS Running buffer | life technologies | NP0001 | gel electrophoresis |
| NuPAGE Sample Reducing Agent (10 ×) | invitrogen | NP0004 | reducing Agent for gel electrophoresis |
| PBS - phosphate buffered saline | Sigma Aldrich | P4417 | buffer |
| SeeBlue Plus2 Prestained Standard Protein Marker | invitrogen | LC5925 | prestained Protein Marker for gel electrophoresis |
| Sodium acetate | Sigma Aldrich | S2889 | ethanol precipitation |
| Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 252859 | buffer |
| Trypsin | Sigma Aldrich | TRYPSEQ-RO (11418475001) | in-gel digestion |
| Vivaspin centrifugal devices (different MWCO) | Sartorius | i.e. VS0101 | buffer exchange, concentration |
| Water for HPLC | Sigma Aldrich | 270733-2.5L-M | solvent |
| Name | Company | Catalog Number | Comments |
| Instruments | |||
| Centrifuge Heraeus Fresco 21, bench-top centrifuge | Thermo Scientific | 75002425 | |
| Flaming / Brown Micropipette puller Model P-1000 | Sutter Instruments | P-1000 | |
| Gelelectrophoresis chamber Xcell SureLock MiniCell | Invitrogen | EI0001 | |
| Gold Coater Quorum Q150R S | Quorum Technologies Ltd. | Q150RS | |
| Horizontal gel shaker Rotamax 120 T | Heidolph Instruments | 544-41200-00 | |
| Q-TOF Ultima mass spectrometer, MS Vision high mass upgrade | Waters (Micromass) | - | |
| reversed-phase C18 analytical column Acclaim PepMap (C18, 75 mm I.D., 50 cm, 3 mm pore size) | Thermo Scientific | 164570 | |
| reversed-phase C18 pre-column (C18, 150 mm I.D., 2 cm, 5 mm pore size) | Thermo Scientific | 164213 | |
| scalpel | Fisher Scientifc | 10463989 | |
| SpeedVac SPD121P vacuum centrifuge | Thermo Scientific | SPD121DP-230 | |
| Thermomixer C | Eppendorf | 5382000015 | |
| Tweezers AA | Sigma Aldrich | Z680184-1EA | |
| Ultrasonic cleaner USC-TH, sonication bath | VWR | 142-0084 | |
| UltiMate Dionex 3000 nano-LC system, coupled to Q-Exactive plus hybrid mass spectrometer (nano-ESI source) | Thermo Scientific | 0726030+ | |
| Name | Company | Catalog Number | Comments |
| Software, Software Tools, Database search | |||
| Mascot | www.matrixscience.com | ||
| Massign | http://massign.chem.ox.ac.uk | ||
| MassLynx | Micromass | ||
| MassMatrix | Xu, H. J Proteome Res. 9 (2010) | ||
| MaxQuant | Cox, J. Nat. Biotechn. 26 (2008) | ||
| pLink | Yang, B. Nat Methods 9 (2012) | ||
| pXtract conversion tool | http://pfind.ict.ac.cn/downloads.html | ||
| UniDec | Marty, M.T. Anal. Chem. 87 (2015) | ||
| XCalibur | Thermo Scientific | ||
| XiNET | http://crosslinkviewer.org | ||
| XLinkX | Liu, F. Curr Op Struct Biol 35 (2015) | ||
| xQuest | Rinner, O. Nat Methods 5 (2008) | ||
| Xvis | https://xvis.genzentrum.lmu.de |
参考文献
- Campbell, I. D. Timeline: the march of structural biology. Nat Rev Mol Cell Biol. 3 (5), 377-381 (2002).
- Liko, I., Allison, T. M., Hopper, J. T., Robinson, C. V. Mass spectrometry guided structural biology. Curr Opin Struct Biol. 40, 136-144 (2016).
- Robinson, C. V. From molecular chaperones to membrane motors: through the lens of a mass spectrometrist. Biochem Soc Trans. 45 (1), 251-260 (2017).
- Lossl, P., van de Waterbeemd, M., Heck, A. J. The diverse and expanding role of mass spectrometry in structural and molecular biology. EMBO J. 35 (24), 2634-2657 (2016).
- Wang, L., Chance, M. R. Protein Footprinting Comes of Age: Mass Spectrometry for Biophysical Structure Assessment. Mol Cell Proteomics. 16 (5), 706-716 (2017).
- Steen, H., Mann, M. The ABC's (and XYZ's) of peptide sequencing. Nat Rev Mol Cell Biol. 5 (9), 699-711 (2004).
- Olsen, J. V., Mann, M. Status of large-scale analysis of post-translational modifications by mass spectrometry. Mol Cell Proteomics. 12 (12), 3444-3452 (2013).
- Schmidt, C., Robinson, C. V. Dynamic protein ligand interactions--insights from MS. FEBS J. 281 (8), 1950-1964 (2014).
- Marcsisin, S. R., Engen, J. R. Hydrogen exchange mass spectrometry: what is it and what can it tell us?. Anal Bioanal Chem. 397 (3), 967-972 (2010).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Leitner, A., Lindner, W. Current chemical tagging strategies for proteome analysis by mass spectrometry. J Chromatogr B Analyt Technol Biomed Life Sci. 813 (1-2), 1-26 (2004).
- Leitner, A., Lindner, W. Chemistry meets proteomics: the use of chemical tagging reactions for MS-based proteomics. Proteomics. 6 (20), 5418-5434 (2006).
- Kiselar, J. G., Chance, M. R. Future directions of structural mass spectrometry using hydroxyl radical footprinting. J Mass Spectrom. 45 (12), 1373-1382 (2010).
- Xu, G., Chance, M. R. Hydroxyl radical-mediated modification of proteins as probes for structural proteomics. Chem Rev. 107 (8), 3514-3543 (2007).
- Liu, F., Heck, A. J. Interrogating the architecture of protein assemblies and protein interaction networks by cross-linking mass spectrometry. Curr Opin Struct Biol. 35, 100-108 (2015).
- Rappsilber, J. The beginning of a beautiful friendship: cross-linking/mass spectrometry and modelling of proteins and multi-protein complexes. J Struct Biol. 173 (3), 530-540 (2011).
- Rinner, O., et al. Identification of cross-linked peptides from large sequence databases. Nat Methods. 5 (4), 315-318 (2008).
- Yang, B., et al. Identification of cross-linked peptides from complex samples. Nat Methods. 9 (9), 904-906 (2012).
- Xu, H., Hsu, P. H., Zhang, L., Tsai, M. D., Freitas, M. A. Database search algorithm for identification of intact cross-links in proteins and peptides using tandem mass spectrometry. J Proteome Res. 9 (7), 3384-3393 (2010).
- Leitner, A., Faini, M., Stengel, F., Aebersold, R. Crosslinking and Mass Spectrometry: An Integrated Technology to Understand the Structure and Function of Molecular Machines. Trends Biochem Sci. 41 (1), 20-32 (2016).
- Heck, A. J. Native mass spectrometry: a bridge between interactomics and structural biology. Nat Methods. 5 (11), 927-933 (2008).
- Sharon, M., Robinson, C. V. The role of mass spectrometry in structure elucidation of dynamic protein complexes. Annu Rev Biochem. 76, 167-193 (2007).
- Goth, M., Pagel, K. Ion mobility-mass spectrometry as a tool to investigate protein-ligand interactions. Anal Bioanal Chem. , (2017).
- Uetrecht, C., Rose, R. J., van Duijn, E., Lorenzen, K., Heck, A. J. Ion mobility mass spectrometry of proteins and protein assemblies. Chem Soc Rev. 39 (5), 1633-1655 (2010).
- Hernandez, H., Robinson, C. V. Determining the stoichiometry and interactions of macromolecular assemblies from mass spectrometry. Nat Protoc. 2 (3), 715-726 (2007).
- Sobott, F., Hernandez, H., McCammon, M. G., Tito, M. A., Robinson, C. V. A tandem mass spectrometer for improved transmission and analysis of large macromolecular assemblies. Anal Chem. 74 (6), 1402-1407 (2002).
- Rostom, A. A., et al. Detection and selective dissociation of intact ribosomes in a mass spectrometer. Proc Natl Acad Sci U S A. 97 (10), 5185-5190 (2000).
- Zhou, M., et al. Mass spectrometry of intact V-type ATPases reveals bound lipids and the effects of nucleotide binding. Science. 334 (6054), 380-385 (2011).
- Uetrecht, C., et al. High-resolution mass spectrometry of viral assemblies: molecular composition and stability of dimorphic hepatitis B virus capsids. Proc Natl Acad Sci U S A. 105 (27), 9216-9220 (2008).
- Morgner, N., et al. Hsp70 forms antiparallel dimers stabilized by post-translational modifications to position clients for transfer to Hsp90. Cell Rep. 11 (5), 759-769 (2015).
- Schmidt, C., Beilsten-Edmands, V., Robinson, C. V. The joining of the Hsp90 and Hsp70 chaperone cycles yields transient interactions and stable intermediates: insights from mass spectrometry. Oncotarget. 6 (21), 18276-18281 (2015).
- Schmidt, C., Beilsten-Edmands, V., Mohammed, S., Robinson, C. V. Acetylation and phosphorylation control both local and global stability of the chloroplast F1 ATP synthase. Sci Rep. 7, 44068 (2017).
- Schmidt, C., et al. Comparative cross-linking and mass spectrometry of an intact F-type ATPase suggest a role for phosphorylation. Nat Commun. 4, 1985 (2013).
- Schmidt, C., Robinson, C. V. A comparative cross-linking strategy to probe conformational changes in protein complexes. Nat Protoc. 9 (9), 2224-2236 (2014).
- Schmidt, C., et al. Surface Accessibility and Dynamics of Macromolecular Assemblies Probed by Covalent Labeling Mass Spectrometry and Integrative Modeling. Anal Chem. 89 (3), 1459-1468 (2017).
- Lakomek, K., Stoehr, G., Tosi, A., Schmailzl, M., Hopfner, K. P. Structural basis for dodecameric assembly states and conformational plasticity of the full-length AAA+ ATPases Rvb1 · Rvb2. Structure. 23 (3), 483-495 (2015).
- Mareya, S. M., Raushel, F. M. A molecular wedge for triggering the amidotransferase activity of carbamoyl phosphate synthetase. Biochemistry. 33 (10), 2945-2950 (1994).
- Shevchenko, A., Wilm, M., Vorm, O., Mann, M. Mass spectrometric sequencing of proteins silver-stained polyacrylamide gels. Anal Chem. 68 (5), 850-858 (1996).
- Olsen, J. V., et al. Parts per million mass accuracy on an Orbitrap mass spectrometer via lock mass injection into a C-trap. Mol Cell Proteomics. 4 (12), 2010-2021 (2005).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol. 26 (12), 1367-1372 (2008).
- Kirshenbaum, N., Michaelevski, I., Sharon, M. Analyzing large protein complexes by structural mass spectrometry. J Vis Exp. (40), (2010).
- Morgner, N., Robinson, C. V. Massign: an assignment strategy for maximizing information from the mass spectra of heterogeneous protein assemblies. Anal Chem. 84 (6), 2939-2948 (2012).
- Marty, M. T., et al. Bayesian deconvolution of mass and ion mobility spectra: from binary interactions to polydisperse ensembles. Anal Chem. 87 (8), 4370-4376 (2015).
- Thoden, J. B., Wesenberg, G., Raushel, F. M., Holden, H. M. Carbamoyl phosphate synthetase: closure of the B-domain as a result of nucleotide binding. Biochemistry. 38 (8), 2347-2357 (1999).
- Rose, R. J., Damoc, E., Denisov, E., Makarov, A., Heck, A. J. High-sensitivity Orbitrap mass analysis of intact macromolecular assemblies. Nat Methods. 9 (11), 1084-1086 (2012).
- Gault, J., et al. High-resolution mass spectrometry of small molecules bound to membrane proteins. Nat Methods. 13 (4), 333-336 (2016).
- Mehmood, S., et al. Mass spectrometry captures off-target drug binding and provides mechanistic insights into the human metalloprotease ZMPSTE24. Nat Chem. 8 (12), 1152-1158 (2016).
- Kaake, R. M., et al. A new in vivo cross-linking mass spectrometry platform to define protein-protein interactions in living cells. Mol Cell Proteomics. 13 (12), 3533-3543 (2014).
- Liu, F., Rijkers, D. T., Post, H., Heck, A. J. Proteome-wide profiling of protein assemblies by cross-linking mass spectrometry. Nat Methods. 12 (12), 1179-1184 (2015).
- Walzthoeni, T., et al. False discovery rate estimation for cross-linked peptides identified by mass spectrometry. Nat Methods. 9 (9), 901-903 (2012).
- Barrera, N. P., Di Bartolo, N., Booth, P. J., Robinson, C. V. Micelles protect membrane complexes from solution to vacuum. Science. 321 (5886), 243-246 (2008).
- Barrera, N. P., et al. Mass spectrometry of membrane transporters reveals subunit stoichiometry and interactions. Nat Methods. 6 (8), 585-587 (2009).
- Laganowsky, A., et al. Membrane proteins bind lipids selectively to modulate their structure and function. Nature. 510 (7503), 172-175 (2014).
- Marcoux, J., et al. Mass spectrometry reveals synergistic effects of nucleotides, lipids, and drugs binding to a multidrug resistance efflux pump. Proc Natl Acad Sci U S A. 110 (24), 9704-9709 (2013).
- Politis, A., Schmidt, C. Structural characterisation of medically relevant protein assemblies by integrating mass spectrometry with computational modelling. J Proteomics. , (2017).
- Hall, Z., Politis, A., Robinson, C. V. Structural modeling of heteromeric protein complexes from disassembly pathways and ion mobility-mass spectrometry. Structure. 20 (9), 1596-1609 (2012).
- Pukala, T. L., et al. Subunit architecture of multiprotein assemblies determined using restraints from gas-phase measurements. Structure. 17 (9), 1235-1243 (2009).
- Politis, A., et al. Topological models of heteromeric protein assemblies from mass spectrometry: application to the yeast eIF3:eIF5 complex. Chem Biol. 22 (1), 117-128 (2015).
- Politis, A., et al. A mass spectrometry-based hybrid method for structural modeling of protein complexes. Nat Methods. 11 (4), 403-406 (2014).
- Fischer, L., Chen, Z. A., Rappsilber, J. Quantitative cross-linking/mass spectrometry using isotope-labelled cross-linkers. J Proteomics. 88, 120-128 (2013).
- Yu, C., et al. Developing a Multiplexed Quantitative Cross-Linking Mass Spectrometry Platform for Comparative Structural Analysis of Protein Complexes. Anal Chem. 88 (20), 10301-10308 (2016).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。