Method Article
Kombination von chemische Vernetzung und Massenspektrometrie intakt Proteinkomplexe, die Architektur des Multi-Untereinheit Protein Baugruppen zu studieren
In diesem Artikel
Zusammenfassung
Die Architektur von Proteinkomplexen ist essentiell für ihre Funktion. Kombination von verschiedenen massenspektrometrischen Techniken erwies sich als leistungsstarke, deren Montage zu studieren. Wir bieten Protokolle für chemische Vernetzung und native Massenspektrometrie und zeigen, wie diese komplementären Techniken helfen, die Architektur des Multi-Untereinheit Protein Baugruppen aufzuklären.
Zusammenfassung
Proteine interagieren Sie mit ihrer Liganden in Form aktiver und dynamischer Assemblys die verschiedenen zellulären Funktionen durchführen. Erläuterung dieser Wechselwirkungen ist daher grundlegend für das Verständnis der zellulären Prozesse. Jedoch viele Proteinkomplexe sind dynamische Assemblys und nicht durch herkömmliche strukturelle Techniken zugänglich. Massenspektrometrie trägt zu der strukturellen Untersuchung dieser Versammlungen, und vor allem die Kombination von verschiedenen massenspektrometrischen Techniken liefert wertvolle Einblicke in deren strukturelle Anordnung.
In diesem Artikel beschreiben wir die Anwendung und Kombination von zwei sich ergänzende massenspektrometrischen Techniken, nämlich chemische Vernetzung gekoppelt mit Massenspektrometrie und native Massenspektrometrie. Chemische Vernetzung umfasst die kovalente Verknüpfung der Aminosäuren in der Nähe durch die Verwendung von chemischen Reagenzien. Nach der Verdauung mit Proteasen vernetzte di-Peptide werden durch Massenspektrometrie identifiziert und Protein Interaktionen Websites werden aufgedeckt. Native Massenspektrometrie ist auf der anderen Seite die Analyse von intakten Proteinen Baugruppen in der Gasphase eines Massenspektrometers. Es zeigt Protein Zusammensetzungen sowie Eiweiß und Ligand Interaktionen. Beide Verfahren liefern daher ergänzende Informationen über die Struktur der Protein-Ligand Baugruppen und deren Kombination erwies sich mächtig in früheren Studien.
Einleitung
Die strukturellen Untersuchung von Protein-Baugruppen ist besonders wichtig für das Verständnis der zellulären Prozesse geworden. Infolgedessen haben viele Techniken entwickelt und verbessert in der Strukturbiologie1. Diese Techniken sind jedoch manchmal in ihrer Anwendung aufgrund von Größe, Flexibilität oder Heterogenität der Proteinkomplexe untersuchten begrenzt. Massenspektrometrie kann diese Herausforderungen bewältigen und daher erwies sich als ein mächtiges Werkzeug in der Strukturbiologie2,3,4,5. Der größte Vorteil der Massenspektrometrie, ist jedoch die Fähigkeit, Proteine auch in komplexen und heterogenen Gemischen6eindeutig zu identifizieren. Zu diesem Zweck Proteine sind in der Regel mit Endoproteinases verdaut und die daraus resultierenden Peptid Mischung wird durch flüssige Chromatographie getrennt und direkt in das Massenspektrometer eluiert. Peptid-Massen werden anschließend bestimmt und Vorläufer Ionen sind für weitere Zersplitterung der Peptide ausgewählt. Proteine werden dann durch Suche Peptid und entsprechende Fragment Massen gegen eine bekannte Datenbank identifiziert. Dieses Verfahren ermöglicht nicht nur die Identifikation der Peptide/Proteine, sondern auch ihre Post-translationalen Modifikationen, die eine Masse Verschiebung der Vorläufer-Peptide und die Durchführung der Änderung7Fragment-Ionen verursachen. Einige der vielen Techniken in der strukturellen Massenspektrometrie basieren auf diesem Prinzip4,8. Z. B. Fuß Kennzeichnung Techniken wie z. B. Wasserstoff Deuterium Austausch9,10, chemische Kennzeichnung Strategien11,12, oder Hydroxyl-radikal Druck13,14 , geben Einblicke in die Oberfläche Zugänglichkeit der Proteine unter bestimmten Bedingungen.
Eine andere Technik ist (chemischen) Vernetzung, an denen die kovalente Verknüpfung der Aminosäuren in der Nähe durch ihre funktionellen Gruppen. Dafür sind chemische Reagenzien, UV-aktivierbare Reagenzien oder Aminosäuren beschäftigt15,16. Nach der Vernetzung, die Proteine sind in der Regel mit Proteasen hydrolysiert und vernetzte di-Peptide werden durch flüssige Chromatographie-gekoppelten Massenspektrometrie analysiert. Die Identifizierung der Vernetzung Produkte von Datenbank-Suche, erfordert jedoch die Verwendung einer speziellen Software die Peptid-Sequenzen von verschiedenen Proteinen und verschiedenen Regionen17,18,19zu verketten. Die Verwendung von chemischen Quervernetzern hat den Vorteil, dass es werden, um fast jedes Protein Komplex von Interesse eingesetzt kann und erfordert keine Einbeziehung der UV-aktivierbare Aminosäuren, die nur erreicht werden kann, wenn das Protein des Interesses an Wirtszellen zum Ausdruck zu bringen. Als solche Vernetzung ist ein vielseitiges Werkzeug und arbeitete erfolgreich in vielen strukturellen Studien auch große Protein Baugruppen20.
Massenspektrometrie von intakten Proteinkomplexe (manchmal genannt "native" Massenspektrometrie), auf der anderen Seite beinhaltet die Analyse von intakten Proteinen und Proteinkomplexen ohne Hydrolyse in Peptide. Es zeigt Zusammensetzung, Heterogenität, Stöchiometrie, Topologie und Untereinheit Interaktionen von Protein-komplexe21,22. In Kombination mit Ionenbeweglichkeit ermöglicht native Massenspektrometrie weitere Bestimmung der ihre Konformation23,24. Dies macht es ein leistungsfähiges Werkzeug für die strukturelle Untersuchung der Proteinkomplexe, die schwierig zu beurteilen, durch herkömmliche strukturelle Techniken sind. Native Massenspektrometrie erfordert jedoch Analyse Puffer, die nicht-kovalente Proteininteraktionen während Elektrospray-Ionisation zu pflegen. Dies geschieht in der Regel durch wässrige, flüchtigen Puffer wie Ammonium Acetat25verwenden. Darüber hinaus sind Instrument Modifikationen nötig, Dissoziation während der Übertragung in die Gasphase des Massenspektrometers26zu verhindern. Auf diese Weise angewendet werden, wurden viele (große) Proteinkomplexe analysiert. Eindrucksvolle Beispiele sind die Studien von intakten Ribosomen27, ATP Synthasen28oder Viren29.
In früheren Studien bewährt sich besonders die Kombination von native Massenspektrometrie und Vernetzung. Zum Beispiel die Zusammensetzungen der Chaperon-komplexe, darunter eine unerwartete Hsp70-Dimer könnte eingeholt werden Massenspektrometrie Experimente der intakten Proteinkomplexe, während chemische Vernetzung die Modalitäten der Proteine in offenbart der Baugruppen30,31. In einer anderen Studie wurden die Effekte von post-translationalen Modifikationen auf eine intakte ATP-Synthase komplex untersucht. Native Massenspektrometrie bot Einblicke in die komplexen Proteinstabilität in das Vorhandensein oder Fehlen von Phosphorylierung und Acetylierung32,33. Eine vergleichende vernetzende Strategie34 deckte dann Konformationsänderungen des Proteins Komplex unter verschiedenen Bedingungen.
Hier bieten wir die Protokolle für Proteinidentifizierung von Massenspektrometrie (chemische) Vernetzung und native Massenspektrometrie, einschließlich Datenanalyse und Interpretation (Abbildung 1). Die Kombination von komplementären Ergebnisse, die von diesen Methoden mit Hilfe von zwei gut charakterisierten Proteinkomplexe ist nachgewiesene35. Unser Protokoll kann auf Protein-Montage angewendet werden, die bei bestimmten Reinheit und Konzentration gereinigt werden können. Der Ansatz ist in einigen Fällen durch Datenanalyse der Vernetzung von Daten, d. h., die Größe der Datenbank begrenzt eingesetzt die bestimmt die gewünschte Suche Raum und Zeit. Darüber hinaus manuellen Validierung der identifizierten Querverbindungen ist oft erforderlich und wird die Ausgabe weiter reduziert. Native Massenspektrometrie wird meist durch die Sample-Qualität, z.B., Puffer begrenzt und Addukte während der Reinigung und die Möglichkeit zum Austauschen von wässrigen und flüchtigen Puffer verwendet. Proteinkomplexe, die in der Regel für die Strukturanalyse gereinigt haben jedoch die Qualität, die für erfolgreiche Analyse mit unsere Protokolle erforderlich.
Protokoll
1. Reinigung von Proteinkomplexen
- Protein-Komplex nach optimierten Standardprotokolle vorzubereiten.
Hinweis: Das Protokoll ist hier mit der RvB1/B2-Komplex von Chaetomium Thermophilum und Carbamoylproxyl-Phosphat-Synthase (CPS) von Escherichia colinachgewiesen. RvB1/B2 und CPS waren wie beschrieben36,37gereinigt. Jedes Protein-Komplex erfordert eine individuelle Reinigung-Protokoll. Das Protokoll entsprechend anpassen. Amin-freie Puffer wie Phosphat-gepufferte Kochsalzlösung (PBS) oder 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic Säure (HEPES) sollte für chemische Vernetzung verwendet werden. Ersetzen Sie den Puffer wenn möglich während der Reinigung.
Achtung: Kein auftragen Sie Methoden oder Reagenzien, die die systemeigene Assembly dem Proteinkomplex zu stören.
2. mass Spectrometry-basierte Proteinidentifikation
-
Gelelektrophorese
Hinweis: Es gibt verschiedene Gel-Systeme zur Verfügung und jedes Labor ein eigene Setup verwendet. Passen Sie die Bedingungen nach dem Gel-System. Tragen Sie Handschuhe und Kittel im gesamten Protokoll wie massenspektrometrische Analyse der häufigsten Schadstoffe Keratine zählen.- 7.5 µL 4 X Probenpuffer und 3 µL 10-fach Reduktionsmittel (Endkonzentration 50 mM Dithiothreitol (DTT)) auf 20 µL Protein Probe anwenden. Verwenden Sie 10 µM CPS oder 2,5 µM RvB1/B2. Spin-down für 1 min bei 16.200 × g und Wärme für 10 min bei 70 ° C.
Hinweis: Wenn mit einem anderen Gel-System stellen Sie die geforderten Mengen und Heiztemperatur. - Bereiten Sie eine 4-12 % Steigung Gel für Elektrophorese. Verdünnen Sie die laufenden Puffer 20 Mal mit Wasser zu und füllen Sie die Elektrophorese-Kammer. Verwendung 2-(N-Morpholino) Ethanesulfonic Säure (MES) Puffer für Proteine von kleineren Molekulargewicht (2-200 kDa) und 3-(N-Morpholino) Propanesulfonic Säure (MOPS) Puffer für größere Proteine (14-200 kDa). Hinzu kommt der Innenkammer 0,5 mL Antioxidans.
Hinweis: Wenn mit einem anderen Gel System bereiten laufenden Puffer und das Gel nach Hersteller Protokollen. - Laden Sie einen geeignete Protein Marker (Pre-gebeizt, ungefärbten, verschiedene molekulare Größen) in dem ersten Hohlraum und laden Sie die Proteinproben in die verbliebenen Hohlräume.
- Separate Proteine für 35 min (MES) oder 50 min (MOPS) bei 200 V.
Hinweis: Stellen Sie die Spannung für verschiedene Gel-Systeme und Elektrophorese-Zeiten. - Um die Protein-Bands zu beflecken, übertragen Sie das Gel in ein Gel, die Färbung Box und decken Sie das Gel mit einem wasserbasierten Coomassie Färbelösung zu. Brüten Sie über Nacht und bei Raumtemperatur auf einem horizontalen Gel-Shaker.
- Destain Sie das Gel die Färbelösung durch Wasser ersetzen. Wiederholen Sie diesen Schritt mehrmals (ca. 3 - 5 Mal), bis der Gel-Hintergrund klar erscheint.
- 7.5 µL 4 X Probenpuffer und 3 µL 10-fach Reduktionsmittel (Endkonzentration 50 mM Dithiothreitol (DTT)) auf 20 µL Protein Probe anwenden. Verwenden Sie 10 µM CPS oder 2,5 µM RvB1/B2. Spin-down für 1 min bei 16.200 × g und Wärme für 10 min bei 70 ° C.
-
In-Gel-Verdauung
Hinweis: Zur Vermeidung von Kontamination verwenden Sie HPLC Grade Lösungsmittel im gesamten Verdauung Protokoll und für alle folgenden Schritte (d.h. massenspektrometrische Analyse). Bereiten Sie alle Lösungen vor und Filtratwasser und Ammonium Bicarbonat.- Schnitt der Protein-Bands, die durch blaue Coomassie visualisiert werden aus dem Gel mit einem Skalpell zu beflecken. Die Protein-Bands in kleine Stücke von ca. 1 mm × 1 mm. Spülen schneiden Sie vorsichtig das Skalpell mit Wasser zwischen verschiedenen Protein-Bändern. Waschen Sie die Gel-Bands mit Wasser und Acetonitril (ACN).
- Senkung der Disulfidbrücken mit DVB-t, Alkylat Cystein Rückstände mit Iodoacetamide und verdauen Proteine mit Trypsin wie oben beschrieben38; in der Regel dient eine Enzym: Protein-Verhältnis von 01:20 bis 1: 100. Verwenden Sie 100 mM Ammonium Bicarbonat während der Verdauung-Protokoll.
- Peptide in zwei Schritten zu extrahieren.
- Zuerst brüten Sie die Gel-Stücke mit Ammonium Bicarbonat und ACN, und sammeln Sie die Peptid-haltigen überstand. Zweitens, inkubieren Sie die Gel-Stücke mit 5 % (V/V) Ameisensäure und ACN. Bei jedem Schritt 15 min inkubieren. Kombinieren Sie beide Überstände. Trocknen Sie die extrahierten Peptide durch Verdunstung der Lösungsmittel in einer Vakuum-Zentrifuge.
Hinweis: Getrocknete Peptide können bei-20 ° C für mehrere Monate gespeichert werden.
- Zuerst brüten Sie die Gel-Stücke mit Ammonium Bicarbonat und ACN, und sammeln Sie die Peptid-haltigen überstand. Zweitens, inkubieren Sie die Gel-Stücke mit 5 % (V/V) Ameisensäure und ACN. Bei jedem Schritt 15 min inkubieren. Kombinieren Sie beide Überstände. Trocknen Sie die extrahierten Peptide durch Verdunstung der Lösungsmittel in einer Vakuum-Zentrifuge.
-
Flüssige Chromatographie-gekoppelten Massenspektrometrie
- Lösen Sie die getrockneten Peptide in 2 % ACN/0.1% Ameisensäure. Lösen sich die Peptide in einem Ultraschall-Bad für 2-3 min und Spin down in einer Zentrifuge bei 16.200 x g für 30 min. Transfer der Proben in Autosampler Vials.
Hinweis: Stellen Sie die Lautstärke entsprechend der Protein-Menge. Für gut gefärbten Coomassie Bands verwenden wir 20 µL. - Spritzen Sie 5 µL der Probe mit dem Nano-LC-MS/MS-System über den Autosampler. Laden Sie die Peptid-Mischung auf eine rückgängig-Phase C18 Pre-Spalte (C18, 150 µm I.D., 2 cm, 5 µm Porengröße) zu entsalzen und konzentrieren sich die Peptide online.
- 0,1 % (V/V) Ameisensäure verwenden, als mobile Phase A und 80 % (V/V) ACN/0.1% Ameisensäure als Mobile B. Separate Peptide auf eine rückgängig-Phase C18 Trennsäule (C18, 75 µm I.D., 50 cm, 3 µm Porengröße Phase) mit einer Steigung von 4-80 % B (mit 0,1 % Ameisensäure) bei 300 nL / über 65 min min.
Hinweis: Eine Nanospray Ionenquelle dient eluierten Peptide in das Massenspektrometer zu übertragen. - (Typisch) MS Nutzungsbedingungen: Sprühen Sie Spannung von 1,6 kV; Kapillare Temperatur von 250 ° C; normalisierte Aufprallenergie von 30. Das Massenspektrometer in datenabhängiges Modus zu betreiben.
- Die MS-Spektren in der Masse Analysator (z.B. Orbitrap) zu erwerben (m/Z 350−1, 600) mit einer Auflösung von 70.000 und einer automatischen Steuerung Ziel von 3 x 106. Wählen Sie aus 20 der intensivsten Ionen HCD Fragmentierung auf eine automatische Gain Kontrolle Ziel von 1 x 10-5. Dynamisch ausgewählte ausschließen zuvor Ionen für 30 S. ausschließen, die einzeln-Ionen sowie Ionen mit unbekannter Ladezustand aufgeladen.
Hinweis: Interne Kalibrierung der Massenspektren erfolgte mittels der Sperre Masse Option39.
- Lösen Sie die getrockneten Peptide in 2 % ACN/0.1% Ameisensäure. Lösen sich die Peptide in einem Ultraschall-Bad für 2-3 min und Spin down in einer Zentrifuge bei 16.200 x g für 30 min. Transfer der Proben in Autosampler Vials.
-
Suche in Datenbank
Hinweis: Es gibt verschiedener Software zur Datenbank durchsuchen. MaxQuant40, ist zum Beispiel frei verfügbar.- Umwandeln Sie Ausgabe-Dateien in .mgf-Dateien mit einem pXtract Konvertierungstool (http://pfind.ict.ac.cn/downloads.html).
- Suche Datenbank mit typischen Suchparameter: Datenbank, derjenigen; Peptid Masse Toleranz, 10 ppm; Fragment Masse Toleranz, 0,5 Da; Enzym Trypsin; verpasste Spaltstellen, 2; Variable Modifikationen, Carbamidomethylation (Cystein) und Oxidation (Methionin).
Hinweis: Ändern Sie die Suchparameter gemäß den experimentellen Einstellungen. - Überprüfen Sie die Datenbank-Suchergebnis. Bewerten Sie Protein-Partitur, Anzahl der Peptide identifiziert, Peptid Partituren und Massengenauigkeit; die Sequenz-Abdeckung kehrt die Protein-Abdeckung identifiziert.
Hinweis: Jede Suchmaschine hat ihre individuelle scoring-Algorithmus. Bewerten Sie das Peptid scoring-System von der Qualität der Tandem-Massenspektren beobachtet. Hauptsignale ein gutes Spektrum weisen (komplett) Ionen-Serie um die identifizierten Peptid zu überprüfen. In der Regel die Peptid-Bewertung basiert auf Wahrscheinlichkeit, d.h.die Peptid-Score ist ein Maß für wie wahrscheinlich die identifizierten Peptidsequenz erhaltenen Spektrum entspricht. Der Protein-Score ergibt sich in der Regel aus Peptid Partituren und wird verwendet, um ein Protein in einer Liste der identifizierten Proteine Rang.
(3) Massenspektrometrie intakt Proteinkomplexe (Native Massenspektrometrie)
- Vorbereitung der Gold-beschichteter Strahler für Elektrospray-Ionisation
- Mithilfe einer Mikropipette Abzieher Nanoflow-Strahler von Glaskapillaren bereits25,41beschrieben vorbereitet. Verwenden Sie Borosilikat-Kapillaren mit einem Innendurchmesser von 0,78 mm.
Hinweis: Durch Veränderung der Parameter für das Ziehen der Nadel, Spitze Form und Größe können geändert und angepasst für die Probe. Kapillaren mit verschiedenen inneren Durchmesser und Wandstärke stehen zur Verfügung. - Beschichten der Glaskapillaren mit leitfähigem Material (z.B., Gold oder Palladium); die Verwendung von einem Sputter Coater erzeugen ein gold Plasma ist üblich. Folgen Sie den Anweisungen des Herstellers zur Beschichtung von guter Qualität zu erhalten.
Hinweis: Die Beschichtung sollte ausreichen, um eine stabile Spray zu erhalten, bei der Anwendung der gemeinsamen kapillarer Spannungen (siehe unten).
- Mithilfe einer Mikropipette Abzieher Nanoflow-Strahler von Glaskapillaren bereits25,41beschrieben vorbereitet. Verwenden Sie Borosilikat-Kapillaren mit einem Innendurchmesser von 0,78 mm.
- Probenvorbereitung für die native Massenspektrometrie
Hinweis: Salze, Reinigungsmittel oder große Mengen an Glycerin sind nicht kompatibel mit Electrospray Ionisierung. Daher ist die Reinigung-Puffer von einem flüchtigen, wässrigen Puffer ausgetauscht. 200 mM Ammoniumacetat wird häufig verwendet. Verwenden Sie Größe Ausgrenzung Spin Spalten oder Ultrafiltration Geräte für Buffer Tausch. In einigen Fällen möglicherweise Buffer Tausch Komplexstabilität oder Aktivität betroffen sein. Bewerten Sie die Massenspektren sorgfältig und überprüfen Sie die Aktivität des Komplexes. Fügen Sie Cofaktoren oder Zusatzstoffe in den Analyse-Puffer, falls erforderlich.- Verwenden Sie Größe Ausgrenzung Spin Spalten für schnelle Buffer Tausch. Entfernen Sie den Speicher-Puffer durch Zentrifugation bei 1.000 x g und 4 ° C für 1 min. Entsorgen der Durchströmung. Waschen Sie durch Zugabe von 500 µL 200 mM Ammoniumacetat, gefolgt durch Zentrifugation dreimal. Laden Sie die Protein-Probe auf die Spalte und die Zentrifuge bei 1.000 x g und 4 ° C für 4 min 20 µL.
Hinweis: Die Konzentration der Proteinkomplex sollte 1-10 µM. Das Verfahren kann wiederholt werden, wenn nicht-flüchtigen Komponenten noch die Analyse stören. - Die Protein-Probe zu konzentrieren und den Austausch des Puffers in der gleichen Experiment zentrifugale Filter verwenden. Verwenden Sie eine Filtration Membran Pore Größe 50 % kleiner als die Größe der Proteine analysiert.
- Übertragen Sie die Protein-Probe in der Filtration-Gerät zu, und fügen Sie 200 mM Ammoniumacetat. Spin-down bei 15.000 x g und die Durchströmung zu verwerfen. Fügen Sie 200 mM Ammoniumacetat hinzu und wiederholen Sie die Zentrifugation. Wiederholen Sie diesen Schritt mehrmals. Folgen Sie den Anweisungen des Herstellers für die Zentrifugation Geschwindigkeit. Führen Sie die Zentrifugation bei 4 ° C.
Vorsicht: Membranproteine sind in der Regel auf der Membran des Geräts Filter auszufällen.
- Übertragen Sie die Protein-Probe in der Filtration-Gerät zu, und fügen Sie 200 mM Ammoniumacetat. Spin-down bei 15.000 x g und die Durchströmung zu verwerfen. Fügen Sie 200 mM Ammoniumacetat hinzu und wiederholen Sie die Zentrifugation. Wiederholen Sie diesen Schritt mehrmals. Folgen Sie den Anweisungen des Herstellers für die Zentrifugation Geschwindigkeit. Führen Sie die Zentrifugation bei 4 ° C.
- Verwenden Sie Größe Ausgrenzung Spin Spalten für schnelle Buffer Tausch. Entfernen Sie den Speicher-Puffer durch Zentrifugation bei 1.000 x g und 4 ° C für 1 min. Entsorgen der Durchströmung. Waschen Sie durch Zugabe von 500 µL 200 mM Ammoniumacetat, gefolgt durch Zentrifugation dreimal. Laden Sie die Protein-Probe auf die Spalte und die Zentrifuge bei 1.000 x g und 4 ° C für 4 min 20 µL.
- Massenspektrometrische Analyse von intakten Proteinkomplexen
Hinweis: Es gibt verschiedene Arten von Massenspektrometern verschiedener Hersteller, die für native Massenspektrometrie, z.B., Quadrupol (Q-ToF) Time-of-Flight Massenspektrometer oder Orbitrap Massenspektrometer geändert werden können. Die nachfolgend beschriebene Protokoll wurde auf ein Q-ToF-Instrument durchgeführt.- Setzen Sie eine Gold-beschichteter Kapillare in den Kapillaren Halter und füllen Sie die Kapillare mit 1-4 µL Protein Probe. Öffnen Sie die Spitze der Nadel mit einer Pinzette.
- Die Kapillaren Halter mit Nano-Elektrospray-Quelle zu verbinden und die Kapillare Position einstellen. Positionieren Sie die Spitze der Kapillare bei 0,5-1,5 cm zur Kegel Öffnung. Verwenden Sie 80-150 L/h Nanoflow Gas zu initiieren das Spray und passen die Gaszufuhr zur Aufrechterhaltung einer stabilen Spray.
- Passen Sie die Parameter in der Melodie-Seite des Q-ToF Instruments. Typische Ausgangsbedingungen sind: Kapillare Spannung, 1,50 kV; Kegel-Spannung, 80 V; HF-Linse 1 Energie, 80 V; Aufprallenergie, 20 V; Aperture 3, 13,6 V. ändern diese Parameter um gute Massenspektren zu erhalten. Start der Akquisition durch Anklicken des Buttons "erwerben" und kombinieren so viele Scans wie möglich ein gutes Massenspektrum zu erhalten.
Hinweis: Wir empfehlen mindestens 100 Scans zu kombinieren.
- Tandem-Massenspektrometrie von intakten Proteinkomplexen
- Das Massenspektrum zu erwerben, wie beschrieben (Protokoll Abschnitt 3.3). Wählen Sie eine Vorläufer-Ion von einem Proteinkomplex.
- MS/MS-Modus in der Akquisition-Datei von MS verwandeln. Der Vorläufer die MS/MS-Auswahl soll Masse.
- Start der Akquisition bei niedrigen Aufprallenergie. Kombinieren Sie mehrere Scans (ca. 20 Scans) um zu überprüfen, Auswahl der richtigen Vorstufe Masse. Die Aufprallenergie zu erhöhen, bis der Proteinkomplex distanziert. Um eine gute Massenspektrum kombinieren mindestens 500 Scans zu erhalten.
Hinweis: Stripped komplexe haben manchmal niedrigen Intensität. Verbindet so viele Scans wie möglich erhöht möglicherweise die Auflösung und Signal-Rausch-Verhältnis.
- In Lösung Dissoziation von intakten Proteinkomplexen
Hinweis: Um zusätzliche Einblick in Protein-Interaktionen innerhalb der Proteinkomplexe kann Dissoziation in-Lösung durchgeführt werden.- Bereiten Sie die Protein-Probe vor, wie beschrieben (Abschnitt 3.2). Lösungsmittel, die Protein-Probe hinzufügen oder ändern des pH-Werts um intakte komplexe in Sub-komplexe zu distanzieren. Typische Lösungsmittel sind Methanol, Isopropanol und ACN; ändern Sie den pH-Wert von Ammoniumacetat durch die Zugabe von Essigsäure oder Ammoniak-Lösung. Massenspektren zu erwerben, wie oben (§ 3.3 und 3.4) beschrieben.
- Variieren Sie die Menge der Lösungsmittel oder pH-Bereich, verschiedenen Sub-komplexe zu generieren. In der Regel die Menge des Lösungsmittels ist 5-50 % (Endkonzentration) und eine typische pH-Bereich 4-9. Beginnen Sie mit einer niedrigen Konzentration von Lösungsmittel (5 %) oder leichte Veränderung des pH-Wertes und erwerben ein Massenspektrum (siehe Abschnitt 3.3).
- Erhöhen Sie die Menge des Lösungsmittels oder ändern des pH-Werts schrittweise bis Sub-komplexe in Lösung generiert werden. Massenspektren zu analysieren, Sub-komplexe zu erwerben.
- Daten zu kalibrieren
Hinweis: Erworbenen Massenspektren sind extern mit Cäsium Jodid (CsI) Lösung kalibriert.- 100 mg CsI in 1 mL Wasser auflösen.
- Erwerben Sie ein Massenspektrum von CsI. Variieren Sie die Aufprallenergie um CsI Cluster über den gleichen m/Z -Bereich als dem Proteinkomplex analysiert oben zu erhalten.
Achtung: CsI schnell Ausscheidungen an der Spitze der Emitter und Verunreinigungen des Kegels. Erwerben Sie nur so viele scannt wie erforderlich, um eine ausreichende Bandbreite zu gewinnen. Entfernen Sie den Emitter aus der Quelle, wenn Sie fertig sind. - Machen Sie eine Kalibrierung Datei unter Verwendung der erworbenen Massenspektrum und ein CsI-Referenz-Datei.
- Gelten Sie Kalibrierung für erworbene Massenspektren.
Achtung: Kalibrierung der Massenspektren möglicherweise eine dauerhafte Veränderung der Rohdaten. Wenn nicht kalibrierten Spektren erforderlich sind, machen Sie eine Sicherungskopie der Datei.
- Datenverarbeitung und-Analyse
Hinweis: Es gibt viele frei verfügbare Software-Werkzeuge zur Datenanalyse der native Massenspektren; zum Beispiel Massign42 oder UniDec43. Das Protokoll unten beschreibt manuellen Datenanalyse mit Hilfe der Gerätesoftware sowie die Verwendung von Massign für komplexe Proben. Diese Software eignet sich besonders für die Analyse von komplexen Massenspektren. Folgen Sie den Anweisungen für die Verwendung des Programms (http://massign.chem.ox.ac.uk/) online zur Verfügung gestellt.- Für die Datenanalyse glatte Spektren durch Glättung Parameter anpassen. Zentroid Spektren durch Anpassung der Parameter. Berechnen komplexe Massen von zwei benachbarten Gipfeln der Protein-Komplex "Peak Umschlag mit Instrument Software-Tools.
Achtung: Zu intensive Glättung möglicherweise Daten verloren gehen (z.B. Verlust von gebundenen Liganden). - Erzeugen Sie für die Analyse mit Massign42eine Peak-Liste für das Massenspektrum. Linearisieren Sie Datenpunkte des Spektrums und glatt. Verwenden Sie die verschiedenen Software-Tools weisen Proteinkomplexe, berechnen komplexen Zusammensetzung oder komplexe Peak Umschläge zu simulieren.
- Für die Datenanalyse glatte Spektren durch Glättung Parameter anpassen. Zentroid Spektren durch Anpassung der Parameter. Berechnen komplexe Massen von zwei benachbarten Gipfeln der Protein-Komplex "Peak Umschlag mit Instrument Software-Tools.
4. chemische Vernetzung gekoppelt mit Massenspektrometrie
Hinweis: Es gibt zahlreiche Querverbindungen Strategien zur Verfügung. Hier beschreiben wir die Verwendung des BIZ (Sulfosuccinimidyl) Suberate (BS3), ein Amin-reaktive Vernetzer das allgemein verwendet ist, um Protein-Protein-Wechselwirkungen zu studieren.
- Auflösen von 1,43 mg BS3 in 100 µL Wasser eine 25 mM-Stammlösung vorbereiten.
Hinweis: Andere Reagenzien wie Disuccinimidyl Suberate (DSS) sind nicht wasserlöslich und werden in der Regel im Dimethyl Sulfoxid (DMSO) aufgelöst. Einige Quervernetzern sind auch in schweren isotopischer beschriftet Formen erhältlich. Einbeziehung der schweren stabilen Isotope erzeugt Peak Paare von vernetzten di-Peptide in MS Spektren, bei der Datenauswertung hilft. Wenn differentiell mit Quervernetzern beschriftet, sind Stammlösungen beider Varianten vorbereitet und gemischt 1:1.
Achtung: Zur Vermeidung von Dissoziation von Proteinkomplexen von DMSO ist eine hoch konzentrierte Stammlösung vorbereitet und verdünnt mit Wasser oder Puffer vor die Vernetzungsreaktion. - Der Proteinkomplex BS3 hinzufügen. Verwenden Sie unterschiedliche Mengen an BS3 von 0,5-5 mM, optimale Vernetzer Konzentration zu identifizieren. Inkubieren Sie die Reaktionsgemischen bei 25 ° C für 1 h in einem Thermomixer. Verwenden Sie 4 bis 12 % Steigung Gele und führen Sie SDS-PAGE um vernetzende Ergebnisse auszuwerten (siehe Abbildung 2 für ein Beispiel).
Hinweis: Optimale Konzentration von BS3 ist erreicht, wenn höhere Molekulargewicht Protein Bänder, die nicht in das nicht-Cross-linked-Steuerelement sichtbar sind, während der SDS-PAGE erzielt werden, während einzelne Untereinheiten noch sichtbar (Abbildung 2 sind). - Wiederholen Sie die Vernetzungsreaktion mit der optimalen BS3-Konzentration. Inkubieren Sie die Reaktionsgemischen bei 25 ° C für 1 h in einem Thermomixer.
Hinweis: Einige Proteinkomplexe sind nicht bei Raumtemperatur stabil. Die Vernetzungsreaktion kann auch auf Eis durchgeführt werden; die Reaktionszeit muss jedoch angepasst werden. -
Stillen Sie die Vernetzungsreaktion durch Zugabe von Amin Puffer (z. B.50-100 mM Tris-Puffer, pH 7.5, Endkonzentration) zu, oder führen Sie Ethanol Niederschlag um jede verbleibende vernetzende Reagenz zu entfernen.
- Fügen Sie Wasser oder Puffer zum Reaktionsgemisch zu einem Endvolumen von 200 µL. Fügen Sie 600 µL eiskaltes Ethanol und 20 µL 3 M Natriumacetat, pH-Wert 5,3 zu erreichen. Gründlich mischen und Inkubation bei-20 ° C für 2 Stunden oder über Nacht.
Hinweis: Alternativ können die Proteine bei 80 ° C für 30 min oder in flüssigem Stickstoff ausgefällt werden. - Spin-down bei 16.200 x g und 4 ° C für 30 min. vorsichtig überstand.
- Waschen Sie die Tablette mit 1 mL eiskaltes 80 % (V/V) Ethanol. Spin-down bei 16.200 x g und 4 ° C für 30 min. vorsichtig überstand. Trocknen Sie das Pellet in einer Vakuum-Zentrifuge.
- Fügen Sie Wasser oder Puffer zum Reaktionsgemisch zu einem Endvolumen von 200 µL. Fügen Sie 600 µL eiskaltes Ethanol und 20 µL 3 M Natriumacetat, pH-Wert 5,3 zu erreichen. Gründlich mischen und Inkubation bei-20 ° C für 2 Stunden oder über Nacht.
- Führen Sie SDS-PAGE der vernetzten Proteine. Schneiden Sie die Gel-Bands und verdauen Sie das Gel Protein zu, wie oben (Abschnitte 2.1 und 2.2) beschrieben.
Hinweis: In Lösung Verdauung kann auch durchgeführt werden, erfordert jedoch in der Regel zusätzliche Trennung Schritte (z.B. Größe Ausgrenzung Chromatographie der verdauten Peptide). -
Führen Sie flüssige Chromatographie-gekoppelten Massenspektrometrie, wie oben (Abschnitt 2.3) beschrieben. Wie vernetzte Peptide sind in der Regel niedrigen Sie reichlich vorhanden, gelten Sie folgenden Varianten für die massenspektrometrische Analyse, analytische Tiefe der vernetzten Probe zu erhöhen.
- Längere Steigungen während flüssige Chromatographie Trennung verwenden (z. B.90 min statt 65 min, siehe oben).
- Die doppelt aufgeladenen Peptide von HCD Fragmentierung ausschließen.
Hinweis: Doppelt geladenen Peptide sind in der Regel intra-cross-linked Peptide ("Schleife Peptide" oder "Typ 1" Cross-links). - Bei differentiell beschriftet vernetzende Reagenzien, verwenden Sie die Option "Peak picking" während der Analyse, d. h., HCD Fragmentierung wird durch das Vorhandensein von Peak Paare von definierten Massendifferenz in den Massenspektren ausgelöst.
Hinweis: Die "Peak picking" Option auf jeder Massenspektrometer möglicherweise nicht.
- Verwendung pLink Software18 zur Identifizierung von vernetzten di-Peptide. Verwenden Sie minimierte Datenbanken zur Identifizierung. Typische Suchparameter sind: instrument Spektren, HCD; Enzym Trypsin; Max. verpasste Spaltstellen, 3; Variable Modifikationen, Oxidation (Methionin) und Carbamidomethylation (Cystein); Vernetzer, BS3; min. Peptid Länge, 4; Max. Peptid-Länge, 100; min. Peptid Masse, 400 Da; Max. Peptid-Masse, 10.000 Da; FDR, 1 %.
Hinweis: Wenn mit differentiell Quervernetzern beschriftet, die Massenzunahme verursacht vom Linker konfiguriert werden muss. Es gibt auch andere häufig verwendeter Software für Cross-Link-ID, z. B., xQuest17, MassMatrix19, oder XlinkX15. - Bewerten Sie die Ergebnisse der Datenbank-Suche durch die Qualität der Fragmentierung Spektren. Akzeptable Spektren von Querverbindungen sollten Ion beide Peptide (mindestens 4 benachbarten Ionen) in einem angemessenen Signal-Rausch-Verhältnis zeigen.
Hinweis: Wenn mit differentiell vernetzende Reagenzien beschriftet, Peak-Paare in MS Spektren als eine zusätzliche Qualitätskontrolle einsetzbar. - Auf Wunsch visualisieren der Vernetzung resultiert Proteinnetzwerke Interaktion mit Software-Tools (z.B.XVis, XiNET). Nutzung bar Grundstücke oder kreisförmigen Grundstücken zur Visualisierung von Protein-Interaktionen.
Hinweis: Beide Software-Tools sind frei verfügbar auf einem Webserver. Anweisungen Sie detaillierte auf den entsprechenden Internetseiten (https://xvis.genzentrum.lmu.de/ und http://crosslinkviewer.org/).
Ergebnisse
Die Strukturanalyse von Proteinen und die komplexe, die sie bilden ist grundlegend für das Verständnis ihrer Funktion. Massenspektrometrie trägt wesentlich zur strukturellen Untersuchung, dass es auf fast jeder Komplex von Interesse unabhängig von ihrer Größe angewendet werden können oder probieren Sie die Heterogenität. Wir veranschaulichen das Protokoll mithilfe von zwei gut charakterisierten Proteinkomplexe; Erstens, die Hetero-Dodecamer RvB1/B2 von C. Thermophilum und zweitens die Hetero-Octameric CPS Komplex aus E. Coli.
Erstens haben wir die Eiweißbausteinen der zwei komplexe identifiziert. Die Proteine wurden durch SDS-PAGE (Abbildung 2) getrennt und Gel Bands wurden aus dem Gel geschnitten. Nach-Gel Verdauung der Proteine wurden die Peptid-Mischung wurde durch flüssige Chromatographie-gekoppelten Massenspektrometrie analysiert und Peptid und Fragment Massen Datenbank durchsuchen. Nach diesen Workflow wir identifiziert alle Protein-Untereinheiten der zwei komplexe mit hohem Vertrauen, d. h., eine hohe Anzahl von Peptiden mit angemessenen Peptid-Scores wurde beobachtet nachgeben hohe Sequenz Abdeckung für alle Protein-Untereinheiten (Tabelle 1 ).
Dann analysierten wir die intakte RvB1/B2-Komplex durch native Massenspektrometrie (Abb. 3A). Das Massenspektrum ergab zwei Arten, eine auf ca. 8.000 m/Z und einer anderen Spezies auf ca. 11.000-12.000 m/Z. Die kalkulierten Massen für diese Arten entsprechen der Hexameric (RvB1)3(RvB2)3 Ring (ca. 310 kDa) und die Dodecameric Doppel-Ring (RvB1)6(RvB2)6 (ca. 620 kDa). Beide Gipfel Serien zeigen zwei Populationen; Diese stammen aus einer Mischung von seinem markiert und untagged RvB2 Untereinheiten in den komplexen. Eine Kristallstruktur für den RvB1/B2-Komplex wurde bisher36 und zeigt die Anordnung der Doppel-Ring (Abb. 3 b). Native Massenspektrum daher bestätigt die Stöchiometrie der intakten Dodecamer und darüber hinaus zeigt einen stabilen Sub-Komplex. Darüber hinaus sind nebeneinander bestehende Populationen identifiziert.
Um Protein Interaktion Websites in der RvB1/B2-Komplex zu identifizieren, vernetzt wir chemisch gereinigte Anlage mit BS3 Vernetzer. Wir titriert zunächst die Höhe der BS3 während die Vernetzungsreaktion die optimale Konzentration bestimmt. BS3 ist Amin-spezifische und kovalent Links Lysin-Seitenketten sowie die N-Termini der Proteine. Die Vernetzungsreaktion folgte SDS-PAGE (Abbildung 2A). Der nicht-Cross-linked Komplex zeigte sowohl RvB1 als auch RvB2 Untereinheiten. Das Reaktionsgemisch BS3 hinzufügen verursacht kovalente Bindung der Proteine, wodurch Protein-Banden bei höherem Molekulargewicht. Das SDS-Gel zeigt, dass BS3 in zunehmendem Maße ergeben höhere Mengen an vernetzten Arten, während nicht-Cross-linked Protein Untereinheiten reduziert werden. Wir schneiden die Protein-Bands aus dem Gel und folgte das Protokoll zur Verfügung gestellt über Protein Interaktion Websites identifizieren. Eine Beispiel-Spektrum an eine vernetzte di-Peptid wird (Abbildung 3) angezeigt. Das Spektrum zeigt y-Ion Serie beide Peptide Bestätigung dieser Protein-Interaktion. Insgesamt erhalten wir 14 Proteininteraktionen, darunter vier Querverbindungen zwischen RvB1 und RvB2-Untereinheiten und zwei Querverbindungen zwischen den beiden Kopien des RvB2 (Tabelle 2). Die Ergebnisse von BS3 Vernetzung werden in eine Interaktion Netzwerk (Abbildung 3D) mit intramolekularer Wechselwirkungen sowie Interaktionen zwischen verschiedenen Untereinheiten visualisiert. Intramolekularer Querverbindungen legen nahe, dass sowohl RvB1 als auch RvB2 Untereinheiten so Falten, dass N und C-terminalen Domänen in unmittelbarer Nähe befinden. Beachten Sie, dass intramolekularer Wechselwirkungen von Inter molekularen Wechselwirkungen von der gleichen Untereinheit in diesem Fall nicht zu unterscheiden sein. Inter molekulare Querverbindungen zwischen den beiden Untereinheiten wurden ebenfalls beobachtet. Von diesen konnten zwei Inter molekulare Querverbindungen zwischen RvB1 und RvB2 in der Struktur, die Validierung des Vernetzung Ansatzes visualisiert werden. Die Inter molekularen Querverbindungen befinden sich in der Kristallstruktur nicht enthalten sind in flexiblen Schlaufen. Wir identifizierten auch zwei Querverbindungen in RvB2, die die gleichen Peptid-Sequenzen enthalten. Diese Querverbindungen können eindeutig als Inter molekulare, wie sie zwei Kopien des gleichen Proteins (Abbildung 3D) stammen müssen klassifiziert werden. Unsere Vernetzung Experimente offenbaren Protein Interaktion Websites innerhalb der Anlage, sondern auch innerhalb der Protein-Untereinheiten, die Einblicke in ihre strukturelle Anordnung, die auch durch die bestehende Kristallstruktur (Abb. 3 b) bestätigt werden konnte.
Der zweite Protein-Komplex, die wir untersucht wurde CPS. Die native Massenspektrum (Abb. 4A) ergab drei Proteinkomplexe zwischen 6.000 und 12.000 m/Z. Die größte Komplex von 640 kDa entspricht der intakten Hetero-Octamer. Die kleinere komplexe repräsentieren zwei Sub-komplexe; Das Dimer von kleinen und großen CPS Untereinheiten (160 kDa) und eine Hetero-Tetramer mit zwei Kopien jeder Untereinheit (320 kDa). Diese Sub-komplexe liefern erste Einblicke in die Protein-Versammlung; d. h., die großen und kleinen Untereinheiten stehen in direktem Kontakt (wie durch die Hetero-Dimer aufgedeckt) und das Tetramer könnte ein Produkt von zwei Dimere. Um weitere Informationen über die strukturelle Anordnung in der intakten CPS Komplex zu gewinnen, führten wir durch Tandem-Massenspektrometrie (MS/MS) von der Hetero-Octamer und Hetero-Tetramer. In beiden Fällen der kleinen Untereinheit dissoziiert aus der Vorstufe darauf hindeutet, dass die kleine Untereinheit sich in der Peripherie der Versammlung (Abbildung 4 b befindet). In der Tat ist die kleine Untereinheit Peripherie in den verfügbaren Kristall-Struktur (Abbildung 4)-44.
Chemische Vernetzung mit der BS3-Vernetzer wurde ebenfalls durchgeführt. Mit steigenden Mengen, wurde die kovalente Verknüpfung der CPS Untereinheiten erweitert. Nach Verdauung der Proteine und Peptide wie oben beschrieben erhielten viele Protein-Interaktionen innerhalb der großen Untereinheit und einer Querverbindung entsteht in der kleinen Untereinheit (Tabelle 2). Außerdem, ähnlich wie bei der RvB1/B2-Komplex, fanden wir zwei Inter molekulare Querverbindungen zwischen den beiden Kopien von großen CPS-Untereinheit. Diese Querverbindungen legen Sie die zwei großen Untereinheiten, die einander an ihren C-terminale Seiten gegenüber. In einer früheren Studie identifiziert strukturelle Massenspektrometrie und computergestützten Modellierung35, kombinieren wir drei zusätzliche Interaktionen in der großen Untereinheit, die am ehesten ihren Ursprung in der Schnittstelle von zwei Kopien der großen Untereinheit validiert durch die Kristallstruktur und die erhaltenen Modell (Abbildung 4 und Tabelle 2). Diese Interaktionen können die Anordnung der CPS-Kern Komplex bestehend aus vier großen Untereinheiten. Allerdings wurden keine Inter Untereinheiten Querverbindungen zwischen dem kleinen und dem großen Untereinheiten beobachtet. Durch die Überprüfung der verfügbaren Kristallstruktur (Abbildung 4), wird deutlich, dass die Interaktion Oberfläche zwischen den tetrameres Kern des Komplexes, bestehend aus der großen Untereinheit und die peripheren kleinen Untereinheiten sehr klein, was erklären könnte das Fehlen von Inter Untereinheit Interaktionen. Dies durch native Massenspektrometrie, die zeigten, dass die kleine Untereinheit distanziert sich leicht aus der intakten komplexe sehr wahrscheinlich durch eine kleine verbindliche Schnittstelle. Protein-Interaktionen in der CPS komplexe chemische Vernetzung und native Massenspektrometrie kombiniert gestatten dennoch Aufzucht ihrer strukturellen Anordnung (Abbildung 4).
Zusammengenommen, ermöglicht die Kombination von native Massenspektrometrie und chemische Vernetzung gepaart mit massenspektrometrische Identifizierung von Peptiden, die vernetzte Rekonstitution der strukturellen Anordnung der beiden Beispiel-komplexe. Während chemische Vernetzung Anordnung der Protein-Untereinheiten, zum Beispiel die Wechselwirkungen zwischen RvB1 und RvB2 oder innerhalb der tetrameres Kern des CPS, offenbart native Massenspektrometrie geliefert Protein Zusammensetzungen der intakt-komplexe und gängigen Subcomplexes. Bei CPS, wofür keine Inter molekularen Wechselwirkungen zwischen den beiden Untereinheiten konnte, durch chemische Vernetzung beobachtet werden, zufolge native Massenspektrometrie jede große Untereinheit mit einer kleinen Untereinheit (Abbildung 4) interagiert. Tandem-Massenspektrometrie schlug die periphere Lage der kleinen Untereinheit in der Anlage und eine kleine Schnittstelle zwischen beiden Untereinheiten.
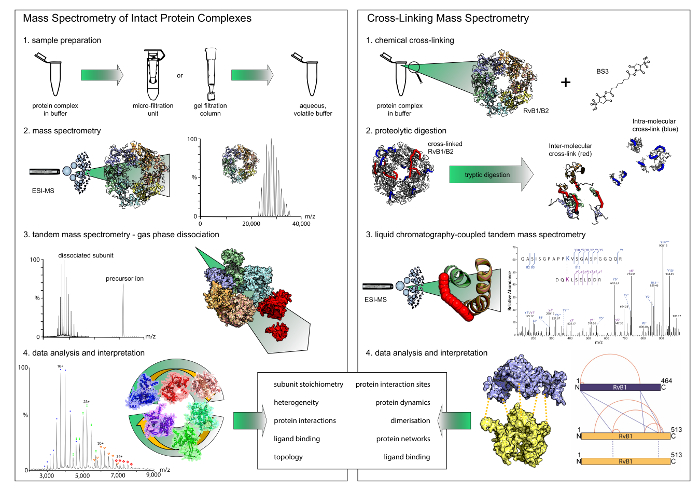
Abbildung 1: Workflow der native Massenspektrometrie und Vernetzung. Beide Techniken liefern ergänzen Ergebnisse. Während Einheimische Massenspektrometrie Zusammensetzungen und Interaktion Module offenbart, gibt Vernetzung Einblicke in Protein Interaktion Websites innerhalb der komplexe. Beachten Sie, dass chemische Vernetzung nur binäre Interaktionen offenbart. (A) der erste Schritt in der nativen Massenspektrometrie ist Buffer Tausch auf einen flüchtigen und wässrigen Puffer mit Filtereinheiten oder Gel Filtration Spalten. Massenspektrometrie von intakten Proteinkomplexe zeigt dann ihre Stöchiometrie. In Tandem-Massenspektrometrie Experimente sind periphere Untereinheiten dissoziiert. (B) für chemische Vernetzung, die Protein-Komplex wird mit einem vernetzende Reagenz inkubiert. Die vernetzte Proteine werden dann in Peptide verdaut, die anschließend durch flüssige Chromatographie-gekoppelten Massenspektrometrie analysiert werden. Bitte klicken Sie hier für eine größere Version dieser Figur.
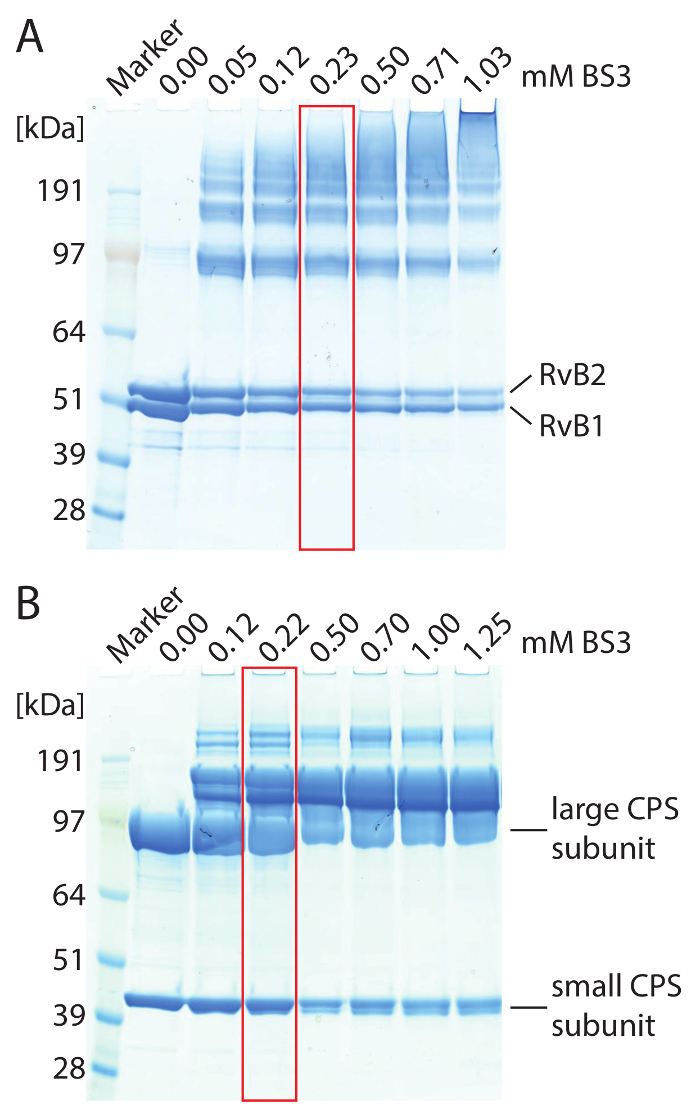
Abbildung 2: SDS-PAGE von RvB1/B2 (A) und CPS (B) komplexe vernetzt. (A) 2,5 µM RvB1/B2 pro Gel Bahn geladen wurden. Die Konzentration der BS3 war vielfältig. Non-Cross-linked RvB1/B2 zeigt die zwei Protein-Untereinheiten auf etwa 50 kDa. Zugabe von BS3 verursacht kovalente Verknüpfung der Protein-Untereinheiten Protein Banden bei höherem Molekulargewicht führt. Die vernetzte Arten wird mit höheren Konzentrationen von BS3 erhöht. Optimale Vernetzung Bedingungen sind (rot) markiert. (B) 10 µM CPS pro Gel Bahn geladen wurden. Der große (90 kDa) und kleine (40 kDa) CPS Untereinheiten sind erhalten. Zugabe von BS3 verursacht kovalente Verknüpfung der Protein-Untereinheiten Protein Banden bei höherem Molekulargewicht führt. Optimale Vernetzung Bedingungen sind (rot) markiert. Bitte klicken Sie hier für eine größere Version dieser Figur.
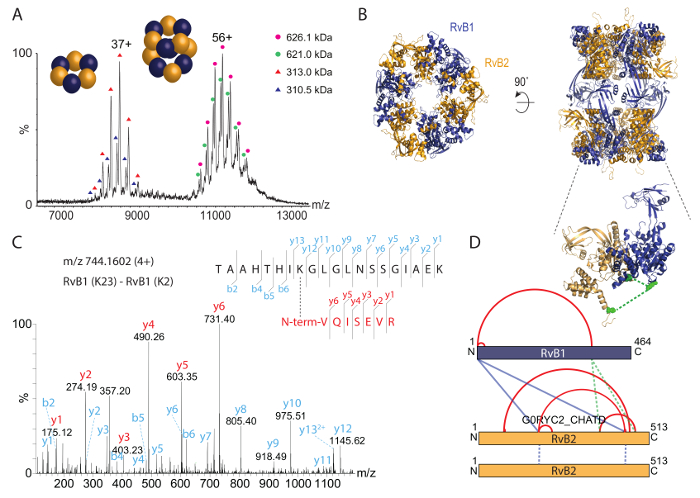
Abbildung 3: Native Massenspektrometrie und chemische Vernetzung der RvB1/B2 Komplex. (A) das native Massenspektrum zeigt zwei Arten von RvB1/B2; die intakte Dodecamer (d.h. (RvB1)6(RvB2)6) bei ca. 11.000 bis 12.000 m/Z und der Hexameric Ring (RvB1)3(RvB2)3 bei ca. 8.000 m/Z. Beide Arten zeigen zwei Populationen aus seinem markiert und untagged RvB2. Das Spektrum wurde von35geändert. (B) der Kristall-Struktur des RvB1/B2 zeigt (PDB-ID 4WVY). Wechsel von RvB1 und RvB2 bilden Untereinheiten zwei Hexameric Ringe. (C) Fragmentierung Spektrum an eine vernetzte di-Peptid. Der N-Terminus des RvB1 war vernetzt mit K23 RvB1. y-Ionen-Serie wurden für beide Peptide (rot und Cyan) erhalten. (D) Intra - und inter - protein-Interaktionen in der RvB1/B2-Komplex erhalten. Intra-cross-Links sind in rot dargestellt und Inter-cross-links in blau angezeigt werden. Der Einsatz zeigt zwei Inter molekulare Querverbindungen zwischen RvB1 und RvB2-Untereinheiten, die in der Kristallstruktur (grün, einfügen) visualisiert werden können. Interaktionen, die ihren Ursprung in zwei RvB2 Kopien werden als blaue gepunktete Linien angezeigt. Bitte klicken Sie hier für eine größere Version dieser Figur.
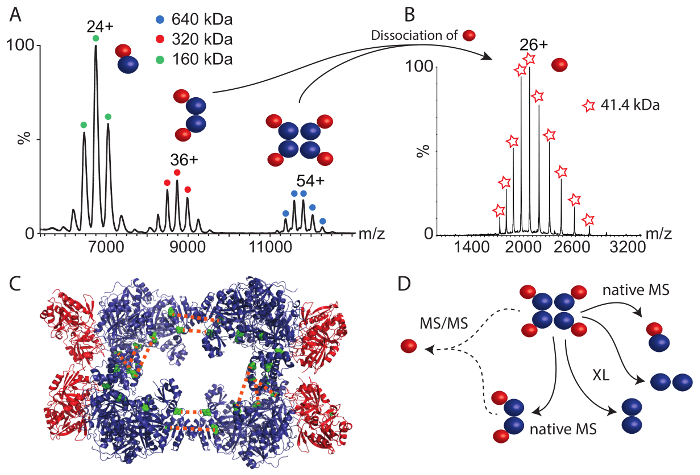
Abbildung 4: Native Massenspektrometrie und chemische Vernetzung der CPS. (A) die native Massenspektrum von CPS zeigt drei komplexe. Die Hetero-Dimer (160 kDa), Hetero-Tetramer (320 kDa) und die Hetero-Octamer (640 kDa). Das Spektrum wurde von35geändert. (B) Tandem-Massenspektrometrie die tetrameres und Octameric CPS komplexe offenbarten Dissoziation von der kleinen Untereinheit des CPS. (C) der Kristall-Struktur des CPS wird (PDB-ID 1BXR) angezeigt. Die großen Untereinheiten bilden einen tetrameres Kern und die kleinen Untereinheiten befinden sich in der Peripherie des Komplexes. Inter molekulare Querverbindungen zwischen den beiden Kopien der großen Untereinheit werden (grün) angezeigt. (D) Interaktionen der großen und kleinen Untereinheiten CPS. Native Massenspektrometrie zeigte Subcomplexes und schlägt eine periphere Lage der kleinen Untereinheit. Chemische Querverbindungen zeigen Arrangements im tetrameres Kern von CPS. Bitte klicken Sie hier für eine größere Version dieser Figur.
Tabelle 1: Datenbank Suchergebnisse. Die Proteine wurden durch flüssige Chromatographie-gekoppelten Massenspektrometrie und Suche Datenbank identifiziert. Die Protein-Namen Beitritt Nummer und Beschreibung sowie Eiweiß Masse erhalten. Die Protein-Partitur, Anzahl der beobachteten Spektren pro Protein und die Anzahl der beobachteten Peptid-Sequenzen werden aufgelistet. Die fünf Peptide mit höchsten Punktzahlen der Maskottchen-Peptide sind für jedes Protein-Untereinheit aufgeführt. Bitte klicken Sie hier, um diese Datei herunterladen.
Tabelle 2: Querverbindungen beobachtet in RvB1/B2 und CPS. Die Untereinheiten der komplexe und vernetzte Rückstände sind gegeben. Die Art der Querverbindung entsteht (Intra- oder Inter molekulare) aus überlappenden Peptid-Sequenzen oder einer früheren Studie35offenbart wurde. Bitte klicken Sie hier, um diese Datei herunterladen.
Diskussion
Protokolle sind Massenspektrometrie-basierte Strukturanalyse von Multi-Untereinheit Proteinkomplexe vorgesehen. In das Protokoll beschriebenen zwei Techniken meist ergänzende Ergebnisse liefern und sind gut geeignet, um Einblicke in die strukturellen Vorkehrungen innerhalb von Protein (-Liganden) komplexe, die schwer durch herkömmliche strukturelle Techniken zu untersuchen sind. Native Massenspektrometrie liefert Einblicke in Protein Zusammensetzungen sowie Protein-Interaktionen durch die Analyse von Subcomplexes und stabilen Interaktion Module. Vernetzung, auf der anderen Seite liefert Informationen über direkten Kontaktstellen. Abhängig von der Vernetzer verwendet eine gewisse Flexibilität kann oder sollten in die Analyse einbezogen werden.
Die zur Verfügung gestellten Protokolle sind im Allgemeinen einfach durchzuführen und nicht zeitaufwendig. Das gesamte Protokoll kann innerhalb einer Woche durchgeführt werden und kann auf fast allen Proteinkomplexen angewendet werden, obwohl ein gewisses Maß von der Protein-Komplex für die erfolgreiche Analyse erforderlich ist. Vorbereitung der Probe ist einfach und erfordert keine speziell gereinigtes Protein-komplexe. Allerdings ist ein Fallstrick Kontamination der Probe während der Probenvorbereitung für Masse Spektrometrie-basierte Proteinidentifikation. Diese Verunreinigungen in den meisten Fällen gehören Keratine, die aus Staub, Haut- oder Haarfarbe stammen. Daher sollten zusätzliche wie das Tragen von Handschuhen und Kittel, Filtrierung wässrigen Puffer und hochreinen Lösungsmitteln während der Probenvorbereitung für Masse Spektrometrie-basierte Proteinidentifikation achten. Andere kontaminierende Proteine wie Chaperone sind in der Regel während der Proteinreinigung, z.B.eingeführt, wenn Affinität-Tags verwenden. In diesen Fällen sollte Proteinreinigung zum Beispiel verbessert werden, indem man Waschschritte. In jedem Fall Protein Verunreinigungen in der Probe sind leicht erkennbar, während die Datenbankrecherche durch Weglassen des Taxonomie-Filters (d.h.gegen Proteine von allen Tierarten suchen). Wenn nur ein paar Peptide eingehalten werden (d.h., eine eiweißarme Abdeckung konnte abgerufen werden), obwohl genügend Probe zur Verfügung steht, es kann notwendig sein, verwenden Sie ein anderes Proteinase während der Verdauung. Im allgemeinen ergibt Trypsin eine ausreichende Anzahl von Peptiden; jedoch in einigen Fällen wie Membranproteine oder Membrandomänen von Proteinen, die Anzahl der Folgen Spaltstellen reduziert und andere Enzyme gezielt hydrophobe Aminosäuren sind eine bessere Wahl.
In Bezug auf die Instrumentierung ist ein besonders modifizierte Instrument für native Massenspektrometrie unterhält nicht-kovalente Wechselwirkungen während der Übertragung in die Gasphase erforderlich. Verschiedene Gerätetypen wurden eingeführt, einschließlich der Q-ToF und Orbitrap Instrumente. Während modifizierte Q-ToF Massenspektrometer seit mehreren Jahren für native Massenspektrometrie im Handel erhältlich sind, letztere wurden erst vor kurzem eingeführt und in den meisten Fällen erfordern spezielle Änderung45. Jedoch die Anwendung des hochauflösenden Instrumente erlaubt Studium Bindung von mehrere Liganden und deren Quantifizierung46,47 und ist vielversprechend für zukünftige Anwendungen.
Um vernetzte di-Peptide durch flüssige Chromatographie-gekoppelten Massenspektrometrie zu identifizieren, können standard-Verfahren mit wenigen Änderungen angewendet werden. Jedoch ist die Suche in der Datenbank der limitierende Faktor Spezialsoftware kann selten befassen sich mit großen Datenbanken sowie reduzierte Datenbanken, in denen die Protein-Untereinheiten der Anlagen erforderlich sind. Neuere Studien verwendeten Masse Spektrometrie spaltbaren Quervernetzern targeting Protein-Interaktionen in der gesamten Zelle Lysates48,49. Die Verwendung von chemischen Quervernetzern, die meist in Tandem-Massenspektrometrie Experimente fragment liefert lineare Peptide (von der Vernetzer geändert), die durch eine weitere Fragmentierung identifiziert werden können und Suche Datenbank lineare Peptide, und dies reduziert Suchzeit und rechnerische Suchraum. Um diese Experimente durchzuführen, ist jedoch ein Ionenfallen Masse Analysator oder ein Hybrid-Massenspektrometer mit einer Ionenfalle erforderlich. Im Allgemeinen als Fehlalarme ein wichtiges Thema sind, sind Massenspektren von vernetzten Peptiden oft manuell durch die Qualität ihrer Fragment Spektren überprüft die Daten Analysezeit enorm erweitert. Robuste scoring-Systeme zu entwickeln, die angewendet werden können, ohne weitere Validierungsschritte daher mögliche zukünftige Anwendungen sind. Eine Möglichkeit, die Datenanalyse zu verbessern und die Zahl der Fehlalarme zu reduzieren war die Einführung der false Discovery Rate Berechnungen und deren Anwendung auf die Vernetzung von Datensätzen50.
Im Allgemeinen können die hier beschriebenen Techniken mit weiteren Massenspektrometrie Techniken ergänzt werden (z.B.kovalente Kennzeichnung) zur Leistungssteigerung von der Analyse. Weitere Änderungen und Verbesserungen der Protokolle können leicht umgesetzt werden. Als solche entwirrt vergleichende vernetzende34 Konformationsänderungen in der Protein-Baugruppe. Weiterentwicklungen in der nativen Massenspektrometrie ermöglichen heute die Analyse der Membran Proteine51,52 und deren Interaktion mit Lipiden28,52,53,54 . Neuentwicklungen von hochauflösenden Massenspektrometer für native Massenspektrometrie wurden erweitert, der Anwendung und Liganden Bindung, z.B., Bindung von Lipiden, Membranproteine, können nun in die Analyse45, einbezogen werden 46. in Kombination mit computergestützten Modellierungsansätze, diese Techniken Strukturmodellen verschiedener Auflösung55liefern können. Wenn keine Kristallstrukturen für die intakten komplexen oder einzelnen Untereinheiten zur Verfügung stehen, kann Massenspektrometrie erste Einblicke in Protein-Interaktionen und die Topologie der unbekannten Anlage liefern. Je nach der verwendeten Techniken und die erzielten Ergebnisse erhalten Sie mit niedriger Auflösung Modelle des unbekannten komplexes56,57,58. Kristallstrukturen oder Homologie Modelle verfügbar sind, kann sogar in der Nähe von Native Modelle59durch die strukturellen Informationen seitens der Massenspektrometrie erzielt werden.
Im Vergleich zu anderen strukturellen Techniken, Massenspektrometrie hat den Vorteil, dass es erfordert geringe Stichprobe Mengen, heterogene Proben bewältigen kann und gilt für Proteinkomplexe unbegrenzter Größe. Darüber hinaus erlaubt Massenspektrometrie die Untersuchung des dynamischen Protein-Systeme. Verschiedene Populationen von Eiweiß oder Protein-Komplex, die in der Lösung vorhanden sind in der Regel gemeinsam analysiert und daher, anders als mit anderen strukturellen Techniken die Auswahl bestimmter Bevölkerungsgruppen erfordern alle Konformationen sind beibehalten während Analyse und sind in einem einzigen Experiment bewertbar. Quantitative vernetzende Ansätze wurden kürzlich eingeführten34,60,61 , sind vielversprechend für zukünftige Anwendungen Konformationsänderungen unter verschiedenen Bedingungen zu beschreiben.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Wir danken unseren Kollegen für hilfreiche Diskussionen. Wir danken auch Ilme Schlichting und Karl-Peter Hopfner für die Bereitstellung von Proteinkomplexen. Wir anerkennen, dass Mittel des Bundesministeriums für Bildung und Forschung (BMBF, ZIK Programm, 03Z22HN22), den Europäischen Fonds für regionale Entwicklung (EFRE, ZS/2016/04/78115) und der MLU Halle-Wittenberg, c.s. und Finanzierung vom Wellcome Trust (109854/658 Z/15/Z), A.P.
Materialien
| Name | Company | Catalog Number | Comments |
| Chemicals, Reagents, Consumables | |||
| 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid (HEPES) | Sigma Aldrich | H4034 | buffer |
| Acetic acid | Sigma Aldrich | 695092 | pH |
| Acetonitrile Optima LC/MS | Fisher Chemicals | A955-1 | solvent |
| Amicon Ultra centrifugal devices (different MWCO) | Millipore | i.e. UFC500396 | buffer exchange, concentration |
| Ammonium acetate solution | Sigma Aldrich | A2706 | native MS |
| Ammonium bicarbonate | Sigma Aldrich | 9830 | in-gel digestion |
| Ammonium solution | Sigma Aldrich | 9859 | pH |
| Bis(Sulfosuccinimidyl)suberate-d0 (BS3-d0) | Thermo Scientific | 21590 | cross-linker |
| Bis(Sulfosuccinimidyl)suberate-d4 (BS3-d4) | Thermo Scientific | 21595 | cross-linker |
| Caesium iodide | Sigma Aldrich | 203033 | calibration |
| Calcium chloride | Sigma Aldrich | C5670 | in-gel digestion |
| Capillaries (1.0 OD × 0.78 ID × 100 L mm) | Harvard Apparatus | 30-0038 | native MS |
| Disuccinimidyl suberate (DSS) | Thermo Scientific | 21655 | cross-linker |
| DL-Dithiothreitol | Sigma Aldrich | D5545 | in-gel digestion |
| DMSO (dimethyl sulfoxide) | Sigma Aldrich | D8418 | solvent |
| Ethanol | Fisher Chemicals | BP2818 | solvent |
| Formic acid Optima LC/MS | Fisher Chemicals | A117-50 | solvent supplement |
| Instant Blue Coomassie staining solution | expedeon | ISB1L | staining solution for gel electrophoresis |
| Invitrogen NuPAGE 4-12% Bis-Tris gels (1 mm, 10 well) | life technologies | NP0323BOX | gel electrophoresis |
| Iodoacetamide | Sigma Aldrich | I1149 | In-gel digestion |
| Isopropyl alcohol, HPLC grade | Fisher Chemicals | P750717 | solvent |
| Methanol | Fisher Chemicals | A456-212 | solvent |
| Micro BioSpin 6 columns | BioRad | 732-6222 | buffer exchange |
| NuPAGE Antioxidant | invitrogen | NP0005 | running buffer, gel electrophoresis |
| NuPAGE LDS Sample Buffer (4 ×) | invitrogen | NP0007 | sample loading buffer (non-reducing) for gel electrophoresis |
| NuPAGE MES SDS Running buffer | life technologies | NP0001 | gel electrophoresis |
| NuPAGE MOPS SDS Running buffer | life technologies | NP0001 | gel electrophoresis |
| NuPAGE Sample Reducing Agent (10 ×) | invitrogen | NP0004 | reducing Agent for gel electrophoresis |
| PBS - phosphate buffered saline | Sigma Aldrich | P4417 | buffer |
| SeeBlue Plus2 Prestained Standard Protein Marker | invitrogen | LC5925 | prestained Protein Marker for gel electrophoresis |
| Sodium acetate | Sigma Aldrich | S2889 | ethanol precipitation |
| Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 252859 | buffer |
| Trypsin | Sigma Aldrich | TRYPSEQ-RO (11418475001) | in-gel digestion |
| Vivaspin centrifugal devices (different MWCO) | Sartorius | i.e. VS0101 | buffer exchange, concentration |
| Water for HPLC | Sigma Aldrich | 270733-2.5L-M | solvent |
| Name | Company | Catalog Number | Comments |
| Instruments | |||
| Centrifuge Heraeus Fresco 21, bench-top centrifuge | Thermo Scientific | 75002425 | |
| Flaming / Brown Micropipette puller Model P-1000 | Sutter Instruments | P-1000 | |
| Gelelectrophoresis chamber Xcell SureLock MiniCell | Invitrogen | EI0001 | |
| Gold Coater Quorum Q150R S | Quorum Technologies Ltd. | Q150RS | |
| Horizontal gel shaker Rotamax 120 T | Heidolph Instruments | 544-41200-00 | |
| Q-TOF Ultima mass spectrometer, MS Vision high mass upgrade | Waters (Micromass) | - | |
| reversed-phase C18 analytical column Acclaim PepMap (C18, 75 mm I.D., 50 cm, 3 mm pore size) | Thermo Scientific | 164570 | |
| reversed-phase C18 pre-column (C18, 150 mm I.D., 2 cm, 5 mm pore size) | Thermo Scientific | 164213 | |
| scalpel | Fisher Scientifc | 10463989 | |
| SpeedVac SPD121P vacuum centrifuge | Thermo Scientific | SPD121DP-230 | |
| Thermomixer C | Eppendorf | 5382000015 | |
| Tweezers AA | Sigma Aldrich | Z680184-1EA | |
| Ultrasonic cleaner USC-TH, sonication bath | VWR | 142-0084 | |
| UltiMate Dionex 3000 nano-LC system, coupled to Q-Exactive plus hybrid mass spectrometer (nano-ESI source) | Thermo Scientific | 0726030+ | |
| Name | Company | Catalog Number | Comments |
| Software, Software Tools, Database search | |||
| Mascot | www.matrixscience.com | ||
| Massign | http://massign.chem.ox.ac.uk | ||
| MassLynx | Micromass | ||
| MassMatrix | Xu, H. J Proteome Res. 9 (2010) | ||
| MaxQuant | Cox, J. Nat. Biotechn. 26 (2008) | ||
| pLink | Yang, B. Nat Methods 9 (2012) | ||
| pXtract conversion tool | http://pfind.ict.ac.cn/downloads.html | ||
| UniDec | Marty, M.T. Anal. Chem. 87 (2015) | ||
| XCalibur | Thermo Scientific | ||
| XiNET | http://crosslinkviewer.org | ||
| XLinkX | Liu, F. Curr Op Struct Biol 35 (2015) | ||
| xQuest | Rinner, O. Nat Methods 5 (2008) | ||
| Xvis | https://xvis.genzentrum.lmu.de |
Referenzen
- Campbell, I. D. Timeline: the march of structural biology. Nat Rev Mol Cell Biol. 3 (5), 377-381 (2002).
- Liko, I., Allison, T. M., Hopper, J. T., Robinson, C. V. Mass spectrometry guided structural biology. Curr Opin Struct Biol. 40, 136-144 (2016).
- Robinson, C. V. From molecular chaperones to membrane motors: through the lens of a mass spectrometrist. Biochem Soc Trans. 45 (1), 251-260 (2017).
- Lossl, P., van de Waterbeemd, M., Heck, A. J. The diverse and expanding role of mass spectrometry in structural and molecular biology. EMBO J. 35 (24), 2634-2657 (2016).
- Wang, L., Chance, M. R. Protein Footprinting Comes of Age: Mass Spectrometry for Biophysical Structure Assessment. Mol Cell Proteomics. 16 (5), 706-716 (2017).
- Steen, H., Mann, M. The ABC's (and XYZ's) of peptide sequencing. Nat Rev Mol Cell Biol. 5 (9), 699-711 (2004).
- Olsen, J. V., Mann, M. Status of large-scale analysis of post-translational modifications by mass spectrometry. Mol Cell Proteomics. 12 (12), 3444-3452 (2013).
- Schmidt, C., Robinson, C. V. Dynamic protein ligand interactions--insights from MS. FEBS J. 281 (8), 1950-1964 (2014).
- Marcsisin, S. R., Engen, J. R. Hydrogen exchange mass spectrometry: what is it and what can it tell us?. Anal Bioanal Chem. 397 (3), 967-972 (2010).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Leitner, A., Lindner, W. Current chemical tagging strategies for proteome analysis by mass spectrometry. J Chromatogr B Analyt Technol Biomed Life Sci. 813 (1-2), 1-26 (2004).
- Leitner, A., Lindner, W. Chemistry meets proteomics: the use of chemical tagging reactions for MS-based proteomics. Proteomics. 6 (20), 5418-5434 (2006).
- Kiselar, J. G., Chance, M. R. Future directions of structural mass spectrometry using hydroxyl radical footprinting. J Mass Spectrom. 45 (12), 1373-1382 (2010).
- Xu, G., Chance, M. R. Hydroxyl radical-mediated modification of proteins as probes for structural proteomics. Chem Rev. 107 (8), 3514-3543 (2007).
- Liu, F., Heck, A. J. Interrogating the architecture of protein assemblies and protein interaction networks by cross-linking mass spectrometry. Curr Opin Struct Biol. 35, 100-108 (2015).
- Rappsilber, J. The beginning of a beautiful friendship: cross-linking/mass spectrometry and modelling of proteins and multi-protein complexes. J Struct Biol. 173 (3), 530-540 (2011).
- Rinner, O., et al. Identification of cross-linked peptides from large sequence databases. Nat Methods. 5 (4), 315-318 (2008).
- Yang, B., et al. Identification of cross-linked peptides from complex samples. Nat Methods. 9 (9), 904-906 (2012).
- Xu, H., Hsu, P. H., Zhang, L., Tsai, M. D., Freitas, M. A. Database search algorithm for identification of intact cross-links in proteins and peptides using tandem mass spectrometry. J Proteome Res. 9 (7), 3384-3393 (2010).
- Leitner, A., Faini, M., Stengel, F., Aebersold, R. Crosslinking and Mass Spectrometry: An Integrated Technology to Understand the Structure and Function of Molecular Machines. Trends Biochem Sci. 41 (1), 20-32 (2016).
- Heck, A. J. Native mass spectrometry: a bridge between interactomics and structural biology. Nat Methods. 5 (11), 927-933 (2008).
- Sharon, M., Robinson, C. V. The role of mass spectrometry in structure elucidation of dynamic protein complexes. Annu Rev Biochem. 76, 167-193 (2007).
- Goth, M., Pagel, K. Ion mobility-mass spectrometry as a tool to investigate protein-ligand interactions. Anal Bioanal Chem. , (2017).
- Uetrecht, C., Rose, R. J., van Duijn, E., Lorenzen, K., Heck, A. J. Ion mobility mass spectrometry of proteins and protein assemblies. Chem Soc Rev. 39 (5), 1633-1655 (2010).
- Hernandez, H., Robinson, C. V. Determining the stoichiometry and interactions of macromolecular assemblies from mass spectrometry. Nat Protoc. 2 (3), 715-726 (2007).
- Sobott, F., Hernandez, H., McCammon, M. G., Tito, M. A., Robinson, C. V. A tandem mass spectrometer for improved transmission and analysis of large macromolecular assemblies. Anal Chem. 74 (6), 1402-1407 (2002).
- Rostom, A. A., et al. Detection and selective dissociation of intact ribosomes in a mass spectrometer. Proc Natl Acad Sci U S A. 97 (10), 5185-5190 (2000).
- Zhou, M., et al. Mass spectrometry of intact V-type ATPases reveals bound lipids and the effects of nucleotide binding. Science. 334 (6054), 380-385 (2011).
- Uetrecht, C., et al. High-resolution mass spectrometry of viral assemblies: molecular composition and stability of dimorphic hepatitis B virus capsids. Proc Natl Acad Sci U S A. 105 (27), 9216-9220 (2008).
- Morgner, N., et al. Hsp70 forms antiparallel dimers stabilized by post-translational modifications to position clients for transfer to Hsp90. Cell Rep. 11 (5), 759-769 (2015).
- Schmidt, C., Beilsten-Edmands, V., Robinson, C. V. The joining of the Hsp90 and Hsp70 chaperone cycles yields transient interactions and stable intermediates: insights from mass spectrometry. Oncotarget. 6 (21), 18276-18281 (2015).
- Schmidt, C., Beilsten-Edmands, V., Mohammed, S., Robinson, C. V. Acetylation and phosphorylation control both local and global stability of the chloroplast F1 ATP synthase. Sci Rep. 7, 44068 (2017).
- Schmidt, C., et al. Comparative cross-linking and mass spectrometry of an intact F-type ATPase suggest a role for phosphorylation. Nat Commun. 4, 1985 (2013).
- Schmidt, C., Robinson, C. V. A comparative cross-linking strategy to probe conformational changes in protein complexes. Nat Protoc. 9 (9), 2224-2236 (2014).
- Schmidt, C., et al. Surface Accessibility and Dynamics of Macromolecular Assemblies Probed by Covalent Labeling Mass Spectrometry and Integrative Modeling. Anal Chem. 89 (3), 1459-1468 (2017).
- Lakomek, K., Stoehr, G., Tosi, A., Schmailzl, M., Hopfner, K. P. Structural basis for dodecameric assembly states and conformational plasticity of the full-length AAA+ ATPases Rvb1 · Rvb2. Structure. 23 (3), 483-495 (2015).
- Mareya, S. M., Raushel, F. M. A molecular wedge for triggering the amidotransferase activity of carbamoyl phosphate synthetase. Biochemistry. 33 (10), 2945-2950 (1994).
- Shevchenko, A., Wilm, M., Vorm, O., Mann, M. Mass spectrometric sequencing of proteins silver-stained polyacrylamide gels. Anal Chem. 68 (5), 850-858 (1996).
- Olsen, J. V., et al. Parts per million mass accuracy on an Orbitrap mass spectrometer via lock mass injection into a C-trap. Mol Cell Proteomics. 4 (12), 2010-2021 (2005).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol. 26 (12), 1367-1372 (2008).
- Kirshenbaum, N., Michaelevski, I., Sharon, M. Analyzing large protein complexes by structural mass spectrometry. J Vis Exp. (40), (2010).
- Morgner, N., Robinson, C. V. Massign: an assignment strategy for maximizing information from the mass spectra of heterogeneous protein assemblies. Anal Chem. 84 (6), 2939-2948 (2012).
- Marty, M. T., et al. Bayesian deconvolution of mass and ion mobility spectra: from binary interactions to polydisperse ensembles. Anal Chem. 87 (8), 4370-4376 (2015).
- Thoden, J. B., Wesenberg, G., Raushel, F. M., Holden, H. M. Carbamoyl phosphate synthetase: closure of the B-domain as a result of nucleotide binding. Biochemistry. 38 (8), 2347-2357 (1999).
- Rose, R. J., Damoc, E., Denisov, E., Makarov, A., Heck, A. J. High-sensitivity Orbitrap mass analysis of intact macromolecular assemblies. Nat Methods. 9 (11), 1084-1086 (2012).
- Gault, J., et al. High-resolution mass spectrometry of small molecules bound to membrane proteins. Nat Methods. 13 (4), 333-336 (2016).
- Mehmood, S., et al. Mass spectrometry captures off-target drug binding and provides mechanistic insights into the human metalloprotease ZMPSTE24. Nat Chem. 8 (12), 1152-1158 (2016).
- Kaake, R. M., et al. A new in vivo cross-linking mass spectrometry platform to define protein-protein interactions in living cells. Mol Cell Proteomics. 13 (12), 3533-3543 (2014).
- Liu, F., Rijkers, D. T., Post, H., Heck, A. J. Proteome-wide profiling of protein assemblies by cross-linking mass spectrometry. Nat Methods. 12 (12), 1179-1184 (2015).
- Walzthoeni, T., et al. False discovery rate estimation for cross-linked peptides identified by mass spectrometry. Nat Methods. 9 (9), 901-903 (2012).
- Barrera, N. P., Di Bartolo, N., Booth, P. J., Robinson, C. V. Micelles protect membrane complexes from solution to vacuum. Science. 321 (5886), 243-246 (2008).
- Barrera, N. P., et al. Mass spectrometry of membrane transporters reveals subunit stoichiometry and interactions. Nat Methods. 6 (8), 585-587 (2009).
- Laganowsky, A., et al. Membrane proteins bind lipids selectively to modulate their structure and function. Nature. 510 (7503), 172-175 (2014).
- Marcoux, J., et al. Mass spectrometry reveals synergistic effects of nucleotides, lipids, and drugs binding to a multidrug resistance efflux pump. Proc Natl Acad Sci U S A. 110 (24), 9704-9709 (2013).
- Politis, A., Schmidt, C. Structural characterisation of medically relevant protein assemblies by integrating mass spectrometry with computational modelling. J Proteomics. , (2017).
- Hall, Z., Politis, A., Robinson, C. V. Structural modeling of heteromeric protein complexes from disassembly pathways and ion mobility-mass spectrometry. Structure. 20 (9), 1596-1609 (2012).
- Pukala, T. L., et al. Subunit architecture of multiprotein assemblies determined using restraints from gas-phase measurements. Structure. 17 (9), 1235-1243 (2009).
- Politis, A., et al. Topological models of heteromeric protein assemblies from mass spectrometry: application to the yeast eIF3:eIF5 complex. Chem Biol. 22 (1), 117-128 (2015).
- Politis, A., et al. A mass spectrometry-based hybrid method for structural modeling of protein complexes. Nat Methods. 11 (4), 403-406 (2014).
- Fischer, L., Chen, Z. A., Rappsilber, J. Quantitative cross-linking/mass spectrometry using isotope-labelled cross-linkers. J Proteomics. 88, 120-128 (2013).
- Yu, C., et al. Developing a Multiplexed Quantitative Cross-Linking Mass Spectrometry Platform for Comparative Structural Analysis of Protein Complexes. Anal Chem. 88 (20), 10301-10308 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten