Method Article
化学架橋と複合体タンパク質複合体の建築を学びそのまま蛋白質の複合体の質量分析法を組み合わせた
要約
タンパク質複合体の建築はその機能は欠かせません。様々 な質量分析技術を組み合わせることをそのアセンブリを勉強する強力な証明しました。化学架橋とネイティブの質量分析のためのプロトコルを提供し、これらの相補的な技術が複数のサブユニット蛋白質アセンブリのアーキテクチャを明らかにする方法を示します。
要約
タンパク質は、様々 な細胞機能を遂行するフォームのアクティブ ・動的アセンブリへの配位子と対話します。したがって、これらの相互作用の解明です細胞プロセスの理解のための基礎。しかし、多くの蛋白質の複合体は動的アセンブリ、従来構造手法からはアクセスできません。質量分析法はこれらのアセンブリの構造研究に貢献し、特に様々 な質量分析技術の組み合わせの構造配置に貴重な洞察力を提供します。
この記事で述べるアプリケーション、2 つの相補的な質量分析技術の組み合わせ、すなわち質量とネイティブの質量と相まって化学架橋します。化学架橋、化学試薬を用いた近接中のアミノ酸の結合リンクが含まれます。プロテアーゼで消化後、架橋・ ディ ・ ペプチドは、質量分析法による識別され、タンパク質の相互作用部位が発見されます。ネイティブの質量分析法は、質量分析計における気相そのまま蛋白質アセンブリの分析を一方です。タンパク質とリガンドの相互作用と同様、蛋白質の結果を明らかにします。両方の方法は、したがってタンパク質-リガンド アセンブリおよびそれらの組み合わせは以前の研究の強力な証明の構造に関する補足情報を提供します。
概要
タンパク質複合体の構造研究は、特に重要な細胞プロセスの理解のためになりました。その結果、多くの場合、技術を開発し、構造生物学1で改善が。ただし、これらのテクニックは時々 調査中蛋白質の複合体の不均一性や柔軟性、サイズにより、アプリケーションで制限されます。質量分析法は、これらの課題に対処することができますそして、したがって、構造生物学2,3,4、5の強力なツールとして浮上しています。質量分析法の最大の利点は、複雑で、異種混合6でもタンパク質を明確に識別する能力。このため、蛋白質は通常 endoproteinases と消化され、得られたペプチド混合物を液体クロマトグラフィーで分離し、質量分析計に直接溶離されます。ペプチッド固まりが決定されその後、ペプチドのフラグメント化の前駆イオンを選択しました。タンパク質は検索ペプチドおよび知られているデータベースに対して対応するフラグメント大衆によって、識別されます。この手順には、ペプチドやタンパク質も前駆体ペプチドと変更7を運ぶフラグメント イオンの質量変化をもたらします彼らのポスト翻訳の修正の識別だけでなくことができます。構造質量分析法における多くの技術のいくつかは、この原則4,8に基づいています。例えば、ラベリングなど水素重水素交換9,10技術、化学ラベリング戦略11,12、またはヒドロキシルラジカル足印刷13,14、特定の条件の下で蛋白質の表面のアクセシビリティに洞察力を与えます。
別の手法は、機能グループを通じて近くのアミノ酸の結合のリンケージを含む架橋 (化学) です。このためアミノ酸や紫外線アクティブ化可能な試薬、化学試薬が採用15,16です。、相互リンク後タンパク質はプロテアーゼと通常加水分解し、架橋・ ディ ・ ペプチドが液体クロマトグラフィー質量分析法によって分析されます。データベース検索、架橋物の同定ただし、様々 なタンパク質と別々 の地域17,18,19のペプチド配列を連結する特殊なソフトウェアの使用が必要です。化学ゲルビーズの使用には、関心のほとんどすべてのタンパク質の複合体を採用することができ、宿主細胞に興味の蛋白質を表現するときにのみ達成することができます紫外線アクティブ化可能なアミノ酸の取り込みを必要としない利点があります。など、汎用性の高いツールです架橋とも大きな蛋白質アセンブリ20の多くの構造研究で採用された正常に。
そのまま蛋白質複合体 ('native' 質量分析法とも呼ばれます)、その一方で、質量は、ペプチドにタンパク質とタンパク質加水分解なしの分析を含みます。組成、不均一性、化学量論、トポロジ、およびタンパク質複合体21,22のサブユニットの相互作用を明らかにします。イオン移動度と組み合わせて、さらにネイティブの質量はその構造23,24の決定をことができます。これは従来構造技術によって評価しにくいタンパク質複合体の構造の調査のための強力なツールになります。ただし、ネイティブの質量分析法は、エレクトロ スプレー イオン化の間に非共有結合蛋白質の相互作用を管理する分析バッファーを必要があります。これは通常アンモニウム酢酸25など水の揮発性のバッファーを使用して実現されます。さらに、楽器の変更が質量分析計26の気相に伝達の間に解離を防ぐために必要です。この方法で適用すると、多くの (大) 蛋白質の複合体を行った。印象的な例は、そのままリボゾーム27、ATP 合成酵素28、またはウイルス29の研究です。
ネイティブの質量と架橋の組み合わせを前の研究で特に成功した証明しました。予想外の Hsp70 ダイマーを含むシャペロン複合体の結果がそのまま蛋白質の複合体の質量分析実験から取得でした化学架橋の蛋白質の手配を明らかにしながら、例えば、アセンブリの30,31。別の研究では、複雑なそのまま ATP のシンターゼのポスト翻訳の修正の効果を調べた。ネイティブの質量分析法は、リン酸化やアセチル化32,33の有無でタンパク質の複雑な安定性への洞察を提供しました。比較架橋戦略34は、異なる条件の下で複雑な蛋白質の構造変化を明らかにしました。
ここでは、質量分析法、(化学) 架橋とデータ分析と解釈 (図 1) を含むネイティブの質量分析法によるタンパク質同定のプロトコルをいたします。2 つのよ特徴付けられたタンパク質複合体の助けを借りて、これらのメソッドから取得した相補的な結果の組み合わせは実証35です。我々 のプロトコルは、特定の純度と濃度に精製することができます任意の蛋白質のアセンブリに適用できます。アプローチはデータ解析データ、すなわちデータベースのサイズを架橋による限られたいくつかのケースで採用、必要な検索空間と時間を決定します。さらに、識別されたクロスリンクの手動の検証が必要、さらに出力を低減します。ネイティブの質量分析、サンプルの質、例えばバッファー制限は主、付加体の水溶液と揮発性のバッファーによって交換する浄化と可能性の時に使用します。ただし、通常構造の分析のための精製タンパク質複合体は私達のプロトコルの解析に成功に必要な品質を持っています。
プロトコル
1. タンパク質複合体の精製
- 最適化された標準プロトコルに従って複雑なタンパク質を準備します。
注: プロトコルはChaetomium thermophilumと大腸菌由来のカルバモイルリン酸シンターゼ (CPS) から、複雑な RvB1/B2 でここで発揮します。RvB1/B2 と CPS を説明36,37として精製しました。すべての蛋白質の複合体、個々 の浄化のプロトコルが必要です。プロトコルを調整します。リン酸緩衝生理食塩水 (PBS) または 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic 酸 (HEPES) などのアミン フリー バッファーを使用して、化学架橋用ください。可能であれば、浄化の間にバッファーを交換します。
注意: は、任意のメソッドまたは蛋白質の複合体のネイティブ アセンブリを邪魔する試薬には適用されません。
2. 質量分析を用いたタンパク質の同定
-
ゲル電気泳動
注: 別のゲル システム利用できるがあるし、すべての研究室では、独自のセットアップ。ジェル システムに従って条件を調整します。ケラチンは質量分析で最も一般的な汚染物質の中で、手袋とプロトコルの中で白衣を着用します。- 7.5 μ L 4 x サンプルバッファーと 3 μ L の 10 倍還元剤 (最終濃度 50 mM ジチオトレイトール (DTT)) を 20 μ L 蛋白質のサンプルに適用されます。10 μ M の CPS または 2.5 μ M RvB1/B2 を使用します。16,200 × g と 70 ° C で 10 分間の熱で 1 分のスピン ・ ダウン
注: とき、必要なボリュームと加熱温度を調整別のゲルを用いたします。 - 4-12% 勾配ゲル電気泳動に備えます。水で 20 倍、連続したバッファーを希釈し、電気泳動室を埋めます。2-(N morpholino) ethanesulfonic 酸 (MES) はタンパク質の分子量の小さい方 (2 200 kDa) と 3 のバッファー - 大きい蛋白質 (14 200 kDa) のため (N morpholino) propanesulfonic 酸 (モップ) のバッファーします。内側のチャンバーに 0.5 mL 抗酸化物質を追加します。
注: とき、連続したバッファーと製造元のプロトコルによるとゲルを準備別のゲルを用いたします。 - 最初の空洞に適したタンパク質マーカー (あらかじめ染色、無染色、異なる分子サイズ) を読み込み、残りの虫歯に蛋白質のサンプルをロードします。
- 別の蛋白質 35 分 (MES) または 200 V で 50 分 (モップ)。
注: は、電気泳動度と異なるゲル系の電圧を調整します。 - タンパク質のバンドを染色するには、ボックスを染色ゲルにゲルを転送し、染色液水性 Coomassie のゲルをカバーします。一晩と水平のゲル シェーカーで室温で孵化させなさい。
- 染色液を水に置き換えることにより脱色します。この手順を繰り返します数回 (約 3-5 回) ゲル背景がはっきり表示されるまで。
- 7.5 μ L 4 x サンプルバッファーと 3 μ L の 10 倍還元剤 (最終濃度 50 mM ジチオトレイトール (DTT)) を 20 μ L 蛋白質のサンプルに適用されます。10 μ M の CPS または 2.5 μ M RvB1/B2 を使用します。16,200 × g と 70 ° C で 10 分間の熱で 1 分のスピン ・ ダウン
-
ゲル内消化
注: 汚染を避けるためには、HPLC グレード溶剤消化力のプロトコルを通じて、すべての次の手順 (すなわち、質量分析計) を使用します。重炭酸アンモニウムと濾液水を使用する前にすべてのソリューションを準備します。- カット ブルー Coomassie によって視覚化される蛋白質バンドをメスを使用してゲルから染色します。慎重にカット小さな断片の約 1 mm × 1 mm. に蛋白質バンドは異なった蛋白質バンドの間に水のメスをすすいでください。水とアセトニ トリル (ACN) ゲルのバンドを洗います。
- DTT とジスルフィド結合を減らす、ヨードアセトアミド、システイン残基のアルキル基を導入、ダイジェスト蛋白質トリプシンと前述したように38;通常 1:20 1: 100 までの酵素: 蛋白比が使用されます。消化力のプロトコルの中に 100 mM 重炭酸アンモニウムを使用します。
- 2 つの手順でペプチドを抽出します。
- まず炭酸水素アンモニウムと ACN、ゲル部分をインキュベートし、ペプチドを含む上清を収集します。第二に、5% (v/v) ギ酸と ACN 入りのゲルを孵化させなさい。各ステップで 15 分間インキュベートします。両方の培養上清を組み合わせます。真空遠心分離の溶媒の蒸発によって抽出されたペプチドを乾燥します。
注: 乾燥ペプチドは-20 ° C で数ヶ月保存できます。
- まず炭酸水素アンモニウムと ACN、ゲル部分をインキュベートし、ペプチドを含む上清を収集します。第二に、5% (v/v) ギ酸と ACN 入りのゲルを孵化させなさい。各ステップで 15 分間インキュベートします。両方の培養上清を組み合わせます。真空遠心分離の溶媒の蒸発によって抽出されたペプチドを乾燥します。
-
液体クロマトグラフィー質量分析法
- 2 %acn/0.1% ギ酸で乾燥ペプチドを溶解します。溶解する超音波浴 2 〜 3 分とスピン中のペプチドを 30 分転送 16,200 × g で遠心分離機のサンプル オートサンプラー バイアル。
注: は、蛋白質量に合わせて音量を調整します。よく染色 Coomassie バンド 20 μ L を使用します。 - オートサンプラーを使用してナノ-クロマトグラフィー-タンデム質量システムにサンプルの 5 μ L を注入します。逆相 C18 前列 (C18、150 μ m の内径、2 cm、5 μ m の細孔径) を脱塩し、オンラインのペプチドを集中にペプチド混合物をロードします。
- 移動相 A と携帯として 80% (v/v) ACN/0.1% ギ酸相 B. 分離逆相 C18 分析カラム (C18、75 μ m 内径、50 cm、3 μ m の気孔のサイズ) にペプチドとして 0.1% (v/v) ギ酸を使用して 300 で 4 80 %b (0.1% ギ酸を含む) のグラデーションを使用して nL/分以上 65 分。
メモ: nanospray イオン源質量分析計に溶出したペプチドを転送する使用されます。 - (典型的な) MS の条件を使用して、: スプレー電圧 1.6 kV;キャピラリー温度 250 ° C;30 の正規化された衝突エネルギー。データ依存型モードで質量分析計を動作します。
- 質量分析器 (例えばorbitrap) の MS スペクトルを取得 (600 m/z 350−1) 70,000 と 3 × 106の自動利得制御ターゲットの解像度を持つ。1 x 10 の5の自動利得制御ターゲットで断片化が HCD の最も強烈なイオンの 20 を選択します。動的に除外する以前選択イオン単独でイオンとしてイオンをチャージが認識されていない状態で起訴 30 s. 除外。
注: 質量スペクトルの内部校正は、ロック オプションの質量39を用いて行われた.
- 2 %acn/0.1% ギ酸で乾燥ペプチドを溶解します。溶解する超音波浴 2 〜 3 分とスピン中のペプチドを 30 分転送 16,200 × g で遠心分離機のサンプル オートサンプラー バイアル。
-
データベース検索
注: ソフトウェアがある異なるデータベースの検索に使用できます。例えば、MaxQuant40は自由に利用可能です。- .Raw ファイルを pXtract 変換ツール (http://pfind.ict.ac.cn/downloads.html) を使用して .mgf ファイルに変換します。
- 典型的な検索パラメーターを使用してデータベース検索を実行: データベース、swissprot;ペプチド質量許容差、10 ppm;フラグメント質量公差、0.5 ダ;酵素、トリプシン;逃された胸の谷間のサイト、2;変数の変更、carbamidomethylation (システイン) と酸化 (メチオニン)。
注: は、実験の設定に従って検索パラメーターを変更します。 - データベース検索結果を検査します。タンパク質のスコア、ペプチドの数、ペプチドのスコア、および質量精度を評価します。シーケンス カバレッジは、識別される蛋白質の範囲を返します。
注意: すべての検索エンジンは、その個々 のスコアリング アルゴリズムです。タンデム質量スペクトルの質によって採点システム ペプチドを評価します。良いスペクトルの主信号機識別されたペプチドを確認する (完全な) イオン シリーズが表示されます。通常ペプチド スコアは確率に基づいて、すなわち、ペプチド スコアは識別されたペプチド配列が得られたスペクトルに一致する可能性はどの程度の測定。タンパク質のスコアは、通常はペプチドのスコアから派生し、蛋白質を識別された蛋白質のリストにランク付けするために使用します。
3. そのまま蛋白質複合体 (ネイティブ質量分析法) の質量
- エレクトロ スプレー イオン化用ゴールド コーティング エミッタの作製
- マイクロ ピペットの引き手を使用して25,41を前述のようにガラス管から nanoflow エミッタを準備します。0.78 mm の内径のホウケイ酸毛細血管を使用します。
注意: 針を引っ張るためのパラメーターを変更すると、先端の形状とサイズ、変更できるサンプルの調整。インナー径および壁厚さの異なる毛細血管があります。 - (、金やパラジウムなどの導電性材料とガラス管をコートします。ゴールドのプラズマを生成するスパッタ コーターの使用は共通です。質の良いコーティングを取得する製造元の指示に従ってください。
注: コーティングは一般的なキャピラリー電圧 (下記参照) を適用する場合は、安定したスプレーを取得する十分なはずです。
- マイクロ ピペットの引き手を使用して25,41を前述のようにガラス管から nanoflow エミッタを準備します。0.78 mm の内径のホウケイ酸毛細血管を使用します。
- ネイティブの質量分析のための試料作製
注: 塩、洗剤、またはグリセロールの大量エレクトロ スプレー イオン化と互換性がありません。したがって、浄化バッファーは、揮発性の水性バッファーによって交換されます。200 mM 酢酸アンモニウムはよく使用されます。バッファー交換のサイズ排除回転カラムまたは限外濾過装置を使用します。場合によっては、複雑な安定性や活動は、バッファー交換による影響可能性があります。質量スペクトルを慎重に評価し、複合体の活動を確認します。必要に応じて補因子や添加物を分析バッファーに追加します。- 高速バッファー交換、サイズ排除回転カラムを使用します。1,000 × g で遠心分離によってストレージ バッファーを削除し、4 ° C で 1 分間流れを破棄します。500 μ L 200 mM 酢酸アンモニウム、続いて遠心分離を追加することによって 3 回を洗浄します。20 μ L の列と 1,000 x g と 4 ° C で 4 分間遠心に蛋白質のサンプルをロードします。
注意: 蛋白質複合体の濃度は 1-10 μ M をする必要があります。プロシージャは、非揮発性成分がまだ分析を妨げるかどうかを繰り返します。 - 蛋白質のサンプルの集中し、同じバッファーを交換するには、実験は、遠心フィルターを使用します。解析蛋白質のサイズより小さい細孔サイズ 50% のろ過膜を使用します。
- ろ過装置に蛋白質のサンプルを転送し、200 mM 酢酸アンモニウムを追加します。15,000 × g でスピンダウンし、流れを破棄します。200 mM 酢酸アンモニウムを加えて遠心分離を繰り返します。この手順を複数回繰り返します。遠心速度について製造元の指示に従ってください。4 ° C で遠心分離を行う
注意:膜蛋白質は膜フィルター デバイスの沈殿する傾向があります。
- ろ過装置に蛋白質のサンプルを転送し、200 mM 酢酸アンモニウムを追加します。15,000 × g でスピンダウンし、流れを破棄します。200 mM 酢酸アンモニウムを加えて遠心分離を繰り返します。この手順を複数回繰り返します。遠心速度について製造元の指示に従ってください。4 ° C で遠心分離を行う
- 高速バッファー交換、サイズ排除回転カラムを使用します。1,000 × g で遠心分離によってストレージ バッファーを削除し、4 ° C で 1 分間流れを破棄します。500 μ L 200 mM 酢酸アンモニウム、続いて遠心分離を追加することによって 3 回を洗浄します。20 μ L の列と 1,000 x g と 4 ° C で 4 分間遠心に蛋白質のサンプルをロードします。
- そのまま蛋白質の複合体の質量分析
注意: ネイティブの質量分析法、例えば、四重極飛行時間 (ToF-Q) 質量分析計または orbitrap 質量分析計の変更できるさまざまなメーカーから質量分析計のさまざまな種類があります。下記プロトコルは、Q ToF の計測器で実行されました。- キャピラリー ホルダー内金で被覆したキャピラリーを置き、蛋白質のサンプルの 1-4 μ L で毛細血管を埋めます。ピンセットで針の先端を開きます。
- ナノ エレクトロ スプレー イオン源とキャピラリーのホルダーを接続し、キャピラリーの位置を調整します。0.5 〜 1.5 で毛細血管の先端を位置円すいオリフィスに cm。80-150 L/h nanoflow ガスを使用してスプレーを開始し、安定したスプレーを維持するためにガスの流れを調整します。
- Q ToF 楽器の曲ページでパラメーターを調整します。典型的な開始条件: 1.50 キャピラリー電圧 kV;コーン電圧 80 V;RF レンズ 1 エネルギー、80 V;衝突エネルギー、20 V;絞り 3、対 13.6 良い質量スペクトルを取得するこれらのパラメーターを変更します。「取得」ボタンをクリックしてして集録を開始し、、良い質量スペクトルを取得することが可能に多くのスキャンを結合します。
注: 少なくとも 100 スキャンを組み合わせることをお勧めします。
- そのまま蛋白質複合体のタンデム質量分析法
- (プロトコル セクション 3.3) 上記のように、質量スペクトルを取得します。蛋白質の複合体の前駆イオンを選択します。
- MS から取得ファイルに MS/MS モードに変更します。前駆体に MS/MS セレクションを設定質量。
- 低衝突エネルギーで集録を開始します。正しい前駆体の選択を確認するいくつかのスキャン (約 20 スキャン) を組み合わせて質量。タンパク質複合体が解離まで衝突エネルギーが増加します。取得するには、良い質量スペクトルは少なくとも 500 スキャンを組み合わせます。
注: ストリップの複合体ある低強度があります。可能な限りとして多くのスキャンを組み合わせると、解像度と信号対雑音比が増加します。
- ソリューションでそのまま蛋白質複合体の解離
注意: 蛋白質複合体の蛋白質の相互作用に追加の洞察力を得るためには、ソリューションに解離が行えます。- (セクション 3.2) 上記のように蛋白質のサンプルを準備します。蛋白質のサンプルに溶剤を追加またはサブの複合体にそのまま複合体を分離するために pH を変更します。典型的な溶媒はメタノール、イソプロパノール、ACN;酢酸やアンモニアの添加による酢酸アンモニウムの pH を変更します。(セクション 3.3、3.4) 上記のように、質量スペクトルを取得します。
- 溶剤や様々 なサブ錯体を生成する pH 範囲の量が異なります。通常、溶剤の量は、5-50% (最終濃度)、典型的な pH 範囲は 4-9。低濃度溶剤 (5%) や pH のわずかな変化、質量スペクトル (セクション 3.3 を参照) を取得します。
- 溶剤の量を増やすかソリューションでサブの錯体を生成するまでに段階の pH を変更します。サブ錯体を分析するマススペクトルを取得します。
- データをキャリブレーションします。
注: 得られたマススペクトルはセシウム (CsI) ヨウ化ソリューションを使用して外部キャリブレーションされています。- 100 mg の 1 mL の水で溶解します。
- CsI のマススペクトルを取得します。上記分析タンパク質複合体と同じm/z範囲に CsI のクラスターを得るためには、衝突エネルギーが異なります。
注意: CsI は、すぐにエミッタの先端を沈殿させるし、円錐を汚染します。多くとしてのみ取得十分なスペクトルを得るために必要なスキャンします。エミッタをしたらソースから削除します。 - 得られた質量スペクトルを使用して校正ファイルと CsI 参照ファイルを作る。
- キャリブレーションを取得した質量スペクトルに適用します。
注意: 質量スペクトルの校正データの永続的な変更があります。スペクトルの非校正が必要な場合は、ファイルのバックアップ コピーを作る。
- データ処理と解析
注意: ネイティブの質量スペクトル; のデータ解析のための多くの自由に利用できるソフトウェア ツールがあります。例えば、Massign42または UniDec43。以下のプロトコルでは、複雑なサンプルのため Massign の使用と同様、計測器ソフトウェアのヘルプとマニュアルのデータ分析について説明します。このソフトウェアは複雑な質量スペクトルの解析に最適です。オンライン プログラム (http://massign.chem.ox.ac.uk/) の使用のための指示に従います。- データ分析のためスペクトル平滑化パラメーターを調整することでスムーズに。パラメーターを調整することによって重心スペクトル。タンパク質複合体の 2 つの隣接したピークから複雑なマスを計算 ' 計測器ソフトウェア ツールを使用してピーク封筒。
注意: データの損失 (例えば、バインドされている配位子の損失) がありますも集中的な平滑化します。 - Massign42解析、質量スペクトルのピークのリストを生成します。そして滑らかなスペクトルのデータ ポイントをリニア化します。蛋白質の複合体を割り当て、複雑な組成を計算または複雑なピーク封筒をシミュレートするさまざまなソフトウェア ツールを使用します。
- データ分析のためスペクトル平滑化パラメーターを調整することでスムーズに。パラメーターを調整することによって重心スペクトル。タンパク質複合体の 2 つの隣接したピークから複雑なマスを計算 ' 計測器ソフトウェア ツールを使用してピーク封筒。
4. 化学架橋結合質量分析法と
注: 多数の架橋戦略利用できるあります。ここでは、ビス (sulfosuccinimidyl) suberate (BS3) 蛋白質蛋白質の相互作用の研究に用いられるアミン反応性架橋剤の使用について述べる。
- 1.43 mg BS3 25 mM ストック溶液を調製する 100 μ L の水に溶解します。
注: disuccinimidyl suberate (DSS) のような他の試薬が水溶性でないと通常ジメチルスルホキシド (DMSO) で溶解しています。いくつかゲルビーズの重い同位体標識フォームも承ります。重い安定同位体の取り込みは、データ評価時に役立ちます MS スペクトルで架橋・ ディ ・ ペプチドのピーク ペアを生成します。特異的標識ゲルビーズした、両方のバリアントの貯蔵液は準備し、1:1 の混合します。
注意: DMSO によるタンパク質複合体の解離を避けるためには、高濃度の原液は準備し、水や架橋反応が進行する前にバッファーで希釈します。 - タンパク質複合体に BS3 を追加します。0.5-5 に至る BS3 の様々 な量を使用して、最適な架橋剤濃度を識別するために mM。1 h、thermomixer で 25 ° C で反応混合物を孵化させなさい。4-12% 勾配ゲルを使用し、(例については図 2を参照) 架橋の結果を評価する SDS ページを実行します。
注: 非架橋コントロールで表示されない、高分子量蛋白質バンドに SDS ページ単一の亜単位はまだ目に見える (図 2)、得られるときに BS3 の最適濃度は達されます。 - 最適の BS3 濃度と架橋反応を繰り返します。1 h、thermomixer で 25 ° C で反応混合物を孵化させなさい。
注: いくつかの蛋白質の複合体は室温で安定しておりません。架橋反応は、氷; で実行することもしかし、反応時間は、調整する必要があります。 -
アミン バッファー (例えば50-100 mM Tris バッファーは、pH 7.5、最終濃度) を追加することによって架橋反応を抑制または削除任意の残留架橋試薬エタノール沈殿を行います。
- 200 μ L。 追加 600 μ L 冷たいエタノール、20 μ L 3 M 酢酸ナトリウム、pH 5.3 の最終的な量に達する反応混合物に水やバッファーを追加します。徹底的に混合し、-20 ° C 2 時間または一晩で孵化させなさい。
注: また、蛋白質が投げ落とされる 80 ° c で 30 分間または液体窒素で。 - 16,200 x g でスピン ・ ダウンと 4 ° C、30 分は、上澄みを慎重に取り外します。
- 1 mL 冷たい 80% (v/v) エタノールとペレットを洗浄します。16,200 x g でスピン ・ ダウンと 4 ° C、30 分は、上澄みを慎重に取り外します。真空遠心分離で沈殿を乾燥させます。
- 200 μ L。 追加 600 μ L 冷たいエタノール、20 μ L 3 M 酢酸ナトリウム、pH 5.3 の最終的な量に達する反応混合物に水やバッファーを追加します。徹底的に混合し、-20 ° C 2 時間または一晩で孵化させなさい。
- 架橋タンパク質の SDS-PAGE を実行します。ゲルのバンドを切り (セクション 2.1 と 2.2) 上記のようにタンパク質のゲルを消化します。
注: ソリューションの消化を実行もことができます、しかし、それは通常 (例えば、消化ペプチドのサイズ排除クロマトグラフィー) の追加の分離手順を必要とします。 -
液体クロマトグラフィー質量分析法は、(セクション 2.3) 上記のように実行します。架橋ペプチドは、通常豊富な低、架橋のサンプルの分析の深さを増加する質量分析に以下のバリエーションを適用します。
- 液体クロマトグラフィー分離中に長いグラデーションを使用 (例えば、65 分ではなく 90 分は上記参照)。
- HCD 断片化から二重荷電ペプチドを除外します。
注: 二重荷電ペプチドは、通常内相互リンクされたペプチド (「ループ ペプチド"または"タイプ 1"架橋) です。 - 差動を使用する場合は架橋試薬のラベル分析、すなわち中に「ピーク ピッキング」オプションを使用して、HCD の断片化、マススペクトル上に定義された質量差のピーク ペアの存在によってトリガーされます。
注:「ピーク ピッキング」オプションはすべて質量分析装置に関する使用できません。
- 使用には、ソフトウェア18架橋・ ディ ・ ペプチドの同定がポロンポロンと鳴る。最小化されたデータベースを使用して、識別のため。典型的な検索パラメーター: 計測スペクトル、HCD;酵素、トリプシン;最大。逃された胸の谷間サイト、3;変数の変更、酸化 (メチオニン) と carbamidomethylation (システイン);架橋剤、BS3;分ペプチドの長さ、4;最大。ペプチドの長さ、100;分ペプチド質量、400 のダ。最大。ペプチドの質量、10,000 Da;ルーズベルト 10 セント、1%。
注: ラベル付けされるゲルビーズと差分を使用して、リンカーによる質量の増加を構成する必要があります。クロスリンクの同定,例えば、xQuest17、MassMatrix19, または XlinkX15の他の一般的に使用されるソフトウェアもあります。 - 断片化スペクトルの質によってデータベースの検索結果を評価します。クロスリンクの許容可能なスペクトル合理的な信号対雑音比で両方のペプチド (少なくとも 4 隣接するイオン) のイオン系列が表示されます。
注: ラベル付けされる架橋試薬と差分を使用して、MS スペクトルのピークのペアは追加の品質管理として使用できます。 - 必要な場合は、ソフトウェア ツール (例えばXVis、XiNET) を使用して蛋白質相互作用ネットワークにおける架橋の結果を視覚化します。プロット バーまたは円形の使用は蛋白質の相互作用の可視化のプロットします。
注: 両方のソフトウェア ツールは、web サーバーで自由に利用できます。それぞれのウェブサイト (https://xvis.genzentrum.lmu.de/と http://crosslinkviewer.org/) の詳細な指示に従います。
結果
タンパク質と錯体形成の構造解析はその機能を理解するため基本的です。質量分析法は、サイズに関係なく興味のほぼすべての複合体に適用することができますや不均一性のサンプル構造の調査にかなり貢献しています。2 つのよ特徴付けられたタンパク質複合体; を使用してプロトコルを例示します。まず、 c. thermophilumと第二に、大腸菌から複雑なヘテロ本 CPS からヘテロ種類 RvB1/B2。
最初に、2 つの複合体のタンパク質成分を同定しました。このため、蛋白質は SDS ページ (図 2) によって分かれていたし、ゲルのバンドがゲルからカットされました。蛋白質のゲルの消化力の後に、ペプチド混合物は液体クロマトグラフィー質量分析法によって分析したペプチドとフラグメントの大衆データベース検索に服従し、。このワークフローに従う我々 は高い自信を持って、すなわち、2 つの複合体のすべての蛋白質の亜単位を識別合理的なペプチドのスコアを持つペプチドの高数 (テーブル 1 のすべてのタンパク質サブユニットの高いシーケンス カバレッジをもたらす観察されました。).
我々 は、ネイティブ質量分析法 (図 3 a) による複雑なそのまま RvB1/B2 を分析しました。質量スペクトルでは、2 つの種、約 8,000 m/zに 1 つずつ、約 11,000-12,000 m/zで別の種を明らかにしました。これらの種の計算の大衆は、6 量体 (RvB1)3(RvB2)3リング (約 310 kDa) と dodecameric ダブル リング (RvB1)6(RvB2)6 (約 620 kDa) に対応します。両方のピーク シリーズは 2 つの細胞集団が表示します。これらは複合体の RvB2 サブユニットを彼タグ付きとタグなしの混合物に属します。RvB1/B2 複雑な結晶構造は36を得られた以前と二重リング (図 3 b) の配置を示しています。ネイティブの質量スペクトルは、したがってそのまま種類の化学量論を確認し、さらに安定したサブ複合体を明らかにします。さらに、共存の個体が識別されます。
RvB1/B2 複合体蛋白質相互作用のサイトを識別するために化学的に BS3 架橋剤と精製の複合体を架橋しました。我々 は最初、最適濃度を決定するための架橋反応中に BS3 の量を滴定。BS3 にはアミンに固有、共有リンク リジン側鎖タンパク質の N 末端が。架橋反応は、SDS ページ (図 2 a) が続いた。非架橋複合体は、RvB1 と RvB2 の両方のサブユニットを示した。BS3 を反応混合物に追加すること、高分子量蛋白質バンドの蛋白質の共有結合連鎖が原因発生します。SDS のゲルは、非架橋タンパク質サブユニットが減少、BS3 の増加する量をもたらす架橋種の高い金額を示しています。ゲルからタンパク質のバンドを切断し、蛋白質の相互作用のサイトを識別するために上記の提供されるプロトコルを後にします。架橋・ ディ ・ ペプチドのスペクトルの例を示す (図 3).スペクトルは、この分子間相互作用を確認する両方のペプチドの y イオン シリーズを示しています。合計で、我々 はサブユニット RvB1 と RvB2 と RvB2 の 2 つのコピー間の 2 つのクロスリンクの間 4 つの架橋を含む 14 の蛋白質の相互作用を得られる (表 2)。架橋 BS3 からの結果は、異なるサブユニット間相互作用と同様に、分子内相互作用を示す相互作用ネットワーク (図 3 D) で視覚化されます。分子内架橋は、RvB1 と RvB2 の両方のサブユニットが、N 末端と C 末端ドメインは近くの方法で折ることをお勧めします。分子内相互作用を同じサブユニットの分子間相互作用からここでは区別できないことに注意してください。2 つのサブユニット間の分子間架橋結合はまた観察されました。、これらの架橋方法を検証する構造で RvB1 と RvB2 の 2 つの分子間架橋を視覚化することができます。他の分子間架橋結合は、結晶構造に含まれていない柔軟なループであります。また同じペプチド配列を含む RvB2 で 2 つのクロスリンクがわかりました。これらの架橋、明確に彼らは同じタンパク質 (図 3 D) の 2 つのコピーから取得する必要があります、分子間に分類されます。架橋実験複合施設内、既存の結晶構造 (図 3 b) も確認できる彼らの構造配置に洞察力を提供する蛋白質の亜単位の中でもタンパク質相互作用部位を明らかにします。
行った 2 番目のタンパク質複合体は、CPS をだった。ネイティブの質量スペクトル (図 4 a) では、6,000 と 12,000 m/zの間の 3 つのタンパク質複合体を明らかにしました。640 kDa の最も大きい複合体は、そのままヘテロ八量体に対応します。小さい体を表す 2 つのサブの複合体;小規模および大規模な CPS サブユニット (160 kDa) とヘテロ テトラマーの各サブユニット (320 kDa) の 2 つのコピーを含むダイマー。これらのサブの複合体蛋白質アセンブリに最初の洞察力を提供します。すなわち、大きく、小さい亜単位は、直接接触 (ヘテロ二量体によって明らかになった) と、四量体二量体 2 つの製品があります。そのまま cps の複雑な構造の配置についての詳細を得るためにヘテロ八量体とヘテロ四量体のタンデム質量分析 (MS/MS) を行った。どちらの場合では、前駆体を示唆しているが、小サブユニット (図 4 b) アセンブリの周辺部にあることから分離した小サブユニット。確かに、小サブユニットは利用可能な結晶構造 (図 4)44周辺です。
BS3 架橋剤を使用して化学架橋も行った。増加する量を使って、CPS のサブユニットの結合の連携が強化されました。タンパク質の消化と前述のペプチドの分析、大サブユニットと小サブユニットの 1 つ架橋内の多くのタンパク質相互作用 (テーブル 2) が得られました。さらに、複雑な RvB1/B2 と同様に、わかったな CPS の大サブユニットの 2 つのコピー間分子間架橋結合 2 つ。これらの架橋結合は、2 つの大サブユニットの C 末端側でお互いに直面して配置します。以前の研究では、我々 はほとんどによって検証大サブユニットの 2 つのコピーのインターフェイスから発する大サブユニットの 3 つの追加操作を識別構造質量と計算モデル35、結晶構造と得られたモデル (図 4および表 2)。これらの相互作用は、大きな 4 つのサブユニットから成る複雑な CPS コアの配置を許可します。しかし、小さく、大きい亜単位間の間サブユニット クロスリンクは認められなかった。利用可能な結晶構造 (図 4) を調べることによって周辺の小サブユニットと大サブユニットから成る複合体の 4 量体のコア間の相互作用の表面はとても小さく、これを説明するかもしれないことが明らかになったサブユニット間相互作用の不在。小サブユニットは容易に小さなバインディング インターフェイスのためほとんど無傷の複合体から解離を示したネイティブの質量分析により確認しています。それにもかかわらず、化学架橋とネイティブの質量分析法を組み合わせた cps の複雑な蛋白質の相互作用を許可の構造配置 (図 4) を推測します。
一緒に取られて、ネイティブの質量分析と化学架橋架橋ペプチドの質量分析による同定と相まっての組み合わせは両方の例の複合体の構造の配置の再構成をことができます。蛋白質の亜単位、例えば相互作用 RvB1 と RvB2 の間または CPS の 4 量体のコア内の配置を明らかにした化学架橋ネイティブ質量無傷の複合体の蛋白質の結果を提供し、流行の subcomplexes。CPS の 2 つのサブユニット間の分子間相互作用が認められなかった化学架橋による場合は、ネイティブの質量分析法は、各大サブユニットが 1 つ小サブユニット (図 4) と対話すること示唆しています。タンデム質量分析は複合体および両方のサブユニット間小さなインターフェイスで小サブユニットの周辺位置を提案しました。
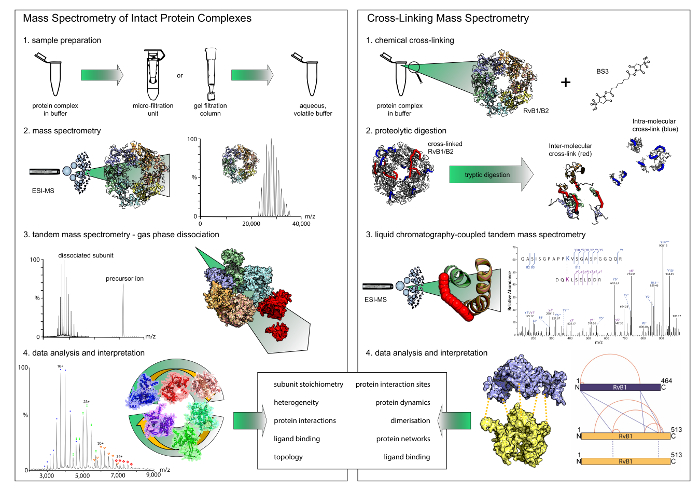
図 1: ネイティブの質量分析および架橋のワークフロー 。両者は相補的な結果を提供します。ネイティブの質量分析法では、結果と相互作用のモジュールを明らかにする、架橋複合体内タンパク質相互作用部位への洞察力を与えます。化学架橋のみとバイナリの相互作用が明らかにすることに注意してください。(A) ネイティブの質量分析の最初のステップはフィルター ユニットまたはゲルろ過カラムを使用して揮発性と水性バッファーにバッファー交換。そのまま蛋白質の複合体の質量は、その化学量論を明らかにします。タンデム質量分析法による実験では, 末梢のサブユニットの関連付けが解除されます。(B) 化学架橋用蛋白質の複合体の架橋試薬と培養です。架橋タンパク質は、ペプチド液体クロマトグラフィー質量分析法による分析はその後に、消化されます。この図の拡大版を表示するのにはここをクリックしてください。
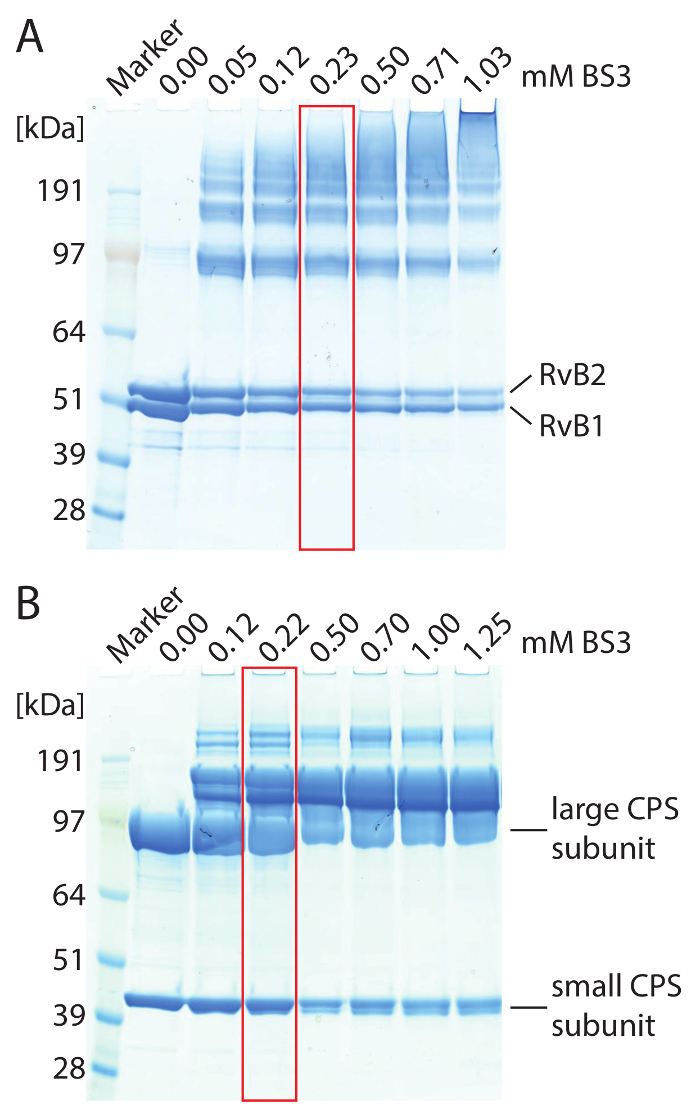
図 2: SDS-PAGE の架橋 RvB1/B2 (A) および CPS (B) 錯体。(A) 2.5 μ M ゲル レーンあたり RvB1/B2 を読み込みました。BS3 の濃度が変化します。非架橋 RvB1/B2 は約 50 kDa の 2 つの蛋白質の亜単位を示しています。BS3 の添加は、高分子量蛋白質バンドの蛋白質の亜単位の共有結合連鎖を発生します。BS3 の高濃度では、架橋の種の量をアップします。最適な架橋条件が強調表示されている (赤) です。ゲル レーン当たり 10 (B) μ M CPS を読み込みました。大きい (90 kDa) と小 (40 kDa) CPS の亜単位が得られます。BS3 の添加は、高分子量蛋白質バンドの蛋白質の亜単位の共有結合連鎖を発生します。最適な架橋条件が強調表示されている (赤) です。この図の拡大版を表示するのにはここをクリックしてください。
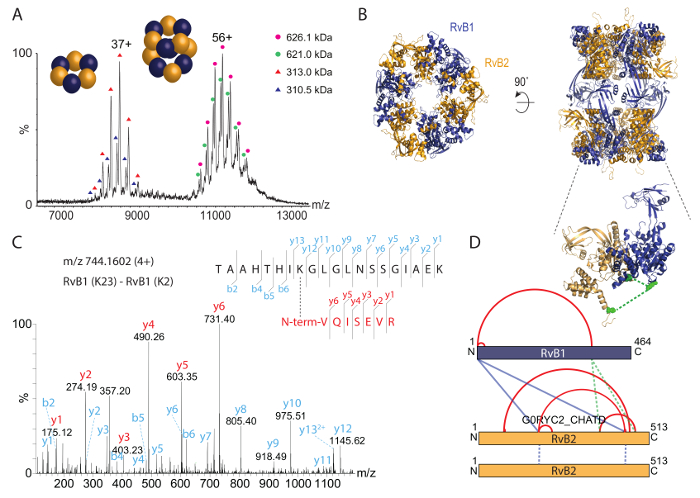
図 3: ネイティブの質量分析法と複雑な RvB1/B2 の化学架橋します。(A) ネイティブの質量スペクトルを明らかに 2 種 RvB1/B2;約 11,000 に 12,000 m/zおよび約 8,000 m/z6 量体リング (RvB1)3(RvB2)3でそのままの種類 (すなわち、(RvB1)6(RvB2)6)。両種は、彼タグ付きとタグなしの RvB2 から生じる 2 つの集団を示します。スペクトルは、35から変更されています。RvB1/B2 の (B) 結晶構造 (PDB ID 4WVY) を示します。RvB1 と RvB2 が交互のサブユニットは 2 つの 6 量体リングを形成します。(C) 架橋・ ディ ・ ペプチドの断片化スペクトル。RvB1 の N 末端だった架橋 RvB1 の K23 と y イオン シリーズ両方のペプチド (赤とシアン) 得られました。(D) 内と間 protein の相互作用 RvB1/B2 複合体で得られました。内相互リンクは赤で示されている inter-cross-links は青で示されます。挿入は、結晶構造 (緑、挿入) で視覚化することができる、RvB1、RvB2 の亜単位の分子間架橋結合 2 つを示しています。2 つの RvB2 コピーから発信された相互作用は、青の点線として表示されます。この図の拡大版を表示するのにはここをクリックしてください。
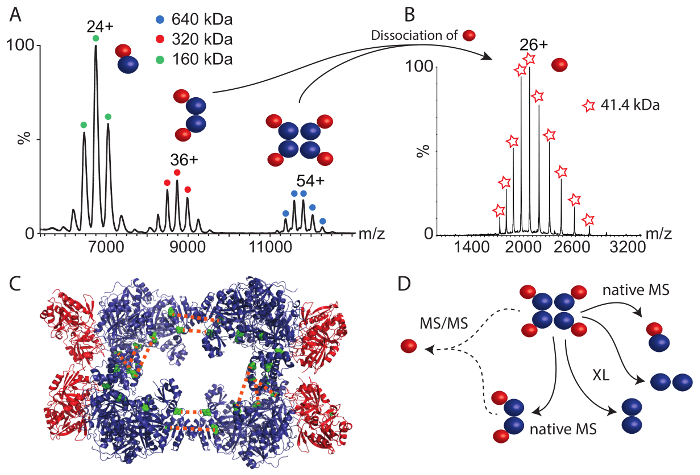
図 4: ネイティブの質量と CPS の化学架橋します。(A) CPS ショーのネイティブの質量スペクトル 3 複合体。ヘテロ ダイマー (160 kDa)、ヘテロ四量体 (320 kDa) と、ヘテロ八量体 (640 kDa)。スペクトルは、35から変更されています。(B) 4 量体および本 CPS 複雑な明らかに解離の CPS の小さい亜単位のタンデム質量分析法。Cps (C) 結晶構造 (PDB ID 1BXR) を示します。大きい亜単位は 4 量体のコアを形成、小サブユニットは、複合体の周辺に位置しています。大サブユニットの 2 つのコピー間の分子間架橋結合は (緑) で表示されます。(D) 大小の CPS サブユニットの相互作用。ネイティブの質量は subcomplexes を明らかにしたし、小サブユニットの周辺位置を示唆しています。化学架橋結合は、CPS の 4 量体の核内配置を示しています。この図の拡大版を表示するのにはここをクリックしてください。
表 1: データベース検索の結果。タンパク質は、液体クロマトグラフィー質量分析法とデータベース検索によって識別されました。タンパク質名受入数、説明とタンパク質質量を与えられています。タンパク質のスコア、タンパク質あたりスペクトルにみられる数観測ペプチド配列の番号が表示されます。マスコット ペプチド スコアの上位 5 つのペプチドは、各蛋白質の亜単位の一覧表示されます。このファイルをダウンロードするここをクリックしてください。
表 2: RvB1/B2 および CPS で観測されたクロスリンク。複合体の架橋残渣の亜単位が与えられます。クロスリンクの種類 (内分子間または) ペプチド配列の重複または以前研究35から明らかになった。このファイルをダウンロードするここをクリックしてください。
ディスカッション
プロトコルは、複数のサブユニット蛋白質の複合体の質量分析を用いた構造解析のため提供しています。プロトコルで説明されている 2 つの手法は、主に相補的な結果を提供し、タンパク質でアレンジを構造に洞察力を得るために適しています (-配位子) 錯体従来構造技術によって研究することは困難であります。ネイティブの質量分析法は、subcomplexes と安定した相互作用モジュールを分析することによってタンパク質の結果として蛋白質の相互作用についての洞察を提供します。架橋、その一方で、直接連絡先のサイト上の情報が得られます。によって使用される架橋剤、特定の柔軟性またはできます分析に含まれるべきであります。
指定されたプロトコルは、一般的に簡単に実行でき、ない時間がかかる。全体のプロトコルは、一週間内で実行することができます、タンパク質複合体量は解析に成功に必要なほぼすべてのタンパク質にも適用できます。サンプル準備は簡単、特に精製したタンパク質複合体を必要としません。ただし、1 つの一般的な落とし穴は、質量分析を用いたタンパク質の同定のための試料調製中のサンプルの汚染です。ほとんどの場合これらの汚染には、ほこり、皮膚や髪から由来するケラチンが含まれます。したがって、手袋、白衣を着てなど余分なケアは、水性バッファーをろ過、高純度溶剤を使用べきである質量分析を用いたタンパク質の同定のためのサンプル準備の間。シャペロンなど他の汚染タンパク質は、親和性の札を使用するとき通常例えば蛋白質の浄化の時に紹介しています。これらのケースで蛋白質の浄化によって改善されるべき、例えば洗濯ステップ数を増やします。いずれの場合も、サンプルの蛋白質汚染容易に識別データベース検索時に分類フィルターを省略することによって (すなわち、すべての種からの蛋白質に対する検索)。場合にのみ、いくつかのペプチドが観察される (すなわち、低たんぱくのカバレッジが得られて) にもかかわらず、十分なサンプルがあり、消化中に異なるプロテアーゼを使用する必要があります。一般的にトリプシンを生成ペプチド; の十分な数しかし、膜タンパク質や膜タンパク質のドメインなどのいくつかの場合、トリプシンの切断点の数を減少、他のターゲットに疎水性アミノ酸の酵素がより良い選択。
計測、面で特に変更された楽器はネイティブ質量気相への移動中に非共有結合相互作用を維持するために必要。Q ToF および orbitrap 楽器を含む、いくつかの楽器の種類が紹介されています。変更 Q ToF 質量分析計は数年からネイティブの質量分析用市販、後者は最近導入され、ほとんどの場合必要があります専門の変更45。しかし、高解像度機器のアプリケーションは複数の配位子とその定量化46,47のバインディングを勉強できる、有望な将来のアプリケーションです。
液体クロマトグラフィー質量分析法による架橋・ ディ ・ ペプチドを識別するためにいくつかの変更と標準的な手順を適用できます。ただし、データベース検索は、制限の要因として、専用のソフトウェアが大規模なデータベースを扱うことがめったとの複合体の蛋白質の亜単位を含んでいる減らされたデータベースが必要です。近年使用される質量分析法による劈開ゲルビーズ全体のセル lysates48,49で蛋白質の相互作用を対象とします。タンデム質量分析の実験でフラグメントの主化学ゲルビーズの使用線形ペプチド (、架橋剤によって変更)、それ以上の分裂によって識別することができますを生成し、線形ペプチドおよびこれのデータベース検索を低減時間と計算検索空間を検索します。しかし、これらの実験を実行するイオン トラップ質量分析計またはイオン トラップを用いたハイブリッド質量分析計です。一般に、偽陽性は重要な問題、架橋ペプチドの質量スペクトルがしばしば手動でデータ解析時間が非常に長く、そのフラグメント スペクトルの質によってに検証されます。さらに検証手順、したがって潜在的な将来のアプリケーションなしに適用できる堅牢なスコアリング システムの開発。データ分析が向上し、偽陽性の数を減らす 1 つの方法は、偽の発見率の計算とデータ セット50を架橋への応用の紹介をされました。
さらに質量分析技術一般に、ここで説明したテクニックで補完することができます (例えば、共有ラベル) 解析からの出力を増加させる。その他の変更とプロトコルの改善は簡単に実装できます。よう比較架橋34は蛋白質の集合の構造変化を解きます。ネイティブの質量分析法のさらなる発展は最近膜蛋白質51,52の解析、脂質28,52,53,54との相互作用を許可します。.ネイティブの質量分析のための高分解能質量分析計の新展開アプリケーションとリガンド結合、例えば脂質膜蛋白質への結合を拡張、今解析45,に含まれることができます。46. 計算モデル化のアプローチと組み合わせて、これらの技術は可変解像度55の構造モデルを提供できます。そのまま複雑なまたは 1 つのサブユニットの結晶構造がない場合、質量分析は蛋白質の相互作用および未知の複雑なトポロジに最初の洞察力を提供できます。不明な複合体の低解像度のモデルによって使用される技術と得られた結果、56,57,58得られます。結晶構造やホモロジー モデルがある場合、質量分析法の構造情報もネイティブに近いモデル59をもたらすことができます。
質量分析法による他の構造技術と比較して利点がある、濃度の低いサンプルが必要です、それは異種のサンプルを扱うことが、蛋白質の複合体のサイズが無制限に適用できます。さらに、質量分析は蛋白質の動的なシステムの調査をことができます。ソリューション内に存在するタンパク質やタンパク質複合体の異なった人口が通常一緒に分析され、そのためとは異なり特定の集団の選択を必要とする他の構造技術とすべての立体構造中が維持されます。分析とは、1 つの実験で課税。定量的な架橋方法は最近導入された34,60,61有望な将来のアプリケーションをさまざまな条件下での構造変化を記述します。
開示事項
著者が明らかに何もありません。
謝辞
役に立つ議論の仲間たちに感謝いたします。我々 はまた蛋白質の複合体を提供するためイルメ シュリヒティングとカール ・ ピーター Hopfner をありがちましょう。我々 は、教育と研究のための中央政府大臣からの資金調達を認める (BMBF、ZIK プログラム、03Z22HN22)、欧州地域開発基金 (EFRE、ZS/2016/04/78115) と c. s.、Wellcome の信頼 (109854/658 から資金に MLU ハレ ・ ヴィッテンベルクZ/15/Z) を兼
資料
| Name | Company | Catalog Number | Comments |
| Chemicals, Reagents, Consumables | |||
| 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid (HEPES) | Sigma Aldrich | H4034 | buffer |
| Acetic acid | Sigma Aldrich | 695092 | pH |
| Acetonitrile Optima LC/MS | Fisher Chemicals | A955-1 | solvent |
| Amicon Ultra centrifugal devices (different MWCO) | Millipore | i.e. UFC500396 | buffer exchange, concentration |
| Ammonium acetate solution | Sigma Aldrich | A2706 | native MS |
| Ammonium bicarbonate | Sigma Aldrich | 9830 | in-gel digestion |
| Ammonium solution | Sigma Aldrich | 9859 | pH |
| Bis(Sulfosuccinimidyl)suberate-d0 (BS3-d0) | Thermo Scientific | 21590 | cross-linker |
| Bis(Sulfosuccinimidyl)suberate-d4 (BS3-d4) | Thermo Scientific | 21595 | cross-linker |
| Caesium iodide | Sigma Aldrich | 203033 | calibration |
| Calcium chloride | Sigma Aldrich | C5670 | in-gel digestion |
| Capillaries (1.0 OD × 0.78 ID × 100 L mm) | Harvard Apparatus | 30-0038 | native MS |
| Disuccinimidyl suberate (DSS) | Thermo Scientific | 21655 | cross-linker |
| DL-Dithiothreitol | Sigma Aldrich | D5545 | in-gel digestion |
| DMSO (dimethyl sulfoxide) | Sigma Aldrich | D8418 | solvent |
| Ethanol | Fisher Chemicals | BP2818 | solvent |
| Formic acid Optima LC/MS | Fisher Chemicals | A117-50 | solvent supplement |
| Instant Blue Coomassie staining solution | expedeon | ISB1L | staining solution for gel electrophoresis |
| Invitrogen NuPAGE 4-12% Bis-Tris gels (1 mm, 10 well) | life technologies | NP0323BOX | gel electrophoresis |
| Iodoacetamide | Sigma Aldrich | I1149 | In-gel digestion |
| Isopropyl alcohol, HPLC grade | Fisher Chemicals | P750717 | solvent |
| Methanol | Fisher Chemicals | A456-212 | solvent |
| Micro BioSpin 6 columns | BioRad | 732-6222 | buffer exchange |
| NuPAGE Antioxidant | invitrogen | NP0005 | running buffer, gel electrophoresis |
| NuPAGE LDS Sample Buffer (4 ×) | invitrogen | NP0007 | sample loading buffer (non-reducing) for gel electrophoresis |
| NuPAGE MES SDS Running buffer | life technologies | NP0001 | gel electrophoresis |
| NuPAGE MOPS SDS Running buffer | life technologies | NP0001 | gel electrophoresis |
| NuPAGE Sample Reducing Agent (10 ×) | invitrogen | NP0004 | reducing Agent for gel electrophoresis |
| PBS - phosphate buffered saline | Sigma Aldrich | P4417 | buffer |
| SeeBlue Plus2 Prestained Standard Protein Marker | invitrogen | LC5925 | prestained Protein Marker for gel electrophoresis |
| Sodium acetate | Sigma Aldrich | S2889 | ethanol precipitation |
| Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 252859 | buffer |
| Trypsin | Sigma Aldrich | TRYPSEQ-RO (11418475001) | in-gel digestion |
| Vivaspin centrifugal devices (different MWCO) | Sartorius | i.e. VS0101 | buffer exchange, concentration |
| Water for HPLC | Sigma Aldrich | 270733-2.5L-M | solvent |
| Name | Company | Catalog Number | Comments |
| Instruments | |||
| Centrifuge Heraeus Fresco 21, bench-top centrifuge | Thermo Scientific | 75002425 | |
| Flaming / Brown Micropipette puller Model P-1000 | Sutter Instruments | P-1000 | |
| Gelelectrophoresis chamber Xcell SureLock MiniCell | Invitrogen | EI0001 | |
| Gold Coater Quorum Q150R S | Quorum Technologies Ltd. | Q150RS | |
| Horizontal gel shaker Rotamax 120 T | Heidolph Instruments | 544-41200-00 | |
| Q-TOF Ultima mass spectrometer, MS Vision high mass upgrade | Waters (Micromass) | - | |
| reversed-phase C18 analytical column Acclaim PepMap (C18, 75 mm I.D., 50 cm, 3 mm pore size) | Thermo Scientific | 164570 | |
| reversed-phase C18 pre-column (C18, 150 mm I.D., 2 cm, 5 mm pore size) | Thermo Scientific | 164213 | |
| scalpel | Fisher Scientifc | 10463989 | |
| SpeedVac SPD121P vacuum centrifuge | Thermo Scientific | SPD121DP-230 | |
| Thermomixer C | Eppendorf | 5382000015 | |
| Tweezers AA | Sigma Aldrich | Z680184-1EA | |
| Ultrasonic cleaner USC-TH, sonication bath | VWR | 142-0084 | |
| UltiMate Dionex 3000 nano-LC system, coupled to Q-Exactive plus hybrid mass spectrometer (nano-ESI source) | Thermo Scientific | 0726030+ | |
| Name | Company | Catalog Number | Comments |
| Software, Software Tools, Database search | |||
| Mascot | www.matrixscience.com | ||
| Massign | http://massign.chem.ox.ac.uk | ||
| MassLynx | Micromass | ||
| MassMatrix | Xu, H. J Proteome Res. 9 (2010) | ||
| MaxQuant | Cox, J. Nat. Biotechn. 26 (2008) | ||
| pLink | Yang, B. Nat Methods 9 (2012) | ||
| pXtract conversion tool | http://pfind.ict.ac.cn/downloads.html | ||
| UniDec | Marty, M.T. Anal. Chem. 87 (2015) | ||
| XCalibur | Thermo Scientific | ||
| XiNET | http://crosslinkviewer.org | ||
| XLinkX | Liu, F. Curr Op Struct Biol 35 (2015) | ||
| xQuest | Rinner, O. Nat Methods 5 (2008) | ||
| Xvis | https://xvis.genzentrum.lmu.de |
参考文献
- Campbell, I. D. Timeline: the march of structural biology. Nat Rev Mol Cell Biol. 3 (5), 377-381 (2002).
- Liko, I., Allison, T. M., Hopper, J. T., Robinson, C. V. Mass spectrometry guided structural biology. Curr Opin Struct Biol. 40, 136-144 (2016).
- Robinson, C. V. From molecular chaperones to membrane motors: through the lens of a mass spectrometrist. Biochem Soc Trans. 45 (1), 251-260 (2017).
- Lossl, P., van de Waterbeemd, M., Heck, A. J. The diverse and expanding role of mass spectrometry in structural and molecular biology. EMBO J. 35 (24), 2634-2657 (2016).
- Wang, L., Chance, M. R. Protein Footprinting Comes of Age: Mass Spectrometry for Biophysical Structure Assessment. Mol Cell Proteomics. 16 (5), 706-716 (2017).
- Steen, H., Mann, M. The ABC's (and XYZ's) of peptide sequencing. Nat Rev Mol Cell Biol. 5 (9), 699-711 (2004).
- Olsen, J. V., Mann, M. Status of large-scale analysis of post-translational modifications by mass spectrometry. Mol Cell Proteomics. 12 (12), 3444-3452 (2013).
- Schmidt, C., Robinson, C. V. Dynamic protein ligand interactions--insights from MS. FEBS J. 281 (8), 1950-1964 (2014).
- Marcsisin, S. R., Engen, J. R. Hydrogen exchange mass spectrometry: what is it and what can it tell us?. Anal Bioanal Chem. 397 (3), 967-972 (2010).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Leitner, A., Lindner, W. Current chemical tagging strategies for proteome analysis by mass spectrometry. J Chromatogr B Analyt Technol Biomed Life Sci. 813 (1-2), 1-26 (2004).
- Leitner, A., Lindner, W. Chemistry meets proteomics: the use of chemical tagging reactions for MS-based proteomics. Proteomics. 6 (20), 5418-5434 (2006).
- Kiselar, J. G., Chance, M. R. Future directions of structural mass spectrometry using hydroxyl radical footprinting. J Mass Spectrom. 45 (12), 1373-1382 (2010).
- Xu, G., Chance, M. R. Hydroxyl radical-mediated modification of proteins as probes for structural proteomics. Chem Rev. 107 (8), 3514-3543 (2007).
- Liu, F., Heck, A. J. Interrogating the architecture of protein assemblies and protein interaction networks by cross-linking mass spectrometry. Curr Opin Struct Biol. 35, 100-108 (2015).
- Rappsilber, J. The beginning of a beautiful friendship: cross-linking/mass spectrometry and modelling of proteins and multi-protein complexes. J Struct Biol. 173 (3), 530-540 (2011).
- Rinner, O., et al. Identification of cross-linked peptides from large sequence databases. Nat Methods. 5 (4), 315-318 (2008).
- Yang, B., et al. Identification of cross-linked peptides from complex samples. Nat Methods. 9 (9), 904-906 (2012).
- Xu, H., Hsu, P. H., Zhang, L., Tsai, M. D., Freitas, M. A. Database search algorithm for identification of intact cross-links in proteins and peptides using tandem mass spectrometry. J Proteome Res. 9 (7), 3384-3393 (2010).
- Leitner, A., Faini, M., Stengel, F., Aebersold, R. Crosslinking and Mass Spectrometry: An Integrated Technology to Understand the Structure and Function of Molecular Machines. Trends Biochem Sci. 41 (1), 20-32 (2016).
- Heck, A. J. Native mass spectrometry: a bridge between interactomics and structural biology. Nat Methods. 5 (11), 927-933 (2008).
- Sharon, M., Robinson, C. V. The role of mass spectrometry in structure elucidation of dynamic protein complexes. Annu Rev Biochem. 76, 167-193 (2007).
- Goth, M., Pagel, K. Ion mobility-mass spectrometry as a tool to investigate protein-ligand interactions. Anal Bioanal Chem. , (2017).
- Uetrecht, C., Rose, R. J., van Duijn, E., Lorenzen, K., Heck, A. J. Ion mobility mass spectrometry of proteins and protein assemblies. Chem Soc Rev. 39 (5), 1633-1655 (2010).
- Hernandez, H., Robinson, C. V. Determining the stoichiometry and interactions of macromolecular assemblies from mass spectrometry. Nat Protoc. 2 (3), 715-726 (2007).
- Sobott, F., Hernandez, H., McCammon, M. G., Tito, M. A., Robinson, C. V. A tandem mass spectrometer for improved transmission and analysis of large macromolecular assemblies. Anal Chem. 74 (6), 1402-1407 (2002).
- Rostom, A. A., et al. Detection and selective dissociation of intact ribosomes in a mass spectrometer. Proc Natl Acad Sci U S A. 97 (10), 5185-5190 (2000).
- Zhou, M., et al. Mass spectrometry of intact V-type ATPases reveals bound lipids and the effects of nucleotide binding. Science. 334 (6054), 380-385 (2011).
- Uetrecht, C., et al. High-resolution mass spectrometry of viral assemblies: molecular composition and stability of dimorphic hepatitis B virus capsids. Proc Natl Acad Sci U S A. 105 (27), 9216-9220 (2008).
- Morgner, N., et al. Hsp70 forms antiparallel dimers stabilized by post-translational modifications to position clients for transfer to Hsp90. Cell Rep. 11 (5), 759-769 (2015).
- Schmidt, C., Beilsten-Edmands, V., Robinson, C. V. The joining of the Hsp90 and Hsp70 chaperone cycles yields transient interactions and stable intermediates: insights from mass spectrometry. Oncotarget. 6 (21), 18276-18281 (2015).
- Schmidt, C., Beilsten-Edmands, V., Mohammed, S., Robinson, C. V. Acetylation and phosphorylation control both local and global stability of the chloroplast F1 ATP synthase. Sci Rep. 7, 44068 (2017).
- Schmidt, C., et al. Comparative cross-linking and mass spectrometry of an intact F-type ATPase suggest a role for phosphorylation. Nat Commun. 4, 1985 (2013).
- Schmidt, C., Robinson, C. V. A comparative cross-linking strategy to probe conformational changes in protein complexes. Nat Protoc. 9 (9), 2224-2236 (2014).
- Schmidt, C., et al. Surface Accessibility and Dynamics of Macromolecular Assemblies Probed by Covalent Labeling Mass Spectrometry and Integrative Modeling. Anal Chem. 89 (3), 1459-1468 (2017).
- Lakomek, K., Stoehr, G., Tosi, A., Schmailzl, M., Hopfner, K. P. Structural basis for dodecameric assembly states and conformational plasticity of the full-length AAA+ ATPases Rvb1 · Rvb2. Structure. 23 (3), 483-495 (2015).
- Mareya, S. M., Raushel, F. M. A molecular wedge for triggering the amidotransferase activity of carbamoyl phosphate synthetase. Biochemistry. 33 (10), 2945-2950 (1994).
- Shevchenko, A., Wilm, M., Vorm, O., Mann, M. Mass spectrometric sequencing of proteins silver-stained polyacrylamide gels. Anal Chem. 68 (5), 850-858 (1996).
- Olsen, J. V., et al. Parts per million mass accuracy on an Orbitrap mass spectrometer via lock mass injection into a C-trap. Mol Cell Proteomics. 4 (12), 2010-2021 (2005).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol. 26 (12), 1367-1372 (2008).
- Kirshenbaum, N., Michaelevski, I., Sharon, M. Analyzing large protein complexes by structural mass spectrometry. J Vis Exp. (40), (2010).
- Morgner, N., Robinson, C. V. Massign: an assignment strategy for maximizing information from the mass spectra of heterogeneous protein assemblies. Anal Chem. 84 (6), 2939-2948 (2012).
- Marty, M. T., et al. Bayesian deconvolution of mass and ion mobility spectra: from binary interactions to polydisperse ensembles. Anal Chem. 87 (8), 4370-4376 (2015).
- Thoden, J. B., Wesenberg, G., Raushel, F. M., Holden, H. M. Carbamoyl phosphate synthetase: closure of the B-domain as a result of nucleotide binding. Biochemistry. 38 (8), 2347-2357 (1999).
- Rose, R. J., Damoc, E., Denisov, E., Makarov, A., Heck, A. J. High-sensitivity Orbitrap mass analysis of intact macromolecular assemblies. Nat Methods. 9 (11), 1084-1086 (2012).
- Gault, J., et al. High-resolution mass spectrometry of small molecules bound to membrane proteins. Nat Methods. 13 (4), 333-336 (2016).
- Mehmood, S., et al. Mass spectrometry captures off-target drug binding and provides mechanistic insights into the human metalloprotease ZMPSTE24. Nat Chem. 8 (12), 1152-1158 (2016).
- Kaake, R. M., et al. A new in vivo cross-linking mass spectrometry platform to define protein-protein interactions in living cells. Mol Cell Proteomics. 13 (12), 3533-3543 (2014).
- Liu, F., Rijkers, D. T., Post, H., Heck, A. J. Proteome-wide profiling of protein assemblies by cross-linking mass spectrometry. Nat Methods. 12 (12), 1179-1184 (2015).
- Walzthoeni, T., et al. False discovery rate estimation for cross-linked peptides identified by mass spectrometry. Nat Methods. 9 (9), 901-903 (2012).
- Barrera, N. P., Di Bartolo, N., Booth, P. J., Robinson, C. V. Micelles protect membrane complexes from solution to vacuum. Science. 321 (5886), 243-246 (2008).
- Barrera, N. P., et al. Mass spectrometry of membrane transporters reveals subunit stoichiometry and interactions. Nat Methods. 6 (8), 585-587 (2009).
- Laganowsky, A., et al. Membrane proteins bind lipids selectively to modulate their structure and function. Nature. 510 (7503), 172-175 (2014).
- Marcoux, J., et al. Mass spectrometry reveals synergistic effects of nucleotides, lipids, and drugs binding to a multidrug resistance efflux pump. Proc Natl Acad Sci U S A. 110 (24), 9704-9709 (2013).
- Politis, A., Schmidt, C. Structural characterisation of medically relevant protein assemblies by integrating mass spectrometry with computational modelling. J Proteomics. , (2017).
- Hall, Z., Politis, A., Robinson, C. V. Structural modeling of heteromeric protein complexes from disassembly pathways and ion mobility-mass spectrometry. Structure. 20 (9), 1596-1609 (2012).
- Pukala, T. L., et al. Subunit architecture of multiprotein assemblies determined using restraints from gas-phase measurements. Structure. 17 (9), 1235-1243 (2009).
- Politis, A., et al. Topological models of heteromeric protein assemblies from mass spectrometry: application to the yeast eIF3:eIF5 complex. Chem Biol. 22 (1), 117-128 (2015).
- Politis, A., et al. A mass spectrometry-based hybrid method for structural modeling of protein complexes. Nat Methods. 11 (4), 403-406 (2014).
- Fischer, L., Chen, Z. A., Rappsilber, J. Quantitative cross-linking/mass spectrometry using isotope-labelled cross-linkers. J Proteomics. 88, 120-128 (2013).
- Yu, C., et al. Developing a Multiplexed Quantitative Cross-Linking Mass Spectrometry Platform for Comparative Structural Analysis of Protein Complexes. Anal Chem. 88 (20), 10301-10308 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved