Method Article
Kimyasal Cross-linking ve çoklu alt birim Protein derlemeler mimarlık okumak için olduğu gibi Protein kompleksleri kütle spektrometresi birleştirme
Bu Makalede
Özet
Protein kompleksleri onların fonksiyonu için temel bir mimaridir. Çeşitli kitle spektrometrik teknikleri birleştirerek kendi derleme çalışması için güçlü olduğunu kanıtladı. Biz kimyasal cross-linking ve yerel kütle spektrometresi protokolleri sağlamak ve nasıl çoklu alt birim protein derlemeler mimarisini aydınlatmak için tamamlayıcı bu teknikleri yardımcı göstermek.
Özet
Proteinlerin çeşitli hücresel işlevlerini gerçekleştirecek formu etkin ve dinamik derlemeler için onların ligandlar etkileşim. Bu etkileşimler elucidating bu nedenle hücresel süreçleri anlamak için esastır. Ancak, birçok protein kompleksleri dinamik derlemeler ve konvansiyonel yapısal teknikleri tarafından erişilebilir değildir. Kütle spektrometresi bu derlemeler yapısal araştırmaya katkıda bulunur ve özellikle çeşitli kitle spektrometrik tekniklerin birleşimi kendi Yapısal düzenleme değerli içgörüler sunar.
Bu makalede, biz uygulama ve iki tamamlayıcı kitle spektrometrik tekniklerin birleşimi tanımlamak, yani kimyasal cross-linking kütle spektrometresi ve yerel kütle spektrometresi ile birleştiğinde. Kimyasal cross-linking amino asitlerin birbirine yakın kovalent bağ kimyasal reaktifler kullanarak içerir. Sonra sindirim proteaz ile çapraz bağlı di-peptidler kütle spektrometresi tarafından tanımlanır ve protein etkileşimleri ele geçen sitelerdir. Yerel kütle spektrometresi Öte yandan bir Kütle Spektrometre gaz aşamasında olduğu gibi protein derlemeler analizidir. Protein stoichiometries yanı sıra protein ve ligand etkileşimleri ortaya koymaktadır. Her iki tekniğin de bu nedenle protein-ligand derlemeler ve onların birlikte önceki çalışmalarda güçlü olduğunu kanıtladı yapısını tamamlayıcı bilgi sunar.
Giriş
Protein derlemeler yapısal incelenmesi özellikle hücresel süreçleri anlamak için önemli hale gelmiştir. Sonuç olarak, birçok teknikleri geliştirilmiş ve Yapısal Biyoloji1' geliştirilmiş. Ancak, bu teknikler bazen onların uygulama boyutu, esneklik veya protein kompleksleri soruşturma altında heterojen nedeniyle sınırlıdır. Kütle spektrometresi bu zorlukları ile başa çıkabilirim ve bu nedenle, Yapısal Biyoloji2,3,4,5güçlü bir araç olarak ortaya çıktı. Kütle spektrometresi, en büyük avantajı ise belirsizliğe yer bırakmadan proteinler bile karmaşık ve heterojen karışımları6' tanımlama yeteneği. Bu amaçla, proteinler genellikle endoproteinases ile sindirmek ve elde edilen peptid karışımı sıvı Kromatografi tarafından ayrılmış ve doğrudan Kütle Spektrometre eluted. Peptid kitleler daha sonra belirlenir ve öncü iyonlar daha fazla peptidler parçalanma için seçilir. Proteinler sonra arama peptid ve bilinen bir veritabanında karşılık gelen parçası kitleler tarafından tanımlanır. Bu yordam sadece peptidler/protein değil, aynı zamanda bir kitle değişim habercisi peptidler ve değişiklik7taşıyan parçası iyonları neden onların translasyonel modifikasyonlar kimliği verir. Bazı yapısal kütle spektrometresi birçok teknikler üzerinde bu ilke4,8temel alır. Örneğin, hidrojen döteryum Satım9,10gibi teknikleri etiketleme, kimyasal etiketleme stratejileri11,12veya radikal hidroksil ayak baskı13,14 , yüzey erişilebilirlik belirli koşullar altında proteinlerin içine anlayışlar verebilir.
(Amino asitlerin birbirine yakın onların fonksiyonel gruplar aracılığıyla kovalent bağ içeren cross-linking kimyasal) başka bir tekniktir. Bunun için istihdam15,16kimyasal reaktifler, UV alınabilen reaktifler veya amino asitler vardır. İlişkilendirerek sonra proteinler genellikle proteaz ile hidrolize ve çapraz bağlı di-peptidler tarafından sıvı Kromatografi birleştiğinde kütle spektrometresi analiz edilir. Cross-linking ürünler arama, veritabanı tarafından kimlik ancak, çeşitli proteinler ve birbirinden farklı bölgeler17,18,19peptid dizileri bir arada özel yazılım kullanımı gerektirir. Kimyasal cross-linkers kullanımı ilgi hemen hemen her protein kompleksi için istihdam ve does değil istemek sadece protein konak hücreleri ilgi ifade edilirken elde edilebilir ve UV alınabilen amino asitler, birleşme avantajı vardır. Bu nedenle, cross-linking çok yönlü bir araçtır ve başarıyla birçok yapısal bile büyük protein derlemeler20çalışmalarda istihdam edildi.
Kütle spektrometresi (bazen olarak adlandırılır 'Yerel' kütle spektrometresi) olduğu gibi protein kompleksleri, diğer taraftan, peptidler sağlam protein ve protein kompleksleri hidroliz olmadan analizini içerir. Kompozisyon, heterojenlik, stoichiometry, topoloji ve alt birim etkileşimleri protein kompleksleri21,22ortaya koymaktadır. İyon hareketlilik ile birlikte, yerel kütle spektrometresi daha da onların uyum23,24tayini sağlar. Bu geleneksel yapı teknikleri tarafından değerlendirmek zordur protein kompleksleri yapısal incelenmesi için güçlü bir araç sağlar. Ancak, yerel kütle spektrometresi kovalent olmayan protein etkileşimleri electrospray iyonlaşma sırasında korumak analiz arabellekleri gerektirir. Bu genellikle sulu, uçucu arabellekleri amonyum asetat25gibi kullanılarak elde edilir. Buna ek olarak, araç değişiklikler ayrılma Kütle Spektrometre26gaz fazına iletim sırasında önlemek gereklidir. Bu şekilde uygulanan, birçok (büyük) protein kompleksleri analiz edildi. Sağlam ribozomlara27, ATP synthases28veya virüs29çalışmaları etkileyici örnekleridir.
Yerel kütle spektrometresi ve cross-linking önceki çalışmalarda özellikle başarılı oldu. Kimyasal cross-linking proteinler arasında düzenlemelerin ortaya iken Örneğin, refakatçi kompleksleri, beklenmedik bir Hsp70 dimer dahil olmak üzere stoichiometries olduğu gibi protein kompleksleri kütle spektrometresi deneylerden elde edilebilir derlemeler30,31. Farklı bir çalışmada, karmaşık bir bozulmamış ATP sentaz üzerinde translasyonel modifikasyonlar etkileri incelenmiştir. Yerel kütle spektrometresi protein kompleksi istikrar anlayışlar fosforilasyon veya asetilasyon32,33içinde sağlanan. Karşılaştırmalı cross-linking strateji34 sonra farklı koşullar altında karmaşık proteinin konformasyon değişiklikler ortaya çıkar.
Burada, protein tanımlama için kütle spektrometresi, (kimyasal) cross-linking ve veri analizi ve yorumu (şekil 1) dahil olmak üzere yerel kütle spektrometresi tarafından protokolleri sağlamak. Tamamlayıcı sonuçları iki iyi karakterize protein kompleksleri yardımıyla bu yöntemlerle elde edilen gösterdiği35birleşimidir. Bizim iletişim kuralı belirli saflık ve konsantrasyon saf herhangi bir protein bütünleştirilmiş uygulanabilir. Yaklaşım veri, Yani, veritabanının boyutunu cross-linking veri analizi tarafından sınırlı bazı durumlarda, gerekli arama uzay ve zaman belirler istihdam edilmektedir. Buna ek olarak, belirlenen çapraz bağlantıları el ile doğrulama kez gereklidir ve çıkış daha da azaltır. Yerel kütle spektrometresi çoğunlukla örnek kalite, örneğin, arabellekleri tarafından sınırlıdır ve adducts onları sulu ve uçucu arabellekleri tarafından Döviz için arıtma ve olasılığını sırasında kullanılan. Ancak, yapısal analiz için genellikle saf protein kompleksleri bizim protokol ile başarılı analiz için gerekli kalite var.
Protokol
1. arıtma Protein kompleksleri
- Protein kompleksi göre en iyi duruma getirilmiş standart protokoller hazırlamak.
Not: Protokol burada RvB1/B2 ile karmaşık Chaetomium thermophilum ve Escherichia coliüzerinden carbamoyl fosfat synthase (CPS) gösterilmiştir. RvB1/B2 ve CPS açıklanan36,37saf. Her protein kompleksi bireysel arıtma iletişim kuralı gerektirir. Protokol buna göre ayarlayın. Amin-Alerjik arabellekleri fosfat tamponlu tuz çözeltisi (PBS) veya 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic asit (HEPES) gibi kimyasal cross-linking için kullanılmalıdır. Arabellek arıtma sırasında mümkünse değiştirin.
Dikkat: herhangi bir yöntemi veya protein kompleksi yerel Meclisi rahatsız reaktifler geçerli değildir.
2. kütle spektrometresi tabanlı Protein tanımlama
-
Jel Elektroforez
Not: Orada farklı jel sistemleri mevcuttur ve her laboratuar kendi kurulum kullanır. Koşullar jel sistemine göre ayarlayın. Olarak keratins en yaygın kirleticiler arasında kitle spektrometrik analizi eldiven ve önlük protokol boyunca giymek.- 7.5 µL 4 x örnek arabellek ve 3 µL 10 x beyaz indirgeyici (son konsantrasyonu 50 mM dithiothreitol (DTT)) 20 µL protein örnek için geçerlidir. 10 µM CPS veya 2.5 µM RvB1/B2 kullanın. Spin aşağı 16,200 × g ve ısı için 70 ° C'de 10 dk 1 dk
Not: Otelde farklı Elektroforez sistemi kullanarak ayarlamak gerekli birimler ve Isıtma sıcaklık. - 4-%12 degrade Jel Elektroforez için hazırlayın. 20 kez su ile çalışan arabellek sulandırmak ve Elektroforez odası doldurun. Kullanım 2-(N-morpholino) ethanesulfonic asit (MES) arabellek için daha küçük molekül ağırlıklı (2-200 kDa) ve 3 proteinler-(N-morpholino) propanesulfonic asit (paspas) arabellek daha büyük proteinler (14-200 kDa). 0.5 mL antioksidan iç odasına ekleyin.
Not: Otelde farklı Elektroforez sistemi kullanarak hazırlamak çalışan tampon ve jel üreticisinin protokolleri göre. - İlk boşluğunda uygun protein işaretleyici (önceden lekeli, günahı, farklı moleküler boyutları) yükleyin ve protein örnekleri kalan boşlukları yüklemek.
- Ayrı proteinler için 35 dk (MES) veya 200 V 50 dk (paspas).
Not: Elektroforez kez ve gerilim farklı jel sistemleri için ayarlayın. - Protein bantları leke için jel kutusu boyama bir jel aktarmak ve jel, su bazlı Coomassie çözüm boyama bir kapak. Bir gecede ve bir yatay jel shaker üzerinde oda sıcaklığında kuluçkaya.
- Jel su ile boyama çözüm değiştirerek destain. Jel arka plan açık görüntüleninceye kadar bu adımı (yaklaşık 3 - 5 kez) birkaç kez yineleyin.
- 7.5 µL 4 x örnek arabellek ve 3 µL 10 x beyaz indirgeyici (son konsantrasyonu 50 mM dithiothreitol (DTT)) 20 µL protein örnek için geçerlidir. 10 µM CPS veya 2.5 µM RvB1/B2 kullanın. Spin aşağı 16,200 × g ve ısı için 70 ° C'de 10 dk 1 dk
-
Jel sindirim
Not: kirlenmesini önlemek için HPLC sınıf çözücüler sindirim Protokolü boyunca ve tüm aşağıdaki adımları (Yani, kitle spektrometrik analizi) için kullanın. Kullanımı ve filtrate su ve amonyum bikarbonat önce tüm çözümleri hazırlayın.- Mavi Coomassie tarafından görüntülenmiştir protein bantları bir neşter kullanarak jel leke kes. Dikkatle kesin küçük parçalara, yaklaşık 1 mm × 1 mm. protein bantları neşter farklı protein bantları arasında su ile durulayın. Jel gruplar Asetonitril (ACN) ve su ile yıkayın.
- Disülfür bağları ile DTT azaltmak, sistein kalıntıları iodoacetamide ile alkylate ve Özet proteinler tripsin ile daha önce38açıklandığı gibi; genellikle bir enzim: protein oranı 1:20 1: 100 kadar kullanılır. 100 mM amonyum bikarbonat sırasında sindirim protokolünü kullanır.
- Peptidler iki adımda ayıklayın.
- İlk amonyum bikarbonat ve ACN jel taşlarla kuluçkaya ve peptid içeren süpernatant toplamak. İkinci olarak, % 5 (v/v) formik asit ve ACN jel taşlarla kuluçkaya. Her adımda 15dk için kuluçkaya. Her iki supernatants birleştirmek. Ayıklanan peptidler solventler vakum santrifüj içinde buharlaşan tarafından kuru.
Not: Kurutulmuş peptidler-20 ° C'de birkaç ay depolanabilir.
- İlk amonyum bikarbonat ve ACN jel taşlarla kuluçkaya ve peptid içeren süpernatant toplamak. İkinci olarak, % 5 (v/v) formik asit ve ACN jel taşlarla kuluçkaya. Her adımda 15dk için kuluçkaya. Her iki supernatants birleştirmek. Ayıklanan peptidler solventler vakum santrifüj içinde buharlaşan tarafından kuru.
-
Sıvı Kromatografi birleştiğinde kütle spektrometresi
- % 2 ACN/0.1% formik asit kurutulmuş peptidler geçiyoruz. Peptidler bir sonication banyoda 2-3 dk ve spin için aşağı 16,200 x g, santrifüj 30 dk. Transfer içinde örnekleri otomatik örnekleyici tüpleri geçiyoruz.
Not: protein miktarı göre ses düzeyini ayarlayın. İyi lekeli Coomassie bantları için 20 µL kullanın. - Otomatik Örnekleyici kullanarak nano-LC-MS/MS sisteme örnek 5 µL enjekte. Peptid karışımı bir ters faz C18 ön sütuna (C18, 150 µm kimlik, 2 cm, 5 mikron gözenek boyutu) desalt ve peptidler online konsantre yükleyin.
- Mobil faz A ve % 80 ACN/0.1% (v/v) formik asit mobil olarak B. ayrı bir ters faz C18 analitik sütunda (C18, 75 µm kimlik, 50 cm, 3 µm gözenek boyutu) peptidler faz olarak % 0,1 (v/v) formik asit kullanmak 4-%80 B (%0,1 formik asit içeren) bir degrade 300 kullanarak nL / dk 65 dk.
Not: Bir nanospray İyon kaynağı eluted peptidler Kütle Spektrometre aktarmak için kullanılır. - (Tipik) MS koşulları: Sprey 1.6 gerilim kV; kapiller sıcaklık 250 ° c; Normalleştirilmiş çarpışma enerjisini 30. Kütle Spektrometre veri bağımlı modunda çalışır.
- MS spectra (örneğin, orbitrap) kitle Çözümleyicisindeki elde (m/z 350−1, 600) 70.000 ve otomatik kazanç kontrolü hedef 3 x 106çözünürlüğe sahip. 20 PİLGRİM6A parçalanma bir otomatik kazanç denetimi target 1 x 105için en yoğun iyonların seçin. Dinamik olarak tut daha önce iyonları 30 s. hariç tek başına iyonlar gibi iyonları tanınmayan şarj durumu ile tahsil için işaretli.
Not: Kilit kitle seçeneği39kullanarak toplu spectra ürününün dahili kalibrasyon gerçekleştirildi.
- % 2 ACN/0.1% formik asit kurutulmuş peptidler geçiyoruz. Peptidler bir sonication banyoda 2-3 dk ve spin için aşağı 16,200 x g, santrifüj 30 dk. Transfer içinde örnekleri otomatik örnekleyici tüpleri geçiyoruz.
-
Veritabanı arama
Not: Veritabanı arama için kullanılabilen farklı yazılım bulunmamaktadır. MaxQuant40, örneğin, serbestçe kullanılabilir.- .Çiğ dosyaları .mgf-pXtract dönüşüm aracını (http://pfind.ict.ac.cn/downloads.html) kullanarak dosyalarına dönüştürün.
- Tipik arama parametrelerini kullanarak veritabanı arama yapmak: veritabanı, swissprot; peptid kitle hoşgörü, 10 ppm; parça kitle hoşgörü, 0.5 Da; enzim, Tripsin; cevapsız bölünme siteleri, 2; değişken değişiklikler, carbamidomethylation (sistein) ve oksidasyon (metionin).
Not: deneysel ayarlara göre arama parametrelerini değiştirin. - Veritabanı arama sonucu kontrol edin. Protein puanı, tanımlanan peptidler sayısını, peptid puanları ve kitle doğruluk değerlendirmek; sıra kapsama tespit protein kapsama verir.
Not: Her arama motoru kendi bireysel puanlama algoritması vardır. Puanlama sistemi gözlenen tandem kitle spectra kalitesi ile peptid değerlendirin. İyi bir spektrum ana sinyalleri tespit peptid doğrulamak için (tam) iyon serisi göstermelidir. Genellikle peptid puanı olasılık tabanlı, Yani, peptid kaç nasıl büyük olasılıkla tanımlanan peptit dizisi elde edilen spektrum maçlar için bir ölçü birimi. Protein puanı peptid puanları genellikle türetilir ve bir protein saptanan proteinler listesinde rütbe için kullanılır.
3. kütle spektrometresi sağlam Protein kompleksleri (yerel kütle spektrometresi)
- Altın kaplı yayıcılar hazırlanması için electrospray iyonlaşma
- 25,41daha önce açıklandığı gibi cam kılcal üzerinden nanoflow yayıcılar hazırlamak için bir micropipette çektirmenin kullanın. Borosilikat kılcal 0,78 mm bir iç çapı ile kullanın.
Not: iğne çekme parametrelerini değiştirerek, ucu şekli ve boyutu değiştirilebilir ve örnek için ayarlanabilir. Kılcal farklı iç çapı ve duvar kalınlığı ile kullanılabilir. - Cam kılcal iletken malzeme (örneğin, altın veya Paladyum); ile kat altın bir plazma üreten bir sputter coater kullanımı yaygındır. Kaliteli kaplama elde etmek için üreticinin yönergeleri izleyin.
Not: Kaplama istikrarlı bir sprey ortak kapiller voltaj (aşağıya bakın) uygularken elde etmek için yeterli olmalıdır.
- 25,41daha önce açıklandığı gibi cam kılcal üzerinden nanoflow yayıcılar hazırlamak için bir micropipette çektirmenin kullanın. Borosilikat kılcal 0,78 mm bir iç çapı ile kullanın.
- Yerel kütle spektrometresi için numune hazırlama
Not: Tuzları, deterjanlar veya gliserol büyük miktarda electrospray iyonlaşma ile uyumlu değildir. Bu nedenle, arıtma arabellek uçucu, sulu tampon tarafından değiştirilir. 200 mM amonyum asetat yaygın olarak kullanılır. Boyutu dışlama spin sütunları veya Ultrafiltrasyon cihazları arabellek alışverişinde kullanmak. Bazı durumlarda, karmaşık istikrar ya da etkinlik arabellek exchange tarafından etkilenebilir. Kitle spectra dikkatle değerlendirin ve karmaşık etkinliği kontrol edin. Kofaktörler ya da katkı maddeleri analiz arabelleğe gerekirse ekleyin.- Boyutu dışlama spin sütunları hızlı arabellek alışverişinde kullanmak. Santrifüjü 1000 x g de depolama arabellek çıkarın ve 1 dk. 4 ° C ile akışı atın. Üç kez Santrifüjü tarafından takip 500 µL 200 mM amonyum asetat ekleyerek yıkayın. 20 µL üstüne belgili tanımlık sütun ve 4 dk 1000 x g ve 4 ° C, santrifüj protein örneği yükleyin.
Not: 1-10 µM protein kompleksi konsantrasyon olmalıdır. Prosedür-ebilmek var olmak geçici olmayan bileşenler analizi hala rahatsız eğer tekrarladı. - Deney protein örnek konsantre ve aynı arabellekte alışverişi için santrifüj filtreleri kullanın. Filtrasyon membran gözenek boyutu % 50 analiz proteinlerin boyutundan küçük kullanın.
- Protein örnek filtrasyon cihazın içine aktarmak ve 200 mM amonyum asetat ekleyin. Spin aşağı de 15.000 x g ve aracılığıyla akış atın. 200 mM amonyum asetat ekleyin ve aralıklarla tekrarlayın. Bu adımı birkaç kez yineleyin. Santrifüjü hız için üreticinin yönergelerini izleyin. 4 ° C'de aralıklarla gerçekleştirmek
Uyarı: Membran proteinlerinin membran filtre aygıt üzerinde çökelti eğilimindedir.
- Protein örnek filtrasyon cihazın içine aktarmak ve 200 mM amonyum asetat ekleyin. Spin aşağı de 15.000 x g ve aracılığıyla akış atın. 200 mM amonyum asetat ekleyin ve aralıklarla tekrarlayın. Bu adımı birkaç kez yineleyin. Santrifüjü hız için üreticinin yönergelerini izleyin. 4 ° C'de aralıklarla gerçekleştirmek
- Boyutu dışlama spin sütunları hızlı arabellek alışverişinde kullanmak. Santrifüjü 1000 x g de depolama arabellek çıkarın ve 1 dk. 4 ° C ile akışı atın. Üç kez Santrifüjü tarafından takip 500 µL 200 mM amonyum asetat ekleyerek yıkayın. 20 µL üstüne belgili tanımlık sütun ve 4 dk 1000 x g ve 4 ° C, santrifüj protein örneği yükleyin.
- Sağlam protein kompleksleri kitle spektrometrik analizi
Not: Orada Kütle Spektrometreleri yerli kütle spektrometresi, örneğin, quadrupole uçuş saat (Q-ToF) Kütle Spektrometre Sistemleri veya orbitrap Kütle Spektrometreleri için değiştirilebilir farklı üreticilerin farklı. Aşağıda açıklanan protokol bir Q-ToF araç üzerinde gerçekleştirildi.- Altın kaplı kılcal kapiller tutucu içinde koyun ve kapiller protein örneği 1-4 µL ile doldurun. İğne ucu bir cımbız ile açın.
- Kapiller sahibi ile nano-electrospray kaynağına bağlanmak ve kapiller pozisyonunu ayarlayýn. Kılcal ucu 0,5-1,5 pozisyon cm koni delik için. 80-150 L/h nanoflow gaz sprey başlatmak ve istikrarlı bir sprey korumak için gaz akışını ayarlamak için kullanın.
- Q-ToF enstrümanın tune sayfasındaki parametreleri ayarlayın. Tipik başlangıç koşulları: kılcal gerilim, 1,50 kV; koni gerilim, 80 V; RF lens 1 enerji, 80 V; çarpışma enerji, 20 V; diyafram 3, V. 13,6 iyi kitle spectra elde etmek için bu parametreleri değiştirmek. "Satın almak" düğmesini tıklatarak satın başlayın ve iyi bir kitle spektrum elde etmek için mümkün olduğunca çok sayıda taramaları birleştirin.
Not: En az 100 taramaları birleştirme önerilir.
- Sağlam protein kompleksleri tandem kütle spektrometresi
- Kütle spektrumu (Bölüm 3.3 Protokolü) açıklandığı gibi elde. Karmaşık bir proteinin bir habercisi iyon seçin.
- --Dan Bayan satın alma dosyasındaki MS/MS moduna değiştirin. MS/MS seçimi habercisi ayarla kitle.
- Düşük çarpışma enerji alım başlayın. Doğru öncü yelpazesi doğrulamak için birkaç taramaları (yaklaşık 20 inceden inceye gözden geçirmek) birleştirmek kitle. Protein kompleksi ayrışıp kadar çarpışma enerji artırmak. İyi bir kitle spektrum elde etmek için en az 500 taramaları birleştirir.
Not: Sökülen kompleksleri bazen düşük yoğunluklu var. Mümkün olduğunca çok sayıda taramaları birleştirerek çözünürlük ve sinyal-gürültü oranı artırabilir.
- Çözüm ayrılma olduğu gibi protein kompleksleri
Not: protein etkileşimleri protein kompleksleri içinde ek anlayış kazanmak için çözüm ayrılma gerçekleştirilebilir.- Protein örnek (Bölüm 3.2) açıklandığı şekilde hazırlayın. Çözücüler protein örnek ekleyebilir veya sağlam kompleksler alt kompleksleri ayırmak için pH değiştirebilirsiniz. Tipik solvent metanol, isopropanol ve ACN. Amonyum asetat pH Asetik asit veya amonyak çözüm eklenmesi ile değiştirin. Kitle spectra elde etmek (Bölüm 3.3 ve 3.4) açıklandığı gibi.
- Solvent veya çeşitli alt konut projeleri oluşturmak için pH aralığı miktarı değişir. Genellikle, solvent miktarı 5-%50 (son konsantrasyonu) ve tipik pH aralığı 4-9. Solvent (% 5) veya hafif değişiklikler olarak pH düşük konsantrasyon başlayın ve kütle spektrumu (bakınız Bölüm 3.3) elde etmek.
- Solvent miktarını artırmak veya alt kompleksleri çözümde oluşturulan kadar kademeli pH değiştirin. Alt konut projeleri analiz etmek için kitle spectra elde etmek.
- Veri ayarlama
Not: Alınan kitle spectra dışarıdan sezyum iyodür (CSI) çözüm kullanılarak kalibre.- 100 mg CSI 1 mL suda çözülür.
- CSI kütle spektrumu almak. Yukarıdaki analiz CSI kümeleri aynı m/z aralığında protein kompleksi olarak elde etmek için çarpışma enerji değişir.
Dikkat: CSI hızlı emitör ucunda precipitates ve koni kirlenmektedir. Sayıda elde yeterli bir spektrum kazanmak için gerektiği şekilde tarar. Yayıcıyı ne zaman tamamlanmak kaynağından çıkarın. - Alınan kütle spektrumu kullanılarak bir kalibrasyon dosyası ve bir olay yeri inceleme uzmanı başvuru dosyası olun.
- Kalibrasyon için alınan kitle spectra uygulanır.
Dikkat: Kalibrasyon kitle spectra ürününün ham veri kalıcı bir değişiklik olabilir. Sigara kalibre spectra gerekiyorsa, dosyanın bir yedek kopyasını yapmak.
- Veri işleme ve analiz
Not: Yerel yığın spectra ürününün veri analizi için birçok serbestçe kullanılabilir yazılım araçları vardır; Örneğin, Massign42 ya da UniDec43. Aşağıdaki iletişim kuralı el ile veri analizi aracı yazılım hem de karmaşık örnekleri için Massign kullanımını açıklar. Bu yazılım karmaşık kitle spectra analiz için çok uygundur. Online programı (http://massign.chem.ox.ac.uk/) kullanım için sağlanan yönergeleri izleyin.- Veri analizi için düzeltme parametreleri ayarlayarak spectra düz. Parametreleri ayarlayarak centroid spectra. İki bitişik doruklarına karmaşık modellerden karmaşık proteinin hesaplamak ' enstrüman yazılım araçları kullanılarak tepe zarf.
Dikkat: Çok yoğun yumuşatma (örneğin, ilişkili ligandlar kaybı) veri kaybına neden olabilir. - Massign42ile analizi için kütle spektrumu için bir zirve listesi oluşturun. Veri noktaları spektrum ve düzgün linearize. Protein kompleksleri atama, karmaşık kompozisyon hesaplamak veya karmaşık tepe zarflar benzetimini yapmak için çeşitli yazılım araçları kullanır.
- Veri analizi için düzeltme parametreleri ayarlayarak spectra düz. Parametreleri ayarlayarak centroid spectra. İki bitişik doruklarına karmaşık modellerden karmaşık proteinin hesaplamak ' enstrüman yazılım araçları kullanılarak tepe zarf.
4. kimyasal kuplajlı kütle spektrometresi ile ilişkilendirerek
Not: Mevcut çok sayıda cross-linking stratejileri vardır. Burada, Bis (sulfosuccinimidyl) suberate (BS3), protein-protein etkileşimlerini incelemek için yaygın olarak kullanılan bir amin-reaktif cross-linker nasıl kullanılacağını açıklar.
- 1.43 mg BS3 25 mM hisse senedi çözüm hazırlamak için 100 µL suda çözülür.
Not: Disuccinimidyl suberate (DSS) gibi diğer reaktifler suda çözünen değildir ve genellikle Dimetil sülfoksit (DMSO) çözülür. Bazı cross-linkers da isotopically etiketli ağır biçimde bulunur. Ağır kararlı izotop birleşmesiyle çapraz bağlı di-peptidler tepe çift veri değerlendirme sırasında yardımcı olan MS spectra oluşturur. Differentially kullanarak etiketli cross-linkers zaman hisse senedi çözümleri her iki türevleri, hazırlanan ve 1:1 karışık.
Dikkat: protein kompleksleri DMSO tarafından ayrılma önlemek için yüksek konsantrasyonlu bir hisse senedi çözüm hazırlanan ve su veya arabellek cross-linking reaksiyon önce ile seyreltilmiş. - BS3 protein kompleksi ekleyin. BS3 0.5-5 arasında değişen miktarlarda kullanmak en uygun cross-linker konsantrasyonu belirlemek için mM. Reaksiyon karışımları için bir thermomixer içinde 1 h 25 ° C'de kuluçkaya. 4-%12 degrade jelleri kullanın ve SDS-sayfa (bir örnek için bkz: Şekil 2 ) ilişkilendirerek sonuçları değerlendirmek için gerçekleştirin.
Not: tek alt birimleri hala görülebilir (Şekil 2) olmakla birlikte çapraz bağlantılı denetiminde görünür değildir, daha yüksek molekül ağırlıklı protein bantları SDS-sayfa sırasında elde edilen zaman BS3 en iyi konsantrasyon ulaşılır. - En iyi BS3 konsantrasyon ile cross-linking tepki yineleyin. Reaksiyon karışımları için bir thermomixer içinde 1 h 25 ° C'de kuluçkaya.
Not: Bazı protein kompleksleri oda sıcaklığında kararlı değil. Cross-linking reaksiyon da buza gerçekleştirilebilir; Ancak, reaksiyon süresi ayarlanması gerekir. -
Amin arabellek (örneğin, 50-100 mM Tris arabellek, pH 7.5, son konsantrasyonu) ekleyerek cross-linking tepki gidermek veya herhangi bir kalıntı cross-linking reaktif kaldırmak için etanol yağış gerçekleştirir.
- Su veya arabellek son hacmi 200 µL. eklemek 600 µL buz gibi etanol ve 20 µL 3 M sodyum asetat, pH 5.3 ulaşmak için tepki karışıma ekleyin. -20 ° c 2 h veya gece kalmak için kuluçkaya ve iyice karıştırın.
Not: Alternatif olarak, proteinler 80 ° C'de 30 dk ya da sıvı azot çöktürülmüş. - Spin aşağı de 16,200 x g ve 4 ° C'de 30 dakika süreyle dikkatle süpernatant kaldırın.
- Pelet 1 mL buz gibi %80 (v/v) etanol ile yıkayın. Spin aşağı de 16,200 x g ve 4 ° C'de 30 dakika süreyle dikkatle süpernatant kaldırın. Vakum santrifüj Pelet kuru.
- Su veya arabellek son hacmi 200 µL. eklemek 600 µL buz gibi etanol ve 20 µL 3 M sodyum asetat, pH 5.3 ulaşmak için tepki karışıma ekleyin. -20 ° c 2 h veya gece kalmak için kuluçkaya ve iyice karıştırın.
- SDS-sayfa çapraz bağlı protein gerçekleştirin. (Bölüm 2,1 ve 2,2) açıklandığı gibi içinde-jel protein sindirimi ve jel bantları kesti.
Not: Çözüm sindirim da gerçekleştirilir, ancak, genellikle ek ayrılık adımları (örneğin, boyut dışlama Kromatografi sindirilir peptidler,) gerektirir. -
Sıvı Kromatografi birleştiğinde kütle spektrometresi (Bölüm 2.3) açıklandığı gibi gerçekleştirin. Çapraz bağlı peptidler genellikle olduğu gibi bol düşük, çapraz bağlı örnek analitik arttırmak için kitle spektrometrik analizi aşağıdaki varyasyonlar geçerli.
- Sıvı Kromatografi ayırma sırasında uzun degradeleri kullanın (örneğin, 90 dk 65 dk, yerine görmek yukarıda).
- İki misli ücret peptidler PİLGRİM6A parçalanma hariç.
Not: İki misli ücret peptidler genellikle Intra-net-çapraz bağlantılı peptidler ("döngü peptidler" veya "tip 1" çapraz bağlantıları) vardır. - Differentially kullanırken reaktifler, cross-linking etiketli analizi, Yanisırasında "tepe seçmek" seçeneği kullanın, PİLGRİM6A parçalanma kitle spectra tanımlanmış kitle fark en yüksek çiftlerini varlığı tarafından tetiklenir.
Not: Her Kütle Spektrometre "Tepe seçmek" seçeneği kullanılamayabilir.
- Kullanım sound'lu çapraz bağlı di-peptidler tanımlanması için yazılım18 . Simge durumuna küçültülmüş veritabanları için kimlik kullanın. Tipik arama parametreleri: araç spectra, PİLGRİM6A; enzim, Tripsin; En fazla. cevapsız bölünme siteleri, 3; değişken değişiklikler, oksidasyon (metionin) ve carbamidomethylation (sistein); Cross-Linker, BS3; min. peptid uzunluğu, 4; En fazla. peptid uzunluğu, 100; min. peptid kitle, 400 Da; En fazla. peptid kitle, 10.000 Da; FDR, % 1.
Not: differentially kullanarak etiketli cross-linkers, bağlayıcı tarafından neden olduğu kitlesel artış yapılandırılması gerekir. Diğer yaygın olarak kullanılan yazılım kullandığında kimliği, örneğin, xQuest17, MassMatrix19, veya XlinkX15da vardır. - Veritabanı Arama sonuçları parçalanma spectra kalitesi ile değerlendirin. Çapraz bağlantıları kabul edilebilir spectra iyon dizi her iki peptidler (en az 4 bitişik iyonları) makul bir sinyal-gürültü oranı göstermelidir.
Not: differentially kullanarak etiketli cross-linking reaktifler, tepe çift olarak MS spectra bir ek kalite denetimi olarak kullanılabilir. - Gerekirse, yazılım araçları (örneğin, XVis, XiNET) kullanarak protein etkileşim ağları cross-linking sonuçlarında görselleştirmek. Kullanım araziler bar ya da dairesel protein etkileşimleri görselleştirme için çizer.
Not: Her iki yazılım araçları web sunucusu üzerinde serbestçe kullanılabilir. Ayrıntılı talimatları ilgili web sitelerinde (https://xvis.genzentrum.lmu.de/ ve http://crosslinkviewer.org/).
Sonuçlar
Yapısal proteinler ve onlar formu kompleksleri onların işlevini anlamak için temel analizidir. Boyutu ne olursa olsun ilgi hemen hemen her karmaşık uygulanabilir veya heterojenite örnek kütle spektrometresi yapısal soruşturma için önemli ölçüde katkıda bulunur. Protokol iki iyi karakterize protein kompleksleri kullanarak örnekler; ilk olarak, hetero-dodecamer RvB1/B2 C. thermophilum ve saniye, hetero-octameric CPS E. colikarmaşık.
İlk kez, iki kompleksleri protein bileşenleri tespit. Bunun için proteinler SDS-PAGE tarafından (Şekil 2) ayrı kaldık ve jel bantları jel kesilmiş. Jel sindirimi proteinlerin sonra peptid karışımı tarafından sıvı Kromatografi birleştiğinde kütle spektrometresi analiz edildi ve peptid ve parça kitleler veritabanı arama için tabi tutuldu. Bu iş akışını takip biz yüksek güven ile Yani, tüm protein alt iki kompleksleri, tespit peptidler makul peptid puanları ile çok sayıda yüksek sıra kapsama tüm protein alt (Tablo 1 için verimli gözlenmiştir ).
Biz o zaman yerli kütle spektrometresi (şekil 3A) tarafından olduğu gibi RvB1/B2 karmaşık analiz edildi. Kütle spektrumu iki tür, bir yaklaşık 8.000 m/z ve başka bir tür yaklaşık 11.000-12.000 m/zsaptandı. Bu türler için hesaplanan kitleler hexameric (RvB1)3(RvB2)3 yüzük (yaklaşık 310 kDa) ve dodecameric çift-ring (RvB1)6(RvB2)6 (yaklaşık 620 kDa) karşılık gelir. Her iki tepe serinin iki popülasyonun gösterisi; Bunlar onun etiketli ve etiketsiz RvB2 alt birimleri komplekslerinde karışımı kaynaklı. RvB1/B2 karmaşık için kristal yapısı daha önce36 elde edildi ve çift-ring (şekil 3B) yerleşimi gösterir. Yerel kitle spektrum olduğu gibi dodecamer stoichiometry bu nedenle onaylar ve ayrıca istikrarlı bir alt kompleksi ortaya koymaktadır. Buna ek olarak, ortak mevcut nüfus tanımlanır.
RvB1/B2 kompleks içinde protein etkileşim siteleri belirlemek için biz kimyasal olarak saf karmaşık BS3 cross-linker ile cross-linked. İlk BS3 miktarı en yüksek konsantrasyonu belirlemek için cross-linking reaksiyonu sırasında titre. BS3 Amin özgü ve kovalent bağlantılar lizin yan zincirleri gibi proteinlerin N termini olduğunu. Cross-linking tepki SDS-PAGE tarafından (şekil 2A) izledi. Çapraz bağlantılı kompleks RvB1 ve RvB2 alt birimleri gösterdi. BS3 reaksiyon karışıma ekleyerek protein bantları daha yüksek molekül ağırlıklı, sonuçlanan proteinlerin kovalent bağ neden oldu. SDS jel çapraz bağlı protein alt azalır iken BS3 miktarda artan çapraz bağlı türler daha yüksek miktarlarda verim gösterir. Daha sonra jel protein grup kesmek ve protein etkileşim siteleri tanımlamak için yukarıda verilen protokolü takip ettim. Bir çapraz bağlı di-peptid bir örnek spektrum (şekil 3 c) gösterilir. Yelpazenin her iki peptidler bu protein etkileşimi teyit y-iyon dizi gösterir. Toplamda, alt birimleri RvB1 ve RvB2 ve RvB2 iki kopyası arasındaki iki çapraz bağlantıları arasında dört çapraz bağlantıları da dahil olmak üzere 14 protein etkileşimler elde edilen (Tablo 2). BS3 sonuçlarından Cross-linking intra moleküler etkileşimleri yanı sıra farklı alt birimleri arasındaki etkileşimleri gösteren bir etkileşim ağı (şekil 3D) içinde görüntülenir. İntra moleküler çapraz bağlantıları tavsiye ederim RvB1 ve RvB2 alt birimleri N ve C terminali etki alanları yakın bir şekilde katlayın. Dikkat, intra moleküler etkileşimler aynı alt birim arası moleküler etkileşim bu durumda seçkin olduğunu. İki alt birimleri arasında arası moleküler çapraz bağlantıları da tespit edildi. Bunlar cross-linking yaklaşım doğrulama yapısı içinde RvB1 ve RvB2 arasında iki arası moleküler çapraz bağlantıları görüntülenmeyecektir. Diğer arası moleküler çapraz bağlantıları kristal yapısı içinde bulunmayan esnek döngüler içinde yer alır. Biz de aynı peptid dizileri içeren iki çapraz bağlantıları RvB2 içinde tespit. Bu çapraz bağlantıları belirsizliğe yer bırakmadan aynı protein (şekil 3D) iki kopyasını kaynaklanan gerekir arası moleküler olarak sınıflandırılabilir. Bizim cross-linking deneyler protein etkileşim siteleri kompleksi içinde aynı zamanda varolan kristal yapısı (şekil 3B) tarafından da teyit kendi Yapısal düzenleme anlayışlar sağlayan protein alt içinde ortaya koyuyor.
Biz okudu ikinci protein kompleksi CPS yapıldı. Yerel kitle spektrum (şekil 4A) üç protein kompleksleri arasında 6000 ve 12.000 m/zsaptandı. 640 kDa en büyük karmaşık olduğu gibi hetero-octamer karşılık gelir. Daha küçük kompleksleri iki alt kompleksleri temsil eder; küçük ve büyük CPS altbirimden (160 kDa) ve her alt birim (320 kDa) iki kopyasını içeren bir heteroseksüel tetramer dimer. Bu alt komplekslerin protein derleme içine ilk anlayışlar teslim; Yani, büyük ve küçük alt birimleri doğrudan temas (hetero-dimer tarafından ortaya gibi) ve tetramer iki dimer bir ürün olabilir. Sağlam CPS karmaşık yapısal düzenleme hakkında daha fazla bilgi elde etmek için tandem kütle spektrometresi (MS/MS) hetero-octamer ve heteroseksüel tetramer gerçekleştirilen. Her iki durumda da habercisi küçük alt birim derleme (4B rakam) çevre içinde yer aldığını öne üzerinden ayrışmış küçük alt birim. Gerçekten de, küçük alt birim kullanılabilir kristal yapısı (şekil 4 c)44periferik.
Kimyasal BS3 cross-linker kullanarak cross-linking da gerçekleştirildi. Artan miktarda kullanarak, CPS alt birimleri kovalent bağ geliştirilmiş oldu. Proteinlerin sindirimi ve yukarıda açıklandığı gibi peptidler analiz sonra birçok protein etkileşimleri büyük alt birim ve küçük alt birim içinde bir hibrid içinde (Tablo 2) elde edilmiştir. Ayrıca, benzer şekilde RvB1/B2 karmaşık, büyük CPS alt birim iki kopyası arasındaki iki arası moleküler çapraz bağlantılar bulduk. Bu çapraz bağlantıları her C-terminal yan yana karşı karşıya iki büyük alt birimleri yerleştirin. Bir önceki çalışmada, yapısal kütle spektrometresi ve sayısal modelleme35, biz hangi büyük olasılıkla iki büyük alt birim tarafından onaylanmış kopyalarını arabiriminden kaynaklanan üç ek etkileşimleri büyük alt birim olarak tespit kristal yapısı ve elde edilen modeli (şekil 4 c ve Tablo 2). Bu etkileşimler CPS çekirdek düzenlenmesi dört büyük alt birimleri oluşan karmaşık olanak sağlar. Ancak, küçük ve büyük alt birimleri arasında hiçbir arası alt birim çapraz bağlantıları tespit edildi. Kullanılabilir kristal yapısı (şekil 4 c) inceleyerek, hangi açıklayabilir büyük alt birim ve periferik küçük alt birimleri oluşan kompleks tetrameric çekirdek arasındaki etkileşim yüzey çok küçük olduğunu ortaya çıkıyor arası alt birim etkileşimleri olmaması. Bu küçük alt birim kolayca sağlam kompleksler küçük bağlama arabirimi nedeniyle büyük olasılıkla gelen ayrışıp gösterdi yerli kütle spektrometresi tarafından doğrulanır. Yine de, CPS karmaşık kimyasal cross-linking ve yerel kütle spektrometresi birlikte protein etkileşimleri kendi Yapısal düzenleme (şekil 4 d) araştırma izin.
Birlikte ele alındığında, yerel kütle spektrometresi ve kimyasal çapraz bağlı peptidler, kitle spektrometrik tanımlaması ile birleştiğinde cross-linking kombinasyonu her iki örnek kompleksleri yapısal düzenlemenin sulandırma sağlar. Kimyasal ilişkilendirerek ortaya protein alt, örneğin etkileşim RvB1 ve RvB2 arasında veya CPS, tetrameric çekirdek içinde düzenlenmesi sırasında yerel kütle spektrometresi protein stoichiometries sağlam kompleksleri, teslim ve yaygın subcomplexes. CPS, kendisi için hiçbir arası moleküler etkileşim iki alt birimleri arasında kimyasal cross-linking tarafından gözlenen durumunda büyük her alt birim bir küçük alt (şekil 4 d) ile etkileşim yerli kütle spektrometresi öneriyor. Tandem kütle spektrometresi önerilen bir karmaşık ve her iki alt birimleri arasında küçük bir arabirim küçük alt birimi periferik yerleşim.
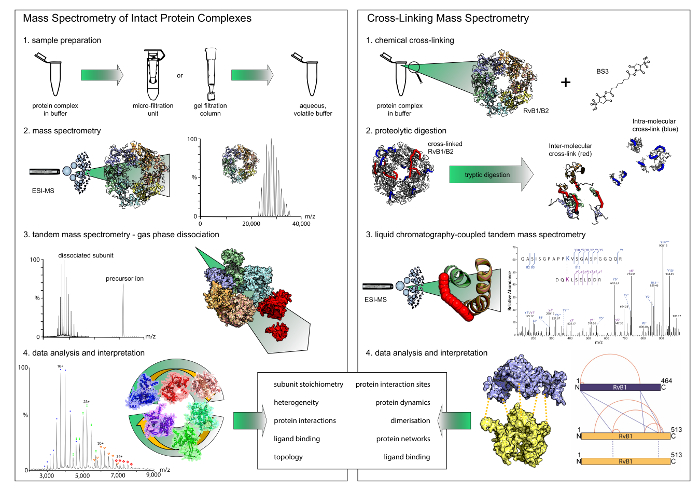
Şekil 1: iş akışı yerel kütle spektrometresi ve cross-linking. Her iki tekniğin de tamamlayıcı sonuçlar. Yerel kütle spektrometresi stoichiometries ve etkileşim modülleri ortaya koymaktadır iken, cross-linking kompleksleri içerisinde protein etkileşim siteleri anlayışlar verir. Not Yalnızca kimyasal cross-linking ikili etkileşimleri ortaya koymaktadır. (A) yerel kütle spektrometresi ilk adımda uçucu ve sulu arabellek filtre cihazları veya jel filtrasyon sütunları kullanarak Exchange'e arabellek olduğunu. Kütle spektrometresi sağlam protein kompleksleri, sonra kendi stoichiometry ortaya koymaktadır. Tandem kütle spektrometresi deneylerde periferik alt birimleri ayrışmış. (B) kimyasal cross-linking için protein kompleksi cross-linking reaktif ile inkübe. Çapraz bağlı protein sonra tarafından sıvı Kromatografi birleştiğinde kütle spektrometresi daha sonra analiz peptidler içine sindirmek. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
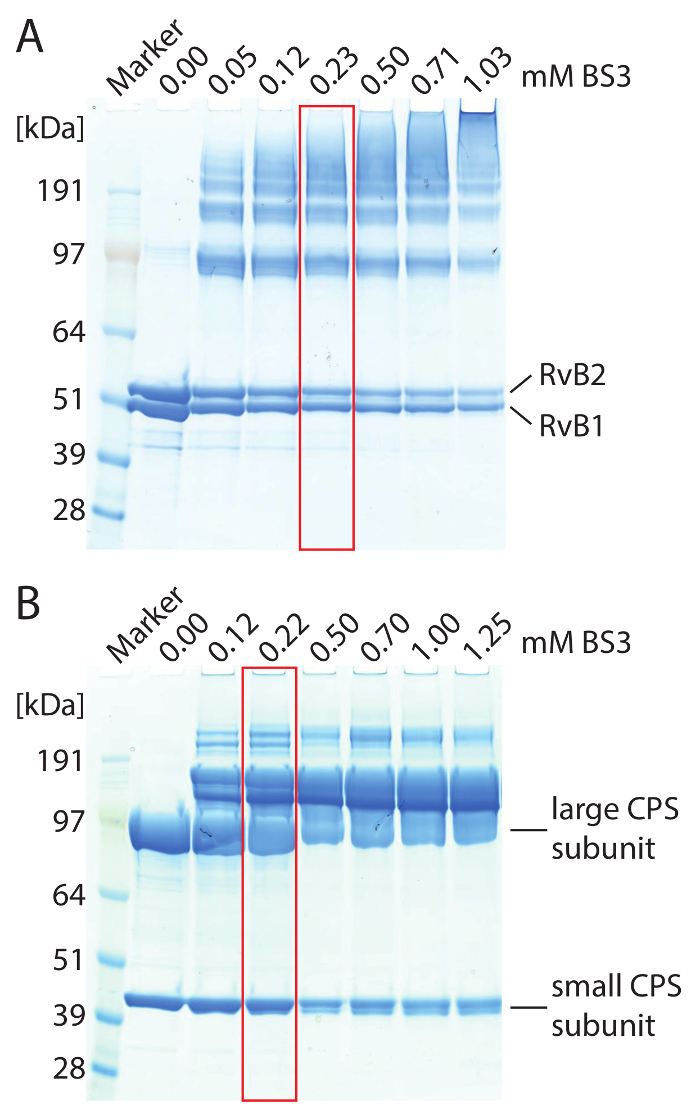
Şekil 2: SDS-sayfa, çapraz bağlı RvB1/B2 (A) ve CPS (B) kompleksleri. (A) 2.5 µM RvB1/B2 jel lane yüklü. BS3 konsantrasyonu çeşitli. Sigara çapraz bağlı RvB1/B2 yaklaşık 50 kDa iki protein alt gösterir. BS3 yanı sıra protein bantları daha yüksek molekül ağırlıklı, sonuçlanan protein alt kovalent bağ neden oldu. Çapraz bağlı türler miktarı ile daha yüksek BS3 konsantrasyonları artar. Optimal koşullar cross-linking vurgulanan (kırmızı) vardır. (B) 10 µM CPS jel lane yüklü. Büyük (90 kDa) ve küçük (40 kDa) CPS alt birimleri elde edilir. BS3 yanı sıra protein bantları daha yüksek molekül ağırlıklı, sonuçlanan protein alt kovalent bağ neden oldu. Optimal koşullar cross-linking vurgulanan (kırmızı) vardır. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
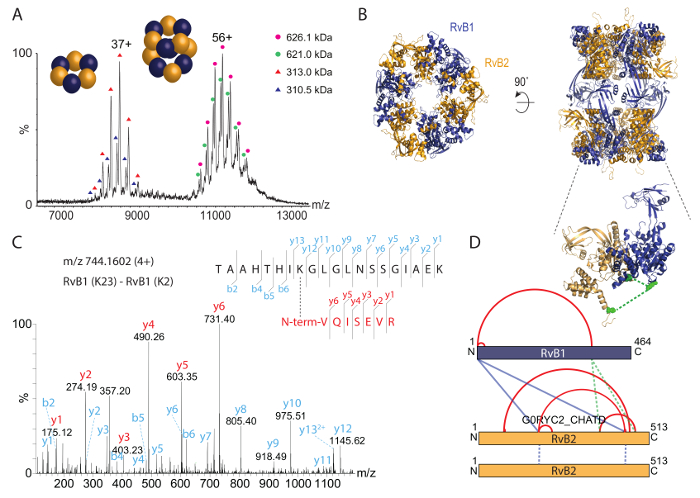
Şekil 3: Yerel kütle spektrometresi ve kimyasal RvB1/B2 karmaşık cross-linking. (A) yerel kitle spektrum RvB1/B2 ortaya iki tür; sağlam dodecamer (Yani, (RvB1)6(RvB2)6) yaklaşık 11.000-12.000 m/z ve hexameric yüzük (RvB1)3(RvB2)3 yaklaşık 8.000 m/z. Her iki tür iki popülasyonun onun etiketli ve etiketsiz RvB2 kaynaklanan göster. Spektrum35değiştirildi. RvB1/B2 (B) kristal yapısı (PDB kimliği 4WVY) gösterilir. RvB1 ve RvB2 alternatif alt birimleri iki hexameric halkalar oluştururlar. (C) bir çapraz bağlı di-peptid parçalanma spektrum. N-terminus RvB1, çapraz bağlı, RvB1. K23 ile her iki peptidler (kırmızı ve mavi) için y-iyon serisi elde edilmiştir. RvB1/B2 karmaşık elde (D) içi ve arası protein etkileşimler. Intra-net çapraz bağlantılar kırmızıyla gösterilir inter-cross-links mavi renkle gösterilir. Eklemek hangi kristal yapısı (yeşil, INSERT) görüntülenmeyecektir RvB1 ve RvB2 alt birimleri arasında iki arası moleküler çapraz bağlantıları gösterir. İki RvB2 kopya--dan kaynaklanan etkileşimleri mavi noktalı çizgiler olarak gösterilmiştir. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
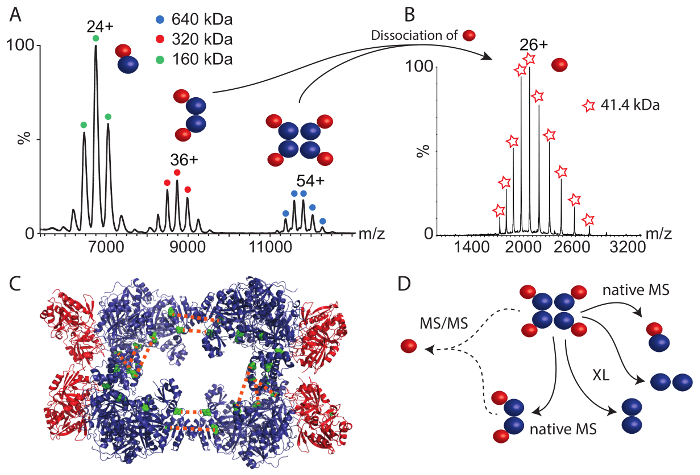
Şekil 4: Yerel kütle spektrometresi ve kimyasal CPS cross-linking. (A) CPS gösterir yerel kütle spektrumu üç kompleksleri. Hetero-dimer (160 kDa), hetero-tetramer (320 kDa) ve hetero-octamer (640 kDa). Spektrum35değiştirildi. (B) tetrameric ve octameric CPS karmaşık ortaya ayrılma bir küçük CPS alt birimi Tandem kütle spektrometresi. (C) kristal yapısı CPS (PDB kimliği 1BXR) gösterilir. Büyük alt birimleri bir tetrameric oluşturmak ve küçük alt birimleri kompleksi çevre içinde yer alır. İki büyük alt birim kopyaları arasındaki arası moleküler çapraz bağlantıları (yeşil) gösterilir. Büyük ve küçük CPS alt birimleri (D) etkileşimleri. Yerel kütle spektrometresi subcomplexes ortaya ve küçük alt birim periferik konumunu göstermektedir. Kimyasal çapraz bağlantıları tetrameric temel düzenlemeler CPS gösterir. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Tablo 1: veritabanı arama sonuçlarını. Proteinler sıvı Kromatografi birleştiğinde kütle spektrometresi ve veritabanı arama tarafından tespit edilmiştir. Protein adları katılım numarası ve açıklaması gibi protein kitle verilir. Protein puanı, protein başına gözlenen spectra sayısı ve gözlenen peptid sıraların sayısını belirtir. En yüksek maskot peptidler puanları ile beş peptidler her protein alt birimi için listelenir. Bu dosyayı indirmek için buraya tıklayınız.
Tablo 2: çapraz bağlantıları gözlenen RvB1/B2 ve CPS. Alt birimleri kompleksleri ve çapraz bağlı artıkları verilir. Hibrid türünü (Intra - veya arası moleküler) çakışan peptid sýra ya da bir önceki çalışma35ortaya çıktı. Bu dosyayı indirmek için buraya tıklayınız.
Tartışmalar
İletişim kuralları kütle spektrometresi tabanlı yapısal analizi çok alt birim protein kompleksleri için sağlanır. İletişim kuralında, açıklanan iki teknikleri, çoğunlukla tamamlayıcı sonuçlar ve protein içinde yapısal düzenlemeler anlayışlar kazanmak için uygundur (-ligand) tarafından geleneksel yapı teknikleri eğitim zor olan kompleksleri. Yerel kütle spektrometresi subcomplexes ve istikrarlı etkileşim modülleri analiz ederek protein stoichiometries yanı sıra protein etkileşimleri içine anlayışlar sunar. İlişkilendirerek, öte yandan, doğrudan ilgili siteler hakkında bilgi verir. Bağlı olarak kullanılan cross-linker, belirli bir esneklik olabilir veya analize dahil.
Genel olarak gerçekleştirmek kolay ve değil zaman alıcı sağlanan kurallarıdır. Tüm iletişim kuralı bir hafta içinde çalıştırılabilir ve belirli bir miktarda protein kompleksi başarılı analiz için gerekli olmasına rağmen hemen hemen tüm protein kompleksleri için uygulanabilir. Numune hazırlama basittir ve özellikle saf protein kompleksleri gerektirmez. Ancak, yaygın bir hatadır kütle spektrometresi tabanlı protein tanımlama için numune hazırlama sırasında örnek kirlenme var. Çoğu durumda bu contaminations toz, deri veya saç kaynaklanan keratins içerir. Bu nedenle, eldiven ve laboratuvar mont giyiyor gibi ilave, Sulu arabellekleri filtrating ve yüksek saflıkta çözücüler kullanılarak kütle spektrometresi tabanlı protein tanımlama için numune hazırlama sırasında özen gösterilmelidir. Benzeşme Etiketler kullanırken chaperones gibi bulaşıcı diğer proteinler protein arıtma sırasında örneğin, genellikle tanıtılmaktadır. Bu gibi durumlarda, protein saflaştırma, örneğin çamaşır adımları artırarak geliştirilmesi. Her durumda, örnekteki protein contaminations kolayca veritabanı arama sırasında taksonomi filtre ihmal olarak tanımlanır (Yani, tüm türlerden proteinler karşı arama). Eğer sadece birkaç peptidler gözlenir (Yani, düşük protein kapsama elde edilebilir), yeterli örnek kullanılabilir olsa bile, farklı bir İndinavir sindirim sırasında kullanmak gerekli olabilir. Genel olarak tripsin peptidler yeterli sayıda verir; Ancak, membran proteinlerinin veya proteinlerin membran etki alanları gibi bazı durumlarda tryptic bölünme sitelerin sayısı azalır ve hidrofobik amino asitler hedefleme diğer enzimler daha iyi bir seçimdir.
Araçları açısından, özellikle değiştirilmiş bir enstrüman gaz fazına aktarmadan kovalent olmayan etkileşimleri tutar yerli kütle spektrometresi gereklidir. Q-ToF ve orbitrap aletler dahil olmak üzere çeşitli araç türleri getirilmiştir. Değiştirilmiş Q-ToF Kütle Spektrometre Sistemleri beri birkaç yıl için yerel kütle spektrometresi ticari olarak mevcut olmakla birlikte, ikinci ancak son zamanlarda kullanılmaya başlanan ve çoğu durumda özel değişiklik45gerektirir. Ancak, uygulama yüksek çözünürlüklü aletleri bağlama birden çok ligandlar ve onların miktar46,47 eğitim izin ve gelecekteki uygulamalar için umut verici.
Çapraz bağlı di-peptidler tarafından sıvı Kromatografi birleştiğinde kütle spektrometresi tanımlamak için birkaç değişiklik ile standart yordamlar uygulayabilirsiniz. Ancak, veritabanı arama sınırlayıcı bir faktör olarak özel yazılım nadiren büyük veritabanları ile başa çıkabilirim ve kompleksleri protein alt içeren azaltılmış veritabanları gerekli olur. Son yıllarda yapılan çalışmalarda kullanılan Kütle spektrometresi yarilabilir cross-linkers protein etkileşimler tüm hücre lysates48,49hedefleme. Tandem kütle spektrometresi deneylerde çoğunlukla parçası kimyasal cross-linkers kullanımı daha fazla parçalanma tarafından tanımlanan, (cross-linker tarafından değiştirilmiş) doğrusal peptidler verimleri ve doğrusal peptidler ve bu veritabanı arama azaltır arama süresi ve Hesaplamalı Arama alanı. Ancak, bu deneyler gerçekleştirmek için bir iyon kapanı kitle Analyzer'ı veya bir melez Kütle Spektrometre ile bir iyon kapanı gereklidir. Yanlış-mutlak önemli bir konu olduğundan, genel olarak, çapraz bağlı peptidler kitle spectra kez el ile veri analiz süresini son derece uzanan kalitesi onların parçası spectra ile doğrulanır. Daha fazla doğrulama adımları bu nedenle potansiyel gelecekteki uygulamalar olmadan, uygulanacak sağlam puanlama sistemlerinin geliştirilmesi. Veri Çözümleme geliştirmek ve yanlış-mutlak sayısını azaltmak için bir yol yanlış bulma oranı hesaplamalarını ve veri kümeleri50cross-linking üzere giriş oldu.
Genel olarak, burada açıklanan teknikler daha fazla kütle spektrometresi teknikleri ile tamamlanmaktadır (örneğin, kovalent etiketleme) analizi çıktısı artırmak için. Diğer değişiklikler ve geliştirmeler iletişim kurallarının kolayca uygulanabilir. Bu nedenle karşılaştırmalı cross-linking34 protein derlemesinde konformasyon değişiklikleri unravels. Daha fazla yerel kütle spektrometresi gelişmeler günümüzde membran proteinleri51,52 analizi ve onların etkileşim lipidler28,52,53,54 ile izin . Yüksek çözünürlüklü Kütle Spektrometreleri yerli kütle spektrometresi için yeni gelişmeler uygulama ve ligand taşıması, örneğin, lipitler membran proteinlerinin için bağlama genişletilmiş var, şimdi analiz45', dahil edilebilir 46. sayısal modelleme yaklaşımlar ile birlikte, bu tekniklerin çözünürlük55değişen Yapısal modeller sunabilirsiniz. Hiçbir Kristal yapıları olduğu gibi karmaşık veya tek alt birimleri için kullanılabilir varsa, kütle spektrometresi ilk yorumlara protein etkileşimleri ve bilinmeyen karmaşık topoloji teslim edebilirsiniz. Kullanılan teknikler ve elde edilen sonuçlar bağlı olarak, düşük çözünürlüklü modeller bilinmeyen kompleks56,57,58elde edilebilir. Kristal yapıları veya homoloji modelleri kullanılabiliyorsa, kütle spektrometresi alınan yapısal bilgi bile yakınındaki yerli modelleri59yol açabilir.
Diğer yapısal teknikleri ile karşılaştırıldığında, kütle spektrometresi avantajı vardır bu düşük örnek gerektiren, türdeş olmayan örnekleri ile başa çıkabilirim ve Sınırsız büyüklükte protein kompleksleri için geçerlidir. Ayrıca, kütle spektrometresi dinamik protein sistemleri araştırma sağlar. Çözüm içinde protein veya protein kompleksi farklı nüfus genellikle birlikte analiz edilir ve bu nedenle, farklı olarak belirli nüfusun seçimi gerektiren diğer yapısal teknikleri ile tüm biçimler sırasında korunur analiz ve olan bir deneyde değerlendirilebilir. Kantitatif yaklaşımlar cross-linking son zamanlarda tanıtılan34,60,61 ve gelecekteki uygulamalar farklı koşullar altında konformasyon değişiklikleri açıklayan için umut verici.
Açıklamalar
Yazarlar ifşa gerek yok.
Teşekkürler
Biz bizim meslektaşları yararlı tartışmalar için teşekkür ederim. Biz de Ilme Schlichting ve Karl-Peter Hopfner protein kompleksleri verdiğiniz için teşekkür ederiz. Biz Federal Bakanlıktan eğitim ve araştırma için fon kabul (BMBF, ZIK programı, 03Z22HN22), Avrupa Bölgesel Kalkınma Fonu (EFRE, ZS/2016/04/78115) ve MLU Halle-Wittenberg C.S. ve Wellcome Trust (109854/658 finansman Z/15/Z) A.P. için
Malzemeler
| Name | Company | Catalog Number | Comments |
| Chemicals, Reagents, Consumables | |||
| 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid (HEPES) | Sigma Aldrich | H4034 | buffer |
| Acetic acid | Sigma Aldrich | 695092 | pH |
| Acetonitrile Optima LC/MS | Fisher Chemicals | A955-1 | solvent |
| Amicon Ultra centrifugal devices (different MWCO) | Millipore | i.e. UFC500396 | buffer exchange, concentration |
| Ammonium acetate solution | Sigma Aldrich | A2706 | native MS |
| Ammonium bicarbonate | Sigma Aldrich | 9830 | in-gel digestion |
| Ammonium solution | Sigma Aldrich | 9859 | pH |
| Bis(Sulfosuccinimidyl)suberate-d0 (BS3-d0) | Thermo Scientific | 21590 | cross-linker |
| Bis(Sulfosuccinimidyl)suberate-d4 (BS3-d4) | Thermo Scientific | 21595 | cross-linker |
| Caesium iodide | Sigma Aldrich | 203033 | calibration |
| Calcium chloride | Sigma Aldrich | C5670 | in-gel digestion |
| Capillaries (1.0 OD × 0.78 ID × 100 L mm) | Harvard Apparatus | 30-0038 | native MS |
| Disuccinimidyl suberate (DSS) | Thermo Scientific | 21655 | cross-linker |
| DL-Dithiothreitol | Sigma Aldrich | D5545 | in-gel digestion |
| DMSO (dimethyl sulfoxide) | Sigma Aldrich | D8418 | solvent |
| Ethanol | Fisher Chemicals | BP2818 | solvent |
| Formic acid Optima LC/MS | Fisher Chemicals | A117-50 | solvent supplement |
| Instant Blue Coomassie staining solution | expedeon | ISB1L | staining solution for gel electrophoresis |
| Invitrogen NuPAGE 4-12% Bis-Tris gels (1 mm, 10 well) | life technologies | NP0323BOX | gel electrophoresis |
| Iodoacetamide | Sigma Aldrich | I1149 | In-gel digestion |
| Isopropyl alcohol, HPLC grade | Fisher Chemicals | P750717 | solvent |
| Methanol | Fisher Chemicals | A456-212 | solvent |
| Micro BioSpin 6 columns | BioRad | 732-6222 | buffer exchange |
| NuPAGE Antioxidant | invitrogen | NP0005 | running buffer, gel electrophoresis |
| NuPAGE LDS Sample Buffer (4 ×) | invitrogen | NP0007 | sample loading buffer (non-reducing) for gel electrophoresis |
| NuPAGE MES SDS Running buffer | life technologies | NP0001 | gel electrophoresis |
| NuPAGE MOPS SDS Running buffer | life technologies | NP0001 | gel electrophoresis |
| NuPAGE Sample Reducing Agent (10 ×) | invitrogen | NP0004 | reducing Agent for gel electrophoresis |
| PBS - phosphate buffered saline | Sigma Aldrich | P4417 | buffer |
| SeeBlue Plus2 Prestained Standard Protein Marker | invitrogen | LC5925 | prestained Protein Marker for gel electrophoresis |
| Sodium acetate | Sigma Aldrich | S2889 | ethanol precipitation |
| Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 252859 | buffer |
| Trypsin | Sigma Aldrich | TRYPSEQ-RO (11418475001) | in-gel digestion |
| Vivaspin centrifugal devices (different MWCO) | Sartorius | i.e. VS0101 | buffer exchange, concentration |
| Water for HPLC | Sigma Aldrich | 270733-2.5L-M | solvent |
| Name | Company | Catalog Number | Comments |
| Instruments | |||
| Centrifuge Heraeus Fresco 21, bench-top centrifuge | Thermo Scientific | 75002425 | |
| Flaming / Brown Micropipette puller Model P-1000 | Sutter Instruments | P-1000 | |
| Gelelectrophoresis chamber Xcell SureLock MiniCell | Invitrogen | EI0001 | |
| Gold Coater Quorum Q150R S | Quorum Technologies Ltd. | Q150RS | |
| Horizontal gel shaker Rotamax 120 T | Heidolph Instruments | 544-41200-00 | |
| Q-TOF Ultima mass spectrometer, MS Vision high mass upgrade | Waters (Micromass) | - | |
| reversed-phase C18 analytical column Acclaim PepMap (C18, 75 mm I.D., 50 cm, 3 mm pore size) | Thermo Scientific | 164570 | |
| reversed-phase C18 pre-column (C18, 150 mm I.D., 2 cm, 5 mm pore size) | Thermo Scientific | 164213 | |
| scalpel | Fisher Scientifc | 10463989 | |
| SpeedVac SPD121P vacuum centrifuge | Thermo Scientific | SPD121DP-230 | |
| Thermomixer C | Eppendorf | 5382000015 | |
| Tweezers AA | Sigma Aldrich | Z680184-1EA | |
| Ultrasonic cleaner USC-TH, sonication bath | VWR | 142-0084 | |
| UltiMate Dionex 3000 nano-LC system, coupled to Q-Exactive plus hybrid mass spectrometer (nano-ESI source) | Thermo Scientific | 0726030+ | |
| Name | Company | Catalog Number | Comments |
| Software, Software Tools, Database search | |||
| Mascot | www.matrixscience.com | ||
| Massign | http://massign.chem.ox.ac.uk | ||
| MassLynx | Micromass | ||
| MassMatrix | Xu, H. J Proteome Res. 9 (2010) | ||
| MaxQuant | Cox, J. Nat. Biotechn. 26 (2008) | ||
| pLink | Yang, B. Nat Methods 9 (2012) | ||
| pXtract conversion tool | http://pfind.ict.ac.cn/downloads.html | ||
| UniDec | Marty, M.T. Anal. Chem. 87 (2015) | ||
| XCalibur | Thermo Scientific | ||
| XiNET | http://crosslinkviewer.org | ||
| XLinkX | Liu, F. Curr Op Struct Biol 35 (2015) | ||
| xQuest | Rinner, O. Nat Methods 5 (2008) | ||
| Xvis | https://xvis.genzentrum.lmu.de |
Referanslar
- Campbell, I. D. Timeline: the march of structural biology. Nat Rev Mol Cell Biol. 3 (5), 377-381 (2002).
- Liko, I., Allison, T. M., Hopper, J. T., Robinson, C. V. Mass spectrometry guided structural biology. Curr Opin Struct Biol. 40, 136-144 (2016).
- Robinson, C. V. From molecular chaperones to membrane motors: through the lens of a mass spectrometrist. Biochem Soc Trans. 45 (1), 251-260 (2017).
- Lossl, P., van de Waterbeemd, M., Heck, A. J. The diverse and expanding role of mass spectrometry in structural and molecular biology. EMBO J. 35 (24), 2634-2657 (2016).
- Wang, L., Chance, M. R. Protein Footprinting Comes of Age: Mass Spectrometry for Biophysical Structure Assessment. Mol Cell Proteomics. 16 (5), 706-716 (2017).
- Steen, H., Mann, M. The ABC's (and XYZ's) of peptide sequencing. Nat Rev Mol Cell Biol. 5 (9), 699-711 (2004).
- Olsen, J. V., Mann, M. Status of large-scale analysis of post-translational modifications by mass spectrometry. Mol Cell Proteomics. 12 (12), 3444-3452 (2013).
- Schmidt, C., Robinson, C. V. Dynamic protein ligand interactions--insights from MS. FEBS J. 281 (8), 1950-1964 (2014).
- Marcsisin, S. R., Engen, J. R. Hydrogen exchange mass spectrometry: what is it and what can it tell us?. Anal Bioanal Chem. 397 (3), 967-972 (2010).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Leitner, A., Lindner, W. Current chemical tagging strategies for proteome analysis by mass spectrometry. J Chromatogr B Analyt Technol Biomed Life Sci. 813 (1-2), 1-26 (2004).
- Leitner, A., Lindner, W. Chemistry meets proteomics: the use of chemical tagging reactions for MS-based proteomics. Proteomics. 6 (20), 5418-5434 (2006).
- Kiselar, J. G., Chance, M. R. Future directions of structural mass spectrometry using hydroxyl radical footprinting. J Mass Spectrom. 45 (12), 1373-1382 (2010).
- Xu, G., Chance, M. R. Hydroxyl radical-mediated modification of proteins as probes for structural proteomics. Chem Rev. 107 (8), 3514-3543 (2007).
- Liu, F., Heck, A. J. Interrogating the architecture of protein assemblies and protein interaction networks by cross-linking mass spectrometry. Curr Opin Struct Biol. 35, 100-108 (2015).
- Rappsilber, J. The beginning of a beautiful friendship: cross-linking/mass spectrometry and modelling of proteins and multi-protein complexes. J Struct Biol. 173 (3), 530-540 (2011).
- Rinner, O., et al. Identification of cross-linked peptides from large sequence databases. Nat Methods. 5 (4), 315-318 (2008).
- Yang, B., et al. Identification of cross-linked peptides from complex samples. Nat Methods. 9 (9), 904-906 (2012).
- Xu, H., Hsu, P. H., Zhang, L., Tsai, M. D., Freitas, M. A. Database search algorithm for identification of intact cross-links in proteins and peptides using tandem mass spectrometry. J Proteome Res. 9 (7), 3384-3393 (2010).
- Leitner, A., Faini, M., Stengel, F., Aebersold, R. Crosslinking and Mass Spectrometry: An Integrated Technology to Understand the Structure and Function of Molecular Machines. Trends Biochem Sci. 41 (1), 20-32 (2016).
- Heck, A. J. Native mass spectrometry: a bridge between interactomics and structural biology. Nat Methods. 5 (11), 927-933 (2008).
- Sharon, M., Robinson, C. V. The role of mass spectrometry in structure elucidation of dynamic protein complexes. Annu Rev Biochem. 76, 167-193 (2007).
- Goth, M., Pagel, K. Ion mobility-mass spectrometry as a tool to investigate protein-ligand interactions. Anal Bioanal Chem. , (2017).
- Uetrecht, C., Rose, R. J., van Duijn, E., Lorenzen, K., Heck, A. J. Ion mobility mass spectrometry of proteins and protein assemblies. Chem Soc Rev. 39 (5), 1633-1655 (2010).
- Hernandez, H., Robinson, C. V. Determining the stoichiometry and interactions of macromolecular assemblies from mass spectrometry. Nat Protoc. 2 (3), 715-726 (2007).
- Sobott, F., Hernandez, H., McCammon, M. G., Tito, M. A., Robinson, C. V. A tandem mass spectrometer for improved transmission and analysis of large macromolecular assemblies. Anal Chem. 74 (6), 1402-1407 (2002).
- Rostom, A. A., et al. Detection and selective dissociation of intact ribosomes in a mass spectrometer. Proc Natl Acad Sci U S A. 97 (10), 5185-5190 (2000).
- Zhou, M., et al. Mass spectrometry of intact V-type ATPases reveals bound lipids and the effects of nucleotide binding. Science. 334 (6054), 380-385 (2011).
- Uetrecht, C., et al. High-resolution mass spectrometry of viral assemblies: molecular composition and stability of dimorphic hepatitis B virus capsids. Proc Natl Acad Sci U S A. 105 (27), 9216-9220 (2008).
- Morgner, N., et al. Hsp70 forms antiparallel dimers stabilized by post-translational modifications to position clients for transfer to Hsp90. Cell Rep. 11 (5), 759-769 (2015).
- Schmidt, C., Beilsten-Edmands, V., Robinson, C. V. The joining of the Hsp90 and Hsp70 chaperone cycles yields transient interactions and stable intermediates: insights from mass spectrometry. Oncotarget. 6 (21), 18276-18281 (2015).
- Schmidt, C., Beilsten-Edmands, V., Mohammed, S., Robinson, C. V. Acetylation and phosphorylation control both local and global stability of the chloroplast F1 ATP synthase. Sci Rep. 7, 44068 (2017).
- Schmidt, C., et al. Comparative cross-linking and mass spectrometry of an intact F-type ATPase suggest a role for phosphorylation. Nat Commun. 4, 1985 (2013).
- Schmidt, C., Robinson, C. V. A comparative cross-linking strategy to probe conformational changes in protein complexes. Nat Protoc. 9 (9), 2224-2236 (2014).
- Schmidt, C., et al. Surface Accessibility and Dynamics of Macromolecular Assemblies Probed by Covalent Labeling Mass Spectrometry and Integrative Modeling. Anal Chem. 89 (3), 1459-1468 (2017).
- Lakomek, K., Stoehr, G., Tosi, A., Schmailzl, M., Hopfner, K. P. Structural basis for dodecameric assembly states and conformational plasticity of the full-length AAA+ ATPases Rvb1 · Rvb2. Structure. 23 (3), 483-495 (2015).
- Mareya, S. M., Raushel, F. M. A molecular wedge for triggering the amidotransferase activity of carbamoyl phosphate synthetase. Biochemistry. 33 (10), 2945-2950 (1994).
- Shevchenko, A., Wilm, M., Vorm, O., Mann, M. Mass spectrometric sequencing of proteins silver-stained polyacrylamide gels. Anal Chem. 68 (5), 850-858 (1996).
- Olsen, J. V., et al. Parts per million mass accuracy on an Orbitrap mass spectrometer via lock mass injection into a C-trap. Mol Cell Proteomics. 4 (12), 2010-2021 (2005).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol. 26 (12), 1367-1372 (2008).
- Kirshenbaum, N., Michaelevski, I., Sharon, M. Analyzing large protein complexes by structural mass spectrometry. J Vis Exp. (40), (2010).
- Morgner, N., Robinson, C. V. Massign: an assignment strategy for maximizing information from the mass spectra of heterogeneous protein assemblies. Anal Chem. 84 (6), 2939-2948 (2012).
- Marty, M. T., et al. Bayesian deconvolution of mass and ion mobility spectra: from binary interactions to polydisperse ensembles. Anal Chem. 87 (8), 4370-4376 (2015).
- Thoden, J. B., Wesenberg, G., Raushel, F. M., Holden, H. M. Carbamoyl phosphate synthetase: closure of the B-domain as a result of nucleotide binding. Biochemistry. 38 (8), 2347-2357 (1999).
- Rose, R. J., Damoc, E., Denisov, E., Makarov, A., Heck, A. J. High-sensitivity Orbitrap mass analysis of intact macromolecular assemblies. Nat Methods. 9 (11), 1084-1086 (2012).
- Gault, J., et al. High-resolution mass spectrometry of small molecules bound to membrane proteins. Nat Methods. 13 (4), 333-336 (2016).
- Mehmood, S., et al. Mass spectrometry captures off-target drug binding and provides mechanistic insights into the human metalloprotease ZMPSTE24. Nat Chem. 8 (12), 1152-1158 (2016).
- Kaake, R. M., et al. A new in vivo cross-linking mass spectrometry platform to define protein-protein interactions in living cells. Mol Cell Proteomics. 13 (12), 3533-3543 (2014).
- Liu, F., Rijkers, D. T., Post, H., Heck, A. J. Proteome-wide profiling of protein assemblies by cross-linking mass spectrometry. Nat Methods. 12 (12), 1179-1184 (2015).
- Walzthoeni, T., et al. False discovery rate estimation for cross-linked peptides identified by mass spectrometry. Nat Methods. 9 (9), 901-903 (2012).
- Barrera, N. P., Di Bartolo, N., Booth, P. J., Robinson, C. V. Micelles protect membrane complexes from solution to vacuum. Science. 321 (5886), 243-246 (2008).
- Barrera, N. P., et al. Mass spectrometry of membrane transporters reveals subunit stoichiometry and interactions. Nat Methods. 6 (8), 585-587 (2009).
- Laganowsky, A., et al. Membrane proteins bind lipids selectively to modulate their structure and function. Nature. 510 (7503), 172-175 (2014).
- Marcoux, J., et al. Mass spectrometry reveals synergistic effects of nucleotides, lipids, and drugs binding to a multidrug resistance efflux pump. Proc Natl Acad Sci U S A. 110 (24), 9704-9709 (2013).
- Politis, A., Schmidt, C. Structural characterisation of medically relevant protein assemblies by integrating mass spectrometry with computational modelling. J Proteomics. , (2017).
- Hall, Z., Politis, A., Robinson, C. V. Structural modeling of heteromeric protein complexes from disassembly pathways and ion mobility-mass spectrometry. Structure. 20 (9), 1596-1609 (2012).
- Pukala, T. L., et al. Subunit architecture of multiprotein assemblies determined using restraints from gas-phase measurements. Structure. 17 (9), 1235-1243 (2009).
- Politis, A., et al. Topological models of heteromeric protein assemblies from mass spectrometry: application to the yeast eIF3:eIF5 complex. Chem Biol. 22 (1), 117-128 (2015).
- Politis, A., et al. A mass spectrometry-based hybrid method for structural modeling of protein complexes. Nat Methods. 11 (4), 403-406 (2014).
- Fischer, L., Chen, Z. A., Rappsilber, J. Quantitative cross-linking/mass spectrometry using isotope-labelled cross-linkers. J Proteomics. 88, 120-128 (2013).
- Yu, C., et al. Developing a Multiplexed Quantitative Cross-Linking Mass Spectrometry Platform for Comparative Structural Analysis of Protein Complexes. Anal Chem. 88 (20), 10301-10308 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır