Method Article
Combinant la réticulation chimique et spectrométrie de masse des Complexes de protéine intacte pour étudier l’Architecture des assemblées de plusieurs sous-unités protéiques
Dans cet article
Résumé
L’architecture des complexes protéiques est essentiel pour leur fonction. Combinant différentes techniques de spectrométrie de masse s’est avérée puissante pour étudier leur assemblage. Nous fournissons des protocoles pour la réticulation chimique et spectrométrie de masse native et montrer comment ces techniques complémentaires contribuer à élucider l’architecture des assemblées de plusieurs sous-unités protéiques.
Résumé
Les protéines interagissent avec leurs ligands aux assemblées de forme active et dynamique qui exercent diverses fonctions cellulaires. Il est donc fondamental pour la compréhension des processus cellulaires d’élucider ces interactions. Cependant, de nombreux complexes de protéine sont des assemblys dynamiques et ne sont pas accessibles par des techniques structurelles conventionnelles. Spectrométrie de masse contribue à l’enquête structurelle de ces assemblées, et particulièrement la combinaison de différentes techniques de spectrométrie de masse fournit des renseignements précieux dans leur agencement structurel.
Dans cet article, nous décrivons l’application et la combinaison de deux techniques complémentaires de spectrométrie de masse, à savoir réticulation chimique couplée à la spectrométrie de masse et spectrométrie de masse native. Réticulation chimique implique la liaison covalente d’acides aminés à proximité en utilisant des réactifs chimiques. Après digestion par les protéases, réticulés di-peptides sont identifiés par spectrométrie de masse et sites des interactions entre protéines sont à découverts. Spectrométrie de masse native est en revanche l’analyse des ensembles de protéines intactes en phase gazeuse d’un spectromètre de masse. Il révèle les stoechiométries de protéines ainsi que des interactions protéiques et de ligand. Les deux techniques fournissent donc des informations complémentaires sur la structure des assemblages de protéine-ligand et leur combinaison s’est avérée puissante lors d’études antérieures.
Introduction
L’enquête structurelle des ensembles de protéines est devenu particulièrement important pour la compréhension des processus cellulaires. Par conséquent, beaucoup, des techniques ont été développés et améliorés en biologie structurale1. Toutefois, ces techniques sont parfois limitées dans leur application en raison de la taille, souplesse ou hétérogénéité des complexes protéine incriminés. Spectrométrie de masse peut faire face à ces défis et, par conséquent, a émergé comme un outil puissant en biologie structurale2,3,4,5. Le plus grand avantage de la spectrométrie de masse, cependant, est la capacité d’identifier sans ambiguïté les protéines même dans des mélanges complexes et hétérogènes,6. À cette fin, les protéines sont digérées habituellement avec les endoprotéinases et le mélange de peptides qui en résulte est séparé par chromatographie liquide et élué directement dans le spectromètre de masse. Masses de peptide sont déterminés par la suite et les ions précurseurs sont choisies pour plus de fragmentation des peptides. Protéines sont identifiées puis par la recherche de peptide et masses correspondantes de fragment contre une base de données connue. Cette procédure permet non seulement l’identification des peptides/protéines, mais aussi leurs modifications post-traductionnelles qui provoquent un déplacement de masse des ions fragments transportant la modification7et les peptides de précurseur. Certaines des nombreuses techniques en spectrométrie de masse structurelle sont basés sur ce principe4,8. Par exemple, étiquetage des techniques telles que l’hydrogène deutérium échange9,10, chimique étiquetage stratégies11,12, ou le radical hydroxyle pied impression13,14 , donner des aperçus de l’accessibilité de surface des protéines sous certaines conditions.
Une autre technique est la réticulation impliquant la liaison covalente d’acides aminés à proximité par le biais de leurs groupes fonctionnels (chimique). Pour ce faire, des réactifs chimiques, réactifs UV-activable ou acides aminés sont indépendants15,16. Après réticulation, les protéines sont hydrolysées habituellement avec des protéases et réticulés di-peptides sont analysés par spectrométrie de masse couplée à la chromatographie en phase liquide. L’identification des produits réticulation par base de données, la recherche, cependant, nécessite l’utilisation d’un logiciel spécialisé qui concatènent des séquences peptidiques de diverses protéines et régions disparates17,18,19. L’utilisation d’agents chimiques a l’avantage qu’elle peut être utilisée pour presque chaque complexe de protéine d’intérêt et ne nécessite pas l’incorporation des acides aminés activable par UV, qui ne peut être atteint en exprimant la protéine d’intérêt dans les cellules hôtes. Par conséquent, la réticulation est un outil polyvalent et a été utilisée avec succès dans de nombreuses études structurales des protéines même de grandes assemblées20.
Spectrométrie de masse des complexes de protéine intacte (parfois appelé « indigène » spectrométrie de masse), d’autre part, implique l’analyse des protéines intactes et des complexes de protéine sans hydrolyse en peptides. Il révèle la composition, l’hétérogénéité, stoechiométrie, topologie et les interactions de sous-unité de protéines complexes21,22. En combinaison avec mobilité ionique, spectrométrie de masse native plus permet la détermination de leur conformation23,24. Ce qui rend un outil puissant pour l’enquête structurelle des complexes protéiques qui sont difficiles à évaluer par des techniques structurelles conventionnelles. Spectrométrie de masse native exige toutefois des tampons d’analyse qui tiennent des interactions non-covalentes de protéines au cours de l’ionisation par électronébulisation. Ceci est généralement obtenu en utilisant des tampons aqueux, volatils tels que de l’acétate d’ammonium25. En outre, les modifications de l’instrument sont nécessaires pour prévenir la dissociation lors de la transmission dans la phase gazeuse du spectromètre de masse à26. Appliqué de cette façon, de nombreux complexes de protéine (grand) ont été analysés. Exemples impressionnants sont les études de ribosomes intact27, ATP synthases28ou virus29.
La combinaison de la spectrométrie de masse native et réticulation s’est avérée particulièrement efficace dans des études antérieures. Par exemple, les stoechiométries de complexes de chaperon, y compris un dimère Hsp70 inattendu, pourraient être obtenues à partir des expériences de spectrométrie de masse des complexes des protéines intactes, pendant réticulation chimique a révélé les arrangements des protéines dans le assemblys30,31. Dans une autre étude, nous avons étudié les effets des modifications post-traductionnelles sur une ATP synthase intact en complexe. Spectrométrie de masse native a fourni des renseignements sur la stabilité des protéines complexes en présence ou en absence de phosphorylation ou l’acétylation32,33. Un comparatif de stratégie réticulation34 révèle alors des changements de conformation de la protéine complexe dans les différentes conditions.
Ici, nous fournissons les protocoles pour l’identification des protéines par spectrométrie de masse, réticulation (chimique) et spectrométrie de masse indigène, y compris l’analyse des données et définitions (Figure 1). La combinaison complémentaire résultats tirés de ces méthodes à l’aide de deux complexes de protéines bien caractérisé est démontrée35. Notre protocole peut être appliqué à tout assemblage de protéines qui peut être épuré à certaine pureté et la concentration. L’approche est parfois limité par l’analyse des données de réticulation des données, c'est-à-direla taille de la base de données Employee, qui détermine le temps et l’espace de recherche requis. En outre, la validation manuelle des réticulations identifiées est souvent nécessaire et réduit encore davantage la sortie. Spectrométrie de masse native est surtout limitée par la qualité de l’échantillon, par exemple, tampons et adduits utilisé au cours de la purification et la possibilité des pour échanger par tampons aqueux et volatiles. Toutefois, des complexes de protéine purifiées pour l’analyse structurale habituellement ont la qualité requise pour l’analyse réussie avec nos protocoles.
Protocole
1. purification de Complexes protéiques
- Préparer la protéine complexe conformément aux protocoles standards optimisés.
Remarque : Le protocole est démontré ici avec le complexe RvB1/B2 de Chaetomium thermophilum et le carbamyl phosphate synthétase (CPS) d’Escherichia coli. RvB1/B2 et CPS ont été purifiés comme décrit36,37. Chaque protéine complexe nécessite un protocole de purification individuelle. Ajuster le protocole en conséquence. Amines libres tampons comme solution saline tamponnée au phosphate (PBS) ou l’acide 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic (HEPES) doivent être utilisés pour la réticulation chimique. Remplacer le tampon durant la purification si possible.
ATTENTION : Ne pas appliquer les méthodes ou les réactifs qui perturbent l’Assemblée native de la protéine complexe.
2. Identification des protéines masse spectrométrie-basé
-
Électrophorèse sur gel
Remarque : Il y a gel de différents systèmes disponibles et chaque laboratoire utilise son propre paramétrage. Ajuster les conditions selon le système de gel. Porter des gants et blouse de laboratoire tout au long du protocole comme kératines sont parmi les contaminants les plus courants d’analyse par spectrométrie de masse.- Appliquer 7,5 µL de tampon d’échantillon x 4 et 3 µL 10 x agent réducteur (concentration finale 50 mM le dithiothréitol (DTT)) à 20 µL d’échantillon protéique. Utiliser 10 µM CPS ou 2,5 µM RvB1/B2. Tournez vers le bas pendant 1 min à 16 200 × g et chauffer pendant 10 min à 70 ° C.
Remarque : En utilisant un système différent de gel quand ajuster les volumes requis et la température de chauffage. - Préparer un gel dégradé de 4 à 12 % d’électrophorèse. Diluer le tampon en cours d’exécution 20 fois avec de l’eau et remplir la chambre d’électrophorèse. Utilisation 2-(N-morpholino) ethanesulfonic acide (MES) tampon pour les protéines de poids moléculaire plus faible (2-200 kDa) et 3 - sulfonique (N-morpholino) (MOPS) tampon pour les protéines de la plus grandes (14-200 kDa). Ajouter antioxydant de 0,5 mL à la chambre intérieure.
Remarque : En utilisant un système différent de gel quand préparer tampon en cours d’exécution et le gel selon les protocoles du fabricant. - Charger un marqueur protéique adapté (préalablement teintés, colorées ou non, différentes tailles moléculaires) dans la première cavité et charger les échantillons de protéine dans les cavités restantes.
- Protéines séparées pendant 35 min (MES) ou 50 min (MOPS) à 200 V.
Remarque : Réglez le temps d’électrophorèse et la tension pour gel de différents systèmes. - Pour souiller les bandes de protéines, transférer le gel dans un gel boîte de coloration et recouvrir le gel avec une solution de coloration de Coomassie à base d’eau. Incuber pendant la nuit et à la température ambiante sur un agitateur de gel horizontal.
- Décolorer le gel en remplaçant la solution colorante avec de l’eau. Répétez cette opération plusieurs fois (environ 3 - 5 fois) jusqu'à ce que le fond de gel apparaît clairement.
- Appliquer 7,5 µL de tampon d’échantillon x 4 et 3 µL 10 x agent réducteur (concentration finale 50 mM le dithiothréitol (DTT)) à 20 µL d’échantillon protéique. Utiliser 10 µM CPS ou 2,5 µM RvB1/B2. Tournez vers le bas pendant 1 min à 16 200 × g et chauffer pendant 10 min à 70 ° C.
-
Digestion en gel
Remarque : Pour éviter toute contamination, solvants HPLC grade dans tout le protocole de la digestion et pour toutes les étapes suivantes (par exemple, l’analyse par spectrométrie de masse). Préparer toutes les solutions avant utilisation et de filtrat de l’eau et de bicarbonate d’ammonium.- Couper les bandes de protéines qui sont visualisées par le bleu de Coomassie tachent provenant du gel à l’aide d’un scalpel. Découpez soigneusement les bandes de protéines en petits morceaux d’environ 1 mm × 1 mm. Rincer le scalpel avec de l’eau entre les bandes de protéines différentes. Laver les bandes de gel à l’eau et l’acétonitrile (ACN).
- Réduire les disulfures avec TNT, alkylat résidus cystéine avec l’iodoacétamide et digérer les protéines avec la trypsine décrite précédemment38; un rapport de l’enzyme : protéine de 01:20 jusqu’au 1/100 est généralement utilisé. Utiliser bicarbonate d’ammonium 100 mM durant le protocole de la digestion.
- Extrait de peptides en deux étapes.
- Tout d’abord Incuber les morceaux de gel avec bicarbonate d’ammonium et d’ACN et recueillir le surnageant contenant du peptide. Deuxièmement, incuber les morceaux de gel avec 5 % (v/v) d’acide formique et ACN. Incuber pendant 15 min à chaque étape. Combiner les deux surnageants. Sécher les peptides extraits par évaporation des solvants dans une centrifugeuse sous vide.
Remarque : Peptides séchées peuvent être stockés à-20 ° C pendant plusieurs mois.
- Tout d’abord Incuber les morceaux de gel avec bicarbonate d’ammonium et d’ACN et recueillir le surnageant contenant du peptide. Deuxièmement, incuber les morceaux de gel avec 5 % (v/v) d’acide formique et ACN. Incuber pendant 15 min à chaque étape. Combiner les deux surnageants. Sécher les peptides extraits par évaporation des solvants dans une centrifugeuse sous vide.
-
Spectrométrie de masse couplée à la chromatographie en phase liquide
- Dissoudre les peptides séchées dans 2 % ACN/0.1% d’acide formique. Dissoudre les peptides dans un bain de sonication pour 2-3 min et un essorage vers le bas dans une centrifugeuse à 16 200 x g pendant 30 min. transfert des échantillons en flacons pour échantillonneur automatique.
Remarque : Réglez le volume selon la quantité de protéines. Pour des bandes de bleu de Coomassie bien colorées, nous utilisons 20 µL. - Injecter 5 µL de l’échantillon dans le système de nano-LC-MS/MS à l’aide de l’échantillonneur automatique. Charger le mélange de peptides sur une phase inverse C18 la précolonne (C18, 150 µm I.D., 2 cm, taille de pore de 5µm) de dessalement et de concentrer les peptides en ligne.
- Utiliser l’acide formique 0,1 % (v/v) comme phase mobile A et 80 % ACN/0.1% (v/v) d’acide formique mobile phase B. séparer les peptides sur une colonne analytique C18 à phase inverse (C18, 75 µm I.D., 50 cm, taille de pore de 3 µm) à l’aide d’un dégradé de 4 à 80 % de B (contenant de l’acide formique 0,1 %) à raison de 300 nL / min pendant 65 min.
Remarque : Une source d’ions de tuyères est utilisée pour transférer des peptides éluées dans le spectromètre de masse. - Utiliser des conditions de MS (typiques) : tension de 1,6 de pulvérisation kV ; capillaire température de 250 ° C ; énergie de collision normalisé de 30. Exploiter le spectromètre de masse en mode dépendant des données.
- Acquérir les spectres MS dans l’analyseur de masse (p. ex., Orbitrap valant) (m/z 350−1, 600) avec une résolution de 70 000 et un objectif de contrôle de gain automatique de 3 x 106. Sélectionnez 20 des ions plus intenses pour la fragmentation HCD sur une cible de contrôle automatique de gain de 1 x 10-5. Dynamiquement exclure précédemment sélectionné des ions pour 30 Exclude s. facturés individuellement des ions ainsi que des ions avec l’état de charge non reconnu.
NOTE : Étalonnage interne des spectres de masse a été réalisée à l’aide du verrou option masse39.
- Dissoudre les peptides séchées dans 2 % ACN/0.1% d’acide formique. Dissoudre les peptides dans un bain de sonication pour 2-3 min et un essorage vers le bas dans une centrifugeuse à 16 200 x g pendant 30 min. transfert des échantillons en flacons pour échantillonneur automatique.
-
La recherche de base de données
Remarque : Il existe différents logiciels pour la recherche de base de données. MaxQuant40, par exemple, est librement disponible.- Convertir les fichiers .raw en .mgf-fichiers à l’aide d’un outil de conversion de pXtract (http://pfind.ict.ac.cn/downloads.html).
- Effectuer la recherche de base de données à l’aide de paramètres de recherche typique : base de données, swissprot ; tolérance masse de peptide, 10 ppm ; tolérance de masse fragment, Da 0,5 ; enzyme, la trypsine ; sites de clivage manquée, 2 ; modifications de variables, carbamidomethylation (cystéine) et l’oxydation (méthionine).
Remarque : Modifier les paramètres de recherche selon les paramètres expérimentaux. - Examinez le résultat de la recherche de base de données. Évaluer le score de protéine, nombre de peptides identifiés, scores de peptide et masse exactitude ; la couverture de séquence renvoie la couverture de la protéine identifiée.
Remarque : Chaque moteur de recherche possède son algorithme de notation individuelle. Évaluer le peptide système de notation de la qualité des spectres de masse en tandem observé. Les principaux signaux d’un spectre de bon devraient montrer série ion (complet) pour vérifier le peptide identifié. Le score de peptide est généralement fondée sur les probabilités, c'est-à-dire, le score de peptide est une mesure pour quelle est la probabilité correspond à la séquence peptidique identifié le spectre obtenu. Le score de protéine est généralement dérivé de scores de peptide et est utilisé pour classer une protéine dans une liste de protéines identifiées.
3. la microanalyse des Complexes des protéines intactes (spectrométrie de masse Native)
- Préparation des émetteurs or-enduit pour l’ionisation par électronébulisation
- Utiliser un micropipettes pour préparer nanoflow émetteurs de capillaires en verre comme décrit plus haut25,41. Utilisez les capillaires borosilicaté avec 0,78 mm de diamètre intérieur.
NOTE : En changeant les paramètres pour tirer l’aiguille, la pointe forme et la taille peuvent être modifiées et ajustés pour l’échantillon. Capillaires avec différent intérieur diamètre et épaisseur de paroi sont disponibles. - Enrober les capillaires en verre avec un matériau conducteur (p. ex., or ou palladium) ; l’utilisation d’une coucheuse par pulvérisation cathodique générer un plasma or est fréquente. Suivez les instructions du fabricant pour obtenir des revêtement de bonne qualité.
Remarque : Le revêtement devrait être suffisant pour obtenir une pulvérisation stable lors de l’application de tensions capillaires commun (voir ci-dessous).
- Utiliser un micropipettes pour préparer nanoflow émetteurs de capillaires en verre comme décrit plus haut25,41. Utilisez les capillaires borosilicaté avec 0,78 mm de diamètre intérieur.
- Préparation des échantillons pour la spectrométrie de masse native
NOTE : Sels, détergents ou grandes quantités de glycérol ne sont pas compatibles avec ionisation par électronébulisation. Par conséquent, le tampon de purification est échangé par un tampon volatil, aqueux. acétate d’ammonium 200 mM est couramment utilisé. Utilisez les colonnes de taille exclusion spin ou dispositifs d’ultrafiltration pour l’échange de la mémoire tampon. Dans certains cas, stabilité complexe ou activité pourrait être affectée par échange de la mémoire tampon. Évaluer les spectres de masse soigneusement et vérifier l’activité du complexe. Ajouter des cofacteurs ou additifs vers le tampon de l’analyse, si nécessaire.- Utilisez les colonnes à centrifuger exclusion taille pour échange tampon rapide. Retirer le tampon de stockage par centrifugation à 1 000 x g et 4 ° C pendant 1 min. jeter le débit à travers. Laver trois fois en ajoutant 500 µL 200 mM d’acétate d’ammonium, suivie par centrifugation. Charger 20 µL de l’échantillon de protéine sur la colonne et centrifuger à 1 000 x g et 4 ° C pendant 4 min.
Remarque : La concentration de la protéine complexe doit être de 1 à 10 µM. La procédure peut être répétée si les composants non volatils troubler encore l’analyse. - Pour concentrer l’échantillon de protéines et d’échanger le tampon de la même expérience utilisez les filtres centrifuges. Utiliser une membrane de filtration de pore taille 50 % inférieure à la taille des protéines analysées.
- Transférer l’échantillon de protéine dans le dispositif de filtration et ajouter 200 mM d’acétate d’ammonium. Tournez en bas à 15 000 x g et jeter le débit à travers. Ajouter 200 mM d’acétate d’ammonium et répétez la centrifugation. Répétez cette opération plusieurs fois. Suivez les instructions du fabricant pour la vitesse de centrifugation. Effectuer la centrifugation à 4 ° C.
Mise en garde : Protéines membranaires ont tendance à précipiter sur la membrane de l’appareil de filtrage.
- Transférer l’échantillon de protéine dans le dispositif de filtration et ajouter 200 mM d’acétate d’ammonium. Tournez en bas à 15 000 x g et jeter le débit à travers. Ajouter 200 mM d’acétate d’ammonium et répétez la centrifugation. Répétez cette opération plusieurs fois. Suivez les instructions du fabricant pour la vitesse de centrifugation. Effectuer la centrifugation à 4 ° C.
- Utilisez les colonnes à centrifuger exclusion taille pour échange tampon rapide. Retirer le tampon de stockage par centrifugation à 1 000 x g et 4 ° C pendant 1 min. jeter le débit à travers. Laver trois fois en ajoutant 500 µL 200 mM d’acétate d’ammonium, suivie par centrifugation. Charger 20 µL de l’échantillon de protéine sur la colonne et centrifuger à 1 000 x g et 4 ° C pendant 4 min.
- Analyse par spectrométrie de masse des complexes de protéine intacte
Remarque : Il y a différents types de spectromètres de masse provenant de fabricants différents qui peuvent être modifiés par spectrométrie de masse native, par exemple, des spectromètres de masse quadripolaire time-of-flight (Q-ToF) ou Orbitrap valant spectromètres de masse. Le protocole décrit ci-dessous a été réalisé sur un instrument Q-ToF.- Mettre un revêtement or capillaire dans le support capillaire et remplir le tube capillaire avec 1-4 µL de l’échantillon de protéine. Ouvrir la pointe de l’aiguille avec une pince.
- Connectez le support capillaire avec la source de nano-electrospray et ajustez la position de capillaire. Positionnez l’extrémité du capillaire à 0,5 à 1,5 cm de l’orifice du cône. Gaz nanoflow de 80 à 150 L/h permet de lancer le spray et régler le débit de gaz pour maintenir une pulvérisation stable.
- Ajustez les paramètres dans la page tune de l’instrument de Q-ToF. Les conditions de départ typiques sont : tension capillaire, 1.50 kV ; tension de cône, 80 V ; Énergie de RF objectif 1, 80 V ; énergie de collision, 20 V ; Aperture 3, 13,6 V. modifier ces paramètres afin d’obtenir des spectres de masse bons. Commencer l’acquisition en cliquant sur le bouton « acquérir » et combiner les scans autant que possible pour obtenir un bon spectre de masse.
Remarque : Nous vous recommandons d’associer au moins 100 pages.
- Spectrométrie de masse des complexes de protéine intacte
- Acquérir le spectre de masse comme décrit ci-dessus (protocole section 3.3). Choisissez un ion précurseur d’un complexe protéique.
- Changement de MS en mode MS/MS dans le dossier d’acquisition. Définir la sélection MS/MS le précurseur de la masse.
- Démarrer l’acquisition à l’énergie de collision faible. Combiner plusieurs balayages (environ 20 analyses) pour vérifier la sélection correcte du précurseur de la masse. Augmenter l’énergie de collision jusqu'à ce que la protéine complexe se dissocie. Pour obtenir un spectre de masse bien combiner au moins 500 numérisations.
NOTE : Complexes dénudées ont parfois de faible intensité. Combinant des scans autant que possible pourrait augmenter le rapport signal-bruit et de la résolution.
- Solution dissociation des complexes de protéine intacte
Remarque : Pour obtenir des indications supplémentaires sur les interactions de protéine dans les complexes de protéine, dissociation en solution peut être effectuée.- Préparation des échantillons de protéines tel que décrit ci-dessus (section 3.2). Ajouter des solvants à l’échantillon de protéines ou changer le pH pour dissocier les complexes intacts dans des complexes. Les solvants typiques sont le méthanol, alcool isopropylique et ACN ; changer le pH de l’acétate d’ammonium par l’addition d’acide acétique ou d’ammoniac en solution aqueuse. L’acquisition de spectres de masse comme décrit ci-dessus (Articles 3.3 et 3.4).
- Varier la quantité de solvant ou de la plage de pH à générer des complexes sous divers. En règle générale, la quantité de solvant est de 5 à 50 % (concentration finale) et une gamme de pH typique est de 4-9. Commencer avec une faible concentration de solvant (5 %) ou de légers changements du pH et d’acquérir un spectre de masse (voir section 3.3).
- Augmenter la quantité de solvant ou de changer le pH par étapes jusqu'à ce que Sub complexes sont produites en solution. Acquérir des spectres de masse pour analyser des complexes.
- Calibrer les données
Remarque : Les spectres de masse acquises sont étalonnés externe à l’aide de solution d’iodure (CsI) de césium.- Dissoudre 100 mg CsI dans 1 mL d’eau.
- Acquérir un spectre de masse de CsI. Varier l’énergie de collision pour obtenir des grappes de la CsI au cours de la même gamme de m/z que le complexe protéique analysées ci-dessus.
ATTENTION : CsI rapidement précipités à la pointe de l’émetteur et contamine le cône. Acquérir seulement autant scanne que nécessaire pour obtenir un spectre suffisant. Supprimer l’émetteur de la source lorsque vous avez terminé. - Rendre un fichier de calibration en utilisant le spectre de masse acquis et un fichier de référence de CsI.
- Appliquer calibrage de spectres de masse acquises.
ATTENTION : Le calibrage des spectres de masse peut être un changement permanent des données brutes. Si non calibrées spectres sont nécessaire, effectuez une copie de sauvegarde du fichier.
- Traitement des données et analyse
Remarque : Il y a beaucoup d’outils logiciels disponibles gratuitement pour l’analyse de données de spectres de masse des indigènes ; par exemple, Massign42 ou UniDec43. Le protocole ci-dessous décrit l’analyse manuelle des données avec l’aide du logiciel de l’instrument ainsi que l’utilisation de Massign pour des échantillons complexes. Ce logiciel est bien adapté pour l’analyse des spectres de masse complexes. Suivez les instructions en ligne pour l’utilisation du programme (http://massign.chem.ox.ac.uk/).- Pour l’analyse des données, lisse les spectres en ajustant les paramètres de lissage. Spectres de centre de gravité en ajustant les paramètres. Calculer les masses complexes de deux pics adjacents de la protéine complexe ' enveloppe de crête à l’aide d’outils logiciels instrument.
ATTENTION : Un lissage trop intensive peut provoquer des pertes de données (p. ex., perte de ligands liés). - Pour l’analyse Massign42, générer une liste de pointe pour le spectre de masse. Linéariser les points de données du spectre et lisse. Utiliser les divers outils logiciels pour assigner des complexes protéiques, composition complexe de calculer ou simuler des enveloppes pic complexe.
- Pour l’analyse des données, lisse les spectres en ajustant les paramètres de lissage. Spectres de centre de gravité en ajustant les paramètres. Calculer les masses complexes de deux pics adjacents de la protéine complexe ' enveloppe de crête à l’aide d’outils logiciels instrument.
4. chimique réticulation couplé à la spectrométrie de masse
Remarque : Il existe plusieurs stratégies de réticulation. Nous décrivons ici l’utilisation du Bis (sulfosuccinimidyl) subérate (BS3), un réticulant amine-réactif qui est couramment utilisé pour l’étude des interactions protéine-protéine.
- Dissoudre 1,43 mg BS3 dans 100 µL d’eau pour préparer une solution stock de 25 mM.
Remarque : Les autres réactifs comme disuccinimidyl subérate (DSS) ne sont pas solubles dans l’eau et sont habituellement dissous dans le diméthylsulfoxyde (DMSO). Certains agents sont également disponibles dans des formes lourdes isotopiquement étiquetés. Incorporation d’isotopes stables lourds génère des paires de pic de réticulé di-peptides dans les spectres MS, qui aide au cours de l’évaluation des données. Lorsqu’à l’aide de façon différentielle labellisé ultraviolets, les solutions mères de ces deux variantes sont préparées et mixte 1:1.
ATTENTION : Pour éviter la dissociation des complexes protéiques de DMSO, une solution très concentrée est préparée et diluée avec de l’eau ou de la mémoire tampon avant la réaction de réticulation. - BS3 s’ajoute le complexe protéique. Utiliser des quantités variables de BS3 allant de 0,5 à 5 mM pour identifier la concentration optimale de réticulant. Incuber les mélanges réactionnels à 25 ° C pendant 1 h dans un thermomixer. Utiliser des gels dégradé de 4 à 12 % et exécuter SDS-PAGE, afin d’évaluer les résultats de réticulation (voir la Figure 2 pour obtenir un exemple).
NOTE : Une concentration optimale de BS3 est atteint lorsque les bandes de protéines de poids moléculaire plus élevés, qui ne sont pas visibles dans le contrôle non réticulé, sont obtenus au cours de la SDS-PAGE, tandis que les sous-unités unique sont encore visibles (Figure 2). - Répétez la réaction croisée avec la concentration optimale de BS3. Incuber les mélanges réactionnels à 25 ° C pendant 1 h dans un thermomixer.
Remarque : Certains complexes de protéines ne sont pas stables à température ambiante. La réaction de réticulation peut également être effectuée sur la glace ; Cependant, le temps de réaction doit être ajustée. -
Étancher la réticulation réaction en ajoutant tampon amine (p. ex., 50-100 mM tampon Tris, pH 7.5, concentration finale) ou effectuer la précipitation d’éthanol pour supprimer tout réactif résiduel de réticulation.
- Ajouter de l’eau ou le tampon au milieu réactionnel pour atteindre un volume final de 200 µL. Ajouter 600 µL glacee éthanol et 20 µL 3M l’acétate de sodium, pH 5,3. Bien mélanger et laisser incuber à-20 ° C pendant 2 h ou toute une nuit.
Remarque : Par ailleurs, les protéines peuvent être précipités à 80 ° C pendant 30 min ou dans l’azote liquide. - Tournez en bas à 16 200 x g et 4 ° C pendant 30 min. Retirer délicatement le surnageant.
- Laver le culot avec 1 mL d’éthanol à 80 % (v/v) glacée. Tournez en bas à 16 200 x g et 4 ° C pendant 30 min. Retirer délicatement le surnageant. Sécher le culot dans une centrifugeuse sous vide.
- Ajouter de l’eau ou le tampon au milieu réactionnel pour atteindre un volume final de 200 µL. Ajouter 600 µL glacee éthanol et 20 µL 3M l’acétate de sodium, pH 5,3. Bien mélanger et laisser incuber à-20 ° C pendant 2 h ou toute une nuit.
- Effectuer le SDS-PAGE des protéines réticulés. Couper les bandes de gel et de digérer le protéines en gel comme décrit plus haut (sections 2.1 et 2.2).
Remarque : La digestion en solution peut également être effectuée, cependant, elle nécessite habituellement des étapes de séparation supplémentaire (p. ex., chromatographie d’exclusion des peptides digérés). -
Effectuer la spectrométrie de masse couplée à la chromatographie en phase liquide, tel que décrit ci-dessus (section 2.3). Réticulé peptides sont généralement peu abondants, appliquez les modifications suivantes à l’analyse par spectrométrie de masse pour augmenter la profondeur analytique de l’échantillon réticulé.
- Utiliser des dégradés plus longtemps pendant la séparation par chromatographie liquide (par exemple, 90 min au lieu de 65 min, voir ci-dessus).
- Exclure les peptides doublement chargés de fragmentation HCD.
NOTE : Peptides doublement chargés sont habituellement intra-cross-linked peptides (« boucle peptides » ou « type 1 » des liens croisés). - Lorsque vous utilisez différentiellement étiquetés réactifs de réticulation, utilisez l’option « pic aléatoire » pendant l’analyse, c'est-à-dire, fragmentation HCD est déclenchée par la présence de paires de pic de la différence de masse définie dans les spectres de masse.
Remarque : Option de la cueillette « pic » n’est peut-être pas disponible sur chaque spectromètre de masse.
- Utilisation pLink logiciel18 pour l’identification des di-peptides réticulés. Utiliser des bases de données réduites pour l’identification. Paramètres de recherche typiques sont : instrument de spectres, HCD ; enzyme, la trypsine ; Max. sites de clivage manquée, 3 ; adaptations variables, oxydation (méthionine) et carbamidomethylation (cystéine) ; réticulant, BS3 ; longueur min. peptide, 4 ; Max. longueur de peptide, 100 ; min. masse de peptide, 400 Da ; Max. masse de peptide, 10 000 Da ; FDR, 1 %.
Remarque : Lorsqu’à l’aide de façon différentielle labellisé ultraviolets, l’augmentation massive causée par l’éditeur de liens doit être configuré. Il y a aussi d’autres logiciels couramment utilisés pour l’identification de réticulation, par exemple, xQuest17, MassMatrix19ou XlinkX15. - Évaluer les résultats de recherche de base de données de la qualité des spectres de fragmentation. Des spectres acceptables des réticulations devraient montrer série ion des deux peptides (ions adjacentes au moins 4) avec un rapport signal-bruit raisonnable.
Remarque : Lorsqu’à l’aide de façon différentielle labellisé réactifs de réticulation, paires de pointe dans les spectres MS utilisable comme un contrôle supplémentaire de la qualité. - Si nécessaire, visualiser les résultats de réticulation dans des réseaux d’interactions protéiques à l’aide d’outils logiciels (p. ex., XVis, XiNET). Utilisation bar placettes circulaires ou parcelles pour la visualisation des interactions entre protéines.
Remarque : Les deux logiciels sont disponibles gratuitement sur un serveur web. Suivre les instructions détaillées sur les sites respectifs (https://xvis.genzentrum.lmu.de/ et http://crosslinkviewer.org/).
Résultats
L’analyse structurale des protéines et les complexes qu’ils forment est fondamentale pour comprendre leur fonction. Spectrométrie de masse contribue considérablement à l’enquête structurelle qu’elle peut être appliquée à presque chaque complexe d’intérêt indépendamment de leur taille ou l’hétérogénéité de l’échantillon. Nous illustrent bien le protocole à l’aide de deux complexes de protéines bien caractérisée ; tout d’abord, l’hétéro-dodécamère RvB1/B2 de c. thermophilum et d’autre part, la CPS hétéro-et complexe d’e. coli.
Tout d’abord, nous avons identifié les constituants protéiques des deux complexes. Pour ce faire, les protéines ont été séparées par SDS-PAGE (Figure 2) et bandes de gel ont été taillés dans le gel. Après la digestion des protéines en gel, le mélange de peptides a été analysé par spectrométrie de masse couplée à la chromatographie en phase liquide et masses de peptide et de fragment ont été soumis à la recherche de base de données. Suite à ce flux de travail, nous avons identifié toutes les sous-unités protéiques des deux complexes avec le degré de confiance élevé, c'est-à-direun grand nombre de peptides avec des scores de peptide raisonnable a été observé ce qui donne une couverture pour toutes les sous-unités protéiques (tableau 1 séquence très ).
Ensuite, nous avons analysé le complexe B2/RvB1 intact par spectrométrie de masse native (Figure 3 a). Le spectre de masse a révélé deux espèces, une à environ 8 000 m/z et une autre espèce à environ 11 000-12 000 m/z. Les masses calculées pour ces espèces correspondent à la hexamériques (RvB1)3(RvB2)3 bague (environ 310 kDa) et le dodécamères double-anneau (RvB1)6(RvB2)6 (environ 620 kDa). Les deux séries de crête montrent deux populations ; ils proviennent d’un mélange de sous-unités de RvB2 son marqués et non marqués dans les complexes. Une structure en cristal pour le complexe RvB1/B2 a été précédemment obtenue36 et montre l’arrangement de la double-anneau (Figure 3 b). Le spectre de masse natif confirme donc la stoechiométrie de la dodécamère intact et révèle en outre un complexe stable sous. En outre, les populations coexistants sont identifiées.
Pour identifier les sites d’interaction de protéine dans le complexe RvB1/B2, nous réticulé chimiquement le complexe purifié avec réticulant BS3. Nous avons tout d’abord titré la quantité de BS3 lors de la réaction de réticulation pour déterminer la concentration optimale. BS3 est amine spécifique et par liaison covalente des chaînes latérales lysine liens ainsi que les extrémités N-terminales des protéines. La réaction croisée a été suivie par SDS-PAGE (Figure 2 a). Le complexe non réticulé a montré des sous-unités fois RvB1 et RvB2. Ajouter BS3 au milieu réactionnel causé une liaison covalente des protéines résultant dans les bandes de protéines à poids moléculaire plus élevé. Le gel SDS montre que des quantités croissantes de BS3 produisent des quantités plus élevées d’espèces réticulés tandis que les sous-unités protéiques non réticulé sont réduites. Nous avons ensuite couper les bandes de protéines du gel et suivi le protocole fourni ci-dessus pour identifier les sites d’interaction de protéine. Un spectre d’exemple d’un di-peptide réticulé apparaît (Figure 3). Le spectre montre la série y-ion des deux peptides confirmant cette interaction de la protéine. Au total, nous avons obtenu des interactions protéine 14, y compris quatre liaisons transversales entre les sous-unités RvB1 et RvB2 et deux liens croisés entre les deux exemplaires de RvB2 (tableau 2). Les résultats de BS3 réticulation sont visualisées dans un réseau d’interactions (Figure 3D) montrant des interactions intra moléculaires ainsi que des interactions entre les sous-unités différentes. Liaisons transversales intra moléculaires suggèrent que les sous-unités fois RvB1 et RvB2 plier de façon que les domaines N - et C-terminaux sont à proximité. Notez qu’interactions intra moléculaires ne peuvent être distinguées de leurs interactions de la sous-unité même dans ce cas. Leurs liens croisés entre les deux sous-unités ont aussi été observés. Parmi ceux-ci, deux liens inter moléculaires croisés entre RvB1 et RvB2 pourraient être visualisées dans la structure de valider l’approche de réticulation. Les autres liaisons transversales inter moléculaires sont situés dans des boucles souples qui ne figurent pas dans la structure cristalline. Nous avons aussi identifié deux liaisons transversales à RvB2 contenant les mêmes séquences peptidiques. Ces réticulations sans ambiguïté peuvent être classées comme inter moléculaires comme elles doivent provenir de deux copies de la même protéine (Figure 3D). Nos expériences de réticulation révèlent des sites d’interaction de protéine au sein du complexe, mais aussi dans les sous-unités de protéine fournit des aperçus de leur agencement structurel qui pourrait également être confirmée par la structure existante de cristal (Figure 3 b).
Le deuxième complexe de protéine que nous avons étudié était CPS. Le spectre de masse natif (Figure 4 a) a révélé trois complexes protéiques entre 6 000 et 12 000 m/z. Le plus grand complexe de 640 kDa correspond à l’hétéro-octamère intact. Les petits complexes représentent deux complexes sous ; le dimère de petites et grandes sous-unités CPS (160 kDa) et un hétéro-tétramère contenant deux exemplaires de chaque sous-unité (320 kDa). Ces complexes sous livrer les premiers aperçus de l’Assemblée de la protéine ; par exemple, les grandes et petites sous-unités sont en contact direct (comme l’a révélé l’hétéro-dimères) et le tétramère peut être un produit de deux dimères. Pour obtenir plus d’information sur l’arrangement structurel dans la complexe de CPS intacte, nous avons effectué la spectrométrie de masse tandem (MS/MS) de l’hétéro-octamère et hétéro-tétramère. Dans les deux cas, la petite sous-unité dissociée le précurseur, ce qui suggère que la petite sous-unité est située dans la périphérie de l’Assemblée (Figure 4 b). En effet, la petite sous-unité est périphérique dans le cristal disponibles structure (Figure 4)44.
Réticulation chimique utilisant le réticulant BS3 a également été effectuée. À l’aide de quantités croissantes, la liaison covalente des sous-unités CPS a été améliorée. Après la digestion des protéines et l’analyse des peptides comme décrit ci-dessus, de nombreuses interactions de protéine au sein de la grande sous-unité et un pontage dans la petite sous-unité ont été obtenues (tableau 2). En outre, tout comme le complexe RvB1/B2, nous avons trouvé deux liens inter moléculaires croisés entre deux copies de la grande sous-unité de la CPS. Ces réticulations placer deux sous-unités grandes face à face à leurs côtés de C-terminal. Dans une étude antérieure, combinant la spectrométrie de masse structurelle et la modélisation informatique35, nous avons identifié trois interactions supplémentaires dans la grande sous-unité, qui très probablement sont créés depuis l’interface de deux copies de la grande sous-unité validés par le la structure cristalline et le modèle obtenu (Figure 4 et tableau 2). Ces interactions permettent l’arrangement du noyau CPS complexe composé de quatre grandes sous-unités. Toutefois, aucun inter-sous-unité des liens croisés entre les petites et les grosses sous-unités ont été observés. En inspectant la structure du cristal disponibles (Figure 4), il devient évident que la surface de l’interaction entre le noyau tétramère de ce complexe, composé de la grande sous-unité et les sous-unités de petites périphériques est très faible, ce qui pourrait expliquer l’absence d’interactions entre sous-unités. Ceci est confirmé par spectrométrie de masse native qui a montré que la petite sous-unité se dissocie facilement des complexes intacts très probables due à une interface de liaison petit. Néanmoins, les interactions de protéine dans le CPS complexe combiné de réticulation chimique et spectrométrie de masse native permettent de déduire leur arrangement structural (Figure 4).
Pris ensemble, la combinaison de la spectrométrie de masse native et réticulation chimique couplée à une identification par spectrométrie de masse de peptides réticulés, permet de reconstitution de l’entente structurelle des deux complexes de l’exemple. Réticulation chimique a révélé l’arrangement des sous-unités protéiques, par exemple les interactions entre RvB1 et RvB2 ou dans le noyau tétramère de CPS, spectrométrie de masse native livré stoechiométries de protéine des complexes intacts et subcomplexes courantes. Dans le cas de la CPS, pour qui aucune leurs interactions entre les deux sous-unités ne pourraient être observées par réticulation chimique, spectrométrie de masse native suggère que chaque grande sous-unité interagit avec une petite sous-unité (Figure 4). Spectrométrie de masse a proposé la situation périphérique de la petite sous-unité dans le complexe et une petite interface entre les deux sous-unités.
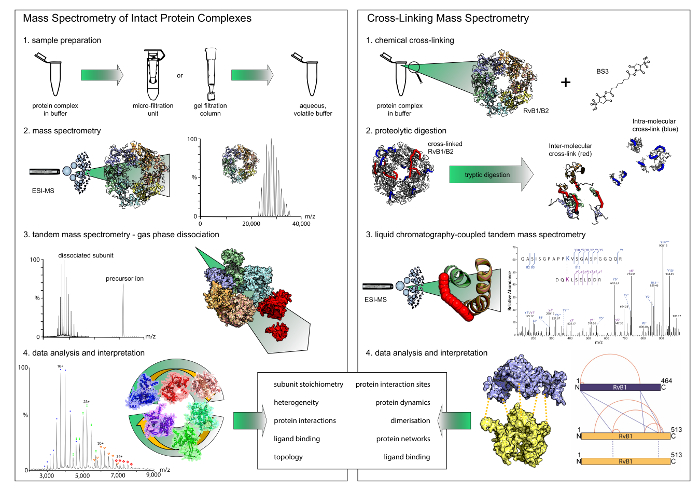
Figure 1 : flux de travail de la spectrométrie de masse native et réticulation. Les deux techniques de livrer des résultats complémentaires. Alors que la spectrométrie de masse native dévoile stoechiométries et modules d’interaction, réticulation donne des indications sur les sites d’interaction de protéine dans les complexes. Notez que seulement de réticulation chimique révèle des interactions binaires. (A) la première étape en spectrométrie de masse native est change de tampon sur un buffer volatil et aqueux à l’aide de blocs filtrants ou colonnes de gel filtration. Spectrométrie de masse des complexes des protéines intactes révèle alors leur stoechiométrie. Dans des expériences de spectrométrie de masse en tandem, sous-unités périphériques sont dissociées. (B) pour la réticulation chimique, le complexe protéique est incubé avec un réactif de réticulation. Les protéines réticulées sont ensuite digérées en peptides qui sont ensuite analysés par spectrométrie de masse couplée à la chromatographie en phase liquide. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
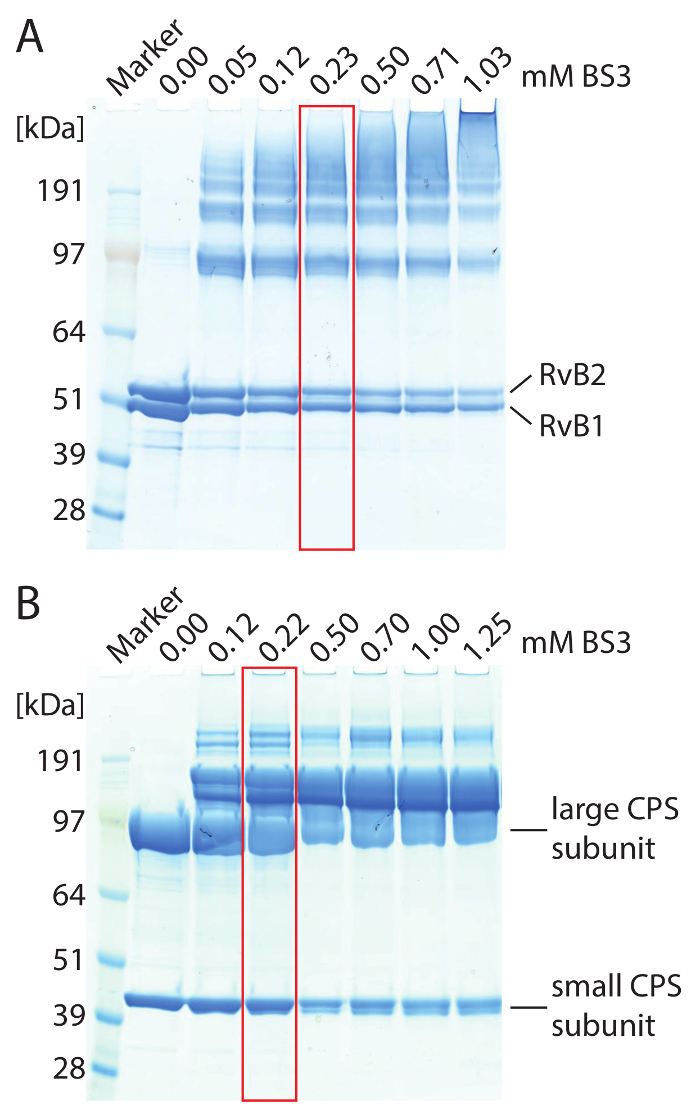
Figure 2 : SDS-PAGE de réticulé complexes RvB1/B2 (A) et CPS b. (A) 2,5 µM RvB1/B2 ont été chargés par voie de gel. La concentration de BS3 a été variée. Non réticulé RvB1/B2 montre les sous-unités de deux protéine à environ 50 kDa. Ajout de BS3 causé une liaison covalente des sous-unités protéine résultant dans les bandes de protéines à poids moléculaire plus élevé. Le montant des espèces réticulés est augmenté avec des concentrations plus élevées de BS3. Des conditions optimales de réticulation sont en surbrillance (rouge). (B) 10 µM CPS ont été chargés par voie de gel. Le grand (90 kDa) et petit (40 kDa) on obtient des sous-unités de la CPS. Ajout de BS3 causé une liaison covalente des sous-unités protéine résultant dans les bandes de protéines à poids moléculaire plus élevé. Des conditions optimales de réticulation sont en surbrillance (rouge). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
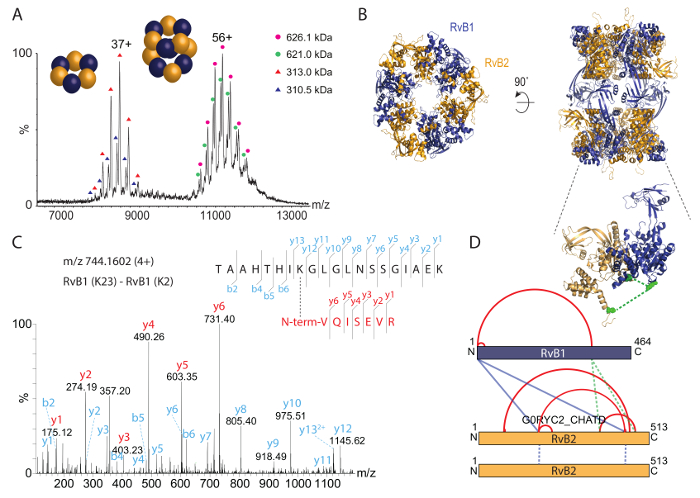
Figure 3 : spectrométrie de masse Native et réticulation chimique des RvB1/B2 complexes. (A), le spectre de masse natif révèle deux espèces de RvB1/B2 ; le dodécamère intact (c.-à-d., (RvB1)6(RvB2)6) à environ 11 000 à 12 000 m/z et le hexamérique anneau (RvB1)3(RvB2)3 , à environ 8 000 m/z. Les deux espèces montrent deux populations résultant de son marqués et non marqués RvB2. Le spectre a été modifié par35. Structure (B), le cristal de RvB1/B2 est montré (PDB ID 4WVY). Alternance de RvB1 et RvB2 sous-unités forment deux anneaux hexamérique. (C) spectre de Fragmentation d’un di-peptide réticulé. L’extrémité N-terminale de RvB1 a été réticulé avec K23 de RvB1. série y-ion ont été obtenus pour les deux peptides (rouge et cyan). (D) Intra - et inter - protein interactions obtenues dans le complexe RvB1/B2. Intra-cross-liens apparaissent en rouge, inter-cross-links sont indiqués en bleu. L’encart montre deux liens inter moléculaires croisés entre les sous-unités RvB1 et RvB2 qui pourraient être visualisées dans la structure cristalline (verte, insertion). Les interactions qui proviennent de deux copies de RvB2 sont affichées sous forme de lignes pointillées bleues. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
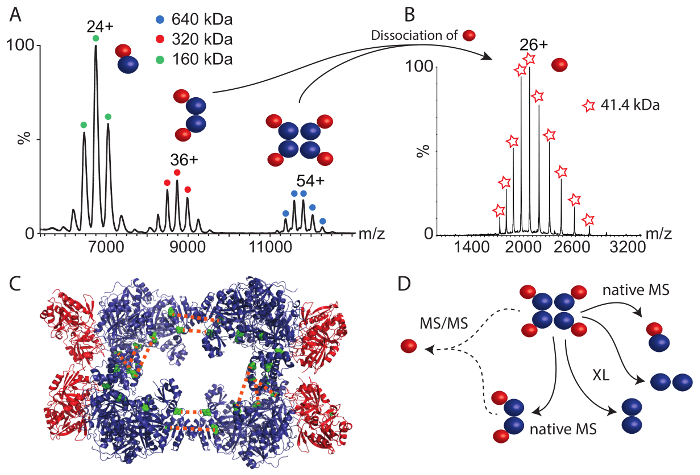
Figure 4 : spectrométrie de masse Native et réticulation chimique des CPS. (A), le spectre de masse natif de CPS montre trois complexes. L’hétéro-dimères (160 kDa), Hétéro-tétramère (320 kDa) et l’hétéro-octamère (640 kDa). Le spectre a été modifié par35. (B) spectrométrie de masse de la dissociation révélée complexe CPS de la petite sous-unité de CPS, tétramère et et. (C), le crystal structure du CPS est montré (PDB ID 1BXR). Les grosses sous-unités forment un noyau tétramère et les petites sous-unités sont situées à la périphérie du complexe. Leurs liens croisés entre les deux copies de la grande sous-unité apparaissent (verts). (D) les Interactions des sous-unités CPS petites et grandes. Spectrométrie de masse native a révélé subcomplexes et suggère une situation périphérique de la petite sous-unité. Réticulations chimiques indiquent des arrangements dans le noyau tétramère de CPS. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Tableau 1 : résultats de recherche de base de données. Les protéines ont été identifiés par spectrométrie de masse couplée à la chromatographie en phase liquide et la recherche de base de données. Les noms de protéine, d’adhésion nombre et description comme protéine de masse sont donnés. Le score de protéine, nombre de spectres observés par les protéines et le nombre de séquences peptidiques observées sont répertoriés. Les cinq peptides avec les meilleurs scores de peptides mascotte sont répertoriés pour chaque sous-unité protéique. S’il vous plaît cliquez ici pour télécharger ce fichier.
Tableau 2 : liaisons transversales observées en RvB1/B2 et CPS. Les sous-unités des complexes et les résidus réticulés sont données. Le type de pontage (intra - ou inter moléculaires) a été révélé de chevauchement des séquences peptidiques ou une précédente étude35. S’il vous plaît cliquez ici pour télécharger ce fichier.
Discussion
Protocoles sont fournis pour l’analyse structurale basé sur la spectrométrie de masse des complexes de plusieurs sous-unités protéiques. Les deux techniques, décrites dans le protocole, pour la plupart donnent des résultats complémentaires et sont bien adaptés pour nous éclairer sur les arrangements structurels au sein de la protéine (-ligand) complexes qui sont difficiles à étudier par des techniques structurelles conventionnelles. Spectrométrie de masse Native offre aperçus de stoechiométries protéines ainsi que des interactions de protéine par l’analyse subcomplexes et modules d’interaction stable. Réticulation, d’autre part, fournit une information sur les sites de contact directs. Selon l’agent utilisé, une certaine souplesse peut ou doit être incluse dans l’analyse.
Les protocoles fournis sont en général facile à effectuer et pas beaucoup de temps. L’ensemble du protocole peut être exécuté dans une semaine et peut être appliqué à presque tous les complexes de protéine, bien qu’une certaine quantité de la protéine complexe est nécessaire pour l’analyse réussie. Préparation de l’échantillon est simple et ne nécessite pas spécifiquement des complexes de protéine purifiée. Toutefois, un piège commun est la contamination de l’échantillon au cours de la préparation des échantillons pour l’identification des protéines basé sur la spectrométrie de masse. Ces contaminations dans la plupart des cas comprennent les kératines qui proviennent de la poussière, la peau et les cheveux. Par conséquent, supplémentaires comme le port de gants et blouses, filtrant les tampons aqueux et l’utilisation de solvants de grande pureté il faut au cours de la préparation des échantillons pour l’identification des protéines basé sur la spectrométrie de masse. Autres protéines contaminantes telles que les accompagnateurs sont généralement introduites au cours de la purification des protéines, par exemple, lors de l’utilisation de balises d’affinité. Dans ces cas, purification des protéines doit être améliorée, par exemple en augmentant les étapes de lavage. Dans tous les cas, des contaminations de protéines dans l’échantillon sont facilement identifiées lors de la recherche de la base de données en omettant le filtre de la taxonomie (c.-à-d.la recherche contre les protéines de toutes les espèces). Si seulement quelques peptides sont observés (c'est-à-dire, une couverture de faible teneur en protéines pourrait être obtenue), même si l’échantillon suffisant est disponible, il pourrait être nécessaire d’utiliser une protéinase différente au cours de la digestion. En général la trypsine donne un nombre suffisant de peptides ; Toutefois, dans certains cas par exemple de protéines membranaires ou domaines membranaires des protéines, le nombre de sites de clivage trypsique est réduit et autres enzymes ciblant des acides aminés hydrophobes sont un meilleur choix.
En ce qui concerne l’instrumentation, un instrument particulièrement mis à jour l’est nécessaire pour la spectrométrie de masse native qui maintient les interactions non covalentes lors du transfert dans la phase gazeuse. Plusieurs types d’instruments ont été introduits, y compris les instruments Q-ToF et Orbitrap valant. Tandis que les spectromètres de masse mis à jour le Q-ToF sont disponibles dans le commerce pour la spectrométrie de masse native depuis plusieurs années, celles-ci ont été introduites que récemment et dans la plupart des cas nécessite la modification spécialisées45. Toutefois, l’application des instruments à haute résolution a permis l’étude de liaison des ligands multiples et leur quantification46,47 et est prometteuse pour les futures applications.
Pour identifier les di-peptides réticulés par spectrométrie de masse couplée à la chromatographie en phase liquide, les procédures standard avec quelques modifications peuvent être appliquées. Cependant, la recherche de la base de données est le facteur limitant car un logiciel spécialisé peut rarement traitent de grandes bases de données et des bases de données réduits contenant les sous-unités protéiques des complexes sont nécessaires. Des études récentes utilisé la spectrométrie de masse-CLIVABLES agents ciblant les interactions de protéine dans la cellule entière lysats48,49. L’utilisation d’agents chimiques qui se fragmentent pour la plupart dans des expériences de spectrométrie de masse tandem cède peptides linéaires (modifiés par l’agent), qui peuvent être identifiés par une nouvelle fragmentation et base de données, la recherche de peptides linéaires et cela réduit temps de recherche et de l’espace de recherche computationnelle. Cependant, pour effectuer ces expériences, un analyseur de masse piège ionique ou un spectromètre de masse hybride avec un piège ionique est requis. En général, comme les faux positifs sont une question importante, des spectres de masse de peptides réticulés souvent se valident manuellement par la qualité de leurs spectres de fragment qui étend énormément les temps d’analyse de données. Conçoit des systèmes de notation robustes qui peuvent être appliquées sans autres étapes de validation sont donc possibles applications futures. Une façon d’améliorer l’analyse des données et de réduire le nombre de faux positifs a été l’introduction de calculs de taux de fausse découverte et leur application aux ensembles de données50de réticulation.
En général, les techniques décrites ici peuvent être complétés avec d’autres techniques de spectrométrie de masse (par exemple, le marquage covalent) pour augmenter le rendement de l’analyse. Autres modifications et améliorations des protocoles peuvent être facilement mis en œuvre. Ainsi comparative réticulation34 dénoue les changements conformationnels dans l’assemblage des protéines. Évolution de la situation en spectrométrie de masse native aujourd'hui permettre l’analyse des protéines de membrane51,52 et leurs interactions avec les lipides28,52,,du5354 . Nouveaux développements de spectromètres de masse haute résolution pour la spectrométrie de masse native ont étendu l’application et de liaison du ligand, par exemple, liaison des lipides aux protéines de la membrane, peuvent maintenant être inclus dans l’analyse de45, 46. en combinaison avec des approches de modélisation informatique, ces techniques peuvent offrir des modèles structuraux de la variation de résolution55. Si aucune structures cristallines ne sont disponibles pour les sous-unités simples ou complexes intactes, spectrométrie de masse peut fournir des premiers aperçus des interactions de protéine et la topologie du complexe inconnue. Selon les techniques utilisées et les résultats obtenus, les modèles basse résolution du complexe inconnu peuvent être obtenus56,57,58. Si les structures cristallines ou modèles d’homologie sont disponibles, les informations structurelles a reçu de la spectrométrie de masse peuvent produire des modèles même quasi native59.
En comparaison avec d’autres techniques structurelles, spectrométrie de masse a l’avantage qu’il nécessite des quantités d’échantillon faible, il peut traiter les échantillons hétérogènes et s’applique à des complexes protéiques de taille illimitée. En outre, la spectrométrie de masse permet l’étude des systèmes dynamiques de protéines. Des populations différentes de la protéine ou une protéine complexe qui existent en solution sont généralement analysées ensemble et donc, à la différence avec d’autres techniques structurelles qui nécessitent la sélection de certaines populations, toutes les conformations sont maintenues pendant analyse et sont imposable dans une expérience. Approches quantitatives de réticulation ont été récemment introduites34,60,61 et sont prometteurs pour de futures applications décrivant les changements conformationnels dans des conditions différentes.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions nos collègues pour discussions utiles. Nous remercions également Ilme Schlichting et Karl-Peter Hopfner pour fournir des complexes protéiques. Nous reconnaissons le financement par le ministère fédéral pour l’éducation et la recherche (BMBF, ZIK programme, 03Z22HN22), le Fonds européen de développement régional (FEDER, ZS/2016/04/78115) et de la Halle-Wittenberg MLU C.S. et le financement du Wellcome Trust (109854/658 Z/15/Z) à A.P.
matériels
| Name | Company | Catalog Number | Comments |
| Chemicals, Reagents, Consumables | |||
| 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid (HEPES) | Sigma Aldrich | H4034 | buffer |
| Acetic acid | Sigma Aldrich | 695092 | pH |
| Acetonitrile Optima LC/MS | Fisher Chemicals | A955-1 | solvent |
| Amicon Ultra centrifugal devices (different MWCO) | Millipore | i.e. UFC500396 | buffer exchange, concentration |
| Ammonium acetate solution | Sigma Aldrich | A2706 | native MS |
| Ammonium bicarbonate | Sigma Aldrich | 9830 | in-gel digestion |
| Ammonium solution | Sigma Aldrich | 9859 | pH |
| Bis(Sulfosuccinimidyl)suberate-d0 (BS3-d0) | Thermo Scientific | 21590 | cross-linker |
| Bis(Sulfosuccinimidyl)suberate-d4 (BS3-d4) | Thermo Scientific | 21595 | cross-linker |
| Caesium iodide | Sigma Aldrich | 203033 | calibration |
| Calcium chloride | Sigma Aldrich | C5670 | in-gel digestion |
| Capillaries (1.0 OD × 0.78 ID × 100 L mm) | Harvard Apparatus | 30-0038 | native MS |
| Disuccinimidyl suberate (DSS) | Thermo Scientific | 21655 | cross-linker |
| DL-Dithiothreitol | Sigma Aldrich | D5545 | in-gel digestion |
| DMSO (dimethyl sulfoxide) | Sigma Aldrich | D8418 | solvent |
| Ethanol | Fisher Chemicals | BP2818 | solvent |
| Formic acid Optima LC/MS | Fisher Chemicals | A117-50 | solvent supplement |
| Instant Blue Coomassie staining solution | expedeon | ISB1L | staining solution for gel electrophoresis |
| Invitrogen NuPAGE 4-12% Bis-Tris gels (1 mm, 10 well) | life technologies | NP0323BOX | gel electrophoresis |
| Iodoacetamide | Sigma Aldrich | I1149 | In-gel digestion |
| Isopropyl alcohol, HPLC grade | Fisher Chemicals | P750717 | solvent |
| Methanol | Fisher Chemicals | A456-212 | solvent |
| Micro BioSpin 6 columns | BioRad | 732-6222 | buffer exchange |
| NuPAGE Antioxidant | invitrogen | NP0005 | running buffer, gel electrophoresis |
| NuPAGE LDS Sample Buffer (4 ×) | invitrogen | NP0007 | sample loading buffer (non-reducing) for gel electrophoresis |
| NuPAGE MES SDS Running buffer | life technologies | NP0001 | gel electrophoresis |
| NuPAGE MOPS SDS Running buffer | life technologies | NP0001 | gel electrophoresis |
| NuPAGE Sample Reducing Agent (10 ×) | invitrogen | NP0004 | reducing Agent for gel electrophoresis |
| PBS - phosphate buffered saline | Sigma Aldrich | P4417 | buffer |
| SeeBlue Plus2 Prestained Standard Protein Marker | invitrogen | LC5925 | prestained Protein Marker for gel electrophoresis |
| Sodium acetate | Sigma Aldrich | S2889 | ethanol precipitation |
| Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 252859 | buffer |
| Trypsin | Sigma Aldrich | TRYPSEQ-RO (11418475001) | in-gel digestion |
| Vivaspin centrifugal devices (different MWCO) | Sartorius | i.e. VS0101 | buffer exchange, concentration |
| Water for HPLC | Sigma Aldrich | 270733-2.5L-M | solvent |
| Name | Company | Catalog Number | Comments |
| Instruments | |||
| Centrifuge Heraeus Fresco 21, bench-top centrifuge | Thermo Scientific | 75002425 | |
| Flaming / Brown Micropipette puller Model P-1000 | Sutter Instruments | P-1000 | |
| Gelelectrophoresis chamber Xcell SureLock MiniCell | Invitrogen | EI0001 | |
| Gold Coater Quorum Q150R S | Quorum Technologies Ltd. | Q150RS | |
| Horizontal gel shaker Rotamax 120 T | Heidolph Instruments | 544-41200-00 | |
| Q-TOF Ultima mass spectrometer, MS Vision high mass upgrade | Waters (Micromass) | - | |
| reversed-phase C18 analytical column Acclaim PepMap (C18, 75 mm I.D., 50 cm, 3 mm pore size) | Thermo Scientific | 164570 | |
| reversed-phase C18 pre-column (C18, 150 mm I.D., 2 cm, 5 mm pore size) | Thermo Scientific | 164213 | |
| scalpel | Fisher Scientifc | 10463989 | |
| SpeedVac SPD121P vacuum centrifuge | Thermo Scientific | SPD121DP-230 | |
| Thermomixer C | Eppendorf | 5382000015 | |
| Tweezers AA | Sigma Aldrich | Z680184-1EA | |
| Ultrasonic cleaner USC-TH, sonication bath | VWR | 142-0084 | |
| UltiMate Dionex 3000 nano-LC system, coupled to Q-Exactive plus hybrid mass spectrometer (nano-ESI source) | Thermo Scientific | 0726030+ | |
| Name | Company | Catalog Number | Comments |
| Software, Software Tools, Database search | |||
| Mascot | www.matrixscience.com | ||
| Massign | http://massign.chem.ox.ac.uk | ||
| MassLynx | Micromass | ||
| MassMatrix | Xu, H. J Proteome Res. 9 (2010) | ||
| MaxQuant | Cox, J. Nat. Biotechn. 26 (2008) | ||
| pLink | Yang, B. Nat Methods 9 (2012) | ||
| pXtract conversion tool | http://pfind.ict.ac.cn/downloads.html | ||
| UniDec | Marty, M.T. Anal. Chem. 87 (2015) | ||
| XCalibur | Thermo Scientific | ||
| XiNET | http://crosslinkviewer.org | ||
| XLinkX | Liu, F. Curr Op Struct Biol 35 (2015) | ||
| xQuest | Rinner, O. Nat Methods 5 (2008) | ||
| Xvis | https://xvis.genzentrum.lmu.de |
Références
- Campbell, I. D. Timeline: the march of structural biology. Nat Rev Mol Cell Biol. 3 (5), 377-381 (2002).
- Liko, I., Allison, T. M., Hopper, J. T., Robinson, C. V. Mass spectrometry guided structural biology. Curr Opin Struct Biol. 40, 136-144 (2016).
- Robinson, C. V. From molecular chaperones to membrane motors: through the lens of a mass spectrometrist. Biochem Soc Trans. 45 (1), 251-260 (2017).
- Lossl, P., van de Waterbeemd, M., Heck, A. J. The diverse and expanding role of mass spectrometry in structural and molecular biology. EMBO J. 35 (24), 2634-2657 (2016).
- Wang, L., Chance, M. R. Protein Footprinting Comes of Age: Mass Spectrometry for Biophysical Structure Assessment. Mol Cell Proteomics. 16 (5), 706-716 (2017).
- Steen, H., Mann, M. The ABC's (and XYZ's) of peptide sequencing. Nat Rev Mol Cell Biol. 5 (9), 699-711 (2004).
- Olsen, J. V., Mann, M. Status of large-scale analysis of post-translational modifications by mass spectrometry. Mol Cell Proteomics. 12 (12), 3444-3452 (2013).
- Schmidt, C., Robinson, C. V. Dynamic protein ligand interactions--insights from MS. FEBS J. 281 (8), 1950-1964 (2014).
- Marcsisin, S. R., Engen, J. R. Hydrogen exchange mass spectrometry: what is it and what can it tell us?. Anal Bioanal Chem. 397 (3), 967-972 (2010).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Leitner, A., Lindner, W. Current chemical tagging strategies for proteome analysis by mass spectrometry. J Chromatogr B Analyt Technol Biomed Life Sci. 813 (1-2), 1-26 (2004).
- Leitner, A., Lindner, W. Chemistry meets proteomics: the use of chemical tagging reactions for MS-based proteomics. Proteomics. 6 (20), 5418-5434 (2006).
- Kiselar, J. G., Chance, M. R. Future directions of structural mass spectrometry using hydroxyl radical footprinting. J Mass Spectrom. 45 (12), 1373-1382 (2010).
- Xu, G., Chance, M. R. Hydroxyl radical-mediated modification of proteins as probes for structural proteomics. Chem Rev. 107 (8), 3514-3543 (2007).
- Liu, F., Heck, A. J. Interrogating the architecture of protein assemblies and protein interaction networks by cross-linking mass spectrometry. Curr Opin Struct Biol. 35, 100-108 (2015).
- Rappsilber, J. The beginning of a beautiful friendship: cross-linking/mass spectrometry and modelling of proteins and multi-protein complexes. J Struct Biol. 173 (3), 530-540 (2011).
- Rinner, O., et al. Identification of cross-linked peptides from large sequence databases. Nat Methods. 5 (4), 315-318 (2008).
- Yang, B., et al. Identification of cross-linked peptides from complex samples. Nat Methods. 9 (9), 904-906 (2012).
- Xu, H., Hsu, P. H., Zhang, L., Tsai, M. D., Freitas, M. A. Database search algorithm for identification of intact cross-links in proteins and peptides using tandem mass spectrometry. J Proteome Res. 9 (7), 3384-3393 (2010).
- Leitner, A., Faini, M., Stengel, F., Aebersold, R. Crosslinking and Mass Spectrometry: An Integrated Technology to Understand the Structure and Function of Molecular Machines. Trends Biochem Sci. 41 (1), 20-32 (2016).
- Heck, A. J. Native mass spectrometry: a bridge between interactomics and structural biology. Nat Methods. 5 (11), 927-933 (2008).
- Sharon, M., Robinson, C. V. The role of mass spectrometry in structure elucidation of dynamic protein complexes. Annu Rev Biochem. 76, 167-193 (2007).
- Goth, M., Pagel, K. Ion mobility-mass spectrometry as a tool to investigate protein-ligand interactions. Anal Bioanal Chem. , (2017).
- Uetrecht, C., Rose, R. J., van Duijn, E., Lorenzen, K., Heck, A. J. Ion mobility mass spectrometry of proteins and protein assemblies. Chem Soc Rev. 39 (5), 1633-1655 (2010).
- Hernandez, H., Robinson, C. V. Determining the stoichiometry and interactions of macromolecular assemblies from mass spectrometry. Nat Protoc. 2 (3), 715-726 (2007).
- Sobott, F., Hernandez, H., McCammon, M. G., Tito, M. A., Robinson, C. V. A tandem mass spectrometer for improved transmission and analysis of large macromolecular assemblies. Anal Chem. 74 (6), 1402-1407 (2002).
- Rostom, A. A., et al. Detection and selective dissociation of intact ribosomes in a mass spectrometer. Proc Natl Acad Sci U S A. 97 (10), 5185-5190 (2000).
- Zhou, M., et al. Mass spectrometry of intact V-type ATPases reveals bound lipids and the effects of nucleotide binding. Science. 334 (6054), 380-385 (2011).
- Uetrecht, C., et al. High-resolution mass spectrometry of viral assemblies: molecular composition and stability of dimorphic hepatitis B virus capsids. Proc Natl Acad Sci U S A. 105 (27), 9216-9220 (2008).
- Morgner, N., et al. Hsp70 forms antiparallel dimers stabilized by post-translational modifications to position clients for transfer to Hsp90. Cell Rep. 11 (5), 759-769 (2015).
- Schmidt, C., Beilsten-Edmands, V., Robinson, C. V. The joining of the Hsp90 and Hsp70 chaperone cycles yields transient interactions and stable intermediates: insights from mass spectrometry. Oncotarget. 6 (21), 18276-18281 (2015).
- Schmidt, C., Beilsten-Edmands, V., Mohammed, S., Robinson, C. V. Acetylation and phosphorylation control both local and global stability of the chloroplast F1 ATP synthase. Sci Rep. 7, 44068 (2017).
- Schmidt, C., et al. Comparative cross-linking and mass spectrometry of an intact F-type ATPase suggest a role for phosphorylation. Nat Commun. 4, 1985 (2013).
- Schmidt, C., Robinson, C. V. A comparative cross-linking strategy to probe conformational changes in protein complexes. Nat Protoc. 9 (9), 2224-2236 (2014).
- Schmidt, C., et al. Surface Accessibility and Dynamics of Macromolecular Assemblies Probed by Covalent Labeling Mass Spectrometry and Integrative Modeling. Anal Chem. 89 (3), 1459-1468 (2017).
- Lakomek, K., Stoehr, G., Tosi, A., Schmailzl, M., Hopfner, K. P. Structural basis for dodecameric assembly states and conformational plasticity of the full-length AAA+ ATPases Rvb1 · Rvb2. Structure. 23 (3), 483-495 (2015).
- Mareya, S. M., Raushel, F. M. A molecular wedge for triggering the amidotransferase activity of carbamoyl phosphate synthetase. Biochemistry. 33 (10), 2945-2950 (1994).
- Shevchenko, A., Wilm, M., Vorm, O., Mann, M. Mass spectrometric sequencing of proteins silver-stained polyacrylamide gels. Anal Chem. 68 (5), 850-858 (1996).
- Olsen, J. V., et al. Parts per million mass accuracy on an Orbitrap mass spectrometer via lock mass injection into a C-trap. Mol Cell Proteomics. 4 (12), 2010-2021 (2005).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol. 26 (12), 1367-1372 (2008).
- Kirshenbaum, N., Michaelevski, I., Sharon, M. Analyzing large protein complexes by structural mass spectrometry. J Vis Exp. (40), (2010).
- Morgner, N., Robinson, C. V. Massign: an assignment strategy for maximizing information from the mass spectra of heterogeneous protein assemblies. Anal Chem. 84 (6), 2939-2948 (2012).
- Marty, M. T., et al. Bayesian deconvolution of mass and ion mobility spectra: from binary interactions to polydisperse ensembles. Anal Chem. 87 (8), 4370-4376 (2015).
- Thoden, J. B., Wesenberg, G., Raushel, F. M., Holden, H. M. Carbamoyl phosphate synthetase: closure of the B-domain as a result of nucleotide binding. Biochemistry. 38 (8), 2347-2357 (1999).
- Rose, R. J., Damoc, E., Denisov, E., Makarov, A., Heck, A. J. High-sensitivity Orbitrap mass analysis of intact macromolecular assemblies. Nat Methods. 9 (11), 1084-1086 (2012).
- Gault, J., et al. High-resolution mass spectrometry of small molecules bound to membrane proteins. Nat Methods. 13 (4), 333-336 (2016).
- Mehmood, S., et al. Mass spectrometry captures off-target drug binding and provides mechanistic insights into the human metalloprotease ZMPSTE24. Nat Chem. 8 (12), 1152-1158 (2016).
- Kaake, R. M., et al. A new in vivo cross-linking mass spectrometry platform to define protein-protein interactions in living cells. Mol Cell Proteomics. 13 (12), 3533-3543 (2014).
- Liu, F., Rijkers, D. T., Post, H., Heck, A. J. Proteome-wide profiling of protein assemblies by cross-linking mass spectrometry. Nat Methods. 12 (12), 1179-1184 (2015).
- Walzthoeni, T., et al. False discovery rate estimation for cross-linked peptides identified by mass spectrometry. Nat Methods. 9 (9), 901-903 (2012).
- Barrera, N. P., Di Bartolo, N., Booth, P. J., Robinson, C. V. Micelles protect membrane complexes from solution to vacuum. Science. 321 (5886), 243-246 (2008).
- Barrera, N. P., et al. Mass spectrometry of membrane transporters reveals subunit stoichiometry and interactions. Nat Methods. 6 (8), 585-587 (2009).
- Laganowsky, A., et al. Membrane proteins bind lipids selectively to modulate their structure and function. Nature. 510 (7503), 172-175 (2014).
- Marcoux, J., et al. Mass spectrometry reveals synergistic effects of nucleotides, lipids, and drugs binding to a multidrug resistance efflux pump. Proc Natl Acad Sci U S A. 110 (24), 9704-9709 (2013).
- Politis, A., Schmidt, C. Structural characterisation of medically relevant protein assemblies by integrating mass spectrometry with computational modelling. J Proteomics. , (2017).
- Hall, Z., Politis, A., Robinson, C. V. Structural modeling of heteromeric protein complexes from disassembly pathways and ion mobility-mass spectrometry. Structure. 20 (9), 1596-1609 (2012).
- Pukala, T. L., et al. Subunit architecture of multiprotein assemblies determined using restraints from gas-phase measurements. Structure. 17 (9), 1235-1243 (2009).
- Politis, A., et al. Topological models of heteromeric protein assemblies from mass spectrometry: application to the yeast eIF3:eIF5 complex. Chem Biol. 22 (1), 117-128 (2015).
- Politis, A., et al. A mass spectrometry-based hybrid method for structural modeling of protein complexes. Nat Methods. 11 (4), 403-406 (2014).
- Fischer, L., Chen, Z. A., Rappsilber, J. Quantitative cross-linking/mass spectrometry using isotope-labelled cross-linkers. J Proteomics. 88, 120-128 (2013).
- Yu, C., et al. Developing a Multiplexed Quantitative Cross-Linking Mass Spectrometry Platform for Comparative Structural Analysis of Protein Complexes. Anal Chem. 88 (20), 10301-10308 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon