需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
应用三色单分子焦虑研究蛋白质相互作用的相关性
摘要
在这里, 我们提出了一个协议, 以获得三色 smFRET 数据和分析与3D 集成隐马尔可夫模型。通过这种方法, 科学家可以从复杂的蛋白质系统中提取动力学信息, 包括协同或相关的相互作用。
摘要
单分子 rster 共振能量转移 (smFRET) 已成为一种广泛应用的生物物理技术, 研究生物分子的动力学。对于许多分子机器, 在一个细胞蛋白质必须与互动伙伴在一个功能周期, 以完成他们的任务。双色到多色 smFRET 的扩展使得同时探测多个相互作用或构象变化成为可能。这不仅为 smFRET 实验增加了一个新的维度, 而且它还提供了一种独特的可能性, 直接研究事件序列, 并在使用固定样本和全内反射荧光检测相关的相互作用显微镜 (显微镜)。因此, 多色 smFRET 是一种多用途的工具, 以定量的方式研究生物大分子化合物, 并在以前无法做到的细节。
在这里, 我们将展示如何克服多色 smFRET 实验对蛋白质的特殊挑战。我们提出了获取数据和提取动力学信息的详细协议。这包括使用3D 集成隐马尔可夫模型 (HMM) 对噪声数据的跟踪选择标准、状态分离和状态轨迹的恢复。与其他方法相比, 动态信息不是从停留时间直方图中恢复的, 而是直接从 HMM 中得到的。最大似然框架允许我们对动力学模型进行批判性评估, 并为利率提供有意义的不确定性。
通过将我们的方法应用于热休克蛋白 90 (Hsp90), 我们可以解开核苷酸结合和蛋白质的全球构象变化。这使我们可以直接观察到 Hsp90 二聚体的两个核苷酸结合袋之间的协同。
引言
许多蛋白质完成他们的作用在动态复合体与其他分子, 由构象变化和瞬态协会介导在宽广的范围的时间表1,2,3。耦合到外部能量源 (例如, ATP) 这些动态交互作用可以在功能循环中导致方向性, 并最终维持单元格中的非平衡稳定状态, 这是生命的先决条件。
为了充分理解这些分子机器, 以结构研究为指导的静态描述是不够的。此外, 重要的是要了解基本的动力学模型和确定的动力学速率常数。一些现有的方法让研究人员研究两个感兴趣的分子之间的二元相互作用的动力学,例如,表面等离子体共振, 具有光谱读数的松弛方法 (例如,跳转或停止流技术), 和核磁共振。但是, 它们的适用性在大多数情况下仅限于简单的双状态系统 (例如,一个绑定和一个未绑定状态), 这是由于批量实验所固有的平均值。在涉及更多的状态或中间体的情况下, 它们只产生速率常数的复杂混合。单分子方法, 如光学或磁性镊子或双色 smFRET,即,一个施主和一个受体荧光, 与表面固定的样品可以恢复所有观察到的构象变化的速率常数。但是, 当涉及到影响多个绑定站点的交互时, 这些方法仍然是有限的, 而关于两个 (或多个) 交互的可能相关性的信息只能通过一组实验的间接结论访问。
多色 smFRET4,5,6,7,8,9提供了一个机会, 直接研究这些组件之间的交互, 在实时和下接近生理情况10。这允许一个调查例如, 一个配体或另一个蛋白质的构象依赖性的捆绑8,9,11。这里介绍的总体方法是在特定位置标记感兴趣的蛋白质, 将一个蛋白质附加到测量室的表面, 并在棱镜型显微镜上跟踪时间的荧光强度 (详见9,12). 不同染料的空间接近度可以通过它们之间的能量转移来确定。标记策略可能因蛋白质而异 (在13中进行了审查), 并且避免了 smFRET 测量中的工件存在14的准则。
由于施主染料可能转移能量对不同的接受者染料在多颜色 smFRET 实验, 所有染料的相对位置不是可接近的从励磁一个染料单独15,16。但与交替激光励磁 (亚历克斯17, 并在18中进行了审查) 结合使用, 该方法提供了所有的时空信息, 分二次和亚纳米分辨率。
在主, 高分辨率结构信息可以通过使用的染料之间的距离计算, 从所有荧光强度的组合在一个多色 smFRET 实验与亚历克斯。然而, 在这里, 我们的重点是状态识别和分离以及动力学模型的提取, 其中多色 smFRET 是必不可少的。当 "只" 的结构确定由三角化是需要的, 一组简单的双色 smFRET 实验的高信噪比可以执行12,19。
我们使用部分荧光 () 作为两个荧光 7之间能量传输的代理。PF是从与双色实验的烦恼效率类似的荧光强度计算得出的:
7之间能量传输的代理。PF是从与双色实验的烦恼效率类似的荧光强度计算得出的:

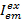 其中, 是在发射通道上的强度与颜色ex之后的em , 而c是波长最长的受体。检测通道在样品室中代表同一位置, 但记录荧光光的不同光谱范围。此协议中使用了相同的激发和发射标识符 (即"蓝色"、"绿色" 和 "红色")。
其中, 是在发射通道上的强度与颜色ex之后的em , 而c是波长最长的受体。检测通道在样品室中代表同一位置, 但记录荧光光的不同光谱范围。此协议中使用了相同的激发和发射标识符 (即"蓝色"、"绿色" 和 "红色")。
由于实验的缺陷, 所测得的荧光强度不仅取决于能量的转移, 而且还依赖于荧光和设置的性质。为了获得两个荧光之间的真实能量传递效率, 必须对测量的强度进行修正。以下过程基于引用9。明显渗漏的校正因子 (lk,即从指定给另一染料的通道中的荧光中检测光子) 和表观伽玛 (ag,即,染料的荧光量子屈服和该通道的检测效率是从单分子的痕迹, 显示一个受体漂白事件。
在所有可能的受体通道上的施主染料的泄漏是从记录的荧光痕迹的所有数据点计算的, 那里的受体染料漂白, 但施主仍然荧光 ():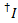
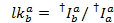
将泄漏直方图的中值作为表观泄漏系数。在对泄漏进行校正后, 视伽玛系数是从同一组跟踪中确定的。它是通过将受体通道中的荧光变化除以施主通道中的发光变化来计算受体染料的漂白:

其中, c再次是最长波长的接受方的检测通道。所产生的分布的中位数被用作表观校正因子。
每个通道的校正强度通过以下方法获得:
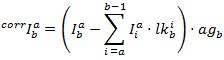
然后根据以下内容计算PF :

在PF所跨越的多维空间中, 可以分隔不同的填充。用多维高斯函数拟合数据, 确定了各状态的位置和宽度。基于所有PF跟踪的一个全局 HMM 的后续优化提供了观察到的动力学的定量描述。即使是微小的变化率是可察觉的。
模型提供一种从嘈杂的时间痕迹集合中推断状态模型的方法。系统被认为是在任意给定时间的一组离散的隐藏状态中的一个, 而实际观测 (即发射) 是这个隐藏状态的概率函数20。在显微镜 smFRET 数据的情况下, 可通过连续高斯概率密度函数对每个状态i的发射概率bi 进行建模。在通常间隔的离散时间点, 从一个到另一个状态的转换可以根据时间不变的过渡概率发生, 并且只取决于当前状态。转换矩阵a包含这些转换概率aij 在所有隐藏状态之间。初始状态分布为时间跟踪的第一个时间点提供特定于状态的概率. 
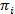 使用最大似然方法, 这些参数可以优化, 以最有效地描述数据的前向向后和鲍姆-韦尔奇算法20,21。这就产生了最大似然估计量。最后, 最有可能产生观测轨迹的状态序列可以用维特比算法来推断。与 smFRET 数据的其他 hmm 分析相反,24、2526我们不使用 hmm 作为数据的 "平滑", 而是从数据集中提取动态状态模型, 而不需要拟合驻留时间直方图27。HMM 分析是做内部脚本使用伊戈尔 Pro。代码的实现基于引用21。我们在我们的网页上提供一个软件套件和模范数据, 以遵循本协议 (https://www.singlemolecule.uni-freiburg.de/software/3d-fret) 的5和6节。可根据要求提供完整的软件。
使用最大似然方法, 这些参数可以优化, 以最有效地描述数据的前向向后和鲍姆-韦尔奇算法20,21。这就产生了最大似然估计量。最后, 最有可能产生观测轨迹的状态序列可以用维特比算法来推断。与 smFRET 数据的其他 hmm 分析相反,24、2526我们不使用 hmm 作为数据的 "平滑", 而是从数据集中提取动态状态模型, 而不需要拟合驻留时间直方图27。HMM 分析是做内部脚本使用伊戈尔 Pro。代码的实现基于引用21。我们在我们的网页上提供一个软件套件和模范数据, 以遵循本协议 (https://www.singlemolecule.uni-freiburg.de/software/3d-fret) 的5和6节。可根据要求提供完整的软件。
在任何检测通道中, pf <-1 或pf > 2 的数据中的时间点被分配给所有状态 (10-200) 的最小发射概率。这可以防止在这些数据点进行人工转换。
根据5.7 步中描述的高斯函数的 3D PF直方图的拟合, 得到了发射概率的参数。这些参数在优化 HMM 的过程中保持固定。
在所提出的方法中, 初始状态分布向量和转换矩阵在全局上用于描述整个跟踪的集合。它们根据引用27的数据集中的所有N分子进行更新。
初始状态分布的起始参数由PF直方图的2D 投影确定 (步骤 5.3), 而转换概率设置为 0.05, 但概率保持在同一状态, 这是选择离开某一状态的概率被归为统一。
一种似然分析方法用于给所有过渡速率的置信区间 (CIs)21,22, 它作为对其不确定性的有意义的估计。为了计算某一特定速率下的 CI 的边界, 将利率的转移概率定为非极大似然的值。这将生成测试模型λ "。给定数据集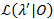 0的可能性的可能性比 (LR) 测试是根据以下内容执行的:
0的可能性的可能性比 (LR) 测试是根据以下内容执行的:
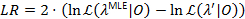
当LR超过 3.841, 95% 位的x2-分布与一个自由度22,23时, 参数的置信度达到了95%。
使用 Hsp90 演示了该方法的威力。这种丰富的蛋白质是在细菌和真核生物中发现的, 是细胞应激反应的一部分28。它是一个有前途的药物靶在癌症治疗29。Hsp90 是在每个子单元30的 N 终端域中具有一个核苷酸绑定口袋的 homodimer。它可以在至少两个全局不同的构象之间进行转换, 一个闭合和一个 N 终端开放, V 形构象19,31,32。二性质直接引起了 Hsp90 两个核苷酸结合位点之间相互作用的问题。
在下面的过程中, 我们提供了一个分步协议的数据获取和分析三色 smFRET 实验酵母 Hsp90 和核苷酸。分析了荧光标记的安培-pnp (amp)、非水解模拟 ATP 的构象依赖性结合。被描述的做法的应用允许研究核苷酸结合和同时 Hsp90 的构象变动和从而显露协同在二个核苷酸结合的口袋 Hsp90 之间。
研究方案
1. 设置和先决条件
- 在棱镜型显微镜上执行多色 smFRET 测量。在参考12中给出了作为朱庇特出版物的双色设置的说明。
- 构造多色显微镜。9中详细介绍了常规布局。
- 使用可切换、二极管泵浦的固态连续波激光器, 使机械百叶窗在励磁路径中的使用不必要。
- 使用不对称的, 拉长的棱镜, 防止从后方反射的励磁光束进入目标。
- 在检测路径中使用2英寸无色非球面熔融石英透镜, 收集尽可能多的光, 并防止自动荧光和像差,例如图像的非中心区域失真。
- 将每个检测路径集中在 EMCCD 的芯片上, 并使用一个单独的镜头。这使得每个检测通道的最佳聚焦。
警告:类3B 激光器用于显微镜。这意味着他们是危险的, 如果眼睛是直接暴露, 但漫反射是无害的。在系统运行前, 根据当地政府的规定, 确保遵守激光安全预防措施。
- 使用 dsDNA 样品预先确定安装和荧光性能的校正因子。
- 使用一个高烦恼 dsDNA 样品为每染料与接受人有最长的励磁波长 (为被提出的设置: Atto488-Atto647N, Atto550-Atto647N, Atto594-Atto647N)。确保 DNA 是另外用生物素修饰的。
- 将样本稀释至 5 nM, TNM 缓冲 (5 mm pH 7.5, 5 毫米氯化钠, 20 mm 氯化镁2) 和 2 mm Trolox (也可使用此缓冲区进行测量)。
- 固定在步骤2.5 和2.7 中描述的 dsDNA Hsp90。
- 计算表观渗漏 (lk) 和表观伽玛 (ag) 的校正因子, 从单分子迹线上显示受体漂白事件。
- 建造一个流室, 这是一个三明治的 peg/生物素 peg 钝化石英幻灯片, 薄膜, 是胶粘剂两边, 和一个封面滑动。为细节协议为石英幻灯片和钝化的清洁参见参考9。
- 使用厚 (3 毫米) 石英幻灯片, 以几何上防止激光散射在石英-甘油-石英界面之间的棱镜和功能化石英幻灯片的收集。
- 使用在非粘合面上喷涂粘合剂的薄 (40 µm) 密封膜。薄膜减少了表面附着分子与物镜之间的距离。加热到80° c 并按上。
- 在顶部放置一个盖子滑动。加热到80° c 并按上。在棱镜上放置流动腔时, 使用一滴甘油。
注: 棱镜和石英玻片的材料以及甘油是指数匹配的。
- 表达 Hsp90 从酿酒酵母以两个单一的半胱氨酸点突变体的形式在位置 D61 或 Q385。添加一个 C 终端盘绕线圈的主题, 以防止二聚体离解在 picomolar 浓度。分别标记突变蛋白, 并交换单体以获得 heterodimers 标记的 Atto488 在氨基酸位置61和 Atto550 在氨基酸位置 3859。
2. 测量
- 启动摄像机软件并设置如下所述的成像参数:
- 设置传感器冷却的温度尽可能低 (-95 ° c 与外部水冷), 以减少暗电流噪音。
- 使用针对单分子记录进行优化的相机设置: 3.3 µs 垂直移位速度、正常垂直时钟电压、17 MHz 16 位水平读出、预放大增益3、电子倍增器增益1000。
- 将捕获的触发设置为 "外部", 并将曝光时间设为 70 ms 记录电影, 其长度为750采集周期。
注意: 当照相机获得时, 关闭房间灯, 以防止 EMCCD 传感器的饱和。
- 在本地 SSD 上为测量创建一个文件夹。在软件设置进入 < 自动保存 > 车手, 启用 < 自动保存 >, 并选择文件格式 "Tiff" 的电影获取。选择 SSD 上的文件夹作为自动保存位置。
- 启动控制声光可调谐滤镜 (AOTF) 的软件, 该软件控制激光器的运行和触发软件, 同步激光器、AOTF、检测路径中的百叶窗和照相机。调整激光功率与 AOTF (约3兆瓦前进入棱镜), 并加载正确的触发模式。
- 安装的样品持有人与棱镜和流室, 附加油管, 放置在一个离心杯进口油管, 并连接出口油管到注射器泵。冲洗室与大约150µL 缓冲, 排列励磁光束和焦点。将任何荧光污染物漂白在表面上, 通过缓慢地沿着幻灯片的完整探测范围移动, 激光功率约10兆瓦, 用于所有激光器。这大约需要1小时。
注: 如果不另行说明, 则使用的缓冲器包含 40 mm HEPES pH 7.5、150 mm 氯化钾和 10 mm 氯化镁2。 - 冲洗约300µL 的 NeutrAvidin 溶液 (0.25 毫克/毫升缓冲) 进入会议厅和孵化1分钟. 冲洗出未绑定的 NeutrAvidin 与缓冲区和冲洗约300µL 的 BSA 溶液 (0.5 毫克/毫升缓冲) 通过会议厅。
注意: 这块剩余表面功能化缺陷通过不吸附 BSA 到表面。 - 固定样品通过装载流动房间与大约150µL 化并且被标记的 Hsp90 在上升的集中 (稀释在缓冲 + 0.5 毫克/毫升 BSA) 直到充足的表面密度被到达, 通常是案件在集中5-下午10点。用大约300µL 缓冲液 + 0.5 毫克/毫升 BSA 清洗未绑定的蛋白质。
- 在缓冲区 + 0.5 毫克/毫升 BSA 中, 150 µL 25 nM-PNP * 的冲洗。让它孵育5分钟, 并重复此步骤一次, 以确保正确的核苷酸浓度。
注: 对于在额外的, 未标记的 amp 的存在的实验, 也添加250µM 功放-pnp。 - 从数据获取开始。对于一个适当的数据量获得约20电影约1.5 小时。
- 将样品室的位置垂直于励磁梁与压电步进, 以改变视野。
- 调整焦点与 z 压电, 控制目标的高度, 如果需要。当测量室没有倾斜时, 这不应该经常是必要的。
- 通过在相机软件中按 < 采取信号 > 的方法准备摄像机的记录, 并使用触发器软件中的 < start > 按钮启动励磁/采集周期。这开始了对荧光强度的获取。
- 通过在设置的所有检测通道的光谱范围内, 首先录制带有荧光珠的电影来执行通道注册。然后, 通过搜索最亮的斑点并确定从高斯拟合到强度剖面的中心位置, 来检测校准影片中的磁珠位置。保存在所有通道中找到的珠子的坐标, 并将 x 和 y 方向的映射偏移量与2D 多项式的度数三9相匹配。
3. 单分子痕迹的选择
- 数据分析是通过内部脚本使用伊戈尔 Pro。通过打开 "iniTIRF. ipf" 并选择正确的实验类型来加载所有必要的脚本。
注意: 在下面的 "< 按钮 >" 中指定菜单或用户界面中的可单击元素。函数调用用引号表示,例如, "打印" Hello 世界 "。这些命令可以粘贴到命令行的伊戈尔 Pro (没有封闭引号)。 - 确保已加载检测通道注册的参数。
- 通过单击 < smFRET 新建 |分析 GUI >。
- 加载影片 (即,存储为16位 TIFF 堆栈的 512 x 512 像素的帧序列), 在各自的通道中保持强度。通过按 < 加载胶片 > 按钮, 然后从所谓的 "主" 和 "从属" 相机中选择一个接一个的文件。
- 通过在某一检测通道中搜索前五帧的总和中的最亮点来识别潜在单分子的位置。从通道映射中计算所有其他检测通道中的相应位置。为了获得荧光强度迹线, 对每个帧的中心位置周围的像素正方形的强度求和。通过按按钮 < 在 GUI 中查找跟踪 > 来执行此项。
注: 正方形的边长 (像素) 由: 2 * < 总和靴 > + 1。 - 对于每个分子, 计算一个联合的原始强度跟踪作为这个点的所有痕迹的总和相同的励磁颜色。根据以下标准评估所有通道中分子的强度分布:
- 一个大致平的高原在联合原始的强度和一个单一漂白步为所有励磁颜色, anticorrelated 行为在适当的检测渠道, 红色荧光的检测 (表明苦恼对一个束缚的放大器-PNP *) 至少一次在跟踪, 并没有在红色荧光的多个步骤, 这将表明存在两个安培-PNP * 绑定到一个 Hsp90 二聚体。
- 保存荧光痕迹的现场, 以进一步分析, 如果这些标准得到满足。通过选择带有光标的跟踪, 然后按 "时间线" 图中的 < 保存 > 按钮来执行此项。手动检查所有三检测通道的强度痕迹, 蓝色激发后, 每部电影大约200分子。
4. 部分荧光痕迹的计算
- 显示所保存的分子的所有荧光强度迹线。使用图表中的游标选择时间范围。
- 选择所有荧光已漂白的时间间隔。平均背景强度是从这个范围计算的, 并减去每个通道的强度跟踪。通过按 < 背景 > 按钮完成此项。
- 选择烦恼效率范围, 其中至少两个染料连接到 Hsp90 (Atto488 和 Atto550) 存在。请确保排除包含闪烁事件的跟踪 (请参见图 3B)。这些事件的特点是一个下降的荧光强度在一个通道没有伴随增加的任何其他渠道。
- 计算PF跟踪。通过按 < PF 计算器 > 按钮执行此项。用于表观泄漏 (lk) 和表观伽玛 (ag) 的预定义校正因子被应用于原始强度, 以纠正照片物理和设置属性。
5. 人口选择和3D 直方图拟合
- 删除在PF跟踪中显示低信噪比的分子。在任何PF跟踪中超过10% 帧的时间间隔 [-1; 2] 的分子将从数据集中移除。通过从命令窗口执行 "RemoveTracesLowSNR ()" 来完成此项。
- 计算PF数据的搁置2D 投影。在范围 [-0.5; 1.5] 中, 以 100 x 100 箱的分辨率绘制过和结束.
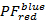
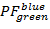
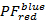 为此, 请执行以下操作:
为此, 请执行以下操作:- "HistFret2D (" r_b "," r_g ", binHist=100)"
- "HistFret2D (" r_b "," g_b ", binHist=100);MoveWindow 553.5, 42.5, 1055.25, 508.25 "
- 确定2D 投影中每个可区分状态的相对总体。
- 将适当的图形带到前面并执行 "panelHist2DCount ()"。
- 按 < 初始化 > 按钮, 在峰值周围绘制一个自由多边形。
- 单击 < 计数 > 按钮。多边形中的数据点数和投影中的数据点总数将打印在命令窗口中。
- 通过执行 "HistFret3D (" g_b "、" r_b "、" r_g ") 来准备PF数据的3D 直方图。
- 将3D 直方图正常化为1的整数。执行以下操作:
- "NewDataFolder/S fit0"
- "重复/O:: FRET:Hist3D, Hist3D"
- "可变/G div = 总和 (Hist3D) * (DimDelta (Hist3D, 0)) ^ 3"
- "Hist3D/= div;打印 div "
- 为3D 高斯拟合提供初始参数, 并准备必要的数据结构。
- 执行 "Gauss3D_initParam ();编辑 W_coef_old。
- 将状态填充添加到参数向量的末尾。
- 执行 "Gauss3D_prepareFit ()"。
注意:W_coef_old 是一个向量, 它保存了适合的初始参数。每状态这意味着和状态填充, 在向量的末尾串联.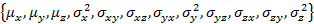 确保协方差矩阵是对称的。
确保协方差矩阵是对称的。
- 将S 3D 高斯函数的总和与 3D PF直方图相匹配, 其中s是可区分状态的数目。
- 执行 "do3D ()"。这可能需要一个小时或更多的正常办公 PC, 取决于初始参数的质量。
- 执行 "postprocessFitMultiGauss3D ();evalFitMultiGauss3D ();编辑 W_coef。
- 显示合适的结果。对于两个2D 投影中的每一项, 请使用以下命令:
- "contourPF3D_new (0);contourPF3D_new (1);contourPF3D_new (2);contourPF3D_new (3);contourPF3D_new (4) "
- "contourPF3D_colorize ()"
6. 3D 集成 HMM 的动力学分析
- 准备一个集成 HMM 运行来提取动力学信息。从数据集中的所有分子中优化一个 HMM。使用上一步中获得的信息来定义 3D PF空间中每个状态的位置和宽度。
- 初始化 hmm 用户界面 (< hmm |初始化 HMM >) 并选择适当数量的状态 (在 Hsp90 数据的情况下, 这意味着 < NumStates > = 5), 输入信号的维数 (< NumDims > = 3) 和输入类型 (< 输入类型 > = "烦恼 3D bgr")。
- 通过让软件收敛 hmm 的可能性来优化 hmm 的参数 (执行 "prepENS_CONVERGE_gB (GetDataFolder (1),-14)"), 直到转换矩阵中与上一个迭代相比的变化低于阈值 (10-14对于每个过渡概率的绝对更改的总和)。这将在正常的办公室 PC 上的一个小时内生成过渡概率的极大似然。
- 重复填充选择、高斯拟合和数据子集的 HMM 优化 (例如,完整数据集的 75%)。如果完整的数据集是从不同的实验中合并而成的, 那么对每个单独的实验也重复优化。子集分析允许估计人工人口选择的不确定性和数据集内的变异性。
- 计算了转换概率的 CI, 报告了数据集的异构性和 HMM 的精度。
- 通过执行 "cd $ (根: path3Dimport +" HMM) 来粗略估计 CI 边界;loop_getCI_estimate_limits ()。
- 通过执行以下操作计算 CI 的确切限制:
- "loop_getCI_HMM_converge (1)"
- "CIresults_conv_new ()"
- "cd:: HMM_CIresult;reportCI_conv () "
- "cd:: cmp_CI_conv;CI_plot2 ("嗯", doAppend=0) "
- 压缩可用的动力学信息以简化解释。
- 收集有关标记的核苷酸停留在 Hsp90 的时间的信息。通过折叠绑定到即插即用的状态 (即, s1, s2和 s3, 请参阅图 2) 并编译驻留时间直方图。执行 "cd $ (根: path3Dimport +" HMM ");collapse_states_get_DT ({01、11、0}) ", 它合并了状态 s 的0和 s4以及 s1、s2和 s3。
- 提取的停留时间从维特比路径的每一个利益状态, 并比较所产生的停留时间直方图为不同的实验条件。执行 "plot_collapsed_DT_Hist (wDTo_01110_record1)"。
- 对于更详细的图片, 折叠具有不同PF但功能相同的状态, 以缓解进一步的数据分析,例如, 在带有标记的 Hsp90 和核苷酸的三色实验中, 状态 S2和 s3可以折叠。执行 "collapse_states_get_DT ({01、22、4})"。
结果
多色 smFRET 测量允许直接检测两个或多个不同的交互站点之间的相关性。这使得技术独特的研究多组分系统, 如蛋白质复合物。我们专注于介绍一个三色的 smFRET 实验, 这是一个例证。
该方法的常规工作流显示在图 1中。第一部分包括在棱镜型 TIRF 显微镜上记录多色 smFRET 数据。图 2A?...
讨论
我们提出的实验程序, 以获得三色 smFRET 数据的复杂蛋白质系统和逐步描述的分析这些测量。这种方法提供了一种独特的可能性, 直接评估多个交互站点或构象变化之间的相关性。
为了在蛋白质上获得合适的多色单分子数据, 在低噪声水平下进行可重复测量是很重要的。这可以通过使用高效可靠的流室9的表面钝化协议来实现。一个足够密度的分子固定在表面, ...
披露声明
作者声明没有利益冲突。
致谢
这项工作由德国研究基金会 (研究所 39/969-1) 和欧洲研究理事会通过《紧急救济协调员赠款协定》681891资助。
材料
| Name | Company | Catalog Number | Comments |
| Setup | |||
| vibration-damped optical table | Newport, Irvine, CA, USA | RS2000 | |
| OBIS 473nm LX 75mW LASER | Coherent Inc, Santa Clara, CA, USA | 1185052 | |
| OBIS 532nm LS 50mW LASER | Coherent Inc, Santa Clara, CA, USA | 1261779 | |
| OBIS 594nm LS 60mW LASER | Coherent Inc, Santa Clara, CA, USA | 1233470 | |
| OBIS 637nm LX 140mW LASER | Coherent Inc, Santa Clara, CA, USA | 1196625 | |
| laser control unit | Coherent Inc, Santa Clara, CA, USA | 1234465 | Scientific Remote |
| aspheric telescope lenses | Thorlabs Inc, Newton, New Jersey, USA | d=25.4mm, f=50mm and f=100mm | |
| CF ex1 | AHF analysentechnik AG, Tübingen, Germany | ZET 473/10 | cleanup filter excitation |
| CF ex2 | AHF analysentechnik AG, Tübingen, Germany | ZET 532/10 | cleanup filter excitation |
| CF ex3 | AHF analysentechnik AG, Tübingen, Germany | ZET 594/10 | cleanup filter excitation |
| CF ex4 | Thorlabs Inc, Newton, New Jersey, USA | FL635-10 | cleanup filter excitation |
| DM ex1 | AHF analysentechnik AG, Tübingen, Germany | ZQ594RDC | dichroic mirror excitation |
| DM ex2 | AHF analysentechnik AG, Tübingen, Germany | 570DCXR | dichroic mirror excitation |
| DM ex3 | AHF analysentechnik AG, Tübingen, Germany | ZQ491RDC | dichroic mirror excitation |
| AOTFnC-Vis | AA Opto-Electronic, Orsay, France | ||
| λ/4 plate | Thorlabs Inc, Newton, New Jersey, USA | AQWP05M-600 | |
| CFI Apo TIRF 100x | Nikon Instruments Inc, Melville, NY, USA | high-NA objective | |
| piezo focus positioner MIPOS 250 CAP | piezosystem jena GmbH, Jena, Germany | Piezo Controller NV 40/1 CLE | |
| piezo stepper | Newport, Irvine, CA, USA | PZA12 | PZC200-KT NanoPZ Actuator Kit |
| achromatic aspheric lenses | Qioptiq Photonics GmbH & Co. KG, Göttingen, Germany | G322-304-000 | d=50mm, f=200mm |
| adjustable optical slit | Owis GmbH, Staufen i. Br., Germany | 27.160.1212 | max. aperture 12 x 12 mm |
| DM det1 | AHF analysentechnik AG, Tübingen, Germany | T 600 LPXR | dichroic mirror detection |
| DM det2 | AHF analysentechnik AG, Tübingen, Germany | H 560 LPXR superflat | dichroic mirror detection |
| DM det3 | AHF analysentechnik AG, Tübingen, Germany | HC BS R635 | dichroic mirror detection |
| BP det1 | AHF analysentechnik AG, Tübingen, Germany | 525/40 BrightLine HC | bandpass filter detection |
| BP det2 | AHF analysentechnik AG, Tübingen, Germany | 586/20 BrightLine HC | bandpass filter detection |
| BP det3 | AHF analysentechnik AG, Tübingen, Germany | 631/36 BrightLine HC | bandpass filter detection |
| BP det4 | AHF analysentechnik AG, Tübingen, Germany | 700/75 ET Bandpass | bandpass filter detection |
| optical shutters detection | Vincent Associates, Rochester, NY, USA | Uniblitz VS25S2T0 | |
| EMCCD iXon Ultra 897 | Andor Technology Ltd, Belfast, Northern Ireland | ||
| digital I/O card, PCIe-6535 | National Instruments, Austin, Texas, USA | ||
| syringe pump | Harvard Apparatus, Holliston, MA, USA | PHD22/2000 | |
| Name | Company | Catalog Number | Comments |
| Flow chamber | |||
| quartz slides | G. Finkenbeiner Inc, Waltham, MA, USA | Spectrosil2000, h=3mm | |
| TEGADERM film | 3M Deutschland GmbH, Neuss, Germany | 1626W | 10 x 12cm |
| spray adhesive | 3M Deutschland GmbH, Neuss, Germany | Photo Mount 050777 | |
| glycerol | Carl Zeiss AG, Oberkochen, Germany | Immersol G | |
| immersion oil | OLYMPUS EUROPA SE & CO. KG, Hamburg, Germany | IMMOIL-F30CC | |
| prism | Vogelsberger Quarzglastechnik GmbH, Hauzenberg, Germany | Suprasil1 | |
| aluminium prism holder | custom built | ||
| hollow setscrews | Thorlabs Inc, Newton, New Jersey, USA | with custom drilling | |
| Tygon S3 E-3603 tubing | neoLab Migge GmbH, Heidelberg, Germany | 2-4450 | ACF00001 |
| PTFE tubing | Bohlender GmbH, Grünsfeld, Germany | S1810-08 | |
| Name | Company | Catalog Number | Comments |
| Sample | |||
| yeast Hsp90 D61C, Q385C_biotin | UniProt ID P02829 | ||
| Maleimide derivatives of Atto488, Atto550 | ATTO-TEC GmbH, Siegen, Germany | ||
| AMP-PNP* | Jena Bioscience, Jena, Germany | γ-[(6-Aminohexyl)-imido]-AMP-PNP-Atto647N | |
| Fluospheres | Thermo Fisher Scientific, Waltham, MA, USA | F8764 | amine-modified, 0.2 μm, yellow-green fluorescent |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Andor Solis | Andor Technology Ltd, Belfast, Northern Ireland | version 4.30 | |
| LabVIEW | National Instruments, Austin, Texas, USA | version 2012, 32bit; misc. hardware control | |
| MDS control software | AA Opto-Electronic, Orsay, France | version 2.03a | |
| Coherent Connection | Coherent Inc, Santa Clara, CA, USA | version 3 | |
| Igor Pro | WaveMetrics Inc, Portland, OR, USA | version 6.37 |
参考文献
- Nooren, I. M. A., Thornton, J. M. Diversity of protein-protein interactions. EMBO J. 22 (14), 3486-3492 (2003).
- Marsh, J. A., Teichmann, S. A. Structure, dynamics, assembly, and evolution of protein complexes. Annu Rev Biochem. 84, 551-575 (2015).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Hohng, S., Joo, C., Ha, T. Single-Molecule Three-Color FRET. Biophys J. 87 (2), 1328-1337 (2004).
- Person, B., Stein, I. H., Steinhauer, C., Vogelsang, J., Tinnefeld, P. Correlated movement and bending of nucleic acid structures visualized by multicolor single-molecule spectroscopy. ChemPhysChem. 10 (9-10), 1455-1460 (2009).
- Lee, J., Lee, S., Ragunathan, K., Joo, C., Ha, T., Hohng, S. Single-molecule four-color FRET. Angew Chem Int Ed. 49 (51), 9922-9925 (2010).
- Ratzke, C., Berkemeier, F., Hugel, T. Heat shock protein 90's mechanochemical cycle is dominated by thermal fluctuations. Proc Natl Acad Sci U S A. 109 (1), 161-166 (2012).
- Ratzke, C., Hellenkamp, B., Hugel, T. Four-colour FRET reveals directionality in the Hsp90 multicomponent machinery. Nat Commun. 5, 4192 (2014).
- Götz, M., Wortmann, P., Schmid, S., Hugel, T. A Multicolor Single-Molecule FRET Approach to Study Protein Dynamics and Interactions Simultaneously. Methods Enzymol. 581, 487-516 (2016).
- Yengo, C. M., Berger, C. L. Fluorescence anisotropy and resonance energy transfer: Powerful tools for measuring real time protein dynamics in a physiological environment. Curr Opin Pharmacol. 10 (6), 731-737 (2010).
- Wortmann,P , ., Götz M, ., Hugel T , . Cooperative Nucleotide Binding in Hsp90 and Its Regulation by Aha1. Biophys J. 113, 1711-1718 (2017).
- Dörfler, T., Eilert, T., Röcker, C., Nagy, J., Michaelis, J. Structural Information from Single-molecule FRET Experiments Using the Fast Nano-positioning System. J Vis Exp. (120), e54782 (2017).
- Stephanopoulos, N., Francis, M. B. Choosing an effective protein bioconjugation strategy. Nature chemical biology. 7 (12), 876-884 (2011).
- Sánchez-Rico, C., Voith von Voithenberg, L., Warner, L., Lamb, D. C., Sattler, M. Effects of Fluorophore Attachment on Protein Conformation and Dynamics Studied by spFRET and NMR Spectroscopy. Chemistry (Weinheim an der Bergstrasse, Germany). , (2017).
- Roy, R., Hohng, S., Ha, T. A practical guide to single-molecule FRET. Nat Methods. 5 (6), 507-516 (2008).
- Lee, N. K., et al. Three-color alternating-laser excitation of single molecules: monitoring multiple interactions and distances. Biophys J. 92 (1), 303-312 (2007).
- Kapanidis, A. N., Lee, N. K., Laurence, T. A., Doose, S., Margeat, E., Weiss, S. Fluorescence-aided molecule sorting: analysis of structure and interactions by alternating-laser excitation of single molecules. Proc Natl Acad Sci U S A. 101 (24), 8936-8941 (2004).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chem Soc Rev. 43 (4), 1156-1171 (2014).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nat Methods. 14, 174-180 (2017).
- Rabiner, L. R. A tutorial on hidden Markov models and selected applications in speech recognition. Proc IEEE. 77 (2), 257-286 (1989).
- Fink, G. A. . Markov Models for Pattern Recognition. , (2014).
- Giudici, P., Ryden, T., Vandekerkhove, P. Likelihood-Ratio Tests for Hidden Markov Models. Biometrics. 56 (3), 742-747 (2000).
- Visser, I., Raijmakers, M. E. J., Molenaar, P. C. M. Confidence intervals for hidden Markov model parameters. Br J Math Stat Psychol. 53 (2), 317-327 (2000).
- McKinney, S. A., Joo, C., Ha, T. Analysis of Single-Molecule FRET Trajectories Using Hidden Markov Modeling. Biophys J. 91 (5), 1941-1951 (2006).
- Bronson, J. E., Fei, J., Hofman, J. M., Gonzalez, R. L., Wiggins, C. H. Learning Rates and States from Biophysical Time Series: A Bayesian Approach to Model Selection and Single-Molecule FRET Data. Biophys J. 97 (12), 3196-3205 (2009).
- Greenfeld, M., Pavlichin, D. S., Mabuchi, H., Herschlag, D. Single Molecule Analysis Research Tool (SMART): an integrated approach for analyzing single molecule data. PLoS ONE. 7 (2), e30024 (2012).
- Schmid, S., Götz, M., Hugel, T. Single-Molecule Analysis beyond Dwell Times: Demonstration and Assessment in and out of Equilibrium. Biophys J. 111 (7), 1375-1384 (2016).
- Taipale, M., Jarosz, D. F., Lindquist, S. HSP90 at the hub of protein homeostasis: emerging mechanistic insights. Nat Rev Mol Cell Biol. 11 (7), 515-528 (2010).
- Trepel, J., Mollapour, M., Giaccone, G., Neckers, L. Targeting the dynamic HSP90 complex in cancer. Nat Rev Cancer. 10 (8), 537-549 (2010).
- Wayne, N., Bolon, D. N. Dimerization of Hsp90 is required for in vivo function. Design and analysis of monomers and dimers. J Biol Chem. 282 (48), 35386-35395 (2007).
- Ali, M. M. U., et al. Crystal structure of an Hsp90-nucleotide-p23/Sba1 closed chaperone complex. Nature. 440 (7087), 1013-1017 (2006).
- Southworth, D. R., Agard, D. A. Species-dependent ensembles of conserved conformational states define the Hsp90 chaperone ATPase cycle. Mol Cell. 32 (5), 631-640 (2008).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proc Natl Acad Sci U S A. 101 (35), 12893-12898 (2004).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An Oxygen Scavenging System for Improvement of Dye Stability in Single-Molecule Fluorescence Experiments. Biophys J. 94 (5), 1826-1835 (2008).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Rognoni, L., Stigler, J., Pelz, B., Ylänne, J., Rief, M. Dynamic force sensing of filamin revealed in single-molecule experiments. Proc Natl Acad Sci U S A. 109 (48), 19679-19684 (2012).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in Surface-Tethered Lipid Vesicles as a New Tool for Single Biomolecule Spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-mode waveguides for single-molecule analysis at high concentrations. Science. 299 (5607), 682-686 (2003).
- Panaretou, B., et al. ATP binding and hydrolysis are essential to the function of the Hsp90 molecular chaperone in vivo. EMBO J. 17 (16), 4829-4836 (1998).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。