JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
3 색 단일 분자 무서 워를 사용 하 여 단백질 상호 작용의 상관 관계를 연구 하
요약
여기, 우리는 프로토콜 3 색 smFRET 데이터 및 3D 앙상블 숨겨진 마르코프 모델의 분석을 제시. 이 방법을 과학자 들은 복잡 한 단백질 시스템, cooperativity 또는 서로 상호 작용을 포함 하 여에서 운동 정보를 추출할 수 있습니다.
초록
단일 분자 포스터 공명 에너지 전달 (smFRET)의 생체 역학을 공부 하는 널리 사용 되 생물 기술 되고있다. 대 한 세포 단백질에 많은 분자 기계는 그들의 작업을 수행 하기 위해 기능 주기에서 상호 작용 협동자와 함께 행동 해야 합니다. 확장 다 색을 두 색 smFRET 가능 하 게 동시에 하나 이상의 상호 작용 또는 구조적 변화를 조사. 이 뿐만 아니라 smFRET 실험에 새로운 차원을 추가 하지만 그것은 또한 직접 이벤트의 시퀀스를 공부 하 고 고정된 샘플 및 총 내부 반사 형광을 사용 하 여 서로 상호 작용을 검출 하는 독특한 가능성을 제공 현미경 (TIRFM)입니다. 따라서, 멀티 컬러 smFRET 이전 unachievable 자세히 바이오 단지 양적 방법으로 공부에 대 한 다양 한 도구입니다.
여기, 단백질에 멀티 컬러 smFRET 실험의 특별 한 도전을 극복 하는 방법을 보여 줍니다. 자세한 프로토콜 데이터를 얻기 위해 및 운동 정보를 추출 하기 위한 선물이. 이 추적 선택 기준, 상태 분리 및 3D 앙상블 숨겨진 마르코프 모델 (HMM)를 사용 하 여 시끄러운 데이터에서 상태 궤적의 복구 포함 됩니다. 다른 방법에 비해 운동 정보는 복구 되지 면만 시간 히스토그램에서 하지만 직접는 흠. 최대 가능성 프레임 워크 운동 모델을 비판적으로 평가 요금에 대 한 의미 있는 불확실성을 제공 하 고 있습니다.
열 충격 단백질 90 (Hsp90) 우리의 메서드를 적용 하 여 우리는 뉴클레오티드 바인딩 및 단백질의 글로벌 구조적 변화를 풀 수 있습니다. Hsp90 이합체의 2 개의 뉴클레오티드 바인딩 포켓 사이 cooperativity를 직접 관찰할 수 있습니다.
서문
많은 단백질 구조적 변화 및 광범위 한 계획1,2,3과도 연결 하 여 중재 하는 다른 분자와 동적 단지에서 그들의 기능을 충족 해야 합니다. 외부 에너지 원 (예를 들어, ATP) 이러한 동적 상호 작용 기능에서 방향으로 이어질 수 있고 궁극적으로 셀, 인생에 대 한 전제 조건에에서 비 평형 안정 상태를 유지 하는 결합.
완전히 이해 분자 기계, 구조 연구에 의해 유도 정적 설명 충분 하지 않습니다. 또한, 기본 운동 모델의 지식을 하 고 운동 속도 상수를 결정 하기 위해 필수적입니다. 여러 가지 기존 방법을 허용 관심, 예를 들면, 표면 플라스몬 공명, 이완 메서드 (예: 점프 또는 중지 흐름 분 광 판독의 2 개의 분자 사이 상호 작용을 바이너리의 역학을 공부 하는 연구원 기술), 및 핵 자기 공명. 그러나, 그들의 적용은 대부분의 경우 간단한 2-상태 시스템 (예를 들어, 하나의 바운드와 언바운드 상태) 평균 하는 대량 실험에 제한. 경우 더 많은 상태 또는 중간체 관련, 그들은 속도 상수만 복잡 한 혼합물을 얻을. 광학 또는 마그네틱 핀셋 또는 2 색 smFRET, 즉, 하나의 기증자 등 표면 움직일 샘플 1 개의 수락자 fluorophore 단일 분자 방법 구조적 변화를 관찰 하는 모든에 대 한 속도 상수를 복구할 수 있습니다. 그러나, 그것은 하나 이상의 바인딩 사이트에 영향을 미치는 상호 작용에 관해서 라면, 이러한 방법을 유지 제한 고 가능한 상관 관계는 2 개 (이상)의 상호 작용에 정보만 실험 집합 간접적 결론을 통해 액세스할 수 있습니다.
실시간에 직접, 및에서 이러한 구성 요소 간의 상호 작용을 공부 하는 기회를 제공 하는 멀티 컬러 smFRET4,5,6,7,,89 근처 생리 조건10. 이 하나는 ligand 또는 다른 단백질8,,911의 구조에 종속적 바인딩 예를 들어 조사를 허용 합니다. 여기에 제시 된 전반적인 접근은 특정 위치, 측정 챔버의 표면에 1 개의 단백질을 연결 하 고 (대 한 자세한 내용은 참조 9 프리즘 형 TIRFM에 시간이 지남에 형광 강도 추적에 대 한 관심의 protein(s)를 , 12). 다른 염료의 공간 근접 다음 에너지 전송 그들 사이에서 확인할 수 있습니다. 전략 라벨 다를 수 있습니다 단백질에서 단백질 ( 13에서 검토) 및 지침을 smFRET 측정에서 아티팩트를 피하기 위해 존재14.
이후 기증자 염료 다른 수락자 염료 멀티 컬러 smFRET 실험에서 에너지를 전송할 수 있습니다, 모든 염료의 상대 위치 한 염료 혼자15,16의 여기에서 액세스가 불가능 합니다. 하지만 레이저 여기 (알렉스17, 그리고 검토에 18) 교체와 함께에서이 방법을 초 및 하위 나노미터의 해상도에서 모든 spatio 시간적 정보를 제공 합니다.
교장, 고해상도 구조 정보 간 염료 거리를 사용 하 여 달성 될 수 있다 알렉스 멀티 컬러 smFRET 실험에서 모든 형광 강렬의 조합에서 계산. 그러나, 여기 우리가에 초점을 어디 다 색 smFRET은 필수 운동 모델의 추출 뿐만 아니라 상태 식별 및 분리. 삼각 측량에 의하여 구조 결심 "만" 원할 때 높은 신호 대 잡음 비율을 간단 하 게 2 색 smFRET 실험의 세트 수행된12,19될 수 있습니다.
우리가 사용 하는 부분 형광 ( ) 두 fluorophores7사이의 에너지 전송에 대 한 프록시로 서. PF 는 2 색 실험의 무서 워 효율 유사한 형광 강도에서 계산 됩니다.
) 두 fluorophores7사이의 에너지 전송에 대 한 프록시로 서. PF 는 2 색 실험의 무서 워 효율 유사한 형광 강도에서 계산 됩니다.

어디, 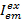 방출 채널 em 에서 강도 색 전,와 여기 이후 이며 c 는 가장 긴 파장을 가진 수락자. 검색 채널 샘플 챔버 하지만 형광 빛의 기록 다른 스펙트럼 범위에서 동일한 위치를 나타냅니다. 여기 및 방출에 대 한 동일한 식별자가이 프로토콜에 사용 됩니다 (즉, "파랑," "녹색," 및 "빨강").
방출 채널 em 에서 강도 색 전,와 여기 이후 이며 c 는 가장 긴 파장을 가진 수락자. 검색 채널 샘플 챔버 하지만 형광 빛의 기록 다른 스펙트럼 범위에서 동일한 위치를 나타냅니다. 여기 및 방출에 대 한 동일한 식별자가이 프로토콜에 사용 됩니다 (즉, "파랑," "녹색," 및 "빨강").
실험적인 단점 때문에 측정 된 형광 강렬은 에너지 전송에 뿐만 아니라 fluorophore 및 설정 속성에 따라 달라 집니다. 두 fluorophores 사이의 진정한 에너지 전송 효율을 얻기 위하여 측정된 농도 수정 해야 합니다. 다음 절차는 참조9를 기반으로 합니다. 명백한 누설 (lk, 즉, 다른 염료에 대 한 지정 된 채널에 fluorophore에서 광자의 탐지) 및 명백한 감마 보정 요인 (ag, 즉, 염료의 형광 양자 수율 및 채널의 검출 효율) 이벤트를 표백 하는 수락자를 표시 하는 단일 분자 추적에서 얻을 수 있습니다.
모든 가능한 수락자 채널에 기증자 염료의 누설은 수락자 염료 표백 하지만 기증자 여전히 형광 기록된 형광 추적에서 모든 데이터 요소에서 계산 (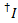 ):
):
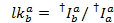
누설 히스토그램의 중간값은 명백한 누설 요소로 사용 됩니다. 보정 후 누설에 대 한, 명백한 감마 요소 추적의 동일한 세트에서 결정 됩니다. 그것은 기증자 채널 수락자 염료의 표백 시에 형광의 변화에 의해 수락자 채널에서 형광의 변화를 나누어 계산 됩니다.

여기서 c 는 다시 긴 파장을 가진 수락자에 대 한 감지 채널입니다. 결과 분포의 중앙값 명백한 보정 요소로 사용 됩니다.
각 채널에서 수정 된 농도 의해 얻을 수 있습니다.
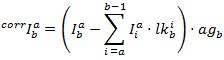
PF 는 다음에 따라 계산 됩니다.

다른 인구 PFs에 다차원 공간에서 분리 될 수 있다. 위치와 각 상태의 폭 피팅 다차원 가우스 기능 데이터에 의해 결정 됩니다. 모든 PF 흔적을 기반으로 한 글로벌 흠의 후속 최적화 관찰된 활동의 양적 설명을 제공 합니다. 속도의 심지어 작은 변화는 감지.
HMMs 시끄러운 시간 추적의 컬렉션에서 상태 모델을 유추 하는 방법을 제공 합니다. 시스템은 어떤 주어진된 시간에 실제 관측 (즉, 방출) 숨겨진된 상태는이 숨겨진된 상태20의 확률 함수 이산 집합 중 하나에 있을 여겨진다. TIRFM smFRET 데이터의 경우는 방출 확률 b나 주 나 당 연속 가우스 확률 밀도 함수에 의해 모델링 될 수 있습니다. 정기적으로 간격 둔된 개별 시간 포인트에서 다른 상태에서 전환 시간 불변 이며만 현재 상태에 따라 전환 확률에 따라 발생할 수 있습니다. 전환 행렬 A 이 전환 확률 ij 모든 숨겨진된 상태 사이 포함 되어 있습니다. 초기 상태 분포  국가 특정 확률을 제공
국가 특정 확률을 제공 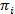 시간 추적의 처음으로 포인트. 최대 가능성 접근을 사용 하 여, 가장 앞으로 뒤로 하 고 바 움-웰 치 알고리즘20,21을 사용 하 여 데이터를 설명 하기 위해 이러한 매개 변수를 최적화할 수 있습니다. 이 최대 가능성 평가 인 (MLE)을 생성합니다. 마지막으로, 가장 가능성이 관측의 궤적 생성 상태 시퀀스 Viterbi 알고리즘 유추 수 있습니다. SmFRET 데이터24,,2526 의 다른 흠 분석 달리 우리가 사용 하지 않는 한 단순한 "부드럽게" 데이터 하지만 필요 없이 데이터 집합에서 추출 운동 상태 모델의 피팅 유지 시간으로 흠 히스토그램27 흠 분석 자체 스크립트 이고르 프로 사용 하 여 이루어집니다. 코드의 구현은 참조21을 기반으로 합니다. 제공 하는 소프트웨어 키트 모범적인 데이터 우리의 웹 페이지에 따라 섹션 5와 6이이 프로토콜 (https://www.singlemolecule.uni-freiburg.de/software/3d-fret)의 순서. 전체 소프트웨어는 요청 시 이용 가능.
시간 추적의 처음으로 포인트. 최대 가능성 접근을 사용 하 여, 가장 앞으로 뒤로 하 고 바 움-웰 치 알고리즘20,21을 사용 하 여 데이터를 설명 하기 위해 이러한 매개 변수를 최적화할 수 있습니다. 이 최대 가능성 평가 인 (MLE)을 생성합니다. 마지막으로, 가장 가능성이 관측의 궤적 생성 상태 시퀀스 Viterbi 알고리즘 유추 수 있습니다. SmFRET 데이터24,,2526 의 다른 흠 분석 달리 우리가 사용 하지 않는 한 단순한 "부드럽게" 데이터 하지만 필요 없이 데이터 집합에서 추출 운동 상태 모델의 피팅 유지 시간으로 흠 히스토그램27 흠 분석 자체 스크립트 이고르 프로 사용 하 여 이루어집니다. 코드의 구현은 참조21을 기반으로 합니다. 제공 하는 소프트웨어 키트 모범적인 데이터 우리의 웹 페이지에 따라 섹션 5와 6이이 프로토콜 (https://www.singlemolecule.uni-freiburg.de/software/3d-fret)의 순서. 전체 소프트웨어는 요청 시 이용 가능.
PF 데이터에서 포인트 시간 <-1 또는 PF > 2 어떤 검색 채널에 모든 국가 (10-200)에 대 한 최소한의 방출 확률 할당 됩니다. 이러한 데이터 요소에 인공 전환이 되지 않습니다.
5.7 단계에서 설명한 대로 방출 확률에 대 한 매개 변수 가우스 기능 3D PF 히스토그램의 적합에서 얻을 수 있습니다. 이 매개 변수는 흠의 최적화 하는 동안 고정 유지 됩니다.
제시 방법에서 초기 상태로 배포 벡터와 전환 매트릭스 사용 됩니다 세계적으로 추적의 전체 앙상블을 설명 하. 그들은 참조27에 따라 데이터 집합에서 모든 N 분자에 따라 업데이트 됩니다.
초기 상태 분포에 대 한 시작 매개 변수 (단계 5.3) PF 히스토그램의 2D 계획에서 결정 됩니다 고 전환 확률 설정 됩니다 확률 제외 0.05 동일한 상태를 유지 하기 등을 선택 하는 특정 상태를 두고 확률 화합과 정규화 됩니다.
가능성 프로 파일링 방법 모든 전환 속도21,22, 그들의 불확실성에 대 한 의미 있는 견적으로 봉사 하는 자신감을 간격 (CIs)에 게 하는 데 사용 됩니다. 특정 비율에 대 한 CI의 범위를 계산 하려면 전환 확률의 MLE 이외의 값으로 고정 됩니다. 이 테스트 모델 λ 생성 '. 가능성의 가능성 비율 (LR) 테스트 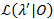 데이터 집합 주어진 0 에 따라 수행 됩니다.
데이터 집합 주어진 0 에 따라 수행 됩니다.
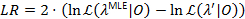
바인딩된 매개 변수 LR 3.841는 x2의 95% 논집을 초과 하는 경우에 도달 하면에 대 한 95% 신뢰-한 자유도22,23분포.
방법의 파워는 Hsp90를 사용 하 여 증명 됩니다. 풍부한 단백질이 박테리아 및 진핵생물에서 발견 되 고 세포질 긴장 응답28의 일부입니다. 그것은 암 치료29유망 약 대상입니다. Hsp90 homodimer 각 소 단위30의 N 맨끝 도메인에 하나의 뉴클레오티드 바인딩 포켓입니다. 그것은 적어도 두 개의 전역 고유 conformations, 폐쇄 하나 하나의 N-터미널 오픈, V 자형 구조19,31,32사이 영상 효과 받을 수 있습니다. 직접 dimeric 자연 Hsp90에 두 개의 뉴클레오티드 바인딩 사이트 사이 상호 작용의 문제를 발생 시킵니다.
다음에, 우리는 데이터 수집 및 효 모 Hsp90 그리고 뉴클레오티드에 3 색 smFRET 실험의 분석에 대 한 단계별 프로토콜을 제공. 붙일 레이블 앰프 PNP의 구조에 종속적 바인딩 (앰프 PNP *, ATP의 비 hydrolyzable 아날로그) 분석 된다. 설명 된 절차의 응용 프로그램 허용 뉴클레오티드 바인딩 및 동시에 연구 Hsp90의 구조적 변화 하 고 그로 인하여 Hsp90의 2 개의 뉴클레오티드 바인딩 포켓 사이 cooperativity를 보여준다.
프로토콜
1. 설치 및 필수 구성 요소
- 프리즘 형 TIRFM에 멀티 컬러 smFRET 측정을 수행 합니다. 2 색상 설정의 설명 정돈 간행물 주어진 대로12참조.
- 멀티 컬러 TIRFM 생성 합니다. 일반 레이아웃은 9에 상세 하다.
- 사용 전환, 다이오드 여기 경로 있는 기계적인 셔터의 사용을 불필요 한 렌더링 솔리드 스테이트 연속파 레이저를 펌핑.
- 목표 입력을 후면에서 여기 광선의 후면 반사를 방지 하는 비대칭, 길쭉한 프리즘을 사용 합니다.
- 2 인치 색 비구 융합 된 실리 카 렌즈를 사용 하 여 최대한 많은 빛을 수집 하 고 자동 형광 및 착오, 예를 들어, 중앙 지역에는 이미지의 왜곡을 방지 하는 검색 경로 있는.
- 별도 렌즈를 가진 EMCCD의 칩에 초점을 각 검색 경로. 각 탐지 채널의 최적의 초점을 맞추고 있습니다.
주의: 클래스 3B 레이저는 TIRFM에 사용 됩니다. 즉, 그들은 위험한 경우는 눈에 직접 노출 확산 반사 유해 하지 않습니다. 시스템을 운영 하기 전에 지방 정부 규정에 따라 안전 조치 준수 레이저를 확인 합니다.
- 미리 dsDNA 샘플을 사용 하 여 설치 및 fluorophore 속성에 대 한 보정 요소를 결정 합니다.
- 각 염료 조합에 대 한 하나의 높은 무서 워 dsDNA 샘플을 사용 하 여 긴 여기 파장을 갖는 수락자와 (제시 설치: Atto488-Atto647N, Atto550 Atto647N, Atto594-Atto647N). DNA는 biotin과 수정 또한 다는 것을 확인 하십시오.
- 5 샘플을 희석 nM TNM 버퍼 (5 mM Tris pH 7.5, 5 m m NaCl, 20 mM MgCl2)와 2 mM Trolox (이 버퍼 사용도 측정).
- Hsp90 2.5, 2.7 단계에 설명 된 대로 dsDNA를 무력화.
- 명백한 누설 (lk)와 수락자 표백 이벤트를 표시 하는 단일 분자 추적에서 명백한 감마 (ag)에 대 한 보정 요소를 계산 합니다.
- 못/비오 틴-말뚝 세 석 영 슬라이드의 샌드위치는 흐름 챔버, 접착 면에 커버 슬립은 박막을 생성 합니다. 석 영의 청소에 대 한 세부 프로토콜에 대 한 슬라이드와 패 시 베이 션 참조9을 참조 하십시오.
- 사용 두께 (3mm) 석 영 기하학적으로 프리즘 및 기능성된 석 영 슬라이드 사이의 석 영 석 영-글리세롤 인터페이스에서 흩어져 레이저 빛의 수집을 방지 하기 위해 슬라이드.
- 한 얇은 (40 µ m)를 사용 하 여 씰링 접착 비 접착 면에 분무는 영화. 박막 표면에 부착 된 분자와 목표 사이 거리를 감소 시킨다. 80 ° C에가 열 하 고 누릅니다.
- 상단에 커버 슬립을 놓습니다. 80 ° C에가 열 하 고 누릅니다. 프리즘을 통해 흐름 챔버를 배치할 때 글리세롤의 방울을 사용 합니다.
참고: 글 리세 린 뿐만 아니라 프리즘 및 석 영 슬라이드 자료는 일치 하는 인덱스.
- Hsp90 D61 또는 Q385 위치에서 두 명의 단일 시스테인 점 돌연변이의 형태로 Saccharomyces cerevisiae 에서 표현 한다. Picomolar 농도에서 이합체의 분리를 방지 하기 위해 C 터미널 코일 코일 모티브를 추가 합니다. 별도로 돌연변이 단백질을 분류 하 고 단위체 아미노산 위치 61에서 Atto488와 Atto550 아미노산 위치 3859에서 표시 하는 heterodimers를 교환 한다.
2입니다. 측정
- 카메라 소프트웨어를 시작 하 고 아래 지정 된 대로 이미지 매개 변수를 설정 합니다.
- 가능 (외부 물 냉각을 가진-95 ° C) 낮은 어두운 전류 잡음 감소를 냉각 하는 센서에 대 한 온도 설정 합니다.
- 최적화 된 카메라 설정을 사용 하 여 단일 분자 기록: 3.3 µs 수직 이동 속도, 정상적인 수직 시계 전압, 17의 16-비트 수평 읽어, 사전 증폭 이득 3, 전자 증 1000의 이득.
- 750 수집 사이클의 길이와 "외부"를 수집 하 고 70 양 기록 영화 노출 시간의 발생을 설정 합니다.
참고: 카메라는 EMCCD 센서의 포화를 방지 하기 위해 취득 하는 때 방 빛을 해제 합니다.
- 측정에 대 한 로컬 SSD에 대 한 폴더를 만듭니다. 소프트웨어에서 설정 < 자동 저장 > 라이더, < 자동 저장 > 활성화 이동한 "Tiff" 영화 수집을 위한 파일 포맷을 선택 합니다. 자동 저장 위치는 SSD에 폴더를 선택 합니다.
- 검색 경로, 및 카메라에서 셔터 제어 acousto 광학 가변 필터 (AOTF), 레이저의 작동을 제어 하는 소프트웨어와 레이저, AOTF, 동기화 트리거 소프트웨어 소프트웨어를 시작 합니다. AOTF (ca. 3 mW 프리즘에 들어가기 전에)와 레이저 파워를 조정 하 고 올바른 트리거 패턴을 로드.
- 프리즘와 흐름 챔버 샘플 홀더 탑재, 튜브를 연결, 입구 배관 microcentrifuge 컵에 놓고 주사기 펌프 출구 튜브를 연결 합니다. 약 150 µ L 버퍼와 챔버를 플러시, 여기를 정렬 광선과 초점. 약 10의 레이저 파워 슬라이드의 완전 한 감지 범위를 따라 천천히 이동 하 여 표면에 어떤 형광 오염 물질을 표백 모든 레이저에 대 한 mW. 이 약 1 시간 걸립니다.
참고: 그렇지 않으면 다르게 진술, 사용된 버퍼 포함 40 mM HEPES pH 7.5, 150 m KCl, m 그리고 10 mM MgCl2합니다. - 실로 NeutrAvidin 솔루션 (버퍼에서 0.25 mg/mL)의 약 300 µ L을 플러시 하 고 챔버를 통해 BSA 솔루션 (버퍼에서 0.5 mg/mL)의 1 분 버퍼와 플러시 약 300 µ L와 언바운드 NeutrAvidin 밖으로 플러시에 대 한 품 어.
참고:이 블록 표면 기능화 남은 표면에 BSA의 불특정 흡착에 의해 결함. - biotinylated의 대략 150 µ L와 흐름 챔버 로드 하 여 샘플을 무력화와 상승 농도 (버퍼 + 0.5 mg/mL BSA에 희석) Hsp90 표시는 일반적으로 농도에 경우 충분 한 표면 밀도 도달할 때까지 5-10 오후. 약 300 µ L 버퍼 + 0.5 mg/mL BSA 언바운드 단백질 세척 한다.
- 25의 150 µ L에 플러시 앰프 PNP nM * 버퍼 + 0.5 mg/mL BSA. 그것은 5 분 동안 품 어 정확한 뉴클레오티드 농도 보장 하기 위해이 단계를 한 번 반복 하자.
참고: 추가, 레이블 없는 앰프 PNP 존재 실험, 또한 추가 250 µ M 앰프 PNP. - 데이터 수집으로 시작 합니다. 데이터의 적절 한 금액에 대 한 약 1.5 시간 걸립니다 약 20 영화 취득.
- 보기의 필드를 변경 하려면 피 스테퍼와 여기 빔에 수직인 샘플 챔버의 위치를 이동 합니다.
- 필요한 경우는 목표의 높이 제어 하는 z-피와 초점을 조정 합니다. 이 해서는 안됩니다 필요한 측정 챔버 성향 없이 탑재 되어 때에 너무 자주.
- 카메라 소프트웨어에서 < 걸릴 신호 >를 눌러 카메라의 녹화를 준비 하 고 트리거 소프트웨어에서 < 시작 > 버튼을 사용 하 여 여기/수집 주기 시작. 이 형광 강도의 수집을 시작합니다.
- 첫 번째 설치의 모든 검색 채널의 스펙트럼 범위에서 형광 방출 표시 형광 구슬과 영화 녹음 채널 등록을 수행 합니다. 다음, 밝은 반점에 대 한 검색 하 여 교정 영화에 구슬 위치를 감지 하 고 중앙 위치는 가우스에서 강도 프로 파일에 맞게 결정 합니다. 모든 채널에서 발견 되 고 x-및 y-방향 학위 39의 2 차원 다항식으로 매핑 오프셋을 맞는 구슬의 좌표를 저장 합니다.
3입니다. 단일 분자 추적의 선택
- 데이터 분석 자체 스크립트 이고르 프로 사용 하 여 이루어집니다. "IniTIRF.ipf"를 열어서 모든 필요한 스크립트를 로드 하 고 올바른 유형의 실험을 선택 합니다.
참고: 다음에, < 버튼 > 메뉴 또는 사용자 인터페이스에서 클릭할 수 있는 요소를 지정 합니다. 함수 호출에 따옴표, 예를 들어, "인쇄" Hello World "" 표시 됩니다. 이 명령은 (바깥쪽 따옴표) 없이 이고르 프로의 커맨드 라인에 붙여넣을 수 있습니다. - 검색 채널 등록에 대 한 매개 변수 로드는 다는 것을 확인 하십시오.
- 클릭 하 여 GUI를 시작 < 새로운 smFRET | 분석 GUI >.
- 각 채널에는 강도 영화 (즉, 16 비트 TIFF 스택을 저장 512 x 512 픽셀 프레임의 시퀀스)를 로드 합니다. < 부하 영화 > 버튼을 누르면 다른 소위 "마스터"와 "슬레이브" 카메라 하나에서에서 파일을 선택 하 여 이렇게 합니다.
- 특정 검색 채널에 처음 다섯 프레임의 합계에 있는 밝은 반점에 대 한 검색 하 여 잠재적인 단일 분자의 위치를 식별 합니다. 채널 매핑에서 다른 모든 검색 채널에서 해당 위치를 계산 합니다. 형광 강도 추적 얻으려면, 각 프레임에 대 한 중앙 위치 주위 픽셀 사각형의 합계. GUI에서 < 찾을 추적 > 버튼을 누르면 이렇게 됩니다.
참고: (에서 픽셀) 광장의 옆 길이 의해 주어진 다: 2 * < 합계 Pxs > + 1. - 각 분자에 대 한 공동 원시 강도 추적 같은 여기 색이 자리의 모든 흔적의 합으로 계산 합니다. 평가 다음과 같은 기준에 대 한 모든 채널에 분자의 강도 프로필:
- 모든 여기 색상, 적절 한 검색 채널에서 anticorrelated 동작, 빨간 형광의 탐지에 대 한 공동 원시 강도 및 단일 표백에 대략 편평한 고원 단계 (무서 워 앰프 PNP 바운드를 나타내는 *) 적어도 한 번 내는 추적, 그리고 두 개의 앰프 PNP의 존재를 나타내는 것이 빨간 형광에 없는 여러 단계 * 1 Hsp90 이합체에 바인딩된.
- 이러한 기준을 충족 하는 경우 추가 분석을 위해의 형광 흔적을 저장 합니다. 커서 추적을 선택 하 고 다음 "타임 라인" 그래프에서 < 저장 > 버튼을 누르면 이렇게 됩니다. 수동으로 블루 여기 영화 당 약 200 분자에 대 한 후에 모든 3 개의 탐지 채널 강도 추적 검사.
4입니다. 부분 형광 흔적의 계산
- 저장 된 분자 중 하나에 대 한 모든 형광 강도 추적을 표시 합니다. 그래프에서 커서를 사용 하 여 시간 범위를 선택 합니다.
- 모든 fluorophores 이미 표백은 시간 간격을 선택 합니다. 평균 배경 농도이 범위에서 계산 하 고 각 채널에서 강도 추적에서 뺍니다. < 배경 > 버튼을 누르면 이렇게 됩니다.
- 적어도 두 염료 Hsp90 연결할 무서 워 효율성 범위 선택 (Atto488 및 Atto550). 깜박이 이벤트를 포함 하는 추적을 제외 확인 ( 그림 3B참조). 이러한 이벤트는 다른 채널에서 동반 증가 없이 한 채널에 드롭 형광 강도 특징.
- PF 추적 계산 합니다. < PF Calc > 버튼을 누르면 이렇게 됩니다. 명백한 누설 (lk) 그리고 명백한 감마 (ag)에 대 한 미리 정의 된 수정 요인 사진-물리에 대 한 해결 하기 위해 원시 강도에 적용 됩니다 및 속성 설정.
5. 인구 선택 및 3 차원 히스토그램 피팅
- PF 추적에 낮은 신호 대 잡음 비율을 표시 하는 분자를 제거 합니다. 분자는 간격 [-1, 2] PF 흔적에는 프레임의 10%를 초과 하는 데이터 집합에서 제거 됩니다. 명령 창에서 "RemoveTracesLowSNR()"를 실행 하 여 이렇게 합니다.
- 공용 폴더 데이터의 범주화 된 2D 계획을 계산 합니다. 플롯
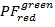 통해
통해 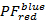 및
및 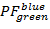 통해
통해 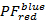 100 x 100 쓰레기통의 해상도 범위 [-0.5; 1.5]에서. 이렇게 하려면 실행:
100 x 100 쓰레기통의 해상도 범위 [-0.5; 1.5]에서. 이렇게 하려면 실행:- "HistFret2D ("r_b","r_g", binHist = 100)"
- "HistFret2D ("r_b","g_b", binHist = 100); MoveWindow 553.5, 42.5, 1055.25, 508.25"
- 2D 계획에 각 구별 국가의 상대 인구를 결정 합니다.
- 앞으로 적절 한 그래프를가지고 하 고 "panelHist2DCount()"를 실행 합니다.
- < 초기화 > 버튼 누르고 피크 주위 직접 다각형을 그립니다.
- < 카운트 > 버튼을 클릭 합니다. 데이터 요소에는 다각형의 수와 프로젝션에 데이터 포인트의 총 수는 명령 창에서 인쇄 됩니다.
- 실행 하 여 공용 폴더 데이터의 3 차원 히스토그램을 준비 "HistFret3D ("g_b","r_b","r_g")".
- 1의 전체를 3 차원 히스토그램을 정상화. 다음 실행:
- "NewDataFolder/S fit0"
- "중복/O:: 무서 워: Hist3D, Hist3D"
- "가변 /G div = sum(Hist3D)*(DimDelta(Hist3D,0)) ^3"
- "Hist3D / div; = 인쇄 사업부 "
- 3 차원 가우스에 대 한 초기 매개 변수 적합 하 고 필요한 데이터 구조를 준비를 제공 합니다.
- "Gauss3D_initParam(); 실행 W_coef_old 편집. "
- 매개 변수 벡터의 끝에 국가 인구를 추가 합니다.
- "Gauss3D_prepareFit()"를 실행
참고: W_coef_old는 적합을 위한 초기 매개 변수를 보유 하는 벡터 이다. 즉 국가 당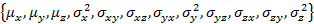 및 벡터의 끝에 연결 상태 인구. 공분산 행렬은 대칭 다는 것을 확인 하십시오.
및 벡터의 끝에 연결 상태 인구. 공분산 행렬은 대칭 다는 것을 확인 하십시오.
- S 구별할 수 주의 수와 3D PF 히스토그램에 S 3 차원 가우스 함수의 합계를 맞습니다.
- "Do3D()"를 실행 이 초기 매개 변수의 품질에 따라 정상적인 사무실 PC에 한 시간 이상 걸릴 수 있습니다.
- "PostprocessFitMultiGauss3D(); 실행 evalFitMultiGauss3D(); W_coef 편집. "
- 맞는 결과 표시 합니다. 두 개의 2D 계획의 각각에 대해 다음 명령을 사용 하 여:
- "contourPF3D_new(0); contourPF3D_new(1); contourPF3D_new(2); contourPF3D_new(3); contourPF3D_new(4) "
- "contourPF3D_colorize()"
6. 운동 분석 3D 앙상블과 흠
- 앙상블 흠 운동 정보를 추출 실행을 준비 합니다. 한 흠 데이터 집합에 있는 모든 분자에서 최적화 되어 있습니다. 이전 단계에서 얻은 정보를 사용 하 여 3D PF 공간에서 위치와 각 상태의 폭 정의 하.
- 흠 사용자 인터페이스 초기화 (< 흠 | 초기화 흠 >) 국가의 적절 한 수를 선택 하 고 (Hsp90 데이터의 경우 즉 < NumStates > = 5), 입력 신호 크기의 숫자 (< NumDims > = 3), 및 입력의 유형이 (< 입력 형식 > = "무서 워 3D bgr").
- 소프트웨어는 흠의 가능성을 수렴 시키는 여는 HMM의 매개 변수를 최적화 (실행 "prepENS_CONVERGE_gB(GetDataFolder(1),-14)") 이전 반복에 비해 전환 매트릭스에 변경 임계값 (10-14이하로 떨어질 때까지 각 전환 확률에 대 한 절대 변경의 합계에 대 한). 이 정상적인 사무실 PC에 한 시간 정도 전환 가능성에 대 한 MLE를 생성합니다.
- 데이터 (예: 전체 데이터 세트의 75%)의 하위 집합에 대 한 인구 선택, 가우스 피팅 및 흠 최적화를 반복 합니다. 전체 데이터 집합은 다른 실험에서 병합 하는 경우 각각 하나의 실험에 대 한 최적화도 반복 합니다. 하위 집합의 분석 수동 채우기 선택 하 고 데이터 집합 내의 가변성의 불확실성을 추정을 허용 합니다.
- 데이터 집합이 고는 흠의 정밀도 대 한 보고서 전환 가능성에 대 한 CI를 계산 합니다.
- "Cd $(루트: path3Dimport +"흠");를 실행 하 여 CI 범위의 대략적인 견적을 얻을 loop_getCI_estimate_limits()입니다. "
- 실행 하 여는 CI의 정확한 한계를 계산:
- "loop_getCI_HMM_converge(1)"
- "CIresults_conv_new()"
- "cd:: HMM_CIresult; reportCI_conv() "
- "cd:: cmp_CI_conv; CI_plot2 ("흠", doAppend = 0) "
- 해석을 단순화 하기 위해 사용할 수 있는 운동 정보 응축
- 레이블이 지정 된 뉴클레오티드 유지 Hsp90 바인딩된 시간에 대 한 정보를 수집 합니다. 앰프-PNP에 바인딩된 상태를 축소 하 여이 작업을 수행 * (즉, S1, S2 와 S3을 또한 그림 2참조) 면만 시간 히스토그램을 컴파일합니다. "Cd $(루트: path3Dimport +"흠");를 실행합니다 collapse_states_get_DT({0,1,1,1,0}) ", S0 와 S4 와 S1, S2및 S3상태는 결합의 거주.
- 관심의 각 상태에 대 한 Viterbi 경로에서 면만 번을 추출 하 고 다른 실험 조건에 대 한 결과 거 시간 히스토그램을 비교. "Plot_collapsed_DT_Hist (wDTo_01110_record1)"를 실행
- 더 자세한 사진에 대 한 뚜렷한 PF 하지만 기능적으로 추가 데이터 분석, 예를 들어, 레이블이 Hsp90 그리고 뉴클레오티드, 미국 S2 와 S 3 색 실험의 경우 쉽게 동일한 상태 축소 3 축소 될 수 있습니다. "Collapse_states_get_DT({0,1,2,2,4})"를 실행
결과
멀티 컬러 smFRET 측정 두 개 이상의 고유한 상호 작용 사이트 간의 상관 관계의 직접적인 탐지를 허용 한다. 이 기술은 다중 구성 요소 시스템, 단백질 복합물을 조사에 고유한 렌더링 합니다. 우리는 여기에, 설명 예 역할 3 색 smFRET 실험의 프레 젠 테이 션에 초점.
방법의 일반적인 워크플로 그림 1에 표시 ...
토론
우리는 복잡 한 단백질 시스템 및이 측정 분석에 대 한 단계별 설명에 대 한 3 색 smFRET 데이터를 얻기 위해 실험 절차를 제시. 이 방법은 직접 여러 상호 작용이 사이트 또는 구조적 변화 간의 상관 관계를 평가 하기 위해 고유한 가능성을 제공 합니다.
단백질에 적합 한 멀티 컬러 단일 분자 데이터를 얻기 위해 그것은 저 잡음 수준에서 재현 가능한 측정을 수행 하는 것이 중요...
공개
저자는 관심 없음 충돌 선언합니다.
감사의 말
이 작품은 독일 연구 재단 (INST 39/969-1)와 ERC 부여 계약 n. 681891 통해 유럽 연구 위원회에 의해 자금을.
자료
| Name | Company | Catalog Number | Comments |
| Setup | |||
| vibration-damped optical table | Newport, Irvine, CA, USA | RS2000 | |
| OBIS 473nm LX 75mW LASER | Coherent Inc, Santa Clara, CA, USA | 1185052 | |
| OBIS 532nm LS 50mW LASER | Coherent Inc, Santa Clara, CA, USA | 1261779 | |
| OBIS 594nm LS 60mW LASER | Coherent Inc, Santa Clara, CA, USA | 1233470 | |
| OBIS 637nm LX 140mW LASER | Coherent Inc, Santa Clara, CA, USA | 1196625 | |
| laser control unit | Coherent Inc, Santa Clara, CA, USA | 1234465 | Scientific Remote |
| aspheric telescope lenses | Thorlabs Inc, Newton, New Jersey, USA | d=25.4mm, f=50mm and f=100mm | |
| CF ex1 | AHF analysentechnik AG, Tübingen, Germany | ZET 473/10 | cleanup filter excitation |
| CF ex2 | AHF analysentechnik AG, Tübingen, Germany | ZET 532/10 | cleanup filter excitation |
| CF ex3 | AHF analysentechnik AG, Tübingen, Germany | ZET 594/10 | cleanup filter excitation |
| CF ex4 | Thorlabs Inc, Newton, New Jersey, USA | FL635-10 | cleanup filter excitation |
| DM ex1 | AHF analysentechnik AG, Tübingen, Germany | ZQ594RDC | dichroic mirror excitation |
| DM ex2 | AHF analysentechnik AG, Tübingen, Germany | 570DCXR | dichroic mirror excitation |
| DM ex3 | AHF analysentechnik AG, Tübingen, Germany | ZQ491RDC | dichroic mirror excitation |
| AOTFnC-Vis | AA Opto-Electronic, Orsay, France | ||
| λ/4 plate | Thorlabs Inc, Newton, New Jersey, USA | AQWP05M-600 | |
| CFI Apo TIRF 100x | Nikon Instruments Inc, Melville, NY, USA | high-NA objective | |
| piezo focus positioner MIPOS 250 CAP | piezosystem jena GmbH, Jena, Germany | Piezo Controller NV 40/1 CLE | |
| piezo stepper | Newport, Irvine, CA, USA | PZA12 | PZC200-KT NanoPZ Actuator Kit |
| achromatic aspheric lenses | Qioptiq Photonics GmbH & Co. KG, Göttingen, Germany | G322-304-000 | d=50mm, f=200mm |
| adjustable optical slit | Owis GmbH, Staufen i. Br., Germany | 27.160.1212 | max. aperture 12 x 12 mm |
| DM det1 | AHF analysentechnik AG, Tübingen, Germany | T 600 LPXR | dichroic mirror detection |
| DM det2 | AHF analysentechnik AG, Tübingen, Germany | H 560 LPXR superflat | dichroic mirror detection |
| DM det3 | AHF analysentechnik AG, Tübingen, Germany | HC BS R635 | dichroic mirror detection |
| BP det1 | AHF analysentechnik AG, Tübingen, Germany | 525/40 BrightLine HC | bandpass filter detection |
| BP det2 | AHF analysentechnik AG, Tübingen, Germany | 586/20 BrightLine HC | bandpass filter detection |
| BP det3 | AHF analysentechnik AG, Tübingen, Germany | 631/36 BrightLine HC | bandpass filter detection |
| BP det4 | AHF analysentechnik AG, Tübingen, Germany | 700/75 ET Bandpass | bandpass filter detection |
| optical shutters detection | Vincent Associates, Rochester, NY, USA | Uniblitz VS25S2T0 | |
| EMCCD iXon Ultra 897 | Andor Technology Ltd, Belfast, Northern Ireland | ||
| digital I/O card, PCIe-6535 | National Instruments, Austin, Texas, USA | ||
| syringe pump | Harvard Apparatus, Holliston, MA, USA | PHD22/2000 | |
| Name | Company | Catalog Number | Comments |
| Flow chamber | |||
| quartz slides | G. Finkenbeiner Inc, Waltham, MA, USA | Spectrosil2000, h=3mm | |
| TEGADERM film | 3M Deutschland GmbH, Neuss, Germany | 1626W | 10 x 12cm |
| spray adhesive | 3M Deutschland GmbH, Neuss, Germany | Photo Mount 050777 | |
| glycerol | Carl Zeiss AG, Oberkochen, Germany | Immersol G | |
| immersion oil | OLYMPUS EUROPA SE & CO. KG, Hamburg, Germany | IMMOIL-F30CC | |
| prism | Vogelsberger Quarzglastechnik GmbH, Hauzenberg, Germany | Suprasil1 | |
| aluminium prism holder | custom built | ||
| hollow setscrews | Thorlabs Inc, Newton, New Jersey, USA | with custom drilling | |
| Tygon S3 E-3603 tubing | neoLab Migge GmbH, Heidelberg, Germany | 2-4450 | ACF00001 |
| PTFE tubing | Bohlender GmbH, Grünsfeld, Germany | S1810-08 | |
| Name | Company | Catalog Number | Comments |
| Sample | |||
| yeast Hsp90 D61C, Q385C_biotin | UniProt ID P02829 | ||
| Maleimide derivatives of Atto488, Atto550 | ATTO-TEC GmbH, Siegen, Germany | ||
| AMP-PNP* | Jena Bioscience, Jena, Germany | γ-[(6-Aminohexyl)-imido]-AMP-PNP-Atto647N | |
| Fluospheres | Thermo Fisher Scientific, Waltham, MA, USA | F8764 | amine-modified, 0.2 μm, yellow-green fluorescent |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Andor Solis | Andor Technology Ltd, Belfast, Northern Ireland | version 4.30 | |
| LabVIEW | National Instruments, Austin, Texas, USA | version 2012, 32bit; misc. hardware control | |
| MDS control software | AA Opto-Electronic, Orsay, France | version 2.03a | |
| Coherent Connection | Coherent Inc, Santa Clara, CA, USA | version 3 | |
| Igor Pro | WaveMetrics Inc, Portland, OR, USA | version 6.37 |
참고문헌
- Nooren, I. M. A., Thornton, J. M. Diversity of protein-protein interactions. EMBO J. 22 (14), 3486-3492 (2003).
- Marsh, J. A., Teichmann, S. A. Structure, dynamics, assembly, and evolution of protein complexes. Annu Rev Biochem. 84, 551-575 (2015).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Hohng, S., Joo, C., Ha, T. Single-Molecule Three-Color FRET. Biophys J. 87 (2), 1328-1337 (2004).
- Person, B., Stein, I. H., Steinhauer, C., Vogelsang, J., Tinnefeld, P. Correlated movement and bending of nucleic acid structures visualized by multicolor single-molecule spectroscopy. ChemPhysChem. 10 (9-10), 1455-1460 (2009).
- Lee, J., Lee, S., Ragunathan, K., Joo, C., Ha, T., Hohng, S. Single-molecule four-color FRET. Angew Chem Int Ed. 49 (51), 9922-9925 (2010).
- Ratzke, C., Berkemeier, F., Hugel, T. Heat shock protein 90's mechanochemical cycle is dominated by thermal fluctuations. Proc Natl Acad Sci U S A. 109 (1), 161-166 (2012).
- Ratzke, C., Hellenkamp, B., Hugel, T. Four-colour FRET reveals directionality in the Hsp90 multicomponent machinery. Nat Commun. 5, 4192 (2014).
- Götz, M., Wortmann, P., Schmid, S., Hugel, T. A Multicolor Single-Molecule FRET Approach to Study Protein Dynamics and Interactions Simultaneously. Methods Enzymol. 581, 487-516 (2016).
- Yengo, C. M., Berger, C. L. Fluorescence anisotropy and resonance energy transfer: Powerful tools for measuring real time protein dynamics in a physiological environment. Curr Opin Pharmacol. 10 (6), 731-737 (2010).
- Wortmann,P , ., Götz M, ., Hugel T , . Cooperative Nucleotide Binding in Hsp90 and Its Regulation by Aha1. Biophys J. 113, 1711-1718 (2017).
- Dörfler, T., Eilert, T., Röcker, C., Nagy, J., Michaelis, J. Structural Information from Single-molecule FRET Experiments Using the Fast Nano-positioning System. J Vis Exp. (120), e54782 (2017).
- Stephanopoulos, N., Francis, M. B. Choosing an effective protein bioconjugation strategy. Nature chemical biology. 7 (12), 876-884 (2011).
- Sánchez-Rico, C., Voith von Voithenberg, L., Warner, L., Lamb, D. C., Sattler, M. Effects of Fluorophore Attachment on Protein Conformation and Dynamics Studied by spFRET and NMR Spectroscopy. Chemistry (Weinheim an der Bergstrasse, Germany). , (2017).
- Roy, R., Hohng, S., Ha, T. A practical guide to single-molecule FRET. Nat Methods. 5 (6), 507-516 (2008).
- Lee, N. K., et al. Three-color alternating-laser excitation of single molecules: monitoring multiple interactions and distances. Biophys J. 92 (1), 303-312 (2007).
- Kapanidis, A. N., Lee, N. K., Laurence, T. A., Doose, S., Margeat, E., Weiss, S. Fluorescence-aided molecule sorting: analysis of structure and interactions by alternating-laser excitation of single molecules. Proc Natl Acad Sci U S A. 101 (24), 8936-8941 (2004).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chem Soc Rev. 43 (4), 1156-1171 (2014).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nat Methods. 14, 174-180 (2017).
- Rabiner, L. R. A tutorial on hidden Markov models and selected applications in speech recognition. Proc IEEE. 77 (2), 257-286 (1989).
- Fink, G. A. . Markov Models for Pattern Recognition. , (2014).
- Giudici, P., Ryden, T., Vandekerkhove, P. Likelihood-Ratio Tests for Hidden Markov Models. Biometrics. 56 (3), 742-747 (2000).
- Visser, I., Raijmakers, M. E. J., Molenaar, P. C. M. Confidence intervals for hidden Markov model parameters. Br J Math Stat Psychol. 53 (2), 317-327 (2000).
- McKinney, S. A., Joo, C., Ha, T. Analysis of Single-Molecule FRET Trajectories Using Hidden Markov Modeling. Biophys J. 91 (5), 1941-1951 (2006).
- Bronson, J. E., Fei, J., Hofman, J. M., Gonzalez, R. L., Wiggins, C. H. Learning Rates and States from Biophysical Time Series: A Bayesian Approach to Model Selection and Single-Molecule FRET Data. Biophys J. 97 (12), 3196-3205 (2009).
- Greenfeld, M., Pavlichin, D. S., Mabuchi, H., Herschlag, D. Single Molecule Analysis Research Tool (SMART): an integrated approach for analyzing single molecule data. PLoS ONE. 7 (2), e30024 (2012).
- Schmid, S., Götz, M., Hugel, T. Single-Molecule Analysis beyond Dwell Times: Demonstration and Assessment in and out of Equilibrium. Biophys J. 111 (7), 1375-1384 (2016).
- Taipale, M., Jarosz, D. F., Lindquist, S. HSP90 at the hub of protein homeostasis: emerging mechanistic insights. Nat Rev Mol Cell Biol. 11 (7), 515-528 (2010).
- Trepel, J., Mollapour, M., Giaccone, G., Neckers, L. Targeting the dynamic HSP90 complex in cancer. Nat Rev Cancer. 10 (8), 537-549 (2010).
- Wayne, N., Bolon, D. N. Dimerization of Hsp90 is required for in vivo function. Design and analysis of monomers and dimers. J Biol Chem. 282 (48), 35386-35395 (2007).
- Ali, M. M. U., et al. Crystal structure of an Hsp90-nucleotide-p23/Sba1 closed chaperone complex. Nature. 440 (7087), 1013-1017 (2006).
- Southworth, D. R., Agard, D. A. Species-dependent ensembles of conserved conformational states define the Hsp90 chaperone ATPase cycle. Mol Cell. 32 (5), 631-640 (2008).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proc Natl Acad Sci U S A. 101 (35), 12893-12898 (2004).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An Oxygen Scavenging System for Improvement of Dye Stability in Single-Molecule Fluorescence Experiments. Biophys J. 94 (5), 1826-1835 (2008).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Rognoni, L., Stigler, J., Pelz, B., Ylänne, J., Rief, M. Dynamic force sensing of filamin revealed in single-molecule experiments. Proc Natl Acad Sci U S A. 109 (48), 19679-19684 (2012).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in Surface-Tethered Lipid Vesicles as a New Tool for Single Biomolecule Spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-mode waveguides for single-molecule analysis at high concentrations. Science. 299 (5607), 682-686 (2003).
- Panaretou, B., et al. ATP binding and hydrolysis are essential to the function of the Hsp90 molecular chaperone in vivo. EMBO J. 17 (16), 4829-4836 (1998).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유