Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Uso del traste de una sola molécula a estudiar la correlación de las interacciones proteína
En este artículo
Resumen
Aquí, presentamos un protocolo para obtener datos smFRET tricolor y su análisis con un conjunto 3D modelo de Markov ocultos. Con este enfoque, los científicos pueden extraer información cinética de sistemas complejos de la proteína, incluyendo cooperatividad o interacción correlacionadas.
Resumen
Transferencia de energía de resonancia de una sola molécula Förster (smFRET) se ha convertido en una técnica ampliamente utilizada biofísica para estudiar la dinámica de las biomoléculas. Para muchas máquinas moleculares en las proteínas de una célula tienen que actuar junto con socios de la interacción en un ciclo funcional para cumplir con su tarea. La extensión de dos colores en multicolor smFRET permite sondear simultáneamente más de una interacción o cambio conformacional. Esto no sólo añade una nueva dimensión a los experimentos smFRET pero también ofrece la posibilidad única para estudiar directamente la secuencia de acontecimientos y detectar interacciones correlacionadas cuando se utiliza una muestra inmovilizada y una fluorescencia de la reflexión interna total microscopio (TIRFM). Por lo tanto, smFRET multicolor es una herramienta versátil para el estudio de complejos biomoleculares en términos cuantitativos y en un detalle previamente inalcanzable.
Aquí, demostramos cómo superar los problemas especiales de varios colores smFRET experimentos con proteínas. Presentamos protocolos detallados para la obtención de los datos y para extraer información cinética. Esto incluye criterios de selección de la traza, separación del estado y la recuperación de las trayectorias de estado de los datos de ruido utilizando un conjunto 3D modelo oculto de Markov (HMM). En comparación con otros métodos, la información cinética no se recupera de los histogramas de tiempo de permanencia pero directamente en el HMM. El marco de la máxima verosimilitud nos permite evaluar críticamente el modelo cinético y proveer incertidumbres significativas para las tasas.
Al aplicar nuestro método a la proteína de choque térmico 90 (Hsp90), somos capaces de desentrañar el enlace de nucleótidos y los globales cambios conformacionales de la proteína. Esto nos permite observar directamente la cooperatividad entre los dos bolsillos de unión de nucleótido del dimer de Hsp90.
Introducción
Muchas proteínas cumplen con su función en dinámicos complejos con otras moléculas, mediadas por cambios conformacionales y asociaciones transitorias en una amplia gama de escalas temporales1,2,3. Junto a una fuente externa de energía (e.g., ATP) estas interacciones dinámicas pueden conducir a direccionalidad en un ciclo funcional y, en definitiva, mantener el estado estacionario de no equilibrio en una célula, el requisito previo para la vida.
Para entender completamente estas máquinas moleculares, no es suficiente una descripción estática guiada por los estudios estructurales. Además, es esencial tener conocimiento del modelo cinético subyacente y para determinar las constantes de tipo cinético. Varios métodos existentes permiten a los investigadores estudiar la dinámica de las interacciones binarias entre dos moléculas de interés, por ejemplo, resonancia de plasmón superficial, métodos de relajación con una lectura espectroscópica (por ejemplo, salto o flujo detenido técnicas) y resonancia magnética nuclear. Sin embargo, su aplicabilidad es en la mayoría de los casos que se limita a simples sistemas de dos Estados (por ejemplo, un límite y un estado independiente) debido a la inherente a los experimentos de mayor promedio. En los casos donde están involucrados más Estados o productos intermedios, rinden sólo una mezcla compleja de las constantes de velocidad. Métodos de una sola molécula como pinzas ópticas o magnéticas o smFRET bicolor, es decir, un donante y un fluoróforo aceptor, con una muestra de superficie inmovilizada pueden recuperar las constantes de velocidad para todos observar cambios conformacionales. Sin embargo, cuando se trata de interacciones que afectan a más de un enlace, estos métodos siguen siendo limitados y la información sobre las interacciones de la posible correlación de los dos (o más) sólo será accesible a través de conclusiones indirectas de un conjunto de experimentos.
SmFRET multicolor4,5,6,7,8,9 ofrece la oportunidad de estudiar la interacción entre estos componentes directamente, en tiempo real y bajo condiciones fisiológicas cerca de10. Esto permite investigar por ejemplo, la Unión dependiente de la conformación de un ligando u otra proteína8,9,11. El enfoque general que se presenta aquí es a etiqueta de la proteína de interés en posiciones específicas, para unir una proteína a la superficie de la cámara de medición y seguimiento de la intensidad de fluorescencia con el tiempo en un tipo de prisma TIRFM (para detalles ver 9 , 12). la proximidad espacial de los diferentes tintes puede ser determinada luego de la transferencia de energía entre ellos. Etiquetado estrategias puede variar de proteína a proteína (revisada en 13) y directrices para evitar artefactos en smFRET medidas existen14.
Ya que un tinte de donantes puede transferir energía al aceptador diferentes tintes en un experimento de varios colores smFRET, la posición relativa de los colorantes no es accesible desde la excitación de un tinte solo15,16. Pero en combinación con alternancia de excitación láser (ALEX17y ha comentado en 18) este método proporciona toda la información espacio-temporal en fracciones de segundos y resolución sub-nanométrica.
En principio, alta resolución información estructural se logra mediante el uso de las distancias entre tintes calcula a partir de la combinación de todas intensidades de fluorescencia en un experimento smFRET multicolor con ALEX. Sin embargo, aquí nos enfocamos en la identificación de estado y separación así como la extracción de modelos cinéticos, donde varios color smFRET es indispensable. Cuando se desea "sólo" determinación estructural mediante triangulación, un conjunto de experimentos de dos colores smFRET más simples con alta relación de señal a ruido puede ser realizado12,19.
Utilizamos el parcial de la fluorescencia ( ) como un proxy para la transferencia de energía entre dos fluoróforos7. El PF es calculado de la intensidad de fluorescencia similar a la eficacia de traste de un experimento de dos colores:
) como un proxy para la transferencia de energía entre dos fluoróforos7. El PF es calculado de la intensidad de fluorescencia similar a la eficacia de traste de un experimento de dos colores:

Donde, 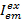 es la intensidad en el canal de emisión em después de excitación con color ex, y c es el aceptante con la longitud de onda más larga. Canales de detección representan la misma posición en la cámara de la muestra pero discos diferentes rangos espectrales de la luz de la fluorescencia. Utilizan el mismo identificador de excitación y emisión en este protocolo (es decir, "azul", "verde" y "rojo").
es la intensidad en el canal de emisión em después de excitación con color ex, y c es el aceptante con la longitud de onda más larga. Canales de detección representan la misma posición en la cámara de la muestra pero discos diferentes rangos espectrales de la luz de la fluorescencia. Utilizan el mismo identificador de excitación y emisión en este protocolo (es decir, "azul", "verde" y "rojo").
Debido a deficiencias experimentales las intensidades de fluorescencia medidos dependen no sólo en la transferencia de energía, sino también de propiedades fluoróforo y configuración. Para obtener la verdadera eficiencia de transferencia entre dos fluoróforos, las intensidades medidas tienen que corregirse. El siguiente procedimiento se basa en la referencia9. Factores de corrección para salida aparente (LC, es decir, la detección de fotones de un fluoróforo en un canal designada para otro tinte) y gamma aparente (ag, es decir, el rendimiento cuántico de fluorescencia del colorante y la eficiencia de la detección del canal) se obtiene de rastros de una sola molécula que muestran un aceptor evento de blanqueo.
La salida del tinte en cada canal de aceptador posible donante se calcula desde todos los puntos de datos en los rastros de fluorescencia grabado donde el tinte del aceptador de la blanqueada pero el donante es todavía fluorescente (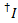 ):
):
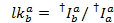
La media del histograma de salida se utiliza como el factor de salida aparente. Después de la corrección de fugas, el factor gamma aparente se determina desde el mismo conjunto de rastros. Se calcula dividiendo el cambio de fluorescencia en el canal del receptor por el cambio de fluorescencia en el canal de donantes sobre la decoloración del tinte aceptador:

Donde c es otra vez el canal de detección para el aceptante con la longitud de onda más larga. La mediana de la distribución resultante se utiliza como el factor de corrección aparente.
Las intensidades corregidas en cada canal se obtienen por:
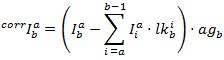
Luego se calcula el PF según:

Diferentes poblaciones pueden ser separadas en el espacio multidimensional atravesado por la s PF. La posición y la anchura de cada Estado se determina introduciendo los datos con funciones Gaussianas multidimensionales. Optimización posterior de un HMM global basado en los restos PF proporciona una descripción cuantitativa de la cinética observada. Incluso pequeños cambios de las tasas son detectables.
HMMs proporcionan una manera de inferir un modelo de estado de una colección de huellas de ruido tiempo. El sistema se considera en uno de una serie de discretos, Estados ocultos en cualquier momento y en la observación real (es decir, la emisión) es una función probabilística de este estado oculto20. En el caso de los datos de TIRFM smFRET, la emisión probabilidades b por el estado puede ser modelados por funciones de densidad de probabilidad gaussiana continua. En los puntos regularmente espaciados de tiempo discreto, las transiciones de uno a otro estado pueden ocurrir según la probabilidad de transición que es tiempo-invariante y sólo depende del estado actual. La matriz de transición A contiene estas probabilidades de transición unij entre todos los Estados ocultos. La distribución de estado inicial  da las probabilidades de estado específico
da las probabilidades de estado específico 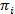 para el primer punto de tiempo de un seguimiento de tiempo. Usando un enfoque de probabilidad máxima, estos parámetros pueden optimizarse para describir mejor los datos con el adelante-atrás y Baum-Welch algoritmos20,21. Esto produce los estimadores de máxima verosimilitud (MLE). Por último, se puede inferir la secuencia de estado que es muy probable que la trayectoria de observaciones con el algoritmo de Viterbi. A diferencia de otros análisis HMM de smFRET datos24,25,26 no utilizamos el HMM como una mera "atenuación" de los datos, pero extracto el modelo cinético de estado del conjunto de datos sin la necesidad para la instalación de tiempo de permanencia histogramas de27. HMM el análisis es hecho con scripts internos con Igor Pro. Aplicación del código se basa en la referencia21. Le ofrecemos un kit de software y los datos de ejemplares en nuestra pagina web para seguir las secciones 5 y 6 del presente Protocolo (https://www.singlemolecule.uni-freiburg.de/software/3d-fret). Software completo está disponible bajo petición.
para el primer punto de tiempo de un seguimiento de tiempo. Usando un enfoque de probabilidad máxima, estos parámetros pueden optimizarse para describir mejor los datos con el adelante-atrás y Baum-Welch algoritmos20,21. Esto produce los estimadores de máxima verosimilitud (MLE). Por último, se puede inferir la secuencia de estado que es muy probable que la trayectoria de observaciones con el algoritmo de Viterbi. A diferencia de otros análisis HMM de smFRET datos24,25,26 no utilizamos el HMM como una mera "atenuación" de los datos, pero extracto el modelo cinético de estado del conjunto de datos sin la necesidad para la instalación de tiempo de permanencia histogramas de27. HMM el análisis es hecho con scripts internos con Igor Pro. Aplicación del código se basa en la referencia21. Le ofrecemos un kit de software y los datos de ejemplares en nuestra pagina web para seguir las secciones 5 y 6 del presente Protocolo (https://www.singlemolecule.uni-freiburg.de/software/3d-fret). Software completo está disponible bajo petición.
Puntos en los datos con PF de tiempo < -1 o PF > 2 en cualquier canal de detección se asigna la probabilidad de emisión mínima para todos los Estados (10-200). Esto evita que las transiciones artificiales en estos puntos de datos.
Los parámetros para las probabilidades de emisión se obtienen a partir del ajuste del histograma PF 3D con funciones Gaussian como se describe en el paso 5.7. Estos parámetros se mantienen fijos durante la optimización de lo HMM.
En el enfoque presentado, el vector de distribución del estado inicial y la matriz de transición se utilizan a nivel mundial para describir todo el conjunto de huellas. Que se actualicen en todas las moléculas de N del conjunto de datos según referencia27base.
Parámetros de inicio para la distribución de estado inicial se determinan las proyecciones 2D del histograma PF (paso 5.3) y las probabilidades de transición estén en 0.05 a excepción de las probabilidades de permanecer en el mismo estado, que se eligen tales que la probabilidad de salir de un determinado Estado se normaliza a la unidad.
Un método de generación de perfiles de probabilidad se utiliza para dar intervalos de confianza (IC) para toda transición tarifas21,22, que sirven como significativas estimaciones de su incertidumbre. Para calcular los límites de la CI para una velocidad específica, la probabilidad de transición de interés se fija en un valor distinto de la EAM. Esto rinde la prueba modelo λ'. Una prueba de la razón (LR) de probabilidad de la probabilidad 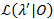 teniendo en cuenta el conjunto de datos 0 se realiza según:
teniendo en cuenta el conjunto de datos 0 se realiza según:
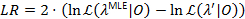
El 95% de confianza limitado por el parámetro se alcanza cuando LR excede de 3.841, el cuantil del 95% de un x2-distribución con un grado de libertad22,23.
El poder del método se demuestra con la Hsp90. Esta proteína abundante se encuentra en bacterias y eucariotas y es parte de la respuesta de estrés celular28. Es un blanco de droga prometedor en el tratamiento de cáncer29. Hsp90 es un homodímero con un bolsillo de unión de nucleótido en el dominio N-terminal de cada subunidad30. Pueden experimentar transiciones entre al menos dos conformaciones distintas a nivel mundial, uno cerrado y uno N-terminal conformación abierta en forma de V,19,31,32. La naturaleza dimérica plantea directamente la cuestión de la interacción entre los dos sitios de unión de nucleótido en Hsp90.
A continuación, proporcionamos un protocolo paso a paso para la adquisición de datos y el análisis de un experimento smFRET tricolor en levadura Hsp90 y nucleótidos. La Unión dependiente de la conformación de la fluorescencia etiquetada AMP-PNP (PNP AMP *, un análogo no hidrolizables de la ATP) se analiza. La aplicación del procedimiento descrito permite el estudio de la Unión de nucleótidos y al mismo tiempo los cambios conformacionales de la Hsp90 y tal modo revela la cooperatividad entre los dos bolsillos de unión de nucleótido de Hsp90.
Protocolo
1. instalación y requisitos previos
- Realizar las mediciones smFRET varios colores en un prisma de tipo TIRFM. Una descripción de una configuración de dos colores como una publicación de Zeus se da en referencia a12.
- Construir un TIRFM multicolor. Una disposición general se detalla en el 9.
- Uso cambiable, diodo bombeó el láser de onda continua de estado sólido, que hacen innecesario el uso de obturadores mecánicos en las vías de excitación.
- Emplean un prisma alargado, asimétrico que evita la reflexión posterior de la viga de la excitación de la parte posterior para entrar en el objetivo.
- Usar lentes acromáticos de 2 pulgadas asférico sílices en los caminos de detección que recogen tanta luz como sea posible y evitar auto-fluorescencia y aberraciones, por ejemplo, distorsiones en las regiones fuera del centro de la imagen.
- Cada trayectoria de detección se centran en el chip de EMCCD con lentes separadas. Esto permite un enfoque óptimo de cada canal de detección.
PRECAUCIÓN: El TIRFM se utilizan láseres de clase 3B. Esto significa que son peligrosos si el ojo está expuesto directamente, pero reflexiones difusas no son dañinos. Asegurar el cumplimiento con láser las precauciones de seguridad según las regulaciones del gobierno local antes de que el sistema es operado.
- Determinar los factores de corrección para las propiedades de configuración y fluoróforo previamente utilizando muestras de dsDNA.
- Utilizar una muestra de dsDNA alto traste para cada colorante en combinación con el aceptador de tener la más larga longitud de onda de excitación (para la configuración actual: Atto488-Atto647N, Atto550-Atto647N, Atto594-Atto647N). Asegúrese de que el ADN además es modificado con una biotina.
- Diluir la muestra a 5 nM con tampón de TNM (5 mM Tris, pH 7,5, 5 mM NaCl, 20 mM de MgCl2) y 2 mM Trolox (uso de este buffer también para medición).
- Inmovilizar el dsDNA descrito para Hsp90 en pasos de 2.5 y 2.7.
- Calcular los factores de corrección para salida aparente (LC) y gamma aparente (ag) de una sola molécula rastros que muestran un evento de blanqueamiento de aceptador.
- Construir una cámara de flujo que es un bocadillo de un portaobjetos de cuarzo apaciguado PEG/biotina-PEG, una fina película que es adhesivo en ambos lados y un cubreobjetos. Para los protocolos de detalle para la limpieza del cuarzo diapositivas y estabilización ver referencia9.
- Cuarzo de espesor (3 mm) de uso las diapositivas para evitar geométricamente la colección de luz láser dispersada en la interfaz de cuarzo-glicerol entre el prisma y el portaobjetos de cuarzo funcionalizados.
- Utilizar una fina (40 μm) película que se rocía con pegamento en el lado no adhesivo. La película delgada reduce la distancia entre las moléculas de superficie adjunta y objetivo. Calentar a 80 ° C y pulse.
- Coloque un cubreobjetos sobre la parte superior. Calentar a 80 ° C y pulse. Utilice una gota de glicerina al colocar la cámara de flujo sobre el prisma.
Nota: Los materiales del prisma y los portaobjetos de cuarzo como el glicerol son emparejados por el índice.
- Expresar Hsp90 de Saccharomyces cerevisiae en la forma de dos mutantes de punto único de cisteína en las posiciones D61 o Q385. Añadir un motivo en espiral-arrolle de terminal C para evitar la disociación de dimer a concentraciones picomolar. Las proteínas del mutante de la etiqueta por separado e intercambiar los monómeros para obtener heterodímeros con Atto488 en la posición del aminoácido 61 y Atto550 en el aminoácido posición 3859.
2. la medida
- Inicie el software de la cámara y configurar los parámetros de imagen tal como se especifica a continuación:
- Ajuste la temperatura para el sensor de enfriamiento tan bajo como sea posible (-95 ° C con enfriamiento de agua) para disminuir el ruido oscuro actual.
- Utilizar ajustes de la cámara que están optimizados para la grabación de una sola molécula: 3.3 μs velocidad de desplazamiento vertical, voltaje normal del reloj vertical, 17 MHz 16 bits horizontal Lee, ganancia pre-amplificación 3, ganancia de multiplicador de electrón 1.000.
- Establecer la activación de la adquisición "Externo" y el tiempo de exposición a películas de expediente de la Sra. 70 con una longitud de 750 ciclos de adquisición.
Nota: Apague la luz de la habitación cuando la cámara está adquiriendo para evitar la saturación del sensor EMCCD.
- Crear una carpeta en la unidad SSD local para la medición. En el software de configuración ir al jinete < Auto-Save > Habilitar < Auto-save > y elegir formato "Tiff" para la adquisición de película. Seleccione la carpeta en el SSD como lugar de guardado automático.
- Inicie el software que controles el acusto-óptico filtro sintonizable (AOTF), el software que controla el funcionamiento de los láseres y el software de gatillo que sincroniza los lasers, AOTF, persianas en la trayectoria de detección y las cámaras. Ajustar la potencia del láser con la AOTF (ca. 3 mW antes de entrar en el prisma) y el patrón de activación correcta de la carga.
- Montar el soporte de la muestra con el prisma y la cámara de flujo, fije el tubo, coloque el tubo de entrada en una taza de microcentrífuga y conectar la tubería de salida a una bomba de jeringa. Limpie la cámara con aproximadamente 150 μL de tampón, alinee la excitación viga y enfoque. Bleach cualquier contaminante fluorescente sobre la superficie desplazando lentamente a lo largo de la gama de detección completa de la diapositiva con una potencia de láser de 10 mW para todos los láser. Esto toma alrededor de 1 h.
Nota: Si no indique lo contrario, el buffer utilizado contiene 40 mM HEPES pH 7.5, 150 mM KCl y 10 mM MgCl2. - Descarga aproximadamente 300 μL de una solución de NeutrAvidin (0.25 mg/mL en buffer) en la cámara e incubar durante 1 minuto Flush out NeutrAvidin independiente con aproximadamente 300 μL de tampón y al ras de una solución de BSA (0.5 mg/mL en tampón) a través de la cámara.
Nota: Este bloque restante funcionalización de superficies defectos por la adsorción inespecífica de BSA a la superficie. - Inmovilizar la muestra por la carga de la cámara de flujo con aproximadamente 150 μL de la biotinilado y etiquetada Hsp90 a concentraciones crecientes (diluidas en tampón + 0,5 mg/mL BSA) hasta que se alcanza una suficiente densidad de superficie, que suele ser el caso a una concentración de 5-22:00. Lave hacia fuera proteína independiente con aproximadamente 300 μL de tampón de + 0,5 mg/mL BSA.
- Ras en 150 μL de 25 nM AMP PNP * buffer + 0,5 mg/mL BSA. Dejar incubar durante 5 minutos y repita este paso una vez para asegurar concentración nucleótido correcto.
Nota: Para los experimentos en presencia de AMP-PNP adicional, sin etiqueta, también agregar μm 250 AMP-PNP. - Comenzar con la adquisición de datos. Para una cantidad adecuada de datos adquirir aproximadamente 20 películas que tarda aproximadamente 1,5 horas.
- Mover la posición de la cámara de muestra perpendicular al haz de excitación con un piezo paso a paso para cambiar el campo de visión.
- Ajuste el enfoque con el z-piezo que controla la altura del objetivo, si es necesario. Esto no debería ser necesario muy a menudo cuando es montada en la cámara de medición sin inclinación.
- Preparar la grabación de las cámaras pulsando < tomar señal > software de la cámara y empezar los ciclos de excitación o adquisición utilizando el botón < Start > en el software de activación. Esto inicia la adquisición de la intensidad de fluorescencia.
- Primera grabación de una película con los granos fluorescentes que muestran la emisión de fluorescencia en el espectro de todos los canales de detección de la configuración para realizar un registro de canal. Luego, detectar posiciones de grano en la película de calibración mediante la búsqueda de los puntos más brillantes y determinar que la posición central de un Gaussian se ajuste al perfil de la intensidad. Guardar las coordenadas de los granos que se encuentran en todos los canales y ajuste tanto el offset mapping en x y en y-dirección con un 2D polinomio de grado tres9.
3. selección de trazas de una sola molécula
- Análisis de datos se realiza con secuencias de comandos internos con Igor Pro. Carga todos los scripts necesarios mediante la apertura de "iniTIRF.ipf" y seleccionar el correcto tipo de experimento.
Nota: en el siguiente botón de < > especifica elementos puede hacer clic en el menú o la interfaz de usuario. Llamadas de función se indican entre comillas, por ejemplo, "Imprimir"Hola mundo"". Estos comandos se pueden pegar a la línea de comandos de Igor Pro (sin las comillas de cierre). - Asegúrese de que los parámetros para el registro de canales de detección se cargan.
- Iniciar la interfaz gráfica haciendo clic en < smFRET nuevo | Análisis de interfaz gráfica de usuario >.
- Carga de las películas (es decir, la secuencia de fotogramas con 512 x 512 píxeles como pilas TIFF de 16 bits) que mantienen la intensidad en los canales respectivos. Para ello, presionar el botón < carga película > y seleccionando los archivos de las cámaras de "master" y "esclavo" llamadas uno tras otro.
- Identificar las posiciones de las moléculas individuales posibles buscando los puntos más brillantes en la suma de los cinco primeros fotogramas en un determinado canal de detección. Calcular las posiciones correspondientes de los otros canales de detección de la asignación de canales. Para obtener el rastro de la intensidad de fluorescencia, en definitiva la intensidad de un cuadrado de píxeles alrededor de la posición central de cada fotograma. Hacer esto presionando el botón < encontrar rastros > en la GUI.
Nota: La longitud del lado de la Plaza (en píxeles) está dada por: 2 * < suma Pxs > + 1. - Para cada molécula, calcular una traza común intensidad cruda como la suma de todos los rastros de este punto con el mismo color de la excitación. Evaluar el perfil de intensidad de la molécula en todos los canales para los siguientes criterios:
- Una meseta más o menos plana en la intensidad conjunta de crudo y un blanqueo solo paso para todos los colores de excitación, anticorrelated comportamiento en los canales de detección apropiado, detección de fluorescencia roja (indicando el traste para un encuadernado AMP-PNP *) al menos una vez en el rastro y no varios pasos en la fluorescencia roja, que indicaría la presencia de dos AMP-PNP * a un dímero de la Hsp90.
- Guardar los rastros de la fluorescencia del punto para su posterior análisis, si se satisfacen estos criterios. Para ello seleccionar la traza con el cursor y pulsando el botón < guardar > en la gráfica de "plazos". Inspeccionar manualmente las huellas de la intensidad en los tres canales de detección después de excitación azul para alrededor de 200 moléculas por película.
4. cálculo de las huellas de fluorescencia parcial
- Mostrar todo rastro de intensidad de fluorescencia para una de las moléculas guardadas. Utiliza los cursores del gráfico para seleccionar intervalos de tiempo.
- Seleccione un intervalo de tiempo donde los fluoróforos se blanquean ya. La intensidad de fondo media es calculada a partir de esta gama y restar de la huella de la intensidad en cada canal. Hacer esto presionando el botón < fondo >.
- Seleccione el rango de eficiencia de traste, donde al menos dos los tintes al Hsp90 (Atto488 y Atto550) están presentes. Asegúrese de excluir trazas que contienen un evento intermitente (ver figura 3B). Estos eventos se caracterizan por una intensidad de fluorescencia de sobreponer en un canal sin un aumento acompañante en cualquier otro canal.
- Calcular los rastros de la PF . Hacer esto presionando el botón < PF Calc >. Los factores de corrección predefinidas para salida aparente (LC) y gamma aparente (ag) se aplican a la intensidad cruda para corregir para foto-físicas y propiedades de configuración.
5. población selección y ajuste de histograma 3D
- Eliminar moléculas que muestran una baja relación señal a ruido en los rastros de la PF . Moléculas que excedan el intervalo [-1; 2] en cualquier rastro PF más del 10% de las tramas se eliminan del conjunto de datos. Para ello ejecutar "RemoveTracesLowSNR()" desde la ventana de comandos.
- Calcular las proyecciones 2D desechadas de los datos de la PF . Parcela
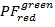 sobre
sobre 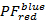 y
y 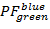 sobre
sobre 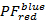 en la gama [-0.5; 1,5] con una resolución de 100 x 100 contenedores. Para ello, ejecutar:
en la gama [-0.5; 1,5] con una resolución de 100 x 100 contenedores. Para ello, ejecutar:- "HistFret2D ("r_b","r_g", binHist = 100)"
- "HistFret2D ("r_b","g_b", binHist = 100); MoveWindow 553.5, 42.5, 1055.25, 508,25"
- Determinar la población relativa de cada estado distinguible en las proyecciones 2D.
- El gráfico correspondiente a la parte delantera y ejecutar "panelHist2DCount()".
- Presione el botón < Init > y traza un polígono de mano alrededor de la cima.
- Haga clic en el botón < cuenta >. El número de puntos de datos en el polígono y el número total de puntos de datos en la proyección se imprimen en la ventana de comandos.
- Prepare un histograma 3D de los datos PF ejecutando "HistFret3D ("g_b","r_b","r_g")".
- Normalizar el histograma 3D a una integral de 1. Ejecute lo siguiente:
- "NewDataFolder/S fit0"
- "Duplicado/O:: traste: Hist3D, Hist3D"
- "Div variable/g = sum(Hist3D)*(DimDelta(Hist3D,0)) ^ 3"
- "Hist3D = div; Imprimir div"
- Proporcionan parámetros iniciales para la gaussiana 3D fit y preparan las estructuras de datos necesarias.
- Ejecutar "Gauss3D_initParam(); Editar W_coef_old".
- Añadir las poblaciones del estado hasta el final del vector de parámetros.
- Ejecutar "Gauss3D_prepareFit()."
Nota: W_coef_old es un vector que contiene los parámetros iniciales para el ajuste. Esto significa que cada estado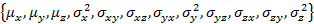 y la población del estado, que se concatena en el extremo del vector. Asegúrese de que la matriz de covarianza es simétrica.
y la población del estado, que se concatena en el extremo del vector. Asegúrese de que la matriz de covarianza es simétrica.
- Ajustar la suma de S 3D funciones Gaussian del histograma 3D de PF , S es el número de Estados distinguibles.
- Ejecutar "do3D()." Esto puede tomar una hora o más en una oficina normal PC, dependiendo de la calidad de los parámetros iniciales.
- Ejecutar "postprocessFitMultiGauss3D(); evalFitMultiGauss3D(); Editar W_coef".
- Mostrar el resultado de forma. Cada una de las dos proyecciones 2D, utilice los siguientes comandos:
- "contourPF3D_new(0); contourPF3D_new(1); contourPF3D_new(2); contourPF3D_new(3); contourPF3D_new(4)"
- "contourPF3D_colorize()"
6. cinético análisis conjunto 3D HMM
- Preparar un conjunto HMM para extraer la información cinética. Un HMM se ha optimizado de todas las moléculas en el conjunto de datos. Utilice la información obtenida en el paso anterior para definir la posición y la anchura de cada estado en el espacio 3D de PF .
- Inicializar la interfaz de usuario HMM (< HMM | Inicio HMM >) y seleccione el número apropiado de Estados (en el caso de los datos de la Hsp90 esto significa < NumStates > = 5), el número de dimensiones de la señal de entrada (< NumDims > = 3) y el tipo de la entrada (tipo de entrada de < > = "Traste bgr 3D").
- Optimizar los parámetros de lo HMM dejando el software convergen la probabilidad de que el HMM (ejecutar "prepENS_CONVERGE_gB(GetDataFolder(1), -14)") hasta que el cambio en la matriz de transición en comparación con la iteración anterior cae por debajo de un umbral (10-14 por la suma del cambio absoluto para cada probabilidad de transición). Esto rinde el MLE para las probabilidades de transición en aproximadamente una hora en una PC normal de oficina.
- Repetir la selección de la población, ajuste gaussiano y la optimización de HMM para subconjuntos de los datos (por ejemplo, el 75% de todo el conjunto de datos). Si el conjunto completo de datos se combinan de diferentes experimentos, repita la optimización también para cada uno de los experimentos individuales. Análisis de subconjuntos permite para estimar la incertidumbre de la selección manual de la población y la variabilidad dentro del conjunto de datos.
- Calcular el CI de las probabilidades de transición, que informe acerca de la heterogeneidad del conjunto de datos y la precisión de lo HMM.
- Obtener una estimación aproximada de los límites del CI por ejecutar "cd $(raíz: path3Dimport +"HMM"); loop_getCI_estimate_limits()."
- Calcular los límites exactos de la IC ejecutando:
- "loop_getCI_HMM_converge(1)"
- "CIresults_conv_new()"
- "cd:: HMM_CIresult; reportCI_conv()"
- "cd:: cmp_CI_conv; CI_plot2 ("HMM", doAppend = 0) "
- Condensar la información cinética disponible para simplificar la interpretación.
- Recopilar información sobre el tiempo de un nucleótido marcado queda obligado a Hsp90. Para ello, colapsando los Estados que están obligados a AMP PNP * (es decir, S1, S2 y S3, ver también figura 2) y compilar el histograma de tiempo de permanencia. Ejecutar "cd $(raíz: path3Dimport +"HMM"); collapse_states_get_DT({0,1,1,1,0}) ", que combina Mora de Estados S0 y S4 como S1, S2y S3.
- Extraer los tiempos de detención de la trayectoria de Viterbi para cada estado de interés y comparar el histograma resultante de tiempo de permanencia para diferentes condiciones experimentales. Ejecutar "plot_collapsed_DT_Hist (wDTo_01110_record1)."
- Para un cuadro más detallado, derrumbamiento Estados que tienen distintos PF pero que son funcionalmente idénticas para facilitar el posterior análisis de los datos, por ejemplo, en el caso de un experimento de tres colores con Hsp90 etiquetados y nucleótidos, Estados S2 y S 3 puede ser derrumbada. Ejecutar "collapse_states_get_DT({0,1,2,2,4})."
Resultados
SmFRET multicolor medidas permiten la detección directa de la correlación entre dos o más sitios de interacción diferentes. Esto hace que la técnica única para investigar sistemas de varios componentes, tales como complejos de la proteína. Nos centramos en la presentación de un experimento smFRET tricolor, que sirve como un ejemplo ilustrativo.
El flujo de trabajo general del método se muestra en la
Discusión
Presentamos el procedimiento experimental para obtener datos smFRET tricolor para un sistema complejo de proteína y una descripción paso a paso del análisis de estas mediciones. Este enfoque ofrece la única posibilidad de evaluar directamente la correlación entre múltiples sitios de interacción o cambios conformacionales.
Para obtener datos de una sola molécula de multicolores adecuados en proteínas es importante realizar mediciones reproducibles en un bajo nivel de ruido. Esto puede ...
Divulgaciones
Los autores no declaran conflictos de interés.
Agradecimientos
Este trabajo es financiado por la Fundación alemana de investigación (INST 39/969-1) y el Consejo Europeo de investigación a través del acuerdo de subvención del CEI n. 681891.
Materiales
| Name | Company | Catalog Number | Comments |
| Setup | |||
| vibration-damped optical table | Newport, Irvine, CA, USA | RS2000 | |
| OBIS 473nm LX 75mW LASER | Coherent Inc, Santa Clara, CA, USA | 1185052 | |
| OBIS 532nm LS 50mW LASER | Coherent Inc, Santa Clara, CA, USA | 1261779 | |
| OBIS 594nm LS 60mW LASER | Coherent Inc, Santa Clara, CA, USA | 1233470 | |
| OBIS 637nm LX 140mW LASER | Coherent Inc, Santa Clara, CA, USA | 1196625 | |
| laser control unit | Coherent Inc, Santa Clara, CA, USA | 1234465 | Scientific Remote |
| aspheric telescope lenses | Thorlabs Inc, Newton, New Jersey, USA | d=25.4mm, f=50mm and f=100mm | |
| CF ex1 | AHF analysentechnik AG, Tübingen, Germany | ZET 473/10 | cleanup filter excitation |
| CF ex2 | AHF analysentechnik AG, Tübingen, Germany | ZET 532/10 | cleanup filter excitation |
| CF ex3 | AHF analysentechnik AG, Tübingen, Germany | ZET 594/10 | cleanup filter excitation |
| CF ex4 | Thorlabs Inc, Newton, New Jersey, USA | FL635-10 | cleanup filter excitation |
| DM ex1 | AHF analysentechnik AG, Tübingen, Germany | ZQ594RDC | dichroic mirror excitation |
| DM ex2 | AHF analysentechnik AG, Tübingen, Germany | 570DCXR | dichroic mirror excitation |
| DM ex3 | AHF analysentechnik AG, Tübingen, Germany | ZQ491RDC | dichroic mirror excitation |
| AOTFnC-Vis | AA Opto-Electronic, Orsay, France | ||
| λ/4 plate | Thorlabs Inc, Newton, New Jersey, USA | AQWP05M-600 | |
| CFI Apo TIRF 100x | Nikon Instruments Inc, Melville, NY, USA | high-NA objective | |
| piezo focus positioner MIPOS 250 CAP | piezosystem jena GmbH, Jena, Germany | Piezo Controller NV 40/1 CLE | |
| piezo stepper | Newport, Irvine, CA, USA | PZA12 | PZC200-KT NanoPZ Actuator Kit |
| achromatic aspheric lenses | Qioptiq Photonics GmbH & Co. KG, Göttingen, Germany | G322-304-000 | d=50mm, f=200mm |
| adjustable optical slit | Owis GmbH, Staufen i. Br., Germany | 27.160.1212 | max. aperture 12 x 12 mm |
| DM det1 | AHF analysentechnik AG, Tübingen, Germany | T 600 LPXR | dichroic mirror detection |
| DM det2 | AHF analysentechnik AG, Tübingen, Germany | H 560 LPXR superflat | dichroic mirror detection |
| DM det3 | AHF analysentechnik AG, Tübingen, Germany | HC BS R635 | dichroic mirror detection |
| BP det1 | AHF analysentechnik AG, Tübingen, Germany | 525/40 BrightLine HC | bandpass filter detection |
| BP det2 | AHF analysentechnik AG, Tübingen, Germany | 586/20 BrightLine HC | bandpass filter detection |
| BP det3 | AHF analysentechnik AG, Tübingen, Germany | 631/36 BrightLine HC | bandpass filter detection |
| BP det4 | AHF analysentechnik AG, Tübingen, Germany | 700/75 ET Bandpass | bandpass filter detection |
| optical shutters detection | Vincent Associates, Rochester, NY, USA | Uniblitz VS25S2T0 | |
| EMCCD iXon Ultra 897 | Andor Technology Ltd, Belfast, Northern Ireland | ||
| digital I/O card, PCIe-6535 | National Instruments, Austin, Texas, USA | ||
| syringe pump | Harvard Apparatus, Holliston, MA, USA | PHD22/2000 | |
| Name | Company | Catalog Number | Comments |
| Flow chamber | |||
| quartz slides | G. Finkenbeiner Inc, Waltham, MA, USA | Spectrosil2000, h=3mm | |
| TEGADERM film | 3M Deutschland GmbH, Neuss, Germany | 1626W | 10 x 12cm |
| spray adhesive | 3M Deutschland GmbH, Neuss, Germany | Photo Mount 050777 | |
| glycerol | Carl Zeiss AG, Oberkochen, Germany | Immersol G | |
| immersion oil | OLYMPUS EUROPA SE & CO. KG, Hamburg, Germany | IMMOIL-F30CC | |
| prism | Vogelsberger Quarzglastechnik GmbH, Hauzenberg, Germany | Suprasil1 | |
| aluminium prism holder | custom built | ||
| hollow setscrews | Thorlabs Inc, Newton, New Jersey, USA | with custom drilling | |
| Tygon S3 E-3603 tubing | neoLab Migge GmbH, Heidelberg, Germany | 2-4450 | ACF00001 |
| PTFE tubing | Bohlender GmbH, Grünsfeld, Germany | S1810-08 | |
| Name | Company | Catalog Number | Comments |
| Sample | |||
| yeast Hsp90 D61C, Q385C_biotin | UniProt ID P02829 | ||
| Maleimide derivatives of Atto488, Atto550 | ATTO-TEC GmbH, Siegen, Germany | ||
| AMP-PNP* | Jena Bioscience, Jena, Germany | γ-[(6-Aminohexyl)-imido]-AMP-PNP-Atto647N | |
| Fluospheres | Thermo Fisher Scientific, Waltham, MA, USA | F8764 | amine-modified, 0.2 μm, yellow-green fluorescent |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Andor Solis | Andor Technology Ltd, Belfast, Northern Ireland | version 4.30 | |
| LabVIEW | National Instruments, Austin, Texas, USA | version 2012, 32bit; misc. hardware control | |
| MDS control software | AA Opto-Electronic, Orsay, France | version 2.03a | |
| Coherent Connection | Coherent Inc, Santa Clara, CA, USA | version 3 | |
| Igor Pro | WaveMetrics Inc, Portland, OR, USA | version 6.37 |
Referencias
- Nooren, I. M. A., Thornton, J. M. Diversity of protein-protein interactions. EMBO J. 22 (14), 3486-3492 (2003).
- Marsh, J. A., Teichmann, S. A. Structure, dynamics, assembly, and evolution of protein complexes. Annu Rev Biochem. 84, 551-575 (2015).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Hohng, S., Joo, C., Ha, T. Single-Molecule Three-Color FRET. Biophys J. 87 (2), 1328-1337 (2004).
- Person, B., Stein, I. H., Steinhauer, C., Vogelsang, J., Tinnefeld, P. Correlated movement and bending of nucleic acid structures visualized by multicolor single-molecule spectroscopy. ChemPhysChem. 10 (9-10), 1455-1460 (2009).
- Lee, J., Lee, S., Ragunathan, K., Joo, C., Ha, T., Hohng, S. Single-molecule four-color FRET. Angew Chem Int Ed. 49 (51), 9922-9925 (2010).
- Ratzke, C., Berkemeier, F., Hugel, T. Heat shock protein 90's mechanochemical cycle is dominated by thermal fluctuations. Proc Natl Acad Sci U S A. 109 (1), 161-166 (2012).
- Ratzke, C., Hellenkamp, B., Hugel, T. Four-colour FRET reveals directionality in the Hsp90 multicomponent machinery. Nat Commun. 5, 4192 (2014).
- Götz, M., Wortmann, P., Schmid, S., Hugel, T. A Multicolor Single-Molecule FRET Approach to Study Protein Dynamics and Interactions Simultaneously. Methods Enzymol. 581, 487-516 (2016).
- Yengo, C. M., Berger, C. L. Fluorescence anisotropy and resonance energy transfer: Powerful tools for measuring real time protein dynamics in a physiological environment. Curr Opin Pharmacol. 10 (6), 731-737 (2010).
- Wortmann,P , ., Götz M, ., Hugel T , . Cooperative Nucleotide Binding in Hsp90 and Its Regulation by Aha1. Biophys J. 113, 1711-1718 (2017).
- Dörfler, T., Eilert, T., Röcker, C., Nagy, J., Michaelis, J. Structural Information from Single-molecule FRET Experiments Using the Fast Nano-positioning System. J Vis Exp. (120), e54782 (2017).
- Stephanopoulos, N., Francis, M. B. Choosing an effective protein bioconjugation strategy. Nature chemical biology. 7 (12), 876-884 (2011).
- Sánchez-Rico, C., Voith von Voithenberg, L., Warner, L., Lamb, D. C., Sattler, M. Effects of Fluorophore Attachment on Protein Conformation and Dynamics Studied by spFRET and NMR Spectroscopy. Chemistry (Weinheim an der Bergstrasse, Germany). , (2017).
- Roy, R., Hohng, S., Ha, T. A practical guide to single-molecule FRET. Nat Methods. 5 (6), 507-516 (2008).
- Lee, N. K., et al. Three-color alternating-laser excitation of single molecules: monitoring multiple interactions and distances. Biophys J. 92 (1), 303-312 (2007).
- Kapanidis, A. N., Lee, N. K., Laurence, T. A., Doose, S., Margeat, E., Weiss, S. Fluorescence-aided molecule sorting: analysis of structure and interactions by alternating-laser excitation of single molecules. Proc Natl Acad Sci U S A. 101 (24), 8936-8941 (2004).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chem Soc Rev. 43 (4), 1156-1171 (2014).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nat Methods. 14, 174-180 (2017).
- Rabiner, L. R. A tutorial on hidden Markov models and selected applications in speech recognition. Proc IEEE. 77 (2), 257-286 (1989).
- Fink, G. A. . Markov Models for Pattern Recognition. , (2014).
- Giudici, P., Ryden, T., Vandekerkhove, P. Likelihood-Ratio Tests for Hidden Markov Models. Biometrics. 56 (3), 742-747 (2000).
- Visser, I., Raijmakers, M. E. J., Molenaar, P. C. M. Confidence intervals for hidden Markov model parameters. Br J Math Stat Psychol. 53 (2), 317-327 (2000).
- McKinney, S. A., Joo, C., Ha, T. Analysis of Single-Molecule FRET Trajectories Using Hidden Markov Modeling. Biophys J. 91 (5), 1941-1951 (2006).
- Bronson, J. E., Fei, J., Hofman, J. M., Gonzalez, R. L., Wiggins, C. H. Learning Rates and States from Biophysical Time Series: A Bayesian Approach to Model Selection and Single-Molecule FRET Data. Biophys J. 97 (12), 3196-3205 (2009).
- Greenfeld, M., Pavlichin, D. S., Mabuchi, H., Herschlag, D. Single Molecule Analysis Research Tool (SMART): an integrated approach for analyzing single molecule data. PLoS ONE. 7 (2), e30024 (2012).
- Schmid, S., Götz, M., Hugel, T. Single-Molecule Analysis beyond Dwell Times: Demonstration and Assessment in and out of Equilibrium. Biophys J. 111 (7), 1375-1384 (2016).
- Taipale, M., Jarosz, D. F., Lindquist, S. HSP90 at the hub of protein homeostasis: emerging mechanistic insights. Nat Rev Mol Cell Biol. 11 (7), 515-528 (2010).
- Trepel, J., Mollapour, M., Giaccone, G., Neckers, L. Targeting the dynamic HSP90 complex in cancer. Nat Rev Cancer. 10 (8), 537-549 (2010).
- Wayne, N., Bolon, D. N. Dimerization of Hsp90 is required for in vivo function. Design and analysis of monomers and dimers. J Biol Chem. 282 (48), 35386-35395 (2007).
- Ali, M. M. U., et al. Crystal structure of an Hsp90-nucleotide-p23/Sba1 closed chaperone complex. Nature. 440 (7087), 1013-1017 (2006).
- Southworth, D. R., Agard, D. A. Species-dependent ensembles of conserved conformational states define the Hsp90 chaperone ATPase cycle. Mol Cell. 32 (5), 631-640 (2008).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proc Natl Acad Sci U S A. 101 (35), 12893-12898 (2004).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An Oxygen Scavenging System for Improvement of Dye Stability in Single-Molecule Fluorescence Experiments. Biophys J. 94 (5), 1826-1835 (2008).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Rognoni, L., Stigler, J., Pelz, B., Ylänne, J., Rief, M. Dynamic force sensing of filamin revealed in single-molecule experiments. Proc Natl Acad Sci U S A. 109 (48), 19679-19684 (2012).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in Surface-Tethered Lipid Vesicles as a New Tool for Single Biomolecule Spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-mode waveguides for single-molecule analysis at high concentrations. Science. 299 (5607), 682-686 (2003).
- Panaretou, B., et al. ATP binding and hydrolysis are essential to the function of the Hsp90 molecular chaperone in vivo. EMBO J. 17 (16), 4829-4836 (1998).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados