È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Usando tre colori singola molecola FRET per studiare la correlazione delle interazioni della proteina
In questo articolo
Riepilogo
Qui, presentiamo un protocollo per ottenere dati di tre colori smFRET e la sua analisi con un ensemble 3D modello di Markov nascosto. Con questo approccio, gli scienziati possono estrarre informazioni cinetiche da sistemi proteici complessi, tra cui il cooperativity o interazioni correlate.
Abstract
Trasferimento di energia di risonanza di singola molecola Förster (smFRET) è diventata una tecnica ampiamente utilizzata biofisica per studiare la dinamica delle biomolecole. Per molte macchine molecolari in una proteine delle cellule devono agire insieme ai partner di interazione in un ciclo funzionale per adempiere il loro compito. L'estensione di due colori di multi-colore smFRET permette di sondare contemporaneamente più di una interazione o cambiamento conformazionale. Questo non solo aggiunge una nuova dimensione agli esperimenti di smFRET, ma offre anche la possibilità di studiare direttamente la sequenza di eventi e di rilevare interazioni correlate quando si utilizza un campione immobilizzato ed una fluorescenza di riflessione interna totale microscopio (TIRFM). Di conseguenza, multi-colore smFRET è un versatile strumento per lo studio biomolecolare complessi in maniera quantitativa e dettagliatamente in precedenza irraggiungibili.
Qui, dimostriamo come superare le sfide speciali di multi-colore smFRET esperimenti sulle proteine. Vi presentiamo protocolli dettagliati per ottenere i dati e per l'estrazione di informazioni cinetiche. Questo include i criteri di selezione traccia, separazione di stato e il recupero delle traiettorie di stato dai dati rumorosi utilizzando un ensemble 3D modello di Markov nascosto (HMM). Rispetto ad altri metodi, le informazioni cinetiche non viene recuperate da istogrammi di tempo di permanenza, ma direttamente dall'HMM. Il quadro di massima verosimiglianza ci permette di valutare criticamente il modello cinetico e fornire significative incertezze per le tariffe.
Applicando il nostro metodo per la proteina di scossa di calore 90 (Hsp90), siamo in grado di districarsi tra l'associazione del nucleotide e globali cambiamenti conformazionali della proteina. Questo ci permette di osservare direttamente la cooperatività tra le due tasche di associazione del nucleotide del dimero Hsp90.
Introduzione
Molte proteine assolvere la loro funzione in dinamici complessi con altre molecole, mediati dai cambiamenti conformazionali e transitorie associazioni su una vasta gamma di scale cronologiche1,2,3. Accoppiato ad una fonte di energia esterna (ad es., ATP) queste interazioni dinamiche possono portare alla direzionalità in un ciclo funzionale e, infine, mantenere il non-equilibrio stazionario in una cella, il prerequisito per la vita.
Al fine di comprendere appieno queste macchine molecolari, una descrizione statica guidata da studi strutturali non è sufficiente. Inoltre, è essenziale avere conoscenza del modello cinetico e per determinare le costanti cinetiche tasso. Diversi metodi esistenti consentono ai ricercatori di studiare le dinamiche delle interazioni binarie tra due molecole di interesse, ad esempio, la risonanza plasmonica di superficie, metodi di rilassamento con una lettura spettroscopica (ad es., salto o flusso interrotto tecniche) e risonanza magnetica nucleare. Tuttavia, la loro applicabilità è nella maggior parte dei casi limitati a semplici sistemi di due stati (ad esempio, un limite e uno stato non associato) a causa della media inerente agli esperimenti di massa. Nei casi in cui siano coinvolti più Stati o intermedi, cedono solo una miscela complessa delle costanti di tasso. Metodi di singola molecola come pinzette ottiche o magnetiche o due colori smFRET, cioè, un donatore e un fluoroforo accettore, con un campione di superficie-immobilizzata possono recuperare le costanti di velocità per tutti osservati cambiamenti conformazionali. Tuttavia, quando si tratta di interazioni che interessano più di un binding sito, questi metodi rimangono limitati e le informazioni sulle interazioni possibili correlazioni di due (o più) saranno sempre accessibili via indirette conclusioni da una serie di esperimenti.
Multi-colore smFRET4,5,6,7,8,9 offre l'opportunità di studiare l'interazione tra questi componenti direttamente, in tempo reale e sotto condizioni quasi-fisiologiche10. Questo permette di indagare, ad esempio, l'associazione dipendente dalla conformazione di un ligando o di un'altra proteina8,9,11. L'approccio globale presentato qui è per etichettare la proteina di interesse alle posizioni specifiche, per allegare una proteina alla superficie della camera di misura e per monitorare l'intensità di fluorescenza nel tempo su un TIRFM di tipo a prisma (per dettagli Vedi 9 , 12). la prossimità spaziale delle tinture diverse quindi può essere determinata dal trasferimento di energia tra di loro. Strategie di etichettatura può variare da proteina a proteina (rivisto in 13) ed esistono linee guida per evitare artefatti nelle misure smFRET14.
Poiché un colorante donatore può trasferire energia al ricettore diversi coloranti in un esperimento di smFRET multi-colore, la posizione relativa di tutti i coloranti non è accessibile dall'eccitazione di uno tintura solo15,16. Ma in combinazione con alternanza di eccitazione laser (ALEX17e ha commentato in 18) questo metodo fornisce tutte le informazioni spazio-temporali a frazioni di secondo e risoluzione sub-nanometrica.
In linea di principio, ad alta risoluzione informazioni strutturali possono essere ottenute utilizzando le distanze di Inter-tintura calcolato dalla combinazione di tutte le intensità di fluorescenza in un esperimento di smFRET multi-colore con ALEX. Tuttavia, qui ci concentriamo sulla identificazione dello stato e separazione così come l'estrazione di modelli cinetici, dove è indispensabile smFRET multi-colore. Quando "solo" determinazione della struttura di triangolazione è voluta, una serie di esperimenti di smFRET di due colori più semplice con alto rapporto segnale-rumore può essere eseguita12,19.
Usiamo la fluorescenza parziale ( ) come un proxy per il trasferimento di energia tra due fluorofori7. Il PF è calcolato dall'intensità di fluorescenza analoga per l'efficienza FRET di un esperimento di due colori:
) come un proxy per il trasferimento di energia tra due fluorofori7. Il PF è calcolato dall'intensità di fluorescenza analoga per l'efficienza FRET di un esperimento di due colori:

Dove, 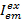 è l'intensità di emissione canale em dopo l'eccitazione con colore ex, e c è il ricettore con la lunghezza d'onda più lunga. Canali di rilevazione rappresentano la stessa posizione nella camera del campione ma registrare diversi campi di spettro della luce di fluorescenza. Lo stesso identificatore per eccitazione e di emissione sono utilizzati nel presente protocollo (cioè, "blu", "verde" e "rosso").
è l'intensità di emissione canale em dopo l'eccitazione con colore ex, e c è il ricettore con la lunghezza d'onda più lunga. Canali di rilevazione rappresentano la stessa posizione nella camera del campione ma registrare diversi campi di spettro della luce di fluorescenza. Lo stesso identificatore per eccitazione e di emissione sono utilizzati nel presente protocollo (cioè, "blu", "verde" e "rosso").
A causa delle carenze sperimentali le intensità di fluorescenza misurata dipendono non solo sul trasferimento di energia, ma anche sulle proprietà fluoroforo e installazione. Per ottenere l'efficienza di trasferimento di vera energia tra due fluorofori, le intensità misurate devono essere corretto. La procedura seguente si basa sul riferimento9. Fattori di correzione per perdita apparente (LC, vale a dire, la rilevazione di fotoni da un fluoroforo in un canale destinata per un altro colorante) e gamma apparente (ag, vale a dire, il rendimento quantico di fluorescenza del colorante e la efficienza di rivelazione del canale) sono ottenuti da tracce di singola molecola che mostrano un accettore candeggio evento.
La dispersione del colorante donatore in ogni canale possibile acceptor viene calcolata da tutti i punti dati dalle tracce di fluorescenza registrata dove l'accettore sbiancato ma il donatore è ancora fluorescente (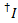 ):
):
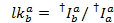
La mediana dell'istogramma perdita viene utilizzata come il fattore di perdita apparente. Dopo la correzione per la perdita, il fattore gamma apparente è determinato dallo stesso insieme di tracce. Viene calcolato dividendo il cambiamento di fluorescenza nel canale ricettore dal cambiamento di fluorescenza nel canale donatore con lo sbiancamento del colorante accettore:

Dove c è ancora una volta il canale di rilevamento per il ricettore con la lunghezza d'onda più lunga. La mediana della distribuzione risultante viene utilizzata come il fattore di correzione apparente.
La corretta intensità in ciascun canale sono stati ottenuti da:
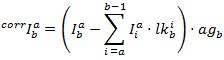
Il PF è quindi calcolato in base alla:

Diverse popolazioni possono essere separati nello spazio multi-dimensionale, attraversato dal s PF. La posizione e la larghezza di ogni stato è determinata inserendo i dati con funzioni gaussiane multi-dimensionale. Successiva ottimizzazione di uno eh globale basato su tutte le tracce PF fornisce una descrizione quantitativa della cinetica osservata. Anche piccoli cambiamenti delle tariffe sono rilevabili.
HMMs consentono di dedurre un modello di stato da una raccolta di tracce tempo rumoroso. Il sistema è considerato di essere in uno di una serie di discreti, stati nascosti in qualsiasi dato momento e l'effettiva osservazione (cioè, l'emissione) è una funzione probabilistica di questo stato nascosto20. Nel caso di dati di smFRET TIRFM, l'emissione probabilità bio a stato possono essere modellati da funzioni di densità di probabilità gaussiana continuo. A intervalli di tempo discreti regolarmente spaziati, le transizioni da uno a un altro stato possono verificarsi secondo la probabilità di transizione che è tempo-invariante e dipende solo dallo stato corrente. La matrice di transizione A contiene queste probabilità di transizione unij tra tutti gli stati nascosti. La distribuzione di stato iniziale  dà le probabilità relative allo stato
dà le probabilità relative allo stato 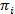 per il primo punto di tempo di una traccia di tempo. Utilizzando un approccio di massima verosimiglianza, questi parametri possono essere ottimizzati per meglio descrivere i dati con l'avanti-indietro e Baum-Welch algoritmi20,21. Questo produce gli stimatori di massima verosimiglianza (MLE). Infine, la sequenza di stato che più probabilmente prodotta la traiettoria delle osservazioni può essere dedotta con l'algoritmo di Viterbi. A differenza di altre analisi HMM del smFRET dati24,25,26 non usiamo il HMM come una mera "smoothing" dei dati ma il modello di stato cinetico l'Estratto dal set di dati senza la necessità per il montaggio di tempo di permanenza istogrammi27. Analisi HMM sono fatto con in-House scripts utilizzando Igor Pro. Implementazione del codice si basa su riferimento21. Forniamo un kit di software e dati esemplari sulla nostra pagina Web per seguire le sezioni 5 e 6 del presente protocollo (https://www.singlemolecule.uni-freiburg.de/software/3d-fret). Software completo è disponibile su richiesta.
per il primo punto di tempo di una traccia di tempo. Utilizzando un approccio di massima verosimiglianza, questi parametri possono essere ottimizzati per meglio descrivere i dati con l'avanti-indietro e Baum-Welch algoritmi20,21. Questo produce gli stimatori di massima verosimiglianza (MLE). Infine, la sequenza di stato che più probabilmente prodotta la traiettoria delle osservazioni può essere dedotta con l'algoritmo di Viterbi. A differenza di altre analisi HMM del smFRET dati24,25,26 non usiamo il HMM come una mera "smoothing" dei dati ma il modello di stato cinetico l'Estratto dal set di dati senza la necessità per il montaggio di tempo di permanenza istogrammi27. Analisi HMM sono fatto con in-House scripts utilizzando Igor Pro. Implementazione del codice si basa su riferimento21. Forniamo un kit di software e dati esemplari sulla nostra pagina Web per seguire le sezioni 5 e 6 del presente protocollo (https://www.singlemolecule.uni-freiburg.de/software/3d-fret). Software completo è disponibile su richiesta.
Tempo punti nei dati con PF < -1 o PF > 2 in qualsiasi canale di rilevamento sono assegnate la probabilità di emissione minima per tutti gli Stati (10-200). Questo previene le transizioni artificiale in questi punti di dati.
I parametri per la probabilità di emissione sono ottenuti dal fit dell'istogramma 3D PF con funzioni gaussiane come descritto al punto 5.7. Questi parametri sono tenuti fissi durante l'ottimizzazione dell'eh.
Nell'approccio presentato, il vettore di distribuzione stato iniziale e la matrice di transizione vengono utilizzati globalmente per descrivere l'intero ensemble di tracce. Essi vengono aggiornati basato su tutti i N molecole dal set di dati secondo riferimento27.
Parametri di avvio per la distribuzione di stato iniziale sono determinati dalle proiezioni 2D dell'istogramma PF (punto 5.3) e le probabilità di transizione sono riportate a 0,05 fatta eccezione per le probabilità di rimanere nello stesso stato, che sono scelti tali che la probabilità di lasciare un certo stato viene normalizzata all'unità.
Un metodo di analisi di probabilità viene utilizzato per dare gli intervalli di confidenza (CIs) per tutte le transizione tariffe21,22, che servono come stime significative per loro incertezza. Per calcolare i limiti dell'IC per un tasso specifico, la probabilità di transizione di interesse è fissata su un valore diverso dal MLE. Questo produce la prova modello λ'. Un test di ratio (LR) probabilità del rischio 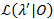 dato il set di dati 0 viene eseguita secondo:
dato il set di dati 0 viene eseguita secondo:
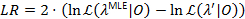
Il 95% di confidenza associato per il parametro è raggiunto quando LR supera 3.841, il quantile 95% di un X2-distribuzione con un grado di libertà22,23.
Il potere del metodo è dimostrato utilizzando la Hsp90. Questa proteina abbondante è trovata nei batteri e negli eucarioti ed è parte della risposta stress cellulare28. È un promettente bersaglio di farmaci nel trattamento di cancro29. Hsp90 è un omodimero con una tasca di legame del nucleotide nel dominio N-terminale di ogni subunità30. Può subire transizioni tra almeno due conformazioni globalmente distinti, uno chiuso e uno N-terminale a forma di V aperta, conformazione19,31,32. La natura dimerica direttamente solleva il problema dell'interazione tra i due siti di legame del nucleotide in Hsp90.
Di seguito, forniamo un protocollo passo-passo per l'acquisizione dati e l'analisi di un esperimento di tre colori smFRET il lievito Hsp90 e del nucleotide. L'associazione dipendente dalla conformazione di fluorescente contrassegnati AMP-PNP (AMP-PNP *, un analogo non hydrolyzable dell'ATP) viene analizzato. L'applicazione della procedura descritta permette lo studio di associazione del nucleotide e allo stesso tempo i cambiamenti conformazionali di Hsp90 e quindi rivela la cooperatività tra le due tasche di associazione del nucleotide di Hsp90.
Protocollo
1. installazione e prerequisiti
- Eseguire le misure di smFRET multi-colore su un TIRFM di tipo a prisma. Una descrizione di una configurazione di due colori come una pubblicazione di Giove è dato riferimento a12.
- Costruire un TIRFM multi-colore. Un layout generale è dettagliato in 9.
- Uso commutabile, pompato a diodi laser onda continua a stato solido, che rendono inutile l'uso di persiane meccaniche nei percorsi di eccitazione.
- Impiegare un prisma asimmetrico, di forma allungato che impedisce la riflessione posteriore del fascio di eccitazione dal lato posteriore per inserire l'obiettivo.
- Utilizzare lenti di 2 pollici acromatico asferiche silice fusa nei percorsi di rilevamento che raccolgono quanta più luce possibile e prevenire auto-fluorescenza e aberrazioni, per esempio, distorsioni nelle regioni fuori centro dell'immagine.
- Concentrarsi ogni percorso di rilevamento sul chip di EMCCD con una lente separata. Questo consente una messa a fuoco ottimale di ogni canale di rilevamento.
Attenzione: Classe 3B laser sono utilizzati nella TIRFM. Questo significa che sono pericolosi se l'occhio è esposto direttamente, ma diffuse riflessioni non sono nocivi. Garantire la conformità con laser precauzioni di sicurezza secondo le normative di governo locale prima di far funzionare il sistema.
- Determinare i fattori di correzione per le proprietà di configurazione e fluoroforo in anticipo utilizzando campioni di dsDNA.
- Utilizzare un campione di dsDNA ad alta-FRET per ogni colorante in combinazione con il ricettore avendo la lunghezza d'onda più lunga di eccitazione (per l'installazione di presentato: Atto488-Atto647N, Atto550-Atto647N, Atto594-Atto647N). Assicurarsi che il DNA è ulteriormente modificato con una biotina.
- Diluire il campione a 5 nM con buffer di TNM (5 mM Tris-HCl pH 7.5, 5 mM NaCl, 20 mM MgCl2) e 2 mM Trolox (utilizzare anche questo buffer per misura).
- Immobilizzare il dsDNA come descritto per Hsp90 ai punti 2.5 e 2.7.
- Calcolare i fattori di correzione per perdita apparente (LC) e gamma apparente (ag) da tracce di singola molecola che mostrano un evento sbiancante accettore.
- Costruire una camera di flusso che è un panino di una diapositiva di quarzo passivato PEG/biotina-PEG, una sottile pellicola che è adesivo su entrambi i lati e un vetrino coprioggetti. Per i protocolli di dettaglio per la pulizia del quarzo diapositive e passivazione vedere riferimento9.
- Uso di spessore (3 mm) quarzo diapositive per geometricamente impedire la raccolta di luce laser sparsi nell'interfaccia di quarzo-glicerolo tra il prisma e la diapositiva di quarzo funzionalizzati.
- Utilizzare un sottile (40 µm) pellicola che è spruzzato con adesivo sul lato non adesivo sigillante. Il film sottile riduce la distanza tra l'obiettivo e di molecole di superficie-collegato. Riscaldare fino a 80 ° C e premere.
- Posto un coprioggetto in alto a sinistra. Riscaldare fino a 80 ° C e premere. Utilizzare una goccia di glicerolo quando mettendo la camera di flusso sopra il prisma.
Nota: I materiali del prisma e le diapositive di quarzo così come il glicerolo sono indice-abbinati.
- Rapidi Hsp90 da Saccharomyces cerevisiae sotto forma di due mutanti di punto singolo cisteina alle posizioni D61 o Q385. Aggiungere un motivo di arrotolato-arrotola C-terminale per evitare la dissociazione del dimero a concentrazioni picomolare. Etichettare le proteine mutanti separatamente e scambiare i monomeri per ottenere eterodimeri etichettati con Atto488 alla posizione dell'amminoacido 61 e Atto550 dell'amminoacido posizione 3859.
2. misurazione
- Avviare il software della fotocamera e impostare i parametri di imaging come specificato di seguito:
- Impostare la temperatura per il sensore di raffreddamento più basso possibile (-95 ° C con raffreddamento ad acqua esterna) per diminuire il rumore di corrente di buio.
- Utilizzare le impostazioni della fotocamera che sono ottimizzate per la registrazione di singola molecola: 3,3 µs spostamento verticale velocità, tensione normale orologio verticale, 17 MHz 16 bit orizzontale leggere, pre-amplificazione guadagno 3, guadagno di moltiplicatore di elettroni 1.000.
- Impostare l'attivazione dell'acquisizione di "Esterno" e il tempo di esposizione di 70 ms. film di Record con una lunghezza di 750 cicli di acquisizione.
Nota: Spegnere la luce di camera quando la fotocamera è l'acquisizione per evitare la saturazione del sensore EMCCD.
- Creare una cartella sul SSD locale per la misurazione. Nel software impostazioni andare al pilota < Auto-Save > abilitare < Auto-save > e scegliere il formato di file "Tiff" per acquisizione di film. Selezionare la cartella sul SSD come percorso di salvataggio automatico.
- Avviare il software che controlli l'acusto-ottico sintonizzabile filtro (AOTF), il software che controlla il funzionamento dei laser e il software di trigger che sincronizza i laser, AOTF, persiane nel percorso di rilevazione e telecamere. Regolare la potenza del laser con il AOTF (ca. 3 mW prima di entrare il prisma) e caricare il pattern di attivazione corretto.
- Montare il supporto del campione con il prisma e la camera di flusso, collegare il tubo, inserire il tubo di aspirazione in una microcentrifuga tazza e collegare il tubo di uscita di una pompa a siringa. A filo della camera con circa 150 µ l di tampone, allineare l'eccitazione fascio e messa a fuoco. Candeggina qualsiasi contaminanti fluorescente sulla superficie spostando lentamente lungo il campo di rilevamento completo della diapositiva con un laser potente di circa 10 mW per tutti i laser. Questa operazione richiede circa 1 h.
Nota: se non diversamente specificato, il buffer utilizzato contiene 40 mM HEPES pH 7.5, 150 mM KCl e 10mm MgCl2. - A filo circa 300 µ l di una soluzione di NeutrAvidin (0,25 mg/mL in tampone) nell'alloggiamento e incubare per 1 min. Flush fuori NeutrAvidin non associato con buffer e a filo circa 300 µ l di una soluzione di BSA (0,5 mg/mL in tampone) attraverso la camera.
Nota: Difetti di questo blocco restanti funzionalizzazione superficiale di adsorbimento non specifico di BSA in superficie. - Immobilizzare l'esempio caricando la camera di flusso con circa 150 µ l della biotinilato ed etichettati Hsp90 alle concentrazioni aumentanti (diluite in tampone + 0,5 mg/mL BSA) fino a quando non viene raggiunta una sufficiente densità di superficie, che di solito è il caso ad una concentrazione di 5-22. Sciacquare la proteina non associata con circa 300 µ l di tampone + 0,5 mg/mL BSA.
- A filo in 150 µ l di 25 nM AMP-PNP * nel buffer + 0,5 mg/mL BSA. Lasciare Incubare per 5 minuti e ripetere questo passaggio una volta per garantire la concentrazione corretta del nucleotide.
Nota: Per gli esperimenti in presenza di ulteriori, senza etichetta AMP-PNP, anche aggiungere 250 µM AMP-PNP. - Iniziare con l'acquisizione di dati. Per una quantità appropriata di dati acquisire circa 20 film che dura circa 1,5 ore.
- Spostare la posizione della camera del campione perpendicolare alla trave di eccitazione con un piezo stepper per modificare il campo visivo.
- Regolare la messa a fuoco con il z-piezo che controlla l'altezza dell'obiettivo, se necessario. Questo non dovrebbe essere necessario troppo spesso quando la camera di misura è montata senza inclinazione.
- Preparare la registrazione delle telecamere premendo < prendere segnale > il software della fotocamera e iniziare i cicli di eccitazione/acquisizione utilizzando il pulsante < Start > nel software trigger. Verrà avviata l'acquisizione dell'intensità di fluorescenza.
- Prima registrazione di un filmato con perline fluorescenti che mostrano l'emissione di fluorescenza nell'intervallo spettrale di tutti i canali di rilevamento del programma di installazione per eseguire una registrazione del canale. Quindi, rilevare posizioni della perla nel film calibrazione ricercando le macchie brillanti e determinare che la posizione centrale da una gaussiana adatta per il profilo di intensità. Salvare le coordinate di perle che si trovano in tutti i canali e montare entrambi l'offset di mappatura in x - e y-direzione con un 2D polinomio di grado tre9.
3. selezione di tracce di singola molecola
- Analisi dei dati avviene con in-House scripts utilizzando Igor Pro. Caricare tutti gli script necessari con l'apertura di "iniTIRF.ipf" e selezionare il corretto tipo di esperimento.
Nota: In seguito, < Button > specifica di elementi selezionabili nel menu o l'interfaccia utente. Chiamate di funzione sono indicate tra virgolette, ad esempio, "Print"Hello World"". Questi comandi possono essere incollati alla riga di comando di Igor Pro (senza le virgolette che lo contiene). - Assicurarsi che i parametri per la registrazione del canale di rilevamento vengono caricati.
- Avviare l'interfaccia grafica facendo clic su < smFRET nuovo | Analisi GUI >.
- Caricare i film (cioè, la sequenza di fotogrammi con 512 x 512 pixel archiviati come pile TIFF a 16 bit) che tengono l'intensità nei rispettivi canali. Eseguire questa operazione premendo il pulsante < carico Film > e selezionando i file dal cosiddetto "Maestro" e "slave" telecamere uno dopo l'altro.
- Identificare le posizioni di potenziale singole molecole tramite la ricerca per le macchie più brillanti nella somma dei primi cinque fotogrammi in un certo canale di rilevamento. Calcolare le posizioni corrispondenti in tutti gli altri canali di rilevamento dalla mappatura canale. Per ottenere la traccia di intensità di fluorescenza, sommare l'intensità di un quadrato di pixel intorno alla posizione centrale per ogni fotogramma. Eseguire questa operazione premendo il pulsante < trovare tracce > nella GUI.
Nota: La lunghezza del lato del quadrato (in pixel) è dato da: 2 * < somma Pxs > + 1. - Per ogni molecola, è necessario calcolare una traccia comune intensità prima come la somma di tutte le tracce di questo posto con lo stesso colore di eccitazione. Valutare il profilo di intensità della molecola in tutti i canali per i seguenti criteri:
- Un grosso modo piatto altopiano dell'intensità di crudo misto e una decolorazione singolo passo per tutti i colori di eccitazione, anticorrelated comportamento nei canali di rilevamento, rilevamento della fluorescenza rossa (che indica FRET per un associato AMP-PNP *) almeno una volta all'interno del traccia e non più passaggi nella fluorescenza rossa, che indicherebbe la presenza di due AMP-PNP * associato a un dimero di Hsp90.
- Se questi criteri sono soddisfatti, salvare le tracce di fluorescenza dello spot per ulteriori analisi. Fare questo selezionando la traccia con il cursore e premendo il pulsante < Salva > nel grafico "timeline". Controllare manualmente le tracce di intensità in tutti i tre canali di rilevamento dopo l'eccitazione blu per circa 200 molecole per il film.
4. calcolo delle tracce parziali di fluorescenza
- Visualizzare tutte le tracce di intensità di fluorescenza per una delle molecole salvate. Utilizzare i cursori nel grafico per selezionare intervalli di tempo.
- Selezionare un intervallo di tempo in cui tutti i fluorofori sono sbiancati già. L'intensità media sfondo viene calcolato da questa gamma e sottratto la traccia di intensità in ogni canale. Fare questo premendo il pulsante < sfondo >.
- Selezionare l'intervallo di efficienza FRET, dove almeno entrambi i coloranti attaccati al Hsp90 (Atto488 e Atto550) sono presenti. Assicurarsi di escludere le tracce che contengono un evento lampeggio (Vedi Figura 3B). Questi eventi sono caratterizzati da un'intensità di fluorescenza di drop-in un canale senza un accompagnamento aumento in qualsiasi altro canale.
- Calcolare le tracce PF . Eseguire questa operazione premendo il pulsante < PF Calc >. I fattori di correzione predefinite di apparente perdita (LC) e gamma apparente (ag) vengono applicati all'intensità crudo per correggere foto-fisiche e proprietà di installazione.
5. popolazione selezione e montaggio di istogramma 3D
- Rimuovere le molecole che mostrano un rapporto segnale-rumore basso nelle tracce PF . Molecole che superano l'intervallo [-1; 2] in qualsiasi traccia PF per oltre il 10% dei fotogrammi vengono rimosse dal set di dati. Fare questo mediante l'esecuzione di "RemoveTracesLowSNR()" dalla finestra di comando.
- Calcolare le proiezioni 2D binned dei dati PF . Trama
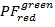 sopra
sopra 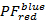 e
e 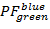 sul
sul 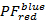 nell'intervallo [-0,5; 1,5] con una risoluzione di 100 x 100 bidoni. A tale scopo, eseguire:
nell'intervallo [-0,5; 1,5] con una risoluzione di 100 x 100 bidoni. A tale scopo, eseguire:- "HistFret2D ("r_b","r_g", binHist = 100)"
- "HistFret2D ("r_b","g_b", binHist = 100); MoveWindow 553,5, 42.5, 1055.25, 508,25"
- Determinare la relativa popolazione di ogni stato distinguibile nelle proiezioni 2D.
- Portare in primo piano il grafico appropriato ed eseguire "panelHist2DCount()".
- Premere il tasto < Init > e disegnare un poligono a mano libera intorno alla vetta.
- Fare clic sul pulsante < Count >. Il numero di punti dati nel poligono e il numero totale di punti dati nella proiezione vengono stampati nella finestra di comando.
- Preparare un istogramma 3D dei dati PF eseguendo "HistFret3D ("g_b","r_b","r_g")".
- Normalizzare l'istogramma 3D per un integrale di 1. Eseguire le seguenti operazioni:
- "NewDataFolder/S fit0"
- "Duplicato/O:: FRET: Hist3D, Hist3D"
- "Variabile /G div = sum(Hist3D)*(DimDelta(Hist3D,0)) ^ 3"
- "Hist3D = div; Stampa div"
- Fornire parametri iniziali per la gaussiana 3D fit e preparano le strutture dei dati necessarie.
- Eseguire "Gauss3D_initParam(); modificare W_coef_old."
- Aggiungere le popolazioni dello stato alla fine del vettore parametro.
- Eseguire "Gauss3D_prepareFit()."
Nota: W_coef_old è un vettore che contiene i parametri iniziali per la vestibilità. Questo significa che per ogni stato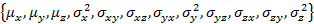 e la popolazione dello stato, che è concatenata alla fine del vettore. Assicurarsi che la matrice di covarianza è simmetrica.
e la popolazione dello stato, che è concatenata alla fine del vettore. Assicurarsi che la matrice di covarianza è simmetrica.
- Misura la somma di S 3D gaussiana funzioni per l'istogramma 3D di PF , con S il numero di stati distinguibili.
- Eseguire "do3D()." Questo può richiedere un'ora o più su un normale ufficio PC, a seconda della qualità dei parametri iniziali.
- Eseguire "postprocessFitMultiGauss3D(); evalFitMultiGauss3D(); modificare W_coef."
- Visualizzare il risultato in forma. Per ognuna delle due proiezioni 2D, utilizzare i seguenti comandi:
- "contourPF3D_new(0); contourPF3D_new(1); contourPF3D_new(2); contourPF3D_new(3); contourPF3D_new(4)"
- "contourPF3D_colorize()"
6. analisi cinetica con 3D Ensemble HMM
- Preparare un ensemble HMM eseguire per estrarre le informazioni cinetiche. Un HMM è ottimizzato da tutte le molecole nel set di dati. Utilizzare le informazioni ottenute nel passaggio precedente per definire la posizione e la larghezza di ogni stato nello spazio 3D PF .
- Inizializzare l'interfaccia di utente HMM (< HMM | Init HMM >) e scegliere il numero appropriato di Stati (nel caso i dati di Hsp90 questo significa < NumStates > = 5), il numero di dimensioni del segnale in ingresso (< NumDims > = 3) e il tipo di input (< Input Type > = "FRET bgr 3D").
- Ottimizzare i parametri del HMM lasciando il software convergono la probabilità che il HMM (eseguire "prepENS_CONVERGE_gB(GetDataFolder(1), -14)") fino a quando il cambiamento nella matrice di transizione rispetto all'iterazione precedente scende sotto una soglia (10 -14 di per la somma della variazione assoluta per ogni probabilità di transizione). Questo produce la MLE per le probabilità di transizione in circa un'ora su un normale PC in ufficio.
- Ripetere la selezione della popolazione, raccordo gaussiana e l'ottimizzazione di HMM per sottoinsiemi di dati (ad esempio, il 75% del set di dati completo). Se il set di dati completo è stata fusa da diversi esperimenti, ripetere l'ottimizzazione anche per ciascuno dei singoli esperimenti. Analisi di sottogruppi permette di stimare l'incertezza della selezione manuale della popolazione e la variabilità all'interno del set di dati.
- Calcolare l'IC per le probabilità di transizione, che una relazione sull'eterogeneità di set di dati e la precisione dell'eh.
- Ottenere una stima approssimativa dei limiti CI eseguendo "cd $(radice: path3Dimport +"HMM"); loop_getCI_estimate_limits()."
- Calcolare i limiti esatti dell'IC eseguendo:
- "loop_getCI_HMM_converge(1)"
- "CIresults_conv_new()"
- "cd:: HMM_CIresult; reportCI_conv()"
- "cd:: cmp_CI_conv; CI_plot2 ("HMM", doAppend = 0) "
- Condensare le informazioni cinetiche disponibili per semplificare l'interpretazione.
- Raccogliere informazioni sul tempo, un nucleotide con etichettato rimane associato a Hsp90. Fare questo comprimendo gli Stati associati a AMP-PNP * (cioè, S1, S2 e S3, vedi anche Figura 2) e compilare l'istogramma del tempo di permanenza. Eseguire "cd $(radice: path3Dimport +"HMM"); collapse_states_get_DT({0,1,1,1,0}) ", che combina la dimora di dichiara S0 e S4 così come S1, S2e S3.
- Estrarre i tempi di permanenza dal percorso di Viterbi per ciascuno stato di interesse e confrontare l'istogramma risultante di tempo di permanenza per diverse condizioni sperimentali. Eseguire "plot_collapsed_DT_Hist (wDTo_01110_record1)."
- Per un quadro più dettagliato, crollo degli Stati che hanno distinto PF ma sono funzionalmente identici per facilitare l'ulteriore analisi dei dati, ad esempio, nel caso di un esperimento a tre colori con Hsp90 con etichetta e del nucleotide, Stati S2 e S 3 può essere compresso. Eseguire "collapse_states_get_DT({0,1,2,2,4})."
Risultati
Multi-colore smFRET misure permettono la rilevazione diretta di correlazione tra due o più siti di interazione distinte. Questo rende la tecnica unica per studiare sistemi multi-componenti, ad esempio complessi proteici. Ci concentriamo sulla presentazione di un esperimento di tre colori smFRET qui, che serve come un esempio illustrativo.
Il flusso di lavoro generale del metodo è illustrato nella Figura 1<...
Discussione
Vi presentiamo la procedura sperimentale per ottenere dati di smFRET tre colori per un sistema complesso della proteina e una descrizione dettagliata dell'analisi di queste misurazioni. Questo approccio offre la possibilità unica di valutare direttamente la correlazione tra più siti di interazione o cambiamenti conformazionali.
Al fine di ottenere dati di singola molecola multi-colori adatti sulle proteine è importante eseguire misurazioni riproducibili a un livello basso rumore. Ciò può ...
Divulgazioni
Gli autori non dichiarano conflitti di interesse.
Riconoscimenti
Questo lavoro è finanziato dalla Fondazione di ricerca tedesca (INST 39/969-1) e il Consiglio europeo della ricerca attraverso l'accordo di sovvenzione del CER n. 681891.
Materiali
| Name | Company | Catalog Number | Comments |
| Setup | |||
| vibration-damped optical table | Newport, Irvine, CA, USA | RS2000 | |
| OBIS 473nm LX 75mW LASER | Coherent Inc, Santa Clara, CA, USA | 1185052 | |
| OBIS 532nm LS 50mW LASER | Coherent Inc, Santa Clara, CA, USA | 1261779 | |
| OBIS 594nm LS 60mW LASER | Coherent Inc, Santa Clara, CA, USA | 1233470 | |
| OBIS 637nm LX 140mW LASER | Coherent Inc, Santa Clara, CA, USA | 1196625 | |
| laser control unit | Coherent Inc, Santa Clara, CA, USA | 1234465 | Scientific Remote |
| aspheric telescope lenses | Thorlabs Inc, Newton, New Jersey, USA | d=25.4mm, f=50mm and f=100mm | |
| CF ex1 | AHF analysentechnik AG, Tübingen, Germany | ZET 473/10 | cleanup filter excitation |
| CF ex2 | AHF analysentechnik AG, Tübingen, Germany | ZET 532/10 | cleanup filter excitation |
| CF ex3 | AHF analysentechnik AG, Tübingen, Germany | ZET 594/10 | cleanup filter excitation |
| CF ex4 | Thorlabs Inc, Newton, New Jersey, USA | FL635-10 | cleanup filter excitation |
| DM ex1 | AHF analysentechnik AG, Tübingen, Germany | ZQ594RDC | dichroic mirror excitation |
| DM ex2 | AHF analysentechnik AG, Tübingen, Germany | 570DCXR | dichroic mirror excitation |
| DM ex3 | AHF analysentechnik AG, Tübingen, Germany | ZQ491RDC | dichroic mirror excitation |
| AOTFnC-Vis | AA Opto-Electronic, Orsay, France | ||
| λ/4 plate | Thorlabs Inc, Newton, New Jersey, USA | AQWP05M-600 | |
| CFI Apo TIRF 100x | Nikon Instruments Inc, Melville, NY, USA | high-NA objective | |
| piezo focus positioner MIPOS 250 CAP | piezosystem jena GmbH, Jena, Germany | Piezo Controller NV 40/1 CLE | |
| piezo stepper | Newport, Irvine, CA, USA | PZA12 | PZC200-KT NanoPZ Actuator Kit |
| achromatic aspheric lenses | Qioptiq Photonics GmbH & Co. KG, Göttingen, Germany | G322-304-000 | d=50mm, f=200mm |
| adjustable optical slit | Owis GmbH, Staufen i. Br., Germany | 27.160.1212 | max. aperture 12 x 12 mm |
| DM det1 | AHF analysentechnik AG, Tübingen, Germany | T 600 LPXR | dichroic mirror detection |
| DM det2 | AHF analysentechnik AG, Tübingen, Germany | H 560 LPXR superflat | dichroic mirror detection |
| DM det3 | AHF analysentechnik AG, Tübingen, Germany | HC BS R635 | dichroic mirror detection |
| BP det1 | AHF analysentechnik AG, Tübingen, Germany | 525/40 BrightLine HC | bandpass filter detection |
| BP det2 | AHF analysentechnik AG, Tübingen, Germany | 586/20 BrightLine HC | bandpass filter detection |
| BP det3 | AHF analysentechnik AG, Tübingen, Germany | 631/36 BrightLine HC | bandpass filter detection |
| BP det4 | AHF analysentechnik AG, Tübingen, Germany | 700/75 ET Bandpass | bandpass filter detection |
| optical shutters detection | Vincent Associates, Rochester, NY, USA | Uniblitz VS25S2T0 | |
| EMCCD iXon Ultra 897 | Andor Technology Ltd, Belfast, Northern Ireland | ||
| digital I/O card, PCIe-6535 | National Instruments, Austin, Texas, USA | ||
| syringe pump | Harvard Apparatus, Holliston, MA, USA | PHD22/2000 | |
| Name | Company | Catalog Number | Comments |
| Flow chamber | |||
| quartz slides | G. Finkenbeiner Inc, Waltham, MA, USA | Spectrosil2000, h=3mm | |
| TEGADERM film | 3M Deutschland GmbH, Neuss, Germany | 1626W | 10 x 12cm |
| spray adhesive | 3M Deutschland GmbH, Neuss, Germany | Photo Mount 050777 | |
| glycerol | Carl Zeiss AG, Oberkochen, Germany | Immersol G | |
| immersion oil | OLYMPUS EUROPA SE & CO. KG, Hamburg, Germany | IMMOIL-F30CC | |
| prism | Vogelsberger Quarzglastechnik GmbH, Hauzenberg, Germany | Suprasil1 | |
| aluminium prism holder | custom built | ||
| hollow setscrews | Thorlabs Inc, Newton, New Jersey, USA | with custom drilling | |
| Tygon S3 E-3603 tubing | neoLab Migge GmbH, Heidelberg, Germany | 2-4450 | ACF00001 |
| PTFE tubing | Bohlender GmbH, Grünsfeld, Germany | S1810-08 | |
| Name | Company | Catalog Number | Comments |
| Sample | |||
| yeast Hsp90 D61C, Q385C_biotin | UniProt ID P02829 | ||
| Maleimide derivatives of Atto488, Atto550 | ATTO-TEC GmbH, Siegen, Germany | ||
| AMP-PNP* | Jena Bioscience, Jena, Germany | γ-[(6-Aminohexyl)-imido]-AMP-PNP-Atto647N | |
| Fluospheres | Thermo Fisher Scientific, Waltham, MA, USA | F8764 | amine-modified, 0.2 μm, yellow-green fluorescent |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Andor Solis | Andor Technology Ltd, Belfast, Northern Ireland | version 4.30 | |
| LabVIEW | National Instruments, Austin, Texas, USA | version 2012, 32bit; misc. hardware control | |
| MDS control software | AA Opto-Electronic, Orsay, France | version 2.03a | |
| Coherent Connection | Coherent Inc, Santa Clara, CA, USA | version 3 | |
| Igor Pro | WaveMetrics Inc, Portland, OR, USA | version 6.37 |
Riferimenti
- Nooren, I. M. A., Thornton, J. M. Diversity of protein-protein interactions. EMBO J. 22 (14), 3486-3492 (2003).
- Marsh, J. A., Teichmann, S. A. Structure, dynamics, assembly, and evolution of protein complexes. Annu Rev Biochem. 84, 551-575 (2015).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Hohng, S., Joo, C., Ha, T. Single-Molecule Three-Color FRET. Biophys J. 87 (2), 1328-1337 (2004).
- Person, B., Stein, I. H., Steinhauer, C., Vogelsang, J., Tinnefeld, P. Correlated movement and bending of nucleic acid structures visualized by multicolor single-molecule spectroscopy. ChemPhysChem. 10 (9-10), 1455-1460 (2009).
- Lee, J., Lee, S., Ragunathan, K., Joo, C., Ha, T., Hohng, S. Single-molecule four-color FRET. Angew Chem Int Ed. 49 (51), 9922-9925 (2010).
- Ratzke, C., Berkemeier, F., Hugel, T. Heat shock protein 90's mechanochemical cycle is dominated by thermal fluctuations. Proc Natl Acad Sci U S A. 109 (1), 161-166 (2012).
- Ratzke, C., Hellenkamp, B., Hugel, T. Four-colour FRET reveals directionality in the Hsp90 multicomponent machinery. Nat Commun. 5, 4192 (2014).
- Götz, M., Wortmann, P., Schmid, S., Hugel, T. A Multicolor Single-Molecule FRET Approach to Study Protein Dynamics and Interactions Simultaneously. Methods Enzymol. 581, 487-516 (2016).
- Yengo, C. M., Berger, C. L. Fluorescence anisotropy and resonance energy transfer: Powerful tools for measuring real time protein dynamics in a physiological environment. Curr Opin Pharmacol. 10 (6), 731-737 (2010).
- Wortmann,P , ., Götz M, ., Hugel T , . Cooperative Nucleotide Binding in Hsp90 and Its Regulation by Aha1. Biophys J. 113, 1711-1718 (2017).
- Dörfler, T., Eilert, T., Röcker, C., Nagy, J., Michaelis, J. Structural Information from Single-molecule FRET Experiments Using the Fast Nano-positioning System. J Vis Exp. (120), e54782 (2017).
- Stephanopoulos, N., Francis, M. B. Choosing an effective protein bioconjugation strategy. Nature chemical biology. 7 (12), 876-884 (2011).
- Sánchez-Rico, C., Voith von Voithenberg, L., Warner, L., Lamb, D. C., Sattler, M. Effects of Fluorophore Attachment on Protein Conformation and Dynamics Studied by spFRET and NMR Spectroscopy. Chemistry (Weinheim an der Bergstrasse, Germany). , (2017).
- Roy, R., Hohng, S., Ha, T. A practical guide to single-molecule FRET. Nat Methods. 5 (6), 507-516 (2008).
- Lee, N. K., et al. Three-color alternating-laser excitation of single molecules: monitoring multiple interactions and distances. Biophys J. 92 (1), 303-312 (2007).
- Kapanidis, A. N., Lee, N. K., Laurence, T. A., Doose, S., Margeat, E., Weiss, S. Fluorescence-aided molecule sorting: analysis of structure and interactions by alternating-laser excitation of single molecules. Proc Natl Acad Sci U S A. 101 (24), 8936-8941 (2004).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chem Soc Rev. 43 (4), 1156-1171 (2014).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nat Methods. 14, 174-180 (2017).
- Rabiner, L. R. A tutorial on hidden Markov models and selected applications in speech recognition. Proc IEEE. 77 (2), 257-286 (1989).
- Fink, G. A. . Markov Models for Pattern Recognition. , (2014).
- Giudici, P., Ryden, T., Vandekerkhove, P. Likelihood-Ratio Tests for Hidden Markov Models. Biometrics. 56 (3), 742-747 (2000).
- Visser, I., Raijmakers, M. E. J., Molenaar, P. C. M. Confidence intervals for hidden Markov model parameters. Br J Math Stat Psychol. 53 (2), 317-327 (2000).
- McKinney, S. A., Joo, C., Ha, T. Analysis of Single-Molecule FRET Trajectories Using Hidden Markov Modeling. Biophys J. 91 (5), 1941-1951 (2006).
- Bronson, J. E., Fei, J., Hofman, J. M., Gonzalez, R. L., Wiggins, C. H. Learning Rates and States from Biophysical Time Series: A Bayesian Approach to Model Selection and Single-Molecule FRET Data. Biophys J. 97 (12), 3196-3205 (2009).
- Greenfeld, M., Pavlichin, D. S., Mabuchi, H., Herschlag, D. Single Molecule Analysis Research Tool (SMART): an integrated approach for analyzing single molecule data. PLoS ONE. 7 (2), e30024 (2012).
- Schmid, S., Götz, M., Hugel, T. Single-Molecule Analysis beyond Dwell Times: Demonstration and Assessment in and out of Equilibrium. Biophys J. 111 (7), 1375-1384 (2016).
- Taipale, M., Jarosz, D. F., Lindquist, S. HSP90 at the hub of protein homeostasis: emerging mechanistic insights. Nat Rev Mol Cell Biol. 11 (7), 515-528 (2010).
- Trepel, J., Mollapour, M., Giaccone, G., Neckers, L. Targeting the dynamic HSP90 complex in cancer. Nat Rev Cancer. 10 (8), 537-549 (2010).
- Wayne, N., Bolon, D. N. Dimerization of Hsp90 is required for in vivo function. Design and analysis of monomers and dimers. J Biol Chem. 282 (48), 35386-35395 (2007).
- Ali, M. M. U., et al. Crystal structure of an Hsp90-nucleotide-p23/Sba1 closed chaperone complex. Nature. 440 (7087), 1013-1017 (2006).
- Southworth, D. R., Agard, D. A. Species-dependent ensembles of conserved conformational states define the Hsp90 chaperone ATPase cycle. Mol Cell. 32 (5), 631-640 (2008).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proc Natl Acad Sci U S A. 101 (35), 12893-12898 (2004).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An Oxygen Scavenging System for Improvement of Dye Stability in Single-Molecule Fluorescence Experiments. Biophys J. 94 (5), 1826-1835 (2008).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Rognoni, L., Stigler, J., Pelz, B., Ylänne, J., Rief, M. Dynamic force sensing of filamin revealed in single-molecule experiments. Proc Natl Acad Sci U S A. 109 (48), 19679-19684 (2012).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in Surface-Tethered Lipid Vesicles as a New Tool for Single Biomolecule Spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-mode waveguides for single-molecule analysis at high concentrations. Science. 299 (5607), 682-686 (2003).
- Panaretou, B., et al. ATP binding and hydrolysis are essential to the function of the Hsp90 molecular chaperone in vivo. EMBO J. 17 (16), 4829-4836 (1998).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon