Method Article
解剖果蝇黑色素组飞行肌肉为奥米克斯方法
摘要
果蝇飞行肌肉是研究转录调节、替代拼接、代谢和肌生物学的有力模型。我们提出了从活pupae中解剖荧光标记飞行肌肉的协议,以生成高富集的样品,非常适合蛋白质组学和深度测序。这些样本可以提供重要的机械见解,肌肉发育的各个方面。
摘要
果蝇飞行肌肉是研究各种过程的有力模型,如转录调节、替代拼接、代谢和肌瘤生物学,这些过程都影响肌肉发育和肌血生成。Omics 数据(如质谱或深度测序生成的数据)可以为这些生物过程提供重要的机械见解。对于这些方法,分析组织特异性样本,提高组分指纹的选择性和特异性是有好处的。在这里,我们提出了一个协议,从活pupae解剖荧光标记飞行肌肉,为组学应用生成高度丰富的肌肉样本。我们首先描述如何在早期脓肿阶段(<48 h 后,Puprium 形成 [APF]) 解剖飞行肌肉,当肌肉可以通过绿色荧光蛋白 (GFP) 标记来识别时。然后,我们描述如何解剖晚期pupae(>48 h APF)或成人的肌肉,当肌肉在解剖显微镜下可以区分时。随附的视频协议将使这些技术要求苛刻的解剖更容易为肌肉和果蝇研究社区。对于RNA应用,我们用不同时间点和不同方法测定可分离的RNA的数量和质量。我们进一步表明,Bruno1 (Bru1) 是肌苷重链(Mhc)拼接的时态变化所必需的,表明解剖的肌肉可用于 mRNA-Seq、质谱和逆转录聚合酶链反应 (RT-PCR)应用。这种解剖方案将有助于促进组织特异性组学分析,并通常可用于研究造血的多种生物学方面。
引言
现代组学技术为肌肉发育和人类肌肉疾病背后的机制提供了重要的见解。例如,对转录组学数据的分析与动物模型中的遗传和生化验证相结合,发现由于对30多个沙科梅的目标网络进行调节,因此失去拼接因子RBM20会导致扩张性心肌病。以前与心脏病相关的基因,包括锡丁1,2,3。
在第二个例子中,来自细胞培养、动物模型和人类患者的研究表明,肌营养不良症是由肌肉盲(MBNL)的固存和CELF14,5的增强引起的RNA调节的中断造成的。MBNL 和 CELF1(也称为 CUGBP1 或 Bruno-Like-Like 2)之间的交叉调控和时间动力学有助于解释肌营养不良症患者持续的胚胎拼接模式。此外,大型错误控制的目标网络有助于解释疾病4、6、7、8的复杂性质。大多数此类研究利用基因模型生物体中的组学方法来了解人类肌肉疾病背后的机制。此外,他们强调首先了解健康肌肉中的时间和组织类型特定基因表达、蛋白质修饰和代谢模式的重要性,以了解患病或老化肌肉的变化。
果蝇黑色素是另一个成熟的遗传模型有机体。沙科梅的结构以及单个沙科梅成分从苍蝇到脊椎动物4、9、10的高度保存,间接飞行肌肉(IFMs)已成为研究的有力模型肌肉发育的多个方面11,12。首先,纤维肌在功能和形态上不同于管状体肌肉11,13,允许研究肌肉类型的特定发育机制。转录因子包括斯帕尔特主要(萨尔姆)14,外登(外联),和霍莫托拉克斯(Hth)15已被确定为纤维素命运调节器。此外,在萨尔姆下游,CELF1同源布鲁诺1号(Bru1,Aret)指导一个纤维素特拼接程序16,17。
其次,IFM是了解肌细胞生成过程的重要模型,从肌细胞融合和肌管附着到肌血生成和沙突成熟9、18、19。第三,果蝇遗传学允许调查单个蛋白质、蛋白质域和蛋白质等各形式对沙科梅形成、功能和生物物理特性的贡献 20、21、22 , 23.最后,IFM模型已经开发用于研究多种人类肌肉疾病,如肌营养不良症、肌营养不良症、肌退行性疾病、行为性病变等24、25、26 ,27,并提供了重要的见解疾病机制和潜在的疗法28,29,30。因此,果蝇是一个有用的模型,以解决肌生成领域的许多开放问题,包括肌肉类型特定转录机制,拼接和染色质调节,以及代谢在肌肉发育中的作用。现代组学技术的应用,特别是结合果蝇中种类繁多的遗传、生化和细胞生物测定,有可能极大地促进对肌肉的理解。发展、衰老和疾病。
IFM是苍蝇中最大的肌肉,在成人31、32的胸腔长度中跨越了近1毫米。然而,这种小尺寸产生挑战,以获得足够的样本,以组织类型特定的方式应用果蝇技术在果蝇。 此外,IFM 是成人肌肉的一部分,在阴极阶段形成。肌细胞引信形成肌管,在Puparium形成(APF)后24小时左右附着在肌腱上,并经历必要的压实步骤,以启动约30 h APF(图1A-D)18、33的肌张力发生。 34.
肌纤维然后生长到跨越整个胸腔的长度,与肌纤维进入初始生长阶段集中在沙科雷添加,直到约48 h APF,然后过渡到成熟阶段,其中沙科梅里增长的长度和宽度,并改造后,通过 72 h APF(图1A-D)32、35建立拉伸激活。纤维成熟开始至少部分由Salm和E2F32、36、37和多个IFM特异性沙科梅蛋白等形体控制,其拼接由Bru1控制。阶段16,17.成熟苍蝇从90-100 h APF关闭。这意味着,为了研究肌肉发育,IFM必须从多个普帕尔时间点中分离出足够的数量、质量和纯度,以便使用omics方法进行分析。
已发布IFM解剖的若干协议。虽然这些协议非常适合其预期应用程序,但没有一种是组学方法的理想选择。保留IFM形态的表扑和成人IFM19、分离IFM纤维进行机械评价31,或利用从低温节38中微分泌的PUpal IFM的表征,其特殊性和时间性和时间性大量人力为组学应用合理获取足够数量的IFM组织。已开发其他协议,用于快速解剖特定成人IFM38,39,因此不适用于普帕尔阶段, 并使用不理想或与RNA分离不兼容的缓冲液.因此,有必要开发新的方法来分离用于生物化学或组学应用的pupalIFM。
在这里,我们提出了一个协议,在pupal阶段解剖IFM,已经成功地用于mRNA-Seq分析从16 h APF到成人阶段16,32。该协议采用绿色荧光蛋白(GFP)标签来识别在普陀和成人发育的所有阶段的IFM,允许在荧光解剖显微镜下进行活体解剖。该方法的劳动密集型程度较低,其吞吐量高于现有的IFM解剖协议。这允许快速分离和冷冻保存样品,经过几轮解剖后产生足够的材料,用于组学方法以及标准逆转录聚合酶链反应 (RT-PCR) 或西方印迹。
我们将协议分为两部分,演示如何在 48 h APF 之前(在早期变形期间,当IFM附件更脆弱时)和 48 h APF 之后(当 pupal 体计划和IFM附件定义明确时)快速剖析 IFM。我们证明,我们可以在所有时间点从解剖IFM中分离出高质量的RNA,并呈现关于RNA分离和逆转录的不同方法的数据。最后,我们以CELF1同源布鲁诺1为例,演示了解剖方案在mRNA-Seq、质谱和RT-PCR中的应用。在布鲁诺1突变IFM的蛋白质组学数据中,我们显示了沙科梅蛋白异种的误读,并检查了Myosin重链(Mhc)的C端拼接事件的布鲁诺1调控。这些结果说明了组学数据如何有助于更深入地理解生物现象,补充遗传和生化实验。
研究方案
1. 暂存普佩
- 在瓶子中升起所需基因型的苍蝇(图1E)。要么对解剖股票进行新的翻转,要么用至少20只雌性处女苍蝇设置一个十字架。保持瓶子,直到苍蝇开始起起。
- 用湿润的画笔收集预皮,并转移到60毫米培养皿中的润湿滤纸(图1F)。
- 性别的pupae,收集适当的性别的实验(图1G)。雄性通过睾丸的存在来识别,睾丸在原本不透明的pupa中显示为半透明球。
- 用时间、日期和基因型标记培养皿,然后将 pupae 年龄化到所需的阶段(图1H)。
注:在温度控制的培养箱中保持十字架/库存和年龄pupae(即,25°C或27°C的RNAi十字架,因为在较高温度下增加Gal4活性会增加击倒效率40)。确保湿度足够高,这样在老化数天时不会干涸。
2. IFM 解剖 48 h APF 之前
- 组装必要的设备,包括两个#5生物学级钳子、移液器、移液器头、干冰和(对于RNA样品)分离试剂(见材料表)。此外,冷黑色解剖盘(见材料表),1x磷酸盐缓冲盐水(PBS)缓冲液,和1.5 mL微离心管在冰上。
- 使用润湿的画笔,将舞台化的pupae转移到一个黑色解剖盘中,里面装满了大约三分之二的冷1x PBS(图2A,B)。移动到荧光解剖显微镜。
注:在 30 分钟的时间内,尽可能多地使用 pupae。根据经验,范围从3~15页不等。有关黑色解剖菜肴的替代品的讨论,请参阅补充方法。 - 使用#5钳子,将其中一个pupae推到黑色解剖盘的底部,并调整显微镜的变焦和对焦,以清楚地看到pupa(图2C)。
- 用一把钳子抓住pupa的前部(图2D),然后用另一个钳子的一个尖头在腹部稍微偏离中心,就在胸腔后面。这保持pupa就位,并防止IFM移动到腹部(图2E)。
注:从此时开始计时解剖长度,一旦 pupal 完整性被打乱。使用定义的解剖长度(例如 20-30 分钟)以尽量减少肌肉死亡和相关转录组和蛋白酶的变化。在这段时间内,尽可能多地解剖苍蝇。 - 使用第一钳子,取出普宝箱的前半部分(图2F)。
- 使用相同的钳子捏在胸腔后面暴露的pupae,并将腹部与胸腔分开(图2G)。
- 使用钳子,轻轻挤压胸腔的前部分(对于 <35 h APF),或撕开胸腔以暴露荧光标记的IFM(图2H)。IFM 很容易从表皮分离,因为早期时间的肌腱附件是脆弱的。用钳子丢弃剩余的尸体,将其推到盘子的另一侧。
- 重复步骤 2.3_2.7,剖析额外的 pupae。
- 用钳子收集IFM纤维,并将其组织成黑色解剖盘底部的一堆(图2I,J)。使用钳子将其推出视野,清除所有碎屑。
注:通过练习,可以近距离接触钳尖,而无需相互接触。此技术可用于松散地抓取 IFM 而不销毁 IFM。其他方法包括用单个尖端或完全闭合的钳子轻轻推动或抬起IFM,或使用IFM服用一些脂肪或其他组织,并去除步骤2.10中所述的脂肪。 - 质量控制IFM肌肉样本,使用钳子从样品中去除非IFM肌肉、脂肪、角质等(图2K,L)。
注:使用Mef2-Gal4,IFM在早期时间点比其他肌肉类型标注得更强烈(图2K,K'),允许根据荧光强度和肌肉形状去除跳跃肌肉和幼虫肌肉。脂肪和角质组织看起来不同,没有标记肌肉特定的荧光标签 (图 2K,K'。有关标有IFM的其他Gal4行,请参阅讨论部分。 - 使用夹紧的移液器尖端,将一堆IFM转移到一个1.5 mL微离心管中,管中装有250μL的冷却1x PBS(图2M-O)。立即转到第 4 节。
注:只需粘在移液器尖端的一侧,即可丢失IFM样本。在收集IFM之前,上下移液缓冲液可以使标准吸头的粘性降低,而具有较低表面张力的硅化或全氟氧(PFA)尖端(见材料表)有助于防止样品损失。
3. 48 h APF 后 IFM 解剖
- 组装必要的设备,包括两#5生物级钳子、精细剪刀、标准玻璃显微镜幻灯片、双棒胶带、移液器、移液器吸头、干冰和(用于RNA)分离试剂(见材料表)。在冰上冷却 1x PBS 和微离心管。
- 使用轻轻润湿的画笔,将暂存的pupae转移到安装在显微镜幻灯片上的双面胶带条(图3A)。将 pupae 放在方向相同的直线上(向下和朝向幻灯片底部)。
注:小心不要在画笔或过滤器上使用过多的水,否则水坑不会粘好。如果pupae不粘住,首先转移到干过滤器或纸巾上,将其擦干。在 30 分钟的时间内,可以解剖尽可能多的 pupae,理想情况下为 ±10 pupae。 - 从 pupal 外壳中取出 pupa。使用钳子来挑逗分开,打开前尖刺上方的 ppal 外壳 (图3B)。
- 轻轻地将一对钳子滑向后部,在钳子移动时切割 ppal 外壳(图 3B')。小心不要破裂底层的pupa。将 pupa 从打开的外壳上解放出来,并立即转移到第二张显微镜幻灯片上的 1x PBS 下降(图 3B",C)。
- 对线路中的所有 pupae 重复步骤 3.3 和 3.4,然后将双杆胶带滑片放在一边。
- 使用精细剪刀,将胸腔的腹部从胸腔上切开,然后推入一个单独的桩中(图3D,D')。对剩余的 pupae 重复上述步骤。
注:开始计时与步骤3.6解剖的长度,一旦pupal完整性被打乱。在20-30分钟内解剖尽可能多的苍蝇,以防止细胞死亡和相关转录组和蛋白细胞的变化。当解剖 1 d 成人或 >90 h pupae 时,通常方便以后的步骤,以额外的去除头部与精细剪刀。 - 使用纸巾,去除大多数1xPBS(一般多云与悬浮脂肪)以及腹部堆(图3E)。在剩余的胸腔中加入一滴新鲜、冷藏的 1x PBS。
- 使用剪刀将胸部切成两半(图3F,F'),以单一运动从头部向下切割纵向体轴。或者,如果头部已拆下,首先将剪刀插入头部连接的位置,并在IFM之间纵向切割胸部的上半部分。然后,以相同的方向切割胸部的腹侧, 用第二个切口。
- 重复步骤 3.7 和 3.8,对所有 pupae 进行解剖,在幻灯片中心附近生成一堆胸腔。确保幻灯片上有足够的冷却 1x PBS,以便截面不会干涸。
注:48 h APF 后,IFM 足够大,可以在标准解剖显微镜下对经过训练的眼睛可见。在协议中的这一点,带有荧光标签的肌肉可以移动到荧光解剖范围,以帮助IFM识别或用于训练目的,但这不是必要的。 - 将 IFM 从胸腔中分离出来。使用#5钳分离其中一个截面(图3G,H)。轻轻地将一个钳子的尖端插入IFM的中间和下方(图3G',H')。静下第一把钳子时,用细剪刀将IFM的一端从角质层和肌腱上剪开。然后,将IFM的另一端从角质层中切开(图3G'',H')。
注:根据第一次IFM切割后胸腔的方向,旋转胸腔180°是很有用的,这样第二个IFM切割更容易执行。 - 用钳子从胸腔中取出IFM束(图3G''',H''),将其转移到PBS气泡的边缘,使用水张力将其保持原位(图3I )。将尸体推到幻灯片的另一侧。对剩余的胸腔切片重复上述步骤,生成一组解剖 IFM。
注:如果IFM不停留在整齐的堆里,用纸巾取出一些1xPBS。小心不要让所有的PBS蒸发,并确保解剖的IFM和双体轴仍然被缓冲覆盖。 - 解剖所有IFM后,快速对解剖的肌肉进行质量控制。使用#5钳子,删除任何可能进入样品的跳跃肌肉或角质碎片(图3J-K'')。
注:跳跃肌肉看起来与IFM不同。如果解剖Mef2-Gal4标记肌肉荧光下,跳跃肌肉有较弱的荧光和不同的形状和质地。在正常光线下,它看起来几乎半透明,而IFM是不透明的乳黄色(图3J-J',K)。 - 使用水张力,捕获(但不要在一对钳子之间)解剖IFM(图3L)。将IFM转移到预装250 μL的冷冻1x PBS的1.5 mL微离心管(图3M)。立即执行第 4 节。
注:当钳子尖端彼此靠近并提起缓冲液溶液时,水张力会导致在钳子尖端之间捕获缓冲气泡。如果IFM也存在于这个气泡中,它们就可以从解决方案中取出,并轻松转移到另一个充满缓冲的容器。重要的是挤压钳子,使提示彼此靠近,而不接触对方,以避免在缓冲气泡中捕获的组织被压住。
4. 对IFM样本进行颗粒和保存
- 在台式离心机中,在2000 x g下将1.5 mL微离心管离心3~5分钟(图4A,B)。
- 使用移液器尖端(图4C)拆下缓冲液。
- 对于RNA应用,将IFM颗粒重新悬浮在所需RNA分离缓冲液的50~100μL中(参见材料表,图4D)。否则,继续执行步骤 4.4。
注:在步骤4.2之后,IFM可以进行干冷冻,用于质谱制剂或用商业试剂盒分离RNA(参见代表性结果)。对于RNA应用,通过立即在分离缓冲液中重新悬浮和冻结IFM颗粒,可以获得更好的结果。 - 在干冰上冷冻样品或液体氮卡冰(图4E)。储存在-80°C,直到准备好后续步骤的样品准备下游分析。
注:冷冻保存后,样品可以储存数月,然后再进行下游调查。
结果
上面提出的解剖方案有助于在Puprium形成(APF)后16小时到成人阶段生成富含IFM的样本。分离的飞行肌肉样本可用于多种应用,并已成功应用于RT-PCR4,17,RNA-Seq16,32,ChIP36,37,西部印迹14,41和质谱实验(见下文)。为了帮助潜在用户解剖基于RNA的应用,我们首先提出我们的结果,强调特别考虑从IFM中分离RNA。为了更广泛地证明我们的解剖协议效用,我们然后用我们关于RNA结合蛋白Bruno1的数据来说明一些可能的_omics应用。
IFM解剖协议产生高质量的RNA
重要的是要提前确定要解剖的苍蝇数量,因为编码mRNA估计只占RNA42总量的1-5%。我们从IFM中从1d成人中分离出平均每只苍蝇总RNA的24~9 ng(图4F和补充图1A),产量通常随着经验而增加。每只苍蝇的总RNA产量相对稳定,在16 h APF、24 h APF、30 h APF、48 h APF、72 h APF和90 h APF(图4F和补充图1B,D,E)下,IFM的量度波动在25纳克左右。这些观察还反映了从污染脂肪、肌腱、气管或其他细胞类型中分离出的任何RNA,这些物质在从早期时间点分离的样品中可能更高。因此,我们从50只苍蝇中从IFM中获取了>1 μg的总RNA,并通常从100-150只苍蝇中解剖IFM,为RNA-Seq样品生成>3μg的总RNA。
RNA分离方法影响已恢复RNA的数量和质量,我们鼓励用户验证其分离方法。例如,虽然使用方法1的分离平均产生IFM总RNA的1143 ~465 ng,从50 1 d成年苍蝇,分离与各种商业试剂盒产生任何地方从186 × 8 ng到1261 × 355 ng的总RNA(图4G和补充图1C。从商业试剂盒中分离出的RNA通常质量良好(图4H和补充图1F),但低回收率表明RNA可能无法有效地从基质中洗脱。RNA的完整性也可能因为使用方法2(图4H,第二图)中的试剂盒而受到损害,这可能是由于缓冲体组成和热处理,导致严重的碎片化,从而影响下游实验。
在分离和处理RNA样品时,观察适当的无RNase技术也很重要。虽然冻融周期和4小时室温孵育不会显著影响RNA完整性谱,但即使是少量的RNA酶也会导致快速RNA降解(图4I和补充方法)。仍然鼓励用户在冰上工作,并限制冻融,以防止RNA水解和破碎。这里没有检测到这种情况,但使用过滤器提示和 DEPC 处理的缓冲液来防止 RNase 污染是绝对必要的。
逆转录的效率也影响着下游应用的成功。我们用我们测试的三个商用RT试剂盒中的两个获得了可靠的结果,这两个试剂盒都放大了核糖体基因rp49的强RT-PCR带(图4J)。然而,RT Kit #2对低表达转录本的检测可能更为敏感,因为我们获得了所有三个生物复制的RNA结合蛋白bru1的更强带(图4J)。综合起来,这些结果表明,高质量的RNA可以从通过这个程序解剖的IFM中分离出来。
分离的IFM产生高质量的mRNA-Seq和蛋白质组学数据
使用根据上述协议在30 h APF、72 h APF 和 1 d 成年苍蝇下解剖的IFM,我们之前表明RNA结合蛋白和CELF1-同源布鲁诺1(Bru1,逮捕,Aret)控制着IFM特异性拼接途径。转录因子斯帕尔特主要(萨尔姆)16.来自空突变体以及具有肌肉特异性布鲁诺1 RNAi(布鲁1-IR)的苍蝇的IFM显示沙科雷生长缺陷,肌苷活性调节不当,最终肌肉纤维过度收缩和损失16,17.下面我们演示了解剖IFM对整个蛋白质组质谱的效用,并表明我们在RNA水平上观察到的一些表达变化在蛋白质水平上也很明显。我们进一步强调了由 Bruno1 发现的Mhc中特定的开发接头事件,说明解剖 IFM 中的 mRNA-Seq 和 RT-PCR 可用于演示替代拼接事件的调节。
根据库质量和深度,mRNA-Seq 数据可以根据基因单位(基因所有外置子的平均读取计数)、单个外置子或拼接结数水平进行分析。与野生型相比,来自bru1-IRIFM的mRNA-Seq数据显示,基因单元16级(图5A)的表达变化较弱。在 72 h APF 时,已经出现了沙科梅基因的趋势,如肌肉 LIM 蛋白在 60A [Mlp60A], 行为蛋白 57B [Act57B], 肌肉特异性蛋白质 300 kDa [Msp300] 或拉伸-Mlck [斯特恩-姆克] , 对适当的肌肉发育非常重要布鲁1-IR肌肉(图5A和补充表1)。然而,我们之前已经表明,在单个外显子水平上,对特定的沙科莫雷基因异构体16有更强的向下调节,这表明布鲁诺1的主要功能是控制替代拼接(补充表1).
使用解剖IFM上的全蛋白体质谱法,我们可以在蛋白质水平上显示类似的调节(图5B和补充表2)。在检测到的1,895个肽组中,524个(28%)其中1 d成人在布鲁1M2突变IFM中调节不当(补充表2)。还观察到Strn-Mlck和Mlp60A蛋白的降量,匹配了mRNA-Seq数据中转录水平的观测结果。尽管映射到特定蛋白质等形体的数据库肽数量有限(有关分析详细信息,请参阅补充方法),对于沙科梅蛋白特罗波辛 1 (Tm1),支持 (向上/TnT), Mhc, 弯曲 (bt/项目) 和帕拉米辛 (Prm) 我们观察一个异形的肽的向上调节和另一个等形的降调(图5B),证实了我们以前对RNA16级类似调节的观察。这表明解剖IFM对mRNA-Seq和蛋白质组学应用都很有用。
作为组学数据如何补充传统方法以增强和扩展生物学洞察力的进一步示例,我们选择专注于Mhc的 C-总站拼接。以前被描述的蛋白质陷阱线称为weeP26被插入Mhc43,44的最终内创中(有关确切位置,请参阅补充方法)。 weeP26包含一个强大的接头接受器,并包含在大概所有Mhc转录本(图 5C)。然而,IFM中标有GFP的蛋白质被合并到M线两侧的两个"点"中,而在腿部肌肉中,它均匀地跨M线合并,在粗细丝上弱化(图5E)。Orfanos和Sparrow在IFM形式中显示了这些"点",由于一个发展Mhc等形开关:在48 h APF之前表示的Mhc等形被GFP标记为在开放读取帧中的weeP26外显子插入,而Mhc48 h APF 后表示的等形未标记,因为weeP26外生在 3' -UTR44中包含在停止科顿下游。
我们的 mRNA-Seq 数据使我们能够更详细地描述 C 端Mhc等形表达式。虽然已报告两个不同的Mhc端接43,44,我们的 mRNA-Seq 数据和当前 Flybase 注释 (FB2019_02) 表明,在Mhc实际上有三种可能的替代拼接事件C-终点(Exon 34-35、34-36 或 34-37)(图 5C),由 RT-PCR (图 5D) 确认。韦P26GFP 插入于 Exon 36 和 37 之间的内创体中;因此,由于Exon 34-35和Exon 34-36等形包含停止子,GFP只能翻译在Exon 34-37等形(导致Exon 34-GFP-37)。进一步我们可以看到所有Mhc等形的时空调节。在IFM中,我们观察到在27°C时从Exon 34-37到Exon 34-35的Mhc等形开关,在27°C(图5E)之间介于30 h APF和48 h APF(图5C,D,F)之间,即使48 h APF(图5E)的免疫荧光尚未看到。腿已经表示埃克森 34-37 和 Exon 34-35 在 30 h APF 的混合物, 和 72 h APF 表示所有三个Mhc等形 (图 5D,F) 的混合物.成人跳跃肌肉(TDT)也表达所有三个Mhc等形(图5F),表明这一般适用于管状躯体肌肉。因此,我们的mRNA-Seq数据通过缩小IFM中Mhc等形开关的时间范围和描述管状肌肉中的Mhc等形使用,从而扩展了以前的发现。
然后检查盐液和布鲁1突变IFM中的Mhc异形调节。在这两种情况下,我们看到WeeP26的管制不当。Salm突变 IFM 未能在后期阶段完成Mhc等形表达和表拷贝腿拼接模式的发育开关,包括 Exon 34-36 事件的增益(图 5F)。这与先前的发现一致,即Salm的丢失导致IFM几乎完全的命运转换到管状肌肉16。Bru1-IR 和布鲁1突变IFM,类似于salm-/- IFM,通过成人阶段(图5E,F)保留Exon 34-37拼接事件,从而产生类似腿部肌肉的WeeP26 GFP标签模式,但它没有获得Exon 34-36事件。这表明,布鲁诺1在IFM中至少需要部分控制Mhc替代拼接中的发育开关,但它表明,在salm-/-上下文中,其他拼接因子也调节不当。此外,本示例还说明了解剖IFM中的RT-PCR和mRNA-Seq数据如何有助于深入了解发育拼接机制和观察到的形态缺陷。
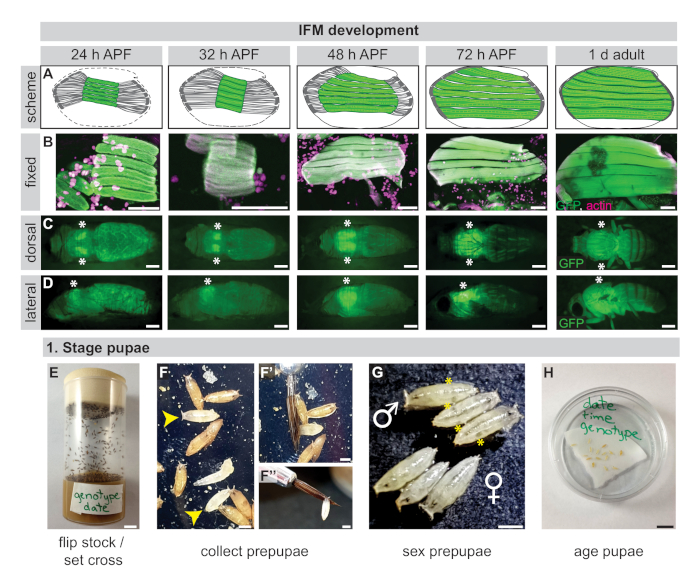
图1:IFM的开发和暂存。(A) IFM在24 h APF、32 h APF、48 h APF、72 h APF 和 1 d 成人在 +32 h APF 和后续纤维生长时显示飞行肌肉(绿色)的压实和后续纤维生长以填充胸腔的图解。肌腱是深灰色的。(B) 固定IFM的共聚焦图像,从打开的图书解剖(24小时,32小时,48小时)19或胸部切片(72小时,1天)染色actin(红胺类黄素,品红色)和GFP(绿色)。(C,D)在活平面中显示完整IFM形态的GFP荧光图像,说明背(C)或侧(D)平面解剖飞行线的完整IFM形态。星号标记IFM位置。(E) 为做好解剖准备,飞行库存应提前3至4天翻转或交叉设置。(F) Prepupae 由其白色(黄色箭头)选择,并使用润湿画笔(F',F'')隔离。(G) 前置应基于睾丸的存在,将女性与男性分开,这些睾丸显示为后置半透明球(黄色星号)。(H) Pupae 在 60 mm 盘子中的湿滤纸上老化。刻度条 = 100 μm (B),1 厘米(C、D、E、H)、1 毫米(F,F'',G)。请点击此处查看此图的较大版本。
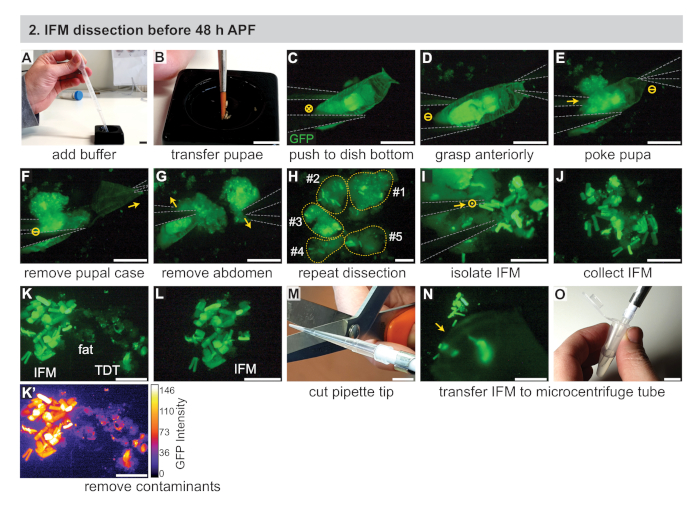
图 2: 在 48 h APF 之前对 IFM 进行解剖。(A) 在带转液器的黑色解剖盘中加入 1x PBS 缓冲液。(B) 使用画笔转移舞台的pupae。(C) 在荧光解剖显微镜下,以可视化 GFP,用#5钳子(以灰色表示)将 pupa 轻轻推到解剖盘的底部。圆圈中的"X"表示图像中的运动。(D,E)抓住前部 (D), 然后戳在胸腔 (E) 后面的 pupae.圆中的短划线表示没有运动。(F,G)用前钳(箭头)拉出腹壳(F),然后取出腹部(G)。(H) 重复 C-G 为几个 pupa.黄色虚线编号表示贡献 pupae。(I, J)使用钳子 (I) 将IFM与周围组织(J)分离。圆中的点表示从页面流出的运动。(K,L)去除污染物,包括脂肪和跳跃(TDT)肌肉(K),以产生一个干净的IFM样本(L)。TDT 具有较低的 GFP 表达式和与 IFM 光纤 (K') 不同的形状。(M、N、O)使用夹紧移液器尖端 (M) 收集解剖 IFM (N) 并将其转移到微离心管 (O)。刻度条 = 1 厘米(A、B、M、O)、1 毫米(C-G)、500 μm(H-L、N)。请点击此处查看此图的较大版本。
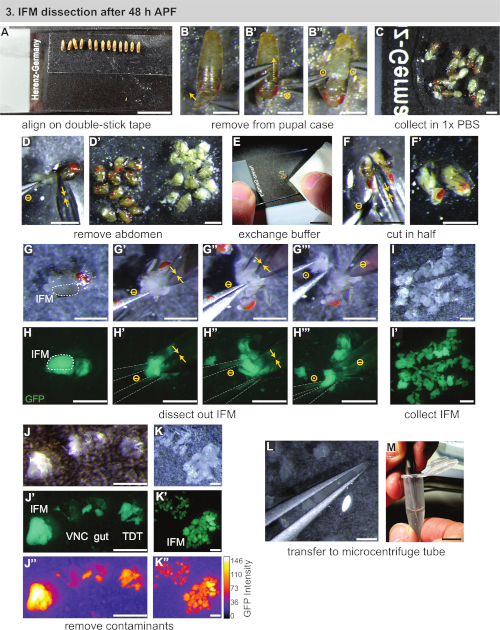
图3:48 h APF后IFM的解剖。(A) 在双棒胶带上对齐 pupae.(B) 通过打开前部 (B),切下壳体(B') 和取出 pupa(B'') 从 pupal 箱中取出 pupae。圆圈符号表示与图 2相同。(C) 将pupae转移到缓冲器中。(D) 用剪刀切割腹部(黄色双箭头)和从胸斧(D') 分离。(E, F)加入清洁缓冲液(E),然后纵向切割一半(F,F')。(G,H)解剖可以在白光(G)或荧光下进行,以可视化GFP(H);在一侧 (G') 切割 IFM,然后另一侧 (G'');用钳子从胸腔中取出(以灰色表示)(G'')。(I,J,K)在缓冲液 (I) 中收集 IFM 并去除污染的腹腔神经线 (VNC)、肠道和跳肌 (TDT) (J),以生成干净的 IFM 样本 (K)。TDT 具有较低的 GFP 表达式和与 IFM 光纤(J''、K')不同的形状。(L,M)使用钳子将IFM(L)转移到微离心管(M)。刻度条 = 1 厘米(A、E、M)、1 毫米(B-D'、F-L)。请点击此处查看此图的较大版本。
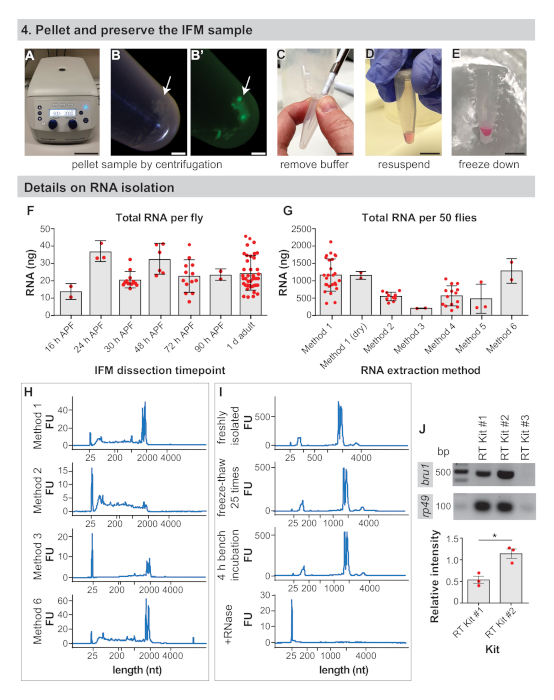
图4:IFM保存和RNA分离细节。(A) IFM 在 2000 x g下通过离心进行5分钟的造粒。(B) IFM 颗粒(箭头)和荧光下颗粒 (B')。(C) 用移液器尖端去除所有缓冲液。(D) 用于RNA提取,在分离缓冲液中重新悬浮颗粒。可以跳过此步骤以干冻结解剖 IFM。(E) 在液氮或干冰上冷冻样品,储存在-80°C。刻度条 = 10 厘米 (A),1 毫米(B,B'),1 厘米(C,D,E)。(F) 每飞在16小时APF、24 h APF、30 h APF、48 h APF、72 h APF、90 h APF和1 d成人时从解剖IFM中获得的总RNA的纳米图(ng)。错误条 = SD. (G) 从从50 1 d成年苍蝇中分离出的RNA总量使用不同的提取方法。误差条 = SD. (H) 代表跟踪不同提取方法后测定RNA完整性。核糖体波段运行略低于2000核苷酸(nt),标记带在25 nt.补充图1中提供的其他轨迹。(I) 新鲜分离的RNA样本(顶部),干冰上25倍的样品冻化25倍(第二块图),在工作台上留下4小时(第三个图)的样本,以及用RNase A(底图)处理的样品的代表性痕迹。注意在添加RNase A. (J) RT-PCR凝胶时RNA完全降解,从试剂盒中贴上bru1和rp49的标签。针对rp49的bru1波段的相对强度如下图所示。错误条形 = SEM(未配对的 t-测试,p = 0.0119)。请点击此处查看此图的较大版本。
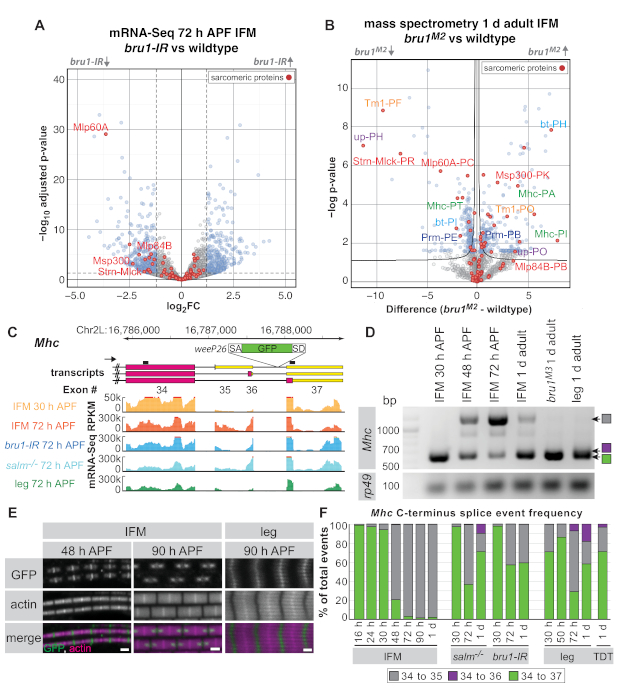
图5:应用IFM解剖来调查布鲁诺1功能在替代拼接中。(A) 在 72 h APF 下解剖 IFM 的 mRNA-Seq 数据(基因单位)的火山图。在布鲁1-IR和野生型IFM(padj <0.05,abs(日志2FC)>1.5)之间显著不同的调控基因以蓝色显示,非显著基因以灰色显示。Sarcomere 蛋白质以红色突出显示,并标记选定的基因。(B) 火山图的整个蛋白酶质谱结果来自1个成人IFM。布鲁M2突变体和野生型(FDR < 0.05)的蛋白质明显不同,以蓝色、非显著蛋白为灰色显示。沙孔蛋白以红色突出显示。与(A)中基因对应的肽被标记为红色。映射到相同蛋白质的不同异体的肽集以相同的颜色标记。(C) Mhc C-termterm 的方案,说明WeeP26基因陷阱的不同转录等形和插入位置(参见插入点的补充方法)。RT-PCR 引录在抄本上方表示为黑线。从 mRNA-Seq 中每千万碱基 (RPKM) 的读取计数显示,从野生类型解剖的 IFM 在 30 h APF(橙色)和 72 h APF(红色)、从布鲁1-IR(蓝色)和萨尔姆-/-(青色)到 72 h APF 时从整个腿(绿色)到 72 h APF.(D) RT-PCR,带针对Mhc的引体,显示IFM中30小时APF和更高时间点之间的等形开关。Exon 34-35 拼接事件仅在布鲁M3突变 IFM 或成人腿部弱观察。(E) 在 48 h APF 和 90 h APF 与 90 h APF 腿部肌肉相比,在野生型 IFM 沙康梅中WeeP26 GFP 定位的共聚焦图像。刻度条 = 1 μm . (F) 从 mRNA-Seq 数据对基因型和时间点的分离结进行定量,并标出。交界读取以特定拼接事件(Exon 34 到 35 灰色、34 到 36 紫色和 34 到 37 以绿色)与共享外显子 34 拼接捐赠者的总数事件的比率表示。请点击此处查看此图的较大版本。
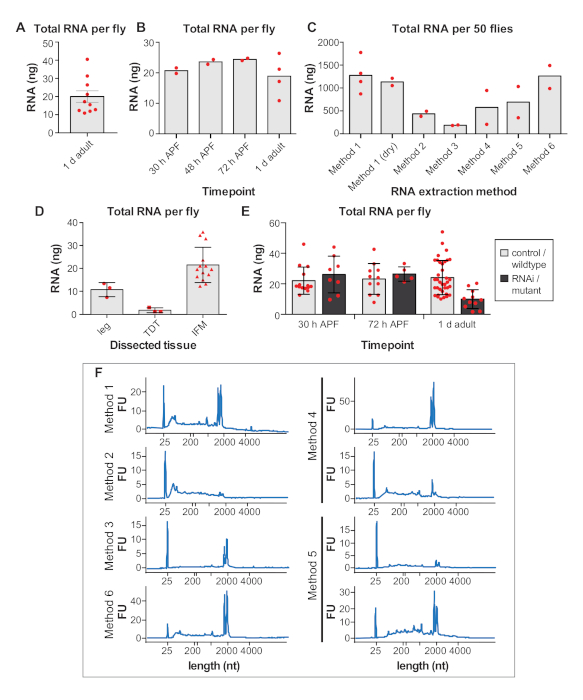
补充图1:( A,B,C) RNA从同一研究人员在同一周内解剖的同一基因型样本中产生。对所有样本进行解剖后,对RNA进行分离并在同一天进行测量。(A) 每1d成人飞行从IFM解剖中获得的总RNA的纳米图(ng)。误差条 = SEM.(B) 每只苍蝇在30小时APF、48 h APF、72 h APF和1 d成人时从解剖IFM获得的总RNA。(C) 从50只1d成年苍蝇中分离出的从IFM中分离的RNA总量,使用不同的提取方法。(D) 每只被解剖的腿、跳跃肌肉 (TDT) 和 IFM 每只苍蝇的总RNA浓度。更多的RNA是从较大的IFM中获得的。误差条 = SD. (E) 从对照组解剖的IFM每飞总RNA浓度与30 h APF、72 h APF和1 d成人的RNAi或突变样本相比。对于突变体,w1118用作野生型控制。突变数据来自布鲁1-IR、salm-/-和另一种RNA结合蛋白突变体。 请注意,对于这些操作,RNA产量在1 d成人下降,由于肌肉萎缩和损失,所以更多的苍蝇需要解剖,以获得足够的量的组学方法。错误条 = SD. (F) 图4G和补充图 1C中所示的 RNA 分离方法显示 RNA 完整性的其他痕迹。请点击此处查看此图的较大版本。

补充方法:详细说明整个文本中使用的方法和试剂,特别是生成图 1A-D、图 4F-K、图 5、补充表 1和补充表2。这些数据激励了解剖方案,并证明了其对RNA分离、mRNA-Seq、RT-PCR和蛋白质组学的效用。请点击此处下载此文件。
| 与图 5 和主文本中的相关段落相关 | |
| 选项卡名称 | 数据摘要 |
| 沙科雷蛋白 | Spletter等人的沙科梅基因列表,Elife 2018;在这里,我们列出了当前的FBgn和基因名称。 |
| SP基因单位_DESeq2_72h | 利用Spletter等人2015年EMBO Rep的数据,我们专门研究了mRNA-Seq数据中的72 h APF中的沙科梅基因。这是从DESeq2分析检测在基因单位水平上的差异表达控制(Mef2-Gal4,UAS-GFM-Gma交叉到w1118)和Mef2-Gal4,UAS-GFM-Gma x Bruno1-IR。以黄色突出显示的行是明显向上或向下调节的基因(高于/低于 log2FC_abs(1.5)的阈值)。这些数据是图 5A 中的红点叠加。对于每个 sarcomere 基因,我们提供标识符信息、DESeq2 中的 log2FC、P 值和调整后的 P 值,以及 DESeq2 规范化表达式计数。 |
| SP 外向子_DEXSeq_72h | 使用Spletter等人的数据EMBO Rep 2015,我们专门研究了在72 h APF的mRNA-Seq数据中的沙科梅雷基因外源使用。这是从DEXSeq分析检测差异外子使用控制(Mef2-Gal4,UAS-GFM-Gma交叉到w1118)和Mef2-Gal4,UAS-GFM-Gma x Bruno1-IR。以黄色突出显示的行是明显向上或向下调节的显子(高于/低于 log2FC_abs(1.5)的阈值)。我们提供外向子和基因标识符信息、DEXSeq 的 log2FC、P 值和调整后的 P 值,以及相关成绩单的列表。 |
| 请注意,许多基因在 DEXSeq 分析中显示一个或多个外显子的调节,通常具有高对数2FC值和低P值/调整P值,而有限的基因列表显示72 h APF的变化。这支持布鲁诺失去对替代拼接调节的强烈影响。 | |
补充表1:在布鲁1-IR与野生型IFMs中,72 h APF mRNA-Seq数据的表,用于识别不同表达基因(通过DESeq2)和外大子(通过DEXSeq)。
| 与图 5B 和主文本中的相关段落相关 | |
| 选项卡名称 | 数据摘要 |
| 佩尔塞乌斯输出 | 这是一个经过处理的数据电子表格,显示用于生成图 5B 的质谱数据。IFM样本来自1d成人对照(w1118)和突变(布鲁诺1-M2)苍蝇。重要列是每个样本的 4 个复制中的每一个转换强度值、t-test 统计量和显著性、肽 D 以及相应的基因名称和 Flybase D。使用 Perseus (FDR<.05) 中的标准设置计算了签名。检测到1859种蛋白质/肽,其中524种(28%)样本之间显著不同。 |
| 降调 | 这些是Perseus输出中所有252种蛋白质/肽,在布鲁诺1-M2突变IFM中向下调节。由于FlybaseID和基因名称已经过时,我们还提供当前的Flybase基因ID和基因名称。 |
| 向上调节 | 这些是Perseus输出中所有272种蛋白质/肽,在布鲁诺1-M2突变IFM中得到调节。由于FlybaseID和基因名称已经过时,我们还提供当前的Flybase基因ID和基因名称。 |
| 请注意,上图 5B 中以红色突出显示的沙科美蛋白位于上述列表中。在补充表 1 中的一个选项卡中提供了被视为沙科酮一部分的基因列表。 | |
补充表2:来自1 d成人的整个蛋白质组质谱数据表,在布鲁M2突变体中识别不同表达的蛋白质和蛋白质异体。野生型IFM。
讨论
在此协议中,我们提出了从早期和晚期对蛋白质、DNA、RNA 或其他大分子进行下游分离的果蝇IFM解剖的基本技术。该协议可以很容易地调整,以解剖从成年苍蝇IFM。我们演示了我们对于mRNA-Seq、蛋白质组学和RT-PCR应用的解剖协议的效用。随着组学技术的不断改进,能够分析起始材料较少且输入浓度较低的样品,这些解剖可能对于许多附加应用变得有价值。由于IFM是人类肌病的既定模型4,24和肌肉类型特定发展9,12,我们设想,例如,IFM丰富的代谢组,染色质构象的研究通过 3C 或 4C,通过 CLiP 相互作用或肌血生成磷-蛋白质组学拼接网络评估。
请务必考虑,这些解剖会产生一个为IFM而不是纯IFM样本富集的样本。由于运动神经元内侧运动、肌腱附件和肌肉纤维的气管入侵,这是不可避免的。生物信息学分析可用于识别IFM富集基因或蛋白质,但需要进一步的实验来证明它们实际上是IFM特异性的。样品纯度可以使用已公布的组织特定标记进行检测,如条纹45(肌腱)、Act79B4、44(管状肌肉)、Act88F15 (IFM) 或 syb46(神经特定)。可能可以使用此类标记将数据集规范化为IFM特定内容,但用户需要注意,用于规范化的基因表达的时态变化,例如IFM特异性基因或图布林,可能会偏袒这种方法。
基因编码的组织特异性标记方法,例如EC标记47,48或PABP标签49,50分离RNA近年来已经开发,这可能有助于获得一个真正的组织特异性RNA样本。然而, EC 标记需要不断喂养苍蝇 47 ,因此不适用于在阶段。PABP标记转录的灵敏度和完整性可能有限制51。FACS分离单个肌肉纤维的方法由于IFM的体积大和合质而变得复杂。INTACT52,53型方法可应用于从IFM中分离特定的亚细胞腔,这可能证明对分离IFM核或线粒体的纯种群有用。对于大多数下游应用,手动解剖仍然是获得完整IFM组织的当前标准。
样品质量取决于解剖过程中的几个关键步骤。解剖技术要求很高,解剖速度和样品纯度随着经验的提高而增加。在没有洗涤剂的情况下在冷冻缓冲液中进行短时间(20-30分钟)的解剖有助于保持样品的完整性,正如之前观察到的小鼠肌腱分离54。IFM在从颗粒中去除所有缓冲液后可以成功干冷冻,但特别用于RNA分离,在隔离缓冲液中冷冻样品往往能产生更好的结果。在RNA或蛋白质分离之前,将多达20个单独解剖的IFM组合起来,允许放大和收集足够的材料,即使从早期时间点或突变体16,32进行下游分析。
对于RNA应用,最关键的步骤可能是RNA本身的分离。二恶亚硝基苯酚-氯仿分离(上述方法1)优于大多数经测试的商业试剂盒,如前所述,55便宜得多。商业试剂盒在RNA分离产量中观察到的变异性与以前的观察结果56,57一致。我们在异丙醇沉淀期间进一步添加糖原,以帮助恢复所有RNA。除了RNA产量之外,验证RNA完整性以确保样品在解剖和分离过程中没有被碎片化或降解非常重要。工作无RNase也是必不可少的。最后,RT-套件的选择会影响逆转录过程的灵敏度。虽然不经常详细讨论,但所有这些点都影响IFM样本的质量和从下游应用程序获得的数据。
几个重要的修改使协议与现有的IFM解剖协议不同。虽然 IFM 免疫荧光的详细解剖方案存在 19 ,但该协议提出了一种不同的解剖方法,允许更快速地分离 IFM 组织。这允许收集大量的IFM组织(相对而言),解剖时间有限,以防止蛋白酶或转录组的变化。其他协议描述成人 IFM 的解剖,用于在单个肌纤维蛋白 39中可视化 GFP 染色或幼虫体壁肌肉 58染色,但它们不涉及在阶段解剖或 RNA 或蛋白质的分离。这种方法也不同于现有的从低温节38中对pupal IFM进行微解剖的协议,后者可以生成更纯净的IFM样本,但劳动强度更大,产生的材料更少。与其他快速成人IFM解剖协议38,39相比,IFM在PBS缓冲液中分离,无需洗涤剂,以限制应力诱导和其他主要表达变化。
该协议的关键进展是包含一个实时的荧光报告器,允许在早期pupal阶段隔离IFM。我们标准使用Mef2-GAL459驱动UAS-CD8::GFP 或 UAS-GFP::Gma60。 这允许对IFM进行差异标记(飞行肌肉比其他脓肿肌肉更贴有强烈标签和形状不同),以及基于GAL4-UAS的操作的性能,例如救援或RNAi实验。也可以结合Mef2-GAL4与浴缸-GAL80ts,以避免RNAi相关的早期杀伤力或与UAS-Dcr2,以提高RNAi效率40。
有额外的GAL4驱动程序或GFP线可用,在肌肉类型特异性,时间表达模式,和驱动器强度19,61,可以使用代替Mef2-GAL4。例如,Act88F-GAL4 首先表示大约 24 h APF,因此不能用于较早的时间点;然而,它强烈标注IFM,并可能有助于避免RNAi相关的早期杀伤力。他-GFP 或Act88F-GFP 标签 IFM,再次具有时间限制,但它们避免了标记表达式的 GAL4 依赖性,并且可能与感兴趣的突变背景结合使用。其他可能的标记行列表有19。还应注意的是,使用转基因和 GAL4/UAS 系统可能会导致基因表达伪影,因此使用适当的控制非常重要,例如,跨越到野生类型背景应变的驱动线,以便此类伪影可能在所有样本中都相同。
在随附的视频中,此详细协议旨在使 pupal IFM 解剖更易于访问,并促进使用组学方法来研究肌肉发育。将果蝇遗传学和细胞生物学的力量与通过解剖IFM获得的生物化学和组学测定相结合,有可能促进对肌发生和肌肉功能的机械理解。未来的研究将转录组和蛋白酶调控的系统级观察与代谢和功能输出联系起来,将有助于更深入地了解肌肉类型的具体发育和肌肉疾病的发病机制。
披露声明
作者没有什么可透露的。
致谢
我们感谢安德烈亚斯·拉杜纳和弗兰克·施诺勒的慷慨支持。我们感谢桑德拉·埃塞尔的出色技术援助和Akanksha Roy生成质谱数据。我们承认布卢明顿和维也纳股票中心提供苍蝇。我们感谢核心设施生物成像在共聚焦成像和Zentrall für蛋白酸化技术分析质谱样本方面的帮助,无论是在LMU生物医学中心(马丁斯里德,DE)。我们的工作得到了德国福森斯Gemeinschaft(MLS,SP 1662/3-1),慕尼黑综合蛋白质科学中心(CIPSM)的支持,在路德维希-马克西米利安大学明琴大学(MLS),弗雷德里希-鲍尔基金会(MLS)和国际马克斯普朗克研究学院(EN)。
材料
| Name | Company | Catalog Number | Comments |
| 5x High Fidelity (HF) buffer | Thermo Fisher | F518L | |
| 60 mm culture dishes | Sigma-Aldrich | Z643084-600EA | Greiner dishes, 60 mm x 15 mM, vented |
| Black dissecting dish (glass) | Augusta Laborbedarf | 42021010 | Lymphbecken, black glass, 4 cm x 4 cm |
| Black silicon dissecting dishes: activated charcoal powder | Sigma-Aldrich | C9157 | Also available from most pharmacies |
| Black silicon dissecting dishes: Sylgard 184 | Sigma-Aldrich | 761036 | Dishes are made by mixing the Sylgard (~50g) with activated charcoal powder (200 mg) and curing it in Petri dishes (~4 60 mm dishes). |
| Blue pestle | Sigma-Aldrich | Z359947-100EA | Any pellet pestle that can sterilized, also can be used with a motor-driven grinder |
| Cell phone camera, Samsung Galaxy S9 | Samsung | SM-G960F/DS | used for photos not taken under a microscope |
| Chloroform | PanReac AppliChem | A3691,0500 | |
| Confocal microscope, Leica SP8X upright confocal | Leica | www.leica-microsystems.com | |
| Confocal microscope, Zeiss LSM 780 inverted confocal | Zeiss | www.zeiss.com | |
| Double stick tape | Scotch/3M | 3M ID 70005108587 | Double-sided tape, available at most office supply handlers |
| Dumont #5 Forceps | Fine Science Tools | 11252-20 | Inox straight tip 11 cm forceps, Biology grade with 0.05 mm x 0.02 mm tip |
| EtOH (100%, RNase free) | Sigma-Aldrich | 32205-M | |
| fluorescent dissecting microscope camera, Leica DFC310 FX camera | Leica | www.leica-microsystems.com | |
| Fluorescent dissecting microscope software, Leica Application Suite (LAS) version 4.0.0 | Leica | www.leica-microsystems.com | |
| Fluorescent dissecting microscope, Leica M165 FC | Leica | www.leica-microsystems.com | |
| Fly: Bru1[M2] | Fly stock; This paper | ||
| Fly: Bru1[M3] | Fly stock; This paper | ||
| Fly: Mef2-GAL4 | Bloomington Stock Center | BDSC:27390 | Fly stock |
| Fly: salm[1] | Bloomington Stock Center | 3274 | Fly stock |
| Fly: salm[FRT] | Fly stock; see Spletter et al., Elife, 2018 | ||
| Fly: UAS-Bru1IR | Vienna Drosophila Research Center | GD41568 | Fly stock, RNAi hairpin |
| Fly: UAS-GFP::Gma | Bloomington Stock Center | BDSC:31776 | Fly stock |
| Fly: UAS-mCD8a::GFP | Bloomington Stock Center | BDSC:5130 | Fly stock |
| Fly: w[1118] | Bloomington Stock Center | 3605 | Fly stock |
| Fly: weeP26 | Fly stock; see Clyne et al., Genetics, 2003 | ||
| GFP detection reagent, GFP-Booster | ChromoTek | gba488-100 | |
| Glycogen | Invitrogen | 10814-010 | |
| Image processing software, Photoshop CS6 | Adobe | www.adobe.com | |
| Isopropanol | Sigma-Aldrich | I9516-25ML | 2-propanol |
| Method 1 (RNA isolation): TRIzol | Life Technologies | 15596018 | Guanidinium isothiocyanate and phenol monophasic solution |
| Method 2 (RNA isolation): Method 1 + TURBO DNA-free Kit | Invitrogen | AM1907 | TRIzol isolation followed by treatment with a kit to remove DNA |
| Method 3 (RNA isolation): Direct-zol RNA Miniprep Plus Kit | Zymo Research | R2070S | RNA isolation in TRIzol, but over commercial columns instead of using phase separation. Recommended DNase treatment performed with Monarch Dnase I in Monarch DNase I Reaction buffer. |
| Method 4 (RNA isolation): RNeasy Plus Mini Kit | Qiagen | 74134 | We used the provided DNase treatment. IFM pellets were homogenized in RTL buffer as suggested for animal tissues. |
| Method 5 (RNA isolation): ReliaPrep RNA Tissue Miniprep System | Promega | Z6110 | We applied the protocol for ‘Purification of RNA from Fibrous Tissues’. |
| Method 6 (RNA isolation): Monarch Total RNA Miniprep Kit | New England Biolabs | T2010G | We applied the protocol for tissues/leukocytes and lysed in 300 μL of RNA Protection Reagent. |
| Microcentrifuge tubes | Thermo Fisher | AM12400 | RNase-free Microfuge Tubes, 1.5 mL |
| Microscope slides | Thermo Fisher | 12342108 | Standard slides, uncharged, 1 mm |
| Microtome blades | PFM Medical | 207500003 | C35 feather 80 mm |
| Monarch DNase I | New England Biolabs | T2004-21 | |
| Monarch DNase I Reaction Buffer | New England Biolabs | T2005-21 | |
| Normal goat serum | Thermo Fisher | 16210072 | |
| OneTaq Polymerase | New England Biolabs | M0480X | |
| Paintbrush | Marabu | 1910000000 | Marabu Fino Round No. 0, or similar brush from any art supply |
| Paraformaldehyde | Sigma-Aldrich | 158127 | |
| PBS buffer (1x) | Sigma-Aldrich | P4417 | Phosphate buffered saline tablets for 1 L solutions, pH 7.4 |
| PFA PureTip Pipette Tips | Elemental Scientific | ES-7000-0101 | Optional substitute for standard pipette tips to reduce sample loss; 100 mL, 0.8 mm orifice |
| Phusion High Fidelity Polymerase | Thermo Fisher | F-530XL | |
| Pipette tips | Sigma-Aldrich | P5161 | Universal 200 mL pipette tips |
| Preomics iST 8x Kit | Preomics | P.O.00001 | peptide preparation kit for mass spectrometry |
| Q Exactive mass spectrometer | Thermo Fisher | 725500 | mass spectrometry was performed at the Protein Analysis Unit of the LMU Biomedical Center |
| Qubit RNA Assay Kit | Life Technologies | Q32855 | |
| Rhodamine-phalloidin | Invitrogen, Molecular Probes | 10063052 | |
| RNA concentration Approach 1 & RNA integrity traces, Bioanalyzer | Agilent Technologies | G2939BA | |
| RNA concentration Approach 2, Nanodrop | Thermo Fisher | ND-2000 | |
| RNA concentration Approach 3, Qubit 4 Fluorometer | Invitrogen | Q33238 | |
| RNA Pico Chips | Agilent Technologies | 5067-1513 | |
| RNase A | Promega | A7937 | |
| RNase-free water, Diethyl pyrocarbonate (DEPC) | Sigma-Aldrich | D5758 | DEPC treat water overnight and then autoclave, to remove all RNase. |
| RT Kit #1: Super Script III Reverse Transcriptase Kit | Invitrogen | 18080-044 | reverse transcription kit |
| RT Kit #2: LunaScript | New England Biolabs | E3010S | reverse transcription kit |
| RT Kit #3: QuantiNova Reverse Transcription Kit | Qiagen | 205410 | reverse transcription kit |
| Slide mounting buffer, Vectashield | Vector Laboratories | H-1200 | containing DAPI |
| Statistical software: GraphPad Prism | GraphPad Prism | www.graphpad.com | |
| Statistical software: Microscoft Excel | Microsoft | Purchased as part of the bundle: Office Home & Student 2019 | |
| Table-top centrifuge | Eppendorf | 5405000760 | Eppendorf Centrifuge 5425 or equivalent |
| Tissue/ Kimwipes | Sigma-Aldrich | Z188956 | Standard tissue wipes |
| Transfer pipette | Sigma-Aldrich | Z350796 | Plastic pipette |
| Triton-X100 | Sigma-Aldrich | T9284-500ML | |
| Vannas spring scissors | Fine Science Tools | 15000-00 | 3 mm cutting edge, tip diameter 0.05 mm, length 8 cm |
| Whatman paper | Sigma-Aldrich | 1004-070 | Filter paper circles, Grade 4, 70 mm |
参考文献
- Rexiati, M., Sun, M., Guo, W. Muscle-Specific Mis-Splicing and Heart Disease Exemplified by RBM20. Genes. 9 (1), 18 (2018).
- Guo, W., et al. RBM20, a gene for hereditary cardiomyopathy, regulates titin splicing. Nature Medicine. 18 (5), 766-773 (2012).
- Guo, W., et al. Splicing Factor RBM20 Regulates Transcriptional Network of Titin Associated and Calcium Handling Genes in The Heart. International Journal of Biological Sciences. 14 (4), 369-380 (2018).
- Nikonova, E., Kao, S. -. Y., Ravichandran, K., Wittner, A., Spletter, M. L. Conserved functions of RNA-binding proteins in muscle. The International Journal of Biochemistry & Cell Biology. 110, 29-49 (2019).
- Wang, E. T., et al. Dysregulation of mRNA Localization and Translation in Genetic Disease. The Journal of Neuroscience. 36 (45), 11418-11426 (2016).
- Wang, E. T., et al. Antagonistic regulation of mRNA expression and splicing by CELF and MBNL proteins. Genome Research. 25 (6), 858-871 (2015).
- Kalsotra, A., et al. A postnatal switch of CELF and MBNL proteins reprograms alternative splicing in the developing heart. Proceedings of the National Academy of Sciences of the United States of America. 105 (51), 20333-20338 (2008).
- Ho, T. H., et al. Muscleblind proteins regulate alternative splicing. The EMBO Journal. 23 (15), 3103-3112 (2004).
- Lemke, S. B., Schnorrer, F. Mechanical forces during muscle development. Mechanisms of Development. 144, 92-101 (2017).
- Iwamoto, H. Structure, function and evolution of insect flight muscle. Biophysics. 7, 21-28 (2011).
- Schnorrer, F., Dickson, B. J. Muscle building; mechanisms of myotube guidance and attachment site selection. Developmental Cell. 7 (1), 9-20 (2004).
- Spletter, M. L., Schnorrer, F. Transcriptional regulation and alternative splicing cooperate in muscle fiber-type specification in flies and mammals. Experimental Cell Research. 321 (1), 90-98 (2014).
- Benoist, P., Mas, J. A., Marco, R., Cervera, M. Differential muscle-type expression of the Drosophila troponin T gene. A 3-base pair microexon is involved in visceral and adult hypodermic muscle specification. Journal of Biological Chemistry. 273 (13), 7538-7546 (1998).
- Schönbauer, C., et al. Spalt mediates an evolutionarily conserved switch to fibrillar muscle fate in insects. Nature. 479 (7373), 406-409 (2011).
- Bryantsev, A. L., et al. Extradenticle and Homothorax Control Adult Muscle Fiber Identity in Drosophila. Developmental Cell. 23 (3), 664-673 (2012).
- Spletter, M. L., et al. The RNA-binding protein Arrest (Bruno) regulates alternative splicing to enable myofibril maturation in Drosophila flight muscle. EMBO Reports. 16 (2), 178-191 (2015).
- Oas, S. T., Bryantsev, A. L., Cripps, R. M. Arrest is a regulator of fiber-specific alternative splicing in the indirect flight muscles of Drosophila. The Journal of Cell Biology. 206 (7), 895-908 (2014).
- Kim, J. H., Jin, P., Duan, R., Chen, E. H. ScienceDirect Mechanisms of myoblast fusion during muscle development. Current Opinion in Genetics & Development. 32, 162-170 (2015).
- Weitkunat, M., Schnorrer, F. A guide to study Drosophila muscle biology. Methods. 68 (1), 2-14 (2014).
- Rai, M., Nongthomba, U., Grounds, M. D. Skeletal Muscle Degeneration and Regeneration in Mice and Flies. Mechanisms of Regeneration. 108, 247-281 (2014).
- Swank, D. M., Wells, L., Kronert, W. A., Morrill, G. E., Bernstein, S. I. Determining structure/function relationships for sarcomeric myosin heavy chain by genetic and transgenic manipulation of Drosophila. Microscopy Research and Technique. 50 (6), 430-442 (2000).
- de Joussineau, C., Bataillé, L., Jagla, T., Jagla, K. Diversification of muscle types in Drosophila: upstream and downstream of identity genes. Current Topics in Developmental Biology. 98, 277-301 (2012).
- Maqbool, T., Jagla, K. Genetic control of muscle development: learning from Drosophila. Journal of Muscle Research and Cell Motility. 28 (7-8), 397-407 (2008).
- Jagla, K., Kalman, B., Boudou, T., Hénon, S., Batonnet-Pichon, S. Beyond mice: Emerging and transdisciplinary models for the study of early-onset myopathies. Seminars in Cell & Developmental Biology. 64, 171-180 (2017).
- Haigh, S. E., et al. Drosophila indirect flight muscle specific Act88F actin mutants as a model system for studying congenital myopathies of the human ACTA1 skeletal muscle actin gene. Neuromuscular Disorders. 20 (6), 363-374 (2010).
- Batonnet-Pichon, S., et al. Myofibrillar Myopathies: New Perspectives from Animal Models to Potential Therapeutic Approaches. Journal of Neuromuscular Diseases. 4 (1), 1-15 (2017).
- Kreipke, R. E., Kwon, Y. V., Shcherbata, H. R., Ruohola-Baker, H. Drosophila melanogaster as a Model of Muscle Degeneration Disorders. Current Topics in Developmental Biology. 121, 83-109 (2017).
- Souidi, A., Zmojdzian, M., Jagla, K. Dissecting Pathogenetic Mechanisms and Therapeutic Strategies in Drosophila Models of Myotonic Dystrophy Type 1. International Journal of Molecular Sciences. 19 (12), 4104 (2018).
- Sparrow, J., Hughes, S. M., Segalat, L. Other Model Organisms for Sarcomeric Muscle Diseases. Advances in Experimental Medicine and Biology. 642, 192-206 (2008).
- Lloyd, T. E., Taylor, J. P. Flightless flies: Drosophila models of neuromuscular disease. Annals of the New York Academy of Sciences. 1184, 1-20 (2010).
- Swank, D. M. Mechanical analysis of Drosophila indirect flight and jump muscles. Methods. 56 (1), 69-77 (2012).
- Spletter, M. L., et al. A transcriptomics resource reveals a transcriptional transition during ordered sarcomere morphogenesis in flight muscle. eLife. 7, 1361 (2018).
- Weitkunat, M., Kaya-Çopur, A., Grill, S. W., Schnorrer, F. Tension and force-resistant attachment are essential for myofibrillogenesis in Drosophila flight muscle. Current Biology. 24 (7), 705-716 (2014).
- Gunage, R. D., Dhanyasi, N., Reichert, H., VijayRaghavan, K. Drosophila adult muscle development and regeneration. Seminars in Cell & Developmental Biology. 72, 56-66 (2017).
- Weitkunat, M., Brasse, M., Bausch, A. R., Schnorrer, F. Mechanical tension and spontaneous muscle twitching precede the formation of cross-striated muscle in vivo. Development. 144 (7), 1261-1272 (2017).
- Zappia, M. P., Rogers, A., Islam, A. B. M. M. K., Frolov, M. V. Rbf Activates the Myogenic Transcriptional Program to Promote Skeletal Muscle Differentiation. Cell Reports. 26 (3), 702-719 (2019).
- Zappia, M. P., Frolov, M. V. E2F function in muscle growth is necessary and sufficient for viability in Drosophila. Nature Communications. 7 (1), 10509 (2016).
- Bryantsev, A. L., et al. Myogenesis in Drosophila melanogaster: Dissection of Distinct Muscle Types for Molecular Analysis. Methods in Molecular Biology. 1889 (5), 267-281 (2019).
- Xiao, Y. S., Schöck, F., González-Morales, N. Rapid IFM Dissection for Visualizing Fluorescently Tagged Sarcomeric Proteins. Bio-Protocol. 7 (22), (2017).
- Kaya-Çopur, A., Schnorrer, F. RNA Interference Screening for Genes Regulating Drosophila Muscle Morphogenesis. Myogenesis. 1889, 331-348 (2019).
- Chechenova, M. B., et al. Functional redundancy and non-redundancy between two Troponin C isoforms in Drosophila adult muscles. Molecular Biology of the Cell. 28 (6), 760-770 (2017).
- Alberts, B. . Molecular Biology of the Cell. , (2017).
- Clyne, P. J., Brotman, J. S., Sweeney, S. T., Davis, G. Green fluorescent protein tagging Drosophila proteins at their native genomic loci with small P elements. Genetics. 165 (3), 1433-1441 (2003).
- Orfanos, Z., Sparrow, J. C. Myosin isoform switching during assembly of the Drosophila flight muscle thick filament lattice. Journal of Cell Science. 126 (1), 139-148 (2013).
- Volohonsky, G., Edenfeld, G., Klambt, C., Volk, T. Muscle-dependent maturation of tendon cells is induced by post-transcriptional regulation of stripeA. Development. 134 (2), 347-356 (2007).
- Estes, P. S., Ho, G. L., Narayanan, R., Ramaswami, M. Synaptic localization and restricted diffusion of a Drosophila neuronal synaptobrevin--green fluorescent protein chimera in vivo. Journal of Neurogenetics. 13 (4), 233-255 (2000).
- Hida, N., et al. EC-tagging allows cell type-specific RNA analysis. Nucleic Acids Research. 45 (15), 138 (2017).
- Thomas, A., et al. A versatile method for cell-specific profiling of translated mRNAs in Drosophila. PLoS ONE. 7 (7), 40276 (2012).
- Yang, Z. Isolation of mRNA from specific tissues of Drosophila by mRNA tagging. Nucleic Acids Research. 33 (17), 148 (2005).
- Jiao, Y., Moon, S. J., Montell, C. A Drosophila gustatory receptor required for the responses to sucrose, glucose, and maltose identified by mRNA tagging. Proceedings of the National Academy of Sciences of the United States of America. 104 (35), 14110-14115 (2007).
- Blazie, S. M., et al. Comparative RNA-Seq analysis reveals pervasive tissue-specific alternative polyadenylation in Caenorhabditis elegans intestine and muscles. BMC Biology. 13 (1), 1775-1821 (2015).
- Henry, G. L., Davis, F. P., Picard, S., Eddy, S. R. Cell type-specific genomics of Drosophila neurons. Nucleic Acids Research. 40 (19), 9691-9704 (2012).
- Deal, R. B., Henikoff, S. The INTACT method for cell type-specific gene expression and chromatin profiling in Arabidopsis thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Grinstein, M., Dingwall, H. L., Shah, R. R., Capellini, T. D., Galloway, J. L. A robust method for RNA extraction and purification from a single adult mouse tendon. PeerJ. 6 (8), 4664 (2018).
- Green, M. R., Sambrook, J. . Molecular Cloning. , (2012).
- Brown, R. A. M., et al. Total RNA extraction from tissues for microRNA and target gene expression analysis: not all kits are created equal. BMC Biotechnology. 18 (1), 16 (2018).
- Ford, K. L., et al. Optimisation of laboratory methods for whole transcriptomic RNA analyses in human left ventricular biopsies and blood samples of clinical relevance. PLoS ONE. 14 (3), 02136855 (2019).
- Ramachandran, P., Budnik, V. Dissection of Drosophila larval body-wall muscles. Cold Spring Harbor Protocols. (8), 5469 (2010).
- Ranganayakulu, G., Schulz, R. A., Olson, E. N. Wingless signaling induces nautilus expression in the ventral mesoderm of the Drosophila embryo. Developmental Biology. 176 (1), 143-148 (1996).
- Dutta, D., Bloor, J. W., Ruiz-Gómez, M., VijayRaghavan, K., Kiehart, D. P. Real-time imaging of morphogenetic movements in Drosophila using Gal4-UAS-driven expression of GFP fused to the actin-binding domain of moesin. Genesis. 34 (1-2), 146-151 (2002).
- Lemke, S. B., Schnorrer, F. In Vivo Imaging of Muscle-tendon Morphogenesis in Drosophila Pupae. Journal of Visualized Experiments. (132), e57312 (2018).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。