Method Article
오믹스 접근법에 대한 드로소필라 멜라노가스터 비행 근육의 해부
요약
초파리 비행 근육은 전사 조절, 대체 접합, 신진 대사 및 메카노생물학을 연구하는 강력한 모델입니다. 우리는 프로테오믹스및 깊은 시퀀싱에 이상적인 고농축 샘플을 생성하기 위해 살아있는 번데기에서 형광 표지된 비행 근육의 해부를 위한 프로토콜을 제시합니다. 이 견본은 근육 발달의 다양한 양상에 중요한 기계론적인 통찰력을 제공할 수 있습니다.
초록
Drosophila 비행 근육은 전사 조절, 대체 접합, 신진 대사 및 기계생물학과 같은 다양한 과정을 연구하는 강력한 모델이며, 이는 모두 근육 발달과 근생 신생에 영향을 미칩니다. 질량 분석 또는 깊은 시퀀싱에 의해 생성된 Omics 데이터는 이러한 생물학적 프로세스에 대한 중요한 기계적 통찰력을 제공할 수 있습니다. 이러한 접근법의 경우, 조직 특이적 샘플을 분석하여 오믹스 지문의 선택성과 특이성을 모두 증가시키는 것이 유익하다. 여기에서 우리는 omics 응용을 위한 높게 풍요로운 근육 견본을 생성하기 위하여 살아있는 pupae에서 형광 표지한 비행 근육의 해부를 위한 프로토콜을 제시합니다. 우리는 먼저 초기 pupal 단계에서 비행 근육을 해부하는 방법을 설명 (&48 번화 증형성 후 시간 [APF]), 근육은 녹색 형광 단백질에 의해 식별 할 때 (GFP) 라벨. 그런 다음 근육이 해부 현미경으로 구별 될 때 늦은 번데기 (>48 h APF) 또는 성인에서 근육을 해부하는 방법을 설명합니다. 동반 된 비디오 프로토콜은 기술적으로 요구하는 해부를 근육과 초파리 연구 커뮤니티에 보다 광범위하게 접근 할 수 있게 합니다. RNA 적용을 위해, 우리는 다른 시점에서 그리고 다른 접근으로 격리될 수 있는 RNA의 양 그리고 질을 분석합니다. 우리는 또한 Bruno1 (Bru1)가 myosin 중계체인(Mhc)접합의 시간적 변화에 필요하다는 것을 보여주고, 해부 된 근육이 mRNA-Seq, 질량 분석 법 및 역전사 폴리머라제 연쇄 반응 (RT-PCR)에 사용될 수 있음을 입증합니다. 응용 프로그램. 이 해부 프로토콜은 조직 특이적 오믹스 분석을 촉진하는 데 도움이 되며 일반적으로 근생의 여러 생물학적 측면을 연구하기 위해 적용될 수 있습니다.
서문
현대 omics 기술은 근육 발달과 인간의 근육 장애의 근본적인 메커니즘에 대한 중요한 통찰력을 제공합니다. 예를 들어, 동물 모델에서 유전적 및 생화학적 검증과 결합된 전사체 데이터의 분석은 결합 인자 RBM20의 손실이 30개 이상의 sarcomere의 표적 네트워크의 조절로 인해 확장된 심근병증을 유발한다는 것을 밝혀냈습니다. 이전에 심장 질환과 관련된 유전자, 티틴을포함1,2,3.
두 번째 예에서, 세포 배양, 동물 모델 및 인간 환자로부터의 연구는 근위축증이 MUSCLEblind(MBNL)의 격리 및 CELF14,5의상류조절로 인한 RNA 조절의 붕괴에 기인한다는 것을 보여주었다. MBNL과 CELF1 (CUGBP1 또는 Bruno-Like 2라고도 함) 사이의 교차 조절 및 시간 적 역학은 근위축증 환자의 지속적인 배아 접합 패턴을 설명하는 데 도움이됩니다. 추가적으로, 잘못 규제된 표적의 큰 네트워크는 질병의 복잡한 본질을 설명하는 것을 돕습니다4,6,7,8. 그 같은 연구 결과의 대다수는 인간 적인 근육 질병의 근본적인 기계장치를 이해하기 위하여 유전 모형 유기체에 있는 omics 접근을 이용합니다. 또한, 그들은 질병이나 노화 근육의 변화를 이해하기 위해 건강한 근육의 시간적 및 조직 유형 특정 유전자 발현, 단백질 수정 및 대사 패턴을 먼저 이해하는 것의 중요성을 강조합니다.
초파리 멜라노가스터는 또 다른 잘 확립된 유전 모델 유기체입니다. 사르코레의 구조와 개별 사르코망 성분은 파리에서 척추동물4,9,10으로매우 보존되며 간접 비행 근육(IFMs)은 연구할 수 있는 강력한 모델이 되었습니다. 근육 발달의 여러 측면11,12. 첫째, 섬유질 비행 근육은 기능적으로 그리고 형태학적으로 관형 신체근육과 구별11,13,근육 형 특정 발달 메커니즘의 조사를 허용. 스팔트 메이저(Salm)14,엑스트라덴티클(Exd), 호모토락스(Hth)15를 포함한 전사 인자는 세브릴라 운명 조절자로 확인되었다. 또한, 살름의 하류, CELF1 호몰로그 Bruno1 (Bru1, Aret)은 세브릴라 특이적 접합 프로그램16,17을지시한다.
둘째, IFM은 근형성 융합 및 myotube 부착부터 근위대 유족발생 및 사코메르 성숙9,18,19에이르기까지 근형성 자체의 과정을 이해하는 중요한 모델이다. 셋째, Drosophila 유전학은 sarcomere 형성, 기능 및 생물 물리학적 특성에 개별 단백질, 단백질 도메인 및 단백질 이소폼에 의한 기여의 조사를허용합니다 20,21,22 ,23. 마지막으로, IFM 모델은 근위축증, 근시근균, 근육퇴행성 질환, 작용병증 등과 같은 다중 인간 근육 장애의 연구를 위해 개발되었으며,24,25,26 ,27, 질병 메커니즘 및 잠재적 치료법에 대한 중요한 통찰력을 제공28,29,30. 따라서, Drosophila는 근육 형 특이적 전사, 접합 및 염색질 조절의 메커니즘뿐만 아니라 근육 발달에서 신진 대사의 역할을 포함하여 근형성 분야에서 많은 열린 질문을 해결하는 유용한 모델입니다. 현대 오믹스 기술의 적용, 특히 다양한 유전적, 생화학적 및 세포 생물학적 분석을 통해 Drosophila에서사용할 수 있는, 근육의 이해를 극적으로 발전시킬 수 있는 잠재력을 가지고 있습니다. 개발, 노화 및 질병.
IFM은 비행에서 가장 큰 근육이며, 성인31,32의흉부 전체 길이에 걸쳐 거의 1mm에 걸쳐 있습니다. 그러나, 이러한 작은 크기는 조직 형 특이적 방식으로 Drosophila에서 오믹 기술을 적용하기에 충분한 샘플을 얻는 도전을 생성한다. 또한 IFM은 푸팔 단계 중에 형성되는 성인 근육의 일부입니다. 근체세포는 근체튜브를 형성하기 위해 융합되며, 이는 puparium 형성 후 약 24시간 동안 힘줄에 부착하고 30h APF(그림 1A-D)18,33,30h APF 주위의 근위병 발생을 개시하는 데 필요한 다짐 단계를 거칩니다. 34.
myofibers는 약 48 h APF까지 sarcomere 추가에 초점을 맞춘 초기 성장 단계를 겪고, 흉부의 전체 길이에 걸쳐 성장, 다음 성숙 단계로 전환, 있는 sarcomeres 길이와 폭에서 성장 하 고 72 h APF(그림 1A-D)32,35에의해 스트레치 활성화를 설정하기 위해 리모델링. 섬유 성숙의 개시는 적어도 부분적으로 Salm 및 E2F32,36,37에의해 통제되고, 그 접합이 Bru1에 의해 통제되는 다중 IFM 특이적인 sarcomere 단백질 이소폼은 이 기간 동안 통합됩니다 단계16,17. 성숙한 파리는 90-100 h APF에서 닫힙입니다. 즉, 근육 발달을 연구하기 위해 IFM은 오믹스 접근법을 사용하여 분석을 용이하게 하기 위해 여러 pupal 타임포인트에서 충분한 양, 품질 및 순도로 격리되어야 합니다.
IFM 해부를 위한 몇 가지 프로토콜이 게시되었습니다. 이러한 프로토콜은 의도된 응용 프로그램에 적합하지만 omics 접근 방식에 이상적인 프로토콜은 없습니다. 푸팔 및 성인 IFM19의면역형광에 대한 IFM 형태를 보존하는 프로토콜, 기계적 평가를 위한 IFM 섬유 분리31,또는 극저온절38에서 푸팔 IFM의 미세해부를 활용하는 프로토콜은 너무 전문화되고 시간및 오믹스 응용 프로그램에 대한 IFM 조직의 충분한 양을 합리적으로 얻기 위해 노동 집약적. 다른 프로토콜은 특히 성인 IFM38,39의신속한 해부를 위해 개발되었으며, 따라서 pupal 단계에 적용되지 않으며, 이상적이지 않거나 RNA 격리와 호환되지 않을 수 있는 버퍼를 사용한다. 따라서, 생화학 또는 오믹 응용을 위한 pupal IFM을 분리하기 위한 새로운 접근법을 개발할 필요가 있다.
여기서 우리는 16h APF에서 성인 단계16,32를통해 mRNA-Seq 분석을 위해 성공적으로 사용된 pupal 단계 동안 IFM의 해부를 위한 프로토콜을 제시한다. 이 프로토콜은 녹색 형광 단백질 (GFP) 라벨을 사용하여 pupal 및 성인 발달의 모든 단계에서 IFM을 식별하여 형광 해부 현미경하에서 살아있는 해부를 허용합니다. 이 접근 방식은 기존 IFM 해부 프로토콜보다 처리량이 높은 노동 집약적입니다. 이를 통해 시료의 신속한 분리 및 동결 보존이 가능하여, 오믹스 접근법뿐만 아니라 표준 역전사 폴리머라제 연쇄 반응(RT-PCR) 또는 서부 블로팅에 대해 여러 차례 해부 후 충분한 물질을 생성할 수 있습니다.
우리는 48 h APF 전(초기 변형 중, IFM 부착이 더 엄격할 때) 및 48시간 APF(푸팔 바디 플랜 및 IFM 부착이 잘 정의된 경우) 모두 IFM을 빠르게 해부하는 방법을 보여주는 두 부분으로 프로토콜을 제시합니다. 우리는 모든 시점에서 해부 된 IFM에서 고품질 RNA를 분리하고 RNA 격리 및 역 전사에 대한 다른 접근법에 대한 데이터를 제시 할 수 있음을 보여줍니다. 마지막으로, CELF1 호몰로그 Bruno1을 예로 사용하여 mRNA-Seq, 질량 분석법 및 RT-PCR에 해부 프로토콜의 적용을 입증한다. 우리는 Bruno1 돌연변이 IFM에서 프로테오믹스 데이터에서 사르코메르 단백질 이소폼의 오발성을 보여주고 Myosin중쇄(Mhc)의C 말단 스플라이스 이벤트의 Bruno1 조절을 조사하였다. 이러한 결과는 omics 데이터가 유전 적 및 생화학 적 실험을 보완하는 생물학적 현상에 대한 깊은 이해를 제공 할 수있는 방법을 보여줍니다.
프로토콜
1. 푸파 준비
- 병에 원하는 유전자형의 파리를 올립니다(그림 1E). 해부 스톡의 신선한 플립을 만들거나 적어도 20 여성 처녀 파리와 십자가를 설정합니다. 파리가 퍼페이트되기 시작할 때까지 병을 유지하십시오.
- 젖은 페인트 브러시로 프리 푸페를 수집하고 60mm 페트리 접시에 젖은 여과지로 옮김(그림 1F).
- 강아지를 성별하고, 실험에 적합한 성별을수집한다(도 1G). 남성은 그렇지 않으면 불투명 한 pupa에 반투명 공으로 나타나는 고환의 존재에 의해 식별됩니다.
- 페트리 접시에 시간, 날짜 및 유전자형을 표시한 다음 번화를 원하는 단계로 나이를 누린다(그림1H).
참고: 온도 제어 인큐베이터에서 십자가/주식 및 나이 퍼프를 유지합니다(즉, RNAi 십자가의 경우 25°C 또는 27°C, 더 높은 온도에서 Gal4 활성이 증가함에 따라 노크다운 효율40). 며칠 동안 숙성할 때는 번데어가 마르지 않도록 습도가 충분히 높은지 확인하십시오.
2. 48 시간 APF 전에 IFM 해부
- 두 개의 #5 생물학 등급 집게, 파이펫, 파이펫 팁, 드라이 아이스 및 (RNA 샘플의경우) 절연 시약을 포함하여 필요한 장비를 조립합니다(재료 표 참조). 또한, 차가운 검은 해부 접시 (재료 표참조), 1 x 인산염 완충 식염수 (PBS) 버퍼, 얼음에 1.5 mL 미세 원심 분리 튜브.
- 젖은 페인트 브러시를 사용하여, 차가운 1x PBS(그림 2A, B)로약 3 분의 2를 가득 검은 해부 접시에 단계적 인 pupae를 전송합니다. 형광 해부 현미경으로 이동합니다.
참고: 30 분 시간 창 내에서 해부 할 수있는 만큼 많은 강아지를 사용하십시오. 경험에 따라, 이것은 3-15 pupae에서 범위. 검은 해부 요리에 대한 대안에 대한 자세한 방법을 참조하십시오. - #5 포셉을 사용하여, 검은 해부 접시의 바닥에 강아지 중 하나를 밀어 명확하게 pupa를 볼 수 현미경 줌과 초점을 조정(그림 2C).
- 한 집게(그림 2D)로강아지의 앞쪽을 잡은 다음 흉부 바로 뒤에 복부의 중앙에서 약간 벗어난 다른 집게의 단일 팁으로 강아지를 찌른다. 이것은 제자리에 있는 pupa를 붙잡고 IFMs가 복부로 이동하는 것을 방지합니다(그림 2E).
참고: pupal 무결성이 중단되는 즉시이 시점에서 해부 길이타이밍을 시작합니다. 근육 사멸 및 관련 전사체 및 단백질 변화를 최소화하기 위해 정의된 해부 길이(예: 20-30분)를 사용합니다. 이 기간에 가능한 한 많은 파리를 해부하십시오. - 첫 번째 집게를 사용하여 푸팔 케이스의 앞쪽 절반을 제거합니다(그림2F).
- 동일한 포셉을 사용하여 흉부 바로 뒤에 노출된 강아지를 꼬집고 흉부에서 복부를 분리합니다(그림2G).
- 집게를 사용하여 흉부 의 앞쪽 부분 (<35 h APF)을 부드럽게 짜내거나 흉부에서 찢어형으로 표시된 IFMs(그림 2H)를노출시십시오. IFM은 초기 시점에서 힘줄 부착이 깨지기 쉽기 때문에 표피에서 쉽게 분리됩니다. 집게를 사용하여 나머지 시체를 버리고 접시의 반대편으로 밀어 넣습니다.
- 2.3-2.7단계를 반복하고, 추가 번데기해 한다.
- 집게로 IFM 섬유를 수집하고 검은 해부 접시의 바닥에 더미로 구성(그림 2I,J). 집게를 사용하여 시야밖으로 밀어 파편을 제거합니다.
참고: 연습으로, 집게 끝은 서로 를 건드리지 않고 가까운 근접으로 가져올 수 있습니다. 이 기술은 IFM을 파괴하지 않고 느슨하게 잡는 데 사용할 수 있습니다. 다른 방법은 부드럽게 밀거나 단일 팁 또는 완전히 닫힌 집게로 IFM을 들어 올리거나, IFM으로 일부 지방 또는 다른 조직을 복용하고 단계 2.10에 설명 된 대로 지방을 제거하는 것을 포함한다. - 품질 관리는 IFM 근육 샘플을, 집게를 사용하여 샘플로부터 비IFM 근육, 지방, 표피 등을 제거한다(도2K,L).
참고: Mef2-Gal4를 사용하면 IFM은 초기 시점에서 다른 근육 유형보다 더 강하게 표시되어(그림 2K, K')형광 강도와 근육 모양에 따라 점프 근육과 애벌레 근육을 제거 할 수 있습니다. 지방 및 표피 조직은 다르게 보이며 근육 특이적 형광 라벨에 의해 표지되지 않습니다(그림 2K, K'). IFM이라고 표시된 다른 Gal4 줄에 대한 토론 섹션을 참조하십시오. - 잘린 파이펫 팁을 사용하여 IFM 더미를 냉각 된 1x PBS(그림 2M-O)의250 μL로 채워진 1.5 mL 미세 원심 분리튜브로 옮니다. 섹션 4로 즉시 진행하십시오.
참고: IFM 샘플은 파이펫 팁의 측면에 달라붙는 것만으로도 손실될 수 있습니다. IFM을 수집하기 전에 버퍼를 여러 번 위아래로 파이펫팅하면 표준 팁이 덜 끈적거리며, 표면 장력이 낮은 실리콘 또는 퍼플루오로알록시(PFA) 팁(재료 표참조)은 시료 손실을 방지하는 데 도움이 될 수 있습니다.
3. IFM 해부 후 48 시간 APF
- 두 개의 #5 생물학 등급 집게, 미세 가위, 표준 유리 현미경 슬라이드, 이중 스틱 테이프, 파이펫, 파이펫 팁, 드라이 아이스 및 (RNA 응용 프로그램의 경우) 격리 시약을 포함하여 필요한 장비를 조립합니다(재료 표참조). 1x PBS 및 미세 원심 분리튜브를 얼음위에 식힙니다.
- 가볍게 젖은 페인트 브러시를 사용하여 단계형 번데기를 현미경 슬라이드에 장착 한 양면 끈적 끈적한 테이프 스트립으로 옮니다(그림 3A). 번데어를 같은 방향(아래쪽과 앞쪽 슬라이드 아래쪽)을 향하도록 줄에 놓습니다.
참고: 페인트 브러시 나 필터에 너무 많은 물을 사용하지 않도록주의하거나, 번데어가 잘 붙어있지 않습니다. 번데기가 달라붙지 않으면 먼저 마른 필터 나 티슈 페이퍼로 옮겨 서 건조시십시오. 30 분 시간 창 내에서 해부 할 수있는 만큼 많은 강아지를 마운트, 이상적으로 ~ 10 번페. - 푸팔 케이스에서 강아지를 제거합니다. 포셉을 사용하여 앞쪽 첨탑 위의 푸팔 케이스를 애타게 하고 엽니다(그림3B).
- 한 쌍의 포셉을 뒤쪽쪽으로 가볍게 밀어 내어 집게가 움직일 때 푸팔 케이스를 절단합니다(그림3B'). 기본 강아지가 파열되지 않도록주의하십시오. 열린 케이스에서 강아지를 해방하고 즉시 두 번째 현미경 슬라이드에 1 x PBS의 방울로 전송(그림 3B", C).
- 줄에 있는 모든 번데기의 경우 3.3 단계와 3.4 단계를 반복한 다음 이중 스틱 테이프 슬라이드를 옆으로 놓습니다.
- 미세 가위를 사용하여, 흉부에서 멀리 pupa의 복부를 잘라 별도의 더미로 밀어(그림 3D, D'). 남은 번데기는 반복한다.
참고: pupal 무결성이 중단되는 즉시 3.6 단계로 해부 길이타이밍을 시작합니다. 세포 사멸 과 관련 전사체 및 프로테오믹 변화를 방지하기 위해 가능한 한 많은 파리를 20-30 분으로 해부하십시오. 1 d 성인 또는 >90 h 번데기를 해부 할 때, 미세 가위로 머리를 추가로 제거하는 것이 종종 편리합니다. - 티슈 페이퍼를 사용하여, 1x PBS의 대다수를 제거 (일반적으로 부유 지방과 흐린) 뿐만 아니라 복부의 더미(그림 3E). 남은 흉부류에 신선하고 차가운 1x PBS 한 방울을 넣습니다.
- 가위를 사용하여 흉부 반으로 자른다(그림3F,F')머리에서 세로 몸체 축을 한 번 의 동작으로 절단합니다. 또는, 머리를 제거 한 경우, 먼저 머리가 부착 된 가위를 삽입하고 IFMs 사이에 흉부의 상단 절반을 세로로 잘라. 그런 다음 흉부의 복부 면을 동일한 방향으로 두 번째 컷으로 잘라냅니다.
- 모든 번데어가 해부될 경우 3.7 단계와 3.8 단계를 반복하여 슬라이드 의 중심 근처에 흉부 반구 더미를 생성합니다. 반구가 마르지 않도록 슬라이드에 1x PBS가 충분히 있는지 확인하십시오.
참고: 48 시간 APF 후, IFM은 훈련 된 눈에 표준 해부 현미경의 밑에 표시 될 정도로 크다. 프로토콜의 이 시점에서, 형광 라벨을 가진 근육은 IFM 식별 또는 훈련 목적을 돕기 위해 형광 해부 범위로 이동될 수 있지만, 이것은 필요하지 않다. - 흉부에서 IFM을 해부합니다. #5 집게를 사용하여 반구 중 하나를분리합니다(그림 3G,H). IFM의 중간 위와 아래에 한 집게의 끝을 부드럽게 삽입합니다(그림3G', H'). 첫 번째 집게를 여전히 유지하면서 미세 한 가위를 사용하여 IFM의 한쪽 끝을 표피와 힘줄에서 멀리 자른다. 그런 다음 IFM의 다른 쪽 끝을 표피에서 자유롭게 자른다(그림3G',H'').
참고: 첫 번째 IFM 컷 후 흉부 방향에 따라 두 번째 IFM 절단이 더 쉽게 수행되도록 흉부 180°를 회전하는 것이 유용합니다. - 포임이 있는 흉부에서 IFM 번들을 제거합니다(그림3G'',H'''),PBS 버블의 가장자리로 이송하여 물 장력을 사용하여 제자리에 고정합니다(그림3I). 슬라이드의 반대쪽으로 시체를 밀어 넣습니다. 나머지 흉부 방수부에 대해 반복하여 해부된 IFM 컬렉션을 생성합니다.
참고: IFM이 깔끔한 더미에 머무르지 않으면 티슈가있는 1x PBS 중 일부를 제거하십시오. 모든 PBS가 증발하지 않도록 주의하고 해부된 IFM과 헴미토락스가 버퍼로 덮여 있는지 확인하십시오. - 모든 IFM을 해부 한 후 해부 된 근육에 대한 품질 관리를 신속하게 수행하십시오. #5 집게를 사용하여 샘플에 들어갈 수 있는 점프 근육 이나 표피 조각을 제거합니다(그림3J-K').
참고: 점프 근육은 IFM과 다르게 나타납니다. Mef2-Gal4 표지 된 근육을 형광 하에 해부하면 점프 근육은 형광이 약하고 모양과 질감이 다릅니다. 정상 광원에서는 IFM이 불투명하고 유백색인 동안 거의 반투명으로나타납니다(그림 3J-J',K). - 물 장력을 사용하여 한 쌍의 집게 사이에 해부된 IFM을 캡처(스퀴시하지 는 않지만)(그림 3L). IFMs를 냉각된 1x PBS의 250 μL로 미리 채워진 1.5 mL 마이크로원심지 튜브로 이송한다(도3M). 섹션 4를 즉시 진행하십시오.
참고: 포셉 팁이 서로 근접하여 버퍼 용액에서 해제되면 물 장력으로 인해 포셉 팁 사이에 버퍼 거품이 포착됩니다. IFM이 이 버블에 존재하는 경우 용액에서 해제하고 다른 버퍼 채워진 리셉터클로 쉽게 전송할 수 있습니다. 버퍼 버블에 포착 된 조직을 macerating 피하기 위해 서로 만지지 않고 서로 가까이 팁을 가지고 집게를 짜내는 것이 중요합니다.
4. 펠릿 및 IFM 샘플 보존
- 1.5 mL 미세원심분리튜브를 탁상원심분리기에서 2,000 x g에서 3-5분 동안 원심분리하여 IFMs를 펠렛(도4A, B).
- 파이펫팁(그림4C)을사용하여 버퍼를제거합니다.
- RNA 적용의 경우, 원하는 RNA 분리 완충액의 50-100 μL에서 IFM 펠릿을 다시 일시 중단합니다(재료 표, 그림 4D참조). 그렇지 않으면 4.4 단계로 진행합니다.
참고: IFM은 질량 분석 준비 또는 상용 키트로 RNA의 격리를 위해 4.2단계 후에 건조 냉동될 수 있습니다(대표적인 결과 참조). RNA 적용의 경우 IFM 펠릿을 즉시 재중단하고 절연 버퍼로 동결하여 더 나은 결과를 얻을 수 있습니다. - 드라이 아이스에 샘플을 동결 하거나 액체 질소에 스냅 동결(그림 4E). 다운스트림 분석을 위한 시료 준비의 후속 단계가 준비될 때까지 -80°C에서 보관하십시오.
참고: 동결 보존 후, 샘플은 다운스트림 조사를 위해 처리하기 전에 몇 달 동안 저장할 수 있습니다.
결과
위에 제시된 해부 프로토콜은 성인 단계까지 puparium 형성 (APF) 후에 16 시간에서 IFM 농축 된 샘플을 생성하는 데 유용합니다. 해부 된 비행 근육 샘플은 여러 응용 프로그램에 사용할 수 있으며 지금까지 RT-PCR4,17,RNA-Seq16,32, ChIP36,37, 서양에 성공적으로 적용되었습니다. 블로팅 14,41 및 질량 분석 실험 (아래 참조). 잠재적인 사용자가 RNA 기반 응용 프로그램에 대해 해부할 수 있도록 먼저 IFM에서 RNA를 격리하기 위한 중요한 고려 사항을 강조하는 결과를 발표합니다. 보다 광범위하게 우리의 해부 프로토콜의 유용성을 입증하기 위해, 우리는 RNA 결합 단백질 Bruno1에 대한 우리의 데이터를 사용하여 가능한 -omics 응용 프로그램의 일부를 설명합니다.
IFM 해부 프로토콜은 고품질 RNA를 산출합니다.
코딩 mRNA는 총 RNA42의1-5%만을 구성하는 것으로 추정되기 때문에 사전에 해부될 파리의 수를 결정하는 것이 중요하다. 우리는 1 d 성인(그림 4F 및 보충 그림 1A)에서해부 된 IFM으로부터 비행 당 총 RNA의 평균 24 ± 9 ng를 얻었으며, 일반적으로 경험으로 수율이 증가합니다. 플라이당 총 RNA의 이 수율은 상대적으로 일정하며, IFM에 대해 16h APF, 24h APF, 30h APF, 48h APF, 72h APF 및 90h APF(도4F 및 보충 도 1B, D,E)에서해부된 IFM에 대해 약 25 ng 변동한다. 이 관측은 또한 이전 시간 점에서 격리된 견본에서 더 높을지도 모르다 지방, 힘줄, 기관 또는 그밖 세포 모형에서 격리된 어떤 RNA든지반영합니다. 따라서, 우리는 50개의 파리로부터 IFM으로부터 총 RNA의 >1 μg를 획득하고 일반적으로 100-150파리로부터 IFM을 해부하여 RNA-Seq 샘플에 대한 총 RNA의 >3 μg을 생성합니다.
RNA 격리 방법은 복구된 RNA의 양과 품질에 영향을 미치며 사용자가 격리 접근법을 검증할 것을 권장합니다. 예를 들어, 방법 1을 사용하여 격리하면 IFM으로부터 총 RNA의 평균 1143±465 ng를 50 1 d 성인 파리로부터 생성하지만, 다양한 상용 키트로 격리하여 총 RNA의 186±8 ng에서 1261±355 ng까지 어디서나 수율을 산출한다(도4G 및 추가 도 1C). 상용 키트로부터 분리된 RNA는 일반적으로 양호한품질(도 4H 및 보충 도 1F)이지만,낮은 회복은 RNA가 컬럼으로부터 효율적으로 용출되지 않을 수 있음을 시사한다. RNA 무결성은 또한 버퍼 체질 및 열 처리로 인해 하류 실험에 영향을 미칠 수있는 심각한 단편화로 이어질 수있는 방법 2(그림 4H, 두 번째 플롯)에서 수행 된 키트를 사용하여 손상 될 수 있습니다.
RNA 샘플을 분리하고 취급할 때 적절한 RNase 없는 기술을 관찰하는 것도 중요합니다. 동결 해동 주기와 4 시간 실온 배양은 RNA 무결성 프로파일에 극적으로 영향을 미치지 않지만 소량의 RNase조차도 빠른 RNA 분해로 이어진다(그림 4I 및 보충 방법). 사용자는 여전히 얼음에서 작업 하 고 RNA 가수 분해 및 단편화를 방지 하기 위해 동결 해동을 제한 하는 것이 좋습니다. 이것은 여기에서 검출되지 않았지만 필터 팁과 DEPC 처리 버퍼를 사용하여 RNase 오염을 방지하는 것은 절대적으로 필수적입니다.
역전사의 효율성은 다운스트림 애플리케이션의 성공에도 영향을 미칩니다. 우리는 리보소말 유전자 rp49 (그림 4J)에대한 강력한 RT-PCR 대역을 증폭시키는 3 개의 상용 RT 키트 중 두 가지를 통해 신뢰할 수있는 결과를 얻었습니다. 그러나, RT 키트 #2 3가지 생물학적 복제물 모두에 대해 RNA 결합 단백질 bru1에 대한 더 강한 밴드를 수득함에 따라 저발성 전사체의 검출에 더 민감할 수있다(도 4J). 종합하면, 이러한 결과는 고품질 RNA가 이 절차로 해부된 IFM으로부터 분리될 수 있음을 보여줍니다.
해부된 IFM은 고품질 mRNA-Seq 및 프로테오믹스 데이터를 생성합니다.
위의 프로토콜에 따라 30h APF, 72 h APF 및 1 d 성인 파리에서 해부된 IFM을 사용하여, 우리는 이전에 RNA 결합 단백질과 CELF1-homologue Bruno1 (Bru1, Arrest, Aret)이 IFM 특이적 접합 경로를 하류로 제어하는 것으로 나타났습니다. 전사 인자 스팔트 전공 (살름)16. IFM은 근육 특이적 bruno1 RNAi (bru1-IR)로 날아다니는 것뿐만 아니라 근육 특이적 브루노1 RNAi(bru1-IR)로 날아가는 것, 미오신 활동의 잘못된 조절, 궁극적으로 근육 섬유의 과수축 및 손실을16,17로 표시합니다. . 아래에서 우리는 전체 프로테오메 질량 분광법에 대한 해부 된 IFM의 유용성을 입증하고 RNA 수준에서 관찰 된 발현 변화 중 몇 가지가 단백질 수준에서도 분명하다는 것을 보여줍니다. 또한, 해부된 IFM의 mRNA-Seq 및 RT-PCR이 대체 스플라이스 이벤트의 규제를 입증하는 데 사용될 수 있음을 보여 주며, Bruno1에 의해 규제되는 것으로 밝혀진 Mhc의 특정 개발 스플라이스 이벤트를 더욱 강조합니다.
라이브러리 품질 및 깊이에 따라 mRNA-Seq 데이터는 유전자 단위 수준(유전자의 모든 엑온에 대한 평균 읽기 개수), 개별 엑온 또는 스플라이스 접합부 수준에서 분석할 수 있습니다. 야생형에 비해 bru1-IR IFMs로부터의 mRNA-Seq 데이터는 유전자 단위 레벨16에서의 발현의 약한 변화를 나타낸다(도5A). 72h APF에서, 이미 60A [Mlp60A]에서 근육 LIM 단백질, 액틴 57B [Act57B], 근육 특이적 단백질 300 kDa [Msp300], 또는 스트레딘-Mlck [Strn-Mlck])와 같은 sarcomere 유전자에 대한 추세가 있다. bru1-IR 근육(그림 5A 및 보충 표 1). 그러나, 우리는 이전에 개별 적인 exons의 수준에, Bruno1의 주요 기능은 대안 접합을 통제하는 것을 건의하는 특정 sarcomere 유전자 이소폼(16)의훨씬 더 강한 하향 조절이 있다는 것을 보여주었습니다(보충 표 1 ).
해부된 IFM에 대한 전체 프로테오메 질량 분석법을 사용하여, 우리는 단백질 수준에서 유사한 조절을 보여줄 수있다(도 5B 및 보충표 2). 검출된 1,895개의 펩티드 그룹 중 524개(28%) 그 중 1 d 성인에서 Bru1M2 돌연변이 IFM에서 잘못 조절됩니다(보충 표 2). Strn-Mlck 및 Mlp60A 단백질의 하향 조절도 관찰되며, mRNA-Seq 데이터의 전사체 수준에서 관찰을 일치시다. 특정 단백질 이소폼에 매핑되는 제한된 수의 데이터베이스 펩티드에도 불구하고(분석 세부 사항에 대한 보충 방법 참조), 사르코메르 단백질 Tropomyosin 1 (Tm1), 지지 (업/TnT), Mhc, 구부러진 (bt/projectin) 및 파라미오신 (Prm) 우리는 하나의 이소폼에서 펩티드의 상승 조절을 관찰하고 다른(그림 5B)RNA 수준16에유사한 규칙의 우리의 이전 관찰을 확인하. 이는 해부된 IFM이 mRNA-Seq 및 프로테오믹스 애플리케이션 모두에 유용하다는 것을 보여줍니다.
omics 데이터가 생물학적 통찰력을 향상시키고 확장하기 위해 전통적인 접근법을 보완할 수 있는 방법의 또 다른 예로, 우리는 Mhc의C-종단에 접합에 집중하기로 결정했습니다. weeP26이라고 하는 이전에 특징이 있는 단백질 트랩 라인은 Mhc43,44의 최종 인트론에 삽입된다(정확한 위치에 대한 보충 방법 참조). weeP26은 강력한 스플라이스 수용체를 포함하고 아마도 모든 Mhc 전사체에 통합된다(도5C). 그러나 IFM에 표시된 GFP 표지 단백질은 M 라인의 양쪽에 두 개의 "점"으로 통합되며 다리 근육에서는 M 라인을 균일하게 통합하고 두꺼운 필라멘트를 가로 질러 약하게 통합됩니다(그림 5E). Orfanos와 참새는 개발 Mhc 이소폼 스위치로 인해 IFM 형태로 이러한 "점"을 보였다: 48 시간 APF 전에 표현 된 Mhc 이소폼은 오픈 판독 프레임에서 weeP26 엑온 인서트으로 GFP 로 표시되어 있으며, Mhc는 48시간 APF 후 발현된 이소폼은 3'-UTR44에서정지 코돈의 하류에 포함되는 weeP26 엑온으로서 표지되지 않은 것이다.
우리의 mRNA-Seq 데이터는 우리가 C 말단 Mhc 이소폼 발현을 더 자세하게 특성화하는 것을 허용했습니다. 두 개의 다른 Mhc 종단이 보고된 동안43,44,우리의 mRNA-Seq 데이터 및 현재 플라이베이스 어노미 (FB2019_02)는 실제로 Mhc에서 세 가지 가능한 대체 스플라이스 이벤트가 있음을 시사합니다. C-종점 (엑시온 34-35, 34-36, 또는 34-37)(그림 5C),이는 RT-PCR에 의해 확인된다(그림 5D). 위P26 GFP는 엑손 36과 37 사이의 인트론에 삽입된다; 따라서, 모두 엑슨 34-35 와 엑슨 34-36 이소폼정지 코돈이 포함되어 있기 때문에, GFP는 엑슨 34-37 아이소폼에서만 번역할 수 있습니다(엑슨 34-GFP-37의 결과). 우리는 더 나아가 모든 Mhc 등소폼의 시간적 및 공간적 조절을 모두 볼 수 있었다. IFM에서, 우리는 27 °C에서 30 시간 APF와 48 시간 APF 사이 엑슨 34-35에엑슨 34-35에서 Mhc 이소폼 스위치를 관찰, 이 48 시간 APF에서 면역 형광에 의해 아직 보이지 않는 경우에도(그림 5E). 다리는 이미 30 h APF에서 엑슨 34-37 및 엑슨 34-35의 혼합물을 표현하고, 72 h APF에 의해 세 개의 Mhc 이소폼(그림 5D,F)을모두 표현한다. 성인 점프 근육 (TDT) 또한 모든 세 Mhc 이소폼을 표현(그림 5F),이것은 관 체 근육에 대 한 일반적으로 사실 제안. 따라서, 우리의 mRNA-Seq 데이터는 IFM에 있는 Mhc 이소폼 스위치를 위한 기간을 좁히고 관 근육에 있는 Mhc 이소폼 사용을 특성화해서 이전 사실 인정의 확장을 허용합니다.
살름 및 브루1 돌연변이체 IFM에서 Mhc 이소폼 조절을 검사한 후. 두 경우 모두 weeP26의잘못된 규제를 보았습니다. Salm 돌연변이 IFM은 Exon 34-36 이벤트의 이득을 포함하여 후기 단계에서 Mhc 등소형 발현 및 표현성 다리 접합 패턴에서 발달 스위치를 완료하지 못한다(그림 5F). 이것은 Salm의 손실이 관 근육에 IFM의 거의 완전한 운명 변환결과16이전 사실 인정과 동의합니다. Bru1-IR 및 bru1 돌연변이 IFM은 살름-/- IFM과 유사하며, 성인 단계(그림5E,F)를통해 Exon 34-37 스플라이스 이벤트를 유지하여 다리 근육을 닮은 weeP26 GFP 라벨링 패턴을 생성하며, 하지만 엑슨 34-36 이벤트를 얻지 못합니다. 이것은 Bruno1이 적어도 부분적으로 Mhc 대안 접합에서 발달 스위치를 제어하기 위해 IFM에서 필요하다는 것을 시사하지만, 추가 접합 인자는 또한 살름-/-컨텍스트에서 잘못 조절된다는 것을 나타낸다. 또한, 이 예제는 해부된 IFM의 RT-PCR 및 mRNA-Seq 데이터가 어떻게 발달 접합 메커니즘과 관찰된 형태학적 결함을 더 깊이 이해하는 데 유용할 수 있는지를 보여줍니다.
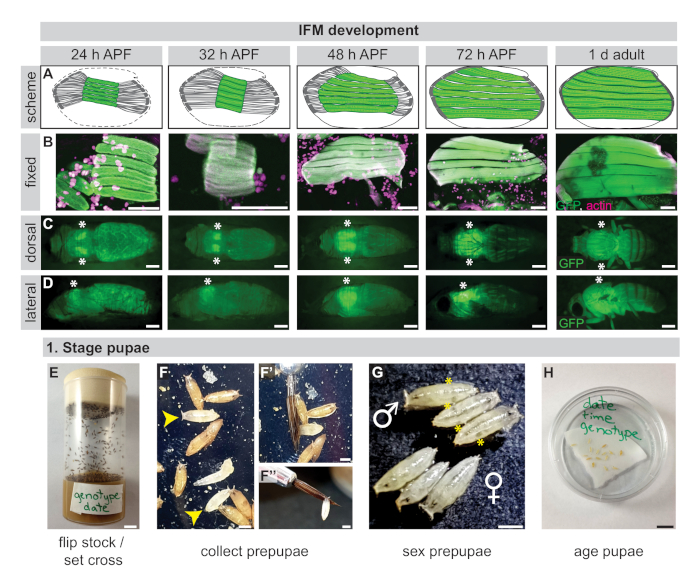
그림 1: IFM 개발 및 번데기의 준비. (a)24시간 APF, 32시간 APF, 48시간 APF, 72시간 APF, 및 1d 성인에서 IFM 개발의 회로도는 ~32시간 APF에서 비행 근육(녹색)의 다짐을 나타내고 이후의 섬유성장으로 흉부들을 채웁니다. 힘줄은 진한 회색입니다. (B)오픈 북 해부 (24 시간, 32 시간, 48 시간)에서 고정 된 IFMs의 공초점 이미지 (24 시간, 32 시간, 48 시간)19 또는 흉부 배미섹션 (72 h, 1 일) 액틴 (로다민 파할로이드, 마젠타) 및 GFP (녹색)에 대해 염색. (C, D) 살아있는 번데프에서 GFP 형광의 이미지는 등측(C) 또는 측측부(D) 평면에서 해부 플라이 라인의 IFM 형태를 그대로 예시한다. 별표는 IFM 위치를 표시합니다. (E)해부를 준비하려면, 플라이 스톡을 뒤집거나 십자가를 3-4일 전에 설정해야 합니다. (F)Prepupae는 흰색 (노란색 화살촉)에 의해 선택되고 젖은 페인트 브러시 (F',F'')를 사용하여 격리됩니다. (G)Prepupae는 후방에 위치한 반투명 공 (노란색 별표)로 나타나는 고환의 존재에 따라 남성과 여성을 분리하기 위해 성관계를 가져야합니다. (H)60mm 접시에 젖은 여과지에 숙성 된 번데어입니다. 배율 막대 = 100 μm (B), 1cm (C, D, E, H), 1mm (F,F',G). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
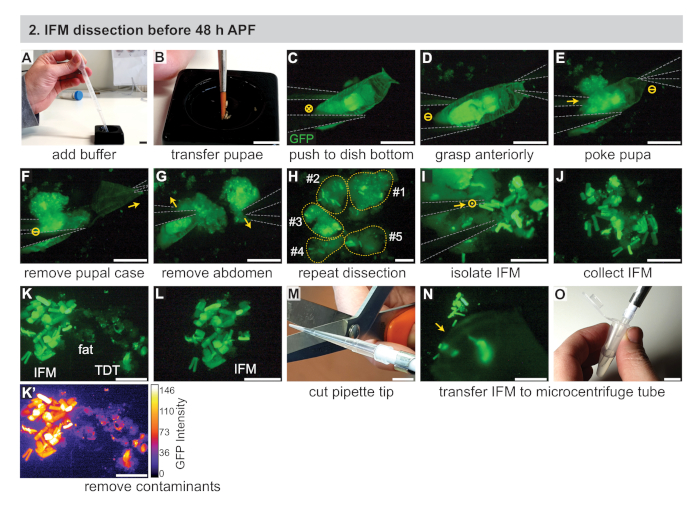
그림 2: 48 시간 APF 전에 IFM의 해부. (A)전사 피펫을 사용하여 검은 색 해부 접시에 1 x PBS 버퍼를 첨가합니다. (B)페인트 브러시를 사용하여 단계형 번데기의 이송. (C)GFP를 시각화하기 위해 형광 해부 현미경하에서, #5 집게 (회색으로 윤곽)를 사용하여 해부 접시의 바닥에 강아지를 부드럽게 밀어. 원의 "X"는 이미지에 모션을 나타냅니다. (D,E) 앞쪽 (D)의 pupae를 잡고, 흉부 (E) 바로 뒤에 있는 강아지를 찌르고. 원의 대시는 움직임을 나타내지 않습니다. (F,G)) 전방 집게(화살표)를 당겨 푸팔 케이스(F)를 제거한 다음 복부(G)를 제거합니다. (H)여러 개의 강아지에 대한 C-G의 반복. 노란색 점선은 기여 pupae를 나타내는 번호가 매겨져 있습니다. (I, J) 집게 (I)를 사용하여 IFM을 주변 조직 (J)으로부터 격리시. 원의 점은 페이지 밖으로 모션을 나타냅니다. (K, L) 깨끗한 IFM 샘플(L)을 생성하기 위해 지방 및 점프(TDT) 근육(K)을 포함한 오염물질을 제거한다. TDT는 IFM섬유(K')와는 GFP 발현이 낮고 형상이 다르다. (M, N, O) 잘린 피펫 팁(M)을 사용하여 해부된 IFMs(N)를 수집하고 미세원심지 튜브(O)로 의 이송한다. 스케일 바 = 1cm (A, B, M, O), 1mm (C-G), 500 μm (H-L, N). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
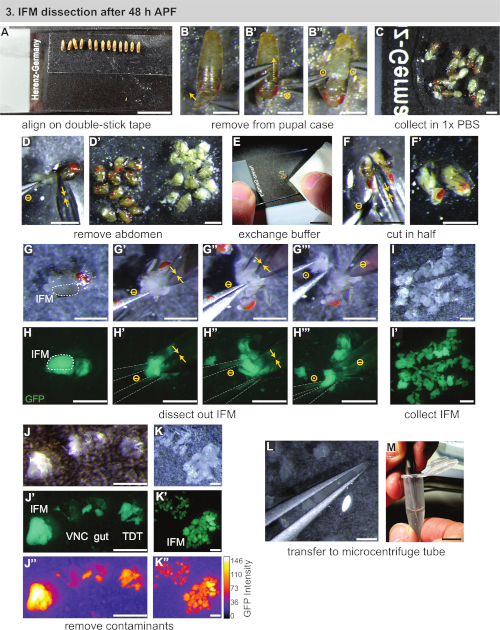
그림 3: 48 시간 APF 후 IFM의 해부. (A)양면 테이프에 번데기 정렬. (B)앞쪽 (B)을 열고 케이스를 등도 절단(B')하고,퍼파(B')를들어 올려 푸팔 케이스에서 푸페를 제거합니다. 원 기호는 그림 2와동일합니다. (C)번데기를 완충으로 옮김. (D)가위로 절단하여 복부를 제거하고 (노란색 이중 화살표) 및 thoraxes(D')에서분리. (E, F) 클린 버퍼 (E)의 추가, 다음 세로 절반에 토락의 절단(F,F'). (G, H) 해부는 백색광 (G) 또는 형광하에 수행되어 GFP (H)를 시각화할 수 있습니다. 한쪽에 IFM의 절단 (G'), 다음 다른 쪽 (G''); 집게 (회색으로 윤곽)와 흉부에서 들어 올린 (G''''). (I,J,K) 완충제(I)에서 IFM을 수집하고 오염된 복부 신경 코드(VNC), 창자 및 점프 근육(TDT)(J)을 제거하여 깨끗한 IFM 샘플(K)을 생성한다. TDT는 IFM 섬유(J', K')와 는 GFP 발현이 낮고 다른 형상을 가지고 있다. (L,M) 민인력을 사용하여 IFMs(L)를 미세원심지 튜브(M)로 이송합니다. 배율 막대 = 1cm (A, E, M), 1mm (B-D', F-L). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
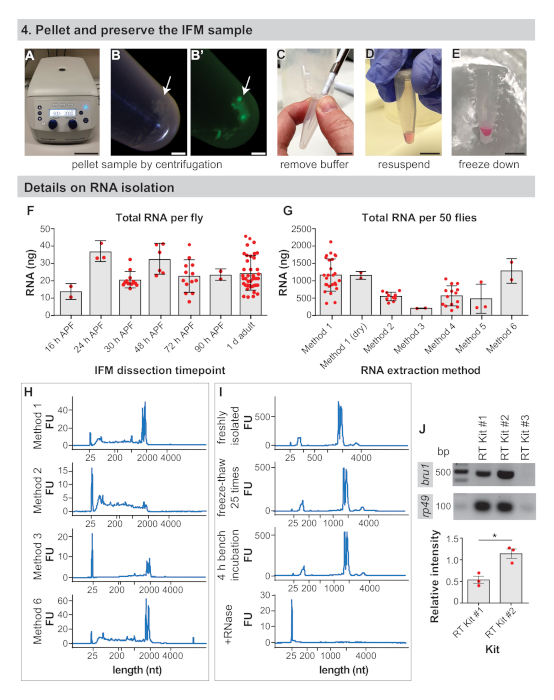
그림 4: IFM 보존 및 RNA 격리 세부 사항. (A)IFM은 2000 x g에서5 분 동안 원심 분리에 의해 펠트됩니다. (B)IFM 펠릿 (화살표) 및 형광 하에 펠릿 (B'). (C)파이펫 팁으로 모든 버퍼를 제거합니다. (D)RNA 추출의 경우, 격리 버퍼에서 펠릿의 재서스펜션. 이 단계는 동결 해부 IFMs로 건너 뛸 수 있습니다. (E)액체 질소 또는 드라이 아이스에서 시료를 동결시키고 -80°C에서 보관하십시오. 배율 막대 = 10cm (A), 1mm (B, B'), 1cm (C, D, E). (F)16h APF, 24h APF, 30h APF, 48h APF, 72h APF, 90h APF 및 1 d 성인에서 비행당 얻어진 해부된 IFM으로부터의 총 RNA의 나노그램(ng). 오차 막대 = SD.(G)IFM으로부터 분리된 총 RNA는 50 1 d성인 파리에서 해부되어 상이한 추출 방법을 사용한다. 오차 막대 = SD.(H)대표적인 추적은 상이한 추출 방법 후에 RNA 무결성을 분석한다. 리보좀 밴드는 2000뉴클레오티드(nt) 및 마커 밴드 25 nt. 보충 도1에서사용할 수 있는 추가 트레이스를 바로 밑에 실행한다. (I)새롭게 단리된 RNA 샘플(상부)의 대표적인 흔적, 드라이 아이스(2차 플롯)에서 25배의 시료 동결-해동, 벤치상에서 4시간 동안 방치된 샘플(제3 플롯), RNase A(하단 플롯)로 처리된 샘플. rNase A.(J)RT-PCR 젤을 브루1 및 rp49에대한 라벨이 붙은 키트에서 추가시 RNA의 완전한 분해를 주의하십시오. rp49에 대해 정규화된 bru1 밴드의 상대 강도는 아래에 플롯됩니다. 오류 막대 = SEM(페어링되지 않은 t-test, p = 0.0119). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
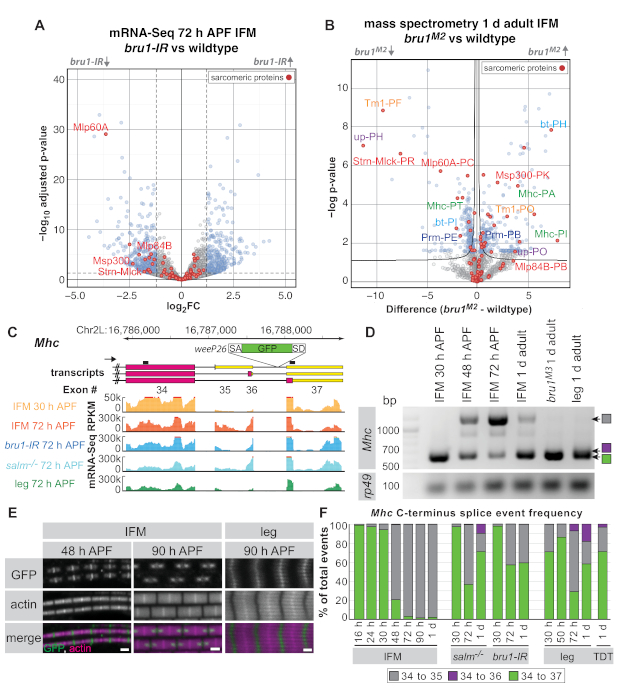
그림 5: 대체 접합에서 Bruno1 함수를 조사하기 위해 IFM 해부를 적용합니다. (a)72시간 APF에서 해부된 IFM으로부터의 mRNA-Seq 데이터(유전자 단위)의 화산 플롯. bru1-IR과 wildtype IFM (padj < 0.05, abs (log2FC) >1.5) 사이에서 현저하게 분별되는 유전자는 파란색으로 표시되고, 회색으로 중요하지 않은 유전자. Sarcomere 단백질은 빨간색으로 강조 표시되고, 선택 유전자는 표지됩니다. (B)전체 프로테오메 질량 분광법의 화산 플롯은 1d 성인 IFM에서 발생합니다. 브루M2돌연변이체와 야생형(FDR < 0.05)의 단백질은 회색의 청색, 중요하지 않은 단백질로 나타났다. 육종 단백질은 빨간색으로 강조 표시됩니다. (A)에 있는 유전자에 대응하는 펩티드는 적색으로 표지됩니다. 동일한 단백질의 상이한 이소폼에 매핑되는 펩티드 세트는 동일한 색상으로 표지된다. (C) weeP26 유전자 트랩의 뚜렷한 전사체 이소폼 및 삽입 위치를 예시하는 Mhc의 C-종단의 계획(삽입 점에 대한 보충 방법 참조). RT-PCR 프라이머는 전사체 위의 검은 선으로 표시됩니다. mRNA-Seq에서 백만 기지당 킬로베이스 당 읽기 카운트는 30 h APF (오렌지) 및 72 시간 APF (빨간색) 및 bru1-IR (파란색) 및 살름-/--에서 72 h APF에서 전체 다리 (녹색)에서 야생형에서 해부 IFMs에 대해 표시됩니다 . (D)30h APF 이후 의 IFM에서 이소폼 스위치를 보여주는 Mhc에 대한 프라이머RT-PCR. 엑슨 34-35 스플라이스 이벤트는 브루M3돌연변이 IFM 또는 성인 다리에서만 약하게 관찰된다. (e)90h APF 다리 근육에 비해 48시간 APF 및 90h APF에서 야생형 IFM 사코메레스에서 weeP26 GFP 국소화의 공초점 이미지. 스케일 바 = 1 μm.(F)표지된 유전자형 및 타임포인트에 대한 mRNA-Seq 데이터로부터의 스플라이스 접합 정량화. 정션 판독값은 특정 스플라이스 이벤트(Exon 34~35,35번 회색, 34~36회, 녹색 34~37개)의 비율로 제공되며, 총 이벤트 수는 엑소온 34 스플라이스 기증자를 공유합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
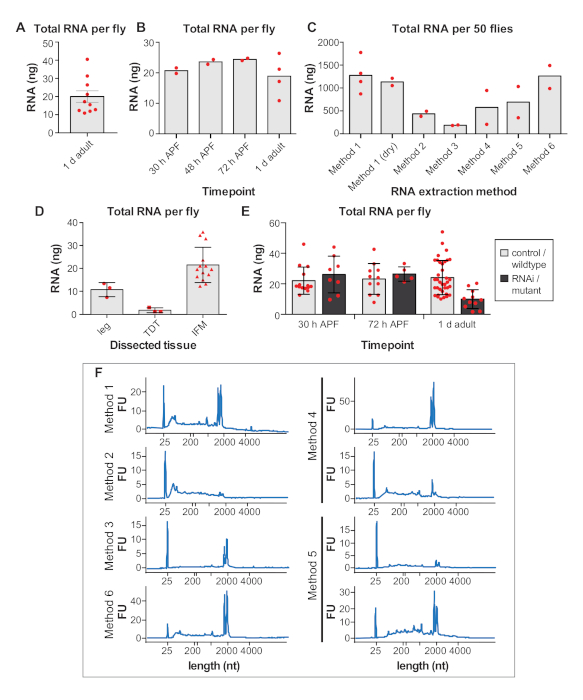
보충 도 1: (A, B, C)RNA는 같은 주에 동일한 연구원에 의해 해부된 동일한 유전자형의 샘플로부터 산출한다. 모든 샘플을 해부한 후, RNA를 분리하고 같은 날 측정하였다. (a) 1 d 성인 비행 당 IFM 해부로부터 얻은 총 RNA의 나노그램 (ng). 오차 막대 = SEM. (B) 30 h APF, 48 h APF, 72 h APF 및 1 d 성인에서 비행당 해부된 IFM으로부터 얻어진 총 RNA. (c) 50 1 d 성인으로부터 해부된 IFM으로부터 분리된 총 RNA는 상이한 추출 방법을 사용하여 파리를 비행한다. (D)해부된 다리, 점프 근육(TDT) 및 IFM으로부터 의 비행당 총 RNA 농도. 더 많은 RNA는 더 큰 IFMs로부터 수득된다. 오차 막대 = SD.(E)IFM의 비행당 총 RNA 농도는 30h APF, 72h APF 및 1 d 성인에서 RNAi 또는 돌연변이 샘플과 비교하여 대조군으로부터 해부된다. 돌연변이체의 경우, w1118을 야생형 대조군으로 사용하였습니다. 돌연변이 데이터는 bru1-IR, 살름-/- 및 다른 RNA 결합 단백질 돌연변이체로부터 컴파일됩니다. 이러한 조작에 대 한, RNA 수율은 근육 위축 및 손실로 인해 1 d 성인에서 감소, 그래서 더 많은 파리 omics 접근에 대 한 충분 한 수량을 얻기 위해 해부 될 필요가. 오차 막대 = SD.(F) 도 4G 및 보충 도 1C에도시된 RNA 격리 방법에 대한 RNA 무결성을 보여주는 추가 트레이스. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

추가 방법: 본 문호 전체에 걸쳐 사용되는 방법 및 시약에 대한 상세한 설명, 특히, 도 1A-D, 도 4F-K, 도 5,보충표 1,및 도표에도시된 데이터를 생성하기 위해서, 및 보충 표 2. 이러한 데이터는 해부 프로토콜에 동기를 부여하고 RNA 격리, mRNA-Seq, RT-PCR 및 프로테오믹스에 대한 그 유용성을 입증합니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
| 기본 텍스트의 그림 5 및 관련 단락과 관련 | |
| 탭 이름 | 데이터 요약 |
| 사르카레 단백질 | Spletter 외. Elife 2018에서 sarcomere 유전자의 목록; 여기에서 우리는 현재 FBgn 및 유전자 이름을 나열합니다. |
| SP 유전자 단위_DESeq2_72h | Spletter 외. EMBO Rep 2015에서 데이터를 사용하여, 우리는 72 시간 APF에 mRNA-Seq 데이터에 있는 sarcomere 유전자를 구체적으로 보았습니다. 이는 대조군(Mef2-Gal4, UAS-GFM-Gma가 w1118로 교차)과 Mef2-Gal4, UAS-GFM-Gma x Bruno1-IR 사이의 유전자 단위 수준에서 차등 발현을 검출하는 DESeq2 분석에서 이다. 노란색으로 강조 표시된 행은 규제된 유전자의 위/아래(log2FC=abs(1.5)의 임계값 위/아래)를 표시합니다. 이러한 데이터는 그림 5A의 빨간색 점 오버레이입니다. 각 sarcomere 유전자에 대해, 우리는 식별자 정보, DESeq2에서 log2FC, P 값 및 조정된 P 값, 뿐만 아니라 DESeq2 정규화된 발현 카운트를 제공합니다. |
| SP 엑손_덱세크_72h | Spletter 외. EMBO Rep 2015에서 데이터를 사용하여, 우리는 72 h APF에서 mRNA-Seq 데이터에서 sarcomere 유전자 엑슨 사용을 구체적으로 보았다. 이는 제어(Mef2-Gal4, UAS-GFM-Gma가 w1118로 교차)와 Mef2-Gal4, UAS-GFM-Gma x Bruno1-IR 간의 차등 엑슨 사용을 검출하는 DEXSeq 분석에서 나온 것입니다. 노란색으로 강조 표시된 행은 규제된 엑온(log2FC=abs(1.5)의 임계값 위/아래)를 위 또는 아래로 표시합니다. 우리는 엑슨 및 유전자 식별자 정보, DEXSeq에서 log2FC, P 값 및 조정 된 P 값뿐만 아니라 관련 전사체의 목록을 제공합니다. |
| 많은 유전자는 DEXSeq 분석에서 하나 이상의 엑슨의 조절을 보여 주며, 종종 높은 log2FC 값과 낮은 P 값 / 조정 P 값으로, 제한된 유전자 목록은 72 h APF에서 변화를 보여줍니다. 이것은 대체 접합의 규칙에 브루노의 손실의 강한 효과를 지원합니다. | |
보충표 1: 72시간 APF mRNA-Seq 데이터에서 차별적으로 발현된 유전자(DESeq2를 통해)및엑소(exonseq를 통해)를식별하는 사르코메르 단백질에 대한 표 는 브루1-IR 대야생형 IFMs이다.
| 기본 텍스트의 그림 5B 및 관련 단락과 관련된 항목 | |
| 탭 이름 | 데이터 요약 |
| 페르세우스 출력 | 이는 도 5B를 생성하는 데 사용되는 질량 분석 데이터를 제시하는 처리된 데이터 스프레드시트입니다. IFM 샘플은 1d 성인 대조군(w1118) 및 돌연변이(bruno1-M2)로부터 파리이다. 중요한 컬럼은 각 샘플에 대한 4개의 복제, t-검정 통계 및 유의성, 펩티드 ID 및 해당 유전자 이름 및 플라이베이스 ID에 대한 각 복제에 대한 형질전환 된 강도 값입니다. 부의미값은 페르세우스(FDR<.05)의 표준 설정을 사용하여 계산되었습니다. 1859개의 단백질/펩타이드가 검출되었으며, 그 중 524개(28%)가 검출되었습니다. 샘플 간에 크게 다릅니다. |
| 하향 조절 | 이들은 모두 252 bruno1-M2 돌연변이 IFM에서 하향 조절되는 페르세우스 출력에서 펩티드입니다. Flybase ID와 유전자 이름이 구식이기 때문에 현재 Flybase 유전자 ID와 유전자 이름을 추가로 제공합니다. |
| 업규제 | 이들은 모두 272 bruno1-M2 돌연변이 IFM에서 upregulated 는 Perseus 출력에서 펩티드. Flybase ID와 유전자 이름이 구식이기 때문에 현재 Flybase 유전자 ID와 유전자 이름을 추가로 제공합니다. |
| 그림 5B에서 빨간색으로 강조 표시된 sarcomere 단백질은 위의 목록에 있습니다. sarcomere의 일부로 간주되는 유전자목록은 보충표 1의 탭 중 하나에서 확인할 수 있다. | |
보충 표 2: 브루M2돌연변이체에서 차별적으로 발현된 단백질 및 단백질 이소폼을 식별하는 1 d 성인으로부터의 전체 프로테오메 질량 분석 데이터 표. 와일드 타입 IFMs.
토론
이 프로토콜에서는, 우리는 단백질, DNA, RNA 또는 그밖 거대 분자의 하류 격리를 위한 초기와 말기 pupae에서 Drosophila IFMs를 해부하는 기본적인 기술을 제시합니다. 이 프로토콜은 성인 파리에서 IFM을 해부하기 위해 쉽게 조정할 수 있습니다. 우리는 mRNA-Seq, 프로테오믹스 및 RT-PCR 애플리케이션에 대한 해부 프로토콜의 유용성을 입증합니다. 시작 재료가 적고 입력 농도가 낮은 샘플을 분석할 수 있는 omics 기술의 지속적인 개선으로 이러한 해부는 많은 추가 응용 분야에서 유용하게 사용될 것입니다. IFM은 인간 근병증에 대한 확립 된 모델이기 때문에4,24 및 근육 유형 특정 개발9,12, 예를 들어 IFM 농축 대사체학, 크로마틴 형태 조사 3C 또는 4C를 통해, CLiP 상호 작용 또는 근위 병의 인광 -프로테오믹스를 통해 네트워크 평가를 접합.
이러한 해부가 순수 IFM 샘플 대신 IFM용 으로 농축된 샘플을 생성한다는 점을 고려하는 것이 중요합니다. 이것은 모터 신경 내심, 힘줄 부착 및 근육 섬유의 기관 침입으로 인해 피할 수 없습니다. 생물정보학 분석은 IFM 농축 유전자 또는 단백질을 확인하기 위하여 이용될 수 있습니다, 그러나 추가 실험은 실제로 IFM 특정하다는 것을 증명하기 위하여 요구됩니다. 샘플 순도는 스트라이프45(힘줄), Act79B4,44(관형 근육), Act88F15(IFM) 또는 syb46(뉴런 특이적)과 같은 출판된 조직 특이적 마커를 사용하여 분석할 수 있다. 이러한 마커를 사용하여 IFM 특이적 콘텐츠로 데이터 세트를 정규화할 수 있지만, 사용자는 정상화를 위해 사용되는 유전자의 발현의 시간적 변화, 예를 들어 IFM 특이적 유전자 또는 튜불린의 경우 이러한 접근법을 편향시킬 수 있다는 경고를 받습니다.
유전자 부호화 조직 특이적 라벨링 방법, 예를 들어 EC-태깅47,48 또는 PABP 라벨링49,50 분리 RNA에 대한 최근 몇 년 동안 개발되었으며, 이는 진정으로 얻을 수 있는 데 도움이 될 수 있다. 조직 특정 RNA 견본. 그러나, EC 태깅은파리(47)의 지속적인 공급을 필요로 하므로 푸팔 단계 에는 적용되지 않는다. PABP 표지된 전사체의 민감도 및 완전성에는51에제한이 있을 수 있다. 개별 근육 섬유를 분리하는 FACS 접근법은 IFMs의 큰 크기와 동기화 특성에 의해 복잡합니다. INTACT52,53 스타일 접근법은 IFM으로부터 특정 세포실 구획을 분리하기 위해 적용될 수 있으며, 이는 IFM 핵 또는 미토콘드리아의 순수한 집단을 분리하는 데 유용할 수 있다. 수동 해부는 대부분의 다운스트림 응용에 대해 그대로 IFM 조직을 얻기 위해 여전히 현재 표준입니다.
시료 품질은 해부 공정의 몇 가지 중요한 단계에 따라 달라집니다. 해부는 기술적으로 까다롭고, 해부 속도와 샘플 순도는 경험에 따라 증가합니다. 세제 없이 냉각된 완충제에서 짧은 시간(20-30분)에 대해 해부하고 즉시 동결하면 마우스 힘줄격리(54)에대해 이전에 관찰된 바와 같이 시료 무결성을 보존하는 데 도움이 된다. IFM은 펠릿에서 모든 버퍼를 제거한 후 성공적으로 건조 동결될 수 있지만, 특히 RNA 격리를 위해, 분리 완충제에서 샘플을 동결하면 더 나은 결과를 얻을 수 있다. 최대 20개의 개별 해부로부터의 IFM은 RNA 또는 단백질 분리 이전에 결합되어 다운스트림 분석을 위해 초기 시점 또는 돌연변이체 16,32에서도충분한 물질을 확장하고 수집할 수 있습니다.
RNA 적용의 경우, 가장 중요한 단계는 RNA 자체의 분리일 수 있다. Guanidinium thiocyanate-페놀-클로로포름 절연(위의 방법 1)은 시험된 대부분의 상용 키트를 능가하며, 앞서 언급한 바와 같이 상당히저렴하다 55. 상용 키트와 RNA 격리 수율에서 관찰된 가변성은 이전관측56,57과일치한다. 우리는 또한 모든 RNA를 복구하는 데 도움이 이소 프로판 올 강수 시 글리코겐을 추가합니다. RNA 수율을 넘어서, RNA 무결성을 확인하여 해부 및 격리 공정 중에 샘플이 단편화되거나 저하되지 않았는지 확인하는 것이 중요합니다. RNase 가 없는 작업도 필수적입니다. 마지막으로, RT 키트의 선택은 역전사 공정의 감도에 영향을 미칠 수 있다. 자세히 설명하지는 않지만 이러한 모든 점은 IFM 샘플의 품질과 다운스트림 응용 프로그램에서 얻은 데이터에 영향을 미칩니다.
몇 가지 중요한 수정은 기존 IFM 해부 프로토콜과 는 별개로 프로토콜을 설정합니다. IFM 면역형광에 대한 상세한 해부 프로토콜이19개존재하지만, 이 프로토콜은 IFM 조직의 보다 신속한 분리를 허용하는 푸팔 해부에 대한 다른 접근법을 제시한다. 이것은 프로테오메 또는 전사체 변경을 방지하기 위하여 제한된 해부 시간을 가진 IFM 조직 (상대적으로 말하기)의 다량의 집합을 허용합니다. 다른 프로토콜은 개별 myofibrils에서 GFP 염색을 시각화하기 위한 성인 IFM의 해부를기술합니다 39 또는 애벌레 바디 벽 근육의 염색을 위해58,그러나 pupal 단계에서 또는 RNA 또는 단백질의 격리를 위해 해부를 다루지 않습니다. 이 접근법은 또한 더 순수한 IFM 샘플을 생성할 수 있지만 노동 집약적이고 더 적은 물질을 생성하는 저온 절38에서pupal IFM의 미세 해부에 대한 기존 프로토콜과 구별된다. 다른 급속성 IFM 해부 프로토콜38,39,IFM은 스트레스 유도 및 기타 주요 발현 변화를 제한하기 위해 세제 없이 PBS 버퍼로 단리된다.
이 프로토콜의 핵심 은 초기 pupal 단계에서 IFM을 격리 할 수 있도록 라이브 형광 리포터를 포함하는 것입니다. 우리는 표준적으로 Mef2-GAL459 운전 UAS-CD8::GFP 또는 UAS-GFP를 사용합니다::Gma60. 이것은 IFM의 차등 라벨링을 허용합니다 (비행 근육은 다른 pupal 근육보다 더 강하게 표시되고 다르게 모양) 뿐만 아니라 GAL4-UAS 기반 조작의 성능, 예를 들어 구조 또는 RNAi 실험. 또한 Mef2-GAL4를 욕조-GAL80ts와 결합하여 RNAi 관련 조기 치사를 피하거나 UAS-Dcr2와 결합하여 RNAi 효율40을증가시킬 수 있다.
Mef2-GAL4대신 사용할 수 있는 근육 유형 특이성, 시간발현 패턴 및 드라이버 강도19,61에서 변화하는 추가 GAL4 드라이버 또는 GFP 라인이 사용 가능합니다. 예를 들어 Act88F-GAL4는 처음에는 24h APF 를 중심으로 표현되므로 이전 타임포인트에는 사용할 수 없습니다. 그러나, 그것은 강하게 IFM를 표시하고 RNAi 관련 초기 치사성을 피하기 위하여 유용할 지도 모릅니다. 그-GFP 또는 Act88F-GFP라벨 IFM, 다시 시간적 제한으로, 그러나 그들은 마커 발현의 GAL4 의존성을 피하고 관심의 돌연변이 배경과 조합하여 유용할 수 있다. 다른 가능한 마커 라인의 목록은 사용할 수있습니다 19. 또한 유전자 및 GAL4/UAS 시스템의 사용은 유전자 발현 아티팩트를 유발할 수 있으므로, 예를 들어 드라이버 라인이 야생 형 배경 변형으로 교차하여 이러한 아티팩트가 아마도 그러하도록 적절한 컨트롤을 사용하는 것이 중요합니다. 모든 샘플에서 동일합니다.
첨부 된 비디오와 함께,이 상세한 프로토콜은 pupal IFM 해부를 보다 쉽게 접근할 수 있도록하고 근육 발달을 연구하기 위한 오믹스 접근법의 사용을 촉진하는 것을 목표로합니다. 해부된 IFM을 통해 접근할 수 있는 생화학 및 오믹스 분석과 Drosophila 유전학 및 세포 생물학의 힘을 결합하는 것은 myogenesis 및 근육 기능의 기계론적인 이해를 발전시키는 잠재력을 가지고 있습니다. 전사체 및 단백질 조절의 시스템 수준 관찰을 신진 대사 및 기능적 출력과 연결하는 향후 연구는 근육 유형 의 특정 개발 및 근육 장애의 발병 기전에 대한 깊은 이해를 제공할 것입니다.
공개
저자는 공개 할 것이 없다.
감사의 말
안드레아스 라두르너와 프랭크 슈노어에게 감사의 말입니다. 우리는 우수한 기술 지원과 질량 분석 데이터를 생성하기위한 아칸샤 로이 산드라 에세르 에게 감사드립니다. 우리는 블루밍턴과 비엔나 스톡 센터가 파리를 제공한 것을 인정합니다. 우리는 LMU 생물 의학 센터 (Martinsried, DE)에서 질량 분광 샘플의 분석을 위한 공초점 화상 진찰과 Zentrallabor für Proteinanalytik에 도움을 위한 핵심 시설 Bioimaging에 감사드립니다. 우리의 작업은 도이치 포르충스 게마인샤프트 (MLS, SP 1662/3-1), 통합 단백질 과학 뮌헨 센터 (CIPSM) 루드비히 막시밀리안 대학 뮌헨 (MLS), 프레데리히 - 바우어 스티퉁 (MLS), 그리고 국제 맥스에 의해 지원되었다 플랑크 연구 학교 (EN).
자료
| Name | Company | Catalog Number | Comments |
| 5x High Fidelity (HF) buffer | Thermo Fisher | F518L | |
| 60 mm culture dishes | Sigma-Aldrich | Z643084-600EA | Greiner dishes, 60 mm x 15 mM, vented |
| Black dissecting dish (glass) | Augusta Laborbedarf | 42021010 | Lymphbecken, black glass, 4 cm x 4 cm |
| Black silicon dissecting dishes: activated charcoal powder | Sigma-Aldrich | C9157 | Also available from most pharmacies |
| Black silicon dissecting dishes: Sylgard 184 | Sigma-Aldrich | 761036 | Dishes are made by mixing the Sylgard (~50g) with activated charcoal powder (200 mg) and curing it in Petri dishes (~4 60 mm dishes). |
| Blue pestle | Sigma-Aldrich | Z359947-100EA | Any pellet pestle that can sterilized, also can be used with a motor-driven grinder |
| Cell phone camera, Samsung Galaxy S9 | Samsung | SM-G960F/DS | used for photos not taken under a microscope |
| Chloroform | PanReac AppliChem | A3691,0500 | |
| Confocal microscope, Leica SP8X upright confocal | Leica | www.leica-microsystems.com | |
| Confocal microscope, Zeiss LSM 780 inverted confocal | Zeiss | www.zeiss.com | |
| Double stick tape | Scotch/3M | 3M ID 70005108587 | Double-sided tape, available at most office supply handlers |
| Dumont #5 Forceps | Fine Science Tools | 11252-20 | Inox straight tip 11 cm forceps, Biology grade with 0.05 mm x 0.02 mm tip |
| EtOH (100%, RNase free) | Sigma-Aldrich | 32205-M | |
| fluorescent dissecting microscope camera, Leica DFC310 FX camera | Leica | www.leica-microsystems.com | |
| Fluorescent dissecting microscope software, Leica Application Suite (LAS) version 4.0.0 | Leica | www.leica-microsystems.com | |
| Fluorescent dissecting microscope, Leica M165 FC | Leica | www.leica-microsystems.com | |
| Fly: Bru1[M2] | Fly stock; This paper | ||
| Fly: Bru1[M3] | Fly stock; This paper | ||
| Fly: Mef2-GAL4 | Bloomington Stock Center | BDSC:27390 | Fly stock |
| Fly: salm[1] | Bloomington Stock Center | 3274 | Fly stock |
| Fly: salm[FRT] | Fly stock; see Spletter et al., Elife, 2018 | ||
| Fly: UAS-Bru1IR | Vienna Drosophila Research Center | GD41568 | Fly stock, RNAi hairpin |
| Fly: UAS-GFP::Gma | Bloomington Stock Center | BDSC:31776 | Fly stock |
| Fly: UAS-mCD8a::GFP | Bloomington Stock Center | BDSC:5130 | Fly stock |
| Fly: w[1118] | Bloomington Stock Center | 3605 | Fly stock |
| Fly: weeP26 | Fly stock; see Clyne et al., Genetics, 2003 | ||
| GFP detection reagent, GFP-Booster | ChromoTek | gba488-100 | |
| Glycogen | Invitrogen | 10814-010 | |
| Image processing software, Photoshop CS6 | Adobe | www.adobe.com | |
| Isopropanol | Sigma-Aldrich | I9516-25ML | 2-propanol |
| Method 1 (RNA isolation): TRIzol | Life Technologies | 15596018 | Guanidinium isothiocyanate and phenol monophasic solution |
| Method 2 (RNA isolation): Method 1 + TURBO DNA-free Kit | Invitrogen | AM1907 | TRIzol isolation followed by treatment with a kit to remove DNA |
| Method 3 (RNA isolation): Direct-zol RNA Miniprep Plus Kit | Zymo Research | R2070S | RNA isolation in TRIzol, but over commercial columns instead of using phase separation. Recommended DNase treatment performed with Monarch Dnase I in Monarch DNase I Reaction buffer. |
| Method 4 (RNA isolation): RNeasy Plus Mini Kit | Qiagen | 74134 | We used the provided DNase treatment. IFM pellets were homogenized in RTL buffer as suggested for animal tissues. |
| Method 5 (RNA isolation): ReliaPrep RNA Tissue Miniprep System | Promega | Z6110 | We applied the protocol for ‘Purification of RNA from Fibrous Tissues’. |
| Method 6 (RNA isolation): Monarch Total RNA Miniprep Kit | New England Biolabs | T2010G | We applied the protocol for tissues/leukocytes and lysed in 300 μL of RNA Protection Reagent. |
| Microcentrifuge tubes | Thermo Fisher | AM12400 | RNase-free Microfuge Tubes, 1.5 mL |
| Microscope slides | Thermo Fisher | 12342108 | Standard slides, uncharged, 1 mm |
| Microtome blades | PFM Medical | 207500003 | C35 feather 80 mm |
| Monarch DNase I | New England Biolabs | T2004-21 | |
| Monarch DNase I Reaction Buffer | New England Biolabs | T2005-21 | |
| Normal goat serum | Thermo Fisher | 16210072 | |
| OneTaq Polymerase | New England Biolabs | M0480X | |
| Paintbrush | Marabu | 1910000000 | Marabu Fino Round No. 0, or similar brush from any art supply |
| Paraformaldehyde | Sigma-Aldrich | 158127 | |
| PBS buffer (1x) | Sigma-Aldrich | P4417 | Phosphate buffered saline tablets for 1 L solutions, pH 7.4 |
| PFA PureTip Pipette Tips | Elemental Scientific | ES-7000-0101 | Optional substitute for standard pipette tips to reduce sample loss; 100 mL, 0.8 mm orifice |
| Phusion High Fidelity Polymerase | Thermo Fisher | F-530XL | |
| Pipette tips | Sigma-Aldrich | P5161 | Universal 200 mL pipette tips |
| Preomics iST 8x Kit | Preomics | P.O.00001 | peptide preparation kit for mass spectrometry |
| Q Exactive mass spectrometer | Thermo Fisher | 725500 | mass spectrometry was performed at the Protein Analysis Unit of the LMU Biomedical Center |
| Qubit RNA Assay Kit | Life Technologies | Q32855 | |
| Rhodamine-phalloidin | Invitrogen, Molecular Probes | 10063052 | |
| RNA concentration Approach 1 & RNA integrity traces, Bioanalyzer | Agilent Technologies | G2939BA | |
| RNA concentration Approach 2, Nanodrop | Thermo Fisher | ND-2000 | |
| RNA concentration Approach 3, Qubit 4 Fluorometer | Invitrogen | Q33238 | |
| RNA Pico Chips | Agilent Technologies | 5067-1513 | |
| RNase A | Promega | A7937 | |
| RNase-free water, Diethyl pyrocarbonate (DEPC) | Sigma-Aldrich | D5758 | DEPC treat water overnight and then autoclave, to remove all RNase. |
| RT Kit #1: Super Script III Reverse Transcriptase Kit | Invitrogen | 18080-044 | reverse transcription kit |
| RT Kit #2: LunaScript | New England Biolabs | E3010S | reverse transcription kit |
| RT Kit #3: QuantiNova Reverse Transcription Kit | Qiagen | 205410 | reverse transcription kit |
| Slide mounting buffer, Vectashield | Vector Laboratories | H-1200 | containing DAPI |
| Statistical software: GraphPad Prism | GraphPad Prism | www.graphpad.com | |
| Statistical software: Microscoft Excel | Microsoft | Purchased as part of the bundle: Office Home & Student 2019 | |
| Table-top centrifuge | Eppendorf | 5405000760 | Eppendorf Centrifuge 5425 or equivalent |
| Tissue/ Kimwipes | Sigma-Aldrich | Z188956 | Standard tissue wipes |
| Transfer pipette | Sigma-Aldrich | Z350796 | Plastic pipette |
| Triton-X100 | Sigma-Aldrich | T9284-500ML | |
| Vannas spring scissors | Fine Science Tools | 15000-00 | 3 mm cutting edge, tip diameter 0.05 mm, length 8 cm |
| Whatman paper | Sigma-Aldrich | 1004-070 | Filter paper circles, Grade 4, 70 mm |
참고문헌
- Rexiati, M., Sun, M., Guo, W. Muscle-Specific Mis-Splicing and Heart Disease Exemplified by RBM20. Genes. 9 (1), 18 (2018).
- Guo, W., et al. RBM20, a gene for hereditary cardiomyopathy, regulates titin splicing. Nature Medicine. 18 (5), 766-773 (2012).
- Guo, W., et al. Splicing Factor RBM20 Regulates Transcriptional Network of Titin Associated and Calcium Handling Genes in The Heart. International Journal of Biological Sciences. 14 (4), 369-380 (2018).
- Nikonova, E., Kao, S. -. Y., Ravichandran, K., Wittner, A., Spletter, M. L. Conserved functions of RNA-binding proteins in muscle. The International Journal of Biochemistry & Cell Biology. 110, 29-49 (2019).
- Wang, E. T., et al. Dysregulation of mRNA Localization and Translation in Genetic Disease. The Journal of Neuroscience. 36 (45), 11418-11426 (2016).
- Wang, E. T., et al. Antagonistic regulation of mRNA expression and splicing by CELF and MBNL proteins. Genome Research. 25 (6), 858-871 (2015).
- Kalsotra, A., et al. A postnatal switch of CELF and MBNL proteins reprograms alternative splicing in the developing heart. Proceedings of the National Academy of Sciences of the United States of America. 105 (51), 20333-20338 (2008).
- Ho, T. H., et al. Muscleblind proteins regulate alternative splicing. The EMBO Journal. 23 (15), 3103-3112 (2004).
- Lemke, S. B., Schnorrer, F. Mechanical forces during muscle development. Mechanisms of Development. 144, 92-101 (2017).
- Iwamoto, H. Structure, function and evolution of insect flight muscle. Biophysics. 7, 21-28 (2011).
- Schnorrer, F., Dickson, B. J. Muscle building; mechanisms of myotube guidance and attachment site selection. Developmental Cell. 7 (1), 9-20 (2004).
- Spletter, M. L., Schnorrer, F. Transcriptional regulation and alternative splicing cooperate in muscle fiber-type specification in flies and mammals. Experimental Cell Research. 321 (1), 90-98 (2014).
- Benoist, P., Mas, J. A., Marco, R., Cervera, M. Differential muscle-type expression of the Drosophila troponin T gene. A 3-base pair microexon is involved in visceral and adult hypodermic muscle specification. Journal of Biological Chemistry. 273 (13), 7538-7546 (1998).
- Schönbauer, C., et al. Spalt mediates an evolutionarily conserved switch to fibrillar muscle fate in insects. Nature. 479 (7373), 406-409 (2011).
- Bryantsev, A. L., et al. Extradenticle and Homothorax Control Adult Muscle Fiber Identity in Drosophila. Developmental Cell. 23 (3), 664-673 (2012).
- Spletter, M. L., et al. The RNA-binding protein Arrest (Bruno) regulates alternative splicing to enable myofibril maturation in Drosophila flight muscle. EMBO Reports. 16 (2), 178-191 (2015).
- Oas, S. T., Bryantsev, A. L., Cripps, R. M. Arrest is a regulator of fiber-specific alternative splicing in the indirect flight muscles of Drosophila. The Journal of Cell Biology. 206 (7), 895-908 (2014).
- Kim, J. H., Jin, P., Duan, R., Chen, E. H. ScienceDirect Mechanisms of myoblast fusion during muscle development. Current Opinion in Genetics & Development. 32, 162-170 (2015).
- Weitkunat, M., Schnorrer, F. A guide to study Drosophila muscle biology. Methods. 68 (1), 2-14 (2014).
- Rai, M., Nongthomba, U., Grounds, M. D. Skeletal Muscle Degeneration and Regeneration in Mice and Flies. Mechanisms of Regeneration. 108, 247-281 (2014).
- Swank, D. M., Wells, L., Kronert, W. A., Morrill, G. E., Bernstein, S. I. Determining structure/function relationships for sarcomeric myosin heavy chain by genetic and transgenic manipulation of Drosophila. Microscopy Research and Technique. 50 (6), 430-442 (2000).
- de Joussineau, C., Bataillé, L., Jagla, T., Jagla, K. Diversification of muscle types in Drosophila: upstream and downstream of identity genes. Current Topics in Developmental Biology. 98, 277-301 (2012).
- Maqbool, T., Jagla, K. Genetic control of muscle development: learning from Drosophila. Journal of Muscle Research and Cell Motility. 28 (7-8), 397-407 (2008).
- Jagla, K., Kalman, B., Boudou, T., Hénon, S., Batonnet-Pichon, S. Beyond mice: Emerging and transdisciplinary models for the study of early-onset myopathies. Seminars in Cell & Developmental Biology. 64, 171-180 (2017).
- Haigh, S. E., et al. Drosophila indirect flight muscle specific Act88F actin mutants as a model system for studying congenital myopathies of the human ACTA1 skeletal muscle actin gene. Neuromuscular Disorders. 20 (6), 363-374 (2010).
- Batonnet-Pichon, S., et al. Myofibrillar Myopathies: New Perspectives from Animal Models to Potential Therapeutic Approaches. Journal of Neuromuscular Diseases. 4 (1), 1-15 (2017).
- Kreipke, R. E., Kwon, Y. V., Shcherbata, H. R., Ruohola-Baker, H. Drosophila melanogaster as a Model of Muscle Degeneration Disorders. Current Topics in Developmental Biology. 121, 83-109 (2017).
- Souidi, A., Zmojdzian, M., Jagla, K. Dissecting Pathogenetic Mechanisms and Therapeutic Strategies in Drosophila Models of Myotonic Dystrophy Type 1. International Journal of Molecular Sciences. 19 (12), 4104 (2018).
- Sparrow, J., Hughes, S. M., Segalat, L. Other Model Organisms for Sarcomeric Muscle Diseases. Advances in Experimental Medicine and Biology. 642, 192-206 (2008).
- Lloyd, T. E., Taylor, J. P. Flightless flies: Drosophila models of neuromuscular disease. Annals of the New York Academy of Sciences. 1184, 1-20 (2010).
- Swank, D. M. Mechanical analysis of Drosophila indirect flight and jump muscles. Methods. 56 (1), 69-77 (2012).
- Spletter, M. L., et al. A transcriptomics resource reveals a transcriptional transition during ordered sarcomere morphogenesis in flight muscle. eLife. 7, 1361 (2018).
- Weitkunat, M., Kaya-Çopur, A., Grill, S. W., Schnorrer, F. Tension and force-resistant attachment are essential for myofibrillogenesis in Drosophila flight muscle. Current Biology. 24 (7), 705-716 (2014).
- Gunage, R. D., Dhanyasi, N., Reichert, H., VijayRaghavan, K. Drosophila adult muscle development and regeneration. Seminars in Cell & Developmental Biology. 72, 56-66 (2017).
- Weitkunat, M., Brasse, M., Bausch, A. R., Schnorrer, F. Mechanical tension and spontaneous muscle twitching precede the formation of cross-striated muscle in vivo. Development. 144 (7), 1261-1272 (2017).
- Zappia, M. P., Rogers, A., Islam, A. B. M. M. K., Frolov, M. V. Rbf Activates the Myogenic Transcriptional Program to Promote Skeletal Muscle Differentiation. Cell Reports. 26 (3), 702-719 (2019).
- Zappia, M. P., Frolov, M. V. E2F function in muscle growth is necessary and sufficient for viability in Drosophila. Nature Communications. 7 (1), 10509 (2016).
- Bryantsev, A. L., et al. Myogenesis in Drosophila melanogaster: Dissection of Distinct Muscle Types for Molecular Analysis. Methods in Molecular Biology. 1889 (5), 267-281 (2019).
- Xiao, Y. S., Schöck, F., González-Morales, N. Rapid IFM Dissection for Visualizing Fluorescently Tagged Sarcomeric Proteins. Bio-Protocol. 7 (22), (2017).
- Kaya-Çopur, A., Schnorrer, F. RNA Interference Screening for Genes Regulating Drosophila Muscle Morphogenesis. Myogenesis. 1889, 331-348 (2019).
- Chechenova, M. B., et al. Functional redundancy and non-redundancy between two Troponin C isoforms in Drosophila adult muscles. Molecular Biology of the Cell. 28 (6), 760-770 (2017).
- Alberts, B. . Molecular Biology of the Cell. , (2017).
- Clyne, P. J., Brotman, J. S., Sweeney, S. T., Davis, G. Green fluorescent protein tagging Drosophila proteins at their native genomic loci with small P elements. Genetics. 165 (3), 1433-1441 (2003).
- Orfanos, Z., Sparrow, J. C. Myosin isoform switching during assembly of the Drosophila flight muscle thick filament lattice. Journal of Cell Science. 126 (1), 139-148 (2013).
- Volohonsky, G., Edenfeld, G., Klambt, C., Volk, T. Muscle-dependent maturation of tendon cells is induced by post-transcriptional regulation of stripeA. Development. 134 (2), 347-356 (2007).
- Estes, P. S., Ho, G. L., Narayanan, R., Ramaswami, M. Synaptic localization and restricted diffusion of a Drosophila neuronal synaptobrevin--green fluorescent protein chimera in vivo. Journal of Neurogenetics. 13 (4), 233-255 (2000).
- Hida, N., et al. EC-tagging allows cell type-specific RNA analysis. Nucleic Acids Research. 45 (15), 138 (2017).
- Thomas, A., et al. A versatile method for cell-specific profiling of translated mRNAs in Drosophila. PLoS ONE. 7 (7), 40276 (2012).
- Yang, Z. Isolation of mRNA from specific tissues of Drosophila by mRNA tagging. Nucleic Acids Research. 33 (17), 148 (2005).
- Jiao, Y., Moon, S. J., Montell, C. A Drosophila gustatory receptor required for the responses to sucrose, glucose, and maltose identified by mRNA tagging. Proceedings of the National Academy of Sciences of the United States of America. 104 (35), 14110-14115 (2007).
- Blazie, S. M., et al. Comparative RNA-Seq analysis reveals pervasive tissue-specific alternative polyadenylation in Caenorhabditis elegans intestine and muscles. BMC Biology. 13 (1), 1775-1821 (2015).
- Henry, G. L., Davis, F. P., Picard, S., Eddy, S. R. Cell type-specific genomics of Drosophila neurons. Nucleic Acids Research. 40 (19), 9691-9704 (2012).
- Deal, R. B., Henikoff, S. The INTACT method for cell type-specific gene expression and chromatin profiling in Arabidopsis thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Grinstein, M., Dingwall, H. L., Shah, R. R., Capellini, T. D., Galloway, J. L. A robust method for RNA extraction and purification from a single adult mouse tendon. PeerJ. 6 (8), 4664 (2018).
- Green, M. R., Sambrook, J. . Molecular Cloning. , (2012).
- Brown, R. A. M., et al. Total RNA extraction from tissues for microRNA and target gene expression analysis: not all kits are created equal. BMC Biotechnology. 18 (1), 16 (2018).
- Ford, K. L., et al. Optimisation of laboratory methods for whole transcriptomic RNA analyses in human left ventricular biopsies and blood samples of clinical relevance. PLoS ONE. 14 (3), 02136855 (2019).
- Ramachandran, P., Budnik, V. Dissection of Drosophila larval body-wall muscles. Cold Spring Harbor Protocols. (8), 5469 (2010).
- Ranganayakulu, G., Schulz, R. A., Olson, E. N. Wingless signaling induces nautilus expression in the ventral mesoderm of the Drosophila embryo. Developmental Biology. 176 (1), 143-148 (1996).
- Dutta, D., Bloor, J. W., Ruiz-Gómez, M., VijayRaghavan, K., Kiehart, D. P. Real-time imaging of morphogenetic movements in Drosophila using Gal4-UAS-driven expression of GFP fused to the actin-binding domain of moesin. Genesis. 34 (1-2), 146-151 (2002).
- Lemke, S. B., Schnorrer, F. In Vivo Imaging of Muscle-tendon Morphogenesis in Drosophila Pupae. Journal of Visualized Experiments. (132), e57312 (2018).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유