Method Article
Dissektion von Drosophila melanogaster Flight Muscles for Omics Approaches
In diesem Artikel
Zusammenfassung
Drosophila Flugmuskel ist ein leistungsfähiges Modell, um Transkriptionsregulation, alternative Spleißung, Stoffwechsel und Mechanobiologie zu studieren. Wir präsentieren ein Protokoll zur Zerlegung von fluoreszierend markierten Flugmuskeln aus lebenden Pupas, um hoch angereicherte Proben zu erzeugen, die ideal für Proteomik und Tiefensequenzierung sind. Diese Proben können wichtige mechanistische Einblicke in verschiedene Aspekte der Muskelentwicklung bieten.
Zusammenfassung
Drosophila Flugmuskel ist ein leistungsfähiges Modell, um verschiedene Prozesse wie Transkriptionsregulation, alternative Spleißung, Stoffwechsel und Mechanobiologie zu studieren, die alle beeinflussen Muskelentwicklung und Myofibrillogenese. Omics-Daten, wie sie durch Massenspektrometrie oder tiefe Sequenzierung erzeugt werden, können wichtige mechanistische Einblicke in diese biologischen Prozesse liefern. Für solche Ansätze ist es vorteilhaft, gewebespezifische Proben zu analysieren, um sowohl die Selektivität als auch die Spezifität der Omics-Fingerabdrücke zu erhöhen. Hier stellen wir ein Protokoll zur Zerlegung von fluoreszierend markierten Flugmuskeln aus lebenden Pupae vor, um hoch angereicherte Muskelproben für Omics-Anwendungen zu erzeugen. Wir beschreiben zunächst, wie man Flugmuskeln in frühen Pupalstadien seziert (<48 h nach Pupariumbildung [APF]), wenn die Muskeln durch die Kennzeichnung von grünem fluoreszierendem Protein (GFP) erkennbar sind. Wir beschreiben dann, wie man Muskeln von späten Welpen (>48 h APF) oder Erwachsenen seziert, wenn Muskeln unter einem Sezierenmikroskop unterscheidbar sind. Das begleitende Videoprotokoll wird diese technisch anspruchsvollen Sezierungen für die Muskel- und Drosophila-Forschungsgemeinschaften breiter zugänglich machen. Für RNA-Anwendungen prüfen wir die Quantität und Qualität von RNA, die zu verschiedenen Zeitpunkten und mit unterschiedlichen Ansätzen isoliert werden können. Wir zeigen weiter, dass Bruno1 (Bru1) für eine zeitliche Verschiebung in der Myosin-Schwerenkette (Mhc) spleißen notwendig ist, was zeigt, dass sezierte Muskeln für mRNA-Seq, Massenspektrometrie und Reverse-Transkriptionspolymerase-Kettenreaktion (RT-PCR) verwendet werden können. Anwendungen. Dieses Sezierprotokoll wird dazu beitragen, gewebespezifische Omics-Analysen zu fördern und kann allgemein angewendet werden, um mehrere biologische Aspekte der Myogenese zu untersuchen.
Einleitung
Moderne Omics-Technologien liefern wichtige Einblicke in die Muskelentwicklung und die Mechanismen, die menschlichen Muskelerkrankungen zugrunde liegen. Beispielsweise hat die Analyse von Transkriptomikdaten in Verbindung mit genetischer und biochemischer Verifikation in Tiermodellen ergeben, dass der Verlust des Spleißfaktors RBM20 aufgrund der Regulierung eines Zielnetzes von mehr als 30 Sarcomere eine erweiterte Kardiomyopathie verursacht. Gene, die zuvor mit Herzerkrankungen in Verbindung gebracht wurden, einschließlich Titin1,2,3.
In einem zweiten Beispiel haben Studien von Zellkultur, Tiermodellen und menschlichen Patienten gezeigt, dass myotonische Dystrophie durch eine Störung der RNA-Regulierung durch Sequestrierung von Muscleblind (MBNL) und Upregulation von CELF14,5verursacht wird. Die interregulierende und zeitliche Dynamik zwischen MBNL und CELF1 (auch CUGBP1 oder Bruno-Like 2 genannt) hilft, die anhaltenden embryonalen Spleißmuster bei myotonischen Dystrophie-Patienten zu erklären. Darüber hinaus hilft das große Netzwerk von falsch regulierten Zielen, die komplexe Natur derKrankheit4,6,7,8zu erklären. Eine Mehrheit dieser Studien nutzt omics Ansätze in genetischen Modellorganismen, um die Mechanismen zu verstehen, die menschlichen Muskelerkrankungen zugrunde liegen. Darüber hinaus unterstreichen sie die Bedeutung des ersten Verständnisses der zeitlichen und Gewebe-spezifischen Genexpression, Proteinmodifikation und Metabolische Muster in gesunden Muskeln, um Veränderungen in kranken oder alternden Muskeln zu verstehen.
Drosophila melanogaster ist ein weiterer etablierter genetischer Modellorganismus. Die Struktur des Sarcomere sowie einzelne Sarkome-Komponenten sind von Fliegen bis wirbeltiere stark konserviert4,9,10, und die indirekten Flugmuskeln (IFMs) sind zu einem leistungsstarken Modell zum Studium geworden mehrere Aspekte der Muskelentwicklung11,12. Erstens unterscheiden sich die fibrillaren Flugmuskeln funktionell und morphologisch von den röhrenförmigen Körpermuskeln11,13, was die Untersuchung muskelspezifischer Entwicklungsmechanismen ermöglicht. Transkriptionsfaktoren wie Spalt major (Salm)14, Extradenticle (Exd) und Homothorax (Hth)15 wurden als fibrillare Schicksalsregulatoren identifiziert. Zusätzlich leitet der CELF1-Homolog Bruno1 (Bru1, Aret) flussabwärts von Salm ein fibrillarspezifisches Spleißprogramm16,17.
Zweitens sind IFMs ein wichtiges Modell für das Verständnis des Prozesses der Myogenese selbst, von Myoblast-Fusion und Myotube-Bindung an Myofibrillogenese und Sarcomere Reifung9,18,19. Drittens ermöglicht die Drosophila-Genetik die Untersuchung von Beiträgen einzelner Proteine, Proteindomänen und Proteinisoformen zur Sarcomere-Bildung, Funktion und biophysikalischen Eigenschaften20,21,22 ,23. Schließlich wurden IFM-Modelle für die Untersuchung mehrerer menschlicher Muskelerkrankungen entwickelt, wie myotonische Dystrophie, myofibrillare Myopathien, Muskeldegenerative Störungen, Aktinopathien, etc.24,25,26 ,27, und haben wichtige Einblicke in Krankheitsmechanismen und mögliche Therapien28,29,30. Daher ist Drosophila ein nützliches Modell, um viele offene Fragen im Bereich der Myogenese zu beantworten, einschließlich Mechanismen der muskelspezifischen Transkription, Spleißen und Chromatinregulation, sowie die Rolle des Stoffwechsels bei der Muskelentwicklung. Die Anwendung moderner Omics-Technologien, insbesondere in Kombination mit der Vielzahl genetischer, biochemischer und zellbiologischer Assays in Drosophila,hat das Potenzial, das Verständnis von Muskel Entwicklung, Alterung und Krankheit.
IFMs sind die größten Muskeln der Fliege und überspannen fast 1 mm über die gesamte Länge des Thorax bei Erwachsenen31,32. Diese kleine Größe stellt jedoch die Herausforderung dar, genügend Proben zu erhalten, um Omics-Technologien in Drosophila auf gewebespezifische Weise anzuwenden. Darüber hinaus sind IFMs Teil der erwachsenen Muskulatur, die während der Pupalstadien gebildet wird. Myoblasten verschmelzen zu Myotuben, die nach der Pupariumbildung (APF) um 24 h an Sehnen anhaften und sich einem Verdichtungsschritt unterziehen, der notwendig ist, um die Myofibrillogenese um 30 h APF zu initiieren (Abbildung 1A-D)18,33, 34.
Die Myofiber wachsen dann, um die gesamte Länge des Thorax zu überspannen, wobei Myofibrils eine erste Wachstumsphase durchlaufen, die sich auf die Sarcomere-Zugabe bis etwa 48 h APF konzentriert und dann in eine Reifephase übergeht, in der Sarkome in Länge und Breite wachsen und umgestaltet, um die Stretch-Aktivierung durch 72 h APF (Abbildung 1A-D)32,35zu etablieren. Der Beginn der Faserreifung wird zumindest teilweise von Salm und E2F32,36,37und mehreren IFM-spezifischen Sarkomre-Protein-Isoformen kontrolliert, deren Spleißen durch Bru1 gesteuert wird, werden dabei eingearbeitet. Phase16,17. Reife Fliegen aus der Nähe von 90-100 h APF. Dies bedeutet, dass IFM zur Untersuchung der Muskelentwicklung mit ausreichender Quantität, Qualität und Reinheit von mehreren Pupal-Zeitpunkten isoliert werden muss, um die Analyse mit omics-Ansätzen zu erleichtern.
Mehrere Protokolle für die IFM-Sektion wurden veröffentlicht. Diese Protokolle eignen sich zwar gut für ihre beabsichtigten Anwendungen, aber keines ist ideal für omics-Ansätze. Protokolle, die die IFM-Morphologie zur Immunfluoreszenz von Pupal- und erwachsenen IFMs19erhalten, IFM-Fasern für die mechanische Auswertung isolieren31oder Mikrodissektion von Pupal-IFM aus Kryosektionen38 nutzen, sind zu spezialisiert und arbeitsintensiv, um ausreichend IFM-Gewebe für Omics-Anwendungen zu erhalten. Andere Protokolle wurden für die schnelle Zerlegung von speziell erwachsenen IFM38,39entwickelt, daher sind sie nicht auf Pupalstadien anwendbar und verwenden Puffer, die nicht ideal sind oder beispielsweise mit der RNA-Isolierung unvereinbar sein können. Daher ist es notwendig, neue Ansätze zu entwickeln, um pupal IFM für Biochemie- oder Omics-Anwendungen zu isolieren.
Hier stellen wir ein Protokoll zur Zerlegung von IFM während der Pupalstadien vor, das erfolgreich für die mRNA-Seq-Analyse von 16 h APF bis zur Erwachsenenstufe16,32verwendet wurde. Das Protokoll verwendet ein grünes fluoreszierendes Protein (GFP) Label, um IFMs in allen Stadien der Pupal- und Erwachsenenentwicklung zu identifizieren, was eine lebende Sezierung unter einem fluoreszierenden Sezierenmikroskop ermöglicht. Der Ansatz ist weniger arbeitsintensiv und hat einen höheren Durchsatz als bestehende IFM-Sektionsprotokolle. Dies ermöglicht eine schnelle Isolierung und Kryokonservierung von Proben, wodurch nach mehreren Sezierensrunden genügend Material für Omics-Ansätze sowie für Standard-Reverse-Transkriptionspolymerase-Kettenreaktion (RT-PCR) oder Western Blotting erzeugt wird.
Wir präsentieren das Protokoll in zwei Teilen und zeigen, wie IFMs sowohl vor 48 h APF (während der frühen Metamorphose, wenn IFM-Anhänge zäher sind) als auch nach 48 h APF (wenn der pupal body plan und IFM-Anhänge gut definiert sind) schnell seziert werden können. Wir zeigen, dass wir qualitativ hochwertige RNA zu allen Zeitpunkten von sezierten IFMs isolieren und Daten zu verschiedenen Ansätzen zur RNA-Isolation und Reverse-Transkription präsentieren können. Schließlich zeigen wir die Anwendung des Dissektionsprotokolls auf mRNA-Seq, Massenspektrometrie und RT-PCR am Beispiel des CELF1-Homologs Bruno1. Wir zeigen Fehlexpression von Sarkome-Protein-Isoformen in Proteomik-Daten von Bruno1 mutant IFM und untersuchen Bruno1 Regulierung des C-Terminal Spleice Ereignis von Myosin schwere Kette (Mhc). Diese Ergebnisse veranschaulichen, wie Omics-Daten ein tieferes Verständnis biologischer Phänomene ermöglichen und genetische und biochemische Experimente ergänzen.
Protokoll
1. Inszenierung der Pupae
- Heben Sie Fliegen des gewünschten Genotyps in Flaschen an (Abbildung 1E). Entweder machen Sie einen frischen Flip des Sezierens oder setzen Sie ein Kreuz mit mindestens 20 weiblichen Jungfraufliegen. Halten Sie Flaschen, bis die Fliegen beginnen zu verpupieren.
- Vorpupae mit einem benetzten Pinsel sammeln und in einer 60 mm Petrischale auf benetztes Filterpapier übertragen (Abbildung 1F).
- Sex die Pupae, sammeln das entsprechende Geschlecht für das Experiment (Abbildung 1G). Männchen werden durch das Vorhandensein von Hoden identifiziert, die als durchscheinende Kugeln in der ansonsten undurchsichtigen Pupa erscheinen.
- Beschriften Sie die Petrischale mit der Uhrzeit, dem Datum und dem Genotyp, und altern Sie dann die Welpen auf das gewünschte Stadium(Abbildung 1H).
HINWEIS: Bewahren Sie Kreuze/Bestände und Alterspupas in einem temperaturgeregelten Inkubator (d. h. 25 °C oder 27 °C für RNAi-Kreuze), da erhöhte Gal4-Aktivität bei höheren Temperaturen die Knock-down-Effizienz erhöht40). Stellen Sie sicher, dass die Luftfeuchtigkeit ausreichend hoch ist, damit die Welpen nicht austrocknen, wenn sie mehrere Tage altern.
2. IFM-Sektion vor 48 h APF
- Montieren Sie die notwendige Ausrüstung, darunter zwei #5 Biologie-Grade-Zangen, eine Pipette, Pipettenspitzen, Trockeneis und (für RNA-Proben) Isolationsreagenz (siehe Tabelle der Materialien). Darüber hinaus sind kalte schwarze Sezierende (siehe Tabelle der Materialien), 1x Phosphat-gepufferter Saline-Puffer (PBS) und 1,5 ml Mikrozentrifugenrohre auf Eis.
- Mit einem benetzten Pinsel, übertragen inszenierte Pupae auf eine schwarze Sesektierungschale gefüllt etwa zwei Drittel mit kalten 1x PBS (Abbildung 2A,B). Wechseln Sie zu einem fluoreszierenden Sezierendes Mikroskop.
HINWEIS: Verwenden Sie so viele Pupae, wie innerhalb eines 30-min-Zeitfensters seziert werden können. Je nach Erfahrung reicht dies von 3-15 Welpen. Siehe Ergänzende Methoden zur Diskussion über Alternativen zu schwarzen Sezieren von Gerichten. - Mit #5 Zange, drücken Sie einen der Pupas auf den Boden einer schwarzen Sezieren schale und passen Sie den Mikroskop-Zoom und Fokus, um deutlich zu sehen, die Pupa (Abbildung 2C).
- Greifen Sie das Vorderteil der Pupa mit einer Zange (Abbildung 2D), dann schlagen Sie die Pupae mit einer einzigen Spitze der anderen Zange leicht aus der Mitte in den Bauch, direkt hinter dem Thorax. Dadurch wird die Pupa an Ort und Stelle und verhindert, dass die IFMs in den Bauch bewegen (Abbildung 2E).
HINWEIS: Beginnen Sie, die Länge der Sezierung von diesem Punkt zu timing, sobald die Pupalintegrität gestört ist. Verwenden Sie eine definierte Sezierender Länge (z. B. 20–30 min), um den Muskeltod und die damit verbundenen transkriptomischen und proteomischen Veränderungen zu minimieren. Sezieren Sie so viele Fliegen wie möglich in dieser Zeit. - Entfernen Sie mit den ersten Zangen die vordere Hälfte des Pupalgehäuses (Abbildung 2F).
- Verwenden Sie die gleichen Zangen, um die exponierten Pupas direkt hinter dem Thorax zu kneifen, und trennen Sie den Bauch vom Thorax (Abbildung 2G).
- Mit den Zangen den vorderen Teil des Thorax (für <35 h APF) vorsichtig drücken oder den Thorax öffnen, um die fluoreszierend beschrifteten IFMs(Abbildung 2H)freizulegen. IFMs lösen sich leicht von der Epidermis, da Sehnenbefestigungen zu frühen Zeitpunkten zerbrechlich sind. Entsorgen Sie den verbleibenden Kadaver mit Zangen, um ihn auf die gegenüberliegende Seite der Schale zu schieben.
- Wiederholen Sie die Schritte 2.3–2.7, sezieren Sie zusätzliche Welpen.
- Sammeln Sie die IFM-Fasern mit Zangen und organisieren Sie sie in einem Stapel am Boden der schwarzen SezierenSchale (Abbildung 2I,J). Entfernen Sie alle Trümmer, indem Sie sie mit Zangen aus dem Sichtfeld schieben.
HINWEIS: Mit der Praxis können Zangenspitzen in die Nähe gebracht werden, ohne sich gegenseitig zu berühren. Diese Technik kann verwendet werden, um IFMs lose zu packen, ohne sie zu zerstören. Alternative Methoden umfassen das sanfte Drücken oder Heben der IFMs mit einer einzigen Spitze oder vollständig geschlossenen Zange oder die Einnahme von Fett oder anderem Gewebe mit dem IFM und das Entfernen des Fettes, wie in Schritt 2.10 beschrieben. - Qualitätskontrolle der IFM-Muskelprobe, mit den Zangen, um Nicht-IFM-Muskeln, Fett, Nagelhaut, etc. aus der Probe zu entfernen (Abbildung 2K,L).
HINWEIS: Mit Mef2-Gal4 ist IFM zu frühen Zeitpunkten stärker als andere Muskeltypen gekennzeichnet(Abbildung 2K,K'),was die Entfernung von Sprungmuskel- und Larvenmuskeln auf der Grundlage der Fluoreszenzintensität und Muskelform ermöglicht. Fett und Nagelhautgewebe sehen anders aus und sind nicht durch ein muskelspezifisches Fluoreszenzetikett gekennzeichnet (Abbildung 2K,K'). Weitere Gal4-Zeilen mit der Bezeichnung IFM finden Sie im Diskussionsabschnitt. - Übertragen Sie den IFMs-Stapel mit einer abgeschnittenen Pipettenspitze in ein 1,5 ml Mikrozentrifugenrohr, das mit 250 l gekühlten 1x PBS gefüllt ist (Abbildung 2M-O). Fahren Sie sofort mit Abschnitt 4 fort.
HINWEIS: IFM-Proben können einfach durch Kleben an der Seite der Pipettenspitze verloren gehen. Pipettierpuffer nach oben und unten mehrmals vor dem Sammeln von IFMs können Standardspitzen weniger klebrig machen, und silikonisierte oder Perfluoralkoxy (PFA) Spitzen (siehe Tabelle der Materialien) mit niedrigeren Oberflächenspannungen können helfen, Probenverlust zu verhindern.
3. IFM-Sektion nach 48 h APF
- Montieren Sie die notwendigen Geräte, darunter zwei #5 Biologie-Grade-Zangen, feine Schere, Standard-Glasmikroskop-Dias, Doppel-Stick-Band, Pipette, Pipettenspitzen, Trockeneis und (für RNA-Anwendungen) Isolationsreagenz (siehe Tabelle der Materialien). Die 1x PBS und Mikrozentrifugenrohre auf Eis abkühlen.
- Übertragen Sie die inszenierten Welpen mit einem leicht benetzten Pinsel auf einen Streifen doppelseitiger Klebebande, die auf einem Mikroskopschlitten montiert sind (Abbildung 3A). Platzieren Sie die Pupae in einer Linie, die in der gleichen Ausrichtung ausgerichtet ist (ventral nach unten und vordernach zum unteren Rand der Folie).
HINWEIS: Achten Sie darauf, nicht zu viel Wasser auf dem Pinsel oder Filter zu verwenden, oder die Pupae wird nicht gut kleben. Wenn Die Welpen nicht kleben, trocknen Sie sie, indem Sie sie zuerst in einen Trockenenoder oder Gewebepapier überführen. Montieren Sie so viele Pupae, wie innerhalb eines 30 min Zeitfensters seziert werden können, idealerweise 10 Pupae. - Entfernen Sie die Pupa aus dem Pupal-Gehäuse. Verwenden Sie Zangen, um auseinander zu necken und öffnen Sie das Pupalgehäuse über den vorderen Spiracles (Abbildung 3B).
- Schieben Sie vorsichtig ein Paar Zangen dorsal in Richtung des Hinterteils, schneiden Sie das Pupal-Gehäuse, wenn sich die Zangen bewegen (Abbildung 3B'). Achten Sie darauf, die zugrunde liegende Pupa nicht zu brechen. Befreien Sie den Pupa aus dem geöffneten Gehäuse und übertragen Sie ihn sofort auf einen Tropfen von 1x PBS auf einem zweiten Mikroskopschlitten (Abbildung 3B",C).
- Wiederholen Sie die Schritte 3.3 und 3.4 für alle Welpen in der Linie, und stellen Sie dann das Doppelknüppel-Dia beiseite.
- Mit der feinen Schere den Bauch der Pupa vom Thorax wegschneiden und in einen separaten Haufen schieben (Abbildung 3D,D'). Wiederholen Sie dies für die verbleibenden Welpen.
HINWEIS: Beginnen Sie mit dem Timing der Sezieren mit Schritt 3.6, sobald die Pupalintegrität gestört ist. Sezieren Sie so viele Fliegen wie möglich in 20-30 min, um den Zelltod und die damit verbundenen transkriptomischen und proteomischen Veränderungen zu verhindern. Bei der Sezieren 1 d Erwachsene oder >90 h Pupae, ist es oft bequem für spätere Schritte, um zusätzlich den Kopf mit der feinen Schere zu entfernen. - Entfernen Sie mit einem Tissuepapier den Großteil der 1x PBS (in der Regel trüb mit hängendem Fett) sowie den Bauchstapel(Abbildung 3E). Fügen Sie einen Tropfen frische, gekühlte 1x PBS zu den verbleibenden Thoraxen hinzu.
- Verwenden Sie die Schere, um den Thorax in die Hälfte zu schneiden (Abbildung 3F,F'),indem Sie vom Kopf die Längskörperachse in einer einzigen Bewegung herunterschneiden. Wenn der Kopf entfernt wurde, legen Sie zunächst die Schere ein, an der der Kopf befestigt war, und schneiden Sie die obere Hälfte des Thorax längs zwischen den IFMs. Schneiden Sie dann die ventrale Seite des Thorax mit einem zweiten Schnitt in der gleichen Ausrichtung.
- Wiederholen Sie die Schritte 3.7 und 3.8, damit alle Welpen seziert werden, wodurch ein Haufen Thoraxhalbsektionen in der Nähe der Mitte der Rutsche erzeugt wird. Stellen Sie sicher, dass genügend gekühlte 1x PBS auf der Rutsche vorhanden sind, damit die Hemisections nicht austrocknen.
HINWEIS: Nach 48 h APF sind IFMs groß genug, um unter einem Standard-Sezierendes Mikroskop für das geschulte Auge sichtbar zu sein. An dieser Stelle des Protokolls können Muskeln mit einem fluoreszierenden Etikett in einen fluoreszierenden Sezierenden Bereich verschoben werden, um bei der IFM-Identifikation oder zu Trainingszwecken zu helfen, aber dies ist nicht notwendig. - Sezieren Sie die IFMs aus dem Thorax. Isolieren Sie einen der Hemisektionen mit den #5 Zangen (Abbildung 3G,H). Legen Sie vorsichtig die Spitzen einer Zange über und unter der Mitte der IFMs ein (Abbildung 3G',H'). Während Sie die ersten Zangen still halten, verwenden Sie eine feine Schere, um ein Ende des IFM von der Nagelhaut und den Sehnen wegzuschneiden. Dann schneiden Sie das andere Ende des IFM frei von der Nagelhaut (Abbildung 3G'',H'').
HINWEIS: Abhängig von der Ausrichtung des Thorax nach dem ersten IFM-Schnitt ist es sinnvoll, den Thorax um 180° zu drehen, damit der zweite IFM-Schnitt einfacher durchzuführen ist. - Entfernen Sie das IFM-Bundle mit Zangen aus dem Thorax (Abbildung 3G''',H'''), und übertragen Sie es an den Rand der PBS-Blase, um es mit Wasserspannung an Ort und Stelle zu halten (Abbildung 3I). Schieben Sie den Kadaver auf die gegenüberliegende Seite der Rutsche. Wiederholen Sie dies für die verbleibenden Thorax-Hemisektionen, wodurch eine Sammlung von sezierten IFMs generiert wird.
HINWEIS: Wenn die IFMs nicht in einem ordentlichen Stapel bleiben, entfernen Sie einige der 1x PBS mit einem Gewebe. Achten Sie darauf, dass nicht alle PBS verdampfen, und stellen Sie sicher, dass die sezierten IFMs und Hemithoraxes durch Puffer abgedeckt bleiben. - Nach der Sezieren aller IFMs führen Sie schnell eine Qualitätskontrolle für den sezierten Muskel durch. Entfernen Sie mit #5 Zangen alle Sprungmuskel- oder Nagelfragmente, die ihren Weg in die Probe gefunden haben könnten (Abbildung 3J-K'').
HINWEIS: Sprungmuskel erscheint anders als IFM. Wenn Mef2-Gal4 beschriftet Muskel unter Fluoreszenz, Sprungmuskel hat eine schwächere Fluoreszenz und eine andere Form und Textur. Bei normalem Licht erscheint es fast durchscheinend, während die IFMs ein undurchsichtiges, milchiges Gelb sind (Abbildung 3J-J'',K). - Mit Wasserspannung, erfassen (aber nicht squish) die sezierten IFMs zwischen einem Paar Zangen (Abbildung 3L). Übertragen Sie die IFMs in ein 1,5 ml Mikrozentrifugenrohr, das mit 250 l gekühlten 1x PBS vorgefüllt ist (Abbildung 3M). Fahren Sie sofort mit Abschnitt 4 fort.
HINWEIS: Wenn Zangenspitzen in einander in die Nähe gebracht und aus einer Pufferlösung gehoben werden, führt die Wasserspannung dazu, dass eine Pufferblase zwischen den Zangenspitzen eingefangen wird. Wenn IFMs auch in dieser Blase vorhanden sind, können sie aus der Lösung gehoben und einfach in eine andere puffergefüllte Buchse übertragen werden. Es ist wichtig, die Zange zu drücken, um die Spitzen in die Nähe zu bringen, ohne sich gegenseitig zu berühren, um zu vermeiden, dass das in der Pufferblase gefangene Gewebe mazeriert wird.
4. Pellet und Konservieren der IFM-Probe
- Pellet die IFMs durch Zentrifugieren des 1,5 ml Mikrozentrifugenrohres für 3–5 min bei 2.000 x g in einer Tischzentrifuge (Abbildung 4A,B).
- Entfernen Sie den Puffer mit einer Pipettenspitze (Abbildung 4C).
- Für RNA-Anwendungen können Sie das IFM-Pellet in 50–100 l des gewünschten RNA-Isolationspuffers aussetzen (siehe Materialtabelle, Abbildung 4D). Fahren Sie andernfalls mit Schritt 4.4 fort.
HINWEIS: IFMs können nach Schritt 4.2 für Massenspektrometriepräparate oder Isolierung von RNA mit kommerziellen Kits trockengefroren werden (siehe repräsentative Ergebnisse). Bei RNA-Anwendungen werden bessere Ergebnisse erzielt, indem das IFM-Pellet im Isolationspuffer sofort wieder ausgesetzt und eingefroren wird. - Probe auf Trockeneis einfrieren oder in flüssigem Stickstoff einfrieren (Abbildung 4E). Bei -80 °C lagern, bis sie für nachfolgende Schritte in der Probenvorbereitung für die nachgelagerte Analyse bereit sind.
HINWEIS: Nach der Kryokonservierung können Proben mehrere Monate gelagert werden, bevor sie für die nachgelagerte Untersuchung verarbeitet werden.
Ergebnisse
Die oben vorgestellten Dissektionsprotokolle sind nützlich, um IFM-angereicherte Proben von 16 h nach Pupariumbildung (APF) bis zum Erwachsenenstadium zu erzeugen. Sezierte Flugmuskelproben können für mehrere Anwendungen verwendet werden und wurden bisher erfolgreich für RT-PCR4,17, RNA-Seq16,32, ChIP36,37, Western angewendet 14,41 und Massenspektrometrieexperimente (siehe unten). Um potenziellen Anwendern zu helfen, für RNA-basierte Anwendungen zu sezieren, stellen wir zunächst unsere Ergebnisse vor, die wichtige Überlegungen speziell für die Isolierung von RNA von IFMs hervorheben. Um den Nutzen unserer Sezierprotokolle weiter zu veranschaulichen, veranschaulichen wir dann anhand unserer Daten zum RNA-bindenden Protein Bruno1 einige der möglichen –omics-Anwendungen.
IFM-Sektionsprotokoll liefert hochwertige RNA
Es ist wichtig, die Anzahl der Fliegen zu bestimmen, die im Voraus seziert werden sollen, da die Codierung mRNA schätzungsweise nur 1–5% der gesamten RNA42ausmachte. Wir erhielten durchschnittlich 24 x 9 ng der gesamten RNA pro Fliege von IFM seziert von 1 d Erwachsenen(Abbildung 4F und ergänzende Abbildung 1A), mit Erträgen in der Regel mit Erfahrung zu erhöhen. Diese Ausbeute der gesamten RNA pro Fliege ist relativ konstant und schwankt um 25 ng für IFM seziert bei 16 h APF, 24 h APF, 30 h APF, 48 h APF, 72 h APF und 90 h APF (Abbildung 4F und Ergänzende Abbildung 1B,D,E). Diese Beobachtungen spiegeln auch jede RNA wider, die aus kontaminierendem Fett, Sehne, Luftröhre oder anderen Zelltypen isoliert ist, die in Proben höher sein kann, die von früheren Zeitpunkten isoliert wurden. So erhielten wir aus 50 Fliegen >1 g Gesamt-RNA aus IFM und sezieren IFM typischerweise von 100 bis 150 Fliegen, um für RNA-Seq-Proben >3 g GESAMT-RNA zu erzeugen.
Die Methode der RNA-Isolation beeinflusst die Quantität und Qualität der zurückgewonnenen RNA, und wir ermutigen Anwender, ihren Isolationsansatz zu validieren. Während beispielsweise die Isolierung mit Methode 1 durchschnittlich 1143 x 465 ng der gesamten RNA aus IFM von 50 1 d erwachsenen Fliegen erzeugt, ergibt sich die Isolierung mit verschiedenen kommerziellen Kits überall von 186 x 8 ng bis 1261 x 355 ng der gesamten RNA(Abbildung 4G und Ergänzende Abbildung 1C). RNA, die aus kommerziellen Kits isoliert ist, ist im Allgemeinen von guter Qualität(Abbildung 4H und Ergänzende Abbildung 1F), aber niedrige Rückgewinnungen deuten darauf hin, dass RNA möglicherweise nicht effizient aus den Spalten eluiert wird. Die RNA-Integrität kann auch durch die Verwendung eines Kits wie in Methode 2(Abbildung 4H, zweites Diagramm) beeinträchtigt werden, wahrscheinlich aufgrund von Pufferkonstitution und Wärmebehandlungen, was zu einer starken Fragmentierung führt, die nachgelagerte Experimente beeinflussen kann.
Es ist auch wichtig, die richtige RNase-freie Technik bei der Isolierung und Handhabung von RNA-Proben zu beachten. Obwohl Gefrier-Tau-Zyklen und eine 4 h Raumtemperatur-Inkubation die RNA-Integritätsprofile nicht dramatisch beeinflussen, führen selbst kleine Mengen RNase zu einem schnellen RNA-Abbau (Abbildung 4I und ergänzende Methoden). Die Benutzer werden immer noch ermutigt, auf Eis zu arbeiten und Frosttau zu begrenzen, um RNA-Hydrolyse und Fragmentierung zu verhindern. Dies wurde hier nicht erkannt, aber die Verhinderung einer RNase-Kontamination durch die Verwendung von Filterspitzen und DEPC-behandelten Puffern ist absolut notwendig.
Die Effizienz der Reverse-Transkription wirkt sich auch auf den Erfolg nachgelagerter Anwendungen aus. Wir haben zuverlässige Ergebnisse mit zwei von drei kommerziellen RT-Kits erhalten, die wir getestet haben, die beide starke RT-PCR-Bänder für das ribosomale Gen rp49 verstärken (Abbildung 4J). RT Kit #2 kann jedoch empfindlicher für den Nachweis von niedrig exprimierten Transkripten sein, da wir stärkere Bänder für das RNA-bindende Protein bru1 für alle drei biologischen Replikationen erhalten haben (Abbildung 4J). Zusammengenommen zeigen diese Ergebnisse, dass hochwertige RNA von IFMs isoliert werden kann, die mit diesem Verfahren seziert werden.
Sezierte IFMs produzieren hochwertige mRNA-Seq- und Proteomikdaten
Mit IFM seziert nach dem obigen Protokoll bei 30 h APF, 72 h APF und von 1 d erwachsenen Fliegen, haben wir zuvor gezeigt, dass das RNA-bindende Protein und der CELF1-Homolog Bruno1 (Bru1, Arrest, Aret) einen IFM-spezifischen Spleißweg nach dem Transkriptionsfaktor Spalt major (Salm)16. IFMs von Nullmutanten sowie Fliegen mit muskelspezifischen bruno1 RNAi (bru1-IR) zeigen sarcomere Wachstumsdefekte, Fehlregulation der Myosinaktivität und letztlich Hyperkontraktion und Muskelfaserverlust16,17 . Im Folgenden zeigen wir den Nutzen von sezierten IFMs für die gesamte Proteom-Massenspektrometrie und zeigen, dass einige der expressionsverändernden Veränderungen, die wir auf RNA-Ebene beobachtet haben, auch auf dem Proteinspiegel sichtbar sind. Wir heben ferner ein spezifisches Entwicklungsspleißereignis in Mhc hervor, das von Bruno1 reguliert wurde und zeigt, dass mRNA-Seq und RT-PCR aus sezierten IFMs verwendet werden können, um die Regulierung alternativer Spleißereignisse zu demonstrieren.
Je nach Bibliotheksqualität und -tiefe können mRNA-Seq-Daten auf der Ebene der Geneinheiten (mittelmittelwerte Lesezahlen über alle Exons eines Gens), einzelner Exons oder Spleißknoten analysiert werden. mRNA-Seq-Daten von bru1-IR-IFMs im Vergleich zum Wildtyp zeigen schwache Veränderungen der Expression auf derGeneinheitsebene 16 (Abbildung 5A). Bei 72 h APF gibt es bereits einen Trend zu Sarcomere-Genen wie Muskel-LIM-Protein bei 60A [Mlp60A], Actin 57B [Act57B], muskelspezifischem Protein 300 kDa [Msp300], oder Stretchin-Mlck [Strn-Mlck]), die wichtig sind, damit die richtige Muskelentwicklung bei bru1-IR-Muskel (Abbildung 5A und Zusatztabelle 1). Wir haben jedoch bereits gezeigt, dass auf der Ebene der einzelnen Exons eine viel stärkere Downregulation spezifischer Sarkomre-Genisoformen16besteht, was darauf hindeutet, dass die Hauptfunktion von Bruno1 darin besteht, alternatives Spleißen zu kontrollieren (Zusatztabelle 1 ).
Mit Der Verwendung der Vollproteom-Massenspektrometrie an sezierten IFMs können wir eine ähnliche Regulierung auf dem Proteingehalt nachweisen (Abbildung 5B und Ergänzende Tabelle 2). Von den 1.895 nachgewiesenen Peptidgruppen wurden 524 (28%) von ihnen sind in Bru1M2 mutiertes IFM bei 1 d Erwachsenen(Zusatztabelle 2) falsch reguliert. Eine Downregulation sowohl des Strn-Mlck- als auch des Mlp60A-Proteins wird ebenfalls beobachtet, da die Beobachtungen auf Transkriptsebene in unseren mRNA-Seq-Daten übereinstimmen. Trotz der begrenzten Anzahl von Datenbankpeptiden, die bestimmten Protein-Isoformen zugeordnet sind (siehe Ergänzende Methoden für Analysedetails), für Sarkome-Proteine Tropomyosin 1 (Tm1), aufrechterhalten (up/TnT), Mhc, bent (bt/projectin) und Paramyosin (Prm) Beobachten Sie die Upregulation von Peptiden aus einer Isoform und die Downregulation einer anderen (Abbildung 5B), was unsere bisherigen Beobachtungen einer ähnlichen Regulation auf der RNA-Ebene16bestätigt. Dies zeigt, dass sezierte IFMs sowohl für mRNA-Seq- als auch für Proteomik-Anwendungen nützlich sind.
Als weiteres Beispiel dafür, wie omics-Daten traditionelle Ansätze zur Verbesserung und Erweiterung biologischer Erkenntnisse ergänzen können, haben wir uns entschieden, uns auf das Spleißen am C-Terminus von Mhczu konzentrieren. Eine zuvor charakterisierte Proteinfallelinie namens weeP26 wird in das endgültige Intron von Mhc43,44 eingefügt (siehe Ergänzende Methoden für die genaue Position). weeP26 enthält einen starken Spleiß-Akzeptor und ist vermutlich in alle Mhc-Transkripte integriert (Abbildung 5C). Das GFP-markierte Protein in IFM ist jedoch in zwei "Punkte" auf beiden Seiten der M-Linie integriert, während es im Beinmuskel gleichmäßig über die M-Linie und schwach über die dicken Filamente einbaut (Abbildung 5E). Orfanos und Sparrow zeigten diese "Punkte" in IFM-Form aufgrund eines Entwicklungs-Mhc-Isoformschalters: Die vor 48 h APF exprimierte Mhc-Isoform ist GFP-gekennzeichnet als die weeP26 Exon-Einsätze im offenen Leserahmen, während der Mhc Isoform nach 48 h APF exprimiert ist unbeschriftet, da das weeP26 Exon nach dem Stop-Codon im 3'-UTR44enthalten ist.
Unsere mRNA-Seq-Daten ermöglichten es uns, die C-Terminal Mhc-Isoformexpression detaillierter zu charakterisieren. Während zwei verschiedene Mhc-Terminationen 43,44berichtet wurden, deuten unsere mRNA-Seq-Daten und die aktuelle Flybase-Anmerkung (FB2019_02) darauf hin, dass es tatsächlich drei mögliche alternative Spleißereignisse am Mhc gibt. C-terminus (Exon 34-35, 34-36 oder 34-37) (Abbildung 5C), die durch RT-PCR bestätigt wird (Abbildung 5D). weeP26 GFP wird in das Intron zwischen Exon 36 und 37 eingefügt; Da also sowohl Exon 34-35 als auch Exon 34-36 Isoformen Stoppcodons enthalten, kann GFP nur in die Exon 34-37 Isoform übersetzt werden (was zu Exon 34-GFP-37 führt). Wir konnten auch sowohl die zeitliche als auch die räumliche Regulierung aller Mhc-Isoformen sehen. In IFM beobachten wir einen Mhc-Isoformwechsel von Exon 34-37 zu Exon 34-35 zwischen 30 h APF und 48 h APF (Abbildung 5C,D,F) bei 27 °C, obwohl dies durch Immunfluoreszenz bei 48 h APF(Abbildung 5E) noch nicht sichtbar ist. Die Beine drücken bereits eine Mischung aus Exon 34-37 und Exon 34-35 bei 30 h APF aus und drücken durch 72 h APF alle drei Mhc-Isoformen aus (Abbildung 5D,F). Adult Jump Muscle (TDT) drückt auch alle drei Mhc-Isoformen aus (Abbildung 5F), was darauf hindeutet, dass dies in der Regel für röhrenförmige somatische Muskeln gilt. So ermöglichen unsere mRNA-Seq-Daten eine Erweiterung früherer Befunde, indem sie den Zeitrahmen für den Mhc-Isoformschalter in IFM verengen und den Mhc-Isoformeinsatz in röhrenförmigen Muskeln charakterisieren.
Anschließend wurde die Mhc-Isoformregulation im salm- und bru1-mutierten IFM untersucht. In beiden Fällen haben wir eine Fehlregulierung von weeP26gesehen. Salm-mutierte IFMs können den Entwicklungsschalter in Mhc-Isoformexpression und Phenokopie-Beinspleißmustern in späteren Stadien nicht abschließen, einschließlich der Verstärkung des Exon 34-36-Ereignisses (Abbildung 5F). Dies stimmt mit früheren Erkenntnissen überein, dass der Verlust von Salm zu einer nahezu vollständigen Verwandlung von IFM in den röhrenförmigen Muskel16führt. Bru1-IR und bru1 mutierteS IFM, ähnlich wie salm-/- IFM, behält das Exon 34-37 Spleißereignis durch Erwachsene(Abbildung 5E,F), was zu einem weeP26 GFP-Etikettierungsmuster führt, das dem Beinmuskel ähnelt, aber es gewinnt nicht das Exon 34-36 Ereignis. Dies deutet darauf hin, dass Bruno1 im IFM notwendig ist, um den Entwicklungsschalter in Mhc alternatives Spleißen zumindest teilweise zu steuern, aber es deutet darauf hin, dass zusätzliche Spleißfaktoren auch im Salm-/-Kontextfalsch reguliert werden. Darüber hinaus zeigt dieses Beispiel, wie RT-PCR- und mRNA-Seq-Daten aus seziertem IFM wertvoll sein können, um ein tieferes Verständnis von Entwicklungsspleißmechanismen und beobachteten morphologischen Defekten zu gewinnen.
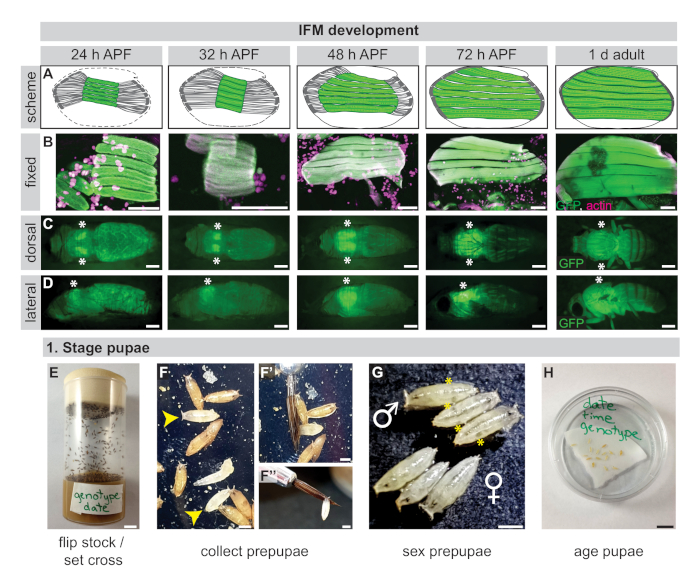
Abbildung 1:IFM-Entwicklung und Inszenierung von Pupas. (A) Schematische IFM-Entwicklung bei 24 h APF, 32 h APF, 48 h APF, 72 h APF und 1 d Erwachsene, die eine Verdichtung der Flugmuskulatur (grün) bei 32 h APF und das anschließende Faserwachstum zeigen, um den Thorax zu füllen. Die Tendons sind dunkelgrau. (B) Konfokale Bilder von festen IFMs aus offenen Buchsektionen (24 h, 32 h, 48 h)19 oder Thoraxhalbsektionen (72 h, 1 Tag), gefärbt für Actin (Rhodamine phalloidin, Magenta) und GFP (grün). (C,D) Bilder der GFP-Fluoreszenz in lebenden Pupas, die eine intakte IFM-Morphologie der Sezierfliegenlinie in der dorsalen (C) oder seitlichen (D) Ebene veranschaulichen. Sternchen markieren den IFM-Standort. (E) Zur Vorbereitung auf Sezierungen sollten Flugbestände umgedreht oder Kreuze 3 bis 4 Tage im Voraus gesetzt werden. (F) Prepupae werden durch ihre weiße Farbe (gelbe Pfeilspitzen) ausgewählt und mit einem benetzten Pinsel (F',F'') isoliert. (G) Prepupae sollten gesext werden, um Weibchen von Männern zu trennen, basierend auf dem Vorhandensein von Hoden, die als hintergründige transluzente Kugeln (gelbe Sternchen) erscheinen. (H) Pupae werden auf benetzten Filterpapier in 60 mm Geschirr gereift. Skalenbalken = 100 m (B), 1 cm (C,D,E,H), 1 mm (F,F'',G). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
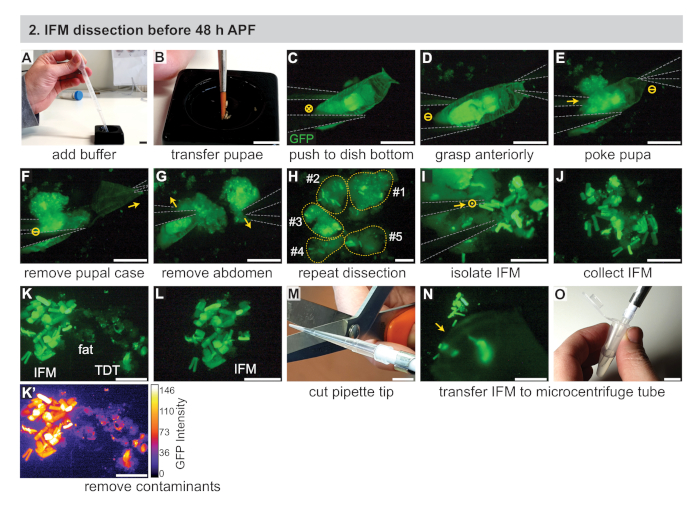
Abbildung 2:Zerlegung von IFMs vor 48 h APF. (A) Zugabe von 1x PBS-Puffer zu einer schwarzen Sezierenvonschale mit einer Transferpipette. (B) Übertragung der inszenierten Welpen mit einem Pinsel. (C) Unter einem fluoreszierenden Sezierendes Mikroskop zur Visualisierung von GFP, sanftes Schieben der Pupa auf den Boden einer Sezschale mit #5 Zangen (grau umrandet). Das "X" im Kreis bezeichnet Bewegung in das Bild. (D,E) Greifen der Pupae vorderlich (D), dann pochen der Pupae direkt hinter dem Thorax (E). Dash im Kreis bezeichnet keine Bewegung. (F,G) Ziehen mit den vorderen Zangen (Pfeil), um den Pupal-Fall (F) zu entfernen, dann Entfernung des Bauches (G). (H) Wiederholung von C-G für mehrere Welpen. Gelbe gepunktete Linien sind nummeriert, die einen Beitrag pupae. (I, J) Verwendung der Zange (I), um IFMs aus dem umgebenden Gewebe (J) zu isolieren. Punkt in einem Kreis bezeichnet die Bewegung a-blatt. (K,L) Entfernung von Verunreinigungen einschließlich Fett und Sprung (TDT) Muskeln (K) um eine saubere IFM-Probe (L) zu erzeugen. TDT hat eine niedrigere GFP-Expression und eine andere Form als IFM-Fasern (K'). (M,N,O) Verwendung einer abgeschnittenen Pipettenspitze (M) zum Sammeln sezierter IFMs (N) und deren Übertragung in ein Mikrozentrifugenrohr (O). Skalenstäbe = 1 cm (A,B,M,O), 1 mm (C-G), 500 'm (H-L,N). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
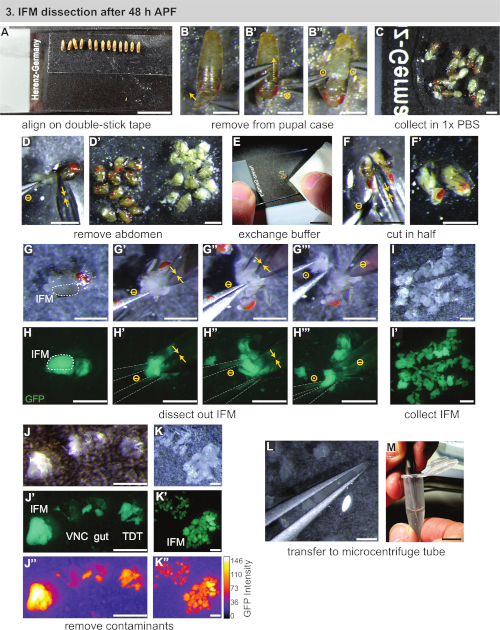
Abbildung 3:Zerlegung von IFMs nach 48 h APF. (A) Ausrichten von Pupae auf Doppelklebeband. (B) Entnahme von Pupilben aus dem Pupal-Gehäuse durch Vordereröffnung (B), Dorsalschneiden (B') und Herausheben der Pupilbe (B''). Kreissymbole, die mit Abbildung 2dargestellt werden. (C) Übertragung von Pupae in Puffer. (D) Entfernung des Bauches durch Schneiden mit Schere (gelbe Doppelpfeile) und Trennung von Thoraxen (D'). (E, F) Zugabe eines sauberen Puffers (E), dann Schneiden der Thoraxen in halblängs (F,F'). (G,H) Dissektionen können unter weißem Licht (G) oder Fluoreszenz durchgeführt werden, um das GFP (H) zu visualisieren; Schneiden der IFMs auf der einen Seite (G'), dann auf der anderen Seite (G''); Heben aus dem Thorax mit Zange (grau umrandet) (G'''). (I,J,K) Sammlung von IFMs im Puffer (I) und Entfernung von kontaminierender ventraler Nervenschnur (VNC), Darm- und Sprungmuskel (TDT) (J) zur Erzeugung einer sauberen IFM-Probe (K). TDT hat eine niedrigere GFP-Expression und eine andere Form als IFM-Fasern (J'', K'). (L,M) Verwendung von Zangen zur Übertragung von IFMs (L) in ein Mikrozentrifugenrohr (M). Skalenbalken = 1 cm (A,E,M), 1 mm (B-D', F-L). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
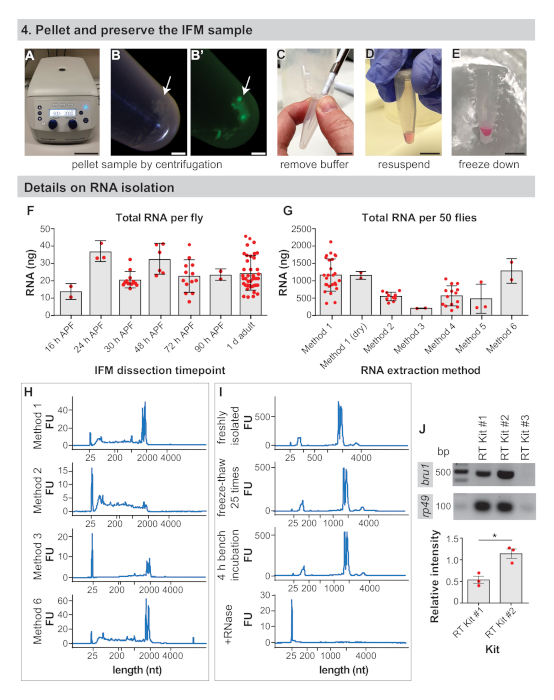
Abbildung 4:IFM-Konservierungs- und RNA-Isolationsdetails. (A) IFMs werden durch Zentrifugation für 5 min bei 2000 x gpelletiert. (B) IFM-Pellet (Pfeil) und Pellet unter Fluoreszenz (B'). (C) Entfernen des gesamten Puffers mit einer Pipettenspitze. (D) Zur RNA-Extraktion, Resuspension von Pellets im Isolationspuffer. Dieser Schritt kann übersprungen werden, um sezierte IFMs trocken zu frieren. (E) Einfrieren der Probe in flüssigem Stickstoff oder auf Trockeneis und Lagerung bei -80 °C. Skalenbalken = 10 cm (A), 1 mm (B,B'), 1 cm (C,D,E). (F) Nanogramme (ng) der gesamten RNA aus seziertem IFM, die pro Fliege bei 16 h APF, 24 h APF, 30 h APF, 48 h APF, 72 h APF, 90 h APF und 1 d Erwachsene erhalten wurden. Fehlerbalken = SD. (G) Gesamt-RNA isoliert von IFM isoliert von 50 1 d erwachsenen Fliegen mit verschiedenen Extraktionsmethoden. Fehlerbalken = SD. (H) Repräsentative Spuren zur Assay-RNA-Integrität nach verschiedenen Extraktionsmethoden. Die ribosomalen Bänder laufen knapp unter 2000 Nukleotide (nt) und das Markerband bei 25 nt. Weitere Spuren sind in Der Ergänzungsabbildung 1verfügbar. (I) Repräsentative Spuren einer frisch isolierten RNA-Probe (oben), einer 25-fach gefrierenden Probe auf Trockeneis (zweites Diagramm), einer Probe, die 4 h auf der Bank (drittes Diagramm) zurückgelassen wurde, und einer mit RNase A (bodenförmigen) Probe. Beachten Sie den vollständigen Abbau der RNA nach Zugabe von RNase A. (J) RT-PCR Gel aus Kits, wie sie für bru1 und rp49gekennzeichnet sind. Die relative Intensität des bru1-Bandes, das gegen rp49 normalisiert ist, wird unten dargestellt. Fehlerbalken = SEM (ungepaartt t-test, p = 0.0119). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
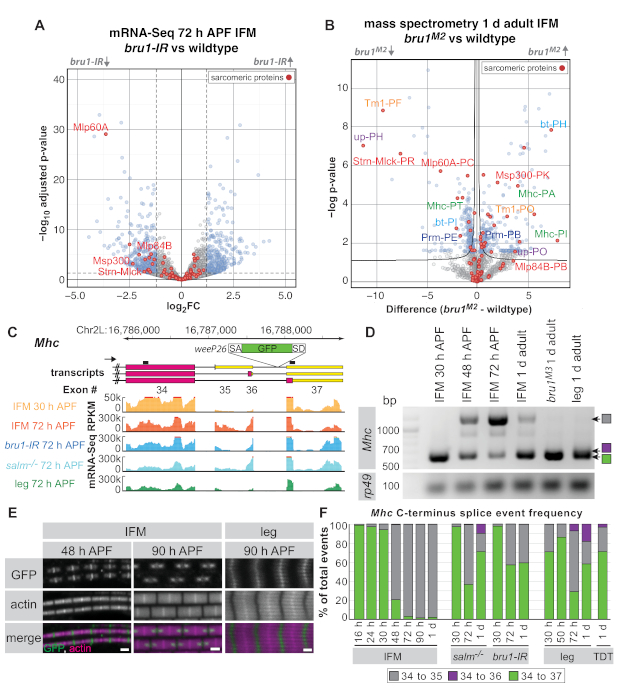
Abbildung 5:Anwendung von IFM-Sektionen zur Untersuchung der Bruno1-Funktion in alternativem Spleißen. (A) Vulkandiagramm von mRNA-Seq-Daten (Geneinheit) von IFMs, die bei 72 h APF seziert wurden. Gene, die signifikant differenziert zwischen bru1-IR und dem Wildtyp IFM (padj < 0.05, abs(log2FC) >1.5) reguliert sind, werden in blau und nicht signifikante Gene in Grau dargestellt. Sarcomere-Proteine sind rot hervorgehoben und ausgewählte Gene werden gekennzeichnet. (B) Vulkandiagramm der gesamten Proteom-Massenspektrometrie ergibt sich aus 1 d erwachsenen IFMs. Proteine, die sich signifikant zwischen bruM2-Mutantenund Wildtyp (FDR < 0,05) unterscheiden, werden in blauen, nicht signifikanten Proteinen in Grau dargestellt. Sarkomische Proteine sind rot hervorgehoben. Peptide, die Dengenen in (A) entsprechen, sind rot gekennzeichnet. Peptide, die verschiedenen Isoformen desselben Proteins zugeordnet sind, werden in derselben Farbe gekennzeichnet. (C) Schema des C-Terminus von Mhc zur Veranschaulichung von unterschiedlichen Transkriptisoformen und Einfügeposition der WeeP26-Genfalle (siehe Ergänzende Methoden für Einfügepunkt). RT-PCR-Primer werden als schwarze Linien über Transkripten bezeichnet. Lesezahlen pro Kilobasis pro Million Basen (RPKM) aus mRNA-Seq werden für IFMs angezeigt, die von Wildtyp bei 30 h APF (orange) und 72 h APF (rot), von bru1-IR (blau) und salm-/- (cyan) bei 72 h APF und vom ganzen Bein (grün) bei 72 h APF seziert werden. . (D) RT-PCR mit Primern gegen Mhc, die den Isoformschalter in IFM zwischen 30 h APF und späteren Zeitpunkten anzeigen. Das Exon 34-35 Spleißereignis wird nur schwach bei bruM3mutiertem IFM oder im Erwachsenenbein beobachtet. (E) Konfokale Bilder der weeP26 GFP Lokalisation in Wildtyp IFM Sarkomen bei 48 h APF und 90 h APF im Vergleich zu 90 h APF Beinmuskel. Skalenbalken = 1 m . (F) Spleißknotenquantififizierung aus mRNA-Seq-Daten für Genotypen und Zeitpunkte, wie sie beschriftet sind. Junction-Lesevorgänge werden als Verhältnis eines bestimmten Spleißereignisses (Exon 34 bis 35 in grau, 34 bis 36 in Violett und 34 bis 37 in Grün) zu den Gesamtzahlsereignissen dargestellt, die den exon 34 Spleißspender teilen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
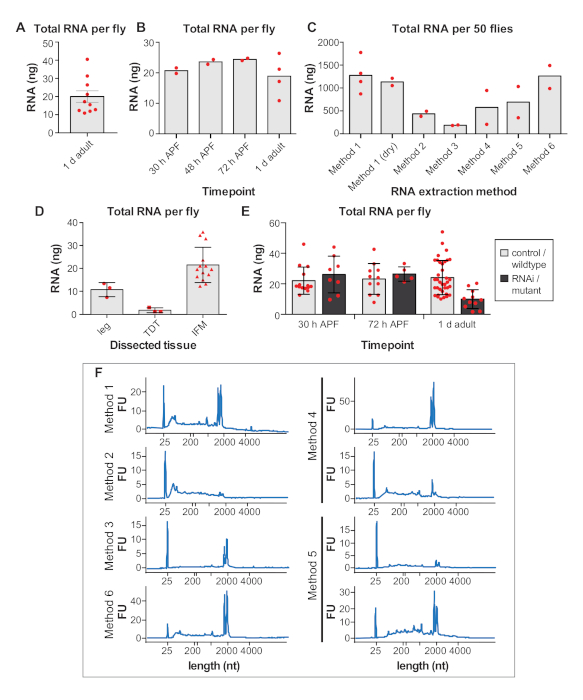
Ergänzende Abbildung 1: (A,B,C) RNA liefert aus Proben desselben Genotyps, die vom gleichen Forscher in derselben Woche seziert wurden. Nachdem alle Proben seziert wurden, wurde die RNA isoliert und am selben Tag gemessen. (A) Nanogramme (ng) der gesamten RNA, die aus IFM-Sektionen pro 1 d erwachsenen Flug gewonnen wird. Fehlerbalken = SEM. (B) Gesamt-RNA aus seziertem IFM pro Fliege bei 30 h APF, 48 h APF, 72 h APF und 1 d Erwachsene. (C) Gesamt-RNA isoliert aus IFM seziert von 50 1 d erwachsenen Fliegen mit verschiedenen Extraktionsmethoden. (D) Die Gesamt-RNA-Konzentrationen pro Fliege von sezierten Beinen, Sprungmuskel (TDT) und IFM. Mehr RNA wird aus den größeren IFMs gewonnen. Fehlerbalken = SD. (E) Die gesamten RNA-Konzentrationen pro Fliege von IFM, die von Kontrollen seziert wurden, verglichen mit RNAi- oder Mutantenproben bei 30 h APF, 72 h APF und 1 d Erwachsenen. Für Mutanten wurde w1118 als Wildtypkontrolle verwendet. Mutante Daten werden aus bru1-IR, salm-/- und einem weiteren RNA-bindenden Proteinmutanten zusammengestellt. Beachten Sie, dass für diese Manipulationen die RNA-Ausbeute in 1 d Erwachsenen aufgrund von Muskelatrophie und Verlust verringert wird, so dass mehr Fliegen seziert werden müssen, um ausreichende Mengen für Omics-Ansätze zu erhalten. Fehlerbalken = SD. (F) Zusätzliche Spuren, die die RNA-Integrität für die in Abbildung 4G und in Der Ergänzungsabbildung 1Cdargestellten RNA-Isolationsmethoden zeigen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Ergänzende Methoden: Eine detaillierte Beschreibung der im gesamten Text verwendeten Methoden und Reagenzien und insbesondere zur Erzeugung der in Abbildung 1A-Ddargestellten Daten, Abbildung 4F-K, Abbildung 5, Ergänzende Tabelle 1und Ergänzende Tabelle 2. Diese Daten motivieren das Sezierprotokoll und demonstrieren seine Nützlichkeit für RNA-Isolation, mRNA-Seq, RT-PCR und Proteomik. Bitte klicken Sie hier, um diese Datei herunterzuladen.
| Im Zusammenhang mit Abbildung 5 und den zugehörigen Absätzen im Haupttext | |
| Tab-Name | Datenzusammenfassung |
| Sarcomere Proteine | Liste von Sarcomere-Genen von Spletter et al. Elife 2018; Hier listen wir den aktuellen FBgn und Gennamen auf. |
| SP-Geneinheiten_DESeq2_72h | Anhand von Daten von Spletter et al. EMBO Rep 2015 haben wir uns speziell die Sarcomere-Gene in den mRNA-Seq-Daten bei 72 h APF angesehen. Dies ist aus der DESeq2-Analyse, die Differentialexpression auf der Geneinheitsebene zwischen der Kontrolle (Mef2-Gal4, UAS-GFM-Gma gekreuzt auf w1118) und Mef2-Gal4, UAS-GFM-Gma x Bruno1-IR erkennt. In gelb hervorgehobene Zeilen sind deutlich nach oben oder unten regulierte Gene (über/unter einem Schwellenwert von log2FC=abs(1.5)). Diese Daten sind die rote Punktüberlagerung in Abbildung 5A. Für jedes Sarcomere-Gen stellen wir Identifikatoren, den log2FC aus DESeq2, den P-Wert und den angepassten P-Wert sowie die normalisierte Expression simt 2 sowie die DeSeq2-Normalexpressionsanzahl zur Verfügung. |
| SP exon_DEXSeq_72h | Anhand von Daten aus Spletter et al. EMBO Rep 2015 untersuchten wir speziell die Verwendung von Sarcomere-Genexon in den mRNA-Seq-Daten bei 72 h APF. Dies ist von der DEXSeq-Analyse zur Erkennung differential exon Verwendung zwischen Steuerung (Mef2-Gal4, UAS-GFM-Gma gekreuzt zu w1118) und Mef2-Gal4, UAS-GFM-Gma x Bruno1-IR. In gelb hervorgehobene Zeilen sind deutlich nach oben oder unten regulierte Exons (über/unter einem Schwellenwert von log2FC=abs(1.5)). Wir liefern Exon- und Gen-Id-Informationen, den log2FC aus DEXSeq, den P-Wert und den angepassten P-Wert sowie eine Liste der zugehörigen Transkripte. |
| Bitte beachten Sie, dass viele Gene die Regulierung eines oder mehrerer Exons in der DEXSeq-Analyse zeigen, oft mit hohen log2FC-Werten und niedrigen P-Werten/Anpassungswerten, während eine begrenzte Liste von Genen Veränderungen bei 72 h APF anzeigt. Dies unterstützt einen starken Einfluss des Verlustes von Bruno auf die Regulierung der alternativen Spleißen. | |
Ergänzende Tabelle 1: Tabelle mit 72 h APF mRNA-Seq-Daten für Sarkome-Proteine, die differenziell exprimierte Gene (über DESeq2) und Exons (über DEXSeq) in bru1-IR vs.wildtype IFMs identifizieren.
| Im Zusammenhang mit Abbildung 5B und den zugehörigen Absätzen im Haupttext | |
| Tab-Name | Datenzusammenfassung |
| Perseus-Ausgabe | Hierbei handelt es sich um eine verarbeitete Datentabelle, in der die Massenspektrometriedaten zur Generierung von Abbildung 5B dargestellt werden. IFM-Proben stammen aus 1 d Erwachsenenkontrolle (w1118) und mutierten (bruno1-M2) Fliegen. Wichtige Spalten sind die transformierten Intensitätswerte für jede der 4 Replikationen für jede Probe, die t-Test-Statistik und Signifikanz, Peptid-IDs und entsprechende Gennamen und Flybase-IDs. Die Anzeige wurde mit Standardeinstellungen in Perseus (FDR<.05) berechnet. Es wurden 1859 Proteine/Peptide nachgewiesen, davon 524 (28%) unterscheiden sich zwischen den Proben erheblich. |
| Downregulated | Dies sind ALLE 252 Proteine/Peptide aus dem Perseus-Ausgang, die in bruno1-M2-Mutant IFM nach unten reguliert werden. Da die Flybase-IDs und Gennamen veraltet sind, geben wir zusätzlich die aktuelle Flybase-Gen-ID und den Gennamen an. |
| Upregulated | Dies sind ALLE 272 Proteine/Peptide aus dem Perseus-Ausgang, die in bruno1-M2-Mutant IFM hochreguliert sind. Da die Flybase-IDs und Gennamen veraltet sind, geben wir zusätzlich die aktuelle Flybase-Gen-ID und den Gennamen an. |
| Bitte beachten Sie, dass die in Abbildung 5B rot hervorgehobenen Sarcomere-Proteine in den obigen Listen enthalten sind. Die Liste der Gene, die als Teil des Sarcomeres betrachtet werden, ist in einer der Registerkarten in Der Ergänzenden Tabelle 1 verfügbar. | |
Ergänzende Tabelle 2: Tabelle der gesamten Proteom-Massenspektrometriedaten von 1 d Erwachsenen, die differenziell exprimierte Proteine und Proteinisoformen in bruM2mutant vsidentifizieren. Wildtyp IFMs.
Diskussion
In diesem Protokoll stellen wir die grundlegende Technik zur Sezieren von Drosophila-IFMs aus früh- und spätstadiumischen Pupas zur nachgelagerten Isolierung von Protein, DNA, RNA oder anderen Makromolekülen vor. Das Protokoll kann leicht angepasst werden, um IFM von erwachsenen Fliegen zu sezieren. Wir demonstrieren den Nutzen unseres Sezierprotokolls für mRNA-Seq-, Proteomik- und RT-PCR-Anwendungen. Mit der kontinuierlichen Verbesserung der omics-Technologien, um die Analyse von Proben mit weniger Ausgangsmaterial und niedrigeren Eingangskonzentrationen zu ermöglichen, werden diese Sezierungen wahrscheinlich für viele zusätzliche Anwendungen wertvoll werden. Da IFMs ein etabliertes Modell für humane Myopathien4,24 und muskelspezifische entwicklung9,12, wir sehen, zum Beispiel, IFM-angereicherte Metabolomik, Untersuchungen der Chromatin-Konformation über 3C oder 4C, Spleißnetzauswertung über CLiP-Wechselwirkungen oder Phospho-Proteomik der Myofibrillogenese.
Es ist wichtig zu berücksichtigen, dass diese Sezierungen eine Probe produzieren, die für IFM angereichert ist, anstatt eine reine IFM-Probe. Dies ist aufgrund der motorischen Neuroneninnervation, Sehnenanhaftungen und trachealen Invasion von Muskelfasern unvermeidbar. Die Bioinformatik-Analyse kann verwendet werden, um IFM-angereicherte Gene oder Proteine zu identifizieren, aber weitere Experimente sind erforderlich, um zu zeigen, dass sie tatsächlich IFM-spezifisch sind. Die Probenreinheit kann mit veröffentlichten gewebespezifischen Markern wie Stripe45 (Tendon), Act79B4,44 (Tubularmuskel), Act88F15 (IFM) oder syb46 (neuronal spezifisch) untersucht werden. Es kann möglich sein, solche Marker zu verwenden, um Datensätze auf den IFM-spezifischen Inhalt zu normalisieren, aber Benutzer werden darauf hingewiesen, dass zeitliche Veränderungen in der Expression von Genen, die für die Normalisierung verwendet werden, zum Beispiel von IFM-spezifischen Genen oder Tubulin, einen solchen Ansatz beeinflussen können.
Genetisch kodierte gewebespezifische Etikettierungsmethoden, z. B. EC-Tagging47,48 oder PABP-Kennzeichnung49,50 zur Isolierung von RNA wurden in den letzten Jahren entwickelt, was dazu beitragen kann, eine gewebespezifische RNA-Probe. EC-Tagging erfordert jedoch eine konstante Fütterung der Fliegen47 und ist daher in den Pupalstadien nicht anwendbar. Die Empfindlichkeit und Vollständigkeit von PABP-markierten Transkriptomen kann Einschränkungenhaben 51. FACS-Ansätze zur Isolierung einzelner Muskelfasern werden durch die große Größe und Synzytialität von IFMs erschwert. INTACT52,53 Stil Ansätze können angewendet werden, um bestimmte subzelluläre Kompartimente von IFMs zu isolieren, die sich als nützlich für die Isolierung reinen Populationen von IFM-Kernen oder Mitochondrien erweisen können. Manuelle Sezierungen sind nach wie vor der aktuelle Standard, um intaktes IFM-Gewebe für die meisten nachgelagerten Anwendungen zu erhalten.
Die Probenqualität hängt von mehreren kritischen Schritten im Sezierprozess ab. Die Sezierungen sind technisch anspruchsvoll, wobei die Seziergeschwindigkeit und die Probenreinheit mit der Erfahrung zunehmen. Das Sezieren für kurze Zeit (20–30 min) im gekühlten Puffer ohne Waschmittel und das sofortige Einfrieren trägt zur Erhaltung der Probenintegrität bei, wie zuvor bei der Isolierung der Maussehne beobachtet wurde54. IFMs können erfolgreich trockengefroren werden, nachdem sie alle Puffer aus dem Pellet entfernt haben, aber speziell für die RNA-Isolierung führt das Einfrieren von Proben im Isolationspuffer tendenziell zu besseren Ergebnissen. IFMs aus bis zu 20 separaten Sezieren werden vor der RNA- oder Proteinisolation kombiniert, so dass auch von frühen Zeitpunkten oder Mutanten16,32, für die nachgelagerte Analyse genügend Material skaliert und gesammelt werden kann.
Für RNA-Anwendungen kann der kritischste Schritt die Isolierung der RNA selbst sein. Guanidinium thiocyanat-phenol-chloroform isolation (Methode 1 oben) übertrifft die meisten kommerziellen Kits getestet und, wie bereits erwähnt, ist deutlich billiger55. Die variabilitätsbeobachtete RNA-Isolationsausbeute mit kommerziellen Kits stimmt mit früheren Beobachtungen56,57überein. Wir fügen auch Glykogen während der Isopropanol-Ausfällung hinzu, um die gesamte RNA wiederherzustellen. Über die RNA-Ausbeute hinaus ist es wichtig, die RNA-Integrität zu überprüfen, um sicherzustellen, dass die Probe während der Sezieren- und Isolationsprozesse nicht fragmentiert oder abgebaut wurde. Es ist auch wichtig, RNase-frei zu arbeiten. Schließlich kann die Wahl des RT-Kits die Empfindlichkeit des Reverse-Transkriptionsprozesses beeinflussen. Obwohl nicht oft ausführlich diskutiert, beeinflussen all diese Punkte die Qualität der IFM-Probe und die Daten aus nachgelagerten Anwendungen.
Mehrere wichtige Änderungen unterscheiden das Protokoll von bestehenden IFM-Dissektionsprotokollen. Obwohl ein detailliertes Dissektionsprotokoll für IFM-Immunfluoreszenz19existiert, stellt dieses Protokoll einen anderen Ansatz für Pupal-Sektionen dar, der eine schnellere Isolierung von IFM-Gewebe ermöglicht. Dies ermöglicht die Sammlung großer Mengen von IFM-Gewebe (relativ gesprochen) mit begrenzten Sezierenvon Zeiten, um Proteom- oder Transkriptomänderungen zu verhindern. Andere Protokolle beschreiben die Zerlegung des erwachsenen IFM zur Visualisierung der GFP-Färbung in einzelnen Myofibrils39 oder zur Färbung von Larvenkörper-Wand-Muskeln58, aber sie sprechen nicht die Sezieren in Pupalstadien oder zur Isolierung von RNA oder Protein an. Dieser Ansatz unterscheidet sich auch vom bestehenden Protokoll für die Mikrodissektion von Pupal-IFMs aus kryosections38, das eine reinere IFM-Probe erzeugen kann, aber arbeitsintensiver ist und weniger Material produziert. Im Vergleich zu anderen schnellen erwachsenen IFM-Sektionsprotokollen38,39werden IFMs im PBS-Puffer ohne Reinigungsmittel isoliert, um die Spannungsinduktion und andere wichtige Expressionsänderungen zu begrenzen.
Der wichtigste Fortschritt in diesem Protokoll ist die Aufnahme eines lebenden, fluoreszierenden Reporters, der die Isolierung der IFMs in frühen Pupalstadien ermöglicht. Wir verwenden standardmäßig Mef2-GAL459 fahren entweder UAS-CD8::GFP oder UAS-GFP::Gma60. Dies ermöglicht eine differenzielle Kennzeichnung von IFM (Flugmuskeln sind stärker gekennzeichnet und anders geformt als andere Pupalmuskeln) sowie die Durchführung von GAL4-UAS-basierten Manipulationen, z. B. Rettungs- oder RNAi-Experimenten. Es ist auch möglich, Mef2-GAL4 mit Wanne-GAL80ts zu kombinieren, um RNAi-assoziierte frühe Letalität zu vermeiden, oder mit UAS-Dcr2, um die RNAi-Effizienz zu erhöhen40.
Es stehen zusätzliche GAL4-Treiber oder GFP-Linien zur Verfügung, die in Muskeltyp-Spezifität, zeitlichem Ausdrucksmuster und Treiberstärke19,61 variieren, die anstelle von Mef2-GAL4 verwendet werden können. Beispielsweise wird Act88F-GAL4 zuerst um 24 h APF ausgedrückt, sodass es nicht für frühere Zeitpunkte verwendet werden kann; Es wird jedoch stark als IFM bezeichnet und kann nützlich sein, um RNAi-assoziierte frühe Letalität zu vermeiden. Him-GFP oder Act88F-GFP Label IFM, wieder mit zeitlichen Einschränkungen, aber sie vermeiden GAL4 Abhängigkeit von Marker-Expression und kann in Kombination mit einem mutierten Hintergrund von Interesse nützlich sein. Listen anderer möglicher Markerlinien sind verfügbar19. Es sollte auch beachtet werden, dass die Verwendung von Transgenen und dem GAL4/UAS-System Genexpressionsartefakte verursachen kann, daher ist es wichtig, geeignete Kontrollen zu verwenden, z. B. die Treiberlinie, die zum Wildtyp-Hintergrundstamm gekreuzt wird, so dass solche Artefakte vermutlich in allen Proben gleich.
Mit dem begleitenden Video soll dieses detaillierte Protokoll die pupalIFM-Sektion zugänglicher machen und den Einsatz von Omics-Ansätzen zur Erforschung der Muskelentwicklung fördern. Die Kopplung der Kraft der Drosophila-Genetik und Der Zellbiologie mit den Biochemie- und Omics-Assays, die durch seziertes IFM zugänglich sind, hat das Potenzial, das mechanistische Verständnis von Myogenese und Muskelfunktion zu fördern. Zukünftige Studien, die Beobachtungen von Transkriptom und Proteomregulation auf Systemebene mit metabolischen und funktionellen Ausgängen verknüpfen, werden ein tieferes Verständnis der muskelspezifischen Entwicklung und der Pathogenese von Muskelerkrankungen liefern.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Wir danken Andreas Ladurner und Frank Schnorrer für die großzügige Unterstützung. Wir danken Sandra Esser für die hervorragende technische Unterstützung und Akanksha Roy für die Generierung der Massenspektrometriedaten. Wir erkennen die Bloomington und Vienna Stock Center für die Bereitstellung von Fliegen an. Wir danken der Core Facility Bioimaging für die Hilfe bei der konfokalen Bildgebung und dem Zentrallabor für Proteinanalytik für die Analyse von Massenspektrometrieproben, beide im LMU Biomedical Center (Martinsried, DE). Unterstützt wurden unsere Arbeiten von der Deutschen Forschungs Gemeinschaft (MLS, SP 1662/3-1), dem Center for Integrated Protein Science Munich (CIPSM) an der Ludwig-Maximilians-Universität München (MLS), der Frederich-Bauer Stiftung (MLS) und dem International Max Planck Research School (EN).
Materialien
| Name | Company | Catalog Number | Comments |
| 5x High Fidelity (HF) buffer | Thermo Fisher | F518L | |
| 60 mm culture dishes | Sigma-Aldrich | Z643084-600EA | Greiner dishes, 60 mm x 15 mM, vented |
| Black dissecting dish (glass) | Augusta Laborbedarf | 42021010 | Lymphbecken, black glass, 4 cm x 4 cm |
| Black silicon dissecting dishes: activated charcoal powder | Sigma-Aldrich | C9157 | Also available from most pharmacies |
| Black silicon dissecting dishes: Sylgard 184 | Sigma-Aldrich | 761036 | Dishes are made by mixing the Sylgard (~50g) with activated charcoal powder (200 mg) and curing it in Petri dishes (~4 60 mm dishes). |
| Blue pestle | Sigma-Aldrich | Z359947-100EA | Any pellet pestle that can sterilized, also can be used with a motor-driven grinder |
| Cell phone camera, Samsung Galaxy S9 | Samsung | SM-G960F/DS | used for photos not taken under a microscope |
| Chloroform | PanReac AppliChem | A3691,0500 | |
| Confocal microscope, Leica SP8X upright confocal | Leica | www.leica-microsystems.com | |
| Confocal microscope, Zeiss LSM 780 inverted confocal | Zeiss | www.zeiss.com | |
| Double stick tape | Scotch/3M | 3M ID 70005108587 | Double-sided tape, available at most office supply handlers |
| Dumont #5 Forceps | Fine Science Tools | 11252-20 | Inox straight tip 11 cm forceps, Biology grade with 0.05 mm x 0.02 mm tip |
| EtOH (100%, RNase free) | Sigma-Aldrich | 32205-M | |
| fluorescent dissecting microscope camera, Leica DFC310 FX camera | Leica | www.leica-microsystems.com | |
| Fluorescent dissecting microscope software, Leica Application Suite (LAS) version 4.0.0 | Leica | www.leica-microsystems.com | |
| Fluorescent dissecting microscope, Leica M165 FC | Leica | www.leica-microsystems.com | |
| Fly: Bru1[M2] | Fly stock; This paper | ||
| Fly: Bru1[M3] | Fly stock; This paper | ||
| Fly: Mef2-GAL4 | Bloomington Stock Center | BDSC:27390 | Fly stock |
| Fly: salm[1] | Bloomington Stock Center | 3274 | Fly stock |
| Fly: salm[FRT] | Fly stock; see Spletter et al., Elife, 2018 | ||
| Fly: UAS-Bru1IR | Vienna Drosophila Research Center | GD41568 | Fly stock, RNAi hairpin |
| Fly: UAS-GFP::Gma | Bloomington Stock Center | BDSC:31776 | Fly stock |
| Fly: UAS-mCD8a::GFP | Bloomington Stock Center | BDSC:5130 | Fly stock |
| Fly: w[1118] | Bloomington Stock Center | 3605 | Fly stock |
| Fly: weeP26 | Fly stock; see Clyne et al., Genetics, 2003 | ||
| GFP detection reagent, GFP-Booster | ChromoTek | gba488-100 | |
| Glycogen | Invitrogen | 10814-010 | |
| Image processing software, Photoshop CS6 | Adobe | www.adobe.com | |
| Isopropanol | Sigma-Aldrich | I9516-25ML | 2-propanol |
| Method 1 (RNA isolation): TRIzol | Life Technologies | 15596018 | Guanidinium isothiocyanate and phenol monophasic solution |
| Method 2 (RNA isolation): Method 1 + TURBO DNA-free Kit | Invitrogen | AM1907 | TRIzol isolation followed by treatment with a kit to remove DNA |
| Method 3 (RNA isolation): Direct-zol RNA Miniprep Plus Kit | Zymo Research | R2070S | RNA isolation in TRIzol, but over commercial columns instead of using phase separation. Recommended DNase treatment performed with Monarch Dnase I in Monarch DNase I Reaction buffer. |
| Method 4 (RNA isolation): RNeasy Plus Mini Kit | Qiagen | 74134 | We used the provided DNase treatment. IFM pellets were homogenized in RTL buffer as suggested for animal tissues. |
| Method 5 (RNA isolation): ReliaPrep RNA Tissue Miniprep System | Promega | Z6110 | We applied the protocol for ‘Purification of RNA from Fibrous Tissues’. |
| Method 6 (RNA isolation): Monarch Total RNA Miniprep Kit | New England Biolabs | T2010G | We applied the protocol for tissues/leukocytes and lysed in 300 μL of RNA Protection Reagent. |
| Microcentrifuge tubes | Thermo Fisher | AM12400 | RNase-free Microfuge Tubes, 1.5 mL |
| Microscope slides | Thermo Fisher | 12342108 | Standard slides, uncharged, 1 mm |
| Microtome blades | PFM Medical | 207500003 | C35 feather 80 mm |
| Monarch DNase I | New England Biolabs | T2004-21 | |
| Monarch DNase I Reaction Buffer | New England Biolabs | T2005-21 | |
| Normal goat serum | Thermo Fisher | 16210072 | |
| OneTaq Polymerase | New England Biolabs | M0480X | |
| Paintbrush | Marabu | 1910000000 | Marabu Fino Round No. 0, or similar brush from any art supply |
| Paraformaldehyde | Sigma-Aldrich | 158127 | |
| PBS buffer (1x) | Sigma-Aldrich | P4417 | Phosphate buffered saline tablets for 1 L solutions, pH 7.4 |
| PFA PureTip Pipette Tips | Elemental Scientific | ES-7000-0101 | Optional substitute for standard pipette tips to reduce sample loss; 100 mL, 0.8 mm orifice |
| Phusion High Fidelity Polymerase | Thermo Fisher | F-530XL | |
| Pipette tips | Sigma-Aldrich | P5161 | Universal 200 mL pipette tips |
| Preomics iST 8x Kit | Preomics | P.O.00001 | peptide preparation kit for mass spectrometry |
| Q Exactive mass spectrometer | Thermo Fisher | 725500 | mass spectrometry was performed at the Protein Analysis Unit of the LMU Biomedical Center |
| Qubit RNA Assay Kit | Life Technologies | Q32855 | |
| Rhodamine-phalloidin | Invitrogen, Molecular Probes | 10063052 | |
| RNA concentration Approach 1 & RNA integrity traces, Bioanalyzer | Agilent Technologies | G2939BA | |
| RNA concentration Approach 2, Nanodrop | Thermo Fisher | ND-2000 | |
| RNA concentration Approach 3, Qubit 4 Fluorometer | Invitrogen | Q33238 | |
| RNA Pico Chips | Agilent Technologies | 5067-1513 | |
| RNase A | Promega | A7937 | |
| RNase-free water, Diethyl pyrocarbonate (DEPC) | Sigma-Aldrich | D5758 | DEPC treat water overnight and then autoclave, to remove all RNase. |
| RT Kit #1: Super Script III Reverse Transcriptase Kit | Invitrogen | 18080-044 | reverse transcription kit |
| RT Kit #2: LunaScript | New England Biolabs | E3010S | reverse transcription kit |
| RT Kit #3: QuantiNova Reverse Transcription Kit | Qiagen | 205410 | reverse transcription kit |
| Slide mounting buffer, Vectashield | Vector Laboratories | H-1200 | containing DAPI |
| Statistical software: GraphPad Prism | GraphPad Prism | www.graphpad.com | |
| Statistical software: Microscoft Excel | Microsoft | Purchased as part of the bundle: Office Home & Student 2019 | |
| Table-top centrifuge | Eppendorf | 5405000760 | Eppendorf Centrifuge 5425 or equivalent |
| Tissue/ Kimwipes | Sigma-Aldrich | Z188956 | Standard tissue wipes |
| Transfer pipette | Sigma-Aldrich | Z350796 | Plastic pipette |
| Triton-X100 | Sigma-Aldrich | T9284-500ML | |
| Vannas spring scissors | Fine Science Tools | 15000-00 | 3 mm cutting edge, tip diameter 0.05 mm, length 8 cm |
| Whatman paper | Sigma-Aldrich | 1004-070 | Filter paper circles, Grade 4, 70 mm |
Referenzen
- Rexiati, M., Sun, M., Guo, W. Muscle-Specific Mis-Splicing and Heart Disease Exemplified by RBM20. Genes. 9 (1), 18 (2018).
- Guo, W., et al. RBM20, a gene for hereditary cardiomyopathy, regulates titin splicing. Nature Medicine. 18 (5), 766-773 (2012).
- Guo, W., et al. Splicing Factor RBM20 Regulates Transcriptional Network of Titin Associated and Calcium Handling Genes in The Heart. International Journal of Biological Sciences. 14 (4), 369-380 (2018).
- Nikonova, E., Kao, S. -. Y., Ravichandran, K., Wittner, A., Spletter, M. L. Conserved functions of RNA-binding proteins in muscle. The International Journal of Biochemistry & Cell Biology. 110, 29-49 (2019).
- Wang, E. T., et al. Dysregulation of mRNA Localization and Translation in Genetic Disease. The Journal of Neuroscience. 36 (45), 11418-11426 (2016).
- Wang, E. T., et al. Antagonistic regulation of mRNA expression and splicing by CELF and MBNL proteins. Genome Research. 25 (6), 858-871 (2015).
- Kalsotra, A., et al. A postnatal switch of CELF and MBNL proteins reprograms alternative splicing in the developing heart. Proceedings of the National Academy of Sciences of the United States of America. 105 (51), 20333-20338 (2008).
- Ho, T. H., et al. Muscleblind proteins regulate alternative splicing. The EMBO Journal. 23 (15), 3103-3112 (2004).
- Lemke, S. B., Schnorrer, F. Mechanical forces during muscle development. Mechanisms of Development. 144, 92-101 (2017).
- Iwamoto, H. Structure, function and evolution of insect flight muscle. Biophysics. 7, 21-28 (2011).
- Schnorrer, F., Dickson, B. J. Muscle building; mechanisms of myotube guidance and attachment site selection. Developmental Cell. 7 (1), 9-20 (2004).
- Spletter, M. L., Schnorrer, F. Transcriptional regulation and alternative splicing cooperate in muscle fiber-type specification in flies and mammals. Experimental Cell Research. 321 (1), 90-98 (2014).
- Benoist, P., Mas, J. A., Marco, R., Cervera, M. Differential muscle-type expression of the Drosophila troponin T gene. A 3-base pair microexon is involved in visceral and adult hypodermic muscle specification. Journal of Biological Chemistry. 273 (13), 7538-7546 (1998).
- Schönbauer, C., et al. Spalt mediates an evolutionarily conserved switch to fibrillar muscle fate in insects. Nature. 479 (7373), 406-409 (2011).
- Bryantsev, A. L., et al. Extradenticle and Homothorax Control Adult Muscle Fiber Identity in Drosophila. Developmental Cell. 23 (3), 664-673 (2012).
- Spletter, M. L., et al. The RNA-binding protein Arrest (Bruno) regulates alternative splicing to enable myofibril maturation in Drosophila flight muscle. EMBO Reports. 16 (2), 178-191 (2015).
- Oas, S. T., Bryantsev, A. L., Cripps, R. M. Arrest is a regulator of fiber-specific alternative splicing in the indirect flight muscles of Drosophila. The Journal of Cell Biology. 206 (7), 895-908 (2014).
- Kim, J. H., Jin, P., Duan, R., Chen, E. H. ScienceDirect Mechanisms of myoblast fusion during muscle development. Current Opinion in Genetics & Development. 32, 162-170 (2015).
- Weitkunat, M., Schnorrer, F. A guide to study Drosophila muscle biology. Methods. 68 (1), 2-14 (2014).
- Rai, M., Nongthomba, U., Grounds, M. D. Skeletal Muscle Degeneration and Regeneration in Mice and Flies. Mechanisms of Regeneration. 108, 247-281 (2014).
- Swank, D. M., Wells, L., Kronert, W. A., Morrill, G. E., Bernstein, S. I. Determining structure/function relationships for sarcomeric myosin heavy chain by genetic and transgenic manipulation of Drosophila. Microscopy Research and Technique. 50 (6), 430-442 (2000).
- de Joussineau, C., Bataillé, L., Jagla, T., Jagla, K. Diversification of muscle types in Drosophila: upstream and downstream of identity genes. Current Topics in Developmental Biology. 98, 277-301 (2012).
- Maqbool, T., Jagla, K. Genetic control of muscle development: learning from Drosophila. Journal of Muscle Research and Cell Motility. 28 (7-8), 397-407 (2008).
- Jagla, K., Kalman, B., Boudou, T., Hénon, S., Batonnet-Pichon, S. Beyond mice: Emerging and transdisciplinary models for the study of early-onset myopathies. Seminars in Cell & Developmental Biology. 64, 171-180 (2017).
- Haigh, S. E., et al. Drosophila indirect flight muscle specific Act88F actin mutants as a model system for studying congenital myopathies of the human ACTA1 skeletal muscle actin gene. Neuromuscular Disorders. 20 (6), 363-374 (2010).
- Batonnet-Pichon, S., et al. Myofibrillar Myopathies: New Perspectives from Animal Models to Potential Therapeutic Approaches. Journal of Neuromuscular Diseases. 4 (1), 1-15 (2017).
- Kreipke, R. E., Kwon, Y. V., Shcherbata, H. R., Ruohola-Baker, H. Drosophila melanogaster as a Model of Muscle Degeneration Disorders. Current Topics in Developmental Biology. 121, 83-109 (2017).
- Souidi, A., Zmojdzian, M., Jagla, K. Dissecting Pathogenetic Mechanisms and Therapeutic Strategies in Drosophila Models of Myotonic Dystrophy Type 1. International Journal of Molecular Sciences. 19 (12), 4104 (2018).
- Sparrow, J., Hughes, S. M., Segalat, L. Other Model Organisms for Sarcomeric Muscle Diseases. Advances in Experimental Medicine and Biology. 642, 192-206 (2008).
- Lloyd, T. E., Taylor, J. P. Flightless flies: Drosophila models of neuromuscular disease. Annals of the New York Academy of Sciences. 1184, 1-20 (2010).
- Swank, D. M. Mechanical analysis of Drosophila indirect flight and jump muscles. Methods. 56 (1), 69-77 (2012).
- Spletter, M. L., et al. A transcriptomics resource reveals a transcriptional transition during ordered sarcomere morphogenesis in flight muscle. eLife. 7, 1361 (2018).
- Weitkunat, M., Kaya-Çopur, A., Grill, S. W., Schnorrer, F. Tension and force-resistant attachment are essential for myofibrillogenesis in Drosophila flight muscle. Current Biology. 24 (7), 705-716 (2014).
- Gunage, R. D., Dhanyasi, N., Reichert, H., VijayRaghavan, K. Drosophila adult muscle development and regeneration. Seminars in Cell & Developmental Biology. 72, 56-66 (2017).
- Weitkunat, M., Brasse, M., Bausch, A. R., Schnorrer, F. Mechanical tension and spontaneous muscle twitching precede the formation of cross-striated muscle in vivo. Development. 144 (7), 1261-1272 (2017).
- Zappia, M. P., Rogers, A., Islam, A. B. M. M. K., Frolov, M. V. Rbf Activates the Myogenic Transcriptional Program to Promote Skeletal Muscle Differentiation. Cell Reports. 26 (3), 702-719 (2019).
- Zappia, M. P., Frolov, M. V. E2F function in muscle growth is necessary and sufficient for viability in Drosophila. Nature Communications. 7 (1), 10509 (2016).
- Bryantsev, A. L., et al. Myogenesis in Drosophila melanogaster: Dissection of Distinct Muscle Types for Molecular Analysis. Methods in Molecular Biology. 1889 (5), 267-281 (2019).
- Xiao, Y. S., Schöck, F., González-Morales, N. Rapid IFM Dissection for Visualizing Fluorescently Tagged Sarcomeric Proteins. Bio-Protocol. 7 (22), (2017).
- Kaya-Çopur, A., Schnorrer, F. RNA Interference Screening for Genes Regulating Drosophila Muscle Morphogenesis. Myogenesis. 1889, 331-348 (2019).
- Chechenova, M. B., et al. Functional redundancy and non-redundancy between two Troponin C isoforms in Drosophila adult muscles. Molecular Biology of the Cell. 28 (6), 760-770 (2017).
- Alberts, B. . Molecular Biology of the Cell. , (2017).
- Clyne, P. J., Brotman, J. S., Sweeney, S. T., Davis, G. Green fluorescent protein tagging Drosophila proteins at their native genomic loci with small P elements. Genetics. 165 (3), 1433-1441 (2003).
- Orfanos, Z., Sparrow, J. C. Myosin isoform switching during assembly of the Drosophila flight muscle thick filament lattice. Journal of Cell Science. 126 (1), 139-148 (2013).
- Volohonsky, G., Edenfeld, G., Klambt, C., Volk, T. Muscle-dependent maturation of tendon cells is induced by post-transcriptional regulation of stripeA. Development. 134 (2), 347-356 (2007).
- Estes, P. S., Ho, G. L., Narayanan, R., Ramaswami, M. Synaptic localization and restricted diffusion of a Drosophila neuronal synaptobrevin--green fluorescent protein chimera in vivo. Journal of Neurogenetics. 13 (4), 233-255 (2000).
- Hida, N., et al. EC-tagging allows cell type-specific RNA analysis. Nucleic Acids Research. 45 (15), 138 (2017).
- Thomas, A., et al. A versatile method for cell-specific profiling of translated mRNAs in Drosophila. PLoS ONE. 7 (7), 40276 (2012).
- Yang, Z. Isolation of mRNA from specific tissues of Drosophila by mRNA tagging. Nucleic Acids Research. 33 (17), 148 (2005).
- Jiao, Y., Moon, S. J., Montell, C. A Drosophila gustatory receptor required for the responses to sucrose, glucose, and maltose identified by mRNA tagging. Proceedings of the National Academy of Sciences of the United States of America. 104 (35), 14110-14115 (2007).
- Blazie, S. M., et al. Comparative RNA-Seq analysis reveals pervasive tissue-specific alternative polyadenylation in Caenorhabditis elegans intestine and muscles. BMC Biology. 13 (1), 1775-1821 (2015).
- Henry, G. L., Davis, F. P., Picard, S., Eddy, S. R. Cell type-specific genomics of Drosophila neurons. Nucleic Acids Research. 40 (19), 9691-9704 (2012).
- Deal, R. B., Henikoff, S. The INTACT method for cell type-specific gene expression and chromatin profiling in Arabidopsis thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Grinstein, M., Dingwall, H. L., Shah, R. R., Capellini, T. D., Galloway, J. L. A robust method for RNA extraction and purification from a single adult mouse tendon. PeerJ. 6 (8), 4664 (2018).
- Green, M. R., Sambrook, J. . Molecular Cloning. , (2012).
- Brown, R. A. M., et al. Total RNA extraction from tissues for microRNA and target gene expression analysis: not all kits are created equal. BMC Biotechnology. 18 (1), 16 (2018).
- Ford, K. L., et al. Optimisation of laboratory methods for whole transcriptomic RNA analyses in human left ventricular biopsies and blood samples of clinical relevance. PLoS ONE. 14 (3), 02136855 (2019).
- Ramachandran, P., Budnik, V. Dissection of Drosophila larval body-wall muscles. Cold Spring Harbor Protocols. (8), 5469 (2010).
- Ranganayakulu, G., Schulz, R. A., Olson, E. N. Wingless signaling induces nautilus expression in the ventral mesoderm of the Drosophila embryo. Developmental Biology. 176 (1), 143-148 (1996).
- Dutta, D., Bloor, J. W., Ruiz-Gómez, M., VijayRaghavan, K., Kiehart, D. P. Real-time imaging of morphogenetic movements in Drosophila using Gal4-UAS-driven expression of GFP fused to the actin-binding domain of moesin. Genesis. 34 (1-2), 146-151 (2002).
- Lemke, S. B., Schnorrer, F. In Vivo Imaging of Muscle-tendon Morphogenesis in Drosophila Pupae. Journal of Visualized Experiments. (132), e57312 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten