Method Article
IR-TEx:专为疟疾病媒阿诺菲斯甘巴病设计的大数据转录组学的开源数据集成工具
摘要
IR-TEx探索了阿诺菲莱斯甘丹鱼物种中与杀虫剂耐药性相关的转录谱。此处提供了有关使用该应用程序的完整说明、用于探索多个转录组数据集的修改,以及使用该框架构建一个交互式数据库,用于从任何组织中提取转录数据(在任何平台中生成)。
摘要
IR-TEx 是一种用 Shiny(R 包)编写的应用程序,它允许探索其表达与Anopheles gambiae蚊子中的杀虫剂抗性表型相关的转录本的表达(以及为其分配函数)。该应用程序可以在线使用或下载和本地使用任何人。可以修改本地应用程序,以添加从多个 -omics 平台生成的新的杀虫剂抗性数据集。本指南演示如何添加新数据集和处理缺失数据。此外,IR-TEx 可以完全轻松地重新编码为使用任何实验数据中的 omics 数据集,使其成为许多研究人员的宝贵资源。该协议以微菌体谷胱甘肽转移酶GSTMS1为例,说明了IR-TEx在确定新的杀虫剂耐药性候选物的效用。该成绩单在来自科特迪瓦和布基纳法索的多个抗热虫种群中进行了调节。共相关转录本的鉴定提供了进一步洞察该基因的假定作用。
引言
通过微阵列平台和RNAeq技术同时测量大量转录文的表达的能力,产生了大量数据集,将成绩单表达与模型和非模型生物体中的特定表型相关联。这些数据集对于研究人员来说是极其丰富的资源,通过在大数据集成方法中结合相关集,可以增强其能力。然而,这种方法仅限于那些具有特殊生物信息学技能的人。这里描述的是一个程序,IR-TEx(以前由Ingham等人1)发布,它写在名为Shiny2的R包中,它允许几乎没有生物信息学训练的用户相对轻松地集成和询问这些数据集。
IR-TEx,发现在http://www.lstmed.ac.uk/projects/IR-TEx,是写探索与杀虫剂耐药性有关的记录在阿诺菲莱斯冈比亚,非洲主要的疟疾病媒1。疟疾是由疟原虫引起的寄生虫病,通过雌性疟原虫叮咬在人类之间传播。事实证明,用杀虫剂攻击蚊媒是预防非洲与疟疾有关的发病率和死亡率的最有效手段。自2000年以来,扩大工具(即长效杀虫蚊帐)也是疟疾病例大幅减少的关键。由于杀虫剂数量非常有限,蚊子的进化压力很大,现在非洲疟疾病媒中抗药性广泛。
此外,靶点突变5和杀虫剂的代谢清除6,7仍然是主要研究的抗药性机制,但其他强效抗药性机制现在出现1。这些新机制中有许多以前没有与杀虫剂耐药性有关,而是通过使用IR-TEx应用程序在多个耐药人群中寻找基因表达的常见模式,随后通过基因组学方法1进行功能验证来检测。
此处介绍的是使用 IR-TEx 的分步方法,无论是在 Web 上还是在本地安装时。该协议描述了如何将新的杀虫剂耐药性数据集集成到现有软件包中,并解释了如何使用缺失的数据进行操作。最后,它描述了如何将该软件与其他与杀虫剂耐药性无关的 -omics 数据集一起使用,从而将不同 -omics 方法的数据组合在一起,同时同时使用缺失值和规范化操作,以便数据具有可比性。
研究方案
1. 使用IR-TEx网络应用程序
- 在 Web 浏览器中运行应用程序
- 通过按照在http://www.lstmed.ac.uk/projects/IR-TEx找到的页面底部的链接打开 IR-TEx Web 应用程序。
- 初始化网页后,单击页面顶部的"应用程序"按钮,该按钮将显示应用程序和相关输出。
- 在成绩单 ID 框中读取与AGAP008212-RA (CYP6M2) 的默认条目相关的每个输出,并符合以下条件:A. coluzzii数据集(i) 暴露于除虫菊杀虫剂或 (ii) 未暴露于任何杀虫剂类,以及相关脚本与 |r=>0.98.
- 探索兴趣记录的表达
- 要选择感兴趣的脚本,请在成绩单 ID 框中输入脚本ID,记住脚本以-RX结尾,具体取决于兴趣的等式形式。
- 通过勾选 (i) 国家/地区的相关框来选择要查询的数据集;(四) 国家/地区/a;(四) 国家/地区/a/a;(四) 国家/地区/地区/地区/a/a;( i) 国家/地区/(二) 接触状况,(三) 兴趣物种;和(iv) 杀虫剂类的兴趣,同时确保这些标准导致 >1 包含数据集(参见 Ingham 等人 1 中的补充表 1)。
注:(iii)指用户感兴趣的An.gambiae物种复合体的成员。目前,关于安·科卢齐伊和阿拉比恩西斯的数据已经掌握。 - 单击选择菜单底部的更新视图,或按"返回",忽略绝对相关性值(现在)。
- 给应用程序更新时间。
- 将第一个图表读为:记录每个数据集中符合步骤 1.2 中所选条件的耐药人群和实验室易感蚊虫群之间的2倍变化,这些记录符合步骤 1.2 中选择的条件。所有数据集的详细信息可以在 Ingham 等人1中找到。
- 阅读下图的信息,如:除更正的 p 值 (Q) 外,每个相关数据集的抗蚊和易感蚊子之间的折叠变化。每行表示微阵列上的单个探测。图形显示的方法已报告之前1。
- 阅读下表,作为感兴趣成绩单重要的实验数,以及符合步骤 1.2 中选择的标准的实验总数。
- 要以选项卡分隔格式下载数据,请单击两个表下的"下载"按钮。这允许用户使用 Excel 等程序以更简单的方式浏览数据。
- 按如下方式解释地图:每个点表示每个数据集中具有抗药性蚊子的近似收集站点,其中兴趣记录以不同方式表示。颜色遵循在应用程序中解释的红绿灯系统 (图 2)。
- 对于步骤 1.2.5 和 1.2.8,通过右键单击、单击"将图像另存为..."并选择适当的文件夹来保存图形输出。
注: 在应用程序出现输出错误的情况下,可能没有数据集与输入的条件匹配。如果发生这种情况,请查看 Ingham 等人 1 中的补充表1。
- 确定感兴趣的假定函数/路径
- 跨多个数据集的脚本表达式模式的相关性(最小 r2输入值)可用于预测脚本函数,并可能从同一通路阐明共调控的脚本。使用 Ingham 等人1( AGAP001076-RA) 的示例;CYP4G16,按照上一节中的步骤1.2.1~1.2.2,选择所有数据集以获得最大功率。
- 在单击"更新视图"之前,将绝对相关性值滑块移动到 0.85,然后单击"更新视图"或"返回"。
- 检查相关表(最底层表),以查找当前显示并与输入的脚本相关的多个脚本(\r= = 0.85)。
- 操作绝对相关性值滑块,并观察最底部图形和表格中的任何变化;步骤 1.3.2 中的输出将保持不变。如图3 (\r= > 0.9, &r= > > 0.8), 降低相关值的严格性将显示更多的脚本,但会引入更多的噪声。
- 阅读下面的图形输出表,该表(除了步骤 1.2.6 中描述的参数外)包含每个脚本的相关值。
- 要以选项卡分隔格式下载数据,请单击"下载"按钮。
- 使用DAVID分析8,可以在下载的脚本ID列表上执行功能浓缩分析。一旦在DAVID网站(在https://david.ncifcrf.gov/找到),选择功能分析。粘贴完整的基因列表,使用基因 ID [不带 -RX 的标识符,通过在系统 ID 右侧插入一列并键入_LEFT(X1,10)(其中 X1 是系统 ID 单元格),可以在 Excel 中完成。选择标识符作为VectorBase_ID和基因列表,然后单击"提交列表"。
- 单击"功能注释聚类"按钮可生成此关联网络中的丰富内容的概览,从而允许将潜在函数分配给脚本。通过查看不同的类别并单击每个类别的+按钮,然后单击"图表"来探索深入的丰富性。
2. 在本地下载和实施 IR-TEx
- 下载并运行 IR-TEx
- 转到在http://github.com/LSTMScientificComputing/IR-TEx找到的链接;然后单击克隆或下载|下载邮编。直接到选择的文件夹,然后解压缩该文件夹中的文件。
- 从http://cran.r-project.org/mirrors.html找到的链接下载相应操作系统的最新版本的R软件。安装程序。
- 下载并安装最新的R Studio软件,再次从http://www.rstudio.com/products/rstudio/download/找到的链接找到相应的操作系统。
- 安装后,打开R 工作室|补充编码文件1,并运行每行来设置IR-TEx系统。
- 成功安装并根据需要更新所有软件包后,请转到文件|打开,找到IR-TEx.R,突出显示并打开。这应该在R工作室的顶部窗口可见。
- 要运行应用,请按窗口右上角的"运行应用"按钮,将弹出第二个窗口,其中将加载应用。加载完成后,单击"打开位于加载窗口右上角的浏览器"的完整功能。
- 将电阻数据集添加到 IR-TEx(使用Anopheles gambiae 15k 安捷伦阵列生成)
- 要将在同一微阵列平台 (A-MEXP-2196) 上生成的新分析数据集添加到可用数据集,请下载应用并找到第 2.1 节中下载的解压缩文件夹。
- 打开附加文件 1,它表示 A-MEXP-2196 1上的 limma 分析的输出。使用 Excel,在列 H1 中,编写Fold_Change,在 H2 中写入{2}B2,其中 B2 是日志折叠更改。在列 H 中应用此方法可生成原始折叠更改。
- 排列附加文件 1,使列 A 是 ID,列 B 是从列 H(复制列 H,突出显示列 B,然后右键单击和粘贴值)的折叠更改,列 C 是调整的 p 值。删除所有其他列并保存为选项卡分隔文件。
- 打开补充编码文件 2并使用步骤 2.2.3 中生成的选项卡分隔表运行。
NEWFILE_FC = c("国家","已使用状态","物种","INSECTICIDE")
NEWFILE_Q = c("国家","已使用状态","物种","INSECTICIDE")
注: 应更改单个引号中的字段以反映新数据集中的信息。接触状态是指在接触杀虫剂(暴露/未暴露)后是否收集了样本。杀虫剂:如果"未曝光",请使用"无"。请参阅Fold_Changes.txt。来自其他示例的元数据。确保拼写一致。 - 打开地理.txt,滚动到最后占用的行,并在下面选择。键入数据集的名称,后跟第 1 列中的Q和NEWFILE_Q、列 2 中样本集网站的纬度和第 3 列中的经度。保存更改。
- 如果使用任何新颖的条目(即冈比亚),这些条目在数据集中无法选择(参见 Ingham 等人补充表 11),则需要将这些条目添加到代码中。为此,请在 RStudio 中打开 IR-TEx.R 并找到 RStudio 指示的线路 26,此时应开始以下操作:
'侧边板(....'。
注: 每个继续行都与步骤 2.2.5 中 Fold_Changes.txt 中数据集名称下方的行中输入的元数据项相关。 - 要添加新颖的元数据,请滚动到所选元数据的行的末尾,然后找到术语"选择*"。紧随其后应为逗号和闭合括号;此时,单击关闭括号内的光标。在最终撇号之后,键入逗号,后跟撇号,后跟新的元数据(例如,"冈比亚"),并保存更改。有关示例,请参阅下文。
"国家输入"("国家输入","选择相关国家",c("布基纳法索","科特迪瓦","喀麦隆","赤道几内亚","赞比亚","坦桑尼亚","苏丹","乌干达',"多哥', "冈比亚",选择"科特迪瓦","科特迪瓦","喀麦隆","赤道几内亚","赞比亚","坦桑尼亚","苏丹","乌干达","多哥") - 运行应用。新的元数据条目应显示为相关标题下的未选中的勾选框。如果用户希望选择它,则应在选择\c(...,如下所示)之后添加它:
"国家输入"("国家输入","选择相关国家",c("布基纳法索","科特迪瓦","喀麦隆","赤道几内亚","赞比亚","坦桑尼亚","苏丹","乌干达',"多哥', "冈比亚",选择"科特迪瓦","科特迪瓦","喀麦隆","赤道几内亚","赞比亚","坦桑尼亚","苏丹","乌干达","多哥","冈比亚") - 要添加未在 A-MEXP-2196 上执行的电阻数据集,请参阅第 3 节。
3. 修改 IR-TEx 以用于不同的数据集
- 跨多个 -omics 平台使用,并继续处理缺少的数据
- 要继续在数据集中执行"0":请查阅数据集源,获取"0"的特定含义。建议(保守)将"0"替换为"NA"。与原始折叠变化 (B/A) 一样,"0"表示实验条件 B 中未检测到的信号。在实验条件 A 显示实质性表达式的情况下,用户可以应用小折叠更改值。
- 打开附加文件2.txt,一个从Uyhelji等人改编而成的RNAeq文件。此文件表示新数据应基于的模板:列 A = 标识符、列 B = 原始折叠更改,以及列 C = 调整的 p 值。使用此文件可以运行以下步骤。
- 运行 R 代码,将标识符匹配到跨平台的单个选项卡分隔文件中,然后组织和规范化数据 (补充编码文件 2)。说明包含在文件中。任何 FILEPATH 将用 MacOS 的"/" 或 Windows 的"//"分隔(从"\"中更改它们,因为它们会出现)。
- 将补充编码文件 2末尾生成的文件输出到步骤 3.1.5 中的首选位置。补充编码文件2将输出一个新的Fold_Changes.txt文件。备份原始文件。
- 执行补充编码文件3中包含的代码。在指定为FILEPATH的文件夹中查找名为FC_distribPlot.png的输出文件。检查日志2折叠更改的分布,以验证日志2折叠更改分布在数据集之间几乎相同。
- 按照步骤 2.2.6 中的说明编辑其他文件并确保新Fold_Changes.txt的兼容性。
- 修改 IR-TEx 以用于全新的数据集
- 在 RStudio 中打开IR-TEx.R并找到以以下开头的行 (23-34):
'选项卡面板('
结尾为:
提交按钮("更新视图",图标("刷新"))
), - 将以下行中的AGAP008212-RA更改为对新数据感兴趣的脚本。
文本输入("文本输入","脚本 ID",值="AGAP008212-RA"), - 找到以下四个选项:
复选框组输入(
可以修改这些选项以表示用户希望筛选新数据的重要元数据。在每个实例中,用户应更改"选择相关国家/地区";选择曝光状态;选择相关物种;并选择杀虫剂类作为数据的代表(即,选择组织类型;选择性别;选择年龄支架;选择疾病状态)。 - 识别与数据集和输入关联的元数据,以在第一个c('之后立即替换现有选项)在每个实例中,选项将包含在语音标记中,并用逗号与下一个选择分开。最终选择后,应关闭支架。选择疾病状态的示例是:
c("感染","未感染","未知") - 选择在打开应用时将选择哪些元数据。可以通过在选择_c('之后修改选项来更改这些选项)来更改这些选项。选择疾病状态的示例是:
选择\c("感染","未感染")
这将指示应用在初始加载时仅选择与这些标准匹配的数据集。 - 要创建新的数据表,请按照Fold_Changes.txt中的布局和第 2 节中的说明进行操作。将元数据更改为步骤 3.2.4 中概述的每个相应的更改,与写入代码完全一样(R 区分大小写)。进入解毒列,输入基因名称,并在成绩单类型列中,输入每个成绩单的基因描述。添加新数据集时,请遵循第 3.2 节。
- 如果映射与实验要求无关,请找到以下代码行,并将"*"放在前面:
第 49-51 行:
br(,br()
带平纳(图输出("地理"),)
文本输出("Geography_legend"),
第 493 行开始:
输出 $地理 <- 渲染图(*
到行 602 结束:
输出$Geography_legend <- 渲染文本(*
粘贴("仅限重要脚本(p",作为.表达式("<="),"0.05":FC > 5 = 红色,FC > 1 = 琥珀色,FC < 1 = 绿色",sep=""
})
- 在 RStudio 中打开IR-TEx.R并找到以以下开头的行 (23-34):
结果
我们使用IR-TEx附带的Fold_Changes.txt文件,比较了在耐抗阿诺菲莱斯·科鲁齐和阿诺菲斯冈比亚数据集中明显不同表达的脚本,与来自科特迪瓦和布基纳法索的易感对照。这产生了18个感兴趣的脚本(表1;此搜索可以使用Excel、R或其他程序执行)。其中两种,一个ATPase(AGAP006879)和β-晶石(AGAP007160),以前曾报道过,前者对除虫菊酯耐药性有显著影响。除了这两个成绩单外,还有两个解毒记录,即GSTMS1(FC = 1.95和1.85)和UGT306A2(FC = 2.29和2.28)。
qPCR验证了其中两个记录(GSTMS1,一个解毒记录;和AGAP009110-RA,一个未知的,蚊子特定的记录,包含β-1,3-葡糖结合域)执行,如前面描述1。分析使用附加文件3中描述的引录集进行,并表明这些记录在来自科特迪瓦(Tiasalé)和另一个布基纳法索(班福拉)的耐多人群中明显受到调节,而实验室中易受实验室影响的N'Gousso(图4A)。
由于两份记录显示每个耐药人群的调节显著上升,因此在LSTM实验室Tiasalé菌群对蚊子进行了RNAi诱导的击倒。这个殖民地起源于科特迪瓦,对公共卫生中使用的所有主要杀虫剂具有抗药性,如前文所述。与GFP注射对照组相比,GSTMS1表达的衰减导致三角甲二甲醚暴露后的死亡率显著增加(p = 0.021),这表明该转录在除虫菊菊阻抗中的重要性(图4B)。相反,AGAP009110-RA击倒导致暴露后死亡率没有显著变化(图4B)。
GSTMS1是一个微体GST,是发现在A.gambia蚊子11的三个之一。虽然Epsilon和delta类的GST成员以前曾被牵连到杀虫剂解毒12,13,14,这是我们所知的第一个证据,表明微体GST在除虫菊抗性15中的作用。为了探索该记录在阿诺菲莱斯甘丹蚊中的假定功能,确定了IR-TEx的表达和相关性。除比奥科岛外,在可用于这些物种的21个数据集中,有20个数据集中,GSTMS1明显多于表达。在每个位置,与易感人群相比,过度表达不到五倍(图5)。
由于微生物类GST作为潜在的杀虫剂解毒剂在很大程度上被忽视,人们对它们在杀虫剂耐药性中的作用知之甚少。通过探索其他抄本的共相关关系,可以通过假定共同监管或参与同一途径来阐明假定函数。为了最大化相关网络中的功率,选择了 IR-TEx 中存在的所有微阵列数据集,并选择了 [r]>0.75 被选中。表 2显示了来自 IR-TEx 的输出。
这些转录本在DAVID的功能注释工具8中富含氧化酶活性和葡萄糖/碳水化合物代谢。葡萄糖-6-磷酸脱氢酶和西他提酮伽马-乳酸酶均维持哺乳动物细胞16、17中的谷胱甘肽水平,因此与谷胱甘肽-S转移酶GSTMS1直接相关。Catalase 是一种快速作用的氧化应激反应剂,可保护细胞免受活性氧物种损害,这是除虫菊酯暴露的副产品。瓦拉西洛韦水酶是一种水酶,在哺乳动物细胞的解毒中可能起一定的作用。CYP4H17也存在于相关网络中。细胞色素p450s是除虫菊杀菌杀虫剂的直接代谢物,这些分解产物可以通过GST进一步代谢。最后,CYP4H17在A.funestus19中与除虫菊抗性有关。综合起来,这些数据有力地支持了GSTMS1在异种生物解毒中的作用。
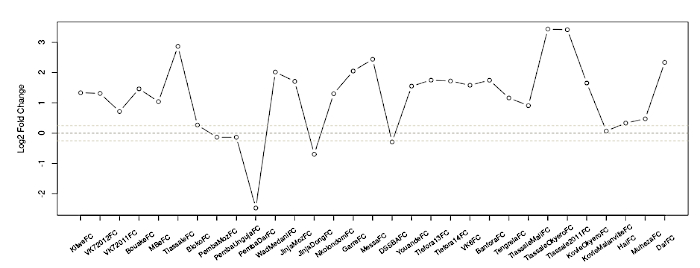
图 1:记录所有数据集中 AGAP002865-RA 的 2 折更改。x 轴详细说明了不同的数据集,可在上一份出版物1中的补充表 1 中找到这些信息,y 轴显示感兴趣记录中的日志2折变化。浅灰色虚线表示显著性近似阈值,此处为 <0.8 折更改或 >1.2 的折数变化。点状黑线表示折数变化 1(即抵抗人群和易感人群之间的表达没有差异)。请点击此处查看此图的较大版本。
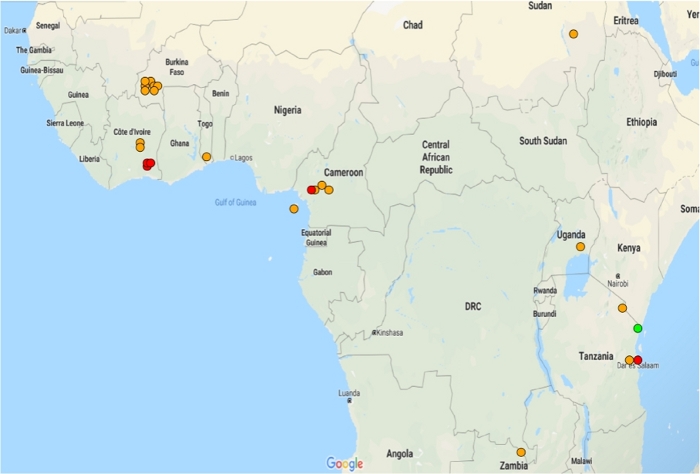
图2:微阵列分布,显示AGAP002865-RA在抗性人群中的显著差异表达。折叠变化在交通灯系统中表示:绿色折叠变化 <1,橙色折叠变化 >1,红色折叠变化 >5。仅显示具有显著(p = 0.05)差异表达式的数据集。请点击此处查看此图的较大版本。
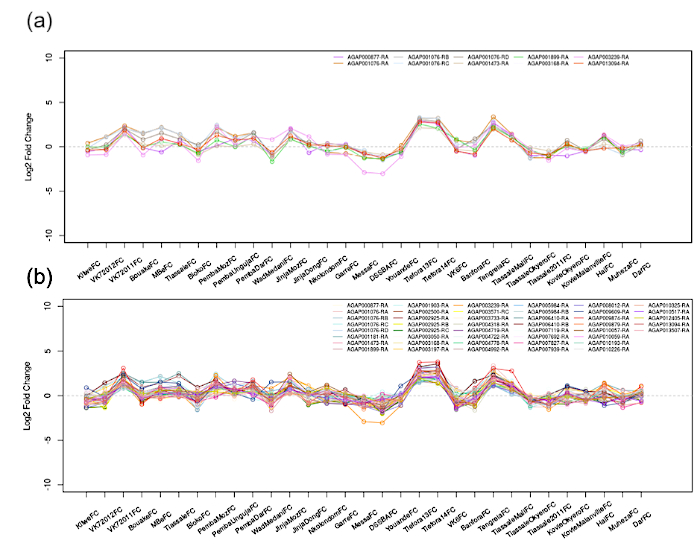
图3:AGAP001076-RA(CYP4G16)的相关网络。在 31 个微阵列数据集的所有脚本中计算成对相关性,并应用用户定义的截止。此处显示的是 (A) [r]> 0.9 和 (B) [r]> 0.8.图上显示的所有脚本都符合此标准,并遵循 AGAP001076-RA 的表达式更改。请点击此处查看此图的较大版本。
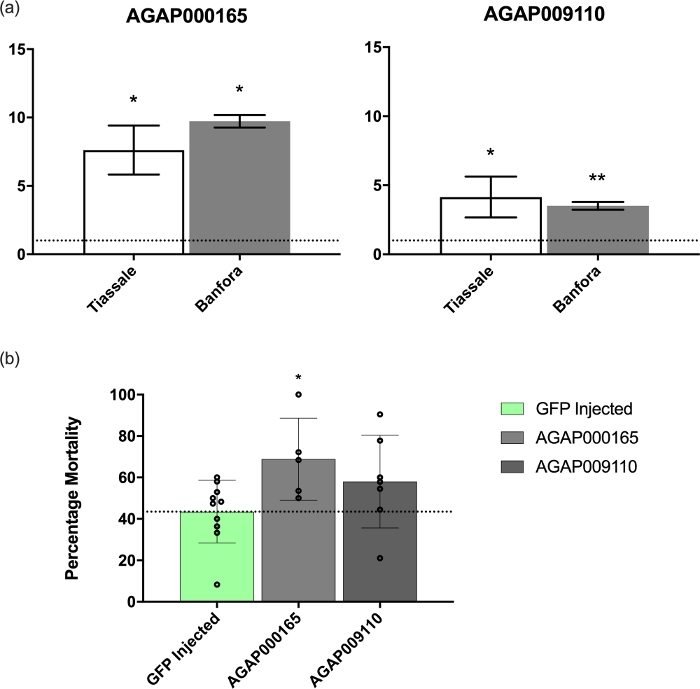
图4:GSTMS1和AGAP009110-RA衰减时的mRNA表达和表型。(A) 在科特迪瓦和布基纳法索的两个耐多抗的安.科卢齐伊种群中,对GSTMS1和AGAP009110-RA的mRNA表达。与实验室易感性安·科鲁齐·恩古索相比,水平进行了比较。由 ANOVA 计算的显著性水平,通过后特的 Dunnett 测试计算。(B) 与GFP注入对照组相比,两个转录本的RNAi诱导衰减。GSTMS1衰减显示三角甲二甲二甲二甲二甲二甲物暴露后的死亡率显著增加(由ANOVA通过特次图基测试计算;[p = 0.05,+p = 0.01])。请点击此处查看此图的较大版本。
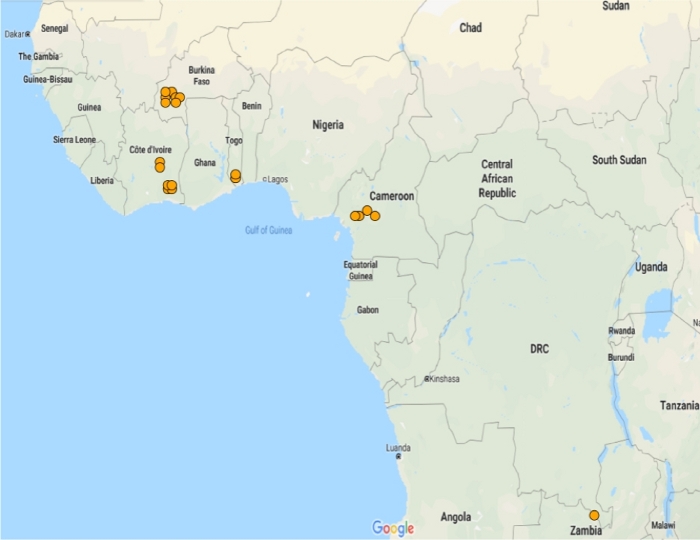
图5:在阿诺菲莱斯·甘比亚和阿诺菲莱斯·科鲁齐伊种群中GSTMS1的表达。显示可用微阵列数据集中GSTMS1显著差异表达式的地图。在 21 个微阵列数据集中,有 20 个数据集的GSTMS1明显存在差异。请点击此处查看此图的较大版本。
| 成绩单 ID | 描述 | 布基纳法索 | 科特迪瓦 |
| AGAP006879-RA | ATPase | 27.94 | 43.05 |
| AGAP007160-RB | 阿-水晶林 | 11.49 | 10.58 |
| AGAP007160-RC | 阿-水晶林 | 11.14 | 10.38 |
| AGAP007160-RA | 阿-水晶林 | 9.78 | 9.84 |
| AGAP009110-RA | 未知 | 9.26 | 5.96 |
| AGAP007780-RA | NADH 脱氢酶 | 10.49 | 3.77 |
| AGAP006383-RA | 寡糖酶复合亚单位β | 3.69 | 5.57 |
| AGAP007249-RB | 飞行 | 4.61 | 3.86 |
| AGAP003357-RA | RAG1激活蛋白1样蛋白 | 4.31 | 4.05 |
| AGAP007249-RA | 飞行 | 4.48 | 3.46 |
| AGAP001998-RA | mRpS10 | 3.46 | 2.85 |
| AGAP007589-RA | UGT306A2 | 2.29 | 2.28 |
| AGAP000165-RA | GSTMS1 | 1.95 | 1.85 |
| AGAP002101-RA | 伊索莱基-tRNA合成酶 | 0.57 | 0.59 |
| AGAP002969-RA | 芦参数-tRNA合成酶 | 0.45 | 0.45 |
| AGAP004199-RA | 溶质载体系列 5(钠耦合单卡苯二酯运输器),成员 8 | 0.35 | 0.48 |
| AGAP004684-RA | rRNA处理蛋白CGR1 | 0.36 | 0.22 |
| AGAP006414-RA | Cht8 | 0.024 | 0.36 |
表1:布基纳法索和科特迪瓦人口在同一折数变化方向上有明显差异。来自两个国家代表安·科鲁齐和安·甘比亚人口的每个数据集的脚本 ID、基因描述和平均折叠变化。
| 相关 | 系统名称 | 成绩单类型 |
| 1 | AGAP000165-RA | GSTMS1 |
| 0.82 | AGAP004904-RA | 过氧化氢 酶 |
| 0.76 | AGAP007243-RA | 26S 蛋白酶监管子单元 8 |
| 0.79 | AGAP008358-RA | CYP4H17 |
| 0.76 | AGAP009436-RA | 瓦拉西克洛韦水酶 |
| 0.75 | AGAP010739-RA | 葡萄糖-6-磷酸盐1-脱氢酶 |
| 0.85 | AGAP011172-RA | 半胱氨酸伽马-莱酶 |
| 0.76 | AGAP012678-RA | 葡萄糖-6-磷酸盐1-脱氢酶 |
表2:与GSTMS1密切相关的成绩单。该表显示了使用 {r} 的 IR-TEx 上GSTMS1的相关网络输出> 0.75.该表显示了斯皮尔曼的关联性、成绩单 ID 和每个共相关成绩单的基因描述。
附加文件 1:在 limma 上分析的 A-MEXP-2196 阵列的输出文件。与GFP控制数组相比,该文件源自Met敲除,在 ArrayExpress (E-MTAB-4043) 和另一个以前的出版物1中详细介绍了。列表示 AGAP 标识符(系统名称)、日志折叠更改 (logFC)、日志表达式值 (AveExpr)、t 统计量 (t)、未校正的 p 值 (P.Value)、调整后的 p 值 (adj)。P.Val)和B统计(B)20。就本文件而言,这些蚊子是来自科特迪瓦的阿诺菲莱斯科卢齐,不接触杀虫剂,采集纬度和经度分别为-5.4和6.0。请点击此处查看此文件(右键单击下载)。
附加文件 2:来自 RNAseq 实验的输出文件。RNAseq分析取自Uyhelji等人9,描述了在暴露于50%盐度时,阿诺菲勒蚊子转录组的变化。此文件改编自出版物的表 S2,包括 AGAP 标识符 (系统 ID)、原始折叠更改 (Fold_Change) 和调整后的 p 值 (q_value)。请点击此处查看此文件(右键单击下载)。
附加文件 3:代表性结果的入门列表。AGAP标识符、基因名称、dsRNA正向、dsRNA反向、qPCR正向和qPCR反向引录集。请点击此处查看此文件(右键单击下载)。
补充编码文件 1。请点击此处查看此文件(右键单击下载)。
补充编码文件2。请点击此处查看此文件(右键单击下载)。
补充编码文件3。请点击此处查看此文件(右键单击下载)。
讨论
大数据转录组学生成数千个转录记录的列表,这些转录记录对于每个实验条件都进行了不同的表达。其中许多实验是在相关生物体和表型上进行的,并且几乎完全被分析为独立的实验。利用这些丰富的数据源,通过全面检查数据,没有理论假设将1)导致新的候选成绩单的识别,2)防止丢弃有价值的数据,只是因为有太多的信息,以验证在体内1。
IR-TEx 为用户提供了有限的生物信息学背景,能够轻松检查多个数据集、可视化数据集中的更改以及下载相关信息1。尽管 IR-TEx 不支持在每个搜索中搜索多个脚本,但用户只需使用 Excel、R 或其他适当的程序即可检查关联的Fold_Changes.txt 文件。IR-TEx的进一步效用源于使用相关网络来预测转录功能,输入具有未知功能的假设蛋白质或转录本,以及使用下游软件搜索富集1。
在本协议中演示的示例中,IR-TEx 根据其原始功能使用。在这里,它允许探索与杀虫剂耐药性相关的记录,并通过映射图形可视化过度和不足表达的分布。感兴趣的记录在体内进行验证,以确定给定转录本的过度表达或表达不足是否有助于观察到的表型1(例如,杀虫剂耐药性)。这里演示了数据集,如前面报道的1所示,数据集可用于假设驱动的方法,以根据具体国家/地区确定感兴趣的脚本。然后,IR-TEx 可用于 1) 浏览脚本的表达式,2) 通过在每个 -omics 数据集中包含的所有脚本中应用成对关联网络来将脚本的功能与上下文相关。在这里,GSTMS1被证明与一些与排毒有关的其他成绩单有共相关。这些数据(连同导致接触杀虫剂后死亡率显著增加的抄本的敲击)表明该记录在异种生物清除中的重要性。
IR-TEx 是探索网络上或使用本地应用的杀虫剂耐药性相关记录的宝贵资源。该协议演示如何针对不同的 -omics 平台以及全新的数据修改 IR-TEx。本指南说明了如何使用 IR-TEx 将来自多个 -omics 平台和数据集的数据与缺失数据集成,以及如何重新编码 IR-TEx,以便对研究转录数据集的任何人有用。
披露声明
作者没有什么可透露的。
致谢
这项工作由MRC技能发展奖学金资助V.I.(MR/R024839/1)和皇家学会挑战赠款(CH160059)到H.R.。
材料
| Name | Company | Catalog Number | Comments |
| Laptop with browser | Any | - | - |
| R Program | The R Project for Statistical Computing | - | https://www.r-project.org/ |
| R Studio | R Studio | - | https://www.rstudio.com/ |
参考文献
- Ingham, V. A., Wagstaff, S., Ranson, H. Transcriptomic meta-signatures identified in Anopheles gambiae populations reveal previously undetected insecticide resistance mechanisms. Nature Communications. 9 (1), 5282(2018).
- Chang, W., Cheng, J., Allaire, J., Xie, Y., McPherson, J. shiny: Web Application Framework for R. , (2017).
- Bhatt, S., et al. The effect of malaria control on Plasmodium falciparum in Africa between 2000 and 2015. Nature. 526 (7572), 207-211 (2015).
- Ranson, H., Lissenden, N. Insecticide Resistance in African Anopheles Mosquitoes: A Worsening Situation that Needs Urgent Action to Maintain Malaria Control. Trends in Parasitology. 32 (3), 187-196 (2016).
- Donnelly, M. J., et al. Does kdr genotype predict insecticide-resistance phenotype in mosquitoes. Trends in Parasitology. 25 (5), 213-219 (2009).
- Stevenson, B. J., et al. Cytochrome P450 6M2 from the malaria vector Anopheles gambiae metabolizes pyrethroids: Sequential metabolism of deltamethrin revealed. Insect Biochemistry and Molecular Biology. 41 (7), 492-502 (2011).
- Müller, P., et al. Field-Caught Permethrin-Resistant Anopheles gambiae Overexpress CYP6P3, a P450 That Metabolises Pyrethroids. PLoS Genetics. 4 (11), 1000286(2008).
- Huang, D., et al. The DAVID Gene Functional Classification Tool: a novel biological module-centric algorithm to functionally analyze large gene lists. Genome Biology. 8 (9), 183(2007).
- Uyhelji, H. A., Cheng, C., Besansky, N. J. Transcriptomic differences between euryhaline and stenohaline malaria vector sibling species in response to salinity stress. Molecular Ecology. 25 (10), 2210-2225 (2016).
- Edi, C. V., Benjamin, K. G., Jones, C. M., Weetman, D., Ranson, H. Multiple-Insecticide Resistance in Anopheles gambiae Mosquitoes, Southern Côte d’Ivoire. Emerging Infectious Diseases. 18 (9), 1508-1511 (2012).
- Ding, Y., Ortelli, F., Rossiter, L., Hemingway, J., Ranson, H. The Anopheles gambiae glutathione transferase supergene family: annotation, phylogeny and expression profiles. BMC Genomics. 4 (1), 1-16 (2003).
- Enayati, A. A., Ranson, H., Hemingway, J. Insect glutathione transferases and insecticide resistance. Insect Molecular Biology. 14 (1), 3-8 (2005).
- Ranson, H., et al. Identification of a novel class of insect glutathione S-transferases involved in resistance to DDT in the malaria vector Anopheles gambiae. The Biochemical Journal. 359, 295-304 (2001).
- Riveron, J. M., et al. A single mutation in the GSTe2 gene allows tracking of metabolically based insecticide resistance in a major malaria vector. Genome Biology. 15 (2), 27(2014).
- Pavlidi, N., Vontas, J., Van Leeuwen, T. The role of glutathione S-transferases (GSTs) in insecticide resistance in crop pests and disease vectors. Current Opinion in Insect Science. 27, 97-102 (2018).
- Salvemini, F., et al. Enhanced glutathione levels and oxidoresistance mediated by increased glucose-6-phosphate dehydrogenase expression. Journal of Biological Chemistry. 274 (5), 2750-2757 (1999).
- Deplancke, B., Gaskins, H. R. Redox control of the transsulfuration and glutathione biosynthesis pathways. Current Opinion in Clinical Nutrition & Metabolic Care. 5 (1), (2002).
- Puente, X. S., López-Otn, C. Cloning and expression analysis of a novel human serine hydrolase with sequence similarity to prokaryotic enzymes involved in the degradation of aromatic compounds. Journal of Biological Chemistry. 270 (21), 12926-12932 (1995).
- Riveron, J. M., et al. Genome-wide transcription and functional analyses reveal heterogeneous molecular mechanisms driving pyrethroids resistance in the major malaria vector Anopheles funestus across Africa. G3: Genes, Genomes, Genetics. 7 (6), 1819-1832 (2017).
- Smyth, G. K. Linear models and empirical bayes methods for assessing differential expression in microarray experiments. Statistical Applications in Genetics and Molecular Biology. 3 (1), 3(2004).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。