Method Article
IR-TEx: Sıtma Vektör Anopheles gambiyae için tasarlanmış Büyük Veri Transkripsiyonları için Açık Kaynak Veri Entegrasyon Aracı
Bu Makalede
Özet
IR-TEx, Anopheles gambiaetüründe insektisit direncine bağlı transkripsiyonel profilleri keşfeder. Burada sağlanan uygulama nın kullanımı için tam talimatlar, birden fazla transkriptomik veri kümeleri keşfetmek için değişiklikler, ve herhangi bir platformda oluşturulan herhangi bir organizmadan transkripsiyonel veri koleksiyonları için interaktif bir veritabanı oluşturmak için çerçeve kullanarak.
Özet
IR-TEx, Anopheles gambiae sivrisineklerinde insektisit direnci fenotipleriyle ilişkilendirilen transkriptlerin ekspresyonunun (ve işlevlerin atanmasının yanı sıra) araştırılmasına olanak tanıyan Parlak (R paketi) ile yazılmış bir uygulamadır. Uygulama çevrimiçi olarak kullanılabilir veya indirilebilir ve herkes tarafından yerel olarak kullanılabilir. Yerel uygulama, birden çok omics platformlarından oluşturulan yeni insektisit direnci veri kümeleri eklemek için değiştirilebilir. Bu kılavuz, yeni veri kümeleri eklemek ve eksik verileri işlemek nasıl gösterir. Ayrıca, IR-TEx, herhangi bir deneysel veriden kullanılan omics veri kümelerine tamamen ve kolayca yeniden kodlanabilir ve bu da onu birçok araştırmacı için değerli bir kaynak haline getirir. Protokol mikrozomal glutatyon transferi kullanarak yeni insektisit direnci adayları nın belirlenmesinde IR-TEx yarar göstermektedir, GSTMS1, bir örnek olarak. Bu transkript Fildişi Sahili ve Burkina Faso'dan gelen birden fazla piretropid dirençli popülasyonda düzenlenir. Eş-ilişkili transkriptlerin tanımlanması, bu genin putatif rolleri hakkında daha fazla bilgi sağlar.
Giriş
Çok sayıda transkriptin ekspresyonunu mikrodizi platformları ve RNAseq teknolojisi aracılığıyla aynı anda ölçebilme yeteneği, hem model hem de model olmayan organizmalarda transkript ifadesini belirli bir fenotiple ilişkilendiren geniş veri kümelerinin oluşmasına yol açmıştır. Bu veri kümeleri araştırmacılar için son derece zengin bir kaynaktır ve bu kaynakların gücü, büyük bir veri tümleştirme yaklaşımında ilgili kümeleri birleştirerek artırılabilir. Ancak, bu metodoloji özellikle biyoinformatik becerileri olanlar ile sınırlıdır. Burada açıklanan bir program, IR-TEx (daha önce Ingham ve ark.1tarafından yayınlanan) parlak2 adlı bir R paketi nde yazılmış ve küçük biyoinformatik eğitimi olan kullanıcıların bu veri kümelerini göreceli kolaylıkla entegre ve sorgulamalarına olanak tanır.
IR-TEx, http://www.lstmed.ac.uk/projects/IR-TExbulunan , Anopheles gambiaeböcek direnci ile ilişkili transkript keşfetmek için yazılmıştır , büyük Afrika sıtma vektör1. Sıtma plazmodium türlerinin neden olduğu parazitik bir hastalıktır, dişi Anopheles sivrisinek ısırıkları ile insanlar arasında bulaşan. Sivrisinek vektörünün böcek öldürücülerle hedef alınması, Afrika'da sıtmaya bağlı morbidite ve mortaliteyi önlemenin en etkili yolu olduğu kanıtlanmıştır. Araçların ölçekleme (yani, uzun ömürlü insektisit ağları) da 2000 yılından bu yana sıtma vakalarında dramatik azalmalar önemli olmuştur3. Mevcut insektisitler çok sınırlı sayıda ile, sivrisinekler üzerinde güçlü evrimsel basınç vardır, ve direnç şimdi Afrika sıtma vektörleri yaygındır4.
Ayrıca, hedef bölge mutasyonları5 ve insektisitlerin metabolik temizliği6,7 direnç birincil çalışılan mekanizmaları kalır, ancak diğer güçlü dirençli mekanizmalar şimdi ortaya çıkmaktadır1. Bu yeni mekanizmaların çoğu daha önce insektisit direnci ile ilişkili değil, ancak IR-TEx uygulaması kullanarak birden fazla dirençli popülasyonlar arasında gen ekspresyonu ortak desenleri aranarak tespit edilmiş ve daha sonra işlevsel genomik yaklaşımlar tarafından doğrulanmış1.
Burada açıklanan ir-TEx kullanarak bir adım-adım yaklaşım, hem web üzerinde ve yerel olarak yüklü. Protokol, yeni insektisit direnci veri kümelerinin mevcut pakete nasıl entegre edilebildiğini ve eksik verilerle nasıl çalıştırılabildiğini açıklar. Son olarak, bu yazılımın insektisit direnciyle ilgisi olmayan diğer -omics veri kümeleriyle nasıl kullanılacağını açıklar, böylece farklı -omics yaklaşımlarından elde edilen verileri birleştirirken aynı zamanda eksik değerlerle ve normalleştirme ile de faaliyet gösterir, böylece veriler karşılaştırılabilir dir.
Protokol
1. IR-TEx web uygulamasını kullanma
- Uygulamayı bir web tarayıcısında çalıştırma
- http://www.lstmed.ac.uk/projects/IR-TExbulunan sayfanın altındaki bağlantıyı izleyerek IR-TEx web uygulamasını açın.
- Web sayfası başolarak ortaya çıktıktan sonra, uygulama ve ilişkili çıktıları görüntüleyen sayfanın üst kısmındaki Uygulama düğmesini tıklatın.
- AGAP008212-RA (CYP6M2)varsayılan girişiile ilgili her çıkışı transkript kimliği kutusunda aşağıdaki koşullarla okuyun: (i) piretropit li böcek öldürücülere maruz kalan veya (ii) herhangi bir insektisit sınıfına maruz olmayan coluzzii veri kümeleri ve |r| ile ilişkili transkriptler| >0.98.
- İlgi nin bir transkriptinin ifadesini keşfetme
- İlginin bir transkriptini seçmek için transkript kimliğini transkript kimliği kutusuna girerek transkriptlerin -RX'de sona erdiğini hatırlayın.
- (i) Ülkeler için ilgili kutuları işaretleyerek sorgulamak için veri kümelerini seçin; (ii) Maruz kalma durumu, (iii) İlgi alanı türleri; ve (iv) İnsektisit sınıfı ilgi alanı, tüm bu kriterlerin >1 ile sonuçlanmasını sağlarken veri seti (ingham ve ark.1'dekiEk Tablo 1'e bakınız).
NOT: (iii) kullanıcının ilgilendiği An. gambiya tür kompleksinin üyesini ifade eder. Şu anda, veri An. coluzzii ve An. arabiensisiçin kullanılabilir. - Seçim menüsünün altındaki Görünümü Güncelleştir'i tıklatın veya Mutlak Korelasyon Değerini yoksayarak Returntuşuna basın (şimdilik).
- Uygulamanın güncelleştirilen zaman vermesini.
- İlk grafiği şu şekilde okuyun: basamak1.2'de seçilen kriterleri karşılayan her veri kümesindeki ilgi nin transkriptinin dirençli popülasyonu ile laboratuvara duyarlı sivrisinek popülasyonu arasındaki log 2 kat değişimi(Şekil 1). Tüm veri kümelerinin ayrıntılarını Ingham ve ark.1'debulabilirsiniz.
- Grafiğin altındaki bilgileri şu şekilde okuyun: Düzeltilmiş p değerlerine (Q) ek olarak, ilgili her veri kümesi için dirençli ve duyarlı sivrisinekler arasındaki kıvrım değişiklikleri. Her satır mikrodizideki tek tek sondaları temsil eder. Grafiksel görüntüleme metodolojisi daha öncebildirilmiştir 1.
- Aşağıdaki ek tabloyu, ilgi nin transkriptinin önemli olduğu deneme sayısı ve adım 1.2'de seçilen ölçütleri eşleşen toplam deney sayısı olarak okuyun.
- Verileri sekme ayrılmış biçiminde indirmek için, iki tablonun altındaki İndir düğmesini tıklatın. Bu, kullanıcının Excel gibi bir programı kullanarak verileri daha kolay bir şekilde keşfetmesini sağlar.
- Haritayı şu şekilde yorumlayın: Her nokta, ilgi nin farklı olarak ifade edildiği her veri kümesindeki dirençli sivrisineklerin yaklaşık toplama alanlarını temsil eder. Renkler uygulamada açıklanan bir trafik ışığı sistemini izler (Şekil 2).
- 1.2.5 ve 1.2.8 adımları için, sağ tıklayarak, resmi kaydet'i ...tıklatarak ve uygun bir klasör seçerek grafik çıktılarını kaydedin.
NOT: Uygulamanın çıktı hatası durumunda, hiçbir veri kümesinin giriş ölçütleri ile eşleşmesi olasıdır. Ingham ve ark.1'de ek tablo 1'i kontrol edin.
- İlgi nin putatif fonksiyonlarını/yollarının belirlenmesi
- Transkriptlerin birden çok veri kümesi ndeki ifade kalıplarının korelasyonları (minimum r2 değeri girişi) transkript işlevini tahmin etmek ve aynı yoltaki birlikte düzenlenmiş transkriptleri açıklığa kavuşturmak için kullanılabilir. Ingham ve ark.1 (AGAP001076-RA; CYP4G16), yukarıdaki bölümde 1.2.1-1.2.2 adımlarını takip ederek maksimum güç için tüm veri kümelerini seçin.
- Güncelleştirme Görünümü'nütıklatmadan önce, Mutlak Korelasyon Değeri kaydırıcısını 0,85'e taşıyın ve Görünümü Güncelleştir'e veya İade'yebasın.
- Şu anda görüntülenen ve girişli transkriptle ilişkili olan (|r| = 0,85) birden çok transkripti bulmak için korelasyon tablosunu (en alttaki tabloyu) inceleyin.
- Mutlak Korelasyon Değeri kaydırıcısını manipüle edin ve en alttaki grafik ve tablodaki değişiklikleri gözlemleyin; adım 1.3.2'deki çıktılar değişmeden kalır. Şekil 3'te (|r| > 0.9, |r| > 0.8)'de gösterildiği gibi, korelasyon değerinin sıkılığını düşürmek daha fazla transkript gösterir, ancak daha fazla gürültü yetecektir.
- (adım 1.2.6'da açıklanan parametrelere ek olarak) her transkript için korelasyon değerini içeren grafik çıktının altındaki tabloyu okuyun.
- Verileri sekmeyle ayrılmış bir biçimde indirmek için İndir düğmesini tıklatın.
- Fonksiyonel zenginleştirme analizi david analizi8kullanılarak indirilen transkript kimlik listesinde yapılabilir. Bir kez DAVID web sitesinde (https://david.ncifcrf.gov/bulunan), Fonksiyonel Analizseçin. X1'in Sistematik Kimlik hücresi olduğu Sistematik Kimliğin sağına bir sütun ekleyerek ve =LEFT(X1,10)yazarak excel'de yapılabilecek -RX olmadan gen kimliklerini [tanımlayıcı-RX olmadan tanımlayıcı] kullanarak tam gen listesini yapıştırın. VectorBase_ID ve gen listesi olarak tanımlayıcıyı seçin ve Listeyi Gönder'itıklatın.
- Bu korelasyon ağında bulunan zenginleştirmelere genel bir bakış sağlamak ve transkripsiyüye potansiyel bir işlev atanmasını sağlamak için İşlevsel Ek Açıklama Kümeleme düğmesini tıklatın. Farklı kategorilere bakarak ve her biri için + düğmelerini tıklatarak ve daha sonra Grafik'itıklatarak derinlemesine zenginleştirmeleri keşfedin.
2. IR-TEx'i yerel olarak indirme ve uygulama
- IR-TEx'i indirme ve çalıştırma
- http://github.com/LSTMScientificComputing/IR-TExbulunan bağlantıya gidin; ve Clone veya download tıklayın | Zip indirin. Doğrudan seçtiğiniz bir klasöre yönlendirin ve dosyanın zip'ini açın.
- http://cran.r-project.org/mirrors.htmlbulunan bağlantıdan uygun işletim sistemi için R yazılımının en son sürümünü indirin. Programı yükleyin.
- Download ve en son R Studio yazılımı yüklemek, yine http://www.rstudio.com/products/rstudio/download/bulunan bağlantıdan uygun işletim sistemi için .
- Kurulduktan sonra, Açık R Studio | Ek kodlama Dosya 1 ve IR-TEx için sistem kurmak için her satırı çalıştırın.
- Tüm paketler başarıyla yüklendikten ve gerektiği gibi güncelleştirildikten sonra Dosya | Açık, IR-TEx.Rbulmak , vurgulamak ve açın. Bu şimdi R Studioüst penceresinde görünür olmalıdır.
- Uygulamayı çalıştırmak için, pencerenin sağ üst kısmındaki Uygulamayı Çalıştır düğmesine basın ve uygulamanın yüklendiği ikinci bir pencere açılır. Yükleme tamamlandıktan sonra, tam işlevsellik için yüklenen pencerenin sağ üst kısmında bulunan Tarayıcıda Aç'ı tıklatın.
- IR-TEx'e direnç veri setleri ekleme (Anopheles gambiae 15k Agilent dizisi kullanılarak oluşturulmuştur)
- Kullanılabilir veri setine aynı mikrodizi platformunda (A-MEXP-2196) oluşturulan yeni bir analiz edilmiş veri kümesi eklemek için uygulamayı indirin ve bölüm 2.1'de indirilen sıkıştırılmamış klasörü bulun.
- A-MEXP-2196 1'dekilimma analizinden elde edilen çıktıyı temsil eden Ek Dosya 1'iaçın. Excel'i kullanarak, H1 sütununda Fold_Changeyazın ve H2'de B2'nin günlük kıvrım değişikliği olduğu =2^B2yazın. Ham kıvrım değişiklikleri üretmek için bunu H sütununa uygulayın.
- A sütunu id, B sütunu H sütunundan (b sütunu B sütununu, b sütununu vurgula, sonra sağ tıklatma ve yapıştırma değerleri) ve C sütunu düzeltilmiş p değeridir. Diğer tüm sütunları silin ve sekme yle sınırlı dosya olarak kaydedin.
- Ek kodlama Dosyası 2'yi açın ve adım 2.2.3'te üretilen sekme-sınırlandırılmış sayfayı kullanarak çalıştırın.
NEWFILE_FC = c('ÜLKE','POZİsYON DURUMU','TÜRLER','Böcek Öldürücü')
NEWFILE_Q = c('ÜLKE','POZİsYON DURUMU','TÜRLer','BÖCEK Öldürücü')
NOT: Tek tırnak işaretleri içindeki alanlar, yeni veri kümesindeki bilgileri yansıtacak şekilde değiştirilmelidir. Maruz kalma durumu, örneklerin insektisit maruziyetinden (maruz/maruz kaldıktan sonra) toplanıp toplanmadığı dır. Böcek öldürücü: eğer 'maruz' ise, 'yok' kullanın. bkz. Fold_Changes.txt. diğer örneklerden meta veriler için. Yazım denetiminin tutarlı olduğundan emin olun. - Açık geography.txt, son işgal satırına kaydırın ve aşağıda seçin. Veri kümesinin adını, ardından Q ve NEWFILE_Q'ı sütun 1'de, örnek toplama alanının enlemini ve sütun 3'teki boylamını yazın. Değişiklikleri kaydedin.
- Veri setinde seçim için uygun olmayan yeni girişler (örneğin, Gambiya) kullanılırsa (bkz. Ingham ve ark. Ek Tablo 11),bunların koda eklenmesi gerekir. Bunu yapmak için RStudio'da IR-TEx.R'yi açın ve RStudio tarafından belirtildiği gibi 26 no'lu satırı bulun ve bu noktada aşağıdakilerin başlaması gerekir:
'kenar çubuğuPanel(....'.
NOT: Devam eden satırların her biri, adım 2.2.5'te Fold_Changes.txt'deki veri kümesi adının altındaki satırlara girilen meta veri öğesiile ilgilidir. - Yeni meta verileri eklemek için, tercih edilen meta veri satırının sonuna kaydırın ve 'selected=' terimini bulun. Hemen ardından bir virgül ve kapalı parantez olmalıdır; bu noktada, kapalı köşeli ayraç içindeki imleci tıklatın. Son apostrophe'den sonra virgül yazın, ardından bir kese işareti, ardından yeni meta veriler (örn. 'Gambiya') ve değişiklikleri kaydedin. Bir örnek için aşağıya bakın.
checkboxGroupInput('CountryInput','Select Relevant Countries',c('Burkina Faso','Cote D'Ivoire','Kamerun','Ekvator Ginesi','Zambiya','Tanzanya',Sudan','Uganda','Togo', 'Gambiya' (Gambiya'),seçilmiş=c('Burkina Faso','Cote D'Ivoire','Kamerun','Ekvator Ginesi','Zambiya','Tanzanya',Sudan','Uganda','Togo')) - Uygulamayı çalıştırın. Yeni meta veri girişi, ilgili başlık altında seçilmemiş bir onay kutusu olarak görünmelidir. Kullanıcı seçilmesini istiyorsa, aşağıda gösterildiği gibi seçili=c(...) sonra eklenmelidir:
checkboxGroupInput('CountryInput','Select Relevant Countries',c('Burkina Faso','Cote D'Ivoire','Kamerun','Ekvator Ginesi','Zambiya','Tanzanya',Sudan','Uganda','Togo', 'Gambiya'),selected=c('Burkina Faso','Cote D'Ivoire','Kamerun','Ekvator Ginesi','Zambiya','Tanzanya',Sudan','Uganda',Togo', 'Gambiya')) - A-MEXP-2196 üzerinde gerçekleştirilmeyen direnç veri kümeleri eklemek için bölüm 3'e bakın.
3. Farklı veri kümeleriyle kullanılmak üzere IR-TEx'i değiştirme
- Birden çok -omics platformlarında kullanma ve eksik verilerle devam
- Veri kümelerinde "0" ile devam etmek için: "0" kelimesinin belirli anlamı için veri kümesi kaynağına başvurun. "0" (konservatif) "NA" ile değiştirilir önerilir. Ham kıvrım değişikliklerinde (B/A) olduğu gibi, "0" deneysel b durumunda tespit edilmemiş bir sinyali gösterir. Deneysel durum A'nın önemli bir ifade göstermesi durumunda, kullanıcı küçük bir kat değişim değeri uygulayabilir.
- Aç Ek Dosya 2.txt, Uyhelji ve ark9uyarlanmış bir RNAseq dosyası . Bu dosya, yeni verilerin temel alınması gereken şablonu temsil eder: A = tanımlayıcı sütunu, B sütunu = ham kıvrım değişikliği ve C = ayarlanmış p değeri sütunu. Aşağıdaki adımları çalıştırmak için bu dosyayı kullanın.
- Tanımlayıcıları platformlar arasında tek bir sekme yle sınırlandıran dosyaya eşleştirmek için R kodunu çalıştırın, ardından verileri düzenleyin ve normalleştirin(Tamamlayıcı Kodlama Dosyası 2). Talimatlar dosya içinde yer almaktadır. Herhangi bir FILEPATH MacOS için "/" veya Windows için "//" ile ayrılır (bunları görünecekleri gibi "\" olarak değiştirin).
- Ek Kodlama Dosyası 2'nin sonunda üretilen dosyayı adım 3.1.5'te kullanılmak üzere tercih edilen bir konuma çıktı. Ek Kodlama Dosyası 2 yeni bir Fold_Changes.txt dosyası çıktı olacaktır. Orijinal dosyayı yedekle.
- Ek kodlama Dosyası 3'teyer alan kodu yürütün. FILEPATHolarak belirtilen klasörde FC_distribPlot.png adlı çıktı dosyasını bulun. Günlük2 kat değişiklik dağılımlarının veri kümeleri arasında neredeyse aynı olduğunu doğrulamak için günlük2 kat değişikliğinin dağılımlarını denetleyin.
- Ek dosyaları yeniden dizin ve yeni Fold_Changes.txtuyumluluğunu sağlamak için adım 2.2.6'daki yönergeleri izleyin.
- Tamamen yeni veri kümeleriyle kullanılmak üzere IR-TEx'i değiştirme
- RStudio'da IR-TEx.R'ı açın ve aşağıdakilerden başlayarak satırları (23-34) bulun:
'sekmesiPanel('
ve biten:
submitButton("View'ı güncelleştir", simge("yenileme"))
), - Aşağıdaki satırlarda bulunan AGAP008212-RA'yı yeni verilere ilgi nin bir dökümüyle değiştirin.
textInput('textInput','Transkript Kimliği',value='AGAP008212-RA'), - Aşağıdakilerden başlayan dört seçeneği bulun:
checkboxGroupInput(
Bu seçenekler, kullanıcının yeni verileri filtrelemek istediği önemli meta verileri temsil edecek şekilde değiştirilebilir. Her durumda, kullanıcı Belirli Ülkeler seçseçeneğini değiştirmelidir; Pozlama Durumunu Seçin; İlgili Türleri Seçin; ve verileri temsil etmek için Insektisit Sınıfı'nı seçin (yani, Doku Türünü Seçin; Cinsiyet seçin; Yaş Braketi Seçiniz; Hastalık Durumunu Seçin). - İlk c'denhemen sonra varolan seçenekleri değiştirmek için veri kümesiyle ilişkili meta verileri belirleyin ve girdiyi belirleyin. Her durumda, seçenekler konuşma işaretleri içinde yer alacak ve bir sonraki seçimden virgülle ayrılır. Son seçimden sonra, parantez kapatılmalıdır. Hastalık Durumunu Seç için bir örnek:
c('Enfekte', 'Enfekte Olmayan', 'Bilinmiyor') - Uygulamayı açtıktan sonra bu meta verilerden hangilerinin seçileceğini seçin. Bunlar seçili=c('. Hastalık Durumunu Seç için bir örnek:
selected=c('Enfekte', 'Enfekte Olmayan')
Bu, uygulamaya yalnızca ilk yüklemede bu ölçütlerle eşleşen veri kümelerini seçmesi talimatını verir. - Yeni bir veri tablosu oluşturmak için Fold_Changes.txt'de bulunan düzeni ve bölüm 2'deki yönergeleri izleyin. Meta verileri, kodda yazıldığı gibi 3.2.4 adımında özetlenen her bir değişiklikle değiştirin (R büyük/küçük harf duyarlıdır). Detoksifikasyon sütununa, giriş gen adlarına ve transkript türü sütununa, her transkript için gen açıklamaları girişi. Yeni veri kümeleri eklerken bölüm 3.2'yi izleyin.
- Eşleme deneysel gereksinimler ile ilgili değilse, aşağıdaki kod satırlarını bulun ve önüne '#' yerleştirin:
Satır 49-51:
br(),br(),
withSpinner(plotOutput("Coğrafya")),
textOutput('Geography_legend'),
Satır 493 başlangıç:
output$Coğrafya <- renderPlot({
Satır 602 bitişiçin:
output$Geography_legend <- renderText({
yapıştır("Sadece Önemli Transkriptler (p", as.expression("<="),"0.05): FC > 5 = Kırmızı, FC > 1 = Amber, FC < 1 = Yeşil",sep=")
})
- RStudio'da IR-TEx.R'ı açın ve aşağıdakilerden başlayarak satırları (23-34) bulun:
Sonuçlar
IR-TEx ile birlikte verilen Fold_Changes.txt dosyasını kullanarak, dirençli Anopheles coluzzii ve Anopheles gambiae veri setlerinde önemli ölçüde ifade edilen transkriptleri Côte D'Ivoire ve Burkina Faso'nun hassas kontrolleriyle karşılaştırdık. Bu ilgi 18 transkript(Tablo 1; bu arama Excel, R veya diğer programlar kullanılarak yapılabilir) elde etti. Bunlardan ikisi, bir ATPAz (AGAP006879) ve α-kristalin (AGAP007160), daha önce bildirilmiştir, eski piretropit direnci üzerinde önemli bir etkiye sahip1. Bu iki transkripte ek olarak, iki detoksifikasyon transkripti, GSTMS1 (FC= 1,95 ve 1,85) ve UGT306A2 (FCμ = 2,29 ve 2,28) mevcuttu.
bu transkriptlerden ikisinin qPCR doğrulaması(GSTMS1, detoksifikasyon transkripti; ve AGAP009110-RA, β-1,3-glukan bağlayıcı etki alanı içeren bilinmeyen, sivrisinek özgü transkript) daha önceaçıklandığıgibi 1 . Analiz, Ek Dosya 3'te açıklanan astar setleri kullanılarak yapıldı ve bu transkriptlerin, laboratuvara duyarlı N'Gousso(Şekil 4A)ile karşılaştırıldığında, Côte D'Ivoire (Tiassalé) ve Burkina Faso'dan (Banfora) çok dirençli bir popülasyonda önemli ölçüde düzenlendiğini gösterdi.
Her iki transkript de dirençli popülasyonların her birinde önemli bir artış gösterdiğinden, RNAi'nin indüklediği nakavt LSTM laboratuvarı Tiassalé kolonisinden sivrisinekler üzerinde gerçekleştirildi. Bu koloni Fildişi Sahili kökenli ve daha önce açıklandığı gibi, halk sağlığı kullanılan böcek tüm büyük sınıflaradayanıklıdır 1,10. GSTMS1 ekspresyonunun zayıflatılması, gfp enjekte edilen kontrollere göre deltamethrin maruziyetinden sonra mortalitede önemli bir artış (p = 0.021) ile sonuçlandı ve bu transkriptin piretropit direncindeki önemini gösterdi(Şekil 4B). Buna karşılık, AGAP009110-RA nakavt maruziyet sonrası mortalitede anlamlı (p = 0.082) bir değişiklik ile sonuçlanmadı(Şekil 4B).
GSTMS1 bir mikrozomal GST ve üç A. gambiya sivrisinek11bulunur biridir. GST'lerin epsilon ve delta sınıflarının üyeleri daha önce insektisit detoksifikasyon12,13,14karıştığı olmasına rağmen, bu pyrethroid direnci mikrozomal GSTs bir rol için bilgimizin ilk kanıtıdır15. Anopheles gambiae sl sivrisineklerde bu transkriptin putatif işlevini araştırmak için IR-TEx'teki ifade ve korelasyon tespit edilmiştir. GSTMS1, Bioko Adası hariç, bu türler için mevcut olan 21 veri kümesinin 20'sinde önemli ölçüde aşırı ifade edildi. Her yerde, aşırı ifade duyarlı popülasyonlara göre beş kattan daha azdı(Şekil 5).
Mikrozomal GST'ler büyük ölçüde potansiyel insektisit detoksiler olarak göz ardı edilmiştir gibi, çok az insektisit direnci rollerini hakkında bilinmektedir15. Diğer transkriptlerin birlikte korelasyonunu araştırarak, putatif fonksiyonlar aynı yollarda birlikte düzenleme veya katılım varsayımı ile açıklanabilir. Korelasyon ağındaki gücü en üst düzeye çıkarmak için, IR-TEx'te bulunan tüm mikrodizi veri kümeleri seçildi ve |r| >0.75 seçildi. Tablo 2, IR-TEx'in çıktısını gösterir.
Bu transkriptler DAVID'in fonksiyonel ek açıklama aracı8oxioredüktaz aktivitesi ve glikoz / karbonhidrat metabolizması zenginleştirilmiştir. Hem glukoz-6-fosfat dehidrogenaz ve sitatohidon gama-lyaz memeli hücrelerinde glutatyon düzeyini korumak16,17 ve böylece doğrudan GSTMS1ile bağlantı , bir glutatyon-S-transferaz. Katalaz, hücreleri piroit maruziyetinin bir yan ürünü olan reaktif oksijen türlerinin hasarından koruyan hızlı etkili bir oksidatif stres yanıtlayıcıdır. Valasiklovir hidrolaz memeli hücrelerinde detoksifikasyon bir rol oynayabilir bir hidrolazolduğunu 18. CYP4H17 korelasyon ağında da mevcuttur. Sitokrom p450'ler piretropid insektisitlerin doğrudan metabolizeleridir ve bu arıza ürünleri GST'ler tarafından daha da metabolize edilebilir. Son olarak, CYP4H17 A. funestus19piretropit direnci karıştığı olmuştur. Birlikte ele alındığında, bu veriler güçlü xenobiyotik detoksifikasyon GSTMS1 için bir rol destekler.
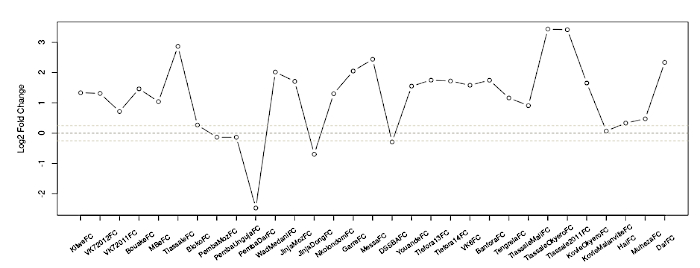
Şekil 1: Tüm veri kümelerinde AGAP002865-RA'nın2 kat değişimini belirtin. X ekseni, önceki biryayındaEk Tablo 1'de bulunan farklı veri kümelerini ayrıntılarıyla, y ekseni ise ilgi nin dökümündeki günlük2 kat değişikliği gösterir. Açık gri noktalı çizgiler, burada <0,8 veya kat değişimi >1.2 olarak ele alınan yaklaşık önem eşiklerini gösterir. Noktalı siyah çizgi 1'in kıvrım değişimini gösterir (yani, dirençli ve duyarlı popülasyonlar arasında ifade farkı yoktur). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
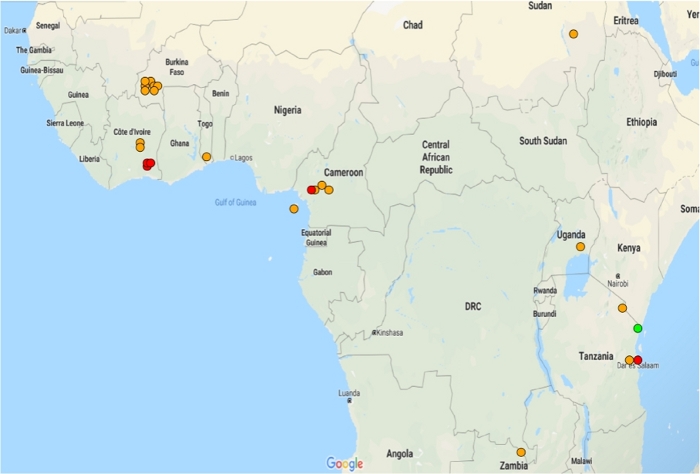
Şekil 2: Dirençli popülasyonlarda AGAP002865-RA'nın önemli diferansiyel ekspresyonu gösteren mikrodizilerin dağılımı. Kat değişiklikleri trafik ışık sisteminde temsil edilir: <1'in yeşil kıvrım değişimi, >1'in turuncu kat değişimi ve >5'in kırmızı kat değişimi. Yalnızca anlamlı (p ≤ 0.05) diferansiyel ekspresyonu olan veri kümeleri gösterilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
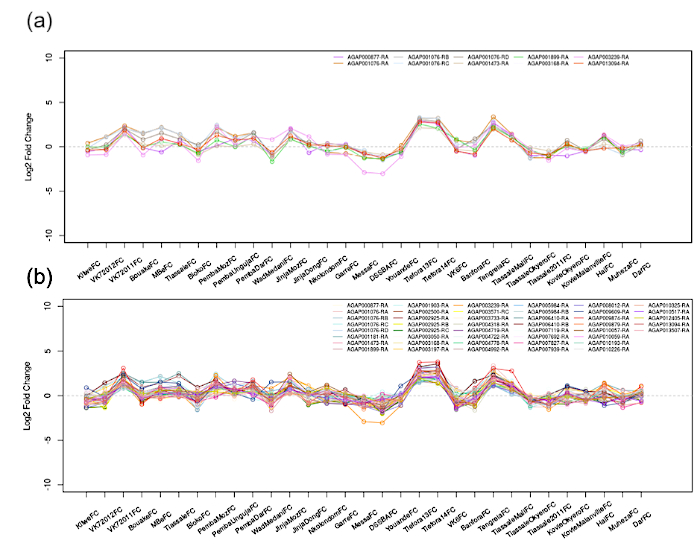
Şekil 3: AGAP001076-RA korelasyon ağları (CYP4G16). Çift yönlü korelasyonlar, 31 mikrodizi veri kümesindeki tüm transkriptler arasında hesaplanır ve kullanıcı tanımlı bir kesme uygulanır. Burada gösterilen (A) |r| > 0.9 ve (B) |r| > 0.8. Grafikte görüntülenen tüm transkriptler bu ölçütü karşılar ve AGAP001076-RA'nın ifade değişikliklerini takip eder. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
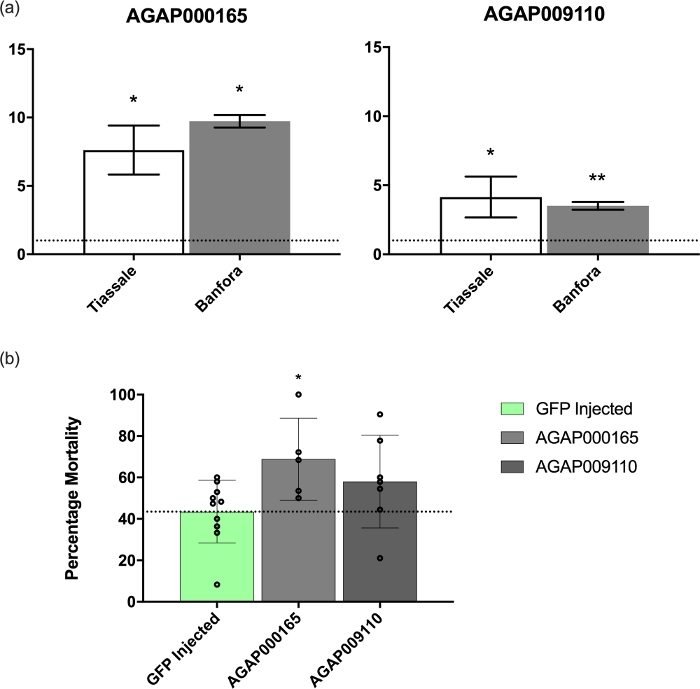
Şekil 4: GSTMS1 ve AGAP009110-RA zayıflaması üzerine mRNA ekspresyonu ve fenotip. (A) GSTMS1 ve AGAP009110-RA'nın côte D'Ivoire ve Burkina Faso'dan gelen iki çok dirençli An. coluzzii popülasyonunda ifade edilmistir. Seviyeler laboratuvara duyarlı An. coluzzii N'Gousso ile karşılaştırıldı. ANOVA tarafından post-hoc Dunnett testi ile hesaplanan önem düzeyleri. (B) GFP enjekte edilen kontrollere göre her iki transkriptin rnai kaynaklı zayıflaması. GSTMS1 zayıflaması deltamethrin maruziyetinden sonra mortalitede önemli bir artış gösterir (ANOVA tarafından post-hoc Tukey testi ile hesaplanır; *p ≤ 0.05, **p ≤ 0.01). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
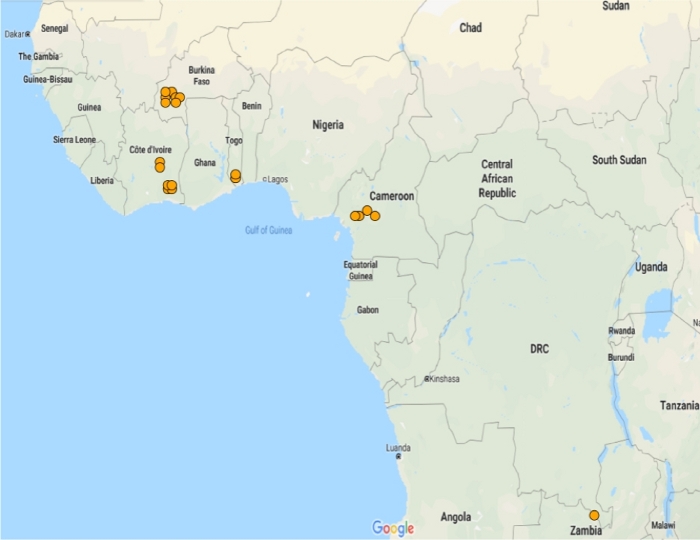
Şekil 5: Anopheles gambiae ve Anopheles coluzzii popülasyonlarında GSTMS1 ifadesi. Kullanılabilir mikrodizi veri kümelerinde GSTMS1'in önemli ölçüde diferansiyel ifadesini gösteren harita. GSTMS1, 21 mikrodizi veri kümesinin 20'sinde önemli ölçüde diferansiyel bulundu. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| Transkript Kimliği | Açıklama | Burkina Faso | Fildişi Sahili |
| AGAP006879-RA | ATPPase | 27.94 | 43.05 |
| AGAP007160-RB | a-kristalin | 11.49 | 10.58 |
| AGAP007160-RC | a-kristalin | 11.14 | 10.38 |
| AGAP007160-RA | a-kristalin | 9.78 | 9.84 |
| AGAP009110-RA | Bilinmeyen | 9.26 | 5.96 |
| AGAP007780-RA | NADH dehidrogenaz | 10.49 | 3.77 |
| AGAP006383-RA | oligosaccharyltransferaz kompleks alt birim beta | 3.69 | 5.57 |
| AGAP007249-RB | Uçuş | 4.61 | 3.86 |
| AGAP003357-RA | RAG1-aktive protein 1-benzeri protein | 4.31 | 4.05 |
| AGAP007249-RA | Uçuş | 4.48 | 3.46 |
| AGAP001998-RA | mRpS10 | 3.46 | 2.85 |
| AGAP007589-RA | UGT306A2 | 2.29 | 2.28 |
| AGAP000165-RA | GSTMS1 | 1.95 | 1.85 |
| AGAP002101-RA | isoleucyl-tRNA synthetase | 0.57 | 0.59 |
| AGAP002969-RA | asparaginyl-tRNA synthetase | 0.45 | 0.45 |
| AGAP004199-RA | solute taşıyıcı aile 5 (sodyum-çift monokarboksilat taşıyıcı), üye 8 | 0.35 | 0.48 |
| AGAP004684-RA | rRNA işleme proteini CGR1 | 0.36 | 0.22 |
| AGAP006414-RA | Cht8 | 0.024 | 0.36 |
Tablo 1: Transkriptler burkina Faso ve Fildişi Sahili popülasyonları arasında aynı kat farklılıkta yön değiştirir. Transkript kimliği, gen açıklaması ve an. coluzzii ve An. gambiyae popülasyonlarını temsil eden iki ülkeden her veri kümesi için ortalama kat değişimi.
| Korelasyon | Sistematik Ad | Transkript Türü |
| 1 | AGAP000165-RA | GSTMS1 |
| 0.82 | AGAP004904-RA | Katalaz |
| 0.76 | AGAP007243-RA | 26S proteease düzenleyici alt birim 8 |
| 0.79 | AGAP008358-RA | CYP4H17 |
| 0.76 | AGAP009436-RA | Valasiklovir hidrolaz |
| 0.75 | AGAP010739-RA | Glikoz-6-fosfat 1-dehidrogenaz |
| 0.85 | AGAP011172-RA | siationin gama-lyaz |
| 0.76 | AGAP012678-RA | Glikoz-6-fosfat 1-dehidrogenaz |
Tablo 2: Transkriptler GSTMS1ile ilişkilidir. Tablo, IR-TEx'teki GSTMS1 korelasyon ağının çıktısını |r| >0.75. Tablo, spearman'ın korelasyon, transkript kimliği ve gen açıklamasını gösterir.
Ek Dosya 1: A-MEXP-2196 dizisinden çıkış dosyası limma üzerinde analiz edildi. Dosya, ArrayExpress (E-MTAB-4043) ve başka bir önceki yayın1'dedaha ayrıntılı olarak açıklanan bir GFP kontrol dizisiyle karşılaştırıldığında Met knockdown'ından kaynaklanır. Sütunlar AGAP tanımlayıcıyı (SystematicName), günlük kıvrım değişikliği (logFC), günlük ifade değerleri (AveExpr), t-istatistik (t), düzeltilmemiş p değeri (P.Value), ayarlanmış p değerini (adj) temsil eder. P.Val) ve B istatistik (B)20. Bu dosyanın amaçları için, sivrisinekler Côte D'Ivoire Anopheles coluzzi ve bir toplama enlem ve boylam ile insektisitler maruz kalmamıştır -5.4 ve 6.0, sırasıyla. Bu dosyayı görüntülemek için lütfen buraya tıklayın (İndirmek için sağ tıklatın).
Ek Dosya 2: RNAseq deneyinden çıkış dosyası. RNAseq analizi Uyhelji ve ark.9'dan alınan ve %50 tuzluluk alabilen Anopheles sivrisineklerinin transkripsiyondaki değişiklikleri açıklar. Bu dosya yayının Tablo S2'sinden uyarlanmıştır ve AGAP tanımlayıcısı (SystematicID), ham kıvrım değişimi (Fold_Change) ve ayarlanmış p değeri (q_value) içerir. Bu dosyayı görüntülemek için lütfen buraya tıklayın (İndirmek için sağ tıklatın).
Ek Dosya 3: Temsilci sonuçları için Astar listesi. AGAP tanımlayıcısı, gen adı, dsRNA ileri, dsRNA ters, qPCR ileri ve her transkript için qPCR ters astar kümeleri. Bu dosyayı görüntülemek için lütfen buraya tıklayın (İndirmek için sağ tıklatın).
Tamamlayıcı kodlama Dosya 1. Bu dosyayı görüntülemek için lütfen buraya tıklayın (İndirmek için sağ tıklatın).
Tamamlayıcı kodlama Dosya 2. Bu dosyayı görüntülemek için lütfen buraya tıklayın (İndirmek için sağ tıklatın).
Tamamlayıcı kodlama Dosyası 3. Bu dosyayı görüntülemek için lütfen buraya tıklayın (İndirmek için sağ tıklatın).
Tartışmalar
Büyük veri transkripsiyonu, her deneysel durum için farklı olarak ifade edilen binlerce transkriptin listesini üretir. Bu deneylerin çoğu ilgili organizmalar ve fenotipler üzerinde gerçekleştirilir ve neredeyse sadece bağımsız deneyler olarak analiz edilir. Verileri bütünsel olarak ve teorik varsayımlar olmadan inceleyerek bu zengin veri kaynaklarını kullanmak 1) yeni aday transkriptlerinin tanımlanmasına yol açacak ve 2) in vivo1'dedoğrulanacak çok fazla bilgi olduğu için değerli verilerin atılmasını engelleyecektir.
IR-TEx, kullanıcılara birden fazla veri kümesini kolayca inceleyebilme, veri kümelerinde ki değişiklikleri görselleştirme ve ilişkili bilgileri indirme becerisine sahip sınırlı bir biyoinformatik arka plan sağlar1. IR-TEx her aramada birden fazla transkript aramayı desteklemese de, kullanıcılar yalnızca Excel, R veya diğer uygun programları kullanarak ilişkili Fold_Changes.txt dosyalarını inceleyebilir. IR-TEx'in daha fazla faydası, transkript işlevini, varsayımsal proteinlerin veya bilinmeyen fonksiyonlara sahip transkriptlerin girişini ve zenginleştirmeleri aramak için downstream yazılımının kullanımından kaynaklanmaktadır1.
Bu protokolde gösterilen örnekte, IR-TEx orijinal işlevine göre kullanılır. Burada, insektisit direnci ile ilişkili transkriptlerin araştırılmasına ve grafiklerin haritalandırılması yoluyla aşırı ve altı ifade dağılımının görselleştirilmesine olanak sağlar. Verilen transkriptlerin aşırı veya az ifadesinin gözlenen fenotip1'e (örneğin, böcek direnci) katkıda bulunup bulunmadığını belirlemek için ilgi dökümleri in vivo olarak doğrulanır. Burada, daha önce bildirilen1,bir veri kümesi nin ülkeye özgü olarak ilgi transkriptlerini tanımlamak için hipotez odaklı bir yaklaşımda kullanılabildiği gösterilmiştir. IR-TEx daha sonra 1) transkriptin ifadesini keşfetmek ve 2) her -omics veri setinde bulunan tüm transkriptler arasında çift yönlü bir korelasyon ağı uygulayarak transkriptin işlevini bağlamsallaştırmak için kullanılabilir. Burada, GSTMS1 detoksifikasyon karıştığı diğer transkript bir dizi ile ilişkili olduğu gösterilmiştir. Bu veriler (insektisit maruziyetinden sonra mortalitede önemli bir artışa yol açan transkriptin yıkılması ile birlikte) ksenobiyotik açıklıkta bu transkriptin önemini göstermektedir.
IR-TEx, web'de böcek direncine bağlı transkriptleri keşfetmek veya yerel uygulamaları kullanmak için değerli bir kaynaktır. Bu protokol, IR-TEx'in farklı omik platformların yanı sıra tamamen yeni veriler için nasıl değiştirilebildiğini gösterir. Kılavuz, birden çok omics platformlarından ve veri kümelerinden gelen verileri eksik verilerle entegre etmek için IR-TEx'in nasıl kullanılacağını ve yalnızca transkripsiyonel veri kümelerini araştıran herkes için yararlı olması için IR-TEx'in nasıl yeniden kodlanacağı ile birlikte gösteriş göstermektedir.
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Bu çalışma, MRC Beceri Geliştirme Bursu ile V.I. (MR/R024839/1) ve Royal Society Challenge Grant (CH160059) tarafından H.R.'a finanse edilmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Laptop with browser | Any | - | - |
| R Program | The R Project for Statistical Computing | - | https://www.r-project.org/ |
| R Studio | R Studio | - | https://www.rstudio.com/ |
Referanslar
- Ingham, V. A., Wagstaff, S., Ranson, H. Transcriptomic meta-signatures identified in Anopheles gambiae populations reveal previously undetected insecticide resistance mechanisms. Nature Communications. 9 (1), 5282(2018).
- Chang, W., Cheng, J., Allaire, J., Xie, Y., McPherson, J. shiny: Web Application Framework for R. , (2017).
- Bhatt, S., et al. The effect of malaria control on Plasmodium falciparum in Africa between 2000 and 2015. Nature. 526 (7572), 207-211 (2015).
- Ranson, H., Lissenden, N. Insecticide Resistance in African Anopheles Mosquitoes: A Worsening Situation that Needs Urgent Action to Maintain Malaria Control. Trends in Parasitology. 32 (3), 187-196 (2016).
- Donnelly, M. J., et al. Does kdr genotype predict insecticide-resistance phenotype in mosquitoes. Trends in Parasitology. 25 (5), 213-219 (2009).
- Stevenson, B. J., et al. Cytochrome P450 6M2 from the malaria vector Anopheles gambiae metabolizes pyrethroids: Sequential metabolism of deltamethrin revealed. Insect Biochemistry and Molecular Biology. 41 (7), 492-502 (2011).
- Müller, P., et al. Field-Caught Permethrin-Resistant Anopheles gambiae Overexpress CYP6P3, a P450 That Metabolises Pyrethroids. PLoS Genetics. 4 (11), 1000286(2008).
- Huang, D., et al. The DAVID Gene Functional Classification Tool: a novel biological module-centric algorithm to functionally analyze large gene lists. Genome Biology. 8 (9), 183(2007).
- Uyhelji, H. A., Cheng, C., Besansky, N. J. Transcriptomic differences between euryhaline and stenohaline malaria vector sibling species in response to salinity stress. Molecular Ecology. 25 (10), 2210-2225 (2016).
- Edi, C. V., Benjamin, K. G., Jones, C. M., Weetman, D., Ranson, H. Multiple-Insecticide Resistance in Anopheles gambiae Mosquitoes, Southern Côte d’Ivoire. Emerging Infectious Diseases. 18 (9), 1508-1511 (2012).
- Ding, Y., Ortelli, F., Rossiter, L., Hemingway, J., Ranson, H. The Anopheles gambiae glutathione transferase supergene family: annotation, phylogeny and expression profiles. BMC Genomics. 4 (1), 1-16 (2003).
- Enayati, A. A., Ranson, H., Hemingway, J. Insect glutathione transferases and insecticide resistance. Insect Molecular Biology. 14 (1), 3-8 (2005).
- Ranson, H., et al. Identification of a novel class of insect glutathione S-transferases involved in resistance to DDT in the malaria vector Anopheles gambiae. The Biochemical Journal. 359, 295-304 (2001).
- Riveron, J. M., et al. A single mutation in the GSTe2 gene allows tracking of metabolically based insecticide resistance in a major malaria vector. Genome Biology. 15 (2), 27(2014).
- Pavlidi, N., Vontas, J., Van Leeuwen, T. The role of glutathione S-transferases (GSTs) in insecticide resistance in crop pests and disease vectors. Current Opinion in Insect Science. 27, 97-102 (2018).
- Salvemini, F., et al. Enhanced glutathione levels and oxidoresistance mediated by increased glucose-6-phosphate dehydrogenase expression. Journal of Biological Chemistry. 274 (5), 2750-2757 (1999).
- Deplancke, B., Gaskins, H. R. Redox control of the transsulfuration and glutathione biosynthesis pathways. Current Opinion in Clinical Nutrition & Metabolic Care. 5 (1), (2002).
- Puente, X. S., López-Otn, C. Cloning and expression analysis of a novel human serine hydrolase with sequence similarity to prokaryotic enzymes involved in the degradation of aromatic compounds. Journal of Biological Chemistry. 270 (21), 12926-12932 (1995).
- Riveron, J. M., et al. Genome-wide transcription and functional analyses reveal heterogeneous molecular mechanisms driving pyrethroids resistance in the major malaria vector Anopheles funestus across Africa. G3: Genes, Genomes, Genetics. 7 (6), 1819-1832 (2017).
- Smyth, G. K. Linear models and empirical bayes methods for assessing differential expression in microarray experiments. Statistical Applications in Genetics and Molecular Biology. 3 (1), 3(2004).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır