Method Article
IR-TEx: Uma ferramenta de integração de dados de código aberto para transcrição de big data projetada para o Vector Anopheles gambiae da malária
Neste Artigo
Resumo
Ir-TEx explora perfis transcricionais relacionados à resistência inseticida na espécie Anopheles gambiae. Estão aqui instruções completas para o uso do aplicativo, modificações para explorar vários conjuntos de dados transcriptômicos e usar a estrutura para criar um banco de dados interativo para coletas de dados transcritas de qualquer organismo, gerados em qualquer plataforma.
Resumo
Ir-TEx é uma aplicação escrita em Shiny (um pacote R) que permite a exploração da expressão de (bem como atribuir funções para) transcrições cuja expressão está associada com fenótipos de resistência a inseticidas em mosquitos Anopheles gambiae. O aplicativo pode ser usado on-line ou baixado e usado localmente por qualquer pessoa. A aplicação local pode ser modificada para adicionar novos conjuntos de dados de resistência a inseticidas gerados a partir de várias plataformas de omics. Este guia demonstra como adicionar novos conjuntos de dados e lidar com dados em falta. Além disso, o IR-TEx pode ser recodificado de forma completa e fácil para conjuntos de dados de uso-ômicos de quaisquer dados experimentais, tornando-o um recurso valioso para muitos pesquisadores. O protocolo ilustra a utilidade do IR-TEx na identificação de novos candidatos à resistência ao inseticida usando a transferência de glutationa microsômica, GSTMS1,como exemplo. Esta transcrição é regulada em várias populações resistentes a piretróides da Costa do Marfim e Burkina Faso. A identificação de transcrições co-correlacionadas fornece uma introspecção mais adicional nos papéis putativos deste gene.
Introdução
A capacidade de medir a expressão de um grande número de transcrições simultaneamente através de plataformas de microarray e tecnologia RNAseq resultou na geração de vastos conjuntos de dados associando a expressão de transcrição com um fenótipo específico em organismos modelo e não modelo. Esses conjuntos de dados são um recurso extremamente rico para os pesquisadores, o poder do que pode ser aumentado combinando conjuntos relevantes em uma abordagem de integração de big data. No entanto, esta metodologia é limitada àqueles com habilidades particulares de bioinformática. Descrito aqui é um programa, IR-TEx (publicado anteriormente pela Ingham et al.1)que está escrito em um pacote R chamado Shiny2 e permite que os usuários com pouco treinamento de bioinformática para integrar e interrogar esses conjuntos de dados com relativa facilidade.
IR-TEx, encontrado em http://www.lstmed.ac.uk/projects/IR-TEx, foi escrito para explorar transcrições associadas com a resistência inseticida em Anopheles gambiae, o principal vetor de malária Africano1. A malária é uma doença parasitária causada por espécies de Plasmodium, transmitida s seres humanos através das picadas de mosquitos anopheles fêmeas. A segmentação do vetor de mosquitos com inseticidas provou ser o meio mais eficaz de prevenir a morbidade e mortalidade relacionadas à malária na África. A ampliação de ferramentas (ou seja, redes inseticidas de longa duração) também tem sido fundamental nas reduções drásticas nos casos de malária desde 20003. Com um número muito limitado de inseticidas disponíveis, há uma forte pressão evolutiva sobre os mosquitos, e a resistência agora é generalizada nos vetores africanos da malária4.
Além disso, as mutações do local alvo5 e a liberação metabólica de inseticidas6,7 continuam a ser os principais mecanismos estudados de resistência, mas outros mecanismos potentes resistentes estão emergindoagora 1. Muitos desses novos mecanismos não foram previamente associados à resistência aos inseticidas, mas foram detectados pela busca de padrões comuns de expressão gênica em várias populações resistentes usando o aplicativo IR-TEx e posteriormente validados funcionalmente por abordagens genômicas1.
Descrito aqui é uma abordagem passo a passo para o uso de IR-TEx, tanto na web e quando instalado localmente. O protocolo descreve como novos conjuntos de dados de resistência a inseticidas podem ser integrados no pacote existente e explica como operar com dados em falta. Finalmente, ele descreve como usar esse software com outros conjuntos de dados -ômicos que não estão relacionados à resistência aos inseticidas, combinando assim dados de diferentes abordagens -ômicos, ao mesmo tempo em que operam com valores ausentes e normalização para que os dados sejam comparáveis.
Protocolo
1. Usando o aplicativo web IR-TEx
- Executando o aplicativo em um navegador da web
- Abra o aplicativo web IR-TEx seguindo o link na parte inferior da página encontrado no http://www.lstmed.ac.uk/projects/IR-TEx.
- Uma vez que a página web tenha inicializado, clique no botão do aplicativo na parte superior da página, que exibirá o aplicativo e as saídas associadas.
- Leia cada saída relacionada à entrada padrão de AGAP008212-RA (CYP6M2)na caixa de identificação transcrição com as seguintes condições: Um. conjuntos de dados coluzzii que são (i) expostos a inseticidas piretróides ou (ii) não expostos a qualquer classe inseticida, e transcrições associadas com uma correlação de |r| E 0,98.
- Explorando a expressão de uma transcrição de interesse
- Para selecionar uma transcrição de interesse, insera a identificação de transcrição na caixa de identificação transcrição, lembrando que as transcrições terminam em -RX dependente de isoforme de interesse.
- Selecione os conjuntos de dados para interrogar, marcando as caixas relevantes para (i) Países; (ii) Status de exposição, (iii) Espécie de interesse; e (iv) Classe de interesse inseticida, tudo ao mesmo tempo garantir que esses critérios resultem em conjunto de dados incluído >1 (ver Tabela Suplementar 1 em Ingham et al.1).
NOTA: (iii) refere-se ao membro do complexo de espécies An. gambiae que o usuário interessado. Atualmente, os dados estão disponíveis para An. coluzzii e An. arabiensis. - Clique na Atualização Veja na parte inferior do menu de seleção ou devoluçãode imprensa, ignorando o valor da correlação absoluta (por enquanto).
- Dê tempo ao aplicativo para atualizar.
- Leia o primeiro gráfico como: log2 vezes a mudança entre uma população resistente e laboratório suscetível a população de mosquitos da transcrição de interesse em cada conjunto de dados que atende aos critérios selecionados na etapa 1.2 (Figura 1). Os detalhes de todos os conjuntos de dados podem ser encontrados em Ingham et al.1.
- Leia as informações abaixo do gráfico como: a dobra muda entre os mosquitos resistentes e suscetíveis para cada conjunto de dados relevante, além dos p-valores corrigidos (Q). Cada linha representa sondas individuais no microarray. A metodologia para a exposição gráfica foi relatada previamente1.
- Leia a tabela adicional abaixo como o número de experimentos em que a transcrição de interesse é significativa, bem como o número total de experimentos que correspondem aos critérios selecionados na etapa 1.2.
- Para baixar os dados em formato separado de guia, clique no botão Download as duas tabelas. Isso permite que o usuário explore dados de forma mais fácil usando um programa como o Excel.
- Interprete o mapa da seguinte forma: cada ponto representa os locais aproximados de coleta de mosquitos resistentes em cada conjunto de dados em que a transcrição do interesse é expressa de forma diferencial. As cores seguem um sistema de semáforo que é explicado no aplicativo(Figura 2).
- Para as etapas 1.2.5 e 1.2.8, salve as saídas gráficas clicando direito, clicando na imagem de Salvar como...e escolhendo uma pasta apropriada.
NOTA: Na instância de um erro de saída pelo aplicativo, é provável que nenhum conjunto de dados corresponda aos critérios inseridos. Verifique a Tabela Suplementar 1 em Ingham et al.1 se isso ocorrer.
- Identificação de funções/vias putativas de transcrição de interesse
- Correlações (valor mínimo r2 inserido) dos padrões de expressão de transcrições em vários conjuntos de dados podem ser usadas para prever a função de transcrição e potencialmente elucidar transcrições coregulares da mesma via. Usando o exemplo de Ingham et al.1 (AGAP001076-RA; CYP4G16), siga os passos 1.2.1-1.2.2 na seção acima, selecionando todos os conjuntos de dados para o poder máximo.
- Antes de clicar na Atualização,mova o controle deslizante de valor de correlação absoluta para 0,85 e clique na Visualização de atualização ou no retorno daimprensa.
- Examine a tabela de correlação (tabela mais inferior) para encontrar as múltiplas transcrições que agora são exibidas e estão correlacionadas (|r| = 0,85) com a transcrição inserida.
- Manipular o controle deslizante de valor de correlação absoluta e observar quaisquer alterações no gráfico e na tabela mais inferiores; as saídas do passo 1.3.2 permanecerão inalteradas. Como mostrado na Figura 3 (|r| > 0.9, |r| > 0.8), abaixar a stringency do valor da correlação mostrará mais transcrições mas introduzirá mais ruído.
- Leia a tabela abaixo da saída gráfica, que (além dos parâmetros descritos na etapa 1.2.6) contém o valor de correlação para cada transcrição.
- Para baixar os dados em um formato separado por guia, clique no botão Download.
- A análise funcional do enriquecimento pode ser executada na lista de identificação de transcrição baixada usando a análise8de DAVID. Uma vez no site da DAVID (encontrado no https://david.ncifcrf.gov/),selecione Análise Funcional. Colar a lista completa de genes, usando identificações gênicas [identificador sem o -RX, que pode ser feito em destaque, inserindo uma coluna à direita da Identificação Sistemática e digitando =ESQUERDA (X1,10), onde X1 é a célula de identificação sistemática]. Selecione o identificador como VectorBase_ID e lista de genes e clique na Lista de Envio.
- Clique no botão de agrupamento de anotação funcional para produzir uma visão geral dos enriquecimentos encontrados nesta rede de correlação, permitindo que uma função potencial seja atribuída à transcrição. Explore enriquecimentos detalhados, examinando as diferentes categorias e clicando nos botões + para cada um e, posteriormente, clicando no Gráfico.
2. Baixar e implementar o IR-TEx localmente
- Download e execução ir-TEx
- Vá para o link encontrado em http://github.com/LSTMScientificComputing/IR-TEx; e clique em Clone ou download | Download Zip. Direto para uma pasta de escolha e descompactar o arquivo em que a pasta.
- Baixe a versão mais recente do software R para o sistema operacional apropriado a partir do link encontrado em http://cran.r-project.org/mirrors.html. Instale o programa.
- Baixe e instale o mais recente software R Studio, novamente para o sistema operacional apropriado a partir do link encontrado no http://www.rstudio.com/products/rstudio/download/.
- Uma vez instalado, aberto R Studio | Arquivo de codificação suplementar 1 e executar cada linha para configurar o sistema para IR-TEx.
- Uma vez que todos os pacotes são instalados com sucesso e atualizados, conforme necessário, vá para o Arquivo | Aberto,localizar IR-TEx.R, destaque, e aberto. Isso agora deve ser visível na janela superior do R Studio.
- Para executar o aplicativo, pressione o botão Run App no canto superior direito da janela, e uma segunda janela aparecerá na qual o aplicativo carregará. Uma vez que o carregamento esteja completo, para funcionalidade completa clique no Open no Navegador localizado no canto superior direito da janela carregada.
- Adicionando conjuntos de dados de resistência ao IR-TEx (gerado usando anopheles gambiae 15k Agilent array)
- Para adicionar um novo conjunto de dados analisado gerado na mesma plataforma de microarray (A-MEXP-2196) ao conjunto de dados disponível, baixe o aplicativo e localize a pasta descompactada baixada na seção 2.1.
- Arquivo Adicional Aberto 1,que representa uma saída de uma análise de limusine em A-MEXP-2196 1. Usando o Excel, na coluna H1, escreva Fold_Change,e em H2, escreva =2^B2, no qual o B2 é a mudança de dobra de registro. Aplique isso ao longo da coluna H para produzir alterações de dobra bruta.
- Organize o Arquivo Adicional 1, de tal forma que a coluna A é a identificação, a coluna B é a alteração da dobra da coluna H (coluna h de cópia, coluna de destaque B e valores de corte e clique direito) e a coluna C é o valor p ajustado. Excluir todas as outras colunas e economizar como um arquivo delimitado a guia.
- Arquivo de codificação suplementar aberto 2 e executado usando a folha delimitada de guiaproduzida na etapa 2.2.3.
NEWFILE_FC = c ('COUNTRY','STATUS DE EXPOSIÇÃO','ESPÉCIE','INSETICIDE')
NEWFILE_Q = c ('COUNTRY','STATUS DE EXPOSIÇÃO','ESPÉCIE','INSETICIDE')
NOTA: Campos dentro de aspas únicas devem ser alterados para refletir informações do novo conjunto de dados. O estado de exposição refere-se a se as amostras foram coletadas após a exposição ao inseticida (exposta/não exposta). Inseticida: se 'não expostos', use 'nenhum'. Veja Fold_Changes.txt. para metadados de outras amostras. Certifique-se de que a ortografia é consistente. - Abra geography.txt,rolar para a linha ocupada final, e selecione abaixo. Digite o nome do conjunto de dados, seguido por Q e NEWFILE_Q na coluna 1, a latitude do site de coleta de amostras na coluna 2 e a longitude na coluna 3. Salve as mudanças.
- Se quaisquer novas entradas forem usadas (ou seja, Gâmbia), que não estão disponíveis para seleção no conjunto de dados (ver Ingham et al. Tabela Suplementar 11),estas precisarão ser adicionadas ao código. Para fazer isso, abra o IR-TEx.R no RStudio e localize a linha 26, conforme indicado pela RStudio, momento em que o seguinte deve começar:
'sidebarPanel (....'.
NOTA: Cada uma das linhas de processo refere-se a um item de metadados inseridos nas linhas abaixo do nome do conjunto de dados em Fold_Changes.txt na etapa 2.2.5. - Para adicionar os novos metadados, role até o final da linha dos metadados de escolha e localize o termo 'selecionado='. Imediatamente após isso deve ser uma comma e suporte fechado; neste ponto, clique no cursor dentro do suporte fechado. Após o apóstrofo final, digite uma comma, seguido por um apóstrofo, seguido pelos novos metadados (por exemplo, 'Gâmbia'), e salvar as mudanças. Veja abaixo um exemplo.
caixa de scaixaGroupInput ('CountryInput','Selecione países relevantes',c('Burkina Faso','Cote D'Ivoire','Camarões','Guiné Equatorial','Zâmbia','Tanzânia''sudan','Uganda','Togo', 'Gâmbia',selected=c('Burkina Faso','Cote D'Ivoire','Camarões','Guiné Equatorial','Zâmbia','Tanzânia','Sudão','Uganda','Togo')) - Executar o aplicativo. A entrada de novos metadados deve aparecer como uma caixa de tiquetaque não selecionada o título relevante. Se o usuário quiser que ele seja selecionado, ele deve ser adicionado após o selecionado =c (..., como mostrado abaixo:
caixa de scaixaGroupInput ('CountryInput','Selecione países relevantes',c('Burkina Faso','Cote D'Ivoire','Camarões','Guiné Equatorial','Zâmbia','Tanzânia''sudan','Uganda','Togo', 'Gâmbia',selected=c('Burkina Faso','Cote D'Ivoire','Camarões','Guiné Equatorial','Zâmbia','Tanzânia','Sudão','Uganda','Togo', 'Gâmbia')) - Para adicionar conjuntos de dados de resistência não realizados no A-MEXP-2196, consulte a seção 3.
3. Modificação do IR-TEx para uso com diferentes conjuntos de dados
- Uso em várias plataformas de omics e prosseguindo com dados em falta
- Para prosseguir com "0" em conjuntos de dados: consulte a fonte do conjunto de dados para o significado específico de "0". Recomenda-se que "0" é (conservadoramente) substituído por "NA". Tal como acontece com as alterações de dobra bruta (B/A), "0" indica um sinal não detectado na condição experimental B. No caso de que a condição experimental A exibe expressão substancial, o usuário pode aplicar um valor de alteração de pequena seleção.
- Arquivo Adicional Aberto 2.txt,um arquivo RNAseq adaptado de Uyhelji et al.9. Este arquivo representa o modelo em que novos dados devem ser baseados: coluna A = identificador, coluna B = alteração de dobra bruta e coluna C = valor p ajustado. Use este arquivo para percorrer as etapas abaixo.
- Executar o código R para combinar identificadores em um único arquivo delimitado por guia em todas as plataformas e, em seguida, organizar e normalizar os dados (Arquivo deCodificação Suplementar 2). As instruções estão contidas no arquivo. Qualquer FILEPATH será separado por "/" para MacOS ou "//" para Windows (alterá-los de "\", como eles aparecerão).
- Saída do arquivo produzido no final do Arquivo de Codificação Suplementar 2 para um local de escolha para uso na etapa 3.1.5. O Arquivo de Codificação Suplementar 2 produzirá um novo arquivo Fold_Changes.txt. Fazer backup do arquivo original.
- Executar o código contido no Arquivo de codificação suplementar 3. Encontre o arquivo de saída chamado FC_distribPlot.png na pasta especificada como FILEPATH. Verifique as distribuições da alteração de log2 vezes para verificar se as distribuições de alteração de log2 vezes são quase idênticas entre os conjuntos de dados.
- Siga as instruções do passo 2.2.6 para eitar arquivos adicionais e garantir a compatibilidade do novo Fold_Changes.txt.
- Modificação do IR-TEx para uso com conjuntos de dados completamente novos
- Abra ir-TEx.R em RStudio e localizar as linhas (23-34) começando com:
'tabPanel('
e terminando em:
submitButton ("Update View", ícone ("atualização"))
), - Alterar o AGAP008212-RA encontrado nas linhas abaixo para uma transcrição de interesse nos novos dados.
textInput ('textInput','Transcript ID',value='AGAP008212-RA'), - Localizar as quatro opções que começam com:
caixa de chequesGroupInput(
Essas opções podem ser modificadas para representar metadados importantes pelos outros. Em cada instância, o usuário deve alterar os países relevantes selecionados; Selecione o statusde exposição; Selecione espécies relevantes; e Selecione classe de inseticida para ser representativo dos dados (ou seja, selecione tipode tecido; Selecione sexo; Selecione faixa etária; Selecione o estado da doença). - Identifique os metadados associados ao conjunto de dados e entrada para substituir as opções existentes imediatamente após o primeiro c('. Em cada caso, as opções serão contidas dentro das marcas de fala e separadas da próxima seleção por uma comma. Após a seleção final, o suporte deve ser fechado. Um exemplo para o status da doença selecionada é:
c('Infectado', 'Não infectado', 'Desconhecido') - Escolha qual desses metadados será selecionado ao abrir o aplicativo. Estes podem ser alterados modificando as opções após selected=c(' Um exemplo para o status da doença selecionada é:
selected=c('Infectado', 'Não infectado')
Isso instruirá o aplicativo a selecionar apenas conjuntos de dados que correspondam a esses critérios no carregamento inicial. - Para criar uma nova tabela de dados, siga o layout encontrado em Fold_Changes.txt e instruções na seção 2. Alterar os metadados para cada alteração respectiva descrita na etapa 3.2.4, exatamente como escrito no código (R é sensível ao caso). Na coluna de desintoxicação, nomes de genes de entrada, e na coluna do tipo transcrição, descrições de genes de entrada para cada transcrição. Siga a seção 3.2 ao adicionar novos conjuntos de dados.
- Se o mapeamento não for relevante para os requisitos experimentais, localizar as seguintes linhas de código e colocar '#' na frente:
Linhas 49-51:
br(),br(),
withSpinner (plotOutput("Geografia")),
textOutput ('Geography_legend'),
Linhas 493 a partir:
saída$Geografia <- renderPlot ({
Para a linha 602 terminando:
saída$Geography_legend <- renderText ({
pasta ("Transcrições significativas Apenas (p", as.expression ("<="),"0.05): FC > 5 = Vermelho, FC > 1 = Amber, FC < 1 = Verde",sep=")
})
- Abra ir-TEx.R em RStudio e localizar as linhas (23-34) começando com:
Resultados
Usando o arquivo Fold_Changes.txt incluído com IR-TEx, comparamos transcrições que foram significativamente expressas de forma diferencial em conjuntos de dados resistentes anopheles coluzzii e Anopheles gambiae a controles suscetíveis da Costa do Marfim e Burkina Faso. Isso rendeu 18 transcrições de interesse (Tabela 1; essa pesquisa pode ser realizada usando excel, R ou outros programas). Dois destes, um ATPase (AGAP006879) e α-cristalina (AGAP007160), foram relatados previamente, com o anterior que tem um efeito significativo na resistência pyrethroid1. Além dessas duas transcrições, duas transcrições de desintoxicação, o GSTMS1 (FCμ = 1,95 e 1,85) e o UGT306A2 (FCμ = 2,29 e 2,28) estavam presentes.
a validação qPCR de duas dessas transcrições (GSTMS1, uma transcrição de desintoxicação; e AGAP009110-RA, uma transcrição desconhecida, específica para mosquitos contendo um domínio de ligação β-1,3 glucano) foram realizadas como descrito anteriormente1. A análise foi realizada usando conjuntos de primer descritos no Arquivo Adicional 3 e mostrou que essas transcrições foram significativamente regulamentadas em uma população multirresistente da Costa do Marfim (Tiassalé) e outra de Burkina Faso (Banfora), em comparação com o n'Gousso ( Figura4A).
Como ambas as transcrições mostraram uma resregulamentação significativa em cada uma das populações resistentes, o knockdown induzido pela RNAi foi realizado em mosquitos da colônia tiassalé do laboratório LSTM. Esta colônia é originária da Costa do Marfim e é resistente a todas as principais classes de inseticida usadas na saúde pública, como descrito anteriormente1,10. Atenuação da expressão de GSTMS1 resultou em um aumento significativo (p = 0,021) na mortalidade após a exposição à deltametrina em comparação com os controles injetados por GFP, demonstrando a importância desta transcrição na resistência piretróide (Figura 4B). Por outro lado, o knockdown aGAP009110-RA não resultou em nenhuma mudança significativa (p = 0,082) na mortalidade após a exposição(Figura 4B).
GSTMS1 é um GST microssômico e é um dos três encontrados em mosquitos A. gambiae 11. Embora os membros das classes de epsilon e delta de GSTs tenham sido previamente implicados na desintoxicação de inseticidas12,13,14,esta é a primeira evidência para o nosso conhecimento para um papel de GSTs microssômicos na resistência piretróide15. Para explorar a função putativa desta transcrição em mosquitos anopheles gambiae sl, a expressão e correlação no IR-TEx foram identificadas. O GSTMS1 foi significativamente superexpresso em 20 dos 21 conjuntos de dados disponíveis para essas espécies, com exceção da Ilha Bioko. Em cada local, a superexpressão foi inferior a cinco vezes em comparação com as populações suscetíveis(Figura 5).
Como GSTs microssômicos têm sido amplamente ignorados como potenciais desintoxicantes de inseticidas, pouco se sabe sobre seu papel na resistência ao inseticida15. Ao explorar a co-correlação de outras transcrições, funções putativas podem ser elucidadas através da assunção de co-regulação ou envolvimento nos mesmos caminhos. Para maximizar o poder na rede de correlação, todos os conjuntos de dados de microarray presentes no IR-TEx foram selecionados e um |r| | de >0.75 foi selecionado. A tabela 2 mostra a saída do IR-TEx.
Estas transcrições são enriquecidas na atividade oxioreductase e no metabolismo da glicose/hidrato de carbono na ferramenta funcional8da anotação de DAVID. Tanto a deshidrogenação de sódio de glicose-6-fosfato quanto a citotia gama-lyase mantêm o nível de glutationa em células de mamíferos16,17 e, assim, se ligam diretamente ao GSTMS1,uma glutationa-S-transferase. Catalase é um socorrista de estresse oxidativo de ação rápida que protege as células de danos reativos de espécies de oxigênio, um subproduto da exposição à piretróide. Valaciclovir hidrolase é uma hidrolase que pode desempenhar um papel na desintoxicação em células de mamíferos18. Cyp4H17 também está presente na rede de correlação. Os p450s cytochrome são metabolizers diretos de insecticidas piretróides, e estes produtos da avaria podem ser metabolizados mais por GSTs. Finalmente, CYP4H17 tem sido implicado na resistência piretróide em A. funestus19. Em conjunto, esses dados apoiam fortemente um papel do GSTMS1 na desintoxicação xenobiótica.
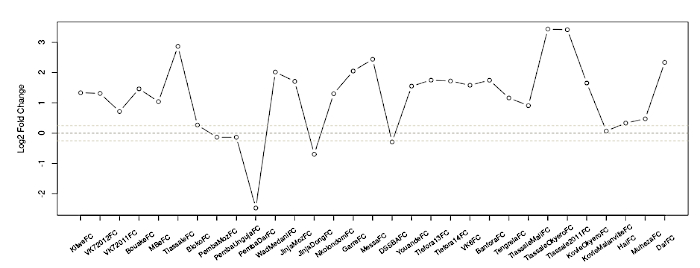
Figura 1: Log2 vezes alteração do AGAP002865-RA em todos os conjuntos de dados. O eixo x detalha os diferentes conjuntos de dados, informações para as quais podem ser encontradas na Tabela Suplementar 1 em uma publicação anterior1,e o eixo y mostra a alteraçãode 2 vezes na transcrição do interesse. As linhas pontilhadas cinza-claro indicam limites aproximados para o significado, tomado aqui para ser uma mudança de dobra de <0.8 ou mudança dobra de >1.2. A linha preta pontilhada indica uma mudança de dobra de 1 (ou seja, nenhuma diferença de expressão entre as populações resistentes e suscetíveis). Clique aqui para ver uma versão maior deste número.
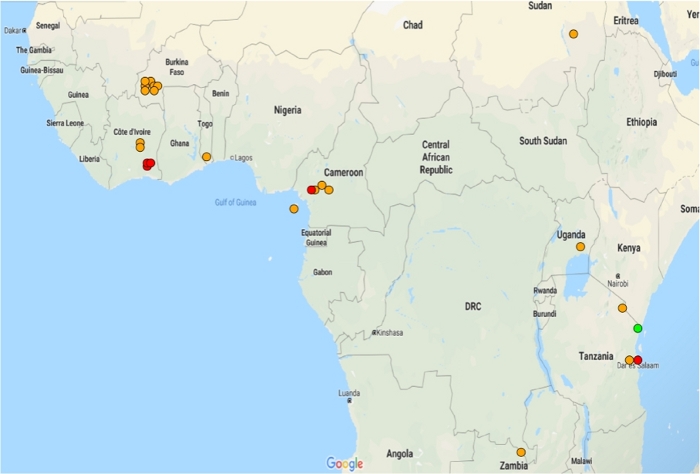
Figura 2: Distribuição de microarrays mostrando expressão diferencial significativa de AGAP002865-RA em populações resistentes. Mudanças de dobra são representadas em um sistema de semáforo: mudança de dobra verde de <1, mudança de dobra laranja de >1 e mudança de dobra vermelha de >5. Apenas conjuntos de dados com expressão diferencial significativa (p≤ 0,05) são mostrados. Clique aqui para ver uma versão maior deste número.
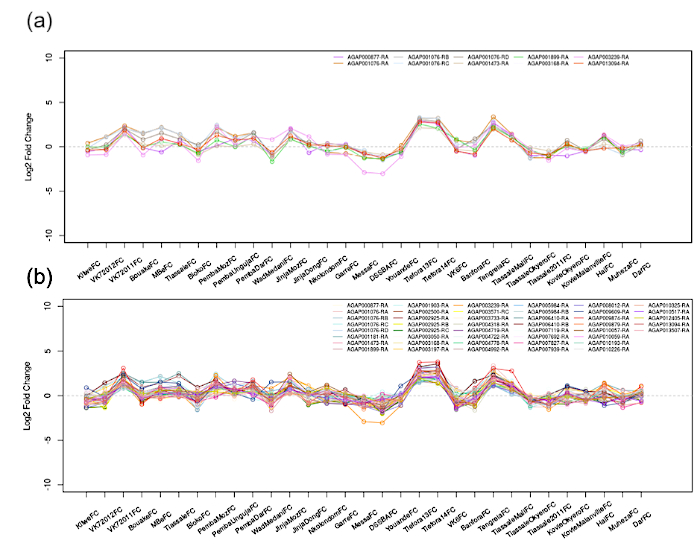
Figura 3: Redes de correlação de AGAP0001076-RA (CYP4G16). As correlações emparelhais são calculadas em todas as transcrições nos 31 conjuntos de dados de microarray, com um corte definido pelo usuário aplicado. Mostrado aqui é (A)|r | > 0,9 e (B) |r| E gt, 0,8. Todas as transcrições exibidas no gráfico atendem a esse critério e seguem as mudanças de expressão do AGAP0001076-RA. Clique aqui para ver uma versão maior deste número.
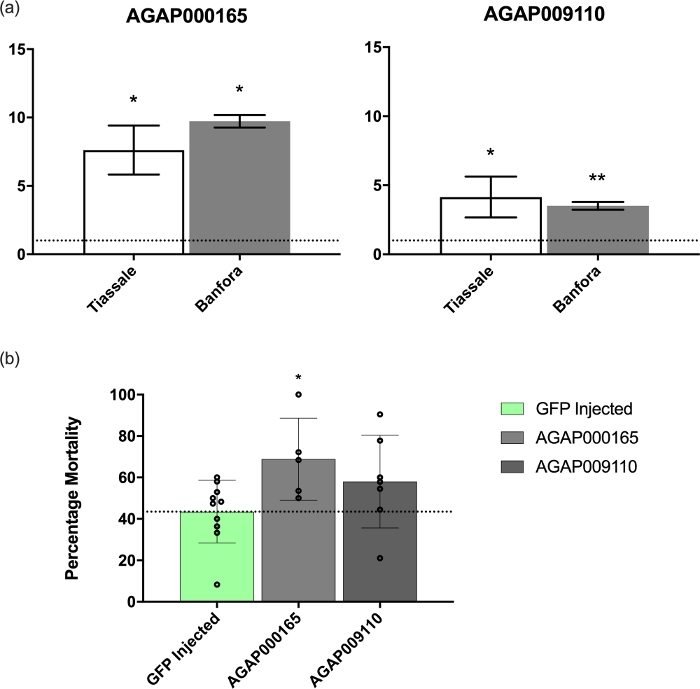
Figura 4: expressão mRNA e fenótipo após atenuação de GSTMS1 e AGAP009110-RA. (A) expressão mRNA de GSTMS1 e AGAP009110-RA em duas populações multirresistentes an. coluzzii da Costa do Marfim e Burkina Faso, respectivamente. Os níveis foram comparados com o laboratório suscetível An. coluzzii N'Gousso. Níveis de significado calculados pela ANOVA com um teste de Dunnett pós-hoc. (B) atenuação induzida pelo RNAi de ambas as transcrições em comparação com os controles injetados pela GFP. A atenuação do GSTMS1 apresenta um aumento significativo na mortalidade após a exposição à deltametrina (calculada pela ANOVA com um teste tukey pós-hoc; *p ≤ 0,05, **p ≤0,01). Clique aqui para ver uma versão maior deste número.
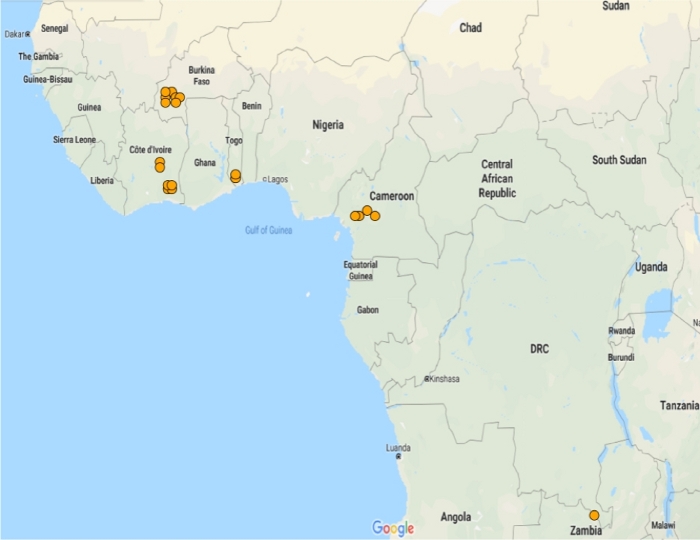
Figura 5: Expressão de GSTMS1 nas populações anopheles gambiae e Anopheles coluzzii. Mapa mostrando a expressão significativamente diferencial do GSTMS1 em conjuntos de dados de microarray disponíveis. O GSTMS1 foi significativamente diferencial em 20 dos 21 conjuntos de dados de microarray. Clique aqui para ver uma versão maior deste número.
| Identificação de transcrição | Descrição | Burkina Faso | Costa do Marfim |
| AGAP006879-RA AGAP006879-RA | Atpase | 27.94 | 43.05 |
| AGAP007160-RB AGAP007160-RB | um cristalina | 11.49 | 10.58 |
| AGAP007160-RC AGAP007160-RC | um cristalina | 11.14 | 10.38 |
| AGAP007160-RA AGAP007160-RA | um cristalina | 9.78 | 9.84 |
| AGAP009110-RA AGAP009110-RA | Desconhecido | 9.26 | 5.96 |
| AGAP007780-RA AGAP007780-RA | NADH dehydrogenase NADH dehydrogenase | 10.49 | 3.77 |
| AGAP006383-RA AGAP006383-RA | oligosaccharyltransferase complexo subunidade beta | 3.69 | 5.57 |
| AGAP007249-RB AGAP007249-RB | Flightin Flightin | 4.61 | 3.86 |
| AGAP003357-RA AGAP003357-RA | Proteína rag1-ativando 1-como a proteína | 4.31 | 4.05 |
| AGAP007249-RA AGAP007249-RA | Flightin Flightin | 4.48 | 3.46 |
| AGAP001998-RA AGAP001998-RA | mRpS10 mRpS10 | 3.46 | 2.85 |
| AGAP007589-RA AGAP007589-RA | UGT306A2 UGT306A2 | 2.29 | 2.28 |
| AGAP000165-RA AGAP000165-RA | GSTMS1 GSTMS1 | 1.95 | 1.85 |
| AGAP002101-RA AGAP002101-RA | synthetase isoleucyl-tRNA | 0.57 | 0.59 |
| AGAP002969-RA AGAP002969-RA | asparaginyl-tRNA synthetase asparaginyl-tRNA synthetase | 0.45 | 0.45 |
| AGAP004199-RA AGAP004199-RA | solute carrier family 5 (sódio acoplado monocarboxylate transportador), membro 8 | 0.35 | 0.48 |
| AGAP004684-RA AGAP004684-RA | proteína de processamento de rRNA CGR1 | 0.36 | 0.22 |
| AGAP006414-RA AGAP006414-RA | Cht8 Cht8 | 0.024 | 0.36 |
Tabela 1: Transcrições diferenciais significativamente na mesma dobra mudam de direção entre as populações de Burkina Faso e Costa do Marfim. Identificação de transcrição, descrição do gene e variação média de cada conjunto de dados dos dois países que representam populações de An. coluzzii e An. gambiae.
| Correlação | Nome sistemático | Tipo de transcrição |
| 1 | AGAP000165-RA AGAP000165-RA | GSTMS1 GSTMS1 |
| 0.82 | AGAP004904-RA AGAP004904-RA | Catalase |
| 0.76 | AGAP007243-RA AGAP007243-RA | 26S protease subunidade reguladora 8 |
| 0.79 | AGAP008358-RA AGAP008358-RA | CYP4H17 CYP4H17 |
| 0.76 | AGAP009436-RA AGAP009436-RA | Hidrolase de Valacyclovir |
| 0.75 | AGAP010739-RA AGAP010739-RA | Glicose-6-fosfato 1-dehydrogenase |
| 0.85 | AGAP011172-RA AGAP011172-RA | cystathionine gama-lyase |
| 0.76 | AGAP012678-RA AGAP012678-RA | Glicose-6-fosfato 1-dehydrogenase |
Tabela 2: Transcrições co-correlacionadas com GSTMS1. A tabela mostra a saída da rede de correlação para GSTMS1 no IR-TEx com |r| de >0.75. A tabela mostra a correlação do Spearman, identificação de transcrição e descrição do gene para cada transcrição co-correlacionada.
Arquivo adicional 1: Arquivo de saída da matriz A-MEXP-2196 analisado na limusine. O arquivo se origina de um knockdown Met em comparação com uma matriz de controle GFP, descrito com mais detalhes no ArrayExpress (E-MTAB-4043) e outra publicação anterior1. As colunas representam identificador AGAP (SystematicName), alteração da dobra de registro (logFC), valores de expressão de registro (AveExpr), t-statistic (t), p-value não corrigido (P.Value), valor p ajustado (adj. P.Val), e B estatística (B)20. Para os propósitos deste arquivo, os mosquitos são Anopheles coluzzi da Costa do Marfim e não estão expostos a inseticidas, com uma latitude de coleta e longitude de -5,4 e 6,0, respectivamente. Clique aqui para ver este arquivo (Clique certo para baixar).
Arquivo adicional 2: Arquivo de saída do experimento RNAseq. Análise rnaseq tomadas de Uyhelji et al.9 descrevendo mudanças no transcriptome de mosquitos Anopheles quando expostos a 50% de salinidade. Este arquivo é adaptado da Tabela S2 da publicação e inclui identificador AGAP (SystematicID), alteração de dobra bruta (Fold_Change) e valor p ajustado (q_value). Clique aqui para ver este arquivo (Clique certo para baixar).
Arquivo adicional 3: Lista Primer para resultados representativos. Identificador AGAP, nome do gene, dsRNA para a frente, dsRNA reverso, qPCR para a frente, e qPCR conjuntos de primer reverso para cada transcrição. Clique aqui para ver este arquivo (Clique certo para baixar).
Arquivo de codificação suplementar 1. Clique aqui para ver este arquivo (Clique certo para baixar).
Arquivo de codificação suplementar 2. Clique aqui para ver este arquivo (Clique certo para baixar).
Arquivo de codificação suplementar 3. Clique aqui para ver este arquivo (Clique certo para baixar).
Discussão
A transcriptômica de Big Data produz listas de milhares de transcrições que são expressas diferencialmente para cada condição experimental. Muitos desses experimentos são realizados em organismos e fenótipos relacionados e são quase exclusivamente analisados como experimentos independentes. A utilização dessas ricas fontes de dados examinando os dados de forma holística e sem suposições teóricas 1) leva à identificação de novas transcrições de candidatos e 2) impede o descarte de dados valiosos simplesmente porque há muita informação para validar in vivo1.
O IR-TEx fornece aos usuários um fundo de bioinformática limitado com a capacidade de examinar facilmente vários conjuntos de dados, visualizar alterações nos conjuntos de dados e baixar as informações associadas1. Embora o IR-TEx não suporte a pesquisa de mais de uma transcrição em cada pesquisa, os usuários podem examinar os arquivos Fold_Changes.txt associados simplesmente usando Excel, R ou outros programas apropriados. Uma utilidade mais adicional de IR-TEx provem do uso de redes da correlação para prever a função do transcript, entrada de proteínas hipotéticas ou transcrições com funções desconhecidas e uso do software downstream para procurarar por enriquecimentos1.
No exemplo demonstrado neste protocolo, o IR-TEx é usado de acordo com sua função original. Aqui, ele permite a exploração de transcrições associadas à resistência aos inseticidas e visualização da distribuição de excesso e sub-expressão através de gráficos de mapeamento. Transcrições de interesse são validadas in vivo para determinar se o excesso ou sub-expressão de transcrições dadas contribui para um fenótipo observado1 (por exemplo, resistência inseticida). Foi demonstrado aqui, como relatado anteriormente1, que um conjunto de dados pode ser usado em uma abordagem orientada por hipóteses para identificar transcrições de interesse em uma base específica do país. O IR-TEx pode então ser usado para 1) explorar a expressão da transcrição e 2) contextualizar a função da transcrição aplicando uma rede de correlação de pares em todas as transcrições contidas em cada conjunto de dados -omics. Aqui, o GSTMS1 mostrou-se co-correlacionado com uma série de outras transcrições implicadas na desintoxicação. Esses dados (juntamente com o knockdown da transcrição que resultou em um aumento significativo na mortalidade após a exposição ao inseticida) demonstram a importância dessa transcrição na liberação xenobiótica.
O IR-TEx representa um recurso valioso para explorar transcrições relacionadas à resistência a inseticidas na web ou usar aplicativos locais. Este protocolo demonstra como modificar o IR-TEx para diferentes plataformas de omics, bem como dados completamente novos. O guia ilustra como usar o IR-TEx para integrar dados de várias plataformas e conjuntos de dados de omics com dados em falta, bem como como recodificar o IR-TEx simplesmente para que seja útil para qualquer pessoa que pesquise conjuntos de dados transcritímicos.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Este trabalho foi financiado por uma Bolsa de Desenvolvimento de Competências do MRC para v.I. (MR/R024839/1) e Royal Society Challenge Grant (CH160059) para H.R.
Materiais
| Name | Company | Catalog Number | Comments |
| Laptop with browser | Any | - | - |
| R Program | The R Project for Statistical Computing | - | https://www.r-project.org/ |
| R Studio | R Studio | - | https://www.rstudio.com/ |
Referências
- Ingham, V. A., Wagstaff, S., Ranson, H. Transcriptomic meta-signatures identified in Anopheles gambiae populations reveal previously undetected insecticide resistance mechanisms. Nature Communications. 9 (1), 5282 (2018).
- Chang, W., Cheng, J., Allaire, J., Xie, Y., McPherson, J. . shiny: Web Application Framework for R. , (2017).
- Bhatt, S., et al. The effect of malaria control on Plasmodium falciparum in Africa between 2000 and 2015. Nature. 526 (7572), 207-211 (2015).
- Ranson, H., Lissenden, N. Insecticide Resistance in African Anopheles Mosquitoes: A Worsening Situation that Needs Urgent Action to Maintain Malaria Control. Trends in Parasitology. 32 (3), 187-196 (2016).
- Donnelly, M. J., et al. Does kdr genotype predict insecticide-resistance phenotype in mosquitoes. Trends in Parasitology. 25 (5), 213-219 (2009).
- Stevenson, B. J., et al. Cytochrome P450 6M2 from the malaria vector Anopheles gambiae metabolizes pyrethroids: Sequential metabolism of deltamethrin revealed. Insect Biochemistry and Molecular Biology. 41 (7), 492-502 (2011).
- Müller, P., et al. Field-Caught Permethrin-Resistant Anopheles gambiae Overexpress CYP6P3, a P450 That Metabolises Pyrethroids. PLoS Genetics. 4 (11), 1000286 (2008).
- Huang, D., et al. The DAVID Gene Functional Classification Tool: a novel biological module-centric algorithm to functionally analyze large gene lists. Genome Biology. 8 (9), 183 (2007).
- Uyhelji, H. A., Cheng, C., Besansky, N. J. Transcriptomic differences between euryhaline and stenohaline malaria vector sibling species in response to salinity stress. Molecular Ecology. 25 (10), 2210-2225 (2016).
- Edi, C. V., Benjamin, K. G., Jones, C. M., Weetman, D., Ranson, H. Multiple-Insecticide Resistance in Anopheles gambiae Mosquitoes, Southern Côte d’Ivoire. Emerging Infectious Diseases. 18 (9), 1508-1511 (2012).
- Ding, Y., Ortelli, F., Rossiter, L., Hemingway, J., Ranson, H. The Anopheles gambiae glutathione transferase supergene family: annotation, phylogeny and expression profiles. BMC Genomics. 4 (1), 1-16 (2003).
- Enayati, A. A., Ranson, H., Hemingway, J. Insect glutathione transferases and insecticide resistance. Insect Molecular Biology. 14 (1), 3-8 (2005).
- Ranson, H., et al. Identification of a novel class of insect glutathione S-transferases involved in resistance to DDT in the malaria vector Anopheles gambiae. The Biochemical Journal. 359, 295-304 (2001).
- Riveron, J. M., et al. A single mutation in the GSTe2 gene allows tracking of metabolically based insecticide resistance in a major malaria vector. Genome Biology. 15 (2), 27 (2014).
- Pavlidi, N., Vontas, J., Van Leeuwen, T. The role of glutathione S-transferases (GSTs) in insecticide resistance in crop pests and disease vectors. Current Opinion in Insect Science. 27, 97-102 (2018).
- Salvemini, F., et al. Enhanced glutathione levels and oxidoresistance mediated by increased glucose-6-phosphate dehydrogenase expression. Journal of Biological Chemistry. 274 (5), 2750-2757 (1999).
- Deplancke, B., Gaskins, H. R. Redox control of the transsulfuration and glutathione biosynthesis pathways. Current Opinion in Clinical Nutrition & Metabolic Care. 5 (1), (2002).
- Puente, X. S., López-Otn, C. Cloning and expression analysis of a novel human serine hydrolase with sequence similarity to prokaryotic enzymes involved in the degradation of aromatic compounds. Journal of Biological Chemistry. 270 (21), 12926-12932 (1995).
- Riveron, J. M., et al. Genome-wide transcription and functional analyses reveal heterogeneous molecular mechanisms driving pyrethroids resistance in the major malaria vector Anopheles funestus across Africa. G3: Genes, Genomes, Genetics. 7 (6), 1819-1832 (2017).
- Smyth, G. K. Linear models and empirical bayes methods for assessing differential expression in microarray experiments. Statistical Applications in Genetics and Molecular Biology. 3 (1), 3 (2004).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados