Method Article
IR-TEx: Una herramienta de integración de datos de código abierto para transcriptomica de Big Data diseñada para las anopheles gambiae de vectores de malaria
En este artículo
Resumen
IR-TEx explora perfiles transcripcionales relacionados con la resistencia a insecticidas en la especie Anopheles gambiae. Aquí se proporcionan instrucciones completas para el uso de la aplicación, modificaciones para explorar varios conjuntos de datos transcriptomáticos y el uso del marco para crear una base de datos interactiva para colecciones de datos transcriptomicos de cualquier organismo, generados en cualquier plataforma.
Resumen
IR-TEx es una aplicación escrita en Shiny (un paquete R) que permite explorar la expresión de (así como asignar funciones a) transcripciones cuya expresión está asociada con fenotipos de resistencia a insecticidas en mosquitos Anopheles gambiae. La aplicación puede ser utilizado en línea o descargado y utilizado localmente por cualquier persona. La aplicación local se puede modificar para agregar nuevos conjuntos de datos de resistencia a insecticidas generados a partir de varias plataformas -omics. Esta guía muestra cómo agregar nuevos conjuntos de datos y controlar los datos que faltan. Además, IR-TEx se puede recodificar completa y fácilmente para utilizar conjuntos de datos omics a partir de cualquier dato experimental, lo que lo convierte en un recurso valioso para muchos investigadores. El protocolo ilustra la utilidad de IR-TEx para identificar nuevos candidatos a la resistencia a los insecticidas utilizando la transferencia de glutatión microsómico, GSTMS1, como ejemplo. Esta transcripción está regulada en múltiples poblaciones resistentes a los piretroides de Costa de Marfil y Burkina Faso. La identificación de transcripciones cocorrelacionadas proporciona más información sobre las funciones putativas de este gen.
Introducción
La capacidad de medir la expresión de un gran número de transcripciones simultáneamente a través de plataformas de microarray y tecnología RNAseq ha dado lugar a la generación de vastos conjuntos de datos que asocian la expresión de transcripción con un fenotipo particular tanto en organismos modelo como no modelo. Estos conjuntos de datos son un recurso extremadamente rico para los investigadores, cuyo poder se puede aumentar combinando conjuntos relevantes en un enfoque de integración de big data. Sin embargo, esta metodología se limita a aquellos con habilidades bioinformáticas particulares. Aquí se describe un programa, IR-TEx (publicado anteriormente por Ingham et al.1) que está escrito en un paquete R llamado Shiny2 y permite a los usuarios con poca formación bioinformática integrar e interrogar estos conjuntos de datos con relativa facilidad.
IR-TEx, encontrado en http://www.lstmed.ac.uk/projects/IR-TEx, fue escrito para explorar transcripciones asociadas con la resistencia a los insecticidas en Anopheles gambiae, el principal vector de malaria africano1. La malaria es una enfermedad parasitaria causada por especies de Plasmodium, transmitida entre humanos a través de las picaduras de mosquitos hembra Anopheles. La orientación al mosquito vector con insecticidas ha demostrado ser el medio más eficaz para prevenir la morbilidad y mortalidad relacionadas con el paludismo en Africa. La ampliación de las herramientas (es decir, mosquiteros insecticidas de larga duración) también ha sido fundamental en las drásticas reducciones de los casos de paludismo desde 20003. Con un número muy limitado de insecticidas disponibles, existe una fuerte presión evolutiva sobre los mosquitos, y la resistencia está ahora generalizada en los vectores de malaria africanos4.
Además, las mutaciones en el sitio objetivo5 y el aclaramiento metabólico de insecticidas6,7 siguen siendo los principales mecanismos estudiados de resistencia, pero ahora están surgiendo otros mecanismos resistentes potentes1. Muchos de estos nuevos mecanismos no se han asociado previamente con la resistencia a los insecticidas, pero se han detectado mediante la búsqueda de patrones comunes de expresión génica en múltiples poblaciones resistentes utilizando la aplicación IR-TEx y posteriormente validados funcionalmente por los enfoques genómicos1.
Aquí se describe un enfoque paso a paso para usar IR-TEx, tanto en la web como cuando se instala localmente. El protocolo describe cómo se pueden integrar nuevos conjuntos de datos de resistencia a insecticidas en el paquete existente y se explica cómo operar con los datos que faltan. Por último, describe cómo utilizar este software con otros conjuntos de datos -omics que no están relacionados con la resistencia a los insecticidas, combinando así datos de diferentes enfoques -omics mientras que también opera con valores que faltan y normalización para que los datos sean comparables.
Protocolo
1. Uso de la aplicación web IR-TEx
- Ejecución de la aplicación en un navegador web
- Abra la aplicación web IR-TEx siguiendo el enlace en la parte inferior de la página que se encuentra en http://www.lstmed.ac.uk/projects/IR-TEx.
- Una vez que la página web se haya inicializado, haga clic en el botón Aplicación en la parte superior de la página, que mostrará la aplicación y las salidas asociadas.
- Lea cada salida relacionada con la entrada predeterminada de AGAP008212-RA (CYP6M2) en el cuadro de identificación de transcripción con las siguientes condiciones: Conjuntos de datos coluzzii que están (i) expuestos a insecticidas piretroides o (ii) no expuestos a ninguna clase de insecticida, y transcripciones asociadas con una correlación de >0.98.
- Explorar la expresión de una transcripción de interés
- Para seleccionar una transcripción de interés, introduzca el ID de transcripción en el cuadro ID de transcripción, recordando que las transcripciones terminan en -RX dependiendo de la isoforma de interés.
- Seleccione los conjuntos de datos que desea interrogar marcando las casillas correspondientes para (i) Países; (ii) Estado de exposición, (iii) Especies de interés; y iv) Clase de interés insecticida, todo ello a la vez que se asegura de que estos criterios den lugar a un conjunto de datos incluido >1 (véase el Cuadro Suplementario 1 en Ingham y otros1).
NOTA: (iii) se refiere al miembro del complejo de especies An. gambiae que el usuario interesado en. Actualmente, los datos están disponibles para An. coluzzii y An. arabiensis. - Haga clic en Actualizar vista en la parte inferior del menú de selección o pulse Retorno, ignorando el valor de correlación absoluta (por ahora).
- Conceda a la aplicación tiempo para actualizar.
- Lea el primer gráfico como: log2 fold change between a resistant population and lab-susceptible mosquito population of the transcript of interest across each dataset that meets the criteria selected in step 1.2 (Figure 1). Los detalles de todos los conjuntos de datos se pueden encontrar en Ingham et al.1.
- Lea la información debajo del gráfico como: el pliegue cambia entre los mosquitos resistentes y susceptibles para cada conjunto de datos relevante, además de los valores p corregidos (Q). Cada fila representa sondas individuales en el microarray. La metodología para la visualización gráfica se ha notificado previamente1.
- Lea la tabla adicional a continuación como el número de experimentos en los que la transcripción de interés es significativa, así como el número total de experimentos que coinciden con los criterios seleccionados en el paso 1.2.
- Para descargar los datos en formato separado por pestañas, haga clic en el botón Descargar debajo de las dos tablas. Esto permite al usuario explorar los datos de una manera más fácil usando un programa como Excel.
- Interpretar el mapa de la siguiente manera: cada punto representa los sitios aproximados de recolección de mosquitos resistentes en cada conjunto de datos en el que se expresa diferencialmente la transcripción de interés. Los colores siguen un sistema de semáforos que se explica en la aplicación (Figura 2).
- Para los pasos 1.2.5 y 1.2.8, guarde las salidas gráficas haciendo clic con el botón derecho, haciendo clic en Guardar imagen como... y eligiendo una carpeta adecuada.
NOTA: En el caso de un error de salida de la aplicación, es probable que ningún conjunto de datos coincida con los criterios introducidos. Compruebe la Tabla Suplementaria 1 en Ingham et al.1 si esto ocurre.
- Identificación de funciones/vías putativas de transcripción de interés
- Las correlaciones (valor r2 mínimo introducido) de los patrones de expresión de transcripciones en varios conjuntos de datos se pueden utilizar para predecir la función de transcripción y potencialmente esclarecer transcripciones coreguladas desde la misma vía. Usando el ejemplo de Ingham et al.1 (AGAP001076-RA; CYP4G16), siga los pasos 1.2.1–1.2.2 de la sección anterior, seleccionando todos los conjuntos de datos para obtener la máxima potencia.
- Antes de hacer clic en Actualizar vista, mueva el control deslizante Valor de correlación absoluta a 0,85 y haga clic en Actualizar vista o pulse Retorno.
- Examine la tabla de correlación (tabla inferior) para buscar las transcripciones múltiples que ahora se muestran y que están correlacionadas con la transcripción de entrada.
- Manipular el control deslizante Valor de correlación absoluta y observar cualquier cambio en el gráfico y la tabla inferior; las salidas del paso 1.3.2 permanecerán sin cambios. Tal y como se muestra en de la Figura 3 (-r-> 0.9, -r-> 0.8), la reducción de la rigenta del valor de correlación mostrará más transcripciones, pero introducirá más ruido.
- Lea la tabla debajo de la salida gráfica, que (además de los parámetros descritos en el paso 1.2.6) contiene el valor de correlación para cada transcripción.
- Para descargar los datos en un formato separado por tabulaciones, haga clic en el botón Descargar.
- El análisis de enriquecimiento funcional se puede realizar en la lista de ID de transcripción descargada utilizando el análisis DAVID8. Una vez en el sitio web de DAVID (que se encuentra en https://david.ncifcrf.gov/),seleccione Análisis funcional. Pegue la lista completa de genes, utilizando identificadores de genes [identificador sin el -RX, que se puede hacer en Excel insertando una columna a la derecha del ID sistemático y escribiendo "IZQUIERDA"(X1,10),donde X1 es la célula de ID sistemático]. Seleccione el identificador como VectorBase_ID y la lista de genes y haga clic en Enviar lista.
- Haga clic en el botón Clustering de anotación funcional para obtener una visión general de los enriquecimientos que se encuentran en esta red de correlación, lo que permite asignar una función potencial a la transcripción. Explore los enriquecimientos en profundidad mirando a través de las diferentes categorías y haciendo clic en los botones + para cada uno y posteriormente haciendo clic en Gráfico.
2. Descarga e implementación de IR-TEx localmente
- Descarga y ejecución de IR-TEx
- Vaya al enlace que se encuentra en http://github.com/LSTMScientificComputing/IR-TEx; y haga clic en Clonar o descargar . Descargar Zip. Directo a una carpeta de su elección y descomprima el archivo en esa carpeta.
- Descargue la última versión del software R para el sistema operativo adecuado desde el enlace que se encuentra en http://cran.r-project.org/mirrors.html. Instale el programa.
- Descargue e instale el software más reciente de R Studio, de nuevo para el sistema operativo adecuado desde el enlace que se encuentra en http://www.rstudio.com/products/rstudio/download/.
- Una vez instalado, abra R Studio ( R Studio) Codificación suplementaria Archivo 1 y ejecute cada línea para configurar el sistema para IR-TEx.
- Una vez que todos los paquetes se hayan instalado y actualizado correctamente según sea necesario, vaya a Archivo . Abra, localice IR-TEx.R, resalte y abra. Esto ahora debería estar visible en la ventana superior de R Studio.
- Para ejecutar la aplicación, presione el botón Ejecutar aplicación en la parte superior derecha de la ventana y aparecerá una segunda ventana en la que se cargará la aplicación. Una vez completada la carga, para obtener la funcionalidad completa, haga clic en Abrir en el navegador situado en la parte superior derecha de la ventana cargada.
- Adición de conjuntos de datos de resistencia a IR-TEx (generado mediante la matriz Anopheles gambiae 15k Agilent)
- Para agregar un nuevo conjunto de datos analizado generado en la misma plataforma de microarray (A-MEXP-2196) al conjunto de datos disponible, descargue la aplicación y busque la carpeta descomprimida descargada en la sección 2.1.
- Abrir archivo adicional 1, que representa una salida de un análisis de limma en A-MEXP-2196 1. Con Excel, en la columna H1, escriba Fold_Changey, en H2, escriba 2 o B2, en la que B2 es el cambio de pliegue de registro. Aplique esto en toda la columna H para producir cambios de plegado sin procesar.
- Organizar archivo adicional 1 de tal forma que la columna A es el IDENTIFICADOR, la columna B es el cambio de pliegue de la columna H (copiar la columna H, resaltar la columna B, luego hacer clic con el botón derecho y pegar valores) y la columna C es el valor p ajustado. Elimine todas las demás columnas y guárdelas como un archivo delimitado por tabulaciones.
- Abra el archivo de codificación suplementaria 2 y ejecute utilizando la hoja delimitada por tabulaciones producida en el paso 2.2.3.
NEWFILE_FC = c('COUNTRY','EXPOSURE STATUS','SPECIES','INSECTICIDE')
NEWFILE_Q = c('COUNTRY','EXPOSURE STATUS','SPECIES','INSECTICIDE')
NOTA: Los campos entre comillas simples deben cambiarse para reflejar la información del nuevo conjunto de datos. El estado de exposición se refiere a si las muestras se recogieron después de la exposición a insecticidas (expuestas/no expuestas). Insecticida: si 'no está expuesto', utilice 'ninguno'. Consulte Fold_Changes.txt. metadatos de otros ejemplos. Asegúrese de que la ortografía sea coherente. - Abra geography.txt, desplácese hasta la fila ocupada final y seleccione a continuación. Escriba el nombre del conjunto de datos, seguido de Q y NEWFILE_Q en la columna 1, la latitud del sitio de la colección de muestras en la columna 2 y la longitud en la columna 3. Guarde los cambios.
- Si se utilizan entradas novedosas (es decir, Gambia), que no están disponibles para su selección en el conjunto de datos (consulte Ingham et al. Supplementary Table 11), deberá agregarse al código. Para ello, abra IR-TEx.R en RStudio y localice la línea 26 como se indica en RStudio, momento en el que debe comenzar lo siguiente:
'sidebarPanel(....'.
NOTA: Cada una de las filas de procedimiento se relaciona con un elemento de metadatos introducidos en las filas debajo del nombre del conjunto de datos en Fold_Changes.txt en el paso 2.2.5. - Para agregar los metadatos nuevos, desplácese hasta el final de la línea de los metadatos de su elección y localice el término 'seleccionado'. Inmediatamente después de esto debe ser una coma y un corchete cerrado; en este punto, haga clic en el cursor dentro del corchete cerrado. Después del apóstrofo final, escriba una coma, seguida de un apóstrofo, seguido de los nuevos metadatos (por ejemplo, 'Gambia') y guarde los cambios. Vea a continuación un ejemplo.
checkboxGroupInput('CountryInput','Select Relevant Countries',c('Burkina Faso','Cote D'Ivoire','Camerún','Guinea Ecuatorial','Zambia','Tanzania','Sudán','Uganda','Togo', 'Gambia'),seleccionado('Burkina Faso','Cote D'Ivoire','Camerún','Guinea Ecuatorial','Zambia','Tanzania','Sudán','Uganda','Togo')) - Ejecute la aplicación. La nueva entrada de metadatos debe aparecer como una casilla de verificación no seleccionada debajo del encabezado correspondiente. Si el usuario desea que se seleccione, debe agregarse después de la seleccionada c(..., como se muestra a continuación:
checkboxGroupInput('CountryInput','Select Relevant Countries',c('Burkina Faso','Cote D'Ivoire','Camerún','Guinea Ecuatorial','Zambia','Tanzania','Sudán','Uganda','Togo', 'Gambia'),seleccionado('Burkina Faso','Cote D'Ivoire','Camerún','Guinea Ecuatorial','Zambia','Tanzania','Sudán','Uganda','Togo', 'Gambia')) - Para agregar datasets de resistencia no realizados en A-MEXP-2196, consulte la sección 3.
3. Modificación de IR-TEx para su uso con diferentes conjuntos de datos
- Uso en múltiples plataformas -omics y proceder con datos que faltan
- Para continuar con "0" en conjuntos de datos: consulte el origen del conjunto de datos para conocer el significado específico de "0". Se recomienda que "0" se sustituya (conservadoramente) por "NA". Al igual que con los cambios de plegado sin procesar (B/A), "0" indica una señal no detectada en la condición experimental B. En el caso de que la condición experimental A muestre una expresión sustancial, el usuario puede aplicar un pequeño valor de cambio de pliegue.
- Abra Archivo adicional 2.txt, un archivo RNAseq adaptado de Uyhelji et al.9. Este archivo representa la plantilla en la que se deben basar los nuevos datos: columna A - identificador, columna B - cambio de plegado sin procesar y columna C - valor p ajustado. Utilice este archivo para ejecutar los pasos siguientes.
- Ejecute el código R para que coincida con los identificadores en un único archivo delimitado por tabulaciones en todas las plataformas y, a continuación, organice y normalice los datos (Archivo decodificación suplementario 2). Las instrucciones están contenidas en el archivo. Cualquier FILEPATH se separará por "/" para MacOS o "//" para Windows (cámbielos de "a", tal y como aparecerán).
- Salida del archivo producido al final del archivo de codificación suplementario 2 en una ubicación de elección para su uso en el paso 3.1.5. El archivo de codificación suplementario 2 generará un nuevo archivo Fold_Changes.txt. Realice una copia de seguridad del archivo original.
- Ejecute el código contenido en Archivo de codificación suplementario 3. Busque el archivo de salida denominado FC_distribPlot.png en la carpeta especificada como FILEPATH. Compruebe las distribuciones del cambio de pliegue del registro2 para comprobar que las distribuciones de cambio de plegado del registro2 son casi idénticas entre los conjuntos de datos.
- Siga las instrucciones del paso 2.2.6 para editar archivos adicionales y garantizar la compatibilidad del nuevo Fold_Changes.txt.
- Modificación de IR-TEx para su uso con conjuntos de datos completamente nuevos
- Abra IR-TEx.R en RStudio y localice las líneas (23–34) empezando por:
'tabPanel('
y terminando en:
submitButton("Update View", icono("refresh"))
), - Cambie el AGAP008212-RA que se encuentra en las siguientes líneas a una transcripción de interés en los nuevos datos.
textInput('textInput','Id. de transcripción',valor'AGAP008212-RA'), - Localice las cuatro opciones que comienzan con:
checkboxGroupInput(
Estas opciones se pueden modificar para representar metadatos importantes por los que el usuario desea filtrar los nuevos datos. En cada caso, el usuario debe cambiar la opción Seleccionar países relevantes; Seleccione Estado de exposición; Seleccione Especies relevantes; y Seleccione Clase de Insecticida para que sea representativa de los datos (es decir, Seleccionar tipo de tejido; Seleccione Sexo; Seleccione Soporte de edad; Seleccione Estado de la enfermedad). - Identifique los metadatos asociados con el conjunto de datos y la entrada para reemplazar las opciones existentes inmediatamente después de la primera c('. En cada caso, las opciones estarán contenidas dentro de las marcas de voz y separadas de la siguiente selección por una coma. Después de la selección final, el soporte debe estar cerrado. Un ejemplo de Seleccionar estado de enfermedad es:
c('Infectado', 'No infectado', 'Desconocido') - Elija cuál de estos metadatos se seleccionará al abrir la aplicación. Estos se pueden cambiar modificando las opciones después de selected-c('. Un ejemplo de Seleccionar estado de enfermedad es:
selected-c('Infectado', 'No infectado')
Esto indicará a la aplicación que seleccione solo los conjuntos de datos que coincidan con estos criterios en la carga inicial. - Para crear una nueva tabla de datos, siga el diseño que se encuentra en Fold_Changes.txt e instrucciones en la sección 2. Cambie los metadatos a cada cambio respectivo descrito en el paso 3.2.4, exactamente como se escribe en el código (R distingue mayúsculas de minúsculas). En la columna de desintoxicación, introduzca los nombres de los genes y, en la columna de tipo de transcripción, introduzca descripciones genéticas para cada transcripción. Siga la sección 3.2 al agregar nuevos conjuntos de datos.
- Si la asignación no es relevante para los requisitos experimentales, localice las siguientes líneas de código y coloque el valor de '' delante:
Líneas 49–51:
br(),br(),
withSpinner(plotOutput("Geography")),
textOutput('Geography_legend'),
Líneas 493 a partir:
output$Geography <- renderPlot(?
Para la línea 602 final:
output$Geography_legend <- renderText(
paste("Significant Transcripts Only (p", as.expression("<-"),""0.05): FC > 5 á Red, FC > 1 á Amber, FC < 1 á Green",sep-"")
})
- Abra IR-TEx.R en RStudio y localice las líneas (23–34) empezando por:
Resultados
Utilizando el archivo Fold_Changes.txt incluido con IR-TEx, comparamos las transcripciones que se expresaron significativamente diferencialmente en conjuntos de datos resistentes de Anopheles coluzzii y Anopheles gambiae con controles susceptibles de Costa de Marfil y Burkina Faso. Esto produjo 18 transcripciones de interés(Tabla 1;esta búsqueda se puede realizar usando Excel, R u otros programas). Dos de ellos, un ATPase (AGAP006879) y -cristalino (AGAP007160), se han reportado previamente, con el primero teniendo un efecto significativo en la resistencia a los piretroides1. Además de estas dos transcripciones, se presentaron dos transcripciones de desintoxicación, GSTMS1 (FC 1,95 y 1,85) y UGT306A2 (FC 2,29 y 2,28).
qPCR validación de dos de estas transcripciones (GSTMS1, una transcripción de desintoxicación; y AGAP009110-RA, una transcripción desconocida, específica de mosquitos que contiene un dominio de unión de -1,3-glucano) como se describió anteriormente1. El análisis se realizó utilizando conjuntos de imprimación descritos en el archivo adicional 3 y mostró que estas transcripciones estaban significativamente reguladas en una población multirresistente de Costa de Marfil (Tiassalé) y otra de Burkina Faso (Banfora), en comparación con el N'Gousso susceptible al laboratorio(Figura 4A).
Como ambas transcripciones mostraron una regulación significativa en cada una de las poblaciones resistentes, se realizó un derribo inducido por el ARNI en mosquitos del laboratorio LSTM de la colonia Tiassalé. Esta colonia tiene su origen en Costa de Marfil y es resistente a todas las principales clases de insecticida utilizados en la salud pública, como se describió anteriormente1,10. La atenuación de la expresión de GSTMS1 dio lugar a un aumento significativo (p - 0,021) en la mortalidad después de la exposición a la deltametrina en comparación con los controles inyectados por GFP, lo que demuestra la importancia de esta transcripción en la resistencia a los piretroides(Figura 4B). Por el contrario, el derribo de AGAP009110-RA no dio lugar a un cambio significativo (p - 0,082) en la mortalidad después de la exposición(Figura 4B).
GSTMS1 es un GST microsómico y es uno de los tres que se encuentran en los mosquitos A. gambiae 11. Aunque los miembros de las clases de épsilon y delta de los GST han estado previamente implicados en la desintoxicación de insecticidas12,13,14, esta es la primera evidencia de nuestro conocimiento para un papel de los GST microsómicos en la resistencia a los piretroides15. Para explorar la función putativa de esta transcripción en los mosquitos Anopheles gambiae sl, se identificó la expresión y correlación en IR-TEx. GSTMS1 se sobreexpresó significativamente en 20 de los 21 conjuntos de datos disponibles para estas especies, con la excepción de la isla de Bioko. En cada ubicación, la sobreexpresión fue inferior a cinco veces en comparación con las poblaciones susceptibles(Figura 5).
Como los GST microsómicos han sido ignorados en gran medida como posibles desintoxicadores de insecticidas, poco se sabe sobre su papel en la resistencia a los insecticidas15. Al explorar la correlación de otras transcripciones, las funciones putativas pueden ser esclarecidas a través de la asunción de la corregulación o la participación en las mismas vías. Para maximizar la potencia en la red de correlación, se seleccionaron todos los datasets de microarray presentes en IR-TEx, y se ha seleccionado un de >0.75 fue seleccionado. El cuadro 2 muestra la salida del IR-TEx.
Estas transcripciones se enriquecen en la actividad de la oxioreductasa y el metabolismo de glucosa/carbohidratos en la herramienta de anotación funcional de DAVID8. Tanto la glucosa-6-fosfato deshidrogenasa como la citationa gamma-liasa mantienen el nivel de glutatión en las células de mamíferos16,17 y por lo tanto se vinculan directamente con GSTMS1,una glutatión-S-transferasa. Catalasa es un respondedor de estrés oxidativo de acción rápida que protege las células del daño reactivo de las especies de oxígeno, un subproducto de la exposición a piretroides. Valacyclovir hydrolase es una hidrolasa que puede desempeñar un papel en la desintoxicación en células de mamíferos18. CYP4H17 también está presente en la red de correlación. Los citocromos p450 son metabolizadores directos de insecticidas piretroides, y estos productos de descomposición pueden ser metabolizados por GST. Finalmente, CYP4H17 se ha implicado en la resistencia a los piretroides en A. funestus19. En conjunto, estos datos apoyan firmemente un papel para GSTMS1 en la desintoxicación xenobiótica.
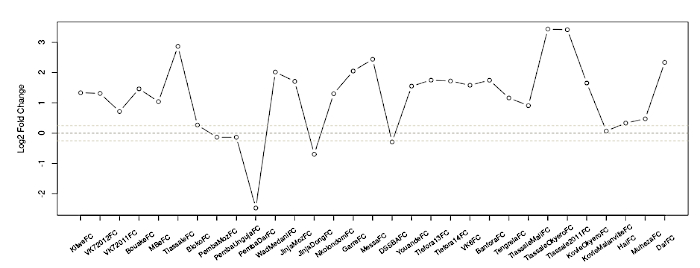
Figura 1: Cambio de pliegue del registro2 de AGAP002865-RA en todos los conjuntos de datos. El eje x detalla los diferentes conjuntos de datos, información para la que se puede encontrar en la Tabla Suplementaria 1 en una publicación anterior1, y el eje Y muestra el cambio de pliegue del registro2 en la transcripción de interés. Las líneas de puntos gris claro indican umbrales aproximados de significancia, tomados aquí para ser un cambio de pliegue de <0.8 o cambio de pliegue de >1.2. La línea negra punteada indica un cambio de pliegue de 1 (es decir, no hay diferencia de expresión entre las poblaciones resistentes y susceptibles). Haga clic aquí para ver una versión más grande de esta figura.
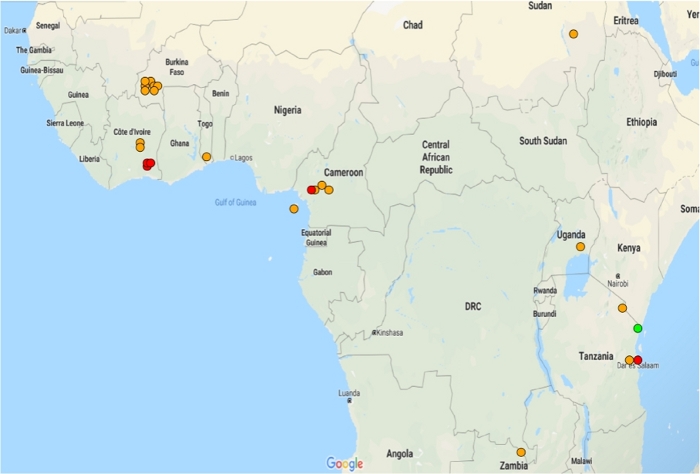
Figura 2: Distribución de microarrays que muestran una expresión diferencial significativa de AGAP002865-RA en poblaciones resistentes. Los cambios de plegado se representan en un sistema de semáforos: cambio de plegado verde de <1, cambio de pliegue naranja de >1 y cambio de pliegue rojo de >5. Solo se muestran los datasets con una expresión diferencial significativa (p - 0,05). Haga clic aquí para ver una versión más grande de esta figura.
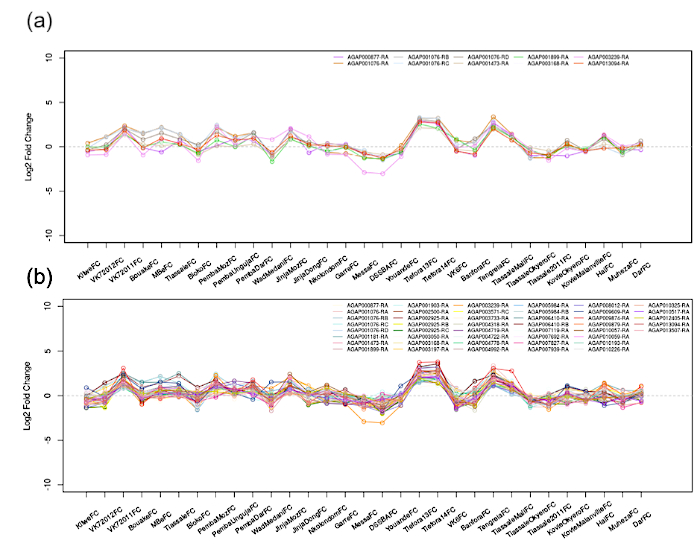
Figura 3: Redes de correlación de AGAP001076-RA (CYP4G16). Las correlaciones por pares se calculan en todas las transcripciones de los 31 conjuntos de datos de microarray, con un corte definido por el usuario aplicado. Aquí se muestra (A) sr. > 0,9 y (B) > 0,8. Todas las transcripciones mostradas en el gráfico cumplen este criterio y siguen los cambios de expresión de AGAP001076-RA. Haga clic aquí para ver una versión más grande de esta figura.
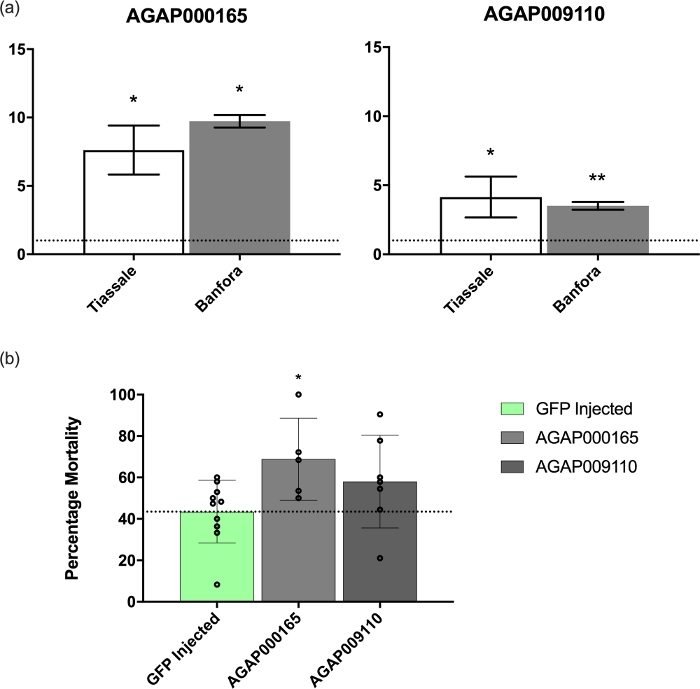
Figura 4: expresión de ARNm y fenotipo al atenuar GSTMS1 y AGAP009110-RA. (A) expresión de ARNm de GSTMS1 y AGAP009110-RA en dos poblaciones multirresistentes de An. coluzzii de Costa de Marfil y Burkina Faso, respectivamente. Los niveles se compararon con el laboratorio-susceptible An. coluzzii N'Gousso. Niveles de significancia calculados por ANOVA con una prueba de Dunnett post-hoc. (B) atenuación inducida por el ARNI de ambas transcripciones en comparación con los controles inyectados por GFP. La atenuación de GSTMS1 muestra un aumento significativo de la mortalidad después de la exposición a la deltametrina (calculado por ANOVA con una prueba de Tukey post-hoc; *p a 0,05, **p a 0,01). Haga clic aquí para ver una versión más grande de esta figura.
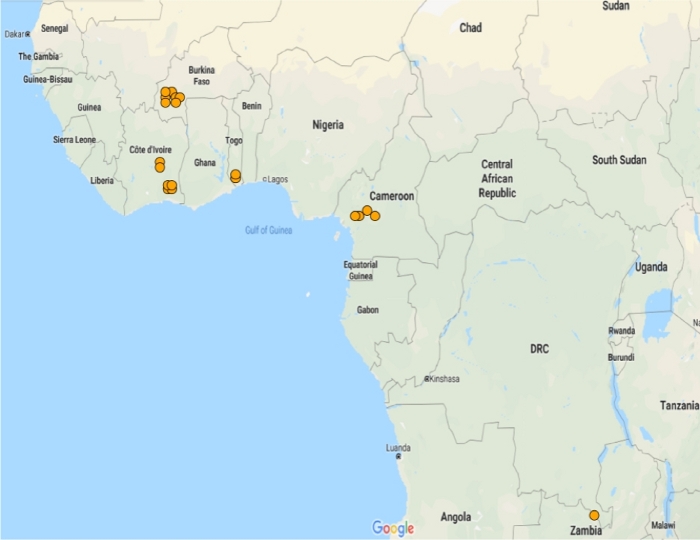
Figura 5: Expresión de GSTMS1 en las poblaciones de Anopheles gambiae y Anopheles coluzzii. Mapa que muestra la expresión significativamente diferencial de GSTMS1 en los datasets de microarray disponibles. Se encontró que GSTMS1 era significativamente diferencial en 20 de los 21 conjuntos de datos de microarray. Haga clic aquí para ver una versión más grande de esta figura.
| ID de transcripción | Descripción | Burkina Faso | Costa de Marfil |
| AGAP006879-RA | Atpasa | 27.94 | 43.05 |
| AGAP007160-RB | a-cristalino | 11.49 | 10.58 |
| AGAP007160-RC | a-cristalino | 11.14 | 10.38 |
| AGAP007160-RA | a-cristalino | 9.78 | 9.84 |
| AGAP009110-RA | Desconocido | 9.26 | 5.96 |
| AGAP007780-RA | NADH deshidrogenasa | 10.49 | 3.77 |
| AGAP006383-RA | oligosapolisilotransferasa complejo subunidad beta | 3.69 | 5.57 |
| AGAP007249-RB | Flightin | 4.61 | 3.86 |
| AGAP003357-RA | Proteína activadora rag1 proteína similar a 1 | 4.31 | 4.05 |
| AGAP007249-RA | Flightin | 4.48 | 3.46 |
| AGAP001998-RA | mRpS10 | 3.46 | 2.85 |
| AGAP007589-RA | UGT306A2 | 2.29 | 2.28 |
| AGAP000165-RA | GSTMS1 | 1.95 | 1.85 |
| AGAP002101-RA | isoleucyl-tRNA sintetasa | 0.57 | 0.59 |
| AGAP002969-RA | asparaginyl-tRNA sintetasa | 0.45 | 0.45 |
| AGAP004199-RA | familia portadora de soluto 5 (transportador de monocarboxilato acoplado en sodio), miembro 8 | 0.35 | 0.48 |
| AGAP004684-RA | proteína procesadora de rRNA CGR1 | 0.36 | 0.22 |
| AGAP006414-RA | Cht8 | 0.024 | 0.36 |
Tabla 1: Transcripciones significativamente diferenciales en la misma dirección de cambio de pliegue entre las poblaciones de Burkina Faso y Costa de Marfil. ID de transcripción, descripción genética y cambio promedio de pliegue para cada conjunto de datos de los dos países que representan las poblaciones de An. coluzzii y An. gambiae.
| Correlación | Nombre sistemático | Tipo de transcripción |
| 1 | AGAP000165-RA | GSTMS1 |
| 0.82 | AGAP004904-RA | Catalasa |
| 0.76 | AGAP007243-RA | Subunidad reguladora de proteasa 26S 8 |
| 0.79 | AGAP008358-RA | CYP4H17 |
| 0.76 | AGAP009436-RA | Valacyclovir hydrolase |
| 0.75 | AGAP010739-RA | Glucosa-6-fosfato 1-deshidrogenasa |
| 0.85 | AGAP011172-RA | cystathionina gamma-lyasa |
| 0.76 | AGAP012678-RA | Glucosa-6-fosfato 1-deshidrogenasa |
Tabla 2: Transcripciones cocorrelacionadas con GSTMS1. La tabla muestra la salida de la red de correlación para GSTMS1 en IR-TEx con el valor de la red de correlación de la red de correlación de la red de correlación de la red de correlación con el valor de la red de correlación de la red de correlación de la red de correlación de de >0.75. La tabla muestra la correlación del Spearman, el ID de transcripción y la descripción del gen para cada transcripción correlacionada.
Archivo adicional 1: Archivo de salida de la matriz A-MEXP-2196 analizada en limma. El archivo se origina a partir de un derribo Met en comparación con una matriz de control GFP, descrita con más detalle en ArrayExpress (E-MTAB-4043) y otra publicación anterior1. Las columnas representan el identificador AGAP (SystematicName), el cambio de plegado de registro (logFC), los valores de expresión de registro (AveExpr), la estadística t (t), el valor p no corregido (P.Value), el valor p ajustado (adj. P.Val), y la estadística B (B)20. A los efectos de este archivo, los mosquitos son Anopheles coluzzi de Costa de Marfil y no están expuestos a insecticidas, con una latitud y longitud de recolección de -5.4 y 6.0, respectivamente. Haga clic aquí para ver este archivo (haga clic con el botón derecho para descargar).
Archivo adicional 2: Archivo de salida del experimento RNAseq. 9describiendo los cambios en el transcriptoma de los mosquitos Anopheles cuando se exponen al 50% de salinidad. Este archivo está adaptado de la tabla S2 de la publicación e incluye el identificador AGAP (SystematicID), el cambio de plegado sin procesar (Fold_Change) y el valor p ajustado (q_value). Haga clic aquí para ver este archivo (haga clic con el botón derecho para descargar).
Archivo adicional 3: Lista de imprimación para resultados representativos. Identificador AGAP, nombre del gen, dsRNA hacia adelante, dsRNA inversa, qPCR forward y qPCR reverse primer sets para cada transcripción. Haga clic aquí para ver este archivo (haga clic con el botón derecho para descargar).
Archivo de codificación suplementario 1. Haga clic aquí para ver este archivo (haga clic con el botón derecho para descargar).
Archivo de codificación suplementario 2. Haga clic aquí para ver este archivo (haga clic con el botón derecho para descargar).
Archivo de codificación suplementario 3. Haga clic aquí para ver este archivo (haga clic con el botón derecho para descargar).
Discusión
La transcriptomica de Big Data produce listas de miles de transcripciones que se expresan diferencialmente para cada condición experimental. Muchos de estos experimentos se realizan en organismos y fenotipos relacionados y se analizan casi exclusivamente como experimentos independientes. El uso de estas fuentes de datos enriquecidas examinando los datos de forma integral y sin suposiciones teóricas 1) conducirá a la identificación de nuevas transcripciones candidatas y 2) evitará el descarte de datos valiosos simplemente porque hay demasiada información para validar in vivo1.
IR-TEx proporciona a los usuarios un fondo bioinformático limitado con la capacidad de examinar fácilmente varios conjuntos de datos, visualizar cambios en los conjuntos de datos y descargar la información asociada1. Aunque IR-TEx no admite la búsqueda de más de una transcripción en cada búsqueda, los usuarios pueden examinar los archivos Fold_Changes.txt asociados simplemente mediante Excel, R u otros programas apropiados. Otra utilidad de IR-TEx se deriva del uso de redes de correlación para predecir la función de transcripción, la entrada de proteínas hipotéticas o transcripciones con funciones desconocidas y el uso de software posterior para buscar enriquecimientos1.
En el ejemplo deeste se muestra en este protocolo, IR-TEx se utiliza según su función original. Aquí, permite la exploración de transcripciones asociadas con la resistencia a los insecticidas y la visualización de la distribución de la sobreexpresión y subexpresión a través de gráficos cartográficos. Las transcripciones de interés se validan in vivo para determinar si la expresión excesiva o subexpresación de determinadas transcripciones contribuye a unfenotipo 1 observado (por ejemplo, resistencia a los insecticidas). Aquí se demostró, como se informó anteriormente1, que un conjunto de datos se puede utilizar en un enfoque basado en hipótesis para identificar transcripciones de interés sobre una base específica de cada país. IR-TEx se puede utilizar para 1) explorar la expresión de la transcripción y 2) contextualizar la función de la transcripción aplicando una red de correlación por pares entre todas las transcripciones contenidas en cada conjunto de datos -omics. Aquí, se demostró que GSTMS1 estaba cocorrelación con una serie de otras transcripciones implicadas en la desintoxicación. Estos datos (junto con el derribo de la transcripción que resultó en un aumento significativo de la mortalidad después de la exposición a insecticidas) demuestran la importancia de esta transcripción en el aclaramiento xenobiótico.
IR-TEx representa un recurso valioso para explorar transcripciones relacionadas con la resistencia a insecticidas en la web o para utilizar aplicaciones locales. Este protocolo demuestra cómo modificar IR-TEx para diferentes plataformas -omics, así como datos completamente nuevos. La guía ilustra cómo utilizar IR-TEx para integrar datos de múltiples plataformas y conjuntos de datos -omics con datos que faltan, así como cómo recodificar IR-TEx simplemente para que sea útil para cualquier persona que investigue conjuntos de datos transcriptomicos.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue financiado por una beca MRC Skills Development Fellowship a V.I. (MR/R024839/1) y Royal Society Challenge Grant (CH160059) a H.R.
Materiales
| Name | Company | Catalog Number | Comments |
| Laptop with browser | Any | - | - |
| R Program | The R Project for Statistical Computing | - | https://www.r-project.org/ |
| R Studio | R Studio | - | https://www.rstudio.com/ |
Referencias
- Ingham, V. A., Wagstaff, S., Ranson, H. Transcriptomic meta-signatures identified in Anopheles gambiae populations reveal previously undetected insecticide resistance mechanisms. Nature Communications. 9 (1), 5282 (2018).
- Chang, W., Cheng, J., Allaire, J., Xie, Y., McPherson, J. . shiny: Web Application Framework for R. , (2017).
- Bhatt, S., et al. The effect of malaria control on Plasmodium falciparum in Africa between 2000 and 2015. Nature. 526 (7572), 207-211 (2015).
- Ranson, H., Lissenden, N. Insecticide Resistance in African Anopheles Mosquitoes: A Worsening Situation that Needs Urgent Action to Maintain Malaria Control. Trends in Parasitology. 32 (3), 187-196 (2016).
- Donnelly, M. J., et al. Does kdr genotype predict insecticide-resistance phenotype in mosquitoes. Trends in Parasitology. 25 (5), 213-219 (2009).
- Stevenson, B. J., et al. Cytochrome P450 6M2 from the malaria vector Anopheles gambiae metabolizes pyrethroids: Sequential metabolism of deltamethrin revealed. Insect Biochemistry and Molecular Biology. 41 (7), 492-502 (2011).
- Müller, P., et al. Field-Caught Permethrin-Resistant Anopheles gambiae Overexpress CYP6P3, a P450 That Metabolises Pyrethroids. PLoS Genetics. 4 (11), 1000286 (2008).
- Huang, D., et al. The DAVID Gene Functional Classification Tool: a novel biological module-centric algorithm to functionally analyze large gene lists. Genome Biology. 8 (9), 183 (2007).
- Uyhelji, H. A., Cheng, C., Besansky, N. J. Transcriptomic differences between euryhaline and stenohaline malaria vector sibling species in response to salinity stress. Molecular Ecology. 25 (10), 2210-2225 (2016).
- Edi, C. V., Benjamin, K. G., Jones, C. M., Weetman, D., Ranson, H. Multiple-Insecticide Resistance in Anopheles gambiae Mosquitoes, Southern Côte d’Ivoire. Emerging Infectious Diseases. 18 (9), 1508-1511 (2012).
- Ding, Y., Ortelli, F., Rossiter, L., Hemingway, J., Ranson, H. The Anopheles gambiae glutathione transferase supergene family: annotation, phylogeny and expression profiles. BMC Genomics. 4 (1), 1-16 (2003).
- Enayati, A. A., Ranson, H., Hemingway, J. Insect glutathione transferases and insecticide resistance. Insect Molecular Biology. 14 (1), 3-8 (2005).
- Ranson, H., et al. Identification of a novel class of insect glutathione S-transferases involved in resistance to DDT in the malaria vector Anopheles gambiae. The Biochemical Journal. 359, 295-304 (2001).
- Riveron, J. M., et al. A single mutation in the GSTe2 gene allows tracking of metabolically based insecticide resistance in a major malaria vector. Genome Biology. 15 (2), 27 (2014).
- Pavlidi, N., Vontas, J., Van Leeuwen, T. The role of glutathione S-transferases (GSTs) in insecticide resistance in crop pests and disease vectors. Current Opinion in Insect Science. 27, 97-102 (2018).
- Salvemini, F., et al. Enhanced glutathione levels and oxidoresistance mediated by increased glucose-6-phosphate dehydrogenase expression. Journal of Biological Chemistry. 274 (5), 2750-2757 (1999).
- Deplancke, B., Gaskins, H. R. Redox control of the transsulfuration and glutathione biosynthesis pathways. Current Opinion in Clinical Nutrition & Metabolic Care. 5 (1), (2002).
- Puente, X. S., López-Otn, C. Cloning and expression analysis of a novel human serine hydrolase with sequence similarity to prokaryotic enzymes involved in the degradation of aromatic compounds. Journal of Biological Chemistry. 270 (21), 12926-12932 (1995).
- Riveron, J. M., et al. Genome-wide transcription and functional analyses reveal heterogeneous molecular mechanisms driving pyrethroids resistance in the major malaria vector Anopheles funestus across Africa. G3: Genes, Genomes, Genetics. 7 (6), 1819-1832 (2017).
- Smyth, G. K. Linear models and empirical bayes methods for assessing differential expression in microarray experiments. Statistical Applications in Genetics and Molecular Biology. 3 (1), 3 (2004).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados